© Emilie Tremblay, 2019
Nouvelle méthode de dépistage de phytopathogènes
fongiques et de plantes au potentiel envahissant par
métabarcodage
Thèse
Emilie Tremblay
Doctorat en microbiologie
Philosophiæ doctor (Ph. D.)
Québec, Canada
NOUVELLE MÉTHODE DE DÉPISTAGE DE
PHYTOPATHOGÈNES FONGIQUES ET DE PLANTES AU
POTENTIEL ENVAHISSANT PAR MÉTABARCODAGE
Thèse
ÉMILIE D. TREMBLAY
Sous la direction de :
Claude Lemieux, directeur de recherche
Guillaume Bilodeau, codirecteur de recherche
iii
Résumé
Les dommages causés par les pathogènes des plantes sont une menace redoutable pour l’environnement, la diversité, ainsi que pour une partie considérable des ressources naturelles forestières et agronomiques telles que les arbres, les plantes et les récoltes. Les endroits situés à proximité des ports d’importation pour le commerce international et des sites de décharge de déchets verts sont considérés comme étant à risque pour l’introduction d’organismes exotiques et indésirables tels que des insectes, des phytopathogènes et des plantes envahissantes. Bien qu’il existe plusieurs méthodes développées et validées selon des standards établis visant à détecter certains genres préoccupants ou espèces ciblées, la plupart d’entre elles sont mal adaptées aux analyses à grande échelle ou sont limitées en ce qui concerne le nombre d’organismes différents pouvant être détectés simultanément. L’objectif principal de ce projet était de développer une méthode de détection nouvelle, plus rapide, à haut débit, hyper sensible et couvrant de plus grandes superficies afin de contribuer à l’amélioration des méthodes de dépistage et de lutte contre les phytopathogènes et les espèces envahissantes. Le projet a tiré avantage d’enquêtes en entomologie d’envergure nationale et préétablies par l’Agence Canadienne d’Inspection des Aliments en réutilisant les liquides de préservation provenant de pièges à insectes. Ces pièges, en plus de pièges à spores et de pièges à granules de pollen issus du butinage par des abeilles, ont été utilisés pour recueillir des échantillons environnementaux à travers le Canada. Le développement d’un pipeline bio-informatique adapté aux types d’organismes recherchés a permis de supporter et d’analyser efficacement les grandes charges de données produites par la plateforme de séquençage de nouvelle génération (SNG) Ion Torrent. De plus, la conception d’amorces de fusion a conféré un pouvoir de multiplexage significatif aux analyses. Le pipeline intégré au métabarcodage a permis d’effectuer la biosurveillance d’entités phytopathogènes fongiques et oomycètes, de plantes envahissantes ainsi que de localiser des régions géographiques d’intérêt où des organismes indésirables ont été trouvés. Les résultats suggèrent l’existence de pathosystèmes entre des insectes xylophages et des maladies fongiques n’ayant jamais été reportés auparavant. De plus, certains pathogènes fongiques et leurs plantes hôtes ont été trouvés dans les échantillons de granules de pollen, et les espèces de plantes identifiées par SNG corroboraient avec l’identification visuelle des plantes ayant été effectuée sur le terrain. Certains des résultats obtenus par métabarcodage ont été validés avec des tests qPCR spécifiques à certaines espèces cibles, ce qui a confirmé le pouvoir et la sensibilité de cette nouvelle méthode. Par exemple,
iv
de minimes quantités de propagules d’espèces de Phytophthora spp. ont été obtenues. Plusieurs espèces faisant partie de genres préoccupants ont été détectées, dont les champignons phytopathogènes Heterobasidion annosum s.s., H. abietinum/H. parviporum, Leptographium spp.,
Ophiostoma spp., Gremmeniella spp. et Geosmithia spp. et les oomycètes phytopathogènes Peronospora spp., Pythium spp. et Phytophthora spp. Ces résultats prometteurs indiquent que des
organismes de réglementation de partout dans le monde pourraient ajouter cette méthode de métabarcodage à leur boîte à outils servant à faire la biosurveillance et la détection d’espèces réglementées. Dans le cas où des zones nécessitant des enquêtes approfondies étaient localisées selon les résultats de métagénomique, des tests qPCR ou tout autre test validé demeurent essentiels, surtout lorsqu’il s’agit d’identifier des espèces critiques comme des ravageurs réglementés. En outre, comme les technologies de séquençage évoluent continuellement, elles produisent des données dont la qualité s’améliore constamment, et ce, à moindre coût. Par conséquent, il est anticipé que la qualité des bases de données sur lesquelles repose le métabarcodage se perfectionne du même coup, permettant également d’augmenter la capacité de résolution de la nouvelle méthodologie décrite.
v
Abstract
Damage caused by plant pathogens represents a devastating threat to the environment, diversity, and a significant part of natural forest and agronomic resources such as trees, plants, and crops. Areas that are in close proximity to international trade ports and green waste disposal facilities are considered high-risk introduction sites for exotic and unwanted organisms such as insects, phytopathogens, and invasive plants. Although there are many standard methods developed to detect numerous specific genera of concern or target species, most are ill-suited for large-scale screening, or are limited in the number of different organisms that can be assessed at a time. The main objective of this project was to develop a new detection method which is fast, high-throughput, highly sensitive, and targets vast survey areas, in order to contribute in the improvement of the methods for screening and battling of phytopathogens and invasive species. Spore traps, insect traps, and honeybee-foraged pollen clusters were used to collect environmental samples across Canada. The project took advantage of entomology surveys conducted by the Canadian Food Inspection Agency by reusing preservative fluids from those insect traps. The development of a bioinformatics pipeline customized for the types of organisms screened allowed for the handling and efficient analysis of the large data loads produced with the Ion Torrent next-generation sequencing (NGS) platform. Additionally, the design of fusion primers conferred the analyses a significant multiplexing power. Integrating the pipeline to metabarcoding allowed for the biosurveillance of fungal and oomycete phytopathogens, as well as invasive plants, and pinpointing geographical regions of concern where unwanted species were found. Results suggest the existence of wood-boring insects and fungal diseases pathosystems never previously reported. In addition, certain fungal pathogens and their plant hosts were detected from the pollen cluster samples, and the plants species identified by NGS corroborated the records of the visual plant inspections performed in the field. Some of the metabarcoding results were validated with some species-specific qPCR assays, which confirmed the power and the sensitivity of this new method. For example, very low levels of some Phytophthora species propagules could be detected. Multiple species within genera of concern were identified, including the plant pathogenic fungi
Heterobasidion annosum s.s., H. abietinum/H. parviporum, Leptographium spp., Ophiostoma spp., Gremmeniella spp., and Geosmithia spp., and the oomycetes Peronospora spp., Pythium spp., and Phytophthora spp. These promising results indicate that regulatory agencies across the world could
vi
for biosurveillance and screening. In the case where areas requiring further inquiries are pinpointed based on the metagenomics results, qPCR or alternate validated assays remain essential, especially to resolve the identification of critical species such as regulated pests. Furthermore, given that constantly evolving sequencing technologies yield increasing quality data continually, and at reduced costs, it is anticipated that the quality of the databases on which metabarcoding relies will improve at the same time, therefore increasing the resolving capacity of the new method described.
vii
Table des matières
Résumé ... iii
Abstract ... v
Table des matières ... vii
Liste des figures ... x
Liste des tableaux ... xii
Remerciements ... xv
Avant-propos ... xix
Introduction ... 1
Chapitre 1: Revue de littérature ... 3
1.1.1 Champignons phytopathogènes ... 5
1.2 Oomycètes ... 6
1.2.1 Oomycètes phytopathogènes ... 7
1.3 Moyens de propagation des agents responsable de maladies fongiques des plantes ... 8
1.3.1 Facteurs environnementaux abiotiques ... 8
1.3.2 Facteurs anthropogéniques ... 8
1.3.3 Insectes vecteurs ... 9
1.3.3.1 Abeilles ... 10
1.4 Rôle de l’Agence Canadienne d’Inspection des Aliments (ACIA) ... 11
1.5 Identification des pathogènes ... 14
1.5.1 Morphologie et culture ... 14
1.5.2 Biologie moléculaire ... 15
1.5.3 Marqueurs génétiques ... 17
1.5.3.1 L’espaceur transcrit interne (ITS) ... 17
1.5.3.2 L’espaceur ATP9-NAD9... 18
1.5.4 Le séquençage et son évolution ... 18
1.5.4.1 Séquençage par terminaison de chaîne (Sanger) ... 18
1.5.4.2 Séquençage à haut débit ... 19
1.5.5 Contexte et choix de la méthode de séquençage dans le cadre du projet ... 20
1.6 Bio-informatique ... 21
1.7 Métagénomique et métabarcodage ... 22
1.8 Cueillette d’échantillons environnementaux ... 24
viii
1.8.2 Pièges à insectes ... 25
1.9 Hypothèses et objectifs du projet ... 25
1.9.1 Hypothèses ... 26
1.9.2 Objectifs ... 26
1.10 Figures ... 29
Chapitre 2: Screening for exotic forest pathogens to increase survey capacity using metagenomics . 32 2.1 Résumé ... 34
2.2 Abstract ... 35
2.3 Introduction ... 36
2.4 Materials and methods ... 40
2.5 Results ... 48 2.6 Discussion ... 52 2.7 Acknowledgements ... 59 2.8 Tables ... 60 2.9 Figures ... 66 2.9 Supplementary Materials ... 68 2.10 Supplementary Tables ... 69 2.11 Supplementary Figures ... 82
Chapitre 3: Next generation sequencing to investigate existing and new insect associations with phytopathogenic fungal propagules ... 92
3.1 Résumé ... 94
3.2 Abstract ... 95
3.3 Introduction ... 96
3.4 Materials and methods ... 99
3.5 Results ... 104 3.6 Discussion ... 109 3.7 Acknowledgements ... 116 3.8 Tables ... 117 3.9 Figures ... 125 3.10 Supplementary Materials ... 128 3.11 Supplementary Tables ... 130 3.12 Supplementary Figures ... 150
Chapitre 4: High-resolution biomonitoring of plant pathogens and bee-foraged plant species using metabarcoding of pollen clusters content collected from a honeybee hive ... 155
ix
4.2 Abstract ... 158
4.3 Introduction ... 159
4.4 Materials and methods ... 161
4.5 Results ... 166 4.6 Discussion ... 173 4.7 Acknowledgements ... 179 4.8 Tables ... 180 4.9 Figures ... 189 4.10 Supplementary Materials ... 197 4.11 Supplementary Tables ... 202 4.12 Supplementary Figures ... 213 Conclusion ... 221
Rappel des hypothèses de recherche ... 221
Principaux résultats ... 221
Contributions ... 227
Perspectives et considérations futures ... 229
x
Liste des figures
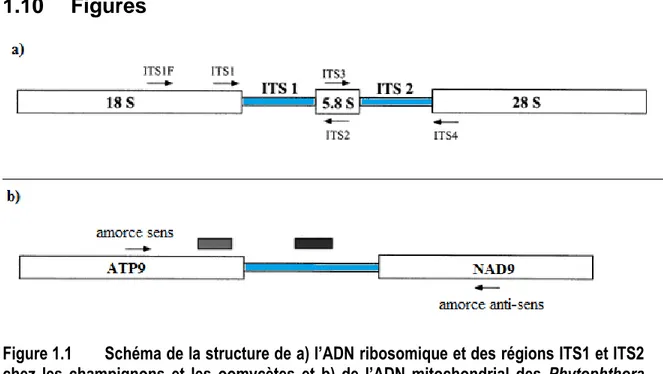
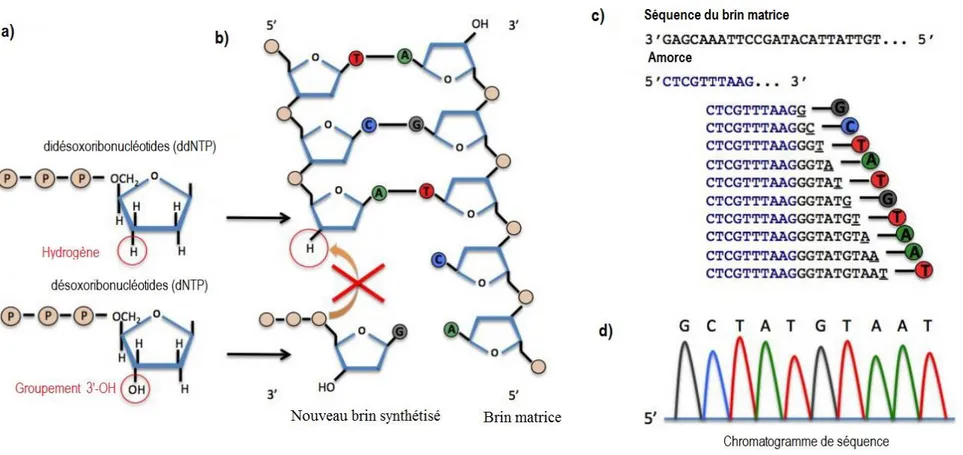
Figure 1.1 Schéma de la structure de a) l’ADN ribosomique et des régions ITS1 et ITS2 chez les champignons et les oomycètes et b) de l’ADN mitochondrial des Phytophthora spp. au niveau des gènes ATP9 et NAD9. Les encadrés blancs représentent les gènes, les encadrés bleus représentent les région intergéniques, les flèches montrent les points d’ancrage de quelques amorces populaires utilisées pour cibler les régions intergéniques illustrées, la boîte grise représente une sonde nucléique spécifique au genre Phytophthora spp. (Bilodeau et al. 2014) et la boîte noire représente une sonde nucléique spécifique à P. ramorum (Bilodeau et al. 2014). Figures adaptées de Larena et al. (1999); Bilodeau et al. (2014). ... 29 Figure 1.2 Processus biochimique du séquençage Sanger impliquant a) des
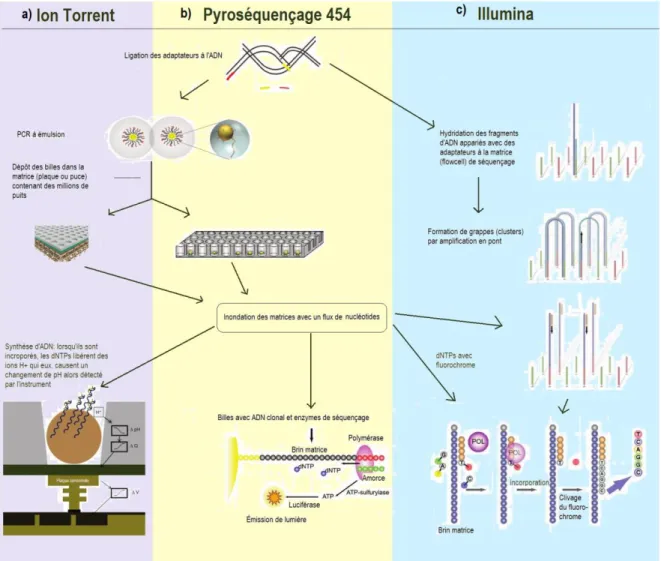
désoxyribonucléotides (forme naturelle) et des nucléotides terminateurs, les didésoxyribonucléotides (groupement hydroxyle remplacé par un atome d’hydrogène) qui, lorsque ajoutés de manière aléatoire, c) préviennent la polymérisation. c) Des séquences de longueurs différentes marquées avec un fluorochrome spécifique au ddNTP sont alors générées et d) lues avec un laser optique afin de déduire la séquence. Figure adaptée de Kircher and Kelso (2010); McGovern (2015). ... 30 Figure 1.3 Schéma simplifié du fonctionnement des plates-formes de séquençage à haut débit
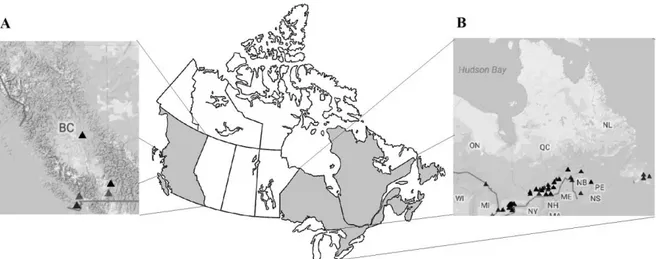
les plus populaires en 2013 a) Ion Torrent, b) pyroséquençage 454 et c) Illumina. Figure adaptée de Mardis (2008); Voelkerding et al. (2009); Life Technologies (2010). ... 31 Figure 2.1 Sampling sites in A the Canadian West Coast and B, Eastern Canada. Adapted from
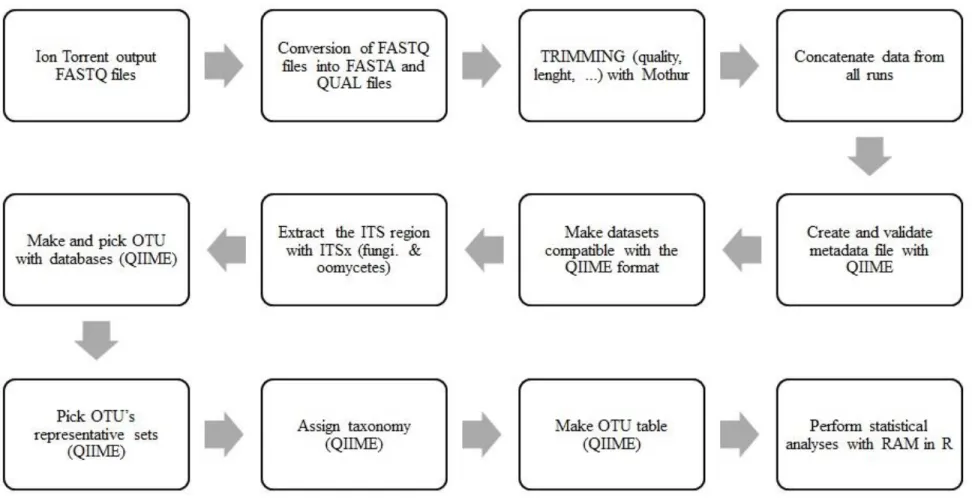
Google Earth. ... 66 Figure 2.2 Bioinformatic pipeline and tools used for next-generation sequencing analysis. ... 67 Figure 3.1 Upset plot to visualize the type of trees from which traps were suspended. The
intersection size number represents the number of times a specific tree combination was found (similar to a Venn diagram), and the set size number corresponds to the number of samples surrounded by a specific type of tree. Most samples were collected from traps placed in forested areas comprising more than one tree species. ... 126 Figure 3.2 Venn diagram of a) fungal species shared or unique to the semiochemical type
employed in insect traps, b) fungal species shared or unique to the semiochemical type employed in insect traps after species subtraction, c) oomycete species shared or unique to the semiochemical type employed in insect traps, d) oomycete species shared or unique to the semiochemical type employed in insect traps after species subtraction. All were obtained by amplifying the ITS1 genic region. ... 127 Figure 4.1 For each pollen pellet sample collected in 2017, abundance of operational taxonomic
unit of fungi detected by the analysis of sequences (ITS1) using the bidirectional fusion primers (ITS1F and ITS2) at the a) phylum (top 5), b) class (top 10), c) order (top 10), d) family (top 15), e) genus (top 15), and f) species (top 30) Levels. n.b. Only the DPS07’s reverse tagged primer (i.e., ITS2) data was analyzed as the ITS1F was discarded because of a suspected contamination. ... 192 Figure 4.2 For each pollen pellet sample collected in 2017, abundance of oomycete operational
taxonomic units detected by analysis of sequences (ITS1) using the bidirectional fusion primers (Omup and Omlo) at the a) order, b) family, c) genus, and d) species (top 30) levels. . 194
xi
Figure 4.3 For each sample collected in 2017, abundance of operational taxonomic units detected from the analysis of plant sequences (ITS2) using the bidirectional fusion primers (ITS4 and SPL) at the a) order (top 10), b) family (top 15), c) genus (top 15), and d) species (top 30) levels. ... 196
xii
Liste des tableaux
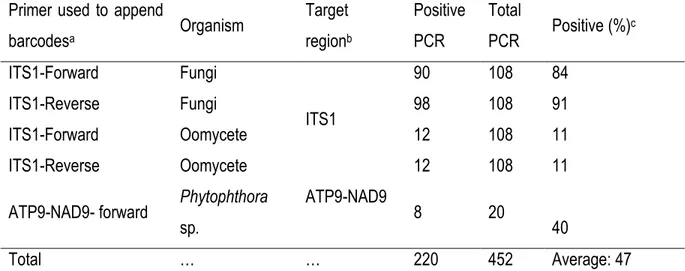
Table 2.1 Presence or absence of amplification as detected by gel electrophoresis using barcoded polymerase chain reaction (PCR) products from 398 environmental samples from targeted organisms, and the percentages of positive reactions obtained. ... 60 Table 2.2 Presence or absence of amplification as detected by gel electrophoresis using
barcoded polymerase chain reaction (PCR) products from 10 mock samples from targeted organisms, and the percentages of positive reactions associated compared with the expected results. ... 60 Table 2.3 Organism sequences and operational taxonomic units (OTU) counts produced from
Ion Torrent Personal Genome Machine next-generation sequencing data output. ... 62 Table 2.4 Saturation values (sequence count) reached through rarefaction curves generated
according to the trap or sample types and observed species number by trap or sample types. .. 63 Table 2.5 Comparison of fungal species of interest found in environmental samples and mock
samples using next-generation sequencing (NGS) and quantitative polymerase chain reaction (qPCR). ... 64 Table 3.1 Presence or absence of amplification as detected by gel electrophoreses using
barcoded PCR products from 108 environmental insect samples by targeted organisms, and the percentages of positive reactions obtained. ... 117 Table 3.2 Operational Taxonomic Units: fungal identification proportion (%) by semiochemical
type at the Phylum taxonomic level using the ITS1 genic region. ... 118 Table 3.3 Operational Taxonomic Units: fungal identification proportion (%) by semiochemical
type at the genus taxonomic level using the ITS1 genic region. ... 119 Table 3.4 Operational Taxonomic Units: fungal identification proportion (%) by semiochemical
type at the species taxonomic level (top 10 species) using the ITS1 genic region. ... 120 Table 3.5 Operational taxonomic units: oomycete identification proportion (%) by
semiochemical type at the Phylum and Genus taxonomic levels using the ITS1 genic region. . 122 Table 3.6 Operational Taxonomic Units: oomycete identification proportion (%) by
semiochemical type at the species taxonomic level (top 10 species) using the ITS1 genic region. ... 123 Table 4.1 List of fungal and oomycetes genera of importance in forest and/or agriculture
phytopathology that were targeted and screened using the custom Perl script metaResultExtractor.pl (Tremblay et al. 2018). Notorious species within the target genus are also listed, most of which are regulated by the Canadian Food Inspection Agency and/or the United States Department of Agriculture - Animal and Plant Health Inspection Service. ... 181 Table 4.2 Summary of amplification bands obtained following electrophoresis ran on the
amplicons generated by PCR with fusion primers. The ITS1 was amplified from fungi and oomycetes, the ITS2 was amplified from plants, and the ATP9-NAD9 was amplified from Phytophthora spp. + means visual band obtained. – means no visual band obtained. (f) = forward fusion primer used. (r) = reverse fusion primer used. ... 187
xiii
Table 4.3 Spearman’s rank correlation (rho) tests (α = 0.05) between the true alpha diversity (Shannon index) of fungi (ITS1), oomycetes (ITS1), and plants (ITS2), and rainfall (total, in mm) or temperature (= T°, average, in °C) recorded. S = significant correlation. NS = Non-significant correlation. ... 188
xiv
À mon père Luc, pour m’avoir enseigné la persévérance et le travail sans répit À ma mère Lise, qui m’a appris à être une femme forte, fière et dévouée À ma sœur Marilyn, ma complice, ma rivale, qui veille à ce que je grandisse avec dignité À ma grand-mère Lucille, qui m’a toujours inspirée de par son amour passionné pour la vie
Et à toi Conrad, mon bel allié, sur qui j’ai su compter à chaque instant
«It required the plague of the potato disease and the example of the Irish famine finally to focus
attention upon the fundamental problem—the relation of the mildew to the sick potato plant, of the smut and rust fungi to the infected grain—the problem of parasitism»
xv
Remerciements
Dans la naïveté du début de la vingtaine, je suis partie de ma province natale, seule, avec le peu de bagages (mon chihuahua et quelques manuels) et de compétences en anglais que j’avais, pour venir habiter à Ottawa. Réalisant à peine l’ampleur de ce que je m’apprêtais à entamer, l’enthousiasme ne manquait point à ce moment.
Guillaume Bilodeau, qui avait accepté de me prendre sous son aile en tant que co-directeur, m’a toujours supportée, poussée, encouragée et défendue. Guillaume, je te remercie d’avoir cru en moi. J’ai parfois eu l’impression que tu croyais plus que moi-même que j’y arriverais au bout du compte. Merci de m’avoir donné tous les outils, les conseils et les réseaux nécessaires pour ce projet et, en partie, pour ma carrière. Merci d’avoir donné la chance à la si petite fille que j’étais alors, car j’ai beaucoup grandi et appris au cours de mon passage dans ton laboratoire. Ton ambition, ta générosité, ta persévérance, ta patience, mais surtout ta passion pour la science sont des qualités contagieuses et on ne peut plus inspirantes. Merci d’avoir insisté pour que je me lance dans le passage accéléré au doctorat. Tu es motivant et c’est ce qui m’a permis de terminer l’un des plus grands accomplissements de ma vie à ce jour.
Également, je remercie Claude Lemieux qui a accepté de me superviser pendant ce voyage. Claude, malgré la distance physique, j’ai toujours senti que vous étiez là pour moi et que vous feriez tout pour m’aider, me guider et m’encourager. Vous êtes une personne exceptionnellement positive et j’ai toujours été reconnaissante de vous avoir comme directeur. Vous êtes extrêmement professionnel, respectueux et vous démontrez un intérêt et un souci des autres remarquables. Tout ce que vous m’avez apporté—outils, conseils, personnes ressources, etc.—était utile, succint, enrichissant, constructif et agréable. Merci de m’avoir acceptée en tant que candidate au doctorat malgré qu’on ne se connaissait que très peu alors. Je vous dois tout mon respect et vous admire.
Je tiens aussi à remercier les autres membres de mon comité d’encadrement, Jean Bérubé et Louis Bernier, qui ont aussi apporté beaucoup au projet tout au long de mon cheminement. L’expertise et la sagesse que vous m’avez apprises sont des outils fondamentaux que j’utiliserai durant toute ma carrière.
xvi
Je remercie aussi Caroline Duchaine, qui a été ma directrice de maîtrise. J’apprécie sincèrement ta présence tout au long de mon doctorat.
Je veux également remercier tous ceux qui m’ont aidée dans le processus de rédaction, incluant mes plus proches collaborateurs: Troy Kimoto, Marc-Olivier Duceppe, Marie-José Côté, Marie-Claude Gagnon, Jean Bérubé, Claude Lemieux, Guillaume Bilodeau et Graham B. Thurston. Notamment, rédiger une publication scientifique n’est pas si simple, et le faire en anglais l’est encore moins. Réviser le travail anglais d’une nouvelle étudiante francophone, je n’en doute point, requiert aussi beaucoup de patience. Vous m’avez toutefois souvent fait la remarque de mes progrès, eh bien c’est grâce à vous tous! Mon ami et collègue Ian King m’a aussi appris beaucoup de subtilités de la langue de Shakespeare et je lui en suis reconnaissante.
Je voudrais également remercier Marie-José Côté pour m’avoir offert la chance de contribuer, de près ou de loin, à des projets de recherches à plusieurs reprises. Entre autres, j’ai beaucoup apprécié contribuer à un domaine d’études autre que la phytopathologie, soit les plantes.
Merci à tous les organismes qui m’ont offert le support financier nécessaire à la réalisation de mon projet de recherche : l’Agence Canadienne d’Inspection des Aliments (ACIA) et sa Stratégie de Partenariat de Recherche (RPS), les Fonds de Développement de Technologies (TD) mandatés de l’ACIA et l’Initiative de Recherche et Développement en Génomique (GRDI) de l’ACIA. J’espère sincèrement que ce projet apportera une contribution positive à l’ACIA… (et aux arbres).
Merci à tous les organismes qui m’ont décerné des bourses afin de m’encourager à poursuivre mon projet de recherche et mes études aux cycles supérieurs: les fonds de soutien à la réussite de la Faculté des Études Supérieures (FESP) de l’Université Laval, la Société de Protection des Forêts contre les Insectes et Maladies (SOPFIM), Ressources Naturelles Canada (RNCan), la Société de Protection des Plantes du Québec (SPPQ), ainsi que le centre d’information Bioinformatics.ca. Chaque fois, les prix reçus sont venus réchauffer mon cœur parce qu’ils m’ont rappelé que les problèmes sur lesquels je travaillais comptent et valent la peine d’être considérés. Merci à Richard Hamelin et ses projets Genome Canada et BioSAFE pour m’avoir permis d’assister à
xvii
une conférence internationale, cette opportunité m’a donné la chance d’élargir mon réseau, mais aussi de goûter aux joies et à l’intensité qui règnent dans de tels événements.
Par-dessus tout, merci à ma tendre famille. J’ai la chance d’entretenir une excellente relation avec chacun de vous, malgré les centaines kilomètres qui nous séparent. Merci à ma maman, Lise, de m’avoir supportée pratiquement tous les dimanches soirs, qui ont parfois été un réel défi moral. Merci de m’avoir préparé des petits pots, cuisiné des biscuits et d’avoir enrichi ma garde-robe pour remplacer mes vieux chandails troués dans mes moments de désespoir (vestimentaires). Merci à mon père Luc pour son support inépuisable. Te rendre fier a toujours été une motivation insatiable. Merci de m’avoir appris, avec le patinage artistique, qu’après être tombée, on doit se relever, se resserrer le chignon, et enchaîner avec la prochaine pirouette: c’était un excellent conditionnement aux montagnes russes que peuvent parfois être les études aux cycles supérieurs. Merci à mes parents pour m’avoir fourni une majeure partie des ressources nécessaires à mon bien-être tout au long de mes, disons-le, longues études. Merci à ma sœur Marilyn pour m’avoir écoutée et porté conseil à maintes reprises, mais aussi pour son support avec de nombreux documents écrits.
Je veux aussi remercier mes amis les plus proches, qui m’ont toujours encouragée et écoutée : Allison, Sabrina, Maryline, Caroline, Mélissa G., Mélissa S., Stéphanie, Raymond, Dana, Marie-Claude, Marc-Olivier et Lisa. Tout au long du processus, j’ai su que je pouvais compter sur vous et je l’apprécie profondément.
Merci aussi à mon plus grand amour, mon partenaire, mon meilleur ami, Conrad Hutter. Au cours de mes études aux cycles supérieurs, tu as fait preuve d’une patience inouïe et d’une force incroyable. Tu ne m’as jamais laissé couler, tu as célébré mes avancements avec moi, et essuyé mes larmes d’autres fois. Tu m’as aussi beaucoup appris à propos des champignons, du fonctionnement à l’ACIA et des compétences informatiques (surtout avec Microsoft Excel). Grâce à toi, je suis devenue une meilleure personne, tout spécialement en termes de patience. Tu as cru en mon potentiel et aussi, tu m’as permis de réaliser des accomplissements personnels plus immédiats durant mon projet, ce qui m’a gardée motivée pour tout le reste : demi-marathons, compétitions de slalom et barrières géantes en ski alpin, compétitions nationales de canoë de sprint, et j’en passe. Il est très clair pour moi que cette aventure a été beaucoup plus agréable avec toi et ta formidable famille à mes côtés.
xviii
Les défis que la vie pourra désormais nous lancer ne m’inquiètent guère puisque, ensemble, nous avons traversé toutes ces étapes en partageant nos buts futurs et les plaisirs quotidiens. Enfin, Bruno et Rose, merci pour votre soutien, vos encouragements et l’énergie positive que vous dégagez en tout temps. Je me sens toujours la bienvenue chez vous, vous êtes une deuxième famille pour moi et vous êtes si souvent venus à ma rescousse, spécialement dans les moments où le temps manquait pour remplir un besoin essentiel: manger.
xix
Avant-propos
Cette thèse de doctorat comporte cinq chapitres, plus précisément, une introduction générale (Chapitre 1), un article scientifique publié (Chapitre 2), deux articles scientifiques soumis, à ce jour, à des revues scientifiques, donc possiblement en attente d’être publiés (Chapitres 3 et 4), et une discussion générale en conclusion (Chapitre 5). Je suis la première auteure de ces trois articles scientifiques.
Le Chapitre 2, intitulé Screening for exotic forest pathogens to increase survey capacity using
metagenomics, est un article accepté pour publication dans la revue Phytopathology en date du 17
juin 2018; doi : 10.1094/PHYTO-02-18-0028-R.
L’article scientifique de ce chapitre présente le concept éprouvé d’une nouvelle approche pour aider les efforts de biosurveillance du gouvernement Canadien. Cette approche permet de dépister des espèces de champignons et d’oomycètes pathogènes de plantes qui représentent un risque majeur pour la biodiversité, l’environnement et les ressources forestières et économiques. La méthode présentée traite plus d’échantillons à la fois, comparativement aux méthodes plus traditionnelles utilisées par les intervenants en phytopathologie. La technologie de séquençage à haut débit ainsi que le pipeline (étapes séquentielles) bio-informatique développé justifient ce rendement plus efficace. Différents types d’échantillonneurs ont été utilisés et placés dans plusieurs provinces canadiennes. Des résultats intéressants ont été obtenus de chacun des types d’échantillonneurs, ce qui a mené à une analyse plus approfondie des résultats des pièges à insectes dans le chapitre 3.
Les coauteurs du chapitre 2 sont Marc-Olivier Duceppe, Jean A. Bérubé, Troy Kimoto, Claude Lemieux et Guillaume Bilodeau. Plus précisément, G. Bilodeau a conceptualisé et supervisé ce projet pilote. Il a collaboré avec T. Kimoto afin d’obtenir des échantillons de pièges à insectes provenant de partout au Canada. T. Kimoto a planifié les protocoles opérationnels, il a mené les enquêtes en entomologie et il a géré les échantillons d’insectes ramassés partout au pays. Enfin, il a révisé cet article. J. Bérubé a fourni les pièges à spores et mené les enquêtes utilisant ces pièges. Il a également révisé cet article. M.-O. Duceppe a contribué au développement du pipeline bio-informatique avec É. Tremblay, ainsi qu’à la révision de ce manuscrit. É. Tremblay a effectué les
xx
analyses formelles au laboratoire et en bio-informatique. Elle a procédé à la collecte d’échantillons à l’aide des pièges JB à Ottawa (Ontario) et à Aylmer (Québec). Elle a fait le développement et l’optimisation de la méthodologie décrite. C. Lemieux a supervisé les travaux et révisé cet article.
Le chapitre 3, qui s’intitule Next-generation sequencing to investigate existing and new insect
associations with phytopathogenic fungal propagules, est un article scientifique qui a été soumis, en
octobre dernier, à la revue scientifique Journal of Fungi dans le cadre d’un numéro spécial intitulé
Fungal-Insect Interactions.
Cet article traite tous les échantillons de liquides préservateurs de pièges à insectes recueillis pendant trois saisons estivales avec l’approche développée dans le chapitre 2. Quatre différents composés sémiochimiques, des phéromones attirant différents groupes d’insectes xylophages, ont été utilisés. L’analyse des échantillons récoltés suite à l’utilisation de chacun de ces composés sémiochimiques a permis de détecter des champignons et des oomycètes modérément pathogènes, des champignons entomopathogènes, ainsi que de nombreux champignons qui font perdre de la valeur au bois. Des pathosystèmes potentiellement nouveaux ont aussi été observés. Chacun des jeux de données des composés sémiochimiques contenaient aussi des espèces retrouvées dans un seul de ces leurres. De plus, la capture accidentelle d’autres insectes, dont plusieurs pollinisateurs, a mené, au chapitre 4, à l’analyse du contenu des granules de pollen récoltés par des abeilles.
Les coauteurs du chapitre 3 sont Jean A. Bérubé, Guillaume J. Bilodeau et Troy Kimoto. Comme cet article a utilisé les mêmes échantillons que ceux ramassés pour le chapitre 2, les rôles de T. Kimoto et G. Bilodeau demeurent les mêmes pour cet article. Il est à noter que T. Kimoto a participé à la rédaction de cet article. De plus, J. Bérubé a contribué à l’analyse des données et à la révision de l’article.
Le chapitre 4 est également un article scientifique qui a été soumis à une revue scientifique le 7 novembre 2018. L’article porte le titre suivant : High-resolution biomonitoring of plant pathogens and
plant species using metabarcoding of pollen pellet contents collected from a honey bee hive. Toujours
xxi
de pollen pour évaluer le potentiel des abeilles à contribuer à la surveillance d’agents phytopathogènes et de plantes au potentiel envahissant.
Les coauteurs du chapitre 4 sont Marc-Olivier Duceppe, Graham B. Thurston, Marie-Claude Gagnon, Marie-José Côté et Guillaume J. Bilodeau. Tous les auteurs ont contribué à l’analyse formelle, à la validation des données et à la révision de l’article. M.-O. Duceppe a également participé aux analyses bio-informatiques. G. Thurston a ramassé les échantillons et a pris soin des abeilles de la ruche, il a procédé aux observations des plantes en floraison sur le terrain, il apporté du support quand est venu le temps des analyses, il a révisé l’article et il a conceptualisé le projet avec M.-C. Gagnon. Quant à M.-C. Gagnon et G. Bilodeau, ils ont supervisé ce projet. M.-C. Gagnon, M.-J. Côté et G. Bilodeau ont fourni les ressources et le matériel nécessaires à la réalisation de ce projet.
Durant mes études supérieures, j’ai collaboré à plusieurs projets. De ce fait, je suis coauteure des articles scientifiques publiés suivants :
Bengtsson-Palme, J., Richardson R. T., Meola M., Wurzbacher C., Tremblay, É. D., Thorell, K., Kanger, K., Eriksson, K. M., Bilodeau, G. J., Johnson, R. M., Hartmann, M., Nilsson, R. H. (2018). Metataxa2 Database Builder: enabling taxonomic identification from metagenomics or
metabarcoding data using any genetic marker. Bioinformatics, DOI: bty482,
https://doi.org/10.1093/bioinformatics/bty482.
Bérubé, J. A., Gagné, P. N., Ponchart, J. P., Tremblay, É. D., Bilodeau, G. J. (2018).
Detection of Diplodia corticola spores in Ontario and Québec based on HighThroughput Sequencing (HTS) methods. Canadian Journal of Plant Pathology, DOI:
10.1080/07060661.2018.1498394.
Roe, A., Torson, A., Bilodeau, G. B., P, Blackburn, G., Cui, M., Cusson, M., Doucet, D., Griess, V., Lafond, V., Paradis, G., Porth, I., Prunier, J., Srivastava, V., Tremblay, E., Uzunovic, A., Yemshanov, D., and Hamelin, R. (2018). Biosurveillance of forest insects: part
I—integration and application of genomic tools to the surveillance of non-native forest insects.
xxii
Bilodeau, P., Roe, A. D., Bilodeau, G., Blackburn, G. S., Cui, M., Cusson, M., Doucet, D., Griess, V. C., Lafond, V. M., Nilausen, C., Paradis, G., Porth, I., Prunier, J., Srivastava, V., Stewart, D., Torson, A. S., Tremblay, E., Uzunovic, A., Yemshanov, D., and Hamelin, R. C. (2018). Biosurveillance of forest insects: part II—adoption of genomic tools by end user
communities and barriers to integration. Journal of Pest Science: 1-12.
Cette recherche était initialement un projet maîtrise. J’ai effectué un passage accéléré au doctorat suite à la première rencontre annuelle que j’ai eue avec le comité d’encadrement en décembre 2014. Cette transition vers le troisième cycle a occasionné le changement d’un des co-directeurs; Caroline Duchaine s’est retirée et c’est Claude Lemieux qui a alors poursuivi la supervision de mon projet avec Guillaume Bilodeau.
Enfin, parmi les nombreux congrès, réunions et autre événements auxquels j’ai participé et où j’ai présenté mon projet de recherche, les plus importants étaient :
le congrès International de Pathologie des Plantes (Boston, 2018);
la réunion annuelle de la Société Canadienne de Phytopathologie de l’Est de l’Ontario (Ottawa, 2018);
la réunion annuelle de la Société Américaine de Phytopathologie, division Nord-Est (Ville de Québec, 2017);
la série de séminaires en recherche sur les plantes de l’ACIA (Ottawa, 2016);
le Café des sciences de l’ACIA (Ottawa, 2016);
la rencontre annuelle de la Société Canadienne de Phytopathologie de l’Est de l’Ontario (Ottawa, 2016);
la réunion annuelle de la Société de Protection des Plantes du Québec (Sainte-Anne-de-Beaupré, 2015);
1
Introduction
L’introduction d’organismes indésirables ou envahissants dans un environnement peut causer des conséquences néfastes, voire irréversibles au sein d’un écosystème. Il s’agit d’un phénomène de plus en plus observé, et ce, pour plusieurs raisons, la principale étant l’augmentation constante du commerce international entre les pays à travers le monde entier. Au Canada, de nombreuses ressources naturelles (ex. grains, plantes horticoles, plantes ornementales, arbres forestiers, etc.) se sont retrouvées menacées ou ont été décimées dans le passé. Des organismes de réglementation exigent des certifications phytosanitaires strictes et spécifiques à certains produits afin de prévenir ou de réduire les dommages occasionnés. À ce jour, des méthodes standards classiques sont couramment utilisées pour identifier les organismes indésirables tels que des plantes envahissantes ou des champignons et des oomycètes phytopathogènes. Cependant, étant donné la vitesse à laquelle ces organismes ravageurs peuvent se propager, il y a un besoin urgent. Une méthode permettant le criblage plus rapide de ceux-ci, à plus grande échelle, et ce, pour un plus grand nombre d’échantillons à la fois est désormais nécessaire.
Cette thèse prouve que l’utilisation de la technologie du séquençage à haut débit permet de faire la détection primaire d’organismes d’intérêt (c.-à.-d. au potentiel phytopathogène) à partir de différents types de pièges installés dans des endroits jugés à risque. La méthode testée a rapporté des résultats prometteurs, c’est pourquoi elle a également été appliquée à la recherche de microorganismes phytopathogènes possiblement transportés par des insectes vecteurs. Un projet pilote a aussi été effectué afin d’évaluer le contenu de granules de pollen pour rechercher des microorganismes indésirables de même que des plantes envahissantes possiblement transportés par des abeilles lors des activités de butinage.
En ce qui concerne le séquençage, il faut dire que les méthodes à haut débit sont attrayantes en raison de leur pouvoir d’analyse. Par contre, elles produisent des charges de données considérables, donc l’interprétation et le traitement de celles-ci représentent un réel défi. Différents marqueurs génétiques peuvent servir à identifier les organismes contenus au niveau du genre et, parfois, de l’espèce. Cependant, l’obtention de résultats plus facilement interprétables doit passer par des analyses bio-informatiques puissantes et complexes. Le traitement bio-informatique consiste à
2
lier une séquence de différentes étapes à effectuer pour lancer différents logiciels à l’aide de scripts exécutant, ensuite, des opérations en boucles ou en parallèle qui allègent et accélèrent la tâche.
Cette revue de la littérature présentera d’abord les principaux organismes à l’étude et certaines caractéristiques spécifiques clés de leur métabolisme. Ensuite, il sera question du rôle de l’Agence Canadienne d’Inspection des Aliments et d’une vue d’ensemble des méthodes traditionnelles, moléculaires et plus nouvelles qui permettent de détecter des organismes d’intérêt. Puis, le rôle déterminant de la bio-informatique dans cette étude sera expliqué. Différents types de pièges permettant de capturer des entités phytopathogènes seront également présentés. Finalement, les hypothèses et objectifs spécifiques de cette thèse seront exposés.
3
4 1.1 Champignons
Le règne des Fungi comprend l’une des plus larges variétés d’espèces sur terre (Feofilova 2001). Les champignons remplissent des rôles écologiques essentiels, notamment celui du recyclage de la matière organique. La paroi cellulaire fongique est constituée principalement de chitine qui forme généralement une structure tubulaire appelée hyphe (Bowman and Free 2006; Stephenson 2010; Carris et al. 2012). La plupart des champignons sont des organismes filamenteux formant un mycélium et qui se reproduisent le plus souvent au moyen de spores disséminées dans l’environnement. Bien que la plupart d’entre eux se reproduisent de manière asexuée, certains peuvent, ou vont occasionnellement le faire de manière sexuée (Stephenson 2010). Selon le groupe fongique et certains facteurs environnementaux imposés, incluant le stress hydrique, le froid et la chaleur, les champignons peuvent produire différents types de structures pour survivre, se disséminer, ou se reproduire—de manière sexuée ou asexuée—, telles des conidiospores (spores asexuées à paroi fine), des chlamydospores (spores asexuées à paroi épaisse issues du gonflement d’hyphes et conçue pour la survie à plus long terme) et des méiospores (spores sexuées) (Kendrick 1985; Prescott et al. 1995; Agrios 2005).
À première vue, les champignons ressemblent aux plantes en raison de leurs structures morphologiques comparables, or, ils ont un métabolisme différent puisqu’ils ne peuvent produire leur propre énergie. En d’autres termes, contrairement aux plantes, ils ne sont pas photosynthétiques. Hétérotrophes, les champignons obtiennent plutôt leurs nutriments à partir de sources de carbone provenant d’autres organismes comme les plantes et les animaux. La digestion du substrat, qui est extracellulaire, est possible grâce au relâchement d’enzymes vers la source d’aliments afin d’accéder aux nutriments essentiels. Les champignons qui se multiplient sur de la matière organique morte sont appelés saprotrophes, tandis que ceux qui utilisent des organismes vivants sont des parasites ou des endophytes. Quant aux champignons endophytes, ils peuvent entretenir une relation symbiotique ou parasitique avec une plante hôte à l’intérieur de laquelle ils prolifèrent (Stephenson 2010; Carris et al. 2012).
Les principaux groupes de champignons sont les ascomycètes, les basidiomycètes, les chytridiomycètes, les zoopagomycètes et les mucoromycètes (Alexopoulos 1996; Blackwell et al. 2006; Webster and Weber 2007; The Royal Botanic Gardens: Kew 2018). Cependant, la
5
classification taxonomique des champignons est très complexe. Autrefois, cette dernière était basée sur les caractéristiques phénotypiques et morphologiques. Aujourd’hui, la taxonomie des champignons repère et utilise des marqueurs génétiques et d’autres méthodes de biologie moléculaire—les réactions en chaîne par polymérase (PCR), les PCR en temps réel (qPCR), le séquençage de nouvelle génération (SNG) et la métagénomique, méthodes décrites dans les sections 1.5.2 à 1.5.4.—qui ont apporté précision, mais aussi complexité et changements constants (Hebert et al. 2003; Yang and Rothman 2004; Shendure and Ji 2008; Shokralla et al. 2012; Katoch and Kapoor 2014; Reina 2017).
1.1.1 Champignons phytopathogènes
Une partie des champignons parasites de plantes sont aussi des pathogènes (Stephenson 2010; Carris et al. 2012). Le règne Fungi est d’ailleurs celui qui inclut le plus grand nombre d’espèces phytopathogènes (Knogge 1996; Carris et al. 2012). En fait, 70% des maladies rencontrées chez les plantes sont à caractère fongique, ce qui représente une véritable menace écologique et économique, car des pertes de récoltes alimentaires, de diversité et de ressources forestières majeures peuvent en découler (Conners 1967; Agrios 2005; Bilodeau et al. 2012; Dean et al. 2012). Les ascomycètes ainsi que les basidiomycètes comprennent la majorité des espèces phytopathogènes (Doehlemann et al. 2017). Les champignons peuvent nuire, voire causer la mort des plantes de diverses façons, notamment en interférant avec leur croissance, ou encore en produisant des toxines (Wolpert et al. 2002; Carris et al. 2012; Ward et al. 2012; Redkar et al. 2015). Les infections fongiques peuvent affecter toutes les parties de la plante, notamment les racines, le système vasculaire, les feuilles et les fruits (Carris et al. 2012). Des exemples typiques de maladies causées par des agents phytopathogènes sont les chancres, les rouilles, les charbons, les pourritures racinaires et les rabougrissements (Vánky 2002; Tisserat et al. 2009; Ward et al. 2012; Redkar et al. 2015). Les champignons phytopathogènes emploient toutes sortes de stratégies pour s’attaquer aux plantes. Par exemple, alors que certains champignons arriveront à contourner le système immunitaire des plantes, d’autres induiront une croissance accrue de l’hôte, ce qui formera des tumeurs (Ward et al. 2012; Redkar et al. 2015). Alors que les champignons biotrophes infectent des tissus vivants, les champignons nécrotrophes, quant à eux, doivent absorber leurs nutriments à
6
travers des tissus ou des cellules de l’hôte dont ils ont causé la mort afin d’assurer leur croissance (Carris et al. 2012; Doehlemann et al. 2017).
Certains champignons sont limités à un hôte en particulier, mais d’autres, comme le champignon hautement polyphage Botrytis cinerea, responsable de la moisissure grise, infectent une vaste gamme de plantes, un aspect qui est souvent associé aux champignons particulièrement dévastateurs (Rodriguez et al. 2009; Giraud et al. 2010; Dean et al. 2012). La coévolution hôte-pathogène entraîne souvent des événements d’adaptation permettant à un hôte-pathogène d’infecter plus d’un groupe de plantes (Norton and Carpenter 1998; Rundle and Nosil 2005). En général, la reconnaissance des effecteurs propres aux phytopathogènes par les plantes se fait au moyen de récepteurs, les protéines R, ce qui déclenche une réaction immunitaire chez la plante, qui elle, relâchera alors des molécules antimicrobiennes (ex. éliciteurs) (Bent and Mackey 2007). Typiquement, à la fin d’un cycle d’infection, les champignons vont produire des spores afin de se disperser et de survivre à plus ou moins long terme dans l’environnement (Madden 1997; Andanson 2010; Carris et al. 2012; Vági et al. 2013).
1.2 Oomycètes
Les oomycètes, tout comme les champignons, sont des organismes filamenteux formant des hyphes et un mycélium. Ils peuvent aussi se reproduire de manière asexuée et sexuée. Ils sont souvent appelés pseudo-champignons parce qu’ils se nourrissent de manière hétérotrophe en tant que saprotrophes ou en tant que parasites, et parce qu’’ils produisent des spores (Fry and Grunwald 2010; Stephenson 2010). D’ailleurs, la production de zoospores flagellées et motiles à l’intérieur des sporanges est l’une des caractéristiques typiques de ces eucaryotes qui vivent principalement dans des environnements humides (e.g. cours d’eau) ou dans le sol (Fry and Grunwald 2010; Stephenson 2010).
La paroi cellulaire des oomycètes est principalement composée de glucanes (Fry and Grunwald 2010). Comme c’est le cas pour les champignons, les mécanismes de défense des plantes contre les oomycètes sont basés sur des interactions entre les gènes R (plante), reconnaissants les effecteurs des agents phytopathogènes (Fry and Grunwald 2010; Stephenson 2010). Or, malgré les
7
ressemblances morphologiques et physiologiques entre les champignons et les oomycètes, les analyses phylogénétiques faites sur ces derniers démontrent que les oomycètes sont tout à fait distincts des « vrais » champignons (White and Dighton 2017).
1.2.1 Oomycètes phytopathogènes
Les oomycètes sont aussi responsables de ravages importants chez les plantes. En effet, plusieurs espèces sont considérées menaçantes puisqu’elles possèdent un large éventail d’hôtes, ce qui favorise leur propagation. Les genres les plus fréquemments rapportés en phytopathologie sont
Phytophthora sp., Pythium sp., Plasmopara sp., Hyaloperonospora sp., et Peronospora sp. (Fry and
Grunwald 2010; Kamoun et al. 2015). Les maladies les plus communément observées sont les mildious, les fontes des semis, les rouilles blanches et les pourritures racinaires (Couture 2008; Carris et al. 2012). Plusieurs espèces de Phytophthora spp. sont particulièrement redoutables parce qu’elles peuvent manipuler le système immunitaire des plantes. Par exemple, les Phytophthora spp. peuvent interférer avec la transcription génique des végétaux grâce à un facteur de transcription silenceur, un phénomène qui peut empêcher l’expression (répression) de gènes de défense chez la plante hôte, ou même causer la mort cellulaire (van West et al. 1999; Fry and Grunwald 2010; Sherwood et al. 2010; Stephenson 2010; Kamoun et al. 2015; Mafurah et al. 2015). La manipulation du système immunitaire des plantes par les oomycètes phytopathogènes est possible grâce à des effecteurs tels que les RXLR (Arg-any amino acid-Leu-Arg) et les CRN (crinkler, crinkling
and necrosis inducing protein). Ces molécules sont aussi connues pour favoriser une adaptation
rapide par l’acquisition de pathogénicité (Mafurah et al. 2015).
Phytophthora ramorum est un exemple notable d’oomycète phytopathogène. En plus d’être
responsable de graves dommages causés aux chênes rouges en Californie, il peut infecter plus d’une centaine d’espèces végétales et provoquer de multiples symptômes selon l’espèce touchée (Rioux et al. 2006; Bilodeau et al. 2009; Kristjansson and Miller 2009). De façon similaire, durant les années 1840, Phytophthora infestans a infecté et ruiné la quasi-totalité des cultures de pommes de terre, causant, du coup, la Grande Famine en Irlande (de Bary 1876; Goss et al. 2014).
8
1.3 Moyens de propagation des agents responsable de maladies fongiques des plantes 1.3.1 Facteurs environnementaux abiotiques
Les champignons et les oomycètes se propagent naturellement sur de courtes et sur de longues distances, un phénomène qui assure la survie de plusieurs espèces (Gage et al. 1999; West and Kimber 2015). Les champignons produisent d’immenses quantités de spores qui se dispersent ensuite dans l’environnement afin de se propager (Brown and Hovmøller 2002). La dispersion par l’air, par l’eau, par le le vent, de même que par des vecteurs pour certains groupes (ex. Ophiostomaceae) sont les principaux moyens empruntés par les champignons et les oomycètes pour envahir un nouvel environnement (Card et al. 2007; Fröhlich-Nowoisky et al. 2009; Fry and Grunwald 2010). De plus, certains types de spores de champignons et d’oomycètes peuvent résister à de nombreux facteurs environnementaux, et ce, sur une longue durée (West and Kimber 2015). De surcroît, lorsqu’un organisme est introduit dans un endroit d’où il n’est pas indigène, il devient exotique et peut acquérir un potentiel envahissant s’il entre en contact avec un hôte compatible et que les conditions sont permissives (Allen and Humble 2002).
1.3.2 Facteurs anthropogéniques
Les spores d’espèces phytopathogènes, y compris celles d’espèces envahissantes ou exotiques, peuvent se propager via l’exportation de plantes ornementales non-sensibles à la maladie via des emballages de bois transportés par cargos ou par le mouvement des insectes ou des animaux (Andanson 2010; Vettraino et al. 2015). De plus, le commerce international du bois est directement lié aux problèmes de forêts décimées par des maladies exotiques, puisque l’introduction accidentelle d’espèces exotiques au potentiel envahissant provient de ces vecteurs de transport (Vettraino et al. 2015). Cette dispersion des spores dans l’environnement par les champignons et par les oomycètes, quoique bien avantageuse pour la propagation de ces microorganismes, représente toutefois un risque élevé d’infection pour les plantes. L’établissement de ces espèces peut aussi être facilité par l’absence ou le délai de réponse des moyens de défense chez les plantes indigènes et par le manque d’organismes compétiteurs sur un terrain nouveau (Allen and Humble 2002). Les monocultures sont des terrains propices à la multiplication des espèces nouvellement arrivées puisqu’il s’agit de grandes superficies où la biodiversité est faible (Brown and Hovmøller 2002). Le déplacement de plantes ou encore de matériel de plantation d’un continent à l’autre entraîne aussi le mouvement et la propagation des maladies (Card et al. 2007; Vettraino et al. 2015). La section 1. 4
9
élabore davantage sur les moyens utilisés pour lutter contre ces invasions indésirables. Il sera question de la gestion des risques phytosanitaires et d’exemples marquants d’introductions dévastatrices de champignons et d’oomycètes au Canada.
1.3.3 Insectes vecteurs
Le transport des agents phytopathogènes par des insectes vecteurs est un phénomène complexe et souvent mal compris. Par contre, certains pathosystèmes sont mieux connus en raison des dommages qu’ils entraînent ou qu’ils ont causés dans le passé (Huang 2003; Maixner 2005; Teale et al. 2011; Kanzaki and Giblin-Davis 2016).
Les mécanismes de transmission, par des insectes vecteurs d’entités phytopathogènes incluant les bactéries, les champignons et les virus, se divisent en deux catégories, soit circulatoire et non-circulatoire. Pour être transmis de manière circulatoire, les phytopathogènes doivent entrer dans l’hémocœle des insectes vecteurs, une composante aux fonctions comparables à celles du sang chez les vertébrés. Il est fréquent que ces microorganismes soient transmis par la salive. Pour ce qui est des vecteurs non-circulatoires, les pathogènes sont transmis par des structures corporelles telles les mycanges chez les scolytes. Les mycanges sont des structures spécialisées qui permettent aux insectes vecteurs d’emmagasiner et de transporter des champignons symbiotiques (Batra 1963; Levieux et al. 1991). Les entités phytopathogènes sont ainsi transmises lorsque, entre autres, des insectes s’alimentent sur une plante (Eigenbrode et al. 2018).
Il a été démontré que les agents phytopathogènes manipulent directement ou indirectement leurs insectes vecteurs afin de favoriser leur dissémination (McArt et al. 2014; Eigenbrode et al. 2018). Par exemple, McLeod et al. (2005) ont démontré que les ormes infectés par les champignons reponsables de la maladie hollandaise de l’orme (Ophiostoma novo-ulmi et O. ulmi) vont produire une molécule organique volatile (sémiochimique) attirant les insectes xylophages. Cette maladie a dévasté des millions d’ormes pratiquement partout en Amérique du Nord suite à son introduction au Canada en 1945 (Allen and Humble 2002). Un moyen connu de prolifération et de propagation de cette maladie est que les larves de scolytes (exotiques et indigènes du continent nord-américain) ayant été en contact physique avec le champignon contaminent, de manière subséquente, des arbres sains lorsqu’elles se nourrissent sous l’écorce des ormes (Allen and Humble 2002). Un autre
10
exemple répertorié seulement à l’extérieur du Canada, est la maladie des mille chancres du noyer (Geosmithia morbida), transportée par le scolyte des pousses du noyer (Pityophthorus juglandis), originaire du Sud-Ouest des États-Unis. Bien connue en raison des ravages qu’elle a causés et qu’elle pourrait induire advenant son introduction au pays, cette maladie a engendré la perte, voire l’élimination, du noyer noir dans certains états américains très près du Canada (ex. Indiana et Ohio) (Tisserat et al. 2009; Juzwik et al. 2015; Juzwik et al. 2016).
1.3.3.1 Abeilles
Plusieurs chercheurs ont étudié les abeilles butineuses pour leur capacité à transmettre des entités phytopathogènes par le transport de granules de pollen recueillis d’une plante à l’autre. Les agents phytopathogènes soupçonnés d’être transmis par le pollen (avec, ou sans insecte pollinisateur) qui ont été les plus étudiés sont les virus (Card et al. 2007; Roberts et al. 2018). Par exemple, le virus de la marbrure du bleuetier (Vaccinium corymbosum) peut se propager au moyen du butinage par les abeilles qui transportent du pollen contaminé (Childress and Ramsdell 1987). Cependant, beaucoup de résultats non concluants ont aussi été rapportés quant à l’association entre les abeilles et certaines infections virales chez les plantes (Card et al. 2007). Il s’agit d’une controverse qui nécessite des recherches plus approfondies. D’autres auteurs ont aussi démontré que les abeilles peuvent agir en tant que vecteurs de bactéries. En l’occurrence, la brûlure bactérienne (Erwinia amylovora) affecte des arbres fruitiers (pommiers, poiriers, etc.) (Johnson et al. 1993; McArt et al. 2014; Pattemore et al. 2014), et le complexe bactérien Pseudomonas syringae affecte, entre autres, les kiwis (Actinidia deliciosa) (Pattemore et al. 2014). Il a même été démontré que les abeilles, lors de leur retour au sein de la colonie, peuvent infecter d’autres butineuses de la ruche, qui elles, pourraient alors potentiellement infecter de nouvelles plantes lors des activités de pollinisation (Pattemore et al. 2014).
Il existe très peu d’études scientifiques qui démontrent la transmission de phytopathogènes fongiques par butinage. Stelfox et al. (1978) ont montré que le champignon de la brûlure de la tête
Sclerotinia sclerotiorum, infectant les crucifères (Brassica sp.), peut être transmis lors des activités
de butinage des abeilles. Également, Huang (2003) a montré que l’abeille découpeuse (Megachile
11
contacts accidentels. Il a aussi été démontré que les champignons phytopathogènes (e.g. Ustilago spp.) peuvent modifier la période de floraison de leur plante hôte pour augmenter les chances de visites d’un insecte pollinisateur vecteur (Jennersten 1988; Jennersten and Kwak 1991; Lara and Ornelas 2003; McArt et al. 2016). Certains champignons plus agressifs (ex. Uromyces pisi) peuvent même induire la production de structures à partir des tissus végétatifs de la plante, imitant les fleurs (c.-à.-d. pseudofleurs), dans le but d’attirer les insectes butineurs et, ainsi favoriser leur propagation (Roy 1993; Pfunder and Roy 2000; McArt et al. 2016).
1.4 Rôle de l’Agence Canadienne d’Inspection des Aliments (ACIA)
En plus de veiller à la protection et à la sécurité des aliments au Canada, l’ACIA joue un rôle très important dans le maintien de la santé des plantes, de l’environnement, des ressources essentielles à la santé humaine, de la biodiversité et de la santé économique du pays (Allen and Humble 2002; Canadian Food Inspection Agency 2018b). La mission de l’ACIA est d’assurer la qualité des aliments, donc de prévenir la propagation de pathogènes responsables de ravages majeurs. À titre d’organisme de réglementation, elle se doit de protéger les produits forestiers, les grains, les cultures, les plantes horticoles, les pommes de terre et les semences qui sont exportés, importés ou vendus au pays. Pour ce faire, des réglementations, des certifications, de même que des normes strictes sont établies au pays (Canadian Food Inspection Agency 2018b). L’ACIA travaille également en partenariat avec d’autres organismes gouvernementaux comme Ressources Naturelles Canada, Agriculture et Agroalimentaire Canada, Service Canadien des Forêts et Santé Canada pour la protection des ressources (Natural Resources Canada 2017).
Bien que les raisons de protéger l’intégrité des aliments soient évidentes, l’importance de la protection des plantes peut sembler plus subtile. L’une des principales raisons pour lesquelles l’ACIA réglemente les importations et les exportations de matériel végétal est que l’introduction de plantes envahissantes et d’organismes nuisibles pour les plantes peut causer des dommages irréversibles suite à leur propagation, et ce, au Canada comme ailleurs (Liebhold et al. 1995; Wallner 1996; Allen and Humble 2002). Les espèces envahissantes sont des espèces exotiques qui s’acclimatent de manière permanente suite à leur introduction dans un environnement où elles prolifèrent. Elles sont responsables de changements importants dans les écosystèmes puisqu’elles peuvent mobiliser les
12
ressources essentielles à d’autres espèces indigènes (Cronk 1995). La loi sur la protection des végétaux a donc pour but de prévenir l’importation et l’exportation d’ennemis des plantes, entre autres, en planifiant des moyens de lutte (e.g. mise en quarantaine, destruction de matériel infecté, interdiction de mouvement du matériel contaminé et restriction des activités) applicables au besoin (The Minister of Justice (Canada) 1990; Agence Canadienne d'Inspection des Aliments 2018a). Une liste de ravageurs, de maladies (ex. insectes, champignons et nématodes) et de plantes exotiques, indigènes ou ayant un potentiel envahissant est établie par l’ACIA (Canadian Food Inspection Agency 2013b; Ressources Naturelles Canada 2018). En constante évolution, cette liste élabore le statut de chacune des espèces y figurant, tout en spécifiant les mesures à prendre pour le traitement du matériel végétal dont il est question (e.g. fruits, plantes ornementales, noix, grains, etc.). Des exemples notables de maladies causées par des champignons exotiques qui ont occasionné des dommages forestiers et économiques irréversibles au pays incluent le chancre européen du mélèze (Lachnellula wilkommi), le chancre du noyer cendré (Ophiognomonia clavigignenti-juglandacearum) et la maladie hollandaise de l’orme (O. novo-ulmi et O. ulmi), expliquée plus en détail dans la section 1.3.3. Le chancre scléroderrien (Gremmeniella abietina), plus spécifiquement la race européenne (plus agressive), malheureusement présent au Canada (Lachance 1979; Laflamme 1987), a été responsable de dommages immenses dans l’état de New York, causant la mort de près de 90% des pins rouges et des pins sylvestres matures à certains endroits (Agence Canadienne d'Inspection des Alimnets 2012). Une maladie encore non-répertoriée au Canada, mais notable pour ses dommages irréversibles, est la maladie des mille chancres (Geosmithia morbida) (discutée dans la section 1.3.3) transportée par un insecte vecteur (Tisserat et al. 2009). En Nouvelle-Écosse, un exemple récent d’introduction d’un insecte indésirable réglementé est le puceron lanigère de la pruche (Adelges
tsugae). La presence de cet insecte a de graves conséquences sur l’environnement, puisque des
habitats terrestres et aquatiques naturels en souffrent grandement (Canadian Food Inspection Agency 2018c). Considérant l’ampleur de l’industrie du bois en Amérique du Nord, il importe de mentionner les conséquences que l’économie subit suite à de telles épidémies. Aux États-Unis, par exemple, les coûts associés aux impacts des maladies envahissantes s’élèvent à des milliards de dollars par année (Pimentel et al. 2000; Pimentel 2002) et plus spécifquement au Canada, l’ACIA estime ces coûts annuels à 20 milliards de dollars pour le secteur forestier et à 2,2 milliards de dollars pour les plantes envahissantes du secteur agricole (Canadian Food Inspection Agency 2014b).
13
Un problème associé à l’introduction de certaines plantes est le potentiel d’une espèce à devenir envahissante. Par exemple, les plantes envahissantes peuvent envahir des zones agricoles, laissant derrière elles des pertes considérables. Tout comme pour les organismes phytopathogènes, l’ACIA réglemente et effectue la surveillance de ces espèces listées en exerçant un contrôle strict sur les importations, sur la manipulation et sur les déplacements de ces végétaux au pays (Canadian Food Inspection Agency 2013b; Agence Canadienne d'Inspection des Aliments 2018b). À titre d’exemple, le kudzu (Pueraria montana), une plante envahissante provenant de l’Asie qui a été introduite aux États-Unis et en Ontario au cours de la dernière décennie, s’avère désormais un problème majeur. La principale raison est que la croissance parfois qualifiée d’incontrôlable de cette vigne grimpante est possible dans à peu près n’importe quel environnement (forêts, bordures de routes, champs agricoles, etc.) (Agence Canadienne d'Inspection des Aliments 2016).
De plus, certaines plantes, bien que présentes au pays, font également partie de la liste des espèces réglementées puisqu’elles sont des hôtes alternes de certains champignons pathogènes. Ainsi, le transport, la vente et la reproduction, en territoire canadien, de l’épine-vinette japonaise (Berberis thunbergii) sont interdits parce qu’il s’agit d’un hôte alterne de la rouille noire des céréales (Puccinia graminis) (Canadian Food Inspection Agency 2014c; Berlin et al. 2017).
Avec la pression constante et grandissante exercée par l’introduction d’espèces exotiques et envahissantes au Canada, l’ACIA bénéficierait à développer des outils diagnostiques de haute performance. Le Canada étant l’un des plus grands pays au monde, l’ACIA a besoin de méthodes de surveillance pouvant supporter l’analyse d’un plus haut débit d’échantillons afin d’enquêter sur l’ensemble de sa superficie (ou, du moins, sur les zones hautement à risque). Pour ce faire, il lui faut un outil qui lui permette de détecter un organisme indésirable présent en très faible quantité, (e.g. quelques spores), avant même qu’il ne puisse s’établir.
Les méthodes d’enquêtes phytosanitaires présentement utilisées par l’ACIA incluent des inspections visuelles sur le terrain afin d’identifier des signes de la présence d’organismes phytpathogènes (Agence Canadienne d'Inspection des Aliments 2018a). La recherche visuelle de plantes envahissantes est faite, en majorité, dans des zones environnantes des installations de
14
manutention des semences et des grains (e.g. les fossés et les terres à proximité et les sites de compostage de déchets), puisque ces lieux sont souvent associés à l’introduction de mauvaises herbes indésirables. Les abords des chemins de fer sur lesquels des trains transportent des grains sont également des endroits ciblés où l’ACIA fait la surveillance de plantes envahissantes (Agence Canadienne d'Inspection des Aliments 2018f). Le personnel de l’ACIA applique aussi différentes méthodes d’échantillonnage. Par exemple, des inspecteurs utilisent des pièges contenant des attractifs chimiques pour attirer et identifier des insectes indésirables. La collecte de matériel provenant d’un hôte suspect ou d’échantillons de sols est également une méthode courante. Le personnel de l’ACIA pratique aussi l’élevage, par exemple, suite à l’échantillonnage d’un hôte soupçonné d’être infecté par un insecte indésirable. Cette méthode de surveillance consiste à fournir un environnement propice à l’émergence de larves dans le but de les identifier (Agence Canadienne d'Inspection des Aliments 2018a).
Il est à noter que les laboratoires de diagnostic de l’ACIA détiennent un mandat de libre accès sur les outils issus des projets de recherche accomplis par des chercheurs de l’ACIA. Un exemple pertinent d’une technologie couramment utilisée par le laboratoire de diagnostic de phytopathologie est le test qPCR (duplex) de Bilodeau et al. (2009), qui dépiste la présence du genre Phytophthora et l’espèce P. ramorum.
1.5 Identification des pathogènes 1.5.1 Morphologie et culture
Les méthodes d’identification des champignons et des oomycètes phytopathogènes évoluent constamment. Historiquement, la morphologie et la mise en culture étaient utilisées pour classifier, identifier et effectuer le diagnostic des agents phytopathogènes (Guarro et al. 1999; Hyde et al. 2011). Les observations microscopiques de structures particulières comme des spores ne fournissent cependant qu’une information limitée et requièrent une expertise poussée (Turin et al. 2000; Raja et al. 2017). Bien qu’il soit souvent possible d’identifier des champignons jusqu’au niveau taxonomique de la famille par morphologie et par culture, ce processus n’est pas aussi simple lorsqu’il s’agit de déterminer le genre et l’espèce, en particulier lorsque de nombreuses espèces sont en jeu (Turin et al. 2000; Geiser 2004; Raja et al. 2017). En effet, certaines caractéristiques