CRISPR-Cas : un nouvel outil pour l’étude des génomes
de bactériophages
Mémoire
Bruno Martel
Maîtrise en microbiologie
Maître ès sciences (M. Sc.)
Québec, Canada
© Bruno Martel, 2014
RÉSUMÉ
Les bactériophages sont maintenant reconnus pour jouer un rôle majeur dans divers écosystèmes. De nouveaux gènes sont fréquemment identifiés dans les génomes de ces bactériophages issus de différentes études environnementales. La majorité de ces gènes ne peuvent être associés à une fonction connue, d’où la nécessité de développer des outils génétiques performants afin mieux comprendre leur rôle dans la biologie des bactériophages virulents.
Dans ce projet de maîtrise, le système CRISPR-Cas de la souche Streptococcus thermophilus DGCC7710 a été utilisé afin de pouvoir étudier des gènes sans fonction connue du bactériophage virulent 2972. Des mutations ponctuelles ainsi que de petites et de grandes délétions ont été réalisées dans le génome de phages virulents en utilisant le système CRISPR-Cas comme pression sélective. Finalement, la méthylation de l’ADN phagique a été démontrée suite à l’insertion d’un gène codant pour une méthyltransférase bactérienne dans le génome du phage 2972.
ABSTRACT
Bacteriophages are now widely recognized as major players in a wide variety of ecosystems. Novel genes are often identified in newly isolated phages as well as in environmental metavirome studies. Most of these novel viral genes have unknown functions but appear to be coding for small, non-structural proteins. To understand their biological role, very efficient genetic tools are required to modify them, especially in the genome of virulent phages.
For this MSc project, specific point mutations and large deletions can be engineered in the genome of the virulent phage 2972 using the Streptococcus thermophilus Cas Type II-A. Furthermore, the CRISPR-Cas engineering system can be used to efficiently introduce a functional methyltransferase gene into a virulent phage genome. Finally, synthetic CRISPR bacteriophage insensitive mutants were constructed by cloning a spacer-repeat unit in a low copy vector illustrating the possibility to target multiple regions of the phage genome.
TABLE DES MATIERES
RÉSUMÉ ... iii
ABSTRACT ... v
TABLE DES MATIERES ... vii
LISTE DES FIGURES ... ix
LISTE DES TABLEAUX ... xi
LISTE DES ABRÉVIATIONS ... xiii
AVANT-PROPOS ... xix
1
INTRODUCTION ... 1
1.1 Les bactéries lactiques ... 1
1.1.1 Caractéristiques générales ... 1
1.1.2 Streptococcus thermophilus ... 2
1.2 Les bactériophages ... 5
1.2.1 Historique ... 5
1.2.2 Classification des bactériophages ... 5
1.2.3 Caudovirales ... 7
1.2.4 Cycle d’infection ... 7
1.2.4.1 Cycle lytique ... 8
1.2.4.1.1 Adsorption des particules virales ... 8
1.2.4.1.2 Entrée de l’ADN viral dans le cytoplasme ... 8
1.2.4.1.3 Réplication du génome ... 9
1.2.4.1.4 Transcription et traduction ... 10
1.2.4.1.5 Assemblage des particules virales ... 10
1.2.4.1.6 Lyse cellulaire ... 12
1.2.4.2 Cycle lysogène ... 13
1.2.5 Phages infectant Streptococcus thermophilus ... 13
1.2.6 Le phage 2972 ... 14
1.3 Mutagénèse dirigée chez les bactériophages ... 17
1.3.1 Historique ... 17
1.3.2 La recombinaison homologue ... 18
1.3.3 Le recombineering ... 19
1.3.4 Le recombineering des bactériophages par électroporation d’ADN ... 20
1.4 Le système CRISPR-Cas ... 22
1.4.1 La diversité des systèmes CRISPR-Cas ... 22
1.4.1.1 La découverte ... 22
1.4.1.2 Les répétitions ... 22
1.4.1.3 Les espaceurs ... 23
1.4.1.4 Les gènes associés au locus CRISPR ... 23
1.4.2 Loci CIRSPR-Cas chez Streptococcus thermophilus... 25
1.4.3 Mécanisme général du système CRISPR-Cas ... 26
1.4.3.1 L’acquisition ... 26
1.4.3.2 Biogénèse des ARNs ... 27
1.4.3.3 Interférence... 28
1.4.4 Échapper au système CRISPR-Cas ... 29
1.5 Le système CRISPR-Cas type II-A chez S. thermophilus DGCC7710 ... 30
1.5.1 Acquisition ... 30
1.5.4 Interférence : Cas9 ... 32
1.6 Système CRISPR-Cas de type II comme outil génétique ... 34
1.6.1 Avant Cas9 ... 34
1.6.2 Optimisation du duplex ARNcr::ARNtracr ... 35
1.6.3 Édition de génome ... 36
1.7 Problématique à l’étude et les objectifs à atteindre ... 38
2
MATÉRIEL ET MÉTHODES ... 39
2.1 Souches bactériennes, phages et conditions de culture. ... 39
2.1.1 Conditions de culture pour les souches bactériennes. ... 39
2.1.2 Propagation des phages ... 39
2.1.3 Titration des lysats de phages ... 40
2.1.4 Détermination de l’efficacité à former des plages de lyse (EOP) ... 41
2.1.5 Cellules compétentes et transformation... 41
2.1.6 Construction des plasmides ... 42
2.2 Détermination de la fréquence des positions mutées chez les CEMs du phage 2972 ... 44
2.2.1 Sélection des BIMs, obtention de phages mutants et criblage ... 44
2.2.2 Criblage des CEMs par séquençage d’amplicons PCR ... 46
2.2.3 Purification des CEMs d’intérêt ... 47
2.3 Résistance au phage 2972 suite à l’expression d’un ARNpré-cr synthétique ... 47
2.3.1 Construction des souches et EOP ... 47
2.4 Édition du génome du phage virulent 2972 ... 47
2.4.1 Mutation ponctuelle isolée dans le phage CEMS91 ... 47
2.4.2 Grande délétion isolée dans le phage CEMS90 ... 47
2.4.3 Délétion menant à un changement de cadre de lecture ... 48
2.4.4 Échange de gènes et expression d’un transgène ... 48
3
RÉSULTATS... 49
3.1 Détermination du profil de mutations dans le proto-espaceur et le PAM. ... 49
3.2 Construction de BIM synthétiques ... 51
3.3 Isolement et caractérisation des CEMs du phage 2972 ... 52
3.4 Édition du génome du phage virulent 2972 ... 54
3.4.1 Preuve de concept ... 54
3.4.1.1 Mutation ponctuelle ... 54
3.4.1.2 Grande délétion ... 56
3.4.2 Inactivation de gène suite à une petite délétion ... 57
3.4.3 Échange de gènes ... 58
4
DISCUSSION ... 61
4.1 Profil des mutations échappatoires du CRISPR1-Cas ... 61
4.2 Construction de BIMs synthétiques ... 61
4.3 Isolement et caractérisation des CEMS du phage 2972... 62
4.4 Édition de génome du phage 2972 ... 62
4.4.1 Preuve du concept ... 62
4.4.2 Inactivation de gène suite à une petite délétion ... 63
4.4.3 Échange de gènes ... 64
5
CONCLUSION ET PERSPECTIVES ... 67
6
RÉFÉRENCES ... 69
LISTE DES FIGURES
Figure 1.1. Voies métaboliques homofermentaire et hétérofermentaire de la dégradation des sucres... 2
Figure 1.2. Observation en microscopie électronique à balayage de S. thermophilus ... 3
Figure 1.3. Morphotypes des virus infectant les procaryotes ... 6
Figure 1.4. Structures types des trois familles de l’ordre Caudovirales ... 7
Figure 1.5. Cycles lytique et lysogène des bactériophages lors de l’infection de son hôte bactérien ... 8
Figure 1.6. Assemblage séquentiel du bactériophage T4. Image modifiée de (San Martín, 2013). ... 11
Figure 1.7. Comparaison de l’organisation génomique des phages infectants S. thermophilus ... 15
Figure 1.8. Modèles des deux voies de réparation HDR ... 19
Figure 1.9. Familles des protéines Cas et leur rôles pour les trois types majeurs de système CRISPR-Cas ... 24
Figure 1.10. Organisation génétique des quatre loci CRISPR-Cas présents dans le génome de la souche S. thermophilus DGCC7710 ... 25
Figure 1.11. Mécanisme général des systèmes CRISPR-Cas ... 27
Figure 1.12. Schéma de la maturation de l’ARNpré-cr en ARNcr ... 32
Figure 1.13. Illustration de l’interférence Cas9 ... 34
Figure 2.1. Représentation schématique des constructions plasmidiques utilisées pour l’édition du génome du phage virulent 2972 ... 45
Figure 2.2. Organisation du génome du phage 2972 ... 46
Figure 3.1. Modèle in silico d’appariement entre le proto-espaceur PS91 dans le génome du phage 2972 (ADN envahisseur) et le duplex ARNcrS91::ARNtracr mature ... 49
Figure 3.2. Fréquence des mutations ponctuelles retrouvées dans la séquence du proto-espaceur et du PAM de phages échappant au CRISPR-Cas ... 51
Figure 3.3. Modélisation générale d’édition de génome pour le phage 2972 selon diverses stratégies... 55
Figure 3.4. Séquences des phages recombinants contournant le CRISPR obtenus sur le BIM S91 ... 56
Figure 3.5. Migration des produits PCR des régions de l’orf33 des phages recombinants ... 57
Figure 3.6. Produits PCR des régions de l’orf33 du phage 2972 sauvage et du phage CEM-LlaDCHIA ... 59
LISTE DES TABLEAUX
Tableau 1.1. Génomes complets des souches de S. thermophilus disponibles ... 3
Tableau 1.2. Gènes du phage 2972 et leurs fonctions prédites ... 16
Tableau 1.3. Organismes modifiés par l’édition de génome ... 36
Tableau 2.1. Liste des souches bactériennes, phages et plasmides utilisés lors de cette étude ... 40
Tableau 2.2. Amorces et oligonucléotides utilisés lors de cette étude ... 43
Tableau 3.1. Efficacité à former des plages du phage 2972 sur différentes souches de S. thermophilus ... 50
Tableau 3.2. EOPs du phage 2972 sur DGCC7710 contenant différentes constructions ... 52
Tableau 3.3. CEMs naturels d’intérêt isolés et leurs caractéristiques ... 53
Tableau 3.4. EOPs du phage 2972 sur les BIMs et les souches utilisées pour l’édition de génome ... 56
LISTE DES ABRÉVIATIONS
A+T Adénine + thymine ADN Acide désoxyribonucléique ADNdb ADN double brinADNsb ADN simple brin ARN Acide ribonucléique ARNcr ARN CRISPR ARNg ARN guide unique ARNm ARN messager
ARNpré-cr ARN pré-messager CRISPR ARNtracr ARN activateur agissant en trans ATP Adénosine triphosphate
ATPase Adénosine-5’-triphosphatase
BIM Mutant résistant aux bactériophages (Bacteriophage Insensitive Mutant) BL Bactérie lactique
°C Degré Celsius
CaCl2 Chlorure de calcium
Cas Gène associé au CRISPR (CRISPR associated gene) cc Centimètre cube
CEM Mutant résistant au CRISPR (CRISPR Escaping Mutant) CLO Cadre de lecture ouvert
CRCHUL Centre de Recherche du Centre Hospitalier de l’Université Laval
CRISPR Petites répétitions palindromiques groupées et régulièrement espacées (Clustered Regularly
Interspaced Short Palindromic Repeats)
dCas9 Cas9-défective
DO600 nm Densité optique à 600 nm
dNTP Désoxyribonucléotide-triphosphate DSB Bris d’ADNdb (Double-Strand Break)
EOP Efficacité à former des plages (Efficacy Of Plating) EPS Exopolysaccarides
G+C Guanine + cytosine
GFP Green Fluorescent Protein
HNH Homing endonuclease
KAc Acétate de potassium kDa Kilodalton
kpb Kilopaires de bases
kV Kilovolts
LB Luria Bertani ou Lysogeny broth
LC-MS/MS Spectroscopie de masse par chromatographie liquide en tandem (Liquid
Chromatography-Tandem Mass Spectrometry)
Mb Mégapaires de bases MgSO4 Sulfate de magnésium
MnCl2 Chlorure de manganèse µF Microfaraday µg Microgramme µl Microlitre µm Micromètre µM Micromolaire ml Millilitre mM Millimolaire
MLST Typage par séquençage de plusieurs loci (Multi-Locus Sequence Typing) MOI Multiplicité d’infection (Multiplicity Of Infection)
MOPS Acide 3-(N-morpholino) propanesulfonique NCBI National Center for Biotechnology Information
NHEJ Raccordement d’extrémités d’ADN non homologues (NonHomologous End Joining)
nm Nanomètre
Ω Ohms
ORF Cadre de lecture ouvert (Open Reading Frame)
PAM Motif adjacent au proto-espaceur (Proto-spacer Adjacent Motif) pb Paire de bases
PCR Réaction en chaîne de la polymérase (Polymerase Chain Reaction) PEG Polyéthylène glycol
pmol Picomole
RbCl Chlorure de rubidium
RBP Protéine de liaison au récepteur (Receptor Binding Protein) RBS Site de liaison du ribosome (Ribosome Binding Site)
R/M Restriction/Modification RNase Ribonucléase
rpm Rotation par minute
SAM Spacer Acquisition Motif
SDS-PAGE Sodium DodecylSulfate-PolyAcrylamide Gel Electrophoresis sp. Espèce (Species)
ssp. Sous-espèce (Subspecies)
TMP protéine étalon (Tape Measure Protein) UFP Unité Formatrice de Plage de lyse
“The most exciting phrase to hear in science, the one that heralds the most discoveries, is not "Eureka!" but 'That's funny...”
AVANT-PROPOS
Une maîtrise en microbiologie demande beaucoup de temps et d’efforts afin de satisfaire à nos propres exigences. Aussi, j’ai dû faire plusieurs sacrifices en tant que mari et père de famille ayant pris sa retraite comme soudeur-assembleur afin d’abreuver ma soif de connaissances pour les sciences et plus particulièrement pour les phages qui sont si extraordinaires. Le mémoire qui suit rapporte les résultats de plusieurs années de travail dans le laboratoire du professeur Sylvain Moineau. Durant mon séjour dans ce laboratoire, d’abord, pour les stages d’été et ensuite pour le travail à temps partiel, j’ai été adopté par une famille hors du commun. D’abord, Sylvain, un professeur et un mentor hors de l’ordinaire que je vois comme le père d’une grande famille, étant prévenant et attentif aux besoins de ses « enfants » afin de leur montrer la bonne attitude à avoir pour la réussite en science. Denise, la « mère » de nous tous, sans qui je n’aurais pas autant appris des méthodes du labo ainsi que certaines de la vie de tous les jours. Finalement, mes frères et sœurs d’adoption avec qui j’ai pu échanger de tout et de rien : Maxim, Marie-Ève, Manuella, Josiane, Molly, Lynn, Simon, Alexander, Geneviève, Rym, Mourad, Rodrigue, Siham, Alfonso, Rodrigo, Pedro, Diego, Sana, Emna et les tout nouveaux membres de la famille Alexia, Marie-Laurence, Caroline, Cas, Marc-Antoine et Giovanni.
Je dois faire un remerciement tout particulier à ma merveilleuse conjointe Mélanie (et mes deux merveilleux enfants) qui m’a permis de faire ce virage à 180° dans ma carrière en plus de supporter mon absence physique, mais plus souvent mentale, et pour son soutien hors de l’ordinaire. Mais, je ne dois pas oublier Julie sans qui ma vie scientifique aurait été un enfer de questions sans réponses ainsi que mon « frère » complémentaire, Maxime, avec qui j’ai pu former un duo de science, de bière et de sport. La recherche universitaire est un monde ouvert sur la science où il ne devrait pas y avoir de compétition, mais seulement de la coopération. Les recherches amènent très souvent plus de questions que de réponses, ce qui peut être tout aussi frustrant qu’euphorique. Un gros merci à Sylvain et Denise qui font tout en leur pouvoir afin de maintenir un esprit d’équipe et de famille au sein du laboratoire, ce qui est, pour moi, la clé de la réussite universitaire. Finalement, je tiens aussi à remercier la Commission canadienne du lait et Novalait inc. pour le soutien financier qu’ils m’ont accordé et aussi le groupe de recherche en écologie buccale pour les locaux, les équipements scientifiques et les activités sociales et sportives qui ont été organisées.
1 INTRODUCTION
1.1 Les bactéries lactiques
1.1.1 Caractéristiques générales
Les bactéries sont ubiquitaires et sont présentes dans tous les environnements connus jusqu’à maintenant. Les bactéries lactiques (BL), quant à elles, sont des microorganismes associés, entre autres, aux plantes (chou, maïs et orge), la viande et les produits laitiers. Les BL sont des bactéries à Gram positif, négatives aux tests de la catalase et de l’oxydase. Généralement, les BL sont retrouvées sous forme de coques, de bâtonnets (sans spore) ou ovoïdes et sont souvent regroupées en paires ou en chaînes. Les BL sont reconnues pour leur capacité à fermenter les sucres en acide lactique (ou lactate) et sont majoritairement aérotolérants, c’est-à-dire qu’elles n’utilisent pas l’oxygène comme accepteur final d’électrons, mais tolèrent la présence d’oxygène (Carr et al., 2002). Leur génome a un faible contenu en G+C. Les BL sont classées dans l’ordre des Lactobacillales dont les genres Aerococcus, Carnobacterium, Tetragenococcus, Enterococcus,
Lactobacillus, Oenococcus, Pediococcus, Leuconostoc, Lactococcus et Streptococcus (Ackermann, 2012).
Depuis des temps immémoriaux, les BL sont utilisées afin de produire de nombreux produits fermentés tels que les vins, les produits carnés ainsi que quelques produits laitiers comme le fromage ou des boissons de lait fermenté. De nos jours, leur utilisation a été optimisée afin de produire à l’échelle industrielle une grande variété de produits fermentés, mais aussi pour des applications diverses comme la biopréservation ainsi que pour leurs propriétés probiotiques (Champagne et Arora, 2009). L’application la plus utilisée pour les BL est la fermentation laitière. Les genres utilisés en Amérique du Nord sont Lactococcus, Streptococcus, Lactobacillus et Leuconostoc. Bien que le genre Bifidobacterium soit de plus en plus utilisé dans l’industrie de la transformation laitière, il n’est pas reconnu comme BL à cause de son génome riche en G+C et sa faible production d’acide lactique. Par contre, Bifidobacterium est ajouté au ferment pour ses propriétés probiotiques et pour l’ajout de saveur aux produits (Champagne, 1998).
Une grande variété de produits à base de lait fermenté se retrouve dans notre alimentation, comme différents types de fromage (Cheddar, Mozzarella, à pâte molle, à pâte ferme et à pâte pressée cuite), le yogourt, la crème sure et le babeurre (Champagne et Arora, 2009). En industrie, les ferments lactiques sont généralement composés de différentes souches bactériennes et sont directement ajoutés au lait pasteurisé, à raison de 107 bactéries/ml de lait, afin d’obtenir une acidification adéquate (Moineau et al., 2002). Les souches
sont sélectionnées en fonction de leurs caractéristiques physiologiques. Par exemple, certains produits laitiers nécessitent des ferments mésophiles (Lactococcus lactis, Leuconostoc sp.) et d’autres des ferments
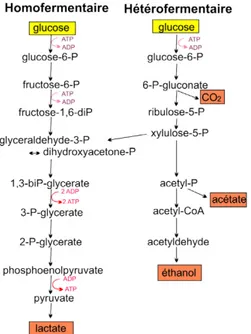
thermophiles (Streptococcus thermophilus, Lactobacillus delbrueckii ssp. bulgaricus). De plus, les souches sont sélectionnées selon le type de fermentation lactique. C’est-à-dire si le produit final de fermentation est exclusivement de l’acide lactique (homofermentaire) ou s’il y a d’autres produits finaux, en plus du lactate (hétérofermentaire), comme du CO2 par exemple (voir Figure 1.1). Finalement, les espèces seront
sélectionnées en fonction du produit alimentaire souhaité.
Figure 1.1. Voies métaboliques homofermentaire et hétérofermentaire de la dégradation des sucres représentées ici
avec le glucose comme exemple. Modifiée de Drider et Prévost, 2009.
1.1.2 Streptococcus thermophilus
Le genre Streptococcus se divise en 55 espèces bien définies et basé sur la séquence de l’ADN 16S spécifique à chacune des espèces. Plusieurs de ces espèces sont reconnues comme des espèces pathogènes autant pour l’humain que pour les animaux, comme S. pneumoniae, S. pyogenes et S. equi. S.
thermophilus est considéré comme un streptocoque viridans du groupe S. salivarius (Facklam, 2002). S. thermophilus était d’abord considérée comme une sous-espèce de S. salivarius, mais des études exhaustives
d’hybridation ADN-ADN ont démontré qu’elle constituait une espèce à part entière (Schleifer et al., 1991) et le tout a ensuite été confirmé par une analyse de Multi-Locus Sequence Typing (MLST) (Hols et al., 2005). L’analyse MLST de plusieurs souches de S. thermophilus isolées de différents produits laitiers a aussi démontré qu’il y a peu de diversité au sein même de l’espèce thermophilus ainsi qu’il existe une corrélation entre la souche, le type de produit laitier et la situation géographique de cette dernière (Bolotin et al., 2004; Hols et al., 2005). Présentement, il y a huit génomes complets de souches de S. thermophilus disponibles sur la base de données du National Center for Biotechnology Information (NCBI) (voir Tableau 1.1). Ses génomes sont homologues, par exemple, 90 % des régions codantes des souches CNRZ1066 et LMG18311 sont
communes démontrant très peu de divergence au sein de l’espèce. Les différences sont majoritairement situées au niveau des gènes codant pour la biosynthèse de polysaccarides extracellulaires, les prophages, la synthèse de bactériocines et l’immunité cellulaire (Bolotin et al., 2004).
Figure 1.2. Observation en microscopie électronique à balayage de la souche S. thermophilus CNRZ1066. Photo prise
par G. Simonson, ASM Microbe Library de NCBI.
Tableau 1.1. Génomes complets des souches de S. thermophilus disponibles.
Souches Taille du
génome (Mb) Contenu G+C Nombre de plasmides Références
ASCC 1275 1,846 39,1 % - (Wu et al., 2014)
CNRZ1066 1,796 39,1 % - (Bolotin et al., 2004)
DGCC7710 1,798 38,9 % - (Barrangou et al., 2013)
JIM 8232 1,929 38,9 % - (Delorme et al., 2011)
LMD-9 1,856 39,1 % 2 (Makarova et al., 2006)
LMG 18311 1,796 39,1 % - (Bolotin et al., 2004)
MN-ZLW-002 1,848 39,1 % - (Kang et al., 2012)
ND03 1,831 39,0 % - (Sun et al., 2011)
Données recueillies de la base de données NCBI.
S. thermophilus est de forme ovoïde et observée en paire ou en chaîne (Hardie et Whiley, 1995). Elle est la
seule espèce du groupe S. mitis à être reconnue comme un organisme Generally Recognized As Safe (GRAS) par la Food and Drug Administration (FDA, http://www.accessdata.fda.gov). En effet, S. thermophilus ne possède aucun gène de virulence associé aux espèces de streptocoques reconnues comme pathogènes (Bolotin et al., 2004). De plus, S. thermophilus est la seule espèce de streptocoques à être volontairement ajoutée dans les ferments. L’industrie de la transformation laitière utilise S. thermophilus pour plusieurs applications, et ce, depuis des décennies (Delorme, 2008).
Streptococcus thermophilus est la deuxième BL en importance dans l’industrie laitière après L. lactis. En effet,
l’apport économique de S. thermophilus est de plus de 40 milliards de dollars par année (Blomqvist et al., 2006). Il est utilisé dans les ferments lactiques thermophiles afin de produire principalement le yogourt, mais aussi plusieurs types de fromage à pâte pressée cuite comme l’Emmental, le Gruyère et le Parmigiano, d’autres types de fromage comme le Cheddar et le Mozzarella ainsi que plusieurs variétés de fromage à pâte
2003; Delorme, 2008). Son rôle principal est de convertir rapidement le lactose en lactate afin de permettre une diminution rapide du pH, ainsi que la production de métabolites importants pour leurs propriétés technologiques (Delorme, 2008).
S. thermophilus peut être utilisée seule ou en combinaison avec différentes espèces de lactobacilles pour la
fabrication de différents fromages (Delorme, 2008). De plus, elle peut aussi être ajoutée à des ferments mésophiles composés de L. lactis afin de permettre de diminuer la concentration de bactéries à inoculer pour la fabrication de Cheddar (Lucey, 2003). Par contre, S. thermophilus est exclusivement combiné avec
Lactobacillus delbrueckii ssp. bulgaricus dans les ferments thermophiles destinés à la fabrication de yogourt
(Auclair et Accolas, 1983). Les deux espèces ont un effet synergique sur la fermentation, la texture et la viscosité du yogourt (Broadbent et al., 2003). Cette synergie symbiotique est d’abord provoquée par la protéolyse de la caséine par des protéases secrétées par les lactobacilles rendant disponibles plusieurs métabolites favorables aux streptocoques. Par la suite, plusieurs streptocoques vont fournir des facteurs de croissance afin de stimuler la croissance des lactobacilles (Champagne, 1998).
Plusieurs souches de S. thermophilus sont aussi sélectionnées pour leur production d’exopolysaccarides (EPS) formés de différents monomères de sucres (hétéropolysaccarides) (De Vuyst et al., 2001). Les EPS permettent entre autres de diminuer la synérèse lors du processus de transformation du lait en yogourt, ou plus précisément de diminuer l’expulsion du lactosérum de la matrice de yogourt (Lamontagne, 2002), et ainsi obtenir une matrice plus visqueuse et plus lisse (Broadbent et al., 2003). Les souches productrices d’EPS sont principalement recherchées dans la production de yogourt afin de remplacer l’ajout d’additifs alimentaires servant à l’ajustement du pourcentage de matières solides nécessaires pour l’obtention d’une matrice adéquate (Cerning, 1995; Lamontagne, 2002). C’est tout le contraire pour la production de plusieurs types de fromage qui nécessite une synérèse afin d’obtenir une matrice plus rigide malgré que certaines souches de
S. thermophilus productrices d’EPS soient utilisées pour la production de Mozzarella (Bahrs et Pruss, 1981;
Hayashi et Nauth, 1995).
Des études ont permis de caractériser plusieurs bactériocines produites par certaines souches de S.
thermophilus. Ces bactériocines sont efficaces contre plusieurs microorganismes pathogènes comme Listeria monocytogenes et Clostridium botulinum. Par contre, seulement certaines de ces souches sont utilisées en
industrie, car certaines bactériocines peuvent aussi affecter les lactobacilles fermentaires (Delorme, 2008). D’un autre côté, S. thermophilus est aussi utilisée comme probiotique grâce à des gènes codant pour l’adhérence, la tolérance à l’acide, la production de bactériocines et l’utilisation du lactose empêchant la colonisation des microorganismes pathogènes (Prajapati et al., 2013). De plus, il a aussi été montré que S.
thermophilus augmente l’efficacité des propriétés probiotiques du genre Bifidobacterium dans un ferment
(Saavedra et al., 1994; Bin-Nun et al., 2005).
1.2 Les bactériophages
1.2.1 Historique
Les bactériophages, communément appelés phages, sont des parasites obligatoires n’ayant aucune activité biochimique à l’extérieur d’une cellule et n’infectent que les bactéries (Ackermann et DuBow, 1987a; Kutter et Sulakvelidze, 2005). Ils sont spécifiques à un nombre très limité d’hôtes et souvent à une seule souche bactérienne. Ils ont été découverts au début du 20e siècle. En 1915, Frederick William Twort avait remarqué
quelques colonies de microcoques d’apparence vitreuse. Lorsqu’il examina ces colonies au microscope, il observa seulement des fragments de cellules bactériennes. Presqu’en même temps, en 1917, Félix d’Hérelle observa la dissolution de Shigella dysentriae, terme qu’il utilisa pour définir la lyse bactérienne (d'Hérelle, 1926; Ackermann et DuBow, 1987a; Kutter et Sulakvelidze, 2005; Calendar, 2006). Contrairement à Twort, Félix d’Hérelle se concentra sur ce phénomène qu’il nommera par la suite la bactériophagie (d'Hérelle, 1926). Malheureusement, l’existence des bactériophages n’était pas bien reçue par la communauté scientifique faute de preuves visuelles directes. Par contre, l’avènement du microscope électronique lors des années 1940 a permis l’observation des premiers bactériophages confirmant ainsi l’existence des phages (Ackermann et DuBow, 1987a).
Suite aux travaux de d’Hérelle, plusieurs scientifiques se sont penchés sur l’étude des bactériophages. Ce qui est fascinant, c’est que les phages sont ubiquitaires comme les bactéries. Ils ont d’abord été isolés dans les excréments des humains et par la suite dans divers environnements, que ce soit sur la terre ou dans l’eau et à des températures et des pressions tant normales qu’extrêmes. Les phages sont maintenant reconnus comme l’entité biologique prédominante sur Terre et leur nombre est estimé à environ 1030 à 1032 phages, soit dix fois plus élevé que le nombre total de bactéries (Breitbart et Rohwer, 2005). Par conséquent, ils jouent un rôle écologique très important en limitant la prolifération des bactéries et en permettant le relargage de nutriments dans l’environnement (Kutter et Sulakvelidze, 2005; Calendar, 2006; Leblanc et al., 2009).
1.2.2 Classification des bactériophages
L’observation en microscopie électronique d’une panoplie de phages a permis de développer une première classification des phages basée sur leur morphologie. Ensuite, plusieurs systèmes de classification ont été proposés pour différencier les types de bactériophages en plus de leur morphologie, soit par leurs acides nucléiques ou encore par les différentes composantes retrouvées dans la capside (Ackermann et DuBow,
1987a; Kutter et Sulakvelidze, 2005; Calendar, 2006). L’utilisation du terme « phage » est réservée au virus infectant les organismes du domaine Eubacteria tandis que le terme « virus » est appliqué aux organismes des domaines Archaea et Eukaryota (Ackermann et Prangishvili, 2012).
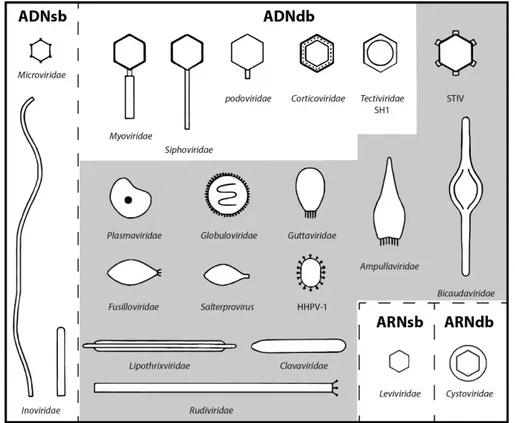
Les premières observations en microscopie électronique ont été recensées en 1959 suite à la description d’une nouvelle méthode de coloration des préparations de virus (Brenner et Horne, 1959; Brenner et al., 1959). Par la suite, une classification basée sur la morphologie des particules virales et la composition en acide nucléique de leur génome a été acceptée en 1967. Elle comportait 111 différents virus observés en microscopie électronique et qui étaient divisés en six morphotypes identifiés de A à F (Bradley, 1967). De nos jours, près de 6300 virus ont été observés en microscopie électronique, dont plusieurs avec des morphologies inusitées (voir figure 1.3). La grande majorité des virus observés, soit 98,5 %, sont des bactériophages probablement en raison des conditions de culture plus faciles en laboratoire pour les bactéries que pour les archaebactéries, qui sont un caractère plutôt extrêmophile (Ackermann et Prangishvili, 2012).
Figure 1.3. Morphotypes des virus infectant les procaryotes. Les lignes pointillées séparent les morphotypes selon la
nature des acides nucléiques qui compose leur génome. Les morphotypes sur fond blanc représentent les bactériophages et certains archaevirus, tandis que les morphotypes sur fond gris représentent les virus infectant seulement le domaine des Archaea. Figure modifiée de (Ackermann et Prangishvili, 2012).
T4 P22 p2
1.2.3 Caudovirales
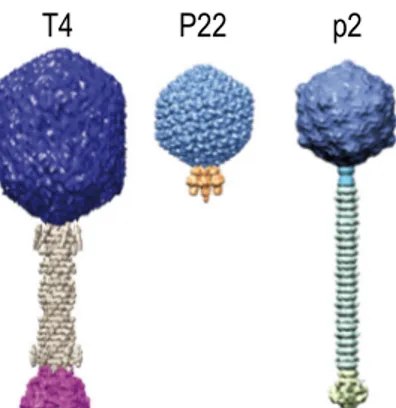
Le plus grand nombre des bactériophages observés font partie de l’ordre des Caudovirales (virus caudés) (Kutter et Sulakvelidze, 2005; Calendar, 2006; Ackermann et Prangishvili, 2012). La morphologie des phages de l’ordre des Caudovirales comprend une capside, renfermant un génome à ADN double brin (ADNdb), attachée à une queue tubulaire, parfois accompagnée d’une plaque basale et des fibres de queue (Ackermann et DuBow, 1987b). La capside des phages caudés est généralement isométrique et est composée de divers arrangements de capsomères prenant des formes d’icosaèdres, d’octaèdres ou encore de dodécaèdres et parfois certaines sont plutôt allongées (Ackermann et Prangishvili, 2012; Prasad et Schmid, 2012). La morphologie des queues est très diversifiée et est la principale caractéristique permettant la division de l’ordre
Caudovirales en trois familles Myoviridae, Siphoviridae et Podoviridae (voir la figure 1.4). La famille des
Myoviridae comprend des phages ayant une queue composée d’une gaine large et contractile recouvrant un
tube central. Le phage T4 est le membre le plus connu et l’un des plus étudiés. Les membres de la famille des
Siphoviridae présentent une longue queue non contractile généralement moins large que celles des membres Myoviridae et le membre le plus connu et étudié est le phage lambda. La famille des Podoviridae regroupe les
phages ayant une queue très courte et non contractile, comme le phage T7 (Ackermann, 2009; King et al., 2012). Les génomes des phages de l’ordre Caudovirales sont présents dans la capside en une seule copie sous forme linéaire, leur taille varie de 18 kpb à plus de 500 kpb et constitue environ la moitié des composantes de la particule virale. Le contenu G+C des génomes de phages est aussi diversifié, soit de 27 % à 72 %, et est généralement similaire au contenu G+C de son hôte bactérien (King et al., 2012).
Figure 1.4. Structures types des trois familles de l’ordre Caudovirales. La famille Myoviridae est représentée par le phage
T4, P22 représente la famille Podoviridae et la famille Siphoviridae est représentée par le phage p2. Figure modifiée de (Veesler et Cambillau, 2011).
1.2.4 Cycle d’infection
Les bactériophages infectent les bactéries selon six grandes étapes générales et communes soit, (1) l’adsorption réversible et irréversible aux cellules, (2) l’entrée du génome viral dans le cytoplasme de l’hôte, (3)
la réplication du génome viral, (4) l’expression des protéines structurales, (5) l’assemblage des particules virales et (6) le largage des particules virales dans l’environnement suite à la lyse cellulaire (voir la figure 1.5) (Labrie et al., 2010).
Figure 1.5. Cycles lytique et lysogène des bactériophages lors de l’infection de son hôte bactérien. Figure modifiée de
Labrie et al., 2010. 1.2.4.1 Cycle lytique
1.2.4.1.1 Adsorption des particules virales
Les phages virulents, c’est-à-dire que l’infection des cellules hôtes mène invariablement à la lyse bactérienne, suivent les six grandes étapes mentionnées ci-haut. Des contacts aléatoires et réversibles se produisent avec des cellules présentes dans le même environnement jusqu’à la reconnaissance d’un ou plusieurs récepteurs accessibles à la surface des cellules. Les récepteurs sont soit des polysaccarides (la chaîne latérale O ou le peptidoglycane), des protéines membranaires ou encore des acides téichoïques et lipotéichoïques (Kutter et
al., 2005). L’attachement initial se produit généralement par l’entremise de fibres de queue ou encore à l’aide
de multimères de protéines de liaison au récepteur (Receptor Binding Protein, RBP). Une fois l’attachement initial achevé, les protéines subissent des changements de conformation irréversibles menant à l’entrée du génome viral à l’intérieur de la cellule grâce à divers processus d’injection ou d’éjection (Kutter et al., 2005; Casjens et Molineux, 2012; Davidson et al., 2012; Leiman et Shneider, 2012).
1.2.4.1.2 Entrée de l’ADN viral dans le cytoplasme
Suite à l’attachement final du phage à son récepteur, les changements de conformation vont permettre la sortie du génome viral via la queue jusqu’au cytoplasme de la cellule infectée. Les membres de la famille
Myoviridae vont utiliser la contraction des multimères protéiques de la queue afin de percer la membrane
externe (bactéries à Gram négatif) comme le ferait une aiguille. Par la suite, les protéines servant à percer le trou se détachent afin d’ouvrir le tube de la queue et permettre la sortie de l’ADN vers le cytoplasme (Leiman et Shneider, 2012). Pour les membres de la famille Siphoviridae, la stratégie est légèrement différente. Pour infecter les bactéries à Gram positif, certains phages doivent d’abord hydrolyser l’épaisse couche de peptidoglycane afin d’accéder aux récepteurs membranaires, et ce, à l’aide de domaines spécifiques présent à la partie distale de la queue. Dans d’autres cas, le récepteur final peut être situé à la surface du peptidoglycane. Dans la majorité des cas, la liaison au récepteur provoque des changements de conformation avec ou sans la présence d’ions divalents, comme le CaCl2, permettant l’ouverture du passage pour l’ADN.
Par contre, la protéine étalon (Tape Measure Protein, TMP) servant d’échafaudage aux protéines de la queue bloque le passage de l’ADN. Donc, la TMP possède souvent des domaines hydrolase permettant de dégrader le peptidoglycane, la bicouche lipidique ou encore des protéines membranaires afin de permettre le libre passage de l’ADN de la capside au cytoplasme (Davidson et al., 2012). D’autres particularités ont été observées chez les membres de la famille Podoviridae, comme la dégradation de la capsule extracellulaire produite par certaines espèces bactériennes. La capsule joue plusieurs rôles, comme par exemple, des facteurs de virulences, résistance à certains antibiotiques ou pour se protéger des phages en masquant le récepteur final (Casjens et Molineux, 2012). Ensuite, l’ADN viral doit être acheminé vers le cytoplasme. L’hypothèse selon laquelle la pression à l’intérieur de la capside serait assez élevée pour permettre l’expulsion complète du génome est de moins en moins plausible. Seulement une portion du génome pourrait être expulsé de cette façon (Casjens et Molineux, 2012; Davidson et al., 2012; Leiman et Shneider, 2012). Il a été démontré que certains phages exposent graduellement leur génome grâce à un couplage avec la transcription des gènes (Molineux, 2001). De plus, des calculs ont démontré que la pression diminue de façon exponentielle au fur et à mesure que le génome sort de la capside, contrairement à l’hypothèse initiale que seule la pression accumulée était suffisante pour l’éjection totale du génome. Par conséquent, différents mécanismes permettant la translocation de l’ADN sont envisageables, comme des canaux membranaires assez grands pour de l’ADN double brins ou encore grâce à une dépolarisation localisée du potentiel membranaire (Casjens et Molineux, 2012; Davidson et al., 2012; Leiman et Shneider, 2012).
1.2.4.1.3 Réplication du génome
Le génome viral d’ADNdb est souvent présent sous forme circulaire dans le cytoplasme (pour la réplication de ce dernier) et linéaire à l’intérieur de la capside. Par conséquent, le génome linéaire est rapidement circularisé dès son entrée dans le cytoplasme en joignant des extrémités cohésives ou par une recombinaison entre des extrémités redondantes. Les génomes circulaires sont ensuite répliqués à l’aide des polymérases bactériennes ou leur propre polymérase. La réplication est soit de type thêta (θ) ou de type cercle-roulant
selon la machinerie de réplication utilisée. La réplication de type cercle-roulant produit de long concatémères contenant plusieurs copies du génome viral (Kutter et al., 2005).
1.2.4.1.4 Transcription et traduction
À l’entrée du génome viral, quelques gènes sont transcrits majoritairement à l’aide des ARN polymérases bactériennes et ce, au moment que la réplication du génome viral. Ces gènes sont dits précoces dû à leur profil d’expression durant l’infection et les promoteurs peuvent être reconnus par les enzymes bactériennes, car aucune enzyme virale n’est présente avant l’infection. Les gènes précoces, ou cadres de lecture ouverts (orfs) précoces, codent généralement pour des protéines impliquées dans la réplication du génome et pour des activateurs de transcription des gènes dits médians ou tardifs, mais codent aussi pour des répresseurs afin de contrôler plus efficacement l’expression de protéines selon les besoins (Josephsen et Neve, 2004). 1.2.4.1.5 Assemblage des particules virales
Les gènes médians et tardifs codent généralement pour la morphogenèse de la capside et de la queue des particules virales (résumé de l’assemblage à la figure 1.6). La morphogenèse est guidée par des protéines d’échafaudage et des protéases spécifiques qui permettent l’assemblage des multiples copies de capsomères ou des multiples copies de la même protéine afin de former des capsides et des queues qui sont généralement assemblées indépendamment (Prasad et Schmid, 2012). À la fin de l’assemblage, la capside est considérée comme une procapside (incomplète) et l’empaquetage de l’ADN viral permet d’obtenir la forme finale de la capside. Plusieurs systèmes d’empaquetage sont utilisés dans la nature, mais deux types particuliers prédominent pour les phages de l’ordre des Caudovirales. Les deux types sont basés sur l’action de la terminase qui se lie à l’ADNdb et à une structure à la base de la capside soit la portale ou le connecteur capside-queue. La terminase est normalement constituée d’une grande et d’une petite sous-unité chacune ayant leur propre fonction. D’abord, la petite sous-unité de la terminase permet la reconnaissance et la liaison des concatémères d’ADNdb à un site spécifique, soit cos ou pac, selon le type de terminase. Par la suite, la grande sous-unité de la terminase se lie à la procapside au niveau de la protéine portale et effectue la translocation de l’ADNdb vers l’intérieur de la procapside grâce à une activité adénosine-5’-triphosphatase (ATPase) qui permet l’hydrolyse des molécules d’adénosine triphosphate (ATP) afin de fournir de l’énergie nécessaire à la translocation. De plus, la grande sous-unité de la terminase effectue la coupure finale au site
cos ou lors que la capside est pleine (type pac). Les terminases de type cos coupent seulement au site cos
faisant en sorte que la progéniture des phages soit homogène par la longueur de son génome et de l'information qu'il contient. Par contre, les terminases de type pac reconnaissent spécifiquement un premier site pac des concatémères. Ensuite, l’ADN est entré dans la procapside jusqu’à ce qu’elle soit remplie au maximum, soit entre 102 % et 110 % du génome, et la terminase coupe l’ADNdb à ce moment. Par conséquent, il y aura présence d’extrémités redondantes parfaites pour la recombinaison lors de la prochaine
infection, et il y aura un décalage dans la séquence génomique dans la prochaine capside et les génomes linéaires de la progéniture sont hétérogènes par rapport aux extrémités redondantes (Kutter et al., 2005; Casjens et Gilcrease, 2009). Une fois l’assemblage indépendant des capsides et des queues achevé, les deux composantes s’auto-assemblent à une même structure de jonction, nommée le cou ou le collier, à la base de la capside (où l’ADN a été encapsidé) et la queue vient par le fait même bloquer l’expulsion prématurée de l’ADN (San Martín, 2013).
1.2.4.1.6 Lyse cellulaire
La lyse cellulaire est une étape cruciale du cycle infectieux. En effet, la lyse doit être extrêmement bien régulée afin de maximiser le nombre de phages matures qui seront largués dans l’environnement. Une lyse hâtive mènerait au largage d’une majorité de phages immatures et non infectieux. Donc, la régulation de la lyse s’opère à différents niveaux, comme par exemple, au niveau de l’expression des gènes de lyse ou encore sur l’activation ou l’inhibition des protéines impliquées dans la lyse. D’ailleurs, si les systèmes de régulation sont mutés de façon à modifier à la hausse ou à la baisse le moment de la lyse, alors une pression sélective très forte pousse les phages à modifier leur génome de manière à ramener un temps de lyse similaire au temps requis par le phage sauvage (Kutter et al., 2005). Les familles de protéines les plus impliquées dans la lyse cellulaire sont l’endolysine et la holine.
Le rôle de l’endolysine est de déstabiliser le peptidoglycane par l’hydrolyse de liens glycosidiques spécifiques selon le genre ou l’espèce de la bactérie infectée. Cette spécificité a permis de classer les endolysine en plusieurs classes. Deux types majeurs d’endolysines ont été décrits jusqu’à présent, l’endolysine cytoplasmique et l’endolysine ancrée sur la face externe de la membrane cytoplasmique. Cette dernière possède un signal de sortie et d’ancrage (signal-anchor-release) à l’extrémité N-terminale qui lui permet d’être secrétée et ancrée sous la forme inactive en attente d’un signal activateur (Young, 2014). Donc, pour que les molécules d’endolysine passent la barrière de la membrane cytoplasmique ou soient activées, d’autres protéines ont un rôle important à jouer, les holines.
La holine possède des domaines transmembranaires qui lui permet de s’insérer dans la membrane cytoplasmique de la bactérie sous forme d’agrégats (Kutter et al., 2005). Lorsque la concentration de holine dans la membrane atteint un certain seuil, les protéines forment un complexe afin de permettre le passage de molécules cytoplasmiques, entre autres, vers la couche de peptidoglycane. Présentement, deux types de holine sont décrits et acceptés, soit les holines qui forment un grand trou (1000 à 3000 holines par trou) qui permettent le passage des endolysines et les pinholines qui forment plutôt des heptamères afin de modifier le potentiel membranaire et permettre la libération des endolysines inactives et ancrées sur la face externe de la membrane cytoplasmique (Young, 2014).
Un troisième groupe de protéines, les spanins, est aussi essentiel afin de permettre aux bactériophages infectant les bactéries à Gram négatif d’obtenir la lyse cellulaire. Les spanins sont secrétées sous une forme inactive sur la face externe de la membrane cytoplasmique. Elles sont activées lors de la dégradation de la couche périplasmique et sont responsables de la dégradation de la membrane externe (Young, 2014).
1.2.4.2 Cycle lysogène
Certains phages sont dits tempérés et peuvent parfois suivre un cycle alternatif dit lysogène. C’est-à-dire qu’une fois le phage adsorbé à la cellule et le génome viral est entré dans le cytoplasme, (A) il est intégré dans le chromosome bactérien sous forme de prophage. Le prophage maintient sa position dans le chromosome grâce à des répresseurs exprimés pendant la lysogénie. Par la suite, (B) le prophage est répliqué en même temps que le chromosome bactérien et il est transmis verticalement dans la population bactérienne. Lorsque la cellule se trouve en situation de stress, comme des radiations UV ou des antibiotiques, (C) le prophage s’excise du chromosome et va reprendre le cycle lytique afin de se propager dans l’environnement (voir la figure 1.5).
1.2.5 Phages infectant Streptococcus thermophilus
Plus de 345 phages infectant S. thermophilus ont été isolés et décrits depuis les premiers phages caractérisés en 1953 et en 1955 (Deane et al., 1953; Kiuru et Tybeck, 1955; Quiberoni et al., 2010). La plupart d’entre eux ont été isolés lors d’études écologiques menées dans des usines de transformation laitière et étalées sur des périodes de temps pouvant s’étendre sur plusieurs années (Quiberoni et al., 2010). En effet, les usines des grandes entreprises de transformation laitière forment des écosystèmes favorisant la sélection et la multiplication de phages par l’utilisation routinière et massive de souches bactériennes spécifiques. Les phages proviennent majoritairement des matières premières, comme par exemple le lait, et la majorité d’entre eux peuvent résister à la pasteurisation (Suárez et al., 2002; Madera et al., 2004; Atamer et al., 2009). De plus, certaines souches lysogènes peuvent mener à l’induction de phages strictement virulents suite à des erreurs d’excision (Bruttin et al., 2002).
Ces études ont aussi démontré que tous les phages de S. thermophilus sont des membres de l’ordre des
Caudovirales et de la famille Siphoviridae. La taille moyenne de la capside des phages de S. thermophilus est
de 60 nm de diamètre. La longueur des queues varie entre 200 et 300 nm et la taille des génomes fluctue entre 29 kpb et 43 kpb (Quiberoni et al., 2010). Lors de ces études environnementales en industrie de la transformation laitière, plusieurs approches ont été utilisées afin de proposer une classification générale des phages dans des groupes ou espèces, comme c’est présentement le cas pour les phages infectant L. lactis (Deveau et al., 2006). Ces approches étaient basées sur la morphologie, le spectre lytique, les profils de restriction et l’homologie génétique, mais aucune d’elles ne semblait optimale (Le Marrec et al., 1997). Il existe une corrélation entre le mécanisme d’empaquetage de l’ADN (pac ou cos), certaines protéines structurales majeures et l’astringence de l’hybridation des ADN génomiques. En effet, tous les phages de type pac ont des génomes homologues tout comme pour les phages de type cos et les profils SDS-PAGE des protéines
structurales démontrent trois bandes majeures (41, 25 et 13 kDa) pour les phages de type pac et deux bandes majeures (32 et 26 kDa) pour les phages de type cos (Le Marrec et al., 1997).
Jusqu’à maintenant, 12 génomes complets de phages infectant S. thermophilus sont disponibles dans la base de données NCBI (http://www.ncbi.nlm.nih.gov) (Le Marrec et al., 1997; Stanley et al., 1997; Desiere et al., 1998; Neve et al., 2003; Lévesque et al., 2005; Deveau et al., 2008; Guglielmotti et al., 2009; Mills et al., 2011). Des comparaisons entre les séquences en acides aminées des orfs des 12 phages illustrent bien l’homologie entre chacun des deux types cos et pac (figure 1.7). Le génome d’un phage infectant S.
thermophilus peut être divisé en quatre grands modules. Le module de lysogénie est toujours présent
quoiqu’incomplet pour les phages virulents. La présence de gènes résiduels laisse croire que certains des phages étaient autrefois tempérés et seraient devenus virulents à la suite d'une erreur d’excision au niveau des gènes nécessaires à l’intégration et au maintien du génome viral dans le chromosome de l’hôte (figure 1.7). Le module de réplication d’ADN et de régulation de la transcription est très diversifié. La portion réplication de l’ADN est divisé en deux classes hautement conservées (Sfi21 et 7201) et qui est probablement transmis par des transferts horizontaux entre les phages durant une co-infection ou par recombinaison avec les génomes de prophages parfois présents dans le génome de l’hôte. De son côté, la portion régulation de la transcription évolue par différents événements d’insertion ou de délétion (figure 1.7). Le troisième module comprend les gènes médians et tardifs liés à la morphogénèse de la particule virale. Ce module est très conservé et peut être classé en trois groupes selon le type d’encapsidation des génomes viraux (voir figure 1.7). Les différences seraient surtout dues à l’accumulation de mutations ponctuelles. Le dernier module regroupe les gènes liés aux fibres caudales (si présentes) et à la lyse cellulaire. Des insertions, délétions et de la recombinaison seraient les causes principales de la diversité rencontrée dans ce module (Brüssow et Kutter, 2005). L’homologie entre les phages infectant S. thermophilus a permis de définir un cœur génomique (ou core genome) pour les phages de type pac et de type cos qui comprend présentement 24 gènes et 26 gènes respectivement (Quiberoni et al., 2010).
1.2.6 Le phage 2972
Le phage 2972 est le modèle d’étude pour les phages de type pac (Quiberoni et al., 2010). Comme tous les phages de S. thermophilus, il est membre de la famille Siphoviridae. Il dispose d’une capside icosaédrique de 55 nm de diamètre et d’une queue non contractile faisant en moyenne 260 nm de longueur. Son génome est le plus petit des 12 génomes disponibles et il contient 34 704 pb et le contenu G+C de 40,15 % est similaire à celui de son hôte S. thermophilus DGCC7710 (38,9 %). Quarante-quatre orfs putatifs ont été identifiés suite à des analyses bioinformatiques. Les orfs sont orientés dans le même sens et 28 d’entre eux ont une fonction connue (voir le tableau 1.2). Huit protéines structurales sont identifiables par des analyses en spectroscopie
de masse (LC-MS/MS) dont les trois protéines majeures associées aux phages de type pac (Lévesque et al., 2005).
Les gènes du phage 2972 sont classés en trois groupes selon leur profil de transcription lors de l’infection. La transcription des gènes orf30 à orf44 et de l’orf1 est précoce et débute dès l’entrée du génome virale dans la cellule. Les transcrits des gènes orf2 à orf12 sont considérés comme médians, tandis que les transcrits des gènes orf13 à orf26 sont tardifs (Duplessis et al., 2005). Ces résultats illustrent bien l’organisation modulaire et séquentielle du génome du phage 2972. De plus, toutes les protéines sont exprimées durant l’infection sauf que les protéines ORF30, ORF39 et ORF41 n’ont pas été détectées malgré que leur gène respectif soit transcrits en ARNm (Duplessis et al., 2005; Young et al., 2012). Les profils de transcription sont en corrélation directe avec les fonctions qui ont été associées aux orfs par les analyses bioinformatiques ainsi que les analyses de LC-MS/MS (Tableau 1.2). Il est donc possible de confirmer les fonctions préalablement associées aux orfs et même de suggérer des fonctions aux orfs sans fonction prédite d’après leur position dans le génome. Par contre, il est toujours impossible de savoir si ces orfs sont essentiels pour la propagation du phage 2972.
Figure 1.7. Comparaison de l’organisation génomique des phages infectants S. thermophilus. Chaque flèche représente
un orf (débutant par un codon de départ jusqu’à un codon d’arrêt). Les orfs de même couleur ont une identité en acides aminés de 70 % et plus et sont reliés par un ombrage gris lorsque possible. Le blanc représente les orfs uniques. Les différents modules des génomes sont représentés par une couleur de fond. Le bleu est pour le module de lysogénie, le
Tableau 1.2. Gènes du phage 2972 et leurs fonctions prédites.
Gènes aa Fonction prédite (domaine) Identification
(BI / SDS ) Exprimée durant l’infectiona
orf1 137 Activateur de transcription (superfamille phage ArpU) BI Oui
orf2 150 Petite sous-unité de la terminase (superfamille terminase_2) BI Oui
orf3/4 411 Grande sous-unité de la terminase (superfamille terminase_3) BI Oui
orf5 501 Portal (superfamille Portal_SPP1) BI / SDS Oui
orf6 297 Protéine mineure de la capside (superfamille phage_Mu_F) BI Oui
orf7 193 Protéine échafaud de la procapside (superfamille DUF4355) BI Oui
orf8 119 Protéine de la capside BI Oui
orf9 348 Protéine majeure de la capside (Phage_cap_E) BI / SDS Oui
orf10 53 Inconnue - Oui
orf11 113 Joint capside-queue (Phage_connect_1, superfamille gp6_gp15_like) BI Oui
orf12 104 Protéine mineure de structure BI Oui
orf13 112 Inconnue (superfamille DUF646) - Oui
orf14 128 Protéine de structure (superfamille DUF3168) BI Oui
orf15 169 Protéine majeure de la queue (superfamille Phage_tail_2) BI / SDS Oui
orf16 117 Assemblage de la queue (DUF3647) BI Oui
orf17 105 Protéine de la queue SDS Oui
orf18 1517 Protéine étalon BI / SDS Oui
orf19 511 Protéine de la pointe BI / SDS Oui
orf20 1605 Protéine de liaison au récepteur BI / SDS Oui
orf21 673 Protéine de la plaque basale (DUF859) BI / SDS Oui
orf22 128 Inconnue (DUF1366) - Oui
orf23 47 Inconnue - Oui
orf24 110 Inconnue - Oui
orf25 108 Holine (superfamille Holin_LLH) BI Oui
orf26/29 281 Endolysine (Amidase_5) BI Oui
orf27 40 Inconnue - Oui
orf28 61 Inconnue - Oui
orf30 43 Inconnue - Non-détectée
orf31 69 Répresseur de transcription (superfamille HtH_XRE, DUF739) BI Oui
orf32 40 Inconnue - Oui
orf33 157 Inconnue (Sipho_Gp157 superfamily) - Oui
orf34 233 Protéine de liaison à l’ADN simple brin (SSAP) / Recombinase BI Oui
orf35 445 Hélicase (DEXDc (ATP-Mg2+), HELICc (Nt-ATP)) BI Oui
orf36 151 Inconnue (DUF669) - Oui
orf37 271 Réplication d’ADN (Domaine Prim-Pol) BI Oui
orf38 505 Primase (Primase C-term superfamily) BI Oui
orf39 106 Inconnue (Domaine VRR_NUC Type III) BI Non-détectée
orf40 83 Inconnue - Oui
orf41 51 Inconnue - Non-détectée
orf42 170 Liaison à l’ADN (superfamille HTH_XRE, superfamille NTP-Ppase) BI Oui
orf43 105 Inconnue (superfamille DUF1372) - Oui
orf44 235 Inconnue (superfamille DUF1340) - Oui
aa : nombre acides aminés, BI : Bioinformatique, SDS : SDS-PAGE_LC-MS/MS
orfx : Fonction inconnue, orfx : protéine identifiée par LC-MS/MS suite à un SDS-PAGE
a Selon (Young et al., 2012)
Deux introns de groupe I ont été observés dans le génome du phage 2972 et ils ont été caractérisés. Le premier intron de 307 pb sépare le gène de la grande sous-unité de la terminase (formée par l’orf3 et l’orf4). Un deuxième intron de 442 pb s’est inséré dans le gène de l’endolysine (formée par l’orf26 et l’orf29) et, de plus, il porte l’orf27 et l’orf28. Les introns sont auto-épissables lors de la transcription, ce qui permet l’expression de le grande sous-unité de la terminase (l’orf3/4) et de l’endolysine (l’orf26/29) fonctionnelles. Le nombre d’orfs du phage 2972 est donc de 40 (Lévesque et al., 2005). Les introns des groupes I et II sont des éléments d’ADN mobiles auto-épissables pouvant s’insérer dans des gènes sans affecter l’expression des
protéines (Edgell et al., 2011). Ils peuvent ainsi se propager en parasitant les génomes comme les génomes de phages infectant S. thermophilus (Foley et al., 2000; Lévesque et al., 2005).
Malgré toutes les études réalisées sur le génome, la biologie et la morphologie du phage 2972, plusieurs orfs demeurent sans fonction putative. En effet, les gènes contenus dans le module de réplication et de régulation de la transcription codent pour de petites protéines non structurales et sont généralement plus diversifiés entre des phages très rapprochés génétiquement, comme c’est le cas pour les phages infectant S. thermophilus (voir figure 1.7).
1.3 Mutagénèse dirigée chez les bactériophages
1.3.1 Historique
Connaître et comprendre la biologie des phages est toujours aussi important et ce depuis près de 50 ans. Les premiers essais de mutations dans les génomes des bactéries et des virus étaient basés sur la sélection phénotypique suite à des mutations aléatoires. Les cellules et les particules virales étaient mises en contact avec de puissants agents mutagènes afin de créer des dommages à l’ADN. Par exemple, une courte exposition aux rayons ultraviolets (UV) cause la formation de dimères de thymine menant à des erreurs lors de la réplication ou encore un agent intercalant comme le bromure d’éthidium menant au même genre d’erreurs de réplication (Marinelli et al., 2008). La sélection des mutants est basée sur un phénotype comme l’auxotrophie d’acides aminés, différentes morphologies coloniales ou des plages de lyse, infection de souches auparavant résistantes ou encore une résistance acquise aux antibiotiques. Ces processus sont longs et onéreux selon la fréquence de la mutation recherchée. De plus, la génétique inversée est nécessaire afin d’identifier les mutations ou les gènes mutés par complémentation ou par analyse de recombinaison avec d’autres mutants (Shortle et al., 1981).
Par la suite, de nouvelles méthodes ont été élaborées afin de faire de la mutagénèse dirigée. La mutagénèse dirigée permet de modifier des régions ciblées de génome. Les méthodes passent par une étape préliminaire
in vitro à l’aide d’enzymes de restriction afin de réaliser des digestions enzymatiques partielles ou complètes
ou encore par d’autres étapes de modifications enzymatiques, mais aussi par l’utilisation de nucléotides analogues. Les ADN sont ensuite transformés dans des cellules afin de compléter le processus de mutation et de tester les effets apportés par les mutations. Des insertions et des délétions, des substitutions de bases et des mutations spécifiques à des sites précis ont été réalisées depuis l’avènement de l’ADN recombinant (Shortle et al., 1981).
1.3.2 La recombinaison homologue
La mutagénèse dirigée utilise les systèmes de réparation d’ADN comme la réparation dirigée par l’homologie (homology-directed repair, HDR) de la cellule. La voie HDR permet de réparer les bris d’ADN souvent double brin (double-strand break, DSB) avec la présence d’une molécule d’ADN homologue à celle brisée afin de réparer le bris par recombinaison homologue (Lieber, 2010). Ce processus est fréquemment sollicité chez les organismes diploïdes. Donc, pour les organismes haploïdes qui ne possèdent qu’une seule copie de leur chromosome, un gabarit de séquence homologue doit être ajouté dans les cellules.
Les systèmes de recombinaison homologue reposent sur deux activités protéiques, soit une activité exonucléase pour la dégradation d’un des deux brins d’ADN ainsi qu’une activité d’appariement de l’ADN afin d’apparier deux brins d’ADN complémentaires. Le processus de recombinaison peut emprunter deux voies soit la voie RecA-dépendante (invasion de l’ADNdb), soit la voie RecA-indépendante (appariement d’ADNsb) (figure 1.8) (Murphy, 2012). La voie RecA-dépendante requiert la liaison de la protéine RecA, protéine liant l’ADNsb (single-strand binding protein, SSB), à l’extrémité 3’ d’un ADNsb de façon à former un filament nucléoprotéique. Par la suite, le filament va envahir l’ADNdb à la recherche d’une séquence complémentaire au brin recouvert par RecA. Une fois la séquence complémentaire trouvée, il se forme une jonction d’Holliday qui est résolue par la résolvase RuvC (figure 1.8). De l’autre côté, la voie RecA-indépendante se fait d’abord par la dégradation d’un des deux brins d’ADN de deux molécules d’ADNdb homologues et une protéine d’appariement d’ADNsb (single-strand annealing protein, SSAP) se lie à l’extrémité d’ADNsb provoqué par l’exonucléase à raison d‘une unité protéique par molécule d’ADN. Les deux unités de SSAP apparient les brins complémentaires ensemble et les trous formés sont réparés par l’ADN polymérase I (Court et al., 2002). La séquence homologue peut être insérée dans un vecteur ou être sous forme linéaire d’ADNdb ou d’ADNsb. Ensuite, le gabarit de recombinaison est directement transformé dans les cellules ou par l’intermédiaire d’un vecteur viral. Par contre, la préparation du plasmide recombinant est longue, coûteuse et l’efficacité de recombinaison est faible chez les procaryotes et nécessite souvent deux évènements de recombinaison (Shortle et al., 1981; Court et al., 2002). Par exemple, les essais de recombinaison dans Escherichia coli nécessitent l’inactivation de la nucléase RecBCD, qui dégrade rapidement l’ADN linéaire transformé, ainsi que des séquences d’homologie de plus de mille paires de bases comparativement aux systèmes de réparation d’ADN par recombinaison homologue efficaces retrouvés chez la levure qui nécessitent que des séquences d’homologie de 50 pb (Ellis et al., 2001; Court et al., 2002).
1.3.3 Le recombineering
L’utilisation de protéines virales a été l’alternative de choix afin de pallier à la faible efficacité de recombinaison chez E. coli. Le recombineering se décrit comme le génie génétique combiné avec les fonctions de recombinaison menée par des protéines de phages par l’intermédiaire de courtes séquences homologues (Copeland et al., 2001; Ellis et al., 2001). Le système de recombinaison Red du phage λ a été d’abord optimisé de manière à inhiber la dégradation de l’ADN linéaire par la nucléase RecBCD ainsi qu’augmenter l’efficacité de la recombinaison homologue (Court et al., 2002). Le système Red est constitué des gènes du phage λ exo, bet et gam placés sous le contrôle d’un promoteur inductible et le tout est inséré à l’intérieur du génome bactérien, ce qui permet une augmentation considérable du taux de cellules recombinantes (Murphy, 1998). Par la suite, plusieurs autres systèmes basés sur les recombinases de phages ont été développés afin de diversifier les genres et les espèces bactériennes modifiables par l’approche de recombineering (Murphy, 2012).
Figure 1.8. Modèles des deux voies de réparation HDR. Figure modifiée de Murphy, 2012.
Certains ont démontré que de courtes séquences homologues étaient suffisantes afin d’obtenir une recombinaison efficace. Par conséquent, de courts produits PCR peuvent être utilisés comme gabarit de recombinaison rendant la méthode plus optimale. De plus, de l’ADNsb linéaire (oligonucléotide) permet l’obtention de meilleurs rendements de recombinaison que l’ADNdb (Ellis et al., 2001; Swaminathan et al.,
2001). D’un autre côté, l’efficacité du recombineering est dépendante du sens de la réplication du génome, c’est-à-dire que si le brin complémentaire à l’oligonucléotide de recombinaison est le brin retardé, alors le rendement est plus grand que pour le brin continu (Costantino et Court, 2003; van Kessel et Hatfull, 2007; Sawitzke et al., 2011). Le recombineering permet de réaliser différents types de mutations comme des insertions, des délétions, des mutations ponctuelles, des inversions et des duplications de gènes en plus de modifier rapidement et efficacement des chromosomes bactériens et des plasmides, tant procaryotes que eucaryotes (Murphy, 2012).
1.3.4 Le recombineering des bactériophages par électroporation d’ADN
L’efficacité et la rapidité du recombineering ont mené au développement de l’approche pour la mutagénèse dirigée des bactériophages tempérés, présents dans le chromosome bactérien ou encore sous forme de chromosomes de phages artificiels (phage artificial chromosome, PAC) (Oppenheim et al., 2004). Par la suite, le recombineering a été adapté afin de modifier le génome des phages virulents qui doivent être modifiés uniquement durant le cycle infectieux. Les expériences menées chez les phages infectant E. coli doivent passer par différentes étapes. D’abord on doit permettre l’adsorption des phages et l’entrée des génomes dans des cellules compétentes. Ensuite, réaliser l'électroporation de l’oligonucléotide de recombinaison, le tout suivi de l’induction thermique du système de recombinaison. Finalement, les plages de lyse doivent être criblées par PCR ou par des analyses de type Southern afin d’identifier les phages recombinants (Thomason
et al., 2009; Marinelli et al., 2012)
Le recombineering des bactériophages par électroporation d’ADN (Bacteriophage Recombineering of
Electroporated DNA, BRED) a été développé d’abord pour l’étude des phages infectant les mycobactéries et
ensuite adapté pour les phages infectant E. coli (Marinelli et al., 2012). Le principe du BRED est similaire au recombineering. Il suffit de co-électroporer un oligonucléotide contenant la mutation souhaitée et l’ADN génomique du phage à modifier dans une souche possédant un système de recombinaison viral, comme par exemple Red pour E. coli ou le système Che9c pour Mycobacterium smegmatis (Oppenheim et al., 2004; Datta et al., 2006; Marinelli et al., 2008; van Kessel et Hatfull, 2008). Malgré la très haute efficacité du recombineering chez les prophages et la possibilité de facilement sélectionner les recombinants, le BRED est difficile à optimiser. Le terme BRED a d’abord été proposé suite aux études faites avec les phages infectant des mycobactéries, qui sont d’ailleurs très différentes des cellules d’E. coli. Les mycobactéries sont plus réfractaires à l’électroporation que les cellules d’E. coli et les constructions plasmidiques contenant des génomes de phages virulents ne sont pas assez stables dans les cellules comparativement à ce qui a été réalisé pour le phage λ (Marinelli et al., 2012). L’approche BRED se divise en trois étapes assez simples; 1) co-électroporation de l’ADN génomique du phage à modifier et du gabarit de recombinaison dans des cellules