UNIVERSITE TOULOUSE III – PAUL SABATIER Ecole doctorale Biologie-Santé-Biotechnologies
THESE
Présentée et soutenue en vue de l’obtention du grade de DOCTEUR DE L’UNIVERSITE TOULOUSE III
Sciences
Spécialité : Immunologie et Maladies infectieuses Le 13 février 2008 Par
Florence LEGRAND-ABRAVANEL
IMPLICATIONS THERAPEUTIQUES
DE LA VARIABILITE GENETIQUE
DES GENOTYPES 4 ET 5
DU VIRUS DE L’HEPATITE C
Directeur de thèse : Jacques IZOPET JURY :Madame le Professeur Françoise LUNEL-FABIANI Président
Madame le Professeur Marie-Edith LAFON Rapporteur
Monsieur le Docteur Czeslaw WYCHOWSKI Rapporteur
Monsieur le Professeur Gilles DUVERLIE Examinateur
Monsieur le Docteur Bernard PIPY Examinateur
A l’issue de ce travail je voudrais adresser ma profonde reconnaissance
Au jury
Mme le Professeur Françoise LUNEL-FABIANI Professeur des Universités – Praticien Hospitalier
Je vous remercie de me faire l’honneur de présider aujourd’hui mon jury de thèse avec la double vision que vous avez de chercheur et de clinicien. Veillez trouver ici l’expression de notre profond respect et de toute notre gratitude.
Mme le Professeur Marie –Edith LAFON
Professeur des Universités – Praticien Hospitalier
Je vous suis grée de participer à mon jury en tant que rapporteur, et de me faire partager votre connaissance de la physiopathologie de l’infection par le HCV.
Mr le Docteur Czeslaw WICHOWSKI Directeur de recherche CNRS
Je vous remercie d’avoir accepté la charge d’être rapporteur de cette thèse et de me faire bénéficier de votre expertise dans le domaine du HCV.
Mr le Professeur Gilles DUVERLIE
Professeur des Universités – Praticien Hospitalier
Je vous suis gré de participer à mon jury. Soyez assuré de mes sincères remerciements.
Mr le Docteur Bernard PIPY Directeur de recherche Inserm
Vous nous faites l’honneur de d’accepter de juger notre travail. Veuillez trouver ici l’expression de notre sincère reconnaissance.
Mr le Professeur Jacques IZOPET
Professeur des Universités – Praticien Hospitalier
Tu m’as fait l’honneur de diriger cette thèse pendant 4 ans. Ton accueil au sein du laboratoire de virologie m’a encouragée à poursuivre dans cette discipline. Tu m’a fais partager tes compétences et ton enthousiasme aussi bien dans le domaine de la recherche qu’au niveau hospitalier. Je te remercie pour ta disponibilité, tes encouragements et ta patience qui nous font avancer au quotidien. La perspective de pouvoir continuer à travailler dans ton équipe est pour moi un très grand plaisir
A Martine, merci pour ta disponibilité et tes précieuses compétences techniques. Je tiens à te remercier pour ta contribution à ce travail.
A Florence, avec qui j’ai eu l’immense plaisir de travailler sur le génotype 4.
A Catherine, Karine, Sabine, Stéphanie, Christophe, Jean-Michel, Marcel. Merci pour votre bonne humeur et votre soutien au quotidien dans de nombreux domaines.
A toute l’équipe du laboratoire de virologie pour leur accueil et leur gentillesse.
A tous mes amis
A toute ma famille pour leurs encouragements et leur patience.
Je dédie cette thèse à Alain, Ariane et Nathan.
Alain je te remercie pour ton soutien inconditionnel et pour chaque instant passé à tes côtés.
Table des matières
INTRODUCTION ...7
I. LE VIRUS DE L’HEPATITE C ...9
1. Le virus ... 9
1.1 Structure de la particule virale et du génome ... 9
1.2 Les protéines virales... 13
2. Cycle réplicatif... 21
2.1 Modèles d’étude du cycle viral ... 21
2.2 Le cycle réplicatif ... 25
II. VARIABILITE GENETIQUE DU HCV ...31
1. Réplication et variabilité génétique ... 31
2. Les quasi-espèces ... 32 3. Les génotypes du HCV ... 33 4. Variabilité et épidémiologie ... 36
III. PHYSIOPATHOLOGIE...40
1. Histoire naturelle ... 40 2. Cibles extra-hépatiques ... 433. La réponse immune lors de l’infection... 45
3.1 Les acteurs de la réponse immune ... 45
3.2 Réponse immune lors de l’infection... 46
4. Variabilité et physiopathologie ... 53
IV TRAITEMENT ...56
1. Molécules antivirales utilisées actuellement... 56
1.1 Les interférons... 56
1.2 La ribavirine ... 66
2. La résistance au traitement... 69
2.1 Efficacité et limite des traitements actuels... 69
2.2 Facteurs viraux ... 72
2.3 Facteurs de l’hôte ... 76
2.4 Facteurs pharmacologiques ... 77
3. Les nouvelles approches thérapeutiques... 79
3.1 Les nouveaux interférons ... 79
3.2 Les analogues de la ribavirine ... 79
3.3 Les molécules bloquant l’entrée du virus... 80
3.4 Les molécules bloquant la traduction du génome ... 81
3.5 Les molécules inhibant la protéase ... 82
3.6 Les molécules inhibant la polymérase... 82
3.7 Les inhibiteurs de la cyclophilline ... 83
OBJECTIFS...85
PARTIE I : SENSIBILITE DES GENOTYPES 4 ET 5 A
L’INTERFERON ALPHA...87
Publication n°1 : J Med Virol 2005 ... ... 87
Publication n°2 : J Infect Dis 2004 ... ... 94
PARTIE II : DIVERSITE DES GENOTYPES 4 ET 5...100
Publication n°3 : J Gen Virol 2005 ... ... 100
Publication n°4 : J Virol 2007 ... ... 110
DISCUSSION ET PERSPECTIVES ...118
REFERENCES BIBLIOGRAPHIQUES...132
INTRODUCTION
Le virus de l’hépatite C (HCV) a été cloné chez un chimpanzé expérimentalement infecté avec le sérum d’un patient atteint d’hépatite non-A non-B (Choo et al., 1989). Il s’agit d’un virus enveloppé, de petite taille, environ 55 nm. Sa capside est isocaédrique. Il possède un génome composé d’un ARN simple brin de polarité positive. L’étude des séquences virales a permis de le classer dans la famille des Flaviviridae, qui comporte 3 genres : Flavivirus, Pestivirus et Hepacivirus. Le HCV est le seul représentant du genre Hepacivirus. Il n’existe qu’un modèle animal d’infection par le HCV, le chimpanzé. Il a fallu attendre 16 ans pour disposer d’un système permettant la propagation du virus et la production de particules virales in
vitro. L’identification d’une souche particulière qui, pour des raisons encore
inconnues, se répliquait à un niveau élevé dans des lignées hépatocytaires humaines a constitué une avancée décisive (Lindenbach et al., 2005; Wakita et al., 2005; Zhong et al., 2005). Ce nouveau système permet à présent l’étude du cycle cellulaire du HCV.
L’infection par le HCV est un problème majeur de santé publique. Selon les estimations de l’Organisation Mondiale de la Santé, 170 millions de personnes sont infectées par HCV (Anonymous, 1999; Lauer & Walker, 2001). La primo-infection se manifeste par une hépatite aiguë souvent asymptomatique. Le passage à la chronicité est fréquent et survient dans environ 70% des cas. Cette persistance du virus conduit le plus souvent à une hépatite chronique active pouvant provoquer une cirrhose et un hépato-carcinome. Des manifestations extra-hépatiques sont également possibles. La cirrhose virale C au stade terminal est un motif principal de transplantation hépatique en Europe et en Amérique du Nord. Le traitement de
l’infection chronique repose actuellement sur l’association de l’interféron-alpha pégylé et de la ribavirine. Ce traitement reste inefficace chez environ 50 % des patients. En conséquence, on s’attend à une augmentation du nombre de patients pouvant présenter des complications, notamment un hépatocarcinome dans les 20 ans à venir (Williams, 2006).
La variabilité génétique du HCV est à l’origine de l’émergence et de la diversification des différents génotypes du virus au cours de l’évolution (Simmonds, 2004). Elle est à l’origine de la distribution en quasi-espèce du virus. Elle est également impliquée dans la physiopathologie de l’infection, aussi bien dans les mécanismes de persistance virale que dans la résistance aux molécules anti-virales. La grande variabilité de ce virus constitue un obstacle à la conception d’un vaccin. L’étude de la variabilité et de ses implications constitue un enjeu important dans la lutte contre le HCV.
I. LE VIRUS DE L’HEPATITE C
1. Le virus
1.1 Structure de la particule virale et du génome
Le HCV est un petit virus enveloppé de 55 à 65 nm de diamètre (Kaito et al., 1994; Shimizu et al., 1996). le HCV possède une capside icosaédrique avec deux glycoprotéines E1 et E2 ancrées dans l'enveloppe dérivée de la bicouche lipidique membranaire de la cellule hôte (Ishida et al., 2001).
Le HCV a été détecté sous différentes formes chez les individus infectés. En effet, il est présent sous forme libre ou associé avec des immunoglobulines ainsi qu’avec des lipoprotéines de haute, faible et très faible densité (Agnello et al., 1999; Hijikata et al., 1993b; Kanto et al., 1994). L’ultracentrifugation en gradient de sucrose a permis d’individualiser deux fractions riches en composants viraux : l’une de densité 1,09-1,10 g/ml, très infectieuse, constituée de lipoprotéines, l’autre de densité 1,22-1,25, faiblement infectieuse, constituée de particules virales associées à des immunoglobulines spécifiques au sein de complexes immuns. En outre, le HCV a été décrit circulant chez les patients sous forme de lipo-viro-particules de faible densité (LVP) (Andre et al., 2002). Ces LVP sont des particules sphériques d’une taille supérieure à 100 nm. Elles possèdent une structure proche des lipoprotéines, très riches en triglycérides contenant des nucléocapsides à l’intérieur et les glycoprotéines d’enveloppe à leur surface (Diaz et al., 2006).
La comparaison des séquences du génome du HCV avec les séquences virales connues a permis de le classer dans la famille des Flaviviridae. Cette famille comporte trois genres : le genre Hepacivirus auquel appartient le HCV, le genre
Pestivirus comprenant des virus responsables d’infections chez l’animal comme le
virus de la peste porcine classique (classical swine fever virus, CSFV) et le virus de la diarrhée virale bovine (bovine viral diarrhea virus, BVDV) et le genre Flavivirus comportant de nombreux virus comme le virus de la dengue, le virus de la fièvre jaune ou le virus de l’encéphalite japonaise. Les GB virus A, B et C appartenant à la famille des Flaviviridae n’ont été classés dans aucun de ces 3 genres.
Le génome viral et les différentes protéines virales ont été caractérisés en détail (Penin et al., 2004). L’organisation du génome est proche de celle des
Pestivirus. Le génome est constitué d'un ARN simple brin de polarité positive
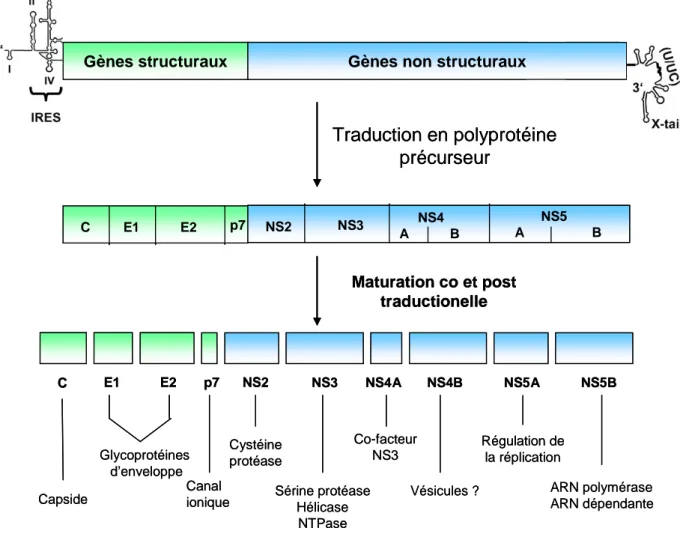
d’environ 9800 nucléotides. Il contient un seul cadre de lecture codant pour une polyprotéine d'environ 3000 acides aminés (Figure1). Le cadre de lecture est encadré par deux régions non codantes. Ces deux régions sont très conservées et la structure de l'ARN est essentielle à la traduction de la polyprotéine et la réplication de l'ARN.
Figure 1 : Diagramme schématique de l’organisation du génome viral
La région en 5' non codante d’environ 341 nucléotides comporte 4 domaines en structure tige boucle numérotés I à IV (Honda et al., 1996). Les domaines II, III et IV constituent le site d’entrée pour la sous-unité 40S du ribosome (IRES, internal ribosomal entry site) et initient la traduction de la polyprotéine. Le codon d'initiation est situé au niveau du domaine IV.
Le cadre ouvert de lecture situé en aval de la région 5’ non codante, comporte 9029 à 9111 nucléotides selon les génotypes. Sa traduction dans les cellules infectées produit une polyprotéine précurseur qui sera clivée pour donner naissance
Gènes structuraux Gènes non structuraux
Traduction en polyprotéine
précurseur
C E1 E2 p7 NS2 NS3 NS4 A B NS5 A B Capside E1 E2 p7 NS2 NS3 NS4A NS4B NS5A NS5B Maturation co et post traductionelle C Glycoprotéines d’enveloppe Canal ionique Cystéine protéase Sérine protéase Hélicase NTPase Co-facteur NS3 Vésicules ? Régulation de la réplication ARN polymérase ARN dépendanteGènes structuraux Gènes non structuraux
Traduction en polyprotéine
précurseur
C E1 E2 p7 NS2 NS3 NS4 A B NS5 A B Capside E1 E2 p7 NS2 NS3 NS4A NS4B NS5A NS5B Maturation co et post traductionelle C Glycoprotéines d’enveloppe Canal ionique Cystéine protéase Sérine protéase Hélicase NTPase Co-facteur NS3 Vésicules ? Régulation de la réplication ARN polymérase ARN dépendanteà dix protéines virales (Grakoui et al., 1993) : les protéines structurales (protéine de capside, E1, E2 et p7) et les protéines non structurales NS2 à NS5B.
La région en 3' comporte environ 230 nucléotides et est constituée de trois domaines. Tout d’abord on trouve une séquence courte d’environ 40 nucléotides très variables, suivie d’une séquence poly(U/UC) de longueur variable, puis d'une séquence très conservée de 98 nucléotides avec une structure en tige boucle appelée région X ou « X-tail » (Kolykhalov et al., 1996; Tanaka et al., 1996). Cette région X et les 52 nucléotides situés en 3’ de la séquence poly(U/UC) jouent un rôle important dans l'initiation de la synthèse du brin négatif au cours de la réplication du génome viral (Friebe & Bartenschlager, 2002; Song et al., 2006; Yanagi et al., 1999).
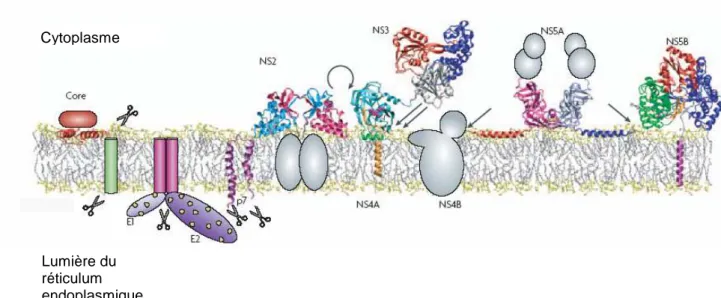
1.2 Les protéines virales
Figure 2 : Structure et association aux membranes des protéines du HCV (d’après Moradpour et al. Nature Reviews, 2007). Les ciseaux indiquent le clivage par les signal peptidases cellulaires, hormis du coté cytoplasmique où le clivage indiqué par les ciseaux se fait avec une signal peptide peptidase. La flèche arrondie indique le clivage par la protéase NS2-NS3. Les flèches droites indiquent le clivage par NS3-NS4A. La structure est représentée à la même échelle que la membrane. Les sphères représentent les domaines protéiques selon leur taille approximative.
La protéine de capside C
La protéine C est clivée à partir de la polyprotéine par une signal peptidase produisant la forme immature de 191 acides aminés. Ensuite le peptide signal présent à la partie C-terminale est clivé par une signal peptide peptidase pour donner naissance à la forme mature de 21 kDa, constituée de 173-179 acides aminés (McLauchlan et al., 2002). La protéine de capside est une protéine en hélice alpha. Cette protéine comporte trois domaines :
Cytoplasme
Lumière du réticulum endoplasmique
- le domaine N-terminal D1 constitué des 117 premiers acides aminés et qui est hydrophile,
- le domaine D2, hydrophobe, constitué de 169 acides aminés
- le domainne D3, hautement hydrophobe, constitué des 20 acides aminés C-terminaux et qui sert de de peptide signal de la protéine E1 dont la plus grande partie est clivée par la signal peptide peptidase (McLauchlan et al., 2002).
Le domaine D1 est riche en acides aminés basiques et semble impliqué dans la liaison avec l’ARN viral et l’oligomérisation. La polymérisation de la protéine de capside constitue la nucléocapside du virus. Les acides aminés de la protéine de capside impliqués dans l’assemblage des virions ont été identifiés récemment (Murray et al., 2007). Le domaine D2 est nécessaire à la conformation de D1 et à la stabilité de la protéine de capside. Ce domaine est absent des Flavivirus et des
Pestivirus mais présent chez le GB virus B (Boulant et al., 2006). La protéine de
capside interfère avec le métabolisme lipidique (Lai & Ware, 2000; McLauchlan, 2000). La partie C-terminale hydrophobe s'associe aux gouttelettes lipidiques dans les cellules de mammifères (Hope & McLauchlan, 2000). Il a été montré que le trafic entre le réticulum endoplasmique où la protéine est synthétisée et les gouttelettes lipidiques est régulé par le clivage du peptide signal. Ce phénomène est très rapide dans une cellule infectée (McLauchlan et al., 2002). La protéine de capside peut interagir avec de nombreuses protéines cellulaires et ainsi pourrait moduler la signalisation cellulaire, la transcription de gènes, la prolifération et la mort cellulaire (McLauchlan, 2000; Tellinghuisen & Rice, 2002).
La protéine F ou ARF (alternative reading frame)
La présence d’une protéine de poids moléculaire de 16 kDa a été montrée et assimilée à une forme tronquée de la protéine de capside. Plus récemment, cette protéine de 16 kDa a été caractérisée comme étant une protéine à part entière du HCV, protéine nommée F pour frameshift. Elle est issue d’un décalage ribosomique du cadre de lecture de type + 1 dans la région N-terminale de la séquence codant la protéine de capside. (Walewski et al., 2001). La détection d'anticorps anti-F chez les
patients infectés suggère que celle-ci est produite pendant l'infection (Bain et al., 2004). D'autres événements de décalage ribosomique ont été décrits (Choi et al., 2003) (Boulant et al., 2003). Leur fonction dans la physiopathologie ou le cycle réplicatif reste à élucider.
Les glycoprotéines d'enveloppe E1 et E2
Les protéines E1 et E2 sont des protéines transmembranaires comportant un ectodomaine de 160 et 334 acides aminés respectivement, et un court domaine C-terminal transmembranaire d'environ 30 acides aminés. Elles sont fortement N-glycosylées. Ces 2 protéines s'associent de façon non covalente pour former un hétérodimère par leur domaine transmembranaire (Deleersnyder et al., 1997; Op De Beeck et al., 2004).
Plusieurs régions hypervariables ont été identifiées dans la séquence de E2 (Weiner et al., 1992). Ces régions diffèrent jusqu’à 80% d’un génotype à l’autre. Les 27 premiers acides aminés de l'ectodomaine de E2 forment la région hypervariable 1 (HVR1) pour laquelle ont été identifiés des anticorps neutralisants dirigés contre le HCV (Farci et al., 1994). Un clone HCV déficient en région HVR1 est infectieux chez le chimpanzé mais de façon très atténuée, renforçant le rôle fonctionnel de ce
domaine dans l'entrée du virus dans les cellules cibles (Forns et al., 2000). Malgré l'importante variabilité de séquence de HVR1, les propriétés physico-chimiques des résidus à chaque position et la conformation générale de HVR1 sont conservées parmi tous les génotypes (Cocquerel et al., 2000). Deux autres régions hypervariables, HVR2 et HVR3, ont été identifiées dans la glycoprotéine E2 de génotype 1. La région HVR2 est constituée de 7 acides aminés consécutifs (position 91-97) présentant jusqu'à 100% de diversité (Kato et al., 1992). La région HVR3 est constituée de 35 acides aminés (position 431-466) et semble jouer un rôle dans la reconnaissance du récepteur et l’entrée cellulaire du virus (Troesch et al., 2006). Les deux glycoprotéines d'enveloppe ont un rôle essentiel dans la fixation et l’entrée du virus dans la cellule cible par liaison au récepteur et dans le mécanisme de fusion avec les membranes cellulaires (Bartosch et al., 2003a).
La protéine p7
La protéine p7 est une petite protéine de membrane de 63 acides aminés. Sa fonction est mal connue. Elle pourrait appartenir à la famille des viroporines (Pavlovic
et al., 2003). Elle se localise au niveau des membranes du réticulum endoplasmique
(Carrere-Kremer et al., 2002). p7 serait indispensable à l’infectiosité du virus et les séquences importantes d’un point de vue fonctionnel sont spécifiques de génotype (Sakai et al., 2003). Ses propriétés de canal ionique font de p7 une cible potentielle de nouvelles drogues antivirales.
La protéine NS2
La protéine NS2 a pour principale fonction le clivage de la polyprotéine à la jonction NS2/NS3. NS2 n’est pas essentielle à la formation du complexe de
réplication (Lindenbach et al., 2005). La structure tridimensionnelle de la protéase NS2-NS3 a récemment montré une structure de type cystéine protéase (Lorenz et
al., 2006). La protéase NS2-NS3 perd son activité après son « auto-clivage » de la
partie NS3 (Franck et al., 2005). Cette protéase est dépendante du zinc et s’insère dans la membrane du réticulum endoplasmique. Son activité peut être inhibée par l’EDTA et stimulée par le zinc (Hijikata et al., 1993a; Pieroni et al., 1997). NS2 pourrait interagir avec la protéine pro-apoptotique spécifique du foie CIDE-B (cell death inducing DFF45-like effector) et inhiber l’apoptose induite par CIDE-B (Erdtmann et al., 2003). Ceci pourrait jouer un rôle dans la persistance virale. Il a été montré récemment que NS2 et p7 ont un rôle dans l’assemblage et la morphogénèse du virus à un stade précoce (Jones et al., 2007).
Les protéines NS3 et NS4A
NS3 est une protéine de 67 kDa qui possède un domaine protéase au niveau des 189 acides aminés N-terminaux et un domaine NTPase/hélicase au niveau des 442 acides aminés C-terminaux.
La fonction sérine protéase de NS3 dépend de la formation d’un hétérodimère stable avec la protéine NS4A, protéine transmembranaire de 54 acides aminés (Failla et al., 1994). Ceci permet d’ancrer la protéase NS3A/4A au niveau des membranes du réticulum endoplasmique. NS4A intervient dans le repliement de NS3 et plus spécifiquement dans la conformation de la partie N-terminale (Wolk et al., 2000). NS4A est également impliquée dans la protection de la protéase contre les dégradations protéolytiques, et permet une activation de l’activité protéase en changeant la conformation du site catalytique. Le complexe NS3/4A assure le clivage de la polyprotéine au niveau des jonctions NS3/NS4A, NS4A/NS4B, NS4B/NS5A et
NS5A/NS5B (Neddermann et al., 1997). La structure tridimensionnelle de NS3 seule ou complexée avec NS4A a été identifiée : elle présente une conformation de chymotrypsine, elle ne possède pas de domaine transmembranaire et sa fonction nécessite aussi la présence de zinc (Kim et al., 1996; Love et al., 1996). L’activité sérine protéase est la cible de nouveaux antiviraux (Perni et al., 2006).
La partie C-terminale de NS3 code pour une hélicase de la superfamille des DexH/D-box hélicases (Tai et al., 1996). Les enzymes de cette famille sont capables de séparer les brins positifs et négatifs de l’ARN lors de la réplication et abolir les structures secondaires de l’ARN afin de permettre l’accès de l’ARN polymérase aux structures très repliées de l’ARN telle que les régions 5’ et 3’ non codantes. L’activité hélicase de NS3 se fait sous forme de dimère (Serebrov & Pyle, 2004). L’activité NTPase qui s’exerce principalement sur l’ATP et le dATP est dépendante du Mg2+ (Frick et al., 2007). Des inhibiteurs de cette fonction ont été étudiés (Boguszewska-Chachulska et al., 2006).
La protéine NS4B
NS4B est une petite protéine hydrophobe de 27kDa, localisée dans la paroi du réticulum endoplasmique et orientée vers le cytoplasme (Hugle et al., 2001). Son expression modifierait les membranes dérivées du réticulum endoplasmique afin de faciliter la formation du complexe de réplication qui associe les protéines structurales et non structurales du HCV (Egger et al., 2002; Gosert et al., 2003). NS4B peut être palmitoylée, et cette modification facilite son oligomérisation et est indispensable à la réplication du HCV (Yu et al., 2006). Différents variants de NS4B peuvent influencer les niveaux de réplication in vitro (Blight, 2007).
La protéine NS5A
NS5A est une protéine relativement hydrophile qui existe sous deux formes : une forme peu phosphorylée de 56 kDa et une forme hyperphosphorylée de 58 kDa. La forme hyperphosphorylée semble associée à des niveaux de réplication plus faible in vitro (Appel et al., 2005).
Seuls deux des trois domaines de NS5A ont été caractérisés sur le plan structural (Liang et al., 2006; Tellinghuisen et al., 2005). Une hélice alpha dans le domaine I permet son ancrage dans la membrane du réticulum endoplasmique. Il a été démontré que NS5A joue un rôle important dans la régulation de la réplication virale en coordonnant l’interaction des protéines non structurales virales et des protéines cellulaires telle que la protéine hVAP-A (human vesicule-associated membrane protein-associated protein A) (Evans et al., 2004; Gao et al., 2004). Cette protéine, localisée dans le réticulum endoplasmique et le Golgi est impliquée dans le trafic intracellulaire de vésicules et interagit à la fois avec NS5A et NS5B (Hamamoto
et al., 2005). NS5A et NS5B se lient également à hVAP-B, dont la déplétion conduit à
une diminution de l’expression de NS5B et de la production d’ARN viral (Hamamoto
et al. 2005).
NS5A influence de nombreux événements cellulaires liés à la persistance et à la pathogénèse du HCV. Ainsi, l’expression de NS5A pourrait altérer les niveaux de calcium intracellulaire, induire un stress oxydatif, et activer STAT-3 et NF-κB (Gong
et al., 2001). NS5A pourrait inhiber les propriétés mitogéniques du facteur de
transcription AP1 (activating protein-1) (Macdonald et al., 2003). NS5A est capable d’inhiber l’apoptose par de multiples mécanismes (Lan et al., 2002; Miyasaka et al., 2003; Nanda et al., 2006; Wang et al., 2006). La protéine NS5A a été particulièrement étudiée en raison de son rôle potentiel dans la réponse au
traitement par l’interféron-alpha (IFN-α). Une étude japonaise a suggéré l’existence d’une région particulière dans NS5A nommée interferon sensitivity determining
region (ISDR) (Enomoto et al., 1995; Enomoto et al., 1996). NS5A pourrait interférer
avec l’activité de la protéine kinase R induite par l’IFN-α in vitro (Gale et al., 1998). Cependant, ces résultats sont très controversés (Boulestin et al., 2003; Duverlie et
al., 1998).
La protéine NS5B
NS5B est une protéine de 68 kDa contenant des motifs caractéristiques des ARN polymérases ARN dépendante. Elle a été étudiée sur le plan biochimique et structural afin de développer des antiviraux (Behrens et al., 1996; Bressanelli et al., 2002; Lesburg et al., 1999). Elle présente une structure 3D en « main droite », avec des doigts, un « pouce » et une « paume ». Le site actif de NS5B a la particularité d’être totalement entouré. Les doigts et le pouce interagissent en de multiples endroits et créent un tunnel dans lequel une molécule d’ARN simple brin est guidée. Un autre tunnel, chargé positivement, permet l’entrée des NTPs et leur passage vers le site actif (Lesburg et al., 1999).
2. Cycle réplicatif
2.1 Modèles d’étude du cycle viral
Le cycle réplicatif du HCV est encore imparfaitement connu. Il a fallu attendre 16 ans après la découverte du HCV pour disposer d’un système permettant la propagation du virus et la formation de particules virales in vitro. Ceci est devenu possible avec l’identification d’une souche particulière, qui pour des raisons encore inconnues, se répliquait à un fort niveau dans des lignées hépatocytaires humaines.
L’incapacité à reproduire le cycle complet du HCV en culture a conduit au développement d’autres procédés artificiels. L’étude des glycoprotéines d’enveloppe a été difficile du fait de leur fort ancrage dans la membrane du réticulum endoplasmique. Des formes solubles de E2 (sE2) ont été produites pour étudier les mécanismes de l’interaction virus-cellule (Michalak et al., 1997). Le domaine transmembranaire d’E2 a été délété, conduisant à la production de formes sécrétées d’E2. L’utilisation des sE2 a permis l’identification des récepteurs d’attachement CD81 et le scavenger Recepteur B de type I.
Des “virus-like” particules ont été produites dans des cellules d’insecte infectées par un baculovirus recombinant codant les protéines structurales du HCV (capside, E1 et E2) et une partie de l’IRES (Baumert et al., 1998). Les protéines produites s’auto-assemblent en particules mais ne peuvent pas se répliquer. Elles sont un modèle de choix pour les études d’attachement. Leurs caractéristiques en font un candidat vaccin potentiel (Baumert et al., 1999; Elmowalid et al., 2007).
Un autre modèle utilise, pour étudier la fixation et l’entrée du virus, un virus de la stomatite vésiculeuse pseudotypé. Ce système est basé sur l’expression des
glycoprotéines d’enveloppe du HCV, E1 et E2, fusionées au domaine transmembranaire de la glycoprotéine G du virus de la stomatite vésiculeuse (Lagging et al., 1998).
Un autre modèle innovant a utilisé des particules retrovirales pseudotypées avec les glycoprotéines E1 et E2 non modifiées du HCV. Cette construction est appelée HCV pseudo-particles (HCVpp) (Bartosch et al., 2003a). Les HCVpp sont produites par co-transfection dans les cellules 293T de trois vecteurs plasmidiques. Le premier exprime E1 et E2, le second les protéines gag et pol rétrovirales, le troisième porte le signal d’encapsidation nécessaire à la formation des particules en amont d’un gène rapporteur (la protéine verte fluorescente, GFP, ou la luciférase). Les cellules 293T sécrètent des particules virales correctement assemblées dans le milieu extracellulaire. Ces HCVpp permettent d’étudier toutes les fonctions virales dépendantes des glycoprotéines. Elles sont neutralisées par les anticorps anti-E2, du sérum humain ou de chimpanzé infecté (Bartosch et al., 2003b; Hsu et al., 2003; Owsianka et al., 2005).
Par analogie aux travaux réalisés sur le poliovirus, des constructions subgénomiques composées seulement des gènes non structuraux du HCV ont été réalisées et leur transfection dans la lignée d’hépatome humain Huh-7 a permis une réplication autonome. La construction prototype de ce système bicistronique contenait les deux régions 5’ et 3’ non codante du HCV, le gène de résistance à la néomycine en remplacement de la région codant pour les protéines structurales et NS2. La région codant pour les autres protéines non structurales était placée sous la dépendance d’une seconde séquence IRES : celle du virus de l’encéphalomyocardite (EMCV) (Lohmann et al., 1999) (Figure 3).
Figure 3 : Diagramme schématique des réplicons subgénomique (A) et génomique (B)
Le premier réplicon subgénomique fut mis au point avec un génotype 1b, la souche Con1. D’autres travaux ont confirmé ces résultats et ont montré qu’un niveau élevé de réplication était lié à des mutations adaptatives localisées dans les régions NS3 et NS5A (Blight et al., 2000). Des clones cellulaires hépatocytaires Huh-7.5, traités par IFN-α afin d’éliminer le HCV, se sont avérés par la suite plus susceptibles à la réplication d’un nouveau réplicon (Blight et al., 2002). Ces éléments ont permis la construction de réplicons du HCV comportant le génome entier, mais aucun réplicon génomique ne permettait la production de particules virales infectieuses. De plus, il a été montré que les mutations adaptatives favorisant la multiplication in vitro étaient en fait délétères pour l’infectiosité in vivo chez le chimpanzé (Bukh et al., 2002).
L’année 2005 aura été déterminante pour la mise au point d’un système cellulaire permettant de reproduire un cycle infectieux complet du HCV in vitro. Une étape essentielle fut la construction d’un clone consensus de génotype 2a, isolé d’un patient japonais présentant une hépatite fulminante, forme clinique exceptionnelle dans les hépatites C. Ce clone fut appelé JFH1. Un réplicon subgénomique de JFH1 présentait des niveaux de réplication plus élevés que les précédents, sans mutation
A
B A
adaptative (Kato et al., 2003). Point crucial, la transfection du génome complet de JFH1 dans les cellules Huh-7 a permis la production de particules virales infectieuses dont l’infectiosité a été démontrée à la fois sur des lignées hépatocytaires et chez le chimpanzé (Wakita et al., 2005). Les titres du virus JFH1 ont été accrus en utilisant les cellules 7.5 (Zhong et al., 2005). Les cellules Huh-7.5 semblent constituer le système le plus favorable à la production virale in vitro. Ces cellules présentent une mutation dans le gène RIG-I (retinoid inducible gene I), conduisant à l’inactivation des réponses immunes innées antivirales (Foy et al., 2005). La culture d’un virus de génotype 1a a pu être réalisée ; mais la production de virus infectieux était inférieure à celle de la souche JFH1 (Yi et al., 2006).
La production de particules virales infectieuses à partir d’un génome HCV chimère résultant du remplacement des gènes C, E1, E2 et NS2 de JFH1 par la séquence correspondante du virus J6 (génotype 2a) a été également décrite (Lindenbach et al., 2005). Ce virus chimère est infectieux chez le chimpanzé et après passage chez cet animal le virus reste infectieux en culture cellulaire (Tscherne et
al., 2006). De plus, la réplication est inhibée par l’IFN-α et les titres viraux obtenus avec ce virus chimère sont plus élevés qu’avec JFH1. Les gènes structuraux de JFH1 semblent donc présenter une plus faible capacité intrinsèque d’assemblage et/ou de libération du virus. Cette hypothèse est étayée par une étude récente où la production en culture de virus chimères intergénotypiques et intragénotypiques a été comparée à celle de JFH1 (Pietschmann et al., 2006). Les titres les plus élevés ont été obtenus avec le recombinant J6-JFH1.
Cependant, malgré tout l’intérêt de la culture conventionnelle pour l’analyse détaillée de la biologie du HCV, les études concernent principalement le génotype 2a ou des constructions dérivées de ce génotype. Le développement de génomes
chimériques, comportant les éléments structuraux et non structuraux de l’ensemble des génotypes, reste un enjeu essentiel.
2.2 Le cycle réplicatif
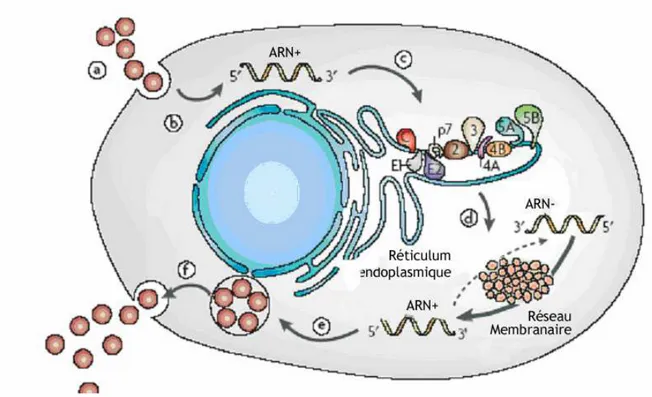
Figure 4 : Le cycle réplicatif du HCV d’après Moradpour et al. 2007 : liaison et internalisation (a), décapsidation dans le cytoplasme (b), traduction de la polyprotéine (c), réplication du génome (d), assemblage (e) et libération des virions (f)
Le cycle cellulaire du HCV débute par l’interaction des glycoprotéines d’enveloppe avec les récepteurs au niveau de la membrane plasmique. Cette interaction avec les récepteurs cellulaires se fait grâce au dimère E1-E2. Plusieurs récepteurs potentiels ont été identifiés :
- la tétraspanine CD81 : elle est exprimée de façon ubiquiste. Le rôle de CD81 dans l’attachement du virus a été montré, dès 1998 (Pileri et al., 1998). Des lignées hépatocytaires HepG2, qui n’expriment pas de CD81, deviennent sensibles à l’infection par transfection de CD81 (Lavillette et al., 2005; Wakita et al., 2005). Ceci n’est pas observé par l’expression de CD81 humain dans des cellules d’autres espèces telles que CHO (Bartosch et al., 2003b). Le CD81 est donc nécessaire mais pas suffisant pour induire l’entré du HCV.
- le récepteur scavenger de classe B et de type I (SR-BI) : il interagit avec la région HVR1 de E2 (Bartosch et al., 2003b). Ce récepteur est notamment responsable de l’assimilation sélective des esters de cholestérol à partir des lipoprotéines de forte densité. La présence de HDL dans le milieu extérieur augmente l’infectivité. Le mécanisme sous-jacent implique le récepteur SR-BI et la région HVR1 de E2 (Bartosch et al., 2005; Meunier et al., 2005). Toutefois, des anticorps dirigés contre SR-BI ne bloquent que partiellement l’entrée du virus suggérant que SR-BI n’est pas le seul récepteur impliqué dans l’attachement du HCV (Barth et al., 2005).
- le récepteur des lipoprotéines de faible densité (LDL) : l’association des particules virales avec des LDL ou des VLDL leur permet d’interagir avec ce récepteur suivi d’une internalisation des virions (Agnello et al., 1999).
- les lectines L-SIGN (Liver/Lymph node specific intracellular adésion molecule-3-grabbing non integrin) ou CD209L et DC-SIGN (Dendritic cell specific intracellular adésion molecule-3-grabbing non integrin) ou CD209 : la glycoprotéine E2 se lie à ces deux récepteurs (Lozach et al., 2003). L-SIGN est exprimée sur les cellules épithéliales des sinusoïdes du foie et DC-SIGN sur les cellules de Küpffer mais pas au niveau des hépatocytes. Ceci suggère que la fixation à ces récepteurs contribue
in vivo à la capture et à la transmission du HCV dans les cellules hépatiques
(Cormier et al., 2004).
- une autre lectine exprimée sur le parenchyme hépatique a été décrit : le récepteur des asialoglycoprotéines (Saunier et al., 2003)
- les glycosaminoglycanes : la protéine E2 se lie aux héparanes sulfates avec une affinité élevée. Ces molécules ont une distribution ubiquiste à la surface des cellules, elles pourraient servir d’attachement à la cellule avant un « transfert » vers un autre recepteur.
- un autre candidat récepteur a été décrit récemment, il s’agit de la claudine-1 (Evans
et al., 2007). Cette protéine impliquée dans les jonctions serrées cellulaires est
fortement exprimée dans le foie (Tsukita & Furuse, 1998). L’utilisation d’anticorps anti-claudine-1 suggère que la claudine-1 agit à un niveau tardif du processus d’entrée, après l’attachement du virus et l’interaction avec un corécepteur tel que le CD81.
- d’autres molécules de la famille des claudines, claudine-9 et 6 sont aussi impliquées dans l’entrée du HCV. Elles sont exprimées au niveau hépatique mais aussi chez des cellules ne comportant pas claudine-1 à leur surface membranaire telles que les cellules mononucléées du sang périphérique (Zheng et al., 2007).
La fixation et l’internalisation des virions mettent probablement en jeu plusieurs molécules. Chaque candidat récepteur serait impliqué ; le rôle de chacun reste à définir et d’autres constituants restent probablement à identifier (Barth et al., 2006). La compréhension de ce mécanisme pourrait permettre le développement de nouvelles molécules bloquant l’entrée du virus comme cela a été réalisé pour le virus de l’immunodéficience humaine. Une application intéressante serait la prévention de la réinfection du greffon après transplantation hépatique.
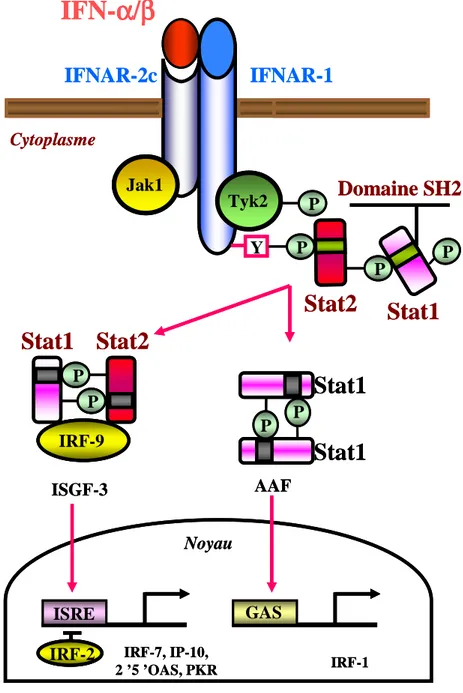
L’étape de fusion des membranes cellulaires avec l’enveloppe du virus se déroule après endocytose par une voie clathrine dépendante comme cela a été montré pour les Flaviviridae (Heinz & Allison, 2000; Hsu et al., 2003). La protéine E1 semble impliquée dans le phénomène de fusion du virus avec les membranes cellulaires (Flint & McKeating, 2000) mais les domaines transmembranaires des deux glycoprotéines semblent aussi impliqués dans cette étape (Ciczora et al., 2007). L’entrée est dépendante de l’acidification au niveau des endosomes tardifs (Tscherne et al., 2006). Après fusion, l’ARN viral est libéré de l’enveloppe et de la capside, puis il est relargué dans le cytoplasme. Comme pour tous les virus à ARN de polarité positive, le génome viral est l’élément central de la traduction, de la réplication et de l’assemblage (Figure 4). La traduction du génome est sous le contrôle de l’IRES (Honda et al., 1996). L’initiation de la synthèse de la polyprotéine débute lors de la formation du complexe entre l’IRES et la sous-unité 40S du ribosome et le recrutement des protéines cellulaires, comme les facteurs d’initiation eIF-2 et eIF-3 et des protéines virales.
La traduction puis le clivage de la polyprotéine a lieu au niveau de structures membranaires et vésiculaires péri-nucléaires. L’ARN polymérase ARN dépendante et les autres protéines non structurales s’associent à des protéines cellulaires de la cellule hôte pour former le complexe de réplication (Gosert et al., 2003; Ishido et al., 1998). Une séquence nucléotidique en tige-boucle très conservée au sein du gène de NS5B, nommée 5BSL3.2, s’apparie avec la région 3’ non codante et cette interaction intervient dans la régulation de la réplication (Friebe et al., 2005). De nombreux facteurs cellulaires affectent la réplication et l’infectiosité du HCV (Randall
et al., 2007). La réplication du génome est influencée par un microARN spécifique du
et est impliqué dans la réplication mais pas la traduction du génome. L’ARN polymérase synthétise un brin négatif à partir du génome, qui sert ensuite de matrice pour la synthèse de brins positifs. Ces brins sont ensuite encapsidés pour former de nouvelles particules virales ou servent de messagers pour la synthèse des protéines virales.
Les étapes ultimes du cycle réplicatif sont elles aussi mal connues. L’assemblage est probablement déclenché par l’intéraction entre la protéine de capside et l’ARN génomique dans sa région 5’ non codante, aboutissant à la formation de nucléocapsides (Shimoike et al., 1999). In vitro, les 124 acides aminés de la partie N-terminale de la protéine de capside suffisent pour former des particules de type nucléocapside (Kunkel et al., 2001). Par analogie avec les Flaviviridae, les nucléocapsides pourraient ensuite s’envelopper par bourgeonnement à l’intérieur du réticulum endoplasmique. Puis les particules virales néoformées seraient excrétées par exocytose. Dans le modèle HCVcc, la réplication d’un génome délété de la séquence de p7 est possible et permet la formation de particules mais elles sont retenues dans le cytoplasme de la cellule. De plus ces particules sont moins infectieuses, ce qui suggère que p7 est critique pour l’assemblage et le relargage des virions (Steinmann et al., 2007).
Un lien entre le cycle du HCV et le métabolisme lipidique a été démontré depuis plusieurs années. Des études d’expression de la protéine de capside dans les lignées cellulaires ont montré que cette protéine se localisait à la surface de gouttelettes lipidiques (Barba et al., 1997). Après clivage, la protéine de capside du HCV reste ancrée dans la membrane du réticulum endoplasmique ou s’associe aux gouttelettes lipidiques grâce à son domaine hydrophobe en position C-terminale
(Hourioux et al., 2007). L’expression de la protéine de capside du HCV dans les systèmes cellulaires in vitro induit une redistribution des gouttelettes lipidiques dans les zones péri-nucléaires. La signification de cette co-localisation n’est pas encore bien comprise. Le clivage de la protéine de capside semble donc requis pour la formation de la particule virale (Ait-Goughoulte et al., 2006). Des mutations dans le domaine D2 de la protéine de capside empêchent l’association aux goutelettes lipidiques et affectent la production de virus (Boulant et al., 2007). La protéine de capside semble recruter les protéines non structurales, l’ARN viral et le complexe de réplication au niveau des goutelettes lipidiques (Miyanari et al., 2007). Ces travaux ont montré que les particules virales s’assemblent et bourgeonnent à proximité des gouttelettes lipidiques. Ainsi les sites de formation de celles-ci pourraient constituer des micro-domaines favorables à l’assemblage des particules virales, en réunissant des facteurs viraux et/ou cellulaires nécessaires aux mécanismes de morphogenèse, ou en excluant les facteurs pouvant l’inhiber. La réplication est stimulée par les acides gras saturés ou mono-insaturés et inhibée par les acides gras poly-insaturés. Ces résultats suggèrent que la fluidité des membranes est importante au fonctionnement du complexe de réplication (Kapadia & Chisari, 2005). Le cycle infectieux pourrait être dépendant de la voie de synthèse des VLDL dans l’hépatocyte humain. Des protéines telles que la MTP (microsomal triglycérides transfert protein), l’apoprotéine B ou l’apoprotéine E interagissent avec les protéines virales au niveau des vésicules de réplication du HCV et l’inhibition par la technique des ARN interférents de ces protéines permet in vitro d’inhiber la production de virus (Chang et al., 2007; Huang et al., 2007).
II. VARIABILITE GENETIQUE DU HCV
La variabilité génétique est une caractéristique commune à tous les virus dont le génome est un ARN ou qui utilisent un ARN comme intermédiaire de réplication. Elle résulte de l’absence d’activité correctrice d’erreur des ARN polymérases virales. La variabilité génétique du HCV est à l’origine de l’émergence et de la diversification des différents génotypes au cours de l’évolution. Elle est également à l’origine de la distribution en quasi-espèce du virus. Elle a été impliquée dans la pathogénie de l’infection et dans la résistance aux molécules anti-virales.
1. Réplication et variabilité génétique
La variabilité du HCV résulte de l’évolution darwinienne du virus. La diversification permanente des populations virales conduit à la compétition entre les différents variants viraux et à la sélection des variants les mieux adaptés à leur environnement. Ce processus est favorisé par l’abondance de la production virale de l’ordre de 1012 virions par jours au cours de l’infection humaine (Neumann et al., 1998) et par le taux élevé de mutations lié au manque de fidélité de l’ARN polymérase virale (Domingo et al., 1998; Duarte et al., 1994). La fréquence des erreurs de la polymérase du HCV est d’environ 10-4 à 10-5 par nucléotide copié. Ces erreurs ne sont pas corrigées car l’ARN polymérase virale est dépourvue d’activité exonucléase 3’-5’. Les mutations s’accumulent donc sur le génome au cours des cycles de réplications successifs. La plupart des mutations étant létales, la majorité des particules virales produites sont défectives. En revanche les mutations
non létales sont transmises à la descendance et s’accumulent au cours du temps. La production et la sélection permanente de nouveaux variants viraux sont à l’origine de l’émergence des génotypes du HCV et de leur diversification au cours de l’évolution (Simmonds, 2001).
2. Les quasi-espèces
Le HCV, comme de nombreux virus à ARN circule chez son hôte, sous la forme d’une quasi-espèce virale, c’est à dire d’un mélange complexe et en équilibre instable de variants viraux génétiquement distincts mais apparentés (Martell et al., 1992). La distribution en quasi-espèces du virus favorise sa survie. En effet la présence simultanée de variants viraux différents et la rapidité avec laquelle de nouveaux variants émergent permettent la sélection rapide et continue des variants les mieux adaptés à l’environnement au sein duquel le virus se réplique (Domingo et
al., 1998; Duarte et al., 1994). La capacité d’adaptation des quasi-espèces virales
aux modifications de l’environnement joue un rôle important dans la physiopathologie de l’infection, aussi bien dans les mécanismes de persistance virale que dans la résistance aux traitements antiviraux ou à la récidive de l’infection après transplantation. Les pressions de sélection entrant en jeu dans ce processus d’adaptation sont de deux ordres :
- les pressions négatives, liées aux contraintes conservatrices sur la séquence du génome ou des protéines virales, résultent de la nécessité de conserver les propriétés fonctionnelles du génome et des protéines pour assurer la survie des variants
- les pressions de sélection positives exercées sur le génome et les protéines virales résultent d’interactions complexes avec les réponses immunes et certaines protéines de l’hôte. Elles évoluent au cours du temps spontanément ou sous l’influence de divers événements extérieurs (infection intercurrente, administration d’antiviraux,…).
3. Les génotypes du HCV
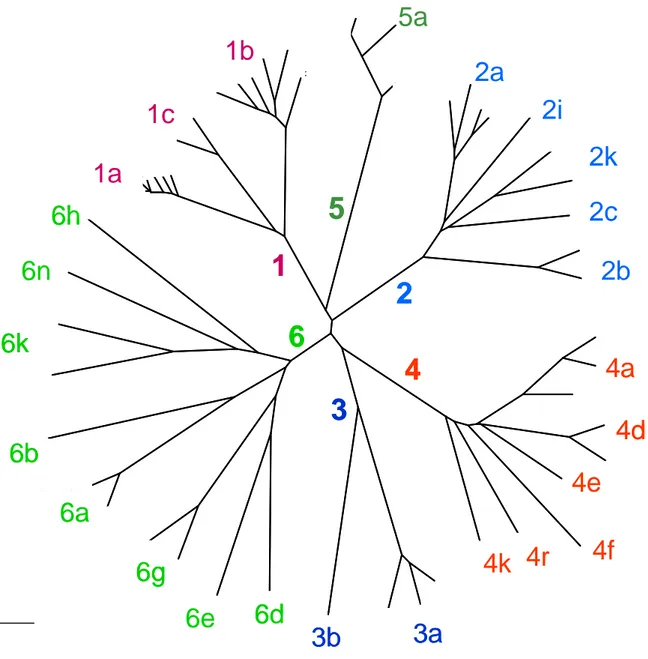
La variabilité du HCV existe à différents niveaux. La comparaison des séquences nucléotidiques de souches virales provenant de différentes régions du monde a permis de classer ces souches dans 6 génotypes, numérotés de 1 à 6. Au sein de chaque génotype, on distingue plusieurs dizaines de sous-types identifiés par une lettre minuscule a, b, c etc (Simmonds et al., 2005) (Figure 4).
En se basant sur le génome complet, le degré de divergence des séquences des différents génotypes est de 30 à 35 %. Les séquences nucléotidiques des sous-types diffèrent les unes des autres d’environ 20 à 25 %. La région la moins variable entre les génotypes est la région 5’ non codante, jouant un rôle essentiel dans la réplication et la traduction.
Figure 5 : Arbre phylogénétique représentant les génotypes du HCV basé sur l’analyse des séquences de la région NS5B.
L’analyse phylogénétique de la séquence du génome viral dans son intégralité est dans l’absolu la technique de référence pour déterminer le génotype et le sous-type. Compte tenu des contraintes techniques et du coût d’une telle approche, ceci
4a-tls1 4a-tls2 4a-Y11604 4d-tls3 4d-tls4 4e M600 4f-tls7 4r-tls5 4k-tls6 3a-D17763 3a-X76918 3a-AF046866 3b-D49374 6d-D84263 6e-DQ314805 6g-D63822 6g-DQ314806 6a-Y12083 6a-AY859526 6b-D84262 6k-D84264 6k-DQ278893 6n-DQ278894 6h-D84265 1a-AF011751 1a-M67463 1a-AX663428 1a-AF271632-NS5B-GC1a-AF5119481a-D10749
1c-AY051292 1c-D14853 1b-AB016785 1b-E05027 1b-AF054247 1b-D63857 1b-D85516 1b-D90208 5a-Y13184 5a H148 5a 741 5a-AF064490 2a-AB047639 2a-AF238481 2a-AX057086 2a-D00944 2i-G2MP103 2k-G2MP017 2k-AB031663 2c-D50409 2b-AF238486 2b-D10988
1a
1c
1b
5a
2i
2a
2k
1
5
2c
2
2b
4a
4d
4
4e
4f
4r
4k
3a
6
3b
3
6d
6e
6g
6a
6b
6k
6n
6h
4a-tls1 4a-tls2 4a-Y11604 4d-tls3 4d-tls4 4e M600 4f-tls7 4r-tls5 4k-tls6 3a-D17763 3a-X76918 3a-AF046866 3b-D49374 6d-D84263 6e-DQ314805 6g-D63822 6g-DQ314806 6a-Y12083 6a-AY859526 6b-D84262 6k-D84264 6k-DQ278893 6n-DQ278894 6h-D84265 1a-AF011751 1a-M67463 1a-AX663428 1a-AF271632-NS5B-GC1a-AF5119481a-D107491c-AY051292 1c-D14853 1b-AB016785 1b-E05027 1b-AF054247 1b-D63857 1b-D85516 1b-D90208 5a-Y13184 5a H148 5a 741 5a-AF064490 2a-AB047639 2a-AF238481 2a-AX057086 2a-D00944 2i-G2MP103 2k-G2MP017 2k-AB031663 2c-D50409 2b-AF238486 2b-D10988
1a
1c
1b
5a
2i
2a
2k
1
5
2c
2
2b
4a
4d
4
4e
4f
4r
4k
3a
6
3b
3
6d
6e
6g
6a
6b
6k
6n
6h
n’est pas réalisable en pratique courante. Différentes techniques alternatives ont été développées reposant sur deux grands principes:
- le typage génomique : caractérisation du polymorphisme d’un fragment du génome
- le typage sérologique : caractérisation d’anticorps spécifiques des différents génotypes
Les tests moléculaires reposent sur l’analyse d’une région du génome viral. Quelque soit la technique utilisée, le choix de la région étudiée est primordial. En effet, la région choisie doit posséder des motifs génétiques spécifiques de type et de sous-type, représentatifs de la diversité du génome complet. Les régions 5’ non codante, capside et NS5B sont les trois régions les plus fréquemment analysées. Les techniques d’hybridation inverse consistent à hybrider les produits de PCR issus de l’amplification d’un fragment du génome sur des bandelettes de nitro-cellulose contenant des sondes d’ADN spécifiques du génotype. Les premiers tests commercialisés basés sur cette technique, analysaient uniquement la région 5’ non codante (Stuyver et al., 1993). Le polymorphisme de cette région permet une bonne discrimination des génotypes mais ne permet pas de distinguer l’ensemble des sous-types en raison de la conservation de la région 5’ non codante (Sandres-Saune et
al., 2003). Une nouvelle version du test basée sur l’analyse par hybridation inverse
des produits de PCR de la région 5’ non codante et de la région de la capside, permet une meilleure discrimination du sous-type (Bouchardeau et al., 2007). La technique de référence pour la détermination du génotype et du sous-type repose sur le séquençage de la région NS5B et la réalisation d’analyses phylogénétiques avec des génomes de référence (Laperche et al., 2006; Sandres-Saune et al., 2003).
4. Variabilité et épidémiologie
Les analyses phylogénétiques des génotypes du HCV ont permis de mieux comprendre l’émergence et la diversification des types et sous-types ainsi que leur distribution mondiale (Figure 5). Le HCV aurait co-évolué avec les populations humaines. L’apparition des génotypes serait plus récente (Smith et al., 1997) (Simmonds, 2001). Le génotype 6, le plus ancien, serait apparu il y a 700 ans, les génotypes 3 et 4, il y a 350 ans et le genotype 2 il y a environ 200 ans. Le génotype 1, le plus récent, serait apparu il y a environ 100 ans (Pybus et al., 2001; Simmonds, 2004).
La répartition géographique des différents génotypes est maintenant bien établie et reflète l’histoire épidémiologique du virus. En Afrique la diversité des génotypes 1, 2 et 4 est considérable (Simmonds, 2001). Ceci est le reflet d’une présence du virus dans les populations africaines depuis de très nombreuses années. Les génotypes 3 et 6 présentent la même diversité génétique en Asie du Sud ou de l’Est (Mellor et al., 1995; Tokita et al., 1994). Cette diversité suggère que le HCV a été endémique en Afrique sub-saharienne et en Asie pendant une période considérable, et que la survenue d’infections dans les pays occidentaux ou les autres pays non tropicaux est un événement relativement récent, lié à des pratiques exposant au virus (Ndjomou et al., 2003; Pybus et al., 2001; Simmonds, 2001). Le génotype 1 présente une large répartition à l’échelle mondiale. C’est l’un des génotypes les plus représenté dans les pays industrialisés. Les sous-types 1a et 1b sont responsables de 60 à 65 % des infections en Europe de l’ouest et infectent la majorité des malades aux Etats-Unis. Le génotype 2 est retrouvé en Europe et au Japon mais est plus rare au Etats-Unis (Cheung, 2000; Dusheiko & Simmonds,
prédominant en Afrique de l’Ouest (Candotti et al., 2003; Mellor et al., 1995; Ruggieri
et al., 1996; Wansbrough-Jones et al., 1998), alors qu’en Afrique centrale les
génotypes 1 et 4 sont prédominants (Bukh et al., 1993b; Mellor et al., 1995; Ndjomou
et al., 2003; Stuyver et al., 1993). L’origine du génotype 5 reste inconnue. Sa
présence était communément considérée comme restreinte à l’Afrique du sud, avec une prévalence qui peut atteindre 40% chez les personnes infectées par le HCV (Smuts & Kannemeyer, 1995). Une étude comparative des souches virales de génotype 5 infectant des sujets belges et sud-africains a montré l’existence de 2 groupes phylogénétiques différents, d’ancienneté identique (Verbeeck et al., 2006). Les techniques de coalescence basées sur le séquençage des régions E1 et NS4B ont permis d’estimer l’âge de l’ancêtre commun à 120 ans pour les deux groupes epidémiologiques. Cet ancêtre commun pourrait provenir d‘Afrique Centrale puisque la Belgique et l’Afrique du Sud n’ont pas eu de rapport direct dans leur histoire, mais ces deux pays ont eu de nombreux échanges au 19ème siècle avec des pays d’Afrique Centrale comme le Congo.
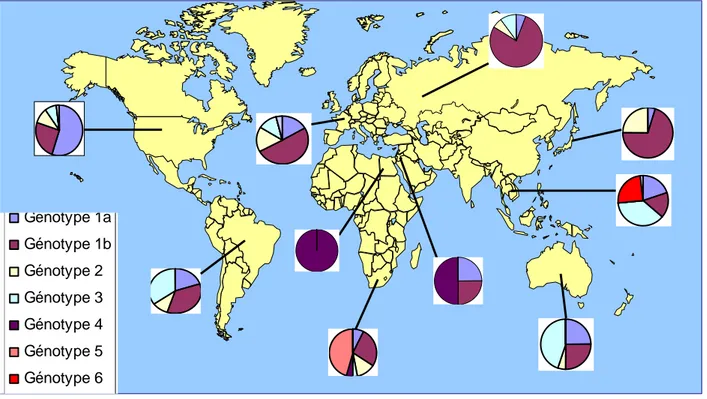
Figure 6 : Distribution géographique des génotypes d’après Zein (Zein, 2000)
La distribution des génotypes varie d'un pays à l'autre, mais les différentes caractéristiques épidémiologiques telles que l'âge, le sexe, et le mode de contamination influencent aussi la répartition des génotypes (voir revues de Zein, 2000 et de Shepard et al. 2005). En France en 1999, les génotypes 1b et 2 étaient plus fréquemment retrouvés chez les femmes que chez les hommes, et chez des patients agés de plus de 50 ans. Alors que les génotypes 1a et 3 étaient plus fréquents chez des patients masculins de moins de 50 ans (Martinot-Peignoux et al., 1999). Les génotypes 1b et 2 sont retrouvés plus fréquemment chez des patients présentant une histoire de transfusion de produits sanguins que chez des patients contaminés par un génotype 1a ou 3. Les études d'épidémiologie moléculaire ont montré une association entre le sous-type 3a et la contamination par toxicomanie intra-veineuse dans de nombreux pays sur le continent européen. Le génotype 3 est
Génotype 1a Génotype 1b Génotype 2 Génotype 3 Génotype 4 Génotype 5 Génotype 6 Génotype 1a Génotype 1b Génotype 2 Génotype 3 Génotype 4 Génotype 5 Génotype 6
plus rare en Afrique et au Japon car cette pratique n'est pas fréquente (Morice et al., 2006). L’Egypte est le pays ou la prévalence du HCV est la plus élevée dans le monde (15 à 20%). Le génotype 4a est responsable de la majorité des infections dans ce pays. Cette diffusion très importante du génotype 4a est liée à la politique de traitement de la shistosomiase par voie parentérale dans les années 1960-1980 (Frank et al., 2000). Le génotype 1a est aussi associé à une contamination par toxicomanie aux Etats Unis et en Europe. Récemment, la diffusion du génotype 4 en Europe a été rapportée dans différentes régions d’Europe (Morice et al., 2001; Sanchez-Quijano et al., 1997; Schröter et al., 2002; van Asten et al., 2004).
III. PHYSIOPATHOLOGIE
1. Histoire naturelle
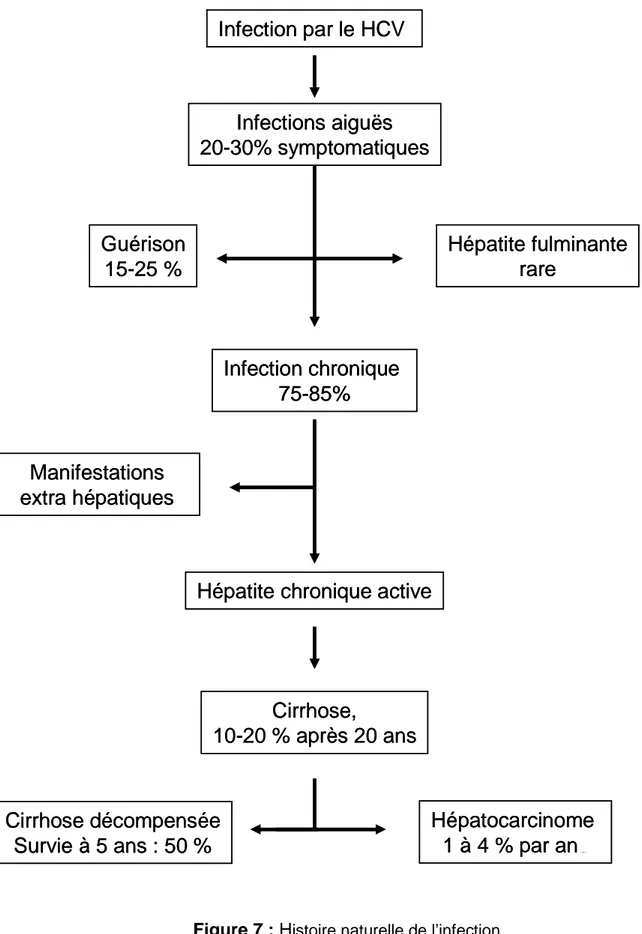
L’histoire naturelle a été bien documentée dans le cadre des hépatites C post-transfusionnelles. Seuls 20-30% des patients avec une infection aiguë vont présenter des symptômes. Ceux-ci apparaissent entre 3 et 12 semaines après la contamination. L’ARN viral pourra être détecté dans le sérum 1 à 2 semaines après une infection (Farci et al., 1991; Thimme et al., 2002). L’activité sérique des aminotransférases augmente 2 à 8 semaines après l’infection. Les anticorps sont détectables dans le sérum 1 à 3 mois après la contamination. Près de 70 % des patients contaminés n’arrivent pas à contrôler l’infection virale après une infection aiguë et de fait évoluent vers une hépatite chronique (Lauer & Walker, 2001). La conséquence majeure de l’infection chronique par le virus HCV est la progression de la fibrose vers la cirrhose et ses complications : hypertension portale, varices oesophagiennes, formation d’ascite et d’œdèmes, hypersplénisme et insuffisance hépatocellulaire. La cirrhose prédispose à l’hépatocarcinome (Poynard et al., 2003b) (Figure 7).
Figure 7 : Histoire naturelle de l’infection
Infection par le HCV
Infections aiguës
20-30% symptomatiques
Hépatite fulminante
rare
Guérison
15-25 %
Infection chronique
75-85%
Manifestations
extra hépatiques
Hépatite chronique active
Cirrhose,
10-20 % après 20 ans
Hépatocarcinome
1 à 4 % par ans
Cirrhose décompensée
Survie à 5 ans : 50 %
Infection par le HCV
Infections aiguës
20-30% symptomatiques
Hépatite fulminante
rare
Guérison
15-25 %
Infection chronique
75-85%
Manifestations
extra hépatiques
Hépatite chronique active
Cirrhose,
10-20 % après 20 ans
Hépatocarcinome
1 à 4 % par ans
Cirrhose décompensée
Survie à 5 ans : 50 %
L’atteinte hépatique est le résultat de la réponse immune suffisamment active pour induire la lyse des cellules hépatiques et la fibrose mais insuffisante pour éradiquer le virus de ses réservoirs (Heydtmann et al., 2001). Deux caractéristiques histologiques sont évaluées lors de l’infection chronique par le HCV : l’activité et la fibrose. L’un des moyens de l’exprimer est le score METAVIR (Bedossa & Poynard, 1996). Ce système évalue les lésions hépatiques en utilisant 2 scores : l’un pour déterminer le niveau de nécrose inflammatoire (A pour activité, de A0 à A3) et l’autre pour déterminer le niveau de fibrose hépatique (F, de F0 à F4). De nombreux facteurs sont corrélés à une progression de la fibrose plus rapide : la durée de l’infection, l’âge, le sexe masculin, une forte consommation d’alcool, une co-infection avec le HIV et le niveau de nécrose (Graham et al., 2001; Massard et al., 2006; Poynard et al., 2003b). Des facteurs métaboliques tels que l’obésité, la stéatose et le diabète sont des cofacteurs récemment décrits dans la fibrogénèse.
Des formes occultes ont été suspectées chez des patients présentant une activité élevée des aminotransférases hépatiques malgré l’absence de détection des anticorps anti-HCV, et de l’ARN HCV par PCR dans le sérum. Chez ces patients, l’ARN HCV a été mis en évidence dans le foie ou dans les cellules mononucléées du sang périphérique (Castillo et al., 2004). La réplication du virus a été objectivée par la recherche du brin négatif du génome HCV dans les cellules mononucléées du sang périphérique (Castillo et al., 2005). Récemment, la même équipe a mis en évidence l’ARN HCV dans le sérum des patients présentant une hépatite C occulte, grâce à des techniques de concentration du virus par ultracentrifugation (Bartolome
et al., 2007). Des études sur gradient d’iodixanol ont montré que l’ARN viral était
présent au niveau des fractions de densité 1,03-1,04 et 1,08-1,19, des fractions similaires à celles de patients présentant une infection chronique classique.
Il est admis que les patients présentant une hépatite C aiguë spontanément résolutive ou considérés comme guéris après un traitement n’étaient plus porteurs du virus. Cette notion a été remise en question en 2004, par deux équipes indépendantes ayant montré chez certains patients la présence de l’ARN viral au niveau du foie et dans les cellules mononucléées du sang périphérique malgrè l’absence d’ARN HCV dans le sérum (Pham et al., 2004; Radkowski et al., 2005a; Radkowski et al., 2005b). Toutefois, la persistance du virus ou la réactivation clinique n’ont pas été observées chez des patients hémodialysés guéris après un traitement anti-HCV, soumis à une immunodépression thérapeutique après transplantation (Kamar et al., 2003).
2. Cibles extra-hépatiques
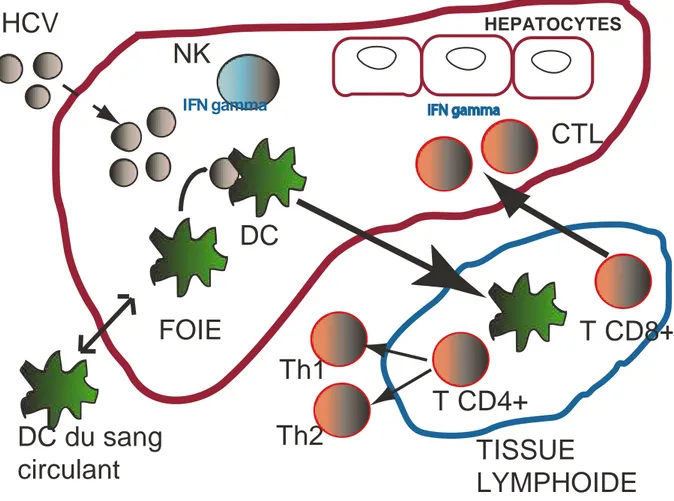
Le tropisme du HCV est essentiellement hépatocytaire mais des sites de réplication extra-hépatiques ont été retrouvés au niveau des cellules mononucléées du sang périphérique tel que les lymphocytes (Bare et al., 2005; Laskus et al., 2000; Shimizu et al., 1992) et les cellules dendritiques (Goutagny et al., 2003), au niveau du système nerveux central (Laskus et al., 2002) et dans la moelle osseuse (Radkowski et al., 2000). Une compartimentation au niveau des cellules mononucléées a été également démontrée (Ducoulombier et al., 2004). Cependant, des études menées par plusieurs laboratoires ont montré que l’entrée des HCVpp dans les cellules mononucléées activées ou non n’était pas détectable (Bartosch et
Les variants isolés dans les différents compartiments corporels d’un même patient diffèrent par leur séquence nucléotidique et leur séquence en acides aminés. Des génotypes différents ont été retrouvés dans les différents compartiments (Roque-Afonso et al., 2005). La compartimentation cellulaire des variants pourrait être liée à un tropisme cellulaire différent. Ce tropisme serait déterminé par la reconnaissance d’une séquence donnée par le complexe récepteur, sa fixation au récepteur et son internalisation spécifique, mais aussi par la capacité des protéines virales à être fonctionnelles dans un contexte cellulaire donné.
Des manifestations extra-hépatiques de l’infection par le HCV ont été décrites. La présence de cryoglobulines mixtes a été retrouvée chez 40 à 80 % des sujets atteints d’hépatite C chronique (Agnello et al., 1992; Lunel et al., 1994; Pawlotsky et
al., 1994). Au sein des cryoprécipités, de fortes concentrations d’ARN viral et de
protéines virales ont été mises en évidence (Agnello et al., 1992). Les manifestations cliniques de ce phénomène sont rares. Il s’agit essentiellement de manifestations cutanées (purpura, lichen plan, syndrome de Raynaud…) neurologiques (paresthésies), rhumatismales (arthralgies) ou rénales (néphropathies glomérulaires). Un lien entre HCV et lymphome a été rapporté dans différentes études (Ferri et al., 1994; Hanley et al., 1996; Hermine et al., 2002; Luppi et al., 1996; Zuckerman et al., 1997). Les lymphomes B non hodgkiniens sont les lymphomes les plus fréquents. Ils semblent plus fréquemment observés en Europe du Sud qu’en Europe du Nord suggérant une contribution de facteurs génétiques ou environnementaux (Matsuo et al., 2004).