L / / 6
THÈSEPRÉSENTÉE
A L'ÉCOLE DES GRADUÉS DE L'UNIVERSITÉ LAVAL
POUR L'OBTENTION
DU GRADE DE MAÎTRE ES SCIENCES (M.SC.)
PAR
DIANE LABBÉ
BACHELIÈRE ES SCIENCES DE L'UNIVERSITÉ LAVAL
CARACTÉRISATION DE SÉQUENCES SIMPLES, FAIBLEMENT RÉPÉTÉES CHEZ LE POULET
ET D'AUTRES ESPÈCES
AVANT-PROPOS
Je voudrais remercier mon directeur de thèse, le Dr. Adolfo Ruiz-Carrillo pour m'avoir guidée tout au long de ce projet. Sa grande disponibilité, ses conseils judicieux issus de sa vaste expérience en ont fait un conseiller précieux.
Je tiens aussi à souligner la contribution de Jean Renaud, notre assistant de recherche dans le laboratoire, d'une part pour la partie du travail qu'il a effectuée et ses conseils techniques, ainsi que pour sa bonne humeur communicative qui a agrémenté mes longues heures d'expéri mentation .
Un merci également à Mamdouh Mikhail de 1 ' Institut de
Recherches Cliniques de Montréal et au Dr. Paul H. Roy du département de biochimie de l'Université Laval pour la patience et la disponibilité qu'ils ont démontré 1 ors de mon apprentissage de l'utilisation des programmes d'ordinateur pour 1‘analyse des séquences.
Je tiens aussi à remercier Francine Samson qui a fait preuve de beaucoup de patience et de minutie pour la dactylographie de cette thèse.
Finalement, je remercie le Conseil de Recherches Médicales du Canada de m'avoir soutenue financièrement pendant ces deux années.
Le présent travail porte sur la caractérisation de séquences simples, composées des tri nucléotides (GGC). Elles ont d'abord été identi fiées dans le génome de poulet avec une sonde d'ADN complémentaire cloné de 1'histone H5 de poulet lors du criblage d'une librairie d'ADN génomique. Plusieurs clones donnant un signal indiquant une homologie partielle avec cette sonde ont été étudiés. Par la séquence de deux d'entre eux, il a été établi que de courtes séquences contenant des répétitions consécutives d'un petit nombre de (GGC) (5 ou 6) en étaient responsables.
L'organisation de ces séquences simples dans le génome a été étudiée par des hybridations sur de 1'ADN génomique. Il s'avère que ce type de séquences simples forment une famille de séquences faiblement répé tées chez le poulet (cent cinquante fois) et également chez toutes les autres espèces eucaryotiques étudiées à savoir le saumon, le canard, le pigeon, la souris et l'humain.
Un polymorphisme dans le nombre de (GGC) consécutifs a été rencontré dans le gêne de 11 histone H5 de poulet par rapport à 1 ‘ADN complémentaire cloné utilisé comme sonde. Ce polymorphisme ainsi que la génération proprement dite de ces séquences pourraient être expliqués par un mécanisme de glissement et de mauvais appariements des répétitions ("slipped mispairing"), lors de la réplication de 1'ADN.
Une étude de la transcription de ce type de séquences a été réalisée chez l'embryon de six jours et les globules rouges immatures de poulet. Dans ces cas, ces séquences simples seraient surtout présentes dans les ARN nucléaires.
Finalement, une recherche par ordinateur dans des banques de séquences d'ADN a permis l'identification de plusieurs gènes porteurs de
(GGC) , pouvant suggérer une variété de rôles biologiques pour ces séquences.
TABLE DES MATIÈRES
Page
AVANT-PROPOS ... i
RÉSUMÉ ... ü TABLE DES MATIÈRES... i i i LISTE DES SYMBOLES ET ABRÉVIATIONS... vi
LISTE DES TABLEAUX... vi i i LISTE DES FIGURES... ix
CHAPITRE 1 INTRODUCTION GÉNÉRALE... 1
1.1 Les séquences répétées dans le génome d'eucaryote. . 3
1.1.1 Séquences fortement répétées : ADN satellites. . 3
1.1.2 Séquences modérément et faiblement répétées, dispersées dans le génome... 3
1.1.2.1 Séquences courtes (de type "SINES"). ... 4
- Séquences simples ... 4
- Famille de séquences Alu... 5
- Autres familles ... 6
1.1.2.2 Séquences longues (de type "LINES"). ... 6
1.1.3 Séquences répétées dans le génome de poulet-, . . 7
1.1.4 Rôles proposés ... 8
CHAPITRE 2 IDENTIFICATION DE SÉQUENCES SIMPLES CHEZ DES CLONES GÉNOMIQUES AYANT UNE HOMOLOGIE PARTIELLE AVEC UN ADN COMPLÉMENTAIRE CLONÉ DE L'HISTONE H5 . . 11
2.1 Introduction... 12
2.2 Matériel et méthodes... 13
2.2.1 Matériel... 13
2.2.2 Méthodes... 13
- Di gestion de 1'ADN avec des enzymes de restriction... 13
- Séparation de 11 ADN par électrophorèse et préparation de fragments... 13
- Transfert de 1 1 ADN... 14
- Marquage de 1 'ADN... 14
Page - Criblage de la librairie d'ADN
génomique... 15
- Cartographie... 16
- Sous-clonage... 16
- Séquence... 17
- Analyse de séquence par ordinateur. . . 18
2.3. Résultats... 20
2.3.1 Criblage de la librairie d'ADN génomique de poulet avec 1'ADN complémentaire cloné de H5 . . 20
2.3.2 Cartographie des clones génomiques xCHVII et XCHIX... 21
2.3.3 Détermination des régions d'homologie des clones XCHVII et xCHIX avec le p541... 21
2.3.4 Séquence des régions d'homologie ... 24
2.3.5 Identification de (GGC)n dans d'autres clones génomiques... 28
2.3.6 Analyse de séquence... 32
2.4 Discussion... 33
CHAPITRE 3 ORGANISATION DES SÉQUENCES SIMPLES DE (GGC)n CHEZ LE POULET ET D'AUTRES ORGANISMES ... 35
3.1 Introduction... 36
3.2 Matériel et méthodes... 39
3.3.1 Matériel... 39
3.3.2 Méthodes... 39
- Courbe de fusion... 39
- Isolement de 11 ADN génomique... 40
- Digestion et hybridation d'ADN génomique... 41
- "Dots" pour les ADN génomiques .... 41
- Recherche de séquences (GGC)n dans la littérature... 43
- Calculs statistiques... 43
3.3 Résultats... 44
3.3.1 Calcul de la probabilité de retrouver au hasard des séquences simples de (GGC)n dans le génome de poulet... 44
3.3.2 Détermination du Tm de l'hybride de 20 pb formé entre le pCHIX0.6H/S et le p541... 44
3.3.3 Organisation et réitération des séquences de (GGC)n dans différents génomes ... 47
Page 3.3.4 Quantification des séquences de (GGC)n dans
différents génomes ... 51
3.3.5 Identification de gènes ayant des séquences de (GGC)n... 55
3.4 Discussion... 58
CHAPITRE 4 TRANSCRIPTION DES SÉQUENCES SIMPLES DE (GGC)n• ... 64
4.1 Introduction... 65
4.2 Matériel et méthodes... 67
4.2.1 Matériel... 67
4.2.2 Méthodes... 67
- Isolement d1 ARN total... 67
- Electrophorèse et transfert d1 ARN ... 68
- Marquage de 1 1 ARN ... .... . 69
- Hybridation des transferts d'ADN et d'ARN... 70
4.3 Résultats. . ... 71
4.3.1 Transcription dans les embryons de poulets de 6 jours... 71
4.3.2 Transcription dans les érythrocytes immatures de poulet... 74
4.4 Discussion... 78
CHAPITRE 5 DISCUSSION GÉNÉRALE... 80
BSA DTT 2-ME SDS DABA PEG NP-40 LB EDTA EGTA Tri s ampr tets D TE
albumine de sérum de boeuf
dithiothréitol
2-mercaptoëthanol
sodium dodécyle sulfate
acide diaminobenzoïque dihydrochlorure
polyéthylène glycol
Noni det P-40
milieu de Luria-Bertani (par litre : 10 g bacto-tryptone, 5 g d'extrait bacto-1evure, 10 g NaCl, pH 7.5)
éthylène diamine tëtraacëtate
1,2-di(2-aminoëthoxy)ëthane-N,N,N',N'tëtra acide acétique
trishydroxymëthylaminomëthane
résistance à l'ampicilline
sensibilité à la tétracycline
solution de Denhardt (IX D = 0.02% BSA - 0.02% Fi col 1 400 - 0.02% polyvinyl pyrrolidone)
TBE : TA SSCPE : SSC : (p/v) : (v/v) : "I'm pb : kb : d .1 . : U : vol . : temp. :
tampon 89 mM Tris-HCl pH 8.3 - 89 mM acide borique - 2 mM NagEDTA
tampon 40 mM Tris-HCl pH 7.8 - 20 mM CHgCOONa - 2 mM NagEDTA
solution sodium-citrate-phosphate-EDTA (IX SSCPE = 0.12 M NaCl - 0.015 M citrate de sodium - 1 mM NagEDTA - 0.013 M NagHPOz;., pH 7.4)
solution sodium-citrate
(IX SSC = 0.15 M NaCl - 0.015 M citrate de sodium, pH 7.4)
(poids/volume)
(volume/volume)
température de fusion (température où 50% des hybrides sont dissociés) paire de bases ki1obase diamètre interne Unité enzymatique volume température
TABLEAU 1 Calcul de la probatilité de retrouver au hasard des séquences simples de (GGC)n dans le génome
de poulet... 45
TABLEAU 2 Nombre de copies des séquences de (GGC)n dans
LISTE DES FIGURES
Page FIGURE 1 Cartes de restriction des insertions des clones
génomiques xCHVII et xCHIX ... 22 FIGURE 2 Détermination de la région d'homologie des clones
génomiques a CH VII et xCHIX avec le p541... 23
FIGURE 3 Cartes de restriction des sous-clones pCHVIII.8H/B
et pCHIX0.6H/S... 25 FIGURE 4 Séquence de 11 insertion de pCHIX0.6H/S... 26 FIGURE 5 Séquence de la région de 247 pb de pCHVII1.8H/B
contenant 11 homologie avec le p541... - . 27 FIGURE 6 Détermination de la région d'homologie de xCHVII
avec le pCHIX0.6H/S... 29 FIGURE 7 Caractérisation de clones génomiques porteurs de
séquences simples de (GGC)n... 30 FIGURE 8 Courbe de fusion de 1'ADN complémentaire cloné de
H5 (p541) avec 1 ‘insertion de pCHIX0.6H/S... 46 FIGURE 9 Organisation de l'insertion de pCHIX0.6H/S dans le
génome de poulet... 48 FIGURE 10 Organisation des séquences simples de (GGC)n dans
différents génomes ... 50 FIGURE 11 Quantification des séquences (GGC)n dans différents
génomes par hybridation de "dots"... 52 FIGURE 12 Séquences de (GGC)n dans des gènes et des
ADN clonés... 56 FIGURE 13 Modèle pour la génération de délétions à partir d'un
mécanisme de glissement et de mauvais appariement
lors de la réplication de 11 ADN... 62 FIGURE 14 Transcription dans les embryons de poulets de
6 jours... 73 FIGURE 15 Transcription dans les érythrocytes immatures
de poulet... 75 FIGURE 16 Hybridation d'un transfert d'ARN d'embryons de
6 jours et d'érythrocytes immatures de poulets
2
De façon générale, la taille du génome des organismes, ou la quantité d'ADN par cellule, augmente avec la complexité de l'échelle évolu tive. Ainsi, une cellule de mammifère contient-elle plusieurs milliers de fois plus d'ADN qu'une bactérie. Chez les organismes supérieurs, une très grande partie de 1'ADN ne code pour aucune protéine ni n'est transcrit en ARN. Par exemple, chez l'humain, le nombre estimé de gênes, approxima tivement de 50 000 à 100 000, apparaît trop faible pour rendre compte des 3 x 1q9 pb (cellule haploïde) existant dans son génome. De plus, une grande partie de 11 ADN du génome des organismes supérieurs apparaît comme une
mosaïque de fragments répétés, quasi identiques à eux-mêmes, jusqu'à des centaines de milliers de fois. Pourquoi tout cet ADN non codant? Pourquoi tant de répétitions?
Britten et Kohne (1965, 1968) ont été les premiers à démontrer 1'existence des séquences répétées chez les organismes supérieurs. C'est à la suite d'expériences portant sur la renaturation de 1'ADN qu'ils furent menés à cette découverte. Ils constatèrent, contrairement à leurs atten tes, que les brins dissociés rencontraient leurs partenaires plus rapide ment dans 1'ADN de vertébré que dans 1'ADN bactérien. Pour expliquer ce paradoxe, une seule explication semblait plausible. L'hypothèse de 1'exis tence de séquences répétées fur alors proposée. L'ADN répétitif représente de 20 à 80% de tout 1'ADN génomique selon 1'espèce considérée.
Grâce aux résultats obtenus suite à 1'utilisation des techni ques de 1'ADN recombinant en génétique moléculaire, 1'organisation des séquences d'ADN répétées dans le génome d'eucaryote est aujourd'hui un peu mieux connue. Aux pages suivantes, un résumé de l'état des connaissances est proposé.
de réitération dans le génome : fortement (1 x 106 et plus), modérément (— 103 - 105) et faiblement répétées (Britten et Khone, 1968; Jelinek, 1982). Alors que les séquences fortement répétées consistent surtout en groupements ("clusters") de répétitions, les séquences modérément et fai blement répétées sont généralement dispersées dans le génome.
1.1.1 Séquences fortement répétées : ADN satellites
Les ADN satellites sont généralement composés de répétitions en tandem soit de quelques paires de bases (séquences simples) ou soit d'u nités formées de sous-répétitions constituant des domaines. Lorsque ces répétitions s'étalent sur de longues distances, cela en fait des séquences hautement répétées. Elles peuvent l'être un million de fois et plus dans le génome. On peut alors généralement les récupérer du reste de 11 ADN d'a près leur séparation caractéristique dans un gradient de densité à l'équi libre, d'où leur nom de satellites. Ces ADN satellites qui peuvent être retrouvés à différents endroits dans le génome sont présents chez les plan tes, les vertébrés et les invertébrés mais ils sont absents chez la levu
re. Quelques-unes de leurs caractéristiques sont 1'association avec l'hë- térochromatine et la réplication tardive dans la phase S du cycle cellulai re. Pour expliquer 1'amplification ou la délétion de ces séquences, des mécanismes de crossing-over inégaux ont été proposés (Smith, 1976).
1.1.2 Séquences modérément et faiblement répétées, dispersées dans le génome
Du point de vue de 1'organisation générale des séquences répé tées dispersées dans le génome, les études de réassociation de 1‘ADN ont indiqué qu'il existe surtout deux types contrastants: Xenopus et Droso phila. Alors que le type Xenopus est caractérisé par un patron de disper sion des séquences répétées au travers de courtes séquences uniques, le mo de Drosophila se rencontre dans les génomes où les répétitions se situent
entre de longs segments d'ADN.
Chez Xenopus, la majorité des séquences uniques de 1 à 2 kb de longueur sont entremêlées de séquences répétées de 0.2 - 0.4 kb. Toute fois, il y a en minorité des séquences uniques plus longues, de 4 kb ou plus, entrecoupées de courts éléments répétés et des séquences réitérées de plus de 1 kb qui n'apparaissent que dans moins de 10% de 1'ADN génomique (Davidson et al., 1975).
Le génome de Drosophila, au contraire, ne contient pas en quantité appréciable de courtes séquences répétées alternant avec des sé quences uniques mais on retrouve plutôt des séquences répétées d'au moins 5.6 kb au travers de séquences uniques de plus de 13 kb (Manning et al., 1975).
L'arrangement de type Xenopus est retrouvé dans la majorité des groupes taxonomiques des animaux et des plantes, à la fois chez les vertébrés et les invertébrés alors que le type Drosophi1 a semble beaucoup moins répandu.
1.1.2.1 Séquences courtes (de type "SINES")
- Séquences simples
Des séquences simples peuvent être retrouvées dispersées dans le génome. Le nombre de répétitions en tandem de l'unité de base peut varier de quelques fois à quelques centaines de fois. Elles se différen cient alors des ADN satellites. En outre, un même type de séquences sim ples (c'est-à-dire des séquences composées-de la même unité) peut exister en plusieurs exemplaires dans le génome. Plusieurs exemples ont été cités dans la littérature. Il ne sera fait mention ici que de quelques-uns afin d'illustrer le propos.
Dans la famille multigénique des gènes d'hi stones chez 1'our sin de mer (S. purpuratus), Sures et collaborateurs (1978) ont trouvé la séquence (CT/GA)27 dans un "spacer" entre les gènes H2A et Kl.
Chez la souris, Nishioka et Leder (1980) ont retrouvé la séquence (CA)3] dans deux gênes d'immunoglobulines de cellules embryon naires et de plasmacytomes. Un fragment génique contenant cette séquence a donné un fort bruit de fond lors de 1'hybridation de transferts d'ADN géno mique.
Chez l'humain, des séquences simples de (TG)n ont été trouvées dans des gènes de globi nés foetales (Slightom et al., 1980) ainsi que dans le gène de 1'actine du muscle cardiaque (Hamada et Kakunaga, 1982). Il y au rait environ cent mille copies de ce type de séquences dans le génome humain.
- Famille de séquences Alu
Parmi les séquences courtes, modérément répétées et dispersées dans le génome, les séquences Alu sont sans aucun doute les mieux caracté risées. Elles ont d'abord été observées par Houck et al. (1979) chez 1'hu main, qui a un patron de dispersion des séquences répétées dans le génome du type Xenopus. La famille Alu de l'humain est composée de dimères de 165 et 135 pb qui sont mis en évidence lorsqu'on coupe avec 11 enzyme de res triction Alu1. Chaque monomère se termine par une séquence de poly (dA) et contient une séquence semblable à 1'origine de réplication du papovavirus (Jelinek et al., 1980). De courtes répétitions directes de 9 à 20 pb bor dent la séquence de 300 pb.
Les séquences Alu sont présentes environ 500 000 fois dans le génome humain (Houck et al., 1979; Rinehart et al., 1981). Lorsqu'on crible une librairie d'ADN génomique avec une sonde Alu, environ 95% des phages hybrident (Tashima et al., 1981), ce qui signifie que les séquences Alu sont effectivement dispersées parmi les séquences uniques. Elles sont conservées chez d'autres espèces c'est-à-dire que l'on a pu dériver une séquence consen sus. On a retrouvé d'autres membres de cette famille entre autres chez les primates et les rongeurs.
Les séquences Alu sont retrouvées environ dix fois plus dans les produits de transcription au niveau nucléaire que cytoplasmique (revue de M. F. Singer, 1982). On retrouve de petits ARNs, c'est le cas du 4.5S
chez la souris et le hamster (Jelinek, 1978b), qui ont de 1'homologie partielle avec les séquences Alu. On retrouve également chez la souris des ARN messagers qui contiennent des séquences de type Alu (Georgiev et al., 1982).
- Autres familles
Beaucoup d'autres types de séquences courtes répétées, se distinguant de la séquence consensus de la famille Alu, commencent à être caractérisés. Qu'il suffise de mentionner qu'il existe chez l'humain la famille Hinf composée d'une unité de 319 pb bordée de répétitions directes de 1.36 pb et de répétitions inversées de 2 pb. Il existerait un faible nombre de copies, soit de cinquante à cent par génome haploïde (Shimizu, 1983). Chez la souris, il existe environ cent mille copies de séquences de type R faite d'une unité de 475 pb. Certaines d'entre elles ont été identifiées dans le voisinage des gênes d'immunoglobulines (Gebhard et al., 1982).
1.1.2.2 Séquences longues (de type "LINES")
Il y a des séquences longues et répétées qui ont des proprié tés analogues aux éléments transposables bactériens. On retrouve ainsi les éléments Copia chez Drosophila et Ty chez la levure qui ont de 5 à 7 kb de longueur. Ils sont bordés par des répétitions directes longues de 300 à 600 pb et par des plus courtes, de quelques paires de bases (Levis et al., 1980; Dunsmuir et al., 1980).
Les familles Kpnl chez les primates et EcoRI chez la souris regroupent des séquences répétées longues. Les éléments de la famille Kpnl affichent un polymorphisme de longueur et ont été observés près des gênes. Quelques-uns d'entre eux sont transcrits (Lerman et al., 1983).
Finalement, un élément d'une famille inconnue de séquences répétées ayant environ 6.4 kb et réitéré de 3 000 à 5 000 fois dans le génome humain a été repéré à 3 kb en aval de l'extrémité 3' du gêne de g-globine humaine (Adams et al., 1980).
1.1.3 Séquences répétées dans le génome de poulet
Le génome de poulet contient seulement 10% de séquences répétées dispersées et 3% d'ADN satellites (Ologsson et Bernardi, 1983a). Seulement quelques génomes étudiés jusqu'à présent n'ont que si peu de
séquences répétées: Drosophila, Chironomus ainsi que 1'abeille A. mellifera en sont des exemples (Manning et al., 1975; Wells et al., 1976; Crain
et al., 1976).
Les travaux du groupe de F. Eden (1978a, b) ont finalement fait la lumière sur des interprétations contradictoires qui avaient été présentées pour 1'organisation générale des séquences dans le génome de poulet. De Jimenez et al. (1974) avaient d'abord proposé un mode de dis persion à courte périodicité. Par la suite, Arthur et Straus (1978) ont estimé la longueur des éléments répétés à 3.4 kb et ont opté pour une orga nisation se rapprochant de celle de Drosophila. . Les résultats des études de réassociation de 1'ADN du groupe de F. Eden démontrèrent qu'environ 40% du génome consiste en séquences modérément répétées d'au moins 2 kb de lon gueur au travers de séquences uniques d'environ 4.5 kb. L'autre moitié du génome comprend des segments d'au moins 17.5 kb qui ne sont interrompus par aucune séquence répétée. Le génome de poulet contient donc des éléments caractéristiques de chacun des types extrêmes d'organisation décrits précé demment à savoir Xenopus et Drosophila. Ces résultats ont d'ailleurs fina lement été confirmés par Arthur et Straus (1983).
Le clonage d'une fraction d'ADN génomique enrichie en séquen ces modérément répétées (Eden, 1980) a permis de retrouver dans un clone deux séquences répétées de longueur et de fréquence de réitération complè tement différentes. Ainsi, l'une a 1.75 kb alors que l'autre a 2.3 kb de longueur. Le fait que des séquences répétées soient voisines démontre qu'en plus d'un mode de dispersion des séquences répétées parmi les séquen ces uniques, il peut y avoir dispersion au travers de d'autres séquences répétées ayant des caractéristiques structurales différentes.
8
Plusieurs types de séquences simples répétées ont été retrou vés au voisinage ou dans les gênes codant pour des protéines de l'oeuf chez le poulet: ovotransferrine, ovalbumine-X (Maroteaux et al., 1983).
D'autre part, Dybvig et collaborateurs (1983) ont rapporté la séquence simple (AGAGG)g2 qui est répétée 2 000 fois dans le génome.
De courtes séquences répétées ayant un certain degré d'homolo gie avec les séquences Alu ont été rapportées et sont appelées CRI (Stumph et al., 1981). Des représentants de la famille CRI ont été retrouvés dans le voisinage d'un gêne codant pour 1'ARN Ul et dans celui du gêne de l'o valbumine. Ces séquences sont formées de 180 pb et le nombre de copies a été estimé à environ 7 000 (Stumph et al., 1981 ; Olofsson et Bernard!, 1983b), ce qui correspond à 0.1% du génome. L‘homologie avec la séquence consensus de la famille Alu des mammifères est de 70% dans certaines régions. Cela indique qu'au moins quelques portions de séquences existaient avant la séparation entre les espèces aviaires et de mammifères. Ce niveau d'homo logie est celui que l'on doit espérer en se basant du point de vue évolutif (Stumph et al., 1981).
1.1.4 Rôles proposés
Pour les ADN satellites, on pense entre autres qu'ils pour raient être des composants structuraux des chromosomes, étant donné qu'ils résident principalement dans leurs portions centromériques et télomériques (Mattoccia et Comings, 1971).
Plusieurs hypothèses d'un rôle général des séquences répétées dispersées ont été formulées pour expliquer leur accumulation et leur per sistance dans le génome d'eucaryote. Ainsi, Britten et Davidson (1969) ont proposé que les séquences répétées puissent agir comme éléments régulateurs de l'expression des gênes étant donné leur dispersion à travers le génome. La quantité d'ADN supplémentaire des organismes supérieurs, par rapport par exemple aux uni cellulaires, comblerait le besoin d'une régulation spéci fique. Ils suggèrent également un rôle évolutif aux séquences répétées comme réservoir de séquences pour créer de nouveaux gênes (Britten et Davidson, 1971) et un rôle dans la régulation de la maturation spécifique des ARN précurseurs (Davidson et Britten, 1979).
les "spacers" dans un modèle d'ADN inutile ("égoïste"). Ils proposent que la réplication de 1'ADN puisse permettre l'accumulation dans le génome de séquences qui n'apportent aucune contribution à l'expression du phénotype mais dont la présence favorise conséquemment 1'accumulation de séquences du même type. Ces séquences pourraient ou non, être transcrites. Cependant, en se basant sur les coûts énergétiques, 1‘ADN inutile s1 accumulerait
davantage dans les régions non transcrites du génome que dans celles qui le sont.
Le modèle de régulation de 1'expression des gênes par les séquences répétées était basé à 1'époque sur un mode d'organisation des séquences de type Xenopus. On sait maintenant que ce type d'arrangement n'est pas unique. Ce modèle dans sa conception originale semble donc être maintenant devenu inadéquat. Quant à celui de 1'ADN inutile, il apparaît lui aussi contestable au fur et à mesure que des rôles sont suggérés pour des séquences qui, à priori ne semblaient apporter aucune contribution à 1'expression du phénotype. D'ailleurs, l'étude des séquences répétées connaît maintenant un regain d'intérêt justifié par ces indications.
Récemment, Hess et collaborateurs (1983) ont suggéré d'après leurs résultats que des séquences Alu présentes dans des gênes de type
g - globi ne chez l'humain puissent affecter le processus de conversion de ces gènes. Plusieurs autres rôles ont été suggérés pour les séquences de type Alu (revu par Jelinek, 1982; Bouchard, 1982) à savoir la régulation de la transcription par de petits ARN: nucléaires qui ont des séquences homo logues à la séquence consensus Alu et un rôle dans la réplication de l'ADN en tant qu'origines de réplication. D'autre part, selon une hypothèse intéressante de Jagadeeswaran et collaborateurs (1981), les séquences Alu pourraient s'insérer au travers des séquences du génome par un mécanisme de rétro transcription, à la manière des éléments transposables. On sait déjà que, du fait de leur mobilité, les éléments transposables sont susceptibles de générer des mutations à leur site d'insertion, pouvant provoquer soit une perte, soit une augmentation d'activité des gênes adjacents.
10
Dans la littérature, il a été fait mention de plusieurs rôles possibles pour les séquences simples. Ainsi, dans certains cas, elles seraient impliquées dans des événements de recombinaison (Sures et al., 1978; Slightom et al., 1980). Certaines séquences semblent modifier loca lement la structure de 1'ADN d'après les résultats obtenus avec la nuclëase SI (Hentschel. 1982; Glikin et al., 1983; Dybvig et al., 1983). Des séquences simples de (TG)n peuvent, d'après des études réalisées in vitro, adopter
une conformation Z dans 1'ADN (Wang et al., 1979; Haniford et Pulleyblank, 1983), nettement différente de la conformation B qui est la plus répandue.
Il ne fait donc aucun doute, compte tenu de l'état des connais sances actuelles, que l'étude des séquences répétées soit d'un intérêt certain. C'est d'ailleurs ce qui nous a motivés à réaliser le travail présenté dans le cadre de cette thèse.
IDENTIFICATION DE SÉQUENCES SIMPLES CHEZ DES CLONES GÉNOMIQUES
12
2.1 Introduction
L'intérêt de notre laboratoire étant entre autres axé sur l'étude de la régulation de 1'expression du gène de 1‘histone H5, une
librairie d'ADN génomique de poulet (construite par Dodgson et al., 1979) a été criblée pour trouver des clones porteurs de ce gène (Ruiz-Carrillo et al., 1983). A cette fin, on a utilisé un ADN complémentaire de 1'histone H5 de poulet, cloné dans pBR322. La taille de 1 ‘insertion du p541 est de 259 pb. Il contient 118 nucléotides dans la portion 5' non traduite de 1‘ARN messager ainsi que les séquences codantes de 46 acides aminés de la portion N-terminale des 189 acides aminés de la protéine (Ruiz-Vazquez et Ruiz-Carrillo, 1982).
Outre des clones contenant le gêne de 1'histone H5 et caracté risés en détail (Ruiz-Carrillo et al., 1983), plusieurs autres clones géno miques ont donné un signal d'hybridation correspondant à une homologie partielle avec la sonde utilisée. Nous avons donc décidé de déterminer les séquences portées par deux de ces clones. Par la suite, le caractère répé titif des séquences trouvées a été illustré avec d'autres clones du même type.
2.2 Matériel et méthodes
2.2.1 Matériel
Toutes les enzymes utilisées proviennent soit de Boëhringer- Mannheim, BRI, Biolabs ou de Life Sciences. Les [a - 32p] dNTPs
(4 x 103 à 6 x 103 Ci /mmole) et [y - 32P] ATP (5 x 103 à 7 x 103 Ci /mmole) ont été achetés de NEN (New England Nuclear) ou d1Amersham. L1 agarose à faible point de fusion vient de BRL. Les filtres de nitrocellulose (BA 85, 0.45 ym) sont de Schleicher & Schüll.
Pour les réactions de séquence, le dimëthyl sulfate (99%) provient de chez Aldrich Chemical Co., 1'hydrazine (95%) d1Eastman Organic Chemicals, la piperidine de Fisher Scientific, la formamide, la pyridine et l'acide formique de BDH (Analar), l'urée de BRL et le BSA de Miles Labora tories. La résine AG501-X8 servant à dëioniser la formamide ainsi que le bisacrylamide et 1'acrylamide ont été achetés chez Bio-Rad.
2.2.2 Méthodes
- Di gestion de 1‘ADN avec des enzymes de restriction
Les digestions de 1'ADN avec des enzymes de restriction sont faites en présence d'un excès de trois fois d'enzyme dans les conditions recommandées par les fabricants. Dans le cas des doubles digestions, 1'ADN est précipité entre chacune d'elles par 1'addition de NaCl en concentration finale de 300 mM et de 2.5 vol. d'éthanol à 95%.
-Séparation de 1'ADN par électrophorèse et préparation de fragments
L'ADN est séparé par électrophorèse sur gel d'agarose variant de 0.5% (p/v) â 1.8% dépendant de la taille des molécules. L'électro phorèse est faite dans le tampon TBE.
14
Les fragments d'ADN servant à la fabrication des sondes sont purifiés sur gel d1 agarose à faible point de fusion, dans le tampon TA. Ces fragments sont ensuite prélevés et extraits par phénolisations suivies de précipitations à 11 éthanol.
Pour la séquence, les fragments marqués radioactivement aux extrémités 3' ou 5' sont préparés sur gel de polyacrylamide à 4% (p/v) acrylamide - 0.14% (p/v) bisacrylamide. Les bandes d'ADN désirées sont excisées et éluëes par agitation à 37°C dans 400 yl du mélange 1 M NaCl - 10 mM EDTA - 2 yg d'ADN d'E. coli sonifië - 3 yg d'ARN-t d'E. coli, de 16 à 20 h.
Pour la séquence, on s'est servi de gels minces (0.4 cm d'épaisseur) (Sanger et Cou!son, 1978) et on a utilisé des concentrations d'acrylamide / bisacrylamide de 5.7%/0.3% et 7.6%/0.4% avec 8.3 M urée.
- Transfert de 1 ‘ADN
Après électrophorèse, les fragments d'ADN sont transférés sur filtre de nitrocellulose selon la méthode de Southern (1975), 1'ADN ayant préalablement été dégradé par dépurination partielle et hydrolyse alcaline
(Wahl et al., 1979).
- Marquage de 1 'ADN
Le marquage des extrémités 3' de 1'ADN est réalisé par l'activité 5‘-3' polymérase de .l'ADN polymérase de T4 selon le protocole adapté de 0'Farrell (1981) en présence de 1 dNTP marqué et des autres dëso- xyribonuclëotides froids. Dans le cas des extrémités franches de 1'ADN, un traitement préalable avec la même enzyme, en l'absence de précurseurs est effectué.
Le marquage des extrémités 5' de 1'ADN par la polynuclëotide kinase de T4 (réaction 5B) est réalisé selon le protocole de Maxam et Gil bert (1980). Dans le cas de l'étape préliminaire de déphosphorylation, le traitement de 1'ADN est effectué dans 45 yl de TE pH 8.0 et 5 yl de 50 mM
Tris-HCl pH 8.0 avec 0.1 U de phosphatase alcaline d'intestin de veau (CIP) par 10 pmoles durant 45 min S 37°C.
Le marquage de 1'ADN par la technique de "nick-translation" avec l'ADN polymérase I d'_E. coli est fait selon Rigby et al. (1977). L'ADN est purifié par passage dans une colonne de Sephadex G-50 (Pharmacia). On obtient généralement une activité spécifique de 1-2 x 10® cpm/yg.
- Hybridation
Les pré-hybridations d'ADN se font dans 3X SSCPE - 10X D (Denhardt, 1966) - 0.1% (p/v) SDS en présence de 50 ug/ml d'ADN d'E. coli sonifié et dénaturé par chauffage dans TE pH 7.5 à 100°C/5 min. Les
hybridations se font dans un milieu de composition identique auquel on rajoute la sonde en excès (1 x 10® cpm/ml de mélange d'hybridation). Les pré-hybridations (2 à 4 h) et les hybridations (16 à 20 h) se font à 67°C. Les filtres sont ensuite lavés, sauf indication contraire, dans les condi tions suivantes: 3X SSCPE - IX D - 0.1% SDS/temp. ambiante/15 min/4 fois; IX SSCPE - 0.1% SDS/65°C/15 min/2 fois; 0.5X SSCPE - 0.1% SDS/65°C/15 min/2 fois ; 0.2X SSCPE - 0.1% SDS/65°C/15 min/1 fois. Les films Kodak XAR5 et les écrans Dupont Cronex Lightening Plus sont utilisés pour 1‘autoradiogra phie a -80°C.
- Criblage de la librairie d'ADN génomique
L'ADN complémentaire cloné de H5, le p541 (Ruiz-Vazquez et Ruiz-Carri 11o, 1982), a été utilisé pour cribler environ 2 x 105 phages de la librairie d'ADN génomique de poulet construite par Dodgson et al.
(1979). Ils ont été criblés selon la méthode de Benton et Davies (1977) et ce, à 0.5X SSC - 0.1% SDS/65°C, en présence de poly G (50 yg/ml). Des
phages positifs des trois classes de signaux d'hybridation (A,B,C; voir p.20) ont été préparés (Ruiz-Carrillo et al., 1983).
L'insertion du pCHIX0.6 H/S (voir p.24) marquée par "nick- translation" a été utilisée pour cribler à 0.5X SSCPE - 0.1% SDS/65°C, des phages ayant donné un signal de type B lors du criblage initial de la librairie.
Cette partie du travail a été réalisée par notre assistant de recherche dans le laboratoire, Jean Renaud. L'ADN des phages positifs avec le pCHIX0.6H/S a été préparé, digéré avec EcoRI, transféré sur filtre de nitrocellulose et hybridé avec les insertions de pCHIX0.6H/S et du p541 marquées par "nick-translation". Les hybrides ont été lavés jusqu'à 0.IX
SSCPE - 0.1% SDS/65°C/7 min dans le cas du pCHIX0.6H/S et jusqu'à 0.2X SSCPE - 0.1% SDS/65°C/15 min pour le
p541.-- Cartographie
Essentiellement, deux méthodes ont été utilisées. Des diges tions simples et doubles avec différentes enzymes de restriction ont été faites de manière à déduire la carte de restriction recherchée d'après la longueur des fragments obtenus. On s'est aussi servi de la méthode des digestions partielles de Smith et Birnstiel (1976) qui permet d'ordonner, après autoradiographie, la position des sites de restriction à partir d'une extrémité marquée.
- Sous-clonage
Les régions d'intérêt des recombinants ACHVII et ACHIX ont été sous-clonées dans pAT153, un dérivé de pBR322 se répliquant à un nombre élevé de copies (Twigg et Sherratt, 1980). Les ligations ont été faites avec 1 U d'ADN ligase de T4 avec 150 ng des fragments Hind III - Sal I de 644 pb de ACHIX ou HindlII - BamHI de 1 800 pb de ACHVII en utilisant 450 ng de pAT153 amputé des fragments correspondants et purifié. Les réactions ont été faites à 4°C dans 20 yl (vol. final), de 16 à 20 h puis à 14°C 1 h, avec 10 mM DÎT - 0.4 mM ATP - 60 mM Tris-HCl pH 7.5 - 6.6 mM MgCl2* La souche E. coli C600 (rec A") a été transformée par la méthode au chlorure de calcium (Mande! et Higa, 1970). La sélection des clones
positifs ampr et tets a été réalisée sur des milieux LB avec ampicilline (100 yg/ml) ou tétracycline (20 yg/ml) selon le cas. L'identification a été complétée par un criblage de l'ensemble des recombinants selon la méthode de Grunstein et Hogness (1975). Les fragments Sali - EcoRI
régions d'intérêt ont été marqués par "nick-translation" et utilisés comme sondes.
Les ADN plasmidiques de plusieurs sous-clones ont été isolés par une méthode rapide mise au point dans notre laboratoire. Le culot de cellules bactériennes en suspension dans 25% sucrose - 50 mM Tris-HCl pH 8.0 est lysé par l'addition, en concentration finale, de 0.6 mg/ml de lysozyme - 0.060 M EDTA - 25 mM Tris-HCl pH 8.0 - 0.05% Triton X-100 à 0°C/10 min. Le mélange est ensuite chauffé à 70°C/15 min. Après centrifugation
(12 000 g/15 min), le surnageant contenant les acides nucléiques est préci pité par l'addition d'un vol. de 20% PEG (6 000) - 20 mM Tris-HCl pH 8.0 - 1 M NaCl â temp, ambiante pendant 30 min. Le culot est dissout dans du TE (10/0.1) pH 8.0 puis traité â la RNase (0.15 mg/ml) S 37°C/30 min. L'ADN est finalement obtenu par précipitation dans de 1'acétate d'ammonium 2 M et 3 vol. d'éthanol â 95% (rendement d'environ 2.5 yg). La taille des insertions a été vérifiée par digestion avec les enzymes appropriées et mi gration sur gel d'agarose.
Les plasmides pCHIX0.6H/S et pCHVIII.8H/B amplifiés ont été isolés en grande quantité selon la procédure des lysats clairs, mise au point dans notre laboratoire (lyse bactérienne contrôlée, effectuée dans 60 mM EDTA - 25 mM Tris-HCl pH 8.0 - 1% (v/v) Triton X-100 avec 0.3 mg/ml de lysozyme) suivi de deux centrifugations consécutives, à 1'équilibre, du culot d'acides nucléiques dans 4.7 M CsCl contenant 0.4 mg/ml de bromure d'ëthidium à 62 000 rpm/4 h (rotor VTi 65). La concentration d'ADN est déterminée par fluorimétrie après réaction avec DABA selon le protocole de Kapp et al. (1974).
- Séquence
La méthode de clivage chimique partiel de 1'ADN ainsi que les techniques s'y rattachant, mises au point par Maxam et Gilbert (1977, 1980) ont été employées. On a utilisé les réactions spécifiques aux guanines, purines, pyrimidines et cytosines. Les stratégies choisies pour sêquencer
18
les 644 pb de 11 insertion de pCHIX0.6H/S ainsi que les 247 pb du fragment Smal - BamHI de pCHVII1.8H/B sont illustrées à la figure 3. Pour 11 inser tion de pCHIX0.6H/S, 89% des 315 pb autour de la région homologue au p541 ont été séquencées deux fois alors que pour le fragment de pCHVIII.8H/B, 72% des 2 brins ont été séquencés 2 fois.
- Analyse de séquence par ordinateur
L'analyse des séquences a été effectuée grâce aux programmes mis au point par Staden (1977, 1978) (BDOVRL, OVRLAP, TRANMT) et ceux du groupe UWGCG ("University of Wisconsin Genetics Computer Group"), principa lement le groupe de J. Devereux (1984) (FIND). Les banques de séquences "GenBank" (Fickett et al., 1982) et EMBL ("European Molecular Biology Laboratory") ont été analysées.
Le programme BDOVRL permet de chercher des homologies pour une séquence donnée avec 11 ensemble des séquences d'une sous-division de la li brairie "GenBank". La séquence recherchée est traitée par l'ordinateur en blocs de comparaison de N nucléotides. On définit également un nombre mi nimal de nucléotides homologues. Il est souhaitable de commencer la re cherche d'une séquence inconnue avec des blocs de séquence de 3 nucléotides et un minimum de 9 pb appariées. Le résultat global de 1‘homologie trouvée avec chacune des séquences d'intérêt dans la banque est donné en pourcen tage. On peut ensuite faire une analyse plus précise d'homologie avec le programme OVRLAP.
Le programme OVRLAP est utilisé pour comparer des séquences afin de trouver des régions homologues avec ou sans mauvais appariement. Il peut être utilisé pour trouver des répétitions internes (directes ou inversées) dans une séquence ou pour comparer deux séquences. Une longueur minimale d'homologie ainsi qu'un nombre de mauvais appariements sont
spécifiés par l'utilisateur et toute homologie satisfaisant ces paramètres est rapportée. Le programme calcule aussi automatiquement le complément
d'une des séquences et le compare à l'autre par la même occasion. Pour les fins de recherche d'identité de pCHIX0.6H/S et pCHVIII.8H/B avec certaines séquences des librairies, on a généralement donné un minimum de 9 pb homo logues et aucun mauvais appariement.
Le programme FIND recherche une chaîne de caractères dans d'autres séquences, par exemple dans certaines portions définies de "GenBank", d'EMBL ou dans toute autre séquence. Ce programme recherche aussi le complémentaire si la chaîne de caractères est formée de nucléo tides. En donnant plusieurs chaînes d'une dizaine de caractères, issues d'une séquence inconnue et en les cherchant dans les banques de séquences, on peut retracer la séquence homologue, si elle existe.
Le programme TRANMT détermine les codons correspondants aux trois cadres de lecture possibles dans la séquence proposée. Il exécute aussi la même opération sur la séquence du brin complémentaire.
2.3 Résultats
2.3.1 Criblage de la librairie d'ADN génomique de poulet avec 11 ADN complémentaire cloné de H5
Environ 2 x 10$ phages représentant environ 2 fois la taille du génome de poulet ont été criblés avec 1 1 ADN complémentaire cloné de H5, le p541. Trois types de signaux ont été obtenus après hybridation puis lavage de la sonde dans des conditions de lavage de 0.5X SSC - 0.1% SDS /65°C. Neuf clones donnant un signal très fort (A), 77 un signal intermé diaire (B) et 516 faibles (C) ont été observés. Les signaux faibles ne provenaient pas d'une hybridation non spécifique puisque, dans les mêmes conditions, des clones recombinants Xgt d'une librairie d'E. coli n'ont donné aucune réponse avec la sonde. Ces résultats ont été obtenus par A. Ruiz-Carrillo.
Par la suite, une analyse par hybridation de "dots", de 1'ADN de recombinants de chacun des 3 types de signaux obtenus a montré qu'après lavage des hybrides dans des conditions de 0.IX SSC - 0.1% SDS/65°C, seuls les signaux de types A et B sont demeurés (A. Ruiz-Carrillo, résultats non pub!iës).
Deux clones de type A ont été caractérisés en détail et se sont avérés contenir des fragments chevauchants du gêne de 1'histone H5 dont la séquence a été déterminée. L'hybridation du p541 avec de l'ADN génomique a permis de conclure qu'il n'y a qu'une copie de ce gêne par génome haploïde (Ruiz-Carrillo et al., 1983). Les clones de type B et C semblaient, quant à eux, contenir des séquences ayant des homologies partielles avec la sonde utilisée, ceux de type B davantage que le type C.
Nous avons d'abord décidé d'identifier la nature des homo logies portées par des clones de type B en sëquençant deux d'entre eux choisis au hasard, les ACHVII et ACHIX.
2.3.2 Cartographie des clones génomiques ACHVII et ACHIX
Les emplacements des divers sites de restriction des clones ACHVII et ACHIX ont été déduits d'après la taille des fragments obtenus par des digestions simples et doubles avec différentes enzymes de restriction. La technique des digestions partielles de Smith et Birnstiel (1976) a servi à compléter ces informations. Les ACHVII et ACHIX comportent respective ment des insertions de 12.8 kb et 18.0 kb de différents morceaux d'ADN à en juger d'après la position des sites de restriction. Ces résultats sont il
lustrés à la figure 1.
2.3.3 Détermination des régions d'homologie des clones ACHVII et ACHIX avec le p541
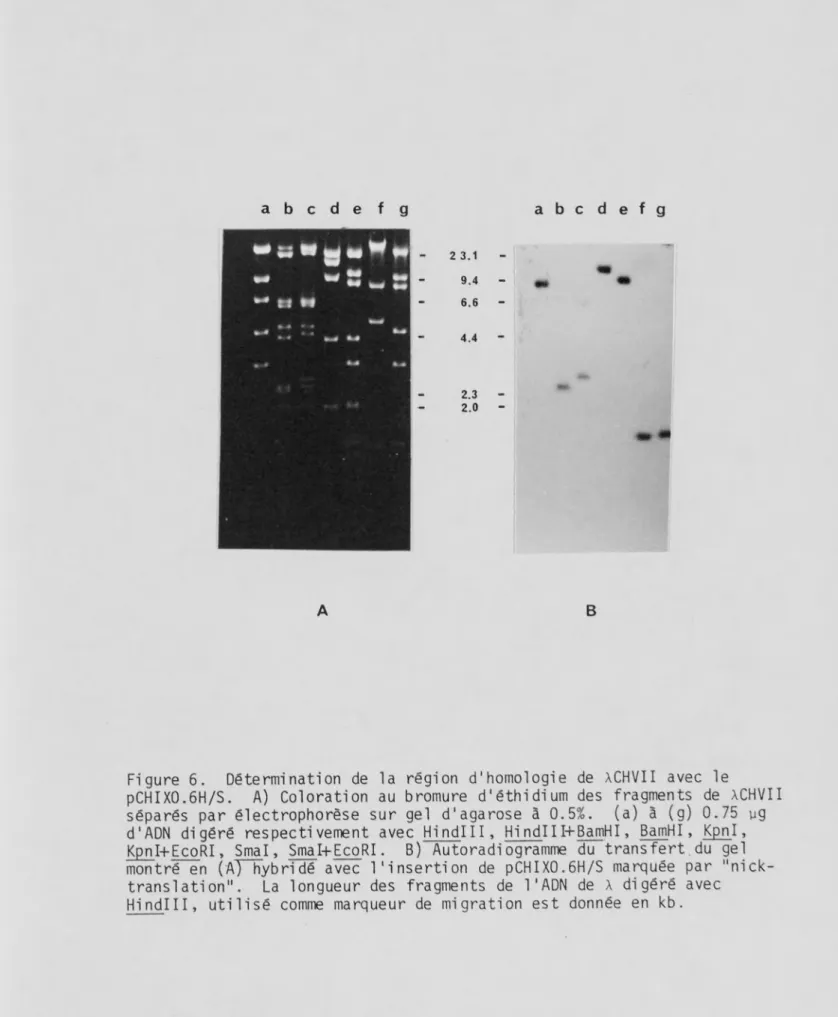
Afin de cerner précisément les régions de ACHVII et ACHIX homologues à 1'ADN complémentaire cloné de H5, ces recombinants ont été digérés avec plusieurs enzymes de restriction dont les sites d'action étaient connus. Les fragments d'ADN séparés par électrophorèse sur gel d'agarose ont été transférés sur filtre de nitrocellulose et hybridés avec l'insertion du p541, marquée par "nick-translation". D'après la figure 2, on observe sur les deux autoradiogrammes qu'une seule bande par canal a hybridé avec la sonde; dans les cas où il y a deux bandes, la bande la plus faible serait due à une digestion partielle. Cela signifie donc que les régions d'homologie ne couvrent qu'une portion de chacun des clones géno miques .
On a ainsi pu déduire que 1'homologie du p541 avec ACHVII était au maximum de 247 pb (fragment Smal - BamHI) et de 644 pb (fragment HindIII - Sal I) avec ACHIX (les longueurs précises ont été déterminées par séquence). Ces régions d'homologie sont montrées sur les cartes de res triction de la figure 1.
# eg o □ • a • g i---1 1 kb g a
/
a g g g gg □ □ g1
/
A ga ■ • B I---1 1 kbFigure 1. Cartes de restriction des insertions des clones génomiques XCHVII et XCHIX. Le rectangle noir représente les régions de XCHVII (A) et XCHIX (B) sous-clonées, contenant les portions homologues â l'ADN complémentaire cloné de H5 (p541). Les symboles utilisés sont: HindiII (□), Smal (O),
BamHI (•), EcoRI (A), Kpnl (■), Sali (y), jonctions synthétiques EcoRI (a).L'enzyme Sali ne coupe pas
XCHVII alors que XCHIX n'est pas coupé par Smal.
ro IX)
Figure 2. Détermination de la région d'homologie des clones génomiques XCHVII et ACHIX avec le p541.
A) Coloration^au bromure d'éthidium des fragments d'ADN séparés par électrophorèse sur gel d'agarose a 0.5%. (a) & (i) 0.5 yg d'ADN de ACHIX digéré respectivement avec EcoRI, EcoRI+SalI, Sali, EcoRI+Hindl11, HindiII, EcoRI+Kpnl, Kpnl, EcoRI+BamHI, BamHI; (j) à (1) 0.5 yg, (m) à (q) 0.75 yg d'ADN de ACHVII digéré respec tivement avec EcoRI, EcoRI+BamHI, BamHI, Smal, EcoRI+Hindl11, Hindl11, EcoRI+KpnI, KpnI. B) Autoradiogrammes des transferts des gels montrés en (A) hybridés avec 1'insertion du p541 marquée par "nick-translation". La longueur des fragments de l'ADN de A digéré avec Hindi 11 utilisé comme marqueur de migration est donnée en kb. ♦quantité moindre, indéterminée, dans le cas des doubles digestions.
rx) co
2.3.4. Séquence des régions d'homologie
Les fragments contenant les régions d'homologie ont par la suite été sous-clonés dans pAT153. Dans le cas de aCHVII, c'est le
fragment HindIII - BamHI de 1.8 kb (figure 1) qui a été sous-cloné pour former le plasmide pCHVIII.8H/B. Le pCHIX0.6H/S a été obtenu par sous- clonage du fragment Hind 111 - Sal I de 0.6 kb issu de ACHIX.
Des cartes de restriction détaillées de ces deux sous-clones apparaissent à la figure 3. Les stratégies de séquence par la méthode de dégradation chimique de Maxam et Gilbert (1977, 1980) y sont également i 11ustrées.
Les séquences des brins porteurs de 1‘homologie avec l'ARN messager de H5 dans l'insertion du pCHIXO.6H/S (644 pb) et dans le fragment BamHI - Smal (247 pb) de pCHVIII.8H/B sont respectivement données aux figu res 4 et 5. Il est à noter que la séquence du fragment de pCHVIII.8H/B est riche en bases G+C par rapport à 1'ensemble du génome qui a une composition moyenne en guanines et en cytosines de 44% d'après Shapiro (Handbook of Biochemistry, 1976, p. 269).
La figure 12 (voir chapitre 3) illustre entre autres la compa raison d'une partie des séquences de pCHIX0.6H/S, de p541 (brin homologue à l'ARN-m) et de pCHVIIl.8H/B. On observe que dans le cas des 2 sous-clones, la région d'homologie avec le p541 ne correspond qu'à 20 pb et qu'elle est majoritairement formée d'une séquence simple de 5 tri nucléotides (GGC). De
plus, le triplet (AGC) borde de chaque côté la séquence simple pour
pCHIXO.6H/S et le p541 alors qu'il ne manque qu'un A du côté droit (figure 12) pour observer la même chose avec pCHVIII.8H/B. Dans le cas du
pCHIX0.6H/S, on peut également observer un sixième (GGC) qui se rajoute aux autres de façon consécutive.
La séquence d'homologie de 20 pb est située dans la partie 5' non traduite de l'ARN messager de H5 et plus précisément à la position -113 en amont du codon d'initiation de la traduction d'après le p541 (Ruiz-Carrill et al., 1983).
g
f
1IT
B H-H
50 pb
Figure 3. Cartes de restriction des sous-clones pCHVIIl .8H/B et pCHIX0.6H/S. Les stratégies pour séquencer le fragment, Smal - BamHI de pCHVIIl.8H/B (A) et l'insertion de pCHIXO.6H/S (B) sont données (-►). Les symboles utilisés sont: HindiII (□), Smal (O), BamHI (•), Sali (▼), Alul (♦), Hpall (v), HaelII (◄), Hinfl (<), Sau3A (x).
ro
10 20 30 40 50 60 TCGACGGGAT CAGGGACTGG AGCCTTTTCA GCTGCCTCGG GGTCTGGCTG GACTGCTGTC
70 80 90 100 110 120
CCAAGGAACC CAAGACGGAC ACCGGGGGAC GGAAGATGGC TGTCGGGAGG CGCGGCGTGG
130 140 150 160 170 180
TGGGTCAGGG CTATAGCAAC AGAAGTGGTG •GCGGCTGCGG CTTGCAGGGG GGCCTGGATG
190 200 210 220 230 240
AGCGGGGTCC AAATGACGGG GGTGGGGGTG GATGCGGCAG CGGCGGCGGC GGCGGCGGCA
250 260 270 280 290 300
GCCTGGACGT TGTGAGCACA GTGTGCCATC TCCCTGTCGT GCTGCACGAT CTGCTGGATG
310 320 330 340 350 360
ATCTCATTCT CTTGGTAGTT GAAAACCCCC GAATTGAGGT CATGCTGGAC TTTATGCAGC
370 380 390 400 410 420
AGAATGGAAT TTTTCTTACC TGTAGAGAAA GTGAAGAGAG AAGAGTCATT CTCATGTGTC
430 440 450 460 470 480
AAAGCAGGGG AGGCTGCCTG CCGAACCTTC CTCAAATTGT TGGCACCGTC TCCGCTGTCA
490 500 510 520 530 540
TTATGGGATC TGGGTACAGT CTCTCTGCTC CAGCAACAAG ATGATAAACA TTATATCTTC
550 560 570 580 590 600
TGCTTTTGTG GTATGATCAG CGGACTTTGA GATCCCAGAG AACACAGTCC TAGTTAGACG
610 620 630 640
GTGGAGTCAG TTTTGTGAAG TGGGGCGGAC TGGTAGCAAA GCTT
Figure 4. Séquence de l'insertion de pCHIX0.6H/S. Elle débute au site SalI et se termine au site Hindlll. Le trait continu repré sente 1'homologie avec le p541 alors que le trait discontinu illus tre les répétitions de (GGC). C'est la séquence du brin porteur de 1‘homologie avec l'ARN messager de H5 qui est donnée.
GGCGCGGACG TCGCGGCGGG GAGGAGCCGG GCCGGGCCGG GGCGGATCGG CGCTCGGCGG
130 140 150 160 170 180
GCGGGCAGGG CACGGCGCGG CGCGATGGCG GGGCCCCGCG GCGGCGCTGC AGCGCGGGGT
190 200 210 220 230 240
CCCACGGAGG ATGTGAGGCG GCGGCCGCGG ACGGCCCCAC CGGGCTCGGA GGGCGGAGAG
GGGTCCC
Figure 5. Sequence de la région de 247 pb de pCHVI11.8H/B contenant 1'homologie avec le p541. Elle débute au site BamHI et s'achève au site Smal. Le trait continu représente 1'homologie avec le p541 alors que le trait discontinu illustre les répétitions de (GGC). C'est la séquence du brin porteur de 1'homologie avec l'ARN messager de H5 qui est donnée.
28
Entre ce qui a été séquencé de pCHIX0.6H/S et pCHVII1.8H/B, il n'y a que 21 pb communes comprenant les 5(GGC) en tandem. Ce résultat est d'ailleurs corroboré par l'hybridation de 1'insertion du pCHIX0.6H/S avec des fragments de restriction de ACHVII tel qu'illustré à la figure 6. Les fragments positifs observés sont les mêmes que pour l'hybridation avec le p541 (figure 2).
2.3.5 Identification de (GGC)n dans d'autres clones génomiques
En plus des deux clones ACHVII et aCHIX précédemment caracté risés, on a recherché d'autres clones génomiques porteurs de séquences simples de (GGC)n. L'insertion du pCHIXO.6H/S a été utilisée pour examiner par criblage des plages de lyse, d'autres phages ayant donné un signal de type B lors du criblage initial avec 1'insertion du p541. Cinq phages positifs ont été obtenus après deux purifications d'un petit nombre de phages ayant donné un signal avec la sonde de pCHIX0.6H/S.
Les ADN recombinants de AII, V, X, XIV et XIX sont tous différents à en juger d'après leurs patrons de digestion avec EcoRI tel qu'ils apparaissent après électrophorèse sur gel d'agarose (figure 7A). On remarque cependant que le AXIX est identique au ACHVII caractérisé anté rieurement (voir figure 2A, canal j).
On a hybridé 1'ADN digéré de chacun de ces clones avec
1'insertion du pCHIX0.6H/S (figure 7B) et celle du p541 (figure 7B ' ). On observe que pour chaque recombinant, c'est le même fragment d'ADN qui hybride avec les deux sondes. Pour AXIV, un deuxième fragment de plus grande taille (environ 5.20 kb) apparaît après surexposition des
autoradiogrammes (résultat non montré). Il est à noter que la bande plus faible qui apparaît dans le canal de AX avec la sonde du p541 (canal c' ) ne correspond à aucune bande sur le gel et serait plutôt issue d'une
contamination; d'ailleurs une expérience analogue a été réalisée et cette bande n'est pas apparue.
a b c d e f g abc d e f g
A B
Figure 6. Détermination de la région d'homologie de XCHVII avec le
pCHIX0.6H/S. A) Coloration au bromure d'éthidium des fragments de XCHVII séparés par électrophorèse sur gel d'agarose à 0.5%. (a) à (g) 0.75 wg d'ADN digéré respectivement avec Hi ndl 11, Hi ndl I I+BamHI, BamHI, Kpnl, KpnI+EcoRI, Smal, Smal+EcoRI. B) Autoradiogramme du transfert du gel montré en (A) hybridé avec l'insertion de pCHIX0.6H/S marquée par "nick-
translation". La longueur des fragments de 1'ADN de X digéré avec Hindl11, utilisé comme marqueur de migration est donnée en kb.
30
a b c d e a b c d e a' b' c" d' e' a' b' c* d' e‘ 2 3.1 9.4 6.6 4.4 2.3 2.0 A B A' B'Figure 7. Caractérisation de dones génomiques porteurs de séquences simples de (GGC)n . A,A') Coloration au bromure d'éthidium des fragments d'ADN de clones génomiques digérés avec EcoRI et séparés par électrophorèse sur gel d'agarose à 0.5%. (a) à (e) Ail (0.5 ug); AV, AX (0.3 pg); AXIV (0.7 pg); AXIX (0.6 pg). (a1) à (e1) Ail, AV, AX, AXIV, AXIX (0.5 pg). B,B') Autora
diogrammes des transferts des gels montrés en (A,A') hybridés avec les inser tions de pCHIXO.6H/S (B) et p541 (B1) marquées par "nick-translation". Les flèches indiquent la position sur les gels des bandes correspondantes ayant hybridé avec les sondes. La longueur des fragments de 1'ADN de A digéré avec
Étant donné qu'il n'y a que 20 pb homologues, dont (GGC)g, entre les insertions de pCHIX0.6H/S et du p541, le fait d'observer un signal avec chacune de ces sondes pour le même fragment d'ADN révèle la présence d'une séquence similaire, sans toutefois qu'elle soit nécessai rement identique. Cependant, on ne peut pas comparer les intensités de la même bande avec les 2 sondes car les gels ne sont pas identiques. Il faut aussi noter que les hybrides n'ont pas subi les mêmes conditions de lavage (lavage jusqu'à 0.1X SSCPE - 0.1% SDS/65°C (figure 7B, pCHIX0.6H/S) et jusqu'à 0.2X SSCPE - 0.1% SDS/65°C (figure 7B', p541) ).
En examinant chacun des 2 autoradiogrammes (figure 7B, B'), on se rend compte que les intensités des différentes bandes ne sont pas les mêmes. Les intensités relatives des bandes examinées les unes par rapport aux autres varient dans certains cas selon la sonde utilisée. On voit par exemple que 1'ADN de AX (c) donne un signal d'intensité comparable à celui de
àV (b) avec la sonde du p541 (fig. 7B‘) alors qu'avec l'autre sonde (fig. 7B),
il donne un signal plus faible par rapport à xV (b). Cela signifie donc des homologies différentes avec chacune des sondes. D'autre part, dans le cas de aXIV (canaux d, d'), les signaux sont nettement plus faibles dans les 2 cas et pourraient donc provenir d'une séquence moins riche en (GGC).
Les résultats de ces hybridations différentielles suggèrent la présence de séquences simples de (GGC)n dans les recombinants analysés. Il est cependant impossible de déterminer le nombre de répétitions consécu tives seulement d'après ces résultats car la méthode de détection est limi tée en raison des sondes utilisées: il y a 5 (GGC) chez le p541 et 6 chez le pCHIX0.6H/S. Lors de 1'hybridation avec ces sondes, un nombre plus grand de répétitions consécutives de (GGC) dans une séquence engendre de mauvais appariements et n'est pas détecté comme tel. Par exemple, une sé quence de (GGC)g ne pourra apparier que 6 de ses (GGC) avec le pCHIX0.6H/S et 5 avec le p541. En outre, un changement d'une base au milieu d'une sé quence de (GGC)g engendrerait de mauvais appariements qui la rendrait plus difficilement détectable avec les sondes utilisées.
32
2.3.6 Analyse de séquence
On a d'abord essayé de déterminer des cadres de lecture ou verts dans les séquences trouvées. On a assigné par ordinateur des codons de traduction correspondant aux trois phases de lecture possibles pour cha cun des deux brins. Dans la séquence d'un des brins de pCHVII1.8H/B, il existe trois cadres de lecture ouverts alors que chez le pCHIX0.6H/S, plu sieurs codons de terminai son ont été rencontrés dans chacun des cadres de lecture et ce, sur les deux brins.
Par la suite, on a regardé si ces séquences pouvaient par ha sard correspondre à quelque chose de connu en effectuant une recherche ex haustive dans les banques de séquences de "GenBank" et EMBL ainsi que dans la littérature. Cependant, elles ne correspondent à aucune séquence connue jusqu'à présent.
signal de type B à l'hybridation avec le p541 sont différents d'après les patrons des digestions enzymatiques obtenus, visibles par électrophorèse sur gel (XXIX est identique à XCHVII). Ils correspondent par conséquent à des portions distinctes du génome de poulet.
Les régions d'homologie de 20 pb des XCHVII et XCHIX avec le p541 comprennent une séquence simple de trinuclëotides (GGC). En outre, les séquences de ces régions sont les mêmes chez les deux clones, ce qui est en accord avec les intensités d'hybridation des fragments d'ADN posi tifs qui sont du même ordre de grandeur (comparer les canaux (a-i) pour XCHIX avec (j-1) pour XCHVII sur le même autoradiogramme à la figure 2).
Des séquences simples formées de (GGC) semblent être répétées plusieurs fois dans le génome de poulet 5 en juger par les hybridations différentielles des sondes de pCHIX0.6H/S et p541 avec d'autres clones de type B provenant du criblage initial de la librairie. On peut dire que le nombre suggéré de répétitions de séquences (GGC)n comme celles portées par les clones de type B pourrait être de 39 puisque les 77 clones de type B obtenus proviennent du criblage de la librairie représentant environ deux fois la taille du génome. De même, il pourrait y avoir 516 * 2 d'où 258 répétitions de séquences de (GGC)n de type C où par rapport à celles des clones de type B, il y aurait moins de (GGC) consécutifs détectables avec la sonde utilisée. Cependant, ces chiffres donnés à partir du criblage de la librairie demeurent très approximatifs. D'abord la représentation géné rale des séquences dans la librairie, surtout si elle a été amplifiée, peut être fort différente de celle du génome. Certaines séquences peuvent être sous ou sur-représentées dépendant de leurs effets sur la viabilité des
34
phages. D'autre part, 11 intensité des signaux observés peut dépendre aussi du nombre de phages présents dans 1 es plages de lyse qui ont donné un si gnal avec la sonde. Cela pourrait donc fausser quelque peu le décompte des clones pour chacun des 3 types de signaux (A, B, C) obtenus lors du cribla ge de la librairie avec 11 insertion du p541. Une meilleure évaluation du nombre d'éléments de la famille (GGC)n dans différents génomes sera faite au chapitre suivant.
Une homologie de 14 pb comprenant (GGC)g (provenant du gène de H5; discuté au chapitre suivant) avec le pCHIX0.6H/S et le pCHVIII.8H/B n'est pas détectable par hybridation dans les conditions habituelles et lavage jusqu'à 0.2X SSCPE - 0.1% SDS/65°C (résultats non montrés). On a vu par contre que les hybridations différentielles avec le pCHIX0.6H/S et le p541 détectent bien une séquence de 20 pb homologues comprenant (GGC)g; c'est le cas de ACHVII. Par conséquent, nous pouvons dire qu'il faut au moins plus qu'une séquence homologue de 14 pb avec (GGC)] pour être détec table dans ces conditions. Les signaux d'hybridation des clones de type B obtenus lors du criblage initial de la librairie et résistant à des condi tions de lavage de 0.IX SSC - 0.1% SDS/65°C répondraient apparemment à ces exigences. Cependant, on ne sait pas si les clones porteurs d'une homolo gie intermédiaire, soit entre 14 et 20 pb auraient donné un signal avec la méthode de détection utilisée. La limite inférieure où on commence à dé tecter un signal d'homologie n'a pas été déterminée.
ORGANISATION DES SÉQUENCES SIMPLES DE (GGC)
3.1 Introduction
Parmi les séquences simples rapportées dans la littérature, il y en a qui ont été caractérisées soit à cause de leur présence dans le voi
sinage ou dans les introns de certains gènes, soit aussi par leur fréquence de répétition dans des génomes homologues ou hétérologues. Nous décrirons ici quelques exemples.
Plusieurs types de séquences simples ont été découverts dans le voisinage ou à 11 intérieur d'une variété de gènes. Ainsi, Slightom et al. (1980) ont trouvé des séquences de 13 à 19 (TG) consécutifs dans le deuxième intron des gènes foetaux G et A de type g-globine chez 11 humain. Apparemment, ces séquences joueraient un rôle dans la conversion intergé nique afin de contrôler l'évolution de ces gènes qui sont des produits de duplication d'un ADN ancestral . Des trinuclêotides (TCC)n et (TCA)n ont été trouvés par Cohen et collaborateurs (1982) à des positions analogues, en amont de 5 gènes d'immunoglobulines Vy apparentés, chez la souris. Ils auraient eux aussi un rôle dans la conversion puisqu'en amont de ces sé quences, ces gènes divergent. Chez l'oursin de mer (S. purpuratus), Sures et al. (1978) ont trouvé dans un "spacer" entre les gènes H2A et Kl, une séquence de (CT/GA)27. En parlant de cette séquence, Kedes (1979) suggère qu'elle pourrait, à cause de son homogénéité, se comporter comme une ferme ture éclair lors de sa dissociation-réassociation. Elle pourrait de ce fait minimiser les crossing-over inégaux hors phase mais permettre ceux qui sont en phase. Ce serait un mécanisme pour la correction et l'amplifica tion des gênes.
D'autres auteurs ont étudié le caractère répétitif des séquen ces simples. Hamada et Kakunaga (1982) ont observé la séquence (TG)25 dans le quatrième intron d'un gêne d'actine du muscle cardiaque humain. Cette séquence est située près de la frontière intron/exon. Avec une sonde synthétique de (dT-dG)n, ils ont déterminé qu'il y avait environ cent mille copies de ce motif dans le génome humain. Ces séquences fortement
répétées pourraient, selon ces auteurs, être importantes dans la mutagënê- se, la recombinaison ou le contrôle de 1'expression génique. À 329 pb de la partie 3' codante de deux gènes d'immunoglobulines V provenant de cellules embryonnaires et de plasmacytomes de souris, Nishioka et Leder (1980) ont trouvé la séquence (CA)3i. Une séquence de composition asymé trique en purines-pyrimidines, (AGAGG)32, a été identifiée quant à elle par Dybvig et al. (1983) dans un clone génomique de poulet de nature inconnue. Ce clone a été obtenu par hybridation avec des sondes d'ADN complémentai res enrichis pour les ARN messagers du choc thermique. Ils estiment à au moins 2 000 le nombre de copies de cette séquence simple d'après la quanti té de plages provenant du criblage de la librairie.
Il a aussi été démontré que ces séquences étaient répandues dans des systèmes biologiques hétérologues. La séquence simple (TG)-|y, présente dans un segment de 251 pb entre les gênes s et g-globines humaines est détectable chez 20% des clones d'une librairie génomique humaine (Mi es te! d et al., 1981). Cette séquence, retrouvée dans les génomes de Xenopus, de saumon, de pigeon, de souris, de levure {S_. cerevisae) et de myxomicête (JD. discoideum), pourrait avoir été conservée ou simplement avoir été générée indépendamment durant l'évolution. Gebhard et Zachau (1983) ont identifié les séquences (TGjgQ, (TTTGCjgQ, (GCCTCT)3q dans le voisinage des gènes VK et CK codant pour les chaînes de type k dans les immunoglobu lines de souris. Le caractère répétitif observé chez la souris est conservé chez le saumon. Finalement, Maroteaux et al. (1983) ont étudié la fréquence de répétitions de séquences simples trouvées dans le voisinage ou dans les gènes codant pour des protéines de l'oeuf chez trois espèces aviaires. Ainsi, les pentanucléotides (GGAAG^y, (GGAAA^g et (GGAGA^g ont été trouvés res pectivement à 2.5 kb en amont de l'extrémité 5' du gène ovalbumine-X du poulet. Ces séquences sont réitérées dans trois génomes aviaires (poulet, faisan, canard) à des degrés qui varient avec le type de séquence et l'espèce considérée.
38
Nous avons voulu étudier 1'organisation et la réitération des séquences simples composées du trinuclëotide (GGC), d'abord chez le poulet puis dans d'autres espèces qui lui sont plus ou moins apparentées afin de voir si elles sont présentes chez d'autres organismes. Pour ce faire, il a d'abord fallu établir les conditions optimales de détection de ces
séquences par hybridation différentielle.
Suite à cela, il s'est avéré intéressant d'identifier parmi les gênes séquencés chez quelques espèces, ceux qui étaient porteurs de ce type de séquences afin d'étudier leur localisation dans le génome.
3.2 Matériel et méthodes
3.2.1 Matériel
Les poulets utilisés sont de type Warren (Québec) et White Leghorn (Allemagne), le canard est de la variété Pékin et le pigeon de type voyageur. Les ADN de foie de souris Bal b/c ainsi que de cellules T24 de carcinomes de vessie humaine ont été généreusement fournis respectivement par B. Turcotte et C. Parent et isolés par traitement à la protéinase K-SDS suivi d'extractions au phénol et chloroforme tel que décrit plus bas. Il en est de même pour 11 ADN de salmonelle (S_. thyphimurium) fournit par A. Ruiz- Carrillo. Quant à 11 ADN de E. coli, il a été isolé par gradients de CsCl
L'ADN de sperme de saumon ainsi que le poly (G) proviennent de Boëhringer-Mannheim.
3.2.2 Méthodes
- Courbe de fusion
Pour mesurer le Tm de l'hybride formé entre 11 insertion du pCHIX0.6H/S et celle du p541, on a d'abord fixé 1'insertion purifiée de pCHIX0.6H/S sur 2 disques de nitrocellulose de 0.9 mm (d.i.) selon un protocole adapté de Kafatos et al. (1979): 1.3 yg d'ADN préalablement dénaturé (0.3 M Na0H/10 min/20°C suivi d'une dilution dans 1 vol. de 2 M CH3COONH4 froid) est déposé sur un disque humidifié dans 20X SSCPE. Le filtre est lavé 2 fois avec 50 yl de 20X SSCPE et il est mis à sécher au four à vide à 80°C/2 h. La pré-hybridation et 1'hybridation des filtres avec l'insertion du p541 marquée aux extrémités 5' avec la polynucléotide kinase de T4 ont été réalisées tel que décrit auparavant (section 2.2.2). On a ensuite fait 4 lavages dans 3X SSCPE - 0.1% SDS - lXD/temp.ambiante/15 min et 2 lavages dans IX SSCPE - 0.1% SDS/65°C/15 min. Les 2 filtres ont été déposés séparément dans des vials de plastique contenant 5 ml de 0.IX SSCPE - 0.1% SDS dans un bain à 45°C et stabilisés pendant 5 min. On a
40
alors commencé la dissociation des hybrides par l'augmentation graduelle de la température. Le liquide de lavage a été prélevé et compté par scintil lation. On a augmenté de 3°C la température du bain et on a ajouté 5 ml de la solution de lavage prê-chauffëe qui a été laissée en contact pendant 2 min avec le filtre. On a répété cela jusqu'à 87°C en parallèle avec un filtre témoin. A la fin, les comptes restants sur le filtre ont également été déterminés par scintillation. La somme des comptes ëluës à chaque température et des comptes restants, corrigés avec la valeur du témoin correspond aux comptes totaux présents sur le filtre avant lavage avec 0.IX SSCPE - 0.1% SOS. La courbe du pourcentage des hybrides dissociés
comptes ëluës corrigés \
_________________________ JX 100 en fonction de la température a été tracée. comptes totaux corrigés/
La déviation standard sur la lecture de la température est de ± 0.5°C alors que la déviation standard moyenne sur le calcul du pourcentage des hybrides dissociés est de - 0.7°C.
- Isolement de 1'ADN génomique
Les ADN génomiques d'érythrocytes de poulet, de canard et de pigeon ont été isolés comme suit. Les érythrocytes provenant de sang hëparinë ont été lavés plusieurs fois dans une solution isotonique (0.15 M NaCl - 10 mM Tri s-HCl pH 7.3) puis lysés dans un grand volume de solution 0.25 M sucrose - 0.14 M NaCl - 0.02 M Tri s-HCl pH 7.5 - 0.01 M EDTA - 0.5%
(v/v) NP-40. Après centrifugation à 800 g/5 min/4°C, la lyse a été reprise lorsque nécessaire. Les noyaux ont été lavés dans le même tampon en absen ce de NP-40, centrifugés à 800 g/5 min/4°C et mis en suspension dans 250 vol. de ce dernier auquel de la protéinase K et du SDS ont été ajoutés pour avoir respectivement des concentrations finales de 50 yg/ml et 1%. L'incu bation a été faite à 37°C de 16 à 20 h. L'étape suivante a consisté à faire plusieurs extractions consécutives, très douces, au phénol/chloro forme de façon à éliminer les protéines. L'ADN a été précipité et laissé gonfler dans du TE puis, précipité à nouveau. Un dosage spectrofluorimé trique au DABA a été réalisé.