CONCEPTION DE NOUVEAUX INHIBITEURS
DE LA MATRIPTASE-2
Par
Méryl-Farelle Oye mintsa mi-mba
Mémoire présenté au département de pharmacologie-physiologie en vue de l’obtention du grade de maître ès en sciences (M.Sc.)
Faculté de Médecine et des sciences de la santé Université de Sherbrooke
Sherbrooke, Québec, Mars 2020
Membres du jury Professeur Éric Marsault
Directeur de recherche
Département de pharmacologie-physiologie Professeur Richard Leduc
Co-directeur de recherche
Département de pharmacologie-physiologie Professeur Jean-Bernard Denault Évaluateur interne au programme Département de pharmacologie-physiologie
Professeure Brigitte Guérin Évaluatrice externe au programme
RÉSUMÉ
Conception de nouveaux inhibiteurs de la matriptase-2 Par
Méryl-Farelle Oye mintsa mi-mba Département de pharmacologie-physiologie
Mémoire présenté à la Faculté de médecine et des sciences de la santé en vue de l’obtention du diplôme de maitre ès en sciences (M.Sc) en pharmacologie, Faculté de médecine et des sciences de la santé, Université de Sherbrooke, Sherbrooke, Québec,
Canada, J1H 5N4
La matriptase-2 est une enzyme appartenant à la famille des protéases à sérine transmembranaires de type II. Elle est localisée à la surface des hépatocytes. Plusieurs études ont montré que cette enzyme agissait comme un régulateur dans l’homéostasie du fer en diminuant les niveaux d’hepcidine, hormone centrale du métabolisme du fer, via le clivage de l’hémojuvéline favorisant le relargage du fer emmagasiné dans les cellules dans la circulation. L’inhibition de cette enzyme ouvre la voie vers le traitement des maladies caractérisées par une surcharge en fer comme la bêta-thalassémie et l’hémochromatose juvénile.
Sur la base des inhibiteurs connus de la matriptase, une enzyme structurellement homologue de la matriptase-2, notre laboratoire avait conçu et synthétisé des inhibiteurs de type peptidomimétiques (P4-P3-P2-P1-kbt) pour la matriptase-2. Ces inhibiteurs sont
caractérisés par un groupement cétobenzothiazole (kbt) en position P1, qui agit comme
piège à sérine, et une séquence (P4-P3-P2) d’acides aminés non-naturels. À l’heure
actuelle, un inhibiteur puissant et sélectif pour matriptase-2 ((H)F-hF-AlloThr-R-kbt) a été caractérisé par notre groupe de recherche.
Le but de ce projet de maitrise était d’identifier de nouveaux inhibiteurs plus puissants et sélectifs pour la matriptase-2 par rapport à la matriptase en modifiant les positions P4, P3
et P2 des inhibiteurs, ainsi que de mieux caractériser la relation structure-activité de cette
classe de composés. Nous nous sommes basés sur les travaux déjà effectués, ainsi que de la structure cristalline de la matriptase en complexe avec un composé de notre librairie ((H)F-(Thizaol-4-yl)A-V-R-kbt). Un modèle d’homologie de la matriptase-2 a ainsi été créé pour mettre en évidence les différences de résidus présents au niveau des pochettes de liaison des deux enzymes.
Cela a servi de point de départ afin d’émettre des hypothèses pour modifier chacune des positions. Ces modifications ont conduit à une librairie de 47 composés permettant d’avoir une meilleure compréhension de la relation structure-activité de ce type d’inhibiteurs sur la matriptase-2. Nous avons caractérisé les composés obtenus en mesurant leurs constantes d’inhibition (Ki) sur matriptase et matriptase-2.
Finalement, dans l’éventualité d’une application thérapeutique des composés de ce type, nous avons envisagé la conception d’une prodrogue afin de réduire les problèmes liés à l’utilisation des peptides (faible distribution, difficultés à franchir les barrières épithéliales, dégradation gastro intestinale, polarité élevée etc.). Nous reportons ici une
méthode de synthèse de cette prodrogue et son évaluation en essais biochimiques sur les enzymes matriptase et matriptase-2.
Mots clés : matriptase-2 ; cétobenzothiaziole ; inhibiteurs ; slow-tight binding ; matriptase ; TTSP ; peptidomimétique ; prodrogue.
SUMMARY
Design of new inhibitors of matriptase-2 By
Méryl-Farelle Oye mintsa mi-mba Department of pharmacology-physiology
Thesis presented to the faculty of medicine and health sciences for the obtention of the degree of Master of Science (M.Sc) in pharmacology, faculty of medecine and health
sciences, Université de Sherbrooke, Sherbrooke, Québec, Canada, J1H 5N4 Matriptase-2 is an enzyme that belongs to the family of type II transmembrane serine protease. It is located on the surface of hepatocytes. Several studies have shown that this enzyme acts as a regulator of iron homeostasis by reducing the levels of hepcidin, the hormone that regulates iron metabolism. This occurs via the clivage of hemojuvelin, which promotes the release of iron stored in the cells into the circulation. Inhibition of this enzyme opens the way for the treatment of diseases characterized by iron overload, such as beta-thalassemia and juvenile hemochromatosis.
Based on known inhibitors of matriptase, an enzyme structurally homologous to matriptase-2, our laboratory had designed and synthesized peptidomimetic-type inhibitors (P4-P3-P2-P1-kbt) for matriptase-2. These inhibitors are characterized by a
ketobenzothiazole group (kbt) at the P1 position, which acts as a serine trap, and a
sequence (P4-P3-P2) of non-natural amino acids. Currently, a potent and selective
inhibitor for matriptase-2 ((H)F-hF-AlloThr-R-kbt) has been characterized by our group. The goal of this project was to identify novel inhibitors that are more potent and selective for matriptase-2 compared to matriptase, by modifying the P4, P3 and P2 positions of
inhibitors. Based on the work already done, we used the crystal structure of matriptase in complex with a compound from our library ((H)F-(Thizaol-4-yl)A-V-R-kbt) to create a homology model of matriptase-2, in order to highlight the differences of residues in the binding pockets of both enzymes.
This was used as a starting point to hypothesize modifications for each position. These modifications led to a library of 47 compounds allowing a better understanding of the structure-activity relationship of this type of inhibitors on matriptase-2. We characterized the compounds obtained by measuring their inhibition constants (Ki) on matriptase and
matriptase-2.
Finally, with the perspective of a therapeutic application of compounds of this type, we have considered the design of a prodrug in order to reduce issues associated with the use of peptides (poor distribution, difficulties in crossing epithelial barriers, gastrointestinal degradation, high polarity, etc.). Here, we report a method of synthesis of this prodrug and its evaluation in biochemical assays on the enzymes matriptase and matriptase-2. Keywords: matriptase-2 ; ketobenzothiazole ; inhibitors ; slow-tight binding ; matriptase ; TTSP ; peptidomimetic; prodrug.
TABLE DES MATIÈRES
Résumé………...i
Summary………..iii
Table des matières………....iv
Liste des tableaux ...………...vii
Liste des figures ...………...viii
Liste des abréviations ………...………...xi
Dédicace ………...xv
Épigraphe ………..…xvi
1 INTRODUCTION ...1
1.1 LES ENZYMES : DES PROTEINES AUX PROPRIETES CATALYTIQUES ... 1
1.1.1 Généralités ... 1
1.1.2 La structure des enzymes ... 2
1.2 LES DIFFERENTES CLASSES D’ENZYMES ... 2
1.3 LES PROTEASES A SERINE ... 6
1.3.1 La structure « trypsin-like » des protéases à sérine ... 7
1.3.2 Les protéases à serine transmembranaires de type II (TTSP) ... 10
1.3.3 La sous-famille matriptase ... 13
2 LA MATRIPTASE-2 ET LE FER ...14
2.1 LE FER DANS L’ORGANISME ... 14
2.2 L’HEPCIDINE : UNE HORMONE REGULATRICE DES NIVEAUX DE FER ... 16
2.3 LA MATRIPTASE-2 DANS L’ORGANISME ... 19
3 LES MALADIES ASSOCIEES A LA SURCHARGE EN FER ...21
3.1 L’HEMOCHROMATOSE ... 21
3.1.1 L’hémochromatose juvénile ... 22
3.1.2 Traitements de l’hémochromatose ... 22
3.2 LES THALASSEMIES ... 24
3.2.1 Les b-thalassémies ... 24
3.2.2 Les traitements des b-thalassémies ... 25
4 LES INHIBITEURS ...26
4.1 LES INHIBITEURS REVERSIBLES ... 26
4.2 LES INHIBITEURS IRREVERSIBLES ... 30
4.3.1 Les inhibiteurs lents (de type « slow ») ... 32
4.3.2 Les inhibiteurs de type « tight » ... 33
4.4 LES INHIBITEURS DE LA MATRIPTASE-2 ... 34
4.4.1 Les inhibiteurs naturels ... 34
4.4.2 Les inhibiteurs synthétiques ... 35
5 LES PRODROGUES ...39 5.1 LE PRINCIPE DE LA PRODROGUE ... 41 6 PROJET DE RECHERCHE ...44 7 MATERIEL ET METHODES ...45 7.1 MODELISATION MOLECULAIRE ... 45 7.2 REACTIFS CHIMIQUES ... 45 7.3 REACTIFS BIOLOGIQUES ... 45 7.4 ÉQUIPEMENTS ... 46
7.4.1 Équipements pour la phase solide ... 46
7.4.2 Appareil de chromatographie ultraperformante couplée à un spectromètre de masse (UPLC-MS) ... 46
7.4.2.1 Chromatographie à haute performance préparative (HPLC-préparative) ...46
7.4.2.2 Lecteur de plaques ...47
7.5 SYNTHESE DU PIEGE A SERINE ... 47
7.6 SYNTHESE DES TRIPEPTIDES ... 50
7.7 SYNTHESE DES TETRAPEPTIDES ... 51
7.8 COMPOSES DE REFERENCE UTILISES ... 52
7.9 SYNTHESE DE LA PRODROGUE ... 54
8 RESULTATS ET DISCUSSION ...63
8.1 ANALYSE DES POCHETTES DE LIAISONS DES ENZYMES ... 63
8.2 MODIFICATIONS EN POSITION P2 ... 79
8.3 MODIFICATIONS EN P2 ET P3 ... 85
8.4 MODIFICATIONS EN POSITION P4 ... 88
8.5 MODIFICATIONS EN POSITIONS P3 ET P4 ... 94
8.6 MODIFICATIONS EN POSITIONS P2,P3 ET P4 ... 96
8.7 ÉVALUATION DE LA PRODROGUE SUR LES ENZYMES MATRIPTASE ET MATRIPTASE-2 IN VITRO ... 107
9 CONCLUSION GENERALE ET PERSPECTIVES ...109
Références ………....111
Annexe A ………….………...……….116
Annexe B ………….……….139
LISTE DES TABLEAUX
Tableau 1 Caractérisation des peptides synthétisés ...71
Tableau 2 Caractérisation des composés #1 à #11 ...79
Tableau 3 Modifications de la position P2 à partir de l’échafaudage 1 ...80
Tableau 4 Modifications des positions P2 et P3 à partir de l’échafaudage 2 ...86
Tableau 5 Modification de la position P4 à partir de l’échafaudage 3 ...89
Tableau 6 Modifications de la position P4 à partir de l’échafaudage 4 ...91
Tableau 7 Modifications des positions P3 et P4 à partir de l’échafaudage 5 ...95
LISTE DES FIGURES
Figure 1 Les différentes cibles thérapeutiques (Adapté à partir de la base de données
ChEMBL, 20185) ...1
Figure 2 Disposition des résidus de l’enzyme et de ceux de son substrat ...3
Figure 3 Différentes classes de protéases (Adapté de Deu et al., 201210) ...5
Figure 4 Différentes classes de protéases (Adapté de Deu et al., 201210) ...6
Figure 5 Passage d’une forme inactive à une forme active ...8
Figure 6 Mécanisme catalytique général des protéases à serine ...9
Figure 7 Structure générale des TTSP ...10
Figure 8 Familles des TTSP (Adapté de Bugge et al., 200920) ...12
Figure 9 Le cycle du fer (Adaptée de Beaumont, 200427) ...14
Figure 10 Homéostasie du fer ...15
Figure 11 Rôle de l’hepcidine dans la régulation du fer (Adaptée de Béliveau, 201239) ...16
Figure 12 Mécanisme d’action de l’hepcidine ...17
Figure 13 Voies de signalisation contrôlant l’expression d’hepcidine ...18
Figure 14 Structure de matriptase-2 (Adapté de Wang et al., 201447) ...19
Figure 15 Domaines structurels de matriptase-2 (Adapté de Ramsay et al., 200822). ...20
Figure 16 Rôle de matriptase-2 à la surface des hépatocytes (Adapté de Béliveau 201239) ...20
Figure 17 Chélateurs de fer ...24
Figure 18 Mécanisme d’inhibition compétitive ...27
Figure 19 Structure du captopril ...27
Figure 20 Mécanisme d’inhibition non-compétitive (Inspirée de Smith et al., 20054) ...28
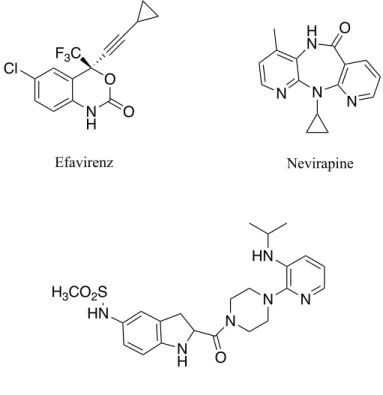
Figure 21 Structures de quelques NNRTI approuvés par la FDA ...29
Figure 22 Mécanisme d’inhibition incompétitive (Inspirée de Smith et al., 20054) ..30
Figure 23 Structure de la camptothécine ...30
Figure 24 Mécanisme d’inhibition irréversible (Inspirée de Smith et al., 20054). ...31
Figure 25 Structure du DFP ...32
Figure 26 Mécanisme proposé pour l’inhibition de type « slow binding » ...32
Figure 27 Structure de la conformycine ...33
Figure 28 Mécanisme proposé pour l’inhibition de type « slow tight binding » - ...33
Figure 29 Structure du SB-23476474. ...34
Figure 30 Structures des inhibiteurs HAI-1 (a) et HAI-2 (b) ...34
Figure 31 Structure de SFTI-1, un peptide isolé des graines de tournesol ...35
Figure 32 Dérivés de benzamidines82,85 ...36
Figure 33 Mécanisme d’inhibition proposé ...37
Figure 34 Composés YYVRkbt et LWWRkbt ...38
Figure 35 Inhibiteurs de matriptase-281 ...39
Figure 36 Structure de l’énalaprilat et de sa prodrogue l’énalapril. ...40
Figure 37 Passage de la prodrogue à la forme active ...40
Figure 38 Structures du captopril et de l’énaprilat ...41
Figure 39 Principe de la prodrogue (Adapté de Rautio et al., 2013) ...41
Figure 40 Structure d’un hexapeptide cyclique (1a) et de sa prodrogue (1b) (Adapté de Weinmüller et al., 201798 ) ...42 Figure 41 Structure d’un autre hexapeptide cyclique (1a’) et de sa prodrogue (1b’)
Figure 42 Synthèse du piège à serine ...48
Figure 43 Synthèse des tripeptides ...50
Figure 44 Synthèse des tétrapeptides ...52
Figure 45 Composé de référence A ...53
Figure 46 Composé de référence B ...53
Figure 47 Composé de référence C. ...54
Figure 48 Synthèse du groupement réactif sur une ornithrine (Synthèse adaptée de Colombo et al., 201280) ...54
Figure 49 Synthèse du de la guanidine avec le groupement Hoc (Synthèse adaptée de Weinmüller et al., 201798) ...58
Figure 50 Synthèse du piège à sérine inactif (Synthèse adaptée de Feichtinger et al., 1998104) ...59
Figure 51 Couplage du piège à sérine avec le tripeptide : Synthèse du composé inactif (Synthèse adaptée de Colombo et al., 201280) ...61
Figure 52 Modèle de la matriptase en complexe avec (H)F-(Thiazol-4-yl)A-V-Rkbt ...63
Figure 53 Modèle d’homologie de matriptase-2 avec (H)F-(Thiazol-4-yl)A-V-R-kbt arrimé dans la pochette de liaison ...64
Figure 54 (A-E) Représentation des composés synthétisés à partir des résultats d’arrimage moléculaire ...66
Figure 55 Criblage des quarante-sept composés sur la matriptase à une concentration de 10 µM ...73
Figure 56 Criblage des quarante-sept composés sur la matriptase-2 à une concentration de 10 µM ...74
Figure 57 Criblage des quarante-sept composés sur la matriptase à une concentration de 100 nM ...75
Figure 58 Criblage des quarante-sept composés sur la matriptase-2 à une concentration de 100 nM ...76
Figure 59 Criblage des quarante-sept composés sur la matriptase à une concentration de 2.50 nM ...77
Figure 60 Criblage des quarante-sept composés sur la matriptase-2 à une concentration de 2.50 nM ...78
Figure 61 Échafaudage 1 ...80
Figure 62 Composé de référence A arrimé dans le modèle d’homologie de la matriptase-2. ...81
Figure 63 Composé #1 arrimé dans le modèle d’homologie de la matriptase-2 ...82
Figure 64 Composé de référence A arrimé dans le modèle d’homologie de la matriptase-2. ...83
Figure 65 Composé #2 arrimé dans le modèle d’homologie de la matriptase-2 ...84
Figure 66 Composé #2 arrimé dans le modèle d’homologie de la matriptase-2 (distances) ...85
Figure 67 Échafaudage 2 ...85
Figure 68 Composé de référence A arrimé dans le modèle homologique de la matriptase-2. ...87
Figure 69 Composé #3 arrimé dans le modèle d’homologie de la matriptase-2. ...88
Figure 70 Échafaudage 3 ...89
Figure 71 Composé de référence B arrimé dans le modèle d’homologie de la matriptase-2. ...90
Figure 72 Composé #5 arrimé dans le modèle d’homologie de la matriptase-2 ...91
Figure 74 Composé de référence A arrimé dans le modèle homologique de la
matriptase-2 ...93
Figure 75 Composé #6 arrimé dans le modèle homologique de la matriptase-2 ...94
Figure 76 Échafaudage 5 ...95
Figure 77 Composé #7 arrimé dans le modèle homologique de la matriptase-2 ...96
Figure 78 Échafaudage 6 ...97
Figure 79 Composé de référence A arrimé dans le modèle homologique de la matriptase-2 ...98
Figure 80 Composé de référence B arrimé dans le modèle homologique de la matriptase-2 ...98
Figure 81 Vue de profile du composé #8 arrimé dans le modèle homologique de la matriptase-2. ...99
Figure 82 Vue de face du composé #8 arrimé dans le modèle homologique de la matriptase-2 ...100
Figure 83 Vue de face du composé #9 arrimé dans le modèle homologique de matriptase-2 ...101
Figure 84 Vue de dessous du composé #9 arrimé dans le modèle homologique de matriptase-2 ...102
Figure 85 Composé #10 arrimé dans le modèle homologique de la matriptase-2 .103 Figure 86 Composé #4 arrimé dans le modèle homologique de la matriptase-2. ..104
Figure 87 Composé #11 arrimé dans le modèle homologique de la matriptase-2 .105 Figure 88 Composé #11 arrimé dans le modèle du cristal de la matriptase ...106
LISTE DES ABRÉVIATIONS Å : angstrom
Abu : acide alpha-aminobutyrique ACN : acétonitrile
AcOEt : acétate d’éthyle
ADN : acide désoxyribonucléique Ala, A : alanine
Alloc : allyloxycarbonyl anh. : anhydre
Arg, R : arginine
Asp, D : acide aspartique
BMP 2, 4, 6 : bone morphogenic protein 2, 4, 6 Boc : tert-butyloxycarbonyl
BSA : bovine serum albumin
CaCo-2 : colon adenocarcinoma cell line CCM : chromatographie sur couche mince CD30 : protéine cluster de différenciation CDA : conjugaison drogue-anticorps Cha : cyclohexylalanine
COX 1, 2 : cyclo-oxygénase 1, 2
CUB : Cls/Clr urchin embryonic growth factor and bone morphogenic protein 1 Cys, C : cystéine
d : déplacement chimique d : doublet
DCM : dichlorométhane
DCYTB : duodenal cytochrome B DFO : desféroxamine DFRA : déférosirox DFP : diisopropyl fluorophosphate DIPEA : N,N-diisopropyléthylamine DMF : N,N-diméthylformamide DMP : Dess-martin periodinane DMSO : dimethyl sulfoxide DMT1 : dimetal transporter 1 Éq : équivalent
F (%) : biodisponibilité
FDA : food and drug administration Fe2+ : ion ferreux
Fe3+ : ion ferrique
Fmoc : Fluorénylméthoxycarbonyle Glu, E : acide glutamique
Gly, G : glycine
GPI : glycosyl-phosphatidylinositol h : heure(s)
H2O : eau
HAI-1 et -2 : hepatocyte growth factor inhibitor type-1 and -2 HAT : human airway trypsine-like protease
HATU : 1-[bis(Diméthylamino)méthylène]-1H-1,2,3-triazolo[4,5-b]pyridinium-3-oxide hexafluorophosphate
HCl : acide chlorhydrique HCP1 : heme carrier protein 1
HFE (gene) : gene codant pour la protéine de regulation du fer homéostatique HFE (protéine) : protéine de regulation du fer homéostatique
HFIP : hexafluoropropan-2-ol His, H : histidine
Hoc : hexyloxylcarbonyl hPhe, hF : homophénylalanine
(H)Phe, (H)F : désaminophénylalanine
HPLC : chromatographie liquide à haute performance Hz : hertz
IL-6 : interleukine-6 Ile, I : isoleucine
IRIDA : iron-refractory iron deficiency anemia i.v : injection par intraveineuse
J : constante de couplage JAK2 : janus kinase 2 k : constante cinétique Kbt : cétobenzothiazole
kDa : kiloDalton (masse moléculaire) Ki : constante d’inhibition
Km : constante de Michaelis l : longueur d’onde
L : litre
L1 : défériprone
LDLA : low density lipoprotein receptor class A Leu, L : leucine Lys, K : lysine m : méta µM : micromolaire m : multiplet M : molaire (mol/L)
MAM : merprin A5 antigen and receptor phosphatase µ Mat : matriptase
Mat-2 : matriptase-2 MeOH : méthanol mg : milligramme
mg/j : milligramme par jour MgSO4 : sulfate de magnésium
mL : millilitre
mL/kg : millilitre par kilogramme mM : millimolaire (mmol/L) MHz : mégahertz
min : minute(s)
MOE : molecular operating environement MS : mass spectrometry
MSTFA : N-Methyl-N-(trimethylsilyl)trifluoroacetamide MW : molecular weight
NaHCO3 : bicarbonate de sodium
NaOH : hydroxyde de sodium Na+-K+ : pompe sodium-potassium Na+-H+ : échangeur sodium-hydrogène
n-BuLi : n-Butyllithium NEt3 : N,N,N-triéthylamine
ng/mL : nanogramme par millilitre nM : nanomolaire (ng/L)
NMM : N-methylmorpholine
NNRTI : non-nucleoside reverse transcriptase inhibitor Nva : norvaline
o : ortho O2 : oxygène
OMS : organisation mondiale de la santé Orn : ornithine
p : para
Papp : perméabilité apparente
PAMPA : parallel artificial membrane permeability assay Pbf : 2,2,4,6,7-Pentamethyldihydrobenzofuran-5-sulfonyl PDB : protein data bank
PE : petrolum ether pH : potentiel hydogène Phe, F : phénylalanine p.o : prise orale
ppm : produit par millions Pro, P : proline
RMN 1H : spectre de résonance magnétique nucléaire du proton rpm : revolutions per minute
RSA : relation structure-activité s : singulet
SA, SB, SC, SE, SF, SG : superfamilles des protéases à sérine SEA : sea urchin protein enterokinase agrin
SCL40A1 : solute carrier family 40 member 1 Ser, S : sérine
SFTI-1 : sunflower trypsin inhibitor 1
SMAD : son of mother against decapentaplegic SRA : group A scavenger receptor
STAT3 : signal transducer and activator of transcription 3 Temp. amb. : température ambiantte
Tf : transferrine
TFA : acide trifluoroacétique Tf2O : anhydride triflique
TFR2 : transferrin receptor 2, gene TfR2 : transferrin receptor 2 THF : téttrahydrofurane Thr, T : thréonine TI : thalassémie intermédiaire TM : thalassémie majeure Tm : thalassémie mineure
TMS : triméthylsilyle tr : temps de rétention Trp, W : tryptophane
TTSP : type two serine protease
UPLC : chromatographie liquide à ultra haute performance Val, V : valine
VIH-1 : virus de l’immunodéficience humaine de type 1 Vmax : vitesse maximale de la réaction
Je dédie mes travaux de recherche à mon défunt oncle Jean-Martin, à mes parents et à mes frères qui ont toujours cru en moi.
Chenon men che saver, dubbiar m'aggrata Dante Alighieri
1 Introduction
1.1 Les enzymes : des protéines aux propriétés catalytiques 1.1.1 Généralités
De manière générale, les enzymes sont définies comme étant des protéines (ou plus rarement des ARN1) qui possèdent des propriétés catalytiques, c’est-à-dire des biomolécules capables d’accélérer des réactions chimiques spécifiques. L’énergie d’activation de la réaction catalysée est abaissée de façon à augmenter la vitesse de réaction et ainsi atteindre l’état final plus rapidement2,3,4. Les enzymes sont capables d’accélérer les réactions chimiques de différentes façons2.
Les quatre principales cibles thérapeutiques dans la découverte du médicament sont les récepteurs, les canaux ioniques, les transporteurs de molécules et les enzymes (Figure 1). Depuis plusieurs années, les enzymes prennent une place très importante dans l’industrie pharmaceutique.
Figure 1 Les différentes cibles thérapeutiques (Adapté à partir de la base de données ChEMBL, 20185) – Parmi les quatre cibles thérapeutiques les plus étudiées, on remarque que les enzymes prennent une grande place suivie des récepteurs couplés aux protéines
41%
22% 7%
4%
26%
Enzymes Recepteurs couplés aux protéines G
Canneaux ioniques Récepteurs nucléaires
Les médicaments qui ciblent les enzymes peuvent se comporter comme des inhibiteurs réversibles ou irréversible (ex : l’aspirine à faible dose inhibe les cyclo-oxygénases COX1 et COX2 irréversiblement)6. On distingue les inhibiteurs compétitifs, qui sont
parfois des analogues du substrat, des inhibiteurs non-compétitifs et incompétitifs3. Les différents types d’inhibiteurs seront abordés au point IV.
1.1.2 La structure des enzymes
Les enzymes se présentent comme des structures dynamiques. Elles sont alors le siège d’un ensemble de variations internes : du mouvement de résidus d’acides aminés individuels à un domaine entier de la protéine, en passant par un groupe de résidus conduisant à un élément de structure secondaire. Ces variations impliquent la formation d’ensembles de structures légèrement différentes les unes des autres. On peut ainsi associer différents états conformationnels des enzymes à différentes phases de leur activité chimique7.
L’environnement (pH, variations de force ionique, de température ou présence de partenaires d’interaction) peut avoir un impact sur l’activité enzymatique. L’impact de ces facteurs est caractérisé par la variation de vitesse de la réaction enzymatique. On nomme inhibiteurs et activateurs les composés qui conduisent respectivement à une diminution et à une augmentation de la vitesse de réaction8.
1.2 Les différentes classes d’enzymes
Chaque cellule possède des enzymes différentes spécialisées dans une réaction pour un substrat ou un groupe de substrats types. La nomenclature d’une enzyme se fait d’après une notation spécifique : le nom de la famille chimique du substrat suivi du nom de la réaction qu’elle catalyse et terminant par le suffixe –ase. De cette façon, on retrouve sept classes d’enzymes : les oxydoréductases, les transférases, les hydrolases, les lyases, les isomérases, les ligases et les translocases8,9.
Les hydrolases catalysent les réactions d’hydrolyse. Parmi cette classe, on retrouve les protéases, représentant 2% du génome humain, qui sont des enzymes clivant les liaisons
peptidiques des protéines ou des peptides. Certaines protéases sont produites sous forme de zymogène, qui est une forme inactive de l’enzyme. Ces zymogènes sont ainsi activés par une coupure protéolytique (clivage) pour libérer l’enzyme fonctionnelle (Figure 2). La forme zymogène d’une protéase permet de limiter son activité dans l’espace et dans le temps et a donc un rôle physiologique important. Par exemple, la trypsine est sécrétée par le pancréas exocrine sous forme de zymogène. C’est seulement lorsque ce zymogène atteint la bordure en brosse de l’intestin grêle qu’il est activé en trypsine par l’entérokinase (une autre protéase membranaire retrouvée uniquement au niveau de la bordure en brosse de l’intestin grêle), permettant alors à la trypsine de remplir sa fonction catalytique. Une activation zymogènique prématurée lors de la maturation de l’enzyme dans les cellules acinaires du pancréas exocrine mène à la pancréatite.
Figure 2 Disposition des résidus de l’enzyme et de ceux de son substrat – Les lettres Px correspondent aux résidus de l’enzyme tandis que Sx correspondent à ceux du substrat.
On a d’une part les exopeptidases qui permettent le clivage des liaisons peptidiques aux extrémités des chaînes. Elles sont divisées en deux catégories : les aminopeptidases et les carboxypeptidases. Les aminopeptidases clivent la liaison entre le premier et le second acide aminé pour libérer le N-terminal et les carboxypeptidases clivent la liaison entre l’avant dernier et le dernier acide aminé afin de libérer le C-terminal. D’une autre part, on retrouve les endopeptidases qui clivent les liaisons peptidiques à l’intérieur des chaînes8.
Les mécanismes d’action des protéases permettent de les classifier en différentes familles mécanistiques (Figure 3) dépendamment des résidus présents dans leurs sites de liaison8,10,11. Ainsi on retrouve les protéases à cystéine, dont certaines possèdent une diade catalytique contenant une cystéine et une histidine dans leur site actif, comme les caspases12 (Figure 4.D). Le soufre joue un rôle de nucléophile sous forme de thiolate13. On retrouve également les protéases à aspartate qui possèdent une diade composée de
N-term Pn P 3 P2 P1 P’1 P’2 P’3 P’n C-term Clivage Sn S3 S2 S1 S’1 S’2 S’3 S’n … … … …
activent conjointement une molécule d’eau qui joue le rôle de nucléophile (exemples : la pepsine ou la protéase du VIH-1). Les protéases à thréonine possèdent, quant à elles, une thréonine dans leur site actif et nécessitent aussi l’intervention d’une molécule d’eau afin d’effectuer leur rôle catalytique (Figure 3.A). Les métalloprotéases ont un cation métallique fixé à la protéine par des chaînes latérales de plusieurs acides aminés (Figure 3.C). Ce cation, qui est généralement un atome de zinc, intervient pour activer une molécule d’eau afin de cliver la chaîne peptidique. Enfin les protéases à sérine ont une triade catalytique qui comprend une sérine, une histidine et une aspartate, le nucléophile est le groupement hydroxyle qui procède à une attaque nucléophile sur le groupement carbonyle de la liaison peptidique (Figure 4.E). Le mécanisme d’action de ces protéases sera abordé au point 1.3.1.
Figure 3 Différentes classes de protéases (Adapté de Deu et al., 201210) – Représentation des différents mécanismes des protéases en fonction de leur classe : A. dans les protéases à thréonine, le mécanisme est enclenché par la présence d’une molécule d’eau. B. Dans les protéases à aspartate, un aspartate capture un atome d’hydrogène provenant d’une molécule d’eau afin d’enclencher le mécanisme. C. Dans les métalloprotéases le cation divalent zinc(II) active la molécule d’eau afin de débuter le mécanisme d’action. A. Protéases à thréonine N O O H O H H H H N H O P1 P’1 H3N O O O H H N H O P1 P’1 H2N O O O H H O P1 H3N O O O H O P1 H2N P’1 + B. Protéases à aspartate P1 O H N P’1 O O H O O OH H P1 OH H N P’1 O O O O O H H P1 O OH OH O O O P’1 H2N C. Métalloprotéases à zinc N HN NNH N NH Zn2+ O HO O H H P1 O H N P’1 N HN NNH N NH Zn2+ O O O H P1 O H N P’1 H N HN NNH N NH Zn2+ O O P1 O OH P’1 H2N
Figure 4 Différentes classes de protéases (Adapté de Deu et al., 201210) – Représentation des différents mécanismes des protéases en fonction de leur classe : D. dans les protéases à cystéine, la déprotonation du groupement thiol est effectuée par une histidine voisine. E. Dans les protéases à sérine, le mécanisme d’action est initié par l’acide aspartique en formant un relais de charge avec les deux autres résidus de la triade catalytique (histidine et serine), permettant de rendre la sérine catalytique plus nucléophile.
Il existe aussi des protéases à domaines multiples, des mécanismes non conventionnels et des exceptions, mais ils ne seront pas abordés dans cet ouvrage.
1.3 Les protéases à sérine
Cette classe d’enzymes est retrouvée chez tous les organismes vivants et représente 30% des protéases. Elles sont impliquées dans un grand nombre de processus biologiques (digestion, réponse immunitaire, coagulation sanguine, maturation des virus, etc).
D. Protéases à cystéine N H O P1 P’1 N H O P1 P’1 S H NH N S HN NH O P1 S N NH H OH O P1 S N NH OH H H2N P’1 O H H E. Protéases à serine N H O P1 P’1 N H O P1 P’1 O H N N H O O O N N H O O H O P1 O N N H O O H OH O P1 O N N H O O OH H H2N P’1 O H H
Il existe toutefois une différence dans la distribution des protéases à sérine chez les organismes vivants. Afin de distinguer les différentes protéases à sérine on parlera de « clans ». Six clans sont référencés dans la littérature : SA, SB, SC, SE, SF, SG ; basés sur la similarité de leurs structures. Chaque groupe est lui-même divisé en familles. On compte plus de 20 familles de protéases à sérine (S1 à S27). Ici, nous nous intéresserons au groupe SA qui est largement représenté chez les eucaryotes et dont les protéases à sérine sont à l’origine d’une grande variété de processus extracellulaires14,15,16.
Dans la nature, il existe sept formes de structures de repliement des protéases selon le réseau polypeptidique17. On distingue alors des formes :
(a) Pepsin-like (b) Retropepsin (c) Trypsin-like (d) Subtilisin-like
(e) Herpes virus (f) Papain-like (g) Thermolysin-like
Les protéases à sérine ont tendance à adopter deux principales structures : « trypsin-like » et « subtilisin-like » mais la plupart vont adopter une conformation « trypsin-like ». Cette structure est le résultat de deux tonneaux b. On retrouve une triade catalytique qui se regroupe à l’interface des deux domaines. La seconde structure de repliement est une tri-couche aba repliée en « sandwich » (repliement Rossmann). Ici, on retrouve la sérine catalytique et l’histidine situées à la fin des hélices adjacentes. Pour une minorité de protéases à sérine, la structure « herpes virus » est retrouvée comme sept brins, pour la plupart anti-parallèles, et comme un tonneau b entourée de sept hélices8,17.
1.3.1 La structure « trypsin-like » des protéases à sérine
Chez l’être humain, la structure « trypsin-like » est adoptée pour six groupes fonctionnels (digestion, coagulation, tryptase, matriptase, kallikreine et granzymes).
L’activation de la plupart des protéases à sérine de ce type structurel nécessite la transformation d’un précurseur inactif de forme zymogène (Figure 5).
Figure 5 Passage d’une forme inactive à une forme active – L’exemple de la conversion du trypsinogène (forme inactive) en trypsine (forme active) après clivage.
Le clivage du précurseur inactif se produit à une position identique pour tous les membres d’une même famille. Le N-terminal nouvellement formé conduit à un changement de conformation dans l’enzyme (Figure 5). Cela passe par la formation d’un appariement d’ions avec un résidu Asp proche de la triade qui permettra l’organisation de la cavité de l’oxyanion et du site de liaison du substrat.
Après activation de l’enzyme s’ensuit un mécanisme catalytique impliquant la triade catalytique His, Asp, Ser18,15. Ces résidus se trouvent à proximité dans l’espace sans pour autant être voisins immédiats dans la séquence primaire.
Trypsinogène Trypsine Entéropeptidase Val-(Asp)4-Lys-Ile 1 6 7 Ile 7 Val-(Asp)4-Lys 1 6
Figure 6 Mécanisme catalytique général des protéases à serine – Le mécanisme d’action des protéases à serine se traduit par une succession d’étapes réversibles où la charge est relayée entre les trois acides aminés de la triade catalytique. L’attaque nucléophile du groupement hydroxyle du résidu Ser se produit en utilisant le résidu His comme base et conduit à la formation d’un lien scissile. L’oxyanion tétrahédrique intermédiaire est stabilisé par les atomes de la structure peptidique qui génèrent une charge positive dans la pochette de liaison. L’acylenzyme formée réagit avec une molécule d’eau afin de former un second oxyanion tétrahédrique. La triade catalytique est reformée.
Lors de ce mécanisme (Figure 6) il est important de noter que le caractère nucléophile de la serine n’est pas assez fort afin de catalyser la réaction chimique. Des ponts hydrogène sont formés entre la sérine et l’histidine afin d’amplifier la nucléophilie. L’aspartate sert à polariser le résidu basique et aligner ce dernier15,16.
Les protéases à sérine de type « trypsin-like » ont une préférence pour les résidus chargés positivement sur la chaîne latérale, tels que l’arginine et la lysine, en position P1 du
O O H N N H O HNC O Asp His Ser Na+ Na+ O O H N N H O C HN O O O H N N OC O O H H H2O Asp His Ser O O H N N H OC O OH O O H N N H Na+ Na+ O HO O Asp His Ser Asp His Ser Asp His Ser -NH2 Oxyanion
Lien scissile Acyl enzyme
Oxyanion tétrahédrique
Addition/élimination Hydrolyse
Addition/élimination
Addition/élimination Na+
substrat. Cette affinité est due à la pochette de liaison où des résidus ayant une charge négative en S1 augmentent la spécificité pour l’arginine et la lysine. À contrario, la
chimotrypsine préfère les résidus aromatiques comme la phenylalanine, la tyrosine et le tryptophane en position P115,16,17.
1.3.2 Les protéases à serine transmembranaires de type II (TTSP)
La plupart des protéases à sérine sont des enzymes sécrétées ou séquestrées dans les organites jusqu’à un signal de régulation qui entraîne leur libération pour permettre d’effectuer leur rôle biologique11,19.
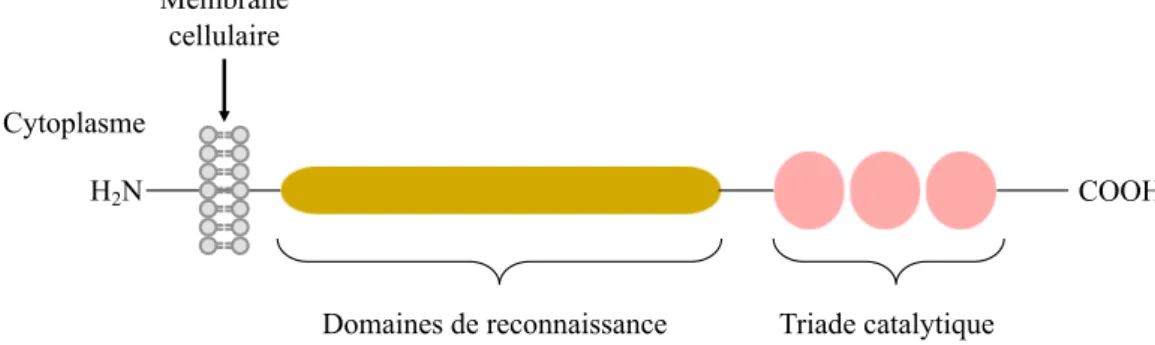
Les TTSP (Figure 7) sont une classe de protéases à sérine dont l’identification et la caractérisation intéressent de nombreux groupes de recherche depuis plusieurs années. Elles sont caractérisées par un domaine N-terminal intracellulaire et un domaine C-terminal extracellulaire20. Ces enzymes se trouvant à la surface des cellules interagissent avec d’autres protéines qui sont elles aussi présentes à la surface cellulaire. Parmi elles, on retrouve des protéines solubles, des composants de la matrice ou encore des protéines des cellules voisines15,16,20.
Figure 7Structure générale des TTSP – Les TTSP possèdent un domaine N-terminal qui traverse la membrane et se retrouve dans le cytoplasme. Les domaines de reconnaissance, catalytique et C-terminal sont extracellulaires.
Les domaines multifonctionnels comprennent les domaines protéolytiques, le domaine transmembranaire et les régions Stem.
H2N COOH
Domaines de reconnaissance Triade catalytique Cytoplasme
Membrane cellulaire
Premièrement, les sites catalytiques des TTSP partagent des séquences d’acides aminés très similaires. Les TTSP sont synthétisées sous forme d’une seule chaîne zymogène et activées par un clivage suivant un résidu arginine ou lysine présent dans le motif d’activation. Une fois l’enzyme clivée, la forme extracellulaire active présente un pont disulfure qui lie le domaine catalytique au reste de la protéine. Pour certaines TTSP, les spécificités de clivage ont été étudiées, indiquant un clivage spécifique après des acides aminés basiques. Cela suggère que les TTSP ont une activité autocatalytique19,20. Le domaine hydrophobe près du N-terminal de chaque TTSP permet d’ancrer la protéase dans la membrane plasmique de façon à ce que le domaine protéolytique soit extracellulaire. On suppose que cette configuration permet de localiser l’activité protéolytique proche du substrat ou de réguler la protéine depuis la surface cellulaire19,20. Par la suite, on retrouve jusqu’à onze domaines structurels dans les régions Stem chez les TTSP. Ces domaines peuvent servir à la régulation et à la liaison. Ils incluent les domaines :
o LDLA (Low Density Lipoprotein receptor class A) o SRA (group A Scavenger Receptor)
o Frizzled : Frizzled domains
o CUB (Cls/Clr Urchin embryonic growth factor and Bone morphogenic protein 1)
o SEA (sea urchin Sperm protein Enterokinase, Argin)
o MAM (a Meprin, A5 antigen, and receptor protein phosphatase µ) o Disulfide noué (disulfide knotted domain)
De toutes les TTSP, l’hepsine est la seule à ne pas posséder de domaine de reconnaissance Stem20.
Finalement, le domaine cytoplasmique des TTSP s’étend de douze acides aminés (HAT) à 112 acides aminés (corine). Ce domaine a la capacité de renforcer les interactions avec le cytosquelette21.
Les domaines cytoplasmiques et transmembranaires contribuent au ciblage des TTSP vers des surfaces cellulaires spécifiques dans les cellules polarisées21.
Le domaine LDLA est le plus courant chez les TTSP. Il permet les interactions protéine-protéine21. Les TTSP sont regroupées en quatre sous-familles (Figure 8). Dans ce travail, nous nous intéresserons plus précisément à la sous-famille matriptase.
Figure 8Familles des TTSP (Adapté de Bugge et al., 200920) – Représentation des différentes familles des TTSP composés de 17 membres ainsi que leurs domaines extracellulaires. N C N C N C N C N C N C N C N C Domaine SEA Domaine CUB Domaine LDLA Domaine Frizzled Domaine SR Domaine MAM Domaine protéase à serine (actif) Domaine protéase à serine (inactif) H S A H D D Sous-Famille Matriptase Sous-Famille Corine Sous-Famille Hepsin/TMPRSS5 Sous-Famille HAT/DESC1 Matriptase Matriptase-2,-3 Polyserase-1 Corine Enteropeptidase TMPRSS5/Spinesin, Hepsin TMPRSS2-4, MSPL HAT, DESC1, TMPRSS11A, TMPRSS11B, TMPRSS11F
1.3.3 La sous-famille matriptase
La sous-famille de la matriptase (Figure 8) est composée de quatre protéases possédant des rôles distincts : la polysérase-1, la matriptase, la matriptase-2 et la matriptase-320. Ces protéases sont localisées dans des tissus différents.
Les matriptase-1, -2 et -3 possèdent des régions stem communes. Cependant, la grande différence structurelle entre ces trois enzymes provient des domaines LDLA. En effet, la matriptase présente quatre de ces domaines tandis que la matriptase-2 et -3 n’en possèdent que trois20,22,23,24. La matriptase a la capacité de s’auto-activer via la séquence d’auto-activation : RQAR-VVGG. L’hypothèse selon laquelle la matriptase agit comme initiateur de cascade de signalisation protéolytique contrôlant la différenciation et le développement épithélial a été émise25. Des études ont ainsi démontré que la matriptase possédait un rôle important dans l’homéostasie des tissus épithéliaux de différents organes. L’expression de cette protéase est importante pour l’intégralité de l’épithélium19.
La polysérase-1 est la seule TTSP à posséder trois domaines serine protéase, un domaine C-terminal et capable de générer des variantes de fonctions avec un domaine SEA et le premier des trois domaines serine protéase. On retrouve cette protéase dans les muscles squelettiques, le foie, le cerveau, le cœur et le placenta14.
La matriptase-3 est relativement exprimée chez la souris avec une forte expression au niveau du cerveau, des yeux, de la peau, des testicules, de la glande salivaire et une faible expression au niveau du cœur, du muscle squelettique, du thymus, des ovaires et de l’utérus, de la prostate. Chez l’homme, la matriptase-3 est sensiblement exprimée de la même façon à l’exception des testicules et des ovaires où l’on retrouve une forte expression de l’enzyme, le cerveau, la trachée, les poumons et les glandes salivaires où l’expression est moins forte14,24.
La matriptase-2 est le quatrième membre de cette famille, et c’est sur cette enzyme que s’est portée notre attention, particulièrement pour son rôle potentiel dans la régulation du fer.
2 La matriptase-2 et le fer
2.1 Le fer dans l’organisme
Le fer est un élément essentiel pour la vie de tous les organismes vivants (plantes, bactéries, animaux et être humain). Principalement apporté par l’alimentation, il permet la formation de l’hémoglobine et de la myoglobine qui auront, respectivement, pour rôle de transporter l’oxygène (O2) depuis les poumons et les bronches vers le reste de
l’organisme, et de stocker l’oxygène. Le fer joue aussi un rôle de cofacteur protéique, qui participe à la production d’énergie via un transfert d’électrons dans la chaîne respiratoire mitochondriale. Finalement il est impliqué dans de nombreux processus métaboliques (réparation et réplication de l’ADN, régulation de l’expression génique , etc)26.
Figure 9 Le cycle du fer (Adaptée de Beaumont, 200427) – Le fer alimentaire est présent sous deux formes (héminique ou non héminique), en fonction desquelles il pénètre l’entérocyte différemment afin de poursuivre son cycle dans l’organisme.
Le fer apporté par l’alimentation est absorbé au niveau du petit intestin (Figure 9). Le fer héminique est capté par le récepteur HCP1 (Heme Carrier Protein 1), lui permettant d’être libéré de son noyau hème (hème oxygénase). Le fer non héminique est réduit en fer ferreux (Fe2+) grâce à la ferriréductase DCYTB (Duodenal Cytochrome B). La protéine DMT1 (Dimetal Transporter 1) permet l’internalisation du fer dans la cellule
Enterocyte
Fer alimentaire
Fer héminique
Fer non héminique
e -DCYTB DMT1 Na+ H+ Fe2+ DMT1 K+ Na+ Hepcidine DMT1 Ferritine Fe2+ Fe3+ Fe2+ Transferrine C6H8O6 Lumière intestinale Sang HCP1 Fe 3+ Fe2+
sous sa forme réduite. Dans la cellule (Figure 9), le fer est stocké par la protéine ferritine d’une part (puis libéré à la mort de la cellule) et d’autre part libéré vers la circulation sanguine. Ce relargage a lieu à l’aide de la ferroportine, transporteur qui va permettre au fer ferreux de franchir le pole basal de la cellule. Il est ensuite oxydé en fer ferrique (Fe3+) par l’hephaestine au niveau de la membrane. L’hepcidine contrôle la sortie du fer des entérocytes en bloquant et dégradant la ferroportine. Son rôle sera abordé plus en détails au point II.1.226,28,29,30.
Le fer libre est toxique car il génère des radicaux libres 28. Pour cette raison il est stocké par la ferritine et relâché dans la circulation sanguine en étant lié à la transferrine (Tf) qui l’acheminera aux tissus31.
Après son absorption, le fer suit un cycle qui comprend transport, utilisation, élimination, recyclage et régulation (Figure 10).
Figure 10 Homéostasie du fer – Après son absorption, le fer est transporté, utilisé, recyclé, régulé et éliminé. Toutes ces étapes constituent l’homéostasie du fer28,32,33,34,35.
Absorption Transport Recyclage Régulation Utilisation Stockage
2.2 L’hepcidine : une hormone régulatrice des niveaux de fer
L’hepcidine est une hormone peptidique synthétisée par le foie (Figure 11) sous forme d’un précurseur de 84 acides aminés. Le passage de la forme de précurseur à l’hormone bioactive fait intervenir une hormone convertase « furin-like » qui clive la pro-hepcidine au niveau de la liaison peptidique du C-terminal, après une séquence polybasique caractéristique34,36. Sous sa forme active, l’hepcidine se présente comme un peptide de vingt-cinq acides aminés (dans le plasma et l’urine), avec une masse de 2.7 kDa37,38. L’hepcidine est sécrétée par les hépatocytes afin de circuler dans le sang sous forme libre puis filtrée par les reins.
Figure 11Rôle de l’hepcidine dans la régulation du fer (Adaptée de Béliveau, 201239)
– La synthèse d’hepcidine s’effectue au niveau du foie (1), l’hepcidine lie la ferroportine se trouvant à la surface des cellules telles que les hépatocytes, les entérocytes et les macrophges. Elle permet ainsi la distribution et la relâche de fer emmagasiné (2), empêche le recyclage issu de la dégradation des érythrocytes (3) et diminue l’absorption de fer dans les entérocytes (4).
L’hepcidine est le plus important régulateur du fer40,41. Elle a pour rôle d’inhiber l’absorption du fer par le duodénum et son relargage dans la circulation sanguine, en empêchant la sortie du fer des entérocytes et des macrophages (Figure 11). L’hepcidine se lie à la ferroportine en formant un complexe hepcidine-ferroportine (Figure 12). Cela entraîne une diminution de la concentration de ferroportine à la membrane plasmique.
Hepcidine P P P Fe2+ Fe2+ Fe2+ FPN FPN FPN X X X 1 2 4 3 Foie Macrophage Entérocyte Hépatocyte
Étant donné que le récepteur membranaire ferroportine permet le transport du fer dans le sang, le complexe formé contrôle les flux de fer présent dans le plasma et celui disponible pour les tissus. La liaison de l’hepcidine à la ferroportine conduit à l’endocytose de cette dernière mais aussi à sa protéolyse dans le lysosome (phosphorylation, internalisation, dégradation). 35
Figure 12 Mécanisme d’action de l’hepcidine – Le transporteur DMT1 permet le transport de fer à travers la membrane apicale. La ferroportine permet au fer de traverser la membrane basolatérale (1). C’est au niveau du foie que le fer va être absorbé et stocké, ce qui provoque la synthèse et l’excrétion de l’hepcidine (2). Après avoir été excrétée, l’hepcidine se lie à la ferroportine (3), le complexe formé est internalisé et dégradé dans le lysosome (4). Suite à cela, le fer entrant dans la cellule est accumulé dans la ferritine (5).
L’expression de l’hepcidine est contrôlée par les niveaux de fer : elle est augmentée en cas de surcharges de fer (une alimentation riche en fer par exemple) et par l’inflammation, elle est diminuée dans les cas d’anémie, manque d’oxygénation et inactivation du gène HFE42,43. Lorsque les niveaux de fer plasmatique sont élevés, l’expression d’hepcidine dans les hépatocytes est assurée par une cascade de signalisation qui fait intervenir la voie BMP (Bone Morphogenic Protein) et SMAD (Son of Mother Against Decapentalegic) (Figure 13). Ainsi, on observe la formation d’un complexe
DMT1 Hepcidine Lysosome Ferritine DMT1 Blocage de la sortie de fer Fer alimentaire (1) (2) (3) (4) (5)
membranaire, l’hémojuvéline (HJV), et aux récepteurs BMPR I et II. Le complexe formé entraîne la phosphorylation de SMAD1/5/8. Parallèlement, un autre complexe hétéromérique se forme entre SMAD1/5/8 et SMAD4. Ce nouveau complexe est ensuite transloqué au noyau et va stimuler la transcription du gène HAMP (Hepcidin Antimicrobial Peptide) qui code pour l’hepcidine. Certaines cytokines inflammatoires, comme l’interleukine 6 (IL-6) favorisent l’expression d’hepcidine en activant la signalisation de JAK2 et la phosphorylation de STAT3, mais la voie de signalisation BMP-SMAD est aussi nécessaire à l’activation de l’expression d’hepcidine29,43,44.
Figure 13 Voies de signalisation contrôlant l’expression d’hepcidine (Adaptée de Falzacappa et al., 200745) – L’expression d’hepcidine est effectuée suite à la transcription du gène HAMP. Après une inflammation, l’interleukine 6 se lie aux récepteurs IL6-R et gP130 pour activer la signalisation de JAK2 permettant la phosphorylation de STAT3 et la transcription du gène HAMP. L’expression de la protéine BMP6 est augmentée par les réserves de fer, la voie BMP-SMAD est activée et conduit à la transcription du gène HAMP. La protéine HFE libre forme un complexe avec TfR2 et conduit à l’expression de l’hepcidine en permettant la phosphorylation de SMAD 1/5/8 pour conduire à la transcription du gène HAMP.
Une fois que les niveaux de fer plasmatique sont rétablis, l’expression d’hepcidine est réduite et l’hepcidine encore présente dans le système sanguin est excrétée par les reins. La diminution de l’expression de cette hormone est effectuée grâce à la matriptase-2.
+ P P P GPI H J V Hepcidine B M P R -I BMP6 B M P R -II BMP6 BMP6 BMP6 BMP6 SMAD 1/5/8 HAMP SMAD4 P SMAD 1/5/8 SMAD 1/5/8 P SMAD4 SMAD 1/5/8 IL6 I L 6 -R g P 1 3 0 JAK2 STAT3 STAT3P INFLAMMATION Fe-Tf HFE TfR2 TfR1 HFE HFE TfR1 -+
2.3 La matriptase-2 dans l’organisme
Comme il a été dit plus tôt, la matriptase-2 (Figure 14) est une TTSP exprimée principalement à la surface des hépatocytes. On la retrouve chez différentes espèces telles que le chien, le mouton, le chimpanzé, la souris et l’être humain où ses caractéristiques structurelles sont conservées22,46. Elle est fortement exprimée dans le foie où elle est
co-exprimée avec l’hémojuvéline à la surface des hépatocytes22. Cette protéine de 811 acides aminés a pour fonction d’inhiber l’expression de l’hepcidine afin de réguler des taux de fer dans l’organisme. Elle est caractérisée par un domaine SEA, deux domaines CUB et trois domaines LDLA suivis d’une triade catalytique (His, Ser, Asp) avant le domaine C-terminal.
Figure 14 Structure de matriptase-2 (Adapté de Wang et al., 201447) – La
matriptase-2 est composée de plusieurs domaines extracellulaires et d’un domaine N-terminal intracellulaire.
Comme la matriptase, la matriptase-2 est synthétisée sous forme de zymogène et activée suite à un auto-clivage et l’activation protéolytique de l’enzyme se produit avec un motif RIVGG22.
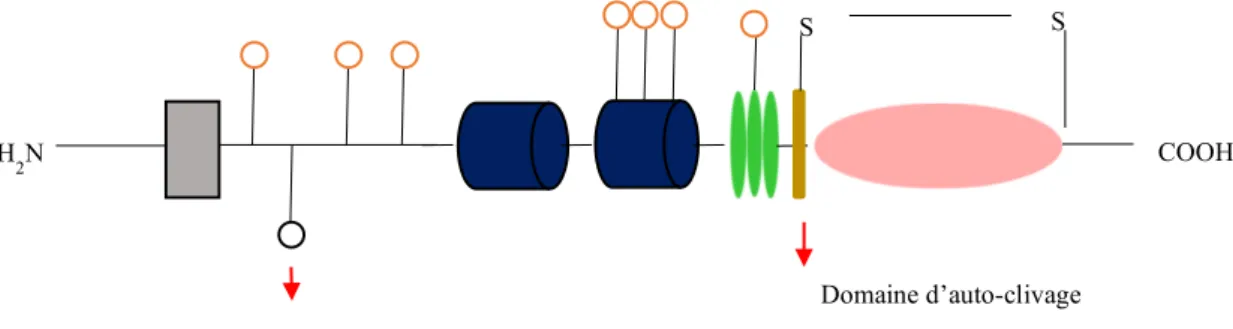
Matriptase-2 possède trente-sept résidus cystéine qui permettent la formation de ponts disulfures. On retrouve ces résidus dans les domaines CUB et LDLA ainsi que des sites consensus de N-glycosylation (Figure 15).
H2N COOH
Domaine SEA Domaine CUB Domaine LDLA
Figure 15Domaines structurels de matriptase-2 (Adapté de Ramsay et al., 200822) - Les sites de N-glycosilation sont présentés en orange et la cystéine non appariée en noir. Le domaine d’auto-clivage se situe à la jonction des domaines pro- et catalytique.
Le domaine catalytique et l’ectodomaine de l’enzyme restent liés via un pont disulfure formé entre les résidus Cys568 et Cys688, formant ainsi un dimère (Figure 15). L’activation de l’enzyme lui permet d’accomplir ses rôles biologiques dans le milieu extracellulaire.
La régulation de l’expression d’hepcidine se produit par clivage de l’hémojuvéline, substrat de la matriptase-2, empêchant ainsi la voie de signalisation BMP/SMAD d’effectuer son rôle48 (Figure 16).
Figure 16 Rôle de matriptase-2 à la surface des hépatocytes (Adapté de Béliveau 201239) – La matriptase-2 permet le clivage de l’hémojuvéline ce qui empêche
l’activation de la voie BMP-SMAD et par conséquent l’expression de l’hepcidine.
SMAD 1/5/8 1 H J V B M P R -I B M P R -II + BMP6 SMAD 1/5/8 SMAD 1/5/8 SMAD4 P P P Hepcidine Matriptase-2 BMP6 HAMP P P BMP6 BMP6 BMP6 GPI S S COOH H2N Cystéine non-appariée Domaine d’auto-clivage
Le contrôle de l’expression d’hepcidine par la matriptase-2 a été démontré dans différentes études à partir d’un modèle murin « Mask »48 (anémie microcytaire et perte progressive du pelage) dont la forme d’anémie menait à des niveaux de fer relativement bas dans le plasma et une consommation excessive, jusqu’à épuisement du fer. Les chercheurs ont pu constater que chez ce modèle, les taux d’hepcidine, dont l’expression est normalement diminuée, étaient élevés, conduisant à une absorption de fer alimentaire moins efficace et réduite. Le gène TMPRSS6, qui code pour la matriptase-2, a été identifié comme responsable de la mutation qui mène à ce phénotype. La mutation produit une matriptase-2 dépourvue de domaine catalytique. Ces études ont permis de démontrer que l’activité catalytique de la matriptase-2 avait un rôle dans la régulation de l’expression d’hepcidine en abaissant ses niveaux d’expression39,47,49,50,51. Une mutation de la
matriptase-2 peut conduire à une expression importante de l’hepcidine et ainsi à une forte anémie (IRIDA, ou iron-refractory iron deficiency anemia) ; ou une faible expression d’hepcidine menant à des maladies caractérisées par une surcharge de fer (β-thalassémie et hémochromatose).
3 Les maladies associées à la surcharge en fer
La surcharge en fer intervient lorsque les réserves en fer présentes dans l’organisme sont augmentées et conduisent à une accumulation dans les organes. Les causes de ces surcharges peuvent être variables : une anomalie génétique, des maladies du foie, ou encore la conséquence d’autres maladies. Dans le dernier cas, on parlera alors de surcharge de fer secondaire.
3.1 L’hémochromatose
L’hémochromatose est une maladie génétique définie par une suraccumulation de fer due à une absorption anormalement excessive du fer alimentaire au niveau du duodénum52,53.
Selon la fondation canadienne du foie, cette pathologie touche 1 canadien sur 32754. Elle
est associée à une mutation génétique qui entraîne la baisse de l’expression de l’hepcidine. On distingue quatre types d’hémochromatose29,52,55.
les porteurs sont homozygotes ; et H63D où les patients sont hétérozygotes C282Y/H63D.
- Le type II est la forme la plus sévère, aussi appelé hémochromatose juvénile. Il est divisé en deux groupes IIA et IIB. Pour ce type d’anomalies, les sites de mutation sont variables. Le type IIA est dû à une mutation de l’hémojuvéline dont le gène est localisé sur le chromosome 18, tandis que le type IIB résulte de la mutation du gène HAMP localisé sur le chromosome 19.
- Le type III est dû à la mutation du gène TFR2 qui code pour le récepteur TfR2. - Le type IV, connu sous le nom de maladie de la ferroportine, est le résultat de la
mutation du gène SLC40A1 codant pour la ferroportine 1.
Parmi les différents types d’hémochromatose décrites, le type I est le plus répandu dans la population. De plus, les types I, II et III sont dus à des mutations à transmission récessive alors que le type IV est à transmission dominante. Dans tous ces types, il est désirable d’abaisser les niveaux de fer circulant.
3.1.1 L’hémochromatose juvénile
L’hémochromatose juvénile est une maladie rare qui, à l’inverse de l’hémochromatose de type I, présente un développement de la surcharge en fer avant l’âge de 30 ans. L’excrétion de fer chez un individu atteint d’hémochromatose ne se produit pas de manière naturelle, entraînant des quantités de fer plasmatique supérieures à 6 mg/jour56.
Des organes tels que le foie, le pancréas, le cœur ou encore l’hypophyse sont affectés par cette maladie qui provoque des lésions anatomiques et fonctionnelles irréversibles. Des maladies secondaires (diabète, troubles cardiaques, dépigmentation de l’épiderme) apparaissent chez les patients faute de traitement55.
3.1.2 Traitements de l’hémochromatose
Actuellement, il existe différents traitements pour la maladie : la phlébotomie (saignée), l’érythraphérèse, ou le traitement par chélation de fer.
La phlébotomie consiste en deux étapes : (1) l’induction pour éliminer la surcharge en fer ; et (2) l’entretien pour maintenir une charge en fer inférieure ou égale à 50ng/mL52,55,57. Lors de la phlébotomie, les patients subissent des saignées
1 à 2 ans en fonction de la surcharge en fer, afin d’atteindre des niveaux de ferritine de 50 ng/mL. La férritinémie et le taux d’hémoglobine sont évalués. L’entretien du maintien de cette charge en fer se poursuit à vie, permettant d’atténuer un grand nombre de complications.
La saignée est un traitement de première intention, il existe malgré tout des contre-indications comme l’anémie, la thalassémie et les cardiopathies sévères non liées à l’hémochromatose, qui imposent la recherche d’alternatives pharmacologiques.
Le traitement par chélation de fer est employé en seconde intention55,58. En effet, les agents chélatants (Figure 17) ont la possibilité d’affecter l’absorption, l’excrétion, ainsi que les voies métaboliques du fer (synthèse de l’hème, liaison de la transferrine à son récepteur au niveau de la membrane cellulaire). Il existe différents agents possédant différents mécanismes d’action : la 8-hydroxyquinoline (lipophile), le maltol, et le 2-hydroxy-4-méthoxypiridine-1-oside (lipophile chez la souris). Les agents chélatants lipophiles conduisent à une surcharge de fer chez les rats, et donc une augmentation de l’absorption de fer alimentaire lors d’une administration orale à long terme. Cet effet est reproduit pour les médicaments chélatants lipophiles (comme le déférasirox (DFRA)). Dans les mêmes conditions, les chélateurs hydrophiles mènent à une baisse de l’absorption de fer (comme le déféroxamine (DFO)), ou une inhibition (2-hydroxy-4-methoxypiridine-1-oside). Ce traitement utilise une combinaison de chélateurs déféroxamine/défériproone (DFO/L1). On l’applique pour les cas d’hémochromatose où la saignée n’est pas recommandée, ainsi que les thalassémies. Son utilisation augmente l’espérance de vie du patient59,57,60,61,62.
Figure 17Chélateurs de fer – Exemple des agents chélatants utilisés comme traitement de l’hémochromatose et des thalassémies
3.2 Les thalassémies
Les thalassémies sont des maladies héréditaires qui affectent l’hémoglobine. Entre 0 et 3% de la population en Amérique est touchée par cette maladie63. On considère deux types de thalassémies selon la chaîne d’hémoglobine affectée : les a- et les b-thalassémies. Ici, le cas des b-thalassémies sera décrit.
3.2.1 Les b-thalassémies
L’altération des chaînes b de l’hémoglobine conduit à la thalassémie. Le gène b-globine à l’origine de la fabrication des chaînes b est muté conduisant à la pathologie. Ainsi, les chaînes b de l’hémoglobine sont produites en faible quantité, ce qui provoque une production insuffisante de l’hémoglobine globale. La maladie se manifeste différemment d’une personne à l’autre en fonction du degré d’altération des gènes b-globine. La production des chaînes b peut être soit réduite, soit nulle avec des symptômes plus ou moins sévères60,64,65. Trois types de b-thalassémies sont référencés :
N OH O O OH N+ -O O HO 8-Hydroxyquinoléine Maltol 2-hydroxy-4-methoxy pyridine-1-oxide HO O N N N OH HO Déférasirox N O OH H2N N H N N N H N OH O O OH O O O OH Défériprone Déféroxamine
(a) La b-thalassémie majeure (TM) : deux gènes b-globine sont altérés, cela se traduit par une anémie.
(b) La b-thalassémie intermédiaire (TI) : deux gènes b-globine sont altérés, et des quantités réduites d’hémoglobine sont fabriquées.
(c) La b-thalassémie mineure (Tm) : un seul gène b-globine est muté. Généralement, il n’y a aucune conséquence sur la santé (porteurs sains).
La mutation sur les deux gènes b-globine conduit à un déséquilibre entre le nombre de chaînes a et le nombre de chaînes b produites. Il y a alors une accumulation des chaînes a dans les globules rouges et les érythrocytes, les endommageant et accélérant leur destruction, ce qui mène à une anémie65.
Bien que cette maladie soit caractérisée par une anémie, les cas cliniques de surcharge en fer chez les personnes thalassémiques sont nombreux. Elle est liée à une plus importante absorption de fer par le système digestif (TI), aux transfusions sanguines (TM) et finalement aux bas niveaux d’hepcidine. L’excédent de fer va être accumulé dans différentes parties du corps, entraînant d’autres pathologies (diabète, ménopause précoce…). Avec le temps, la surcharge en fer n’est plus une conséquence de la maladie mais devient le principal problème65.
3.2.2 Les traitements des b-thalassémies
Les traitements des b-thalassémies varient en fonction du type de pathologie. Il s’agit d’abord de corriger l’anémie à l’aide de transfusions sanguines régulières pour maintenir un taux d’hémoglobine correcte. Dans certains cas, la splénectomie est pratiquée pour éliminer la rate qui recycle les globules rouges. Cette pratique comporte des risques car les patients sont plus vulnérables aux infections microbiennes. La prise d’acide folique (vitamine B9) constitue le dernier traitement. Le seul traitement qui guérisse la maladie est la greffe de moelle osseuse, qui permet la production de globules rouges sans anomalies64,65.
En conclusion, les traitements actuels pour les maladies associées aux surcharges de fer sont soit difficiles à contrôler, soit associés à des effets indésirables lourds. Par conséquent, tel qu’indiqué ci-dessus (Fig. II.8), l’inhibition de la matriptase-2 apparait comme une alternative crédible visant à contrôler les niveaux d’hepcidine. Plusieurs
modes d’inhibition d’enzymes existent à ce titre et seront couverts dans la section suivante.
4 Les inhibiteurs
Comme mentionné dans I, il existe dans la nature des inhibiteurs qui permettent de bloquer l’activité catalytique d’une enzyme. Ils peuvent se diviser en deux groupes distincts, à savoir les inhibiteurs réversibles ou irréversibles4.
4.1 Les inhibiteurs réversibles
Les inhibiteurs réversibles forment des interactions non covalentes avec l’enzyme. Ces interactions permettent aux deux entités de se dissocier. On considère trois catégories d’inhibiteurs réversibles : réversibles non-compétitifs, réversibles incompétitifs et réversibles compétitifs2,3,4.
1. L’inhibition compétitive (Figure 18) : L’inhibiteur se lie à l’enzyme libre sur le même site de liaison que le substrat, sur un site allostérique bloquant la liaison du substrat à l’enzyme ou encore sur un site distant en bloquant l’accès au site de liaison de l’enzyme. Ainsi, il y a compétition entre le substrat et l’inhibiteur. L’enzyme ne peut pas être liée à la fois au substrat et à l’inhibiteur. Le captopril (Figure 19) est un inhibiteur compétitif qui cible l’enzyme de conversion de l’angiotensine, utilisé comme traitement pour l’hypertension artérielle3.
Figure 18Mécanisme d’inhibition compétitive – En absence d’inhibiteur le substrat (S) se lie à l’enzyme (E) sur un site spécifique (1) pour former le complexe enzyme-substrat (ES). Lorsqu’il y a un inhibiteur (I), celui-ci peut se lier à l’enzyme sur le site de liaison du substrat (2), sur un site allostérique de l’enzyme (3) ou encore un site distant (4) pour former le complexe enzyme-inhibiteur (EI).
Figure 19 Structure du captopril – Le captopril empêche la conversion de l’angiotensine I en angiotensine II.
2. L’inhibition non-compétitive (Figure 20) : Ce type d’inhibiteur affiche une affinité de liaison pour l’enzyme libre mais aussi pour l’enzyme liée au substrat (complexe enzyme-substrat). L’affinité enzyme-inhibiteur se définit à l’aide de deux constantes de dissociations : un Ki pour le complexe EI, et un autre (aKi)
pour le complexe enzyme-substrat-inhibiteur.
- Si a = 1, l’affinité de l’inhibiteur pour l’enzyme est égale à celle du
ES S S E E E E I I I I (1) (2) (3) (4) I I