Caractérisation du système anti-phage AbiQ de
Lactococcus lactis
Mémoire
Maxime Bélanger
Maîtrise en microbiologie
Maître ès sciences (M. Sc.)
Québec, Canada
© Maxime Bélanger, 2014
Résumé
L’utilisation de mécanismes anti-phages est l’une des stratégies pour prévenir et contrôler la
contamination par les bactériophages qui peuvent causer des pertes importantes pour l’industrie
de la transformation laitière. AbiQ est un mécanisme d’avortement de l’infection phagique de
type toxine-antitoxine (type III) isolé d’une souche de Lactococcus lactis. Des études précédentes
ont démontrées que la toxine ABIQ (protéine) coupe son antitoxine (ARN non codant) in vivo,
et que la protéine virale ORF38 du lactophage P008 joue un rôle essentiel dans l’activité
anti-phage. Dans cette étude, nous nous sommes intéressés au mode d’action d’AbiQ via trois
objectifs spécifiques : caractériser l’effet de différentes mutations de l’antitoxine, analyser le
comportement viral et cellulaire en présence d’AbiQ et définir le rôle de la cible/activateur viral
ORF38. Nos résultats démontrent qu’il est possible d’optimiser l’efficacité anti-phage par
modification de l’antitoxine et suggèrent que l’ORF38 interagit avec l’ARN antitoxine
permettant la libération de la toxine
Abstract
Antiphage mechanisms represent one of the strategies available to prevent or control
contamination by virulent phages, which are a major risk of fermentation failure in the dairy
industry. The lactococcal phage abortive infection system AbiQ belongs to type III
toxin-antitoxin system. It has been previously demonstrated that the toxin ABIQ (protein) cleaves his
antitoxin (non-coding RNA) in vivo, while the protein ORF38 from phage P008 plays an essential
role in the antiphage activity. In this study, we investigated the mode of action of AbiQ through
three specific objectives: describe the effect of specific modifications in the antitoxin, analyse
the viral and cellular behaviours in presence of AbiQ and determine the role of the phage P008
target/activator ORF38. Our results show that we can optimise the antiphage activity of AbiQ
through mutation in the antitoxin and suggest that ORF38 can bind the antitoxin RNA to favour
the release of the toxin.
Table des matières
Résumé ... iii
Abstract ... v
Liste des tableaux ... ix
Liste des figures ... xi
Liste des abréviations ... xiii
Avant-propos ... xix
Introduction ... 1
Bactéries lactiques ... 1
Lactococcus lactis ... 2
Bactériophages ... 6
Classification des phages ... 7
Lactophages ... 9
Cycle de réplication ... 11
Mécanismes anti-phages ... 15
1.
Inhibition de l'adsorption ... 15
2.
Bloquer l'injection de génome viral ... 17
3.
Coupure des acides nucléiques exogènes ... 17
4.
Avortement de l'infection (Abi) ... 20
Systèmes d'avortement de l'infection chez L. lactis ... 22
Systèmes toxine-antitoxine (TA) ... 27
TA de type I ... 29
TA de type II ... 30
TA de type III ... 33
TA de type IV ... 34
TA de type V ... 34
AbiQ ... 35
Hypothèse de travail... 37
Matériel et méthodes ... 39
Résultats ... 53
Objectif 1 : Étude de la spécificité d’antiQ ... 53
Analyses bio-informatiques ... 53
Détermination du site de coupure et d’initiation de la transcription (5’ RACE PCR) ... 54
I.
Caractérisation des mutants Δ répétitions (antiQ) ... 58
II.
Caractérisation des mutants ponctuels (Mut antiQ) ... 60
Objectif 2 : Étude de la réplication virale et cellulaire en présence du système AbiQ ... 64
Croissance de L. lactis IL1403 en présence d’AbiQ ... 65
Courbes de croissance des phages P008 et P008-Q12 (AbiQ
+) ... 65
Analyse de la traduction d’abiQ en cours d’infection ... 66
Objectif 3 : Caractérisation de la protéine virale ORF38 (phage P008) et de son rôle dans
l’activité du système AbiQ ... 68
Expression d’ORF38 chez L. lactis (système NICE) ... 68
Expression d’ORF38-GST chez E. coli ... 69
Stratégie de clonage en bloc (orf37-orf39) ... 70
Test d’interaction entre ORF38 et antiQ ... 72
Discussion ... 73
Conclusion ... 85
Bibliographie... 87
Liste des tableaux
Tableau 1.1. Tableau comparatif des caractéristiques génomiques des souches de Lactococcus lactis ssp.
lactis et ssp. cremoris. ... 4
Tableau 1.2. Classification des groupes de lactophages ... 9
Tableau 1.3. Regroupement des mécanismes Abi de lactocoques ... 22
Tableau 2.1. Souches, plasmides et phages utilisés dans la cadre du projet ... 40
Tableau 2.2. Tableau des amorces utilisées dans la cadre du projet ... 44
Tableau 2.3. Matrice partielle d’expérience pour l’expression d’ORF38 (système NICE) ... 49
Tableau 2.4. Matrice partielle d’expérience pour l’optimisation de la stabilité d’ORF38 ... 52
Tableau 3.1.1. Efficacité anti-phage des mutants Mut Δ répétitions antiQ contre P008 ... 58
Tableau 3.1.2. Fréquence d’obtention des clones mutants ponctuels antiQ ... 61
Tableau 3.1.3. Résultats cumulatifs des activités anti-phage (EOP) et endoribonucléique pour les différents mutants antiQ ... 62
Liste des figures
Figure 1.1. Image de Lactococcus lactis ssp. cremoris MG1363 par microscopie à balayage ... 3
Figure 1.2. Schéma des systèmes d’expression utilisés pour ce projet ... 5
Figure 1.3. Morphologie des bactériophages ... 8
Figure 1.4. Alignements des génomes de phages 936 modèles ... 11
Figure 1.5. Cycle lytique (A) et lysogénique (B) de réplication des phages ... 12
Figure 1.6. Représentation tridimensionnelle des protéines formant la plaque basale du phage p2 ... 13
Figure 1.7. Mode de fonctionnement des systèmes CRISPR-Cas ... 19
Figure 1.8. Schéma des cinq types de systèmes toxine-antitoxine ... 28
Figure 3.1.1. Schéma de l’opéron AbiQ (A). Séquence de l’opéron AbiQ (B) ... 53
Figure 3.1.2. Produits PCR du 5’ RACE et contrôles migrées sur gel d’agarose ... 55
Figure 3.1.3. Schéma issu du séquençage de l’extrait RACE-AbiQ. ... 55
Figure 3.1.4. Séquençage de 15 clones pBS-KS (RACE-AbiQ) ... 56
Figure 3.1.5. Représentation graphique LOGO de l’alignement de trois séquences de coupure par ABIQ ... 57
Figure 3.1.6.. ... 57
Figure 3.1.7. Hybridation de type Northern ciblant antiQ dans les souches L. lactis IL1403 Δ répétitions (Mut 1,8r et Mut 3,8r) et pNZ123-AbiQ ... 59
Figure 3.1.8. Hybridation de type Northern ciblant antiQ et le gène abiQ dans les souches L. lactis IL1403 Δ répétitions (Mut 1,8r et Mut 3,8r) et pNZ123-AbiQ en cours d’infection par le phage P008 ... 60
Figure 3.1.9. Représentation des mutations ciblées dans antiQ ... 61
Figure 3.1.10. Comparaison des structures prédites des ARNs antiQ ... 63
Figure 3.1.11. Analyse de la structure en pseudonoeud d’antiQ chez le mutant AbiQ-(A32C) ... 64
Figure 3.2.1. Courbes de croissance (DO600nm) de L. lactis IL1403 pNZ123-AbiQ (AbiQ+) ou pNZ123 (AbiQ-) en présence et absence du phage P008 ou le mutant P008-Q12 ... 65
Figure 3.2.2. Courbes de croissance virale (P008 ou P008-Q12) pendant l’infection de L. lactis pSRQ928 (AbiQ+) ou IL1403 pNZ123 (AbiQ-) ... 66
Figure 3.2.3. Résultats d’immunobuvardage Western (anti-his) ciblant la protéine ABIQ(His) durant une infection de L. lactis IL1403 AbiQ+ par le phage P008 ou P008-Q12 ... 67
Figure 3.3.1. Test d’expression d’ORF38-His chez la souche L. lactis NZ9000 (Système NICE) ... 69
Figure 3.3.2. Résultats de l’expression et purification de GST-ORF38 sur colonne d’affinité (Glutathione) ... 70
xiii
Liste des abréviations
2-D En 2-dimensions
5’RACE PCR « Rapid Amplification of cDNA Ends » (Amplification rapide d’extrémités d’ADN complémentaire 5’)
aa Acide aminé
A Base adénine
Abi « Abortive infection » (Avortement de l’infection)
AbiQ Opéron AbiQ
abiQ Gène abiQ
ABIQ Protéine ABIQ (toxine)
ADN Acide désoxyribonucléique
ADNc ADN complémentaire
ADNdb ADN double brin
Amp Ampicilline
antiQ ARN non codant (antitoxine)
ARN Acide ribonucléique
ARNm ARN messager
ARNr ARN ribosomal
ARNt ARN de transfert
ATP Adénosine tri-phosphate
BL bactérie lactique
BSA « Bovine serum albumin » (Albumine de sérum bovin)
C Base cytosine
C-ter Extrémité C-terminale
Cm Chloramphénicol
CRISPR-Cas Clustered Regularly Interspaced Short Palindromic Repeats – CRISPR associated
cpm Coups par minute
(k)Da (Kilo)Dalton
DO600nm Densité optique 600nm
DoE « Design of experiments » (Plan des expériences) DTT Dithiothréitol
EDF « Extracellular death factor » (Facteur extracellulaire de mort) EDTA Acide éthylène diamine tétraacétique
EOP « Efficiency of plating » (Efficacité à formé des plages de lyse) enf « Enforces » (Structure forcée)
FPLC « Fast protein liquid chromatography » (Chromatographie liquide rapide des protéines)
FT Flow through
G Base guanine
g Temps de génération
GF « Gel filtration » (colonne de filtration sur gel)
GMP Guanosine monophosphate
GST Glutathione S-transferase
His (6H) Étiquette 6Histidine
HPLC « High-performance liquid chromatography » (Chromatographie liquide à haute performance)
H-T-H « Helix-Turn-Helix » (Hélice-tour-Hélice) IPTG Isopropyl β-D-1-thiogalactopyranoside kDa Kilodalton
MCS « Multiple cloning site » (Site multiple de clonage) min Minute
mfe « Minimum free energy » (Énergie libre minimum) ml Millilitres
MLST « Multilocus sequence typing » (Typage par séquençage de plusieurs loci) mm Millimètre
MOI « Multiplicity of infection » (Multiplicité de l’infection)
N-ter Extrémité N-terminale
NEB Compagnie, New England Biolabs
NICE « Nisine controlled gene expression system » (Système d’expression inductible à la nisine) nt Nucléotide
NTase
GTP-spécifique Transférase de nucléotide spécifique au guanosine tri-phosphate. O/N « Over night » (Toute la nuit)
orf « Open reading frame » (Cadre de lecture ouvert)
p/V Poids par volume
PAM « Protospacer adjacent motif » (Motif adjacent au protoespaceur) Km Kanamycine
(k)(M)pb (kilo) (million) de paire de bases
PBS « Phosphate buffered saline » (Tampon phosphate)
PCR « Polymerase chain reaction » (Réaction en chaîne de la polymérase)
pI Point isoélectrique
PIB Produit intérieur brut Pip Phage infection protein
PLE phage-inducible chromosomal island-like element PPR Production de protéine recombinante
PTS « Phosphotransferase system » (Système phosphotransférase) PSK « postsegregational killing » (Mortalité post-ségrégationnelle)
PVDF Polyfluorure de vinylidène
R Résistant
R/M Système de restriction-modification
RBP « Receptor binding protein » (Protéine de laison au récepteur) RBS « Ribosome binding site » (Site de liaison au ribosome) RF « RNase-Free» (Sans ARNase)
RT « Reverse transcriptase » (Transcriptase inverse)
s Sensible
s Seconde SDS Sodium dodecyl sulfate
SDS-PAGE « SDS polyacrylamide gel electrophoresis » (Gel de polyacrylamide SDS) SEC-MALS Size-Exclusion Chromatography + Multi-Angle Static Light Scattering SPR « Surface plasmon resonance » (Résonance des plasmons de surface) SSB « Single-strand binding protein » (Protéine de liaison à l’ADN simple brin) ssp. Sous-espèce
T Base thymine
TB Terrific broth
TMP « Tail major protein » (Protéine majeure de la queue)
Tp Température pièce
TSS « Transcription start site » (Site d’initiation de la transcription)
TT Transférase terminale
U Base uracile
UFC Unité formant des colonies UFP Unité formant des plages de lyse
xv UNAG UDP-N-acetyl-D-glucosamine
UP Upstream promoter element
V0 Volume mort
V/V volume/volume W/V « weight/volume » (poids/volume) (n)(µ)(m) g (nano) (micro) (milli)-gramme (n)(µ)(m) m (nano) (micro) (milli)-mètre (n)(µ)(m) M (nano) (micro) (milli)-molaire
%G-C Pourcentage des bases guanine et cytosine
«
Science never solves a problem
without creating ten more
»
(George Bernard Shaw)
Avant-propos
Ma maîtrise en quelques mots… un projet passionnant et inspirant qui m’a permis de définir mes limites et de les surpasser. Les jours plus difficiles, ainsi que les petits moments « eurêka » de la recherche m’ont permis d’acquérir énormément d’expérience aux niveaux social et scientifique, expérience qui s’avère essentiel au développement personnel et professionnel. Au cours de ces années, plusieurs personnes m’ont accompagné dans mon cheminement et m’ont permis d’atteindre mes objectifs, jusqu’à compléter mon programme de maîtrise.
Tout d’abord, merci à mon directeur de recherche, professeur Sylvain Moineau pour sa confiance, son support et son enthousiasme qui m’ont beaucoup inspiré. Aussi, je conserve les mêmes mots pour son bras droit Denise Tremblay qui a souvent fait office de ressource scientifique de référence. Également, je ne peux passer sous silence Julie Samson qui a été mon mentor et modèle dès mon début au laboratoire, et ce même jusqu’à aujourd’hui.
Merci aussi à Bruno pour les nombreuses discussions scientifiques et les bières du vendredi. Également, j’aimerais remercier les membres de mon comité aviseur pour leurs conseils formateurs, ainsi que les membres du laboratoire de Québec et de Marseille qui ont fait de ma maîtrise une expérience plaisante et enrichissante.
Merci à ma famille et mes amis pour votre support constant tout au long de ce projet. Merci à Anton qui, peut-être même sans le savoir, m’a permis de franchir les obstacles jusqu’à l’université, et ainsi pouvoir découvrir le domaine passionnant de la microbiologie. Finalement, j’aimerais remercier ma copine Fannie qui malgré son train de vie acharné, a toujours trouvé du temps pour m’aider à me dépasser, à découvrir et à avancer.
Introduction
Bactéries lactiques
Les travaux de nombreux chercheurs tels que Leeuwenhoek, Fracastoro, Jenner, Pasteur et Koch ont permis de mettre en place les bases de la microbiologie actuelle (Prescott et al., 2002). Toutefois, l’homme utilisait déjà des microorganismes bien avant que ceux-ci soient observés et caractérisés. Une étude a d’ailleurs mis en évidence l’existence d’un breuvage fermenté datant de 7000 ans av. J.-C (McGovern et
al., 2004). Les connaissances acquises depuis ce temps ont permis de domestiquer certains
microorganismes qui jouent désormais des rôles primordiaux dans les domaines pharmaceutique, environnemental et alimentaire. Un exemple est l’industrie laitière qui est parmi les plus importantres industries au Canada. Cette industrie génère près de 15 milliards de dollars annuellement et emploie plus de 215 000 personnes. À lui seul, le secteur de la transformation laitière génére 8 milliards de dollars au PIB. Trente six pourcent des revenus associés à ce secteur (2,6 milliards de dollars) proviennent de la province du Québec (ÉcoRessources Consultants, 2011).
En 2010, les usines de transformation laitière ont transformé plus de 2,5 milliards de litres de lait (http://cilq.ca/industrie/statistiques-sur-lindustrie-laitiere/, 2014) dont 82% sont transformés par trois grandes entreprises : Agropur, Saputo et Parmalat (http://www.lait.org/fr/leconomie-du-lait/la-transformation.php, 2014). Chaque jour, ces entreprises utilisent plus de 1010 bactéries par litre de lait afin
de le transformer en produits dérivés comme du fromage ou du yogourt (Moineau et al., 2002). Les bactéries lactiques (BL) sont utilisées afin d’acidifier le lait et de changer ses propriétés physico-chimiques (Boucher et Moineau, 2001). Cette acidification permet également l’inhibition de croissance de certains contaminants (Klaenhammer, 1988). Ces bactéries peuvent être isolées de différents environnements allant des mamelles d’animaux (Verdier-Metz et al., 2012) aux légumes asiatiques (Chen et al., 2013b). Les BLs forment un groupe hétérogène qui contient exclusivement des membres appartenant au phylum Firmicutes. Elles possèdent tous la capacité de métaboliser le lactose et produire de l’acide lactique. Ces bactéries se subdivisent selon leur type de fermentation hétéro- ou homofermentaires. Le premier regroupe les bactéries produisant de l’acide lactique, mais également d’autres acides et alcools à partir des sucres. Pour leur part, les BLs homofermentaires métabolisent les sucres en pyruvate, puis en lactate comme unique produit final de la glycolyse (Axelsson, 2004). Les bactéries homofermentaires représentent le groupe le plus utilisé dans l’industrie. Outre leur capacité d’utilisation des sucres, la température de croissance et la morphologie cellulaire sont d’autres caractéristiques qui ont été utilisées par Orla-Jensen pour la classification des BLs au début du 20e siècle (Orla-Jensen, 1921; Axelsson, 2004). Les techniques modernes ont permis de préciser leur classification par l’utilisation notamment de
l’identité d’ARNr 16S des bactéries lactiques. Parmi les 12 genres de BL, seuls les genres Lactobacillus,
Lactococcus, Leuconostoc et Streptococcus sont utilisés pour la transformation laitière (Champagne et Arora,
2009). La sélection du type de bactérie lactique est faite en fonction des caractéristiques enzymatiques qui dictent le goût, la saveur, la texture ainsi que la vitesse de fermentation (Mierau et al., 1997; Smit et al., 2005). D’ailleurs, les produits dérivés de fermentation comme l’aldéhyde et le diacétyl, les produits de la protéolyse de la caséine par les protéases bactériennes ainsi que les exopolysaccharides permettent la fabrication de produits laitiers fermentés aux propriétés recherchées (Mierau et al., 1997; Geis, 2005; Smit
et al., 2009).
De façon plus spécifique, le genre Lactococcus est divisé en neuf espèces dont seulement deux (lactis et
raffinolactis) sont associés au domaine laitier (Cai et al., 2011). L’espèce lactis est la seule des lactocoques
utilisée par l’industrie laitière. Les autres espèces sont retrouvées sur des végétaux (plantarum, fujiensis,
taiwanensis et formosensis), isolées de boue activée (chungangensis) ou constituent des pathogènes de poissons
(piscium et garvieae) (Cai et al., 2011; Chen et al., 2013a; Chen et al., 2014).
Lactococcus lactis
Les ferments industriels utilisés pour la production de fromages, de babeurre et de crème sure sont le plus souvent constitués de différentes souches de Lactococcus lactis (Teuber, 1995). L. lactis fut la première bactérie à être isolée en culture pure par Joseph Lister en 1873 (Lister, 1878). Jusqu’en 1985, cette bactérie portait le nom de Streptococcus lactis (Schliefer et al., 1985). Morphologiquement, elle à l’aspect d’un coque de 0,5 à 1,5 µm de diamètre qui s’organise en paire ou en courte chainette (figure 1.1). Sa température optimale de croissance est de 30°C. De plus, L. lactis est une bactérie anaérobie aérotolérante puisqu’elle n’utilise pas l’oxygène naturellement comme accepteur final d’électron. Toutefois, elle peut utiliser la respiration aérobie en présence d’hème (Duwat et al., 2001).
Figure 1.1. Image de Lactococcus lactis ssp. cremoris MG1363 par microscopie à balayage (Grossiord et al.,
2003).
L. lactis est divisé en quatre espèces (ssp.) (cremoris, hordniae, lactis et tructae) où seulement les
sous-espèces cremoris et lactis sont utilisées par l’industrie laitière (Latorre-Guzman et al., 1977; Schliefer et al., 1985; Pérez et al., 2011; Von Wright, 2012). La sous-espèce lactis est fréquemment utilisée dans la production de certains fromages frais (Jamet, 2009) alors que la ssp. cremosis est davantage utilisée dans la fabrication de cheddar (Vedamuthu et al., 1966). Cette dernière produit une plus haute concentration de certains composés volatiles recherchés pour ce type de fromage (Fernández et al., 2011). Ces deux sous-espèces sont très similaires au niveau génétique (Identité >99% pour ARN16S et les gènes conservés
rpoB et recA), mais peuvent être distinguées par des caractéristiques métaboliques spécifiques. En effet, la
ssp. cremoris se distingue de lactis par son incapacité à croitre à 40°C, sa croissance en présence de 4% NaCl ainsi que son aptitude à hydrolyser l’arginine (Holt et al., 1994; Von Wright, 2012).
À ce jour, les génomes de onze souches de L. lactis sont disponibles dans la banque de données GenBank (NCBI, http://www.ncbi.nlm.nih.gov/genome/browse/). Les caractéristiques génétiques de ces souches sont présentées dans le tableau 1.1. Tout comme pour les autres bactéries lactiques, leur génome est faible en bases guanine et cytosine (36% G-C). Le nombre moyen de gènes est de 2552 pour des génomes d’environ 2,5 Mpb chacun. Également, ces souches possèdent entre zéro et huit plasmides différents qui sont impliqués dans plusieurs fonctions variées. Ils possèdent, entre autres, la majorité des gènes nécessaires à la production de bactériocines, la résistance aux phages ainsi que le transport et le métabolisme du lactose (Teuber, 1995). Parmi elles, seules les souches L. lactis ssp. lactis IO-1, CV56, KF147 et L. lactis ssp. cremoris KW2 n’ont pas été isolées de l’environnement laitier (Siezen et al., 2010; Gao et al., 2011; Kato et al., 2012; Kelly et al., 2013). La provenance de la souche influence généralement
sa capacité à métaboliser les composantes de son environnement. En effet, les souches d’origine laitière possèdent d’avantage de gènes impliqués notamment dans la protéolyse des caséines du lait et la résistance aux phages (O'Sullivan et al., 2009).
Tableau 1.1. Tableau comparatif des caractéristiques génomiques des souches de Lactococcus lactis ssp.
lactis et ssp. cremoris. Souche Génome (Mpb) G+C (%) Nombre de gènes Nombre de plasmides Numéro d’accession (GenBank) Références L. lactis ssp. lactis
IL1403 2,37 35,3 2425 - AE005176 (Bolotin et al., 2001; Linares et
al., 2010)
KF147 2,64 34,9 2662 1 CP001834 (Siezen et al., 2010)
CV56 2,52 35,1 2549 5 CP002365 (Gao et al., 2011)
IO-1 2,42 35,1 2319 - AP012281 (Kato et al., 2012)
KLDS
4.0325 2,59 35,4 2662 3 CP006766
(Yang et al., 2013)
L. lactis ssp.
cremoris
MG1363 2,53 37,7 2597 - AM406671 (Wegmann et al., 2007)
NZ9000 2,53 37,7 2594 - CP002094 (Linares et al., 2010)
A76 2,58 35,9 2845 4 CP003132 (Bolotin et al., 2012)
SK11 2,60 35,8 2742 5 NC_008527 (Makarova et al., 2006)
UC509.9 2,46 35,8 2401 8 CP003157 (Ainsworth et al., 2013)
KW2 2,43 36,7 2278 - CP004884 (Kelly et al., 2013)
Isolées du secteur laitier, les souches L. lactis ssp. cremosis MG1363 et L. lactis ssp. lactis IL1403 sont des organismes modèles dans l’étude des bactéries à Gram positif (Bolotin et al., 2001; Wegmann et al., 2007; Linares et al., 2010). Les souches MG1363 et IL1403 sont dérivées respectivement des souches NCDO712 et IL594 dont leur plasmides naturels ont été éliminés via un traitement aux UV et/ou un curage-protoplastique (Gasson, 1983; Chopin et al., 1984). D’ailleurs, il a été démontré que la perte des plasmides de L. lactis MG1363 permet d’augmenter sa stabilité génétique améliorant ainsi cette bactérie comme outil pour des analyses d’ADN recombinant (Gasson, 1983). La stabilité génomique de ces souches pourrait également s’expliquer par leur forme diploïde lorsque mis en culture à croissance lente (Michelsen et al., 2010).
L’étude de ces souches a permis d’approfondir nos connaissances sur L. lactis et a mené au développement de souches « réusinées » avec des caractéristiques de fermentation et de productivité augmentées (de Vos et Hugenholtz, 2004). Ces études ont aussi favorisé le développement de systèmes de production de protéines recombinantes (PPR) efficacent chez les bactéries à Gram positif, incluant L. lactis (de Vos et
al., 1997; de Vos, 1999).
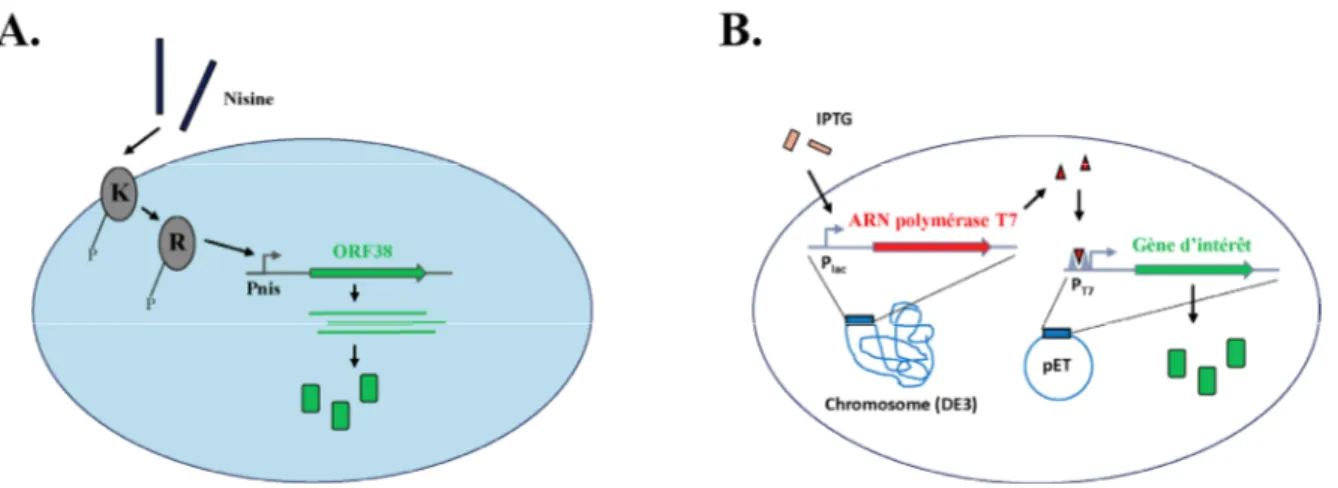
Le système NICE (NIsine Controlled gene Expression system) est sans aucun doute le système d’expression de protéines recombinantes le mieux caractérisé chez cette bactérie (Mierau et Kleerebezem, 2005). La nisine (nisA) est une bactériocine qui induit un enchainement de phosphorylation qui permet l’activation du promoteur Pnis, et la transcription du gène cloné en aval (figure 1.2-A). Les gènes
régulateurs nisK (récepteur s’autophosphorylant) et nisR (activateur du promoteur) peuvent être introduits dans le chromosome de la souche bactérienne utilisée pour l’expression ou sur un plasmide, alors que le promoteur inductible est situé sur le plasmide de clonage du gène d’intérêt (de Ruyter et al., 1996). La souche L. lactis ssp. cremoris NZ9000 est une souche dérivée de L. lactis MG1363 contenant les gènes régulateurs nisK/nisR dans son chromosome. Fait intéressant, la souche NZ9000 a une meilleure capacité à incorporer le glucose que MG1363 et ce phénomène serait causer par la présence de six mutations ponctuelles ayant été identifiées dans des gènes jouant des rôles dans le système phosphotranférase (PTS) spécifique à la cellobiose, la synthèse de chaperonnes et le métabolisme des acides aminés (Linares et al., 2010). Le système NICE a également été adapté pour d’autres bactéries à Gram positif (Kleerebezem et
al., 1997; Pavan et al., 2000).
Figure 1.2. Schéma des systèmes d’expression utilisés pour ce projet. (A) Système NICE chez L. lactis
Les systèmes Zirex (Mu et al., 2013) et SICE (Benbouziane et al., 2013) permettent également la production de protéines recombinantes chez L. lactis. Ces systèmes d’expression chez les bactéries à Gram positif représentent des alternatives intéressantes aux systèmes classiques développés chez Escherichia coli, mais restent tout de même généralement moins efficaces (Morello et al., 2008; Chen, 2012). Chez E. coli, le système d’expression le plus utilisé est le système T7. Le gène codant pour l’ARN polymérase du phage T7 est placé sous le contrôle du promoteur lacUV5 inductible à l’IPTG présent dans la souche d’expression (DE3). À l’induction, l’ARN polymérase T7 permet l’induction du promoteur en amont du gène d’intérêt (figure 1.2-B) (Studier et Moffatt, 1986; Terpe, 2006).
Que ce soit dans le secteur de PPR ou dans l’industrie laitière, l’altération des fermentations et des productions conduit à des pertes financières liées à la diminution des rendements ou à la modification de la qualité des produits finaux. Parmi les nombreux facteurs qui peuvent influencer la croissance bactérienne lors des productions industrielles, la présence de bactériophages est un des principaux agents causal, affectant 0,1 à 10 % des fermentations laitières (Moineau et Lévesque, 2005). De plus, l’incidence des phages cause des problèmes de croissance cellulaire récurrents pour les cultures procaryotes de production de protéines recombinantes.
Bactériophages
Les bactériophages (ou phages) sont des virus bactériens. Ils constituent les entités biologiques les plus abondantes et les plus diversifiées sur terre (Brüssow et Kutter, 2005). Leur nombre total est estimé à plus de 1030, soit 10 à 15 fois plus que le nombre de bactéries (Suttle, 2005). Tout comme leur hôte, les
phages sont retrouvés dans tous les environnements connus sur Terre. Certains phages ont même été retrouvés dans des environnements extrêmes comme des étendues d’eau salées et des sources thermales (Rice et al., 2001; Porter et al., 2007). Bien qu’ils soient ubiquitaires, les phages sont retrouvés en plus grande majorité dans les océans, estimés à plus de 108 phages par millilitre d’eau (Bergh et al., 1989;
Wommack et Colwell, 2000). Leur prédominance est représentative de la présence de leur hôte et varie énormément en fonction du temps et de l’espace (Wommack et Colwell, 2000). Selon plusieurs experts, le rôle des phages serait d’ailleurs de contrôler les populations bactériennes et ainsi conserver l’équilibre géochimique et écologique des écosystèmes (Wommack et Colwell, 2000; Suttle, 2007). Les bactériophages permettent la dégradation de 20-40% de la communauté microbienne à chaque jour (Suttle, 1994).
La découverte de ces entités biologiques remonte au début du 20ième siècle. Les scientifiques Frederick
des cultures de Micrococcus et Shigella (Prescott et al., 2002). Toutefois, seul d’Hérelle a poursuivi ses recherches sur cet agent viral qu’il appellera bactériophage. Cette découverte resta controversée jusqu’à l’avènement de la microscopie électronique en 1940 qui a permis l’observation directe de ces virus (Ackermann, 2011).
Les phages sont des virus généralement simples au niveau génétique ce qui fait d’eux d’excellents modèles pour l’étude des interactions hôte-virus autant pour les procaryotes que pour les eucaryotes. D’ailleurs, c’est en raison de sa petite taille que le premier génome d’ADN simble brin à avoir été complètement séquencé fût celui du phage phiX174 (5 386 nt) (Sanger et al., 1977). Cinq ans plus tard, la séquence d’un premier génome à ADN double brin fut publié, celle du phage modèle lambda ( ) (Sanger et al., 1982). À ce jour, on compte plus de 2 500 phages dont la séquence de leur génome est disponible dans les banques de données publiques (http://www.ncbi.nlm.nih.gov/nuccore). Les recherches sur les phages ont permis, entre autres, le développement d’outils moléculaires indispensables à ce jour. À titre d’exemple, les méthodes courantes de clonage utilisent les systèmes de restriction qui ont d’abord été découverts pour leur efficacité anti-phage (Meselson et Yuan, 1968). Une autre méthode met à profit la capacité lysogénique du phage lambda pour permettre des clonages rapides et efficaces à des sites d’insertions spécifiques (Walhout et al., 2000). Également, des promoteurs viraux sont fréquemment utilisés pour la production de protéines recombinantes afin de maximiser la transcription des gènes d’intérêts. Les phages peuvent aussi constituer des outils intéressants pour d’autres applications comme le «phage display» ou la thérapie par les phages ciblant des bactéries pathogènes (McAuliffe et al., 2007).
Classification des phages
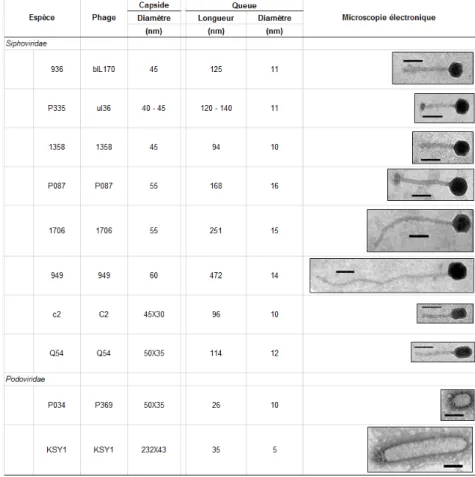
La très grande majorité des phages sont constitués de trois éléments principaux. (1) Une tête protéique appelée capside. Celle-ci contient l’information génétique (2) du phage sous forme d’ADN ou d’ARN, simple ou double brins. Le troisième élément est une queue (3), également de nature protéique, qui est retrouvée chez 96% des phages connus à ce jour (Ackermann, 2003; Ackermann et Prangishvili, 2012). Ces caractéristiques morphologiques représentent les bases de la nomenclature phagique actuelle. Jusqu’à présent, plus de 6000 phages ont été observés au microscope électronique (Ackermann, 2007; Ackermann, 2009; Ackermann et Prangishvili, 2012). Tous ces résultats permettent de mettre en évidence la grande diversité des bactériophages retrouvés sur la planète (figure 1.3.). De tous les phages observés, les phages atypiques (sans queue) ne représentent que 4% et ceux-ci sont divisés en trois groupes morphologiques différents : polyhédrale, filamenteux ou pléomorphe (Ackermann, 2009). Certains de ces phages rares possèdent une enveloppe lipidique ce qui n’est pas le cas chez les phages caudés (Ackermann, 2003).
Figure 1.3. Morphologie des bactériophages. Adaptée de (Ackermann, 2007).
Le ICTV (International Committee on Taxonomy of Viruses) regroupe les phages en 14 familles (ICTV Master Species list 2013). D’un autre côté, certains experts les regroupes en 10 familles (Ackermann, 2007; Ackermann et Prangishvili, 2012). Les nouvelles méthodes de caractérisation des virus telles le séquençage et la protéomique virale pourrait éventuellement mener au remaniement des taxons actuels (Lavigne et al., 2009).
L’ordre des Caudovirales contient tous les phages caudés (96%) et se subdivise en trois grandes familles selon leur type de queue. Les Myoviridae ont une longue queue contractile (25% des Caudovirales), les
Podoviridae ont une petite queue non contractile (14% des Caudovirales) et les Siphoviridae possèdent une
longue queue non contractile (61% des Caudovirales) (Maniloff et Ackermann, 1998). Ces phages possèdent une capside symétrique et une queue hélicoïdale d’une taille allant de 10 et 800 nm. De plus, ils ont tous un génome d’ADN double brin d’une taille variant entre 17 à 700 kpb (Ackermann, 2009). Fait intéressant, les phages caudés seraient les plus vieux virus, expliquant ainsi la diversité génétique à l’intérieur de cet ordre (Ackermann, 2003).
Lactophages
Dans l’industrie de la transformation laitière, les mêmes souches bactériennes de choix sont constamment utilisées afin de maximiser les rendements de l’entreprise et la fidélité des consommateurs. Depuis quelques décennies, l’utilisation massive de ces bactéries a conduit à l’émergence de nouveaux phages qui infectent spécifiquement ces souches d’intérêt (Rousseau et Moineau, 2009). Leur incidence sur les rendements et la qualité des fromages constitue la raison principale pour expliquer l’intérêt marqué des chercheurs pour les phages reliés au domaine laitier. Uniquement dans le laboratoire du Prof. Sylvain Moineau, plus de 125 nouveaux phages de lactocoques en provenance de l’industrie laitière québecoise ont été isolés depuis 15 ans.
Les phages de Lactococcus lactis ont tous une queue non contractile et la majorité d’entre eux appartiennent à la famille des Siphoviridae. La classification actuelle subdivise les lactophages en 10 groupes génétiques distincts (tableau 1.2.) (Deveau et al., 2006). Parmi eux, les groupes 936, c2 et P335 sont, en ordre d’importance, les plus souvent associés à des problèmes de fermentation dans plusieurs pays du monde (Rousseau et Moineau, 2009).
Tableau 1.2. Classification des groupes de lactophages. Adaptée de (Deveau et al., 2006).
Une méthode de type multiplex PCR ciblant des gènes conservés est disponible et permet de classer rapidement les nouveaux phages à l’intérieur d’un de ces trois groupes prédominants (Labrie et Moineau, 2000). L’évaluation de l’incidence et de la variabilité des populations phagiques lors de fermentation industrielle constitue le premier pas dans le contrôle de ces contaminants. L’étude plus approfondie des phages les plus problématiques, tel ceux du groupe des 936, a d’ailleurs permis de développer des modèles de recherche et des outils de détection efficaces pour contrer les phages dans cette industrie.
Chez le groupe 936, certains phages sont bien caractérisés et constituent des modèles d’études : p2 (Sciara
et al., 2010), P008 (Loof et al., 1983), sk1 (Chandry et al., 1997) et bIL170 (Labrie et Moineau, 2000;
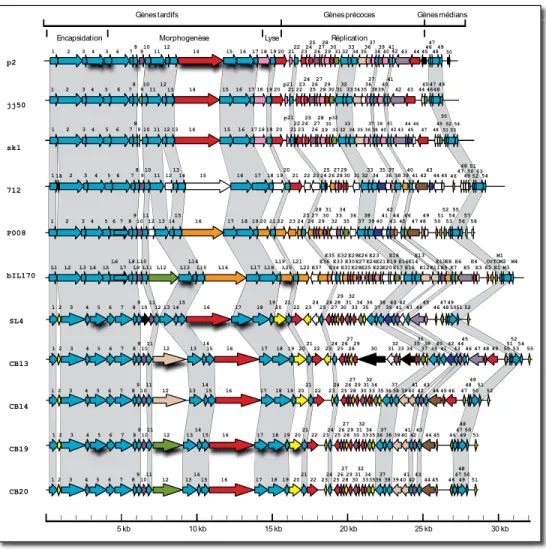
Crutz-Le Coq et al., 2002; Dupont et al., 2005). Crutz-Les phages 936 possèdent une capside icosaédrique et une queue non contractile(Siphoviridiae), leur génome variant entre 25 et 40 kpb (Brøndsted et Hammer, 2006). Le nombre total de phages de type 936 dont le génome a été séquencé s’élève aujourd’hui à 52 (http://www.ncbi.nlm.nih.gov/). Récemment développée dans le laboratoire du Prof. S. Moineau, une nouvelle méthode MLST (« Multilocus Sequence Typing ») permet la différenciation des phages 936 par comparaison de séquence nucléotidique de cinq gènes conservés (Moisan et Moineau, 2012). Tel que démontré à la figure 1.4., les phages 936 forment un groupe génétiquement homogène. Un génome-cœur composé de 33 gènes a été identifié et est constitué majoritairement de gènes codant pour des protéines structurales. Au contraire, les produits de gènes précoces sont en générales peu conservés entre les phages (Rousseau et Moineau, 2009).
Figure 1.4. Alignements des génomes de phages 936 modèles. Les ORFs de même couleur sont ceux
qui ont plus de 80% d’identité en acide aminé alors que les ombrages gris reliant les génomes démontrent les régions conservés chez tous les génomes des 936. Traduite de (Rousseau et Moineau, 2009).
Cycle de réplication
Que ce soit des phages de lactocoques ou infectant d’autres bactéries, tous les phages se répliquent exclusivement dans leur hôte. Les phages peuvent utiliser deux types de cycle de réplication. Les phages sont dit virulents s’ils suivent le cycle lytique de réplication, ou tempérés, s’ils utilisent le cycle lysogénique (figure 1.5.). Chez les lactophages, les phages du groupe 936 et c2 sont virulents alors que les P335 sont virulents ou tempérés (Bissonnette et al., 2000).
5 kb 10 kb 15 kb 20 kb 25 kb 30 kb 712 jj50 P008 sk1 bIL170 p2 SL4 CB13 CB14 CB19 CB20
Gènes tardifs Gènes précoces Gènes médians
Encapsidation Morphogenèse Lyse Réplication
1 2 3 4 5 6 7891011 121314 15 16 17 18 192021 22 23 24252627282930 31 32333435363738 394041 424344 45 48 4950 51 5253 47 54 46 1A 1 2 3 4 5 6 78910111213 14 15 16 17 18 19 20 21 2223 24 2526 27 282930 313233 343536 37 383940 41 42 43 444546474849 1 2 3 4 56 7 89101112 13 1415 16 17 18 19 20 21 22 23 24252627 28 2930 31 3233 34 3536373839 4041 42 4344454647 48495051 52 5354 55 565758 L1 L2 L3 L4 L5 L6 L7L8L9L10L11 L12 L13L14L15 L16 L17 L18L19L20L21L22 E37 E35 E34E33 E32 E31E30 E29 E28E27 E26 E25E24 E23
E22E21E20E19 E18
E17E16E15E14 E13 E12E11E10E9E8 E36 E7E6E5E4E3E2OrfOE1 M1 M2 M3M4 15 16 17 18 19 20 21222324 25 26 28 293031 323334 35 3637383940 41 42 4344454647 484951525354 p32 27 p21 1 2 3 4 5 6 789 10 11 12 13 14 50 1 2 3 4 5 6 78910111213 14 15 16 17 18 19 20 21222324 25 2627 28 293031 323334 3536 37 3839404142 43 44 4546 47 4849 1 2 3 4 5 6 7 89101112 13 1415 16 17 1819202122 232425262728 29 3031 32 3334353637383940414243 4445464748495051 52 13 1415 16 17 18 19 202122 23242526 27 2829 30 313233 3435 36 3738394041424344 45 48 49 5051 52 53 47 54 46 55 1 2 3 4 5 6 7 891011 12 23242526 27 28293031 32 333435 363738 39 4041424344 45 48 49 505152 47 46 13 1415 16 17 18 19 202122 1 2 3 4 5 6 7 891011 12 23242526 27 28293031 32 333435 363738 39 4041424344 45 48 495051 47 46 131415 16 17 18 19 202122 1 2 3 4 5 6 7 891011 12 23242526 27 28293031 32 333435363738 39 4041424344 45 48 495051 47 46 131415 16 17 18 19 202122 1 2 3 4 5 6 7 891011 12 p21 50
Figure 1.5. Cycle lytique (A) et lysogénique (B) de réplication des phages. Adaptée de (Labrie et al., 2010).
Au cours du cycle lytique, les phages utilisent les cellules bactériennes comme des usines de productions virales. En contrepartie, les phages tempérés vont plutôt intégrer leur génome à l'intérieur du chromosome bactérien au moyen d’une intégrase et de séquences d’intégrations spécifiques. Ce génome viral intégré est appelé prophage. La réplication du chromosome bactérien amène du même coup la dissémination du prophage dans les cellules filles, et donc la propagation du phage dans l’environnement. Certaines protéines de répression sont exprimées constitutivement à partir du prophage et permette le maintien du génome virale à l’intérieur du chromosome bactérien. Toutefois, l'induction par certains stress tels les rayons ultra-violets (UV), un stress oxydatif, la température et la Mitomycine C peuvent mener à la déstabilisation de cet état (Little, 1984). Le prophage est alors excisé pour ensuite entrer dans le cycle lytique de réplication.
Que ce soit pour le cycle lytique ou lysogénique, le point de départ de l’infection est le même. Les phages se retrouvent dans l’environnement sous forme inerte appelée virion et vont débuter leur cycle de réplication au moment de la mise en contact avec la bactérie. Malgré les diverses opinions, ce n'est pas clair si la rencontre phage/hôte est uniquement stochastique ou si elle est guidée par différente forces d’interactions. Le phage va donc s'attacher à un ou plusieurs récepteurs présents à la surface cellulaire : protéines membranaires, polysaccharides, pilis, flagelles, acides téicoïques ou lipotéicoïques liés au
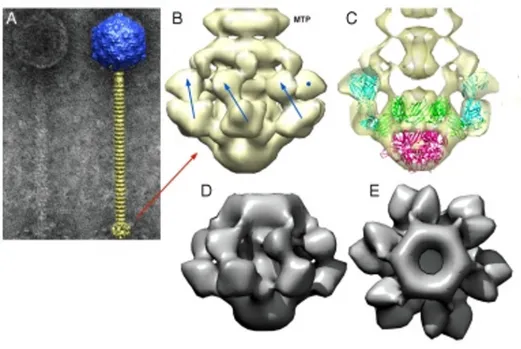
peptidoglycanes (Kutter et al., 2005). Chez les phages caudés, cette interaction se fait principalement entre le(s) récepteur(s) et la plaque basale à l'extrémité de leur queue. Cette dernière est composée de plusieurs protéines jouant un rôle essentiel dans l'adsorption et l'éjection de l'ADN viral lors de l'infection (figure 1.6.). Également, la protéine majeure de la queue (MTP) pourrait participer à la reconnaissance phage/hôte en permettant un attachement primaire et réversible à la surface de la bactérie (Pell et al., 2010).
Figure 1.6. Représentation tridimensionnelle des protéines formant la plaque basale du phage p2. (A)
Image en microscopie électronique et schématisée de p2. (B, C) Plaque basale avec la première MTP. Les flèches (ou points) bleues représentent les RBP. (D, E) Réprésentation par Cryo-EM de la plaque basale. Adaptée de (Sciara et al., 2010).
Pour les phages de lactocoques, seulement la protéine bactérienne membranaire Pip (phage infection protein) a été caractérisée au niveau moléculaire. Cette protéine est essentielle pour l'infection des phages à tête allongée du groupe c2 (Geller et al., 1993), mais n'est pas nécessaire pour l'infection par les phages à capside isométrique 936 et P335 (Kraus et Geller, 1998).
Les plaques basales sont constituées de plusieurs monomères de la protéine de liaison au récepteur (RBP).
Des études sur la RBP du phage p2 (groupe 936) ont permis de mettre en évidence la liaison RBP au glycérol/sucre suggérant une liaison des RBP aux acides lipoteichoïques in vivo (Tremblay et al., 2006). Ces RBP s'assemblent en trimère dans la plaque basale, et sont constituées de trois domaines : 1. épaule
en N-terminale, 2. cou et 3. tête en C-terminale. La tête serait essentielle à la spécificité du récepteur (Spinelli et al., 2006). La liaison RBP au récepteur est irréversible et s'avère nécessaire pour l’étape subséquente, l'éjection de l'ADN viral. Une étude récente a aussi démontré l'importance de la composition en sucres formant la pellicule entourant les cellules de L. lactis ssp. cremoris, pour l'adsorption des phages du groupe 936 (Mahony et al., 2013). Il est évident que le type de récepteur est, du moins en grande partie, déterminant pour le spectre lytique des phages de L. lactis.
L’étape suivante est l’entrée de l’ADN viral à l’intérieur de la cellule bactérienne. L'éjection de l’ADN phagique est dépendante du phage avec un maximum de 3000-4000pb/s, soit environ 40 fois plus rapide que la transformation (Letellier et al., 2003). Deux hypothèses existent pour expliquer ce phénomène (Molineux et Panja, 2013). Dans un premier temps, la pression totale à l’intérieur de la capside constituerait une force suffisante pour injecter directement l'ADN, à la façon qu'un coup de revolver à bout portant. Le deuxième modèle fait appel au concept d'osmose où l'eau entrant (en provenance de l'environnement et/ou du cytoplasme) dans la capside conduirait à la sortie de l'ADN par manque d'espace. Les phages peuvent utiliser l'ATP, le potentiel membranaire et/ou la transcription et traduction pour l’éjection de leur génome (Molineux et Panja, 2013). Le coliphage T7 utilise le potentiel membranaire comme source d'énergie. Chez ce phage, certaines protéines (présente dans le virion) vont former un canal pour conduire l’ADN. Après l’entrée de 1 kb, l’ARN polymérase T7 lie le promoteur et tire l'ADN à l’intérieur de la cellule. (Zavriev et Shemyakin, 1982; Moffatt et Studier, 1988; Kemp et al., 2004). Dès son entrée dans le cytoplasme, l'ADN viral linéaire est vite recircularisé par ses extrémités cohésives (cos-type) ou redondantes (pac-type) permettant ainsi d'éviter la dégradation du génome par les exonucléases bactériennes. À titre indicatif, tous les lactophages appartenant au groupe des 936 sont de type cos (extrémités cohésives de 11 nt) (Rousseau et Moineau, 2009). L’ADNdb circulaire formé est ensuite transcrit par des ARN polymérases ADN dépendant d'origine bactérienne. Toutefois, quelques phages comme le coliphage T7 utilisent leur propre ARN polymérases qui sont nettement plus efficaces dans la transcription génique (Hausmann, 1976; Kutter et al., 2005).
La transcription des gènes se fait de façon chronologique où les gènes précoces sont transcrits en premier. L'étude des profils de transcription de deux lactophages appartenant au groupe 936 (sk1 et P008) a démontré que les gènes précoces sont transcrits respectivement entre 2-5 et 2-10 minutes à partir du début de l'infection (Chandry et al., 1994; Samson et al., 2013a). Bien que la majorité des gènes précoces codent pour des protéines aux fonctions hypothétiques (80%), celles-ci semblent être impliquées dans la prise de contrôle de la cellule (Hatfull et Hendrix, 2011). Pour leur part, les gènes médians codent pour des protéines jouant un rôle dans la réplication virale. La réplication produit de long concatémères d’ADN (5-8 génomes) qui seront séparés lors de l'encapsidation (Kutter et al., 2005). Pour le phage sk1, ces gènes
sont transcrits entre 7 à 10 minutes (Chandry et al., 1994). Finalement, il y a transcription des gènes tardifs qui codent pour les protéines de structure, d'assemblage et de lyse cellulaire. Chez les phages caudés, les capsides et les queues sont produites et assemblées séparément un de l'autre (Kutter et al., 2005). L'ADN viral est d’abord encapsidé sous l'action des terminases qui sont liées à la procapside. Ces enzymes permettent également la coupure du concatémère d’ADN et ainsi l'insertion d'un génome unique et linéaire, ce qui forme la capside virale (Kutter et al., 2005). Le virion mature est ensuite formé grâce aux protéines du collet (portale) qui lie la queue.
La lyse cellulaire constitue la dernière étape du cycle lytique. Différentes protéines jouant un rôle dans la lyse ont déjà été identifiées chez les lactophages. Parmi elles, la holine forme des pores membranaires qui permettent à l'endolysine d'atteindre le peptidoglycane, et d'induire la lyse cellulaire et le relargage des nouveaux virions (Catalão et al., 2013). Pour les lactophages, un cycle de réplication moyen dure 50 minutes, et permet de relarguer environ 100 nouveaux virions (données non publiées).
Mécanismes anti-phages
Les phages sont toujours présents dans l’environnement et représentent une menace constante pour les bactéries. Ce stress constant a mené ces bactéries à développer une panoplie de mécanismes afin de résister à l’infection par les phages (Labrie et al., 2010). Ces mécanismes anti-phages sont divisés en quatre groupes en fonction du moment où ils interviennent au cours du cycle lytique de réplication. Pour une revue complète, le lecteur peut consulter cette revue (Labrie et al., 2010).
1. Inhibition de l'adsorption
Comme mentionnée précédemment, l’adsorption du phage à la cellule est essentielle à la réplication virale. L'idée est donc fort simple, prévenir la reconnaissance de la bactérie par le phage. Pour ce faire, les bactéries peuvent induire des modifications directes ou indirectes sur les récepteurs reconnus par les bactériophages.
Une des stratégies bactériennes est la synthèse d’une matrice extracellulaire ayant pour effet de masquer les récepteurs de surface reconnus par les phages. À titre d'exemple, il a été démontré que la bactérie E.
coli K1 capsulée était en mesure de résister à l'infection par le phage T7 et que cette résistance est perdue
lors de l'élimination de cette capsule (Scholl et al., 2005). Chez certains phages de lactocoque, l'interférence d'adsorption serait causée par l'augmentation de lipides dans les acides téichoïques et la production de galactose et/ou rhamnose à la surface extracellulaire (Garvey et al., 1995). La résistance associée à ces
polymères extracellulaires n'est toutefois pas très efficace (Deveau et al., 2002; Mahony et al., 2013). En effet, certains phages possèdent des enzymes capables de dégrader ces exopolysaccharides, leur permettant ainsi d'atteindre leur récepteur spécifique (Sutherland et al., 2004).
Les bactéries peuvent également produire un compétiteur ayant une forte affinitée pour le récepteur du phage. Un exemple intéressant fait intervenir le peptide antimicrobien microcin J25 qui est connu pour l'inhibition de croissance bactérienne en conditions faibles en nutriments. Sous ce stress, le peptide J25 produit se lie au transporteur de fer FhuA (récepteur du phage), compétitionnant ainsi l'infection par les coliphages comme T1, T5 et Phi80 (Destoumieux-Garzón et al., 2005).
Le phénomène de variation de phase se caractérise par une modification à haute fréquence et variable dans le temps, de certains constituants cellulaires. Ces changements sont induits par recombinaison de sites spécifiques, mésappariement de séquences répétées ou modifications épigénétiques (Henderson et
al., 1999). Certaines bactéries comme Bordetella spp. peuvent utiliser la variation de phase pour modifier
leur composition de surface, et ainsi résister au stress phagique (Uhl et Miller, 1996). En effet, le phage BPP-1 peut infecter efficacement la bactérie uniquement dans la phase Bvg+ (virulence), moment où son
récepteur spécifique est exprimé (l'autotransporteur de pertactine (Prn)) (Beier et Gross, 2008). D’un autre côté, certains phages mutants ont acquis la capacité d'infecter les cellules n'exprimant pas ce récepteur (Bvg-). Les taux d'infection sont toutefois nettement inférieurs (Liu et al., 2002). Également,
certains phages de Bordetella peuvent contourner cette résistance en utilisant des « diversity-generating retroelements » (DGR) (Liu et al., 2002). Ces éléments génétiques permettent des substitutions nucléotidiques dans une région variable du gène phagique mtd « Major tropism determinant » et modifie grandement son tropisme (Doulatov et al., 2004). Des analyses génomiques ont permis d'identifier d'autres DGRs provenant de génomes phagiques, mais également de génomes bactériens (Medhekar et Miller, 2007; Simon et Zimmerly, 2008).
Bien qu'une multitude de stratégies puissent être utilisées pour bloquer l'adsorption, celles-ci peuvent être contournées par les phages (Labrie et al., 2010).
2. Bloquer l'injection de génome viral
Les bactéries peuvent utiliser les systèmes d'exclusion de type superinfection (Sie) comme deuxième barrière de défense contre les phages. Ceux-ci sont composés de protéines qui bloquent spécifiquement l'entrée de l'ADN viral, produisant ainsi une immunité spécifique contre certains phages. Cette stratégie est utilisée par les prophages afin de prévenir l'infection par un autre phage, laquelle conduirait à la mort de la cellule dans lequel il est intégré. À ce jour, peu de mécanismes Sie ont été caractérisés. Parmi eux, le système Sie2009 isolé de L. lactis fut identifié à l'intérieur du génome d'un phage tempéré nommé Tuc2009 (McGrath et al., 2002). Les protéines de ce système s'associent à la membrane pour prévenir l'entrée de l'ADN (McGrath et al., 2002). D’autres Sie de lactocoques ont une activité spécifique contre certains sous-groupes de phages chez les 936 (Mahony et al., 2008).
Bien que rare, il existe des mécanismes Sie n'étant pas codés par des prophages. Le Sie imm/sp (coliphage T4) est un exemple qui a d’ailleurs été très bien caractérisé. Respectivement, les protéines virales imm et sp changent la conformation des sites d'injection et inhibent l'activité du lysozyme T4, ce qui prévient ainsi la dégradation du peptidoglycane (Lu et al., 1993; Lu et Henning, 1994). L'activité de ce système permet de prévenir l'infection par d'autres phages de types T-paires au cours de la réplication du phage T4 (Lu et Henning, 1994).
3. Coupure des acides nucléiques exogènes
A. Systèmes de restriction-modification
Une fois l'ADN viral à l'intérieur de la cellule, celui-ci peut faire face aux systèmes de restriction-modification (R/M). Ces systèmes font intervenir 2 à 3 protéines qui occupent les fonctions de méthylase ou de reconnaissance/restriction. La méthylase permet de modifier l'ADN de l'hôte en ajoutant un groupement méthyle à un site spécifique. Cela a pour effet de protéger l’hôte puisque l’ADN méthylé n’est pas reconnu ou coupé par l'enzyme de restriction (Pingoud et al., 2005). Ces systèmes de défense sont répandus chez les bactéries et les archée dans pas moins de 95 et 99,5% des cas, respectivement (Roberts et al., 2010). À titre d'exemple, les souches de laboratoire L. lactis ssp. cremoris MG1363 et L. lactis ssp. lactis IL1403 contiennent chacun trois systèmes R/M sur leur chromosome. D'autres souches telles
L. lactis ssp. Lactis CV56, qui contient 5 plasmides, possède plusieurs R/M plasmidiques et
chromosomiques (Roberts et al., 2010; Gao et al., 2011).
Il existe quatre types de système R/M (I-IV) qui sont définis selon leur nécessité d'un cofacteur spécifique, la nature des sites de reconnaissance, leur site de coupure et la structure des enzymes (Roberts et al., 2003).
La majorité des enzymes de restriction décrites à ce jour appartiennent au R/M de type II, malgré que les analyses génomiques suggèrent une incidence de seulement 45% (Roberts et al., 2003). Cette différence s'explique par les projets d'identification/caractérisation de R/M qui ciblent plus spécifiquement les types II en raison de leur utilité dans les technologies de l'ADN recombinant (Pingoud et al., 2005). Les R/M type II sont habituellement homodimères et coupent l'ADN à l'intérieur, ou près, des sites de reconnaissance (courtes séquences palindromique) en présence de Mg++ comme cofacteur (Pingoud et
al., 2005).
Le système LlaDCH-4 (isolé de L. lactis ssp. cremoris DCH-4) fut le premier système de L. lactis pour lequel la séquence de reconnaissance et la structure génétique ont été identifiées (Moineau et al., 1995). Fait intéressant, ce système est fort efficace contre les groupes de phages prédominants en industrie laitière, soit c2, P335 et 936 (Moineau et al., 1995). Plusieurs autres R/M de lactocoques ont également été isolés et caractérisés au cours des années (Roberts et al., 2010).
De leur côté, les phages ont développé différentes stratégies afin de contourner les systèmes de restriction-modification. Considérant que l'efficacité du système est directement proportionnelle au nombre de sites de reconnaissance, certains phages accumulent des mutations ponctuelles afin de réduire leurs séquences de reconnaissance (Krüger et al., 1988; Wilson et Murray, 1991). Également, il a été démontré que certains lactophages peuvent acquérir le gène codant pour une méthylase dans leur génome, et ainsi s'auto protéger contre l'action de l'endoribonucléase du même système R/M (Hill et al., 1991; McGrath et al., 1999). Pour sa part, le coliphage T7 utilise une autre stratégie qui est tout aussi ingénieuse. Le gène 0.3 code pour la protéine Ocr qui permet la séquestration des enzymes de restriction en imitant la structure en B de l'ADNdb viral (Walkinshaw, 2002). Finalement, certains phages comme T4 utilisent des bases inhabituelles ou la glycosylation pour constituer leur ADN génomique (Krüger et Bickle, 1983; Labrie et al., 2010). L'essentiel des stratégies d'adaptation des phages sont développées plus en détail dans la revue de Samson et al. (Samson et al., 2013b).
b. CRISPR-Cas
Les systèmes CRISPR-Cas (« Clustered Regularly Interspaced Short Palindromic Repeats – CRISPR associated genes ») sont très répandus chez les bactéries, et ubiquitaires chez les archées. Une étude ciblant 102 bactéries lactiques a permis d'identifier 66 systèmes CRISPR-Cas distribués dans 26 souches différentes (Horvath et al., 2008). Malgré l'abondance, un seul système actif chez L. lactis a pu être identifié jusqu'à présent, et celui-ci est retrouvé sur un plasmide (Millen et al., 2012).
Ces systèmes sont composés d'un locus CRISPR et de gènes associés appelés cas (Jansen et al., 2002). Ces locus contiennent des séquences répétées d'environ 40 nt qui sont séparées par des espaceurs variables
non répétés de 21 à 72 nt de longueur (Barrangou et Horvath, 2012). Les espaceurs sont issus d'ADN exogène, généralement de plasmides ou d'ADN de phage (proto-espaceurs) (Garneau et al., 2010). Contrairement aux mécanismes décrits précédemment, le système CRISPR-Cas constitue une immunité dite adaptative. L'incorporation de ces espaceurs, dans la majorité des cas en 5' du locus (près de la région leader), constitue la première étape du système appelé l'adaptation (Fineran et Charpentier, 2012). La deuxième étape est la synthèse du transcrit ARN pré-crispr qui sera plus tard coupé et maturé par certaines protéines Cas (Terns et Terns, 2011). Les transcrits d'ARNcr sont utilisés dans la reconnaissance de séquence complémentaire (proto-espaceur) ainsi que la coupure de ces ARN exogènes (étape d’interférence). Les systèmes CRISPR-Cas sont présentement séparés en trois types et en 10 subdivisions en fonction notamment de leur séquence répétée et des protéines Cas (Makarova et al., 2011). Les systèmes CRISPR-Cas de types I et II ont besoin d'une séquence PAM (motif adjacent au proto-espaceur) (Mojica et al., 2009). Cette séquence est importante pour l'efficacité du système contre les phages (Deveau
et al., 2008). La figure 1.7. démontre les différentes étapes des systèmes CRISPR-Cas : l'adaptation,
synthèse de l’ARN pré-cr et l'interférence.
Les phages peuvent contourner ce type de système de résistance par différentes stratégies fort intéressantes. De façon similaire à d'autres stratégies décrites plus tôt, le phage peut acquérir une résistance par mutation ponctuelle. Celle-ci peut être retrouvée dans la séquence PAM ou directement dans le proto-espaceur, inhibant ainsi l'interférence (Deveau et al., 2008). Cette stratégie est toutefois peu efficace considérant le nombre très élevé de proto-espaceurs, et la distribution quasi complète de ceux-ci sur le génome phagique (Deveau et al., 2008). Il a été décrit récemment que certains phages tempérés infectant le pathogène Pseudomonas aeruginosa possèdent des gènes anti-crispr dont les protéines agiraient au niveau du complexe d'interférence (Bondy-Denomy et al., 2013). De plus, des analyses bio-informatiques suggèrent que ces gènes pourraient être facilement transférés horizontalement puisqu'on les retrouvent sur des éléments d’ADN mobiles putatifs (Bondy-Denomy et al., 2013).
Tout récemment, Seed et al. ont démontré la présence d'un système CRISPR-Cas fonctionnel chez un phage de Vibro cholerae (Seed et al., 2013). Ce dernier cible le «phage-inducible chromosomal island-like element » (PLE) qui procure normalement une résistance phagique (Seed et al., 2013).
4. Avortement de l'infection (Abi)
Malgré la panoplie de systèmes antiphages décris, et leur rapidité d'adaptation, les phages trouvent toujours un moyen de les contourner et de se répliquer. Pour cette raison, les bactéries possèdent également des systèmes de résistance de « dernière chance » connus sous le nom de mécanismes d’avortement de l'infection (Abi). Contrairement aux systèmes décrits plus tôt, les Abis causent la mort bactérienne des cellules infectées et de cette façon préviennent la réplication et dissémination des phages. Outre la mort cellulaire, les Abis peuvent être identifiés par une diminution de l’efficacité à former des plages de lyse (EOP) et une diminution de la taille de ces plages (Josephsen et Neve, 2004). Jusqu'à présent, très peu d'Abis ont été caractérisés en détail dans la littérature.
Chez E. coli, on retrouve le système à deux composantes Rex dont les gènes (rexA et rexB) sont retrouvés sur le prophage de souches lambda-lysogéniques (Molineux, 1991). La protéine RexA joue un rôle dans la reconnaissance de l'infection virale en identifiant la présence d'un complexe d'ADN-protéines comme intermédiaires de réplication ou de recombinaison durant l’infection (Snyder, 1995). Une fois reconnu, le senseur (RexA) active le régulateur de réponse (RexB). RexB est une protéine transmembranaire formant des canaux ioniques qui mènent à la perte du potentiel membranaire et à l’abaissement du niveau d'ATP (Snyder, 1995). Fait intéressant, cette protéine (RexB) permet également d'inhiber l'activité des systèmes toxine-antitoxine Phd-Doc et MazEF, contrôlant ainsi directement la survie bactérienne (Engelberg-Kulka et al., 1998). Les phages pouvant contourner le système Rex possèdent une mutation dans le gène
motA, un coactivateur transcriptionnel qui agit sur la dépolarisation de la membrane en modifiant l'affinité
de l'ARN polymérase bactérienne (Hinton, 2010).
Le système d'exclusion T7 dépendant du plasmide F utilise une stratégie similaire pour le contrôle de l'infection virale. En effet, une suite d'événements mène à l'altération de la perméabilité membranaire, ce qui cause une perte d’ATP (Schmitt et al., 1991). De plus, ce système affecte significativement la synthèse des macromolécules ainsi que l'injection du génome virale (Molineux, 1991; García et Molineux, 1995). Chez le phage T7, les protéines issues des gènes gp1.2 et gp10 (impliquées respectivement dans le contrôle de l'énergie lors du cycle de réplication et la synthèse de la protéine majeure de capside) sont séquestrées par l'interaction avec la protéine membranaire de l'hôte PifA (Condreay et Molineux, 1989; Nakai et Richardson, 1990). Des mutations dans ces gènes viraux peuvent permettre de contourner la résistance (Molineux, 1988). Il a aussi été démontré qu'une protéine transmembranaire de l'hôte (FxsA) peut inhiber l'efficacité du système en interagissant directement avec PifA (Wang et al., 1999; Cheng et al., 2004). Toujours chez E. coli, les systèmes Abi PrrC et Lit/Gol sont également bien caractérisés. Ceux-ci agissent par l'inhibition de la machinerie traductionnelle afin de prévenir l'infection par les phages (Uzan et Miller, 2010). La protéine PrrC permet la coupure de l'ARN de transfert Lysine (ARNt Lys) en ciblant sa région
anticodon, prévenant ainsi la synthèse de protéines chez les phages sans polynucléotides kinase ou ARN ligase comme T4 (Snyder, 1995). L'activité de PrrC est régulée par l'association d'une enzyme de restriction « prrD-encoded restriction enzyme », dont le gène est d'ailleurs retrouvé dans la même cassette génétique (Snyder, 1995). Le peptide Stp permet la déstabilisation de l'interaction, le relâchement de PrrC, et donc l'avortement de l'infection virale (Snyder, 1995; Uzan et Miller, 2010).
Le système Lit/Gol implique le gène lit retrouvé sur le prophage défectueux e14 intégré dans le génome de la souche E. coli K12 (Hill et al., 1989b). Ce gène code pour protéine (Lit) possédant un domaine métalloprotéinase zinc dépendant qui, une fois activé, coupe le facteur d'élongation EF-Tu dans une région conservée chez la bactérie et même chez l'humain (Bergsland et al., 1990; Yu et Snyder, 1994; Georgiou et al., 1998). Le peptide viral Gol, présent dans la protéine majeure de capside, permet l’activation de Lit (Bingham et al., 2000). La modification de certains acides aminés clés permet de prévenir l'interaction, toutefois une activité résiduelle est observable à haute concentration protéique (Georgiou et
Systèmes d'avortement de l'infection chez L. lactis
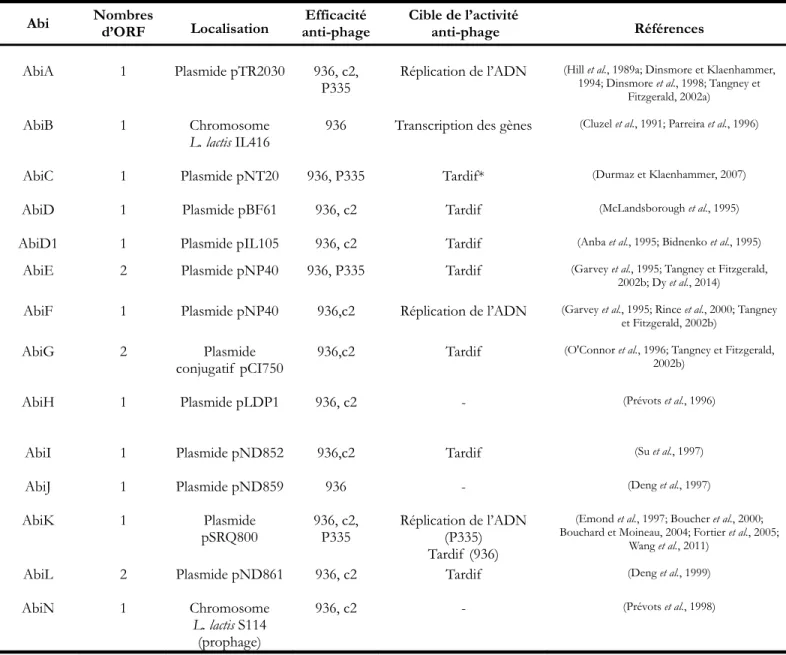
Jusqu'à présent, un nombre impressionnant de 23 Abis ont pu être identifiés dans différentes souches de
L. lactis (Chopin et al., 2005; Durmaz et Klaenhammer, 2007; Holubova et Josephsen, 2007; Haaber et al.,
2008). Un résumé de ces Abis est présenté dans le tableau 1.3.
Tableau 1.3. Regroupement des mécanismes Abi de lactocoques. Adaptée de (Chopin et al., 2005).
Abi Nombres d’ORF Localisation anti-phageEfficacité Cible de l’activité anti-phage Références
AbiA 1 Plasmide pTR2030 936, c2,
P335 Réplication de l’ADN
(Hill et al., 1989a; Dinsmore et Klaenhammer, 1994; Dinsmore et al., 1998; Tangney et
Fitzgerald, 2002a)
AbiB 1 Chromosome
L. lactis IL416 936 Transcription des gènes
(Cluzel et al., 1991; Parreira et al., 1996)
AbiC 1 Plasmide pNT20 936, P335 Tardif* (Durmaz et Klaenhammer, 2007)
AbiD 1 Plasmide pBF61 936, c2 Tardif (McLandsborough et al., 1995)
AbiD1 1 Plasmide pIL105 936, c2 Tardif (Anba et al., 1995; Bidnenko et al., 1995)
AbiE 2 Plasmide pNP40 936, P335 Tardif (Garvey et al., 1995; Tangney et Fitzgerald, 2002b; Dy et al., 2014)
AbiF 1 Plasmide pNP40 936,c2 Réplication de l’ADN (Garvey et al., 1995; Rince et al., 2000; Tangney et Fitzgerald, 2002b)
AbiG 2 Plasmide
conjugatif pCI750 936,c2 Tardif
(O'Connor et al., 1996; Tangney et Fitzgerald, 2002b)
AbiH 1 Plasmide pLDP1 936, c2 - (Prévots et al., 1996)
AbiI 1 Plasmide pND852 936,c2 Tardif (Su et al., 1997)
AbiJ 1 Plasmide pND859 936 - (Deng et al., 1997)
AbiK 1 Plasmide
pSRQ800 936, c2, P335 Réplication de l’ADN (P335) Tardif (936)
(Emond et al., 1997; Boucher et al., 2000; Bouchard et Moineau, 2004; Fortier et al., 2005;
Wang et al., 2011)
AbiL 2 Plasmide pND861 936, c2 Tardif (Deng et al., 1999)
AbiN 1 Chromosome L. lactis S114
(prophage)
936, c2 - (Prévots et al., 1998)