Caractérisation structurale et de la liaison membranaire
de la RGS9-1 Anchor Protein (R9AP)
Thèse
Sarah Bernier
Doctorat en biologie cellulaire et moléculaire
Philosophiæ doctor (Ph. D.)
Caractérisation structurale et de la liaison
membranaire de la RGS9-1 Anchor Protein
(R9AP)
Thèse
Sarah Bernier
Sous la direction de :
Résumé
La vision est rendue possible grâce à la conversion du signal lumineux en un signal électrique par la cascade de phototransduction visuelle, qui implique plusieurs protéines incluant la phosphodiestérase (PDE). L’inactivation des différentes protéines de la phototransduction est primordiale pour que les photorécepteurs retrouvent leur sensibilité aux changements d’intensité lumineuse. Au cours de ce processus, un complexe de protéines incluant la R9AP (RGS9-1 Anchor Protein) inactive la PDE. La R9AP permet l’ancrage d’un complexe protéique à la membrane des disques des photorécepteurs via son segment C-terminal hydrophobe. Des mutations au niveau de la séquence de la R9AP mènent à une maladie appelée bradyopsie qui se caractérise notamment par une photophobie et une difficulté à suivre des objets en mouvement. Cette maladie peut être causée par la perte de la liaison membranaire de la R9AP en raison de mutations menant à une modification de la séquence en acides aminés de son segment C-terminal. La liaison membranaire de la R9AP joue donc un rôle majeur dans l’inactivation de la PDE. Par contre, aucune donnée de liaison membranaire et structurale n’est disponible pour cette protéine. Nous avons donc initié la caractérisation de la structure et de la liaison membranaire de différentes formes de la R9AP, soit la protéine avec et sans son segment C-terminal (∆TM, R9AP∆TM) ainsi que le segment C-terminal seul. Afin d’obtenir la R9AP pure, nous avons cloné, surexprimé et purifié la R9AP∆TM en fusion avec différentes étiquettes de solubilisation/purification. Les protéines recombinantes ont été produites à l’aide d’un système d’expression bactérien. En plus de permettre d’obtenir la R9AP pure pour caractériser sa structure et sa liaison membranaire, ces travaux ont significativement contribué à faire avancer les connaissances à propos de l’utilisation des étiquettes de purification/solubilisation en fusion avec une protéine d’intérêt. En effet, nous avons effectué une étude systématique pour étudier l’impact de la conception des protéines de fusion sur la solubilité, l’expression et la purification de protéine d’intérêt. Il s’agit de la première étude systématique évaluant l’effet du positionnement et de l’identité des différentes
étiquettes de purification/solubilisation sur ces paramètres. Également, la production des protéines recombinantes a permis d’identifier un site alternatif d’initiation de la traduction dans la séquence de l’étiquette GST (glutathione S-transférase) qui cause l’expression d’une protéine de fusion tronquée. Cette observation aura certainement un fort impact compte tenu de l’utilisation répandue de l’étiquette GST.
Concernant les résultats obtenus avec la R9AP, des données de centrifugation ont montré que la protéine complète est beaucoup moins soluble que la R9AP∆TM, ce qui suggère un rôle important du segment C-terminal hydrophobe dans la solubilité de cette protéine. Ainsi, seulement la structure et la liaison membranaire de la R9AP∆TM ont pu être investiguées dans le cadre de cette thèse. Des mesures par dichroïsme circulaire et spectroscopie infrarouge ont montré que la R9AP∆TM ainsi que le peptide C-terminal sont majoritairement constitués d’hélices alpha, ce qui appuie les prédictions structurales de cette protéine et un rôle d’ancrage membranaire de son segment C-terminal. Également, les mesures de liaison membranaire à l’aide des monocouches de Langmuir ont montré que ce segment C-terminal seul possède une forte affinité pour la majorité des phospholipides qui sont représentatifs de la composition lipidique des photorécepteurs. En revanche, la R9AP sans son segment transmembranaire a montré une faible affinité pour la majorité de ces phospholipides. Ainsi, nos travaux suggèrent fortement que la spécificité de liaison membranaire de la R9AP est en majorité dictée par son segment C-terminal, ce qui supporte son rôle important dans l’ancrage du complexe protéique aux membranes des disques des photorécepteurs et dans la bradyopsie.
Abstract
Visual phototransduction involves many proteins including phosphodiesterase, which leads to photoreceptor hyperpolarization and then signal transmission to the brain. Inactivation of the different proteins involved in this process is essential such that photoreceptors remain sensitive to changes in light intensity. In the course of this inactivation, a protein complex including R9AP (RGS9-1 Anchor Protein) inactivates phosphodiesterase (PDE). R9AP anchors a protein complex to disk membranes of the photoreceptor outer segments most likely by use of its C-terminal hydrophobic domain. Mutations in the coding sequence of R9AP lead to a visual disease called bradyopsia, which results in problems with adjusting to light variations and difficulties to follow moving objects. This disease can be caused by the loss of the membrane binding of R9AP as a result of mutations that modify the amino acid sequence of its C-terminal domain. Membrane binding of R9AP thus plays a major role in the inactivation process of PDE. However, membrane binding and structural data are still lacking for this particular protein. We have thus initiated the characterization of the structure and membrane binding of R9AP, including the full-length protein, R9AP without its terminal domain (R9AP∆TM), as well as its C-terminal domain alone.
In order to get pure R9AP, we have cloned, overexpressed and purified R9AP∆TM in fusion with solubility-enhancing/purification tags. Recombinant proteins were expressed using a bacterial expression system. This study allowed us to develop a procedure to obtain pure R9AP∆TM as well as to significantly improve our understanding of the use of fusion proteins. Indeed, we have performed a systematic analysis of the impact of the design of fusion proteins on their solubility, expression and purification. This study was the first one to evaluate the effect of both the identity and the position of the tags on the solubility, expression and purification of proteins of interest. Also, the production of R9AP∆TM recombinant proteins allowed us to identify an alternative translation initiation site in the coding sequence of the GST (glutathione S-transferase) tag, which results in the expression of a truncated fusion
protein. This finding will certainly have an important impact when considering the extensive use of the GST tag.
Results have shown that the R9AP∆TM protein is much more soluble than the full-length protein, which suggests a major role of the C-terminal domain of R9AP for its solubility. Thus, the structure and the membrane binding of R9AP∆TM have been investigated within the scope of this thesis. Infrared spectroscopy as well as circular dichroism measurements have allowed determining that R9AP∆TM as well as the C-terminal domain adopt an alpha-helical structure, which is in good agreement with both the predicted structure of R9AP and the transmembrane role of its C-terminal domain. Also, Langmuir monolayer measurements revealed that the C-terminal segment has a high affinity for most of the phospholipids found in photoreceptor membranes. In contrast, R9AP∆TM has a low affinity for these phospholipids. Thus, our results demonstrate that the membrane binding of R9AP is highly dependent on its C-terminal segment, which is consistent with its important role in anchoring the protein complex to disk membranes of the photoreceptor outer segments and in bradyopsia.
Table des matières
Résumé ... iii
Abstract ... v
Table des matières ... vii
Liste des figures ... xi
Liste des tableaux ... xviii
Liste des abréviations ... xx
Remerciements ... xxiii Avant-propos ... xxvi Introduction ... 1 L’œil ... 1 La rétine ... 2 Les photorécepteurs ... 4
• Structure des cônes et des bâtonnets ... 6
• Fonction des photorécepteurs ... 7
• Environnement membranaire du segment externe des bâtonnets (SEB) ... 10
La phototransduction visuelle ... 12
• Activation de la phototransduction visuelle ... 12
• Inactivation de la phototransduction visuelle ... 15
• Régénération du pigment visuel ... 18
Le complexe protéique RGS9/Gb5/R9AP ... 19
• Caractéristiques de la RGS9-1 ... 20
• Caractéristiques de la Gb5L ... 22
• La R9AP : état d’avancement des connaissances ... 23
• Participation du complexe RGS9-1/Gb5L/R9AP dans la bradyopsie .... 26
Procédures expérimentales... 28
• Production de protéines recombinantes ... 28
o Surexpression chez E. coli ... 30
o Les protéines de fusion ... 36
- Les étiquettes de solubilisation ... 36
- Les étiquettes de purification ... 37
- Les sites de clivage ... 39
o Les protéines membranaires ... 41
o Les propriétés des phospholipides ... 43
o Les systèmes membranaires modèles ... 44
o Les monocouches de Langmuir : mesures de pression de surface et détermination de la pression d’insertion maximale ... 46
• Analyses spectroscopiques de la structure secondaire ... 49
o Dichroïsme circulaire (CD) ... 49
o Spectroscopie infrarouge par réflexion totale atténuée (ATR) ... 50
o Spectroscopie infrarouge de réflexion-absorption par modulation de polarisation (PM-IRRAS) ... 54
• Spectroscopie de fluorescence ... 56
o Principe ... 56
o Interaction membranaire par des mesures de fluorescence ... 58
o Atténuation de la fluorescence ... 59
Contexte, hypothèse et objectifs des travaux de recherche ... 60
• Clonage, surexpression et purification de la R9AP avec et sans segment C-terminal ... 62
• Étude de la liaison membranaire du segment C-terminal de la R9AP ... 63
• Caractérisation de la structure secondaire et de la liaison membranaire de la R9AP bovine sans segment C-terminal ... 64
Chapitre 1 : Systematic analysis of the expression, solubility and purification of a passenger protein in fusion with different tags ... 65
1.1 Résumé en français ... 66
1.2 Abstract ... 67
1.3 Introduction ... 68
1.4 Materials and methods ... 69
1.5 Results ... 82 1.6 Discussion ... 96 1.7 Conclusions ... 101 1.8 Acknowledgements ... 102 1.9 References ... 103 1.10 Supplementary data ... 111
Chapitre 2 : Identification of an alternative translation initiation site in the sequence of the commonly used Glutathione S-Transferase tag ... 113
2.1 Résumé en français ... 114
2.2 Abstract ... 115
2.4 Acknowledgements ... 122
2.5 References ... 123
Chapitre 3 : Structure and binding of the C-terminal segment of R9AP to lipid monolayers ... 125 3.1 Résumé en français ... 126 3.2 Abstract ... 127 3.3 Introduction ... 128 3.4 Experimental section ... 130 3.5 Results ... 133 3.6 Discussion ... 151 3.7 Conclusion ... 159 3.8 Acknowledgements ... 159 3.9 References ... 161 3.10 Supplementary data ... 166
Chapitre 4 : Structure and membrane binding of truncated RGS9-1 Anchor Protein... 176
4.1 Résumé en français ... 177
4.2 Abstract ... 178
4.3 Introduction ... 179
4.4 Materials and methods ... 180
4.5 Results ... 188 4.6 Discussion ... 202 4.7 Acknowledgments ... 205 4.8 References ... 207 4.9 Supplementary data ... 212 Conclusions et perspectives ... 216
Utilisation des protéines de fusion pour la surexpression et la purification de protéines d’intérêt ... 216
• La conception des protéines de fusion a un impact sur l’expression, la solubilité et la purification des protéines ... 216
• Identification d’un site alternatif d’initiation de la traduction dans la séquence codante de l’étiquette de purification/solubilisation GST ... 220
• La spécificité de la liaison membranaire de la R9AP dépend de son
segment C-terminal ... 222
• Caractérisation de la structure secondaire de la R9AP ... 227
Perspectives des travaux de recherche ... 230
Bibliographie ... 237
Annexe 1. Comparison between the behavior of different hydrophobic peptides allowing membrane anchoring of proteins ... 265
A1.1 Résumé en français ... 266
A1.2 Abstract ... 267
A1.3 Contents ... 268
A1.4 Supplementary information ... 327
Annexe 2. How to decipher protein and peptide selectivity for lipids in monolayers ... 336
A2.1 Résumé en français ... 337
A2.2 Abstract ... 338
A2.3 Introduction ... 339
A2.4 Materials and methods ... 340
A2.5 Results and discussion ... 342
A2.6 Conclusion ... 353
A2.7 Acknowledgments ... 353 -
Liste des figures
Introduction
Figure I.1. Représentation schématique de l’œil humain et de ses différentes
structures. ... 2
Figure I.2. Schéma de la rétine humaine. ... 3
Figure I.3. Distribution des cônes et des bâtonnets dans la rétine neurale. ... 5
Figure I.4. Représentation schématique des photorécepteurs de type cône et bâtonnet de la rétine. ... 6
Figure I.5. Structure de la rhodopsine et son organisation dans la membrane des disques des bâtonnets. ... 8
Figure I.6. Spectre d’absorption des pigments visuels des cônes (bleu, vert et rouge) et du pigment visuel des bâtonnets (rhodopsine). ... 9
Figure I.7. Schéma et nomenclature des phospholipides. ... 10
Figure I.8. Activation de la phototransduction visuelle. ... 13
Figure I.9. Mécanismes d’inactivation de la phototransduction visuelle ... 16
Figure I.10. Cycle visuel des rétinoïdes. ... 18
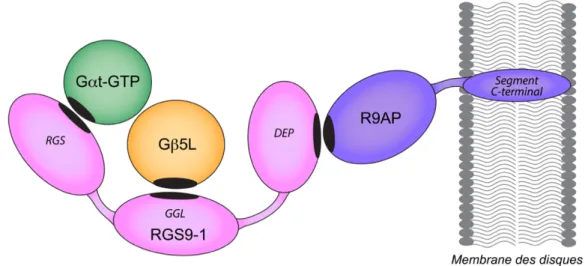
Figure I.11. Complexe RGS9-1/Gb5L/R9AP impliqué dans l’inactivation de la phosphodiestérase. ... 21
Figure I.12. Structure de l’hétérodimère RGS9/Gb5 (PDB 2PBI). ... 22
Figure I.13. Représentation schématique de la structure de la R9AP. ... 23
Figure I.14. Mutations dans la séquence codante de la R9AP qui causent la bradyopsie en conséquence d’un décalage du cadre de lecture. ... 28
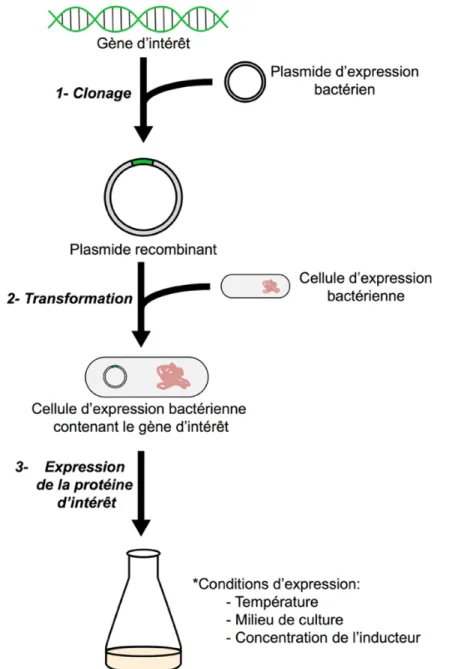
Figure I.15. Schéma présentant les principales étapes pour l’expression de protéines recombinantes chez E. coli. ... 31
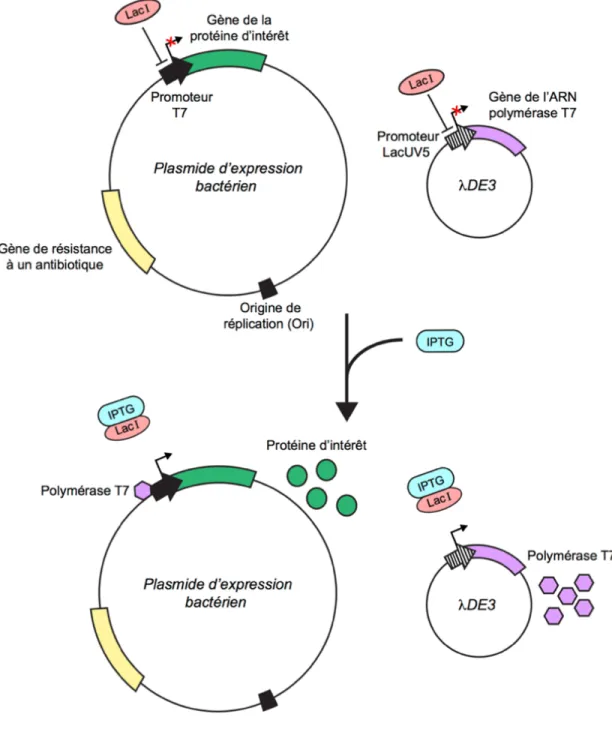
Figure I.16. Aperçu des mécanismes moléculaires de l’induction de l’expression de la protéine d’intérêt suite à l’ajout d’IPTG. ... 32
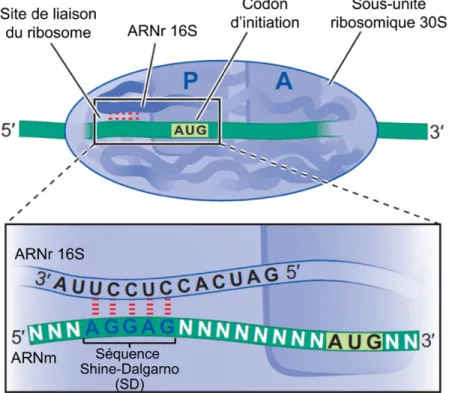
Figure I.17. Site de liaison du ribosome pour l’initiation de la traduction chez E. coli. ... 33
Figure I.18. Représentation schématique d’une membrane biologique constituée de lipides et de protéines membranaires. ... 41
Figure I.19. Prévalence de la localisation des acides aminés dans une hélice a transmembranaire. ... 42
Figure I.20. Modèles membranaires utilisés pour étudier les interactions protéines-lipides. ... 45
Figure I.21. Monocouches de Langmuir. ... 48
Figure I.22. Polarisation circulaire de la lumière et obtention des différences d’absorption (spectre CD). ... 49
Figure I.23. Spectres CD caractéristiques des structures secondaires protéiques ... 50 Figure I.24. Représentation schématique du principe de l’ATR. ... 51 Figure I.25. Origine des bandes amide I et II observées par spectroscopie
infrarouge. ... 52 Figure I.26. Représentation schématique du montage expérimental d’un
PM-IRRAS. ... 54 Figure I.27. Simulations de spectres PM-IRRAS (région amide I et II) d’hélices
a pures à différents angles en monocouche à l’interface air/eau. ... 55 Figure I.28. Diagramme de Jablonski simplifié. ... 56 Figure I.29. Spectres d’absorption et d’émission des acides aminés
aromatiques (Phe, Tyr et Trp) dans l’eau à pH neutre. ... 57 Figure I.30. Effet de l’environnement du tryptophane sur son spectre d’émission
... 58 Figure I.31. Atténuation de la fluorescence d’un résidu tryptophane enfoui (W1)
ou exposé à la surface (W2) d’une protéine. ... 60 Figure I.32. Hypothèse et objectif de recherche... 62
Chapitre 1
Figure 1.1. Schematic representation of all constructs. ... 72 Figure 1.2. Overview of the cloning procedure of the expression plasmids used
to prepare the different fusion proteins. ... 75 Figure 1.3. SDS-PAGE analysis of the expression and solubility of the
GST-bR9AP (A), MBP-GST-bR9AP (B), His10-bR9AP (C), His10-GST-bR9AP (D), GST-His10-bR9AP (E), GST-bR9AP-His10 (F), His10-MBP-bR9AP (G), MBP-His10-bR9AP (H) and MBP-His6-bR9AP fusion proteins (I). ... 84 Figure 1.4. SDS-PAGE analysis of the expression of (A) human and (B) bovine
R9AP expressed using the pET-11a plasmid in fusion with His10-GST at the N-terminus. ... 86 Figure 1.5. SDS-PAGE of the purification of the GST-bR9AP (A) and the
MBP-bR9AP (B) fusion proteins as well as both SDS-PAGE and western blot analyses of the purification of the His10-bR9AP fusion protein using an anti-His antibody (C). ... 88 Figure 1.6. SDS-PAGE of the purification by IMAC of bR9AP expressed using
the pGEX-4T-1 plasmid in fusion with both (A) His10-GST and (B) GST-His10 at the N-terminus as well as (C) GST at the N-terminus and His10 at the C-terminus. ... 91 Figure 1.7. SDS-PAGE of the purification by IMAC of bR9AP expressed using
the pMAL-c2X plasmid in fusion with (A) His10-MBP, (B) MBP-His10 and (C) MBP-His6 at the N-terminus. ... 94
Figure 1.S1. SDS-PAGE of the purification of the supernatant of GST-bR9AP-His10 fusion protein by IMAC. ... 112
Chapitre 2
Figure 2.1. A) SDS-PAGE showing the full-length and truncated fusion protein in different fractions of the expression (Uninduced and Induced with IPTG) and after purification of the GST-bR9AP fusion protein. B) Western blot with the anti-GST polyclonal antibody was performed to visualize wild-type GST before (Induced) and after purification (Purified) using a GSTrap column. ... 117 Figure 2.2. Partial representation of the predicted two-dimensional mRNA
secondary structure of A) GST and B) GST-bR9AP fusion protein using the software RNAfold from the ViennaRNA suite ... 119 Figure 2.3. A) Amino acid and DNA sequences of the region bearing the
potential translation initiation site of wild type (WT) and mutated GST from the commercial vector pGEX-4T-1 (WT(pGEX-4T-1) and Mutated(pGEX-4T-1), respectively). ... 120
Chapitre 3
Figure 3.1. Secondary structure predictions of the human and bovine peptides .. 135 Figure 3.2. Circular dichroic spectra of the human and bovine peptides in HFIP
(solid line) and H-MeOH (dashed line). ... 136 Figure 3.3. Infrared spectra of the human (black) and bovine (pink) peptides. .... 138 Figure 3.4. Typical example of the determination of a MIP value using the
human peptide in the presence of a DSPC monolayer. ... 141 Figure 3.5. Histograms presenting the values of MIP (A-B-C-D) and synergy
(E-F-G-H) of the human and bovine peptides in the presence of different phospholipid monolayers. ... 143 Figure 3.6. Determination of MIP of the human (A) and bovine (B) peptides in
the presence a DSPC monolayer in different physical states. ... 146 Figure 3.7. PM-IRRAS spectra of the human and bovine peptides in the
absence or in the presence of a lipid monolayer. ... 148 Figure 3.8. Spectra (A-B) of the antisymmetric νas(CH2), and symmetric νs(CH2),
stretching modes of the methylene groups of DSPC phospholipids in the absence (black) and in the presence (red) of the human (A) and the bovine (B) peptide. ... 151
Figure 3.9. Infrared spectra of the bovine peptide in the presence of a monolayer (black) or of a bilayer (pink) of DOPC. ... 152 Figure 3.S1. Eisenberg plot of the human and bovine peptides obtained using
the Heliquest software. ... 166 Figure 3.S2. Infrared spectra of the bovine peptide in H-MeOH prior (black) and
after (pink) evaporation of the solvent. ... 167 Figure 3.S3. Infrared spectra of the bovine peptide in H-MeOH in the presence
and in the absence of 50 mM of β-mercaptoethanol. ... 168 Figure 3.S4. Spectral decomposition of the spectra, from Figures 3.3B and C,
of the human (A-B) and bovine (C-D) peptides solubilized in HFIP (A-C) or in H-MeOH (B-D). ... 169 Figure 3.S5. Circular dichroic spectra of the human and bovine peptides
measured at 21 °C (black) and at 58 °C (pink). ... 170 Figure 3.S6. Simulations of the PM-IRRAS spectra of the amide I and II bands
for pure α-helices at different tilt angles, θ, between the helical axis and the normal of the monolayer. ... 171 Figure 3.S7. Comparison between the PM-IRRAS spectra of the human (A) and
bovine (B) peptides in the absence and in the presence of a DSPC monolayer. ... 171 Figure 3.S8. 3D structural model of the human (A) and bovine (B) peptides. ... 172 Figure 3.S9. Histograms of the values of MIP (A) and synergy (B) of the human
and bovine peptides in the presence of different polyunsaturated phospholipid monolayers. ... 173 Figure 3.S10. Comparison between the PM-IRRAS spectra of the human (A)
and bovine (B) peptides in the absence of a phospholipid monolayer (black) and in H-MeOH (pink). ... 174 Figure 3.S11. Comparison between the PM-IRRAS spectra of the human
(black) and bovine (pink) peptides in the presence of a DSPC monolayer. .. 175
Chapitre 4
Figure 4.1. Secondary structure prediction of R9AP with different online tools .... 189 Figure 4.2. Three-Dimensional (3D) structural model of R9AP obtained using
I-TASSER ... 190 Figure 4.3. Circular dichroism (A) and infrared (B) spectra of R9AP∆TM. ... 191 Figure 4.4. Circular dichroism spectra of the wild-type (WT), W79- and
W173-R9AP∆TM proteins (black, blue and pink curves, respectively) ... 192 Figure 4.5. Emission spectra (A) and Stern-Volmer fluorescence quenching
curves (B) of the wild-type (WT), W79- and W173-R9AP∆TM proteins (black, blue and pink, respectively) ... 194
Figure 4.6. Emission spectra (A) and fluorescence quenching curves (B) of the C-terminal peptide of R9AP in the absence (solid line, black) or the presence of DOPC SUV (dashed line, black) as well as (C) in the presence of brominated DSPC (DSPC-Br) lipid vesicles (dashed line, orange) ... 196 Figure 4.7. Determination of the center of mass of R9AP∆TM upon binding to
POPC (black curve) or POPC:POPS (85:15, orange curve) large unilamellar vesicles (LUVs). ... 199 Figure 4.8. Histograms of the values of MIP (A-D) and synergy (E-H) of
R9AP∆TM in the presence of different phospholipid monolayers. ... 200 Figure 4.S1. Effect of the storage temperature and time on the stability of
R9AP∆TM ... 212 Figure 4.S2. Determination of the saturating surface concentration of R9AP∆TM
for the monolayer measurements. ... 213 Figure 4.S3. Circular dichroism spectra of the (A) wild-type, (B) W79- and (C)
W173-R9AP∆TM proteins at different temperatures from 20 to 100 °C. ... 213 Figure 4.S4. Determination of the center of mass of W79- (n) and
W173-R9AP∆TM (u) upon binding to POPC (black curve) and POPC:POPS (85:15, orange curve) LUVs. ... 214 Figure 4.S5. Three-Dimensional (3D) structural model of R9AP obtained using
I-TASSER. ... 215
Conclusions et perspectives
Figure C.1. Histogramme présentant les valeurs de PIM et de synergie de la R9AP∆TM (gris) et du segment C-terminal (vert) en présence de différents types de phospholipides représentatifs de la composition membranaire des disques des SEB ... 223 Figure C.2. Description de différents spectres pouvant être enregistrés par
RMN. ... 228 Figure C.3. Analyse par SDS-PAGE (A) et par immunobuvardage de type
Western à l’aide d’un anticorps anti-GST (B) de l’expression de la R9AP
humaine et bovine avec (R9AP) et sans (R9AP∆TM) leur segment transmembranaire en fusion avec la GST en N-terminal ... 231 Figure C.4. Comparaison entre les acides aminés exposés à plus de 35% à la
surface de la R9AP humaine et bovine en fonction de leurs caractéristiques chimiques ... 232 Figure C.5. Analyse par immunobuvardage de type Western de l’expression (A)
et de la solubilisation (B) de la protéine de fusion MBP-R9AP-His10 à l’aide d’anticorps anti-R9AP et anti-His ... 235
Annexe 1
Figure A1.1. Predicted secondary structure of the peptides obtained using the I-TASSER server. ... 273 Figure A1.2. Circular dichroism and infrared spectra of the RDH8-Cter (A-B),
RDH11-Nter (C-D), LRAT-Cter (E-F) and R9AP-Cter (G-H) peptides in different solvents. ... 275 Figure A1.3. Comparison between the difference in molar ellipticity of the
RDH8-Cter, RDH11-Nter, LRAT-Cter and R9AP-Cter peptides calculated from the circular dichroism spectra shown in Figure A1.2. ... 277 Figure A1.4. Instrument and procedure to prepare lipid monolayers and to study
peptide binding using the Langmuir model membrane system... 281 Figure A1.5. PM-IRRAS spectra of the R9AP-Cter peptide measured either
after spreading the peptide (or the DOPE-peptide mixture) at the air-water interface or after injecting the peptide into the subphase of the DOPE monolayer. ... 283 Figure A1.6. Determination of the saturating surface concentration of peptides
for the monolayer measurements. ... 284 Figure A1.7. Determination of the maximum insertion pressure and the synergy
of peptides in the presence of a DOPE monolayer. ... 286 Figure A1.8. Schematic representation of the instrument used to perform
polarization modulation infrared reflection absorption spectroscopy (PM-IRRAS) measurements... 288 Figure A1.9. Comparison between two different ways to treat PM-IRRAS
spectra of a peptide bound to a lipid monolayer. ... 290 Figure A1.10. PM-IRRAS spectra of the peptides in monolayers... 291 Figure A1.S1. Helical wheel representation of RDH8-Cter (residues 281-312),
RDH11-Nter (residues 2-35), LRAT-Cter (residues 201-230), R9AP-Cter (residues 207-235), Endophilin-Nter (residues 2-19) and GMAP-210-Nter (residues 2-37) peptides. ... 329 Figure A1.S2. Eisenberg plot of the peptides obtained by the Heliquest
generated data. ... 330 Figure A1.S3. A) Comparison between the infrared spectra of the RDH11-Nter
peptide solubilized in HFIP and of the R9AP-Cter peptide dissolved in methanol after the evaporation of HFIP (H-MeOH). Comparison between the infrared spectra of the RDH11-Nter, LRAT-Cter and R9AP-Cter peptides solubilized in HFIP (B) and in H-MeOH (C). All spectra have been normalized to facilitate their comparison. ... 331 Figure A1.S4. PM-IRRAS spectra simulation of the amide I and II region for
pure α-helices at different tilt angles, q, between the helical axis and the normal to the interface. ... 332 Figure A1.S5. A) Comparison between the PM-IRRAS spectra of the
a DOPE monolayer. B) Comparison between the PM-IRRAS spectra of the RDH11-Nter and R9AP-Cter in the absence of a phospholipid monolayer. . 333 Figure A1.S6. A) Comparison between the infrared spectra of the RDH8-Cter
peptide in solution (methanol, MeOH) and in the presence of a DOPE monolayer. B) Comparison between the infrared spectra of the RDH11-Nter peptide in solution (HFIP) and in the presence of a DOPE monolayer. C) Comparison between the infrared spectra of the LRAT-Cter peptide in solution (HFIP or H-MeOH) and in the presence of a DOPE monolayer. D) Comparison between the infrared spectra of the R9AP-Cter peptide in solution (H-MeOH) or in the presence of a DOPE monolayer. ... 334 Figure A1.S7. Comparison between the infrared spectra of peptides in solution
and in the absence of a phospholipid monolayer ... 335
Annexe 2
Figure A2.1. Typical example of the determination of a MIP value using Cter-R9AP in the presence of a DSPE monolayer. ... 343 Figure A2.2. (A) Commercially available apparatus (DeltaPi4, Kibron Inc) using
4 microtroughs of 500 µL each. The multiwell plate is either made of Teflon or of glass with the top covered with Teflon (shown here). (B) Closer view of some of the wells of the multiwell plate allows to more appropriately see the cylindrical surface pressure detector made of platinum ... 344 Figure A2.3. MIP and synergy of RP2 in the presence of different phospholipid
monolayers ... 345 Figure A2.4. Histograms of (A) MIP, (B) synergy and (C) ΔΠ0 of RP2 in the
presence of a monolayer of DPPC at 23 °C in the liquid-expanded (LE ; 0−8 mN/m) and liquid-condensed (LC ; 15−35 mN/m) states. ... 348 Figure A2.5. Determination of the binding parameters of RP2 upon binding to
monolayers of oxidized and intact DDPC at 23 °C ... 349 Figure A2.6. Histograms of the values of (A) MIP, (B) synergy and (C) ΔΠ0 of
tLRAT obtained in the presence of different phospholipid monolayers. ... 350 Figure A2.7. Histograms presenting the values of (A) MIP and (B) synergy of
the Cter-R9AP peptide in the presence of different phospholipid monolayers. ... 352
Liste des tableaux
Introduction
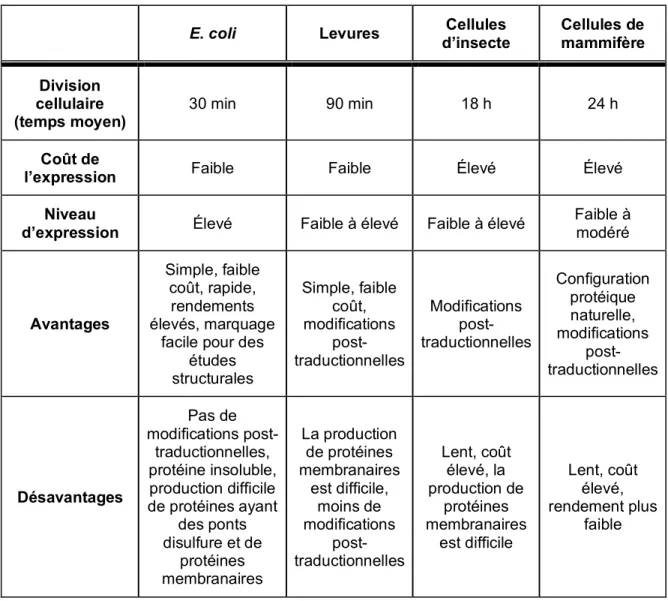
Tableau I.1. Composition lipidique des membranes des SEB (%). ... 11 Tableau I.2. Comparaison des systèmes d’expression pour la production de
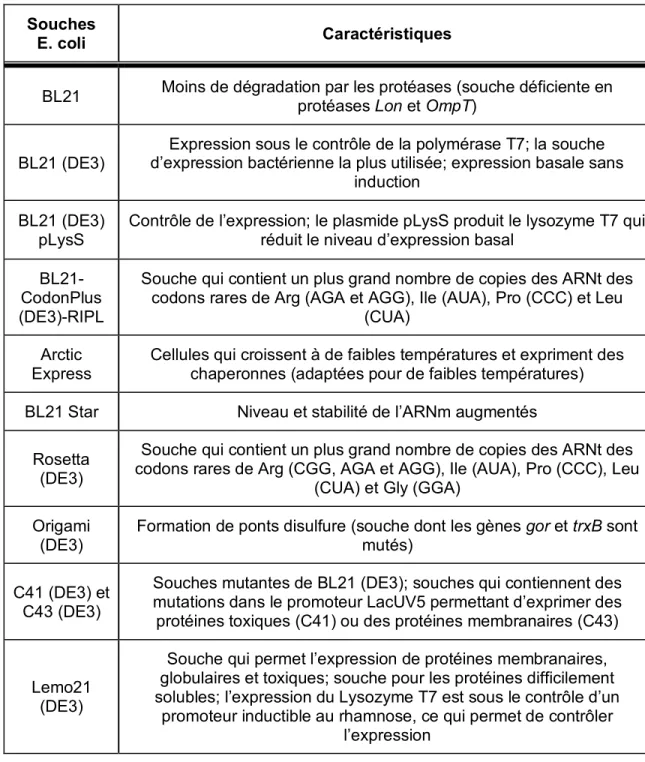
protéines recombinantes. ... 29 Tableau I.3. Caractéristiques des différentes souches de E. coli utilisées pour
l’expression de protéines recombinantes. ... 35 Tableau I.4. Étiquettes de solubilisation parmi les plus utilisées. ... 36 Tableau I.5. Étiquettes de purification parmi les plus utilisées. ... 38 Tableau I.6. Protéases les plus utilisées pour cliver les étiquettes en fusion avec
une protéine d’intérêt. ... 40 Tableau I.7. Association des fréquences d’absorption de la bande amide I
obtenues par spectroscopie IR avec différents types de structure secondaire. ... 53 Tableau I.8. Paramètres de fluorescence des acides aminés aromatiques dans
l’eau à pH neutre. ... 57
Chapitre 1
Table 1.1. Summary of the cloning of the recombinant plasmids used for the expression of the different fusion proteins. ... 74 Table 1.2. Relative expression and solubility of the fusion proteins as well as
their purity after purification. ... 85 Table 1.S1. Summary of the cloning of all the intermediates used for the cloning
procedure. ... 111
Chapitre 3
Table 3.1. Analysis of the amino acid content of the human and bovine peptides ... 134 Table 3.2. Comparison of the secondary structure content of the human and
bovine peptides in HFIP and H-MeOH obtained by CDSSTR analysis of the circular dichroic spectra (CD) and by the decomposition of the spectra measured by infrared spectroscopy (IR)... 137
Annexe 1
Table A1.1. Analysis of the amino acid content of the selected peptides ... 271 Table A1.S1. Comparison between the secondary structure content of the
different peptides using CDSSTR analysis of the circular dichroism spectra... 327 Table A1.S2A. Analysis of the amino acid content of rhodopsin transmembrane
helices. ... 328 Table A1.S2B. Comparison between the analysis of the amino acid content of
the transmembrane a-helical segments of rhodopsin and of the hydrophobic peptides selected in this review (see Table A1.1). ... 328
Liste des abréviations
5’-GMP 5’-guanosine monophosphate DP Variation de la pression de surface
lEm Longueur d’onde d’émission
lEx Longueur d’onde d’excitation
Pe Pression d’équilibre
Pi Pression de surface initiale
AL Lumière polarisée circulaire gauche AR Lumière polarisée circulaire droite
Arr Arrestine
ATR Spectroscopie infrarouge par réflexion totale atténuée bR9AP∆TM R9AP bovine sans segment transmembranaire
CBP Calmodulin-binding peptide
CD Dichroïsme circulaire
CRBP-1 Cellular retinol binding protein-1 DEP Disheveled, Egl-10, Pleckstrin
DRM Detergent-resistant membranes E. coli Escherichia coli
EntK Entérokinase
EPR Épithélium pigmentaire rétinien FXa Factor Xa
Gat Sous-unité a de la transducine
Gat-GTP Sous-unité a de la transducine liée au GTP Gat-GDP Sous-unité a de la transducine liée au GDP
Gabgt Transducine
Gb5 Type 5 G protein b subunit
Gb5L Long splice isoform of the type 5 G protein b subunit
GAP GTPase-Activating Protein
GC Guanylate cyclase
GCAP 1 et 2 Protéines activatrices de la guanylate cyclase 1 et 2
GDP Guanosine diphosphate
GEF Guanine nucleotide Exchange Factors
GGL domain G protein gamma-like domain
GMPc Guanosine monophosphate cyclique GPCR Récepteurs couplés aux protéines G
GST Glutatione S-transferase
GST-bR9AP∆TM R9AP bovine sans segment transmembranaire en fusion avec une étiquette GST
GTP Guanosine triphosphate
GUV Giant unilamellar vesicle
Hisn Étiquette de purification de plusieurs acides aminés histidines
IMAC immobilized metal affinity chromatography
IR infrarouge
IRBP Interphotoreceptor retinol binding protein LRAT Lécithine rétinol acyltransférase
LUV Large unilamellar vesicle
MBP Maltose binding protein
NusA N-utilization substrance
p Faisceau IR polarisé parallèlement
PC Phosphocholine
PDB Protein Data Bank
PDE Phosphodiestérase
PE Phosphoéthanolamine
PIM Pression d’insertion maximale
PM-IRRAS Spectroscopie infrarouge de réflexion-absorption par modulation de polarisation
polyHis Étiquette de purification de plusieurs histidines
PS Phosphosérine
R Rhodopsine
R* Rhodopsine activée
R9AP RGS9-1 Anchor Protein
R9AP∆TM R9AP sans segment transmembranaire
RDH5 Rétinol déshydrogénase 5
RDH8 Rétinol déshydrogénase 8
Rec Recoverine
RGS9 Regulator of G protein signaling
RK Rhodopsine kinase
Rp Réflectivité de la polarisation parallèle
RPE65 Retinal pigment epithelium-specific 65 kDa protein
Rs Réflectivité de la polarisation perpendiculaire s Faisceau IR polarisé perpendiculairement
S/N Ratio signal/bruit
SD Séquence de Shine-Dalgarno
SDS Docédylsulfate de sodium
SEB Segment externe des bâtonnets
Strep-II Streptavidin-binding peptide
SUMO Small ubiquitin-modifier
SUV Small unilamellar vesicle TA proteins Tail-anchored proteins
TEV Tobacco etch virus protease
Thr Thrombine
Tm Température de transition de phase Trx Thioredoxin
« The will to win, the desire to succeed, the urge to reach your full potential... these are the keys that will unlock the door to personal excellence »
Remerciements
Cette thèse est l’achèvement de six années de dur labeur. Je n’apprendrai rien à personne en disant que le succès d’un doctorat passe inévitablement par de nombreux échecs. J’ajouterais également que, pour reprendre les paroles d’un certain Abraham Lincoln, « le succès c’est d’aller d’échec en échec sans jamais perdre son enthousiasme ». Ainsi, il va de soi de remercier chacune des personnes qui ont contribué au succès de cette thèse en me permettant de conserver mon enthousiasme durant ces années d’études.
Mes premiers remerciements vont à mon directeur de thèse, Christian. Merci d’avoir cru en la jeune étudiante ambitieuse que j’étais en 2010. Merci pour votre travail acharné, votre disponibilité, votre grande écoute et votre compréhension. Vous avez grandement contribué à mon évolution scientifique et personnelle en m’encourageant entre autres à exploiter autant mes forces que mes faiblesses. J’aspire sincèrement à devenir un aussi bon mentor que vous l’avez été pour moi. Un merci tout spécial à notre merveilleuse professionnelle de recherche sans qui notre quotidien ne serait pas pareil, non seulement parce qu’elle est une ressource de connaissances sans fin en plus d’être constamment à notre disponibilité, mais également parce qu’elle est un rayon de soleil contagieux. Line, merci de nous transmettre ton amour pour la recherche, tu es véritablement une personne inspirante. J’aspire à être une collègue de travail aussi agréable, patiente (!!) et dévouée que toi. Ta personnalité ensoleillée et ton énergie me manqueront. Merci d’avoir été présente toutes ces années pour partager mes émotions (!!), aussi joyeuses que malheureuses et autant personnelles que professionnelles.
Je tiens sincèrement à remercier ceux qui ont accepté de prendre le temps de réviser ma thèse : Dr Nicolas Doucet, Dr Michel Guertin et Dr Sylvain Guérin. C’est véritablement un honneur pour moi de vous compter parmi mon jury de thèse et de pouvoir ainsi bonifier mon écrit grâce à vos commentaires.
Je tiens également à remercier ma formidable équipe de recherche. Merci à chacun des membres de cette équipe : à mes anciens collègues (Habib, Élodie, Mustapha, Audrey-Anne, Kim et André), aux étudiants de premier cycle que j’ai supervisés (Alexandre B., Sarah, Charlotte et MAM) et ceux que j’ai côtoyés (Samuel, Véronique et Marie-Ève), ainsi qu’à mes collègues actuels (Marie-Ève, Charlotte, Sarah, Marc-Antoine et Alexandre V.). Vous avez tous contribué, chacun à votre façon, à agrémenter mon quotidien tout en me faisant grandir en tant que personne et scientifique. Un merci tout particulier à Élodie et Habib qui ont grandement contribué aux balbutiements de ma jeune carrière scientifique. Vous m’avez inspiré de belles valeurs personnelles et professionnelles, merci. Finalement, j’offre une mention spéciale à MEG et sa franchise parfois déconcertante, à Sarah et son rire contagieux, ainsi qu’à Charlotte et ses propos colorés remplis de sous-entendus. Je vous souhaite beaucoup de succès J. Vous allez me manquer les filles!
Merci à tous les membres du CUO-recherche et plus particulièrement aux personnes avec qui j’ai passé plus de temps (Alicia, Marie-Christine, Anne-Sophie et Léo). J’ai également une pensée spéciale pour ceux dont le départ a laissé un grand vide : Olivier Rochette-Drouin et son humour ainsi que Laurence Trudel-Vandal et son inébranlable bonne humeur. J’espère que nos chemins se croiseront de nouveau un de ces jours!
Merci également à Marie, ma complice de thèse qui est une collègue devenue une amie. Marie, merci pour ton soutien quotidien et d’avoir partagé beaucoup plus que de la science avec moi. Grâce à toi je connais maintenant quelques expressions à éviter lorsque j’irai vous visiter en France, Thierry et toi (tu vois, finalement je n’ai rien compris du tout!!). Ta présence et tes bricolages maison vont me manquer. Je te souhaite une carrière à la hauteur de ton potentiel. -xxx-
Je tiens également à remercier mes amies et ma famille. Maman, papa, Lisa et Joe, vous avez toujours cru en moi et vous m’avez toujours encouragé à me surpasser.
Vous contribuez directement à mes succès. Merci pour votre amour inconditionnel. Je vous aime ©
À mes amies les plus proches, Marie, Claudie, Lorenzo et Émi, merci pour vos encouragements constants et votre émerveillement à propos de mon parcours académique. Merci d’être autant à l’écoute et de me changer les idées quand j’en ai besoin. Je vous aime ©
Finalement, les derniers et non les moindres, mes âmes sœurs : ma meilleure amie Mel et mon copain Jules. Merci à vous deux d’avoir partagé mes joies et mes chagrins. Votre présence apaise tous mes maux. Merci de votre amour et de votre écoute. Mel, ta présence quotidienne depuis maintenant plus de dix ans m’est très précieuse. Merci d’être ma plus grande admiratrice, de m’encourager et de me faire relativiser. Tu me permets d’évoluer. Je t’aime mon amie ©.
Jules, mon chum, mon hockeyeur préféré, j’ai du mal à trouver les mots pour exprimer tout ce que tu m’apportes…merci pour ton soutien, ton écoute, ta compréhension et ton amour. Merci de m’encourager autant dans les moments faciles que dans les moments difficiles. Ta présence à mes côtés est réconfortante et énergisante. Tu contribues chaque jour à ce que je sois une meilleure personne. Merci d’avoir accepté les sacrifices qu’un doctorat implique, d’avoir mis des projets sur la glace en attendant la fin de mes études (hockey, glace… la pognes-tu?! Il fallait au moins que je fasse un jeu de mots poche! ahah). J’ai hâte qu’on réalise tous nos projets. Merci de faire partie de ma vie, mon amour, ensemble on forme une merveilleuse équipe. Je t’aime ©©.
Avant-propos
Cette thèse de doctorat est rédigée sous forme d’insertion d’articles. Par conséquent, les chapitres 1 à 4 sont des articles rédigés en anglais qui seront soumis ou qui ont été publiés dans des journaux scientifiques avec comité de lecture. Les articles dans les annexes 1 et 2 ont également été publiés en anglais sous forme de revue avec résultats expérimentaux et de chapitre de livre, respectivement. Les chapitres 1 à 4 sont précédés d’une introduction et suivis des conclusions de la thèse. La bibliographie de l’introduction et de la conclusion est regroupée à la fin de la thèse, avant les annexes. Les travaux présentés dans cette thèse n’auraient été possibles sans la contribution importante de mes collègues. Cette section détaillera donc l’apport de chacun de mes collèges ainsi que ma contribution pour chacun des articles présentés dans cette thèse.
Chapitre 1
L’article présenté dans le chapitre 1 ayant pour titre « Systematic analysis of the
expression, solubility and purification of a passenger protein in fusion with different tags » a été publié dans le journal Protein Expression and Purification en juillet 2018.
En tant que première auteure de cet article, j’ai effectué la totalité des manipulations, j’ai préparé toutes les figures et j’ai rédigé une première version complète du manuscrit. J’ai aussi élaboré la majorité des protocoles avec l’aide de ma collègue Line Cantin et j’ai effectué l’analyse des résultats. J’ai également contribué à la rédaction de la version finale, à la réponse aux examinateurs et aux révisions du manuscrit. Dans l’ensemble, ma contribution à cet article s’élève à environ 85%.
Chapitre 2
L’article présenté dans le chapitre 2 intitulé « Identification of an alternative
translation initiation site in the sequence of the commonly used Glutathione S-Transferase tag » a été publié sous forme de short communication dans le Journal of Biotechnology en septembre 2018. En tant que première auteure de cet article,
bio-informatiques présentées dans la figure 2 qui ont été effectuées par Louis-Philippe Morency. J’ai aussi préparé les figures, analysé les résultats et rédigé une première version complète du manuscrit dont je détiens l’idée originale. J’ai également contribué à la rédaction de la version finale et à la réponse aux examinateurs. Tous les auteurs ont contribué aux relectures avant que je produise la version finale conjointement avec mon superviseur Christian Salesse. Dans l’ensemble, ma contribution à cet article s’élève à environ 80%.
Chapitre 3
L’article présenté dans le chapitre 3 s’intitulant « Structure and binding of the
C-terminal segment of R9AP to lipid monolayers » a été publié dans le journal Langmuir en janvier 2015. En tant que première auteure de cet article, j’ai effectué
la totalité des manipulations, j’ai préparé toutes les figures et j’ai rédigé une première version complète du manuscrit. J’ai également significativement contribué à l’élaboration des protocoles et à l’analyse des résultats avec l’aide de mon collègue Habib Horchani et de mon superviseur Christian Salesse. J’ai aussi contribué à la rédaction de la version finale, à la réponse aux examinateurs et aux révisions de l’article. Dans l’ensemble, ma contribution à cet article s’élève à environ 85%.
Chapitre 4
Le manuscrit présenté dans le chapitre 4 ayant pour titre « Structure and membrane
binding of truncated RGS9-1 Anchor Protein » sera soumis très prochainement dans
le journal Biochemistry. En tant que première auteure, j’ai préparé les figures et rédigé la première version complète du manuscrit. J’ai effectué la majorité des manipulations (figures 1-5 et 7), dont l’étape critique d’élaboration du protocole de purification de la protéine. Les autres manipulations ont été effectuées par Sarah Roy (figure 6), une étudiante de premier cycle que j’ai supervisée pendant l’été 2015, et Marc-Antoine Millette (figure 8), un étudiant de premier cycle sous ma supervision lors de son stage à l’été 2017. J’ai également élaboré la majorité des protocoles et analysé les résultats avec l’aide de ma collègue Line Cantin ainsi qu’une de nos collaboratrices du Portugal, Ana Coutinho, et de mon superviseur Christian Salesse.
Tous les auteurs contribueront aux relectures avant que je produise, conjointement avec Christian Salesse, la version finale qui sera soumise. Dans l’ensemble, ma contribution à ce manuscrit s’élève à environ 70%.
Annexe 1
L’article de revue présenté dans l’annexe 1 contient une quantité importante de résultats expérimentaux. Il a pour titre « Comparison between the behavior of
different hydrophobic peptides allowing membrane anchoring of proteins » et il a été
publié dans le journal Advances in Colloid and Interface Science en janvier 2014. Ma contribution en tant que co-première auteure a été de produire tous les résultats avec le peptide de la R9AP (figures 2G et H, 3, 5, 6, 7 et 10D), ce qui correspond à environ 25% de l’ensemble des données. J’ai également effectué les analyses pour produire le tableau 1 et les prédictions de la structure secondaire des peptides présentées dans les figures 2B, D, F et H. J’ai aussi activement participé à la production des autres tableaux et figures. Les résultats obtenus avec le peptide de la RDH8 (figures 2A et B, 3, 7 et 10A) ainsi que la majorité des résultats avec le peptide de la RDH 11 (figures 2C et D, 3, 7 et 10B) ont été obtenus par Mustapha Lhor. Les résultats avec le peptide de la LRAT (figures 2E et F, 3 et 10C) ont été obtenus par Sylvain Bussières à l’exception des mesures de liaison membranaire (figure 7) qui ont été effectuées par Mustapha Lhor. Habib Horchani a quant à lui produit toutes les figures finales. La participation de tous les auteurs a été nécessaire pour l’élaboration des protocoles et l’analyse des résultats. L’article a été rédigé en totalité par Christian Salesse et tous les auteurs ont participé aux relectures et à la révision de la revue. Dans l’ensemble, ma contribution à cet article s’élève à environ 30%.
Annexe 2
Le chapitre de livre présenté dans l’annexe 2 intitulé « How to decipher protein and
peptide selectivity for lipids in monolayers » a été publié en tant que « Chapter 6 »
dans le livre Recent Progress in Colloid and Surface Chemistry with Biological
de produire toutes les figures finales en plus des résultats présentés dans les figures 1 et 7. Les résultats de la RP2 (figures 3, 4 et 5) ont été obtenus par Éric Demers à l’exception de ceux présentés dans la figure 6 qui ont été obtenus par Sylvain Bussières. Line Cantin a contribué à l’élaboration des protocoles conjointement avec chacun des auteurs ayant effectué les manipulations. Le chapitre de livre a été écrit en totalité par Christian Salesse et tous les auteurs ont participé aux relectures du manuscrit. J’ai contribué aux révisions conjointement avec Christian Salesse. Dans l’ensemble, ma contribution s’élève à environ 40%
Introduction
La vision est un sens essentiel qui implique des processus biologiques complexes. Plus spécifiquement, l’œil contient des cellules spécialisées, les photorécepteurs, qui nous permettent de distinguer les couleurs et les contrastes des objets qui nous entourent. Un disfonctionnement au niveau de ces cellules peut engendrer des maladies plus ou moins sévères de la vision, allant du daltonisme à la cécité. L’introduction présente un survol de la vision d’un point de vue anatomique et moléculaire, une brève description des procédures expérimentales ainsi que le contexte et les objectifs de mes travaux de recherche.
L’œil
L’œil est l’organe essentiel à la vision. À l’âge adulte, l’œil possède un diamètre d’environ 2,5 cm [1] et un poids moyen de 7 grammes [2]. L’œil est formé de 3 tuniques [1] : 1) la tunique fibreuse composée de la cornée et de la sclère, 2) la tunique vasculaire (uvée) qui comprend la choroïde, le corps ciliaire ainsi que l’iris, et, finalement, 3) la tunique interne (rétine) qui est composée d’une partie nerveuse (dont font partie les photorécepteurs) et d’une partie pigmentaire (Figure I.1). Le cristallin est la lentille qui divise l’œil en un segment antérieur et un segment postérieur. Le segment antérieur comprend la zone de l’œil entre la cornée et le cristallin où on retrouve aussi l’humeur aqueuse, alors que le segment postérieur inclut la région entre le cristallin et la chroroïde ainsi que l’humeur vitrée. Ainsi, lorsque la lumière est focalisée sur la rétine, celle-ci traverse, dans l’ordre, la cornée, l’humeur aqueuse, le cristallin puis l’humeur vitrée avant d’atteindre les photorécepteurs qui sont situés dans la partie nerveuse de la rétine [1]. C’est cependant le cristallin qui, grâce à sa très grande élasticité, permet de focaliser précisément la lumière sur la rétine au niveau de la macula, une zone qui est très concentrée en photorécepteurs, en particulier les cônes (voir section « La rétine », p.2).
L’œil humain réagit à la lumière visible qui correspond à la portion du spectre électromagnétique entre 400 et 700 nm [3]. Lors de son parcours dans l’œil, la lumière est filtrée par les différentes structures avant d’atteindre la rétine. Par exemple, les pigments du cristallin éliminent les photons de plus haute fréquence, ce qui permet d’obtenir une image plus nette en plus de protéger la rétine des dommages causés par des photons de trop grande énergie [3].
Figure I.1. Représentation schématique de l’œil humain et de ses différentes structures. Figure adaptée de celle qui a été fournie par la compagnie Merck.
La rétine
La rétine est la structure de l’œil impliquée dans la conversion du signal lumineux en influx nerveux. Tel que mentionné dans la section « L’œil », la rétine est composée de deux parties : la partie nerveuse, aussi appelée rétine neurale, et la partie pigmentaire ou épithélium pigmentaire rétinien (EPR). Seulement la partie nerveuse de la rétine joue un rôle direct dans la vision. En effet, la rétine neurale qui constitue la couche interne de la rétine comprend un empilement de cellules
nerveuses et gliales (Figure I.2) [1, 4]. Lorsque la lumière est focalisée sur la rétine, celle-ci traverse successivement les cellules ganglionnaires, amacrines, bipolaires, horizontales et de Müller avant d’atteindre les photorécepteurs de type cône ou bâtonnet. Les photorécepteurs captent cette lumière puis la convertissent en signaux qui sont transmis aux cellules ganglionnaires par l’intermédiaire des cellules bipolaires [1]. Le potentiel d’action généré suite à l’absorption de la lumière est transmis au cortex visuel du cerveau via les axones des cellules ganglionnaires qui forment le nerf optique.
Figure I.2. Schéma de la rétine humaine. Figure adaptée de [4].
Bien que l’EPR n’ait pas de rôle direct dans la vision, cette couche unicellulaire qui constitue la couche externe de la rétine joue un important rôle de soutien aux photorécepteurs [1, 4]. En effet, les cellules de l’EPR participent entre autres à la régénération du pigment visuel des photorécepteurs et au renouvellement des
photorécepteurs en phagocytant la portion apicale de leur segment externe, en plus d’échanger des nutriments et des métabolites avec les photorécepteurs [5]. L’EPR contient également de la mélanine qui est importante pour capter la lumière non absorbée par les photorécepteurs, ce qui permet d’éviter la réflexion de la lumière, ce qui résulte ainsi en une vision plus nette des détails.
Les photorécepteurs
Il existe 2 types de photorécepteurs : les cônes et les bâtonnets [3]. Les cônes sont responsables de la vision photopique ou diurne, alors que les bâtonnets sont à l’origine de la vision scotopique ou vision de nuit. En effet, les cônes nous permettent de voir en couleur avec une grande précision, tandis que les bâtonnets nous permettent de voir les contrastes avec un éclairage plus faible. Les bâtonnets sont pour leur part très sensibles, mais possèdent une faible résolution (image floues), alors que les cônes sont environ 100 à 1000 fois moins sensibles que les bâtonnets, mais nous confèrent une acuité visuelle élevée [6].
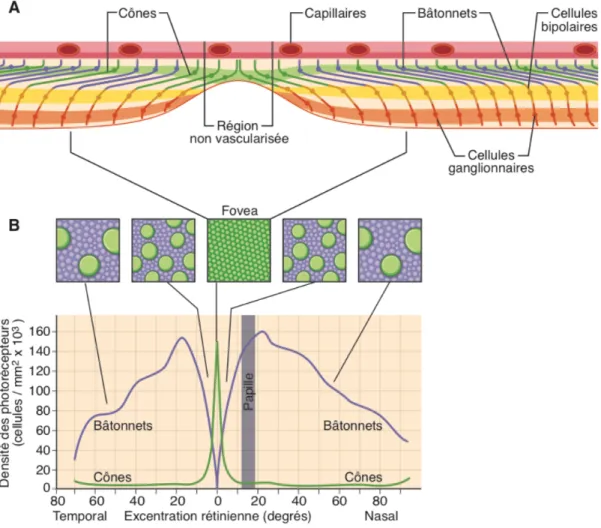
Ces différences sont entre autres dues à la distribution particulière des photorécepteurs dans la rétine (Figure I.3). En effet, les cônes sont concentrés dans la macula, mais plus particulièrement au centre de celle-ci, une zone appelée la fovea. La fovea est une région où on observe une petite dépression (environ 1,2 mm de diamètre), ce qui cause le déplacement des cellules ganglionnaires et bipolaires en périphérie de la fovea (Figure I.3A). Ainsi, cette zone exclusivement constituée de cônes reçoit la lumière directement, sans que celle-ci ne traverse les autres couches de la rétine, ce qui améliore considérablement l’acuité visuelle étant donné qu’il n’y a pas de diffusion de la lumière. Les bâtonnets sont quant à eux répartis dans toute la rétine, mais leur densité diminue à mesure qu’on se rapproche de la macula. Ainsi, les bâtonnets sont très peu présents dans la macula et absents de la fovea (Figure I.3B). Par ailleurs, les bâtonnets sont environ 20 fois plus nombreux que les cônes dans la rétine. En effet, on dénombre environ 110 millions de bâtonnets et 6,5 millions de cônes dans chaque œil [7].
Figure I.3. Distribution des cônes et des bâtonnets dans la rétine neurale. Les
cônes sont concentrés dans la fovea (A), une région de la rétine qui ne contient pas de bâtonnets (B). Figure adaptée de [4].
En plus de la distribution particulière des cônes et des bâtonnets dans la rétine, les circuits neuronaux des cônes et des bâtonnets sont différents, ce qui contribue également à leur différence de sensibilité et d’acuité visuelle [3]. En effet, les cônes de la fovea font synapse avec une seule cellule ganglionnaire par l'intermédiaire d’une seule cellule bipolaire, ce qui permet une acuité visuelle maximale [8]. Cependant, dans le reste de la rétine, les cônes et les bâtonnets font synapse avec leurs cellules bipolaires spécifiques, mais convergent vers les mêmes cellules ganglionnaires. De plus, les cellules bipolaires des bâtonnets reçoivent les signaux
de plusieurs bâtonnets à la fois. Ainsi, les bâtonnets sont des photorécepteurs extrêmement sensibles, mais ce système est cependant de très faible résolution en raison de la convergence des signaux.
Structure des cônes et des bâtonnets
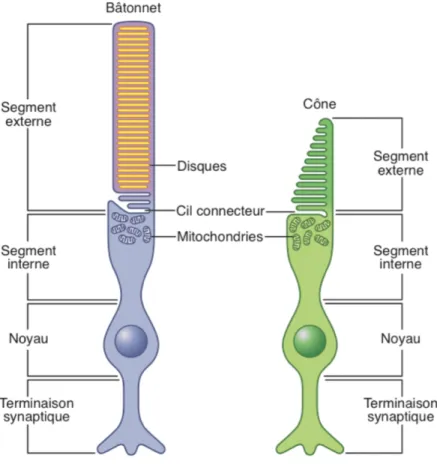
Malgré les importantes différences dans leur sensibilité et leur acuité visuelle, les cônes et les bâtonnets possèdent une morphologie très semblable. En effet, ces deux types de photorécepteurs sont constitués d’un segment externe, d’un segment interne, d’un noyau et d’une terminaison synaptique (Figure I.4).
Figure I.4. Représentation schématique des photorécepteurs de type cône et bâtonnet de la rétine. Figure adaptée de [4].
Le noyau contient l’information génétique de la cellule et la terminaison synaptique permet la transmission du signal entre les photorécepteurs et les cellules bipolaires.
Le segment interne contient l’appareil de Golgi, les mitochondries et le réticulum endoplasmique et est donc responsable de l’activité métabolique des photorécepteurs [9]. Le segment interne et le segment externe sont connectés par un petit segment étroit appelé cil connecteur. Ce sont les segments externes des photorécepteurs qui sont responsables de la conversion du signal lumineux en signal électrique. À noter que les photorécepteurs sont orientés de sorte que la portion apicale de leurs segments externes est en contact avec les cellules de l’EPR. Ainsi, lorsque la lumière est focalisée sur la rétine, le signal lumineux traverse toutes les composantes des photorécepteurs avant d’atteindre le segment externe. Suite à l’absorption du photon, un signal électrique est généré par l’entremise des protéines du segment externe, ce qui entraîne l’arrêt de la sécrétion du neurotransmetteur glutamate au niveau de la terminaison synaptique (voir section « La phototransduction visuelle », p.12 pour les détails de la phototransduction visuelle). Les segments externes des cônes et des bâtonnets possèdent cependant une morphologie différente, comme on peut le constater dans la Figure I.4. En premier lieu, la forme de leur segment externe est différente. En effet, plus on se dirige du côté apical du segment externe des cônes, plus la largeur diminue, alors que le segment externe des bâtonnets (SEB) possède la même largeur sur toute sa longueur. Ces différences sont à l’origine du nom de ces photorécepteurs. Également, le segment externe des cônes est formé par le repliement de sa membrane plasmique [10], alors que celui des bâtonnets est composé d’un empilement d’environ 2 000 disques membranaires indépendants de la membrane plasmique [4].
Fonction des photorécepteurs
Le mécanisme de transduction du signal a été largement étudié chez les bâtonnets puisque, à l’inverse des cônes, il est possible d’obtenir une quantité importante de SEB à partir de rétines d’animaux comme le bovin [11-13]. Par ailleurs, il n’y a qu’un seul type de bâtonnet alors qu’il y a trois types de cônes (bleu, vert et rouge). Même
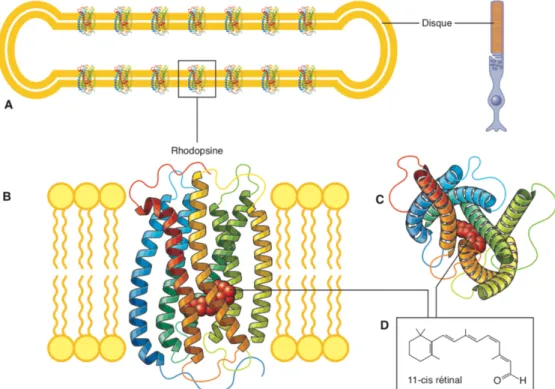
si la fonction des bâtonnets a été davantage étudiée que celle des cônes, on sait toutefois que ces deux types de photorécepteurs contiennent une quantité importante de pigments photosensibles dans la membrane de leur segment externe. En effet, il existe quatre types de pigments visuels différents dans la rétine. La rhodopsine est le seul pigment visuel des bâtonnets (Figure I.5A-C), alors que les cônes bleus, rouges et verts possèdent chacun un pigment visuel différent. Même si les pigments visuels sont différents chez les cônes et les bâtonnets, leur structure générale reste la même [14]. En effet, ils sont constitués d’une portion protéique nommée opsine et d’un chromophore photosensible, le 11-cis rétinal (Figure I.5D). Les opsines possèdent sept segments transmembranaires et font partie de la famille des récepteurs couplés aux protéines G (GPCR) [15].
Figure I.5. Structure de la rhodopsine et son organisation dans la membrane des disques des bâtonnets. A) Représentation schématique d’un disque des
bâtonnets avec le pigment visuel rhodopsine incorporé dans la membrane. B) Structure de la rhodopsine (pdb : 1F88; [16]) liée à la bicouche lipidique des disques. On distingue les 7 hélices a transmembranaires de la rhodopsine et son chromophore, le 11-cis rétinal. C) Structure de la rhodopsine, vue du dessus. D) Structure du 11 cis-rétinal. Figure adaptée de [4].
Les trois types de cônes sont présents dans des proportions bien spécifiques. En effet, on dénombre la moitié moins de cônes verts que de cônes rouges et 32 fois moins de cônes bleus que de cônes rouges (proportion bleu/vert/rouge : 1/16/32) [4]. Les pigments visuels des cônes bleus, verts et rouges possèdent respectivement un maximum d’absorption à 445, 535 et 575 nm [4] (Figure I.6). En comparaison, le maximum d’absorption de la rhodopsine est situé à 500 nm. Aussi, on peut constater dans la Figure I.6 que le spectre d’absorption des pigments visuels des cônes couvre l’ensemble des longueurs d’ondes de la lumière visible. L’Homme peut ainsi distinguer plus de 300 couleurs avec une résolution allant jusqu’à 2 nm [4].
Figure I.6. Spectre d’absorption des pigments visuels des cônes (bleu, vert et rouge) et du pigment visuel des bâtonnets (rhodopsine). Figure adaptée de [4].
La vision dépend donc de l’absorption de la lumière par les pigments visuels. En considérant que les pigments visuels sont des protéines transmembranaires, la morphologie particulière des segments externes des photorécepteurs, qui inclut un empilement de ~2 000 disques, permet de rassembler un important contenu lipidique et, de surcroît, une quantité impressionnante de pigments visuels dans un petit volume. En effet, on retrouve environ 52 000 rhodopsines dans la membrane d’un seul disque, ce qui correspond à 10.4 x 107 rhodopsines pour un seul bâtonnet [17], ce qui en fait donc la protéine la plus abondante dans le segment externe. D’ailleurs, la rhodopsine occupe environ 50% du volume membranaire des disques
[18]. Cette densité très élevée du pigment visuel dans les bâtonnets permet donc l’absorption d’une quantité importante de lumière.
Environnement membranaire du segment externe des bâtonnets (SEB)
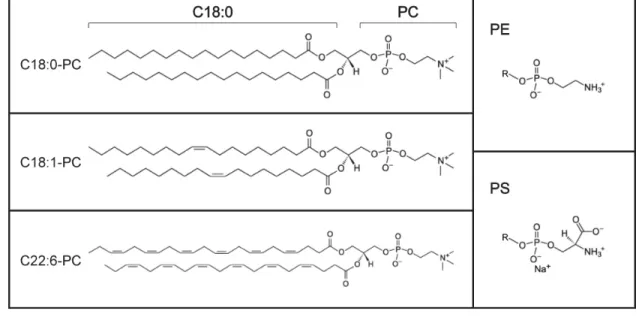
Les membranes des disques des bâtonnets sont constituées de 50% de protéines et 50% de lipides (%m/m) [19]. Plus spécifiquement, les phospholipides comptent pour 80% des lipides alors que le cholestérol correspond à environ 5%. Les phospholipides, aussi appelés phosphoglycérolipides, sont des molécules amphiphiles composés d’une tête polaire et de deux chaînes acyles hydrophobes. Les chaînes acyles des phospholipides peuvent être saturées, insaturées ou polyinsaturées. Les principaux phospholipides qui composent les membranes des SEB sont de type phosphoéthanolamine (PE), phosphocholine (PC) et phosphosérine (PS) (45, 38 et 14% respectivement [20]) (Figure I.7).
Figure I.7. Schéma et nomenclature des phospholipides. Les chaînes acyles
sont nommées par le nombre de carbones qu’elles contiennent suivi du nombre d’insaturations. Par exemple, des chaînes acyles de 22 carbones et 6 insaturations sont nommées C22:6. Les phospholipides de type phosphocholine (PC) et phosphoéthanolamine (PE) ont une charge nette neutre, alors que la phosphosérine (PS) est chargée négativement.
De plus, les membranes des SEB sont de loin celles qui sont les plus insaturées à être connues jusqu’à maintenant [19, 21]. En effet, plus de 60 mol% des chaînes acyles des phospholipides des SEB sont polyinsaturées (Tableau I.1). Ainsi, la majorité de ces phospholipides possèdent soit deux chaînes acyles insaturées (43 % des PS et 24% des PE et des PC) ou une chaîne saturée et une autre insaturée (75, 58 et 57% des PE, PC et PS, respectivement) [20]. Cependant, un nombre significatif de phospholipides PC possède deux chaînes acyles saturées (18%).
Chaîne acyle (45%) PE (38%) PC (14%) PS C16:0 19,6 10,7 1,2 C16:1 1,2 1,1 0,6 C18:0 17,3 22,3 18,2 C18:1 3,7 3,9 1,6 C18:2 1,5 1,3 0,4 C20:3 0,7 0,8 0,6 C20:4 3,4 2,4 1,0 C22:4 1,1 0,9 4,0 C22:5 2,5 2,7 5,2 C22:6 41,9 50,6 49,2 C24:4 0,3 0,1 6,1 C24:5 1,0 0,9 9,4
Tableau I.1. Composition lipidique des membranes des SEB (%). Adapté de
[22].
Aussi, il est important de noter que les membranes sont des bicouches lipidiques et que la composition lipidique de chacun des feuillets interne et externe qui forment la bicouche peut être différente [23]. En effet, on retrouve davantage de phospholipides PE et PS sur le feuillet externe des disques (en contact avec le cytosol des SEB), alors que les phospholipides PC sont situés préférentiellement
sur le feuillet interne [24-27]. Également, il a été rapporté que les membranes des SEB contiennent des detergent-resistant membranes (DRM) [28, 29]. Il s’agit de microdomaines qui sont résistants à l’action d’un détergent non-ionique spécifique et ayant une composition lipidique enrichie en phospholipides saturés et en cholestérol, mais réduite en phospholipides à chaînes acyles insaturées. Les DRM sont donc moins fluides que le reste de la membrane. Il a également été observé que les phospholipides de type PE et PS sont moins abondants dans les DRM [28]. Les DRM sont considérés comme des plateformes de signalisation très importantes [30-32]. Par exemple, dans les disques des bâtonnets, le ratio lipide/protéine est 3 fois plus élevé dans l’environnement lipidique spécifique des DRM comparativement au reste des membranes des SEB [28]. Il a été estimé que 8% des lipides des segments externes des photorécepteurs se retrouvent dans des DRM [29]. D’ailleurs, la majorité des protéines des SEB sont soit transmembranaires ou associées aux membranes [33]. Ainsi, bien que ce ne soit pas encore étudié en détail, il semble que les DRM pourraient jouer un rôle important dans la phototransduction visuelle. Pour l’instant, il semble cependant que la présence des DRM ne favorise pas l’activation de la phototransduction, même si quelques-unes des protéines de la phototransduction visuelle ont été retrouvées associées aux DRM [28, 29, 34-36].
La phototransduction visuelle
Activation de la phototransduction visuelle
Lors de la cascade de phototransduction visuelle, plusieurs protéines sont successivement activées suite à l’absorption d’un photon par la rhodopsine (Figure I.8). Ultimement, la conversion de la lumière induit l’hyperpolarisation des photorécepteurs, un signal qui est ensuite transmis au cerveau pour produire la vision.
Figure I.8. Activation de la phototransduction visuelle. R : rhodopsine. R* :
rhodopsine activée. Gabgt : transducine. Gat : sous-unité a de la transducine. Gat-GTP : Gat liée au Gat-GTP. PDE : phosphodiestérase. Figure adaptée de [4].
Plus spécifiquement, la cascade de phototransduction visuelle est initiée via le chromophore de la rhodopsine, le 11-cis rétinal. En effet, lorsque le 11-cis rétinal capte un photon, il s’isomérise en tout-trans rétinal [37], ce qui mène à un changement conformationnel de la rhodopsine [14, 38]. La rhodopsine passe alors par plusieurs intermédiaires conformationnels, notamment la bathorhodopsine, la lumirhodopsine et la métarhodopsine I, jusqu’à ce qu’elle atteigne sa conformation active, la métarhodopsine II. Cette dernière conformation lui permet de lier et d’activer la transducine [39]. La transducine est une protéine G composée de 3 sous-unités (Gabgt). Comme pour la majorité des protéines G, la sous-unité a de la transducine (Gat) inclut son domaine GTPase. La Gat peut donc être liée au guanosine diphosphate (GDP) ou au guanosine triphosphate (GTP) selon que la transducine est inactive ou active, respectivement. En effet, l’activation des protéines G dépend de l’échange du GDP pour un GTP via l’action d’une GEF (Guanine nucleotide Exchange Factors) pour une protéine G monomérique et via l’action d’une GPCR lorsque la protéine G est hétérotrimérique, comme la transducine [40]. Tel que mentionné précédemment, la rhodopsine appartient à la famille des GPCR. Ainsi, l’interaction entre la forme activée de la rhodopsine et la transducine induit l’échange du GDP de la Gat pour un GTP, rendant ainsi la