L’inhibition de l’activité transcriptionnelle de PRDM6
diminue la migration et l’invasion de cellules de cancer
ovarien épithélial.
Mémoire
Jean-François Pelletier
Maîtrise en médecine expérimentale
Maître ès sciences (M.Sc.)
Québec, Canada
© Jean-François Pelletier, 2014
Résumé
La base moléculaire de la progression du cancer ovarien épithélial (COE) est encore peu comprise. Lors d’une étude antérieure, nous avons déterminé PRDM6 comme gène potentiellement hypométhylé dans les tumeurs de cancer ovarien de haut stade et grade comparés à des tissus normaux. L’hypométhylation n’a pas pu être confirmée, mais la surexpression de ce gène dans les tumeurs de COE laisse croire que PRDM6 peut avoir une implication dans ce cancer. L’expression de PRDM6 fut diminuée dans la lignée COE SKOV3 par la technique de l’interférence à l’ARN et des études fonctionnelles furent effectuées. La diminution de l’expression de PRDM6 entraine la diminution significative de la migration, l’invasion et la formation de colonie qui sont des facteurs nécessaires à la carcinogénèse. De plus, une analyse de l’expression génique a permis de confirmer la sousexpression de gènes associés au développement du cancer dans les lignées dont l’expression de PRDM6 est diminuée.
Abstact
The molecular base of the progression of epithelial ovarian cancer (EOC) is still poorly understood. In a previous study, we identified the gene PRDM6 as potentially hypomethylated in high grade and stage EOC tumors compared to normal tissue. The hypomethylation could not be confirmed, but the overexpression of this gene in EOC tumors suggests that PRDM6 might have some implications in this type of cancer. Using RNA interference, PRDM6 expression was decreased in the EOC cell line SKOV3 and functional studies were performed. Decreased expression of PRDM6 induced a significant decrease in migration, invasion and colony formation factors that are necessary for carcinogenesis. In addition, an analysis of gene expression confirmed the underexpression of genes associated with the development of cancer in cell lines whose PRDM6 expression was decreased.
Table des matières
Résumé ... III Abstact ... V Table des matières ... VII Liste des tableaux ... XI Liste des figures ... XIII Liste des abréviations ... XV Avant-propos ... XVII 1.0 Introduction ... 1 1.1 Cancer ovarien ... 1 1.1.1 Facteurs de risque ... 1 1.1.1.1 Généraux ... 2 1.1.1.2 Activité de reproduction ... 2 1.1.1.3 Incidences familiales ... 2
1.1.2 Origine, histotypes, stade et progression du cancer ovarien ... 2
1.1.2.1 Types histologique de cancer ovarien épithélial (COE) ... 3
1.1.2.1.1 Séreux ... 3 1.1.2.1.2 Mucineux ... 4 1.1.2.1.3 Endométrioïdes ... 4 1.1.2.1.4 Cellules claires ... 4 1.1.2.2 Stade et progression ... 4 1.1.2.3 Grade de la tumeur ... 5 1.1.3 Clinique ... 6 1.1.3.1 Symptômes... 6 1.1.3.2 Détection ... 6 1.1.3.3 Traitements ... 7 1.1.3.3.1 Chirurgie ... 7 1.1.3.3.2 Chimiothérapie ... 7 1.1.3.3.3 Autres traitements ... 8
1.2 Régulation épigénétique du génome ... 8
1.2.1 Historique et émergence ... 8
1.2.4 Méthylation de l’ADN ... 10
1.2.4.1 Mécanisme de méthylation ... 11
1.2.4.2 Méthylation dans le développement ... 12
1.2.4.3 Méthylation dans le cancer ... 12
1.2.4.4 Hypométhylation ... 12
1.2.4.5 Hyperméthylation ... 13
1.2.4.6 Application clinique de la méthylation ... 14
1.2.4.6.1 Biomarqueurs épigénétiques précoces de diagnostic ... 14
1.2.4.6.2 Biomarqueurs épigénétiques pronostiques ... 14
1.2.4.6.3 Biomarqueurs épigénétiques de prédiction à la chimiothérapie ... 15
1.3 Méthylation dans le cancer ovarien ... 15
1.3.1 Hyperméthylation ... 15
1.3.2 Hypométhylation ... 16
1.4 Méthyltransférases ... 16
1.4.1 Méthyltransférases de l’ADN ... 16
1.4.2 Méthyltransférases des protéines ... 17
1.4.3 PR domain containing (PRDM) ... 17
1.4.3.1 PR domain containing 6 (PRDM6) ... 17
2.0 Hypothèses et objectifs ...19
3.0 Matériels et méthodes ...21
3.1 Patientes et échantillons de tissus ... 21
3.2 Lignées cellulaires ... 21
3.3 Extraction d’ADN génomique ... 21
3.4 Extraction d’ARN ... 22
3.5 Conversion de l’ADN génomique par traitement au bisulfite ... 22
3.6 Bisulfite-Sequencing Polymerase chain reaction (BSP) ... 22
3.7 Construction du «tissue microarrays» (TMAs) ... 23
3.8 Immunohistochimie (IHC) ... 23
3.9 Conception du shARN ... 24
3.10 Transfection stable ... 24
3.11 Reverse transcriptase PCR ... 24
3.13 PCR quantitative en temps réel (RT-PCR quantitative) ... 25
3.14 Immunobuvardage ... 25
3.15 Prolifération ... 26
3.16 Formation de colonies... 26
3.17 Migration dans une chambre de Boyden ... 26
3.18 Invasion dans une chambre de Boyden ... 27
3.19 Essaie de toxicité cellulaire au cisplatin et au paclitaxel ... 27
3.20 Technologie de micropuce de l’ADN ... 28
3.20.1 Procédure de marquage... 28
3.20.2 Hybridation des échantillons marqués ... 28
3.20.3 Numérisation et extraction des données obtenues ... 28
3.20.4 Analyse des données recueillies ... 29
3.21 Analyses statistiques ... 29
4.0 Résultats ... 31
4.1 Identification de nouveaux gènes hypométhylés dans le COE par analyse de la méthylation de l’ADN par «Methylated DNA ImmunoPrecipitation» (MeDIP) couplé à une micropuce spécifiques aux îlots CpG humains. ... 31
4.2 Analyse de la méthylation du gène PRDM6 par BSP ... 32
4.3 Analyse de l’expression protéique du gène PRDM6 dans diverses échantillons de tissus normaux et tumoraux (TMA) ... 34
4.4 Analyse de l’expression protéique du gène PRDM6 dans diverses lignée cellulaire du COE . 36 4.5 Modulation de l’expression de PRDM6 par interférence à l’ARN ... 37
4.6 Analyse de la suppression du gène PRDM6 par RT-PCR semi-quantitative ... 37
4.7 Analyse de la suppression du gène PRDM6 par RT-PCR quantitative ... 38
4.8 Analyse de la suppression du gène PRDM6 par immunobuvardage ... 39
4.9 Analyses fonctionnelles ... 40
4.9.1 Essai de prolifération cellulaire ... 40
4.9.2 Essai de formation de colonies ... 41
4.9.3 Essai de migration cellulaire... 42
4.9.4 Essai d’invasion cellulaire ... 43
4.9.5 Analyse de la cytotoxicité du Cisplatin et du Paclitaxel sur les cellules ... 45
4.10 Analyse de l’expression génique après suppression du gène PRDM6 dans la lignée cellulaire SKOV3 par analyse de micropuces à ADN ... 45
4.10.1 Groupes fonctionnels choisis selon l’expression différentielle des gènes après
suppression de PRDM6 ... 46
4.10.2 Comparaison des principaux réseaux d’interactions géniques différentiellement surexprimés et sousexprimés ... 48
4.10.3 Analyse de la principale voie de signalisation touchée par la suppression de PRDM6 . 51 5.0 Discussion ...53
6.0 Conclusions ...59
7.0 Perspectives...61
Liste des tableaux
Tableau 1 : Stade de développement des tumeurs ovariennes classifiées selon le système FIGO. ... 5 Tableau 2 : Grade de développement des tumeurs ovariennes classifiées selon le système AJCC. ... 6 Tableau 3 : Tableau de gènes sélectionnés montrant une variation significative d’expression suite à la
Liste des figures
Figure 1 : Carte mondiale représentant l’incidence du cancer de l’ovaire par 100 000 femmes. ... 1 Figure 2 : Coupes histologiques des quatre principaux types histopathologiques du cancer épithélial de l’ovaire A : Adénocarcinome séreux ovarien B : Adénocarcinome endométrioïdes ovarien C : Adénocarcinome
mucineux ovarien D : Adénocarcinome ovarien de cellules claires. ... 3 Tableau 1 : Stade de développement des tumeurs ovariennes classifiées selon le système FIGO. ... 5 Tableau 2 : Grade de développement des tumeurs ovariennes classifiées selon le système (AJCC). ... 6 Figure 3 : Résumé de la voie de synthèse des miARNs de l’expression du transcrit primaire jusqu’à la
dégradation de l’ARNm ciblé. ... 10 Figure 4 : Schémas du contrôle épigénétique de la transcription des gènes par les altérations de la chromatine A : L’hyperacéthylation des histones permet la formation de l’euchromatine entrainant la transcription du gène B : L’hypoacéthylation des histones permet la formation de l’hétérochromatine ne permettant pas la
transcription du gène. ... 10 Figure 5 : Modification de la cytosine en 5-méthylcytosine par l’ajout d’un groupement méthyl en position 5 par la Dnmt. Le donneur du groupement méthyl est le SAM. ... 11 Figure 6 : Conséquences du changement de méthylation chez les gènes métastasiques et chez les gènes suppresseurs de tumeur dans le cancer non invasif et invasif. ... 13 Figure 7 : Représentation visuel, par le logiciel «Integrative Genome Viewer» (IGV), de la différence de méthylation de PRDM6 entre les différents groupes de tissus testés. A : Îlot CpG numéro 66. B : Îlot CpG numéro 121. La couleur rouge représente une diminution de la méthylation de la région analysée tandis que la couleur verte représente une augmentation de celle-ci. Le noir représente un résultat mitigé. ... 32 Figure 8 : Représentation visuelle des résultats obtenus lors du séquençage des amplicons suite au BSP pour l’îlot CpG 121. Les lignes 1 à 5 représentent les échantillons normaux tandis que les lignes 6 à 17
représentent les échantillons tumoraux. ... 33 Figure 9 : Analyse statistique des résultats obtenus lors du séquençage des amplicons suite au BSP pour l’îlot CpG 121. ... 34 Figure 10 : Analyse de l’expression protéique de PRDM6 par IHC dans différentes tumeurs de COE... 35 Figure 11 : Courbe Kaplan-Meier de survie des patientes. ... 36 Figure 12 : Immunobuvardage de la protéine PRDM6 et du gène contrôle β-actin pour diverses lignées cellulaires de cancer ovarien. ... 37 Figure 13 : Niveau d’expression du gène PRDM6 par RT-PCR semi-quantitative et du gène contrôle GUSB dans différents clones. ... 38 Figure 14 : Vérification de l’efficacité de la modulation de PRDM6 chez les clones choisis lors de la RT-PCR semi-quantitative par RT-PCR quantitative. ... 39 Figure 15 : Immunobuvardage de la protéine PRDM6 et du gène contrôle β-actin pour les clones choisis lors de la RT-PCR quantitative. ... 40 Figure 16 : Analyse de l’effet de la suppression de PRDM6 sur temps de doublement des différentes lignées cellulaires en utilisant la technologie xCelligence... 41 Figure 17 : Analyse de l’effet de la suppression de PRDM6 des différentes lignées cellulaires sur la formation de colonies après 14 jours. ... 42 Figure 18 : Effet de l’inhibition de PRDM6 sur la migration des différentes lignées cellulaires par la technique de chambre de Boyden. ... 43
Figure 19 : Effet de l’inhibition de PRDM6 sur l’invasion des différentes lignées cellulaires par la technique de chambre de Boyden. ... 44 Figure 20 : Analyse de la cytotoxicité des agents chimiothérapeutiques (Cisplatin et Paclitaxel) sur les
différentes lignées cellulaires étudiées. ... 45 Tableau 3 : Tableau de gènes sélectionnés montrant une variation significative d’expression suite à la
suppression du gène PRDM6 dans la lignée cellulaire SKOV3. ... 47 Figure 21 : Réseau d’interaction génique après analyse de surexpression (2-fold). Les gènes marqués de rouge sont surexprimés. ... 49 Figure 22 : Réseau d’interaction génique après analyse de sousexpression (2-fold). Les gènes marqués de vert sont sousexprimés. ... 50 Figure 23 : Voie de signalisation Wnt/β-catenin après analyse de sousexpression (2-fold). Les gènes marqués de vert sont sousexprimés. ... 51
Liste des abréviations
ADN Acide désoxyribonucléique ADNc ADN complémentaireADNg ADN génomique
AJCC American Joint Committee on Cancer ARN Acide ribonucléique
ARNm ARN messager miARN Micro-ARN
siARN Petit ARN interférent ncARN ARN non codant
BSP «Bisulfite-Sequencing Polymerase chain reaction» COE Cancer ovarien épithéliale
COSBG Carcinomes ovariens séreux de bas grade COSHG Carcinomes ovariens séreux de haut grade CpG Cytosine-phosphate-Guanosine
DNMT ADN méthyltransférase FBS «Fetal bovine serum»
FFPE «Formalin fixed, paraffin-embedded»
FIGO Fédération Internationale de Gynécologie et d'Obstétrique IHC Immunohistochimie
IPA «Ingenuity Pathways Analysis» LC Lignée cellulaire
MeDIP «Methylated DNA ImmunoPrecipitation»
MTS «3-(4,5-dimethylthiazol-2-yl)-5-(3-carboxymethoxyphenyl)-2-(4-sulfophenyl)-2H-tetrazolium» dNTP Mélange des quatre désoxyribonucléotides
OMS Organisation mondiale de la santé PBS Tampon phosphate salin
PCR Polymerase Chain Reaction RT-PCR Reverse-Transcriptase PCR SAM «S-adénosyl-L-méthionine»
SPSS «Statistical Package for the Social Sciences» TMA «Tissue macroarray»
Avant-propos
Avant de commencer, j’aimerais remercier certaines personnes qui m’ont permis d’accomplir tout ce travail durant les deux dernières années.
Dr. Dimcho Bachvarov qui m’a laissé la chance de faire ce projet.
Magdalena pour le lot de connaissance qu’elle m’a transmise durant ces années. Mamadou et Zhi Qiang qui furent pour moi comme des grands frères.
Bich pour ses excellents conseils.
Mes parents qui m’ont toujours poussé à faire ce que je voulais dans la vie. Mes amis avec qui il était si facile de se changer les idées.
Finalement, ma copine Valérie qui m’a toujours poussée à continuer et sur qui je peux toujours compter lorsque ça ne va pas.
1.0 Introduction
1.1 Cancer ovarien
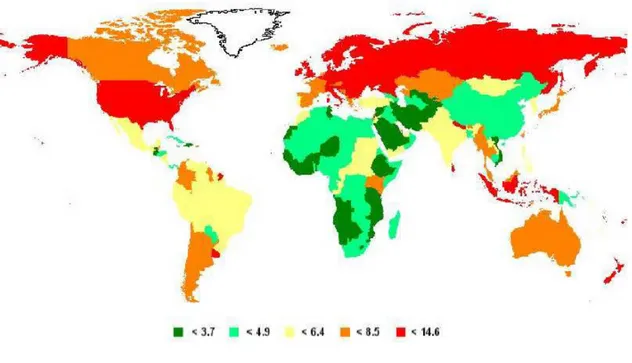
Au Canada, environ 70% des femmes diagnostiquées d’un cancer de l’ovaire mourront de cette maladie (1). En effet, en 2012, ce cancer aura été le cinquième cancer le plus meurtrier ayant causé approximativement 1 750 décès (~5% des décès totaux associés au cancer ou 7/100 000) (1). Toujours au Canada, le cancer de l’ovaire occupe le septième rang des cancers qui affectent le plus les femmes avec approximativement 2 600 nouveaux cas diagnostiqués (~3 des nouveaux diagnostiques de cancer ou 11/100 000) en 2012 (1). La probabilité, pour une femme, de développer le cancer de l’ovaire au cours de sa vie est d’environ 1 sur 69 et la probabilité d’en mourir est de 1 sur 92 (1). L’incidence de ce cancer dans les pays industrialisés est relativement semblable (2) (Figure1) ce qui peut être explicable par les divers facteurs de risques associés à ce cancer.
Figure 1 : Carte mondiale représentant l’incidence du cancer de l’ovaire par 100 000
femmes.
1.1.1 Facteurs de risque
Les facteurs de risque associés au cancer ovarien demeurent relativement peu connus et ont surtout été étudiés dans les cas de tumeurs épithéliales puisque ce sont les tumeurs les plus fréquemment rencontrées (3). Cependant, certains facteurs susceptibles d’influencer l’incidence de ce cancer ont été mis en évidences
au cours des années. Il est possible de classifier ces facteurs sous trois principales catégories, ceux qui sont généraux, ceux reliés à l’activité reproductrice et ceux provenant de l’incidence familiale.
1.1.1.1 Généraux
Le facteur de risque connu ayant la plus grande incidence pour le cancer de l’ovaire est l’âge. En effet, le risque de développer ce cancer augmente drastiquement lorsque la femme a atteint l’âge de 50 ans et l’incidence de la maladie augmente drastiquement chez les femmes ménopausées (4). Certaines études démontrent aussi une augmentation du risque d’environ 40% chez les femmes mesurant plus de 1,7m comparées à celles mesurant moins de 1,6m (5). Il ne semble pas exister de liens entre l’alimentation et l’apparition du cancer ovarien chez une femme, mais les femmes obèses (indice de masse corporelle supérieur à 30) serait jusqu’à 75% plus à risque de développer la maladie (6).
1.1.1.2 Activité de reproduction
Il a été démontré que la grossesse diminue le risque, et que ce risque diminue avec chaque grossesse ultérieure (7). L’utilisation de contraceptifs oraux pour une période d’au moins cinq ans est aussi liée à la réduction du risque de développer le cancer de l’ovaire d’environ 20% (8). Inversement, une femme n’ayant jamais accouché aurait un risque accru de développer la maladie (9).
1.1.1.3 Incidences familiales
L’hérédité constitue également un risque majeur puisqu’elle est impliquée dans 5 à 15% des cas de cancer ovarien (10, 11). Le risque relatif d’une femme, dont la mère a déjà été atteinte du cancer de l’ovaire, de développer cette maladie est de 4,6 (12). De plus, les femmes diagnostiquées pour le cancer du sein de type héréditaire possédant la mutation des gènes BRCA1 ou BRCA2 sont très susceptibles de développer un cancer de l’ovaire. En effet, on estime que 90% des cancers ovariens d’origine héréditaire sont attribués à une mutation fonctionnelle dans les gènes BRCA1/2 (10). Ces deux gènes confèrent respectivement une augmentation du risque de développer le cancer ovarien de l’ordre de 50% et de 20% (13).
1.1.2 Origine, histotypes,
stade et progression du cancer ovarien
Le cancer ovarien peut être divisé en trois catégories distinctes de tumeurs qui proviennent respectivement de cellules originaires différentes. Les tumeurs du stroma et des cordons sexuels se développant à partir du tissu conjonctif (~5% des cas) (14), les tumeurs germinales se développant à partir des cellules qui produisent les ovules (~5% des cas) (14) et les tumeurs épithéliales qui se développent à partir des cellules épithéliales situées à la surface de l’ovaire (~90% des cas) (3). Les tumeurs épithéliales sont les plus fréquemment diagnostiquées chez les patientes et sont celles qui ont été étudiée lors de ma recherche.
1.1.2.1 Types histologique de cancer ovarien épithélial (COE)
Le cancer ovarien épithélial peut être classifié en fonction du type cellulaire le composant en quatre principaux sous-types soit le séreux, le mucineux, l’endométrioïdes et celui provenant de cellules claires (15, 16). Ces sous-types de COE sont si différents, non seulement au niveau de leur arrangement cellulaire spécifique (Figure 2), mais aussi au niveau des caractéristiques génétiques moléculaires (17) et des profils protéiques (18). Ces caractéristiques sont si distinctes entre elles que ces quatre sous-types sont considérés comme étant des maladies complètement différentes. Il existe aussi des tumeurs moins fréquentes telles le carcinome épidermoïde, les tumeurs mixtes mésodermiques (carcinosarcomes), les tumeurs de Brenner et les tumeurs transitionnelles. Dans cette section, nous nous attarderons seulement sur les quatre principaux sous-types de cancer ovarien épithélial (16).
Figure 2 : Coupes histologiques des quatre principaux types histopathologiques du cancer
épithélial de l’ovaire A : Adénocarcinome séreux ovarien B : Adénocarcinome
endométrioïdes ovarien C : Adénocarcinome mucineux ovarien D : Adénocarcinome
ovarien de cellules claires.
1.1.2.1.1 Séreux
Ce sous-type est celui qui est le plus fréquemment rencontré au niveau clinique représentant environ 50% des COE malins (19). Les tumeurs séreuses sont elles-mêmes divisées en deux catégories soit les carcinomes ovariens séreux de bas grade (COSBG) et les carcinomes ovariens séreux de haut grade (COSHG). En effet, aucune étude n’a été en mesure de démontrer l’existence d’une progression des tumeurs COSBG en COSHG
ce qui en fait deux catégories différentes de cancer (20). Les tumeurs de haut grade sont les plus fréquemment retrouvées (~90% des carcinomes séreux) (21). L’inactivation du gène BRCA1 est observée dans les tumeurs de catégorie COSHG par mutation germinale ou somatique ou encore par méthylation du promoteur ce qui n’est pas observé dans les tumeurs de bas grade (22). De plus, les COSHG présentent aussi un nombre d’aberrations chromosomiques plus important ainsi que des mutations dans le gène TP53 ce qui est très rarement observé chez les COSBG (23). Des mutations dans les gènes KRAS et BRAF sont caractéristiques des tumeurs COSBH et sont très rarement observés dans les COSHG (24).
1.1.2.1.2 Mucineux
Les carcinomes de type mucineux comptent pour approximativement 5-10% des cancers de l’ovaire (19). Dans la majorité des cas (~80%) elles sont bénignes et ont un pronostic favorable (3). Tout comme dans le cas des COSBG, les tumeurs mucineuses présentent souvent dans mutations dans le gène KRAS (25).
1.1.2.1.3 Endométrioïdes
Le sous-type endométrioïde compte pour approximativement 5-10% des cancers de l’ovaire (3). Contrairement aux tumeurs mucineuses, les tumeurs endométrioïdes rencontrées au niveau clinique sont majoritairement (~80%) considérées comme étant malignes (3, 19, 26). Au niveau des altérations génétiques, ces tumeurs possèdent souvent des mutations somatiques des gènes bêta-catenin et PTEN (27). De plus, dans les tumeurs de haut grade, TP53 est muté dans environs 90% des cas (28). Malgré tout, il faut souligner que les tumeurs endométrioïdes malignes sont considérées comme ayant un meilleur pronostic que les tumeurs malignes séreuses et mucineuses (3, 29).
1.1.2.1.4 Cellules claires
Les tumeurs provenant de cellules claires représentent environ 5-10% des tumeurs ovariennes (3). Contrairement aux autres sous-types, les mutations de TP53 (28, 30, 31), de KRAS, de BRAF (31) et de BRCA (32) sont plutôt rare. Cependant, le taux de survie 5 ans après le diagnostic des patientes atteintes d’une tumeur de cellules claires sont beaucoup moins bons que les autres types histopathologiques ce qui peut être expliqué par le fait que les tumeurs de cellules claires sont plus résistantes à la chimiothérapie (33). De plus, les tumeurs bénignes et les cas limites qui sont des tumeurs de potentiel malin bas couramment nommés «borderline» dans la littérature pour cet histotype sont rarement observés, la majorité des tumeurs de ce type étant malignes (34).
1.1.2.2 Stade et progression
L’utilisation du terme stade, dans le cas du cancer ovarien, réfère à la taille de la tumeur et à son degré de propagation dans l’organisme. En effet, le stade décrit le degré de propagation de la tumeur primaire aux
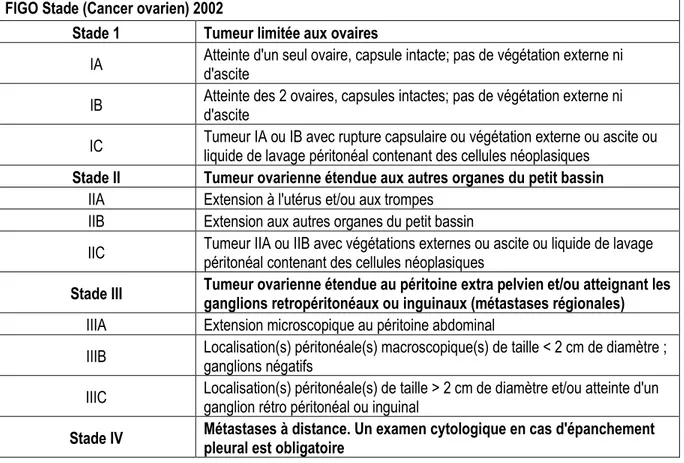
organes locaux. Les tumeurs ovariennes peuvent se disséminer dans le péritoine et la cavité abdominale via les ascites, un liquide contenant des cellules cancéreuses, les lymphocytes et les cellules mésothéliales (35, 36). Puisque le degré de propagation du cancer dans l’organisme est un facteur pronostique significatif (37), le système de stade a été créé par la Fédération Internationale de Gynécologie et d'Obstétrique (38) (FIGO) (Tableau 1). Cette classification divise les cancers de l’ovaire selon quatre stades distinctifs dont les taux de survie respectifs sont 93 % pour le stade I, 70 % pour le stade II, 37 % pour le stade III et 25 % pour le stade IV (37).
Tableau 1 : Stade de développement des tumeurs ovariennes classifiées selon le système
FIGO.
FIGO Stade (Cancer ovarien) 2002
Stade 1 Tumeur limitée aux ovaires
IA Atteinte d'un seul ovaire, capsule intacte; pas de végétation externe ni d'ascite IB Atteinte des 2 ovaires, capsules intactes; pas de végétation externe ni d'ascite IC Tumeur IA ou IB avec rupture capsulaire ou végétation externe ou ascite ou liquide de lavage péritonéal contenant des cellules néoplasiques
Stade II Tumeur ovarienne étendue aux autres organes du petit bassin
IIA Extension à l'utérus et/ou aux trompes IIB Extension aux autres organes du petit bassin
IIC Tumeur IIA ou IIB avec végétations externes ou ascite ou liquide de lavage péritonéal contenant des cellules néoplasiques
Stade III Tumeur ovarienne étendue au péritoine extra pelvien et/ou atteignant les ganglions retropéritonéaux ou inguinaux (métastases régionales)
IIIA Extension microscopique au péritoine abdominal
IIIB Localisation(s) péritonéale(s) macroscopique(s) de taille < 2 cm de diamètre ; ganglions négatifs IIIC Localisation(s) péritonéale(s) de taille > 2 cm de diamètre et/ou atteinte d'un ganglion rétro péritonéal ou inguinal
Stade IV Métastases à distance. Un examen cytologique en cas d'épanchement pleural est obligatoire
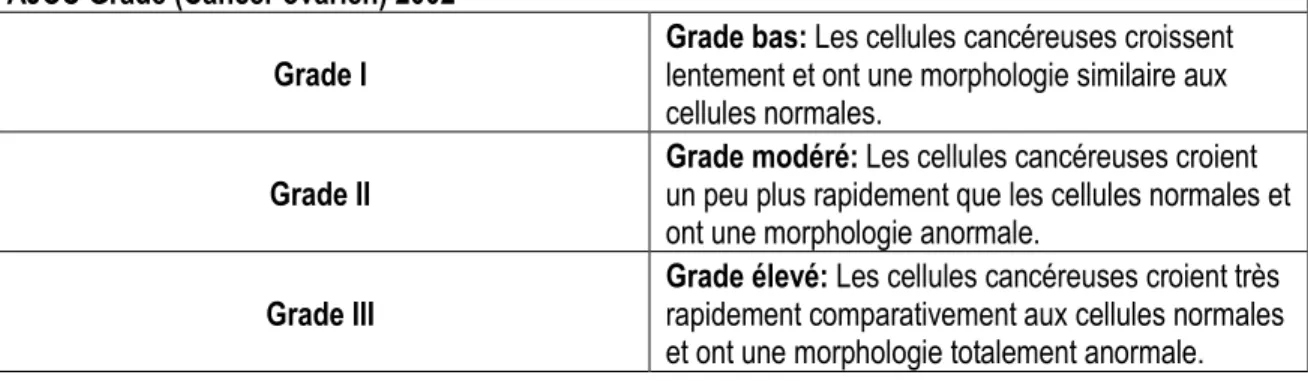
1.1.2.3 Grade de la tumeur
Contrairement au système de classification par le stade qui s’attarde à la progression de la tumeur à l’intérieur de la patiente, la classification par le grade considère la vitesse de croissance et le degré de différenciation de la cellule exclusivement (39). Il existe trois types de grade pour le cancer ovarien (Tableau 2) et il est plus probable que le cancer se propage si son grade est élevé (3).
Une nouvelle classification en fonction des grades à récemment été proposée. Elle divise la gradation en deux groupes au lieu de trois qui sont les tumeurs de bas grade et celles de haut grade (20). Les tumeurs de bas
grade démontrent une atypie nucléaire minimale et un index mitotique faible tandis que les tumeurs de haut grade, elles, démontrent une atypie nucléaire et un index mitotique élevé (20). Cette nouvelle méthode de gradation permet l’obtention de valeurs pronostiques significatives, mais les difficultés liées à sa reproductibilité limitent son application en clinique (40).
Tableau 2 : Grade de développement des tumeurs ovariennes classifiées selon le système
(AJCC).
AJCC Grade (Cancer ovarien) 2002
Grade I Grade bas: Les cellules cancéreuses croissent lentement et ont une morphologie similaire aux cellules normales.
Grade II Grade modéré: Les cellules cancéreuses croient un peu plus rapidement que les cellules normales et ont une morphologie anormale.
Grade III Grade élevé: Les cellules cancéreuses croient très rapidement comparativement aux cellules normales et ont une morphologie totalement anormale.
1.1.3 Clinique
Dans cette section, l’accent sera porté sur les enjeux cliniques qu’implique le cancer ovarien. On y parlera des symptômes qu’occasionnent ce cancer et les méthodes de détection couramment utilisées par les cliniciens pour le détecter. Puis, une section portera sur les méthodes utilisées afin de traiter les patientes atteintes de cette maladie dans l’espoir d’arriver à une rémission complète.
1.1.3.1 Symptômes
Il n’y a aucun symptôme spécifique au cancer ovarien. Les symptômes les plus fréquemment observés chez les patientes sont la distension abdominale, l’urination fréquente, les ballonnements, la constipation, les nausées, l'anorexie ou une sensation de satiété précoce (41, 42). La plupart de ces symptômes sont occasionnés par la présence d’une masse pelvienne prenant de l’expansion dans la cavité abdominale des patientes. La masse peut être détectée lors de l’examen pelvien et est indicative de la présence d’une tumeur ovarienne (33). Un examen plus approfondi de la région pelvienne par échographie transvaginale est effectué lorsque le cancer de l’ovaire est suspecté (33).
1.1.3.2 Détection
L’absence de symptôme spécifique à ce cancer rend sa détection précoce très difficile. De plus, aucun programme de dépistage n’est disponible afin de faciliter sa détection contrairement au cancer du sein et au cancer du col utérin. L’obtention d’un nouveau marqueur sérique pour le diagnostic précoce du cancer ovarien est une priorité (43). Pour le moment, le marqueur biologique antigène tumoral 125 (CA 125) demeure le seul
indicateur de la progression ou la régression de ce cancer (44). Malheureusement, il ne peut pas être utilisé dans une optique de test diagnostique puisque son niveau dans le sang est variable en fonction de certaines conditions féminines telles la menstruation ou la grossesse (45). Cependant, la protéine épididymaire humaine de type 4 (HE4) commence à être reconnue comme étant aussi un marqueur biologique intéressant (46). Il faut aussi souligner que certaines études recommandent l’utilisation de groupes de biomarqueurs afin d’obtenir un diagnostic plus fiable (47, 48).
1.1.3.3 Traitements
En général, le traitement du cancer de l’ovaire s’effectue en deux temps. Premièrement, le clinicien procède à la chirurgie cytoréductive afin de retirer le plus de masse tumorale possible. Ensuite, la patiente devra habituellement subir un traitement de chimiothérapie afin de se débarrasser des cellules cancéreuses toujours présentes après la chirurgie. Il existe aussi des traitements plus marginaux utilisés en derniers recours. Le choix du traitement dépend de plusieurs facteurs tels le stade et le grade de la maladie, de l’histopathologie ainsi que de l’âge et la santé globale de la patiente (49).
1.1.3.3.1 Chirurgie
La chirurgie permet d’identifier le stade et le grade de la tumeur ce qui permet au médecin de faire un meilleur diagnostic et d’identifier si d’autres traitements seront nécessaires et elle permet aussi d’enlever la majeure partie de la masse tumorale (33). Un meilleur pronostique est d’ailleurs associé aux patientes où la masse résiduelle mesure moins de 1 cm. Il existe plusieurs types de chirurgies différentes en fonction de la tumeur devant laquelle se retrouve le chirurgien. Les principales chirurgies pratiquées en clinique sont l’hystérectomie totale (ablation de l’utérus, le col utérin, les trompes de Faloppe et les ovaires), la salpingo-ovarectomie (ablation des trompes de Faloppe et des ovaires), l’omentectomie (ablation de l’epiploon) ou la lymphadénectomie (ablation des ganglions)(33). Bien que ce ne soit pas une pratique fréquente, une ablation chirurgicale préventive des ovaires peut être pratiquée chez la patientes ayant plusieurs antécédents familiaux de cancer de l’ovaire et cancer du sein (50).
1.1.3.3.2 Chimiothérapie
Habituellement, la chimiothérapie est utilisée suite à la chirurgie. L’utilisation de ces médicaments vise à détruire les cellules cancéreuses résiduelles. Cependant, la chimiothérapie n’est pas spécifique aux cellules cancéreuses et touche aussi les cellules normales ce qui cause divers effets secondaires tels l’anémie (baisse de la quantité de globule rouge), la leucopénie (baisse de la quantité de globule blanc), la thrombocytopénie (baisse de la quantité de plaquettes sanguines), des syndromes gastro-intestinaux (nausée, vomissement, diarrhée, etc.) et une perte temporaire des cheveux sans toutefois oublier le risque de perte de fertilité des patientes (51).
Les lignes directrices pour le traitement recommandent que la chimiothérapie standard devrait inclure un mélange de dérivés du platine et du taxane comme le carboplatine et le paclitaxel (33). D’autres médicaments, tel le topotecan, peuvent aussi être utilisés afin d’améliorer le résultat de la chimiothérapie. La fréquence, le dosage, la voie d’injection et les composés utilisés lors de la chimiothérapie sont déterminés par différents facteurs (le type de cancer, le stade et le grade de la maladie, la réponse au traitement, la santé globale de la patiente, etc.) (52). Plus de 80% des patientes répondent au traitement initial (53), mais la majorité de celles étant atteinte de la maladie à un stade avancé feront une rechute. Les guérisons des patientes après récidives sont très rares et la prise en charges de celles-ci se fait souvent dans une unité de soin palliatif (33).
1.1.3.3.3 Autres traitements
Étant donné que la guérison des patientes après une récidive est très rare, certaines thérapies émergentes ont été développées. Trois principales stratégies ont été étudiées au cours des dernières années soit la thérapie génique, la thérapie hormonale et le traitement par inhibiteurs. La thérapie génique s’attarde à l’inhibition de la machinerie cellulaire spécifique aux cellules cancéreuses (54). La thérapie hormonale, elle, vise principalement à contrôler la croissance et le développement de la tumeur. C’est une avenue particulièrement intéressante, car c’est une thérapie déjà utilisé dans le traitement du cancer du sein (55). Le traitement par inhibiteur se concentre sur l’inhibition des voies de réparation cellulaire ce qui augmenterait l’efficacité des traitements de chimiothérapies (56). Malgré tout, puisque chaque histotype est considéré comme étant une maladie spécifique et que les aberrations au niveau de leurs voies de signalisations cellulaire ne sont pas partagées entre elles, il est difficile de concevoir une thérapie ciblant l’ensemble des histotypes. Il est primordial de trouver de nouvelles cibles spécifiques à un histotype précis. Les données déjà recueillis sur les thérapies émergentes nous laisse croire qu’elles pourraient aisément être employées seules ou en combinaison avec des traitements existants afin d’augmenter le nombre de patientes atteignant la rémission complète.
1.2 Régulation épigénétique du génome
1.2.1 Historique et émergence
C’est en 1942 qu’on entend parler pour la première fois du terme «épigénétique» (57). Cette appellation fut proposée par Conrad Waddington pour représenter la branche de la biologie étudiant les interactions s’effectuant entre les gènes et leur environnement définissant le phénotype final d’un individu. L’épigénétique est un concept qui fut mis de côté lors de la découverte de la double-hélice d’ADN. L’avènement du séquençage complet de certains génomes dans les années 90 et l’incapacité de les déchiffrés remirent l’épigénétique en avant plan. De nos jours, l’épigénétique se définit plutôt comme l’étude des changements héréditaires dans la fonction des gènes sans altération de la séquence d’ADN. Récemment, il a été possible
d’associer les mécanismes épigénétiques à certaines maladies non cancéreuses (58), mais aussi à plusieurs cancers différents (59-61) dont le cancer ovarien (62).
Trois principaux mécanismes épigénétiques ont été découvert et étudié jusqu’à maintenant. Premièrement, l’expression des micro-ARNs (miARN), puis les modifications post-traductionnelles des queues d’histones et finalement la méthylation de l’ADN (63).
1.2.2 Expression des micro-ARNs
Les miARNs font partie du groupe des ARN non codants (ncARNs) et ont habituellement une longueur de 20 à 22 nucléotides. Ils sont impliqués en tant que répresseurs de la traduction de l’ARNm en protéine (64, 65). On évalue que ce mécanisme participe à la régulation de tous les processus cellulaire en régulant l’expression d’environ 60% des gènes codant pour les protéines (66). Le mécanisme d’action des miARNs, en résumé, est le suivant. Premièrement, un transcrit primaire est synthétisé par l’ARN polymérase II. Ce transcrit est modifié par Drosha (67) et son cofacteur DGCR8 (68). Il en résulte une molécule d’ARN plus petite qui migre vers le cytoplasme à l’aide du transporteur nucléaire Exportin-5 (69). Le transcrit est ensuite traité par la protéine Dicer et la protéine de liaison à l’ARN double brin TRBP afin de produire le miARN fonctionnel (70). Le miARN est ensuite chargé dans le complex RISC composé de Dicer, TRBP et de la protéine Argonaute. Ce complexe cible les ARNm par complémentarité des séquences de la région 3’ non transcrite et les ARNm reconnus sont détruits (71) (Figure 3).
Figure 3 : Résumé de la voie de synthèse des miARNs de l’expression du transcrit primaire
jusqu’à la dégradation de l’ARNm ciblé.
La dérégulation de l’expression de certains miARNs a un impact oncologique important. Par exemple, la régulation des miARNs miR-15 et miR-16 est très souvent perturbée dans le cas de la leucémie lymphoïde chronique (72). Une des causes les plus fréquentes de la perte de l’expression de miRNAs suppresseurs de tumeurs est l’hyperméthylation des îlots CpG de leur transcrit primaire (73).
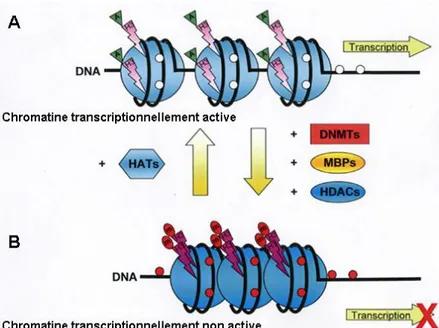
1.2.3 Modification de la chromatine
Il existe deux formes de compaction de la chromatine chez l’homme. La première est l’euchromatine qui est la forme relâchée permettant la transcription active des gènes. Elle est souvent associée à l’hyperacétylation des histones particulièrement au niveau de la lysine 9 de l’histone (74). La deuxième forme de chromatine est l’hétérochromatine qui, elle, est plus compacte rendant la transcription des gènes impossible. Elle est souvent associée à l’hypoacétylation des histones dont la lysine 9 de l’histone H3 qui est méthylé (75) (Figure 4).
Figure 4 : Schémas du contrôle épigénétique de la transcription des gènes par les altérations
de la chromatine A : L’hyperacéthylation des histones permet la formation de
l’euchromatine entrainant la transcription du gène B : L’hypoacéthylation des histones
permet la formation de l’hétérochromatine ne permettant pas la transcription du gène.
1.2.4 Méthylation de l’ADN
La méthylation de l’ADN se caractérise par l’ajout d’un groupement méthyl sur un nucléotide soit l’adénine, la thymine, la guanine ou la cytosine. Cependant, la base le plus fréquemment méthylé est la cytosine et ce, indépendamment de l’espèce. En effet, la méthylation de l’ADN est un mécanisme conservé à travers
l’évolution (76). Les procaryotes ont la capacité de méthyler l’adénine et la cytosine. La méthylation de l’adénine joue un rôle dans la réparation et l’initiation de la réplication de l’ADN tandis que la méthylation de la cytosine protège l’ADN des enzymes de restriction (77). Chez les plantes, la cytosine est la seule base pouvant être méthylée et a comme rôle l’inactivation des éléments transposables, des allèles parentaux, des transgènes et des paramutations (78). Chez l’homme, seule une cytosine (C) suivi d’une guanine (G) (CpG) peut être méthylé (79). Les rôles de cette méthylation ressemblent à ceux des plantes. Toutefois, elle est aussi impliquée dans la régulation de nombreux gènes.
1.2.4.1 Mécanisme de méthylation
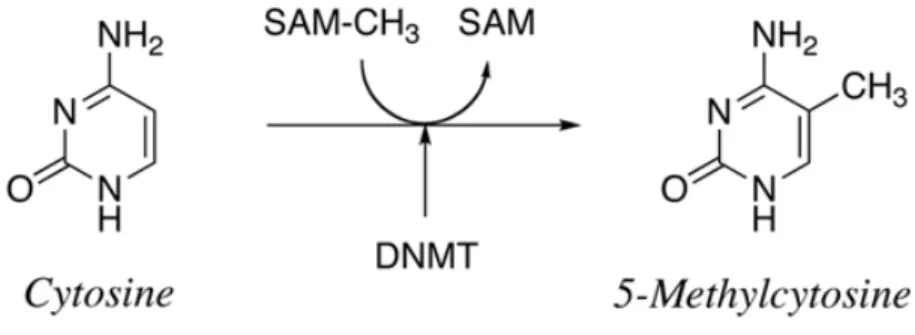
Si l’on se concentre spécifiquement sur l’espèce humaine, ce sont les ADN méthyltransférases (DNMTs) qui sont responsable de la génération et de l’entretient de la méthylation présente sur les cytosines de l’ADN (80). C’est un lien covalent qui associe le groupement méthyl à la cytosine et cette réaction est réalisée en présence du donneur de groupement méthyl la S-adénosyl- L-méthionine (SAM). Ce métabolite est synthétisé à partir de la méthionine, de la choline et d’autres co-facteurs. La réaction de méthylation est réalisée par transfert du groupement méthyl du SAM vers le carbone en position cinq de la cytosine (81). (Figure 5)
Figure 5 : Modification de la cytosine en 5-méthylcytosine par l’ajout d’un groupement
méthyl en position 5 par la Dnmt. Le donneur du groupement méthyl est le SAM.
Jusqu’à ce jour, quatre types d’ADN méthyltransférases ont été identifiés (82). La première est la DNMT1 qui est responsable du maintien de la méthylation après la réplication de l’ADN en intervenant au niveau de la phase de synthèse (S) du cycle cellulaire (83). La deuxième et troisième sont les DNMT3A et DNMT3B. Ce sont les enzymes permettant de réaliser la méthylation de novo en permettant l’ajout de groupements méthyls sur les deux brins de l’ADN non méthylé (83). La dernière est la DNMT2 qui ne possède qu’une faible activité catalytique in vitro (82).
1.2.4.2 Méthylation dans le développement
La méthylation du génome est un élément essentiel lors du développement de l’embryon. En effet, le traitement de cellules par un agent déméthylant a induit une différentiation cellulaire spontanée ce qui démontre l’implication de la méthylation dans le contrôle de la différenciation cellulaire (84).
Chez l’embryon de la majorité des mammifères, quelques heures après la fécondation, le génome provenant du père est activement déméthylé tandis que celui de la mère est déméthylé passivement par un mécanisme dépendant de la réplication (85). Par la suite, pendant l’implantation de l’embryon, la méthylation est restaurée par des mécanismes de novo. À cette étape, l’embryon ne sera pas viable s’il porte une mutation entrainant le dysfonctionnement des DNMTs. Une deuxième vague de reprogrammation de la méthylation survient, lorsque l’embryon est bien implanté, faisant disparaitre les groupements méthyles sur les gènes des cellules germinales primordiales (86). La méthylation sera rétablie, en fonction du sexe, sur les loci maternels ou paternels. Chez le mâle ce phénomène se produit dès la naissance tandis qu’il se produira plutôt dans les ovocytes matures chez la femelle. Ce mécanisme joue un rôle dans le phénomène d’empreinte parentale (87). Bien qu’ils soient tous deux nécessaire au développement de l’embryon, il existe une différence fonctionnelle entre les génomes d’origine maternelle et paternelle. En effet, pour certains gènes un seul allèle est exprimé tandis que l’autre est réprimée par le phénomène de régulation par méthylation. L’absence de la copie d’intérêt conduira alors à de nombreuses maladies comme le syndrome de Prader-Willi (88).
Afin d’assurer une uniformité de l’expression des gènes situés sur les allosomes au niveau des chromosomes X, un des deux chromosomes X est inactif chez les mammifères femelles. On peut observer diverses marques associés à la répression génique sur le chromosome X inactivé tel un fort niveau de méthylation de l’ADN, un faible niveau d’acétylation des histones, un faible niveau de méthylation de la lysine 4 de l’histone H3, un fort niveau de la méthylation de la lysine 9 de l’histone H3 et la présence d’une variante d’histone nommé la macroH2A exclusive à ce chromosome inactivé (89).
1.2.4.3 Méthylation dans le cancer
Dans les cancers, l’intégralité du patron de méthylation des cellules normales est bouleversée. Dans certains cas, le groupement méthyl du 5-méthylcytosine sera perdu, ce phénomène est reconnu comme étant une hypométhylation de l’ADN. Dans d’autres cas, il y aura ajout d’un groupement méthyl sur la cytosine ce qui est plutôt reconnu comme étant une hyperméthylation de l’ADN (61, 90, 91).
1.2.4.4 Hypométhylation
Tel que mentionné précédemment, l’hypométhylation aberrante des séquences de l’ADN est ciblée aux séquences qui sont méthylées dans le tissu normal. Bien que ce phénomène n’ait pas été grandement étudié
(92, 93), il est connu qu’un tel changement dans le patron de méthylation des cellules contribue à l’instabilité génomique ainsi qu’à l’activation transcriptionnelle ponctuelle d’oncogènes. L’hypométhylation semble être un évènement précoce de la tumorogénèse, y compris dans les tumeurs pré-malignes (94, 95).
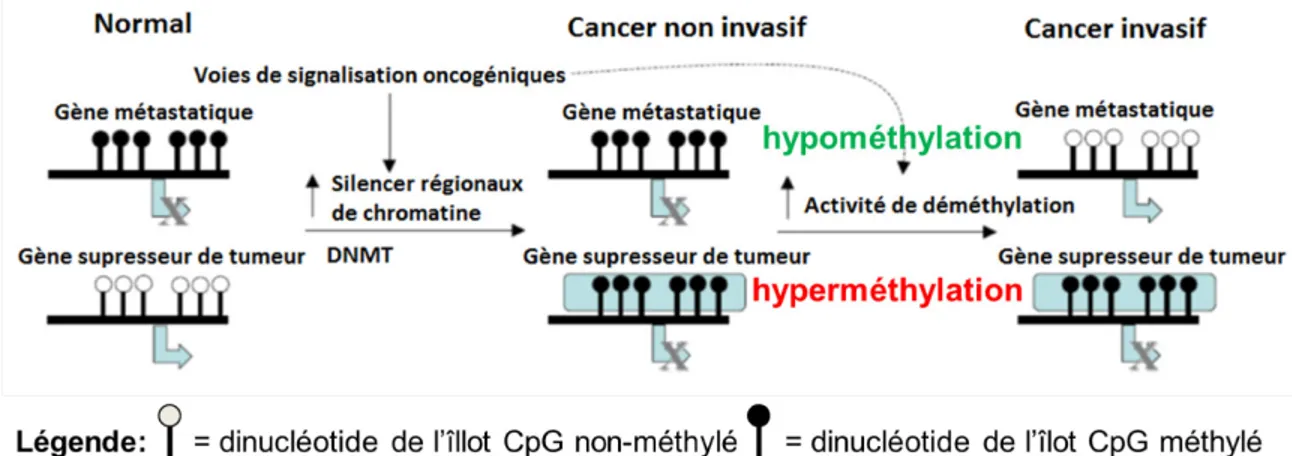
L’hypométhylation de la région promotrice de certains gènes joue aussi un rôle important dans l’invasion tumorale, dans la métastase et la mortalité chez différents cancers (96) (Figure 6). Par exemple, l’hypométhylation, dans le cancer ovarien, des satellites Sat2 augmente avec la progression tumorale (97). On peut considérer cet évènement comme étant un marqueur de mauvais pronostic pour ce type de tumeur. De plus, certaines études ont démontré, dans le cancer ovarien, que le niveau d’hypométhylation est fortement lié au stade de la maladie ainsi qu’au grade tumoral et que la fréquence des évènements de déméthylation augmente avec la progression tumorale (98).
Cependant, il n’est pas encore démontré hors de tout doute que l’hypométhylation est la cause du cancer plutôt qu’une conséquence résultant d’effets secondaires du développement tumoral (99).
Figure 6 : Conséquences du changement de méthylation chez les gènes métastasiques et
chez les gènes suppresseurs de tumeur dans le cancer non invasif et invasif.
1.2.4.5 Hyperméthylation
Contrairement à l’hypométhylation, l’hyperméthylation se produit lorsqu’un groupement méthyl est ajouté sur la cytosine. L’hyperméthylation des ilôts CpG a été beaucoup plus étudié que l’hypométhylation. Ceci a permis de découvrir qu’elle conduit souvent à l’inactivation des gènes suppresseurs de tumeur impliqués dans plusieurs voies de signalisation comme la réparation de l’ADN (MGMT, MLH1, DAPK) (100), la régulation du cycle cellulaire (CDKN2A) (100, 101), l’apoptose (RASSF1A, CASP8) (101, 102), le métabolisme de la carcinogène, la réponse hormonale et l’adhérence cellulaire (103-105). Malgré tout, dans la plus part des cas, les effets fonctionnels des gènes réprimés par l’hyperméthylation sur la pathogenèse du cancer de l'ovaire ne
sont pas connus. Il a été démontré que la méthylation des gènes SFRP1, 2, 4, 5, SOX1, PAX1 et LMX1A est plus élevé dans le cancer ovarien que dans les tissus «borderlines» ou normaux (106). De plus, plusieurs études ont démontré qu’il était possible de reconnaitre les différents sous-types ovariens en fonction du patron d’hyperméthylation (107, 108).
1.2.4.6 Application clinique de la méthylation
Les différences observées dans le patron de méthylation des différents sous-types de cancer ovarien comparés entre eux ainsi qu’aux tissus ovariens normaux offrent plusieurs applications cliniques intéressantes. Premièrement, il est possible d’utiliser ces différences en tant que biomarqueur chez la patiente. On peut alors diviser ces biomarqueurs en trois catégories distinctes soit ceux pouvant permettre un diagnostic précoce de la maladie, ceux pouvant indiquer un pronostic et ceux permettant de prédire la réponse qu’aura la patiente face à la chimiothérapie. Ces applications seront discutées dans les prochaines lignes.
1.2.4.6.1 Biomarqueurs épigénétiques précoces de diagnostic
Afin d’assurer la réussite du traitement du cancer de l’ovaire, il est primordial d’obtenir un diagnostic précoce ce qui est présentement quasi impossible étant donné l’apparition tardive des symptômes et l’absence de modalités de dépistage adéquates (109). L’identification de biomarqueurs détectables dans les liquides biologiques pourrait faciliter la détection de ce cancer à un stade précoce de la maladie. Les marqueurs spécifiques de l’ADN méthylé peuvent être détectés dans le sérum, le plasma et le liquide péritonéal des patientes atteintes d’un cancer de l'ovaire (110, 111). Bien sûr, l’analyse de la méthylation d’un seul gène n’est pas suffisamment spécifique pour permettre le diagnostic du cancer ovarien, mais en utilisant une multitude de gènes, il est possible d’y arriver (112). Par exemple, en analysant la méthylation de six gènes suppresseurs de tumeurs (BRCA1, RASSF1A, APC, p14 arf, p16 ink4a et DAPK) il est possible de déterminer si une patiente est atteinte d’un cancer ovarien avec 100% de spécificité et 82% de sensibilité (110).
Bien qu’il existe encore plusieurs limitations, comme la sensibilité, à l’utilisation des tests de méthylation, la détection de marqueurs épigénétiques dans les fluides corporels des patientes semble être une approche prometteuse pour la détection précoce du cancer ovarien.
1.2.4.6.2 Biomarqueurs épigénétiques pronostiques
Présentement, ce sont les facteurs clinico-pathologiques tels le stade et le grade de la tumeur, le type histologique ainsi que la maladie résiduelle après la chirurgie initiale qui sont utilisés comme facteur pronostiques du cancer ovarien (113, 114). L’utilisation de marqueurs de pronostiques utilisant la méthylation pourrait permettre de compéter les facteurs classiques et améliorer l’indice pronostique de chaque patiente. Par exemple, la méthylation de HOXA11 est liée à une tumeur résiduelle post-chirurgicale et est associée à un
mauvais pronostic (115) tandis que le gène FBXO32 est associé à une survie sans progression à court terme (116). L’utilisation de ces nouveaux marqueurs pronostiques pourrait permettre un traitement plus personnalisé pour chaque patiente.
1.2.4.6.3 Biomarqueurs épigénétiques de prédiction à la chimiothérapie
Puisque chaque type de tumeur possède un patron de méthylation relativement semblable, il pourrait être possible d’y associer une réponse potentielle au traitement (107, 108). La résistance à la chimiothérapie est l’un des principaux obstacles à la survie des patientes atteintes du cancer ovarien. Plusieurs modifications épigénétique de la méthylation des gènes ont déjà été étudiées en lien avec la résistance à la chimiothérapie (117). Par exemple, l’hyperméthylation du gène MLH1 est associée à la résistance aux dérivés platine dans les lignées cellulaires de cancer de l'ovaire (118). Il y a aussi l’exemple de la méthylation des gènes REST et TUBB3 qui ont été liés à la résistance au paclitaxel dans des lignées cellulaires de cancer de l'ovaire (119). Finalement, on peut noter que la surexpression de la protéine MAL (myelin and lymphocyte protein) par hypométhylation de la région promotrice était associée à un mauvais pronostic et à la résistance au platine dans le cancer de l’ovaire (120). Par conséquent, le gène MAL semble être un marqueur potentiel de la sensibilité au platine. L’utilisation de ces biomarqueurs pourrait non seulement améliorer le niveau de vie des patientes qui n’auront pas à subir un traitement qui ne fonctionnera probablement pas, mais permettra aussi au gouvernement d’économiser d’importantes sommes d’argent en éliminant les traitements inutiles.
1.3 Méthylation dans le cancer ovarien
Lors de mes études graduées, il me fut possible de travailler sur un article publié au tout début de l’année 2013 (121). Les prochaines lignes résumeront les découvertes associées à cet article et en quoi elles ont influencé mon projet de recherche à la maîtrise.
1.3.1 Hyperméthylation
Pour ce qui est de l’hyperméthylation, les résultats obtenus tendent à démontrer qu’il y a génération, tant dans les tumeurs de potentiel malin bas «borderline» que dans les tumeurs de stade avancé, d’une forte hyperméthylation de novo de gènes homéotiques ainsi que d’autres gènes impliqués dans la régulation développementale. Ce phénomène a déjà été observé dans d’autres types de cancer comme celui du poumon (122), cerveau (123), sein (124), colorectal (125), prostate (126) et les lymphomes (127). De plus, nous avons observé la présence de certains gènes (HOXA9, HOXA10, SOX1, PAX1 et LMX1A) ayant un potentiel de biomarqueur épigénétique pronostic ou diagnostic étant hyperméthylés (128).
1.3.2 Hypométhylation
Contrairement à l’hyperméthylation qui était observé même dans les tumeurs à potentiel malin bas, l’hypométhylation n’est présente que dans les tumeurs ovariennes séreuses de haut grade (G3). Ceci fut confirmé en comparant le profil de méthylation de cultures cellulaires primaires dérivées de tumeurs ovariennes séreuses de deux patientes avant et après chimiothérapie.
À notre connaissance, c’était la première fois que quelqu’un observait une hypométhylation massive de l’ADN dans les tumeurs séreuses ovariennes de stade avancé. Tel que mentionné précédemment, l’hypométhylation est associée à l’expression de nombreux oncogènes fonctionnellement associés à une augmentation de la progression et du potentiel malin de la tumeur dont l’augmentation de l’invasion tumorale et des métastases (96). D’ailleurs, une autre équipe de recherche est arrivée à des résultats semblables en utilisant une méthodologie similaire, mais cette fois sur des échantillons de cancer du foie. Ils ont identifié 3 700 gène déméthylés chez les tumeurs qui sont principalement impliqués dans la croissance cellulaire, l’adhésion et la communication cellulaire, la mobilité et l’invasion (129).
C’est en analysant les données obtenues pour l’hypométhylation que nous avons généré une liste de gène hypométhylés ayant un potentiel oncogénique. Le gène PRDM6 faisant partie de la famille des PRDM qui sont des méthyltransférases d’histones faisait partie de cette liste. Il a été sélectionné comme candidat potentiellement hypométhylé ayant une implication oncogénique dans le cancer ovarien. Les caractéristiques de ce gène et sa famille protéique seront présentés dans la section suivante.
1.4 Méthyltransférases
Les méthyltransférases sont des enzymes dont le rôle est de transférer un groupement méthyl à partir d’un donneur vers un receveur (130). Habituellement, le groupement donneur de cette enzyme est le S-adénosyl méthionine (130). Les receveurs les plus fréquents sont les acides nucléiques de l’ADN (cytosine) et les acides aminés des protéines (130). On peut classifier les méthyltransférases en deux groupes distincts soit les méthyltransférases de l’ADN et les méthyltransférases des protéines.
1.4.1 Méthyltransférases de l’ADN
Comme leur nom le suggère, les méthyltransférases de l’ADN transfèrent les groupements méthyl sur les acides nucléiques de l’ADN. Il existe trois catégories d’enzymes ayant une activité méthyltransférase qui sont connus, mais dans le règne animal la seule présente est la C5-méthylcytosine transférase (131). Tel que discuté précédemment, chez l’homme ces enzymes sont nommées les DNA méthyltransférases et sont
essentiellement actives au niveau des ilots CpGs (82). Leurs actions sont essentielles pour la bonne régulation génique.
1.4.2 Méthyltransférases des protéines
Puisqu’il existe un grand nombre de protéines différentes qui peuvent être modifiées par l’ajout d’un groupement méthyl, il existe une panoplie de méthyltransférase de protéines (132, 133). Cependant, celles qui ont le plus d’influence au niveau épigénétique sont les méthyltransférases des histones. Tout comme les méthyltransférases de l’ADN, celles qui sont spécifiques aux histones ont une grande influence biologique au niveau de l’expression génique, la stabilité du génome, le développement de l’embryon, etc. (134).
1.4.3 PR domain containing (PRDM)
À ce jour, 17 gènes de la famille PRDM ont pu être identifiés (135). Récemment, cette famille a commencé à susciter beaucoup d’intérêt puisqu’il a été démontré que les gènes en faisant parti ont une implication dans la différenciation cellulaire et semblent être impliqués dans plusieurs maladies chez l’homme (135). Les protéines transcrites par ces gènes sont des méthyltransférases d’histones (136). De plus, certaines études semblent indiquer que certains membres de cette famille de gènes pourrait avoir un potentiel oncogénique ou suppresseur de tumeurs (137-140).
1.4.3.1 PR domain containing 6 (PRDM6)
PRDM6, aussi connu sous le nom de PRISM (PR domain in smooth muscle) fait partie de cette famille de méthyltransférase d’histones (141). Il existe deux transcrits différents pour ce gène soit le transcrit long et le transcrit court. Le transcrit court n’est pas traduit en protéine et ne semble pas avoir de rôle biologique connu. Le transcrit long, par contre, est traduit en une protéine de 595 acides aminés (135).
Les études sont très contradictoires quant au rôle de méthyltransférase d’histone de cette protéine in vitro. Certaines affirment qu’elle effectuerait spécifiquement la méthylation de la lysine 20 de l’histone 4 (141) tandis que d’autres affirment que ce n’est pas le cas (142).
Le mécanisme d’action de cette protéine a été étudié chez les cellules du muscle lisse. PRDM6 aurait la propriété d’inhiber la différentiation cellulaire chez les cellules musculaires lisses permettant une prolifération accrue (141). Inversement, chez les cellules endothéliales, il aurait un rôle de répresseur de la prolifération cellulaire ainsi que de la survie et de la différenciation (142). Il est possible que ces résultats plutôt contradictoires soient causés par l’analyse de différents variants de la protéine (135).
Finalement, une étude a aussi mise en évidence l’expression de PRDM6 lors de la neurogenèse du système nerveux centrale chez les souris. PRDM6 pourrait influencer la différenciation et la spécialisation des neurones via la voie de Notch (143).
2.0 Hypothèses et objectifs
L’hypométhylation des ilots CpG peut entraîner l’activation de la transcription de certains gènes. Ce mécanisme anormal de régulation peut survenir chez les gènes impliqués dans la carcinogénèse comme les oncogènes, les gènes régulant le cycle cellulaire, les gènes de proliférations cellulaire et plusieurs autres (96). L’hypothèse de départ de mon projet de maîtrise est que certains gènes impliqués dans la migration, l’invasion et la formation de métastases dans le COE pourraient être activés par l’hypométhylation de leur région promotrice contribuant à la progression tumorale.
Le premier objectif était de confirmer, dans une cohorte indépendante, l’hypométhylation du gène PRDM6 qui est potentiellement hypométhylé dans les tumeurs de cancer ovarien de haut stade et grade comparés à des tissus normaux selon les résultats obtenus par immunoprécipitation de l’ADN méthylé (MeDIP) couplé à une micropuce spécifique aux ilôts CpG humains effectué précédemment (121). Nous avons supposé que ce gène avait un potentiel oncogénétique dans le cancer ovarien épithéliale.
Le deuxième objectif était de confirmer le rôle d’oncogène de PRDM6 dans l’étiologie du COE en effectuant des essaies fonctionnels in vitro dans les lignées cellulaires du COE.
Finalement, nous avons aussi supposé que PRDM6 pouvait être au centre de certains mécanismes de régulation pouvant être lié à la progression, l’invasion et la migration cellulaire.
Le dernier objectif était de déterminer quelles molécules pourraient être influencées par le niveau d’expression de PRDM6 dans le COE.
3.0 Matériels et méthodes
3.1 Patientes et échantillons de tissus
Les échantillons de tissus, congelés et conservés dans des blocs de paraffine (FFPE), de 117 tumeurs de cancer ovarien séreux ont été obtenus à l’hôpital Hôtel-Dieu de Québec, Québec, Canada. Parmi ces échantillons il y avait 13 échantillons «borderline», 52 échantillons d’adénocarcinomes de haut grade et 52 tissus de métastases ovariennes. Aucune patiente n’avait reçu de traitement de chimiothérapie avant leur chirurgie respective. Toutes les tumeurs ont été classifiées histologiquement par une équipe de pathologiste selon les critères définis par l’organisation mondiale de la santé (OMS) (144). La progression de la maladie a aussi été évaluée selon les recommandations du «Gynecology Cancer Intergroup» (144). Les traitements de chimiothérapies ont été complétés et la réponse individuelle face à celui-ci est connue pour chaque patiente. La survie sans récidive des patientes a été définie comme étant le temps entre le jour de la chirurgie et la première observation de progression de la maladie, de récidive ou la mort de la patiente. Nous avions aussi accès aux tissus de 13 échantillons d’ovaires normaux provenant de femmes ayant subi une ablation des ovaires pour cause n’ayant aucun lien avec une pathologie. Cette étude a été approuvée par le comité d’éthique sur la recherche clinique de l’Hôtel-Dieu de Québec et toutes les patientes ont signé un formulaire de consentement éclairé.
3.2 Lignées cellulaires
La lignée cellulaire (LC) SKOV3, dérivée du COE, a été cultivée dans du milieu RPMI (Wisent, Saint-Bruno, QC, CAN) avec 10 % de sérum de veau foétal (FBS) (Wisent, Saint-Bruno, QC, CAN) et 1 % de Pénicilline/Streptomycine (Wisent, Saint-Bruno, QC, CAN) tel que décrit par le passé (145). Les autres lignées cellulaires utilisées ont été cultivées dans les conditions déterminées précédemment au laboratoire (145-147).
3.3 Extraction d’ADN génomique
Le tissu a été soigneusement disséqué et la section tumorale identifiée lors de l’analyse de la coupe histologique. L’ADN génomique (ADNg) a été extrait en suivant les directives du protocole «DNeasy® Blood & tissue Kit» (QIAGEN). Environ 20 mg de tissus ont été coupés en petit morceau et placés dans un tube eppendorf de 1,5 mL contenant du buffer ATL et 10 mg de protéinase K (GibcoBRL) pour un volume total de 200 µL. Le tout a été incubé de 12 à 18 heures à 56°C. La concentration d’ADN a été obtenue par lecture avec un spectrophotomètre (Nanodrop® ND-1000).
3.4 Extraction d’ARN
L’ARN a été extrait en suivant les directives du protocole «RNeasy® Plus mini Kit» (QIAGEN). Elle a été effectuée à partir d’un pétris de 100 mm contenant une culture cellulaire étant à confluence. Les cellules ont été placées dans un tube eppendorf avec du tampon RLT contenant 1 % v/v de ß-mercaptoéthanol (Sigma-Aldrich, St-Louis, MS, USA) pour un volume total de 600 µL. Le tout a été vortexé et passé à la seringue (aiguille de 20-gauge) environ 10 fois pour obtenir un bon lysat. La concentration a été obtenue par lecture avec un spectrophotomètre (Nanodrop® ND-1000) et la qualité de l’ARN mesuré avec le Bioanalysateur Agilent 2100.
3.5 Conversion de l’ADN génomique par traitement au bisulfite
La quantité d’ADNg utilisé avec le «MethylDetectorTM Bisulfite Modification Kit» (Active Motif) a été de 500 ng par échantillon. La sulfonation des cytosines non-méthyles fut effectuée en incubant le mélange d’ADNg, d’hydroquinone et de tampon de conversion dans un thermocycleur (T-Gradient Biometra) à 94°C pendant trois minutes suivi de l’étape de conversion à 50°C pendant neuf heures. L’ADN sulfoné fut désulfoné et purifié suivant le protocole «MethylDetectorTM». La conversion a été vérifiée par PCR en utilisant le gène p16 comme gène contrôle. L’amplification PCR a été effectuée en suivant le protocole de «MethylDetectorTM». Le résultat de l’amplification PCR a été vérifiée sur gel d’agarose 2,5 % dans du tampon TBE 1X afin de confirmer la réussite de la conversion.3.6 Bisulfite-Sequencing Polymerase chain reaction (BSP)
La séquence de PRDM6 (NM_001136239.1) provient du site «Human Genome Browser» (www.genome.ucsc.edu) et a été utilisée afin d’identifier les îlots CpG présents pour ce gène dont l’hypométhylation avait initialement été observé en utilisant le logiciel «Integrative Genomic Viewer» (IGV). L’analyse de l’ilôt CpG choisi a été effectuée avec le logiciel «MethylPrimer Express V1.0» (Applied Biosystems) afin de déterminer la séquence d’intérêt pour la BSP.
Quatre microlitres d’ADNg converti par réaction ont été utilisé pour les analyses de la méthylation par BSP. Deux PCR consécutives ont été nécessaires pour obtenir un amplicon ayant une qualité acceptable pour le séquençage. Une dilution 1 dans 100 a été effectuée à partir de la première amplification PCR pour procéder à la deuxième amplification PCR (PCR imbriquée). La PCR imbriquée a été faite afin d’éliminer les contaminations produites par les amplifications provenant de mésappariements inattendus des amorces à des séquences non spécifiques. La Taq polymèrase (Feldan Québec, QC, CAN) a été utilisé lors de la PCR. La PCR primaire (amorce sens : TAGAGGGAGAGATGTAGAGAGATT et amorce anti-sens :
ACCCATCTTTAAATAAACTCCC) a été fait dans un volume de 50 µL suivant les conditions suivantes : une première étape de dénaturation à 95°C pendant 5 minutes, suivi de 30 cycles de 94°C pendant 30 secondes, 58,3°C pendant 30 secondes, et 72°C pendant 30 secondes, et une dernière étape d’élongation à 72°C pendant 10 minutes. La PCR imbriquée (amorce sens : GGAGGAAGAAGTTTGGAATT et amorce anti-sens : ACCCATCTTTAAATAAACTCCC) a été effectuée en suivant le même protocole que la première PCR.
Les échantillons ont été purifiés en suivant le protocole «PCR Purification kit» (Feldan, Québec, QC, CAN) et la concentration a été vérifiée avec un spectrophotomètre (Nanodrop® ND-1000). Les échantillons ont été dilués à 1 ng/mL avant d’être envoyé à la Plate-forme d'Analyses Génomiques de l'Université Laval (http://www.bioinfo.ulaval.ca/seq/fr/index.php ) afin de procéder au séquençage. Les données obtenues ont été analysés avec le logiciel Jalview (http://www.jalview.org/).
3.7 Construction du «tissue microarrays» (TMAs)
Le TMA a été construit en se basant sur les directives passées (146). Brièvement, un bloc FFPE représentatif de chaque échantillon de tissus normal ou de tumeur ovarienne a été choisi pour la préparation du TMA. Pour chaque échantillon, trois carottes de tissus d’une grosseur de 0,6 mm ont été prélevées. Les échantillons ont été placés à 1,0 mm de distance les unes des autres à l’aide d’un «Tissue Arrayer» MTA-II (Beecher Instruments, Sun Prairies, WI, USA). Les carottes de tissus ont été insérés dans un des quatre blocs de paraffine récepteurs selon un patron prédéterminé où les tissus d’un même bloc donneur ne se retrouvaient pas les uns à côté des autres afin d’éviter les biais de coloration et d’évaluation lors de l’analyse de l’immunohistochimie. Finalement, des coupes des blocs de TMAs d’une largeur de cinq microns ont été déposées sur lame afin de procéder à la coloration par immunohistochimie.
3.8 Immunohistochimie (IHC)
L’immunohistochimie a été effectuée en se basant sur les directives passées(146, 147). Afin que l’anticorps et l’antigène puissent être mis en contact, les lames de TMA ont subis une déparaffinassions avant d’être submergés dans un tampon Tris-EDTA à pH 9,0 (10 mM Tris Base, 1 mM EDTA et 0,05 % Tween20) (Labmat, Québec, QC, CAN) chauffé à 95°C pour une durée de 30 minutes. L’activité des peroxydases endogènes aux tissus a été bloquée par un traitement au H2O2 3 % pour une période de 10 minutes. Ensuite, une incubation d’une durée d’une heure à température pièce avec l’anticorps primaire anti-PRDM6 (dilution 1 : 50) (Abnova, Taipei City, Taiwan). Ceci fut suivi par une deuxième incubation avec l’anticorps secondaire biotinylé (Dako, Carpinteria, CA, USA) couplé à la streptavidine. La réaction permettant la révélation de la coloration a été provoquée par l’addition de 3-3’ diaminobenzidine (DAB) (Dako, Carpinteria, CA, USA) suivit d’une contre coloration à l’hématoxyline (Leica Biosystems, Concord, ONT, CAN). Finalement, l’expression
protéique a été déterminée de façon semi-quantitative par un pathologiste selon le système de notation de l’intensité du signal suivant : absent (0); faible (1); modéré (2) ou fort (3).
3.9 Conception du shARN
La séquence cible a été déterminer en combinant les résultats obtenus par trois logiciels différents (siRNA at WhiteHead http://sirna.wi.mit.edu/home.php, siRNA Wizard v3.1 http://www.sirnawizard.com/ et GeneLink http://www.genelink.com/sirna/shRNAi.asp). Trois séquences étaient communes aux logiciels : GTTCAGTACAGGTCGAATATA, CAGCATCTCTGGGAGATATAT et TACCGAGCCTGTATAGATATC.
3.10 Transfection stable
Le vecteur de transfection utilisé était le «pSilencer 4.1 puro vector» (Ambion, Austin, TX, USA) dans lequel nous avons inséré chacune des séquences spécifiques à l’ARNm de PRDM6. Parallèlement, nous avons aussi inséré une séquence non spécifique dans un de ces vecteurs qui servira de contrôle. Une journée avant la transfection, nous avons ensemencé dans chaque puits d’une plaque six puits 2 X 105 cellules. Le lendemain la transfection a été effectuée avec 4 µg d’ADN plasmidique et 6 µL de réactif à transfection «TurboFect» (Thermo Scientific, Ottawa, ON, CAN). Après 48 heures d’incubation nous avons procédés à la sélection des clones stables avec un traitement à la puromycine (Wisent, Saint-Bruno, QC, CAN) à la concentration de 1 µg/mL pour une période de deux semaines avant de procéder à la récolte de clones individuels.
3.11 Reverse transcriptase PCR
L’extraction d’ARN a été effectuée tel que mentionné au point 3.5
Pour l’ARN ayant été utilisé lors de la PCR semi-quantitative, la synthèse du premier brin d’ADN complémentaire (ADNc) a été effectuée en utilisant 2 µg d’ARN dans un volume total de 10 µl et 1 µl d’amorce oligo dT15 (50 ng/µl). Ce mélange a été incubé à 70°C pour une période de 10 minutes avant d’être refroidi sur glace pendant 3 minutes. Les réactifs suivants ont été rajoutés : 4 µl de «5xFirst Strand buffer» (Life Technologies, Burlington, ON, CAN), 2 µl de dithiothréitol (DTT) à 0,1 M (Life Technologies, Burlington, ON, CAN), 1 µl de dNTPs et 1 µl de «RNA guard» (Life Technologies, Burlington, ON, CAN). Le tout a été amené à une température de 42°C (pour 2 minutes) et 1 µl de «SuperScript RT» (Life Technologies, Burlington, ON, CAN) a été ajouté et le mélange complet a été incubé à 42°C pendant une heure et désactivé par incubation à 70°C pendant 15 minutes.