Caractérisation génomique et transcriptomique de la
microspore embryogénique chez l'orge
Thèse
Sébastien Bélanger
Doctorat en biologie végétale
Philosophiæ doctor (Ph. D.)
Caractérisation génomique et
transcriptomique de la microspore
embryogénique chez l’orge
Thèse
Sébastien Bélanger
Sous la direction de :
François Belzile, directeur de recherche
Pierre-Étienne Jacques, codirecteur de recherche
Résumé
L’androgenèse est une biotechnologie végétale utilisée pour fixer le bagage génétique des plantes en une seule génération. Elle repose sur la capacité d’un grain de pollen immature, la microspore, à restaurer sa totipotence cellulaire végétale et se dédifférencier puis s’engager dans la voie de l’embryogenèse. Or, on observe que la capacité de la microspore à s’engager dans l’embryogenèse est génétiquement variable. En dépit des nombreux avantages qu’elle présente, l’androgenèse entraîne souvent une conséquence indésirable, soit une distorsion de la ségrégation (DS) chez les populations issues de cette biotechnologie. Ma thèse porte sur l’étude (i) du transcriptome de la microspore en transition entre la voie de développement du grain de pollen et celle de l’androgenèse et (ii) de la DS pour déterminer quand la DS survient dans le processus et où elle affecte le génome. J’ai utilisé l’orge comme espèce modèle pour mon étude.
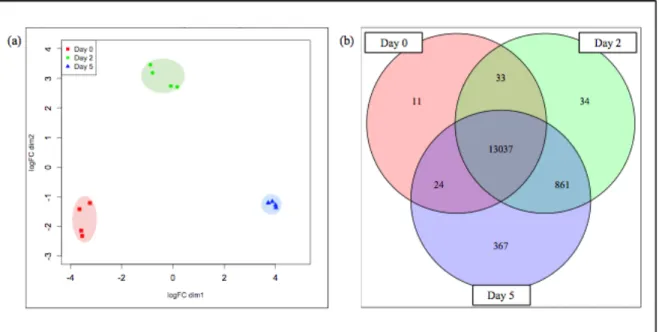
L’analyse transcriptomique a été réalisée sur la microspore isolée de l’anthère à trois stades, soit avant (jour 0) et immédiatement après (jours 2 et 5) l'application d'un traitement de stress visant à induire la voie de l’androgenèse. Je me suis intéressé à deux catégories de gènes; soit les gènes exprimés exclusivement à un stade précis et les gènes exprimés différemment lors de l’initiation de l’androgenèse. J’ai pu identifier des gènes exprimés exclusivement dans la microspore au jour 0 (11), 2 (34) ou 5 (367). Au jour 5, j’ai constaté l’induction de nombreux gènes codant pour des FT et des gènes impliqués dans la synthèse ou la transduction du signal de nombreux régulateurs de croissance. L’analyse des gènes exprimés différemment m’a permis d’identifier certains processus métaboliques qui sont activés/réprimés lors du passage de la microspore du jour 0 au jour 2 et du jour 2 au jour 5. Les gènes exprimés exclusivement à un stade précis du développement pourraient servir de marqueurs moléculaires indicateurs de la performance en androgenèse pour optimiser les protocoles de culture.
Ensuite, la DS a été étudiée par une approche de génotypage à l’échelle génomique. J’ai d’abord développé une méthodologie d’analyse génotypique novatrice, reproductible et précise pour étudier la fréquence allélique sur un
échantillon en composite. Cette méthode m’a permis d’étudier la fréquence allélique chez 1) des échantillons de microspores (avant et après l’application d’un stress), 2) des embryons et 3) des plantes régénérées. J’ai montré que la DS survenait à la fois lors du développement des embryons et la régénération des plantes. Aucune DS n’a été observée chez les échantillons de microspores. Mes résultats montrent que la sélection engendrant la DS s’opère au cours de la culture in vitro. Toujours à l’aide de cette méthode de génotypage en composite, j’ai identifié et comparé la fréquence et l’étendue de la DS chez 12 populations de lignées haploïdes doublées (HD). Dans cette seule étude, un plus grand nombre de populations de lignées HD (12) ont été caractérisées que ce qui avait été fait dans la somme de toutes les études antérieures dans la littérature scientifique. Nous avons montré que les régions de distorsion de ségrégation différaient beaucoup quant à leur position, leur étendue et l’amplitude de la distorsion d’un croisement à l’autre. La connaissance de ces allèles permettrait de prédire le potentiel androgénique des génotypes dans un programme de sélection.
Mes travaux de thèse élèvent à l’ère des « omics » la recherche chez la microspore d’orge dans le système de l’androgenèse. À une échelle sans précédent, mon étude transcriptomique explore et décrit les changements d’expression génique au cours de la transition développementale de la microspore. Mon étude génomique identifie quand s’exerce la sélection engendrant la distorsion de la ségrégation des allèles dans ce système et décrit quelles régions chromosomiques sont affectées par cette distorsion. À la lumière de mes résultats, dans le dernier chapitre, je propose certaines pistes de recherche pour poursuivre l’étude des mécanismes moléculaires dirigeant la transition développementale de la microspore en androgenèse et pour développer des outils de génotypage afin d’utiliser la DS comme un outil d’amélioration génétique.
Abstract
Androgenesis is a plant biotechnology used to fix the genetic background of plants in a single generation. This is based on the ability of an immature pollen grain, the microspore, to restore its totipotency, to dedifferentiate and then to engage in the path of embryogenesis. However, it is observed that the ability of the microspore to engage in embryogenesis is genetically variable. Despite the many desirable attributes of androgenesis, an undesirable side-effect is the segregation distortion (SD) encountered in populations resulting from this biotechnology. My thesis focuses on (i) the study of the transcriptome of microspores undergoing a developmental transition from the pollen-grain pathway towards embryogenesis and (ii) to identify when SD arises in the process and in which genomic regions it occurs. I used barley as a model species for my studies.
Transcriptomic analysis was performed on microspores isolated from anthers at three stages corresponding to the microspore before (day 0) and immediately after (days 2 and 5) the application of a stress treatment aimed at inducing embryogenesis. I was interested in two categories of genes: those expressed exclusively at a specific stage of microspore development and those that were differentially expressed during the initiation of androgenesis. I was able to identify genes expressed exclusively in the microspore on day 0 (11), 2 (34) or 5 (367). On day 5, I found the induction of many genes encoding transcription factors (TFs) in addition to genes involved in the synthesis or signal transduction of many growth regulators. The analysis of differentially expressed genes allowed me to identify certain metabolic processes that were activated/repressed during microspore development from day 0 to 2 and from day 2 to 5. Genes expressed exclusively at a specific stage of development could serve as molecular markers indicative of the performance in androgenesis to optimize isolated microspore culture protocols. Then, SD was studied using a whole-genome genotyping approach. I first developed an innovative, reproducible and accurate genotypic analysis methodology to determine allelic frequency on pooled samples. This method was then used to estimate allelic frequencies in samples of microspores (before and
after the application of stress), embryos and regenerated plants. I showed that SD arises during both the development of embryos and the regeneration of plants. No SD was observed in samples of microspores. My results show that the selective forces promoting SD act during in vitro culture. Still using the same genotyping method performed on pooled samples, I identified and compared the frequency and extent of SD in 12 populations of doubled haploid lines (DH). A greater number of DH (12) populations were characterized in my study alone than the sum of all previous studies in barley. I showed that segregation distortion regions greatly differ in their position, extent, and magnitude in different DH populations. Knowledge of these alleles would be useful to predict the androgenic potential of a genotype in a breeding program.
My dissertation has allowed research into barley microspores, or more widely androgenesis, to enter into the “omics” era. On an unprecedented scale, my transcriptomic study explores and describes the gene expression changes that occur during the developmental transition that the microspore undergoes in the course of androgenesis. My genomic study identifies when the selection (producing SD) arises in this system and describes which chromosomal regions are affected by this distortion. In light of my findings, in the final chapter I propose some lines of research to further study the molecular mechanisms driving the developmental transition from microspores to embryos and to develop genotyping tools to use SD as a genetic improvement tool.
Table des matières
Résumé ... iii
Abstract ... v
Table des matières ... vii
Liste des tableaux ... xi
Liste des figures ... xii
Liste des abréviations ... xvi
Remerciements ... xvii
Avant-propos ... xviii
Introduction générale ... 1
Chapitre I ... 3
Revue bibliographique ... 3
1.1 Reproduction, évolution et agriculture ... 4
1.2 L’embryogenèse ... 5
1.3 L’androgenèse ... 8
1.3.1 Biologie, physiologie et contrôle moléculaire de l’androgenèse ... 10
1.3.1.1 L’acquisition du potentiel embryogénique ... 11
1.3.2.2 L’initiation de la division cellulaire et la formation des embryons .. 13
1.3.2 Limites de l’androgenèse ... 17
1.3.2.1 La réponse génétique à l’androgenèse observée est variable ... 17
1.3.2.2 La distorsion de ségrégation observé chez les populations d’haploïdes doublées ... 18
1.3.3 Facteurs limitant la compréhension des mécanismes moléculaires de dirigeant l’androgenèse ... 20
1.4 L’orge est un bon modèle pour étudier l’androgenèse ... 21
1.5 Les outils technologies pour étudier l’androgenèse à l’ère des « omics » ... 23
1.5.1 L’étude de l’ARN ... 23
1.5.2 L’étude de l’ADN ... 26
Chapitre II ... 31
Differential expression profiling of microspores during the early stages of isolated microspore culture using the responsive barley cultivar Gobernadora 31 2.1 Résumé ... 33
2.2 Abstract ... 34
2.3 Introduction ... 35
2.4 Materiel & methods ... 37
2.4.1 Plant materials ... 37
2.4.2 Cellular fixation and microscopy of microspores ... 38
2.4.3 RNA isolation, library construction and sequencing ... 38
2.4.4 Data processing, analysis of differential gene expression and gene clustering ... 39
2.4.5 Gene functional annotation ... 40
2.5 Results and discussion ... 40
2.5.1 Isolation and purification of microspores at three key stages of IMC ... 40
2.5.2 Transcriptomes at three stages of isolated microspore culture ... 43
2.5.3 Genes exclusively expressed in microspores at day 5 provided highlight about metabolism governing the embryogenesis induction ... 45
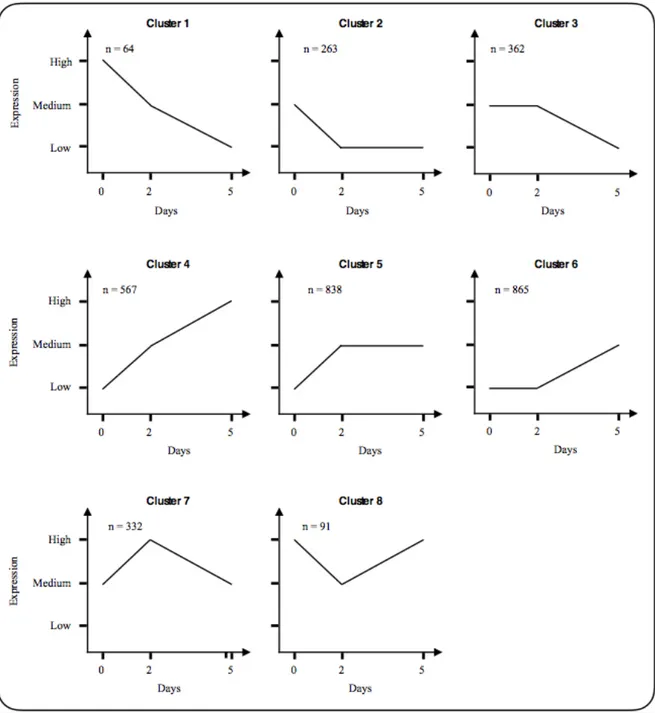
2.5.4 DEG analysis and clustering ... 48
2.5.5 Metabolisms controlled by differentially expressed genes in microspore from day 0 and 2 ... 49
2.5.6 Metabolisms controlled by differentially expressed genes in microspore from day 2 and 5 ... 53
2.6 Concluding remarks ... 59
2.7 Acknowledgments ... 59
Chapitre III ... 61
Genotyping-by-Sequencing on Pooled Samples (Pool-GBS) and its Use in Measuring Segregation Bias during the Course of Androgenesis in Barley ... 61
3.1 Résumé ... 63
3.2 Abstract ... 64
3.3 Introduction ... 65
3.4.1 Plant material ... 67
3.4.2 Genomic DNA isolation, GBS library construction and sequencing ... 68
3.4.3 SNP discovery and genotype calling ... 69
3.4.4 SNP filtering ... 70
3.4.5 Genetic mapping of SNPs, estimation of allelic frequencies and segregation bias analysis ... 71
3.4.6 Impact of pool size on SNP depth of coverage and on the variability of estimated allelic frequencies ... 71
3.4.7 Comparison of allelic frequencies estimated individually vs. in pools and between pool replicates ... 72
3.5 Results ... 72
3.5.1 Optimized strategy for estimating allelic frequencies based on the number of reads ... 72
3.5.2 Accuracy of estimated allelic frequencies ... 75
3.5.3 Validation of the Pool-GBS approach ... 78
3.5.4 Importance of uniform pools ... 80
3.5.5 Segregation biases arising during androgenesis in barley ... 81
3.6 Discussion ... 83
3.6.1 Simple and efficient GBS methodology to estimate allelic frequencies in pooled samples ... 83
3.6.2 Importance of uniform pools ... 86
3.6.3 Pooled genotyping is extremely cost-effective ... 86
3.6.4 Segregation bias occurs mainly during the final phases of androgenesis ... 87
3.7 Acknowledgments ... 88
Chapitre IV ... 89
Extent and Overlap of Segregation Distortion Regions in Twelve Barley Crosses Determined via a Pool-GBS Approach ... 89
4.1 Résumé ... 91
4.2 Abstract ... 92
4.4 Materiel and methods ... 95
4.4.1 Plant material ... 95
4.4.2 Genomic DNA isolation, GBS library construction and sequencing ... 96
4.4.5 SNP discovery, genotype calling and SNPs filtering ... 97
4.4.6 Genetic mapping of SNPs, estimation of allelic frequencies and segregation analysis ... 97
4.5 Results ... 98
4.5.1 Anchoring numerous SNPs on the barley consensus map using Pool-GBS ... 98
4.5.2 Extent of segregation distortion in twelve segregating populations ... 99
4.5.3 Extent of overlap of SDRs in 12 segregating populations ... 100
4.5.4 Segregation distortion in reciprocal crosses ... 103
4.6 Discussion ... 109
4.6.1 Extent of segregation distortion ... 109
4.6.2 Extent of overlap between SDRs ... 110
4.7 Acknowledgements ... 112
Chapitre V ... 124
Discussion générale et conclusions ... 124
Liste des tableaux
Table 2.1 Subset of genes that were newly and specifically in microspores at day
5. ... 46
Table 2.2 List of up-regulated genes in microspores from day 0 to day 2. ... 51
Table 2.3 List of down-regulated genes in microspores from day 0 to day 5. ... 54
Table 2.4 List of up-regulated genes in microspores from day 2 to day 5. ... 56
Table 4.1 Characteristics of the segregating populations used in this work. ... 100
Table 4.2 Extent of SD in 12 barley crosses. SD was measured in terms of the number of SDRs, the percentage of markers showing biased segregation as well as the total genetic distance and percentage of the genetic map affected by SD. ... 101
Table 4.3 Location, magnitude and direction of SD in 12 barley populations obtained via androgenesis ... 105
Liste des figures
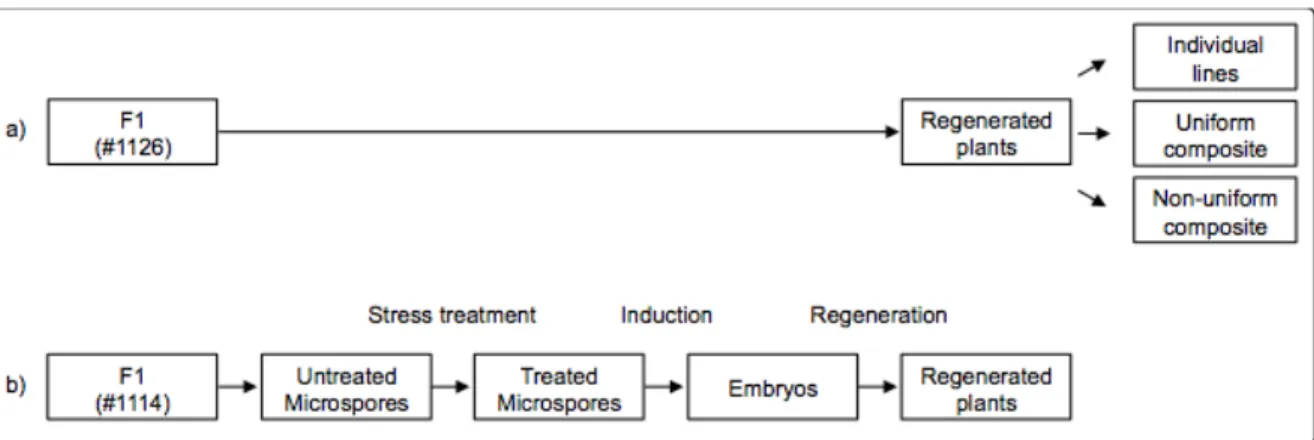
Figure 1.1 Les voies de développements embryogéniques chez les plantes. Cette figure est adaptée de l’article de revue de Maraschin et al. (2005). ... 7 Figure 1.2 Illustration de la fixation génétique par autofécondation ou par embryogenèse gamétique chez un croisement biparental. ... 8 Figure 1.3 Le système biologique de l’androgenèse. Le système biologique de l’androgenèse (a) montre la déviation développementale de la microspore où elle quitte la voie de formation du grain de pollen pour celle de l’embryogenèse qui permet la régénération d’une plante haploïde doublée. Ce système biologique se divise en trois phases développementales (b) correspondant à l’acquisition du potentiel embryogénique, l’initiation des divisions cellulaires et la formation des structures d’un embryon. Cette figure est adaptée de l’article de Maraschin et al. (2006). ... 10 Figure 2.1 Representative samples of microspores obtained from the cv. Gobernadora at the three stages of early isolated microspore culture. (a) Mid-late to late uninucleate microspores (day 0), (b) enlarged microspores (day 2) and (c) star-like microspore to multi-cellular structure microspore phenotype (day 5). Images were captured using a Zeiss Apoptome microscope under UV laser illumination (excitation of 390/22 nm and emission of 460/50 nm) at a 20x magnification without staining (left) and with DAPI staining (right). ... 42 Figure 2.2 Variation in gene expression profiles within and between microspore samples. An MDS plot shows the high degree of uniformity among replicates of the same stage as well as the distinctness between stages (a) and a Venn diagram illustrates the high degree of overlap among genes expressed in the different cell types (b). ... 43 Figure 2.3 Schematic clusters representing different patterns of gene expression among differentially expressed genes (DEGs). Clusters were intuitively generated by integrating each gene expression pattern (expression of a gene can go up, down or remain unchanged) at both developmental transitions (from day 0 to day 2 and from day 2 to day 5) producing a total of eight different expression patterns. 50 Figure 3.1 Source of the plant materials used in this work. Two F1 barley plants
were used to perform androgenesis. (a) Three samples were collected from the DH progeny resulting from the cross HV779STP07 × Oceanik (#1126: individual lines, uniform composite (“pool”) and non-uniform composite. (b) Four samples were collected during androgenesis performed on the Myriam × UL163 (#1114) cross. These samples consisted of populations of microspores before treatment, microspores after stress treatment, embryos, and regenerated plants. ... 68 Figure 3.2 Schematic diagram of the analytical strategy explored in this work. (a) For the individual analysis (i.e., line by line), genotyping by sequencing (GBS) data were generated on 76 barley DH lines using separate barcodes and each line was
analyzed individually. b) The reads from the individual analysis (a) were combined and 125 million reads were split into five equal sets of 25 million reads to be used as replicates for the artificial pool-based strategy. c) For the artificial pool-based strategy, five pools containing a number of reads ranging from 5 million to 25 million were generated from each replicate and analyzed separately. In summary, five artificial pools were tested per replicate for a total of 25 pools. ... 70 Figure 3.3 Impact of pool size and depth of coverage on the number of SNPs. The number of SNPs obtained was determined at four depths of coverage (≥10, 50, 100 and 200 reads/SNP) for each pool size (5 to 25M). A regression line was computed for each depth of coverage. ... 74 Figure 3.4 Variation of the estimated allelic frequencies at various read depths. The coefficient of variation for the estimated allelic frequencies obtained using each pool size (5 to 25M) at four minimum depths of coverage (≥10, 50, 100 and 200 reads) was measured in five replicates. A regression line was computed for each depth of coverage. ... 75 Figure 3.5 Pairwise correlation between allelic frequencies measured individually or in artificial pools. The allelic frequencies estimated through the analysis of 76 individual DH lines and from artificial pools analyzed at five depths of sequencing (5M to 25M reads) and four depths of coverage (≥10, 50, 100 and 200 reads/SNP) were compared by computing Pearson’s correlation coefficient (r). ... 77 Figure 3.6 SD on chromosome 2H measured using four different approaches. Allelic frequencies were estimated a) individually using a total of 125M reads from 76 barley DH lines, b) on an artificial pool of 15M reads, c) on an artificial pool of 15M reads, and d) on a uniformly sampled pool of 15M reads. The x-axis shows the position (in cM) of markers or marker bins along the chromosome and the y-axis shows the estimated allelic frequency. Each symbol indicates the allelic frequency of a parental allele (black circle: HV779STP07; red circle: Oceanik) at each SNP (a and b) or averaged over all SNPs located within a 5-cM bin (c and d). ... 78 Figure 3.7 Impact of sample uniformity for Pool-GBS analysis. These graphs show the estimated allelic frequencies on chromosome 7H in true pools of 15M reads resulting from the sampling of DH lines in a non-uniform (left) or uniform (right) fashion. The x-axis shows the position (in cM) of marker bins along the chromosome and the y-axis shows the estimated allelic frequency. Each symbol indicates the allelic frequency of a parental allele (black circle: HV779STP07; red circle: Oceanik) averaged over all SNPs located within a 5-cM bin. The dashed blue horizontal lines indicate the critical allelic frequencies above/below which segregation is declared significant. ... 81 Figure 3.8 SD regions of differing extent and magnitude in regenerated plants and embryos. SD was measured on chromosome 1H (bottom panel) and 6H (top) in both embryos (left) and regenerated plants (right). The x-axis shows the position (in cM) of marker bins (embryos) or individual markers (plants) along the
chromosome and the y-axis shows the estimated allelic frequency. In the case of marker bins, each symbol indicates the allelic frequency of a parental allele (black circle: Myriam; red circle: UL163) averaged over all SNPs located within a 5-cM bin. The dashed blue horizontal lines indicate the critical allelic frequencies above/below which segregation is declared significant. ... 84 Figure 4.1 Course of isolated microspore culture leading to the production of segregating populations of either plants (a) or embryos (b) used in this work. ... 96 Figure 4.2 Distribution and overlap of SDRs observed across 12 barley populations derived from androgenesis. The line to the left of the schematized chromosome (grey bar) indicates the genetic position (in cM), coloured bars illustrate the location and span of SD regions while a black oval indicates the position of the marker showing the largest distortion (SDR peak) observed in 12 barley populations. ... 103 Figure 4.3 Distorted chromosomal regions among twelve segregating populations in barley. The x-axis shows the position (in cM) along each chromosome and the y-axis shows the number of populations in which markers at this position exhibited a significant distortion in their segregation. Along each chromosome, the number of populations exhibiting distortion within successive 1.0-cM windows was determined. ... 107 Figure 4.4 Distribution and overlap of SDRs observed in two barley populations (#1114A and #1114B) derived from reciprocal crosses. The line to the left of the schematized chromosomes indicates the genetic position (in cM), coloured bars illustrate the location and span of SD regions while a black oval indicates the position of the marker showing the largest distortion (SDR peak) observed in crosses. ... 108 Supplementary Figure 4.1 Distribution of SNP markers and their allelic frequency across all chromosomes and populations. The genetic map position of the markers on the barley consensus map (x-axis, in cM) and the estimated allelic frequency at each locus (y-axis) are plotted. Each dot shows the actual allelic frequency estimated based on the read counts supporting each allele. The solid curved lines show the regression that best fits the variation in allelic frequency. Red and green are used to identify the parental origin of the alleles while the dotted red lines indicate the values above or below which SD was significant (q-value <0.01). ... 119 Supplementary Figure 4.2 Distribution and overlap of SDRs observed across DH barley populations of this study and previous studies. The line to the left of the schematized chromosome (grey bar) indicates the genetic position (in cM). Coloured bars illustrate the location and span of SDRs while a black oval indicates the position of the marker showing the largest distortion (SDR peak) observed in 12 barley population of this work. Markers exhibiting a significant SD in three previously studied populations (Igri x Franka in Graner et al. 1991; TX9425 x Franklin and Yerong x Franklin in Li et al. 2010) were positioned (when possible) on the same genetic map and show the location of previously reported SDRs (dotted lines). ... 123
Liste des abréviations
ADN Acide DésoxyriboNucléique
AFLP Amplified Fragment Length Polymorphism ARN Acide RiboNucléique
ARNm ARN Messager
ARN-seq Séquençage de l’ARN CA Culture d’Anthère
CMI Culture de Microspores Isolées DArT Diversity arrays technology DEG Differentially expressed gene DH Doubled haploid DS Distorsion de Ségrégation FT Facteur de Transcription GBS Genotyping By Sequencing GO Gene ontology HD Haploïde Doublé
KEGG Kyoto Encyclopedia of Genes and Genomes RDS Région de Distorsion de Ségrégation
RFLP Restriction Fragment Length Polymorphism SD Segregation Distortion
SDR Segregation Distortion Region SNP Single Nucleotide Polymorphism SSR Simple Sequence Repeats Pfam Protein Families
Remerciements
Merci M. Belzile. J’ai aimé vous côtoyer. Au-delà de vos qualités professionnelles, j’estime vos qualités humaines. Votre écoute, votre empathie et votre humour ont été fort agréables. François, merci de m’avoir permis de me réaliser.
Merci Pierre-Étienne Jacques. Merci d’avoir accepté la codirection de ma thèse. Tu as toujours été généreux de tes connaissances, ton expérience et ton temps. Merci Pierre-Étienne.
Merci Blake Meyer. Merci de m’avoir accueilli dans votre laboratoire comme l’un de vos étudiants. Avec moi, vous avez été généreux de vos connaissances, votre expérience et votre temps. Merci Blake.
Merci à Suzanne Marchand et Patricio Esteves. Votre assistance a été précieuse et inestimable pour produire mon matériel biologique. Votre rigueur, vos efforts et votre dévouement m’ont permis d’avoir des données de qualité. Cela a été agréable de collaborer avec vous! Merci Suzanne et Patricio!
Merci à Martine Jean. Merci de m’avoir encadré et formé à la biologie moléculaire. J’ai aimé nos discussions impromptues tenues aussi bien dans les corridors que dans nos bureaux. Merci Martine.
Merci à mes parents; Gaétan et Angèle. Votre accompagnement moral, émotionnel et financier a été source de réconfort lors des moments difficiles. J’ai souvenir du jour où je vous ai annoncé que j’avais quitté mon emploi et que je retournais à l’université. J’annonçais que mon cursus débutait par le rattrapage de cours du niveau de cinquièmes secondaires. Vous avez répondu : fonce, tu es capable! Merci pour votre consentement moral. Merci papa et maman!
Merci à Sophie Chabot. Te rencontrer a été l’électrochoc et la source d’inspiration nécessaire à mon retour aux études. Ton parcours de vie m’a inspiré dans la poursuite de mes rêves. Tu as positivement influencé mes choix de vie. Je m’en souviendrai. Merci Sophie!
Avant-propos
Cinq chapitres composent ma thèse. Le chapitre I est une revue bibliographique qui synthétise les connaissances actuelles sur l’androgenèse, les connaissances acquises, les inconnus, la pertinence de l’orge comme espèce modèle et les approches technologiques pour étudier le transcriptome et le génome. Le chapitre V est une conclusion générale. Les références bibliographiques utilisées pour l’introduction et la conclusion sont détaillées à la fin de la thèse.
La référence du chapitre II est : Bélanger S., Marchand S., Jacques P. É., Meyers B., Belzile F. (2018). Differential expression profiling of microspores during the early stages of isolated microspore culture using the responsive barley cultivar Gobernadora. G3: Genes, Genomes, Genetics, g3-200208.
SB et FB ont développé l’idée du projet. SM a produit les échantillons cellulaires. SB a fait la biologie moléculaire et les analyses bio-informatiques. SB BM, PEJ et FB ont participé à l’interprétation des résultats. SB et FB ont rédigé le manuscrit. Tous les auteurs ont fait une révision critique du manuscrit.
La référence du chapitre III est : Bélanger S., Esteves P., Clermont I., Jean M., Belzile F. (2016a). Genotyping-by-sequencing on pooled samples and its use in measuring segregation bias during the course of androgenesis in barley. The Plant Genome, 9(1).
SB et FB ont développé l’idée du projet. PE et IC ont produit le matériel biologique. SB a fait la biologie moléculaire, imaginé la méthodologie analytique et réalisé les analyses bio-informatiques. SB, MJ et FB ont participé à l’interprétation des résultats. SB et FB ont rédigé le manuscrit. Tous les auteurs ont fait une révision critique du manuscrit.
La référence du chapitre IV est : Bélanger S., Clermont I., Esteves P., Belzile F. (2016b). Extent and overlap of segregation distortion regions in 12 barley crosses determined via a Pool-GBS approach. Theoretical and Applied Genetics, 129(7), 1393-1404.
SB et FB ont développé l’idée du projet. IC et PE ont produit le matériel biologique. SB a fait la biologie moléculaire et réalisé les analyses bio-informatiques. SB et FB ont participé à l’interprétation des résultats. SB et FB ont rédigé le manuscrit. Tous les auteurs ont fait une révision critique du manuscrit.
Introduction générale
L’ensemble de processus biologiques par lequel une espèce se perpétue se nomme la reproduction. Deux formes de reproduction sont observées chez les eucaryotes, soit la reproduction asexuée et sexuée. Alors que la reproduction asexuée donne essentiellement un clone de la plante-mère, la reproduction sexuée brasse le bagage génétique permettant d’augmenter la diversité de l’information génétique. À titre individuel, les membres d’une même espèce ont le même nombre de gènes. Cependant, les versions de ces gènes (les allèles) diffèrent. La reproduction sexuée brasse les allèles entre les individus de sexe opposé et combine les allèles donnant une gamme d’assortiments génique distincts. Au plan évolutif, cela permet à l’espèce de s’adapter à un environnement changeant (p. ex. l’évolution d’une nouvelle souche d’un agent pathogène) et assurer sa pérennité dans le temps. La reproduction sexuée est évolutivement avantageuse pour l’espèce (par l’association des meilleurs allèles), mais peut être un cul-de-sac évolutif pour l’individu par l’association d’allèles défavorables.
Conceptuellement, la reproduction sexuée débute par la formation d’un zygote unicellulaire dans l’ovaire lorsque les gamètes mâle (le noyau spermatique du grain de pollen) et femelle (l’ovule) fusionnent. Ce zygote évolue dans un programme de division et différenciation cellulaire pour former un embryon. Lorsque morphologiquement complet, l’embryon comprend les primordiums méristématiques desquels les organes aériens (tige et feuille) et souterrain (racine) de la plante se développeront après la germination (Soriano et al. 2013). La voie de développement menant à la formation d’un embryon se nomme l’embryogenèse.
Une fascinante caractéristique du règne végétal est la totipotence cellulaire qui permet aux plantes de former des embryons à partir de cellules autres que le zygote. Selon l’origine de l’explant, deux types d’embryogenèse se pratiquent en culture in vitro chez les plantes (Soriano et al. 2013). L’une est l'embryogenèse somatique. Cette embryogenèse est induite à partir de tissus végétatifs et génère
essentiellement des clones de la plante-mère (Soriano et al. 2013). L’autre est l’embryogenèse gamétique induite chez les cellules gamétiques mâle ou femelle (Bohanec, 2009; Reynolds, 1997; Segu-Simarro, 2010; Soriano et al. 2013). Cette voie embryogénique a pour avantage d’être dérivée de cellules post-méiotiques, c’est-à-dire des cellules représentant la descendance haploïde de la plante-mère. En culture in vitro, ces cellules doublent (doublement spontané ou induit chimiquement) leurs chromosomes et empruntent la voie de l’embryogenèse. À terme, ces embryons régénéreront des plantes dites haploïdes doublées (HD). Chez les espèces autogames, cela signifie que les méioses et les fécondations subséquentes produiront des graines ayant un patrimoine génétique en tout point identique à celui de la plante-mère. Ces plantes sont dites homozygotes ou fixées. En théorie, nous pouvons produire les HD à partir des gamétophytes mâles ou femelles. En pratique, l’embryogenèse gamétique se pratique principalement à partir du gamétophyte mâle immature (nommé la microspore). Des protocoles de production existent pour bon nombre d’espèces. Néanmoins, la performance de l’embryogenèse gamétique est fortement dépendante de l’espèce et du génotype pour une espèce donnée (Soriano et al. 2013). Soriano et al. (2013) rapportent deux principaux goulots d’étranglement à surmonter (i) le manque ou l’inefficacité de l’induction de l’embryogenèse et (ii) la mauvaise régénération des embryons en plantes. À ces goulots d’étranglement, les allèles aux gènes améliorant le développement de la microspore en androgenèse sont sélectionnés. Cette sélection se manifeste par la distorsion de la ségrégation allélique dans la descendance. C’est-à-dire que les allèles sélectionnés sont transmis à la génération suivante plus souvent qu’attendu. Dans une perspective d’accroître la performance de l’embryogenèse gamétique, il semble important de mieux comprendre (i) quels sont les gènes permettant à la microspore de quitter la voie de développement du grain de pollen pour s’engager dans celle l’embryogenèse et (ii) quand s’exerce la sélection allélique dans le processus et où cette sélection survient chez le génome de l’organisme. C’est dans ce contexte que s’inscrit ma thèse de doctorat.
Chapitre I
1.1 Reproduction, évolution et agriculture
Selon la vision évolutionniste de Darwin, la reproduction sexuée permet l’évolution de l’information génétique sous la pression de sélection exercée par le milieu naturel. À terme, une grande diversité génétique (intraspécifique et interspécifique) s’accumule au fil des générations dans un milieu naturel qui est changeant, hétérogène et instable. En revanche, l’homme pratique l’agriculture dans un milieu physiquement homogène où les conditions y sont maximalement contrôlées et uniformisées. Les paysages naturel et agricole divergent donc fortement. L’assortiment génique résultant de l’évolution en milieu naturel ne répond donc pas aux conditions de culture du milieu agricole. Or, l’assortiment génique « optimal » dans le contexte agricole ne peut pas se trouver directement dans la nature. Un effort de sélection et d’amélioration génétique est nécessaire pour développer des variétés répondant aux besoins agricoles. L’idée générale d’un programme d’amélioration génétique se résume, grossièrement, en quelques étapes (i) sélectionner deux individus ayant les caractères agronomiques recherchés, mais complémentaires (ii) croiser ces deux individus (iii) fixer génétiquement les allèles chez la descendance en ségrégation et (iv) sélectionner les individus montrant la combinaison de caractères recherchés dans cette descendance.
En soi, les programmes d’amélioration génétique reposent sur les propriétés de la reproduction sexuée et donc implicitement sur la méiose. La méiose est un processus de double division cellulaire qui s’organise dans une cellule diploïde (2n) pour former les gamètes haploïdes (1n). Contrairement à la mitose, la méiose produit des cellules différentes et uniques bien qu’elles descendent de la même cellule. Deux propriétés de la méiose sont utiles pour l’amélioration, soit (i) la ségrégation indépendante des chromosomes (brassage interchromosomique) et (ii) la recombinaison d’une paire de chromosomes (brassage intrachromosomique). Les propriétés de la méiose permettent de regrouper les allèles recherchés chez deux individus distincts au sein d’un seul et même individu, qu’ils proviennent du même chromosome (brassage intrachromosomique) ou de deux chromosomes distincts (brassage interchromosomique).
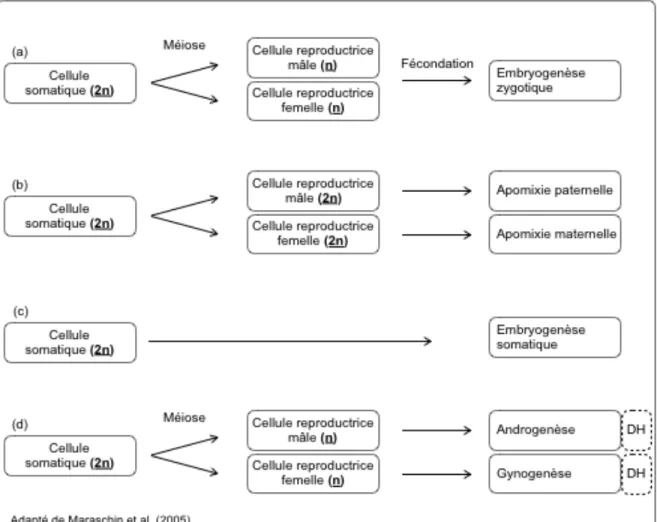
Le processus naturel veut que le gamète mâle féconde le gamète femelle pour former l’embryon hybride d’une graine. Cet hybride aura à la fois le bagage génétique paternel et maternel. Les améliorateurs procéderont à une succession d’autofécondations pour fixer le bagage génétique, évaluer la descendance en champs et sélectionner les plantes ayant les caractères génétiques recherchés. Le principal problème est le temps requis (6-8 ans) pour fixer le bagage génétique et débuter la sélection.
1.2 L’embryogenèse
Au terme de la méiose, les systèmes reproducteurs mâles et femelles produisent respectivement le grain de pollen et l’ovule. En reproduction sexuée (Figure 1.1a), un zygote unicellulaire se forme dans l’ovaire par la fusion des gamètes mâles et femelles. Ce zygote évolue dans un programme de division cellulaire et d’histodifférenciation pour former un embryon. Lorsque morphologiquement complet, l’embryon comprend les primordiums méristématiques des organes aériens (tige et cotylédon/feuille) et souterrain (racine) pouvant se développer après la germination (Soriano et al. 2013). Cette voie de développement menant à la formation d’un embryon se nomme l’embryogenèse et semble avoir évolué pour devenir la stratégie la plus efficace de reproduction chez les organismes multicellulaires supérieurs incluant le règne des plantes (Maraschin et al. 2005). Une caractéristique fascinante du règne végétal est la totipotence cellulaire végétale qui permet aux plantes de former des embryons à partir de cellules autres que le zygote (Soriano et al. 2013). Ce phénomène peut se manifester in vivo dans un cadre normal de développement ou encore être induit en culture in vitro. La figure 1.1 donne un aperçu des types cellulaires à partir desquels un embryon peut se développer. In vivo, l’apomixie maternelle (Figure 1.1b) réfère à la formation asexuée d’une graine à partir des tissus maternels d’un ovule sans qu’il y ait eu de méiose ou de fécondation (Koltunow, 1993; Maraschin et al. 2005). L’apomixie paternelle (Figure 1.1b) réfère à la formation d’un embryon à partir d’un grain de pollen non-réduit (Maraschin et al. 2005; Pichot et al. 2001). In vitro, deux
principaux types d’embryogenèse s’observent selon l’origine de l’explant. L’une d’elles est l’embryogenèse somatique (Figure 1.1c) induite chez des tissus végétatifs et qui génère des plantes de ploïdie et composition génétique identique à la plante-mère (Gaj, 2001; Gaj, 2004; George et al. 2008; Soriano et al. 2013; Zimmerman, 1993). L’autre se nomme « embryogenèse gamétique » (Figure 1.1d). Cette embryogenèse est induite chez les cellules gamétiques mâle ou femelle (Bohanec, 2009; Reynolds, 1997; Seguí-Simarro, 2010; Soriano et al. 2013). Le développement d’un embryon à partir d'ovules non fécondés (in vivo ou in vitro) réfère à la gynogenèse (Maraschin et al. 2005) alors que l'androgenèse réfère au développement d'embryons (in vitro) à partir de grains de pollen immatures (Maraschin et al. 2005). Comme l’anthère d’une fleur contient un très grand nombre de gamétophytes mâles alors que l’ovule contient un unique gamétophyte femelle, l'androgenèse est l’approche privilégiée (Soriano et al. 2013). Quoi qu’il en soit, ces deux voies embryogéniques ont pour avantage de dériver de cellules post-méiotiques, c’est-à-dire des cellules représentant une descendance haploïde au sein de laquelle les allèles sont en ségrégation. En culture in vitro, ces cellules doublent leurs chromosomes et empruntent la voie de l’embryogenèse. À terme, les embryons régénéreront des plantes dites haploïdes doublées (HD). Ces plantes sont homozygotes (génétiquement fixées) et peuvent être utilisées directement pour faire les travaux de sélection en champs.
Des chercheurs ont tâché de développer des applications biotechnologiques pour chacune de ces formes alternatives d’embryogenèse. Des recherches ont porté sur l'apomixie comme moyen de propagation clonale des génotypes recherchés, mais sans succès chez la majorité des espèces cultivées (Bicknell et Koltunow, 2004; Maraschin et al. 2005). En revanche, la propagation clonale a été développée avec succès pour l’embryogenèse somatique. À titre d’exemple, les génotypes de pommes de terres exemptes de virus sont clonés par l’approche de la culture de méristème. Cependant, cette approche n’est d’aucun intérêt pour une perspective d’amélioration génétique.
Figure 1.1 Les voies de développements embryogéniques chez les plantes. Cette figure est adaptée de l’article de revue de Maraschin et al. (2005).
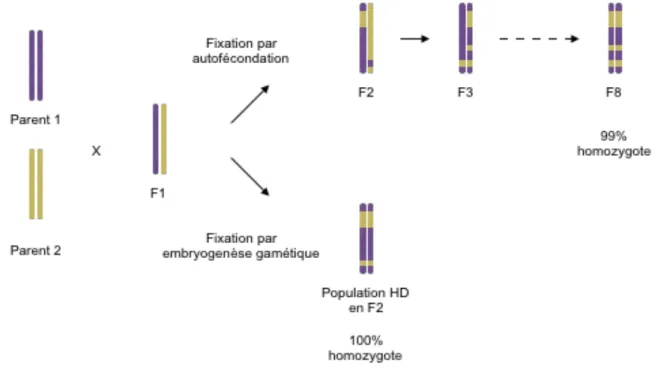
La voie de l’embryogenèse gamétique a été appliquée avec succès dans les programmes d’amélioration génétique. Nous avons précédemment introduit l’importance des gamètes produits par la méiose au sein de l’hybride (la plante F1)
pour recombiner au sein d’un seul et même individu les caractères (les allèles) agronomiques provenant de génotypes distincts. Par l’approche conventionnelle (soit l’embryogenèse zygotique), les génotypes issus de la méiose chez la F1
subissent une succession d’autofécondations pour fixer le bagage génétique et obtenir des lignées homozygotes (Figure 1.2). L’avantage principal est de maximiser le brassage du patrimoine génétique en cumulant les évènements de recombinaison survenant au fil des méioses. Le temps requis est le principal inconvénient puisque de 6 à 8 générations (années) sont nécessaires pour fixer le
bagage génétique et débuter la sélection au champ (Figure 1.2). En revanche, l’embryogenèse gamétique fixe le bagage génétique à 100% en une seule génération et raccourcit significativement le temps requis pour développer de nouvelles variétés (Figure 1.2).
Figure 1.2 Illustration de la fixation génétique par autofécondation ou par embryogenèse gamétique chez un croisement biparental.
1.3 L’androgenèse
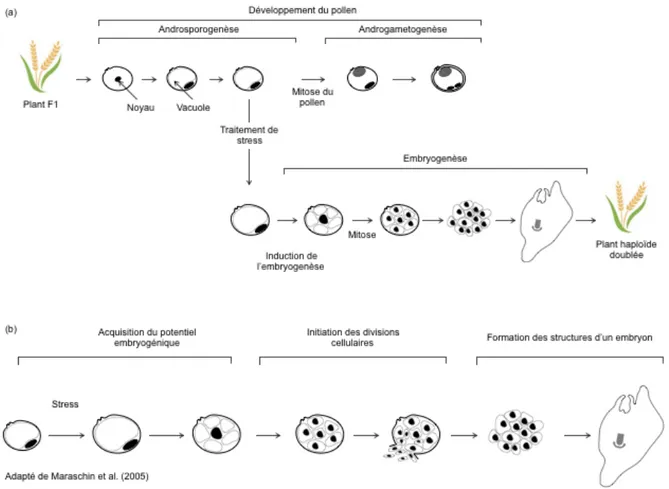
L’androgenèse se pratique soit par la culture d’anthère (CA) ou la culture de microspore isolée (CMI). La CMI montre des avantages par rapport à la CA comme : (i) la paroi de l'anthère ne peut avoir aucun impact négatif sur les microspores (ii) aucun embryon ne peut dériver des tissus somatiques de l’anthère (iii) le développement des embryons augmente (iv) la disponibilité des nutriments nécessaires au développement de la microspore est accrue et (v) le suivi du développement des embryons est facilité (Ferrie et Caswell 2011). Ensemble, ces avantages permettent la production d’un plus grand nombre de plantes vertes par épi par l’approche de la CMI comparativement à celle de la CA (Ferrie et Caswell 2011). Davies (2003) rapporte six facteurs de succès pour la CMI, soit (i) les
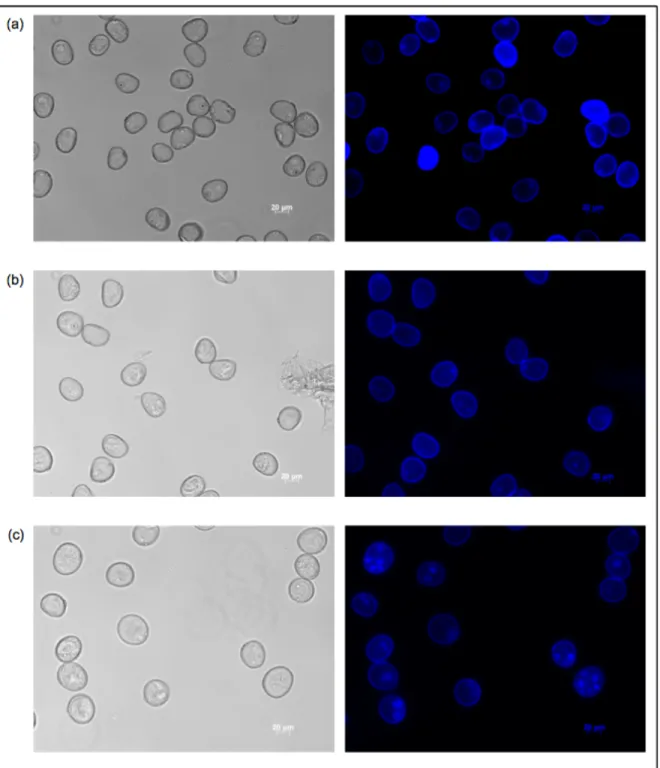
conditions de croissance de la plante-mère (ii) le stade de développement des microspores (iii) le stress appliqué aux microspores (iv) la composition du milieu de culture (v) les conditions de culture et (vi) le génotype. La recherche a permis de développer des protocoles de CMI pour bon nombre d’espèces. À titre d’exemple, Maluszynski et al. (2003) ont publié un manuel décrivant des protocoles pour 33 espèces et répertorié des articles scientifiques rapportant des travaux chez 226 espèces supplémentaires (Ferrie et Caswell 2011). Ces protocoles ont permis l’enregistrement et la commercialisation de nombreuses variétés pour des cultures comme le blé (Triticum aestivum L.), l'orge (Hordeum vulgare L.), le triticale (x Triticosecale Wittm.), le riz (Oryza sativa L.), Brassica spp., l’aubergine (Solanum melongena L.), le poivron (Capsicum annuum L.), l’asperge (Asparagus officinalis L.) et le tabac (Nicotiana tabacum L.) (Thomas et al. 2003; Ferrie et Caswell 2011). Techniquement, la CMI se résume en trois étapes. Premièrement, la récolte de la microspore au stade de développement propice à l’androgenèse. Le stade propice correspond à la cellule issue de la sporogénèse et donc le produit de la méiose. La microspore au stade propice se caractérise par son noyau unique, son ensemble de chromosomes haploïdes, ses chloroplastes fonctionnels et sa plasticité cellulaire (Figure 1.3a). Deuxièmement, un stress est appliqué à la microspore. Seul ou combiné, il est d’usage d’utiliser des stress de température (chaud ou froid), d’osmose et de dénutrition sur une période de temps variable (2, 4, 10 ou 30 jours) (Shariatpanahi et al. 2006). L’objectif du traitement de stress est de provoquer chez la microspore une dédifférenciation cellulaire et rétablir la totipotence cellulaire via une reprogrammation génétique. Troisièmement, la microspore est mise culture in vitro sur un milieu nutritif pour induire l’embryogenèse et la formation de l’embryon (Figure 1.3a). Les embryons sont par la suite transférés sur un autre milieu de culture permettant la régénération (germination) de l’embryon en plante. Pour simplifier l’écriture, notez qu’ultérieurement, le terme androgenèse désignera le système biologique de l’embryogenèse gamétique pratiqué par la méthode de la CMI. Dans le cas contraire, la méthode de production des HD sera spécifiée.
Figure 1.3 Le système biologique de l’androgenèse. Le système biologique de l’androgenèse (a) montre la déviation développementale de la microspore où elle quitte la voie de formation du grain de pollen pour celle de l’embryogenèse qui permet la régénération d’une plante haploïde doublée. Ce système biologique se divise en trois phases développementales (b) correspondant à l’acquisition du potentiel embryogénique, l’initiation des divisions cellulaires et la formation des structures d’un embryon. Cette figure est adaptée de l’article de Maraschin et al. (2006).
1.3.1 Biologie, physiologie et contrôle moléculaire de l’androgenèse
L’androgenèse s’amorce par l’interruption des activités de transcription de l’ADN et traduction des protéines chez la microspore afin d’arrêter la formation du pollen (Maraschin et al. 2005). La microspore se reprogramme génétiquement, rétablit sa totipotence cellulaire et se différencie (Elhiti et al. 2013). Ensuite, la microspore exprime un nouveau programme génétique et s'engage dans une nouvelle voie de développement, en l'occurrence l'embryogenèse (Seifert et al. 2016). Maraschin et al. (2005) ont défini cette transition développementale comme (i) l’acquisition du
potentiel androgénique (ii) l’initiation des divisions cellulaires et (iii) la formation des structures de l’embryon (Figure 1.3b). Alors que l’androgenèse et l’embryogenèse somatique de distinguent par l’origine de l’explant, je note qu’au plan développemental ces deux formes d’embryogenèse sont fort semblables. Par la revue bibliographique du sujet, je note que la majorité des études ont été faites sur l’embryogenèse somatique pour la caractérisation des déterminants génétiques contrôlant les transitions développementales. Ici, je ferai un résumé des connaissances acquises pour la voie de l’androgenèse en priorité et les connaissances en l’embryogenèse somatique seront utilisées comme complément d’information.
1.3.1.1 L’acquisition du potentiel embryogénique
L’acquisition du potentiel embryogénique (Figure 1.3b) réfère à la dédifférenciation cellulaire et au rétablissement de la totipotence cellulaire. Le processus de la dédifférenciation cellulaire permet le retour à l’état élémentaire (semblable à une cellule méristématique) d’une cellule spécialisée (en l’occurrence la microspore). Ce processus de dédifférenciation permet à la cellule de rétablir sa totipotence cellulaire définie comme la compétence de se différencier de nouveau en n’importe quelle cellule spécialisée. Cette étape débute par l’application du stress ou d’une combinaison de stress (Ferrie et Caswell 2011). Les principaux stress utilisés sont la température, l’osmose ou la dénutrition (Figure 1.3b).
Des analyses d’expression génique ont été faites pour définir les événements moléculaires précoces accompagnant l'acquisition du potentiel embryogénique des microspores chez le blé (Sánchez-Díaz et al. 2013; Seifert et al. 2016), l’orge (Vrinten et al. 1999; Muñoz-Amatriaín et al. 2006; Maraschin et al. 2006) et le colza (Joosen et al. 2007; Malik et al. 2007). L’étude transcriptomique la plus exhaustive à ce jour est celle de Seifert et al. (2016). Par le séquençage de l’ARN, ils ont interrogé la microspore de blé avant et après le traitement de stress en plus de la microspore en culture. Leur principal intérêt de recherche était l’expression des gènes lorsque la microspore est mise en culture. Seifert et al. (2016) n’ont donc pas rapporté les résultats quant aux gènes exprimés lors de l’acquisition du
potentiel embryogénique de la microspore et aucune donnée supplémentaire n’est disponible. Pour les travaux menés chez le colza (Joosen et al. 2007; Malik et al. 2007), les microspores semblent avoir été récoltées tardivement alors que la microspore était déjà engagée dans la mitose. L’expression précoce des gènes associés à l’acquisition du potentiel androgénique n’a donc pu être capturée. Marachin et al. (2006) ont réalisé chez l’orge l’étude la mieux conçue. Premièremement, l’expérimentation soustrait l’expression des gènes impliqués dans le développement normal du gamétophyte (la microspore binucléée) de l’expression spécifique à l’acquisition du potentiel androgénique (la microspore après l’application du traitement de stress). Deuxièmement, les microspores échantillonnées après l’application du stress ne sont pas engagées dans le développement sporophytique. Bien que limités à un faible nombre de gènes (une puce ADN de 1421 EST), les travaux de Maraschin et al. (2006) demeurent à mes yeux l’étude la plus aboutie pour décrire l’acquisition du potentiel androgénique. Maraschin et al. (2006) ont montré que sous stress les gènes impliqués dans le développement normal du grain de pollen sont réprimés. Ces gènes étaient impliqués dans la division cellulaire, la synthèse de l’amidon et la production d’énergie. À l’opposé, Maraschin et al. (2006) ont observé l’induction de la transcription de gènes impliqués dans les métabolismes de l’hydrolyse de l’amidon et des sucres, la protéolyse, la réponse au stress, l’inhibition de la mort cellulaire programmée et la signalisation cellulaire. Bien qu’informatifs, les travaux de Maraschin et al. (2006) ne semblent pas avoir clairement abouti à l'identification de gènes régulateurs clés contrôlant l'acquisition du potentiel embryogénique.
Pour que la dédifférenciation cellulaire et le rétablissement de la totipotence cellulaire s’opèrent, intuitivement, la microspore doit modifier son programme d’expression génique. Cette attente suggère l’intervention de mécanismes moléculaires capable de (i) réprimer la transcription génique initialement destinée à former le grain de pollen et (ii) promouvoir l’expression de gènes pour engager le changement de voie développementale vers l’embryogenèse. La modification épigénétique est un puissant mécanisme moléculaire capable de diriger ces changements pour réprimer/induire l’expression des gènes soit par la modification
de la chromatine, des histones ou de l’ADN. Des travaux ont récemment étudié certaines modifications épigénétiques chez la microspore dans la période d’acquisition du potentiel androgénique. Récemment, d’intéressantes découvertes ont été publiées quant aux modifications épigénétiques par les mécanismes de modification de l’ADN et des histones chez l’orge et le colza respectivement. Chez l’orge, El-Tantawy et al. (2014) ont contrasté la méthylation de l’ADN chez la microspore en développement dans les voies développementales du gamétophyte et de l’embryogenèse. Globalement, ils ont observé une hypométhylation de l’ADN chez la microspore en développement dans la voie de l’embryogenèse comparativement à celle du gamétophyte (El-Tantawy et al. 2014). Dans la voie de l’embryogenèse, le niveau de méthylation augmente à partir du stade de structure multicellulaire (El-Tantawy et al. 2014). Chez le colza, les histones ont été étudiées pour les marques de méthylation et d’acétylation connues pour respectivement réprimer et promouvoir la transcription des gènes. Fait intéressant, la reprogrammation des microspores et l’initiation de l’embryogenèse s’accompagnent une diminution du niveau de méthylation de H3K9 (c’est-à-dire la lysine en position 9 (K9) sur l’histone 3 (H3)) (Berenguer et al. 2017). Les travaux de Li et al. (2014) ont montré que l’inhibition de l'activité des histones désacétylases entraîne une hyperacétylation des histones H3 et H4, résultant en une forte augmentation de la proportion de cellules qui passent du pollen à la croissance embryogène. Ces résultats montrent que l’acquisition du potentiel androgénique est corrélée avec des modifications épigénétiques promouvant l’expression génique comme l’hypométhylation de l’ADN, la déméthylation de H3K9 et hyperacétylation des histones H3 et H4. Ensemble, ces résultats suggèrent que les mécanismes épigénétiques reprogramment la microspore durant la phase d’acquisition du potentiel embryogénique.
1.3.2.2 L’initiation de la division cellulaire et la formation des embryons
Comme le nom l’indique, l’initiation de la division cellulaire (Figure 1.3b) est l’étape ou la microspore opère une succession de divisions mitotiques, et ce à l’intérieur de la paroi cellulaire de la microspore. À terme, la paroi cellulaire de la microspore
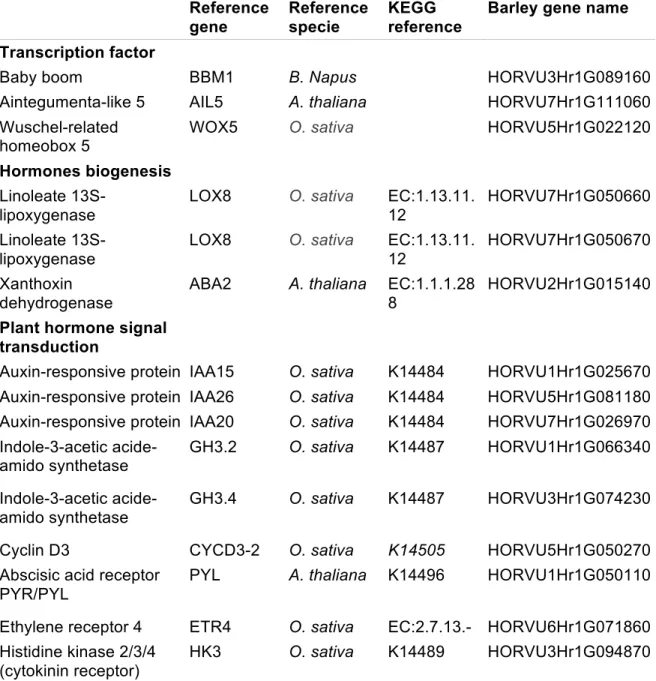
se brise et une masse globulaire nommée « structure multicellulaire » s’observe. Cette structure multicellulaire s’engage dans une différenciation cellulaire programmée pour former des structures embryogéniques. À l’étape de la formation des embryons, une série de divisions cellulaires asymétriques donne lieu à la différenciation d’une stèle vasculaire, un axe apical-basal et d’un épiderme (Maraschin et al. 2005). Sur le plan de la régulation génique, ces deux phases développementales sont difficiles à distinguer comme les gènes sont exprimés aux deux. Cela dit, une catégorie de gènes clés a été identifiée pour initier la division cellulaire (Maraschin et al. 2005; Nowak et Gaj 2016). Ce sont les facteurs de transcription (FT). Mieux décrit en embryogenèse somatique, il semble qu'un ensemble de FT membres des familles MYB, MADS, AP2/ERF, bHLH, C2H2, WRKY, NAC et HB soient impliqués dans l'induction de l’embryogenèse (Nowak et Gaj 2016). Je ne passerai pas en revue tous ces FT. Je prendrai pour exemple les gènes BABY BOOM (BBM), LEAFY COTYLEDON (LEC1, LEC2 et FUS3), WUSCHEL (WUS) et AGAMOUS-like 15 (AGL15).
Le gène BBM est membre de la famille AINTEGUMENTA-LIKE (AIL) et code un TF ayant un domaine protéique AP2/ERF (Nowak et Gaj 2016). BBM est connu pour s’exprimer dans les tissus en division et impliqué dans différents processus de développement incluant l'embryogenèse (Boutilier et al. 2002; Nowak et Gaj 2016). BBM a d’abord été décrit chez les structures multicellulaires du colza (Boutilier et al. 2002) en androgenèse puis ensuite chez A. thaliana en embryogenèse somatique (Tsuwamoto et al. 2010). Un homologue proche de BBM, AINTEGUMENTA-like 5 (AIL5) est exprimé dans les embryons en développement et les embryons (Radoeva and Weijers 2014). Tous deux, ces gènes sont capables d'induire la formation d'embryons lorsqu'il sont surexprimés de manière ectopique (Boutilier et al. 2002; Radoeva et Weijers 2014). Fait intéressant, Boutilier et al. (2002) rapportent que cette embryogenèse spontanée ne fonctionne pas lorsque la feuille-hôte a dépassé un certain stade de développement et de différenciation cellulaire. Ce résultat montre qu'un état cellulaire peu différencié est nécessaire pour que BBM déclenche la voie de développement embryogénique. Cela renforce l’idée qu'une période de
dédifférenciation cellulaire doit précéder pour induire l’embryogenèse dans les systèmes biologiques de l'androgenèse et de l’embryogenèse somatique.
Initialement décrits dans le système de l’embryogenèse zygotique chez A. thaliana, les FT LEC1, LEC2 et FUS3 ont été observés et décrits en embryogenèse somatique à certains stades de développement embryogénique (Harada, 2001). Comme les gènes BBM et AIL5, la surexpression de LEC1 et LEC2 déclenche une embryogenèse somatique des tissus végétatifs (Maraschin et al. 2005), mais pas FUS3. Harada (2001) a proposé que les gènes LEC jouent un rôle régulateur clé pour coordonner les transitions de la phase d'acquisition des compétences embryogéniques à la phase de morphogenèse et de maturation de l'embryogenèse.
Le gène WUS est un FT membre de la famille homeobox normalement exprimé dans le centre organisateur du méristème apical avec pour fonction de maintenir les cellules méristématiques adjacentes dans un état indifférencié (Mayer et Jürgens 1998 ; Mayer et al. 1998; Schoof et al. 2000 ; Radoeva et Weijers 2014). Il semble jouer un rôle essentiel dans le maintien des cellules en état de prolifération et dans la réactivité aux autres signaux de développement (Gallois et al. 2002; Smertenko et Bozhkov 2014; Nowak et Gaj 2016) de même que dans la transition végétative-embryonnaire en embryogenèse somatique chez A. thaliana (Zuo et al. 2002; Smertenko et Bozhkov 2014). Chez les plantes surexprimant WUS, la formation d'embryons somatiques est induite comme pour BBM, LEC1 et LEC2. Membre de la famille des FT au domaine MADS, AGL15, a été rapporté comme étant transloqué dans le noyau au début des divisions cellulaires au cours de l'embryogenèse zygotique et somatique, de l'apomixie et de l'androgenèse (Perry et al. 1999; Maraschin et al. 2005). Ce FT agit à la fois comme un activateur et un répresseur de la transcription (West et al. 1997; Nowak et Gaj 2016) et semble augmenter le développement de l’embryon en embryogenèse somatique (Nowak et Gaj 2016). Contrairement aux précédents FT, AGL15 ne semble pas induire l’embryogenèse à lui seul. Il est connu pour augmenter la capacité à induire
l’embryogenèse en réponse aux autres déclencheurs et s’exprimer principalement chez l’embryon en développement (Heck et al. 1995; Radoeva et Weijers 2014). Ma revue de littérature relève que l’étude transcriptomique la plus exhaustive portant sur la microspore engagée dans la voie embryogénique est celle faite par Seifert et al. (2016). Parmi les FT décrits en embryogenèse zygotique ou somatique, seuls les homologues des gènes BBM et AIL5 ont été rapportés en androgenèse chez le blé. Il est intéressant de noter que, tous deux, ce sont des FT membres de la famille AP2/ERF. Parmi l’ensemble des études portant sur l’androgenèse, indifféremment des espèces, nous n’avons observé aucune mention de l’expression des gènes AGL15, LEC1, LEC2, FUS3 ou WUS alors qu’ils jouent un rôle majeur en embryogenèse somatique. Ce constat pose une interrogation. Cela est-il dû à une incapacité quelconque de détecter ces FT ou bien n’ont-ils pas de rôle actif dans le système biologique l’androgenèse?
Fait intéressant, l’application ectopique de BBM ou de WUS induit l’embryogenèse somatique sans aucun ajout de régulateur de croissance au milieu de culture chez A. thaliana (Zuo et al. 2002 ; Boutilier et al. 2002 ; Nowak et Gaj 2016). Pourtant, le développement somatique d’un embryon est induit par les régulateurs de croissance chez la majorité des espèces, y compris A. thaliana (Mordhorst et al. 1997; Mordhorst et al. 1998; Boutilier et al. 2002). Boutilier et al. (2002) suggère que BBM pourrait agir en stimulant la production des hormones ou en augmentant la sensibilité des cellules à ces substances. Depuis, ces FT membres de la famille AIL et WUS sont connus pour moduler l’expression de régulateurs de croissance impliqués dans le développement (auxine) de l’embryon et la signalisation (auxine, acide abscissique, acide gibbérellique et acide jasmonique) (Nowak et Gaj 2016). Ce résultat montre un étroit noeud interactif entre ces FT et les métabolismes hormonaux. Nous n’avons trouvé aucune étude transcriptomique donnant une vue d’ensemble de l’expression de ces FT ou des régulateurs de croissance chez une même espèce en androgenèse.
1.3.2 Limites de l’androgenèse
1.3.2.1 La réponse génétique à l’androgenèse observée est variable
L’androgenèse est devenue un outil biotechnologique utilisé chez un grand nombre d’espèces (Ferrie et Caswell 2011). Malgré cela, des espèces d’intérêt agronomique ou scientifique demeurent récalcitrantes à ce processus d’embryogenèse de la microspore (Soriano et al. 2013). Parfois, la microspore répond de manière variable à l’embryogenèse chez différents génotypes au sein d’une espèce pourtant reconnue comme performante en androgenèse (Soriano et al. 2013). La capacité de la microspore à former des embryons serait donc dépendante de l’espèce et du génotype en culture. Donc, les protocoles doivent souvent être développés ou affinés au cas par cas pour surmonter deux limites principales qui sont (i) l’incapacité ou l’inefficacité à induire l’embryogenèse chez les microspores (Soriano et al. 2013) et (ii) la mauvaise régénération des embryons en plantes (Germanà 2006; Soriano et al. 2013). D’autres rapportent en plus un important problème d’albinisme chez les espèces de monocotylédones (Kumari et al. 2009; Torp et Andersen 2009). Dans une perspective d’augmenter la performance de l’androgenèse, accroître notre compréhension des déterminants génétiques dirigeant ces deux transitions développementales semble incontournable. Des travaux ont étudié ces deux goulots d’étranglement. Nous avons une compréhension relativement bonne des gènes clés contrôlant l’initiation de la division cellulaire et son développement ultérieur. En revanche, nous connaissons bien peu les évènements précoces à l’étape de l’acquisition du potentiel embryogénique. Nous savons que des mécanismes de contrôle épigénétique interviennent pour rétablir la totipotence cellulaire des microspores. Par contre, aucune étude n’a identifié de gène(s) clé(s) capable(s) de réguler cette acquisition de potentiel embryogénique. L’identification de tels gènes aurait pour avantage le développement de bons marqueurs moléculaires permettant de déterminer le potentiel androgénique des variétés.
1.3.2.2 La distorsion de ségrégation observé chez les populations d’haploïdes doublées
En principe, un individu hétérozygote produit un nombre égal de gamètes portant l’un et l’autre allèle à un locus donné. Supposons qu’aucune sélection génétique ne s’exerce en androgenèse. Comme les plantes HD dérivent directement des produits méiotiques de la plante F1, la fréquence allélique attendue dans la
population est de 1:1 selon l’attente mendélienne. Dans la pratique, ce ratio n’est parfois pas respecté. Ce phénomène s’appelle la distorsion de ségrégation (DS) et se définit comme une déviation de la fréquence allélique par rapport à l’attente mendélienne (Diouf et Mergeai 2012). Chez les populations HD, cette distorsion est attribuée à la présence (à proximité des marqueurs moléculaires biaisés) d’un gène dont un des deux allèles confère un avantage, lequel entraîne une transmission plus fréquente de ce dernier au sein de la descendance HD (Sayed et al. 2002). Étonnement, aucune étude n’a encore déterminé quand survient la DS au cours de l’androgenèse.
Dans les programmes d’amélioration génétique, la DS est indésirable puisqu’elle modifie la fréquence des allèles en ségrégation et peut réduire les chances d'obtenir l’événement de recombinaison allélique recherché. Certaines études ont rapporté l’observation de la DS, cependant, l’objectif premier de ces populations en ségrégation était la cartographie génétique. Rarement, la DS a directement fait l’objet d’une étude. L’étude la plus exhaustive a été faite par Li et al. (2010) chez l’orge. Li et al. (2010) ont analysé quatre populations HD (dérivées de l’androgenèse ou de la gynogenèse) avec plus de 500 marqueurs par population. Les travaux de Li et al. (2010) contrastent avec les précédentes études réalisées sur une seule population HD et avec moins de 100 marqueurs ADN (Graner et al. 1991; Manninen 2000; Sayed et al. 2002). En somme, les études montrent qu'une proportion importante des marqueurs est sujette à la DS dans les populations HD alors qu’environ 40 % (Graner et al. 1991; Manninen 2000; Sayed et al. 2002) et 25 % (Li et al. 2010; Manninen 2000) des marqueurs sont sous l’effet de la DS à un seuil de signification de p ≤ 0,05 et p ≤ 0,01 respectivement. Ces valeurs sont
supérieures à celles observées chez des populations issues de l’autofécondation (en génération F2) où la proportion de marqueurs biaisés se situe autour de 15 %
(Manninen 2000; Sayed et al. 2002). Graner et al. (1991) ont observé une augmentation du taux de DS lorsqu’un parent de l’hybride répond mieux à la culture in vitro. Cela renforce l’idée que la DS reflète la sélection d’un allèle à un gène favorable à la performance à l’androgenèse. Bien qu’informatif, cette observation ne permet aucunement de déterminer quand la pression sélective s’exerce en androgenèse. Pour déterminer quand survient l’effet de sélection en androgenèse, il semble incontournable de mesurer la fréquence allélique à différents stades de développement cellulaire pendant l'androgenèse.
La DS observée chez les populations HD semble avoir fait l’objet de plus d’études chez l’orge que chez les autres espèces. Une problématique s’observe. Il est difficile d’évaluer le chevauchent des régions de distorsion de ségrégation (RDS) en raison du nombre limité de populations étudiées, de la faible densité de couverture des marqueurs et de la diversité des types de marqueurs moléculaires utilisés. Lorsque nous compilons l’ensemble des études significatives faites chez l’orge, un total de 31 RDS semble avoir été signalé sur un total de six populations en ségrégation chez des orges d’un même type (orges à deux rangs de printemps). L’étude de six populations au total ne représente qu’un très petit échantillon de la ressource génétique de l'orge. Ajoutons que pour ces travaux, les RDS ont été simplement définies comme couvrant une certaine partie d'un chromosome sans préciser l'endroit où la distorsion maximale a été observée. Le simple fait d'observer un chevauchement entre deux RDS ne constitue pas une assise solide pour soutenir que le locus « causal » (celui sur lequel agit une pression sélective pendant l'androgenèse) est le même. À cela s’ajoute le nombre de marqueurs. Par exemple, les travaux de Sayed et al. (2002) et ceux de Li et al. (2010) ont respectivement utilisé 43 et plus de 500 marqueurs. Il n’est pas raisonnable de comparer l’étendue des RDS pour les marqueurs positionnés sur ces cartes génétiques. Enfin, les types de marqueurs utilisés pour réaliser ces études étant très variés (RFLP, SSR, DArTs, SNP); il n’est pas banal de placer