HAL Id: hal-02803176
https://hal.inrae.fr/hal-02803176
Submitted on 5 Jun 2020HAL is a multi-disciplinary open access archive for the deposit and dissemination of sci-entific research documents, whether they are pub-lished or not. The documents may come from teaching and research institutions in France or abroad, or from public or private research centers.
L’archive ouverte pluridisciplinaire HAL, est destinée au dépôt et à la diffusion de documents scientifiques de niveau recherche, publiés ou non, émanant des établissements d’enseignement et de recherche français ou étrangers, des laboratoires publics ou privés.
l’identification de marqueurs génétiques chez les chevaux
d’endurance en France
Fanny Baste
To cite this version:
Fanny Baste. Approche du métier d’Ingénieur de Recherche au travers l’identification de marqueurs génétiques chez les chevaux d’endurance en France. Sciences du Vivant [q-bio]. 2012. �hal-02803176�
APPROCHE DU METIER D’INGENIEUR DE
RECHERCHE AU TRAVERS
L’IDENTIFICATION DE MARQUEURS
GENETIQUES CHEZ LES CHEVAUX
D’ENDURANCE EN FRANCE
REMERCIEMENTS
Je tiens à remercier ma tutrice de stage, Anne Ricard, pour m’avoir permis de le réaliser, pour son aide et ses conseils.
Je remercie aussi ma tutrice ENSAT, Zulma Vitezica.
Je remercie Christèle Robert-Granié pour l’aide qu’elle m’a apporté et ses explications qui m’ont permis d’avancer dans mon étude.
Je remercie tout l’équipe de la SAGA pour leur accueil et leur gentillesse.
SOMMAIRE
Abstract ... 1
Introduction ... 2
I. Le contexte du stage ... 3
A. l’INRA, un centre de recherche dynamique ... 3
1. L’INRA national ... 3
2. Le centre de Toulouse ... 5
3. La Station d’amélioration génétique des animaux ... 5
B. Les Haras nationaux ... 6
II. La recherche de marqueurs génétiques chez les chevaux d’endurance ... 7
A. Présentation de la discipline et du projet ... 7
1. Généralités sur l’endurance ... 7
2. Le projet GenEndurance... 8 3. L’étude ... 9 B. Matériel et méthodes... 10 1. Matériel ... 10 2. Méthodes ... 14 A. Résultats ... 16
1. Explication des seuils utilisés ... 16
2. Annonce des résultats ... 18
3. Mise en relation des SNP détectés entre caractères ... 22
B. Discussion ... 23
C. conclusion ... 24
III. Analyse du stage ... 24
A. L’etude et les enseignements reçus ... 24
B. Son inscription dans mon projet professionnel ... 25
Conclusion ... 26 Bibliographie ... I Table des illustrations ... II Annexes ... III Annexe 1 : Exemple d’analyse de variance pour le calcul de l’indice de performance pour le critère de Taille III Annexe 2 : QQplot pour les critères présentés dans le rapport pour les deux modèles ... III Annexe 3 : Exemple de résultats obtenus pour La longueur corporelle ... VI
1
ABSTRACT
In the equestrian world, various disciplines exist. As a horseman, it is possible to do jumping, dressage, cross-country. These are the famous ones. But other disciplines can be as interesting and exciting as the ones previously quoted. Among them, there is endurance.
Endurance consists in running through a set distance for a couple horseman/horse. The distance depends on the level of the race. The speed can be free or imposed. International contests are organized several times during the year in order to enable different nations to confront their couples.
The main purpose of endurance races is not only to run through the distance as fast as possible but also to control as much as possible the effort produced by the horse so it stays in a really good condition. This one is checked many times before, during and after the race. The difficulty, thus, set in the rider’s acquaintance about his or her horse. Obviously, it is also important to develop the ability of the horse to run through important distances: its endurance.
In order to reach this, it is possible to use selected horse breeds that show abilities for this discipline. A selection program is used by the breeders to improve the ability of their horses but it is possible to know if the horse will have great results only after it is involved in free-speed races (that is to say when it is old enough).
The National Studs and the National Institute of Agronomical Research have thus established a shared research project in order to bring out genetic traits that enable the carriers of those traits to have great or poor results in endurance races. The project has been called GenEndurance. Many horses in many different races have been and still are submitted to diverse inspections such as blood sample, weighing, morphometric measurements, appraisal of gaits… The blood sample is used to genotype the horse. Thus, databases have been created to centralize all this information and are available to the project.
The current report presents the study I realized under the GenEndurance project. Thanks to SAS software and self-made computer programs, databases about horses’ performances in endurance races and morphologic measurements have been analyzed. The aim of my study was to bring out genetic markers that could have a link with the genetic area which is responsible for good or poor performances phenotype.
2
INTRODUCTION
Depuis les débuts de l’agriculture, l’Homme a appliqué une sélection de plus en plus forte sur les animaux qu’il utilise et élève. Avec l’amélioration des pratiques agricoles et techniques de sélection, l’élevage a évolué vers une utilisation d’animaux toujours plus performants pour les critères d’intérêt.
A partir de l’arrivée de la mécanisation dans les campagnes, l’utilisation du cheval dans une optique agricole s’est peu à peu éteinte. Aujourd’hui, il est essentiellement utilisé comme animal de compagnie, de loisir ou comme partenaire dans les compétitions. Cependant, la sélection des animaux les plus performants ne s’est pas arrêtée pour autant, pour cette espèce. Les critères d’intérêt ont simplement dérivé de la robustesse, résistance aux intempéries… à des critères plus fins de vitesse, puissance, endurance...
La Fédération Française d’Equitation supervise chaque année de nombreuses compétitions dans diverses disciplines. Parmi celles-ci, on trouve l’endurance. La FFE la défini comme suit : « L’endurance équestre pratiquée individuellement ou en équipe est caractérisée par des épreuves d’extérieur courues à vitesse imposée ou libre sur un itinéraire balisé avec des examens vétérinaires validant la capacité du poney/cheval à parcourir de longues distances. » (1) Les capacités physiques des chevaux et l’entraînement sont donc des facteurs-clés de réussite dans les compétitions.
Dans une optique de sélection plus poussée pour cette discipline, les Haras Nationaux ont mis en place, en collaboration avec l’INRA (Institut National de la Recherche Agronomique), un projet appelé GenEndurance. Ce projet consiste à génotyper les chevaux qui participent aux compétitions officielles et à enregistrer leurs performances. La partie recherche du projet est dirigée par Anne Ricard, ingénieur de recherche en génétique quantitative à l’INRA. Cette section consiste à mettre en évidence des marqueurs génétiques de performance. Ces marqueurs pourront ensuite être utilisés pour déceler au plus tôt les chevaux ayant des prédispositions génétiques pour de bonnes performances en endurance.
Ce rapport présente, dans un premier temps, les partenaires du projet : l’Institut National de la Recherche Agronomique et plus particulièrement le centre de Toulouse et les Haras Nationaux. Dans un second temps, sont exposées la méthode de recherche de marqueurs et l’analyse des résultats obtenus. Enfin, une analyse du stage et de son inscription dans mon projet professionnel est proposée.
3
I. LE CONTEXTE DU STAGE
A. L’INRA, UN CENTRE DE RECHERCHE DYNAMIQUE
1. L’INRA NATIONAL
L’Institut National de la Recherche Agronomique est un organisme de recherche scientifique publique, placé sous la tutelle du Ministère de l’Enseignement supérieur et de la Recherche et celle du Ministère de l’Agriculture, de l’Agroalimentaire et de la Forêt. Fondé en 1946, il est le premier centre européen de recherche agronomique. Il agit sur trois grands axes : l’alimentation, l’agriculture et l’environnement. Ces axes représentent des enjeux majeurs pour la société actuelle. En effet, cette dernière aspire à avoir accès à une alimentation saine et de qualité, à développer une agriculture plus compétitive et durable et, de plus en plus, à vivre dans un environnement préservé et valorisé.
L’INRA se donne pour missions de produire et diffuser des connaissances scientifiques, de concevoir des innovations et des savoir-faire pour la société, d’éclairer, par son expertise, les décisions des acteurs publics et privés, de développer la culture scientifique et technique, de participer au débat science/société et de former à la recherche et par la recherche. Pour ce faire, des acteurs autres que les chercheurs de l’INRA sont engagés dans la recherche. On trouve donc des acteurs socio-économiques comme des entreprises, des organisations collectives agricoles, des collectivités territoriales ou encore les pouvoirs publics.
Ainsi, l’INRA s’inscrit dans une démarche d’excellence qui le propulse au 2ième rang mondial et au 1ier rang européen pour les publications scientifiques agricoles. Il est engagé dans des partenariats avec d’importants instituts de recherche dans le monde, des universités et l’enseignement agronomique et vétérinaire. De ce fait, il collabore et échange avec la communauté scientifique internationale.
4
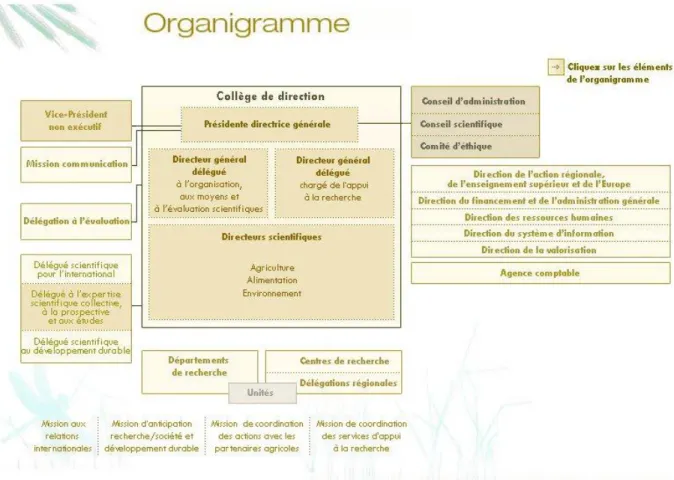
L’institut est très structuré, comme le montre l’organigramme de la Figure 1. Il est organisé en 19 centres, répartis sur tout le territoire national (cf. Figure 2). Chaque centre a ses thématiques de recherche spécifiques. (2)
Figure 1 : Organigramme de l'Institut National de la Recherche Agronomique (3)
5
2. LE CENTRE DE TOULOUSE
La ville de Toulouse a vu s’installer, en 1900, la première plateforme de recherche agronomique dans la région : la Station œnologique créée en collaboration avec le Ministère de l’Agriculture. Devenue la Station régionale d’agronomie et d’œnologie, elle s’implante en 1970 sur le site d’Auzeville et s’appelle désormais le Centre de Recherche INRA de Toulouse. (5) Depuis cette date, il n’a cessé de se développer. Ainsi, le centre de Toulouse s’articule aujourd’hui autour de 15 unités de recherche dont 12 en partenariat avec d’autres établissements d’enseignement supérieur et de recherche, 5 unités expérimentales, 2 unités sous contrat, 3 unités de service, 1 unité d’appui à la recherche et 1 structure transversale.
Les recherches au sein du centre se structurent en 5 thématiques :
- Génome et amélioration des productions : améliorer les animaux d’élevage et la santé des plantes pour répondre à la demande sociétale concernant la quantité et la qualité des produits ;
- Sécurité sanitaire des aliments : développer les connaissances sur la santé des animaux d’élevage jusqu’à la transformation des produits dans le but de mettre sur le marché des aliments sains ;
- Transformation des produits agricoles : utilisation des déchets agricoles pour produire de l’énergie, des produits chimiques, des matériaux innovants plus respectueux de l’environnement ;
- Economie de l’environnement et des marchés : améliorer les connaissances des filières et du marché agricole, assurer une veille sur les sujets sensibles pour créer des outils d’aide à la décision, contribuer aux débats publics et anticiper les demandes sociétales ;
- Environnement, territoire et société : comprendre les systèmes agricoles en place et leurs relations avec l’environnement pour améliorer leur durabilité. (6)
3. LA STATION D’AMELIORATION GENETIQUE DES ANIMAUX
La Station d’Amélioration Génétique des Animaux (SAGA) s’inscrit notamment dans la thématique d’amélioration des productions. Elle contribue à la connaissance du déterminisme génétique des caractères d’intérêt pour la production mais aussi au développement d’aide à la gestion des populations animales. Les espèces étudiées principalement sont les lapins, les petits ruminants tels que les ovins et les caprins, les palmipèdes gras et les équins. Ces derniers sont étudiés par Anne Ricard, ingénieur de recherche à l’INRA pour les Haras Nationaux. (7)
6
B. LES HARAS NATIONAUX
Créés en 1665, les Haras Nationaux ont évolué jusqu’à être, aujourd’hui, les premiers interlocuteurs des propriétaires d’équidés, éleveurs, et professionnels de la filière équine. Ils assurent divers services autant auprès des professionnels de la filière qu’auprès des non-professionnels. Fusionnés depuis le 1ier Février 2010 avec l’Ecole Nationale d’Equitation, ils sont devenus l’Institut Français du Cheval et de l’Equitation (IFCE), l’opérateur public unique pour accompagner la professionnalisation de la filière équine.
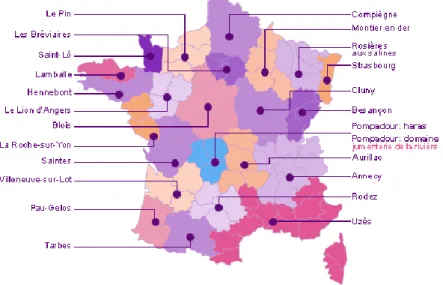
Ils couvrent l’ensemble du territoire français ainsi qu’il est possible de le voir sur la Figure 3.
Figure 3 : Répartition des Haras Nationaux sur le territoire français (8)
Ils ont pour missions, au travers de l’IFCE, de promouvoir l’élevage des équidés et les activités liées au cheval. Ils mettent notamment en place des partenariats avec les organisations socioprofessionnelles, les collectivités locales et les associations dans le but d’assurer le rayonnement de l’équitation. (9)
Grâce à leur association avec l’Ecole Nationale d’Equitation de Saumur, ils mettent à disposition entre autres, les services et savoirs faires suivants :
- La formation, sous forme de stages, aux métiers de l’équitation, de l’élevage et aux métiers connexes ;
- Le sport de haut-niveau pour favoriser l’élévation du niveau des cavaliers et des chevaux ; - L’identification des équidés via un numéro S.I.R.E notamment pour assurer la traçabilité des
7
- La régulation économique et génétique de la filière par la mise en œuvre d’une politique d’orientation de l’élevage et d’utilisation des équidés ;
- La recherche pour améliorer les connaissances générales et spécialisées sur le cheval mais aussi pour favoriser le développement des entreprises équines ;
- La documentation pour favoriser l’accès à la connaissance à travers des médiathèques, les sites internet et les maisons d’édition ;
- La filière internationale pour défendre la filière française à l’étranger. (10)
Plus spécifiquement, les Haras soutiennent et participent, avec l’INRA au projet GenEndurance dans le cadre de la recherche pour l’amélioration des connaissances en endurance et en génétique équine.
II. LA RECHERCHE DE MARQUEURS GENETIQUES CHE Z LES CHEVAUX D’ENDURANCE
A. PRESENTATION DE LA DISCIPLINE ET DU PROJ ET
1. GÉNÉRALITÉS SUR L’EN DURANCE
L’endurance est une discipline de plein-air (cf. Figure 4) où le couple cavalier-cheval parcourt une distance déterminé à un rythme soutenu. Elle a pour particularité de demander une parfaite maîtrise de l’effort fourni par le cheval et donc une très bonne connaissance de sa monture et de ses capacités de la part du cavalier. En effet, le classement repose non seulement sur le temps mis à parcourir la distance demandée mais aussi sur l’état de santé du cheval tout au long de la course. Pour cela, des contrôles vétérinaires obligatoires pour tous les coureurs sont répartis tout le long du parcours et peuvent entraîner la disqualification du couple en cas de doute concernant la bonne santé du cheval. Les distances parcourues sont de 20 km à 160 km pour les plus expérimentés. Il existe deux types d’épreuves. Les épreuves à vitesse libre et celles à vitesse imposée. Pour ces dernières, le classement s’effectue suivant la vitesse mais aussi le rythme cardiaque du cheval après l’effort. Ce sont en général des courses de 20 à 60 km.
Chaque course se déroule de la façon suivante :
- Avant le départ, le cavalier doit récupérer son dossard, la carte du parcours, le « road book » pour son équipe. Il est informé de la situation de l’aire de contrôlé vétérinaire, des points d’eau, des difficultés particulières du parcours et du nombre exact de kilomètres à parcourir.
Figure 4: Photo du test d'effort de Compiègne le
8
En effet, il existe une tolérance de 2 à 15 km en fonction du type d’épreuve. Cette information est très importante pour la gestion de l’effort du cheval mais aussi pour calculer la vitesse moyenne à maintenir pendant la course et la fenêtre horaire ainsi disponible pour terminer dans le temps imparti.
- La deuxième étape est le contrôle vétérinaire au cours duquel des tests d’usage sont fait pour vérifier que le cheval est apte à faire la course. Les tests sont, pour l’essentiel, la mesure de la fréquence cardiaque, la vérification d’absence de blessure, du niveau d’hydratation et des allures.
- La troisième étape est le départ de la course. Suivant le type d’épreuve, le départ se fait en groupe (pour les épreuves à vitesse libre) ou en décalé (pour les épreuves à vitesse imposée notamment). Des échauffements sont bien évidemment effectués pour préparer les athlètes à l’effort.
- La course se déroule sous forme de boucles. Suivant la distance à parcourir et le lieu de la course les boucles peuvent varier de l’une à l’autre. Tous les 5 kms environ, l’équipe suiveuse rejoints le couple cavalier-cheval pour les rafraîchir. A la fin de chaque boucle, un arrêt d’une heure est imposé pendant lequel le chronomètre est arrêté. Un contrôle vétérinaire est effectué 30 minutes après l’arrivée du couple.
- Lorsque la ligne d’arrivée de la dernière boucle est franchi, le temps d’arrivée est relevé et le dernier contrôle vétérinaire effectué 30 minutes après l’heure d’arrivée. (11)
2. LE PROJET GENENDURAN CE
La performance sur de longues distances se traduit donc par une capacité d’effort et une vitesse de récupération importantes. La sélection de chevaux pour l’endurance c’est donc focalisée sur ces points particuliers. Récemment, les avancées et découvertes de la science ont permis le développement de techniques de pointe pour la sélection génétique des animaux. Un projet a donc été mis en place entre l’équipe de Biologie Intégrative et de Génomique Equine de l’INRA, l’unité de Biologie Intégrative des Adaptations à l’Exercice et l’Ecole Vétérinaire d’Alfort. Leur objectif est de déterminer les combinaisons de gènes et les caractéristiques phénotypiques, notamment les caractéristiques biochimique, métabolique, morphologique et des allures, favorables ou défavorables à la performance en course d’endurance. Le projet est financé par les fonds « Encouragement aux Projets Equestres Régionaux Ou Nationaux » (EPERON), de l’Institut Français du Cheval et de l’Equitation (IFCE) et de l’Association nationale française du Cheval Arabe (ACA). L’intérêt direct pour les cavaliers participant au projet est de connaître le poids et la hauteur au garrot de leur cheval, d’obtenir une caractérisation des allures et à terme, un profil génomique de leur cheval. L’intérêt
9
pour la discipline est de mettre en évidence des critères réellement associés à la performance en course pour améliorer les techniques de pointage, avoir un profil génétique et phénotypique plus précis d’un cheval performant et enfin de mettre en place un système de détection d’aptitude génétique à la course d’endurance chez les jeunes chevaux. (12)
3. L’ETUDE
Le principe de la recherche de QTL (Quantitative Trait Locus) est le suivant : - Disposer d’une carte génétique,
- Effectuer le génotypage pour des marqueurs bien répartis sur la carte,
- Réaliser le phénotypage pour les caractères au sein de familles recombinantes,
- Rechercher la liaison génotype au marqueur/phénotype au caractère quantitatif pour localiser la région qui contient le QTL. (13)
Mon travail concerne exclusivement la recherche de liaison génotype au marqueur/phénotype au caractère quantitatif.
Les marqueurs utilisés dans mon étude sont des SNP. Lorsque, sur l’ADN, un segment diffère par une seule paire de base, on dit qu’il y a une variation du type polymorphisme pour un nucléotide. On appelle ces différences des SNP (Single Nucleotide Polymorphism). Ils sont stables, constants et répartis uniformément sur l’ensemble du génome. Ces caractéristiques en font des marqueurs génétiques fiables et très utilisés. De nombreuses méthodes sont aujourd’hui disponibles pour détecter ces différences d’un nucléotide. Celle utilisée pour le projet GenEndurance est la puce à ADN. De 40 000 à 80 000 séquences d’ADN peuvent être fixées sur une puce. Ce sont des méthodes d’analyse de l’ADN à haute densité. Elles permettent non seulement de détecter des SNP mais aussi de comparer des génomes, d’identifier des gènes et de caractériser un génome. (14) Leur utilisation est très répandue en génétique animale notamment.
Les phénotypes au caractère quantitatif étudiés sont les mesures morphologiques et les performances des chevaux en course d’endurance. Les performances semblent avoir une héritabilité relativement élevée. En effet, ces héritabilités sont valables pour la mesure faite dans une épreuve et non pas pour la synthèse des performances annuelles ou pour la carrière entière d’un cheval. De plus, pour comparaison, en Concours de Saut d’Obstacle, pour le même genre de critère l’héritabilité n’est que de 0,16. Concernant la vitesse et le classement des chevaux, l’héritabilité s’élève respectivement à 0,26 et 0,24. Alors que, pour la distance, elle n’est que de 0,06 ce qui n’est pas aussi élevé mais intéressant. Un cheval peut donc avoir avec seulement 3 courses, une précision pour
10
l’estimation de sa valeur génétique de 0,41, ce qui est important. Concernant la valeur d’héritabilité des critères morphologiques considérée pour l’étude, elle a été fixée à 0,25. En effet, je n’ai pas de référence précise sur les mesures telles qu’elles ont été réalisées et sur la population de chevaux d’endurance. De plus l’effectif, avec une structure familiale pauvre (peu de familles avec des demi-frères), ne permet pas d’estimer ces héritabilités sur le fichier étudié. Une valeur moyenne a donc été choisie.
Le but de l’étude est donc de mettre en évidence des marqueurs de performance afin de pouvoir sélectionner jeunes, des chevaux montrant des capacités « génétiques » à l’endurance. L’enjeu est important puisque, les jeunes chevaux ne peuvent participer qu’à des courses à vitesse imposée ce qui ne permet pas de vraiment sélectionner les chevaux les plus rapides. Les chevaux présentent donc des performances exploitables à partir de 7 ans. Aucun critère de sélection précoce qui soit corrélé génétiquement avec la réussite à l’âge adulte n’existe à ce jour pour l’endurance. Pouvoir sélectionner plus jeune les chevaux performants en endurance présente donc un intérêt majeur pour la discipline.
Les performances étudiées sont la vitesse, la distance et le classement en compétition officielle. Sont aussi étudiés des caractères morphologiques tels que la taille, le périmètre thoracique, la longueur corporelle et le poids. D’autres caractères tels que le pli cutané, la note d’état corporel, la taille au garrot et à la croupe, la longueur, le vide sous sternal, l’omoplate, l’humérus, le radius, le métacarpe, la phalange de l’antérieur, l’angle de l’épaule, la longueur de l’épaule, la longueur de l’humérus, l’angle du genou, le coxae, le sacre, le fémur, le tibia, le métatarse et la phalange du postérieur ont été analysés mais, par souci de taille de rapport, seuls les détails d’un exemple de résultat sont présentés en annexe. L’analyse a cependant été faite pour l’ensemble des résultats.
B. MATERIEL ET METHODES
1. MATÉRIEL
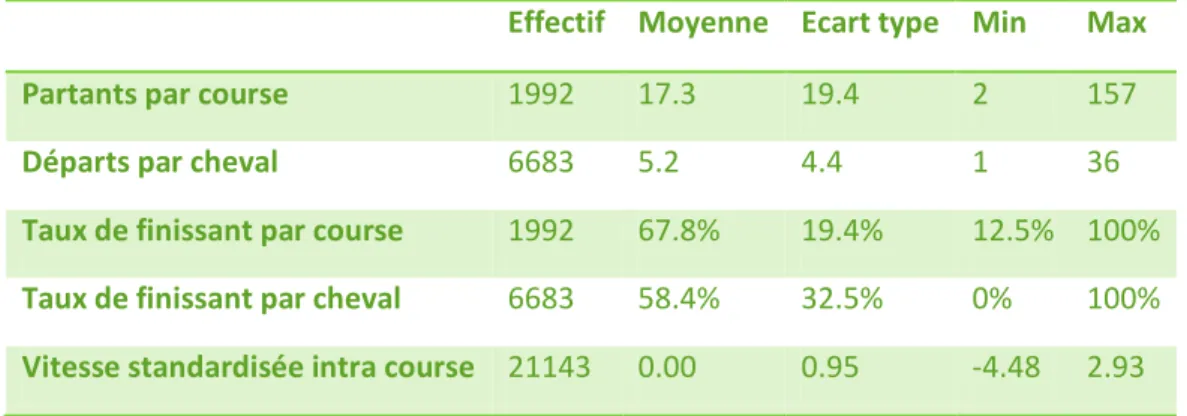
Les Tableaux 1 et 2 ci-dessous permettent de situer l’étude dans l’environnement de la discipline en présentant le nombre de partants et de finissants par course et suivant la distance à parcourir pour les compétitions officielles de 2002 à 2010. On peut remarquer, dans le Tableau 1, que les valeurs données sont très variables : le nombre de partants par course est en moyenne de 17,3 mais l’écart-type est de 19,4. De même pour le nombre de départs par cheval, il est en moyenne de 5,2 mais l’écart-type vaut 4,4. Ceci peut être expliqué par l’âge des chevaux : logiquement, les jeunes chevaux auront un nombre de départs plus faible. Enfin, on peut se demander pourquoi les
11
taux de finissants par course et par cheval sont aussi faibles. Ceci peut être lié au fait qu’en cas de doute concernant l’état de santé du cheval lors d’un contrôle vétérinaire, le couple est disqualifié.
Tableau 1: Données élémentaires des courses d’endurance donnant lieu à l’élaboration d’indices de performance
Effectif Moyenne Ecart type Min Max Partants par course 1992 17.3 19.4 2 157
Départs par cheval 6683 5.2 4.4 1 36
Taux de finissant par course 1992 67.8% 19.4% 12.5% 100%
Taux de finissant par cheval 6683 58.4% 32.5% 0% 100%
Vitesse standardisée intra course 21143 0.00 0.95 -4.48 2.93
Dans le Tableau 2, le nombre de chevaux finissants suivant la distance de la course est très variable : de 30 à 13 028. Ces données sont à mettre en relation avec le nombre d’épreuve organisées par course. On remarque que les épreuves de 90 km sont les plus nombreuses : plus de 1 590 courses ont été organisées entre 2002 et 2010.
Tableau 2: Distribution des distances des courses
Distance (km) Nombre d’épreuve Nombre de chevaux finissants 80 2 30 90 1591 13028 100 21 286 110 9 128 120 126 3242 130 121 2475 140 33 696 150 11 103 160 78 1155
En 2011, 575 chevaux ont été prélevés lors de 9 concours officiels. Ils ont été génotypés. Après contrôle qualité, 574 génotypages se sont révélés exploitables sur les 575. Cependant les performances de ces chevaux ne m’ont pas été communiquées pour les intégrer dans mon étude. Une équipe a été chargée de relever les informations et de faire les mesures nécessaires à l’étude pour chaque compétition.
Les résultats des compétitions officielles de 2002 à 2010 donnent lieu à l’élaboration d’indices de performances qui synthétisent les performances réalisées à partir de 3 critères élémentaires : la vitesse mesurée dans la course quand le cheval finit l’épreuve, une variable nommée classement qui indique si le cheval est finissant ou s’il a été éliminé avant la fin de l’épreuve
12
(0 ou 1) et la distance de l’épreuve (90 à 160 km) comme critère de difficulté. Toutes les courses à vitesse libre de 90 km et plus sont prises en compte.
Parmi les 574 chevaux génotypés en 2011, 467 ont un indice de performance 2002-2010. En effet, certains chevaux n’ont couru qu’en 2011, les indices de cette année-là ne sont pas encore officiellement publiés et d’autres n’ont participé qu’à des courses pour jeunes chevaux qui n’entrent pas dans le calcul des indices de performance (courses à vitesse imposée sur de plus faibles distances).
Les données concernent 34 473 résultats de courses réalisées dans 1992 courses par 6 683 chevaux différents. Seuls 21 143 des 34 473 chevaux partants ont fini la course et permis le recueil de la vitesse moyenne sur la course. Les vitesses, avant d’être traitées, sont standardisées intra course (moyenne 0, écart type 1).
Les indices de performance sont des synthèses des performances réalisées. Les performances sont expliquées par un modèle incluant des effets du milieu (âge, sexe, course sauf pour la distance) et la valeur sportive du cheval (effet aléatoire dont la variance est estimée). L’indice de performance est la valeur sportive estimée par ce modèle. Les indices sont ensuite standardisés pour une présentation plus aisée. Un indice global est proposé comme somme pondérée des 3 indices élémentaires avec une pondération de 30% pour la vitesse, 30% pour le classement et 40% pour la distance.
Les génotypages ont été réalisés grâce à une puce Illumina de 65K à LABOGENA (laboratoire d’analyses génétiques animales situé à l’INRA de Jouy en Josas). Après différents tests de qualité (taux d’attribution d’un génotype > 80% sur l’échantillon, fréquence allélique de l’allèle le moins fréquent > 5%, respect de l’équilibre de Hardy Weinberg avec un test dont la p-value>10-8) 50 234 SNP ont été conservés sur les 65 157 initiaux. Les données de génotypage se présentent sous forme d’un tableau présentant pour chaque cheval génotypé : son numéro afin de le reconnaître et une liste de 50 234 de 0, 1 ou 2. Chaque caractère correspond à un état du SNP : le 0 et le 2 les homozygotes et le 1 l’hétérozygote. Ainsi chaque génome est ainsi décrit précisément aux SNP utilisés.
Les Tableaux 3 et 4 permettent de comparer les performances des chevaux génotypés par rapport aux performances de tous les chevaux participants aux épreuves d’endurance en compétition officielle. On remarque que les chevaux « sélectionnés » pour le génotypage, ont, en moyenne, des performances plus élevées que la moyenne de la population totale. Par exemple, concernant la vitesse, tous les partants des courses ont une vitesse moyenne de 94,5 alors que les chevaux génotypés ont une vitesse moyenne de 104,7. Cependant, en regardant l’écart-type, le minimum et
13
le maximum des performances, on peut voir que la variance est grande. On peut donc considérer que la moyenne de l’échantillon n’est pas trop élevée pour dire qu’il est représentatif de la population totale.
Tableau 3: Données élémentaires sur les indices de performances calculés à partir des performances 2002 à 2010 de tous les partants des courses
Critère Effectif* Moyenne Ecart type Min Max Indice global 6673 99.2 18.9 45 171
Vitesse 6673 94.5 34.6 -11 213
Classement 6673 101.5 32.0 -17 230
Distance (km) 6673 100.1 10.5 80 158
*10 chevaux partants ne figurent pas dans les chevaux indexés car absents du fichier généalogique
Tableau 4: Données élémentaires sur les indices de performances calculés à partir des performances 2002 à 2010 de tous les chevaux génotypés
Effectif Moyenne Ecart type Min Max Indice global 476 107.0 20.1 57 163
Vitesse 476 104.7 36.7 1 198
Classement 476 110.4 34.9 0 209
Distance (km) 476 106.0 12.2 84 152
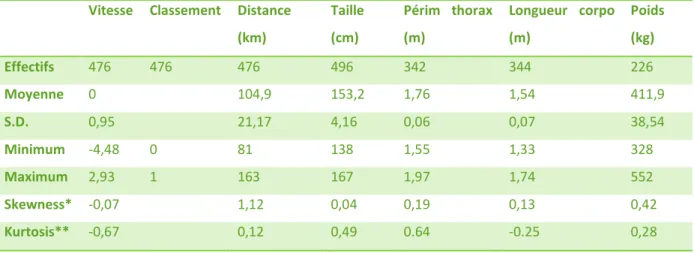
Des statistiques élémentaires pour les principaux critères étudiés sont données dans le Tableau 5. Par exemple, la taille moyenne des chevaux mesurés est de 1,53 m, les chevaux participant aux courses d’endurance sont donc en moyenne relativement petits. Le nombre de chevaux utilisés pour chaque analyse est détaillé dans le Tableau 6.
Tableau 5 : Statistiques élémentaires pour les critères de performances étudiés Vitesse Classement Distance
(km) Taille (cm) Périm thorax (m) Longueur corpo (m) Poids (kg) Effectifs 476 476 476 496 342 344 226 Moyenne 0 104,9 153,2 1,76 1,54 411,9 S.D. 0,95 21,17 4,16 0,06 0,07 38,54 Minimum -4,48 0 81 138 1,55 1,33 328 Maximum 2,93 1 163 167 1,97 1,74 552 Skewness* -0,07 1,12 0,04 0,19 0,13 0,42 Kurtosis** -0,67 0,12 0,49 0.64 -0.25 0,28
14
Tableau 6 : Effectifs de l'analyse des SNP
Vitesse Classement Distance Taille Périm thorax Longueur corpo Poids
Effectifs de l’analyse 467 475 467 328 326 328 122*
* On peut remarquer le faible effectif pour certaines variables, ceci doit être pris en compte lors de l’analyse des résultats.
2. MÉTHODES
Pour chaque performance, un indice (ou pseudo-performance) a été calculé. Celui-ci représente la performance corrigée pour les effets fixes. Comme la performance se répète, la moyenne corrigée pour les effets fixes est pondérée selon le nombre de courses. Concernant les données morphologiques, elles ont été simplement corrigées pour les effets d’environnement.
Les effets fixes considérés sont les suivants :
- Effet de l’âge en différentes classes suivant la performance étudiée ; - Effet de la race en 2 classes (les chevaux avec du sang arabe et les autres) ; - Effet du concours en 10 classes (suivant le lieu et la date) ;
- Effet du niveau d’épreuve en 18 classes (suivant la classification officielle de la FFE).
Les performances corrigées ont été obtenues grâce à des analyses de variances. Les détails des modèles utilisés pour ces calculs sont présentés avec un exemple dans l’Annexe 1.
Les marqueurs de performances significatifs pour chacune d’entre elles sont mis en évidence à l’aide de deux modèles. Le premier calcule le coefficient de régression de l’effet d’un allèle de référence au SNP et l’effet polygénique et ce pour chaque SNP. Le second fait le même calcul mais pour des haplotypes et non plus des allèles. Lorsqu’on s’intéresse à des locus suffisamment proches pour qu’ils aient un taux de recombinaison très faible, on définit ces « zones » du génome comme des haplotypes. Pour chaque SNP, on a 2 allèles possibles. On définit un des allèles comme étant celui de référence. On définit le génotype d’après le nombre d’allèles de référence. Par exemple si les 2 allèles sont A et B, si l’allèle de référence est A, le cheval AA a pour géntoype 2, le AB a pour génotype 1, le BB a pour génotype 0. De même, pour chaque haplotype, un système de notation semblable est utilisé : ce sont les états cachés des haplotypes. Chaque état caché permet de définir précisément le génotype au niveau de l’haplotype.
15
L’écriture mathématique du premier modèle est la suivante :
Où :
- est le vecteur des pseudo-performances des chevaux de l’échantillon (nx1),
- le vecteur des effets fixes du génome au SNP étudié, on a avec l’effet du génotype 0, l’effet du génotype 1 et l’effet du génotype 2. On peut donc avoir - le vecteur des effets aléatoires génétiques polygéniques (mx1). où est la
matrice de parenté généalogique
- le vecteur des résiduelles (nx1).
- une matrice d’incidence qui relie les performances au génotype du cheval
- une matrice d’incidence qui relie la performance (parmi les 1010) au cheval (parmi les 6582). On a pour le cheval identifié dans le fichier des performances et dans le
fichier des généalogies. Tous les autres termes sont nuls. Ainsi les colonnes de correspondant à des chevaux n’ayant pas de génotype et pas de performance ne sont que des colonnes de 0.
Le second modèle la même écriture mathématique : Mais :
- est le vecteur des effets fixes des haplotypes au SNP étudié ; le nombre d’haplotypes étudiés peut être très important (théoriquement avec 2 SNP, on a 4 possibilités, avec 3 SNP on a 8 possibilités etc…, mais en pratique le nombre d’haplotypes observés est inférieur au nombre de combinaisons possibles)
- une matrice d’incidence qui relie les performances aux haplotypes. Chaque performance a deux haplotypes. On a pour le cheval ayant l’haplotype et dans tous les
autres cas.
Le deuxième modèle a pour avantage de prendre en compte l’information provenant du déséquilibre de liaison entre les SNP. En effet, lors de la transmission des caractères, les SNP successifs seront plus à même d’être transmis ensemble et présentent donc une dépendance. Une analyse haplotypique est donc plus proche de la réalité biologique mais elle nécessite des manipulations de données et une estimation des effets peu fréquents dans la population étudiée qui est difficile.
16
Le but est de mettre en évidence les marqueurs les plus significatifs pour chaque performance étudiée. Pour cela, j’ai choisi des seuils permettant d’isoler les marqueurs présentant une p-valeur très faible. Ceci permet de sélectionner ceux qui sont le plus susceptibles d’avoir vraiment un effet sur la performance étudiée. La façon dont est construite l’étude ne permet pas de prendre des seuils « classiques » (1% ou 1‰). En effet, 50 234 tests sont réalisés pour chaque caractère. Avec ces seuils, on obtiendrait par hasard plus de 502 ou 50 tests significatifs ce qui serait un nombre trop important pour être interprétés. Les seuils choisis sont donc beaucoup plus exigeants du type 10-4 voire 10-5. Ces seuils permettent d’obtenir un nombre de tests significatifs beaucoup plus restreint. Ceci est d’autant plus vrai que les 50 234 tests ne sont pas indépendants.
A. RESULTATS
1. EXPLICATION DES SEUILS UTILISÉS
A) LES PERFORMANCES
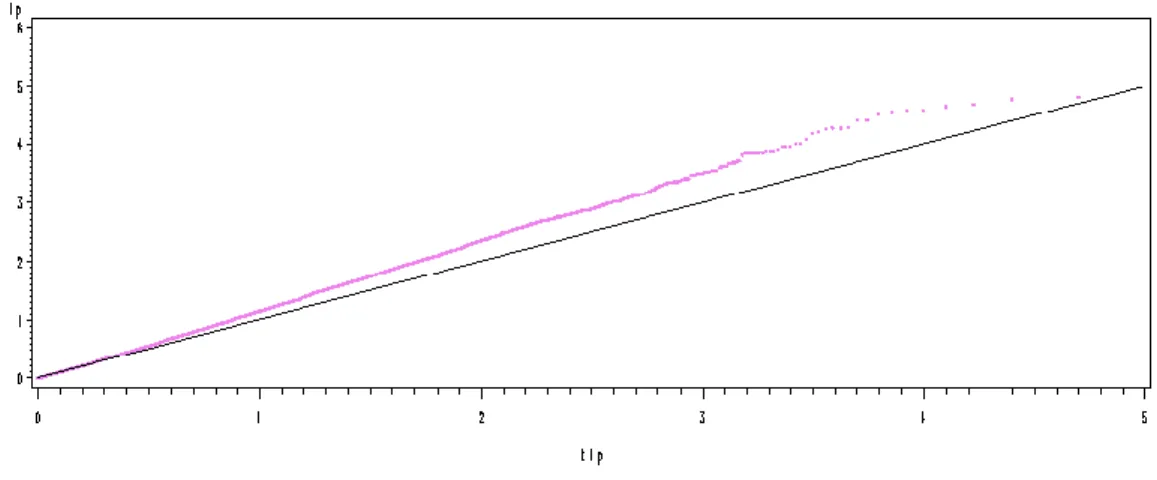
Pour sélectionner le seuil le plus pertinent, des QQplot ont été utilisés. Ils permettent de comparer deux distributions, une observée et une théorique afin d’évaluer si la première correspond à la deuxième. La distribution observée montre les p-valeurs sous l’hypothèse nulle de distribution uniforme (s’il n’y a pas de QTL, on a autant de chance d’être proche du début que de la fin de la distribution du test, ceci signifie qu’en terme de p-valeur, tout est équiprobable). Grace au QQplot, on met en regard de ces p-valeurs une distribution uniforme théorique. On souhaite une distribution uniforme entre 0 et 1 (pour les p-valeurs) ou entre 0 et 4 ou 5 (pour les -log10). Celle qui correspond le mieux à nos attentes est la distribution qui consiste à faire 50 234 points de 1/50234 à 50234/50234.
17
Par exemple, la Figure 5 ci-dessous montre le QQplot obtenu pour le critère de classement analysé par la première méthode. On peut considérer que le seuil lp ≥ 4 est pertinent. Ceci correspond à des p-valeurs ≤ 10-4.
Figure 5: QQplot du critère "classement" pour la première méthode
Pour le critère de distance, on peut observer sur la Figure 6 ci-dessous que la distribution est légèrement bombée pour la distance : le nombre p-valeurs basses, ou de –log10 élevées, est plus important que ce qui est attendu. On peut donc considérer un seuil plus faible. Il possible qu’il y a un problème dans l’analyse de la distance.
Figure 6: QQPlot pour le critère « distance » pour la première méthode
Tous les QQplots réalisés sont donnés en Annexe 2 et les détails d’un des résultats sont donnés en Annexe 3.
18
2. ANNONCE DES RESULTATS
A) LES PERFORMANCES
(1) LA VITESSE
Au seuil p < 10-4, trois SNP potentiellement significatifs ont été mis en évidence avec le modèle mixte sur le chromosome 5. Cependant, deux d’entre eux ont des valeurs de test identiques et sont très proches. On pense pouvoir considérer que ces deux SNP sont en DL (Déséquilibre de Liaison) complet (r²=1) donc que deux QTL ont été détectés. Le pic le plus élevé avec le modèle mixte est atteint autour de 626 cM (p-value 7,06.10-5).
Au seuil p < 10-4, un seul QTL a été détecté avec le modèle uni-QTL par haplotype sur le chromosome 7. Le pic est donc atteint autour de 23 cM (p-value 9,81.10-5). Ceci souligne le fait que les deux modèles ne donnent pas du tout les mêmes résultats. On remarque que pour un seuil de 10-4, le nombre de faux positifs espéré est de soit 5 SNP mis en évidence qui pourraient être des faux positifs.
(2) LE CLASSEMENT
Pour le critère de classement, au seuil p < 10-4, sept SNP ont été mis en évidence avec le modèle mixte sur les chromosomes 3, 7 et 18. Quatre SNP semblent être en DL complet deux à deux, ce qui correspond à cinq QTL détectés. Le pic le plus élevé avec le modèle mixte est atteint autour de 725 cM, soit sur le chromosome 7 (p-value 3,87.10-6).
Au seuil de p < 10-4, un seul QTL a été détecté avec le modèle uni-QTL par haplotype sur le chromosome 6. Le pic est donc atteint autour de 704 cM (p-value 9,06.10-5).
(3) LA DISTANCE
Concernant le critère de distance, au seuil p < 10-4, vingt SNP ont été mis en évidence avec le modèle mixte sur les chromosomes 1, 2, 4, 6, 7, 10, 14, 20, 25 et 31. Trois d’entre eux semblent être en DL complet avec trois autres. Ceci correspond à dix-sept QTL potentiels. Au seuil p < 10-5, un seul QTL potentiel est détecté. Il correspond au pic le plus élevé avec le modèle mixte qui est atteint autour de 45 cM, soit sur le chromosome 1 (p-value 4,01.10-6).
Au seuil p < 10-4, quarante-deux SNP ont été mis en évidence avec le modèle uni-QTL par haplotype sur les chromosomes 3, 6, 9, 11, 13, 14, 18, 20 et 25. Deux SNP semblent être en DL complet avec
19
deux autres ce qui réduit le nombre de QTL détectés à quarante. Au seuil p < 10 -5, aucun SNP n’est mis en évidence. Le pic le plus élevé avec le modèle haplotypique est atteint autour de 377 cM, soit sur le chromosome 3 (p-value 1,20.10-5). Ces nombres semblent relativement élevés lorsqu’on les compare aux nombres obtenus pour les deux autres critères de performance. On peut donc se demander s’il n’y pas eu un problème lors de l’analyse des données pour ce critère.
B) LA MORPHOLOGIE
Les mêmes analyses ont été faites pour les critères morphologiques.
(1) LA TAILLE
Au seuil p < 10-4, dix-huit SNP ont été mis en évidence avec le modèle mixte sur les chromosomes 3, 4, 5, 10, 11, 16 et 20. Il en résulte quinze QTL potentiels après prise en compte des SNP susceptibles d’être en déséquilibre de liaison. Le pic le plus élevé avec le modèle mixte est atteint autour de 1047 cM, soit sur le chromosome 10 (p-value 1,60.10-5).
Au seuil p < 10-4, aucun SNP n’a été mis en évidence. Au seuil p < 2.10-4, cinq SNP ont été mis en évidence avec le modèle uni-QTL par haplotype sur les chromosomes 10, 15 et 18. On a donc potentiellement quatre QTL. Le pic le plus élevé avec le modèle haplotypique est atteint autour de 1341 cM, soit sur le chromosome 15 (p-value 1,08.10-4).
(2) LE PERIMETRE THORACIQUE
Au seuil p < 10-5, trois QTL ont été détectés avec le modèle mixte sur les chromosomes 14, 18 et 24. Le pic le plus élevé avec le modèle mixte est atteint autour de 1301 cM, soit sur le chromosome 14 (p-value 3,24.10-6).
Au seuil p < 10-5, aucun QTL n’a été détecté. Au seuil p < 3.10-5, deux QTL ont été détectés avec le modèle uni-QTL par haplotype sur les chromosomes 3 et 25. Le pic le plus élevé avec le modèle haplotypique est atteint autour de 1986 cM, soit sur le chromosome 25 (p-value 2,36.10-5).
(3) LA LONGUEUR CORPORELLE
Au seuil p < 10-5, trois QTL ont été détectés avec le modèle mixte sur les chromosomes 4, 8 et 11. Le pic le plus élevé avec le modèle mixte est atteint autour de 1110 cM, soit sur le chromosome 11 (p-value 4,25.10-7).
20
Au seuil p < 10-5, aucun QTL n’a été détecté. Il en va de même pour le seuil p < 10-4. Au seuil p < 3,5.10-4, cinq QTL ont été détectés avec le modèle uni-QTL par haplotype sur les chromosomes 4, 13 et 27. Le pic le plus élevé avec le modèle haplotypique est atteint autour de 2104 cM, soit sur le chromosome 27 (p-value 1,73.10-4).
(4) LE POIDS
Au seuil p < 10-5, neuf QTL ont été détectés avec le modèle mixte sur les chromosomes 4, 18, 30 et 31. Le pic le plus élevé avec le modèle mixte est atteint autour de 2212 cM, soit sur le chromosome 30 (p-value 4.10-7).
Au seuil p < 10-5, aucun QTL n’a été détecté. Au seuil p < 7,5.10-5, trois QTL ont été détectés avec le modèle uni-QTL par haplotype sur les chromosomes 4 et 11. Le pic le plus élevé avec le modèle haplotypique est atteint autour de 434 cM, soit sur le chromosome 4 (p-value 1,78.10-5).
C) CONCLUSION
Ainsi une comparaison entre les modèles a pu être faite. Les résultats obtenus par les deux modèles ne présentent pas souvent de QTL communs pour les critères étudiés. Ceci reflète la difficulté de modéliser rigoureusement la transmission des caractères et le lien génotype-phénotype.
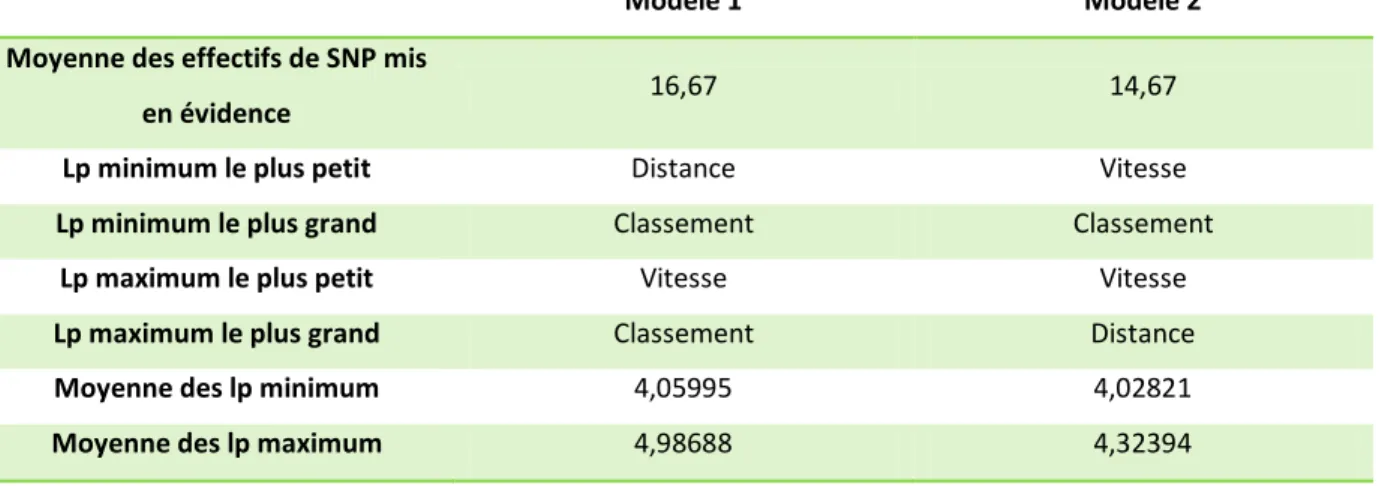
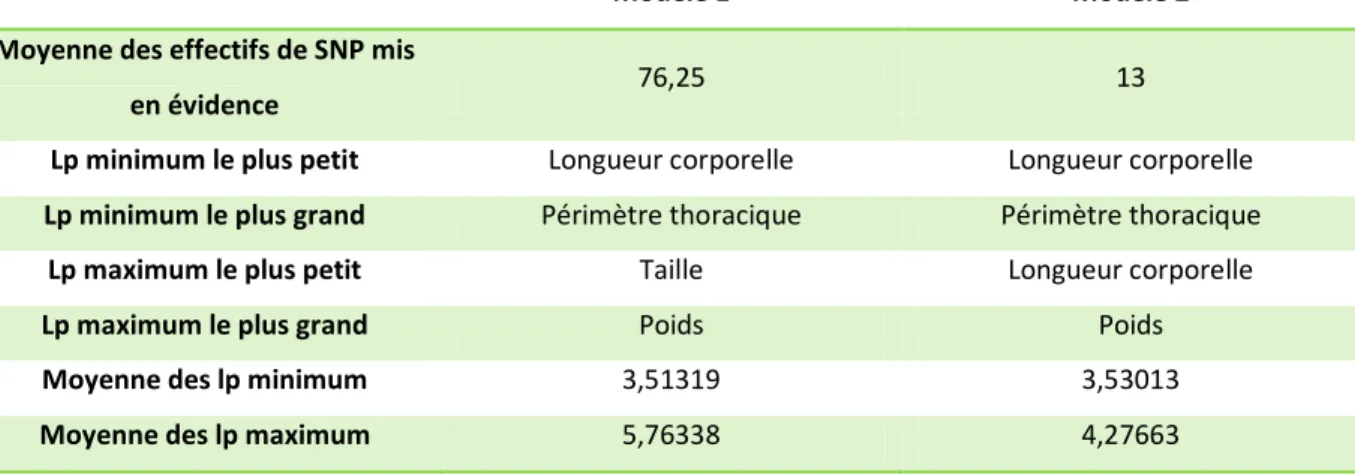
Les Tableaux 7, 8 et 9 ci-dessous permettent de visualiser la différence entre les modèles. Le nombre de SNP détecté est très variable d’un critère à l’autre et d’un modèle à l’autre. Par exemple, 3 SNP sont détectés par le modèle mixte pour la vitesse alors qu’un seul est détecté par le modèle haplotypique. D’un autre côté, 20 SNP sont détectés pour la distance par le modèle mixte et 42 par le modèle haplotypique.
21
Tableau 7 : Comparaison des variables et des modèles
Variables
Modèle 1 mixte Modèle 2 haplotypique
Borne inférieure choisie Nombre de SNP mis en évidence Lp minimum Lp maximum Borne inférieure choisie Nombre de SNP mis en évidence Lp minimum Lp maximum Performances Vitesse 4 3 4,07374 4,15113 4 1 4,00855 4,00855 Classement 4 7 4,09331 5,41279 4 1 4,04282 4,04282 Distance 4 20 4,01281 5,39673 4 42 4,03325 4,92046 Morphologie Taille 3,5 40 3,51331 4,79539 3,5 11 3,53210 3,96738 Périmètre thoracique 3,5 104 3,51850 5,48926 3,5 19 3,52622 4,62764 Longueur corporelle 3,5 38 3,50953 6,37129 3,5 5 3,50169 3,76120 Poids 3,5 123 3,51140 6,39757 3,5 17 3,56051 4,75031
Tableau 8 : Comparaison des modèles pour les critères de performance
Modèle 1 Modèle 2
Moyenne des effectifs de SNP mis
en évidence 16,67 14,67
Lp minimum le plus petit Distance Vitesse
Lp minimum le plus grand Classement Classement
Lp maximum le plus petit Vitesse Vitesse
Lp maximum le plus grand Classement Distance
Moyenne des lp minimum 4,05995 4,02821
22
Tableau 9: Comparaison des modèles pour les critères de morphologie
Modèle 1 Modèle 2
Moyenne des effectifs de SNP mis
en évidence 76,25 13
Lp minimum le plus petit Longueur corporelle Longueur corporelle
Lp minimum le plus grand Périmètre thoracique Périmètre thoracique
Lp maximum le plus petit Taille Longueur corporelle
Lp maximum le plus grand Poids Poids
Moyenne des lp minimum 3,51319 3,53013
Moyenne des lp maximum 5,76338 4,27663
La comparaison intra-caractère entre méthodes montre bien que les deux méthodes donnent des résultats très différents. Cela peut s’expliquer par le fait que les SNP séparent les chevaux en trois groupes : 0, 1 et 2 allèles alors que les états cachés remodèlent entièrement ces groupes. Le problème réside dans la difficulté de comprendre et la confiance qu’il est possible d’accorder à ces résultats.
3. MISE EN RELATION DES SNP DETECTES ENTRE CARACTERES
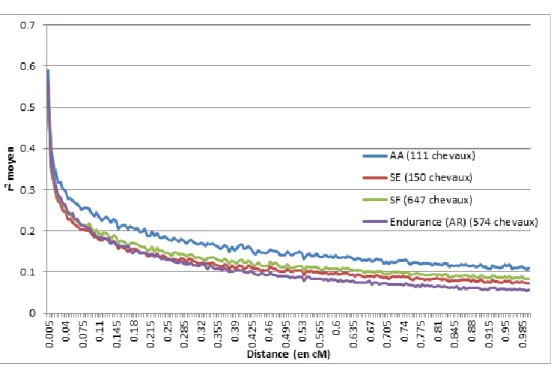
Afin de trouver d’évaluer plus finement les ressemblances/différences entre les modèles, la distance entre les SNP détectés par les deux modèles pour chaque SNP a été calculée. En effet, grâce à la position en cM donnée lors de l’analyse, des distances entre SNP très faibles ont pu être mises en évidence. Par exemple, pour le critère de longueur corporelle, le SNP BIEC2_889778 est situé à 0,43 cM du SNP BIEC2_845678 détecté pour le critère de poids. On peut penser qu’une distance réduite entre les SNP leur permet d’avoir une probabilité plus importante d’être transmis ensemble. On peut confirmer cette hypothèse grâce à l’utilisation du déséquilibre de liaison. En effet, comme le montre la Figure 7 suivant, plus la distance inter-SNP est faible plus le DL est grand. Par conséquent, des SNP proches en distance auront plus de chance d’être transmis ensemble à la génération suivante. Pour l’exemple précédemment cité, le r² moyen est de 0,1 ce qui n’est pas très élevé mais intéressant.
23
Figure 7: Graphique d'évolution du déséquilibre de liaison suivant la distance entre les SNP
Comme signalé dans l’introduction de cette partie, par souci de non-redondance, les autres résultats sont donnés en Annexe 3.
B. DISCUSSION
Les résultats des analyses montrent finalement que peu de cohérence. Ceci ne les discréditent as complètement mais cela prouve la difficulté qui réside dans la modélisation du vivant grâce aux mathématiques.
On peut noter pour chaque critère étudié que ce soit pour les performances ou les critères de morphologie, que le nombre potentiel de faux positifs attendu est relativement élevé par rapport au nombre de SNP mis en évidence par les modèles.
On ne peut donc affirmer que les SNP mis en évidence par ces méthodes sont réellement des QTL. En effet, le fait que l’étude porte sur des caractères quantitatifs est une difficulté non négligeable. Les caractères quantitatifs étant régis par une multitude de faibles effets de gènes.
Du côté de la performance des modèles, on peut mettre en évidence un déséquilibre entre eux. En effet, le modèle mixte paraît être plus performant que le modèle haplotypique puisque des tests plus significatifs ont pu être mis en avant. Ceci est cependant étonnant puisque, comme souligné plus haut, le modèle haplotypique est construit de façon à se rapprocher de la réalité biologique plus que le modèle mixte. De plus, ce peut être dû à un biais de la méthode. On a aussi
24
souligné la difficulté d’estimer les effets peu fréquents pour ce modèle. On peut donc déduire que les estimations réalisées ne correspondent pas aussi bien à la réalité qu’on le pensait.
C. CONCLUSION
En conclusion de l’étude, on peut dire que, parmi les variables citées ici, le poids a les valeur de log10(p-value) les plus élevées pour les deux modèles. Le modèle mixte, malgré la définition plus proche de la réalité du modèle haplotypique, peut être considéré comme plus performant parce qu’il donne en général des valeurs de log10(p-value) plus élevées et détecte un plus grand nombre de SNP. Mais ceci peut être dû à un biais de la méthode.
Au niveau des performances, c’est le classement qui a le log10(p-value) le plus élevé pour le modèle mixte et la distance pour le modèle haplotypique. On peut donc déduire que ces deux critères sont plus liés aux caractéristiques génétiques que la vitesse. Cette conclusion paraît logique puisque la sélection en endurance vise à faire naître des animaux plus « endurants » et non pas plus rapides.
Il sera intéressant de rechercher des gènes majeurs à proximité des SNP sélectionnés. Par manque de temps, malheureusement, toutes les variables disponibles n’ont pas pu être analysées, mais les résultats obtenus et les programmes utilisés pourront servir d’aide à l’analyse des variables manquantes.
III.
ANALYSE DU STAGE
A. L’ETUDE ET LES ENSEIGNEMENTS REÇUS
Ce stage m’a permis d’approcher le quotidien d’un ingénieur de recherche en génétique animale. J’ai ainsi pu participer à des réunions d’équipe présentant le travail de chacun et l’avancée des projets en cours et des réunions de groupe de travail pour réfléchir collectivement aux problèmes rencontrés par chacun au niveau méthodologique. J’ai pu apprécier l’intérêt du travail en groupe et du partage de connaissance entre spécialistes de différents domaines.
Au travers de mes activités, j’ai approfondi mes capacités de raisonnement et d’initiative concernant l’analyse de données. En effet, j’ai, en premier lieu, homogénéisé les fichiers de données afin de les rendre utilisables pour l’analyse et j’ai pu tester mes propres hypothèses d’analyse. L’utilisation du logiciel SAS m’a permis d’appliquer mes connaissances en mathématiques et informatique. Grâce à cette opportunité, j’ai pu apprécier la difficulté d’un travail de recherche et la satisfaction qu’apporte une activité intellectuelle intense.
25
De plus, je travaillais seule sur la partie recherche de marqueurs génétiques du projet Genendurance et je savais que mon travail serait utilisé comme base pour la suite du projet. Ces responsabilités ont été une vraie source d’intérêt et de motivation tout au long de mon stage.
Enfin, j’ai pu appliquer et approfondir mes connaissances en génétique animale et plus particulièrement en génétique équine.
B. SON INSCRIPTION DANS MON PROJET PROFESSIONNEL
Mon intérêt pour ce stage était d’autant plus grand que, étant cavalière, je porte une attention particulière au milieu professionnel du cheval. De plus, j’oriente mon projet professionnel vers la sélection et la génétique dans les productions équines.
Ce stage m’a donc à la fois permis de m’initier au monde de la recherche mais aussi de me faire entrer dans le milieu professionnel de la filière équine en France.
26
CONCLUSION
Le projet GenEndurance s’inscrit dans une logique de sélection des jeunes chevaux pour les courses d’endurance. Grâce au concours de propriétaires faisant participer leurs chevaux aux tests et aux relevés de mesures, des bases de données ont été créées pour satisfaire les besoins du projet.
Le stage réalisé à l’INRA d’Auzeville m’a permis de participer au projet au travers l’étude de la génétique des participants aux courses officielles d’endurance. Cette étude consiste à rechercher des marqueurs génétiques de performances grâce à l’utilisation de données génotypiques et phénotypiques. Les premières sont obtenues par génotypage à l’aide de puce à ADN et les deuxièmes par mesures à l’aide du système Equimétrix®. Les conclusions de mon activité dans le cadre de ce projet sont mitigées. En effet, les modèles mathématiques utilisés sont complexes. De plus, ils ne correspondent pas parfaitement à la réalité puisque la modélisation de l’influence de la génétique sur le phénotype des caractères quantitatifs est très difficile.
Cependant, le travail effectué lors de ce stage servira pour la suite du projet. Les programmes informatiques créés pourront être utilisés pour les futures données et le raisonnement appliqué aux futures analyses.
La bonne réussite du projet permettra de mettre en place un système de sélection des jeunes chevaux porteurs des caractères génétiques influençant la performance en course. Ainsi, une sélection plus forte sera appliquée et une évolution rapide et intéressante devrait avoir lieu concernant l’élevage des chevaux d’endurance.
I
BIBLIOGRAPHIE
1. FFE. Présentation de l'endurance. Fédération Française d'Equitation. [En ligne] http://www.ffe.com.
2. L'INRA en bref. INRA. [En ligne] 3 Novembre 2011. [Citation : 4 Juillet 2012.] http://www.inra.fr/l_institut/l_inra_en_bref.
3. Organigramme. INRA. [En ligne] [Citation : 4 Juillet 2012.] http://www.inra.fr/l_institut/organisation/organigramme. 4. Consulter les offres. INRA. [En ligne] [Citation : 4 Juillet 2012.] http://www.inra.fr/drh/cr2008/bdd/cr1/centres-cr1.html.
5. INRA. Le centre INRA de Toulouse Midi-Pyrénées - 40 ans d'histoire (1970-2010). 2010. 6. —. Centre de recherche - Toulouse Midi-Pyrénées. 2011.
7. —. Intranet INRA. Station d'amélioration génétique des animaux. [En ligne] [Citation : 20 Juillet 2012.] https://germinal.toulouse.inra.fr.
8. La garde des Haras Nationaux. Les chevaux, votre métier ??? [En ligne] [Citation : 4 Juillet 2012.] http://lecheval77.free.fr/photo_11.html.
9. Evolution des Haras Nationaux. Les Haras Nationaux - Institut Français du Cheval et de l'Equitation. [En ligne] 1 Septembre 2011. [Citation : 4 Juillet 2012.]
http://www.haras- nationaux.fr/information/accueil-equipaedia/culture-et-patrimoine/histoire-des-haras/les-haras-nationaux/evolution-des-haras-nationaux.html.
10. Services et savoirs-faires. Institut National du Cheval et de l'Equitation. [En ligne] 2010. [Citation : 5 Juillet 2012.] http://www.ifce.fr/services-et-savoir-faire/?L=nzmpusqrnebvch.
11. Endurance (équitation). Wikipédia. [En ligne] 12 Août 2012. [Citation : 4 Septembre 2012.] http://fr.wikipedia.org/wiki/Endurance_(%C3%A9quitation).
12. GenEndurance. Participez au projet GenEndurance. Projet de Recherche GenEndurance. [En ligne] 18 Mai 2011. [Citation : 30 Mai 2012.] http://genendurance.over-blog.com/.
13. Enseignements de LBOP Gen 3. Laboratoire de Génétique & Evolution des populations végétales -
Université de Lille. [En ligne] 6 Février 2012. [Citation : 26 Juillet 2012.]
http://gepv.univ-lille1.fr/downloads/enseignements/L3-S5/L3-S5-Cuguen-Genet_quanti_3.pdf.
14. Masselot, Monique. SNP. Université Pierre & Marie Curie. [En ligne] [Citation : 9 Août 2012.] http://www.edu.upmc.fr/.
15. Code rural - Article R653-14. Legifrance. [En ligne] 22 Janvier 2010. [Citation : 5 Juillet 2012.] http://www.legifrance.gouv.fr/affichCodeArticle.do;jsessionid=A38A8C64853C4239D42C4016FBA20 075.tpdjo15v_1?idArticle=LEGIARTI000021744331&cidTexte=LEGITEXT000006071367&dateTexte=20 110718.
II
TABLE DES ILLUSTRATIONS
Figure 1 : Organigramme de l'Institut National de la Recherche Agronomique (3) ... 4
Figure 2 : Répartition des centres INRA sur le territoire français (4) ... 4
Figure 3 : Répartition des Haras Nationaux sur le territoire français (8) ... 6
Figure 4: Photo du test d'effort de Compiègne le 16 juin 2012 (12)... 7
Figure 5: QQplot du critère "classement" pour la première méthode ... 17
Figure 6: QQPlot pour le critère « distance » pour la première méthode ... 17
Figure 7: Graphique d'évolution du déséquilibre de liaison suivant la distance entre les SNP... 23
Tableau 1: Données élémentaires des courses d’endurance donnant lieu à l’élaboration d’indices de performance... 11
Tableau 2: Distribution des distances des courses ... 11
Tableau 3: Données élémentaires sur les indices de performances calculés à partir des performances 2002 à 2010 de tous les partants des courses ... 13
Tableau 4: Données élémentaires sur les indices de performances calculés à partir des performances 2002 à 2010 de tous les chevaux génotypés ... 13
Tableau 5 : Statistiques élémentaires pour les critères de performances étudiés ... 13
Tableau 6 : Effectifs de l'analyse des SNP ... 14
Tableau 7 : Comparaison des variables et des modèles ... 21
Tableau 8 : Comparaison des modèles pour les critères de performance ... 21
III
ANNEXES
ANNEXE 1 : EXEMPLE D’ANALYSE DE VARIANCE POUR LE CALCUL DE L’INDICE DE PERFORMANCE POUR LE CRITERE DE TAILLE
Class Levels Values
Sexe 3 F H M
Cnais 11 1997 1998 1999 2000 2001 2002 2003 2004 2005 2006 2007
Crace 2 1 2
Cconc 9 1 2 3 4 5 6 7 8 9
Cepr 18 AUTRE EIO2EY ENA1 ENA1G ENAE ENAEG ENCHAE ENI1E ENI2E ENI3E ENMA1G ENMAE ENMAEG ENSM7A JC4R40 JC5F60 JC6F90 JC6F9L
Number of Observations Read 500 Number of Observations Used 496
Source DF Sum of Squares Mean Square F Value Pr > F Model 38 1435,648044 37,780212 2,43 <.0001
Error 457 7111,690666 15,561686
Corrected Total 495 8547,338710
R-Square Coeff Var Root MSE taille_mes Mean 0,167964 2,574927 3,944830 153,2016
Source DF Type III SS Mean Square F Value Pr > F Sexe 2 634,7712700 317,3856350 20,40 <.0001
cnais 10 304,1754250 30,4175425 1,95 0,0365
crace 1 242,3770477 242,3770477 15,58 <.0001
cconc 8 97,4633215 12,1829152 0,78 0.6180
cepr 17 103,5219673 6,0895275 0,39 0.9870
Le tableau ci-contre montre que les effets du concours et du niveau d’épreuve ne sont pas significatifs. On va donc corriger la variable pour les effets de l’année de naissance, du sexe et de la race.
IV
The GLM Procedure Least Squares Means
Adjustment for Multiple Comparisons: Tukey-Kramer
Least Squares Means for effect cnais Pr > |t| for H0: LSMean(i)=LSMean(j)
Dependent Variable: taille_mes
i/j 1997 1998 1999 2000 2001 2002 2003 2004 2005 2006 2007 1997 1 0,9911 0,4977 0,6462 0,2503 0,3620 0,6346 0,9953 1 0,9967 1998 0,9864 0,4503 0,5822 0,2046 0,3119 0,5480 0,9950 1 0,9983 1999 0,9763 0,9967 0,9064 ,09731 0,9972 1 0,9917 0,8647 2000 1 1 1 1 1 0,8350 0,4181 2001 1 1 1 1 0,9071 0,5387 2002 1 1 1 0,7872 0,3387 2003 1 1 0,8461 0,4082 2004 1 0,8987 0,5206 2005 0,9732 0,7808 2006 1 2007
V
Après suppression des effets du concours et du niveau de l’épreuve, on obtient :
Class Levels Values
Sexe 3 F H M
Cnais 11 1997 1998 1999 2000 2001 2002 2003 2004 2005 2006 2007
Crace 2 1 2
Number of Observations Read 500 Number of Observations Used 496
Source DF Sum of Squares Mean Square F Value Pr > F Model 13 1179,398552 90,722966 5,93 <.0001
Error 482 7367,940158 15,286183
Corrected Total 495 8547,338710
R-Square Coeff Var Root MSE taille_mes Mean 0,137984 2,552032 3,909755 153,2016
Source DF Type III SS Mean Square F Value Pr > F Sexe 2 627,0019441 313,5009721 20,51 <.0001
cnais 10 370,7627453 37,0762745 2,43 0,0080
crace 1 277,7177484 277,7177484 18,17 <.0001
VI
The GLM Procedure Least Squares Means
Adjustment for Multiple Comparisons: Tukey-Kramer
Least Squares Means for effect cnais Pr > |t| for H0: LSMean(i)=LSMean(j)
Dependent Variable: taille_mes
i/j 1 2 3 4 5 6 7 8 9 10 11 1 1 0,9990 0,5600 0,7870 0,3261 0,5984 0,5771 0,9936 1 0,9996 2 0,9970 0,4927 0,7234 0,2706 0,5244 0,5036 0,9849 0,9998 1 3 0,9613 0,9975 0,8856 0,9861 0,9824 1 1 0,8818 4 1 1 1 1 0,9344 0,74150 0,1302 5 0,9999 1 1 0,9953 0,9290 0,2447 6 1 1 0,7688 0,4555 0,0339 7 1 0,9657 0,7776 0,0995 8 0,9570 0,7571 0,0907 9 1 0,6780 10 0,9438 11
VII
Après classification des années de naissance, on obtient :
Class
Levels
Values
Sexe
3
F
H
M
Cnais
3
1999
2000
2005
Crace
2
1
2
Number of Observations Read 500 Number of Observations Used 496
Source DF Sum of Squares Mean Square F Value Pr > F Model 5 1095,617574 219,123515 14,41 <.0001
Error 490 7451,721136 15,207594
Corrected Total 495 8547,338710
R-Square Coeff Var Root MSE taille_mes Mean 0,128182 2,545464 3,899692 153,2016
Source DF Type III SS Mean Square F Value Pr > F Sexe 2 632,4979553 316,2489776 20,80 <0,0001
cnais 2 286,9817676 143,4908838 9,44 <0,0001
crace 1 290,2233297 290,223397 19,08 <0,0001
VIII
The GLM Procedure
Least Squares Means
Adjustment for Multiple Comparisons: Tukey-Kramer
Least Squares Means for effect cnais
Pr > |t| for H0: LSMean(i)=LSMean(j)
Dependent Variable: taille_mes
i/j
1
2
3
1
0,0020
0,9504
2
0,0007
3
La procédure GLM utilisée avec les classes définies ci-dessus permet de calculer l’indice de performance pour chaque cheval. C’est cet indice qui est utilisé dans les modèles mathématiques pour mettre en relation le phénotype (indice de performance) et le génotype (SNP) des chevaux.
III
ANNEXE 2 : QQPLOT POUR LES CRITERES PRESENTES DANS LE RAPPORT POUR LES DEUX MODELES
Critères Modèle Mixte Modèle haplotypique
Vitesse
IV
Distance
Taille
V
Longueur corporelle
Périmètre thoracique
VI
ANNEXE 3 : EXEMPLE DE RESULTATS OBTENUS POUR LA LONGUEUR CORPORELLE
L
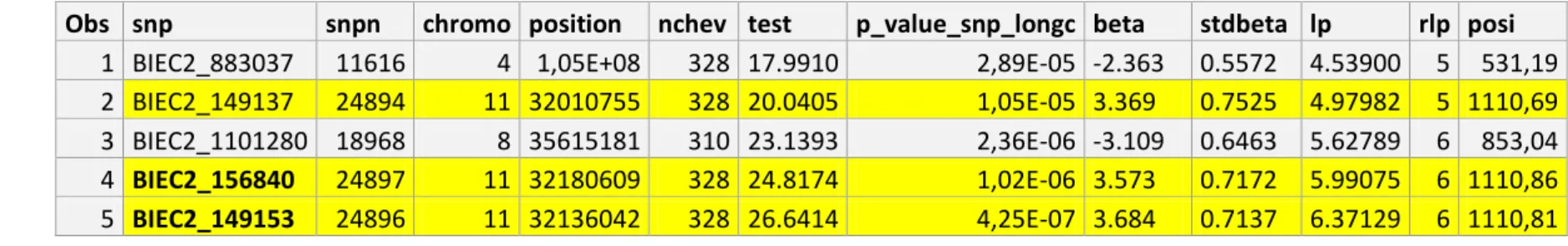
ONGUEUR CORPORELLETableau 10 : SNP détectés par le modèle mixte pour la longueur corporelle
Obs snp snpn chromo position nchev test p_value_snp_longc beta stdbeta lp rlp posi
1 BIEC2_883037 11616 4 1,05E+08 328 17.9910 2,89E-05 -2.363 0.5572 4.53900 5 531,19
2 BIEC2_149137 24894 11 32010755 328 20.0405 1,05E-05 3.369 0.7525 4.97982 5 1110,69
3 BIEC2_1101280 18968 8 35615181 310 23.1393 2,36E-06 -3.109 0.6463 5.62789 6 853,04
4 BIEC2_156840 24897 11 32180609 328 24.8174 1,02E-06 3.573 0.7172 5.99075 6 1110,86
5 BIEC2_149153 24896 11 32136042 328 26.6414 4,25E-07 3.684 0.7137 6.37129 6 1110,81
Les SNP proches
On peut dire que les SNP BIEC2_149137, BIEC2_156840 et BIEC2_149153 (en jaune) sont proches.
Les QTL
Concernant les p-values, au vu de la Figure 8 ci-contre, ont été considérés comme significatifs, les SNP présentant une p-value < 3.10-5. Soit, en se reportant au Tableau 10 ci-dessus, cinq SNP sont considérés comme des QTL*. Leurs p-values variant de 2,89.10-5 à 4,25.10-7.
Les communs avec d’autres critères
BIEC2_156840 et BIEC2_149153 communs avec « Omoplate » (modèle mixte).
*Les faux positifs
Moins de 2 SNP sont susceptibles d’être de faux positifs. 0,00E+00
5,00E-06 1,00E-05 1,50E-05 2,00E-05 2,50E-05 3,00E-05 3,50E-05