Caractérisation des petites protéines de stress/small
heat shock proteins du cyanophage S-ShM2
(HspSP-ShM2) et de son hôte Synechococcus WH7803
(HspS-WH7803)
Mémoire
Maxime Bourrelle-Langlois
Maîtrise en biologie cellulaire et moléculaire
Maître ès science (M. Sc.)
Québec, Canada
Résumé
Les petites protéines de stress/Small heat shock proteins (sHsps) sont des chaperons moléculaires ATP-indépendants ubiquitaires retrouvées chez les procaryotes et eucaryotes. Elles sont dynamiques structurellement et la majorité d’entre elles possèdent la capacité de former de gros complexes oligomériques. Également, elles protègent les cellules du stress protéotoxique causé par divers facteurs de stress abiotiques en prévenant l’agrégation des protéines dénaturées et en promouvant leur repliement par les chaperons moléculaires ATP dépendants tels que Hsp70/DnaK. Récemment, la présence de gènes de sHsp (HspSP-ShM2) chez des virus marins et plus précisément chez des cyanophages infectant le genre Synechococcus sp. et Prochlorococcus sp ont été caractérisés in silico. Au niveau de sa séquence, la sHsp de 18 kDa de Synechococcus sp. montre un domaine alpha crystallin de 92 acides aminés hautement conservé au sein des sHsps, une région C-terminale contenant le motif CAM canonique de type (L-X-I/L/V) et une région N-terminale relativement courte. Nous avons établi grâce à la chromatographie par exclusion stérique (SEC) et le système de Fast Protein Liquid Chromatography (FPLC) sa capacité à former des complexes oligomériques de haut poids moléculaires (600 kDa et 200kDa). De plus, nous avons démontré qu’elle prévient l’agrégation de la citrate synthase, la malate dehydrogenase et la luciférase en condition de stress thermique suggérant qu’elle possède une faible spécificité et un large spectre de protéines clientes/substrats. La prévention complète de l’agrégation a été obtenue à différents ratios (sHsp:substrat) selon le substrat, ce qui indique qu’il y aurait possiblement des interactions différentes et uniques avec chacun d’eux. Nous avons ensuite mis en évidence la formation d’hétéro-oligomères stables et solubles entre HspSP-ShM2 et son substrat dans les mêmes conditions, ce qui est en accord avec les caractéristiques des sHsps en général. Quant à elle, la sHsp de la cyanobactérie Synechococcus WH7803 (HspS-WH7803) possède un poids moléculaire de 15 kDa et montre une capacité à former des tétramères (60 kDa) sur essai de SEC par FPLC en présence de Triton™X-100 pour le maintien de sa solubilité. Contrairement à
HspS-ShM2, HspS-WH7803 ne démontre aucune activité protectrice sur l’agrégation des substrats mentionnés précédement à différents ratios molaires. Finalement, des analyses par SEC/FPLC suggèrent la formation de complexes hétéro-oligomériques entre HspSP-ShM2 et celle de son hôte, HspS-WH7803 de Synechococcus WH7803. Cette interaction entre les sHsps pourrait soit optimiser ou inhiber l’activité de chaperon moléculaire et la réponse au stress de son hôte dans le but de
Abstract
Small heat shock proteins (sHsps) are ubiquitous ATP-independent molecular chaperones found in prokaryotes and eukaryotes. They are structurally dynamic and most of them have the ability to form large oligomeric complexes and to protect cells from proteotoxic stresses by preventing aggregation of non-native proteins and promoting their refolding via ATP-dependent chaperones such as Hsp70/DnaK. Recently, the presence of a sHsp gene (HspSP-ShM2) in marine viruses has been reported using bioinformatics tools. More precisely, the gene has been found in cyanophages infecting cyanobacteria of the genre Synechococcus sp. and Prochlorococcus sp. The Synechococcus phage sHSP has a MW of 18 kDa and shows the highly conserved core alpha crystalline domain of 92 amino acids and relatively short N- and C-terminal arms, the later containing the classical CAM domain (L-X-I/L/V). We established its oligomeric profile using a size exclusion chromatography (SEC) and Fast Protein Liquid Chromatography (FPLC) system and demonstrated its ability to form large oligomeric complexes in native conditions (600 kDa and 200kDa). Furthermore, we report its capacity to prevent the aggregation of citrate synthase, malate dehydrogenase and luciferase suggesting that it has a weak specificity and wide range of protein substrates. The complete prevention of aggregation was achieved at different ratios (sHsp:substrate) depending on the substrate indicating that the sHSP may have different and unique interactions with each of its clients. We also showed the formation of a stable and soluble hetero-oligomeric complex of the phage sHSP and its substrates under heat stress, which is in accordance with the characteristics of sHSP in general. The cyanobacteria Synechococcus WH7803 15 kDa sHSP (HspS-WH7803) shows the ability to form tetramers in the presence of Triton™X-100 for the
maintenance of its solubility using the SEC/FPLC method. For its capability to prevent the aggregation of different substrates, HspS-WH7803 demonstrates no chaperon like activity in all the assays and molar ratios used. Finally, SEC/FPLC results indicate the possible formation of a hetero-oligomeric complex between the sHSP of the phage and the one from its host Synechococcus WH7803 (HspS-WH7803). This interaction could either optimize the chaperone activity and the stress response of its host or inhibit the host sHSP to facilitate the viral life cycle.
Table des matières
Résumé ... iii
Abstract ... v
Table des matières ... vii
Liste des figures ... ix
Liste des abréviations et des sigles ... xi
Remerciements ... xiii
Introduction ... 1
1.1 Les cyanobactéries ... 2
1.1.1 Cycle du carbone et photosynthèse chez les cyanobactéries... 3
1.1.2 Fixation de l’azote chez les cyanobactéries ... 5
1.1.3 Le picophytoplancton Synechococcus sp. ... 7
1.1.3.1 Synechococcus WH7803 ... 8
1.2 Virus marins ... 9
1.2.1 Cyanophages de Synechococcus sp. ... 10
2.1 Protéines de choc thermique ... 13
2.1.1 Petites protéines de stress thermiques (sHSPs) ... 15
2.1.1.1 Séquence et structure des sHSPs ... 15
2.1.1.2 Les sHSPs et la réponse aux stress (HSR) ... 19
2.1.1.3 Survol des autres fonctions cellulaires des sHSPs ... 22
2.1.1.4 sHSPs et la HSR chez les cyanobactéries ... 25
2.1.2 Découverte de séquences de sHSPs chez des cyanophages et cyanobactéries ... 28
Objectifs de travail ... 31
Matériels et méthodes ... 33
3.1 Clonage des gènes des sHSPs dans le vecteur pETHSUK ... 34
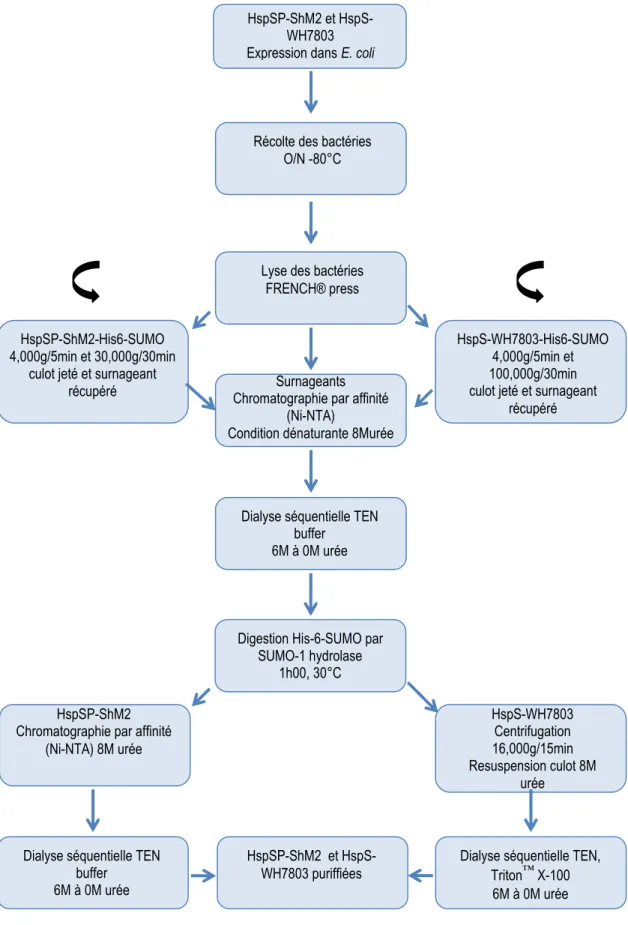
3.2 Expression et purification des sHSPs ... 35
3.3 Essais in vitro de prévention d’agrégation de substrats en condition de stress thermique ... 42
3.4 Analyse de l’état oligomérique par chromatographie par exclusion stérique (SEC) par Fast Protein Liquid Chromatographie (FPLC) et gels SDS-PAGE ... 43
Résultats ... 45
4.2 HspSP-ShM2 forme des homo-oligomères en condition native ... 50
4.3 HspSP-ShM2 est un chaperon efficace formant des hétéro-oligomères (sHSP/substrats) solubles lors de stress thermique ... 52
4.3.1 Essais in vitro d’activité chaperon par prévention d’agrégation de protéines sous stress thermique ... 52
4.3.1.1 HspSP-ShM2 prévient l’agrégation de la citrate synthase ... 52
4.3.1.2 HspSP-ShM2 prévient l’agrégation et maintien la luciférasesoluble ... 54
4.3.1.3 HspSP-ShM2 prévient l’agrégation et maintien la solubilité de la L-malate dehydrogenase ... 56
4.3.2 HspSP-ShM2 forme des complexes hétéro-oligomériques avec la MDH en période stress thermique ... 58
4.4 HspS-WH7803 forme des homo-oligomères en présence de Triton™ X-100 ... 61
4.5 Une interaction entre la petite HSP virale (HspSP-ShM2) et celle de son hôte (HspS-WH7803) dépend de la concentration de Triton™ X-100 ... 62
4.5.1 Absence de complexes hétéro-oligomériques en condition Triton™ X-100 0.23 mM et essais de prévention d’agrégation en présence de TritonTM X-100... 62
4.5.2 Formation d’hétéro-oligomères entre HspS-WH7803 et HspSP-ShM2 en présence réduite (0.138 mM) de Triton™ X-100 ... 68
Discussion... 73
Conclusion ... 85
Liste des figures
Figure 1. Participation du phytoplancton dans le cycle du carbone Figure 2. Photographie d’une cellule de Synechococcus sp WH8102
Figure 3. Photographie par microscopie électronique du phage wV8 démontrant la morphologie typique d’un
myovirus
Figure 4. Représentations 3D des structures cristallisées et superposition des ACD de monomère d’HspB1 et
αB-crystalline humaines
Figure 5. Représentation schématique de l’organisation de la séquence primaire d’une sHSP, mécanismes et
structures de la dimérisation des monomères et représentation 3D de la structure quaternaire de MjHsp16.5, SpHsp16 et TaHsp16.9
Figure 6. Modèle de l’activité de chaperon moléculaire des sHSPs
Figure 7. Modèle 3D du monomère de la sHSP du cyanophage, HspSP-ShM2, aligné à un dimère de la
Hsp16.9 du blé
Figure 8. Carte du vecteur pETHSUK
Figure 9. Séquence peptidique de HspSP-ShM2-His6-SUMO et HspS-WH7803-His6-SUMO résultant du
clonage et expression à partir du vecteur pETHSUK et représentation schématique et comparative de la longueur des domaines de HspSP-ShM2 et HspS-WH7803
Figure 10. Représentation schématique des étapes générales de l’expression et purification in vitro de
HspSP-ShM2 et HspS-WH7803
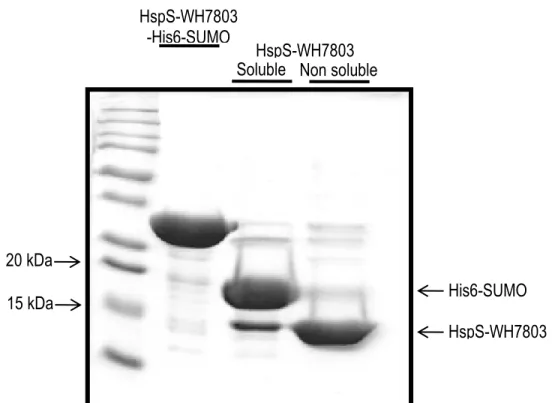
Figure 11. Analyse sur gel SDS-PAGE 12% du contenu protéique de la fraction soluble et non soluble suite à
la digestion de l’étiquette His6-SUMO et centrifugation de HspS-WH7803 (15 kDa)
Figure 12. Vérification de l’absence de l’étiquette His6-SUMO et de la présence de HspSP-ShM2 (18 kDa) de
la fraction purifiée par migration sur gel SDS-PAGE 12% et immunobuvardage
Figure 13. Analyse du poids moléculaire apparent de HspSP-ShM2 (18 kDa) et HspS-WH7803 (15 kDa) sur
gel SDS-PAGE et vérification de la pureté des produits purifiés par chromatographie par affinité
Figure 14. HspSP-ShM2 se présente sous un profil oligomérique polydisperse en condition native avec une
forme majoritaire de 600 kDa et une forme minoritaire de 200 kDa
Figure 15. Prévention de l’agrégation de la citrate synthase (CS) en condition de stress thermique par
HspSP-ShM2 à différents ratios molaires
Figure 16. Prévention de l’agrégation et maintien de la solubilité de la luciférase (LUC) en condition de stress
thermique par HspSP-ShM2 à différents ratios molaires
Figure 17. Prévention de l’agrégation et maintien de la solubilité de la malate déhydrogénase (MDH) en
condition de stress thermique par HspSP-ShM2 à différents ratios molaires
Figure 18. HspSP-ShM2 en condition de stress thermique forme des complexes hétéro-oligomériques avec le
substrat MDH de poids moléculaires supérieurs aux homo-oligomères de HspSP-ShM2 en condition native
Figure 19. HspS-WH7803 se présente sous une forme oligomérique de 60 kDa en condition native
Figure 20. HspS-WH7803 et HspSP-ShM2ne présentent aucun état hétéro-oligomérique lorsqu’en présence de Triton™X-100 à 0.23 mM (0.0144%)
Figure 21. Prévention de l’agrégation de substrats protéiques en condition de stress thermique par
HspSP-ShM2 et HspS-WH7803 en présence de 0.23 mM de Triton™ X-100
Figure 22. HspS-WH7803 et HspSP-ShM2 se présentent sous deux états potentiels d’hétéro-oligomères lorsque l’on diminue la quantité de TritonTm X-100
Figure 23. Alignement des gels SDS-PAGE effectués avec les fractions récupérées lors des essaies de SEC
avec HspS-WH7803 : HspSP-ShM2 condition TritonTmX-100 0.23 mM (haut) et 0.138 mM (bas)
Tableau 1. Séquence de nucléotides des amorces forward et reverse pour l’amplification et le clonage des
gènes hspSP-ShM2, hspS-WH7803 et MYC-hspSP-ShM2 dans le vecteur pETHSUK par le Gibson
Liste des abréviations et des sigles
ACD alpha crystallin domain ADN acide désoxyribonucléique ADP adénosine-5'-diphosphate ARN acide ribonucléique
ARNm acide ribonucléique messager ATP adénosine-5'- triphosphate ATPase domaine d'hydrolyse de l'ATP Bag3 Bcl-2 associated anthanogene 3 Bax Bcl2-associated X protein Bcl-2 B-cell lymphoma 2 BSA bovine serum albumin
C cytosine
Ca+ calcium
CAM C-terminal anchoring module
CHIP chaperone associated ubiquitin ligase
Cl- chlore
CO2 dioxyde de carbone
CS citrate synthase CTD C-terminal domain
EDTA Ethylenediaminetetraacetic acid ERK extracellular signal-regulated kinase FPLC fast protein liquid chromatography
G guanine
H+ proton d'hydrogène
H2 dihydrogène / hydrogène moléculaire
H-/K-/N-box élements de séquence régulatrice de l'expression génique
His histidine
His19/33/34 kinases associées aux membranes H2O monoxyde dihydrogène
HrcA protéine régulatrice de l'expression génique en condition de stress thermique H2S sulfure d’hydrogène
HSF1 heat shock factor 1 HSP heat shock protein sHSP small heat shock protein HSR heat shock response
IPTG isopropyl-B-D-thiogalactoside
kb kilo base
kDa kilo Dalton
LUC luciférase
Mg2+ magnésium
MGlcDG monoglucosyldiacylglycérol
MW molecular weight
N2 diazote / azote moléculaire
Na+ sodium
NaCl chlorure de sodium
NADP+ nicotinamide adénosine dinucléotide phosphate
NADPH nicotinamide adénosine dinucléotide phosphate réduit NEF nucleotide exchange factor
NH3 ammoniac
NH4+ ammonium
Ni-NTA nickel nitrilotriacetic acide
NO2- nitrite
NO3- nitrate
dNTP deoxynucléotide NTD N-terminal domain
O2 dioxygène / oxygène moléculaire
PAR-2 protease-activated receptor 2 PARP poly(ADP- ribose) polymerase PCR polymerase chain reaction PMSF phenylmethanesulfonyl fluoride RNAT ribonucleic acid thermosensor SEC size exclusion chromatography
Smac second mitochondria-derived activator of caspases
SII1130 protéine régulatrice de l'expression génique en condition de stress thermique SDS sodium dodecyl sulfate
SigB/D/E facteur de transcription sigma S2O32- thiosulfate
SUMO small ubiquitin related modifier Tris trisaminomethane
UPR unfolded protein response 5'UTR 5' untranslated région UV-B ultraviolet B
Remerciements
Ce projet de maîtrise n’aurait pu être complété sans le support et l’aide de près ou de loin de plusieurs personnes que je tiens à remercier sincèrement. Tout d’abord, je remercie le Professeur Robert M. Tanguay, directeur de recherche, pour m’avoir accepté et permis d’effectuer mes études de deuxième cycle au sein du laboratoire de recherche. Les années passées à travailler avec votre équipe m’ont permis d’acquérir beaucoup d’expérience sur le plan scientifique et professionnel qui me sera grandement utile dans le futur.
Ensuite je voudrais remercier la Dre Geneviève Morrow pour tout le temps passé à répondre à mes questions aussi insignifiantes soient-elles et pourm’avoir aidé à surmonter les impasses auxquelles j’ai fais face que ce soit théorique ou pratique. Je tiens à souligner la précieuse compagnie de tous mes collègues Afrooz Dabbaghizadeh, Mohamed Taha Moutaoufik et Vincent Roy ainsi que les anciens membres que j’ai côtoyés Idalia Castro, Dre Francesca Angileri et le Dre Ornella Pellerito. Toutes les discussions, les conseils et l’aide de votre part ont permis de rendre mon séjour dans le laboratoire d’autant plus agréable et enrichissant sur le plan culturel et scientifique.
Je voudrais également souligner la gentillesse du Dr Rong Shi pour m’avoir permis d’utiliser son système de Fast Protein Liquid Chromatography et Jérémie Hamel pour avoir donné de son temps à l’opération de l’appareil. Grâce à cela j’ai pu obtenir des résultats de grande qualité qui représentent une majeure partie de mes travaux de maîtrise. Merci aussi au Dr Halim Maaroufi pour les précieux conseils et aide au niveau théorique. Financièrement, je voudrais remercier le regroupement québécois de recherche sur la fonction, la structure et l’ingénierie des protéines (PROTEO) et la Faculté de médecine de l’Université Laval pour m’avoir octroyé des bourses d’étude supérieure. Merci également à Stephen Weeks pour le vecteur pETHSUK.
Enfin je tiens à remercier mes ami(e)s, anciens et présents colocataires pour votre présence, les rires et le temps passé ensemble qui a su rendre mon parcourt universitaire des plus plaisant. Finalement, mon père Claude, ma mère Lise, mon frère Nicolas et leur conjoint(e) Linda, Guy et Kim, je ne pourrai jamais assez vous remercier et vous démontrer à quel point je suis reconnaissant et heureux pour tout ce que vous avez fait pour moi ces dernières années. Vos constants encouragements et votre support m’ont donné une raison de ne jamais lâcher et de me surpasser sans cesse. Ce que j’ai pu accomplir en tant qu’étudiant n’est qu’une extension de votre amour et foi en moi et je vous le dois en grande partie. Merci d’avoir toujours cru en moi.
1.1 Les cyanobactéries
Les cyanobactéries anciennement appelées algues bleu-vert et aussi connues sous le nom de cyanophytes sont des organismes photoautotrophes seulement ou photoauto- et photohétérotrophes appartenant au domaine des bactéries apparues il y a environ 3,5 milliards d’années (Brock et al. 1973 ; Olson, 2006 ; Noffke et al. 2008). Ces organismes photosynthétiques forment depuis leur apparition l’une des biomasses les plus importante de la planète et puisqu’il s’agit d’un phylum très ancien elles sont considérées comme étant les responsables de l’oxygénation initiale de l’atmosphère de la planète (Schopf et al. 1975 ; Rasmussen et al. 2008). Non seulement elles ont permis l’évolution des espèces aérobies, elles ont aussi formé des symbioses avec plusieurs organismes (lichens, coraux, éponges, racines d’angiospermes, bryophytes) (Adams et al. 2012), dont celle menant à l’apparition et l’évolution du chloroplaste chez les organismes eucaryotes photosynthétiques (Falcon et al. 2010). Les cyanobactéries sont séparées en un groupe hétérogène de cinq ordres (Choococcales, Pleurocapsales, Oscillatoriales, Nostocales et Stiggonematales) et peuvent être soit unicellulaires ou présentes sous forme d’amas ou de colonies (filamenteux), et soit posséder ou non la capacité de fixer l’azote (Fogg, 1956 ; Berman-Frank et al. 2003 ; Bothe et al. 2010). De surcroît, elles se sont adaptées et ont colonisé plusieurs environnements terrestres et aquatiques, agissant souvent comme espèces pionnières et jouant un rôle écologique et biogéochimique très important (Whitton et al. 2000). Nous les retrouvons en plus grande quantité dans les océans (Synechococcus, sp.), étangs et mares d’eau douce (Nostoc, Anabaena, Microcystis aerugenosa, Oscllatoria sp., Nodularia spunigena) et nous pouvons aussi les observer dans des environnements extrêmes tels que les lacs gelés de l’antarctique (Calothrixparetina, Nostoc sp., Synechococcus sp., Phormidium frigidum), les déserts (Gloeocapsa sp.), les étangs et mares salés (Gleothece sp., Plectonema sp.) (Rajaram et al. 2014). Leur grande efficacité à utiliser différentes sources de nutriment (Vermaas, 2002) dont certaines non organiques (CO2 et N2 atmosphérique)
pour y extraire les composés désirés et produire leur réserve d’énergie grâce à l’énergie solaire leur a permis de connaître ce succès évolutif. De plus, plusieurs espèces possèdent la caractéristique de pouvoir s’adapter quant à la production d’énergie à différentes conditions environnementales telles que l’anoxie et l’absence de rayons solaires (Moezelaar et al. 1996).
Ainsi, de par leur distribution, biomasse, leur capacité photosynthétique et pour plusieurs espèces, de fixation de l’azote, leurs rôles majeurs dans les écosystèmes et cycles biogéochimiques
de l’azote, oxygène et carbone, l’étude des cyanobactéries présente un grand intérêt écologique et économique.
1.1.1 Cycle du carbone et photosynthèse chez les cyanobactéries
Les cyanobactéries font partie du groupe des phytoplanctons et sont donc des organismes photosynthétiques produisant leurs réserves de carbohydrates grâce à l’énergie solaire et au dioxyde de carbone atmosphérique dissout dans l’eau (Vermaas, 2002). Le processus photosynthétique s’organise et s’opère au niveau de la membrane du thylakoide. Contrairement aux eucaryotes photosynthétiques, le thylakoide cyanobactérien n’est pas compartimenté en granules et stroma, mais il s’agit plutôt d’un repliement de la membrane externe (van de Meene et al. 2006 ; Liberton et al. 2013). Le thylakoide contient les phycobilisomes, excepté chez le genre Proclorochococcus, ainsi que deux photosystèmes (PI et PII) contenant la chlorophylle a et/ou b et les caroténoïdes (Nogales et al. 2012 ; Chang et al. 2015). Le phycobilisome est un complexe multiprotéique (phycobiloprotéines) dont le corps contient le pigment allophycocyanine et des antennes contenant les pigments phycocyanin et phycoerythrin ainsi que des pigments accessoires dépendamment de l’espèce. Suite à l’acquisition d’électrons provenant de la photolyse de molécules d’eau, les phycobilisomes, grâce aux phycobiliprotéines, captent et acheminent l’énergie solaire aux photosystèmes qui en extrait l’énergie. Cette cascade énergétique est utilisée pour obtenir des molécules d’oxygène (O2), des protons, de l’ATP et du NADPH nécessaire à la production d’énergieet la synthèse de biomolécules via les cycles anabolisants.
L’oxygène est relâché dans l’environnement tandis que l’ATP et le NADPH sont acheminés vers le cycle de Calvin pour la production des carbohydrates. Lorsqu’elles se trouvent en condition anaérobique et sans lumière, certaines espèces utilisent d’autres accepteurs d’électrons tels que le sulfure d’hydrogène (H2S), le dihydrogène / hydrogène moléculaire (H2) ou le thiosulfate (S2O32-).
Dans d’autres cas, certaines espèces utilisent leur réserve de glycogène et/ou effectuent la fermentation au dépend de leur réserve de carbohydrates pour s’assurer d’une production maintenue d’énergie malgré l’absence de la lumière (Moezelaar et al. 1996).
Le thylakoide cyanobactérien présente une seconde caractéristique unique soit de pouvoir effectuer la respiration cellulaire et la photosynthèse conjointement au niveau de sa membrane (Vermaas, 2002 ; Hohmann-Marriott et al. 2011). En effet, les cyanobactéries ne présentent pas de séparation spatiale des deux processus comme observée chez les organismes photosynthétiques
eucaryotes où la respiration cellulaire s’effectue dans la mitochondrie tandis que le processus de photosynthèse prend lieu dans le chloroplaste. Le thylakoide cyanobactérien présente au niveau de sa membrane des systèmes de transport des électrons et des gradients de protons créés pour le processus de respiration cellulaire et la production d’ATP et celui de la photosynthèse.
Dans le but d’y produire des réserves organiques de carbone avec l’énergie provenant de la photosynthèse et la respiration, les cyanobactéries extraient le dioxyde de carbone (CO2)
atmosphérique pour la production de carbohydrates (C3H6O3) grâce au cycle de Calvin. On retrouve
pour ce cycle anabolique plusieurs enzymes ainsi que le NADPH et l’ATP provenant des photosystèmes. Pour optimiser l’extraction du carbone du CO2 et par conséquent le cycle, elles
concentrent le CO2 et la RUBISCO dans un microcompartiment appelé le carboxysome (Kerfeld et al.
2010). La RUBISCO est responsable de la première étape de la fixation du carbone atmosphérique et représente le facteur limitant du processus (Long et al. 2007). Du coup, elle permet d’optimiser l’efficacité de l’enzyme et le processus de réduction du CO2 en carbohydrates.
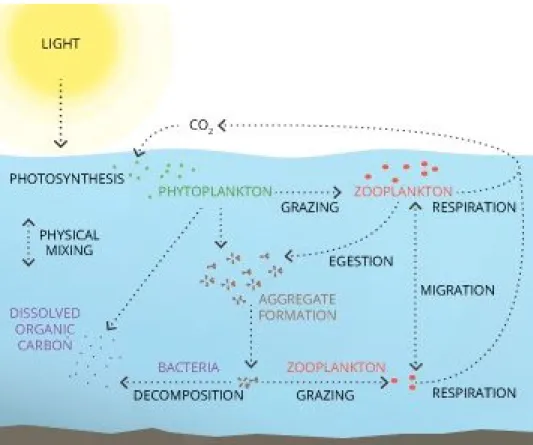
Ainsi, les cyanobactéries utilisent l’énergie solaire pour y transformer le dioxyde de carbone, carbone inorganique, en carbone organique (Fig.1). Cette source de carbone organique rendue accessible par les cyanobactéries est très importante d’un point de vue écologique et biogéochimique. Leur distribution à l’échelle de la planète et leur forte biomasse fait en sorte qu’elles jouent un rôle important et contribuent de façon significative à la productivité primaire globale (Li, 1994). À elles seules, les cyanobactéries sont responsables de la moitié de la fixation globale du carbone (Mitschke et al. 2011). Ce passage du carbone par les cyanobactéries est l’un des points importants de son cycle biogéochimique, donc de la pompe biologique et par conséquent de celui de l’oxygène (Ciais et al. 2013). De part son activité photosynthétique, elles contribuent grandement à l’oxygénation de l’atmosphère, et elles permettent de tamponner l’accumulation du dioxyde de carbone dans l’atmosphère et engendrent son retour vers les fonds océaniques sous sa forme organique et dissoute lors de la mort des cellules.
Du coup, les organismes phytoplanctoniques sont d’excellentes sources de nutriments organiques et représentent la fondation de la chaine alimentaire aquatique. Ils sont le premier chainon de cette chaîne aquatique complexe permettant le passage du carbone et de l’azote aux chaines trophiques supérieures et plus particulièrement aux organismes hétérotrophes herbivores. Une perte de ces organismes mènerait à un effondrement du réseau alimentaire aquatique et par conséquent des chaines trophiques terrestres.
Figure 1. Participation du phytoplancton dans le cycle du carbone. Fixation du carbone atmosphérique
par le phytoplancton grâce à l’énergie solaire et la photosynthèse. Ensuite, le carbone est comsommé par la chaine alimentaire ou décomposé jusqu’à son retour sous forme soluble dans l’océan ou de sédiment sur le plancher océamique.
Source : Fundamentals of environmental measurements, http://www.fondriest.com/environmental-measurements/parameters/water-quality/algae-phytoplankton-chlorophyll/ .
1.1.2 Fixation de l’azote chez les cyanobactéries
L’azote est le deuxième nutriment essentiel à la vie sur terre et se présente majoritairement sous une forme non assimilable par la majorité des espèces soit le diazote / azote atmosphérique (N2). Chez les cyanobactéries ce macronutriment affecte directement leur capacité à fixer le carbone
ainsi que leur croissance (Scanlan, 2003). Selon l’espèce, elles peuvent utiliser différentes sources d’azotes telles que le nitrate (NO3 -), le nitrite (NO2 -), l’ammonium (NH4+), l’urée et autres sources
organiques tandis que d’autres peuvent aussi ou uniquement fixer l’azote atmosphérique faisant partie de ce que l’on appelle les diazotrophes (Lucas et al. 2008 ; Kumar et al. 2010 ; Latysheva et al. 2012 ; Khanna et al. 2015). Les diazotrophes sont des organismes bactériens pouvant croître et survivre avec comme seul apport d’azote l’azote atmosphérique (N2) qu’ils fixent et convertissent en
une source d’azote organique soit l’ammoniac (NH3). Il s’agit donc d’importants biocatalyseurs du
(Vitousek et al. 2002). Les espèces qui utilisent les sources de nitrate, nitrite et organique dissoutes dans l’océan sont généralement côtières du à l’accessibilité des nutriments. Les espèces diazotrophes quant à elles sont plutôt en pleine mer où les sources de nutriments en général se font rares.
Selon les espèces fixatrices d’azote, différentes stratégies sont utilisées pour optimiser le processus afin de minimiser l’exposition de la nitrogénase à l’oxygène (Newton, 2007 ; Bothe et al. 2010). Cet enzyme est responsable de la fixation et conversion de l’azote atmosphérique (N2) en
ammoniac (NH3) et peut être endommagé de façon irréversible par l’oxygène. Chez les organismes
diazotrophiques la photosynthèse et la fixation de l’azote s’effectuent dans l’environnement cellulaire. Ainsi, la nitrogénase peut facilement se retrouver en présence d’oxygène. Pour s’assurer de son bon fonctionnement l’enzyme doit être séquestré dans un environnement anaérobique (Huang et al. 2005 ; Nicolaisen et al. 2009) et plusieurs stratégies sont employées par les espèces de cyanobactéries diazotrophes telles que la séparation spatiale, temporelle ou la formation d’hétérocystes qui est un type cellulaire spécialisé pour la fixation de l’azote (Fay, 1992 ; Schmitz et al. 2001 ; Ananyev et al. 2008 ; Ohki et al. 2009 ; Kumar et al. 2010 ; Sandh et al. 2014). Cette source azotée organique est principalement utilisée pour la synthèse de biomolécules tel que l’ADN et les protéines.
Fait notable les cyanobactéries possèdent aussi la capacité d’assimiler le phosphore et de le stocker sous forme de polyphosphate à l’intérieur de la cellule (Paerl et al. 2001). Ainsi, cette capacité de fixer l’azote atmosphérique et d’emmagasiner des réserves de phosphore leur permet de survivre et croître dans des environnements pauvres en nutriments et de dominer en terme de biomasse les autres organismes aquatiques eucaryotes et procaryotes photosynthétiques ou non.
Tout comme pour le carbone, la fixation de l’azote par les cyanobactéries est d’une énorme importance écologique, puisqu’elle permet à l’azote atmosphérique d’être assimilable pour les plantes et organismes herbivores hétérotrophes (Tilman, 1982). Pour les plantes, l’accessibilité à ce nutriment qui est pour la majorité limitant permet la colonisation de plusieurs environnements considérés hostiles à la végétation. Du coup, la végétation qui en résulte devient la base des chaines alimentaires animalières et maintient plusieurs chaines complexes. Nous pouvons observer cet important apport écologique et économique des cyanobactéries dans quelques régions où il y a utilisation des cyanobactéries fixatrices d’azote comme engrais telles que dans les rizières en Amérique du sud et en Asie (Irisarri et al. 2001 ; Dey et al. 2010) supportant les populations de ces régions.
1.1.3 Le picophytoplancton Synechococcus sp.
Le picophytoplancton regroupe les organismes phytoplanctoniques eucaryotes et procaryotes dont le diamètre est ≤ à 3μm (Stockner, 1988 ; Vaulot et al. 2008). Ces organismes sont retrouvés dans certains écosystèmes d’eau douce (Stockner et al. 1986 ; Weisse, 1993) et plus particulièrement dans la zone photique mésotrophe et oligotrophe des océans où ils dominent en terme de biomasse (Landry et al. 1996 ; Binder et al. 1995, 1996) et de production primaire (Charpy et Blanchot, 1998 ; DuRand et al. 2003). Chez les procaryotes, les organismes appartenant au picophytoplancton font partie du phylum des cyanobactéries et sont séparés en deux genres, Proclorochoccus et Synechococcus.
Les espèces appartenant au genre Synechococcus (Fig.2), dont fait partie l’espèce à l’étude Synechococcus WH7803, sont les cyanobactéries contenant des phycobilisomes les plus abondantes des océans avec quelques individus à plusieurs centaines de milliers d’individus par millilitre (Waterbury et al. 1986 ; Waterbury et Rippka 1989 ; Herdman et al. 2001). Elles appartiennent à l’ordre des Chroococcales, elles sont unicellulaires, et certaines sontdiazotrophes et se présentent sous une forme de coque. Leur classification et phylogénie se fait principalement selon leur localisation, le contenue de l’ADN en G+C, leur besoin en sels minéraux, les principaux pigments de leur appareil photosynthétique et si elles sont motiles ou non. Certaines espèces sont photoautotrophes obligatoires tandis que d’autres sont à la fois photoautotrophes et hétérotrophes pouvant utiliser le carbone organique dissout dans leur milieu. Le genre Synechococcus se caractérise par une division par fission binaire et selon deux modes soit bimodal ou multimodal quant à la distribution de l’ADN. Selon l’espèce, différents facteurs tels que la photopériode, la température et l’accessibilité de certains nutriments régulent leur cycle cellulaire (Vaulot et al. 1996 ; Jacquet et al. 2001).
Elles habitent quelques environnements d’eaux douces, mais se retrouvent principalement dans la zone trophique des océans, allant des zones arctiques 70°N aux zones sub- et tropicales de l’hémisphère sud 50°S (Legendre et al. 1999 ; Zubkov et al. 2000). Leur abondance saisonnière dans ces régions, parfois mésotrophiques, est due à plusieurs propriétés soit leur capacité photosynthétique, de fixation et utilisation de plusieurs sources d’azote (ammonium, nitrite, nitrate et urée), leur petite taille et leur grand ratio surface/volume leur procurant à la fois un faible besoin nutritionnel et une meilleure capacité d’acquisition des nutriments (Scanlan, 2003). Quant à leur
capacité photosynthétique, elles possèdent de la chlorophylle a et leurs phycobilisomes possèdent un centre avec de l’allophycocyanin et des antennes avec des phycocyanins et/ou des phycoerythrins comme différents pigments accessoires. Également, elles sont très bien adaptées à l’acquisition des rayons solaires selon la niche écologique où elles se trouvent (Scanlan, 2003). Certaines espèces sont capables d’adaptation chromatique, donc de moduler le nombre et la composition de leur phycobilisomes pour maximiser leur acquisition des rayons solaires disponibles. Ainsi, à leur tour elles deviennent une excellente source de nutriments pour les brouteurs tels que le zooplancton, et sont un élément essentiel et vital aux différentes chaines alimentaires aquatiques.
Figure 2. Photographie d’une cellule de Synechococcus WH8102. La barre correspond à 0.5 micron.
Source : John Waterbury, Woods Hole oceanographic institute, http://genome.jgi-psf.org/synw8/synw8.home.html
1.1.3.1 Synechococcus WH7803
Synechococcus WH7803 est une espèce côtière et marine non motile (Waterbury et al. 1986 ; Moore et al. 2002 ; Scanlan, 2003). Elle possède un pourcentage moyen de nucléotides G+C dans leur génome de 61,3 % et une capacité d’utiliser le nitrite, nitrate et l’ammonium comme source d’azote. Il s’agit d’une espèce photoautotrophe obligatoire qui possède de la chlorophylle a et des phycoerythrins comme pigments principaux pour capter la lumière. Ses besoins en sodium (Na+),
calcium (Ca+), chlore (Cl-) et magnésium (Mg2+) pour soutenir sa croissance sont élevés. Elle se
divise à un rythme d’environ une division par jour selon le mode bimodal puisque deux pics de distribution de l’ADN correspondant à la phase G1 et G2 sont observables dans une même journée (Liu et al. 1999).
1.2 Virus marins
Les virus marins ont longtemps été sous estimés quant à leur abondance, leurs rôles écologiques et leur implication dans le cycle biogéochimique du carbone, de l’azote et de l’oxygène. Cependant, depuis une trentaine d’années plusieurs travaux de recherche ont été effectués nous permettant d’apprécier avec justesse leur importance et contribution à l’échelle de la planète. Les virus marins sont les particules avec contenu génétique les plus abondantes des océans avec 10 millions de ces particules par millilitre d’eau (Bergh et al. 1989). Le nombre de virus est estimé à 4 x 1030, ce qui représente une population 15 fois plus élevée que celle des bactéries et archae (Suttle,
2007). Ainsi, il n’est pas étonnant de les caractériser comme l’une des principales causes de mortalité chez les microorganismes procaryotes et eucaryotes marins hétérotrophes et photoautotrophes (Proctor et al. 1988, 1990 ; Suttle, 2005, 2007). En effet, selon certaines estimations les virus tuent environ 20 % de la biomasse des microorganismes océaniques par jour et jusqu’à 40% pour les procaryotes marins.
Leur distribution et abondance sont dépendantes de celles de leurs hôtes et de leur productivité (Cochlan et al. 1993 ; Clasen et al. 2008). En effet, une diminution de leur présence est observée plus on s’éloigne des côtes et plus on s’enfonce en profondeur ainsi qu’une régulation journalière (Winter et al. 2004) et saisonnière (Sandaa et al. 2006) de leur abondance.
Ainsi, de part leur abondance et leur activité lytique, les virus influencent grandement les différents processus dans lesquels les organismes procaryotes et eucaryotes marins sont impliqués. Chez les organismes photosynthétiques, les virus diminuent la capacité générale des communautés à fixer le carbone et par le fait même régulent la productivité primaire et la quantité de CO2
atmosphérique (Suttle et al. 1990, 1992 ; Danovaro et al. 2011). De plus, ils contribuent à la circulation des nutriments tels que le carbone, l’azote, le phosphore et le fer qui sont parfois rares et limitant pour la croissance des espèces océaniques (Suttle, 2005, 2007). Une partie du retour de ces composés sous leur forme organique et dissoute permet aux individus hétérotrophes de s’en accaparer pour leur métabolisme tandis qu’une autre partie retourne vers les fonds océaniques contribuant au phénomène de pompe biologique.
Les organismes viraux jouent aussi un rôle majeur au niveau de l’écologie des différentes populations en contribuant à leur régulation et au maintien de la diversité de la communauté (Wommack et al. 2000 ; Thingstad et al. 2000, 2014). Les espèces les plus dominantes et abondantes sont plus susceptibles d’être infectées et lysées. Ceci permet de réduire leur population
et de permettre aux organismes moins dominants d’augmenter ou de maintenir la taille de leur population puisque les nutriments et l’espace deviennent accessibles. Également, ceci permet de maintenir une diversité génétique au sein des communautés d’une même espèce (Muhling et al. 2005). En effet, les individus infectés présentent un génotype différent des autres puisqu’ils sont reconnus par le virus dû à la reconnaissance de récepteurs spécifiques. Ainsi, la mort de ces individus permet de diversifier les génotypes au sein de la population. Malgré le fort taux de mortalité par infection virale, il faut noter que beaucoup d’individus au sein d’une population seront résistants, ce qui permet cette coexistence entre les virus et microorganismes (Waterbury et al. 1993 ; Sullivan et al. 2003). Ces observations à la fois d’une forte infection virale et d’une grande résistance sont expliquées par une coévolution antagoniste résultant en une course à l’armement entre les microorganismes et les virus (Buckling et al. 2002 ; Marston et al. 2012). D’une part, l’un acquiert la résistance et l’autre s’adapte et développe la capacité d’infecter de nouveau et ainsi de suite avec l’acquisition de différents processus ou mécanismes adaptatifs selon l’espèce et l’individu/particule. Encore une fois, ceci résulte en une diversité génomique et phénotypique entre les individus dont certains seront de nouveau plus susceptibles à l’infection par un type viral tandis que d’autres seront moins sensibles (Rodriguez-Brito et al. 2010 ; Avrani et al. 2011). Parfois, cette diversification génétique est le résultat de la phase lysogénique du virus et du phénomène de transfert horizontal de gènes ce qui permet l’échange de gènes entre les virus et leurs hôtes pouvant mener à l’acquisition de nouvelles résistances et caractéristiques fonctionnelles, ou au potentiel d’infection de nouvelles espèces (Canchaya et al. 2003).
1.2.1 Cyanophages de Synechococcus sp.
Les cyanophages sont des virus marins infectant les cyanobactéries qui appartiennent à l’ordre des Caudovirales et soit à la famille des Podoviridae, des Siphoviridae, des Myoviridae ou à celle des virus marins sans queue (Breitbart et al. 2002, 2004 ; Angly et al. 2006 ; Williamson et al. 2008, Brum et al. 2013). Chaque famille de l’orde des Caudovirales est reliée au bactériophage de type T4 et leur génome, de 20 à 500 kb, présente une grande homologie de séquence et un ordre de gènes similaire avec celui des phages entériques (Pope, 2007 ; Millard et al. 2009 ; Huang et al. 2012). Ils sont ubiquitaires, abondants dans l’océan (Sullivan et al. 2003 ; Williamson et al. 2008) et la majorité d’entres eux infectent le picophytoplancton du genre Synechococcus et Prochlorococcus et autres cyanobactéries et bactéries marines (Suttle, 2000). Ils permettent le recyclage et le retour
d’environ un quart du carbone fixé par ces cyanobactéries photosynthétiques vers sa forme organique et dissoute dans les océans (Wilhelm et al. 1999).
Les cyanophages ont une morphologie (Mann et al. 2005 ; Suttle, 2005) et un cycle viral (Duckworth, 1987 ; Weinbauer, 2004 ; Rakhuba et al. 2010) typique de ceux des bactériophages. Ils possèdent une capside où l’on retrouve les acides nucléiques, une queue tubulaire de longueure variable avec une gaine contractile, des fibres caudales, de l’ADN linéaire double brin. Plusieurs ont la capacité d’infecter plusieurs hôtes d’espèces différentes (Fig.3) (Safferman et al. 1983 ; Suttle, 2000). Quant au cycle général des cyanophages, l’absorption est médiée par l’interaction irréversible entre les fibres caudales du phage et un récepteur protéique ou glycoprotéique spécifique présent à la surface externe de la membrane cellulaire de l’hôte. Il s’agit du déterminant crucial de la reconnaissance du virus à son hôte. Suite à l’absorption, le génome viral subit une décapsidation et le virus intègre son génome à l’hôte par dégradation locale de la membrane cellulaire et une injection du génome par contraction de la gaine à travers la membrane plasmique. Les myovirus peuvent intégrer leur génome à celui de leur hôte ou entrer plutôt immédiatement dans un cycle lytique. Le cycle lytique est caractérisé par une redirection du métabolisme de l’hôte pour la production de nouveaux phages. Il s’agit de la production des constituants physiques du virus dont ceux des capsides, les acides nucléiques, la queue contractile, les fibres caudales, les protéines accessoires pour la maturation de la capside et l’encapsidation des acides nucléiques. Une fois les phages assemblés et une certaine quantité atteinte, il y a lyse des bactéries. La lyse est caractérisée par une perméabilisation de la membrane de la cellule hôte par différents enzymes menant éventuellement à l’éclatement de la bactérie. Chez les cyanophages, un seul enzyme ayant ce rôle a été identifié et il s’agit de la holin (Pope et al. 2007). Dans certains cas, les virus sont relâchés par bourgeonnement ou extrusion, donc indépendamment de la lyse. Ce type de relâchement mène parfois à une infection chronique puisque les bactéries ne meurent pas et sont propices à une autre infection. Il s’agit dans ce cas d’un pseudolysogène.
Chez les phages tempérés ou lysogéniques, il y a intégration du génome viral dans celui de son hôte et celui-ci entre dans un état quiescent (Brussow et al. 2004). À ce moment, l’ADN viral s’appelle prophage puisqu’il a le potentiel de devenir un phage et la cellule hôte devient lysogène. L’ADN d’origine virale est répliqué en même temps que le génome de son hôte et est transmis aux cellules filles. Éventuellement, différents facteurs qui sont défavorables à la croissance et la survie de la cyanobactérie peuvent activer la transcription des gènes pour permettre le déclenchement de la
phase lytique. Lors de cette intégration du génome il est possible d’observer, tel que mentionné précédemment, des évènements de transfert horizontal de gènes entre le virus et son hôte (Canchaya et al. 2003). Conséquemment, des gènes d’origine bactérienne se retrouvent dans le génome du virus suite à une intégration aléatoire et/ou à la transcription non fidèle des gènes viraux. Ces gènes une fois dans le génome et encapsidés dans le virus peuvent être silencieux ou être exprimés et fonctionnels affectant directement ou indirectement le cycle viral.
Figure 3. Photographie par microscopie électronique du phage wV8 démontrant la morphologie typique d’un myovirus. Il est possible d’observer la capside, la queue tubulaire avec gaine contractile (tête
de fleche pleine) et les fibres caudales (tête de fleche vide). La barre correspond à 100 nm (tiré de Villegas, 2009).
Parmi les transferts horizontaux de gènes documentés, il y a ceux des « High light inducible genes », des gènes codant pour des composantes protéiques centrales du photosystème II (PSII), les protéines D1 et D2 et gènes du métabolisme du carbones cyanobactérien. Ces gènes présents chez certains cyanomyovirus (Sullivan et al. 2010), cyanopodovirus (Labrie et al. 2013) et cyanosiphoviruse (Huang et al. 2012) présentent une forte homologie de séquence avec ceux de la cyanobactérie hôte, ce qui supporte l’hypothèse d’une acquisition possible par transfert horizontal de gènes. Ces découvertes réalisées chez plusieurs phages marins grâce à des études génomiques ont
été suivies par des études de caractérisation fonctionnelle in vivo démontrant l’impact positif de l’expression de ces protéines virales sur le « fitness » (succès reproducteur et valeurs adaptatives) des cyanobactéries (Mann et al. 2003 ; Lindell et al. 2005 ; Clokie et al. 2006 ; Sullivan et al. 2006). Fonctionnellement, D1 et D2 forment un dimère au cœur du PSII et lient les pigments et cofacteurs nécessaires pour la photolyse de l’eau fournissant l’oxygène moléculaire et les électrons au système tandis que les « High light proteins » dissipent l’excès d’énergie solaire qui peut endommager le système. En effet, l’irradiation solaire peut endommager de façon irréversible D1 et D2 qui sont les composantes les plus sensibles du PSII. Lorsque abimées et dysfonctionnelles celles-ci sont soit prises en charge par les chaperons moléculaires ou remplacés efficacement suite à la synthèse de nouvelles protéines. En période d’irradiation intense il se produit le phénomène de photoinhibition causée par l’incapacité de la cellule à remplacer ces composants résultant en un arrêt de la photosynthèse. Il est proposé que ces gènes viraux sont exprimés au sein de la cyanobactérie et fournissent les protéines à leur hôte ce qui empêche la photoinhibition et permet le maintien de son efficacité photosynthétique, donc de sa production énergétique. Ces gènes photosynthétiques sont sous les mêmes régions promotrices que les gènes de la capside et se font de pair lorsqu’il y a activation de la transcritption des gènes viraux. Ceci permet de maintenir le pool des protéines correspondantes lorsque la machinerie transcriptionnelle et traductionnelle est séquestrée pour la production de virus. En outre, le virus promeut ainsi la survie de son hôte et «s’assure» qu’il produit l’énergie nécessaire pour son propre cycle.
2.1 Protéines de choc thermique
C’est en 1962 que débutent les recherches sur la réponse au stress grâce à l’observation par Ritossa de gonflements sur les chromosomes polytènes des glandes salivaires chez des larves de Drosophila buckii ayant subi un choc thermique (Ritossa, 1962). Des travaux subséquents ont permis d’identifier ces gonflements comme étant des sites de transcription actifs d’ARNm codant pour des protéines de choc thermique dont les chaperons moléculaires (HSP) (Tissières et al. 1974). Ceci fut suivi d’une série d’études menant à l’identification et la caractérisation de la réponse au stress thermique / heat shock response (HSR), un processus complexe hautement conservé chez les organismes vivants et auquel participent activement les protéines de choc thermique.
Les HSPs se divisent en cinq familles selon leur poids moléculaire apparent sur gel SDS-PAGE et leur homologie de séquence. Quatre de ces familles dont celles des Hsp100 (ClpA, ClpB,
ClpE, ClpX et ClpY chez les bactéries), Hsp90 (HptG), Hsp70/Hsc70 (Dnak) et Hsp60 (GroEL) comportent des chaperons ATP dépendantes, et une seule famille la famille des petites protéines de stress / small Heat shock proteins (sHSP), regroupe des chaperons ATP-indépendants. Ces protéines peuvent être exprimées de façon constitutive et/ou induite et se localisent dans le cytosol et/ou les autres compartiments cellulaires (Morimoto, 2008 ; Richter et al. 2010 ; Gidalevitz et al. 2011 ; Saibil, 2013). L’activation dirigée de leur expression suite à un stimulus est principalement associée à leur rôle dans le maintien de la protéostasie cellulaire (Walter et al. 2002 ; Bukau et al. 2006 ; Buchberger et al. 2010 ; Richter et al. 2010 ; Haslbeck et al. 2015a,b). La protéostasie cellulaire qui représente l’homéostasie protéique se caractérise par le maintien de l’équilibre entre la synthèse de nouvelles protéines et la dégradation, via le protéosome, de celles qui sont endomagées et potentiellement toxiques pour la cellules. Parmi les perturbateurs de la protéostasie et activateurs de la HSR, il y a de façpn générale les stress environnementaux, chimiques et physiologiques et plus précisement le stress thermique, oxydatif (Kalmar et al. 2009), les métaux lourds (Heckathorn et al. 2004), certaines infections virales et bactériennes (Zugel et al. 1999), les maladies génétiques affectant la conformation des protéines (Wilhelmus et al. 2007, 2009), les inhibiteurs chimiques de la respiration cellulaire et la progression tumorale ou transformation oncogénique (Tang et al. 2015). Ces stress ont pour effet, entre autre, de compromettre directement ou indirectement le maintien de la protéostasie en perturbant l’intégrité structurelle et fonctionnelle des protéines cellulaires. Ces protéines dénaturées voient leurs domaines hydrophobes être exposés et ont le potentiel d’interagir avec plusieurs constituants cellulaires et de former des agrégats de type amyloïdes. Ces interactions aléatoires et agrégats sont toxiques pour la cellule pouvant causer beaucoup de dommages jusqu’à mener à la mort cellulaire et sont associés à plusieurs maladies telles que les cataractes, myopathies, maladies neurodégénératives et cancers (Clark et al. 2000 ; Benndorf et al. 2004 ; Mymrikov et al. 2011 ; Basha et al. 2012 ; Tang et al. 2015). Dans le but de maintenir la protéostasie et ultimement l’homéostasie cellulaire, les organismes activent la Heat Shock Response et la Unfolded Protein Response (UPR), réponse organelle spécifique, (Ron et al. 2007 ; Richter et al. 2010 ; Hartl et al. 2011) résultant en l’expression des HSPs et de la machinerie de dégradation. En condition physiologique, les HSPs sont aussi impliquées dans le contrôle de la protéostasie cellulaire en assistant aux étapes de la biosynthèse et du métabolisme des protéines (Hartl et al. 2002 ; Bukau et al. 2006 ; Morimoto, 2011).
2.1.1 Petites protéines de stress thermiques (sHSPs)
2.1.1.1 Séquence et structure des sHSPsLes sHSPs sont présentes chez tous les groupes du vivant avec un nombre de gènes codants pour ces protéines qui varie selon l’organisme allant de un seul à douze chez certaines bactéries, dix chez l’humain, seize chez C. elegans, douze chez la drosophile et vingt chez les plantes supérieures (Kriehuber et al. 2010). Elles représentent la famille de HSPs la moins conservée en termes de séquence et poids moléculaire avec une moyenne de 161 acides aminés et un poids variant entre 12 kDa et 42 kDa (De Jong et al. 1993, 1998 ; Kriehuber et al. 2010). Un trait commun important, cependant, est la présence du domaine caractéristique et représentatif des sHSPs soit le domaine α-crystallin (ACD) flanqué d’une part d’un domaine N-terminale (NTD) et de l’autre, lorsque présente, d’un court domaine C-terminale (CTD) (Fig.4A). Typiquement, les sHSPs peuvent former des oligomères de différentes tailles qui sont importants pour leur fonction de chaperon moléculaire (Fig.4D) et dont le nombre de sous unités dépend de la sHSP et des conditions physiologiques (Kim et al. 1998 ; van Montfort et al. 2001a,b ; Haslbeck et al. 2005 ; Basha et al. 2012 ; Hilton et al. 2012, 2013 ; Delbecq et al. 2013 ; Hanazono et al. 2013 ; Heirbaut et al. 2015). Ces oligomères sont très dynamiques quant à leur conformation, structure et état oligomérique et dépendent des interactions transitoires entre les différentes régions (NTD, ACD et CTD) (Basha et al. 2012 ; Hilton et al. 2012, 2013 ; Haslbeck et al. 2015a,b ; Heirbaut et al. 2015). Construits à partir d’interactions entre des dimères et formant des complexes de 12 à plus de 32 sous unités, les sHSP présentent un profil d’états oligomériques soit monodisperse ou polydisperse. Parmi les sHSPs qui se présentent selon un profil polydisperse, certaines possèdent une distribution plus uniforme entre les formes oligomériques allant de 10 à 50 sous unités comme l’αB-crystalline (Aquilina et al. 2003 ; Baldwin et al. 2011) tandis que d’autres présentent des formes majoritaires et minoritaires d’états oligomériques tels que Hsp26 de Saccharomyces cerevisae (Benesch et al. 2010) et Arc1 de M. tuberculosis (Kennaway et al. 2005). De plus, plusieurs sHSPs possèdent la capacité de s’associer à d’autres sHSPs et de former des hétéro-oligomères dans certaines conditions (Sun et al. 2004 ; Fontaine et al. 2005). Pour certaines sHSPs, ceci régule et adapte leur activité de chaperons moléculaires et leur spécificité de clients.
La région N-terminale (NTD) présente une grande variabilité en terme de conservation et longueur de séquence avec une moyenne de 56 résidus. Elle est plus courte chez les virus et les procaryotes que chez les organismes eucaryotes (Kriehuber et al. 2010 ; Heirbaut et al. 2015). C’est
au niveau de cette région qu’il est possible observer des sites de modification post-traductionnelle, d’oxydation, glycation, déamination, d’attachement de methylglyoxal et spécialement des sites de phosphorylation (Fig.4A) (Garrido et al. 2012). Ces modifications sont le résultat de la réponse à plusieurs facteurs tels que le stress thermique, les agents oxydants, les cytokines inflammatoires et les mitogènes dans le but de moduler leur activité. Le statut de phosphorylation de plusieurs sHSPs, spécialement chez les mammifères, permet de réguler leur équilibre d’association/dissociation des sous unités et par conséquent leur activité chaperon. Hsp27/HspB1 (Rogalla et al. 1999), αB-crystallin/HspB5 (Kato et al. 1998) et Hsp20/HspB6 (Beall et al. 1999) sont parmi les sHSPs pouvant être phosphorylées. Ces modifications peuvent aussi avoir comme effet de stabiliser la formation d’homo- et d’hétéro-oligomère en association avec différents substrats tels que d’autres sHSPs (Parcellier et al. 2005 ; Sun et al. 2006). De plus, la phosphorylation de résidus précis module la distribution intracellulaire de quelques sHSPs, comme c’est le cas pour Hsp27/HspB1 (Garrido et al. 1998 ; Adhikari et al. 2004) et αB-crystallin/HspB5 (den Engelsman et al. 2004) pour lesquelles la phosphorylation permet leur translocation au noyau. De surcroît, c’est dans le NTD qu’on observe des séquences de localisation canonique comme pour la Hsp27 de Drosophila (DmHsp27) étant nucléaire (Michaud et al. 2008), la Hsp22 de Drosophila (DmHsp22) (Morrow et al. 2000) et Hsp28 de Toxoplasma gondii (de Miguel et al. 2005) toutes deux mitochondriales et particulièrement chez les plantes où l’on observe plusieurs sHSPs organelles-spécifiques. Également, des études de mutagénèse dirigée chez des sHSPs indépendantes de la phosphorylation ont permis d’associer cette région au maintien de l’activité chaperon et à la stabilisation thermodynamique des gros oligomères (Haslbeck et al. 2004 ; Stromer et al. 2004 ; Basha et al. 2006, 2013 ; Jehle et al. 2011 ; Hilton et al. 2012 ; Haslbeck et al. 2015a ; Heirbaut et al. 2015). Outre la composition en acides aminés, la longueur du NTD influence aussi le profil oligomérique des sHSPs. En effet, il est proposé que c’est par des contacts avec le NTD, entre autre, qu’il y a liaison au substrat et que la formation d’oligomère de l’ordre de 24 mères et plus est possible (Haslbeck et al. 2015a ; Heirbaut et al. 2015). Également, les variations observées dans cette région seraient responsables des différences dans le spectre de substrat des sHSPs et particulièrement chez les sHSPs d’un même organisme (Hilton et al. 2012 ; Haslbeck et al. 2015a ; Heirbaut et al. 2015). Cependant, la flexibilité, le dynamisme et par conséquent le manque de structure cristallisée de cette région ne nous permet pas d’établir toutes les interactions précises permettant au NTD d’influencer le comportement fonctionnel et structurel des sHSPs.
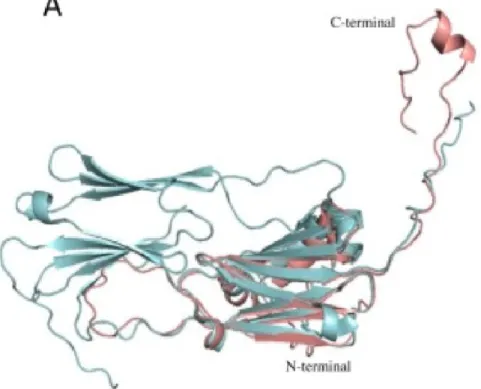
Le domaine α-crystallin (ACD), pour sa part, possède une longueur de séquence relativement conservée de 94 acides aminés en moyenne mais une faible conservation de la séquence en acides aminés malgré qu’elle soit la plus conservée des trois régions des sHSPs (Kriehuber et al. 2010). Le motif le plus souvent observé au niveau de cette région est le motif A-x-x-x-n-G-v-L retrouvé à la fin du domaine ACD (De Jong et al. 1998 ; Narberhaus, 2002). Malgré cette diversité de séquence, la structure tridimensionnelle résultante est très bien conservée chez tous les membres de cette famille de protéines (Fig.4). Cette structure ressemblant à celle d’une immunoglobuline est composée de 7 brins β nommés β2 à β9 formant un sandwich de feuillets β. Chacun des deux feuillets, arrangés de façon antiparallèle, est composé de trois brins β connectés par des boucles interdomaines (Fig.4) (van Montfort et al. 2001a,b, 2002 ; Delbecq et al. 2013). L’un des résidus le mieux conservé de cette région est l’arginine (R120) au niveau du brin β7 important pour la dimérisation des sHSPs. Ainsi, l’ACD est responsable de la dimérisation des sHSPs (Fig. 5B et 5C) ce qui représente la première étape de la formation des oligomères, mais ne peut à lui seul permettre la formation de structure oligomérique au delà du dimère (Bagneris et al. 2009 ; Baranova et al. 2011). Il existe deux mécanismes de dimérisation connu, l’un est observé chez les plantes, bactéries, archae et levures (Fig. 4B) et l’autre chez le reste du vivant (Fig.5C). Celui observé chez les plantes, bactéries, archae et levures est le résultat de l’échange entre chacun des deux monomères de leur brin β6 stabilisant l’association des monomères (Fig.5B) (Van Montfort et al. 2001a,b). Chez les autres organismes vivants, ce que l’on observe est une fusion des brins β6 et β7 d’un même monomère fournissant l’interface nécessaire pour les interactions avec le brin de fusion du monomère adjacent orienté antiparallèlement (Fig.5C) (Delbecq et al. 2013).
Figure 4. Représentations 3D des structures cristallisées et superposition des ACD de monomère d’HspB1 (vert, PDB : 4MJH) et αB-crystalline (violet, PDB : 2WJ7) humaines. Conservation de la
structure tridimensionnelle composé de sept brins β2 à β9 reliés par des boucles inter-domaines formant un sandwich de feuillets β de deux feuillets anti-parallèles. (Tiré de Baranovaet al. 2011)
La région C-terminale (CTD) est plutôt courte avec en moyenne dix résidus et ne dépassant pas les 20 acides aminées (Kriehuber et al. 2010). Elle se sépare en deux segments, la queue et l’extension C-terminale, séparés par le motif CAM. Le motif CAM (C-terminal Anchoring Module) est une séquence typiquement représentée par le motif I-x-I et I-x-I/V et située pour les vertébrés dans une séquence palindromique d’acides aminés (Fig.5A) (Poulain et al. 2010 ; Hilton et al. 2013). C’est ce motif, entre autre, qui permet et dirige la formation d’oligomères de tailles moyennes. Les chaines latérales des résidus du motif interagissent avec les dépressions hydrophobiques formées par les brins β4/β8 des autres dimères et stabilisées grâce à des forces de van der Walls et des ponts d’hydrogène (Treweek et al. 2010 ; Baldwin et al. 2011 ; Delbecq et al. 2012 ; Heirbaut et al. 2015). Tout comme pour le NTD des études de mutagénèse dirigée dans le CTD ont permis d’impliquer cette région dans la reconnaissance et la liaison du substrat, malgré que les interactions exactes ne soient toujours pas connues (Treweek et al. 2007).
À l’équilibre et en condition physiologique, ces protéines sont très dynamiques au niveau de leurs conformations quaternaires et structures oligomériques. Ce qui semble évident, malgré le manque d’informations sur les interactions, est que les trois régions sont nécessaires et que la formation d’oligomères se fait de façon hiérarchique (Haslbeck et al. 2015a). Tandis que le ACD permet la dimérisation, le motif CAM du CTD engendre la formation de tétramères ou d’hexamères qui par la suite s’associent en gros oligomères grâce aux contacts avec le NTD (van Montfort et al. 2001a,b ; Jehle et al. 2011 ; Basha et al. 2012 ; Haslbeck et al. 2015a). Cette oligomérisation hiérarchique et les interactions entre les trois régions et particulièrement par le NTD et le CTD permettent une régulation de leur activité et un dynamisme structurel en réponse aux changements externes ou internes à l’organism (ou cellules) à leur environnement. Ce dynamisme caractérisé par un échange constant de sous unités procure pour la majorité un profil oligomérique polydisperse et la présence constitutive d’états sub-oligomériques et de dimères (Fig.6). Le dimère, qui est le résultat de l’association de deux monomères, est le bloc de base pour la formation des oligomères et représente la forme active et compétente à la reconnaissance des substrats et sa présence en
continuité, conséquence du fort dynamisme structurel, permet une activation rapide pour s’ajuster ou répondre aux besoins cellulaires lorsque la cellule en a besoin.
Figure 5. Représentation schématique de l’organisation de la séquence primaire d’une sHSP, mécanismes et structures de la dimérisation des monomères et représentation 3D de la structure quaternaire de MjHsp16.5, SpHsp16 et TaHsp16.9. (A) Organisation des domaines des sHSPs. NTD (vert foncé avec pointillés), ACD (vert pâle) et le CTD (vert foncé avec le motif conservé I-x-I en forme de cylindre et le reste en pointillés). Tel qu’indiqué jusqu’à trois sites de phosphorylation existent chez certaines sHSPs. (B) Structure de l’échange du brin β6 lors de la dimérisation. (C) Structure de l’interface formée par la fusion du brin β6 et β7 de l’ACD permettant la dimérisation de la sHSP humaine αB-crytalline (solid-state NMR, PDB ID2KLR; (Jehle et al. 2011)). (D) Comparaison à l’échelle de structures oligomériques cristallisées. MjHsp 16.5, M. jannaschii Hsp16.5 représentant un 24mères (Kim et al. 1998). SpHsp16, Schizosaccharomyces
pombe Hsp16 représentant un 16mères (Hanazono et al. 2013). TaHsp16.9, Tritium aestivum Hsp16.9 représentant un 12mères (van Montfortet al. 2001b). Les dimères, blocs de base, sont représentés en bleu poudre, turquoise, rouge et orange. (Tiré de Haslbecket al. 2015b).
2.1.1.2 Les sHSPs et la réponse aux stress (HSR)
Les sHSPs, de part leur activité de chaperons moléculaires ATP-indépendants, constituent la première ligne de défense contre différents stress (Richter et al. 2010 ; Basha et al. 2012 ; Haslbeck et al. 2005, 2015b ; Fu, 2014). Elles se présentent sous formes constitutives et/ou fortement induites en période de stress et interagissent avec les protéines en voie de dénaturation pour empêcher leur agrégation irréversible (Fig.6) (Richter et al. 2010 ; Basha et al. 2004a, 2012 ; Haslbeck et al. 2005, 2015b ; Fu, 2014). L’activation de leur activité de chaperon moléculaire se fait par un déplacement de
leur équilibre et profil oligomérique qui amène un changement conformationnel et/ou une accélération de la vitesse d’échange des sous unités. Cette activation est stimulée par quatre facteurs : soit le stress lui-même tel que l’augmentation de la température, l’apparition de protéines dénaturées, la formation d’hétéro-oligomères (sHSP : substrat) et la phosphorylation chez les sHSPs eucaryotes (Bova et al. 2002 ; Sobott et al. 2002 ; Fu et al. 2004 ; Hilton et al. 2012 ; Fu, 2014 ; Haslbeck et al. 2015a). Ainsi, cette perception précoce du stress et des protéines non natives leur permet d’agir comme senseurs et contribue à l’activation et au maintien de la HSR (McHaourab et al. 2009).
Selon le modèle proposé, le dynamisme au niveau de la conformation quaternaire des sHSPs leur permet d’adopter une conformation dite ouverte/excitée par des changements de la conformation tertiaire des sous unités. La conformation ouverte permettrait de déstabiliser les interactions inter-oligomèriques et de libérer les domaines de reconnaissance aux substrats (Franzmann et al. 2008 ; McHaourab et al. 2009 ; Stengel et al. 2010 ; Basha et al. 2010 ; Hilton et al. 2012 ; Haslbeck et al. 2015a ; Heirbaut et al. 2015). Dans certains cas, cette libération des domaines par le changement conformationnel serait suffisante pour la reconnaissance des substrats. De façon plus générale, cette conformation ouverte représente la première étape du déplacement du profil oligomérique en stimulant la dissociation des oligomères par l’affaiblissement de l’ensemble des interactions inter-oligomèriques. Cette dissociation de sous unités permet le relâchement d’espèces sub-oligomériques plus petites (tétramères/hexamères) et de dimères dont les régions NTD et CTD devenues libres pourront interagir de façon stable et transitoire avec les substrats (Lentze et al. 2004 ; Benesch et al. 2008 ; Jaya et al. 2009 ; Heirbaut et al. 2015). Bref, le dynamisme structurel des sHSPs stimulé en période de stress est observé d’une part selon la conformation de la structure quaternaire au sein d’un même oligomère et de l’accélération des échanges de sous unités résultant en une population de plusieurs états oligomériques actifs que l’on appelle protomères.
La reconnaissance du substrat par les sHSPs se fait grâce à l’exposition des domaines hydrophobes en début de processus de dénaturation du substrat et mène à la formation d’hétéro-oligomères de poids supérieur aux d’hétéro-oligomères natifs qui sont composés de sHSPs et protéines non natives. Ces complexes forment un réservoir de protéines mal repliées et les maintiennent dans un état soluble et compétent au repliement (Ehrnsperger et al. 1997 ; Lee et al. 1997 ; Haslbeck et al. 1999 ; Stromer et al. 2003 ; Friedrich et al. 2004 ; Stengel et al. 2010, 2012). Cette prévention de l’agrégation, peu importe le substrat, doit se faire en cours de processus de dénaturation puisque les