Caractérisation de la structure génétique de populations
et évaluation de ses déterminismes environnementaux
chez le grand brochet (Esox lucius) dans le système lac
Ontario – fleuve Saint-Laurent.
Mémoire
Geneviève OUELLET-CAUCHON
Maîtrise en biologie
Maître ès sciences (M.Sc.)
Québec, Canada
© Geneviève Ouellet-Cauchon, 2013
iii
Résumé
Nous avons documenté la génétique du paysage du grand brochet (Esox lucius) en nous basant sur l’analyse de 22 microsatellites, incluant 10 nouveaux marqueurs développés ici, dans le système lac Ontario – fleuve Saint-Laurent. La structure génétique des populations était globalement très faible mais spatialement variable ; le niveau moyen de différenciation génétique dans la section amont (Ontario) de l’aire d’étude était trois fois plus important que celui observé dans le secteur aval (Québec). Vingt variables environnementales furent considérées et les différentes masses d’eau, la présence de barrages et la stabilité interannuelle du niveau d’eau s’avérèrent positivement associées à l’intensité de la différenciation génétique. Une importante variation du niveau d’eau influe sur la qualité et la localisation annuelle des habitats de reproduction du grand brochet, l’instabilité locale de l’habitat prenant la forme d’une variation interannuelle du niveau d’eau semble localement inhiber la structure génétique de populations, probablement en empêchant la philopatrie.
Mots clés : Esox lucius, grand brochet, génétique du paysage, structure génétique de
populations, variation environnementale, stabilité de l’habitat, marqueurs microsatellites, gestion, conservation.
v
Abstract
In this study, we documented the landscape genetics of northern pike (Esox lucius) based on the analysis of 22 microsatellites, comprising 10 markers developed for the purpose of this study, in the Lake Ontario – St. Lawrence River system. Population genetic structure over the whole study area was globally very weak but spatially variable with mean level of differentiation in the upstream (Ontario) section of the studied area being three-fold higher than observed in the downstream (Québec) sector. Twenty environmental variables were considered and different water masses, dams’ presence and inter-annual water level stability were positively associated to the extent of genetic differentiation. Since high water level variation impacts on yearly quality and localization of northern pike spawning habitats, local habitat instability which is under the form of inter-annual water level variation seems to locally impede population genetic structure, perhaps by inhibiting philopatry behavior.
Keywords: Esox lucius, northern pike, landscape genetics, population genetic structure,
environmental variation, habitat stability, microsatellite markers, management, conservation.
vii
Table des matières
Résumé ... iii
Abstract ... v
Table des matières ... vii
Liste des tableaux ... ix
Liste des figures ... xi
Liste des abréviations et des sigles ... xiii
Remerciements ... xvii
Avant-propos ... xxi
Introduction générale ... 1
Concepts ... 1
Conservation et diversité génétique ... 1
Structure génétique des populations ... 2
Génétique du paysage ... 3
Le grand brochet ... 4
Biologie et écologie de l’espèce ... 4
Bilan des connaissances en génétique des populations ... 5
Système à l’étude ... 6
Problématique ... 6
Objectifs ... 7
Objectif 1. Développement de nouveaux marqueurs microsatellites pour le grand brochet et test sur les autres ésocidés (chapitre 1) ... 8
Objectif 2. Mise au point de 22 marqueurs microsatellites pour le grand brochet (chapitre 1) ... 8
Objectif 3. Étude de la structure de populations sur l’ensemble du système (chapitre 2) ... 8
Objectif 4. Caractérisation de l’habitat du système lac Ontario – fleuve Saint-Laurent (chapitre 2) ... 9
Objectif 5. Analyse de génétique du paysage (chapitre 2) ... 9
CHAPITRE 1 : Novel microsatellite markers for northern pike (Esox lucius) and cross-amplification across all Esox species ... 11
Résumé ... 13
Abstract ... 14
Acknowledgements ... 17
CHAPITRE 2 : Spatially variable extent of population structure in northern pike (Esox lucius) explained by landscape features ... 21
viii
Résumé ... 23
Abstract ... 25
Introduction ... 26
Material and methods ... 29
Sample collection ... 29
Genetic data ... 29
Intra-population diversity and structure ... 30
Population structure ... 30
Landscape genetics ... 31
Results ... 33
Intra-population diversity and structure ... 33
Population structure ... 33
Landscape genetics ... 35
Discussion ... 36
Landscape genetics ... 36
Population structure and genetic diversity ... 39
Acknowledgements ... 43 Conclusion générale ... 59 Bibliographie ... 63 Annexe 1. ... 72 Annexe 2. ... 73 Annexe 3. ... 75 Annexe 4. ... 78
ix
Liste des tableaux
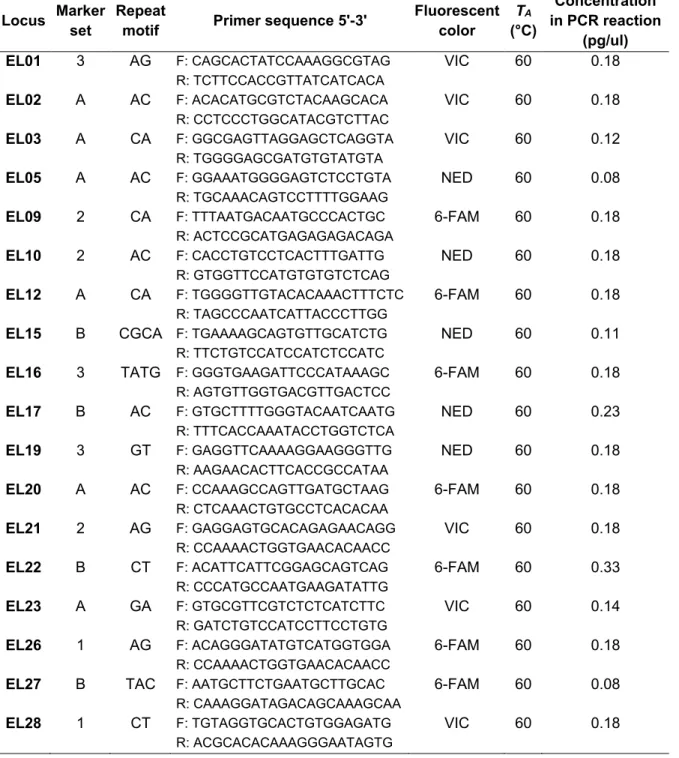
Table 1.1. Microsatellite markers amplification characteristics. Marker sets A and B were amplified in multiplexes and sets 1-3 were amplified independently then pooled for electrophoresis ... 18 Table 1.2. Microsatellite markers characteristics in Esox lucius from North America and other Esox species ... 19 Table32.1. Sample statistics related to each sampling site and analyzed at 22 microsatellite loci.. . 44
Table42.2. Microsatellite loci characteristics. ... 47 Table52.3. Environmental variables included in landscape genetics analyses. Within description is specified the treatment of variables to build distance matrices ... 49 Table62.4. Mantel tests results of pairwise Fst matrix coupled with environmental variables
matrices and identification of variables selected for subsequent multivariate analysis ... 51 Table72.5. Multiple regression on distance matrices (MRM) model on environmental variables results with Pearson’s correlation coefficient and pairwise Fst matrix as a response variable. A) Full
model on selected variables by Mantel's tests (see Table 1). B) Final model with significant
xi
Liste des figures
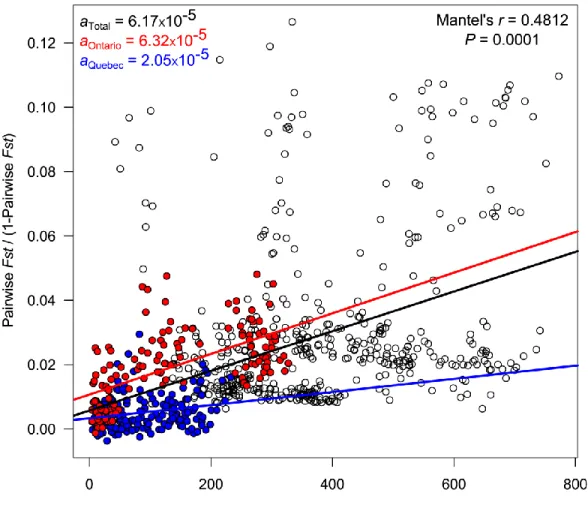
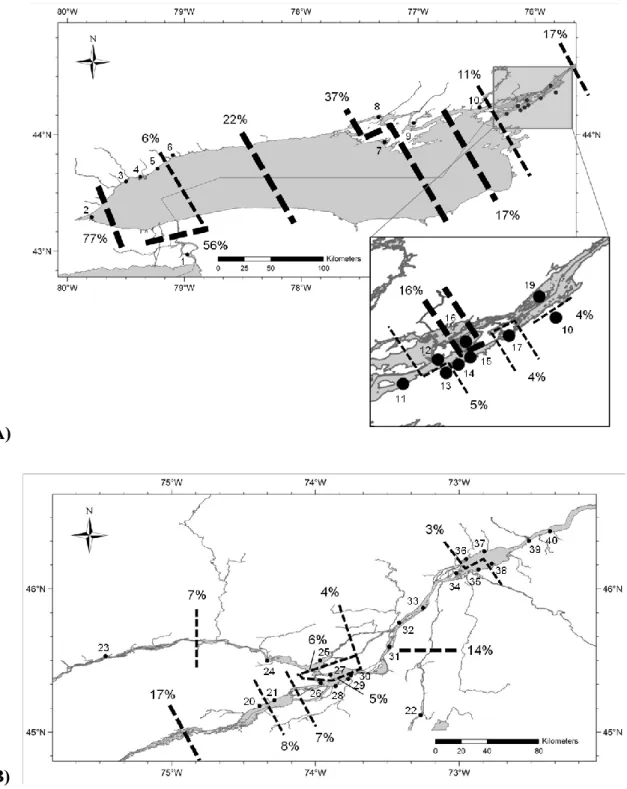
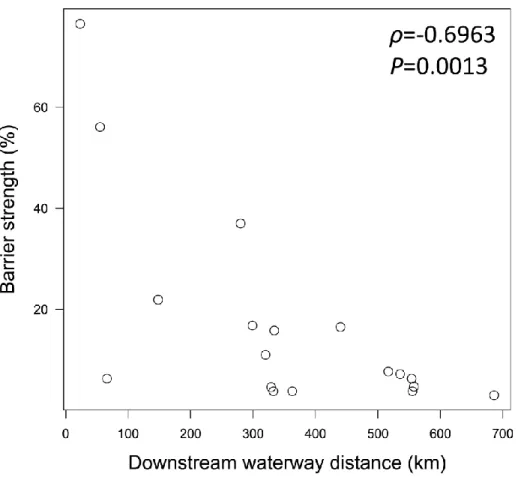
Figure 2.1. Sampling sites in St. Lawrence River and two of its tributaries (Ottawa River and Richelieu River), as well as Lake Ontario and one of its tributary (Niagara River). ... 53 Figure 2.2. Isolation by distance relationships. ... 54 Figure 2.3. A) First 20 barriers in Ontario sector and in B) Quebec sector inferred by BARRIER. Lines thickness is function of barriers’ relative strength (indicated in percentage) ... 55 Figure 2.4. Distribution of the 20 first barriers localization within Lake Ontario and St. Lawrence River system as a function of their relative strength and waterway distance from the upstream farthest point (Hamilton basin, site 2). ... 56 Figure 2.5. Sliding windows of sites located in a 50 km window that moves following water flow across system with a 25 km step. A) Mean inter-annual water level variation coefficients and B) mean pairwise Fst values between sites as a function of middle downstream waterway distance from
the upstream farthest point (Hamilton basin, site 2) of each window. Mean pairwise Fst values
between sites as a function of mean inter-annual water level variation coefficients for C) above and D) below Moses-Saunders regulation dam. ... 57 Figure 2.6. Modelled suitable spawning habitats for northern pike within Lake St. Pierre in lower St. Lawrence River for two extreme water levels scenarios, A) in 1965 where water level was low and B) in 1997 where water level was high. Blue lines and red lines illustrate flood plain upper limit at maximum and minimum water levels, respectively. Green color indicates suitable spawning habitats, from high quality habitats in dark from low quality habitats in light. C) Overlap of
xiii
Liste des abréviations et des sigles
AMOVA Analyse de la variance moléculaire (analysis of molecular variance)
Apr Richesse allélique privée moyenne sur tous les loci standardisée pour la plus petit taille d’échantillonnage (mean private allelic richness)
Ar Richesse allélique moyenne sur tous les loci standardisée pour la plus petit taille d’échantillonnage (mean allelic richness)
b Coefficient standard de régression partielle du modèle MRM (standard partial regression coefficient)
EST Marqueurs de séquences exprimées (expressed sequence tags)
F Valeur du pseudo-test de Fisher du modèle MRM (Fisher's pseudo-test value) F Amorce « sens » (foward primer)
FDR « False discovery rate »
Fis Coefficient de consanguinité (inbreeding coefficient) Fst Indice de différenciation (differentiation coefficient) HE Hétérozygotie attendue (expected heterozygozity) HO Hétérozygotie observée (observeded heterozygozity)
ρ Coefficient de corrélation de Spearman (Spearman’s correlation coefficient) HWE Équilibre de Hardy-Weinberg (Hardy-Weinberg equilibrium)
IBD Isolation par la distance (isolation-by-distance) LD Déséquilibre de liaison (linkage disequilibrium)
MRM Régression multiple sur matrices de distance (multiple regression on distance matrices)
NA Nombre d’allèles par locus (number of alleles)
PCR Réaction de polymérase en chaîne (polymerase chain reaction)
r Coefficient de corrélation du test de Mantel (Mantel’s correlation coefficient) R Amorce « anti-sens » (reverse primer)
R2 Coefficient de détermination multiple du modèle MRM (coefficient of
multiple determination)
xv À mes parents, Françoise et Bernard
xvii
Remerciements
En premier lieu, je tiens à remercier Louis Bernatchez, mon directeur, pour la confiance qu’il m’a démontrée en me laissant mener à terme ce magnifique projet. Il a m’a procuré un soutien constant en démontrant de la compréhension et de la patience tout au long de mon cheminement d’apprentissage. Je remercie aussi Marc Mingelbier, mon co-directeur, qui travaille au sein du Ministère du Développement durable, de l’Environnement, de la Faune et des Parcs du Québec (MDDEFP). Il m’a donné l’opportunité de collaborer avec le Ministère sur un projet de grande envergure. Je lui suis très reconnaissante pour tout son travail de coordination de la campagne d’échantillonnage. Je remercie également Frédéric Lecomte, du MDDEFP, pour son assistance dans mon apprentissage des méthodes de travail efficaces et pour son implication dans nos brainstorms d’idées. Je remercie Charles Perrier, candidat post-doctoral au laboratoire Bernatchez, pour son temps donné avec générosité, sa passion scientifique et sa vision originale qui m’ont permis de mieux comprendre et d’enrichir ce projet. Je remercie chaleureusement Guillaume Côté, professionnel de recherche au laboratoire Bernatchez, pour sa patience sans limites et son soutien apporté dans mon apprentissage au laboratoire et pour son support moral auquel je dois beaucoup. Merci à Lucie Papillon pour son aide et ses conseils techniques sans failles. Je remercie également Émilie Leclerc, diplômée à la maîtrise du laboratoire Bernatchez, pour le généreux partage de son savoir. Un grand merci à Mélanie Dionne du MDDEFP pour ses judicieux conseils et son enthousiasme face au projet.
Pour leur support dans l’obtention des échantillons de grand brochet du système lac Ontario – fleuve Saint-Laurent, merci à Philippe Brodeur, Yves Mailhot, Chantal Côté, Pierre Dumont, Daniel Hatin, Mélissa Larochelle et à l'essentielle équipe des techniciens (William Cayer-Blais, Yanick Soulard, Geneviève Richard, Nicolas Harnois, Vanessa Cauchon), tous du MDDEFP. Merci à John M. Farrell et Chris Barry du SUNY College of Environmental Science and Forestry, Gavin Christie, Alastair Mathers, Chris Wilson et Jim Hoyle du Ontario Ministry of Natural Resources (OMNR), Ian Kelsey et Jeff McNiece du Central Lake Ontario Conservation Authority (CLOCA), Susan Doka et Nicholas Mandrak du Departement of Fisheries and Oceans Canada (DFO), Rodger Klindt du New York State Department of Environmental Conservation (NYS DEC) ainsi qu’aux pêcheurs
xviii
commerciaux et aux pourvoyeurs qui ont généreusement collaboré dans la campagne d’échantillonnage. Merci à tous les autres qui ont mis la main à la pâte au cours de la campagne d’échantillonnage; vous êtes si nombreux que je ne peux tous vous nommer. Pour le partage d’échantillons de tissus des espèces d’ésociés, je remercie grandement Julien April du laboratoire Bernatchez, Michael Hansen (Aarhus University), Chenghui Wang (Shanghai Ocean University), Gregory Maes (KU Leuven) et Andres Lopez (University of Alaska Museum).
Pour la partie préliminaire du développement des amorces et l’annotation des marqueurs, un grand merci à Eric Normandeau, professionnel de recherche au laboratoire Bernatchez. Je remercie chaleureusement Marie-Hélène Perreault pour son enrichissante collaboration dans le travail de laboratoire. Je suis reconnaissante envers sa contagieuse bonne humeur et son travail acharné et impeccable duquel j’ai énormément appris. Merci Marie-Hélène, ce fut un immense plaisir. Je remercie également Amélie Gilbert, stagiaire technicienne finissante, pour son aide apportée au travail de laboratoire. Je remercie Geneviève Richard, technicienne au MDDEFP, pour la production de cartes de la localisation des habitats de fraie du grand brochet (Figure 6 au chapitre 2). Je remercie Sébastien Boutin, étudiant au doctorat du laboratoire de Nicolas Derome, pour son assistance statistique et son soutien d’analyse informatique en plus de ses commentaires sur ce mémoire. Merci à Glenn Yannick, stagiaire post-doctoral au laboratoire Bernatchez et à Antoine Bernatchez pour l’aide à la programmation informatique. Merci à Vicky Albert du MDDEFP pour ses conseils sur la standardisation du génotypage. Je remercie Eliane Valiquette, Vincent Bourret, Pierre-Alexandre Gagnaire, Caroline Côté, Bérénice Bougas, Scott Pavey, Anne-Marie Dion-Côté, Fabien Lamaze et tous les autres membres rencontrés au laboratoire Bernatchez pour les échanges enrichissants. Je remercie Julie Turgeon, Julian Dodson et Nadia Aubin-Horth pour leur participation à mes comités d’évaluation et pour leurs précieux commentaires sur le projet.
Finalement, je remercie les sources de financement de ce projet, soit le Conseil de Recherches en Sciences Naturelles et en Génie du Canada (CRSNG) et le MDDEFP. Je remercie également mes sources de financement personnelles : le CRSNG, le Fonds de
xix Recherche du Québec Nature et Technologies (FQRNT), le Fonds de soutien à la Maîtrise du Département de Biologie de l’Université Laval et Québec-Océan.
Sur une note plus personnelle, je remercie mes parents Françoise et Bernard pour leur support inconditionnel apporté tout au long de cette aventure. Vous formez la meilleure équipe! Enfin, je remercie mes amis et amies passionnés qui se sont intéressés au déroulement de ce projet. Merci pour votre énergie positive !
xxi
Avant-propos
Cette aventure a pris racine dans ma participation au travail de terrain du projet de la truite arc-en-ciel au Québec mené par Isabel Thibault au sein du laboratoire du Dr Julian Dodson à l’Université Laval. C’est au cours de cet été que j’ai été piquée d’un amour pour les poissons et que j’ai décidé de m’investir dans la protection de la faune aquatique. Ainsi, après avoir travaillé quelques mois dans l’environnement du laboratoire du Dr Louis Bernatchez, je me suis résolue à demander à ce dernier s’il était prêt à me confier un projet de maîtrise appliqué à la conservation dans son laboratoire. C’est à mon grand plaisir qu’il m’a proposé de réaliser ce projet de recherche en collaboration avec le Ministère des Ressources Naturelles et de la Faune du Québec (MRNF), maintenant actif sous le nom de Ministère du Développement durable, de l’Environnement, de la Faune et des Parcs du Québec (MDDEFP).
Ce mémoire est l’aboutissement de trois enrichissantes années d’apprentissage de méthodes de travail efficaces et d’organisation et de gestion de temps, en plus de l’accroissement d’une maturité scientifique et de l’acquisition de connaissances variées. Ce document inclut deux chapitres présentés sous la forme d’articles scientifiques (en anglais) dont je suis l’auteure principale. Cette étude porte sur la caractérisation génétique des populations de grand brochet (Esox lucius) du système du lac Ontario et du fleuve Saint-Laurent situé au nord-est de l’Amérique du Nord.
Dans le contexte du premier chapitre, de nouveaux marqueurs microsatellites pour le grand brochet furent développés à l’aide de marqueurs de séquences exprimées (Expressed Sequence Tags, EST) et des tests d’amplification croisée de ces marqueurs furent appliqués sur les autres espèces d’ésocidés (genre Esox). Cet article scientifique a été co-écrit avec mon directeur Louis Bernatchez, Eric Normandeau qui est professionnel de recherche au laboratoire Bernatchez et mon co-directeur Marc Mingelbier du Ministère du Développement durable, de l’Environnement, de la Faune et des Parcs (MDDEFP). Eric Normandeau a effectué les analyses informatiques du développement de 28 nouvelles paires d’amorces et il a accompli les tests préliminaires d’amplification sur le grand brochet et dirigé l’annotation de ces marqueurs. J’ai ensuite réalisé des tests approfondis d’amplification de ces 28 marqueurs sur le grand brochet et les autres espèces d’ésocidés.
xxii
Par la suite, j’ai effectué le génotypage de ces marqueurs et les analyses statistiques correspondantes. Enfin, j’ai rédigé l’article scientifique avec la collaboration des co-auteurs, lequel sera soumis sous peu au journal Conservation Genetics Resources.
Ces microsatellites ont été ajoutés aux marqueurs préexistants dans la littérature pour former une banque dans laquelle nous avons sélectionné les marqueurs les plus appropriés pour réaliser les analyses du deuxième chapitre. À proprement parler, ce chapitre porte sur la caractérisation génétique des populations de grand brochet (Esox lucius) et l’étude de ses déterminismes environnementaux dans le système du lac Ontario et du fleuve Saint-Laurent. Cet article scientifique a été co-écrit avec mon directeur Louis Bernatchez, mon co-directeur Marc Mingelbier et Frédéric Lecomte du MDDEFP. Dans un premier temps, avec la concertation du MDDEFP et de mon directeur, j’ai élaboré les objectifs du projet. Ensuite j’ai établi des hypothèses de recherche en accord avec ma recherche bibliographique. Marc Mingelbier a planifié et coordonné la majeure partie de la campagne d’échantillonnage et j’ai coordonné et exécuté une petite partie de cette dernière. J’ai effectué le travail de laboratoire avec l’assistance de Marie-Hélène Perrault et Amélie Gilbert. J’ai effectué la totalité des analyses statistiques et j’ai rédigé l’article scientifique avec la collaboration des co-auteurs, lequel sera soumis prochainement au journal Transactions of the American Fisheries Society.
En résumé, dans ce mémoire sont insérés deux chapitres qui présentent le format typique d’articles scientifiques en langue anglaise. Ces chapitres sont précédés d’une introduction et ils sont suivis d’une conclusion en langue française pour former le corps du mémoire.
1
Introduction générale
Concepts
Conservation et diversité génétique
La biologie de la conservation fait la promotion du maintien de la biodiversité à plusieurs niveaux, de la diversité des écosystèmes jusqu’à la diversité génétique (Primack 2006). La diversité génétique est ainsi perçue comme le matériel brut de l’évolution adaptative (McNeely et al. 1990). La conservation de la diversité génétique d’une espèce ou d’une population lui confère l’opportunité de maintenir son potentiel évolutif, i.e. sa capacité à persister par adaptation en réponse aux variations environnementales de son milieu (Frankham et al. 2010), telles que les changements climatiques ou des modifications anthropogéniques apportées à l’habitat. Ainsi, dans le domaine de la conservation biologique, la variation génétique a été élevée à un statut prioritaire et a été reconnue par l’IUCN (World Conservation Union) comme l’une des trois composantes de la diversité biologique à conserver (McNeely et al. 1990).
Dans le but de préserver la biodiversité et de diminuer le taux d’extinction actuel, la conservation de la diversité génétique utilise des outils moléculaires pour mettre en œuvre des plans de gestion permettant aux espèces ou aux populations, qui se comportent comme des entités dynamiques et distinctes, de s’adapter aux changements environnementaux (Frankham et al. 2010). La démarche globale consiste à estimer l’ampleur de la divergence entre les populations en quantifiant la diversité génétique intra- et inter-populationnelle dans l’optique de déterminer l’organisation hiérarchique de la diversité génétique des populations d’une espèce. Ce processus permet d’identifier et de localiser les unités évolutives significatives (ESU : evolutionary significative units, Ryder, 1986) et/ou les unités de gestion (MU : management units, Palsbøll et al., 2007). Dans un contexte d’exploitation durable des espèces, comme par exemple dans le contexte des pêcheries, un des principaux enjeux de gestion est l’identification des populations locales (Carvalho & Hauser 1995) et la compréhension de l’influence des processus génétiques et écologiques sur cette structure (Manel et al. 2003). Idéalement, dans un système donné pour une espèce donnée, les unités populationnelles devraient correspondre aux unités de gestion (dénommées « unités de stock » en gestion des pêcheries) qui correspondent aux groupes
2
de poissons exploités dans une région spécifique ou alors par une méthode spécifique (Carvalho & Hauser 1995; Reiss et al. 2009). Dans l’élaboration des plans de gestion, l’étude de la structure génétique des populations permet donc la définition de ces unités qui sont utilisées dans l’établissement des priorités des mesures de préservation de la diversité génétique entre les populations (Lande 1988; Waples 1989; Moritz 1994; Fraser & Bernatchez 2001). Cette étape est primordiale dans le cas des populations exploitées, car elle permet de mieux prévoir les conséquences des interventions humaines sur la diversité génétique.
Structure génétique des populations
Les individus d’une espèce sont généralement regroupés en populations locales par un processus d’isolement reproducteur partiel où ces populations sont soumises aux quatre forces évolutives ayant une influence sur leur composition génétique de façon indépendante. La mutation, la dérive génique et la sélection directionnelle naturelle (qui favorise l’adaptation locale aux conditions environnementales) mènent à la différenciation génétique des populations locales tandis que le flux génique (causé par la migration) et la sélection balancée s’opposent à cette différenciation (Slatkin 1987). Notamment, le flux génique tend à homogénéiser les fréquences alléliques entre les populations et son intensité détermine les effets relatifs de la sélection et de la dérive génique (Balloux & Lugon-Moulin 2002). Ces forces exercent des pressions de différentes intensités entre les groupes reproductifs et elles engendrent ainsi des compositions génétiques distinctes pour chacune des populations. Lors d’une étude de structure génétique de populations, c’est cette variation génétique entre les groupes qui permet de discriminer les différentes populations, qui, par définition, présentent une connectivité limitée entre elles.
La connectivité entre les populations est déterminée par la dispersion (le mouvement d’individus entre l’aire natale et l’aire ou prend place la première reproduction ou le mouvement entre deux aires de reproduction successives) des individus émigrants et immigrants. Les barrières environnementales, les processus historiques et les traits d’histoire de vie (ex : stratégies de reproduction) peuvent moduler l’intensité du flux génique (transfert de matériel génétique entre les populations par la reproduction d’individus immigrants avec les individus résidents) entre les populations et influer sur le
3 patron et la magnitude de la structure génétique des populations (Balloux & Lugon-Moulin 2002). Également, les populations sont souvent génétiquement différenciées selon le principe d’isolation par la distance, car l’étendue de la répartition des espèces est généralement plus large que la capacité de dispersion d’un individu. Les populations à proximité sont génétiquement davantage similaires que les populations plus distantes (Balloux & Lugon-Moulin 2002), comme par exemple selon le modèle stepping-stone (Kimura & Weiss 1964).
Génétique du paysage
L’étude classique de la structure génétique des populations soulève un questionnement sur les mécanismes de structuration de la diversité génétique des populations. La « génétique du paysage », ou landscape genetics (Manel et al. 2003), permet de déterminer l’ampleur et les causes de la distribution de la variation génétique spatio-temporelle et d’effectuer la correspondance entre la structure génétique des populations et l’hétérogénéité environnementale. La génétique du paysage est une discipline née d’un amalgame entre la structure génétique des populations, l’écologie du paysage (étude des interactions entre les patrons spatiaux et les processus écologiques) et des analyses spatiales (Manel et al. 2003; Storfer et al. 2007). Cette nouvelle aire de recherche permet de tester l’influence relative des variations biotiques et abiotiques des conditions environnementales du milieu sur le flux génique, les discontinuités génétiques (Guillot et al. 2005) et la structure génétique des populations (Manel et al. 2003; Holderegger & Wagner 2006, 2008) et de déterminer l’échelle à laquelle ils agissent (Gaggiotti et al. 2009).
La génétique du paysage est devenue un outil d’investigation dans l’étude des processus biologiques évolutifs fondamentaux tels la dynamique des métapopulations et les éléments écologiques sous-jacents à la divergence des populations (Storfer et al. 2007). En outre, la génétique du paysage peut s’appliquer à l’identification des barrières anthropogéniques qui diminuent le flux génique ou la diversité génétique, à prédire les conséquences des alternatives de gestion proposées sur la variation génétique et la connectivité des populations et à identifier les corridors biologiques potentiels pour l’implantation de réserves écologiques (Storfer et al. 2007). Les études de génétique du paysage en milieu aquatique sont de plus en plus nombreuses mais demeurent cependant davantage centrées
4
sur de petits réseaux ramifiés de ruisseaux avec leurs tributaires. Par conséquent, le projet présenté ici, appliquant l’approche de la génétique du paysage à l’étude du grand brochet (Esox lucius) dans le système lac Ontario – fleuve Saint-Laurent, représentera une des plus importantes études de structuration génétique chez le grand brochet réalisée à ce jour et une des études les plus élaborées de la génétique du paysage en milieu aquatique d’eau douce.
Le grand brochet
Le grand brochet, Esox lucius Linnaeus 1758, est une espèce de poisson d’eau douce appartenant à la famille des ésocidés qui présente une répartition circumpolaire dans l’hémisphère Nord (Craig 1996). Le grand brochet se retrouve dans 45% des eaux douces d’Amérique du Nord (Carlander et al. 1978) et est ainsi considéré comme ubiquitaire sur le continent (Craig 1996). Dans la plupart des pays, le grand brochet a une valeur commerciale et récréative (Crossman & Casselman 1987; Raat 1988).
Biologie et écologie de l’espèce
Le grand brochet est un prédateur-clé piscivore commun des eaux peu profondes des systèmes d’eau douce (Craig 1996, 2008). Les principaux critères de sélection de l’habitat du grand brochet sont la température de l’eau (modérément élevée), la densité de végétation (modérément dense), la concentration en oxygène dissous (élevée pour température), la profondeur de l’eau (modérément faible) et la turbidité (faible) (Casselman & Lewis 1996). Cependant, l’espèce peut tolérer une grande diversité de conditions environnementales. En effet, elle présente une grande variation en ce qui a trait aux traits d’histoire de vie, ce qui pourrait témoigner d’adaptations locales. Le grand brochet tient un rôle important dans la structuration des communautés aquatiques ; entre autres, il régule fortement la composition, l’abondance et la distribution de plusieurs espèces de poissons, incluant ses conspécifiques, par la prédation, la compétition et le cannibalisme (revu par Craig 2008).
Le grand brochet atteint la maturité sexuelle entre deux et six ans, à une taille se situant entre 26 et 51 cm (Craig 1996). Cette espèce fraye hâtivement au printemps (vers le mois d’avril (Craig 1996), pendant la période de crue, dans les milieux humides inondés. La période de fraye s’étale sur approximativement deux semaines, avec un pic de fraye de un à deux jours. Le brochet montre une préférence de fraye dans les eaux peu profondes à faible
5 courant, où la température augmente plus rapidement qu’ailleurs dans le fleuve et où la végétation est prépondérante (Mingelbier et al. 2008).
Des évidences pointent vers une certaine fidélité du brochet au site natal (philopatrie) et au site de reproduction selon des études de génétique des populations (Miller et al. 2001), de travaux de télémétrie (Koed et al. 2006) et de capture-marquage-recapture (Karås & Lehtonen 1993; revu par Craig 2008). Bosworth et Farrell (2006) ont cependant soulevé que le degré de fidélité au site natal de fraye demeure indéfini, mais ils ont identifié une divergence génétique significative entre des sites de forte proximité. De plus, le grand brochet ne semble pas se disperser à grande échelle et semble plutôt utiliser le site de reproduction adéquat le plus près (revu par Craig 2008). Des études en rivière sur le mouvement des individus supportent l’hypothèse que le brochet serait principalement sédentaire (mais non exclusivement) et n’effectue pas de mouvements sur de grandes distances (Craig 1996, 2008; Rosell & MacOscar 2002; Koed et al. 2006; Vehanen et al. 2006). Koed et ses collaborateurs (2006) ont rapporté que les femelles et les individus de plus grande taille parcourent les plus grandes distances. La dispersion des larves pourrait être considérée comme négligeable, puisque les larves se fixent généralement à la végétation des sites de fraye (revu par Craig 1996). À une taille d’environ 20 mm seulement, les jeunes brochets migrent de l’aire de ponte vers des milieux à végétation plus éparse (Franklin & Jr 1963; Massé & Dumont 1991; revu par Craig 1996) et quittent les aires de fraye qui commencent à s’assécher. Ainsi, les jeunes de l’année peuvent se disperser à petite échelle (Cucherousset et al. 2009). Cependant, aucune étude n’a directement investigué leurs déplacements sur une période s’étalant sur plus de quelques semaines. En somme, certaines évidences d’un comportement de philopatrie ainsi qu’une faible dispersion apparente témoignent d’une probable connectivité limitée entre les populations et ceci indiquerait donc la nécessité de planifier la gestion du brochet en considérant les stocks distincts (Craig 2008).
Bilan des connaissances en génétique des populations
Le grand brochet présente une forte différenciation génétique entre les populations à grande échelle géographique, mais, parallèlement, présente de faibles niveaux de polymorphisme, même à des loci microsatellites (Senanan & Kapuscinski 2000; revu par Miller & Senanan
6
2003). La forte différenciation génétique et la faible variabilité génétique au sein de l’espèce a été attribuée à 3 éléments non mutuellement exclusifs : 1) le flux génique restreint entre les populations de différentes masses d’eau ; 2) de faibles tailles effectives de population (dues à son statut de grand prédateur) combinées à de faibles ratios de taille effective par rapport à la taille réelle des populations (Ne/N) ; 3) des goulots d’étranglement survenus durant la dernière glaciation et des effets fondateurs associés à la recolonisation postglaciaire, surtout en Amérique du Nord (Miller & Kapuscinski 1997; Senanan & Kapuscinski 2000; Miller & Senanan 2003; Maes et al. 2003; revu par Jacobsen et al. 2005).
Système à l’étude
L’aire d’étude correspond à la portion nord du lac Ontario et au Saint-Laurent fluvial. Le système présente des variations interannuelles du débit en fonction des conditions climatiques (Mingelbier et al. 2004). La plaine inondable du système est sensible aux variations du débit et donc aux variations du niveau d’eau (Mingelbier et al. 2004). La répartition géographique des sites potentiels de fraye du grand brochet dans le système correspond de manière globale à la surface de la plaine d’inondation (Mingelbier et al. 2004, 2008). Il y a coïncidence de la période de reproduction du grand brochet avec la crue printanière du système, ce qui permet au brochet d’utiliser la plaine d’inondation comme habitat de fraye (Jude & Pappas 1992; Mingelbier et al. 2004). Le niveau d’eau du système en période de crue contrôle l’accès des individus reproducteurs aux frayères de la plaine inondable et module la superficie disponible des habitats de reproduction du grand brochet (Mingelbier & Morin 2005). Également, après la fraye, le niveau de l’eau module le degré d’immersion des œufs et des larves (Mingelbier et al. 2008), donc détermine la survie des premiers stades de vie. Le retrait printanier des eaux peut donc causer une forte mortalité quand les embryons et les larves se retrouvent en milieu asséché (Dumont & Fortin 1977; Fortin et al. 1982)
Problématique
Depuis le milieu du XIXe siècle, le long du fleuve Saint-Laurent et des Grands Lacs, les activités anthropiques ainsi que la diminution du niveau d’eau du fleuve ont causé la destruction de plus de 70% des milieux humides (Höök et al. 2001) qui correspondent à
7 l’habitat de reproduction du grand brochet. Également, le régime hydrologique du fleuve subit des variations spatio-temporelles, selon ses différents secteurs et suivant le climat (Mingelbier et al. 2004). Lors des événements de faible hydraulicité suivant plusieurs années consécutives, on dénote une perte notable de superficie des habitats printaniers du grand brochet (jusqu’à 17% par année dans le fleuve Saint-Laurent, Mingelbier et al. 2004) et les impacts associés sont cumulatifs. Cette perte d’habitat peut devenir limitante pour la fraye du grand brochet dans les secteurs plus sensibles aux variations du débit (Mingelbier et al. 2004) qui sont davantage sujets à une plus forte variation interannuelle du niveau d’eau. Pour mieux comprendre les conséquences de cette fluctuation spatio-temporelle de l’habitat de reproduction sur les populations de grand brochet dans le système, l’étude de l’impact de la variation du niveau d’eau sur la structure des populations de grand brochet devient pertinente relativement à d’autres facteurs environnementaux susceptibles d’affecter cette structure. Aucun plan de gestion spécifique n’existe actuellement pour le grand brochet dans le fleuve Saint-Laurent et le lac Ontario. Une étude précédente a révélé que la structure de populations de la perchaude (Perca flavescens) du fleuve Saint-Laurent ne correspondait pas exactement au découpage hydrologique utilisé dans le cadre de gestion de celle-ci (Leclerc et al. 2008). Conséquemment, il était important de délimiter les populations du grand brochet du système lac Ontario – fleuve Saint-Laurent et de caractériser leur connectivité en vue de mettre en pratique une meilleure gestion de l’espèce dans le système.
Objectifs
L’objectif général du projet consistait en une analyse génétique du grand brochet (Esox lucius) à l'échelle du paysage dans le système lac Ontario – fleuve Saint-Laurent dans le dessein de déterminer l'extension des différentes populations de cette espèce ainsi que l’ampleur de la connectivité entre celles-ci, en plus de viser à comprendre l’influence de l’hétérogénéité environnementale sur la structure génétique observée en utilisant une approche de génétique du paysage. Le but ultime de l’étude était de permettre la planification de la gestion des populations de grand brochet en considérant les secteurs du système limitants pour la reproduction à l’aide d’unités de gestion pertinentes.
8
Objectif 1. Développement de nouveaux marqueurs microsatellites pour le grand brochet et test sur les autres ésocidés (chapitre 1)
Comme le grand brochet démontre une faible variation génétique aux marqueurs microsatellites préexistants dans la littérature (revu dans Miller & Senanan 2003), nous visions dans un premier temps à développer de nouveaux marqueurs dans le but d’enrichir la banque actuelle. Afin de déterminer si ces marqueurs étaient potentiellement utilisables lors de futures études génétiques des autres espèces d’ésocidés, ces marqueurs furent testés chez les cinq autres espèces du genre Esox, soit le brochet de l’Amur (E. reichertii) d’Eurasie, le brochet d’Amérique (E. americanus americanus), le brochet vermiculé (E. americanus vermiculatus), le maskinongé (E. masquinongy) et le brochet maillé (E. niger) d’Amérique du Nord. Également, ces marqueurs furent testés chez le grand brochet d’Europe.
Objectif 2. Mise au point de 22 marqueurs microsatellites pour le grand brochet (chapitre 1)
À partir de la banque de marqueurs bonifiée lors de la réalisation du premier objectif, ce second objectif visait la mise au point le développement et l’optimisation de kits de marqueurs microsatellites polymorphes pour le grand brochet dans le but de les utiliser dans cette étude comme unité de base de la mesure de la variation génétique. L’utilisation de marqueurs microsatellites fut préconisée pour leur neutralité et leur degré relativement élevé de polymorphisme (par rapport à l’ensemble des marqueurs disponibles ; Wan et al. 2004) et leur caractérisation selon un protocole d’analyse fiable et rapide (Chistiakov et al. 2006). Également, puisque le grand brochet est une espèce présentant un faible niveau de polymorphisme (revu dans Miller & Senanan 2003), l’utilisation d’un grand nombre de marqueurs a été privilégiée pour l’obtention d’un bon pouvoir de résolution dans les analyses statistiques.
Objectif 3. Étude de la structure de populations sur l’ensemble du système (chapitre 2)
Le troisième objectif était de documenter la structure de populations du grand brochet sur l’ensemble du système lac Ontario – fleuve Saint-Laurent en réalisant une analyse de la variation génétique observée au niveau des 22 marqueurs microsatellites. La diversité génétique du grand brochet dans le système a été caractérisée et l’importance de l’isolation
9 par la distance a été évaluée. La détermination de la localisation spatiale de potentielles barrières au flux génique fut effectuée afin de définir la connectivité des populations. L’ampleur de la différenciation génétique des différentes localités du système a été comparée à celle de sites externes géographiquement connectés, incluant certains tributaires du fleuve Saint-Laurent (rivière Richelieu et rivière des Outaouais) et du lac Ontario (Rivière Niagara). Notre prédiction stipulait une faible différenciation génétique retrouvée dans le système à l’instar de celle retrouvée chez le grand brochet dans des systèmes hautement connectés.
Objectif 4. Caractérisation de l’habitat du système lac Ontario – fleuve Saint-Laurent (chapitre 2)
L’objectif suivant consistait à construire une base de données géospatiales détaillée des caractéristiques abiotiques de l’environnement sélectionnées pour l’étude de génétique du paysage du grand brochet dans la section aval du fleuve Saint-Laurent (secteur du Québec), de l’extrémité ouest du lac Saint-François jusqu’aux environs de Trois-Rivières.
Objectif 5. Analyse de génétique du paysage (chapitre 2)
Le dernier objectif consistait à identifier les facteurs environnementaux qui interagissent pour façonner le patron et la magnitude de la structure génétique du grand brochet dans le fleuve Saint-Laurent dans le but de vérifier l’importance relative de la variation du niveau d’eau dans l’explication de la structure de populations en lien avec les autres facteurs. Cette analyse a été réalisée à l’échelle de la section aval du fleuve Saint-Laurent (secteur du Québec) seulement puisque les données environnementales disponibles n’étaient pas aussi détaillées pour le lac Ontario que pour cette région, et aussi à cause de la présence d’un grand trou d’échantillonnage entre la section amont et la section aval du fleuve Saint-Laurent, ce qui est inapproprié pour les analyses de génétique du paysage (Manel et al. 2003). Selon nos prédictions, notamment, l’hypothèse centrale impliquait dans les secteurs davantage sujets à de fortes variations interannuelles du niveau une plus faible différentiation génétique causée par la perturbation de la dynamique de reproduction. Également, il était attendu que la différentiation génétique soit plus élevée de part et d’autre des différents barrages présents dans le système. Finalement, on s’attendait à ce que la
10
présence de différentes masses d’eau aux caractéristiques physicochimiques divergentes coïncide avec une différenciation génétique plus élevée.
11
CHAPITRE 1 : Novel microsatellite markers for
northern pike (Esox lucius) and cross-amplification
across all Esox species
Geneviève Ouellet-Cauchon1*, Eric Normandeau1, Marc Mingelbier2 and Louis Bernatchez1
1 Université Laval, Institut de Biologie Intégrative et des Systèmes (IBIS), 1030 Avenue de la
Médecine, Québec (Québec), G1V 0A6, Canada
2 Ministère du Développement durable, de l’Environnement, de la Faune et des Parcs du Québec
(MDDEFP), Service de la Faune Aquatique, 880 chemin Sainte-Foy, Québec (Québec), G1S 4X4, Canada
13
Résumé
Dans le cadre de l’investigation de la génétique du paysage de l’espèce, nous avons développé 17 nouveaux microsatellites dérivés de marqueurs de séquences exprimées (« EST sequences ») chez le grand rochet de l’Amérique du Nord, Esox lucius. Nous avons également testé l’amplification croisée chez les populations européennes de la même espèce de même que chez cinq autres espèces du genre Esox. Sur 30 individus des trois populations nord américaines, une déviation significative de l’équilibre d’Hardy-Weinberg a été détectée mais aucun déséquilibre de liaison entre toutes les paires de loci n’a été observé. Une moyenne de 6,88 allèles par locus a été détectée et une hétérozygotie attendue moyenne de 0,49 a été obtenue, mais la diversité génétique était fortement variable selon les loci (NA s’échelonnait entre deux et 23 allèles et HE variait entre 0,033 and 0,950). Tous les loci furent amplifiés avec succès pour E. lucius d’Amérique du Nord et d’Europe, de même que dans le cas du brochet de l’Amur, E. reichertii. Entre huit et 11 loci furent amplifiés avec succès pour toutes les espèces d’ésocidés et, d’un point de vue général, quatre des 17 loci furent amplifiés avec succès sur toutes les espèces.
Mots-clés : Esox lucius, grand brochet, microsatellites liés aux marqueurs de séquence
14
Abstract
We developed 17 novel microsatellite markers derived from EST sequences in North American northern pike, Esox lucius, in order to investigate the species’ landscape genetics. We also tested cross-amplification on European populations of the same species as well as other (N = 5) Esox species. One significant deviation from Hardy-Weinberg equilibrium was detected out of 30 individuals from three North American populations. However, no linkage disequilibrium among all loci pairs was observed. A mean of 6.88 alleles per locus was detected, with a mean expected heterozygozity of 0.49, and allelic genetic diversity was highly variable across loci (NA ranging between two and 23 alleles and HE between 0.033 and 0.950). All loci were successfully amplified on E. lucius from North America and Europe, as well as in the Amur pike, E. reichertii. Between eight and 11 loci were successfully amplified in other Esox species and, overall, four out of 17 loci were successfully cross-amplified on all species.
15 Northern pike (Esox lucius) is a nearly ubiquitous fish established in most of the northern hemisphere’s freshwater bodies and presents a “least concern” status according to UICN (Freyhof & Kottelat 2008). Nevertheless, this species is heavily exploited by recreational and commercial fisheries in most regions (Craig 1996) and suffers from habitat alterations (Casselman & Lewis 1996). Northern pike populations have been shown to go through a global decline (Casselman & Lewis 1996; Lorenzoni et al. 2002; Westin & Limburg 2002; Launey et al. 2003; Jacobsen et al. 2005; Lucentini et al. 2006, 2009; Smith et al. 2007). To achieve proper conservation and management of that resource, better knowledge of the species’ population genetic diversity and structure must be acquired.
Compared to other fish species, including the European northern pike, North American northern pike presents a distinctly lower level of genetic diversity and polymorphism (DeWoody & Avise 2000). Hence, most microsatellite markers previously developed for Esox lucius lacked polymorphism when used on North American northern pike (Miller & Senanan 2003). To overcome this problem, we developed 17 novel polymorphic microsatellite markers for North American northern pike.
We first developed 17 pairs of primers derived from EST sequences found within the cGRASP EST database (Table 1). Based on blast searches done in the Blast2Go software (Conesa et al. 2005) using the nr blast database, only EL21 had known annotation, which corresponded to the Danio rerio solute carrier organic anion transporter family member 2b1 gene. Microsatellite markers were amplified on 30 E. lucius individuals captured from three locations in St. Lawrence River in North America (Gentilly, 46°24'2"°N, -72°22'8"°W; St-Jean stream, 45°22'11"°N, -73°46'19"°W; Thousand Islands, 44°25'16"°N, -75°51'55"°W). Cross-amplification was tested on all five Esox species (northern pike from Europe [E. lucius, N = 20], Amur pike [E. reichertii, N = 17] from Eurasia, redfin pickerel [E. americanus americanus, N = 10], grass pickerel [E. americanus vermiculatus, N = 10], muskellunge [E. masquinongy, N = 10] and chain pickerel [E. niger, N = 10] from North America). Two markers sets (A and B), which featured respectively six and four markers, were designed and optimized in multiplexes with fluorescent forward primers (Table 1). The 10 other markers (sets 1-2-3) were individually amplified using M13 universal primers (Schuelke 2000) and subsequently pooled in three multiplexes (1 to 3, Table 1) for
16
genotyping. Multiplexed microsatellite markers were amplified using Bio-Rad IQ Supermix and individual M13 markers were amplified using GoTaq Flexi DNA Polymerase. PCR cycles started with a 3 min denaturation step at 95°C, followed by 35 cycles with 30s of denaturation at 95°C, 30s of hybridization at 56 °C or 60°C and 30s of elongation at 72°C, and were completed with a final 30 min elongation step at 60°C. PCR products were pooled with formamide and ROX size standards from Applied Biosystems. The electrophoresis of amplified loci was completed using an Applied Biosystems 3130xl Genetic Analyzer and the raw data were treated with DATA COLLECTION v3.1.1. Genotypes were resolved using GENEMAPPER v.4.1 and automated scoring manual verification. Number of alleles per locus (NA), observed and expected heterozygozity (HE, HO), allele range size, Fis values, linkage disequilibrium (LD) tests and Hardy-Weinberg equilibrium (HWE) tests (both on all the three northern pike populations and over each of them), were computed with GENEPOP v.4.2 (Raymond & Rousset 1995). For multiple tests, Bonferroni corrections were applied (Rice 1989).
For North American E. lucius, allele sizes ranged between 111 and 376 bp (Table 2). Only one significant deviation from HWE (Table 2) was observed for EL16 among the three populations. No significant deviations from HWE or from LD were observed in individual populations. The average level of genetic variation was moderate (mean NA = 6.88, mean HE = 0.49, mean HO = 0.51; Table 2) and highly variable across loci (NA ranged between two and 23 alleles, HE and HO ranged between 0.033 and 0.950 and 0.033 and 0.967, respectively (Table 2). Fis values varied between -0.06 and 0.13, except for EL16 which revealed a strong deficit in heterozygotes with a value of 0.65 (Table 2) and most likely presents null alleles. All loci were successfully amplified in both E. lucius from North America and Europe and E. reichertii from Eurasia (Table 2). Among the other species, the number of successfully amplified loci varied between eight and 11. Moreover the quality of amplification was generally lower than observed in E. lucius and E. reichertii. Overall, four out of 17 loci were successfully cross-amplified on all Esox species for the tested PCR conditions (Table 2). Given the scarcity of microsatellite markers currently available for members of the genus Esox, these new polymorphic microsatellite markers should be very useful to assist the study of lowly polymorphic E. lucius populations in North America and
17 in other regions of the world. These markers could also benefit studies of other Esox species.
Acknowledgements
Funding for this project was provided by the Natural Sciences and Engineering Research Council of Canada (NSERC) collaborative project grant accorded to Louis Bernatchez and from the Ministère du Développement durable, de l’Environnement, de la Faune et des Parcs du Québec (MDDEFP). Funding from NSERC, Fonds de Recherche du Québec Nature et Technologies (FQRNT), Fonds de soutien à la Maîtrise from Département de Biologie of Université Laval and Québec-Océan (QO) financially supported Geneviève Ouellet-Cauchon during her M.Sc. This project is a contribution to the research program of QO. We thank John M. Farrell (SUNY College of Environmental Science and Forestry), Michael M. Hansen (Aarhus University), Julien April (Université Laval), Erling Holm (Royal Ontario Museum), Mike Douglas (Prairie Research Institute), Philippe Brodeur and Suzanne Lepage (MDDEFP), Chenghui Wang (Shanghai Ocean University), Gregory Maes (KU Leuven) and Andres Lopez (University of Alaska Museum) for providing tissue samples. We thank Guillaume Côté, Lucie Papillon, Marie-Hélène Perrault and Amélie Gilbert from Université Laval for laboratory assistance.
18
Table 1.1. Microsatellite markers amplification characteristics. Marker sets A and B were
amplified in multiplexes and sets 1-3 were amplified independently then pooled for electrophoresis. TA: annealing temperature.
Locus Marker set
Repeat
motif Primer sequence 5'-3'
Fluorescent color TA (°C) Concentration in PCR reaction (pg/ul) EL01 3 AG F: CAGCACTATCCAAAGGCGTAG VIC 60 0.18
R: TCTTCCACCGTTATCATCACA
EL02 A AC F: ACACATGCGTCTACAAGCACA VIC 60 0.18 R: CCTCCCTGGCATACGTCTTAC
EL03 A CA F: GGCGAGTTAGGAGCTCAGGTA VIC 60 0.12 R: TGGGGAGCGATGTGTATGTA
EL05 A AC F: GGAAATGGGGAGTCTCCTGTA NED 60 0.08 R: TGCAAACAGTCCTTTTGGAAG
EL09 2 CA F: TTTAATGACAATGCCCACTGC 6-FAM 60 0.18 R: ACTCCGCATGAGAGAGACAGA
EL10 2 AC F: CACCTGTCCTCACTTTGATTG NED 60 0.18 R: GTGGTTCCATGTGTGTCTCAG
EL12 A CA F: TGGGGTTGTACACAAACTTTCTC 6-FAM 60 0.18 R: TAGCCCAATCATTACCCTTGG
EL15 B CGCA F: TGAAAAGCAGTGTTGCATCTG NED 60 0.11 R: TTCTGTCCATCCATCTCCATC
EL16 3 TATG F: GGGTGAAGATTCCCATAAAGC 6-FAM 60 0.18 R: AGTGTTGGTGACGTTGACTCC
EL17 B AC F: GTGCTTTTGGGTACAATCAATG NED 60 0.23 R: TTTCACCAAATACCTGGTCTCA
EL19 3 GT F: GAGGTTCAAAAGGAAGGGTTG NED 60 0.18 R: AAGAACACTTCACCGCCATAA
EL20 A AC F: CCAAAGCCAGTTGATGCTAAG 6-FAM 60 0.18 R: CTCAAACTGTGCCTCACACAA
EL21 2 AG F: GAGGAGTGCACAGAGAACAGG VIC 60 0.18 R: CCAAAACTGGTGAACACAACC
EL22 B CT F: ACATTCATTCGGAGCAGTCAG 6-FAM 60 0.33 R: CCCATGCCAATGAAGATATTG
EL23 A GA F: GTGCGTTCGTCTCTCATCTTC VIC 60 0.14 R: GATCTGTCCATCCTTCCTGTG
EL26 1 AG F: ACAGGGATATGTCATGGTGGA 6-FAM 60 0.18 R: CCAAAACTGGTGAACACAACC
EL27 B TAC F: AATGCTTCTGAATGCTTGCAC 6-FAM 60 0.08 R: CAAAGGATAGACAGCAAAGCAA
EL28 1 CT F: TGTAGGTGCACTGTGGAGATG VIC 60 0.18 R: ACGCACACAAAGGGAATAGTG
19
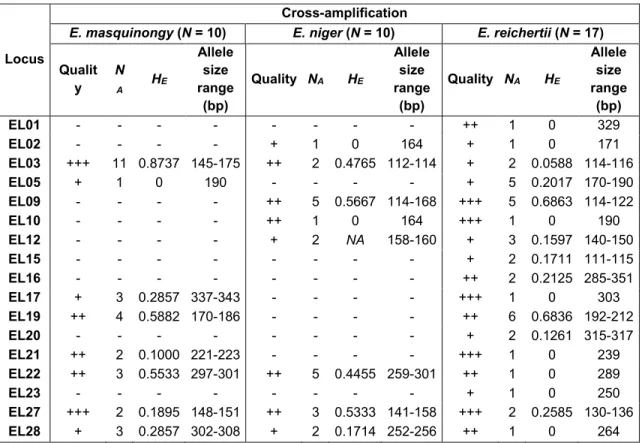
Table 1.2. Microsatellite markers characteristics in Esox lucius from North America and other Esox species. NA: Number of alleles; HO/HE: Observed/expected heterozygozity; Quality (of amplification): +++ 100% success, ++ ≥50% success, + <50% success, - no amplification.
Locus
E. lucius (North America, N = 30)
Cross-amplification
E. lucius (Europe, N = 20) E. americanus americanus
(N = 10) E. americanus vermiculatus (N = 10) Quality Allele size range (bp) NA HE HO Fis HWE P- Quality NA HE Allele size range (bp) Quality NA HE Allele size range (bp) Quality NA HE Allele size range (bp) EL01 +++ 317-327 5 0.6283 0.6333 -0.01 0.4444 ++ 3 0.3778 335-343 - - - - EL02 +++ 197-211 6 0.5113 0.5000 0.02 0.3771 +++ 7 0.8256 183-207 +++ 2 0.1895 176-182 +++ 3 0.5316 180-184 EL03 +++ 126-142 7 0.4763 0.5000 -0.05 0.5985 +++ 6 0.6308 118-134 +++ 1 0 120 +++ 1 0 118 EL05 +++ 176-192 7 0.6090 0.6000 0.02 0.7941 +++ 6 0.7026 164-192 + 4 0.2600 178-190 - - - - EL09 +++ 120-134 11 0.8576 0.8333 0.03 0.8118 +++ 8 0.7013 134-150 +++ 2 0.3462 114-115 ++ 3 0.4059 114-116 EL10 +++ 188-192 2 0.0333 0.0333 0.00 NA +++ 5 0.5030 202-214 ++ 1 0 164 - - - - EL12 +++ 148-162 6 0.4215 0.4000 0.05 0.7208 +++ 3 0.1845 144-166 + 3 0.2429 154-158 + 4 0.2000 150-160 EL15 +++ 111-131 6 0.4503 0.4000 0.11 0.2235 +++ 2 0.4662 115-123 ++ 3 0.2556 115-123 + 1 0 123 EL16 +++ 317-341 5 0.5921 0.9667 -0.65 0.0000 +++ 2 0.4824 347-351 - - - - EL17 +++ 329-341 4 0.3452 0.3333 0.03 0.7253 +++ 8 0.7905 329-249 + 1 0 337 + 1 0 337 EL19 +++ 178-186 4 0.2678 0.2333 0.13 0.4944 +++ 4 0.4526 180-192 - - - - EL20 +++ 308-376 23 0.9503 0.9667 -0.02 0.7249 +++ 13 0.9282 320-360 - - - - + 2 NA 345-347 EL21 +++ 227-253 7 0.3537 0.3667 -0.04 0.6268 +++ 5 0.6081 241-257 - - - - EL22 +++ 295-303 4 0.4311 0.5000 -0.16 0.7637 +++ 5 0.6654 267-297 ++ 7 0.5364 259-301 +++ 2 0.1000 265-285 EL23 +++ 246-267 8 0.6616 0.7000 -0.06 0.4514 +++ 7 0.6814 275-290 - - - - EL27 +++ 152-173 7 0.4746 0.4667 0.02 0.7074 +++ 6 0.6689 158-173 +++ 2 0.3368 155-167 +++ 3 0.5684 161-167 EL28 +++ 256-270 5 0.1898 0.1667 0.12 0.2208 +++ 9 0.7859 282-326 +++ 1 0 260 +++ 1 0 250
20
Table 1.2. Microsatellite markers characteristics in Esox lucius from North America and other Esox species. NA: Number of alleles; HO/HE: Observed/expected heterozygozity; Quality (of amplification): +++ 100% success, ++ ≥50% success, + <50% success, - no amplification.
Locus
Cross-amplification
E. masquinongy (N = 10) E. niger (N = 10) E. reichertii (N = 17)
Qualit y N A HE Allele size range (bp) Quality NA HE Allele size range (bp) Quality NA HE Allele size range (bp) EL01 - - - ++ 1 0 329 EL02 - - - - + 1 0 164 + 1 0 171 EL03 +++ 11 0.8737 145-175 ++ 2 0.4765 112-114 + 2 0.0588 114-116 EL05 + 1 0 190 - - - - + 5 0.2017 170-190 EL09 - - - - ++ 5 0.5667 114-168 +++ 5 0.6863 114-122 EL10 - - - - ++ 1 0 164 +++ 1 0 190 EL12 - - - - + 2 NA 158-160 + 3 0.1597 140-150 EL15 - - - + 2 0.1711 111-115 EL16 - - - ++ 2 0.2125 285-351 EL17 + 3 0.2857 337-343 - - - - +++ 1 0 303 EL19 ++ 4 0.5882 170-186 - - - - ++ 6 0.6836 192-212 EL20 - - - + 2 0.1261 315-317 EL21 ++ 2 0.1000 221-223 - - - - +++ 1 0 239 EL22 ++ 3 0.5533 297-301 ++ 5 0.4455 259-301 ++ 1 0 289 EL23 - - - + 1 0 250 EL27 +++ 2 0.1895 148-151 ++ 3 0.5333 141-158 +++ 2 0.2585 130-136 EL28 + 3 0.2857 302-308 + 2 0.1714 252-256 ++ 1 0 264
21
CHAPITRE 2 : Spatially variable extent of
population structure in northern pike (Esox lucius)
explained by landscape features
Geneviève Ouellet-Cauchon1*, Marc Mingelbier2, Frédéric Lecomte2 and Louis Bernatchez1
1 Université Laval, Institut de Biologie Intégrative et des Systèmes (IBIS), 1030 Avenue de la Médecine,
Québec (Québec), G1V 0A6, Canada
2 Ministère du Développement durable, de l’Environnement, de la Faune et des Parcs du Québec (MDDEFP),
Service de la Faune Aquatique, 880 chemin Sainte-Foy, Québec (Québec), G1S 4X4, Canada
23
Résumé
Un nombre croissant d’études ont investigué l’influence de facteurs environnementaux contemporains sur la structure génétique de populations, mais un facteur qui jusqu’à maintenant est demeuré peu étudié est la stabilité de l’habitat. A cette fin, un modèle d’aire d’étude approprié est le système lac Ontario – fleuve Saint-Laurent qui présente localement divers degrés de variation interannuelle du niveau d’eau. Dans cet article, nous documentons la génétique du paysage du grand brochet (Esox lucius), un poisson fortement exploité qui fraie dans les plaines d’inondation, en se basant sur l’analyse de près de 3000 individus provenant de 40 sites d’échantillonnage en utilisant 22 marqueurs microsatellites dans le système lac Ontario – fleuve Saint-Laurent. La structure génétique sur l’ensemble de l’aire d’étude était globalement très faible (Fst = 0.0208) mais spatialement variable avec un niveau moyen de différenciation dans la section amont (Ontario) de l’aire d’étude étant trois fois plus importante (Fst = 0.0297) que dans le secteur aval (Québec, Fst = 0.0100). Vingt variables environnementales furent considérées et un modèle de régression multiple sur matrices de distances (R2 = 0.6397, P < 0.001) a révélé que les masses d’eau (b =
0.3617, P < 0.001) et la présence de barrages (b = 0.4852, P < 0.005) ont réduit la connectivité et conséquemment augmenté la structure génétique de populations. De plus, la stabilité interannuelle du niveau d’eau était positivement associée à l’intensité de la différenciation génétique (b = 0.3499, P < 0.05). Comme une importante variation du niveau d’eau influe sur la qualité et la localisation annuelle des habitats de reproduction de grand brochet, l’instabilité locale de l’habitat sous la forme de la variation interannuelle du niveau d’eau semble localement empêcher la structure génétique de populations, probablement en empêchant le comportement de philopatrie. La gestion du grand brochet devrait être conséquemment reconsidérée dans une perspective de métapopulation, particulièrement dans les régions qui présentent une importante variation du niveau d’eau.
24
Mots-clés : structure génétique de populations, Esox lucius, variation environnementale,
25
Abstract
A growing number of studies have been investigating the influence of contemporary environmental factors on population genetic structure, yet one factor that has been poorly studied is habitat stability. To this end, an appropriate model study area is Lake Ontario – St. Lawrence River system that locally presents diverse degrees of inter-annual water level variation. In this paper, we document the landscape genetics of northern pike (Esox lucius), a heavily exploited fish that spawns in flood plains, based on the analysis of nearly 3000 individuals from 40 sampling sites using 22 microsatellite markers in the Lake Ontario – St. Lawrence River system. Genetic structure over whole study area was globally very weak (Fst = 0.0208) but spatially variable with mean level of differentiation in the upstream (Ontario) section of the studied area being three-fold higher (Fst = 0.0297) than observed in the downstream (Québec) sector (Fst = 0.0100). Twenty environmental variables were considered and a multiple regression on distance matrices model (R2 = 0.6397, P < 0.001)
revealed that water masses (b = 0.3617, P < 0.001) and the presence of man-made dams (b = 0.4852, P < 0.005) reduced connectivity, thus enhancing population genetic structure. Moreover, inter-annual water level stability was positively associated to the extent of genetic differentiation (b = 0.3499, P < 0.05). Since high water level variation impacts on yearly quality and localization of northern pike spawning habitats, local habitat instability which is under the form of inter-annual water level variation seems to locally impede population genetic structure, perhaps by inhibiting philopatry behavior. Thus, northern pike management should be carefully reconsidered in a metapopulation perspective, especially within sensitive areas that present high water level variation.
Keywords: population genetic structure, Esox lucius, environmental variation, habitat
26
Introduction
Landscape genetic studies have mostly focused on long-term processes that structure populations. Recently, a growing number of studies have been focusing on quantifying the influence of natural and anthropic contemporary environmental changes that may alter population genetic structure. Since landscapes are dynamic, environmental features may be continuously altered by different levels of temporal landscape fluctuations. Environment temporal instability has been previously suggested to impact on biological diversity (reviewed in Fjeldså & Lovett 1997), population size and persistence (Nelson et al. 1987; Ostergaard et al. 2003), reproductive success (Titus & Mosegaard 1992) and dispersal (Baggiano et al. 2011; Messier et al. 2012). Since habitat stability level influence various aspects of population dynamics, its impact should be integrated in conservation strategies and in management plans of exploited species (Ostergaard et al. 2003).
Temporal landscape unsteadiness has also been shown to affect the temporal stability of population genetic structure, namely in insects (Baggiano et al. 2011), amphibians (Fitzpatrick et al. 2009), mammals (Messier et al. 2012) and freshwater fishes (Garant et al. 2000; Ostergaard et al. 2003). Most of these studies assessed temporal stability of genetic structure in a uniformly unstable habitat over the studied range of the species. In contrast, to our knowledge, for one species and in a single connected system, no study has investigated the consequences on population genetic structure of a range of different levels of habitat stability in relation to other environmental factors.
Due to its two components that greatly differ from their stability dynamic, Lake Ontario – St. Lawrence River system from the Great Lakes basin in north eastern North America represent a particularly relevant study site to investigate the impact of spatially variable habitat stability on freshwater fish population genetic structure (Figure 2.1). More specifically, Lake Ontario, the easternmost and the smallest of the Great Lakes, with a length of 311 km and a width of 85 km and totalling 19 000 km2 (Rukavina & Boyce
2012), receive most of its water from Great Lakes via the Niagara River and discharges into the St. Lawrence River. The St. Lawrence River, with a mean stream flow of about 12 600 m3/s near Quebec city (Gingras 1997), stretches on 1197 km (Marsh 2012) comprising