MARIE-EVE DUPUIS
CARACTERISATION DU MODE
D'ACTION DU SYSTÈME CRISPR1/CAS
DE STREPTOCOCCUS THERMOPHILUS
Mémoire présenté
à la Faculté des études supérieures et postdoctorales de l'Université Laval dans le cadre du programme de maîtrise en microbiologie
pour l'obtention du grade de Maître es Sciences (M. Se.)
DEPARTEMENT DE BIOCHIMIE, DE MICROBIOLOGIE ET DE BIO-INFORMATIQUE
FACULTÉ DES SCIENCES ET DE GÉNIE UNIVERSITÉ LAVAL
QUÉBEC
2011
RÉSUMÉ
Streptococcus thermophilus est une bactérie utilisée pour la fabrication industrielle de yogourts et de fromages et elle est sujette aux infections phagiques. Plusieurs mécanismes de défense contre les phages sont connus, dont le système CRISPR/Cas. Les loci CRISPR sont constitués de courtes séquences d'ADN répétées entrecoupées de séquences variables, les espaceurs. Des gènes cas et une région promotrice sont associés aux loci et la séquence homologue chez le phage est appelée le protoespaceur.
Pour ce mémoire, l'effet du système CRISPRl/Cas de la souche S. thermophilus DGCC7710 sur l'ADN phagique a été étudié. Ainsi, il a été prouvé que l'ADN phagique est clivé dans les protoespaceurs par le système CRISPRl/Cas. Puis, l'analyse des différents espaceurs démontre que les motifs associés aux protoespaceurs sont plus permissifs qu'anticipé. Finalement, la méthylation de l'ADN phagique ne semble pas affecter l'immunité CRISPR/Cas ou l'acquisition d'espaceurs.
ABSTRACT
Streptococcus thermophilus is a lactic acid bacterium used to manufacture yogurt and some Italian- and Swiss-type cheeses. Most S. thermophilus strains are phage sensitive. At least, five general groups of natural phage resistance mechanisms are described in the literature. The most recent is called CRISPR/Cas and it is found in many bacterial genomes. The CRISPR locus is composed by short repeats interspaced by variable sequences (called spacers) that can target and cleave foreign nucleic acids (protospacers).
During this M. Sc. project, the mode of action of the CRISPRl/Cas system of the strain S. thermophilus DGCC7710 was studied. First, viral DNA cleavage was shown during phage infection and the cleavage occurs within the protospacer sequence. Then, the protospacer-associated motif was also analysed and found to be more permissive than previously reported. Finally, the combination of CRISPR/Cas and restriction-modification systems was investigated and found to be compatible.
AVANT-PROPOS
La rédaction d'un mémoire est l'aboutissement de plusieurs mois de travail et de recherche, mais aussi de beaucoup de patience, de détermination et, il faut l'avouer, de beaucoup de plaisir. Voici donc le rapport écrit de mon passage au laboratoire du professeur Moineau. Durant mon séjour en tant que stagiaire d'été et d'étudiante à la maitrise, j'ai eu l'occasion de côtoyer des collègues merveilleux : les actuels (en ordre de paillasses : Bruno, Maxim, Alfonso, Denise, Siham, Julie, Maxime, Lynn, Mourad et Rodrigue), les anciens (Hélène, Simon, Geneviève, Audrey, Rym et Josiane) et les stagiaires (Céline, Daniella, Jeannot, Mariangeles et Ilhem).
Je dois un merci particulier à Josiane pour m'avoir formée à mon arrivée, à Manuela pour le partage si agréable du bureau ensoleillé et à mon copain Nicolas pour avoir supporté mes nombreux retards, mes absences et mes délais pas si courts de « scientifique ». La recherche universitaire c'est aussi parfois l'aboutissement de plusieurs frustrations, d'échecs et d'incompréhension. Je tiens donc à remercier l'esprit d'équipe et l'ambiance générale du labo pour le soutien dans ces moments moins agréables. Un gros merci à Sylvain qui travaille très fort pour maintenir ce climat d'entraide et vive les activités para-labo! Finalement, un dernier remerciement pour le soutien financier de Novalait et de la Commission canadienne du lait, ainsi qu'au groupe de recherche en écologie buccale pour avoir été pendant ces quatre dernières années une seconde famille ...
Alors à tous, je vous dis « bisou-bye-bye » et on se voit au prochain camping...
Marie-Ève (also known as MED)
« Les amis sont comme les étoiles; nous ne pouvons toujours les voir, mais nous savons pourtant qu 'ils sont là. »
Un signet madelinot
« Toutes nos connaissances, passées, présentes et à venir ne sont rien au regard de ce que nous ne saurons jamais. »
Evgueni Evtouchenko, Les baies sauvages de Sibérie
TABLE DES MATIERES
RÉSUMÉ ii ABSTRACT iii AVANT-PROPOS iv TABLE DES MATIÈRES vi LISTE DES FIGURES ix LISTE DES TABLEAUX x LISTE DES ABRÉVIATIONS xii
1. INTRODUCTION 1 1.1 Les bactéries lactiques 1 1.1.1 Les caractéristiques générales 1
1.1.2 Le contexte de la transformation laitière 1 1.2 La bactérie Streptococcus thermophilus 2
1.2.1 Les caractéristiques générales 2 1.2.2 Les séquences génomiques disponibles 3
1.2.3 Le genre Streptococcus 5 1.2.4 L'utilisation dans le domaine de la transformation laitière 6
1.2.4.1 La fabrication de fromages 7 1.2.4.2 La fabrication de yogourts 7
1.3 Les bacteriophages 8 1.3.1 L'historique de la découverte des bacteriophages 8
1.3.2 La classification des virus bactériens 9 1.3.3 Le cycle de multiplication virale 10 1.3.3.1 Les phages virulents et le cycle lytique 11
1.3.3.2 Les phages tempérés et le cycle lysogénique 15 1.3.4 L'impact en contexte de la transformation laitière 16 1.4 Les bacteriophages infectant Streptococcus thermophilus 17 1.4.1 Les caractéristiques des phages isolés infectant S. thermophilus 17
1.4.2 Les phages virulents 2972 et DT1 infectant S. thermophilus 19 1.5 Les mécanismes de résistance aux bacteriophages chez les bactéries lactiques 24
1.5.1 Les moyens de défense anti-phages 24 1.5.2 Les mécanismes de restriction-modification (RM) 26
1.5.2.1 L'historique 26 1.5.2.2 Les généralités 27 1.5.2.3 Les types de systèmes RM 27
1.5.2.4 Les RM et les phages 29 1.5.2.5 Les RM et Streptococcus thermophilus 29
1.6 Les systèmes CRISPR/Cas 33 1.6.1 L'historique de la découverte des systèmes CRISPR/Cas 33
1.6.2 Les éléments du système 34
1.6.2.1 Les répétitions 34 1.6.2.2 Les espaceurs 35 1.6.2.3 Les loci 36 1.6.2.4 Les gènes cas 36 1.6.2.5 La région leader 38 1.6.2.6 Les éléments exogènes 38 1.6.2.7 Les systèmes CRISPR/Cas de S. thermophilus 39
1.6.3 Les étapes du mécanisme d'action 41 1.6.3.1 L'adaptation par l'acquisition d'espaceurs 41
1.6.3.2 La biogenèse des petits ARN 44
1.6.3.3 L'interférence 46 1.7 Le mode d'action du locus CRISPRl/Cas chez S. thermophilus DGCC7710 48
1.7.1 La problématique de l'étude 48 1.7.2 Les objectifs du projet 49 2. MATÉRIEL ET MÉTHODES 50 2.1 Les phages et les souches bactériennes utilisés 50
2.2 L'obtention de BIM CRISPR1+ESP 50
2.2.1 L'isolement de BIM CRISPR+ESP résistants à l'infection phagique 50
2.2.2 La détection des BIM CRISPR1+ESP 50
2.2.3 L'analyse des espaceurs acquis 52 2.3 La replication de l'ADN phagique durant l'infection d'une souche de S. thermophilus . 53
2.3.1 Suivi temporel de l'infection d'une souche de S. thermophilus par le phage 2972 53
2.3.2 L'extraction de l'ADN total pendant l'infection 53
2.3.3 La digestion de l'ADN 54 2.3.4 Le transfert de l'ADN sur membrane 54
2.3.5 La synthèse de sondes pour les hybridations de type Southern 55 2.3.6 L'hybridation de type Southern et la détection de l'ADN phagique 56 2.4 La détermination du site de clivage du système CRISPRl/Cas de 5". thermophilus 58
2.4.1 L'infection des souches bactériennes par le phage virulent 2972 58 2.4.2 L'extraction et la digestion de l'ADN total pendant l'infection 58
2.4.3 La migration et la récupération de l'ADN total 59 2.4.4 La ligature intramoléculaire et l'amplification de l'ADN par réaction PCR 59
2.4.5 L'analyse des séquences des fragments de clivage 62 2.5 L'effet de la méthylation de l'ADN phagique sur l'efficacité du système
CRISPRl/Cas de 5". thermophilus 63 2.5.1 L'obtention des souches RM^ et RM" 63 2.5.2 L'obtention de lysats de phages à ADN méthylé ou non méthylé 65
2.5.3 L'évaluation de l'efficacité des systèmes étudiés 65 2.5.4 L'effet de la méthylation de l'ADN sur la capacité d'infection des phages 66
2.5.5 La comparaison avec un RM natif 66 2.6 L'effet de la méthylation de l'ADN phagique sur l'étape d'acquisition d'espaceurs 67
2.6.1 L'obtention et la caractérisation des BIM 67 2.6.2 L'amplification des unités RE du locus CRISPR1 de S. thermophilus DGCC7710 .... 67
2.6.3 L'analyse de la fréquence d'obtention de BIM-GATC 68
3. RÉSULTATS 70 3.1 La caractérisation des nouveaux BIM CRISPRl+ESPde S. thermophilus 70
3.2 La replication de l'ADN phagique en contexte d'infection 70 3.3 La détermination du site de clivage du système CRISPRl/Cas de S", thermophilus 74
3.4 L'efficacité des systèmes RM LlaDCHI et CRISPRl/Cas de la souche S. thermophilus
DGCC7710 76 3.5 L'effet de la méthylation de l'ADN phagique sur l'étape d'interférence du système
CRISPRl/Cas de la souche S. thermophilus DGCC7710 77 3.6 L'efficacité des systèmes RM301 et CRISPRl/Cas de la souche S. thermophilus
SMQ-301 78 3.7 L'effet de la méthylation de l'ADN phagique sur l'étape d'acquisition d'espaceurs du
système CRISPRl/Cas de la souche 5". thermophilus DGCC7710 79
4. DISCUSSION 82
4.1 La caractérisation des nouveaux BIM CRISPR+ESP 82
4.1.1 L'activité des loci CRISPR 82 4.1.2 Le mécanisme d'insertion et la nature des espaceurs acquis 84
4.1.3 La tolérance des systèmes CRISPR/Cas 85 4.2 La replication de l'ADN phagique en contexte d'infection 86
4.3 La détermination du site de clivage du système CRISPRl/Cas de S. thermophilus 87
4.4 L'efficacité des systèmes RM et CRISPRl/Cas de S. thermophilus 88 4.5 L'effet de la méthylation de l'ADN phagique sur l'étape d'interférence du système
CRISPRl/Cas de la souche S. thermophilus DGCC7710 90 4.6 L'effet de la méthylation de l'ADN phagique sur l'étape d'acquisition d'espaceurs
du système CRISPRl/Cas de la souche S. thermophilus DGCC7710 91
5. CONCLUSIONS ET PERSPECTIVES 94
6. BIBLIOGRAPHIE 96
LISTE DES FIGURES
Voies métaboliques des bactéries lactiques 2 Photo de Streptococcus thermophilus observée en microscopie
électronique à transmission 3 Adhésion des cellules de 5*. thermophilus à celles de L. bulgaricus dans
une co-culture liquide en phase mi-exponentielle de croissance 8 Morphotypes observés et noms des familles des virus infectant les
Prokaryota 10 Principales étapes de multiplication des phages de l'ordre des
Caudovirales 12 Organisation génomique de 11 phages infectant S. thermophilus dont la
séquence génomique est disponible dans GenBank 20 Génomes phagiques représentatifs des deux principaux groupes de
bacteriophages infectant 5. thermophilus 21 Photo du phage virulent 2972 observé en microscope électronique à
transmission 23 Photo du phage virulent DT1 observé en microscope électronique à
transmission 23 Schématisation des quatre systèmes CRISPR/Cas retrouvés chez la
souche 5. thermophilus DGC77110 40 Schématisation générale du mécanisme d'action du système CRISPR/Cas
contre les bacteriophages 42 Détails de la détermination du site de clivage de l'ADN du phage 2972
par le système CRISPRl/Cas de S. thermophilus 60 Détermination de l'effet du système CRISPRl/Cas de la souche
S. thermophilus DGCC7710 sur l'ADN du phage 2972 73 Détermination des sites de clivage du système CRISPRl/Cas de la
souche S. thermophilus DGCC7710 sur l'ADN du phage 2972 76 Plasmides utilisés dans l'étude et positionnement des protoespaceurs 81 Compréhension actuelle de l'étape d'interférence du système
CRISPRl/Cas de la souche S. thermophilus DGCC7710 89 Figure 4.2 Schématisation de la dynamique d'interaction entre les systèmes
CRISPRl/Cas et RM chez S. thermophilus 93 Figure 1.1 Figure 1.2 Figure 1.3 Figure 1.4 Figure 1.5 Figure 1.6 Figure 1.7 Figure 1.8 Figure 1.9 Figure 1.10 Figure 1.11 Figure 2.1 Figure 3.1 Figure 3.2 Figure 3.3 Figure 4.1 IX
LISTE DES TABLEAUX
Tableau 1.1 Liste des souches bactériennes de l'espèce Streptococcus thermophilus
dont la séquence génomique complète est disponible dans GenBank 4 Tableau 1.2 Liste des bacteriophages infectant l'espèce 5*. thermophilus dont la
séquence génomique complète est disponible dans GenBank 18 Tableau 1.3 Détails des composantes de système RM répertoriés chez les quatre
souches de S. thermophilus dont la séquence génomique complète est
disponible 31 Tableau 1.4 ' Détails des composantes des systèmes RM plasmidiques répertoriées
chez les autres souches de S. thermophilus 31 Tableau 1.5 Détails des composantes des systèmes RM chromosomiques répertoriées
chez les autres souches de S. thermophilus 32 Tableau 2.1 Liste des BIM CRISPR1+ESP de S. thermophilus utilisés dans la présente
étude .' 51 Tableau 2.2 Liste des amorces utilisées pour analyser par réaction PCR le contenu
des loci CRISPR1 et CRISPR3 des souches S. thermophilus DGCC7710
etSMQ-301 52 Tableau 2.3 Liste des souches de S. thermophilus utilisées lors des infections dans
le temps par le phage virulent 2972 54 Tableau 2.4 Détails des enzymes de restriction utilisées en fonction des souches de
5". thermophilus infectées avec le phage virulent 2972 55 Tableau 2.5 Détails des amorces nécessaires à la synthèse des sondes utilisées lors
des hybridations de type Southern 56 Tableau 2.6 Liste des souches isogéniques de S. thermophilus ayant servi à l'infection
par le phage 2972 pour la détermination du site de clivage du système
CRISPRl/Cas 58 Tableau 2.7 Détails des sites de clivage utilisés pour produire les fragments pour la
ligature intramoléculaire de l'ADN 59 Tableau 2.8 Liste des amorces ayant servies à l'amplification de l'ADN par réaction
de PCR inverse et au séquençage des amplicons obtenus 61-62 Tableau 2.9 Liste des BIM CRISPR1+ESP utilisés pour l'obtention de souches de
S. thermophilus contenant les plasmides pNZ123 ou pSRQ707 64 Tableau 2.10 Liste des BIM CRISPR1+ESP utilisés pour l'étude de l'effet de la
méthylation de l'ADN phagique sur l'étape d'interférence du système
CRISPRl/Cas 64 Tableau 2.11 Résumé des calculs à effectuer pour évaluer les EOP du phage 2972 en
présence des systèmes (CRISPR/Cas, RM) seuls ou combinés 65 Tableau 2.12 Résumé des calculs à effectuer pour évaluer les EOP du phage DTI en
Tableau 2.13 Liste des amorces utilisées pour analyser par réaction PCR l'effet de la méthylation sur l'étape d'acquisition d'espaceurs du système
CRISPRl/Cas de S. thermophilus DGCC7710 68 Tableau 3.1 Détails des BIM analysés en fonction de la souche sauvage et des
caractéristiques des espaceurs, des acquisitions et des protoespaceurs 71 Tableau 3.2 Résultats de la détermination des sites de clivage des protoespaceurs du
phage 2972 par le système CRISPRl/Cas de la souche S. thermophilus
DGCC7710 75 Tableau 3.3 Valeurs d'EOP pour le phage 2972 sur les différentes souches à l'étude
en présence des systèmes RM LlaDCHI et CRISPRl/Cas seuls ou
combinés 77 Tableau 3.4 Rapports des titres du phage 2972 pour évaluer l'effet de la méthylation
de l'ADN phagique sur le système CRISPRl/Cas de la souche
S. thermophilus DGCC7710 et les BIM dérivés 78 Tableau 3.5 EOP du phage DTI sur la souche S. thermophilus SMQ-301 et un
BIM dérivé 79 Tableau 4.1 Détails sur le nombre de motifs CRISPR1 et CRISPR3 pour les 11 phages
LISTE DES ABREVIATIONS O Abi ADN ARN ARNi ARNm BIM BIM CRISPR BIM-GATC B\M::cas9~ BIM: :csn2~ BL C cas Cas Cascade COG crARN CRISPR CRISPR1 CRISPR2 CRISPR3 CRISPR4 CRISPRdb CRP Da db DIG D.O. DR E. coli EOP +ESP Phage
Avortement de l'infection (Abortive Infection) Acide désoxyribonucléique
Acide ribonucléique Interférence à l'ARN ARN messager
Bactérie résistante aux phages (Bacteriophage Insensitive Mutant) BIM de type CRISPR ayant acquis un espaceur
BIM de type CRISPR ayant acquis un espaceur qui contient un site GATC
BIM dont le gène cas9 a été inactivé BIM dont le gène csn2 a été inactivé Bactérie lactique
Composante de contrôle (C) des systèmes RM
Gène associé aux CRISPR (CRISPR-Associated gene)
Produit protéique résultant de la traduction des ARNm des gènes cas Complexe protéique retrouvé chez Escherichia coli (CRISPR-Associated Complex for Antiviral Defense)
Classe de groupes orthologues de protéines (Cluster of Orthologous Groups of proteins)
ARN CRISPR (CRISPR RNA)
Système composé de courtes répétitions palindromiques régulièrement espacées (Clustered Regularly Interspaced Short Palindromic Repeats) Nom donné au locus CRISPR 1 chez Streptococcus thermophilus Nom donné au locus CRISPR2 chez Streptococcus thermophilus Nom donné au locus CRISPR3 chez Streptococcus thermophilus Nom donné au locus CRISPR4 chez Streptococcus thermophilus Base de données sur les systèmes CRISPR/Cas (CRISPR Database) Protéine récepteur de l'AMPc (cAMP Receptor Protein)
Dalton Double-brin Digoxygénine Densité optique
Répétitions directes (Direct Repeats) Escherichia coli
Efficacité à former des plages de lyse (Efficiency ofPlaquing)
EPS Exopolysaccharide
FDA Agence fédérale américaine des produits alimentaires et médicamenteux (Food and Drug Administration) GC Contenu en guanine et en cytosine
GenBank Base de données génétiques créée au Centre national pour l'information biotechnologique (NCBI, National Center for Biotechnology
Information)
GRAS Généralement reconnu comme sécuritaire (Generally Recognized As Safe)
H. volcanii Haloferax volcanii
ICTV Comité international de taxonomie des virus (International Committee on Taxonomy of Viruses)
in silico Expression qualifiant des recherches effectuées au moyen d'outils informatiques
in vitro Expression qualifiant des recherches effectuées en dehors de l'organisme vivant ou de la cellule
in vivo Expression qualifiant des recherches effectuées avec un organisme vivant ou une cellule
L. bulgaricus Lactobacillus delbrueckii sous-espèce bulgaricus
LCTR Long groupe de séquences répétées en tandem (Long Cluster of Tandem Repeats)
L. lactis Lactococcus lactis
LPS Lipopolysaccharide
LTRR Longues séquences répétées en tandem (Long Tandemly Repeated Repetitive sequences)
M Composante de modification (M) des systèmes RM MCP Protéine majeure de la capside (Major Capsid Protein) MOI Multiplicité d'infection (Multiplicity of Infection)
Mpb Mégapaires de base, soit 1 million de paires de bases (pb) MTP Protéine majeure de la queue (Major Tail Protein)
N. D. Donnée non disponible NI Non infecté
nt Nucleotide
ORF Cadre de lecture ouvert (Open Reading Frame)
PAM Motif adjacent aux protoespaceurs (Proto-spacer Adjacent Motif) pb Paire de bases
PCR Réaction de polymérisation en chaîne (Polymerase Chain Réaction) PDG Pepdidoglycane
PEM Pré-crARN R RAMP RBP RC RE REBASE Ref. RM RM* RM" RNase S sb S. epidermidis S. mitis SPIDR S. pneumoniae S. pyogenes SRSR S. salivarius ssp. S. suis S. thermophilus S. uberis S. vestibularis TA TMP tracr-ARN TREP U ufc VR2
Bactérie réfractaire à la transformation plasmidique (Plasmid Interfering Mutants)
Longs ARN précurseurs des crARN matures Composante de restriction (R) des systèmes RM
Protéine mystérieuse associée aux séquences répétées (Repeat Associated Mysterious Protein)
Protéine de liaison au récepteur (Receptor-Binding Protein) Mécanisme de type « cercle roulant » (Rolling circle) Répétition-espaceur
Base de données sur les enzymes de restriction (Restriction Enzymes database)
Référence
Restriction-modification
Souche possédant un système RM actif Souche ne possédant pas de système RM actif Ribonucléase
Composante de spécificité (S) des systèmes RM Simple-brin
Staphylococcus epidermidis Streptococcus mitis
SPIDR (Spacers Interspersed Direct Repeats) Streptococcus pneumoniae
Streptococcus pyogenes
Courtes répétitions régulièrement espacées (Short Regularly Spaced Repeats) Streptococcus salivarius Sous-espèce Streptococcus suis Streptococcus thermophilus Streptococcus uberis Streptococcus vestibularis Température ambiante
Protéine étalon de la queue (Tape Measure Protein)
ARN activant en trans le système CRISPR (Trans-Activating CRISPR RNA)
Répétitions en tandem (Tandem Repeats) Unité
Unité formatrice de colonie
Région variable #2 (Variable Région #2)
1 INTRODUCTION
1.1 Les bactéries lactiques
LtL.l..)r^._^d__^__§_I.Q__?J.S^Str9}.._
Les bactéries lactiques (BL) sont des microorganismes unicellulaires très répandus dans la nature. Elles regroupent des espèces bactériennes à Gram-positif et sont retrouvées dans le sol, les litières, les fourrages, sur les mamelles des animaux et dans le lait qu'elles produisent, ainsi que dans le tube digestif de l'homme. Le grand groupe des BL comprend plusieurs genres bactériens,, dont Aerococcus, Carnobacterium, Enterococcus, Lactobacillus (entres autres les espèces acidophilus, delbrueckii, casei, helveticus, lactis, plantarum et rhamnosus), Lactococcus (espèces cremoris et lactis), Leuconostoc (espèces cremoris, dextranicum et lactis), Pediococcus (espèces acidilactici et pentosaceus) et Streptococcus (espèce thermophilus) (Carr et al., 2002). Ces bactéries peuvent être de formes sphériques, ovoïdes ou de bâtonnets, mais elles partagent toutes la capacité de produire de l'acide lactique (ou lactate) comme produit de fermentation à partir des hydrates de carbone, dont le lactose. Les voies métaboliques des BL sont résumées et présentées à la figure 1.1. Cette capacité métabolique leur vaut une importance industrielle et économique particulière dans le domaine de la fermentation alimentaire et surtout laitière.
LL_..Le contexte deJafr^sfonnatjon laitière
Domestiquées par l'homme depuis des millénaires, les BL sont utilisées pour la fabrication d'aliments divers, comme le saumurage des légumes, la fabrication du vin ou du pain, le saurissage des poissons et des viandes, etc. (Drider et Prévost, 2009). Elles sont notamment reconnues et utilisées pour l'obtention de produits laitiers fermentes, tels que le yogourt, le fromage, le babeurre, la crème sûre et le kéfir. Les BL permettent d'améliorer la conservation des aliments grâce à leurs propriétés acidifiantes en empêchant la prolifération des microorganismes d'altération. Les différents produits finaux et intermédiaires de fermentation déterminent aussi l'obtention de saveurs et de textures particulières. Les souches de BL sont sélectionnées en fonction d'un grand nombre de propriétés métaboliques et technologiques et elles sont ensuite incluses dans des ferments
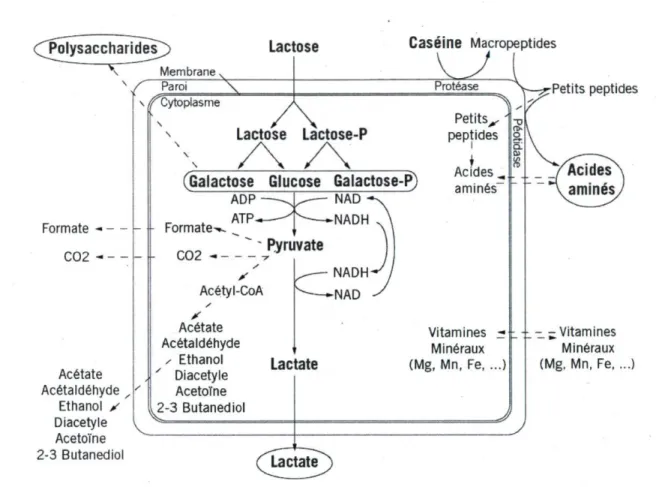
commerciaux. Ceux-ci peuvent être de nature définie ou non selon si les souches qu'ils contiennent sont caractérisées et ils serviront à ensemencer le lait au début des fermentations. Lactose Membrane Formate - — C02 -Paroi /^Cytoplasme _ _ : Caséine Macropeptides Lactose Lactose-P (Galactose Glucose Galactose-P) Formate-»-Pyruvate |_ C02 . _* Acétyl-CoA Acétate Acétaldéhyde . Ethanol Diacetyle Acetoïne w2-3 Butanediol NAD NADH NADH--NAD peptides Acides „ | aminés Vitamines _■ Minéraux (Mg, Mn, Fe, ...) Petits peptides - - Vitamines Minéraux (Mg, Mn, Fe, ...) Acétate Acétaldéhyde . Ethanol y Diacetyle Acetoïne 2-3 Butanediol
Figure 1.1 - Voies métaboliques des bactéries lactiques. Figure tirée du Centre national interprofessionnel de l'économie laitière (CNIEL, 2009).
Lactate
1.2 La bactérie Streptococcus thermophilus
.L2.1_.Les.caractéristiques générales
Streptococcus thermophilus est une bactérie qui appartient à l'ordre des Lactobacillales de la division des Firmicutes. Elle fait partie des BL thermophiles; elle a une température optimale de croissance comprise entre 42 °C et 45 °C, mais peut supporter des températures allant de 22 °C à 52 °C (Bergey et al., 1984; Chandan, 2006). Ce streptocoque a une taille comprise entre 0,7 et 0,9 pm, n'est pas motile et ne possède pas les
enzymes catalase et oxydase. Bien que cette bactérie soit communément isolée dans l'environnement laitier, son environnement naturel n'est pas connu (Bergey et al, 1984; Hardie et Whiley, 1992). Au microscope, elle prend la forme de cellules sphériques (de coques) et se présente en paires ou en chaînes de longueur variable (voir la figure 1.2). Du point de vue métabolique, S. thermophilus est un organisme anaérobie facultatif et chimioorganotrophe fermentaire. Ce streptocoque peut produire de l'acide lactique à partir du lactose, du sucrose et du glucose et certaines souches peuvent aussi fermenter le fructose et, plus rarement, le galactose (Hols et al, 2005).
Figure 1.2-Photo de Streptococcus thermophilus observée en microscopie électro-nique à transmission. Figure tirée de Science Photo Library (Scimat, B236/154). Agrandissement inconnu.
L2:2..Les séquences.gé^
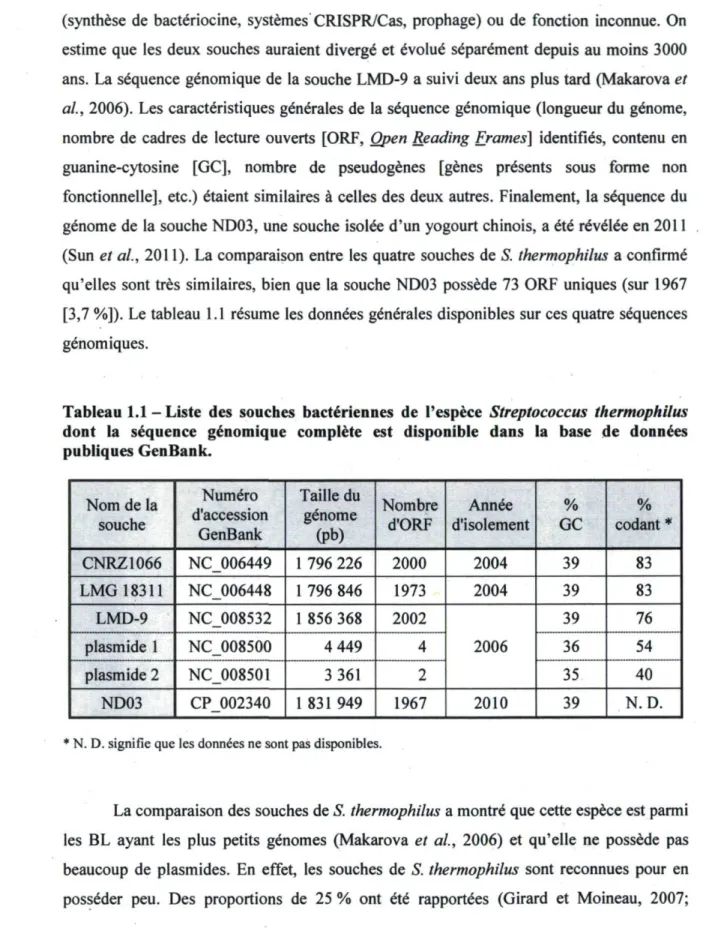
Orla-Jensen fut le premier en 1919 à classer Streptococcus thermophilus comme une espèce à part entière de streptocoques et il aura fallu 85 ans avant que les premières séquences génomiques ne soient obtenues. Au moment de l'écriture de ce mémoire, quatre séquences génomiques complètes de souches de S. thermophilus sont disponibles dans la base de données publiques GenBank. Les deux premières, celles des souches CNRZ1066 et LMG 18311, ont été publiées en 2004 (Bolotin et al, 2004). L'analyse des séquences de taille d'environ 1,8 millions de paires de bases (Mpb) chacune ont montré que les deux souches partagent plus de 90 % de leurs séquences codantes. Les différences observées concernent des gènes liés à la production d'exopolysaccharides (eps, rps), d'immunité
(synthèse de bactériocine, systèmes CRISPR/Cas, prophage) ou de fonction inconnue. On estime que les deux souches auraient divergé et évolué séparément depuis au moins 3000 ans. La séquence génomique de la souche LMD-9 a suivi deux ans plus tard (Makarova et al, 2006). Les caractéristiques générales de la séquence génomique (longueur du génome, nombre de cadres de lecture ouverts [ORF, Open Reading Frames] identifiés, contenu en guanine-cytosine [GC], nombre de pseudogènes [gènes présents sous forme non fonctionnelle], etc.) étaient similaires à celles des deux autres. Finalement, la séquence du génome de la souche ND03, une souche isolée d'un yogourt chinois, a été révélée en 2011 (Sun et al, 2011). La comparaison entre les quatre souches de S. thermophilus a confirmé qu'elles sont très similaires, bien que la souche ND03 possède 73 ORF uniques (sur 1967 [3,7 %]). Le tableau 1.1 résume les données générales disponibles sur ces quatre séquences génomiques.
Tableau 1.1 - Liste des souches bactériennes de l'espèce Streptococcus thermophilus dont la séquence génomique complète est disponible dans la base de données publiques GenBank. Nom de la souche Numéro d'accession GenBank Taille du génome (Pb) Nombre
d'ORF d'isolement Année GC % codant * % CNRZ1066 NC_006449 1 796 226 2000 2004 39 83 LMG 18311 NC_006448 1 796 846 1973 2004 39 83 LMD-9 NC_008532 1 856 368 2002 2006 39 76 plasmide 1 NC_008500 4 449 4 2006 36 54 plasmide 2 NC_008501 3 361 2 2006 35 40 ND03 CP_002340 1 831 949 1967 2010 39 N.D.
* N. D. signifie que les données ne sont pas disponibles.
La comparaison des souches de S*, thermophilus a montré que cette espèce est parmi les BL ayant les plus petits génomes (Makarova et al, 2006) et qu'elle ne possède pas beaucoup de plasmides. En effet, les souches de S. thermophilus sont reconnues pour en posséder peu. Des proportions de 25 % ont été rapportées (Girard et Moineau, 2007;
Rizzotti et al, 2009; Somkuti et Steinberg, 1986; Turgeon et al, 2004) et les souches n'en possèdent jamais plus de deux (Turgeon et al, 2004). Parmi les souches dont le génome a été séquence, seule la souche LMD-9 possède des plasmides. Au total, une vingtaine de séquences plasmidiques de 5". thermophilus ont été rapportées, dont dix qui sont disponibles dans la base de données publiques GenBank. Les tailles sont comprises entre 2,6 et 9,5 Kpb, codant pour deux à neuf ORF et ont des contenus en GC de 32 % à 38 %. Fait intéressant, les plasmides de S. thermophilus ont été retrouvés préférentiellement chez des souches non industrielles (Turgeon et Moineau, 2001). Cela s'explique par le fait que les plasmides cette espèce codent rarement pour des phenotypes d'importance industrielle.
L2:3..Le:g^nK_Strep_tococcus
Dans le grand groupe des streptocoques, S. thermophilus appartient au groupe des Viridans, qui comprend entre autres les espèces S. salivarius et S. vestibularis. S. thermophilus a été classée comme une sous-espèce de S. salivarius jusqu'en 1991, alors que Schleifer et al. ont montré clairement la distinction entre les deux espèces. Certaines équipes ont émis l'hypothèse que S. thermophilus aurait tout de même partagé un ancêtre commun avec S. salivarius il y a de cela IO7 générations (Bolotin et al, 2004). La
divergence évolutive coïnciderait avec l'apparition de l'élevage des ruminants, et donc de la domestication des BL pour l'alimentation humaine à partir de leur lait. Le streptocoque thermophile se serait ainsi acclimaté à sa nouvelle niche écologique.
Lors de cette adaptation, le génome de S. thermophilus aurait subit d'importantes modifications qui expliquent sa place si particulière parmi les streptocoques (Bolotin et al, 2004). D'abord, il aurait acquis des gènes lui permettant d'utiliser le sucre du lait, le lactose. La comparaison des génomes de souches de S. thermophilus avec ceux d'autres souches de streptocoques a montré que les premières possèdent un transporteur spécifique au lactose qui n'est pas retrouvé chez aucune autre espèce du genre. Ensuite, plusieurs gènes, dont certains codent pour d'autres systèmes de transport des sucres (comme le glucose et le fructose), sont devenus désuets pour S. thermophilus et sont maintenant absents ou présents sous forme de pseudogènes dans les génomes. Cette BL metabolise donc un nombre plus restreint de sucres par rapport aux autres streptocoques. De cette
analyse, il a finalement été suggéré que S. thermophilus, en s'adaptant à ce nouvel environnement, a perdu le caractère pathogène commun aux autres streptocoques (Hardie et Whiley, 1992). Parmi ceux-ci se trouvent S. pneumoniae, l'agent causal de la pneumonie, S. pyogenes, causant des infections, telles que les pharyngites, le rhumatisme articulaire et la fasciite nécrosante, et S. mutons, impliquée dans les caries dentaires. 5*. thermophilus, quant à lui, est le seul représentant de ce genre bactérien qui origine des produits laitiers, et c'est aussi le seul qui soit reconnu sécuritaire (GRAS, Generally Recognized As Safe) par la FDA (Food and Drug Administration).
Par ces analyses comparatives, les chercheurs ont montré que les gènes identifiés chez S. thermophilus sont à 80 % orthologues aux gènes des autres streptocoques (~ 1500 ORF). Cela démontre que les streptocoques partagent entre eux tout de même une partie « substantielle de leur physiologie et de leur métabolisme » (Bolotin et al, 2004). Les souches de S. thermophilus possèdent aussi un grand nombre de gènes devenus non fonctionnels après des événements de mutations, de deletions ou d'insertion. De plus, aucun gène de résistance aux antibiotiques n'a encore été identifié et les souches analysées semblent sensibles à beaucoup d'agents antimicrobiens. Tous ces éléments permettent à ce streptocoque lactique de conserver sa place privilégiée parmi les BL alimentaires.
L.2.4. .L'utiUsation d.MS.Ie domainedejatransfo
Streptococcus thermophilus est la BL la plus importante pour la production de produits laitiers fermentes après Lactococcus lactis. Elle est nécessaire dans la fabrication commerciale et traditionnelle de fromages, de yogourts et de plusieurs autres laits fermentes, comme le dahi, le skyr et le leben (Prajapati et Nair, 2003). Au Canada, plus de 300 719 000 kg de yogourts et 115 129 000 kg de mozzarella ont été produits en 2010 (Infolait). Au total, IO21 cellules vivantes de S. thermophilus sont ingérées par l'humanité
chaque année (Hols et al, 2005) et la valeur commerciale annuelle de produits alimentaires le contenant est évaluée à plus de 40 milliards de dollars (Blomqvist et al, 2006). Son aptitude à survivre à de hautes températures industrielles et ses capacités fermentaires et probiotiques expliquent pourquoi son utilisation, seule ou combinée, est aussi répandue.
1.2.4.1 ht\ febriçgtfoq dç frPBMgff
Lors de la fabrication fromagère, les BL utilisent le lactose du lait et le transforment en acide lactique par la fermentation. Cette acidification provoquée par les ferments lactiques, combinée à l'ajout de présure, permet la précipitation des protéines du lait. Ce coagulum (le caillé) est séparé de la phase aqueuse du lait (le lactosérum) et il peut être consommé directement après Pégouttage ou être affiné. Bien que des BL soient présentes naturellement dans le lait, elles sont souvent en quantité insuffisante pour une acidification adéquate (Chandan, 2006). Le lait est donc, la plupart du temps, pasteurisé afin de détruire sa flore initiale et les ferments sont ensuite ajoutés à une concentration finale de 107
cellules par millilitre (ml) de lait (Moineau et Lévesque, 2005). Les produits désirés et la technologie fromagère utilisée déterminent les caractéristiques des souches qui composent les ferments. Les fromages nécessitant l'utilisation de souches de S. thermophilus sont principalement la mozzarella, et aussi l'emmental, le gruyère et le parmesan (Delorme, 2008).
1-2.4.2..La fabrication.de jogourts
Le terme yogourt réfère à l'utilisation symbiotique de souches de S. thermophilus et de Lactobacillus delbrueckii sous-espèce bulgaricus (L. bulgaricus) et sa consommation fait partie de la tradition alimentaire de plusieurs parties du monde depuis des siècles. Dans ce type de fermentation, la croissance de chaque espèce stimule celle de l'autre. En début de fermentation, ce sont surtout les streptocoques qui agissent : ils acidifient le lait, diminuent le potentiel d'oxydoréduction et produisent également du formate comme produit métabolique. Ces trois éléments permettent ensuite aux lactobacilles de prendre la relève. La formation du coagulum est terminée lorsque l'acidification est suffisante. Le yogourt est ensuite refroidi et ne nécessite pas d'égouttage comme les fromages. Fait intéressant, dans l'étude de Bolotin et al (2004), il a été suggéré que S. thermophilus aurait acquis des gènes utiles dans l'environnement laitier en côtoyant d'autres BL, dont L. bulgaricus. Des études en microscopie ont d'ailleurs révélé que ces deux bactéries vivent en étroite association (voir la figure 1.3). Ce rapprochement spatial favoriserait ainsi les échanges de gènes même si elles sont éloignées au point de vue évolutif.
"**$Q!_3
S. thermophilus [ " * * $ & $ y •"-•■-■ »s__ *H__ -V
e
**
L. bulgaricusFigure 1.3 - Adhésion des cellules de S. thermophilus (les coques) à celles de L. bulgaricus (les bâtonnets) dans une co-culture liquide en phase mi-exponentielle de croissance. Figure tirée de Bolotin et al. (2004).
1.3 Les bacteriophages
.L.3.1..L'historique dejadéçouyerte.desMçtériophages
L'usage massif des BL en usine a amené une demande sans cesse grandissante de performance et de productivité des souches composant les ferments. Un des critères essentiels dans le choix des bactéries sélectionnées est leur résistance contre les virus bactériens, les bacteriophages (ou simplement phages). Ces phages furent découverts par Frederick Twort en 1915, puis fortement étudiés par Félix d'Hérelle à partir de 1917 (Summers et Abedon, 2006). Twort avait observé la présence d'un agent transmissible qui affectait la croissance cellulaire sans pouvoir toutefois l'identifier. D'Hérelle, quant à lui, saisit la nature virale de ces nouveaux microorganismes et il démontra la capacité de lyse d'une culture liquide par un « agent invisible qui se multiplie à l'infini » (Summers, 2005). La présence et la nature de cet agent fut confirmée avec l'arrivée de la microscopie électronique en 1940 (Ackermann, 2007). Ce fut dans les années trente que l'on montra pour la première fois que les productions laitières pouvaient être affectées par la présence de phages (Whitehead, 1953). De nos jours, on évalué entre 0,1 et 10% le nombre de fermentations lactiques affectées par l'infection phagique (Moineau et Lévesque, 2005). La caractérisation des phages est maintenant devenue un outil essentiel pour contrôler les populations virales dans les usines laitières.
.L__2..La classification des virus bactériens
Il est reconnu que les virus bactériens forment le groupe d'entités biologiques le plus abondant sur la Terre (nombre d'entités total estimé entre IO30 et IO32) et qu'ils sont
retrouvés partout où l'on retrouve des bactéries (Kutter et Sulakvelidze, 2005). Historiquement, la première classification fut établie en 1943 (Ruska, 1943), mais plus de cinquante ans plus tard, la classification virale actuelle n'inclut toujours pas tous les phages (Ackermann, 2009). La méthode la plus courante se base sur les caractéristiques structurales des particules virales, appelées le morphotype, qui sont observées en microscopie électronique. À ce jour, plus de 5500 phages ont été observés (Ackermann, 2007) et ils peuvent prendre des formes diverses (symétriques, cubiques, filamenteuse ou pléomorphes). Essentiellement, ces virus sont composés d'acides nucléiques (le génome) et de protéines (la capside). Au total, plus de 70 critères sont utilisés pour la caractérisation des virus. Ils concernent la nature du génome (ADN ou d'ARN; simple-brin [sb] ou double-brin [db]; circulaire ou linéaire), la structure de la particule virale, les tests d'inactivation, la physiologie, le type de replication et d'assemblage, etc.
Le Comité international de taxonomie des virus (ICTV, International Committee on Taxonomy of Viruses), regroupe actuellement les virus de bactéries et d'archées en 15 familles (dont deux communs aux bactéries et aux archées) et 39 genres différents (dont trois communs); les bacteriophages à eux seuls regroupent 10 familles et 29 genres. Les morphotypes de l'ordre, des familles et des genres de virus procaryotiques sont présentés à la figure 1.4. Un seul ordre est reconnu, celui des Caudovirales qui regroupe plus de 5300 phages à queue, soit 96 % des virions observés. Ils possèdent une capside de forme icosaédrique ou allongée et contiennent tous de l'ADNdb. L'ordre est divisé en trois familles selon si les virus ont des queues contractiles (Myoviridae; six genres), courtes (Podoviridae; quatre genres) ou longues (Siphoviridae; huit genres). Ces derniers sont les plus nombreux avec 3229 observations (61 %). Ces phages caudés ont en moyenne des tailles de 30 à 160 nm pour la capside et de 10 à 800 nm pour la queue (Ackermann, 2009). Tous les phages infectant S. thermophilus isolés à ce jour appartiennent à la famille des Siphoviridae. Les plus gros phages, les phages « jumbo », possèdent des génomes énormes pouvant aller jusqu'à 498 Kpb dans le cas du phage G infectant Bacillus megaterium (Hendrix, 2009). Concernant les phages sans queue, très peu de membres ont été identifiés
et ils sont de formes et de natures variées (Guttman et al., 2005). Des exemples de phages
sont fournis pour chacune des familles de la figure 1.4.
ADNsb
.
O
Micronridae (♦174) Inoviridae (M13)ADNdb
Tectmridae (PRD1) C ortie oviridae (PM2) Caudovirales iQ 9 9
Podoriridae (T7) Styoviridae Siphoviridae (T4) (X) Plasmavmdae « Globuloviridae » x» (L2) (PSV) \ [ v^ _ ^ ^ V ^ ^ - * ^ « S171 » Gutta-nridae Salterproviruf' Fuselloviridae (ST1V) (Hisl) (SSV1)0
ARNsb
O
LevMridae (MS2)ARNdb
CystO'iridae (*6) (SNDV) « Ampullaviridae (ABV) » Biiiiiitlm iriilm »> (ATV) Z. iphothrixi'iridae (TTV1) Rudmridae (SIRV-1)Figure 1.4 - Morphotypes observés et noms des familles des virus infectant les Prokaryota. Les noms en bleu sont les familles de phages infectant les Bacteria, ceux en vert sont les familles de phages infectant les Archaea et les noms en noir sont les familles communes aux deux domaines. Les noms entre guillemets ne sont pas des familles encore reconnues par l'ICTV et l'astérisque représente le nom du genre unique d'une famille sans nom. Un exemple de phage appartenant à chacune des familles est fourni entre parenthèses. Un seul ordre est reconnu par le comité, soit celui des Caudovirales qui comprend les familles Myoviridae, Siphoviridae et Podoviridae. Tous les phages infectant S. thermophilus isolés jusqu'à ce jour appartiennent à la famille des Siphoviridae. Figure modifiée d'Ackermann (2007).
L3.3..Le cycle, de:multiplication virale
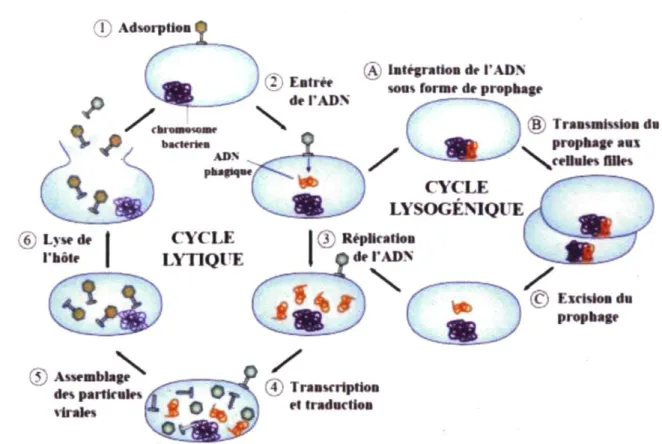
Les virus sont des parasites obligatoires, c'est-à-dire qu'ils ne peuvent pas se reproduire sans prendre le contrôle d'un organisme hôte. Au cours de leur multiplication, la plupart des virus vont provoquer la mort de leur cellule hôte lors de la libération de virions
matures. On distingue deux types de virus selon leur type de multiplication : les virus virulents, qui vont compléter leur cycle de multiplication jusqu'à la libération d'autres virions (le cycle lytique), et les virus tempérés, qui auront en plus le choix de demeurer de manière latente dans leur hôte (le cycle lysogénique). Une schématisation des principales étapes de la multiplication virale est présentée à la figure 1.5. Les cycles seront détaillés dans les sections suivantes en fonction des phages caudés, car ce sont eux qui sont les plus fréquemment observés, les plus étudiés et aussi ceux utilisés dans cette étude.
L.3. :3.:A.. Les. phages virulents, et le cycle, lytique
Le cycle lytique débute lorsqu'un phage s'adsorbe à un récepteur, situé à la surface bactérienne, à partir d'une protéine de liaison au récepteur fixée à l'extrémité de la queue (Kutter et a l , 2005). Cette étape de liaison est aléatoire et peut être réversible sans une liaison secondaire. De plus, le récepteur bactérien peut grandement varier d'une espèce à une autre : les protéines, les lipopolysaccharides (LPS), les flagelles, les pili sexuels et les capsules peuvent servir de récepteur bactérien (Guttman et a l , 2005). Des ions de calcium et de magnésium seraient nécessaires dans certains cas pour augmenter l'affinité de liaison (Beliaev et al, 1988; Capra et al, 2006; Cvirkaite-Krupovic et a l , 2010; Landry et Zsigray, 1980; Pham et al, 2009; Steensma et Blok, 1979). Le spectre lytique représente donc l'ensemble des souches qu'un phage peut infecter; il s'agit souvent d'un groupe restreint de souches d'une même espèce ou, plus rarement, d'espèces différentes (Guttman et a l , 2005).
Une fois le phage fixé et bien positionné, des enzymes s'affairent à dégrader le peptidoglycane (PDG). Celles-ci sont situées à l'extrémité de la queue et permettent le passage de l'ADN jusqu'à la membrane cellulaire. La capside et la queue resteront liées à la surface de la cellule hôte lorsque l'ADN du phage sera injecté à l'intérieur. Il passera d'abord au travers du canal du complexe portai de la capside, puis dans celui de la queue.
Par la suite, le génome se circularisera ou demeurera linéaire selon le cas. L'échange de matériel entre la capside et le cytoplasme cellulaire peut se faire à très grande vitesse, soit jusqu'à 4000 pb par seconde pour le phage T4 (Letellier et al, 2004). À titre comparatif, la vitesse de transfert lors de la conjugaison ou de la transformation naturelle
0 Adtorptioa • V____ ) ® E » t i . Eitrte dr l'ADN ^ _ / \
^(<t^
y
A Intefjralion d«- I ADN tons fornr dr prophageC__w
pbafique ADN B Tr_«%mission da - prophagr i n >»yf rriMM flllrs CYCLEV J L y LYSOGÉMQIE,
(g L>»r de l'hoir CYCLE LYTIQUE ; j Rrpliralion © ElcWoDdu prophage (5) Assrmblagr^ drs partkolr\ M l . l l .-Figure 1.5 - Principales étapes de multiplication pour les phages de l'ordre des Caudovirales. Les bacteriophages peuvent être virulents ou tempérés. Les phages virulents se multiplient rapidement une fois que 1'adsorption à la cellule hôte est effectuée (1). Il s'ensuit le relâchement d'un nombre plus ou moins important de virions matures qui cause la mort cellulaire (6). Les phages tempérés, quant à eux, auront le choix après l'entrée de l'ADN de poursuivre le cycle lytique ou d'effectuer le cycle lysogénique (A). Lors de la lysogénie, les virus peuvent s'intégrer au chromosome bactérien (comme illustré en A) ou être présents sous forme plasmidique. Le prophage sera transmis aux cellules filles lors de la multiplication bactérienne (B). Figure modifiée de Labrie et al. (2010).
est de 100 pb/s. La dynamique d'entrée de l'ADN phagique n'est pas encore bien comprise. Dans certains cas, la pression contenue dans la capside semble suffire à l'injection complète de l'ADN. Dans d'autres cas, comme celui du phage T7 (40 Kpb), l'entrée de l'ADN serait couplée au processus de transcription (Kemp et al, 2004; Molineux, 2001) : sans transcription, la vitesse n'est que de 75 pb/s. Le phage T5 (121 Kpb), quant à lui, injecte d'abord 8 % de son génome et n'injectera le reste que lorsque deux protéines phagiques seront traduites (Letellier et al., 2004).
Une fois l'ADN entré et circularise, la polymerase bactérienne est utilisée pour la replication à partir d'une ou plusieurs origines de replication (ori) phagique. La replication
peut être, selon les phages, de type « cercle roulant » (RC, Rolling Circle), de type thêta (0) ou comprendre les deux types (Catalano, 2000). Pour le mode RC, l'un des deux brins d'ADN sert de modèle et permet la production du matériel génétique, souvent sous forme de longs concatémères. Dans le cas du mode thêta, il y a replication de l'ADN à partir de l'origine (ori), puis formation de deux fourches de replication menant à la structure 0. Souvent simultanément à la replication, la production d'ARNm par l'ARN polymerase bactérienne se fait à partir de promoteurs phagiques. Cette transcription est généralement divisée en trois phases selon les modules de transcription, soit ceux des gènes précoces, des gènes médians et des gènes tardifs (Kutter et a l , 2005). Les protéines associées aux premiers permettent de prendre le contrôle de l'hôte et de protéger le génome phagique. Les gènes médians sont plutôt impliqués dans la replication de l'ADN du phage. Finalement, les gènes tardifs codent pour la morphogenèse des particules phagiques. Plusieurs protéines produites sont dites structurales étant donné qu'elles sont directement retrouvées dans la structure du virion mature. Selon les phages, l'ARN polymerase bactérienne pourra être modifiée pour la transcription des autres modules de transcription. Certains produiront un nouveau facteur sigma, d'autres des protéines liant l'ADN et d'autres encore une ARN polymerase d'origine phagique (Kutter et al, 2005).
Les particules virales sont, d'une part, assemblées en combinant une molécule d'ADN à une capside préformée (les procapsides). L'ensemble forme alors les nucléocapsides. D'autre part, les queues sont produites parallèlement, puis ajoutées à la nucléocapside pour former les particules complètes. D'autres éléments structuraux peuvent compléter la particule virale, comme des collets ou des fibres caudales. La capside est constituée de nombreuses unités d'une même ou de deux protéines (MCP, Major Capsid Protein) qui se regroupent pour former des ensembles structurels identiques, les capsomères. Des protéines d'échafaudage (scaffold proteins) interagissent avec les MCP pour assurer l'assemblage approprié de la capside. Les capsomères s'assemblent alors à partir du complexe de la protéine portale.
L'encapsidation de l'ADN concatémérique se fait différemment selon si le génome du virus contient des sites cos ou des sites pac (Rao et Feiss, 2008). Dans tous les cas, les
mécanismes d'encapsidation nécessitent l'action d'une protéine multifonctionnelle composée de deux sous-unités (la petite et la grande), la terminase ATPase (Catalano, 2000; Rao et Feiss, 2008). Lors de la translocation de l'ADN, elle se lie au concatémère, clive à un premier site, puis s'attache au complexe du portail. Elle utilise alors l'énergie d'hydrolyse de l'ATP pour faire entrer l'ADN dans la capside. Il a été montré chez certains phages qu'une molécule d'ATP est nécessaire pour chaque deux paires de bases (Guo et al, 1987; Morita et al, 1993). Une fois le génome entré, la terminase clive à un second site. Des taux d'encapsidation moyen de 119 pb/s et de 140 pb/s ont été estimés pour le phage lambda (k) (Yang et al, 2009) et pour le phage T7 (Cerritelli et al, 1997). Durant l'encapsidation de type cos, l'enzyme reconnaît des sites cos et clive l'ADN. Le clivage génère des extrémités cohésives qui faciliteront la recircularisation lors de l'injection ultérieure de l'ADN. En ce qui concerne les virus utilisant les sites pac, la terminase clive au premier site pac du concatémère et l'enzyme arrêtera l'encapsidation qu'une fois la capside totalement remplie (le mécanisme de la tête pleine). Ce mécanisme est même utilisé chez certains phages sans utiliser des sites de reconnaissance spécifiques (Black, 1989). Par la suite, la terminase, toujours liée au concatémère, rejoindra une autre procapside. Pour les phages de type pac, il y aura toujours présence d'une redondance dans le génome encapsidé (généralement de 2 % à 10 % du génome [Rao et Feiss, 2008]). La portion répétée permettra la recombinaison homologue comme processus de recircularisation de l'ADN, une fois celui-ci injecté dans le cytoplasme bactérien. Le nombre de procapsides remplies par concatémère varie : deux à trois pour X,, trois à quatre pour Tl et PI, et trois à cinq pour P22 (Black, 1989).
Les queues des virus, parallèlement assemblées aux capsides, sont formées d'un long tube central qui laissera passer l'ADN lors de l'injection dans la cellule hôte. Ce tube est formé de protéines identiques (les protéines majeures de la queue [MTP, Major Tail Protein]) qui s'alignent autour d'un complexe initiateur composé de la protéine étalon (TMP, Tape Mesure Protein) et des protéines de la plaque basale, dont la protéine de liaison au récepteur [RBP, Receptor- Binding Protein]. Cet alignement se fait à partir de l'extrémité interagissant avec la bactérie (l'extrémité distale) jusqu'à celle liée à la capside (l'extrémité proximale) (Kutter et al, 2005). C'est la TMP qui déterminera la longueur de la
queue. Une fois la formation caudale terminée, les complexes sont liés aux nucléocapsides, scellant la capside définitivement.
Au terme du processus, qui peut prendre plusieurs minutes à plusieurs heures selon les phages caudés, des virions matures seront relâchés dans l'environnement extracellulaire. Cette étape fait appel à la lyse cellulaire, dans laquelle deux protéines sont impliquées : la holine (Wang et al, 2000; Young 2002) et l'endolysine (Fishetti, 2010). Elles effectuent respectivement la perméabilisation de la membrane cytoplasmique et la dégradation du PDG à travers les pores formés par la holine. La lyse cellulaire nécessite une synchronisation parfaite afin d'optimiser le nombre de particules virales produites. Les phages ayant de petits génomes à ADN ou à ARN n'ont pas ce système lytique à deux composantes; ils interfèrent plutôt dans la synthèse du PDG (Fishetti, 2010). Fait à noter, les phages à ADN filamenteux, comme ceux de la famille des Inoviridae, ne sont pas libérés suite à une lyse cellulaire, mais le sont plutôt directement au travers de l'enveloppe bactérienne (phénomène appelé extrusion). Les virus de la famille des Plasmaviridae utilisent quant à eux le bourgeonnement. Dans ces deux cas, les bactéries survivent et continuent à se diviser malgré la production de phages, bien que leur croissance puisse être tout de même ralentie (Ackermann, 2005).
L.3:3.2..Lesphage.s..tempéré.s..et..le.çyç
Les virus, une fois l'ADN injecté, peuvent aussi demeurer sous forme latente (les prophages) dans les souches bactériennes. Ce phénomène est appelé la lysogénie et consiste en la présence d'un génome viral intégré dans le chromosome bactérien ou, parfois, présent dans la cellule sous forme plasmidique (Guttman et al, 2005). La bactérie qui contient un prophage est alors dite lysogène. Un virus possédant un module fonctionnel de lysogénie dans sa séquence génomique peut ainsi effectuer ce cycle lysogénique. Le cas échéant, l'intégration dans le chromosome s'effectue à des sites spécifiques nécessitant l'action d'une recombinase phagique (l'intégrase), et des protéines de l'hôte. Le phage X, par exemple, possède les sites attP qui permettent l'intégration aux sites attB de sa souche hôte (-'Escherichia coli (Groth et Calos, 2004). Le virus intégré est maintenu dans cet état par un répresseur qui se fixe à des endroits précis sur l'ADN du prophage. Il empêche ainsi toute
autre transcription virale, que ce soit du prophage ou d'un nouveau phage ayant infecté la souche bactérienne et qui serait lui aussi spécifique à ce répresseur. Durant le cycle lysogénique, le virus est répliqué en même temps que son hôte et est transmis aux cellules filles. Ce n'est que lorsque l'environnement sera défavorable à la croissance bactérienne que le phage réamorcera son cycle lytique, menant à la production de virions, puis à la mort cellulaire. Cet état de prophage peut être très stable, mais il a été montré qu'il est possible de les forcer à s'exciser (par induction) en utilisant les rayons UV par exemple (Lederberg, 1951). L'état latent, combiné au taux naturel de mutations de la bactérie, peut aussi mener à la présence des prophages défectifs dans les génomes bactériens (Casjens, 2003).
L3.4..L'impact;en.contexte.de la.transfo
Dans plusieurs fermentations industrielles, la propagation d'une infection phagique peut avoir des effets négatifs importants. De fait, lorsque le taux de lyse cellulaire est trop important (> IO4 phages/ml [Moineau et Lévesque, 2005]), le goût et la texture des produits
fermentes sont touchés et cela peut représenter des pertes économiques importantes. Cette multiplication rapide et massive des phages se traduit par une mort rapide de la culture bactérienne et empêche de terminer la production. Cela préviendra, par exemple, l'acidification adéquate du produit fermenté. Avec les années, les usines ont développé différentes stratégies de contrôle des phages afin de réduire les risques d'échecs fermentaires. Les souches bactériennes utilisées dans les ferments sont ainsi choisies selon leur résistance aux phages et une rotation des ferments est aussi effectuée dans les usines afin de réduire la propagation phagique. La sensibilité phagique peut varier en fonction des BL impliquées. Par exemple, pour des raisons encore mal comprises, les souches de S. thermophilus sont plus sujettes à l'infection phagique que les lactobacilles (Chandan, 2006).
Il faut aussi savoir que les sources de contaminations phagiques sont nombreuses. Comme toutes les bactéries peuvent être théoriquement sujettes à l'infection phagique, le lait utilisé pour la fermentation peut lui-même contenir des phages (Madera et al, 2004). De plus, comme plusieurs phages résistent à la pasteurisation (Chopin, 1980), ils ne sont pas nécessairement éradiqués lors de ce traitement thermique. La propagation phagique est
alors possible si les souches utilisées sont sensibles aux virus présents. Parallèlement, l'environnement industriel peut contribuer à la propagation des phages, que ce soit par la production (les cuves de fermentation, le lactosérum, les produits finaux), l'air, les surfaces, le personnel, etc. Un mauvais contrôle de la population phagique peut donc mener à une grande dissémination. Le choix des souches bactériennes est aussi un élément important : si les souches contiennent des prophages, ceux-ci peuvent s'exciser naturellement et se propager. Fait intéressant, certaines souches possédant des prophages sont tout de même utilisées, car la lyse bactérienne provoquée par les prophages excisés libère des enzymes contribuant à la flaveur des produits (Feirtag et McKay, 1987; O'Sullivan et al, 2000).
1.4 Les bacteriophages infectant Streptococcus thermophilus
L4:.l.._.Le.s„çaraçtér^
Il a été rapporté que plus de 345 phages de S. thermophilus ont été isolés de cultures de ferments industriels (Quiberoni et al, 2010). Ces phages montrent généralement une grande adaptation pour une prolifération rapide en milieu industriel, avec entres autres de courts temps de latence (20 à 40 minutes), des tailles de progéniture importantes (190 à 600 phages relargués par cellule infectée) et une résistance aux températures élevées. Tous les phages infectant S. thermophilus isolés jusqu'à maintenant appartiennent à la famille des Siphoviridae (à queue non contractile) de l'ordre des Caudovirales (à ADNdb). La plupart des phages isolés sont de type virulent (cycle lytique), mais quelques phages tempérés (cycle lysogénique) ont été isolés ou identifiés dans les génomes bactériens (Quiberoni et al, 2010). Parmi les quatre souches bactériennes dont le génome complet est disponible, seule la souche CNRZ1066 possède un prophage (Bolotin et al, 2004). Cependant, des gènes ayant des homologies avec des gènes de phages sont présents chez les trois autres souches de S. thermophilus : quatre gènes chez LMG18311, huit chez LMD-9 et 10 chez ND03. Il est donc possible que ces souches bactériennes aient déjà contenu des prophages.
Les phages infectant S. thermophilus forment un groupe très homogène pour lequel au moins deux sous-groupes existent. Ces phages sont ainsi distingués selon le nombre de protéines majeures de structure et leur mode d'encapsidation de l'ADN phagique. Le
premier groupe comprend les phages ayant deux protéines majeures (26 et 32 KDa) et un mode de type cos, alors que le second comprend trois protéines majeures (13, 25 et 41 KDa) et un mode de type pac (Le Marrec et al, 1997). Dans la base de données publiques GenBank, 11 séquences génomiques complètes de phages de S. thermophilus sont disponibles (voir le tableau 1.2).
Tableau 1.2 - Liste des bacteriophages infectant l'espèce .S", thermophilus dont la séquence génomique est disponible dans la base de données publiques GenBank.
Nom du phage Numéro d'accession GenBank Taille du
génome Nombre d'ORF d'isolement Année GC % provenance Pays de
O1205 NC_004303 43 075 57 1997 38 France DTI NC_002072 34 815 45 1999 39 Canada Sfil9 NC_000871 37 370 45 1999 38 France Sfi21 NC_000872 40 739 50 1999 37 France Sfill NC_002214 39 807 53 2000 38 France 7201 NC_002185 35 466 46 2000 39 Pays-Bas 2972 NC_007019 34 704 44 2006 40 France 858 NC_010353 35 543 46 2008 39 France 5093 NC_012753 37 184 48 2009 38 N.D. ALQ13.2 NC_013598 35 525 44 2009 39 Argentine Abc2 NC_013645 34 882 48 2009 38 Argentine
* N. D. signifie que les données ne sont pas disponibles.
Les phages Abc2 (Guglielmotti et al, 2009), DT1 (Tremblay et Moineau, 1999), Sfil9 (Brussôw et al, 1994; Desière et al, 1998), Sfi21 (Brussôw et al, 1994; Desière et al, 2001) et 7201 (Stanley et al, 2000) forment le groupe des phages cos, alors que ceux de type pac sont ALQ13.2 (Guglielmotti etal, 2009), O1205 (Fayard et al, 1993; Stanley et al, 1997), Sfill (Brussôw et al, 1994; Lucchini et al, 1998), 2972 (Lévesque et al, 2005) et 858 (Barrangou et al, 2007; Deveau et al, 2008). La figure 1.5 montre l'alignement des génomes de phages dont la séquence complète est disponible, alors que le
tableau 1.2 résume les données disponibles sur ces onze séquences génomiques. Quiberoni et al. ont d'ailleurs identifié en 2010 les gènes faisant partie des « génomes centraux » (core genome) partagés entre tous les phages infectant S. thermophilus ou seulement ceux d'un même sous-groupe; la figure 1.6 illustre ces génomes centraux en comparant les phages modèles 2972 (de type pac) et DT1 (de type cos).
L.4.:2....Les.phages yx^W^J^Z~^J^.X.^S!C^i.SiJl%lS^£hilM
Le phage 2972 a été isolé en France en 1999 d'une fermentation de yogourt utilisant la souche S. thermophilus DGCC7710 productrice d'EPS (aussi publiée sous le nom RD534). Le génome phagique de 34,7 Kpb (44 ORF) a été rendu disponible en 2005 (NC_007019 [Lévesque et al, 2005]). Il s'agit du plus petit génome parmi les séquences disponibles de phages infectant S. thermophilus. Le phage possède une capside isométrique de 55 nm de diamètre et une longue queue non contractile de 260 nm (voir la figure 1.8). Diverses analyse ont permis d'établir qu'il s'agit d'un phage de type pac : il possède trois protéines majeures de structure (de 10, 29 et 40 KDa) et produit des fragments d'ADN sous-molaires de faible intensité lors de la digestion enzymatique de son génome (Labonté, 2005). Il a été supposé que le phage 2972 dérivait d'un phage tempéré, car il possède des éléments de lysogénie (par exemple, un gène codant pour la protéine Cro). Ce phage s'est bien adapté à sa souche hôte (Deveau et al, 2008; Duplessis, 2005) puisqu'il possède un taux d'adsorption variant entre 90% et 97% (après respectivement 15 et 10 minutes d'infection), a un temps de latence de 40 minutes et libère près de 200 phages par cellule infectée (à 42°C). Parmi les souches testées, le phage 2972 n'infecte que sa souche hôte.
Le phage DTI a quant à lui été isolé d'une production canadienne de mozzarella en 1996 (Tremblay et Moineau, 1999) qui utilisait la souche SMQ-301. Ce génome phagique a une taille de 34,8 Kpb, comprend 46 ORF et est disponible dans la base de données publiques GenBank (NC002072). Il possède une capside icosaédrique de 60 nm et une longue queue de 260 nm par 8 nm (figure 1.9). Il appartient aux phages cos et possède deux protéines majeures de structure (26 et 32 KDa). Son spectre lytique comprend cinq des vingt-et-une souches qui ont été testées (Tremblay et Moineau, 1999) et une infection relâche 276 ± 36 phages et pour un temps de latence de 25 minutes à 42°C. Une étude
M = :
SI Si
3 z 3 RI.1 I
l
n
El l i i l 51
s 1 ;i
203£ - o o 5 a -s -<_ r OO
ft 2 3
aS _
s s
CJ JE e M § fi 22 5. £3 a> t_ »u __ = U ._S3
t
ri
. 2 113
| 2M
h§
h?.
r» aAS.
§ "a ^ **» /-^ r . i ? <c *•* ^ u_ *a* _m. OO S ^ IT) « m-1 0 0 TO u f _ V a _ a> a» S =3 -o& ^ 3
*» _r" D , < 'ml __ C <_> a c_ <D "fl o t _ CI _< «J __ u __ £ **■*' M O O 2 fl(N p B MJJ T—I"S "^ -s
c °
ca m en c :S -^ o a. _ (U & _ ■ + - ( N B M « 4. W d ^ 3 t_3 <C QJ •^•-3 M J= <_■ ci -s 3 . . . QJp a s
M * &«
3s
S D O • O os «
S S u c _ — £ H , _ Q [N c t_> f** QJ G '-ti 2 D. _. 3 OJ 3 Qo c _o __ (U TD ox O i / . Bî C u o oo S. fl ' 5 <U _. « .S fl x E o c & ■ " _ _ 'OJ 3 H ^ **"* c_ P-00 • ^ H )**~\ 3 +■*• « ca -fl o O, fl.(N fl y. O Ji ° N ca 0> 3 ca 3 _ ox O i / . U "O c_ (U C o.-s QJ _ ■o as .«a b "-> o c C <y _ 35 •2 o -glff-8
C _ f-» SJ o S ca - s :is
.sro
__ § co ^ S u u i_ S w o S fe __ B u .fl ï**** p. «S a _T __ s a M j - 3 fa U 2 - o _3 "3 x CJ •u ■M«S
So
«n S £P " -C u fl. O i aj >-V >Â ^
y S, O H S- > > (U * ^ S «J § ^ O Va U M ca D. _çj N (U J 5 CJ _ -OJË
OJ va C O c j ca va OJ - a'I
c OJ - 4 J C <u 75. QJ C 3 OJ _ r o « o r . <ca _ OT fN rsl G M g M U a « c — 2. o __ -A "*5 C_5 rO) oo - T . bl) e O fl — Ë ° ,fl "N fl ^ 'fl —^ S a *m C c oII
S T3 ca u — -o«S
« -5 o QJ _ QJs e
I — c **. Q J QJ L . 3 — 0 0 21parue en 2001 (Duplessis et Moineau, 2001) a montré que Yorfl8 est impliqué dans le spectre lytique du phage et que c'est la région variable VR2 qui est plus particulièrement responsable de cette spécificité. Binetti et al. (2005) ont d'ailleurs développé une méthode de détection des phages de S. thermophilus de type PCR (Polymerase Chain Réaction) utilisant cette région pour l'amplification de l'ADN. Le phage DTI possède également une protéine de type Cro; il dériverait ainsi lui aussi d'un phage ancêtre de type tempéré (Tremblay et Moineau, 1999).
En 2005, Duplessis et al. ont publié des analyses d'expression temporelle des gènes des phages 2972 et DT1. Il s'agit de la seule étude transcriptionnelle pour un phage de type pac (2972) et la première pour les phages de type cos (DT1) depuis celles de Ventura et al (2002a, 2002 b, 2004) pour les phages Sfil9 et Sfi21. Les transcriptomes à l'état de prophage des phages Sfi21 et O1205 (pac) ont aussi été publiés (Ventura et al, 2002b). Les analyses des phages 2972 et DT1 ont permis d'associer chacun de leurs gènes à l'un des trois modules généraux de transcription identifiés. Ainsi, DT1 possède 19 gènes précoces (transcrits au maximum entre 0-2 min), 12 gènes médians (2-7 min) et 12 gènes tardifs (7-22 min), alors que 2972 possède 16 gènes précoces (0-7 min), 11 gènes médians (7-(7-22 min) et 14 gènes tardifs (12-22 min). L'expression globale des gènes est similaire pour les deux phages, mais celle de DT1 est plus rapide que celle du phage 2972. Cela peut être dû entre autres à une transcription plus efficace pour DT1, à une différence due au mode d'encapsidation ou à des différences au niveau des souches hôtes. De plus, chacun des deux phages possède trois gènes appelés gènes tardifs #2; il s'agit de gènes ayant des profils de transcription différents des autres gènes tardifs, car ils sont exprimés plus tôt, mais de façon beaucoup plus graduelle. Il s'agit entre autres des gènes codant pour la holine et l'endolysine. Cette transcription temporelle particulière doit être adaptée au processus de lyse cellulaire qui doit être parfaitement synchronisé (voir la section 1.3.3.1) et qui nécessite une accumulation progressive des transcrits et des protéines. Ce phénomène n'a cependant pas été observé pour les phages Sfil9 et Sfi21.
Figure 1.8 - Photo du phage virulent 2972 observé en microscope électronique à transmission. Figure modifiée de Lévesque et al. (2005).
Figure 1.9 - Photo du phage virulent DT1 observé en microscope électronique à transmission. Figure tirée du site web du centre de référence pour virus bactériens Félix d'Hérelle de l'Université Laval (www.phage.ulaval.ca/).
1.5 Les mécanismes de résistance aux bacteriophages chez les bactéries lactiques LS. 1 Leg moyens -je défense anti-pfr»gç-*j
Les BL possèdent des systèmes de défense contre les phages (Labrie et al, 2010; Stern et Sorek, 2011; Sturino et Klaenhammer, 2004b), qui sont divisés selon leurs effets sur le cycle de multiplication phagique. Ils comprennent l'inhibition de l'adsorption, l'inhibition de l'injection de l'ADN, les systèmes de restriction-modification (RM), les systèmes CRISPR/Cas et les systèmes d'avortement de l'infection (Abi, Abortive Infection). Tous ces mécanismes ciblent une ou des étapes de la replication virale. Dans le cas de celle de l'adsorption, l'inhibition peut provenir d'une modification du récepteur bactérien ou du camouflage de celui-ci par des composantes extracellulaires. L'injection de l'ADN peut être bloquée bien que le mécanisme ne soit pas bien compris. Certaines bactéries vont pouvoir aussi agir sur l'ADN à l'aide des systèmes RM et CRISPR/Cas (voir les sections 1.5.2 et 1.6). Ces derniers peuvent, dans certains cas, cibler plutôt l'ARN. Finalement, les systèmes Abi regroupent plusieurs mécanismes agissant entre le moment de l'entrée de l'ADN et la fin du cycle phagique (Chopin et al, 2005). Ceux-ci vont prévenir la propagation phagique en provoquant la mort cellulaire. Ce suicide altruiste permet ainsi à la population de résister à la multiplication phagique.
En ce qui concerne S. thermophilus, peu de mécanismes de défense naturels ont été identifiés. L'absence de plasmide peut expliquer, entre autres, cette rareté étant donné que les mécanismes RM et Abi sont souvent encodes par des plasmides. Chez S. thermophilus, aucun mécanisme ciblant le blocage de l'entrée de l'ADN n'ont été identifiés. Chez certains phages, l'anti-récepteur est connu, mais le récepteur bactérien n'a pas encore été identifié. Ensuite, les mécanismes Abi agissent à différents moments du cycle lytique : replication du génome, transcription, traduction, encapsidation et assemblage des particules phagiques. Une vingtaine de systèmes Abi a été identifié chez la BL Lactococcus lactis, mais un seul système de type Abi a été supposé chez S. thermophilus (Larbi et al, 1992); cependant, il n'a été ni clone ni caractérisé. Ainsi, les systèmes RM et CRISPR demeurent les systèmes les plus présents et les mieux documentés pour la bactérie S. thermophilus. Étant donné le propos de l'étude, les systèmes RM et CRISPR seront détaillés aux sections 1.5.2 et 1.6 respectivement.