T
T
H
H
È
È
S
S
E
E
En vue de l'obtention du
D
D
O
O
C
C
T
T
O
O
R
R
A
A
T
T
D
D
E
E
L
L
’
’
U
U
N
N
I
I
V
V
E
E
R
R
S
S
I
I
T
T
É
É
D
D
E
E
T
T
O
O
U
U
L
L
O
O
U
U
S
S
E
E
Délivré par l'Université Toulouse III - Paul SabatierDiscipline ou spécialité : Biologie Cellulaire - Biologie Moléculaire
JURY
Dr. Pierre-Emmanuel GLEIZES, DR CNRS, Professeur UPS - Président Dr. Jean-Noël FREUND, DR INSERM - Rapporteur
Dr. Olivier JEAN-JEAN, DR CNRS - Rapporteur Dr. Théophile OHLMANN, DR INSERM - Rapporteur
Dr. Christiane SUSINI, DR INSERM
Dr. Stéphane PYRONNET, CR 1 - Directeur de thèse
Ecole doctorale : Biologie-Santé-Biotechnologies de Toulouse Unité de recherche : INSERM U858, I2MR
Directeur(s) de Thèse : Dr. Stéphane Pyronnet
Rapporteurs : Dr. Jean-Noël FREUND, Dr. Olivier JEAN-JEAN, Dr. Théophile OHLMANN
Présentée et soutenue par Amandine Alard Le 22 octobre 2009
Titre : Régulation de la dégradation protéasome-dépendante du facteur d'initiation de la traduction, eIF4G.
Régulation de la dégradation du facteur d’initiation de la traduction eIF4G par le protéasome.
eIF4G possède un rôle essentiel dans le processus d’initiation de la traduction. Protéine d’échafaudage du complexe eIF4F, elle permet le recrutement de la petite sous-unité du ribosome en 5’ des ARN messagers. Deux homologues fonctionnels d’eIF4G ont été décrits, eIF4GI et eIF4GII. Ils possèdent cependant certaines fonctions propres, et subissent des mécanismes de régulation différents.
Dans un premier temps, nous avons montré qu’eIF4GI interagit avec la NAD(P)H quinone oxydoréductase 1, NQO1. Possédant des fonctions de neutralisation du stress oxydatif par réduction des quinones, elle est également impliquée dans la régulation de la dégradation, ubiquitine-indépendante, de certaines protéines par le protéasome 20S. eIF4GI est stabilisée en présence d’inhibiteurs du protéasome et semble donc subir une dégradation protéasome-dépendante. Par ailleurs, l’utilisation d’un inhibiteur de NQO1, le dicoumarol, entraine une disparition d’eIF4GI, disparition abolie par la lactacystine, un inhibiteur irréversible du protéasome. L’invalidation de NQO1 par la technologie des siRNAs, entraine une perte d’eIF4GI, et à l’inverse, l’augmentation des quantités cellulaires de NQO1 par transfection de son ADNc permet de stabiliser eIF4GI. Par ailleurs, des expériences de co-immunoprécipitation nous ont permis de démontrer qu’eIF4GI se dissocie préalablement du complexe eIF4F avant d’être dégradée par le protéasome. Dans une situation plus physiologique, la régulation de la stabilité d’eIF4GI par le protéasome semble être un mécanisme important lors d’un stress oxydatif.
Nous avons également démontré une interaction directe entre la Polo-like kinase (Plk) 2 et eIF4GII. Cette interaction entraine une dégradation protéasome-dépendante et ubiquitine-indépendante d’eIF4GII. De plus, nous avons identifié un résidu dans la partie N-terminal d’eIF4GII, la sérine 267, dont la phosphorylation abolit la liaison de Plk2 et protège ainsi eIF4GII d’une dégradation par le protéasome. In vitro, l’interaction entre eIF4GII et le protéasome 26S, mais pas le protéasome 20S, est augmentée lorsqu’eIF4GII est préalablement liée à Plk2, suggérant que Plk2 agit comme un adaptateur spécifique d’eIF4GII pour le protéasome 26S. Plk2 ayant été décrite comme une cible transcriptionnelle de p53, l’utilisation d’agents génotoxiques, activant p53, entrainent une dégradation d’eIF4GII dépendante du protéasome et de Plk2. Nous proposons alors un modèle selon lequel Plk2 contrôle l’initiation de la traduction protéique en modulant la quantité cellulaire d’eIF4GII.
Nos travaux ont ainsi pu mettre en évidence deux mécanismes originaux de régulation de la dégradation par le protéasome des facteurs d’initiation de la traduction eIF4GI et II, et donc de régulation de l’initiation de la traduction protéique. Nous avons aussi décrit un activateur de cette dégradation, la Polo-like kinase 2, ainsi qu’un inhibiteur, la NAD(P)H quinone oxydoréductase, NQO1.
Sommaire
Avant propos : ... 8
\ÇàÜÉwâvà|ÉÇ U|uÄ|ÉzÜtÑ{|Öâx
\ÇàÜÉwâvà|ÉÇ U|uÄ|ÉzÜtÑ{|Öâx
\ÇàÜÉwâvà|ÉÇ U|uÄ|ÉzÜtÑ{|Öâx
\ÇàÜÉwâvà|ÉÇ U|uÄ|ÉzÜtÑ{|Öâx
... 9
CHAPITRE 1 : Plk2 et la famille des Polo-like kinases : ... 10
I : Introduction générale :... 10
II : Structure des Polo-like Kinases : ... 12
A : Le domaine PBD : ... 12
B : Fonctions du PBD : ... 14
1 : Localisation cellulaire : ... 14
2 : Reconnaissance du substrat : ... 14
3 : Inhibition de l’activité kinase par liaison intramoléculaire : ... 16
C : Séquence de localisation nucléaire : ... 16
III : Régulation des Plks : ... 17
A : Régulation transcriptionnelle : ... 17
B : Régulation de l’activité kinase : ... 19
Phosphorylations : ... 19
a : Phosphorylations de la boucle d’activation ou T-loop : ... 19
b : Déphosphorylation du T-loop : ... 20
C: Dégradation par le protéasome: ... 20
IV : Principales fonctions des Plks au cours du cycle cellulaire : ... 21
A : Principales fonctions de Plk1 : ... 21
1 : Fonctions Mitotiques : ... 21
a : Entrée en mitose : ... 21
b : Mise en place du fuseau mitotique et maturation des centrioles : ... 21
c : Régulation de la cohésion des chromatides sœurs et de la fonction des kinétochores : ... 22
d : Sortie de mitose : ... 22
e : Cytokinèse : ... 22
2 : Activation des points de contrôle du cycle cellulaire ou checkpoints : ... 23
a : Point de contrôle mitotique : ... 23
b : Stress génotoxiques et point de contrôle de la phase S : ... 23
c : Plk1 et p53 : ... 23
B : Principales fonctions de Plk3 : ... 23
1 : Fonctions mitotiques : ... 24
a : Entrée en mitose : ... 24
b : Régulation de l’entrée en phase S : ... 24
c : Fragmentation de l’appareil de Golgi : ... 24
3 : Réparation de l’ADN : ... 24
4 : Activation des checkpoints : ... 24
C : Principales fonctions de Plk4 : ... 25
1 : Fonctions mitotiques : ... 25
a : Duplication des centrioles : ... 25
b : Sortie de mitose : ... 26
c : Plk4 et p53 : ... 26
V : Plk2 dans tous les détails… ... 26
A : Expression dans des cellules en culture : ... 26
B : Expression tissulaire : ... 27
2 : Tissus tumoraux : ... 27
C : Fonctions de Plk2 relatives au cycle cellulaire : ... 27
1 : Duplication des centrioles : ... 28
2 : Activation des checkpoints : ... 28
a : Plk2 et p53 : ... 28
b : Checkpoint de la phase S : ... 28
c : Checkpoint mitotique : ... 29
D : Localisation cellulaire : ... 29
E : Fonctions originales de Plk2 dans les neurones post-mitotiques : ... 30
VI : Polo-like kinases et Cancer : ... 30
A : Plk1 et cancer : ... 30
1 : Expression dans les tissus tumoraux : ... 30
2 : Plk1 est un facteur de mauvais pronostic : ... 31
B : Plk2 et cancer : ... 31
C : Plk3 et cancer : ... 32
D : Plk4 et cancer : ... 33
E : Thérapeutiques anticancéreuses basées sur les Plks : ... 33
VII : Polo-like kinases et protéasome : ... 34
A : Dégradation de Plk1 : ... 34
1 : Plk1 possède une D-box : ... 34
2 : Plk1 est ubiquitinylée par le complexe APC/C : ... 35
3 : Plk1 est également ubiquitinylée par Chfr : ... 35
B : Dégradation de Plk2 : ... 35
C : Dégradation de Plk3 : ... 36
D : Dégradation de Plk4 : ... 36
E : Protéines dont la dégradation est induite par les Plks : ... 37
1 : Dégradations protéasome-dépendantes induites par Plk1 : ... 37
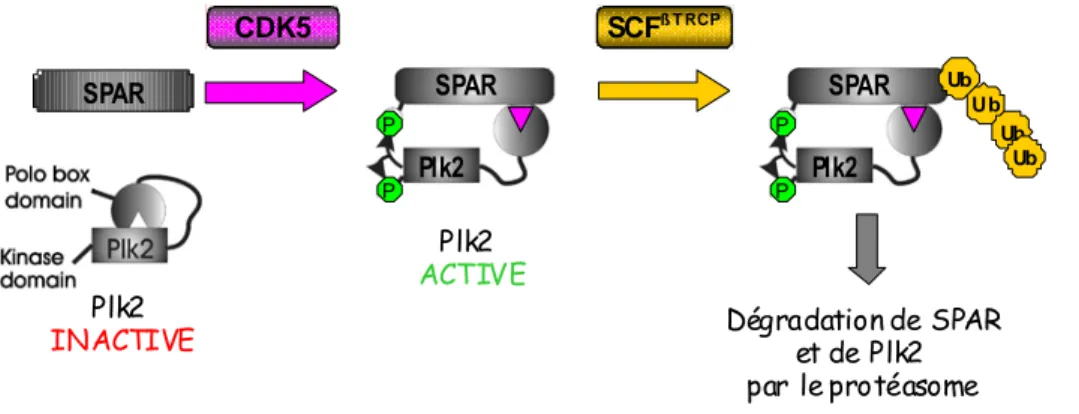
2 : Plk2 et SPAR : ... 37
F : Plk1 interagit avec le protéasome et régule son activité : ... 38
VIII : Fonctions des Plks en dehors du cycle cellulaire : ... 38
A : Plks et régulation de l’entrée en méiose : ... 39
B : Plks et mise en place de divisions asymétriques : ... 39
1 : Expression de Plk2 et Plk3 dans le cerveau : ... 40
2 : Plk2 et régulation de la plasticité synaptique : ... 41
CHAPITRE 2 : Les protéasomes : ... 45
I : Introduction générale :... 45
II : La Structure des Protéasomes :... 46
A : Le protéasome 20S : ... 46
1 : Structure moléculaire : ... 46
2 : Structures alternatives : ... 48
3 : Structure inactive, fermée : ... 49
4 : L’ouverture de la chambre catalytique : ... 50
B : Structure et fonctions de la sous-unité régulatrice 19S : (voir figure 1) ... 50
1 : La base : ... 51
2 : Le lid : ... 52
C : Les autres sous-unités régulatrices : ... 52
1 : Le PA28αβ ou 11S : ... 52
2 : Le régulateur nucléaire PA28γ ou REGγ : ... 53
3 : PA200 chez les mammifères ou Blm10 chez les levures : ... 54
D : Les inhibiteurs du protéasome : ... 54
1 : PI31 et PR39 : ... 54
2 : Les inhibiteurs synthétiques ou bactériens : ... 55
III : L’ubiquitinylation des protéines : ... 56
B : Mécanisme classique de la voie d’ubiquitinylation des protéines : ... 58
C : La grande famille des molécules proches de l’ubiquitine : ... 60
1 : UBLs (Ubiquitin-like proteins) ou UBL modifiers : ... 60
2 : Les domaines ULDs ou Integral Ubiquitine-Like Domains : ... 61
D : L’hydrolyse de l’ubiquitine et les DUBs (Deubiquitinating enzymes) : ... 62
E : L’ubiquitine chez les procaryotes : ... 63
1 : Conséquences d’une mono-ubiquitinylation : ... 64
2 : Conséquences d’une poly-ubiquitinylation : ... 65
IV : La dégradation par le protéasome, ubiquitine-indépendante : ... 67
A : Introduction : ... 67
B : Activité endoprotéolytique du protéasome : ... 68
L’exemple de la maturation d’YB-1 : ... 68
C : La dégradation par défaut du protéasome 20S : ... 69
V : NQO1 et le protéasome 20S : ... 71
A : Généralités concernant NQO1 : ... 71
B : Fonctions de détoxification de NQO1 : ... 72
C : Induction de NQO1 : ... 72
D : Inhibiteurs de NQO1 : ... 73
1 : Le dicoumarol : ... 73
2 : La curcumine : ... 73
E : NQO1 et cancer : ... 73
1 : Propriétés anti-oncogéniques de NQO1 : ... 73
a : Propriétés anti-oxydantes : ... 73
b : Bio-activation de quinones anti-tumorales : ... 74
c : Stabilisation de p53 : ... 74
2 : Absence de NQO1 et susceptibilité accrue à face au cancer : ... 74
3 : Surexpression de NQO1 dans les cancers : ... 75
F : NQO1 et protection de la dégradation par défaut du protéasome 20S : ... 76
1 : La dégradation de p53 : ... 76 a : Dégradation ubiquitine-dépendante :... 77 b : Dégradation ubiquitine-indépendante : ... 78 NQO1 et p53 : ... 78 NQO2 et p53 : ... 79 2 : La dégradation de p73 : ... 79 3 : La dégradation de l’ODC : ... 80
a : Dégradation dépendante de l’Antizyme-1 (AZ-1) : ... 80
b : NQO1 et ODC : ... 81
4 : La dégradation de p33 : ... 82
5 : Conclusions : ... 83
CHAPITRE 3 : eIF4G et l’initiation de la traduction protéique : .... 85
I : Introduction générale :... 85
II : L’initiation de la traduction cap-dépendante chez les eucaryotes : .... 86
A : Structures des ARNm impliquées dans l’initiation de la traduction cap-dépendante : ... 88 1 : La coiffe : ... 88 2 : La queue poly(A) : ... 88 B : La formation du complexe 43S : ... 88 Le complexe ternaire : ... 89 C : Le complexe eIF4F : ... 89 1 : eIF4A : ... 90 2 : eIF4E : ... 90
3 : 4E-BP1 et inhibition de la formation du complexe eIF4F : ... 90
III : Rôles d’eIF4G dans l’initiation de la traduction cap-dépendante : ... 91
A : Structure générale d’eIF4G : ... 92
1 : Formation du complexe eIF4F : ... 94
a : Interaction avec eIF4E : ... 94
b : Interaction eIF4G et eIF4A : ... 94
eIF4G positionne intelligemment eIF4A : ... 94
eIF4G stimule l’activité ARN hélicase d’eIF4A : ... 95
2 : Recrutement de la petite sous-unité du ribosome en 5’ de l’ARNm : 95 4 : Circularisation des ARNm : ... 96
C : Rôle d’eIF4G dans la traduction IRES-dépendante :... 97
D : Les homologues d’eIF4G et protéines apparentées : ... 99
1 : eIF4GII : ... 99 2 : p97/NAT1/DAP5 : ... 100 3 : Paip1 : ... 101 4 : DUG : ... 102 5 : Off-schedule : ... 102
IV : Régulation d’eIF4G : ... 102
A : Expression d’eIF4G et ses Isoformes : ... 103
B : Phosphorylation d’eIF4G : ... 105
1 : Phosphorylation d’eIF4GI : ... 106
B : Phosphorylation d’eIF4GII : ... 108
C : Clivages d’eIF4G : ... 109
1 : Modifications d’eIF4G lors de l’apoptose : ... 109
a : Le clivage d’eIF4GI et d’eIF4GII : (Voir figure 7) ... 109
b : Phosphorylation apoptoptique d’eIF4GI : ... 112
2 : Clivages lors de l’infection virale : ... 112
a : Clivage par les protéases virales 2A : ... 114
b : Clivage par la protéase L du virus FMDV : ... 115
c : Clivage par d’autres protéases virales : ... 116
V : Rôle du protéasome dans la dégradation des facteurs d’initiation du
complexe eIF4F: ... 117
A : Dégradation ubiquitine-dépendante d’eIF4E : ... 117
B : La dégradation de 4E-BP1 : ... 118
1 : Dégradation ubiquitine-dépendante : ... 118
2 : Processus de maturation de 4E-BP1 par le protéasome : ... 118
C : Dégradation ubiquitine-indépendante d’eIF4G et d’eIF3a : ... 119
D: Régulation de la dégradationd’eIF4GI par p53 : ... 121
VI : eIF4G et cancers : ... 121
A : Surexpression d’eIF4G dans les cancers : ... 121
B : Phosphorylation d’eIF4GI dans les cellules cancéreuses : ... 122
C : L’effet d’une surexpression d’eIF4G sur les cellules : ... 122
e°áâÄàtàá XåÑ°Ü|ÅxÇàtâå
e°áâÄàtàá XåÑ°Ü|ÅxÇàtâå
e°áâÄàtàá XåÑ°Ü|ÅxÇàtâå
e°áâÄàtàá XåÑ°Ü|ÅxÇàtâå ... 125
RESULTATS EXPERIMENTAUX ... 126
ARTICLE 1 ... 126
Keywords: translation initiation, eIF4G, NQO1 ... 126
ARTICLE 1 : ... 127
La quinone oxydoréductase NQO1 protège eIF4GI d’une dégradation par le
protéasome ... 127
INTRODUCTION : ... 127
Keywords: translation initiation, eIF4G, NQO1 ... 128
ABSTRACT ... 128
INTRODUCTION ... 129
RESULTS ... 130
Oxidative stress provokes eIF4GI degradation by the proteasome . 139
DISCUSSION ... 140
MATERIALS AND METHODS ... 142
Cells and cell culture. ... 142
Compounds. ... 142
Plasmids, siRNAs and transfections. ... 142
Co-immunoprecipitation and Western-blotting. ... 142
ODC activity. ... 143
Quantification of western-blotting signals. ... 143
ACKNOWLEDGMENTS ... 143
REFERENCES ... 143
CONCLUSIONS ET PERSPECTIVES : ... 146
1 : Régulation de l’interaction : ... 146
2 : NQO1 et eIF4GII : ... 146
3 : Dégradation d’eIF4GI libre :... 147
4 : Dégradation par le protéasome et fragments de clivages d’eIF4G : ... 147
5 : Circonstances physiologiques ? ... 148
RESULTATS EXPERIMENTAUX ... 149
ARTICLE 2 ... 149
Plk2-targeted proteasomal destruction of eIF4GII ... 149
Keywords : translation initiation, eIF4G, PLk2, proteasome ... 149
ARTICLE 2 : ... 150
La Polo-like kinase Plk2 induit une dégradation protéasome-dépendante du
facteur d’initiation de la traduction eIF4GII ... 150
INTRODUCTION : ... 150
Plk2-targeted proteasomal destruction of eIF4GII ... 151
Abstratct ... 151
Luciferase activities. Dual luciferase activities were measured as
previously described
25. ... 168
Surface Plasmon Resonance. ... 168
Acknowledgments ... 168
References ... 168
CONCLUSIONS ET PERSPECTIVES : ... 171
1 : Plk2 et eIF4GI : ... 171
2 : Circonstances physiologiques de la dégradation d’eIF4GII : ... 171
3 : Réversion partielle de la dégradation par le Mg132 : ... 172
4 : Quelle est la kinase qui phosphoryle la sérine S267 d’eIF4GII : ... 173
5 : Comment Plk2 induit une dégradation protéasome–dépendante d’eIF4GII ? ... 174
VÉÇvÄâá|ÉÇ Z°Ç°ÜtÄx xà cxÜáÑxvà|äxá
VÉÇvÄâá|ÉÇ Z°Ç°ÜtÄx xà cxÜáÑxvà|äxá
VÉÇvÄâá|ÉÇ Z°Ç°ÜtÄx xà cxÜáÑxvà|äxá
VÉÇvÄâá|ÉÇ Z°Ç°ÜtÄx xà cxÜáÑxvà|äxá ... 175
CONCLUSION GENERALE et PERSPECTIVES ... 176
Modèle global de dégradation d’eIF4G dans les cellules : ... 176
Régulation de la dégradation d’eIF4G dans les cellules cancéreuses : ... 177
Plk2 et cellules cancéreuses : ... 177
NQO1 et cellules cancéreuses : ... 178
e°y°ÜxÇvxá U|uÄ|ÉzÜtÑ{|Öâxá
e°y°ÜxÇvxá U|uÄ|ÉzÜtÑ{|Öâxá
e°y°ÜxÇvxá U|uÄ|ÉzÜtÑ{|Öâxá
e°y°ÜxÇvxá U|uÄ|ÉzÜtÑ{|Öâxá ... 179
TÇÇxåx‹
TÇÇxåx‹
TÇÇxåx‹
TÇÇxåx‹ ... 205
4E-BP1 is a target of Smad4 essential for TGF
ββββ
-mediated inhibition of cell
proliferation ... 206
Avant propos :
La thèse que vous vous apprêtez à lire a pour objectif de présenter les résultats expérimentaux exposés dans la partie II.
Ces résultats concernent la dégradation protéasome-dépendante du facteur d’initiation eIF4G, dégradation régulée par deux protéines, Plk2 et NQO1.
Ainsi, l’introduction bibliographique se découpe en 3 chapitres. Dans une première partie seront présentées les Polo-like kinases et principalement Plk2. Suivra un chapitre consacré aux dégradations des protéines par le protéasome, dépendantes de leur ubiquitinylation, ou régulées par la protéine NQO1.
Enfin, l’initiation de la traduction protéique et les facteurs d’initiation eucaryotes eIF4GI et eIF4GII seront présentés dans un dernier et troisième chapitre.
Par la suite, les résultats expérimentaux seront présentés sous la forme de deux articles, introduits et discutés :
Article 1 : La Polo-like kinase Plk2 induit une dégradation protéasome-dépendante du facteur d’initiation de la traduction eIF4GII.
Article 2 : La quinone oxydoréductase NQO1 protège eIF4GI d’une dégradation par le protéasome.
Enfin, une conclusion générale et des perspectives à ces travaux seront présentées dans une dernière partie.
\ÇàÜÉwâvà|ÉÇ U|uÄ|ÉzÜtÑ{|Öâx
\ÇàÜÉwâvà|ÉÇ U|uÄ|ÉzÜtÑ{|Öâx
\ÇàÜÉwâvà|ÉÇ U|uÄ|ÉzÜtÑ{|Öâx
\ÇàÜÉwâvà|ÉÇ U|uÄ|ÉzÜtÑ{|Öâx
CHAPITRE 1 : Plk2 et la famille des Polo-like kinases :
I : Introduction générale :
Les Polo-like kinases sont des sérines-thréonines kinases impliquées dans la progression du cycle cellulaire. Elles ont également été décrites comme des éléments de réponse des à stress génotoxiques, activant les points de contrôle du cycle cellulaire (Dai W., et al., Oncogene 2005).
Elles doivent leur nom, polo, au phénotype observé chez la mouche à fruit, ou Dosophila Melanogaster. En effet, c’est dans ce modèle que le premier membre de cette famille de protéines kinases, Polo, fut identifié. Des mutations du gène polo dans ce même modèle entrainent de nombreuses anomalies au niveau des pôles des fuseaux de microtubules (Sunkel CE. and Glover DM., J.Cell Sci 1988). Quelques années plus tard, Llamazares et al. démontrent que le gène polo permet l’expression d’une protéine kinase (Llamazares S., et al., Genes Dev., 1991). Par la suite, un rôle mitotique majeur lui a été attribué (Fenton B. and Glover DM., Nature, 1993).
Décrits comme des régulateurs clés du cycle cellulaire, au même titre que les Cycline-Dependent Kinases ou CDKs, les Polo-like kinases sont très conservées lors de l’évolution. En effet, des homologues de structure et de fonction de ces protéines, sont retrouvés des levures, aux vertébrés (Barr FA., et al., Nat. Rev. Mol. Cell Biol., 2004). (Voir la Figure 1)
A ce jour, aucune protéine kinase présentant une homologie avec les Polo-like kinases n’a été décrite chez les plantes, les bactéries ou les archébactéries.
Chez les mammifères, la famille des Polo-like kinases compte 4 membres :
- Plk1, la plus étudiée des membres de la famille. Elle est un important régulateur mitotique, méiotique et de la cytokinèse (Archambault V. and Glover DM., Nat. Rev. Mol. Cell Biol., 2009).
- Plk2 ou Snk, (Serum inducible kinase), initialement décrite comme un régulateur de la phase G1 du cycle cellulaire, phase où elle a été décrite comme catalytiquement active (Ma S., Mol. Cell Biol., 2003), elle est également impliquée dans une réponse au stress et semble être une cible transcriptionnelle de p53 (Shimizu-Yoshida Y., Biochem. Biophys. Res. Commun., 2001) (Burns TF., et al., Mol. Cell Biol., 2003).
- Plk3 est impliquée dans la réponse des cellules à des stress génotoxiques (Archambault V. and Glover DM., Nat. Rev. Mol. Cell Biol., 2009). Initialement découverte
dans le milieu des années 1990 comme un facteur de croissance des fibroblastes murins, ou Fnk (FGF inducible kinase)
(Donohue PJ., et al., J. Biol. Chem., 1995),
son homologue humain a été découvert l’année suivante et nommé Prk (Proliferation-related kinase) (Li B., et al., J. Biol. Chem., 1996).- Plk4 ou Sak (Snk/Plk-akin kinase), membre original de la famille de par sa structure, elle a été originellement clonée chez la souris, en tant qu’homologue de la kinase de Drosophile, Polo. Son implication dans la prolifération cellulaire a alors été évoquée (Fode C., et al., PNAS, 1994).
Selon les espèces, différentes nomenclatures sont utilisées pour présenter les Plks. Ainsi, on retrouve les Plkx1-3 chez le Xénope, Plc1-3 chez C. elegans, polo chez D. melanogaster, et chez les levures, Plo1p pour S. Pombe et Cdc5 pour S. cerevisae. (Voir figure 1)
Cette famille de protéines kinases présente des fonctions essentielles dans la progression du cycle cellulaire mais aussi dans la réponse des cellules à différents stress génotoxiques. Les protéines kinases Plk2 et Plk3 possèdent quant à elles des fonctions
Or ganismes Plk Fonctions principales proposées Mammifères Plk1 Rôles principaux en mitose et
cytokinèse
Plk2 / Snk Réponse aux dommages de
l’ADN, régulation de la plasticité synaptique, Rôle en G1/S Plk3 / Fnk/ Prk Réponse aux dommages de
l’ADN, régulation de la plasticité synaptique, Rôle en G2/M Plk4 / Sak Sortie de mitose
Xenopus laevis Plx1 Entrée et sortie de mitose
Plx2 Non déterminées Plx3 Non déterminées
Drosophila melanogaster Polo Régulation de la mitose et
cytokinèse
Plk4 Duplication des centrioles
Caenorhabditis elegans Plc1, Plc2, Plc3 Nécessaire à la dissociation de
l’enveloppe nucléaire
Schizosaccharamyces pombe Plo1p Régulation de la mitose et
cytokinèse
Saccharomyces cerevisiae Cdc5 Régulation de la méiose, mitose
et cytokinèse Figure 1 : Les membres de la famille des p olo-like kinases :
originales dans les neurones post-mitotiques. Les fonctions relatives à chacune des Plks seront détaillées plus en aval
.
II : Structure des Polo-like Kinases :
Les Plks possèdent deux caractéristiques structurales très bien conservées au cours de l’évolution. Outre leur domaine catalytique en N-terminal, elles possèdent un domaine C-terminal particulier, composé de deux boîtes en tandem, dites « polo ». La région comprise entre les deux boîtes Polo diverge largement entre les différentes Plks. (Voir la Figure 2)
A : Le domaine PBD :
La partie C-terminale des Plks comprenant, les deux boîtes Polo et le domaine de jonction reliant ces deux dernières, est appelé PBD pour Polo Box Domain et possède des fonctions particulières dans la cellule (Lowery DM., et al., Oncogene, 2005).
Le domaine compris entre le domaine catalytique et le PBD est quant à lui appelé le Linker. (Voir la Figure 2)
Seule Plk4 diffère des trois autres membres de la famille des Plks par son domaine C-terminal. En effet, possédant une forte homologie avec les autres membres de la famille concernant le domaine kinase, son C-terminal ne compte quant à lui qu’une seule boîte Polo. Cette dernière confère à Plk4 des propriétés particulières. En effet, elle permet la
Linker
Adapté de :
Structure and func tion of Polo-like kinas es
(Lowery DM., et al., Oncogene, 2005)
Figu re 2 : Str ucture géné rale de s Plk s :
L es Plks possèdent une structure très conservée lors de l’évol uti on et en tre les di fférents membres de la fami lle. Ell es possèdent e n effet, un domaine Kinase en N-terminal et un d omaine appel é PBD, pour Po lo-Box Domai n, en C-termi nal, comptant l e plus so uvent de ux b oîtes Pol o, (ou PB pour Polo Box). La région comprise entre l e doma ine k inase et le PBD, p eu conservée entre les di fférentes Pl ks, est appel ée le linker (Numérotation b asé e sur la séque nce de Plk1 humaine)
dimérisation de la protéine kinase, in vitro et lors de sa surexpression dans les cellules (Leung GC., et al., Nat. Struct. Biol., 2002). Participant à une localisation correcte de la protéine, l'effet de cette dimérisation sur l'activité kinase de Plk4 n'est pas démontré.
L’alignement des structures des Plks de différentes espèces et des 4 Plks de mammifères sont présentées dans la figure 3.
La structure cristallographique du domaine PBD de Plk1 a été résolue, et montre que le PBD possède un site de liaison pour un résidu phopho-sérine ou phospho-tyrosine, situé entre les deux boîtes polo. L’histidine 538 et la lysine 540 sont par ailleurs indispensables à cette interaction (nomenclature de Plk1) (Elia AE., et al., Cell, 2003).
Des alignements de séquences révèlent que les boites Polo ne sont retrouvées sur aucune autre protéine connue, suggérant que les Plks possèdent des fonctions cellulaires exclusives.
Figure 3 : Représentation schématique des Polo-like kinases :
Le nombre de droite dénote la longueur de c haque protéine en acides aminés.
Le domaine en bleu, représente le domaine kinas e; PBD1 et PBD2, en vert, s ont les deux boîtes polo. On remarque que Plk4 ne c ompte qu’une s eule boîte polo.
Ad apté de :
Polo-like kinases, an intr oduction
B : Fonctions du PBD :
Le domaine PBD des Plks est critique pour la localisation cellulaire et l’interaction de ces protéines kinases avec leurs substrats. Il possède également une fonction importante dans le mécanisme d’activation des Plks.
Dans des cellules de mammifères, la surexpression des domaines PBD isolés provoque un arrêt pré-anaphasique (Seong YS., et al., J. Biol. Chem., 2002), et bloque la cytokinèse des levures à bourgeon (Song S., et al., J. Cell Biol., 2001). Ces phénotypes peuvent être expliqués par un rôle dominant négatif des domaines PBD isolés, empêchant la protéine kinase de remplir ses fonctions mitotiques propres.
1 : Localisation cellulaire :
Lee KS. et al. en 1998, démontrent que des mutations ponctuelles dans le domaine PBD de Plk1 entraînent une délocalisation de la kinase et la rendent incapable de complémenter des mutants thermosensibles de Cdc5 chez la levure (Lee KS., et al., PNAS, 1998). Les bases moléculaires de la fonction du PBD ont été mises en évidence en 2003. En effet, Elia et al. ont démontré que le PBD est un domaine capable d’interagir avec une protéine préalablement phosphorylée sur une sérine ou une thréonine (Elia AE., et al., Science, 2003).
Le motif consensus optimal reconnu par le PBD des Plks a été décrit : Ser-[pSer/pThr]-[Pro/X]. Ce site est par ailleurs le site consensus de phosphorylation par les Cdks ou les MAP kinases, suggérant que ces dernières puissent être à l’origine des phosphorylations « primaires », et donc de la création de sites de liaison au PBD des Plks, sur des protéines d’ancrage participant à la localisation cellulaire des Plks (Elia AE., et al., Science, 2003).
2 : Reconnaissance du substrat :
La liaison d’un substrat préalablement phosphorylé sur le PBD entraine une variation de conformation de la kinase, libérant alors son domaine catalytique (Elia AE., et al., Cell, 2003). Ensuite, la phosphorylation de Plk1 sur sa Thréonine 210, bloquerait la liaison intramoléculaire entre son PBD et son domaine catalytique (Jang YJ., et al., PNAS, 2002). En effet, la liaison intramoléculaire ne peut se mettre en place qu’avec des formes non-phosphorylées de Plk1 et non avec des mutants de Plk1 dans lesquels la thréonine 210
a été remplacée par un acide aminé acide (Jang YJ., et al., J. Biol. Chem., 2002). (Voir §, II, B, 3 et figure 4)
Deux modèles de phosphorylation, "en série" ou "distributive", sûrement complémentaires, ont alors été proposés (Lowery DM., et al., Oncogene, 2005).
Dans le modèle d’activation dit "en série", la kinase, activée par liaison d’un phosphopeptide sur son PBD et autophosphorylation de sa T210, peut alors également phosphoryler la protéine liée à son PBD et qui a permis son activation. Par exemple, Wee1 est un substrat de Plk1 et sa phosphorylation est fortement stimulée par une phosphorylation préalable de Wee1 par Cdk1 (Watanabe N., et al., PNAS, 2004).
Dans le second modèle de phosphorylation "distributive", la libération du domaine kinase se réalise grâce à une phospho-protéine qui n’est pas un substrat potentiel des Plks. Cependant, ainsi activée, la kinase Plk peut phosphoryler tout substrat à sa portée (Elia AE., et al., Cell, 2003). (Voir figure 4)
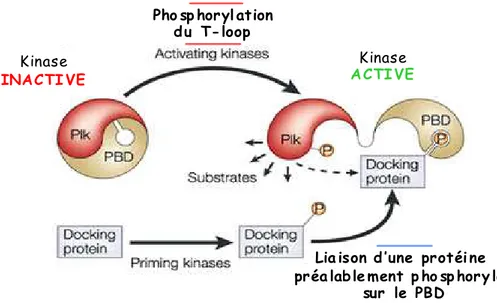
Pho sp horyl ation d u T-loop
Lia ison d ’une protéi ne préa lab le ment p ho sp hory lée
sur le PBD
Kinase
INACTI VE
Kinase
A CTI VE
Figure 4 : M odèle d ’activ ation des Plks :
Un e li aison in tramol é cu lair e du PB D et d u domai ne ki na se main ti ent la ki nas e da ns une confor mation i nactiv e. La ph osphoryl ati on d u T -loo p, ai ns i que l a l iai son d’ une pr otéi ne pr éal abl em ent pho sp ho ry lée sur le P BD, perm e tten t une v ari ation de con for mation, et acti vent l a kinase .
Pl k ainsi ac tiv ée, p eu t phos pho ryl er la protéi ne l iée à s on PB D, da ns un mo dèl e de phosphor yl ati on process iv e, ( ) e t p eut éga le me nt phop ho r yler de s su bstrats à proxi mi té da ns un mo dèl e de p hosph oryl ati on di stri bu tiv e . ( )
Adapt é d e:
Pol o-l ik e ki nas es an d the orc hestra ti on o f cel l d ivi sio n (Barr F A., et al ., Na t. Rev. M ol. Cell Biol. , 2004)
Des exemples de liaison sur des peptides phosphorylés ne manquent pas. Nous pouvons ainsi citer le domaine SH2 (Src Homoly Domain) des kinases de la famille Src, et PTB (PhosphoTyrosine Binding), ou des protéines de signalisation (Shc, IRS-1), qui interagissent avec des motifs type Tyrosine phosphorylée (Pawson T., et al., Trends Cell Biol., 2001). Il existe aussi des domaines "Forkhead-associated" et "double tryptophane" (WW), retrouvé par exemple sur la protéine Pin1, qui interagissent quant à eux avec des motifs pS (phospho-Sérine) ou pT (phospho-Thréonine) (Yaffe MB. and Elia AE., Curr. Opin. Cell Biol., 2001).
3 : Inhibition de l’activité kinase par liaison intramoléculaire :
Dans sa forme inactive, Plk1 adopte une conformation "repliée" dans laquelle son domaine catalytique est inhibé par interaction intramoléculaire avec son PBD (Jang YJ., et al., PNAS, 2002). Des expériences de délétions de différents domaines de Plk1 ont démontré cette interaction intramoléculaire inhibitrice. En effet, un mutant de Plk1 délété de sa partie C-terminale, le PBD, possède une activité kinase augmentée de plusieurs fois, suggérant que le PBD inhibe le domaine catalytique de la kinase (Lee KS. and Erikson RL., Mol. Cell Biol., 1997) (Mundt KE., et al., Biochem. Biophys. Res. Commun., 1997). Ce processus d’inhibition de l’activité kinase par liaison intramoléculaire est conservé pour Plk2. En effet, sa partie C-terminale inhibe son domaine catalytique (Burns TF., et al., Mol. Cell Biol., 2003).
C : Séquence de localisation nucléaire :
Plk1 se localise dans le cytoplasme et dans le noyau des cellules. Elle contient une séquence de localisation nucléaire bipartite impliquée dans son import nucléaire. En effet, les régions 134-136 et 143-146 sont toutes les deux nécessaires et suffisantes pour entrainer la localisation nucléaire de Plk1 dans les cellules Hela (Taniguchi E., et al., J. Biol. Chem., 2002).
Bien que ces séquences soient conservées pour Plk2 et Plk3, leur rôle dans leur localisation cellulaire n’a pas été démontré. Il semblerait même que Plk2 ait une localisation plutôt cytoplasmique (Kauselmann G., et al., EMBO J., 1999). La structure protéique de Plk4 quant à elle, présente uniquement le second cluster basique (van de Weerdt BC. and Medema RH., Cell Cycle, 2006).
La figure 5 présente les séquences de localisation nucléaire, prédictives ou démontrées, retrouvées sur les 4 Plks de mammifères.
III
: Régulation des Plks :
L’activité des Plks est régulée au niveau transcriptionnel, ainsi que par des phénomènes de phosphorylation et de protéolyse. Ces kinases sont également régulées au niveau spatial par des interactions protéiques favorisant la localisation cellulaire de la kinase au niveau de son site d’action.
A : Régulation transcriptionnelle :
Les transcrits de Plk1 présentent un pic d’expression lors de la transition G2/M du cycle cellulaire. Les facteurs de transcription de la famille des forkhead (FKH-TFs pour Forkhead transcription factors), par exemple FoxM1, stimulent la transcription de Plk1 dans les cellules de mammifères et les levures. En effet, l’expression des FKH-TFs augmente l’expression de Plk1 dans les cellules NHI3T3. De plus, des expériences de précipitation de la chromatine ont montré une interaction directe entre ce facteur de transcription et le promoteur de Plk1 (Alvarez B., et al., Nature, 2001) (Laoukili J., et al., Nat. Cell Biol., 2005).
F igur e 5 : St ructures particulières présentes sur les Plks (NLS et séquences PEST) :
Sont représentées : - les NLS prédictives ou démontrées. Aucune séquence NLS n’a pour l’instant été évoquée concernant Plk4. - les séquences PEST potentielles et démontrées. Aucune PESTn’a été prédite concernant Plk1. Par contre, cette der nière possède une boîte de destruction ou D-Box ( RKPL en position 336).
NLS potentielle NLS démontr ée
Plk4
Polo Box
PEST démontr ée PEST potentielle
L ég en de :
Adap té de :
Polo-like k inase 3 is r equired for e ntry into S phase(Zimmerman W. and Rikson R., PNAS, 2003)
Finding Plk3(Zimmerma n W. and R ikson R., Cell Cycle, 2007)
Plk4 and mitotic fidelity(Swallow CJ., et al., Oncogene , 2005
O rdered proteolysis in anaphase inactivat es Plk1 to contribute to proper mitot ic exit in human cells(Lind on C. and Pin es J., J. Cell Biol., 2004)
De plus, FoxM1 est un substrat de Plk1. En effet, ce facteur de transcription interagit avec le domaine PBD de Plk1, grâce à la phosphorylation préalable de Foxm1 par Cdk1, et répondant au schéma classique d’activation des Plks (voir § II, B, 2 et 3). Ensuite, Plk1 phosphoryle directement FoxM1 lors de la transition G2/M du cycle cellulaire, ce qui augment l’activité transcriptionnelle de FoxM1, nécessaire pour l’expression de régulateurs mitotiques clés, comme la cycline B (Laoukili J., et al., Nat. Cell Biol., 2005), la phosphatase Cdc25 et Plk1 elle-même (Wang IC., et al., Mol. Cell Biol., 2005).
A l’inverse, la transcription de Plk1 en phase G1 du cycle cellulaire est réprimée par un élément appelé CDE/CHR (cell-cycle-dependent element/cell cycle homology region). Ces séquences sont également présentes sur les promoteurs de gènes spécifiques de la phase G2/M du cycle cellulaire comme la cycline B, Cdk1, ou Cdc25C. Les connaissances relatives aux facteurs de transcription interagissant avec ces séquences sont très limitées (Martin BT., et al., Cell Cycle, 2006). Cependant p53 et sa cible transcriptionnelle p21Cip1, décrits comme des régulateurs négatifs de la transcription de Plk1 (Incassati A., et al., Oncogene 2006), semblent être de bons candidats (Martin BT., et al., Cell Cycle, 2006).
Un travail récent a démontré, dans des cellules humaines de carcinome alvéolaire, que la transcription des gènes codant pour les protéines kinases Plk1 et Plk3 sont sous la dépendance d’un promoteur répondant au facteur de transcription E2F1; facteur également impliqué dans la transcription de nombreux gènes importants pour la progression du cycle cellulaire. L’analyse des promoteurs de Plk1, Plk3 et Plk4 a montré que E2F1 est un trans-activateur de ces promoteurs (Tatequ M., et al., J. Genet. Genomics, 2008).
Concernant Plk2, plusieurs travaux impliquent p53 dans la régulation transcriptionnelle de Plk2. En effet, un élément de réponse à ce suppresseur de tumeur a été retrouvé sur le promoteur de Plk2 (Shimizu-Yoshida Y., Biochem. Biophys. Res. Commun., 2001) (Voir § V, C, 2, a). Ces données indiquent que Plk2 peut exercer des fonctions différentes des autres Plks. Par ailleurs, des analyses en Northern blot montrent que la surexpression du récepteur ERRγ (Estrogen Related-Receptor gamma) entraîne l’induction de Plk2, suggérant que ce récepteur nucléaire puisse être un élément transactivateur du promoteur de Plk2. De plus, des expériences de précipitation de la chromatine ont démontré une interaction directe entre ERRγ et le promoteur de Plk2 (Park YY., et al., Biochem. Biophys. Res. Commun., 2007).
B : Régulation de l’activité kinase :
L’activité protéine kinase des Plks est contrôlée par deux mécanismes : - Des phosphorylations, notamment sur son T-loop, ou boucle d’activation.
- La liaison d’un substrat phosphorylé sur la partie C-terminale des Plks, ou PBD, présentée précédemment dans le paragraphe II, B 2 et 3)
Phosphorylations :
L’activation des différentes Plks, nécessite une phosphorylation directe. Deux résidus conservés entre différentes espèces, la thréonine 210, et la sérine 137, (numérotation en acides aminés de la forme humaine de Plk1), ont été démontrés comme essentiels dans ce processus. En effet, la mutation de ces deux résidus en acide aspartique, mimant la charge positive d’un phosphate, et donc la phosphorylation de ces résidus, favorise l’activation de la kinase. A l’inverse, une mutation de la thréonine 210 en acide aminé non phosphorylable, comme la valine, empêche l’activation de Plk1 (Lee KS. and Erikson RL., Mol. Cell Biol., 1997) (Quian YW., et al., Mol. Cell Biol., 1999) (Jang YJ., et al., J. Biol. Chem., 2002).
Les régulateurs de Plk1, chez les mammifères, sont relativement peu connus par rapport à la longue liste des fonctions de cette kinase. Dans d’autres espèces, quelques kinases ont été identifiées et on peut citer par exemple, Cdk1, MAPK et PKA (van de Weerdt B. and Medema RH., Cell Cycle, 2006).
Chez le xénope, une kinase appelée Polo-like kinase kinase-1 et la PKA ont été démontrées comme des kinases activatrices de Plx1 (Kelm O., et al., J. Biol. Chem., 2002).
Dans les cellules humaines, un rôle potentiel mais controversé de Cdk1 a été évoqué (van de Weerdt B. and Medema RH., Cell Cycle, 2006). Plus récemment, le rôle de la kinase Aurora A dans l’activation de Plk1 a été plus clairement démontré (Seki A., et al., Science, 2008).
a : Phosphorylations de la boucle d’activation ou T-loop :
La Thr210 se retrouve, dans un domaine particulier appelé le T-loop ou boucle d’activation. Des phosphorylations sur ce domaine, sont un mécanisme répandu d’activation de différentes protéines kinases : les Plks elles-mêmes (autophosphorylation), Aurora A/B, PKA, Cdk1/2, Erk1/2, Mek1, Akt/P (Johnson LN., et al., Cell, 1996).
La mutation de la thréonine 236 de Plk2 en acide glutamique, mimant sa phosphorylation, permet aussi l’activation de la kinase (Ma S., et al., Mol. Cancer Res., 2003), suggérant
alors que le mécanisme d’activation de Plk1 par phosphorylation du résidu correspondant de son T-loop est un mécanisme d’activation conservé dans la famille des Plks.
Deux études indépendantes ont identifié Aurora A comme kinase responsable de la phosphorylation de Plk1 sur sa Thr210 (Macurek L., et al., Nature, 2008) (Seki A., et al., Science, 2008) et BORA, un adaptateur d'Aurora A, est nécessaire à l' activation de Plk1. A l’inverse, Plk1 induit la dégradation protéasome-dépendante de BORA (Chan EH., et al., Chromosoma, 2008) suggérant un mécanisme de rétro-contôle de l'activité de Plk1.
Par ailleurs, une absence de BORA ou de Aurora A, n’empêche pas l’activation de Plk1, mais la retarde (Seki A., et al., Science, 2008), suggérant que d’autres kinases responsables de la phosphorylation de la thréonine 210 de Plk1 restent à découvrir.
En revanche, les autres Plks, Plk2-4, étant majoritairement actives en phase G1 ou S du cycle cellulaire, phase durant lesquelles, le complexe Aurora A/BORA est inactif, les kinases responsables de la phosphorylation de leur boucle d’activation sont encore inconnues (Archambault V. and Glover DM., Nat. Rev. Mol. Cell Biol., 2009).
b : Déphosphorylation du T-loop :
Les Plks étant activées par phosphorylation de la Thréonine 210 de leur boucle d’activation, la déphosphorylation de ce résidu devrait, en principe, inhiber l’activité des Plks. La protéine phosphatase 1, ou PP1, et son adaptateur MYPT1, ont récemment été démontrés comme des antagonistes de Plk1. La déplétion des cellules en MYPT1 résulte en une augmentation de la phosphorylation de la Thréonine 210 de Plk1 et PP1-MYPT1 sont capables de déphosphoryler Plk1 in vitro (Yamashiro S., et al., Dev. Cell, 2008).
MYPT1 n’étant pas conservé chez les invertébrés, de futures études devraient permettre l'identification de nouvelles phosphatases de la boucle d’activation des Plks.
C: Dégradation par le protéasome:
La dégradation des protéines impliquées dans le contrôle du cycle cellulaire est un mécanisme général utilisé par la cellule pour leur inactivation. En sortie de mitose, la cycline B est par exemple dégradée par le système ubiquitine protéasome (Peters JM., Nat. Rev. Mol. Cell Biol., 2006).
Les 4 Plks de mammifères sont dégradées par le protéasome (Archambault V. and Glover DM., Nat. Rev. Mol. Cell Biol., 2009). Les détails de ces dégradations seront présentés plus loin, dans la partie VII de ce chapitre.
IV : Principales fonctions des Plks au cours du cycle cellulaire :
Dans ce paragraphe seront décrites les fonctions principales des kinases Plk1, Plk3 et Plk4. Les fonctions relatives à Plk2 seront détaillées dans le paragraphe V, exclusivement consacré à cette protéine.
A : Principales fonctions de Plk1 :
Exprimée dans les tissus présentant une forte prolifération (foie, placenta, ovaires, rate, testicules) (Alberts GF. and Winkles JA., Cell Cycle, 2004 ), Plk1, présente de nombreuses fonctions, relativement bien conservées chez tous ses orthologues, en relation avec la progression au cours du cycle cellulaire (Archambault V. and Glover DM., Nat. Rev. Mol. Cell Biol., 2009).
Plk1 est également dramatiquement surexprimée dans de nombreuses tumeurs humaines (Takai N., et al., Oncogene 2005). Des petites molécules, inhibitrices de Plk1, sont en cours de développement et promettent des avancées intéressantes en thérapie anti-cancéreuse (Strebhardt K., et al., Nature Rev. Cancer, 2006).(voir partie 6)
1 : Fonctions Mitotiques :
Les fonctions mitotiques dans lesquelles est impliquée Plk1 sont brièvement résumées ci-dessous :
a : Entrée en mitose :
Plk1 participe à l’activation du MPF (Mitosis Promoting Factor) en phosphorylant et activant la phosphatase activatrice du MPF, CDC25C. De plus, elle phosphoryle et entraine la dégradation de Wee1 et Myt1, les kinases inhibitrices du MPF (Kumagai A., et al., Science., 1996).
b : Mise en place du fuseau mitotique et maturation des centrioles :
La présence d'un fuseau mitotique monopolaire est l’une des caractéristiques phénotypiques retrouvées chez le premier mutant de polo de Dosophila Melanogaster (Sunkel CE. and Glover DM., J. Cell Sci., 1988). Plk1 est responsable de la maturation du centrosome en G2/M (Lowery DM., et al., Oncogene, 2005). En effet, Plk1 se localise au niveau du centrosome grâce à son interaction avec une protéine centrosomale, la Cenexin (Soung NK., et al., Mol. Cell Biol., 2006).
c : Régulation de la cohésion des chromatides sœurs et de la fonction des kinétochores :
Chez les mammifères comme chez la levure, Plk1 présente une localisation centromérique, au niveau des kinétochores, grâce à son interaction avec différentes protéines (PB1P1, INCENP, BUBR1, BUB1…) (Qi W., et al., Mol. Cell Biol., 2006).
Plk1 phosphoryle SCC1 (mitotic Sister Chromatid Cohesin 1), ce qui a pour effet de promouvoir le clivage de la cohésine par la séparase, clivage nécessaire à la séparation des chromatides sœurs lors de l’anaphase (Alexandru G., et al., Cell, 2001).
Cependant, les fonctions des Plks au niveau des kinétochores diffèrent en importance et en nature selon les espèces. Cette faible conservation de fonction suggère que les fonctions de régulation des kinétochores ne sont pas la raison d'être de ces protéines kinases.
d : Sortie de mitose :
La sortie de mitose nécessite l'inactivation du complexe CDK1-cycline B, inactivation qui se réalise grâce à la dégradation protéasome-dépendante de la cycline mitotique par l'anaphase promoting complex (APC/C), son E3 ubiquitine ligase. En mitose, Plk1 active l’APC/C. En effet, elle phosphoryle Emi1, inhibiteur de l'APC/C. Cette phosphorylation est reconnue comme un signal de dégradation, et entraîne l'ubiquitinylation de cet inhibiteur et sa dégradation par le protéasome (Moshe Y., et al., PNAS, 2004) (Hansen DV., et al., Mol. Biol. Cell, 2004). Chez le xénope, Plx1 régule de la même façon Emi2. De plus, la phosphorylation préliminaire de ce dernier par CamKII favorise l'interaction entre Plx1 et Emi2 (Rauh NR., et al., Nature, 2005). Chez les levures, les Plks activent également les processus de sortie de mitose : MEN (Mitose Exit Network) chez S. cerevisae et SIN (Septation Initiation Network) chez S. Pombe (Simanis V., et al., J. Cell Sci., 2003).
e : Cytokinèse :
L’implication des Plks dans le processus de cytokinèse a été montrée pour la première fois chez la levure à fission, S. Pombe. En effet, la surexpression de Plo1 à n’importe quelle phase du cycle cellulaire entraîne le phénomène de septation et des mutants de plo1 ne forment pas de septum de division (Ohkura H., et al., Genes Dev., 1995). Malgré leurs différences au niveau architectural, les voies de signalisation moléculaires régulant la cytokinèse sont partiellement conservées entres les levures, les mouches, et les vertébrés. Dans tous ces organismes, les Plks sont essentielles à ce processus. La stimulation de la cytokinèse est probablement la fonction cellulaire la plus
universellement conservée des Plks. Plk1 est par exemple impliquée dans l’activation d’une Rho GTPase stimulant la contraction de l’anneau d’actine et de myosine (Petronczki M., et al., Dev. Cell, 2007).
2 : Activation des points de contrôle du cycle cellulaire ou checkpoints :
a : Point de contrôle mitotique :
La fonction de Plk1 est liée au point de surveillance mitotique du cycle cellulaire. Le but de ce contrôle est de retarder l'entrée en anaphase, c’est à dire la séparation des chromatides sœurs, jusqu'à ce que tous les kinétochores présentent un attachement bipolaire au fuseau de microtubules. Plk1 crée un phosphoépitope sur les kinétochores qui présentent un ancrage défectueux des microtubules mitotiques (Ahonen LJ., et al., Curr. Biol., 2005). Plk1 pourrait également participer à la séquestration cytoplasmique de la phosphatase activatrice du MPF, CDC25C, en la phosphorylant sur sa sérine 198 (Qian YW., et al., Mol. Biol. Cell, 2001).
b : Stress génotoxiques et point de contrôle de la phase S :
Plk1 est inactivée chez l’homme en réponse à des dommages sur l’ADN et de façon Chk1 dépendante (Smits VA., et al., Nat. Cell Biol., 2000) (van Vugt MA., et al., J. Biol. Chem., 2001). Son inactivation se retrouve au niveau transcriptionnel suite à un traitement des cellules avec des rayonnements ionisants (Ree AH., et al., Oncogene, 2003). Plk1 pourrait aussi jouer un rôle positif dans ce checkpoint. En effet, elle serait capable de phosphoryler la thréonine 68 de Chk2, acide aminé également modifié par ATR et qui entraîne une activation de Chk2.
c : Plk1 et p53 :
Dans les cellules de mammifères en culture, Plk1 interagit physiquement avec le domaine de liaison à l’ADN de p53, inhibant par cette interaction la transcription des cibles de ce suppresseur de tumeur (p21, BAX, MDM2) et donc sa capacité à bloquer le cycle cellulaire ou à induire l’apoptose (Ando K., et al., J. Biol. Chem., 2004).
B : Principales fonctions de Plk3 :
Plk3 est avant tout une kinase activée en réponse à différents stress cellulaires. Ainsi, des cellules embryonnaires de souris invalidées pour le gène de plk3, les MEF Plk3-/-, présentent une sensibilité accrue à l’hypoxie (Yang Y., et al., Cancer Res., 2008).
1 : Fonctions mitotiques :
a : Entrée en mitose :
Plk3 phosphoryle la sérine 191 de CDC25C et participe ainsi à son activation en favorisant sa translocation nucléaire (Bahassi el M., et al., Oncongene, 2004).
b : Régulation de l’entrée en phase S :
Après ajout de sérum, les cellules invalidées pour le gène plk3 et bloquées en Go, sont incapables d’entrer en phase S et s’arrêtent en G1. Plk3 serait indispensable à la transition G1-S. En effet, Plk3 est nécessaire à l’expression de la cycline E, fonctionnelle en phase S (Zimmerman WC., et al., PNAS, 2007).
c : Fragmentation de l’appareil de Golgi :
L’expression ectopique de Plk3 induit la fragmentation de l’appareil de Golgi. Plk3 serait une cible de MEK1 dans la cascade de signalisation impliquée dans ce phénomène (Ruan Q., et al., Exp. Cell Res., 2004) (Xie S., et al., Oncogene, 2004).
3 : Réparation de l’ADN :
Plk3 participerait directement aux mécanismes de réparation de l’ADN en régulant l’activité de la DNA polymérase δ. En effet, la plus grosse des sous-unités de cette polymérase, interagit directement avec Plk3. Des essais kinases in vitro montrent que Plk3 est capable d'en phosphoryler la sérine 60. Cependant la fonction physiologique de cette interaction ainsi que de cette phosphorylation restent encore non élucidées (Xie S., et al., Oncogene, 2005).
4 : Activation des checkpoints :
Plk3 est activée grâce au facteur ATM, en réponse à des dommages sur l’ADN ou des stress du fuseau mitotique. Dans ces conditions, cette kinase phosphoryle la sérine 20 de p53, le stabilisant et favorisant ainsi son activité transcriptionnelle (Xie S., et al., J. Biol. Chem., 2001a). Des ARNi dirigés contre Plk3 abolissent la phosphorylation de p53 sur ce site en réponse à un stress oxydatif (Wang L., et al., J. Biol. Chem., 2008). De plus, une surexpression de Plk3 par transfection dans des lignées cellulaires humaines, provoque leur entrée en apoptose (Conn CW., et al., Cancer Res., 2000).
Plk3 jouerait un rôle dans l’arrêt du cycle cellulaire et dans l’induction de l’apoptose par différents mécanismes dont probablement l’induction des voies de signalisation dépendantes de p53 (Xie et al., J. Biol. Chem., 2001b) (Bahassi el M., et al., Oncogene, 2002). En effet, Plk3 phosphoryle CDC25C sur sa sérine 216. Ce résidu, est également phosphorylé par les kinases des points de contrôle du cycle cellulaire, Chk1 et Chk2, en réponse à un stress génotoxique. La phosphorylation de cette sérine 216 crée un site de liaison pour la protéine 14-3-3 et entraîne ainsi la séquestration inactivatrice de CDC25C dans le cytoplasme. Par conséquent, le complexe CDK1-Cycline B ne peut être activé, bloquant les cellules à la transition G2/M du cycle cellulaire (Ouyang B., et al., Oncogene, 1999) (Ouyang B., et al., J. Biol. Chem., 1996 et 1997).
C : Principales fonctions de Plk4 :
Plk4 est une kinase de la famille des Plks, structurellement originale. En effet, contrairement aux trois autres membres de cette famille, Plk4 ne possède qu’un seul domaine polo (Lowery DM., et al., Oncogene, 2005).
L’invalidation du gène codant pour Plk4 chez la souris provoque une létalité embryonnaire. Les embryons Plk4-/- présentent une taille inférieure aux animaux sauvages du même âge avec une proportion de cellules en mitose et en apoptose augmentée, suggérant un arrêt du cycle cellulaire par activation d’un des points de contrôle du cycle cellulaire et induction des voies de signalisation apoptotiques (Hudson JW., et al., Curr Biol., 2001).Les souris hétérozygotes présentent quant à elles une induction des cancers hépatiques et pulmonaires (Ko MA., et al., Nature Genetics, 2005).
1 : Fonctions mitotiques :
a : Duplication des centrioles :
Deux études démontrent l’implication de Plk4 dans la duplication des centrioles dans des cellules humaines et chez D.Melanogaster. En effet, la quantité cellulaire de Plk4 semble directement corrélée au nombre de centrioles par cellule. Une inactivation de Plk4 entraîne une réduction du nombre de centrioles et à l’inverse, ils sont en nombre anormalement élevé lors d’une surexpression de cette protéine kinase (Bettencourt-Dias M., et al., Curr. Biol., 2005) (Habedanck R., et al., Nat. Cell. Biol., 2005).
b : Sortie de mitose :
L’invalidation génétique de Plk4 provoque une accumulation des cellules dans des phases tardives de la mitose. Ces cellules possédant par ailleurs des taux importants de cycline mitotique B1. Le complexe APC/C, responsable de la dégradation de cette cycline, et favorisant donc la fin du processus mitotique, se localise au même endroit que Plk4. Celle-ci pourrait alors être impliquée dans la dégradation par le protéasome de Plk4 par exemple en phosphorylant la cycline B1 et en induisant ainsi sa reconnaissance par son E3 ubiquitine ligase (Hudson JW., et al., Curr Biol., 2001).
c : Plk4 et p53 :
L’expression de Plk4 est régulée par p53. En effet, l’induction de ce suppresseur de tumeur entraîne une diminution de l’expression de Plk4, médiée par le recrutement du répresseur de transcription, HDAC (histone Déacétylase). Par ailleurs, la répression de son expression contribuerait à l’induction de l'apoptose médiée par p53 (Li J., et al., Neoplasia, 2005).
V : Plk2 dans tous les détails…
Plk2, également appelée Snk, pour Serum inducible kinase, est à ce jour la plus mystérieuse, originale, et surtout la moins étudiée des membres de la famille des Plks. Elle semble, contrairement aux autres Plks, ne pas présenter de rôle majeur concernant la progression du cycle cellulaire. Cependant, elle présente des fonctions importantes dans les neurones post-mitotiques et dans la régulation de la plasticité synaptique.
A : Expression dans des cellules en culture :
Plk2 a été initialement identifiée au début des années 1990 comme un gène de réponse précoce au sérum ou à un traitement aux esters de phorbol (PMA) dans les fibroblastes de souris, les cellules NIH3T3 (Simmons DL., et al., Mol. Cell Biol. 1992). Induite par une stimulation au sérum dans les cellules de mammifères son expression protéique est transitoire. Elle présente une demi-vie très courte de 15 minutes (Ma S., et al., Mol. Cancer Res., 2003). Plk2 a alors été décrite comme un régulateur de la phase G1 du cycle cellulaire et son activité catalytique est effective durant cette phase (Ma S., et al., Mol. Cancer Res., 2003).
Cependant, Plk2 n’est pas seulement un élément de réponse à une stimulation par le sérum mais est également impliquée dans une réponse au stress. En effet, Plk2 possède
un élément de réponse à p53 dans son promoteur et est induite notamment lors de stress génotoxiques (Shimizu-Yoshida Y., Biochem. Biophys. Res. Commun., 2001) (Burns TF., et al., Mol. Cell Biol, 2003) (Voir § V, C, 2)
B : Expression tissulaire :
1 : Tissus normaux :
Plk2 est exprimée de façon tissu-spécifique. Son ARNm a été retrouvé dans le cerveau, le cœur, le poumon, les testicules, la glande mammaire, l’utérus et la rate. Contrairement à Plk1, elle serait absente des tissus à forte prolifération comme le thymus, le foie, le rein ou l’intestin (Simmons DL., et al., Mol. Cell Biol. 1992).
Par ailleurs, dans le cerveau de rongeurs l’expression des transcrits de Plk2 est augmentée suite à une stimulation de l’activité synaptique (Kauselmann G., et al., EMBO J., 1999).
2 : Tissus tumoraux :
L’expression de Plk2 dans les tissus tumoraux est peu répertoriée.
Un travail de Syed N et al., révèle que l’expression de cette kinase est fréquemment perdue dans les lignées cellulaires ainsi que des tumeurs primaires de lymphomes B. Cette perte d’expression est liée à une hyperméthylation du promoteur de Plk2, première description d’un événement d’inactivation épigénétique relatif à une Plk dans un cancer. Aucune mutation de la séquence codante de Plk2 n’a été retrouvée dans ces cellules tumorales (Syed N., et al., Blood, 2006).
C : Fonctions de Plk2 relatives au cycle cellulaire :
Contrairement aux rôles bien définis de Plk1 au cours de la division cellulaire, ceux de Plk2 restent mystérieux.
L’expression de Plk2 n’est pas détectable dans des cellules traitées au nocodazole, donc arrêtées en prométaphase de la mitose, suggérant une absence de fonction de Plk2 en mitose (Ma S., et al., Mol Cancer Res., 2003).
Des souris invalidées pour le gène Plk2, plus petites à la naissance, présentent une croissance post-natale comparable à celle de souris sauvages, suggérant un rôle cellulaire physiologique de Plk2 restreint à la période embryonnaire de croissance cellulaire. De plus, les MEF Plk2-/-, présentent un retard à l’entrée en phase S du cycle cellulaire (Ma S., et
al., Mol. Cell. Biol., 2003). Ces observations, d’un phénotype peu modifié, peuvent être en accord avec une possible compensation fonctionnelle réalisée par les autres Plks.
1 : Duplication des centrioles :
La surexpression de Plk2 par transfection, ainsi que l’invalidation de la protéine kinase par l’utilisation de la technologie de l’ARN interférant, influent sur le nombre de centrioles, suggérant un effet de Plk2 dans le mécanisme de duplication des centrioles dans des cellules de mammifères. L’utilisation d’un mutant catalytiquement inactif de Plk2 a permis de démontrer que son activité kinase est indispensable à ce processus (Warnke S., et al., Curr. Biol., 2004).
Par ailleurs, le domaine PBD de Plk2 est nécessaire à la localisation centrosomique de la protéine et des mutations de sites critiques des boîtes polo entrainent une perte de la localisation centrosomique de la kinase ainsi qu’une absence de duplication centriolaire (Cizmecioglu O., et al., Cell Cycle, 2008).
2 : Activation des checkpoints :
a : Plk2 et p53 :
L’ARNm de Plk2 augmente rapidement dans des cellules humaines thyroïdiennes soumises à une irradiation aux rayons X. Un élément de réponse aux radiations, homologue d’un élément de liaison à p53, a été identifié sur le promoteur de Plk2 (Shimizu-Yoshida Y., et al., Biochem. Biophys. Res. Commun., 2001).
Dans une deuxième étude, Burns TF. et al. ont montré que l’induction de l’ARNm de Plk2 dans des cellules humaines exposées à des rayonnements UV ou à des stress génotoxiques est dépendante de l’expression cellulaire de la protéine p53. En effet, l'induction de Plk2 n’est pas retrouvée dans des cellules provenant de souris irradiées mais invalidées pour le gène p53. Une interaction de p53 sur le promoteur de Plk2 a également été démontrée par des expériences d’immunoprécipitation de la chromatine (Burns TF., et al., Mol. Cell Biol, 2003).
b : Checkpoint de la phase S :
Les stress génotoxiques, ou les dommages à l’ADN, entraînent l’activation du point de contrôle de la phase S du cycle cellulaire. Les protéines impliquées dans la transmission du signal relatif à l’activation de ce contrôle sont entre autre, ATM, ATR, Chk1 et 2, Plk3
(Xie S., et al., J. Biol. Chem., 2001). L’effecteur principal touché suite à cette activation est le gardien du génome, p53.
Des cellules H460, de carcinomes pulmonaires à grandes cellules, sauvages pour le gène p53, ont été invalidées pour le gène plk2 de façon stable. En réponse à un stress réplicatif, ces cellules présentent des dommages à l’ADN et une proportion de cellules apoptotiques plus importante. Il semblerait donc que des cellules Plk2-/- ne soient pas capables de mettre en place une réponse cellulaire normale face à ce type de stress.
Par ailleurs, Plk2 interagit avec des protéines de ce point de contrôle (Chk1, Chk2, et p53). Il semblerait donc que Plk2 soit indispensable aux voies d’activation du point de contrôle de la phase S, dépendantes de p53 (Matthew EM., et al., Cell Cycle, 2007).
c : Checkpoint mitotique :
Plk2 est induite lors de l’activation du point de contrôle du fuseau mitotique du cycle cellulaire. Cette activation est dépendante de p53 et permettrait aux cellules de prévenir les catastrophes mitotiques, qui se produisent dans les cellules dont le gène de Plk2 est invalidé, et lorsqu'elles sont traitées par des poisons du fuseau mitotique, type Nocodazole (Burns TF., et al., Mol. Cell Biol., 2003).
D : Localisation cellulaire :
Des cellules Cos-7 ont été transfectées avec des plasmides permettant l’expression de Plk2 recombinante, en fusion avec la GFP (Green Fluorescent Protein). Des analyses au microscope confocal ont démontré une expression cytoplasmique de cette protéine et plus précisément une localisation périnucléaire. Une analyse plus précise a pu mettre en évidence que la région occupée par Plk2 correspondait au centrosome, localisation en accord avec la fonction de cette kinase dans la duplication des centrioles lors de la phase G1/S du cycle cellulaire (Ma S., et al., Mol. Cell Biol., 2003).
Une seconde étude a également validé cette localisation cellulaire dans des cellules humaines d’ostéosarcomes (U20S), et dans les cellules de hamster (CHO), transfectées avec un plasmide permettant l’expression de Plk2 en fusion avec l’antigène HA (Hémagglutinine) (Warnke S., et al., Current Biol., 2004).
Des stimulations électriques suffisantes pour induire une plasticité synaptique à long terme dans l’hippocampe, structure anatomique impliquée dans les phénomènes de mémorisation, induisent la transcription de Plk2. Ainsi induite, Plk2 se localise principalement au niveau du corps cellulaire et des dendrites des neurones ainsi activés,