© Eliel Brochu, 2018
Métagénomique, culturomique et sélectomique
recombinante pour la caractérisation de gènes de
résistance aux antibiotiques dans le microbiote
intestinal humain
Mémoire
Eliel Brochu
Maîtrise en microbiologie-immunologie - avec mémoire
Maître ès sciences (M. Sc.)
76
Métagénomique, culturomique et sélectomique
recombinante pour la caractérisation de gènes de
résistance aux antibiotiques dans le microbiote
intestinal humain
Mémoire
ELIEL BROCHU
Sous la direction de :
Michel G. Bergeron, directeur de recherche
Ann Huletsky, codirectrice de recherche
iii
Résumé
Le microbiote intestinal humain est un important réservoir de gènes de résistance aux antibiotiques qui demeure toutefois méconnu. Dans cette étude, nous avons exploré les gènes de résistance du microbiote intestinal de participants sains avant et après une exposition à l’antibiotique cefprozil. Trois approches ont été utilisées pour caractériser ces gènes et examiner leur altération par les antimicrobiens : la métagénomique, la culturomique et la sélectomique recombinante. Le séquençage métagénomique et la culturomique ont permis d’identifier plusieurs gènes de résistance aux β-lactamines et à d’autres antibiotiques. Cependant, la culturomique a permis d’identifier ces gènes chez un plus grand nombre de participants. La culturomique a mis en évidence la présence de gènes de résistance à la vancomycine de type vanD dans les microbiotes de 46% des participants comparativement à 8% avec le séquençage métagénomique. La culturomique a aussi montré que l’exposition in
vitro et in vivo à une β-lactamine stimule l’émergence des gènes vanD. La sélectomique
recombinante, qui repose sur la construction de banques d’expression à partir de l’ADN des bactéries, a été utilisée pour caractériser de manière fonctionnelle les gènes de résistance aux β-lactamines chez les bactéries cultivables de l’intestin. Elle a permis d’identifier et caractériser cinq β-lactamases différentes dont deux montrant une activité à spectre étendu. La majorité des gènes de β-lactamases étaient associés à d’autres gènes de résistance et/ou des éléments mobiles. Ces travaux ont montré que la culture favorise l’identification de gènes non détectés par le séquençage métagénomique et que la sélectomique recombinante est un outil puissant pour caractériser les fonctions des gènes. Cette étude a aussi révélé que la prise d’une β-lactamine communément utilisée peut influencer l’abondance des bactéries qui contiennent des gènes de résistance à un antibiotique d’une autre classe, comme la vancomycine, un antibiotique de dernier recours dans l’arsenal des antimicrobiens.
iv
Abstract
The human intestinal microbiota is an important and poorly known antibiotic resistance genes reservoir. In this study, we explored resistance genes from the microbiota of healthy volunteers before and after exposure to the β-lactam cefprozil antibiotic. Three approaches were used to characterise resistance genes in the human microbiota and examine alteration by antimicrobials: metagenomics, culturomics and recombinant selectomics. Metagenomic and culturomic sequencing of intestinal microbiota enabled identification of several genes for resistance to β-lactams and other antibiotics. However, culturomics allowed identification of these genes in more participants than metagenomics. Culturomics highlighted the presence of the clinically important vancomycin resistance vanD-like genes in the microbiota of about 46% of participants compared to 8% with metagenomics. Culturomics also showed that in vitro and in vivo β-lactams exposition stimulates the emergence of vanD genes. Recombinant selectomics, which is based on the construction of expression libraries made with bacterial DNA, was also used to functionally characterise β-lactam resistance genes from the cultivable intestinal bacteria. It allowed identification and characterisation of five different β-lactamases including two with an extended-spectrum activity. The majority of β-lactamases genes was associated with other resistance genes and/or mobile elements. This study demonstrated that culture favors the identification of genes undetected by direct metagenomic sequencing and selectomics was a powerful tool to characterise gene functions. It also demonstrated that intake of a commonly used antibiotic of the β-lactam family can influence the abundance of bacteria containing resistance genes to an antibiotic from another class, such as vancomycin, which is a last resort antibiotic.
v
Table des matières
Résumé ... iii
Abstract ... iv
Table des matières ... v
Liste des tableaux ... ix
Liste des figures ... x
Liste des abréviations ... xii
Remerciements ... xvi
Avant-Propos ... xviii
Chapitre 1 Introduction générale ... 1
1.1. Historique ... 2
1.1.1. Les maladies infectieuses à travers l’histoire ... 2
1.1.2. Découverte des antibiotiques ... 3
1.1.3. Impact des antibiotiques ... 3
1.2. Antibiotiques ... 4
1.2.1. Mode d’action ... 4
1.2.2. β-lactamines ... 5
1.2.2.1. Mode d’action des β-lactamines ... 5
1.2.3. Vancomycine ... 6
1.2.3.1. Mode d’action de la vancomycine ... 6
1.3. Résistance des bactéries aux antibiotiques ... 8
1.3.1. Problématique... 8
1.3.2. Impact de la résistance ... 9
1.3.3. Résistance naturelle ... 10
1.3.4. Résistance acquise génétiquement ... 10
1.3.4.1. Mutation ... 10
1.3.4.2. Acquisition de gènes ... 10
1.3.4.2.1. Mécanismes de transfert de gènes ... 11
1.3.4.2.1.1. Conjugaison ... 11
1.3.4.2.1.2. Transformation naturelle ... 11
1.3.4.2.1.3. Transduction ... 11
1.3.4.2.2. Plasmides ... 12
vi
1.3.4.2.4. Intégrons ... 12
1.3.5. Mécanismes de résistance aux antibiotiques ... 13
1.3.5.1. Trois différents mécanismes ... 13
1.3.5.1.1. Altération de la perméabilité membranaire ... 13
1.3.5.1.2. Altération des cibles ... 13
1.3.5.1.3. Inactivation enzymatique ... 14
1.3.5.2. Mécanismes de résistance aux β-lactamines ... 14
1.3.5.2.1. β-lactamases ... 15
1.3.5.3. Mécanismes de résistance à la vancomycine ... 16
1.3.5.3.1. Gène de résistance à la vancomycine de type VanD ... 16
1.4. Ère post-antibiotique? ... 19
1.4.1. Pourquoi si peu de nouveaux antibiotiques? ... 19
1.4.2. Y a-t-il des alternatives aux nouveaux antibiotiques? ... 20
1.4.3. Que peut-on faire pour ralentir l’émergence de résistance? ... 20
1.5. Microbiote ... 22
1.5.1. Microbiote intestinal humain ... 22
1.6. Approches d’analyse des gènes de résistance aux antibiotiques dans le microbiote intestinal humain ... 24
1.6.1. Métagénomique ... 25
1.6.2. Culturomique ... 26
1.6.3. Sélectomique recombinante ... 28
Chapitre 2 Problématique et objectifs de recherche ... 30
2.1. Problématique ... 31
2.2. Objectifs de recherche ... 31
2.2.1. Objectif général ... 31
2.2.2. Objectifs spécifiques ... 32
Chapitre 3 Characterisation of β-lactam resistance genes from human anaerobic cultivable intestinal microbiota by recombinant selectomics... 33
3.1. Avant-propos ... 34
3.2. Résumé ... 36
3.3. Abstract ... 37
3.4. Introduction ... 38
3.5. Material and methods ... 39
Ethics approval ... 39
vii
Recombinant selectomics ... 40
Sample culture for recombinant selectomics ... 40
DNA extraction and purification for recombinant selectomics ... 40
Construction of recombinant plasmids libraries ... 40
Screening of recombinant plasmids libraries... 41
Amplification of resistant clones inserts ... 42
Sequencing ... 42
Sequence homology search in databases ... 42
Construction of E. coli host strain CCRI-22768 ... 42
Verification of the new E. coli host strain CCRI-22768 ... 43
Extraction of plasmids from selected resistant clones ... 44
Antimicrobial susceptibility testing ... 44
Metagenomic and Culturomic Sequencing ... 46
DNA extraction for metagenomics ... 46
Sample culture for culturomics ... 46
DNA extraction for culturomics ... 47
Sequencing ... 47
Reads assembly ... 47
Comparison with metagenomic and culturomic approaches ... 47
3.6. Results ... 47
Verification of the new E. coli host strain CCRI-22768 ... 47
Recombinant selectomics ... 48
Resistance phenotype ... 48
Comparison with metagenomic and culturomic approaches ... 49
3.7. Discussion ... 50
3.8. Conclusions ... 54
3.9. Acknowledgments ... 55
3.10. Conflicts of interest ... 55
3.11. References ... 55
Chapitre 4 Characterisation of vancomycin-resistance vanD-like genes from human intestinal microbiota by metagenomics and culturomics ... 71
4.1. Avant-propos ... 72
4.2. Résumé ... 74
4.3. Abstract ... 75
viii
4.5. Material and methods ... 77
Ethics approval ... 77
Participants’s recruitment and samples collection ... 77
DNA extraction for metagenomics ... 78
Sample culture for culturomics ... 78
DNA extraction for culturomics ... 78
Quality verification of the purified DNA ... 79
Sequencing ... 79
Reads assembly ... 79
Search of vanD-like genes in sequencing data ... 79
Comparison with reference sequences ... 80
Comparison of metagenomic and culturomic approaches and the different culture conditions at different sampling times ... 80
Characterisation of genetic contexts of vanD-like genes ... 81
4.6. Results ... 81
Comparison of metagenomic and culturomic approaches and the different culture conditions at different sampling times ... 81
Phylogenetic comparison of the VanD proteins ... 81
Analysis of the different vanD loci ... 82
4.7. Discussion ... 84 4.8. Conclusions ... 90 4.9. Acknowledgments ... 91 4.10. Conflicts of interest ... 91 4.11. References ... 91 Chapitre 5 Conclusion ... 103
Combinaison de la métagénomique, de la culturomique et de la sélectomique recombinante pour une meilleure vue d’ensemble du résistome intestinal humain .... 104
Avenir des antibiotiques et prédiction de la résistance ... 106
ix
Liste des tableaux
Table 3.1. β-lactams (11) selected to perform functional screening. Table 3.2. Resistance phenotype of the E. coli CCRI-22768 host strain.
Table 3.3. Summary of antibiotic-resistance genes and potential mobile elements recovered
from functional screening of genomic libraries derived from non-selective anaerobic cultures of fecal samples from participants P1 at D0 and D7.
Table 3.4. Summary of antibiotic-resistance genes and potential mobile elements recovered
from functional screening of genomic libraries derived from non-selective anaerobic cultures of fecal samples from participants P4 at D0 and D7.
Table 3.5. Resistance phenotypes of the clones from P1D0. Table 3.6. Resistance phenotypes of the clones from P1D7. Table 3.7. Resistance phenotypes of the clones from P4D0. Table 3.8. Resistance phenotypes of the clones from P4D7.
Table 3.9. Comparison of metagenomics, culturomics, and recombinant selectomics for
detection of β-lactamases.
Table 4.1. Partial or complete VanD proteins identified in the 24 participants studied
before the antibiotic intake (D0) and/or after 7 days of antibiotic exposure (D7) in metagenomic and culturomic sequencing data.
Table 4.2. Closest protein and amino acid identity percentage (%) for the VanD proteins
x
Liste des figures
Figure 1.1. Représentation schématique des cibles des antibiotiques ainsi que les
mécanismes utilisés par les bactéries pour leur résister.
Figure 1.2. Représentation schématique de la pénicilline (lactamine) et de son noyau
β-lactame.
Figure 1.3. Représentation schématique de la vancomycine et de ses sept acides aminés. Figure 1.4. Représentation schématique de l’inactivation enzymatique de la pénicilline
(β-lactamine) par une β-lactamase qui vient hydrolyser le noyau β-lactame de l’antibiotique.
Figure 1.5. Représentation schématique de la diminution de l’affinité envers la
vancomycine par l’incorporation du dipeptide D-Ala-D-Lac.
Figure 1.6. Représentation schématique des différents gènes constituant le locus vanD. Figure 1.7. Facteurs influençant le microbiote intestinal humain.
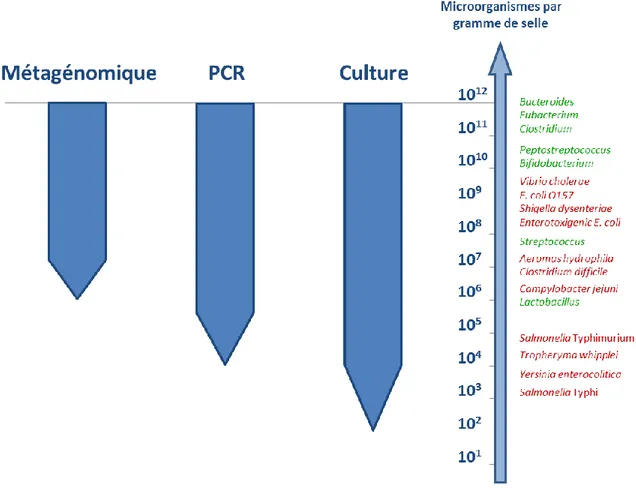
Figure 1.8. Trois approches utilisées pour caractériser le résistome intestinal humain. Figure 1.9. Profondeur de détection des microorganismes dans 1 gramme de selle par les
méthodes de métagénomique, de PCR et de culture.
Figure 3.1. Screening of recombinant plasmids libraries.
Figure 3.2. Electrophoresis on 1% agarose gel of amplified fragments from the E. coli
CCRI-22768 strain and three other E. coli control strains.
Figure 3.3. Scar sequence of the deletion of the kanamycin cassette performed by
FLP-FRT system in the E. coli JW4111 strain from the Keio collection for construction of the E.
coli CCRI-22768 strain.
Figure 4.1. Number of participants (among the 24 studied) for which a vanD-like gene was
identified in metagenomic and culturomic sequencing data independently of antibiotic exposure.
Figure 4.2. Number of participants (among the 18 exposed participants) having a partial
and/or complete vanD-like gene identified before the antibiotic intake (D0) and after 7 days of antibiotic exposure (D7) in culturomic sequencing data.
Figure 4.3. Phylogenetic neighbor-joining tree of the complete VanD proteins identified
with metagenomics (M) and culturomics (C) with the 7 known VanD subtypes of enterococci, other known VanD, and VanA and VanB as an outgroup.
xi
Figure 4.4. Schematic representation of the different genes constituting the vanD loci
(containing complete vanD genes) found in metagenomic and culturomic samples and identity with closest loci homologs.
Figure 5.1. Sélection de la résistance par un antibiotique et évitement de la résistance par la
sélection inverse.
Note : Certains éléments des figures 1.1, 1.8 et 3.1 proviennent du service Servier Medical
Art (http://www.servier.com/Powerpoint-image-bank). Ces éléments ont été modifiés et intégrés à un ensemble d’éléments formant les figures résultantes. Servier Medical Art par Servier fournit des milliers d’éléments médicaux qui peuvent être copiés individuellement ou utilisés en combinaison. Ces éléments sont disponibles pour être utilisés dans des présentations ou bien pour illustrer des publications scientifiques. Servier Medical Art est sous la license «Creative Commons Attribution 3.0 Unported (CC BY 3.0)». Cette licence permet de distribuer, remixer, arranger et adapter l’œuvre, même à des fins commerciales, tant que l’auteur de l’œuvre, dans ce cas-ci Servier Medical Art, soit cité (https://creativecommons.org/).
xii
Liste des abréviations
aa Acide aminé / amino acids ADN Acide désoxyribonucléique ARN Acide ribonucléique
AMP Ampicilline
ATCC American Type Culture Collection
BHI Brain heart infusion
BSA Bovine serum albumin
C Carbone
°C Degré Celsius CAZ Ceftazidime
CCRI Collection du Centre de Recherche en Infectiologie CLOX Cloxacilline
CLSI Clinical & Laboratory Standards Institute
CMI Concentration minimale inhibitrice CPR Cefprozil
CQDM Consortium Québécois sur la découverte du médicament CRO Ceftriaxone
CZ Céfazoline
D0 Day 0 (Before the antibiotic intake)
D7 Day 7 (After the 7 days of antibiotic intake)
D-Ala-D-Ala Dipeptide D-alanyl-D-alanine D-Ala-D-Lac Dipeptide D-alanyl-D-lactate D-Ala-D-Ser Dipeptide D-alanyl-D-sérine DMSO Diméthylsulfoxyde
dsDNA Double-stranded DNA
EDTA Ethylenediaminetetraacetic acid
EMR Entérobactérie multirésistante (EMR) ERG Entérocoque résistant aux glycopeptides ESBL Extended spectrum β-lactamase
EUCAST European Committee on Antimicrobial Susceptibility Testing
FEP Céfépime
FLP Flippase recombinase FOX Céfoxitine
FRT FLP recognition target
Gb Gigabase Gram- Gram négatif Gram+ Gram positif
xiii IS Insertion sequence KAN Kanamycine Kb Kilobase L Litre LB Luria broth
LSPQ Laboratoire de santé publique du Québec MBL Metallo β-lactamase
MEM Méropénem mg Milligramme
MgCl2 Chlorure de magnésium
MHT Modified Hodge test
MIC Minimal inhibitory concentration
Min Minute mL Millilitre
mM Millimolaire / Millimolar
N Azote
NaCl Chlorure de sodium / Sodium chloride
NCBI National Center for Biotechnology Information
ng Nanogramme nr Non-redundant
nt Nucléotide
OMS Organisation mondiale de la santé ORF Open reading frame
P Participant Pb Paire de bases
PBP Penicillin-binding protein
PBS Phosphate-Buffered Saline
PBSc Phosphate-Buffered Saline with cysteine
PCR Polymerase chain reaction
PIP Pipéracilline
PLP Protéine liant la pénicilline P/T Pipéracilline et Tazobactam RPM Rotation par minute
s Seconde
S1 Selection 1
S2 Selection 2
SARM Staphylococcus aureus résistant à la méticilline
SDD Susceptible-dose dependent
TC Tétracycline TIC Ticarcilline
xiv UFC Unité formatrice de colonies µg Microgramme
µL Microlitre µM Micromolaire
USD United States dollar (dollar américain)
xv
«Le plus grand ennemi de la connaissance n’est pas l'ignorance, mais l'illusion de la connaissance.»
Stephen Hawking
«The greatest enemy of knowledge is not ignorance,
it is the illusion of knowledge.»
xvi
Remerciements
Les études supérieures en recherche sont comparables à un voyage. Comme dans tout voyage, le chemin est parfois parsemé d’embûches que l’on doit affronter. Toutefois, ce sont ces difficultés ou ces énigmes intellectuelles qui nous amènent à se questionner et à développer de nouvelles compétences. C’est en avançant de plus en plus sur le chemin académique et en approfondissant toujours nos connaissances que l’on constate que nos connaissances sont bien limitées. Il y a tant à apprendre et tant de mystères encore non-résolus qu’il est impossible de tout connaître et de tout comprendre. Cependant, nous en sortons grandis et plus expérimentés et notre curiosité nous incite à persévérer et toujours vouloir en connaître davantage. Comme dans tout voyage, les gens qui nous accompagnent tout au long de notre parcours ainsi que ceux que l’on rencontre en chemin contribuent fortement au bon déroulement de notre expérience et à notre avancement.
Au cours de ma maîtrise, j’ai eu la chance d’être entouré d’une équipe de scientifiques d’exception, mais aussi d’humains extraordinaires. J’ai cheminé dans une équipe où les membres sont tous différents, mais se complètent parfaitement. C’est, je crois, ce qui fait la force de notre équipe. Je remercie toutes ces personnes qui ont contribué de près ou de loin à mon avancée en tant que scientifique.
Je tiens tout d’abord à remercier mon directeur de recherche, Dr Michel G. Bergeron, de m’avoir accueilli dans son équipe, premièrement comme stagiaire, puis comme étudiant à la maîtrise. Dr Bergeron est pour moi une source d’inspiration, non seulement pour ses accomplissements professionnels, mais aussi pour sa passion et son humanisme qui font de lui un rassembleur hors du commun.
Je tiens également à remercier ma codirectrice de recherche, Dre Ann Huletsky, qui tout au long de ma maîtrise m’a guidé dans mon parcours avec beaucoup d’attention, d’énergie et de bienveillance. Le reflet de son perfectionnisme m’a permis de me surpasser et d’atteindre mes objectifs. Avoir été sous sa codirection a été une expérience très formatrice et je lui en suis très reconnaissant.
xvii
Je dois aussi remercier Dominique Boudreau pour son soutien tout au long de mon parcours à la maîtrise. Il a toujours été présent pour m’aider et me conseiller tant avec sérieux qu’avec humour. Sa disponibilité et sa générosité en font un atout important dans une équipe.
Je remercie également Dr. Maurice Boissinot, qui dès mon arrivée comme stagiaire, m’a enseigné la rigueur scientifique et m’a poussé à toujours me questionner. Je le remercie également pour m’avoir permis d’étendre mes compétences et de m’avoir donné l’opportunité d’accéder au monde du réseautage scientifique.
Finalement, je remercie tous mes collègues et amis, par ordre alphabétique, Martine Bastien, Marthe Bernier, Ève Bérubé, Luc Bissonnette, Rana Daher, Johanne Frenette, Martin Gagnon, Marie-Claude Hélie, Julie Lamoureux, Éliane Lavergne, Amin Ahmed Ouameur, Frédéric Raymond, Jean-Luc Simard et Marilyse Vallée.
Je suis très fier du travail accompli avec vous. Bien qu’une maîtrise soit un défi intellectuel exigeant, j’ai eu beaucoup de plaisir tout au long de ma formation et c’est grâce à vous. Ce fut un réel bonheur de vous côtoyer tout au long de ma maîtrise. Merci!
xviii
Avant-Propos
Ce mémoire comporte deux articles, soit les chapitres 3 et 4, dont les travaux et la rédaction ont été réalisés avec la collaboration de coauteurs. Les informations concernant le statut d’auteur de l’étudiant, le rôle de l’étudiant dans la préparation de l’article, les coauteurs ayant participé à la préparation de l’article, le statut de l’article et certains commentaires pertinents précèdent chacun des deux articles.
1
Chapitre 1
2
1.1. Historique
1.1.1. Les maladies infectieuses à travers l’histoire
Les maladies infectieuses font partie intégrante de l’histoire de l’humanité. Plusieurs évidences de ce phénomène ont été rapportées, que ce soit par l’étude des fossiles des premiers humains descendant des singes, des corps momifiés de l’Égypte ancienne ou des corps humains conservés et préservés par la glace des montagnes de Russie et des Alpes (Cockburn, 1971; Maixner et al., 2016). Les maladies infectieuses comme la lèpre, la tuberculose et la syphilis sont plus faciles à rapporter puisqu’elles apportent des cicatrices à l’ossature. Les corps bien préservés et l’étude des fèces anciennes permettent également de trouver des lésions réalisées sur les organes et certains tissus par des bactéries ainsi que des œufs de parasites dans les fèces (Cockburn, 1971). À mesure que l’humain évoluait dans son mode de vie, les maladies infectieuses le touchant évoluaient aussi. L’arrivée de la sédentarisation, grâce à l’agriculture et la domestication des animaux, l’augmentation de la population et une centralisation par l’urbanisation ont fortement influencé l’évolution de ces maladies, augmentant les risques de transmission entre l’humain et les animaux et entre humains (Cockburn, 1971; Fauci & Morens, 2012; Heymann & Rodier, 2001). Ainsi, de grandes épidémies et pandémies ont affecté les civilisations à travers l’histoire. Pensons notamment à la tuberculose causée par la bactérie Mycobacterium tuberculosis qui affecta les populations humaines dès la préhistoire et qui atteignit son sommet au 18e et 19e siècle en Europe et en Amérique du Nord. Il y a aussi la peste, causée par la bactérie Yersinia
pestis, qui tua le tiers de la population européenne au milieu du 14e siècle. Pensons également au virus de la variole qui infecta pratiquement toute la population de l’Europe jusqu’à la fin du 18e
siècle (Cockburn, 1971; Daniel, 2006; Gage & Kosoy, 2005). Au début du 19e siècle, des expériences de transmission de maladies infectieuses chez les animaux ainsi que le développement de meilleurs microscopes ont permis un incroyable progrès quant à la compréhension des maladies infectieuses. Une des plus grandes percées favorisant l’avancement de la médecine revient à Robert Koch qui a réussi vers la fin du 19e siècle à démontrer l’association entre un microorganisme cultivable et une maladie spécifique. Koch a d’ailleurs établi des critères permettant d’affirmer le rapport de causalité qui sont encore utilisés aujourd’hui. Ces critères sont appelés les postulats de Koch (Daniel,
3
2006; Fauci & Morens, 2012). Ces avancements ont orienté le diagnostic de ces maladies ainsi que le développement de traitements spécifiques à certains pathogènes (Fauci & Morens, 2012). Des vaccins contre certaines maladies infectieuses ont été produits vers la fin des années 1870, mais il faut attendre le 20e siècle pour vivre le grand essor de la vaccination et l’apparition de substances chimiques particulières connues aujourd’hui sous le nom d’antibiotiques qui eurent un impact immense sur la santé humaine (Davies & Davies, 2010; Fauci & Morens, 2012).
1.1.2. Découverte des antibiotiques
En 1928, Alexander Fleming observa une contamination par une moisissure sur une culture de Staphylococcus datant de deux semaines, provenant d’une blessure infectée. Cependant, la moisissure semblait affecter la culture de Staphyloccoccus, tuant les bactéries se situant à proximité. C’est ainsi qu’Alexander Fleming découvrit le premier antibiotique qu’il nomma la pénicilline en référence à la moisissure observée qui était Penicillium notatum. L’ère moderne des antibiotiques était ainsi lancée (Allison, 1974; Fleming, 1945).
1.1.3. Impact des antibiotiques
Les antibiotiques ont transformé la médecine moderne, contribuant à sauver des millions de vies (Davies & Davies, 2010; Ventola, 2015a). Ils ont d’ailleurs eu un rôle crucial lors de la deuxième guerre mondiale, aidant à contrôler les infections d’origine bactérienne chez les soldats blessés (Ventola, 2015a). De plus, leur utilisation permet de prévenir et traiter les infections lors de transplantation d’organes, de traitements de chimiothérapies, de maladies chroniques, de chirurgies et autres. Leur usage a par le fait même contribué à élever l’espérance de vie humaine à travers le monde (Ventola, 2015a). Ils ont ainsi révolutionné le traitement des maladies infectieuses et l’histoire humaine (Davies & Davies, 2010; Yao & Moellering, 2011). Jusqu’à ce jour, des centaines d’antibiotiques ont été produits, commercialisés et utilisés comme antibioprophylaxie afin de prévenir les infections et comme antibiothérapie pour traiter les infections (Yao & Moellering, 2011).
4
1.2. Antibiotiques
Les antibiotiques sont des substances chimiques produites naturellement par des actinomycètes, des champignons et des bactéries ou bien synthétisées chimiquement qui interfèrent avec différentes fonctions physiologiques et biochimiques des cellules bactériennes afin de les tuer (bactéricide) ou d’inhiber leur croissance (bactériostatique) (Mascaretti, 2003c; Wright, 2010).
1.2.1. Mode d’action
Les antibiotiques ciblent ainsi différentes fonctions structurales et métaboliques des cellules bactériennes. Leurs mécanismes d’action consistent principalement à inhiber la synthèse de la paroi cellulaire, à endommager la membrane cellulaire, à empêcher la synthèse de l’ADN, de l’ARN ou des protéines et à inhiber le métabolisme de l’acide folique ou vitamine B9 (Figure 1.1).
Figure 1.1. Représentation schématique des cibles des antibiotiques ainsi que les mécanismes utilisés par les bactéries pour leur résister. Figure adaptée de Wright, 2010.
5
Les antibiotiques sont ainsi relativement non toxiques pour l’humain puisque ces cibles sont présentes chez les cellules procaryotes, comme les bactéries, mais absentes ou différentes chez les cellules eucaryotes dont l’humain est constitué (Mascaretti, 2003c; Wright, 2010).
1.2.2. β-lactamines
Les β-lactamines représentent la famille d’antibiotique la plus utilisée en antibioprophylaxie et en antibiothérapie et sont l’objet du chapitre 3 de ce mémoire. Cette famille d’antibiotiques bactéricides est constituée de cinq groupes, soit les pénicillines, les céphalosporines (1ère à 5e génération), les monobactames, les carbapénèmes et les inhibiteurs de β-lactamases. Les β-lactamines sont caractérisées par la présence d’un noyau β-lactame (Figure 1.2) (Mascaretti, 2003c; Rice & Bonomo, 2011; Wright, 2010; Yao & Moellering, 2011).
Figure 1.2. Représentation schématique de la pénicilline (β-lactamine) et de son noyau β-lactame. Figure adaptée de Mascaretti, 2003d.
1.2.2.1. Mode d’action des β-lactamines
Les β-lactamines inhibent la synthèse de la paroi cellulaire des bactéries à Gram négatif (Gram-) et à Gram positif (Gram+) en inhibant la synthèse du peptidoglycane qui est le principal composé de la paroi cellulaire. Elles ont une structure homologue au motif dipeptide D-alanyl-D-alanine (D-Ala-D-Ala) de l’unité pentapeptidique du peptidoglycane qui est le substrat des enzymes D,D-peptidases, plus communément connues sous le nom de protéines liant la pénicilline (PLPs). Les PLPs permettent la réticulation des
6
peptidoglycanes à la fin de la synthèse de la paroi. Les β-lactamines vont donc imiter le motif D-Ala-D-Ala et être liées par les PLPs au lieu du peptidoglycane normal, inhibant ainsi la synthèse de la paroi cellulaire, ce qui entraîne la mort de la cellule (Mascaretti, 2003c; Mascaretti, 2003d; Wright, 2010; Yao & Moellering, 2011).
1.2.3. Vancomycine
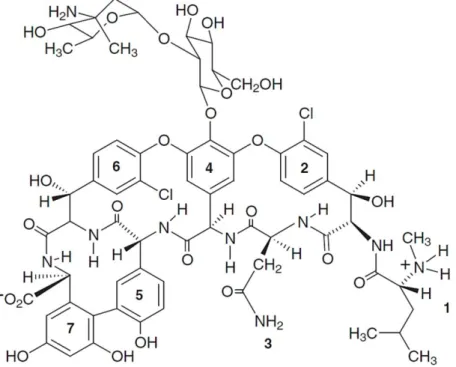
La vancomycine est un antibiotique produit par la bactérie Streptomyces orientalis (Yao & Moellering, 2011) et est l’objet du chapitre 4 de ce mémoire. Elle est un glycopeptide bactéricide qui est très utilisé pour le traitement d’infections sévères causées par des bactéries à Gram+ (Domingo et al., 2005a). Sa structure est composée de sept acides aminés organisés structurellement pour former un peptide cyclique (Figure 1.3) (Mascaretti, 2003c).
Figure 1.3. Représentation schématique de la vancomycine et de ses sept acides aminés. Figure adaptée de Mascaretti, 2003c.
1.2.3.1. Mode d’action de la vancomycine
La vancomycine inhibe la synthèse de la paroi cellulaire des bactéries à Gram+ en empêchant la synthèse de la chaîne du peptidoglycane. Elle se lie au dipeptide terminal
D-7
Ala-D-Ala du pentapeptide précurseur de la chaîne du peptidoglycane. La synthèse de la paroi cellulaire est donc arrêtée, entraînant la mort cellulaire (Domingo et al., 2005a; Domingo et al., 2007; Yao & Moellering, 2011). La vancomycine est inefficace contre les bactéries à Gram- puisqu’elle ne peut pas atteindre sa cible, les précurseurs du peptidoglycane. Chez les bactéries à Gram-, ceux-ci sont situés entre les membranes externes et internes, dans l’espace périplasmique. La taille trop volumineuse de la vancomycine (Figure 1.3) ne lui permet donc pas d’utiliser les porines pour atteindre sa cible (Rice & Bonomo, 2011).
8
1.3. Résistance des bactéries aux antibiotiques
1.3.1. Problématique
« […] I would like to sound one note of warning. […] There may be a danger, though, in underdosage. It is not difficult to make microbes resistant to penicillin in the laboratory by exposing them to concentrations not sufficient to kill them, and the same thing has occasionally happened in the body. […] negligent use of penicillin changed the nature of the microbe. »
Alexander Fleming, 1945 (Fleming, 1945) Comme il a été mentionné précédemment, Alexander Fleming a découvert la pénicilline en 1928, mais dès 1940, avant l’introduction même de la pénicilline comme traitement des maladies infectieuses, une enzyme s’attaquant au nouvel antibiotique a été découverte. Cette enzyme a été appelée à l’époque pénicillinase, mais ce type d’enzyme porte maintenant le nom de β-lactamase (Davies & Davies, 2010). Par ailleurs, la résistance à la vancomycine chez les entérocoques a été observée pour la première fois en 1988. Depuis ce temps, la résistance aux antibiotiques a émergé à travers le monde entier, causant un problème clinique majeur (Fang et al., 2007). Les bactéries possèdent une très grande capacité d’adaptation, leur permettant d’être très rapide dans l’exploitation de nouvelles opportunités et ainsi de résister aux antibiotiques (Heymann & Rodier, 2001). Les avertissements de Fleming n’ont pas été pris en considération, du moins, pas suffisamment. Les antibiotiques ont été utilisés à profusion comme traitement clinique des maladies infectieuses ainsi que dans le domaine alimentaire pour l’élevage des animaux de ferme destinés à la consommation humaine (Lebreton et al., 2013; Sommer et al., 2009). De plus, leur prescription était souvent inadaptée, présentant des dosages et des durées de traitement inadéquats (Sommer et al., 2010; Sommer et al., 2009). Cet abus a ainsi créé une forte pression sélective engendrant une accumulation de résistance aux antibiotiques chez les bactéries (Jernberg et al., 2010; Levy, 2001). En effet, l’utilisation des antibiotiques a forcé les bactéries à s’adapter à leur présence et à y résister par différents moyens (Kirchner et al., 2014). Une fois la résistance acquise chez les bactéries, elles effectuent une
9
dissémination verticale aux nouvelles générations qui conservent cette résistance. Cependant, une dissémination horizontale est aussi possible par le transfert ou le partage de cette résistance avec d’autres bactéries, comprenant les pathogènes (Wright, 2010). La résistance ainsi que la multirésistance ont ainsi connues une très forte croissance chez les pathogènes humains au cours des dernières décennies, rendant le traitement des infections, auparavant bien contrôlées, beaucoup plus difficile (Forsberg et al., 2012; Sommer et al., 2009). Cette évolution continue, amplifiant le problème au niveau de crises sévères qui nous affligent présentement (Forsberg et al., 2012; Levy, 2001).
1.3.2. Impact de la résistance
L’utilité des antibiotiques n’est plus à prouver, ayant contribué fortement à l’avancement médical et ayant littéralement révolutionné la santé publique (Spellberg et al., 2008). Ainsi, la croissance actuelle de la résistance aux antibiotiques amène un impact majeur, augmentant considérablement les taux de morbidité et de mortalité ainsi que les coûts associés aux infections (Cohen, 1992). En effet, la résistance conduit à des traitements non appropriés et inefficaces. Le taux de mortalité et la durée du séjour à l’hôpital sont ainsi deux fois supérieurs chez un patient infecté par une bactérie résistante comme une entérobactérie multirésistante (EMR), un Staphylococcus aureus résistant à la méticilline (SARM) ou un entérocoque résistant aux glycopeptides (ERG) en comparaison au taux de mortalité et à la durée du séjour chez un patient infecté par la même souche bactérienne sensible. Les coûts sont ainsi fortement augmentés (French, 2010). Aux États-Unis, le séjour à l’hôpital d’un patient atteint d’une infection bactérienne résistante sera prolongé de 6,4 à 12,7 jours, ayant un impact négatif pour le patient, mais aussi au niveau des coûts qui y sont associés. Les coûts médicaux estimés pour un patient ayant une infection bactérienne présentant une résistance sont de 18 588$ à 29 069$ USD. Ainsi, aux États-Unis, les coûts occasionnés par les infections bactériennes résistantes sont de 20 milliards de dollars par année pour les soins de santé. De plus, ces patients sont dans l’incapacité de travailler, causant 35 milliards de dollars de pertes par année associées à la non productivité (Golkar et al., 2014).
10
1.3.3. Résistance naturelle
Les bactéries peuvent être parfois naturellement résistantes à certains agents antibactériens. On appelle ce phénomène une résistance intrinsèque (Mascaretti, 2003b). Ces bactéries possèdent certains gènes dans leur génome leur conférant cette résistance (Davies & Davies, 2010). La capacité à résister est naturelle. Les bactéries du sol vivent dans des environnements présentant des toxines, des molécules bioactives et autres substances qui les forcent à évoluer afin d’interagir avec elles et de maintenir un contrôle de leurs effets. Cependant, les pathogènes humains ne sont généralement pas exposés à ces environnements (Wright, 2010). Ils doivent donc trouver une autre façon d’acquérir cette résistance.
1.3.4. Résistance acquise génétiquement
Les bactéries peuvent acquérir une résistance aux antibiotiques de façon génétique. Les deux principaux mécanismes impliquent des mutations et/ou l’acquisition de gènes de résistance (Mascaretti, 2003b; Rice & Bonomo, 2011).
1.3.4.1. Mutation
Pour produire leur effet bactéricide ou bactériostatique, les antibiotiques ciblent souvent une protéine ayant une fonction essentielle chez certaines bactéries. Ces interactions entre antibiotiques et leurs cibles sont spécifiques. Ainsi, une mutation d’une seule base dans le gène codant pour cette protéine causant un changement d’acide aminé impliqué dans le site de liaison de l’antibiotique peut perturber cette interaction et provoquer une résistance à l’antibiotique. Rappelons que ce type de mutation ponctuelle se produit environ 1 fois par 108 unités formatrices de colonies (UFC) chez les bactéries (Rice & Bonomo, 2011). La pression exercée par la présence d’antibiotiques favorise le nouveau mutant qui peut transmettre sa résistance verticalement à la génération future ou de façon horizontale à d’autres bactéries (Mascaretti, 2003b; Rice & Bonomo, 2011).
1.3.4.2. Acquisition de gènes
Les bactéries peuvent acquérir des gènes provenant de cellules non parentales leur conférant une résistance aux antibiotiques. Ce phénomène est très important pour l’évolution bactérienne (Mascaretti, 2003b; Smillie et al., 2011). Il existe trois mécanismes
11
permettant ce transfert de matériel génétique : 1) la conjugaison, 2) la transformation naturelle et 3) la transduction. De plus, ce transfert devient possible par l’intermédiaire de différents éléments, notamment les plasmides, les éléments transposables ainsi que les intégrons (Davies & Davies, 2010; Mascaretti, 2003b; Rice & Bonomo, 2011).
1.3.4.2.1. Mécanismes de transfert de gènes 1.3.4.2.1.1. Conjugaison
La conjugaison bactérienne est un mécanisme nécessitant un contact direct entre la cellule donneuse et la cellule réceptrice pour effectuer le transfert de matériel génétique. De plus, la synthèse d’un pilus sexuel est nécessaire pour les bactéries à Gram-. Les gènes permettant sa synthèse sont situés dans la région tra du plasmide. C’est ce pilus, présent chez la cellule donneuse, qui permet l’appariement spécifique entre les deux cellules par contact avec le récepteur de la cellule réceptrice, créant un tunnel dans lequel passera un des brins d’ADN du plasmide conjugatif. La synthèse des brins complémentaires des deux simples brins d’origine du plasmide est effectuée et il peut y avoir recircularisation des plasmides ou recombinaison homologue. La conjugaison est plus fréquente chez les bactéries à Gram-, mais il existe aussi des bactéries à Gram+ utilisant ce mécanisme. Il peut également y avoir des transposons conjugatifs induisant la conjugaison (Mascaretti, 2003a; Mascaretti, 2003b; Rice & Bonomo, 2011).
1.3.4.2.1.2. Transformation naturelle
La transformation naturelle consiste en l’absorption par une cellule bactérienne d’un fragment d’ADN présent dans le milieu. L’ADN est ensuite intégré au chromosome de la cellule par recombinaison homologue. Cependant, ce ne sont pas toutes les bactéries qui sont naturellement compétentes, c’est-à-dire capable d’effectuer la transformation. Il existe toutefois certaines bactéries à Gram- et à Gram+ qui le sont, notamment Neisseria
gonhorrhoeae qui est une bactérie à Gram- et Streptococcus pneumoniae qui est une
bactérie à Gram+ (Mascaretti, 2003a; Rice & Bonomo, 2011).
1.3.4.2.1.3. Transduction
La transduction est un mécanisme par lequel les bactéries utilisent les virus qui les infectent, soit les bactériophages, pour partager leur matériel génétique. Les bactériophages
12
infectant les bactéries se reproduisent à l’intérieur de ces dernières utilisant un cycle lytique. Au fil de l’assemblage des phages, l’ADN des virus est incorporé dans les capsides des bactériophages. Cependant, il arrive que certains fragments d’ADN bactérien soient aussi incorporés dans les capsides des virus. Les bactériophages vont par la suite infecter d’autres bactéries, introduisant par le fait même certains gènes bactériens provenant de leurs infections passées. Il peut ainsi y avoir recombinaison homologue du fragment d’ADN ou recircularisation dans le cas d’un plasmide (Mascaretti, 2003a; Rice & Bonomo, 2011).
1.3.4.2.2. Plasmides
Les plasmides sont présents chez plusieurs bactéries. Ce sont de petites unités d’ADN circulaire autonomes et distinctes de l’ADN chromosomique. Les plasmides peuvent apporter une résistance à certains antibiotiques à leurs cellules bactériennes hôtes. Ils sont alors appelés plasmides ou facteurs R. Ces plasmides contiennent ainsi un ou plusieurs gènes de résistance aux antibiotiques et peuvent être transmis principalement par conjugaison. Par ailleurs, les gènes de résistance portés par les plasmides peuvent être associés à des transposons ou des intégrons (Mascaretti, 2003a).
1.3.4.2.3. Éléments transposables
Les éléments transposables sont des segments d’ADN qui ont la capacité de faire la transposition, c’est-à-dire de se déplacer d’un endroit à l’autre sur un réplicon, par exemple un chromosome bactérien ou un plasmide. Ces éléments peuvent aussi utiliser le mécanisme de conjugaison, permettant ainsi de se déplacer d’un chromosome bactérien vers le chromosome d’une autre bactérie. Les formes les plus simples sont des séquences d’insertion (IS), qui sont de petits éléments génétiques ne contenant que le matériel nécessaire à la transposition. Les éléments transposables plus grands, les transposons, contiennent tout le matériel nécessaire à leur transposition ainsi que des séquences de gènes supplémentaires, notamment des gènes de toxines ou des gènes de résistance aux antibiotiques (Mascaretti, 2003a; Rice & Bonomo, 2011).
1.3.4.2.4. Intégrons
Les intégrons sont des unités génétiques qui ont la capacité de capturer des éléments mobiles qu’on appelle cassettes de gènes. Ces cassettes comportent un gène qui est souvent
13
un gène de résistance aux antibiotiques. Les intégrons sont composés du site d’insertion des cassettes, un gène codant pour une protéine intégrase, qui permet une recombinaison spécifique pour intégrer ou exciser les cassettes, ainsi qu’un promoteur pour la transcription des gènes des cassettes (Mascaretti, 2003a).
1.3.5. Mécanismes de résistance aux antibiotiques
Les antibiotiques ciblent différentes fonctions structurales et biochimiques des cellules bactériennes pour les tuer ou inhiber leur croissance. Les bactéries ont développé des mécanismes de défense leur permettant de déjouer l’action des antibiotiques et ainsi de leur résister (Wright, 2010).
1.3.5.1. Trois différents mécanismes
Il existe trois mécanismes leur permettant de résister à l’action des antibiotiques. Ceux-ci sont l’altération de la perméabilité membranaire, l’altération des cibles ainsi que l’inactivation enzymatique (Figure 1.1) (Rice & Bonomo, 2011).
1.3.5.1.1. Altération de la perméabilité membranaire
L’altération de la perméabilité membranaire consiste à bloquer l’accès aux cibles des antibiotiques. Cette altération peut être causée par un mécanisme d’efflux ou par la modification, la diminution ou la perte des porines. Le mécanisme d’efflux actif est caractérisé par la présence de protéines agissant comme pompes. Une fois localisé à l’intérieur de la cellule, l’antibiotique est éjecté par une protéine de la grande famille des pompes. Ces pompes peuvent être présentes chez les bactéries à Gram- comme à Gram+ et sont généralement situées dans la membrane cytoplasmique (Rice & Bonomo, 2011; Wright, 2010). D’autre part, les porines sont des protéines membranaires formant des canaux qui permettent à des molécules comme les petits antibiotiques de traverser la membrane externe des bactéries à Gram- pour atteindre la membrane cytoplasmique (Rice & Bonomo, 2011).
1.3.5.1.2. Altération des cibles
L’altération des cibles consiste à modifier la cible de l’antibiotique chez les cellules bactériennes afin d’empêcher leur interaction et contrer l’effet de l’antibiotique.
14
L’interaction entre un antibiotique et sa cible est souvent spécifique. Ainsi, une altération mineure de la cible empêche la liaison de l’antibiotique qui devient inefficace (Rice & Bonomo, 2011).
1.3.5.1.3. Inactivation enzymatique
L’inactivation enzymatique constitue une défense très spécifique et évoluée contre les antibiotiques. Les cellules bactériennes produisent des enzymes qui vont inactiver les antibiotiques de façon à ce qu’ils soient inefficaces. Les antibiotiques perdent leur effet et ne peuvent plus interagir avec leurs cibles. Les bactéries ayant cette capacité deviennent ainsi résistantes envers les antibiotiques (Rice & Bonomo, 2011; Wright, 2010).
1.3.5.2. Mécanismes de résistance aux β-lactamines
L’étude des gènes de résistance aux β-lactamines est l’objet du chapitre 3 de ce mémoire. Les mécanismes de résistance à cette classe d’antibiotiques seront décrits dans cette section. Les trois mécanismes de résistance aux antibiotiques décrits précédemment s’appliquent à la résistance aux β-lactamines qui font partie de la famille d’antibiotiques la plus utilisée. L’altération de la perméabilité par des mécanismes d’efflux actif permet aux bactéries d’éjecter les β-lactamines. L’altération de la perméabilité membranaire par la diminution du nombre de porines ou bien la modification ou la perte de celles-ci empêche les β-lactamines de pénétrer à l’intérieur de la cellule. L’altération de la cible est aussi un mécanisme utilisé pour contrer les β-lactamines. Dans ce cas-ci, les cibles sont les PLPs. On peut observer l’altération des PLPs sous quatre formes, soit une production augmentée des PLPs, l’acquisition de PLPs étrangères, des mutations des PLPs par recombinaison avec de l’ADN étranger et finalement des mutations ponctuelles des PLPs. Dans le premier cas, les PLPs sont en surnombre rendant les β-lactamines inefficaces tandis que dans les trois autres cas, les β-lactamines ne peuvent plus interagir avec les PLPs (Mascaretti, 2003d; Rice & Bonomo, 2011). Cependant, le mécanisme de résistance aux β-lactamines le plus répandu chez les bactéries est l’inactivation enzymatique par la production d’enzymes spécifiques nommées β-lactamases (Jacoby, 1994; Mascaretti, 2003d).
15
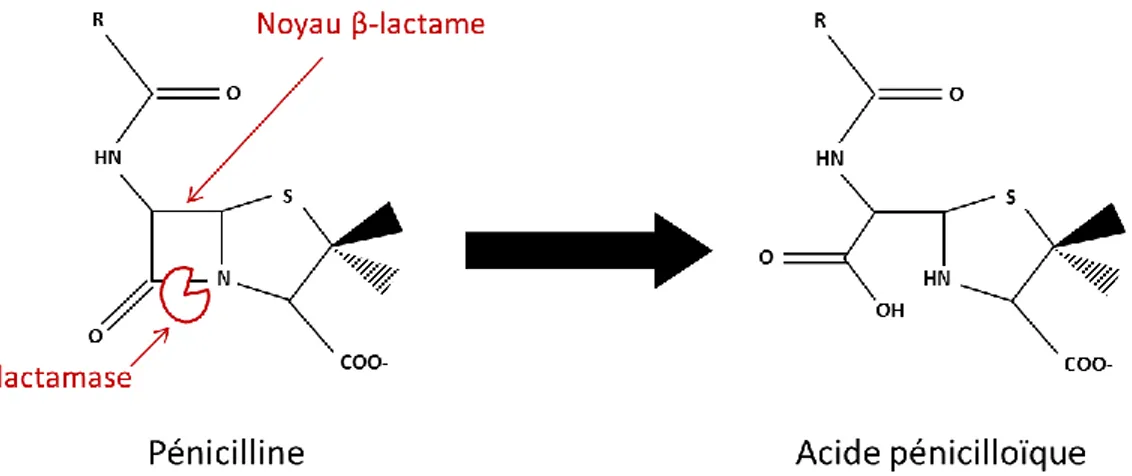
1.3.5.2.1. β-lactamases
Comme il a été mentionné précédemment, la production de β-lactamases est le mécanisme le plus communément utilisé pour résister aux β-lactamines, en particulier chez les bactéries à Gram- cliniquement importantes (Bush & Jacoby, 2010; Jacoby, 1994; Mascaretti, 2003d). Les lactamases permettent de contrecarrer l’effet létal des β-lactamines sur les bactéries (Handal et al., 2004). Elles hydrolysent le noyau β-lactame des β-lactamines en ajoutant une molécule d’eau entre le lien carbone (C) et azote (N), permettant ainsi l’ouverture du noyau et rendant l’antibiotique inapte à l’inhibition de sa cible, les PLPs (Figure 1.4) (Bush & Jacoby, 2010; Mascaretti, 2003b; Mascaretti, 2003d). Les β-lactamases peuvent être produites de manière constitutive ou inductible (Mascaretti, 2003d; Rice & Bonomo, 2011). De plus, les gènes codant pour celles-ci peuvent être situés sur le chromosome bactérien, sur un plasmide, sur un transposon ou même sur un intégron présents dans des cassettes de gènes. La possibilité de transfert de ces gènes en est donc augmentée (Handal et al., 2004; Rice & Bonomo, 2011). Les β-lactamases sont sécrétées dans l’espace périplasmique chez les bactéries à Gram- et dans le milieu extérieur chez les bactéries à Gram+ (Mascaretti, 2003d). L’étude des gènes codant pour ces enzymes devient donc importante pour choisir les traitements appropriés chez les patients présentant une infection causée par une bactérie productrice de β-lactamases (Bush & Jacoby, 2010).
Figure 1.4. Représentation schématique de l’inactivation enzymatique de la pénicilline (β-lactamine) par une β-lactamase qui vient hydrolyser le noyau β-lactame de l’antibiotique. Figure adaptée de Mascaretti, 2003d.
16
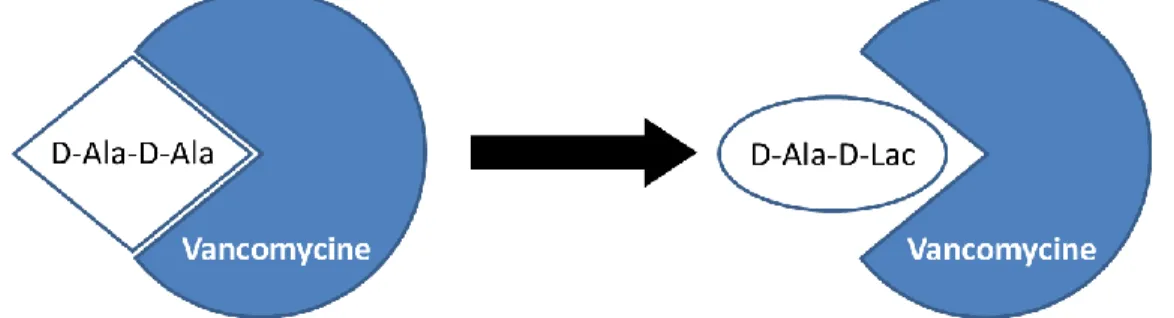
1.3.5.3. Mécanismes de résistance à la vancomycine
L’étude des gènes de résistance à la vancomycine, principalement de type VanD est l’objet du chapitre 4 et les mécanismes de résistance à cet antibiotique seront décrits dans cette section. Le mécanisme de résistance utilisé par les bactéries pour contrer l’effet bactéricide de la vancomycine est l’altération de sa cible (Courvalin, 2006; Rice & Bonomo, 2011; Wright, 2010). La vancomycine a une forte interaction avec le dipeptide terminal D-Ala-D-Ala du pentapeptide précurseur du peptidoglycane. La résistance est ainsi causée par la présence d’opérons produisant certaines enzymes qui vont altérer la synthèse du dipeptide terminal et conduire à la synthèse des lactate (D-Ala-D-Lac) ou D-alanyl-D-sérine (D-Ala-D-Ser) (Courvalin, 2006). Cette altération est effectuée par plusieurs types d’opérons différents présents chez les bactéries (Rasheed & Tenover, 2011; Werner et al., 2008). À ce jour, dix types d’opéron impliqués dans la résistance à la vancomycine ont été caractérisés. Les opérons permettant la synthèse du D-Ala-D-Lac sont vanA, vanB, vanD,
vanF et vanM tandis que ceux produisant le D-Ala-D-Ser sont vanC, vanE, vanG, vanL et vanN (Depardieu et al., 2015; Fraimow et al., 2005; Rubinstein & Keynan, 2014). La
synthèse d’un dipeptide terminal modifié cause une forte diminution de l’affinité envers la vancomycine, conduisant à la résistance de la cellule bactérienne envers cet antibiotique (Figure 1.5) (Domingo et al., 2005b).
Figure 1.5. Représentation schématique de la diminution de l’affinité envers la vancomycine par l’incorporation du dipeptide D-Ala-D-Lac. Figure adaptée de
Courvalin, 2006.
1.3.5.3.1. Gène de résistance à la vancomycine de type VanD
Le premier cas décrit de résistance à la vancomycine de type VanD a été observé en 1991, dans un hôpital de New-York, aux États-Unis. La résistance a été retrouvée chez la bactérie
17
Enterococcus faecium qui a été isolée à partir d’un échantillon d’urine (Ostrowsky et al.,
1999). La résistance à la vancomycine de type VanD est caractérisée par son expression constitutive chez les entérocoques apportant une résistance modérée aux glycopeptides, soit une concentration minimale inhibitrice (CMI) de 64 à 128 µg/ml pour la vancomycine et de 4 à 64 µg/ml pour la téicoplanine (Depardieu et al., 2004; Domingo et al., 2007). La résistance est causée par la présence de l’opéron vanD localisé sur le chromosome bactérien uniquement. Malgré la présence d’un gène intD codant pour une intégrase localisée près de l’opéron vanD, des expériences ont montré que l’opéron vanD ne peut être transféré in vitro chez d’autres entérocoques (Domingo et al., 2007; Werner et al., 2008). L’opéron vanD est composé de 6 gènes : vanRD, vanSD, vanYD, vanHD, vanD et vanXD (Figure 1.6) (Courvalin,
2006; Xu, X. et al., 2010).
Figure 1.6. Représentation schématique des différents gènes constituant le locus vanD.
Figure adaptée de Courvalin, 2006.
Comme il a été mentionné précédemment, l’opéron vanD cause de la résistance par la synthèse du D-Ala-D-Lac. Les 6 gènes de l’opéron jouent chacun un rôle dans l’expression de la résistance. La synthèse du D-Ala-D-Lac nécessite une déshydrogénase, VanHD, qui
convertit le pyruvate en D-Lac. Par la suite, le précurseur se terminant par D-Ala est enlevé par la D,D-dipeptidase VanXD hydrolysant le dipeptide D-Ala-D-Ala produit par la ligase
Ddl de la bactérie hôte. La D,D-carboxypeptidase VanYD permet de cliver le D-Ala
terminal des précurseurs pentapeptidiques du peptidoglycane qui ont échappé à l’hydrolyse de la D,D-dipeptidase VanXD. La ligase, soit VanD, permet ainsi de joindre les deux
peptides D-Ala et D-Lac formant ainsi le dipeptide D-AlaD-Lac. Les gènes vanHD, vanD et
vanXD sont donc essentiels à la résistance (Courvalin, 2006; Depardieu et al., 2004;
Depardieu et al., 2003). Présentement, sept allèles du gène vanD ont été décrits, soit vanD1 à vanD7 (Boyd et al., 2000; Boyd et al., 2004; Boyd et al., 2016; Boyd et al., 2006; Casadewall & Courvalin, 1999; Dalla Costa et al., 2000; Ostrowsky et al., 1999). VanSD est
18
une histidine kinase agissant comme détecteur et VanRD est un régulateur agissant comme
activateur de la transcription. Ces deux protéines réunies permettent la régulation de l’expression de la résistance de type VanD (Depardieu et al., 2004; Depardieu et al., 2003).
19
1.4. Ère post-antibiotique?
L’émergence croissante de résistance et multi-résistance aux antibiotiques chez les bactéries pathogènes devient un problème particulièrement préoccupant. D’autant plus que le nombre d’agents antibactériens en développement ne cesse de diminuer. Certains pathogènes sont maintenant résistants à pratiquement, voire tout l’arsenal d’antibiotiques disponibles sur le marché. Tous ces éléments réunis laissent planer la menace prochaine d’une nouvelle ère post-antibiotique, où notre capacité à traiter les infections d’origine bactériennes serait révolue (Gibson et al., 2015; Heymann & Rodier, 2001; Schulz zur Wiesch et al., 2010).
1.4.1. Pourquoi si peu de nouveaux antibiotiques?
Devant un problème de cette envergure, le besoin de nouveaux agents antibactériens est urgent. Pourtant, le développement de nouveaux antibiotiques est en chute depuis trois décennies (Ventola, 2015a). La cause de cette diminution se rapporte à un ensemble de facteurs tous en lien avec l’économie (Spellberg et al., 2008; Ventola, 2015a; Wright, 2010). Tout d’abord, le développement de nouveaux antibiotiques ne représente plus un bon investissement pour l’industrie pharmaceutique. En effet, les antibiotiques sont des médicaments utilisés pour traiter définitivement une infection d’origine bactérienne et leur utilisation est restreinte à de courtes périodes de temps dépassant rarement deux semaines (Spellberg et al., 2008). Leur production devient ainsi beaucoup moins rentable par rapport au marché des traitements des maladies chroniques comme le diabète, les maladies psychiatriques, l’asthme et bien d’autres. La valeur actuelle nette d’un nouvel antibiotique est de 50 millions de dollars, tandis que la valeur actuelle nette approximative d’un médicament traitant une maladie neuromusculaire est de 1 milliard de dollars, ce qui est beaucoup plus alléchant pour les compagnies pharmaceutiques. De plus, le prix de vente d’un antibiotique est relativement bas en comparaison avec, par exemple, les traitements de chimiothérapies pour les cancers (Ventola, 2015a). Par ailleurs, les scientifiques, microbiologistes et infectiologues recommandent la restriction des antibiotiques. Les médecins, par souci de diminution de la pression sélective afin d’éviter l’émergence de nouvelles résistances, continuent à prescrire les anciens agents antibactériens et conservent
20
les nouveaux antibiotiques comme derniers recours, ce qui n’incite pas les compagnies pharmaceutiques à en développer de nouveaux. Finalement, l’apparition de résistance est inévitable et une compagnie développant un nouvel antibiotique prend toujours le risque d’une apparition rapide de cette résistance après la mise en marché, diminuant ainsi drastiquement les profits attendus (Ventola, 2015a).
1.4.2. Y a-t-il des alternatives aux nouveaux antibiotiques?
Il existe certaines alternatives au développement de nouveaux antibiotiques, notamment l’utilisation d’anciens antibiotiques qui n’ont pas été sélectionnés pour être commercialisés, ayant une toxicité plus élevée. Ce choix devient maintenant nécessaire pour traiter certaines infections causées par des pathogènes particulièrement résistants (Boucher et al., 2009). Il est aussi possible de modifier les agents antibactériens existants ou bien de les utiliser de façon cyclique afin de prolonger l’efficacité de l’antibiotique utilisé (Davies & Davies, 2010). Une autre alternative est la combinaison de deux agents antibactériens pour augmenter l’efficacité. Cependant, ces méthodes ne permettent pas d’éliminer la pression sélective (Cohen, 1992). Il y a aussi d’autres stratégies à étudier, comme les vaccins qui sont à caractère préventif, les bactériophages, soit des virus s’attaquant aux bactéries, les activateurs de l’immunité innée (Wright, 2010) ainsi que les antitoxines qui vont agir directement sur les toxines produites par certaines bactéries, comme le Clostridium difficile (Marozsan et al., 2012). Le diagnostic rapide, non seulement des bactéries, mais également des toxines produites par les bactéries, prend ainsi toute son importance. Les modificateurs de la réponse biologique pourront également être utilisés afin de réduire la réponse inflammatoire excessive notamment par la diminution de la libération des cytokines pro-inflammatoires (Duong et al., 1998).
1.4.3. Que peut-on faire pour ralentir l’émergence de résistance?
Nous sommes complètement dépendants des antibiotiques et leur utilisation est nécessaire au succès des traitements des maladies infectieuses, mais aussi au succès des chirurgies par la prévention des infections post-opératoires (Davies & Davies, 2010). L’émergence de résistance aux antibiotiques est inévitable et la population de la planète entière est touchée par ce problème croissant (Heymann & Rodier, 2001). Cependant, certains actes peuvent
21
être réalisés pour ralentir et limiter l’émergence de nouvelles résistances. Tout d’abord, il est primordial d’implanter un système de surveillance mondial afin d’assurer un meilleur contrôle des maladies infectieuses et de la résistance (Heymann & Rodier, 2001). De plus, les antibiotiques doivent être utilisés de façon contrôlée et adéquate. Un guide de prescription des agents antibactériens devrait être instauré afin d’améliorer les prescriptions pour qu’elles soient précises et appropriées à l’infection d’origine bactérienne à traiter (Davies & Davies, 2010; French, 2010; Ventola, 2015b). Il devrait ainsi y avoir une surveillance rigoureuse dans les hôpitaux et cliniques, mais également en agriculture, pour assurer un contrôle des thérapies effectuées chez les animaux de ferme ainsi que l’abolition de l’utilisation d’antibiotiques dans leur alimentation (Davies & Davies, 2010). Par ailleurs, un meilleur contrôle quant à la transmission des maladies infectieuses influencerait systématiquement l’émergence de résistance (French, 2010). Ainsi, afin de prévenir la transmission dans les hôpitaux, une meilleure gestion physique des lieux doit être établie, comprenant une désinfection accrue des locaux et du matériel ainsi que le dépistage de porteurs asymptomatiques. Il est aussi essentiel d’améliorer la précision et la rapidité des outils de diagnostic des maladies infectieuses (Boissinot & Bergeron, 2002; Ventola, 2015b). Ces actions permettraient ainsi de ralentir l’émergence de nouvelles résistances. Finalement, la recherche d’alternatives aux antibiotiques est souhaitable, mais la recherche de nouveaux agents antibactériens ne doit pas s’arrêter, car ceux-ci restent indispensables au maintien de la qualité des soins de santé (Davies & Davies, 2010). De nouvelles stratégies sont présentement discutées et utilisées pour découvrir de nouveaux antibiotiques. Il y a notamment la recherche de nouvelles cibles, d’inhibiteurs de la résistance et de la virulence ainsi que de nouvelles techniques de culture des micro-organismes, utilisant des milieux plus naturels qui reflètent davantage l’environnement des bactéries et qui comprennent les facteurs potentiels qui pourraient être nécessaires à la production d’antibiotiques (Ventola, 2015b; Wright, 2014). Aussi, de nouvelles sources d’antibiotiques naturels sont examinées. Des sources qui étaient jusqu’à maintenant moins connues ou étudiées, comme les forêts tropicales, les bactéries marines, les bactéries vivant dans des environnements extrêmes, etc. (Ventola, 2015b).
22
1.5. Microbiote
Le microbiote représente l’ensemble des micro-organismes, bactéries, archaebactéries, champignons et virus, peuplant un environnement distinct (Morgan & Huttenhower, 2012). Ainsi, l’humain possède son propre microbiote puisque toute la surface et les cavités du corps sont tapissées de micro-organismes, principalement des bactéries (Highlander et al., 2011; Morgan & Huttenhower, 2012). Cependant, la plupart des bactéries du corps humain résident dans son intestin, qu’on qualifie de microbiote intestinal humain (Gill et al., 2006; Qin et al., 2010; Wang et al., 2015; Xu, J. & Gordon, 2003).
1.5.1. Microbiote intestinal humain
Le microbiote intestinal humain présente une très forte densité de bactéries. En effet, l’intestin d’un adulte est l’hôte d’environ 1013
-1014 bactéries, ce qui correspond à une à dix fois le nombre de cellules nucléées du corps humain (Clemente et al., 2012; de Vries et al., 2011; Highlander et al., 2011; Quigley, 2010; Sender et al., 2016; Xu, J. & Gordon, 2003). Il y a d’ailleurs plus de bactéries dans notre intestin que d’étoiles dans notre galaxie (1011 étoiles) (NASA, 2015). Le microbiote intestinal normal est dominé par des bactéries anaérobies qui sont de 100 à 1000 fois plus abondantes que les bactéries aérobies et aérobies facultatives (Quigley, 2010). De plus, le microbiote intestinal est composé d’environ 1000 espèces bactériennes différentes qui se séparent en seulement quelques phyla (Xu, J. & Gordon, 2003). Les deux principaux phyla sont les Bacteroidetes qui sont des bactéries à Gram- et les Firmicutes dont la majorité ont une structure de paroi cellulaire à Gram+ (Cheng et al., 2012; Cho & Blaser, 2012; Coyne et al., 2014; Highlander et al., 2011; Jernberg et al., 2010; Marathe et al., 2014; Qin et al., 2010; Sommer et al., 2010; Stentz et al., 2015). Ceux-ci représentent plus de 90% du microbiome intestinal humain (Eckburg et al., 2005; Qin et al., 2010). Le microbiome correspond à l’ensemble des gènes codés par les micro-organismes du microbiote (Clemente et al., 2012). D’autre part, le microbiote intestinal humain occupe des fonctions importantes pour le corps humain. Il aide à la digestion par la synthèse d’acides aminés et de vitamines et contribue au maintien du système immunitaire, favorisant la maturation des cellules immunitaires et le
23
développement des fonctions immunitaires (Clemente et al., 2012; Gill et al., 2006; Highlander et al., 2011; Morgan & Huttenhower, 2012).
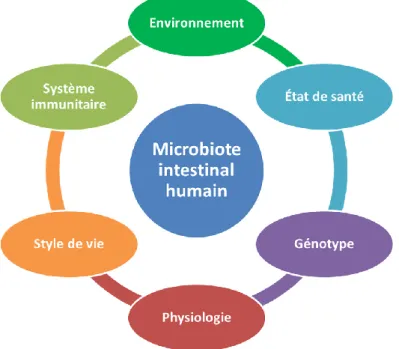
Par ailleurs, le microbiote intestinal humain peut varier selon plusieurs facteurs de l’hôte, notamment son environnement, son état de santé, son génotype, sa physiologie, son style de vie et son système immunitaire (Figure 1.7) (Turnbaugh et al., 2007). L’exposition aux antibiotiques peut également avoir un grand impact sur le microbiote intestinal. En effet, les antibiotiques réduisent la population commensale utile à l’humain, augmentant la possibilité de colonisation du microbiote par des pathogènes opportunistes (par exemple
Clostridium difficile), en plus de favoriser l’émergence de bactéries résistantes aux
antibiotiques (de Vries et al., 2011; Jernberg et al., 2010; Sommer & Dantas, 2011). D’ailleurs, le microbiote intestinal humain comporte une grande quantité de bactéries qui sont considérées comme un réservoir de gènes de résistance aux antibiotiques (de Vries et al., 2011; Fouhy et al., 2014; Hu et al., 2013; Raymond et al., 2016; Tian et al., 2012). Le microbiote intestinal devient par le fait même un environnement propice aux échanges de gènes de résistance entre bactéries, incluant les pathogènes (Karami et al., 2007). L’étude des gènes de résistance présents dans le microbiote intestinal humain est donc très importante afin de mieux comprendre le phénomène de résistance aux antibiotiques.
Figure 1.7. Facteurs influençant le microbiote intestinal humain. Figure adaptée de
24
1.6. Approches d’analyse des gènes de résistance aux
antibiotiques dans le microbiote intestinal
humain
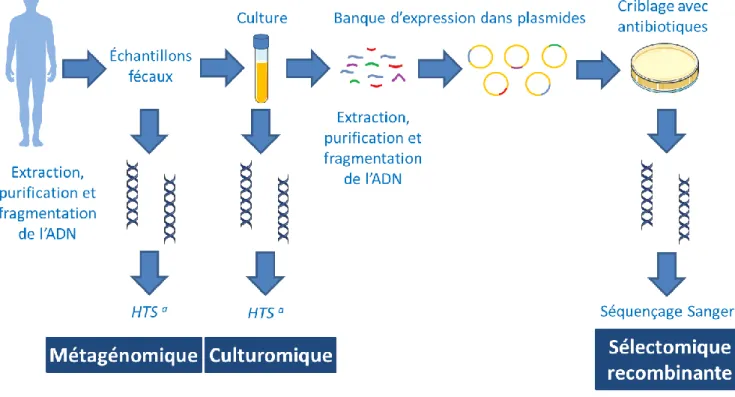
Trois approches peuvent être utilisées pour analyser les gènes de résistance chez les bactéries du microbiote intestinal humain (résistome) : la métagénomique, la culturomique et la sélectomique recombinante. Ces trois approches ont été utilisées dans ce projet de maîtrise et sont expliquées plus en détails ici. La métagénomique est une approche indépendante de la culture des bactéries, tandis que la culturomique et la méthode de sélectomique recombinante utilisée dans ce mémoire en sont dépendantes (Figure 1.8) (Morgan & Huttenhower, 2012).
Figure 1.8. Trois approches utilisées pour caractériser le résistome intestinal humain. a