Étude de l’intégration chromosomique de l’herpèsvirus
humain de type 6 et impact de son infection sur la
reconnaissance des dommages aux télomères
Mémoire
Shella Gilbert-Girard
Maîtrise en microbiologie-immunologie
Maître ès sciences (M.Sc.)
Québec, Canada
© Shella Gilbert-Girard, 2016
Étude de l’intégration chromosomique de l’herpèsvirus
humain de type 6 et impact de son infection sur la
reconnaissance des dommages aux télomères
Mémoire
Shella Gilbert-Girard
Sous la direction de :
iii
Résumé
L’herpèsvirus humain de type 6B (HHV-6B) infecte près de 100% des humains et établit une latence pour le reste de la vie de l’hôte. L’épidémiologie d’HHV-6A est moins connue. Ces deux virus sont capables d’intégrer leur génome dans les télomères des chromosomes humains. Lorsque cette intégration a lieu dans une cellule germinale et qu’il y a fécondation, ceci mène à un individu portant une copie du génome viral dans chacune des cellules de son corps. Cette condition, appelée inherited chromosomally-integrated HHV-6 (iciHHV-6), résulte en une transmission du génome viral intégré à 50% des descendants et est retrouvée chez environ 1% des humains. Malgré cette importante fréquence dans la population, l’intégration de HHV-6, de même que son infection et son effet sur ses cellules hôtes, demeurent peu caractérisés. Dans ce travail, nous avons étudié l’effet de l’infection d’HHV-6A et HHV-6B sur la réponse de dommages à l’ADN (DDR). Nous avons observé une forte réponse DDR localisée dans les compartiments de la réplication virale (RC), colocalisant avec une grande quantité d’ADN télomérique d’origine virale. De plus, les niveaux des ARNm des gènes TRF1, TRF2 et TPP1, membres du complexe shelterin protégeant les télomères, étaient surexprimés dans les cellules infectées. Cette augmentation se traduit par une surexpression de la protéine TRF2, recrutée aux séquences télomériques virales dans les RC. Finalement, nous avons étudié l’effet de BRACO-19, un inhibiteur de la télomérase, sur l’intégration d’HHV-6A et sa persistance. En utilisant un essai d’intégration récemment mis au point dans notre laboratoire, nous avons démontré que l’inhibition de la télomérase menait à une diminution significative de la fréquence de cellules porteuses d’intégration. Ce travail apporte de nouvelles connaissances sur l’infection de HHV-6 et sur son mécanisme d’intégration potentiel, de même que la première observation d’une possible participation des protéines du complexe shelterin dans l’infection de HHV-6.
iv
Abstract
Human herpesviruse 6B (HHV-6B) is a very prevalent virus that infect nearly 100% of humans and establish a life-long latency. Much less is known regarding HHV-6A epidemiology. Both viruses can integrate their genome into the telomeres of human chromosomes. When this integration occurs in a germinal cell, it can lead to an individual carrying one copy of the viral genome in every cell of its body. This condition, called inherited chromosomally-integrated HHV-6 (iciHHV-6), will then be transmitted to 50% of offspring and is found in approximately 1% of individuals across the world. Despite being so frequent, much remains to be studied on HHV-6 infection and integration processes. In this work, we studied the effects of viral infection on DNA damage response (DDR) signaling. We observed a DDR located in viral replication compartments (RC), together with a large amount of telomeric sequences that we confirmed to be of viral origin. In addition, mRNAs coding for TRF1, TRF2 and TPP1, members of the shelterin complex protecting telomeres, were upregulated during infection. Consequently, TRF2 protein was overexpressed and relocated to viral telomeric sequences in RC. Lastly, we’ve examined the effects of BRACO-19, a compound that affects telomerase activity, on HHV-6 integration and persistence. Using an integration assay recently developed in our laboratory, we could demonstrate that in the presence of the telomerase inhibitor, the frequency of cells containing integrated HHV-6 was significantly reduced. This work brings new knowledge regarding HHV-6 infection and its potential integration mechanism, as well as the first observation of a possible participation of the shelterin complex proteins during HHV-6 infection.
v
Table des matières
Résumé ... iii
Abstract ... iv
Table des matières ... v
Liste des tableaux ... vii
Liste des figures ... viii
Liste des abréviations ... ix
Remerciements ... xi
Avant-Propos ... xii
Chapitre 1 : Introduction ... 1
1.1 L’herpèsvirus humain de type 6 ... 1
1.1.1 Origine et classification ... 1
1.1.2 Maladies associées à HHV-6 ... 2
1.1.3 Le cycle des herpèsvirus ... 4
1.1.4 Le génome de HHV-6 ... 7
1.2 L’intégration chromosomique de HHV-6 ... 8
1.2.1 Intégration chromosomique chez les herpèsvirus ... 8
1.2.2 Forme héritée de ciHHV-6 ... 9
1.2.3 Conséquences sur la santé ... 12
1.2.4 Réactivation du virus intégré ... 15
1.2.5 Mécanisme d’intégration ... 15
1.3 Les télomères et l’entretien des chromosomes ... 19
1.3.1 La structure des télomères ... 19
1.3.2 Le complexe shelterin ... 20
1.3.3 Les G-quadruplexes et leurs ligands ... 22
1.3.4 La télomérase et ALT ... 24
1.3.5 La réparation des bris d’ADN ... 25
1.3.6 Virus et détection de dommages à l’ADN ... 29
Chapitre 2 : Hypothèses et objectifs des travaux ... 30
Chapitre 3 : L’infection de HHV-6 déclenche une réponse de dommage à l’ADN et recrute TRF2 aux télomères viraux ... 31
3.1 Résumé ... 31
3.2 Abstract ... 33
3.3 Introduction ... 34
3.4 Materials and Methods ... 36
3.5 Results ... 41 3.6 Discussion... 45 3.7 Acknowledgments ... 48 3.8 Funding ... 48 3.9 Author Contributions ... 48 3.10 Bibliography ... 49 3.11 Figures ... 52 3.12 Supplementary Data ... 64
Chapitre 4 : Activité de la télomérase et intégration chromosomique de HHV-6 ... 65
4.1 Résumé ... 65
4.2 Abstract ... 67
vi
4.4 Materials and Methods ... 70
4.5 Results ... 74 4.6 Discussion... 77 4.7 Acknowledgments ... 79 4.8 Author contributions ... 79 4.9 Bibliography ... 80 4.10 Figures ... 83 Chapitre 5 : Discussion ... 90 5.1 DDR et TRF2 dans les RC de HHV-6 ... 90
5.2 Inhibition de la télomérase et intégration de HHV-6 ... 92
Chapitre 6 : Conclusion ... 95
vii
Liste des tableaux
Chapitre 1
Tableau 1 : Recensement des cas de iciHHV-6 publiés dans la littérature. ... 12
Chapitre 4
Table 1 : Effects of BRACO-19 (B19) on HHV-6A integration frequency in Hela and U2OS cells ... 87
Table 2 : Effects of BRACO-19 (B19) on chromosomal integration of HHV-6 into Hela, MCF-7 and U2OS cells ... 89
viii
Liste des figures
Chapitre 1
Figure 1 : Arbre phylogénétique de la famille des herpèsvirus humains. ... 2
Figure 2 : Schématisation du cycle lytique de HHV-6. ... 6
Figure 3 : Représentation schématique du génome de HHV-6. ... 8
Figure 4 : Intégration de HHV-6 dans les chromosomes de cellules somatiques et germinales. ... 11
Figure 5 : Représentation des risques potentiels associés à iciHHV-6. ... 14
Figure 6 : Schéma du génome de HHV-6 intégré dans un chromosome cellulaire. ... 16
Figure 7 : Schématisation de l’intégration chromosomique de HHV-6 dans un chromosome par recombinaison homologue. ... 17
Figure 8 : Télomère et complexe shelterin. ... 21
Figure 9 : Structure d’un G4 et interaction avec le BRACO-19. ... 23
Figure 10 : Modèle de réparation d’un bris d’ADN double-brin par NHEJ et HR. ... 28
Chapitre 3
Figure 1 : Activation of the DDR during HHV-6A infection of U2OS cells ... 52Figure 2 : U2OS cells infected with HHV-6A (U1102 strain) ... 53
Figure 3 : Colocalization of viral proteins, 53BP1 and telomeric signals ... 54
Figure 4 : Detection of viral temoleres using telomeric probe ... 55
Figure 5 : Localization of TRF2 at viral replication compartments ... 56
Figure 6 : TRF2 binds to viral DNA ... 57
Figure 7 : MBP-TRF2 binds telomeric motifs ... 59
Figure 8 : MBP-TRF2 bind viral TMR ... 60
Figure 9 : Expression shelterin complex mRNAs in uninfected and HHV-6A-infected HSB2 cells ... 61
Figure 10 : TRF2 expression during HHV-6A infection ... 62
Figure 11 : Increased TRF2 expression in HHV-6A-infected U2OS cells ... 63
Supplementary figure 1 : MBP-TRF2 does not bind non-telomeric motifs ... 64
Chapitre 4
Figure 1 : Binding of BG4 antibody to various DNA oligonucleotides. ... 83Figure 2 : Association and dissociation curves of ligands (BG4 and BRACO-19) to the DNA oligos by surface plasmon resonance. ... 84
Figure 3 : Growth of HHV-6B-infected PBMCs (A) and HHV-6B-infected MOLT-3 cells (B) in the presence of BRACO-19. ... 85
Figure 4 : Effect of BRACO-19 on HHV-6B (Z29 strain) infection of Molt-3 cells (A) and PMBC (B). . 86
ix
Liste des abréviations
53BP1 p53-binding protein 1
A Adénine
AAV (-2) Adeno-associated virus (-2)
ADN Acide désoxyribonucléique
ALT Alternative lengthening of telomeres
ARNm Acide ribonucléique messager
ATM Ataxia telangiectasia mutated
ATP Adénosine Triphosphate
ATR Ataxia telangiectasia and Rad3 related
BIR Break-induced repair
C Cytosine CD46/134 Cluster of Differenciation 46/134
ChIP Chromatin immunoprecipitation
ciHHV-6 chromosomally integrated HHV-6
CSE Cellules souches embryonnaires
CSH Cellules souches hématopoïétiques
CST CTC1-STN1-TEN1 complex
Ct Cycle threshold
ddPCR digital droplet polymerase chain reaction
DDR DNA damage response
D-Loop Displacement loop
DNA Desoxyribonucleic acid
DNA-Pkcs DNA-dependent protein kinase catalytic subunit
DRL/R Direct Repeat Left/Right
DSB Double-Strand Break
EBV Epstein-Barr Virus
ELISA Enzyme-linked immunosorbent assay
G Guanine G4 G-quadruplex gH/gL/gQ1/gQ2/gp102 Glycoprotéine H/L/Q1/Q2/102
H2AX (γH2AX) Histone H2A, membre X (H2AX phopshorylée)
HBLV Human B lymphotropic virus
HCMV Human Cytomegalovirus
HHV-6A/B (-7, -8) Human Herpesvirus 6A/B (7, 8)
HR Homologous recombination (recombinaison homologue)
HSV-1/2 Herpes Simplex Virus 1/2
iciHHV-6 inherited chromosomally integrated HHV-6
ICP0 Infected-cell protein 0
IE (1/2) Immediate Early (1/2)
impTMR Imperfect telomeric repeats
MBP Maltose-binding protein
MDV Marek’s Disease Virus
MFI Mean fluorescence intensity
x
MRN Mre11-Rad50-NBS1
MTT 3-(4-5-dimethylthiazol-2-yl)-2,5-diphenyltetrazolium bromide
NHEJ Non-Homologous End Joining
ORF Open reading frame
Pac1/2 Cleavage and Packaging elements
PBMC Peripheral Blood Mononuclear Cells
PIKKs Phosphoinositide 3-kinase related protein kinases
PML Promyelocytic leukemia
PNA Peptide nucleic acid
POT1 Protection of telomere 1
qPCR quantitative polymerase chain reaction
RAP1 Repressor-activator protein
RC Replication compartment
RNF8/168 Ring finger protein 8/168
ROS Reactive oxygen species
RPA Replication protein A
RT Reverse transcriptase
SIDA Syndrome d’immunodéficience acquise
SPR Surface Plasmon Resonance
T Thymine
T-circles Telomeric circles
TERC Telomerase RNA component
TERRA Telomeric repeat-containing RNA
TERT Telomerase reverse transcriptase
TIN2 TRF-interacting nuclear protein 2
T-Loop Telomeric loop
TMPyP4 5,10,15,20-tetrakis(N-methyl-4-pyridyl)porphyrin
TMR Telomeric Repeats
TPP1 (initiales de TINT1, PTOP et PIP1)
TRF1/2 Telomeric repeat binding factor 1/2
T-SCE Telomere-Sister chromatid exchange
U (94) Région Unique (gène 94)
UV Ultraviolet
VIH Virus de l’immunodéficience humaine
xi
Remerciements
Je tiens tout d’abord à remercier le Dr Louis Flamand pour m’avoir accueillie dans son laboratoire et pour m’avoir soutenue et encadrée tout au long de mon projet. Sa patience, sa compréhension et son implication en tant que directeur m’ont beaucoup aidé à avancer et à bâtir la confiance nécessaire pour poursuivre mes études.
Je voudrais aussi remercier Annie Gravel et Isabelle Dubuc, non seulement pour m’avoir guidée pas à pas dans le laboratoire et avoir été de précieuses ressources toujours présentes en cas de besoin, mais aussi pour leur personnalité radieuse. Grâce à vous, travailler au laboratoire a toujours été plaisant. Merci aussi à Vanessa Collin pour les conversations et les encouragements qui ont aidé à passer au travers des jours moins faciles. Entre étudiantes, on se comprend!
Merci aussi à toutes les autres personnes travaillant au laboratoire qui m’ont aidé avec une expérience, un appareil ou tout simplement pour avoir rendu le travail plus agréable par votre présence. Un merci particulier à Guillaume Paré, Marc Boisvert, Maria Fernandes et Julie-Christine Lévesque pour leur aide.
Finalement, merci à mes parents pour avoir toujours été présents, même à distance, pour me soutenir. Votre appui inconditionnel est ce qui m’aide à aller de l’avant. Merci aussi à mes sœurs pour votre support et pour me donner deux si bons exemples de personnes qui savent foncer et mordre dans la vie. Je termine avec un gros merci au reste de ma famille et à mes amis pour tout simplement faire partie de mon entourage et m’aider à garder la tête sur les épaules.
xii
Avant-Propos
Le présent projet porte sur l’étude de l’intégration chromosomique de l’herpèsvirus humain de type 6 et sur l’effet de son infection sur les mécanismes de réparation de l’ADN des cellules infectées. Deux articles sont insérés et au chapitre 3 et 4.
Le premier article joint à ce mémoire et correspondant au chapitre 3 traite de l’influence de l’infection de HHV-6 sur la réponse aux dommages à l’ADN de la cellule et sur le complexe protéique shelterin protégeant les télomères cellulaires. Il est question notamment de la protéine TRF2 et de son interaction avec l’ADN viral. Cet article est présentement en rédaction finale en vue de publication.
Le deuxième article présenté au chapitre 4 porte sur l’effet de BRACO-19, une drogue inhibitrice de la télomérase, sur l’intégration chromosomique de HHV-6 et le maintien de cette intégration dans différentes lignées cellulaires. Cet article a été soumis au Journal of Virology.
Louis Flamand a conçu et dirigé le projet et les expériences pour les deux articles. Il a participé à l’interprétation des résultats et réalisé les analyses statistiques. Il a également corrigé les articles et participé à leur rédaction. Annie Gravel a réalisé certaines des manipulations présentées dans les deux articles et elle a participé à l’interprétation des résultats de la majorité des expériences. Nina Wallasheck et Benedikt Kaufer ont réalisé le FISH sur les cellules iciHHV-6 dans le deuxième article au chapitre 4. Sara Artusi, dirigée par Sara Richter, a effectué les tests de toxicité de BRACO-19 sur plusieurs lignées cellulaires et a réalisé les expériences sur l’effet de cette drogue sur l’ADN intracellulaire de HHV-6 dans les cellules infectées. Ces résultats sont également présentés dans le deuxième article.
J’ai écrit les manuscrits des deux articles et j’ai fait la mise au point et la réalisation de la majorité des manipulations dont il y est question dans chacun d’eux. J’ai analysé et interprété les résultats et effectué certaines des analyses statistiques.
1
Chapitre 1 : Introduction
1.1 L’herpèsvirus humain de type 6
1.1.1 Origine et classificationLes herpèsvirus forment une grande famille de virus enveloppés à ADN double-brin divisée en trois sous-groupes. Les alphaherpèsvirus, comprenant le virus de l’herpès simplex (Figure 1), infectent une grande variété de cellules et s’établissent en latence dans les neurones. Les gammaherpèsvirus, qui comptent le virus Epstein-Barr (EBV), demeurent en latence dans les lymphocytes, alors que les betaherpèsvirus persistent dans les macrophages et les lymphocytes. Ces deux dernières sous-familles ont une réplication plus lente que les alphaherpèsvirus [1]. L’herpèsvirus humain de type 6 (HHV-6) appartient à la sous-famille des betaherpèsvirus, avec HHV-7 et le cytomégalovirus humain (HCMV). Il existe deux espèces différentes de HHV-6, soit HHV-6A et HHV-6B, qui ont d’abord été considérées comme formant une même espèce. En 2012, ces deux virus ont été reconnus comme appartenant à deux espèces distinctes sur la base de leurs différences biologiques, épidémiologiques et immunologiques par l’International Committee on Taxonomy of Viruses [2]. Avec HHV-7, HHV-6A et B sont les seuls représentants du genre des roséolovirus. HHV-6A a d’abord été découvert en 1986, lorsqu’il a été isolé de cellules mononucléaires du sang périphérique (PBMC) de patients atteints du syndrome d’immunodéficience acquise (SIDA) et de désordres lymphoprolifératifs [3]. Il a tout d’abord été nommé HBLV pour Human B lymphotropic virus. Par la suite, de nombreux spécimens de HHV-6 ont été isolés dans différentes régions à travers le monde et toutes les souches retrouvées ont pu être ensuite classées en deux groupes différents, soit A et B [4-7]. Les deux virus sont lymphotropiques, bien qu’ils infectent également d’autres types cellulaires et présentent un tropisme différent pour les lignées de lymphocytes in vitro et une réactivité distincte aux anticorps monoclonaux [2]. L’ADN de ces deux virus contient également des sites de restriction aux endonucléases différents et, puisqu’il n’y a eu aucune observation de recombinaison entre les deux virus, ils semblent occuper des niches différentes in vivo [2, 8].
2
Figure 1 : Arbre phylogénétique de la famille des herpèsvirus humains.
Cette famille comprend neuf virus répartis en trois sous-familles. Les gammaherpèsvirus incluent le virus Epstein-Barr (EBV) et l’herpèsvirus de type 8, responsable du sarcome de Kaposi. Les alphaherpèsvirus comprennent le virus de la varicelle/zona (VZV) et les virus de l’herpès simplex de type 1 et 2 (HSV-1 et -2). La sous-famille des betaherpèsvirus est formée du cytomégalovirus (HCMV), de l’herpèsvirus de type 7 (HHV-7) et des herpèsvirus de type 6A et B (HHV-6A et -6B). Inspirée de [9, 10].
1.1.2 Maladies associées à HHV-6
HHV-6B est l’agent étiologique de la roséole (roseola infantum ou exanthem subitum), aussi connue sous le nom de sixième maladie [7, 11, 12]. Il s’agit d’une maladie infantile caractérisée par une haute fièvre durant quelques jours, parfois suivie d’éruptions cutanées maculopapulaires situées principalement sur le thorax. Cette maladie passe souvent inaperçue car elle est asymptomatique dans plusieurs cas. La roséole est généralement bénigne, mais peut mener occasionnellement à des complications telles que des crises d’épilepsie, de la détresse respiratoire, des encéphalites, des otites, etc [8, 13]. Elle est responsable de 10 à 45% des admissions aux urgences d’enfants avec une maladie fébrile et de 1% des hospitalisations d’enfants [13]. Le fait que HHV-6B peut affecter ainsi différents systèmes suggère que ce virus peut atteindre plusieurs organes et possiblement y rester en latence.
Famille des Herpesviridae humains
Alphaherpèsvirus Gammaherpèsvirus Betaherpèsvirus HCMV HHV-7 HHV-6B HHV-6A EBV HHV-8 VZV HSV-1 HSV-2
3
Il s’agit d’un virus ubiquitaire et plus de 90% de la population mondiale est infectée et contracte la roséole dans les deux premières années de vie, lorsque la protection offerte par les anticorps maternels s’est atténuée [8, 14].
Suite à l’infection primaire, le virus demeure en latence toute la vie de l’hôte, entre autres dans les lymphocytes, les monocytes et probablement divers organes et cette latence est interrompue périodiquement par des réactivations [8, 15]. Lors de ces réactivations, HHV-6B se réplique dans les glandes salivaires et est sécrété dans la salive, moyen par lequel le virus se transfère vraisemblablement des porteurs sains aux enfants [16-18]. Les individus en santé ne sont pas affectés par les réactivations du virus, car elles sont contrôlées par le système immunitaire. Cependant, HHV-6B peut également se réactiver chez des individus immunodéprimés, suite à une greffe ou chez des patients atteints du SIDA par exemple. Le virus se réactive dans 20 à 30% des cas de greffe d’organe solide et il se réactive très fréquemment, dans 30 à 50% des cas, lors de greffes de cellules souches hématopoïétiques (CSH) [19, 20]. La réactivation du virus a lieu généralement dans les deux à quatre semaines suivant l’opération et elle est associée à de nombreuses complications dont des rejets de greffe, des réactions de greffon contre l’hôte, des réactivations du HCMV, des encéphalites pouvant être mortelles, des hépatites, une mortalité plus élevée suite à la greffe, etc. [21-26]. Les greffes de sang de cordon sont particulièrement sujettes à une réactivation de HHV-6B et aux complications qui s’en suivent [24]. Les cas de greffe de CSH de cordons ombilicaux suivis d’une infection de HHV-6 peuvent présenter un taux de 10% d’encéphalite limbique, caractérisée par une forte morbidité et mortalité [27].
Malgré une haute fréquence de réactivation et les nombreuses complications qu’engendre le virus, il n’existe pas de médicament spécifique ou de traitement standard adapté à HHV-6B ni aucun des roséolovirus. L’absence de preuve directe d’association cause à effet entre HHV-6 et les diverses maladies auxquelles il a été associé n’encourage pas le développement de nouvelles thérapies ou l’étude en profondeur de l’efficacité des drogues déjà en circulation. Les cas de réactivation de HHV-6B sont normalement traités avec des antiviraux déjà très utilisés tels que ganciclovir. Cette drogue démontre une relativement bonne efficacité contre HHV-6B, mais des cas de virus développant une résistance ont été observés [28, 29]. Des traitements au foscarnet, utilisé contre le HCMV dans les cas de résistance au ganciclovir, ont aussi été tentés, parfois en combinaison avec d’autres antiviraux. Ce
4
médicament fait preuve d’une bonne efficacité, bien qu’il cause certains effets secondaires en limitant l’utilisation [19, 30, 31]. L’usage d’autres drogues est envisagé, mais ces dernières sont d’abord synthétisées dans le but de neutraliser un autre virus comme le HCMV. À ce jour, aucun traitement antiviral ne vise spécifiquement HHV-6 et la création de drogues à large spectre demeure la meilleure option pour combattre ce virus.
Les complications liées à HHV-6A sont nettement moins connues. Il semblerait que l’infection primaire est asymptomatique et que le virus est acquis plus tard dans la vie dans la majeure partie du monde [32], à l’exception de l’Afrique subsaharienne, où HHV-6A infecte plus fréquemment les enfants que HHV-6B [33]. HHV-6A n’est habituellement pas retrouvé chez les enfants atteints de la roséole [12, 34] et il ne se réplique pas dans les glandes salivaires. Il n’est donc probablement pas transmis de la même façon que HHV-6B. Une association a été faite entre HHV-6A et un syndrome fébrile semblable à la roséole, parfois accompagné d’autres symptômes, en République de la Zambie [35]. Des études ont aussi démontré que HHV-6A pourrait être impliqué dans la thyroïdite de Hashimoto, de même que dans la sclérose en plaques [36, 37]. Il pourrait également être un facteur aggravant dans les infections du virus de l’immunodéficience humaine (VIH) [38, 39]. HHV-6A a été détecté dans divers organes chez des individus sains et malades sans que des associations certaines ne soient établies entre le virus et des maladies. Il parait régulièrement impliqué dans divers problèmes de santé, mais aucun lien sûr de cause à effet n’a été établi. Cependant, bien qu’il se réactive moins souvent que HHV-6B suite à une greffe, HHV-6A semble être retrouvé plus fréquemment dans le système nerveux de patients atteints de maladies nerveuses post-greffes [2]. Ainsi, ce virus pourrait bien être un agent causatif, ou du moins participatif, dans plusieurs maladies, en particulier celles touchant le système nerveux, mais les preuves de telles associations sont encore incomplètes.
1.1.3 Le cycle des herpèsvirus
Le cycle de vie des herpèsvirus comporte deux stades distincts. Tout d’abord la phase productive ou lytique, au cours de laquelle l’ADN du virus est répliqué et des virions sont produits. Le virus se lie à la membrane d’une cellule cible par interactions avec des protéines de surface spécifiques et des récepteurs cellulaires (Figure 2). HHV-6A reconnaît la protéine cellulaire CD46, retrouvée sur toutes les cellules nucléées, alors que HHV-6B se lie plutôt à CD134, un récepteur des lymphocytes T [40,
5
41]. Les deux virus se lient aux récepteurs cellulaires par l’intermédiaire d’un complexe de glycoprotéines, gH-gL-gQ1-gQ2, qui comporte quelques différences entre les deux virus, en particulier en ce qui concerne les protéines gQ1 et gQ2 [42, 43]. L’enveloppe de l’herpèsvirus fusionne ensuite avec la membrane de la cellule ou entre par endocytose. Le tégument, une région formée de protéines et remplissant l’espace entre l’enveloppe et la capside du virus, se dissout, libérant la capside [44]. Celle-ci se rend jusqu’au noyau à l’aide des microtubules et du moteur moléculaire de dynéine du cytoplasme de la cellule [45]. Elle injecte par un pore nucléaire l’ADN viral, qui sera transcrit à l’aide de la machinerie de transcription de la cellule [32].
Au cours de cette phase, les herpèsvirus expriment différents gènes de façon séquentielle pour réguler les activités de la cellule et leur propre réplication. Les gènes viraux peuvent être divisés en trois catégories dépendamment du temps auquel ils sont exprimés au cours de l’infection. Les gènes précoces immédiats (IE pour Immediate Early) sont exprimés en premier, au tout début de l’infection. Ce sont des gènes codant pour des protéines régulatrices contrôlant, entre autres, la transcription des gènes précoces (Early). Ces derniers sont nécessaires pour la réplication de l’ADN viral, l’accumulation d’ARN messagers (ARNm) dans le cytoplasme et l’expression des gènes tardifs (Late). Ceux-ci sont régulés différemment des autres gènes et dépendent de la réplication de l’ADN viral [1]. Nombre d’entre eux codent pour des glycoprotéines qui formeront l’enveloppe du virus. Les herpèsvirus utilisent à la fois leurs facteurs viraux et des facteurs cellulaires pour compléter leur cycle. La transcription est assurée par la polymérase à ARN II cellulaire, mais les herpèsvirus possèdent leur propre machinerie de réplication d’ADN [1, 46]. Ainsi, HHV-6 contient des gènes codant pour une polymérase à ADN, des hélicases et des primases, une protéine de liaison à l’origine de réplication et une autre de liaison à l’ADN simple brin, des protéines de réparation de l’ADN, etc [47]. La réplication du génome du virus a lieu dans des compartiments de réplication virale (RC) se formant à travers le noyau de la cellule. Dans le cas de HHV-6, l’ADN viral est répliqué par la polymérase virale de façon circulaire en continu, par un mécanisme dit « rolling-circle », et le produit résultant est un concatémère, une longue molécule d’ADN double-brin contenant plusieurs copies du génome. Le concatémère est ensuite clivé en plusieurs segments contenant chacun le génome complet du virus et ces derniers sont répartis dans des capsides [32, 48]. Les capsides traversent ensuite la membrane nucléaire interne, puis externe, par bourgeonnement, ce qui leur fait acquérir temporairement une membrane sans glycoprotéines qu’elles perdent aussitôt en sortant du noyau [32]. Les protéines du tégument s’assemblent autour des
6
capsides au cours de leur transport jusqu’au site d’enveloppement où les protéines de l’enveloppe sont présentes, mais le site exact de la tégumentation n’est pas connu [45, 49]. L’enveloppe, contenant les glycoprotéines nécessaires à l’attachement du virus à ses cellules cibles, est assemblée autour des capsides et les virions matures sont complétés, puis relâchés dans l’espace extracellulaire. Certains virus sortent par exocytose, bourgeonnement ou, comme HHV-6 dans ses cellules de prédilection, par lyse de la cellule, ce qui libère tous les virions [50].
Figure 2 : Schématisation du cycle lytique de HHV-6.
Les étapes du cycle sont expliquées en détails dans la section 1.1.3 ci-dessus. Les lettres IE, E et L représentent les gènes Immediate Early, Early et Late, respectivement. Inspirée de [32, 49].
La seconde phase du cycle est la latence, pendant laquelle il n’y a pas production de virus. Les herpèsvirus sont bien connus pour persister en latence dans certaines cellules de l’hôte pour le reste de la vie de ce dernier. Lors de la phase de latence, ces virus se maintiennent normalement sous forme d’épisomes circulaires et extrachromosomiques dans les noyaux des cellules hôtes. À ce stade, l’expression des gènes viraux est minimale, permettant au virus de passer inaperçu. Les épisomes
7
sont maintenus par la machinerie de réplication de la cellule [32]. In vivo, HHV-6 établit probablement sa latence dans les monocytes et macrophages [15], mais il a été montré qu’il peut entrer en latence dans une variété de lignées cellulaires telles que des cellules souches hématopoïétiques [51], des cellules myéloïdes [52], des astrocytes [53] et des oligodendrocytes [54], et il ne s’agit fort probablement que d’une liste partielle. Les réservoirs in vivo du virus n’ont pas encore été complètement définis. En latence, HHV-6 n’exprime pas les transcrits spécifiques au cycle lytique, mais des transcrits de latence sont néanmoins exprimés, provenant par exemple des gènes IE1/IE2, leur ARNm étant alors épicés spécifiquement pour la latence [55]. L’expression du gène U94 a aussi été observée et il pourrait être impliqué dans l’établissement ou le maintien de la latence, car il inhibe l’expression virale pendant cette phase [56-58].
1.1.4 Le génome de HHV-6
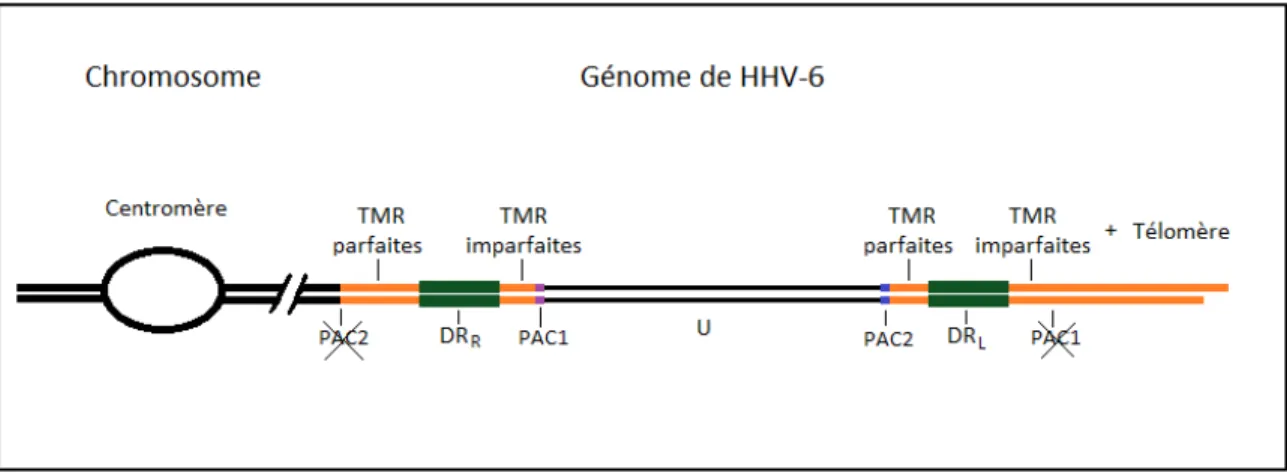
Les herpèsvirus ont un large génome de 120kb à 250kb. HHV-6 possède un génome d’une longueur d’environ 159 kpb pour HHV-6A et environ 162 kpb pour HHV-6B, avec de légères divergences entre les différentes souches [8, 59]. Au centre du génome se trouve une séquence unique (U) d’approximativement 145 kpb, contenant l’origine de réplication du génome, flanquée par deux séquences identiques de répétitions directes (DRL et DRR) d’environ 9 kpb chacune (Figure 3) [47,
59-61]. La composition de ces régions en guanines et cytosines varie entre 40% dans U et 60% dans les DR. Les extrémités de chaque DR se terminent par les séquences pac1 (56 pb) et pac2 (80 pb) impliquées dans le clivage et l’empaquetage du génome du virus pendant l’infection [47, 48]. Dans les deux DR, pac2 est adjacent à une série de répétitions parfaites du motif TTAGGG (TMR pour telomeric repeats), qui est typiquement retrouvé à la fin des chromosomes humains dans la région nommée télomère. Quelques-unes de ces répétitions télomériques se retrouvent aussi réparties dans la région U [59]. De l’autre côté des DR, attenant à pac1, se trouve une autre série de répétitions télomériques, cette fois entrecoupées d’autres motifs répétés, nommées répétitions télomériques imparfaites (impTMR) [62]. La longueur des TMR de HHV-6 varie selon les souches et est généralement longue de 15 à plus de 180 copies de répétitions télomériques (environ 90pb à 1,1 kpb) [63].Les deux espèces de HHV-6 partagent 90% d’homologie de séquence et certains gènes semblent très conservés, notamment U94 avec plus de 95% d’homologie, mais les deux virus possèdent tout de même des régions de plus grande hétérogénéité, dont le DRL et sa jonction avec U et le gène IE1, qui est la
8
possédant le génome le plus semblable à ceux de HHV-6A et B. HCMV partage aussi plusieurs similarités, surtout dans la région U, mais son génome est plus long et plus riche en guanines et cytosines [47]. HHV-6 a plusieurs glycoprotéines qui sont conservées dans tous les herpèsvirus, dont gH et gL qui jouent un rôle dans l’attachement de HHV-6 à ses cellules cibles [61].
Figure 3 : Représentation schématique du génome de HHV-6.
Il est composé d’une région unique (U) d’environ 145kpb flanquée par deux séquences identiques de répétitions directes (DRL et DRR). Les DRs contiennent chacun deux séquences
d’empaquetage (pac1 et pac2), des séquences de répétitions télomériques parfaites (TMR) et imparfaites (impTMR) et plusieurs cadres de lecture ouverts (ORF) (pas représentés). Tirée de [64].
1.2 L’intégration chromosomique de HHV-6
1.2.1 Intégration chromosomique chez les herpèsvirusL’intégration chromosomique du génome viral est un mécanisme utilisé par plusieurs virus, comme les rétrovirus et les virus adéno-associés (AAV) et qui permet l’entrée en latence. Cependant, ce n’est généralement pas le cas des herpèsvirus, qui établissent normalement une latence en maintenant leur ADN sous la forme d’un épisome dans le noyau de leur cellule hôte (section 1.1.3). Une latence par intégration chromosomique représente donc une façon inhabituelle de procéder pour ce type de virus. Plusieurs observations d’insertions de fragments de génome de certains herpèsvirus dans des
9
chromosomes cellulaires ont été faites, mais il s’agit surtout de fragments provenant de virus défectueux qui ne peuvent mener à la réplication d’un produit viral [65]. Quelques herpèsvirus peuvent toutefois intégrer leur génome entier dans des chromosomes, soit le virus de la maladie de Marek (MDV), EBV et HHV-6. MDV est un alphaherpèsvirus oncogène causant des lymphomes des lymphocytes T chez la volaille. Le génome de ce virus, tout comme celui de HHV-6, comprend des régions répétées contenant des séquences de répétitions télomériques (TTAGGG)n [66]. Suite à une infection, MDV s’intègre souvent et ne semble pas avoir de préférence pour un chromosome en particulier, mais il s’intègre davantage dans les télomères à la fin des chromosomes [67]. Il semblerait que l’intégration chromosomique de ce virus est directement reliée à sa capacité oncogénique. Une fois intégré, le génome de MDV peut être répliqué, ce qui résulte en la formation de nouvelles copies simple brin de l’ADN du virus [68]. EBV, appartient plutôt à la sous-famille des gammaherpèsvirus et il s’agit d’un virus oncogène ubiquitaire dont l’infection primaire, asymptomatique chez les enfants, cause la mononucléose chez les jeunes adultes. Il infecte les lymphocytes B et les cellules épithéliales et est également impliqué, entre autres, dans le développement des lymphomes de Burkitt et de Hodgkin, de même que dans le développement du cancer du nasopharynx [69]. EBV peut intégrer son génome dans différents sites des chromosomes, souvent, mais pas exclusivement, dans les régions contenant plusieurs répétitions, riches en guanines et ne codant pour aucun gène. Il a été observé que l’intégration ne se fait pas au hasard et semble se trouver dans certaines sections précises [70]. Pour EBV, tout comme pour MDV, l’intégration chromosomique est responsable du pouvoir carcinogène du virus. Une fois intégré, il semble que le virus ne puisse pas se réactiver, bien qu’il y ait expression de certains gènes [65].
1.2.2 Forme héritée de ciHHV-6
L’intégration chromosomique de HHV-6 (ciHHV-6) est nettement plus fréquente que celles de MDV et EBV. Elle a été observée pour la première fois dans le génome de PBMC par Luppi et al [71, 72]. De nombreuses autres observations de telles intégrations ont ensuite été faites, confirmant la récurrence du phénomène [73-75]. Depuis, cette intégration, possible pour HHV-6A et B, a été observée chez plusieurs familles portant le génome entier du virus dans l’un de leurs chromosomes et le transmettant de génération en génération [76, 77]. Il a été démontré in vitro que HHV-6 peut s’intégrer dans de nombreuses lignées cellulaires. Il est donc probable qu’à la suite d’une infection lytique chez l’humain,
10
quelques cellules infectées portent l’intégration du virus dans l’un de leurs chromosomes. Lorsque cette intégration chromosomique a lieu dans une cellule germinale (Figure 4), elle peut être transmise au descendant, qui porte alors une copie du génome du virus dans chacune des cellules de son corps et peut le transmettre comme l’un de ses propres gènes à 50% de ses descendants, selon la loi de Mendel [78, 79]. D’ailleurs, il a été observé que HHV-6 est le virus le plus souvent présent dans le sperme de donneurs sains et qu’il peut s’attacher à l’acrosome des spermatozoïdes, ce qui pourrait constituer la voie d’entrée du virus dans un ovule, pour ensuite s’y intégrer [80]. Cette intégration chromosomique transmise de parent à enfant est nommée « inherited chromosomally integrated HHV-6 » (iciHHV-HHV-6). Approximativement 1% de la population mondiale est iciHHV-HHV-6+, ce qui représente plus de 70 millions d’individus [65, 81]. Le virus le plus fréquemment retrouvé dans le génome des humains est iciHHV-6B, qui représente environ 75% des cas d’intégration chromosomique de HHV-6, alors qu’iciHHV-6A est retrouvé dans 25% des cas, un taux assez élevé considérant la fréquence à laquelle il est retrouvé en infection active dans les humains [81]. Les deux virus peuvent s’intégrer dans différents chromosomes (Tableau 1), mais l’intégration a toujours lieu dans une région située aux extrémités des chromosomes nommée le télomère (section 1.3.1) [75].
11
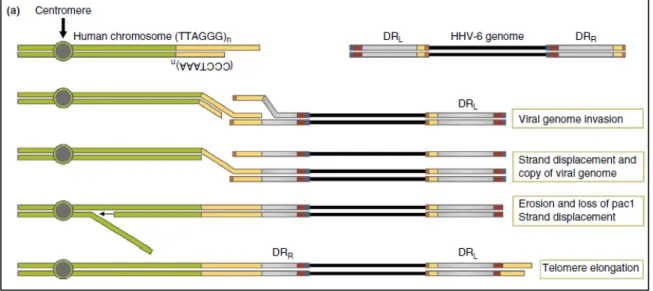
Figure 4 : Intégration de HHV-6 dans les chromosomes de cellules somatiques et germinales.
Section supérieure : Au cours de l’infection primaire de HHV-6, le virus infecte des cellules somatiques (lymphocytes, monocytes, etc.) où il intègre son génome et demeure ainsi en latence pour le reste de la vie de l’hôte. Il n’y a pas transmission du virus intégré aux descendants. Section inférieure : HHV-6 peut infecter des cellules germinales (spermatozoïde ou ovule) et s’y intégrer. L’intégration est alors transmise à 50% des descendants. Tirée de [64].
12
Espèce Nombre de cas/ localisation chromosomique Références
A 1/10q26.3 [75] 2/17p13.3 [75, 77, 82, 83] 1/18q23 [77] 29/ N/A [76, 79, 84-91] B 1/1q44 [92, 93] 2/9q34.3 [75, 83] 1/11p15.5 [75, 82, 83] 5/17p13.3 [71, 73, 75, 83, 94] 1/18p11.3 [87] 2/19q13.4 [75, 83] 3/22q13 [77, 79] 1/22q13 et 1q44 [74, 78] 51/ N/A [77, 79, 84, 85, 88, 89, 91, 95, 96] N/A 1/1q44 [97] 1/9q [98] 1/22q [99] 2/ N/A [100, 101]
a Les cas d’une même famille sont rapportés comme un événement unique. N/A, non-disponible
Tableau 1 : Recensement des cas de iciHHV-6 publiés dans la littérature.
Cas de iciHHV-6 et sites chromosomiques de l’intégration rapportés dans la littérature en 2010. Traduit de [65].
1.2.3 Conséquences sur la santé
Les individus iciHHV-6+ peuvent aisément être identifiés en mesurant le nombre relatif de copies de génome de HHV-6 par cellule, ce qui peut être fait en utilisant une PCR en temps réel (qPCR) ou une PCR digitale en micro-compartiments (ddPCR) [102, 103]. Les individus ne portant pas iciHHV-6 qui ont été infectés à l’enfance par HHV-6 et conservent toujours le virus en latence dans certaines cellules ont normalement de 60 à 80 copies de l’ADN du virus par million de cellules. Les individus portant iciHHV-6, puisqu’ils ont une copie du génome viral par cellule, ont approximativement un million de copies de HHV-6 par million de cellules [104]. Lorsqu’une telle quantité de copies virales est détectée chez un patient, elle peut être faussement interprétée comme une infection active du virus, pendant laquelle la quantité d’ADN viral en circulation est plus élevée. Cela est particulièrement important lors d’une greffe où le donneur ou le receveur porte iciHHV-6. La détection de l’ADN du virus peut mener à un traitement antiviral non nécessaire pouvant avoir des conséquences néfastes sur le patient, d’où
13
l’importance pour les praticiens de s’assurer de bien différencier une infection lytique de HHV-6 d’une intégration chromosomique lorsqu’un taux élevé de copies de HHV-6 est détecté. Ceci est possible en vérifiant si de l’ADN de HHV-6 est détectable dans les follicules des cheveux par exemple [81, 105] ou en usant de tests plus sensibles pouvant distinguer un cas de iciHHV-6 d’une infection active [106].
Outre la possibilité d’un faux diagnostic d’infection lytique, les conséquences sur la santé des individus portant iciHHV-6 ne sont pas encore bien définies. La condition n’est pas létale et, bien que plusieurs études aient rapporté quelques possibles associations entre iciHHV-6 et certaines pathologies, jusqu’à tout récemment, aucune maladie n’avait été directement associée au virus intégré. Des études récentes suggèrent qu’iciHHV-6 pourrait bel et bien contribuer au développement de certaines maladies. Pellet et al. ont regroupé les résultats de plusieurs études indépendantes et ont observé que les individus iciHHV-6+ étaient plus représentés dans la population malade (souffrant de diverses maladies) que dans la population en santé [81]. En 2015, Flamand et al. ont analysé l’ADN de 20 000 Québécoises et Québécois pour y trouver les individus iciHHV-6+ et vérifier si certaines maladies étaient plus représentées dans cette population. Cette étude a démontré une association entre iciHHV-6 et l’angine de poitrine. En effet, les individus iciHHV-iciHHV-6+ sont trois fois plus à risque de développer la maladie [107].
L’intégration de HHV-6 dans les chromosomes présente plusieurs risques potentiels, dont l’effet de l’intégration elle-même dans un chromosome. L’insertion d’une séquence étrangère dans le génome humain peut avoir des conséquences telles que de l’instabilité chromosomique, l’activation de certains gènes cellulaires ou le rétrécissement du chromosome suite à la résection de l’ADN situé après l’intégration (Figure 5) [65]. Il est possible que la présence du génome du virus dans les cellules n’ait aucun effet, mais, comme mentionné à la section 1.1.3, des transcrits viraux sont exprimés pendant la phase de latence et ont été détectés dans des lignées cellulaires portant ciHHV-6 [55, 56, 58]. L’expression spontanée d’une protéine virale, IE1, a aussi été observée dans certaines cellules ciHHV-6+, malgré l’absence d’infection lytique (Gravel et al, en soumission). La détection de ces transcrits ou protéines par le système immunitaire dans un individu iciHHV-6 pourrait causer une réaction en apparence “auto-immune” contre les cellules exprimant ces antigènes viraux. Dans la situation où les antigènes HHV-6 sont exprimés dans les cellules endothéliales, cette inflammation chronique et à long terme pourrait mener à des troubles du système cardiovasculaire, ce qui pourrait expliquer pourquoi
14
iciHHV-6 serait associé à l’angine de poitrine [107]. Un autre risque potentiel d’iciHHV-6 est bien sûr la possibilité d’une réactivation complète du virus avec production de virion (section 1.2.3).
Figure 5 : Représentation des risques potentiels associés à iciHHV-6.
(1) Aucune transcription de gènes viraux. (2) Expression de gènes viraux, réplication de l’ADN viral et production de virions. (3) Expression de certains gènes de HHV-6. (4 et 5) Impact de l’intégration de HHV-6 sur la fonction et l’architecture des télomères et la stabilité chromosomique. (6) Activation en trans et/ou en cis de gènes cellulaires à la suite de l’intégration. (7) Tolérance immunitaire grâce à l’expression de gènes de HHV-6 pendant l’embryogenèse. (8) Destruction de tissus ou de cellules exprimant des antigènes de HHV-6 (provenant de HHV-6 intégré) par des mécanismes de défense immunitaire développés en réponse à une infection naturelle de HHV-6. Tirée de [65].
15 1.2.4 Réactivation du virus intégré
L’intégration chromosomique de HHV-6 a d’abord été considérée comme un cul-de-sac évolutif ne permettant pas au virus de reprendre son cycle lytique. Suite à l’intégration, le génome du virus perd certaines séquences à ses extrémités normalement utiles à son cycle lytique [77]. La possibilité d’une réactivation de ciHHV-6 était, jusqu’à récemment, un sujet de controverse dans la littérature. Cependant, de plus en plus de preuves s’accumulent montrant que HHV-6 peut s’exciser des chromosomes et commencer un cycle lytique de novo in vitro et in vivo, ce qui indique que cette intégration est bien un moyen d’entrer en latence [77, 108-111]. Une étude en particulier, rapportant un cas de réactivation de iciHHV-6 in vivo chez un enfant sévèrement immunodéprimé atteint d’un déficit immunitaire combiné sévère lié à l’X a offert une preuve très convaincante de la possibilité d’une excision du virus intégré pouvant mener à une infection active [109]. En effet, chez cet enfant, la réactivation du virus intégré a causé une infection lytique avec pathologies qui a dû être traitée à l’aide d’antiviraux [109]
Un possible mécanisme de réactivation de ciHHV-6 impliquerait l’excision du virus de sa forme intégrée par la formation d’un cercle télomérique (T-circle), une molécule d’ADN circulaire qui se détache du chromosome par un événement de recombinaison homologue (HR) [108, 112]. Dans ce cas-ci, la recombinaison aurait lieu entre les DR du virus et les T-circles contiendraient alors le génome complet de HHV-6 avec un seul DR. Ces molécules d’ADN pourraient ensuite être répliquées et des concatémères pourraient être créés et permettre la reprise du cycle lytique [64]. Il ne s’agit toutefois que d’une supposition et le mécanisme précis de l’excision du HHV-6 une fois qu’il est intégré est encore largement inconnu.
1.2.5 Mécanisme d’intégration
Alors même que l’on tente de découvrir comment iciHHV-6 arrive à se réactiver, le mécanisme précis de l’intégration elle-même demeure très peu compris. Plusieurs hypothèses ont été suggérées et l’observation des sites d’intégration a permis de faire certaines déductions. Tel que mentionné plus haut, bien qu’il s’intègre dans plusieurs chromosomes différents, ciHHV-6 n’est retrouvé que dans les télomères, situés à la fin des chromosomes [65, 73-75, 77]. Les sites d’intégration de iciHHV-6 ont été analysés et il a été découvert que la jonction entre le télomère et le génome viral se trouve dans la
16
séquence des TMR parfaites du virus, à l’extrémité de la région du DRR (Figure 6) [77]. La région pac2
semble être perdue pendant l’intégration. De plus, à l’autre extrémité du génome viral, la séquence des TMR imparfaites du DRL est allongée par des répétitions télomériques supplémentaires et la région
pac1 est perdue [112, 113]. Cela suggère que les impTMR à l’extrémité du DRL servent de nouveau
télomère suite à l’intégration et sont prolongées par la télomérase dans les cellules germinales, permettant au chromosome de retrouver un télomère de longueur suffisante (voir section 1.3.4) [114]. Toutefois, il semble que cet ajout de répétitions télomériques ne compense pas la perte du télomère naturel, car il a été observé que le télomère portant l’intégration de HHV-6 est souvent le plus court [112]. Un télomère trop court est synonyme d’instabilité chromosomique pouvant mener à la mort cellulaire (voir section 1.3.1) et cela pourrait constituer une raison pour laquelle iciHHV-6 a un effet négatif sur la santé.
Figure 6 : Schéma du génome de HHV-6 intégré dans un chromosome cellulaire.
La jonction entre le chromosome et l’ADN viral se trouve aux TMR parfaites à l’extrémité du DRR. La séquence de pac2 est perdue lors de l’intégration. La séquence pac1 du DRL est
également perdue et de nouvelles répétitions télomériques sont ajoutées aux TMR imparfaites du DRL. Adaptée de [112].
Avec ces observations faites sur la forme intégrée du virus, un mécanisme en particulier a été reconnu comme très probable pour l’intégration chromosomique de HHV-6. L’orientation et les séquences manquantes du virus intégré sont compatibles avec une intégration par HR entre les TMR du virus et les télomères chromosomiques [77, 112, 115]. La perte de pac2 au niveau du DRR est un signe que
17
l’intégration se fait en aval de ce site, dans les TMR virales. De plus, il a été démontré que ces séquences télomériques dans le génome du virus sont essentielles pour son intégration dans les chromosomes [116], ce qui constitue une preuve en faveur d’une intégration par HR. Les TMR sont d’ailleurs aussi utiles à l’intégration chromosomique dans le cas de MDV. Des TMR sont également retrouvées dans plusieurs autres herpèsvirus (HHV-2 équin, HHV-1 à 3 cyprinide, etc) [117]. Des intégrations chromosomiques dans les télomères n’ont été observées que pour HHV-6A et B et MDV, mais il n’est pas impossible que d’autres virus possédant des TMR soient capables de s’intégrer. Cependant, posséder des TMR à ses extrémités pourrait ne pas être suffisant pour permettre l’intégration chromosomique. HHV-7 possède lui aussi de telles séquences, mais aucune intégration de HHV-7 n’a été observée [118]. Il infecte cependant très peu de cellules comparativement à HHV-6 et il est possible qu’il n’infecte pas les cellules germinales, ce qui rend l’intégration chromosomique plus rare et plus difficile à observer [64].
Figure 7 : Schématisation de l’intégration chromosomique de HHV-6 dans un chromosome par recombinaison homologue.
Celle-ci prend naissance entre le télomère du chromosome et les TMR parfaites du DRR. Le
génome viral et copié et à la division cellulaire suivante, son extrémité distale est érodée et perd la séquence pac1. Les TMR imparfaites du DRL peuvent ensuite être reconnues comme un
18
En regard des preuves disponibles, la HR semble un mécanisme d’intégration probable pour HHV-6, mais cette hypothèse n’indique pas les protéines impliquées et le mécanisme exact permettant la HR. Pour tenter d’expliquer cette recombinaison entre les TMR virales et chromosomiques, plusieurs mécanismes ont été suggérés. Par exemple, l’intégration de HHV-6 pourrait s’effectuer grâce à un mécanisme cellulaire de réparation de l’ADN nommé réplication induite par un bris (BIR). Ce mécanisme permet de réparer les cassures n’ayant qu’une extrémité, à la fourche de réplication par exemple, ou d’allonger les télomères en absence de télomérase (voir section 1.3.5). Si l’extrémité simple-brin 3’ du télomère parvenait à s’insérer dans le génome de HHV-6 au niveau des TMR, BIR pourrait être activée et une copie du génome pourrait être ajoutée au télomère. Suite à cela, comme supposé plus tôt, le génome du virus pourrait être dégradé jusqu’aux TMR qui seraient alors utilisées comme modèle pour ajouter de nouvelles répétitions télomériques [115]. Les protéines nécessaires au processus d’intégration ne sont pas connues, mais dans le cas où l’intégration s’effectuerait par BIR, aucune protéine virale ne serait essentielle. D’autres mécanismes ont aussi été proposés et certaines protéines virales ont été soupçonnées de jouer un rôle dans l’intégration chromosomique de HHV-6, en particulier U94. En effet, il s’agit d’une protéine de HHV-6 qui possède plusieurs caractéristiques pouvant être utiles à un tel processus comme une activité exonucléase et hélicase, une liaison à l’ADN préférentiellement aux séquences télomériques et une capacité d’hydrolyser l’ATP [119]. De plus, la séquence génique codant pour U94 partage 24% d’homologie avec la protéine REP68/78 du parvovirus adéno-associé de type 2 (AAV-2), qui est essentielle pour l’intégration d’AAV-2 dans le chromosome 19 [120-122]. U94 peut également substituer REP68/78 dans un mutant où cette protéine est absente, signifiant qu’U94 a aussi les fonctions nécessaires pour aider à une intégration chromosomique [123]. HHV-6A et HHV-6B sont d’ailleurs les seuls herpèsvirus humains à posséder une telle protéine, autant de raisons qui permettaient de supposer qu’U94 pourrait participer à l’intégration du virus. Toutefois, il a été montré récemment que l’intégration est possible et tout aussi fréquente, même en l’absence de U94, ce qui indique fortement qu’elle n’est pas nécessaire à l’intégration [124]. Aucune autre protéine virale n’a été démontrée nécessaire à l’intégration chromosomique à ce jour. Sachant que HHV-6 peut se réactiver de son état latent intégré et causer une pathologie, il est important de comprendre comment une telle excision est possible et pour cela, caractériser le mécanisme d’intégration est nécessaire.
19
1.3 Les télomères et l’entretien des chromosomes
1.3.1 La structure des télomèresLes chromosomes des vertébrés sont terminés par une région nommée télomère composée d’une série de répétitions TTAGGG qui, bien que très hétérogène, s’étire normalement sur environ 8 à 13 kpb de long chez l’humain [125]. À l’extrémité de cette région se trouve une section simple brin en 3’ d’environ 30 à 500 nucléotides [126-128]. Les télomères jouent un rôle essentiel dans le maintien de l’intégrité chromosomique et dans la détermination du potentiel réplicatif des cellules. En effet, à chaque division cellulaire les extrémités des chromosomes ne sont pas complètement répliquées à cause de la nature semi-conservative de la réplication de l’ADN et de l’incapacité de l’ADN polymérase à répliquer l’ADN de 3’ vers 5’ [129]. Pour éviter que de l’information génétique ne soit perdue et que le chromosome ne devienne instable, chaque fin de chromosome est protégée par un télomère qui sert de zone tampon qui rétrécit à chaque cycle cellulaire jusqu’à l’atteinte d’une longueur critique qui déclenche la sénescence ou l’apoptose de la cellule via les voies ATR (ataxia telangiectasia and Rad3 related) ou ATM (ataxia telangiectasia mutated) [114, 130, 131]. Si le télomère est raccourci au point d’avoir moins de 13 répétitions télomériques, il n’est plus protégé et devient instable [132]. Ce mécanisme permet d’empêcher que les chromosomes deviennent instables ou accumulent trop de mutations en se débarrassant des cellules trop vieilles, c’est-à-dire, lorsque l’un de leurs télomères devient trop court. Ainsi, en absence de mécanismes d’élongation de télomères, les cellules somatiques ont un nombre de divisions limité et leur potentiel de réplication est proportionnel à la longueur de leur télomère le plus court [133, 134]. En effet, c’est le télomère le plus court qui détermine la durée de vie d’une cellule, pas la longueur moyenne de tous les télomères.
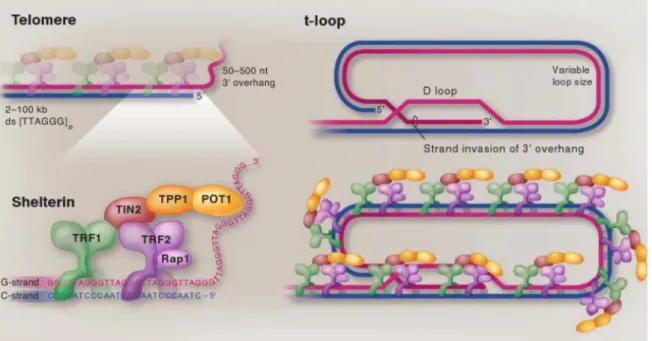
Les télomères jouent également un autre rôle de première importance. Pour éviter que l’extrémité du chromosome soit reconnue comme un bris d’ADN double-brin (DSB), la section simple brin en 3’ se replie et se lie à la portion double brin du télomère pour former la T-Loop (section 1.3.2) [135, 136]. Cette conformation est possible grâce à un complexe protéique, le complexe shelterin, formé de six protéines qui s’attachent aux télomères et en stabilisent les extrémités en plus d’activement réprimer les mécanismes de reconnaissance de dommage à l’ADN [130].
20 1.3.2 Le complexe shelterin
Le complexe shelterin (Figure 8) est composé des protéines TRF1 (Telomeric Repeat binding Factor 1), TRF2 (Telomeric Repeat binding Factor 2), RAP1 (Repressor-activator protein), TIN2 (TRF-interacting nuclear protein 2), POT1 (Protection of Telomere 1) et TPP1 (combiné des noms précédents attribués à cette protéine) [137]. TRF1 et TRF2, qui partagent 30% d’homologie, sont responsables de l’attachement du complexe à la section double brin du télomère [138]. Toutes deux forment des homodimères se liant avec une grande affinité à l’ADN double-brin de façon spécifique à la séquence TTAGGG, mais ne forment pas d’hétérodimères [139-142]. TRF1 est impliquée dans la réplication des répétitions télomériques et évite à la fourche de réplication d’être bloquée [143]. Cette protéine est aussi un régulateur de la longueur des télomères, une fonction qu’elle exécute avec la tankyrase [144]. TRF1 pourrait aussi jouer un rôle dans la répression de la voie ATR en phase S [145]. TRF2 joue un rôle très important dans la protection des télomères contre les mécanismes de réparation de l’ADN en réprimant la jonction d’extrémités non homologues (NHEJ) [146, 147]. Elle réprime également la HR avec l’aide de POT1 et d’un autre complexe, Ku70/80 [130, 148]. TRF2 est également impliquée dans la répression de la voie ATM et de l’arrêt du cycle cellulaire [146, 149]. Cette protéine est de première importance dans le maintien de l’intégrité des chromosomes, qu’elle protège des fusions par les télomères en plus de remplir bien d’autres rôles [150-152]. Tout comme TRF1 et TRF2, POT1 possède aussi la capacité de se lier directement à l’ADN par une reconnaissance spécifique du motif TTAGGG, mais elle se charge plutôt de la liaison avec la section simple brin des télomères [153, 154]. Elle est aussi impliquée dans la protection contre la réponse aux dommages à l’ADN (DDR) et réprime la voie ATR [146, 155]. TPP1 ne s’attache pas directement à l’ADN, elle interagit avec POT1 et relie cette dernière au reste du complexe. Cette liaison avec TPP1 augmente substantiellement l’affinité de POT1 pour l’ADN [137]. Il a été suggéré que ces deux protéines jouent un rôle dans la régulation de la longueur des télomères et agissent comme facteur de processivité de la télomérase [156-159]. TIN2 ne se lie pas non plus à l’ADN, mais elle est importante dans la stabilisation du complexe par ses liaisons avec TRF1, TRF2 et TPP1 et elle aide ces protéines à accomplir leurs rôles respectifs [160-163]. L’absence de TPP1 ou TIN2, qui relient POT1 au reste du complexe, provoque l’activation de la voie ATR [130, 145]. Finalement, RAP1 est très conservée de la levure à l’humain et elle possède plusieurs domaines de liaison à l’ADN et aux protéines et ses fonctions ne se limitent pas aux télomères [164]. Elle n’est pas nécessaire pour la formation de la T-Loop, mais joue un rôle dans la protection
21
des télomères contre la NHEJ et la HR [165, 166]. Elle se lie préférentiellement aux jonctions d’ADN, bien que faiblement, mais n’a pas de reconnaissance d’une séquence spécifique [167].
Figure 8 : Télomère et complexe shelterin.
Trois protéines reconnaissent spécifiquement la séquence télomérique TTAGGG et interagissent directement avec l’ADN : TRF1 et TRF2 dans la séquence double brin, POT1 dans la séquence simple brin. TIN2 et TPP1 relient POT1 à TRF1 et TRF2 et RAP1 est lié au complexe par une interaction avec TRF2. Le complexe est très abondant et recouvre le télomère. Tirée de [130].
Bien entendu, shelterin n’est pas le seul complexe interagissant avec les télomères, sans compter l’ARN non-codant TERRA (telomeric repeat-containing RNA), qui se lie à shelterin et est aussi impliqué dans le maintien de l’intégrité des télomères [152]. Par exemple, le complexe CST (CTC1-STN1-TEN1) se lie aussi à la section simple brin des télomères indépendamment de POT1 et offre une protection supplémentaire en plus d’avoir lui aussi un effet sur l’activité de la télomérase [168, 169]. Ku70/80 est un autre complexe se retrouvant aux télomères par une interaction avec TRF1 [148]. Ce complexe est impliqué dans le mécanisme de réparation NHEJ, mais exerce également un effet protecteur contre la HR aux télomères [148, 170]. Son rôle dans la NHEJ est vraisemblablement inhibé par shelterin [171].
22
Malgré la présence d’autres protéines aux télomères, shelterin est un complexe de première importance en ce qui concerne l’intégrité des télomères et il est tout à fait concevable qu’il soit affecté par un événement tel que l’intégration chromosomique d’un génome viral dans un télomère.
1.3.3 Les G-quadruplexes et leurs ligands
Outre la T-Loop, les télomères peuvent s’organiser en plusieurs conformations, parmi celles-ci les G-quadruplexes (G4). Il s’agit de structures secondaires que les brins d’ADN ou d’ARN peuvent former dans les régions riches en guanines comme certains promoteurs oncogéniques et les télomères [172]. Un G4 est formé de plusieurs quartets planaires de guanines qui s’empilent les uns sur les autres (Figure 9). Les guanines sont reliées entre elles par des ponts hydrogène entre leur face Hoogsteen et Watson-Crick et sont stabilisées par un cation monovalent (ex : potassium, sodium, magnésium) situé au centre du canal formé par les guanines [173, 174]. Un G4 peut se former à partir d’un nombre variable de quartets et ceux-ci peuvent être composés d’un ou de plusieurs brins d’ADN (jusqu’à concurrence de quatre brins), qui peuvent chacun être dans différentes orientations. De plus, les autres nucléotides séparant les guanines peuvent former des boucles aux dimensions et aux propriétés variables et les rainures dans la structure des G4 servant de zones d’interaction avec des ligands peuvent également avoir différentes propriétés. Toutes ces possibilités de structure confèrent aux G4 une grande diversité de conformation [175]. Ces structures ont une influence sur de nombreuses activités dont la régulation des télomères, l’expression de certains gènes et la réplication de l’ADN [176]. En effet, il a été démontré que les G4 peuvent bloquer la fourche de réplication et causer de l’instabilité. Lors de la réplication, la séparation des brins d’ADN donne une opportunité à des G4 de se former. Ils doivent être désassemblés par une hélicase pour permettre le passage de la polymérase [177, 178]. Une possible fonction des G4 serait d’aider à la protection des télomères contre la dégradation, potentiellement en entrant en interaction avec le complexe shelterin, comme le suggère l’observation que RAP1 se lie aux G4 in vitro et en promeut la formation [176, 179]. De plus, les G4 sont très fréquents dans les promoteurs (ex : c-myc), ce qui pourrait indiquer un rôle dans le contrôle de leur expression [180, 181].
23
Figure 9 : Structure d’un G4 et interaction avec le BRACO-19.
(A) : Interaction des guanines et d’un cation dans le quartet d’un G4. (B) : Différentes possibilités d’orientation et de nombre de brins d’ADN dans un G4. Tiré de [181]. (C) : Structure de deux G4 (bleu et rouge) en complexe avec deux molécules du ligand BRACO-19 (jaune) avec la surface des interactions de Van der Waals du ligand visible en jaune. Tirée de [182].
Plusieurs drogues ont été synthétisées dans le but de lier et stabiliser les G4 dans leur conformation pour affecter les activités que les G4 contrôlent. Ces ligands s’attachent aux extrémités des G4, s’intercalent entre les quartets ou se lient aux boucles, aux rainures ou à la structure même des G4, dépendamment de leur nature et de leur conformation, mais ne modifient pas la structure des G4 [183]. Parmi ces drogues, on compte les porphyrines, les anthraquinones, les analogues de l’acridine et plusieurs autres. L’une des porphyrines les plus étudiées est TMPyP4 (5,10,15,20-tetrakis(N-methyl-4-pyridyl)porphyrin), qui se lie aux boucles extérieures ou aux extrémités des G4 dans une proportion de deux ligands pour un G4 [182, 184]. Un autre ligand des G4 bien connu est BRACO-19, qui appartient à la famille des analogues de l’acridine. Il s’agit d’une acridine 3,6,9-tri-substituée créée spécifiquement pour fixer les G4 au niveau des télomères dans le but d’inhiber l’activité de la
24
télomérase dans les cellules cancéreuses [185]. BRACO-19 s’insère entre deux G4 (Figure 9), se liant de façon asymétrique à l’extrémité 3’ d’un G4 et à la tétrade TATA en 5’ du G4 suivant, et ses trois substituants ont été synthétisés pour interagir chacun avec une rainure le long du squelette des G4 [182, 186]. BRACO-19 se lie à ses cibles avec une grande spécificité et a démontré une capacité à inhiber la télomérase dans des cellules cancéreuses, y causant des fusions entre les extrémités des chromosomes [186, 187].
1.3.4 La télomérase et ALT
Comme mentionné à la section 1.3.1, dans la plupart des cellules somatiques, les télomères se raccourcissent à chaque division cellulaire et il en résulte que les cellules ont un nombre limité de divisions possibles [130]. Cependant, il en va autrement des cellules souches embryonnaires (CSE) et des cellules cancéreuses, qui sont dites immortelles car elles ont un potentiel de réplication théoriquement infini. Cela est possible par l’action de la télomérase ou d’un mécanisme d’allongement des télomères alternatif (ALT). La télomérase est une enzyme possédant une sous-unité catalytique de transcriptase inverse (TERT) et une sous-unité d’ARN contenant la séquence répétitive télomérique (TERC) [114]. Elle a d’abord été découverte par Greider et Blackburn, qui ont également proposé son mécanisme par la suite [188, 189]. Elle prolonge le brin 3’ des télomères en y ajoutant des répétitions TTAGGG par son activité de transcriptase inverse. Elle n’a pas besoin d’amorce pour cela grâce à sa sous-unité constituée d’un brin d’ARN guide lui servant de modèle pour sa polymérisation. La machinerie de polymérisation du brin tardif peut ensuite compléter le brin 5’ des télomères en utilisant la nouvelle séquence télomérique comme guide [190]. Grâce à cette enzyme, les cellules souches peuvent rallonger leurs télomères à chaque division cellulaire et conserver ainsi des télomères suffisamment longs pour ne pas déclencher la sénescence ou l’apoptose. Les CSE expriment la télomérase en grande quantité, mais les cellules souches chez les adultes ne l’expriment par suffisamment pour complètement contrecarrer le raccourcissement des télomères, ce qui explique le vieillissement. Les cellules somatiques n’expriment que très peu ou pas la télomérase [191, 192]. Cependant, les cellules peuvent retrouver l’immortalité en réactivant la télomérase au moyen de mutations oncogéniques dans le promoteur de la télomérase. C’est le cas dans environ 90% des cancers. Les cellules cancéreuses qui acquièrent cette modification peuvent se diviser de façon excessive sans que jamais leurs télomères ne deviennent trop courts, entraînant la formation de tumeurs [193].
25
Bien que la plupart des cellules cancéreuses utilisent la télomérase pour maintenir leurs télomères, certaines développent une stratégie différente et se servent d’un mécanisme ALT. De tels mécanismes sont souvent basés sur la HR, qui n’est pas aussi réprimée dans les cellules exprimant ALT que dans les cellules à la télomérase réactivée ou dans les cellules non-immortelles [147, 194]. Des événements de HR sont donc plus fréquents dans les cellules utilisant ALT et mènent à la formation de cercles d’ADN télomérique extra-chromosomiques, suite à la résolution normalement réprimée de la jonction de la T-Loop des télomères par HR [195-197]. Il a été proposé que l’allongement des télomères dans les cellules exprimant ALT pourrait avoir lieu par recombinaison entre les télomères et ces cercles d’ADN télomérique (T-circles) [197], ou encore par HR entre deux télomères de différents chromosomes [198]. Ce mécanisme d’allongement des télomères n’est pas parfaitement bien défini, mais il semble que la HR allongeant les télomères ne se produit pas à chaque division cellulaire, mais lorsqu’un télomère atteint une longueur critique trop courte. Les cercles d’ADN télomérique formés dans les cellules utilisant ALT peuvent avoir plusieurs formes, mais sont majoritairement formés d’ADN double-brin télomérique [194]. En plus de posséder beaucoup de T-circles (qui ne sont toutefois pas uniques aux cellules avec ALT), les cellules utilisant ALT sont reconnaissables par le fait qu’elles n’expriment pas la télomérase et ont des télomères très longs et hétérogènes [196, 199]. Une autre caractéristique de ces cellules est qu’elles ont des corps nucléaires PML (Promyelocytic leukemia) nommés ALT-associated PML bodies qui contiennent les protéines PML, TRF2, TRF1, le facteur de réplication A, Rad51 et Rad52 [200]. Toutefois, la fonction de ces corps n’est pas encore établie et on ignore s’ils sont nécessaires au mécanisme ALT, bien que certaines études penchent en ce sens [194].
1.3.5 La réparation des bris d’ADN
Les mécanismes de la DDR doivent être réprimés aux télomères pour protéger l’intégrité des chromosomes (section 1.3.2). Cependant, ces mécanismes sont très importants dans la protection des chromosomes lorsque survient véritablement un bris à l’ADN. Suite à un dommage à l’ADN, la réparation du bris suit normalement trois étapes, soit la détection du bris, le recrutement des facteurs de réparation au site du dommage et la réparation elle-même [201]. Ces étapes sont principalement régulées par des modifications post-transcriptionnelles. ATM et ATR, deux kinases de la famille des PIKKs (phosphoinositide 3-kinase related protein kinases), sont particulièrement importantes dans la