Étude du rôle de l’ARN Tuna dans le contexte de
pluripotence des cellules souches
Mémoire
Gaultier Héricher
Maîtrise en biologie cellulaire et moléculaire - avec mémoire
Maître ès sciences (M. Sc.)
Étude du rôle de l’ARN Tuna dans le contexte de
pluripotence des cellules souches
Mémoire de Maîtrise ès Science
Biologie Cellulaire et Moléculaire – Faculté de MédecineUniversité Laval. Québec, QC, CANADA
Gaultier HERICHER©
Sous la direction de :
Résumé
Le développement embryonnaire est un processus complexe finement régulé spatio-temporellement par de nombreux acteurs, qu’ils soient ADN, ARN ou protéines. De récents résultats suggèrent un rôle clef dans le développement et les maladies pour une sous-catégorie des ARNs impliqués dans ces mécanismes, les longs ARNs non-codants (lncRNAs). Caractérisés par leur incapacité à produire des protéines, ils sont difficiles à étudier par leur manque de conservation en séquence et leur expression tissu-spécifique. Ici, nous étudions un lncRNA conservé, Tuna (Tcl1 Upstream Neuron-Associated lncRNA), nécessaire au maintien de l’état pluripotent des cellules souches et essentiel à leur différenciation en neurones. Toutefois, les mécanismes dans lesquels cet ARN est impliqué restent inconnus. Pour y répondre, nous avons analysé des données de séquençage d’ARN de différenciation neuronale d’ESCs murines. Nous avons identifié deux nouvelles isoformes spécifiquement exprimées dans les ESCs, sht.Tuna et alt.Tuna. Cette dernière est le résultat de l’épissage alternatif de l’exon 1 de Tuna. Cet épissage alternatif est également observé chez l’humain, démontrant une conservation du traitement de l’ARN entre la souris et l’Homme. Ces deux isoformes sont plus courtes d’environ 1,5kb à l’extrémité 3’ que le transcrit prédit de Tuna (full.Tuna). En effet, l’isoforme full.Tuna n’a pas pu être détectée dans les ESCs, et la surexpression de sht.Tuna a permis d’améliorer la reprogrammation vers l’état pluripotent. Ceci suggère que le rôle de Tuna est mécanistiquement différent dans les ESCs et les neurones. D’autre part, Tuna présente une région hautement conservée (~200bp) contenant un potentiel cadre de lecture pour un peptide de 48 acides-aminés, détectable par surexpression de constructions tagguées FLAG. La mutation du codon AUG de cette séquence codante a abrogé l’effet de l’ARN sur la reprogrammation. Ceci implique un rôle du peptide dans l’acquisition de la pluripotence. Par ailleurs, Tuna a été détecté dans les fractions poly-ribosomales et cytoplasmiques d’ARN, supportant son éventuel potentiel codant. Ensemble, ces résultats démontrent que l’épissage alternatif et le potentiel codant d’un locus propre à un lncRNA est complexe et que cet ARN pourrait avoir de multiples fonctions dépendantes de l’état cellulaire.
Abstract
Embryonic development is a complex process finely regulated in time and place by numerous actors such as DNA, RNA, and proteins. Emerging evidence suggests a key role in development and disease for a subcategory of RNAs implicated in those mechanisms, the long non-coding RNAs (lncRNAs). Characterized by their incapacity to produce proteins, they have proven to be challenging to study due to the lack of sequence conservation and their tissue-specific expression. Here, we focus on a conserved lncRNA, Tuna (Tcl1 Upstream Neuron-Associated lncRNA), that is required for maintaining embryonic stem cells (ESCs) in an undifferentiated state but is also essential for their differentiation towards a neuronal cell fate. However, the mechanism behind this dual role remains largely unknown. To address this, we analyzed available RNA-seq data on mouse ESC differentiation towards neurons. We identified two novel isoforms specifically expressed in ESCs, sht.Tuna and alt.Tuna. The latter isoform was the result of alternative splicing of the exon 1 of Tuna. This alternative splicing was also observed in human ESCs demonstrating a conserved processing of the RNA between mouse and human cells. Both new isoforms were ~1.5kb shorter at the 3'-end than the predicted full transcript of Tuna (full.Tuna). In fact, we failed to detect the full.Tuna isoform in ESCs, and overexpression of sht.Tuna isoform enhanced reprogramming to a pluripotent stem cell state. This suggests that the role of Tuna is mechanistically different in ESCs than in neurons. Besides, Tuna also contains a highly conserved region (~200bp) harboring a predicted 48-amino-acids coding sequence that is detectable upon overexpression if FLAG-tagged. Mutating the start codon of this peptide's coding sequence abrogated the enhanced reprogramming effect. This infers a role for the peptide in the acquisition of a pluripotent state. Moreover, Tuna was detected in poly-ribosomal and cytoplasmic RNA fractions further supporting a peptide coding potential. Taken together, our results demonstrate that alternative splicing and coding potential of a particular lncRNA locus is complex and that a lncRNA may have multiple functionality depending on cell state.
Table des matières
Résumé ... ii
Abstract ... iii
Table des matières ... iv
Liste des figures ... vii
Liste des abréviations ... viii
Remerciements ... xi
Introduction ... 1
1. La pluripotence des cellules souches ... 2
1.1. Propriétés des cellules souches de l’embryon ... 2
1.2. Régulation du réseau de gènes de la pluripotence des ESCs ... 2
1.3. Les cellules souches pluripotentes induites (iPSCs) ... 4
2. Les ARNs non-codants dans la régulation génique ... 5
2.1. Les microARNs ... 5
2.2. Les longs ARN non-codants (lncRNAs) ... 6
2.3. Fonctions des lncRNAs dans la régulation génique ... 6
2.4. Les lncRNAs dans le contrôle du destin cellulaire ... 8
3. La conservation des lncRNAs ... 9
3.1. La conservation synténique ... 9
3.2. La conservation en séquence ... 10
3.3. La conservation en structure secondaire ... 11
3.4. La conservation en fonction ... 11
4. Le potentiel codant des lncRNAs... 12
4.1. De nombreux lncRNAs sont associés aux ribosomes ... 12
4.2. Certains lncRNAs encodent pour des peptides fonctionnels ... 13
4.3. La multifonctionnalité des lncRNAs ... 14
5.1. Tuna est conservé chez les vertébrés ... 15
5.2. Tuna : un lncRNA impliqué dans l’embryogenèse ... 16
5.3. Tuna : un lncRNA impliqué dans le contrôle de la pluripotence ... 16
5.4. Tuna : un lncRNA impliqué dans la différenciation neuronale ... 18
6. Objectifs de recherche ... 18
Chapitre 1. Caractérisation de l’ARN Tuna ... 26
1. Avant-propos... 27
2. Résultats ... 27
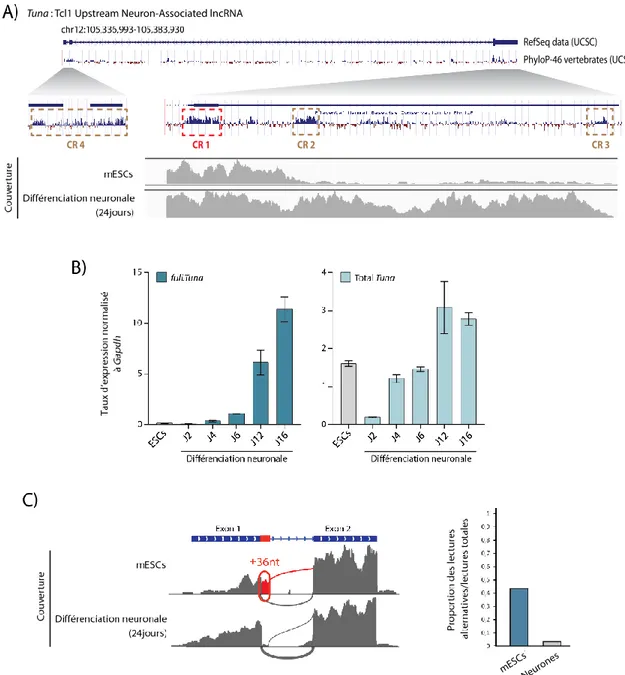
2.1. Tuna est conservé chez les vertébrés sur des régions spécifiques ... 27
2.2. Tuna est exprimé sous différentes isoformes dépendamment de l’état cellulaire ... 27
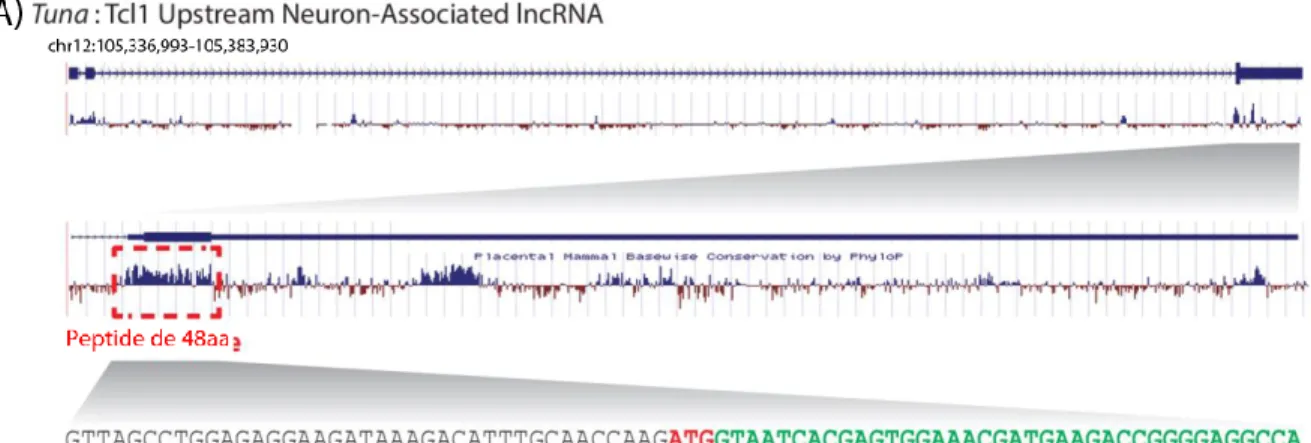
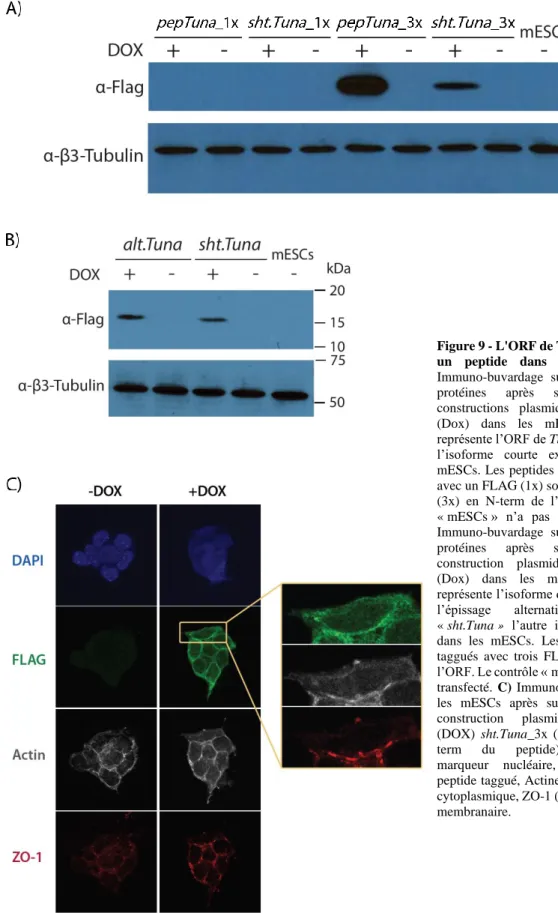
2.3. Tuna présente une séquence codante pour un peptide ... 29
2.4. Régulation de la stabilité de Tuna par NMD ... 31
3. Conclusion du chapitre ... 32
4. Figures ... 35
5. Matériel et Méthodes ... 39
5.1. Culture cellulaire ... 39
5.2. Différenciation neuronale de cellules souches de l’embryon ... 40
5.3. Transfection de mESCs ... 40
5.4. Extraction d’ARN et RT-qPCR ... 40
5.5. Fractionnement cellulaire ... 41
5.6. Profilage de polysomes ... 41
5.7. Extraction de protéines et immuno-buvardage ... 41
5.8. Immunofluorescence ... 43
Chapitre 2. Étude fonctionnelle de Tuna dans les cellules souches pluripotentes ... 44
1. Avant-propos... 45
2.2. Le potentiel codant de Tuna semble au cœur de sa fonction ... 46 3. Conclusion du chapitre ... 48 4. Figures ... 50 5. Matériel et Méthodes ... 52 5.1. Culture cellulaire ... 52 5.2. Transfection de NPCs ... 52 5.3. Reprogrammation de NPCs ... 53 5.4. Cytométrie en flux ... 53 Discussion générale ... 54
1. Le lncRNA ou les lncRNAs Tuna ? ... 55
2. La conservation, un indice important sur la fonction de Tuna ... 56
3. Codant et/ou non-codant ? Telle est la question. ... 58
Annexe 1 – Ensembl Genome Browser (Tuna) et amorces ... 61
Annexe 2 – Constructions plasmidiques tagguées FLAG ... 62
Annexe 3 – Principe CRISPR-inhibition et guides ARN ... 63
Annexe 4 – Gènes de pluripotence (RT-qPCR) ... 64
Liste des figures
Figure 1 - Régulation de la pluripotence des ESCs et différenciation. ... 20
Figure 2 – Exemples de mécanismes de régulation par les lncRNAs... 21
Figure 3 - Identification de nouveaux lncRNAs impliqués dans l’embryogénèse.. ... 22
Figure 4 - L'importance de linc-birc6 pour le bon développement du cerveau.. ... 23
Figure 5 - La perturbation de l'expression de Tuna affecte celle des gènes de pluripotence et de différenciation.. ... 24
Figure 6 - Tuna est impliqué dans le contrôle de la pluripotence et de la différenciation neuronale des mESCs. ... 25
Figure 7 - Tuna est conservé chez les vertébrés et présente de multiples isoformes en fonction de l'état cellulaire. ... 35
Figure 8 - Tuna possède un ORF et est associé aux ribosomes. ... 36
Figure 9 - L'ORF de Tuna encode pour un peptide dans les mESCs. ... 37
Figure 10 - Détection des isoformes de Tuna………..……38
Figure 11 - Le knock-down de Tuna affecte l'expression des gènes de pluripotence…………..50
Figure 12 - Concepts de la reprogrammation de mNPCs……….50
Figure 13 - Le potentiel codant de sht.Tuna est essentiel à sa fonction………51
Liste des abréviations
2i 2 inhibiteurs : CHIR et PDO3ADN Acide Désoxyribo-Nucléique ADNc ADN complémentaire
ADNg ADN génomique
ALU (caractérisé chez) Arthrobacter luteus AMPc Adénosine Monophosphate cyclique ARN Acide Ribo-Nucléique
ARNm ARN messager ARNr ARN ribosomal
ASOs Anti-Sense Oligonucleotides (Oligonucléotides antisens) b-FGF basic Fibroblast Growth Factor
CHIR Chiron (inhibiteur de GSK-3)
ChIP-Seq Chromatin Immuno-Precipitation Sequencing (Immunoprécipitation de chromatine suivie de séquençage d’ADN)
ChIRP Chromatin Isolation by RNA Purification CLIP Cross-linking and Immunoprecipitation CR Conserved Region (Région conservée)
CRISPR Clustered Regularly Interspaced Short Palindromic Repeats DAPI 4',6-diamidino-2-phénylindole
DMEM Dulbecco/Vogt Modified Eagle minimal Medium DMS Diméthyle Sulfate
Dox Doxycycline
ECL Électrochimiluminescence EDTA Éthylènediaminetétraacétique EGF Epidermal Growth Factor ESCs Embryonic Stem Cells
FBS Fetal Bovin Serum (Sérum de veau fétal)
Fendrr Foxf1 adjacent non-coding developmental regulatory RNA Fgf4 Fibroblast Growth Factor 4
FNMD Facteurs du NMD Foxf1 Forkhead box protein F1
FW Forward
GCNF Germ Cell Nuclear Factor GFP Green Fluorescent Protein gp130 Glycoprotein 130
GSK-3 Glycogen Synthase Kinase 3
H19 H19 Imprinted Maternally Expressed Transcript hnRNP A1 Heterogeneous Nuclear Ribonucleoprotein A1 hnRNP-K Heterogeneous Nuclear Ribonucleoprotein K HOTAIR HOX Transcript Antisense RNA
HOTTIP HOXA Transcript at the distal tip HOXA Homeobox A cluster
HOXB-AS3 Homeobox B cluster Anti-sense 3 HOXC Homeobox C cluster
HOXD Homeobox D cluster HUR Human antigen R
iPSCs induced Pluripotent Stem Cells (Cellules Souches Pluripotentes induites) IRES Internal Ribosome Entry Site
JNK c-Jun N-terminal Kinase Klf4 Kruppel Like Factor 4
KD Knock-down
KO Knock-out
KRAB Krüppel Associated Box KSR Knockout Serum Replacement LIF Leukemia inhibition factor LIFR LIF Receptor
LncRNA Long non-coding RNA (Long ARN non-codant)
LincRNA Long intergenic non-coding RNA (Long ARN non-codant intergénique) linc-RoR long intergenic non-coding RNA, regulator of reprogramming
LSD1 Lysine K-specific demethylase 1A MAPK Mitogen-Activated Protein Kinase
MAPKK MAPK Kinase
MEFs Mouse Embryonic Fibroblasts
MEK Autre nom de MAPKK
mESCs mouse Embryonic Stem Cells (Cellules souches de l’embryon de souris)
microARN ARN de 20-25nt impliqués dans la régulation de la stabilité et traduction des ARNm mir-145 microRNA 145
MLL1 Myeloid/lymphoid or mixed-lineage leukemia 1 mTORC1 mammalian Target of Rapamycin Complex 1 Myc MYC Proto-oncogene
MyoD Myoblast determination protein 1
MOs Morpholinos
Nanog Tir Na Nog (référence à la terre mythologique celtique nommée « ever young ») NCL Nucleolin
NGS Next Generation Sequencing (Séquençage de Nouvelle Génération) NMD Non-sense Mediated Decay (Dégradation médiée par le non-sense) NMDI14 NMD Inhibitor #14 (composé chimique inhibiteur du NMD #14) NPCs Neuro-Progenitor Cells (Cellules neuro-progénitrices)
NSCM Neural Stem Cell Media (milieu de culture des cellules souches neurales)
nt Nucléotides
Oct4 Octamer-binding Transcription factor 4 ORF Open Reading Frame (Cadre de lecture ouvert) OSKM Oct4-Sox2-Klf4-Myc (facteurs de Yamanaka) p38MAPK p38 mitogen-activated protein kinase
PDO3 PDO325901 (inhibiteur de MAPKK) PIC Protease Inhibition Cocktail
PKA Protein Kinase cAMP-dependent (Protéine Kinases AMPc dépendantes) PKM1 Pyruvate Kinase Isozyme M1
PKM2 Pyruvate Kinase Isozyme M2 Poly(A) Polyadénylation
Pou5f1 POU domain class 5 transcription factor 1 PRC2 Polycomb Repressive Complex 2
PTBP1 Polypyrimidine Tract Binding Protein 1 Qsp quantité suffisante pour
RACE Rapid Amplification of cDNA Ends
RBPs RNA Binding Proteins (Protéines liant l’ARN) RE Réticulum Endoplasmique
REV Reverse
Rex1 Reduced Expression Protein 1 RIPA Radioimmunoprecipitation assay RISC RNA-Induced Silencing Complex RT Rétro-transcription
Seq Séquençage
SHAPE Selective 2’-hydroxyl acylation analyzed by primer extension sgRNA Single Guide RNA (ARN guide unique)
shRNA short hairpin RNA (court ARN en épingle à cheveux) Sox2 SRY-Box transcription factor 2
SPAR Small regulatory Polypeptide of Amino-acid Response SINE Short Interspersed Nuclear Element
SNC Système Nerveux Central SRA Steroid Receptor Activator SRAP SRA protein
STAT3 Signal transducer and activator of transcription 3 STAU1 Staufen, RNA Binding Protein, Homolog 1
TBS Tris-Buffered Saline (Solution saline de tampon au Tris) TINCR Tissue Differentiation-Inducing Non-Protein Coding RNA TNFa Tumor Necrosis Factor alpha
Tris Trisaminométhane
TrypLE Enzyme de dissociation de cellules
TSS Transcription Starting Site (Site d’initiation de la transcription) Tuna/Tunar Tcl1 Upstream Neuron-Associated lncRNA
TxrG Trithorax protein regulatory Group
UTR Untranslated Region (Région non-traduite)
WNT association de « Wingless » et « Integration » en anglais (impliqué dans la morphogénèse chez Drosophila melanogaster)
Remerciements
En premier lieu, je souhaite remercier mes proches, mes amis, ma famille, qui m’ont soutenu à tous les points de vue tout au long de mon parcours d’études supérieures et tout particulièrement au moment de ma décision d’effectuer mon retour à la maîtrise.
Je remercie également l’Université Laval pour la qualité de l’enseignement et de la formation qui est proposée aux étudiants, notamment au sein de la faculté de médecine et plus particulièrement dans le programme de Biologie Cellulaire et Moléculaire.
Je remercie mes collègues, mes amis, Gabriel Khelifi, Victoire Fort, Emeline Lelong, Valérie Watters et Gaëlle Bourriquen. Merci d’avoir été d’exceptionnels compagnons de route tant d’un point de vue scientifique qu’humain. Tristement, je ne pense pas retrouver un tel plaisir à travailler en équipe à l’avenir que de partager ses moments avec vous.
Je souhaite également remercier tous les autres étudiants d’autres laboratoires avec qui j’ai noué des liens d’amitié. Même s’il m’est difficile de tous vous citer ici, je tiens à vous faire part de tout le bien que m’a apporté votre rencontre et les échanges/les moments que j’ai partagé avec vous. Finalement, j’aimerais remercier mon directeur de recherche, Dr. Samer Hussein, pour avoir cru en mon potentiel et pour son implication totale dans la formation de ses étudiants. Je remercie le Dr. Hussein pour son accessibilité, son ouverture d’esprit mais également pour les nombreuses opportunités qu’il m’a permis de saisir et qui m’ont permis de grandir scientifiquement et personnellement. Je suis convaincu que l’état d’esprit et les valeurs scientifiques que j’ai eu la chance d’acquérir se reflèteront et influenceront sur mes futures expériences professionnelles.
1. La pluripotence des cellules souches
1.1. Propriétés des cellules souches de l’embryon
Lors de l’embryogénèse qui suit la fécondation, les cellules souches pluripotentes sont essentielles, car elles déterminent le devenir de l’embryon. En effet, elles apparaissent dès le jour 4 au stade du blastocyste chez la souris1,2. Elles composent la masse cellulaire interne, protégée par les
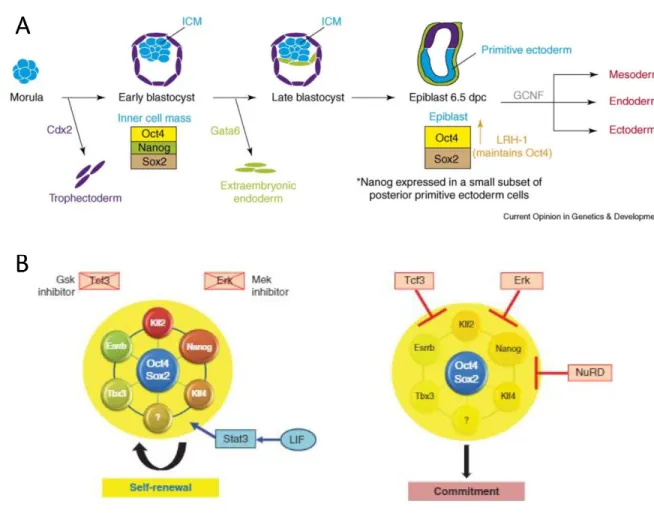
cellules du trophoblaste qui forment la couche externe de l’embryon. Les cellules souches de l’embryon ou « ESCs » (Embryonic Stem Cells) sont les précurseurs de l’ensemble des types cellulaires d’un organisme. En effet, elles ont la capacité de se différencier et de générer les trois feuillets embryonnaires, l’endoderme, l’ectoderme et le mésoderme, qui respectivement se développeront pour donner le tube digestif/les voies respiratoires, le système nerveux/organes externes/muqueuses et les os/muscles/derme3 (Figure 1.A).
Contrairement à la masse cellulaire interne dans laquelle les cellules passent par un état de pluripotence transitoire pour inévitablement entrer en différenciation, les ESCs peuvent être isolées et maintenues presque indéfiniment en culture in vitro grâce à leur capacité de renouvellement3,4. Pour
maintenir de telles cellules, il est nécessaire d’inhiber leur différenciation en stimulant l’expression des gènes associés à la pluripotence et en inhibant celle des gènes associés à leur différenciation vers les types cellulaires des trois feuillets embryonnaires. Les mESCs (cellules souches embryonnaires de souris) sont cultivées en présence de LIF (Leukaemia inhibitory factor) qui promeut leur auto-renouvellement, de CHIR99021 (inhibiteur de GSK3β, abrégé CHIR) et de PD0325901 (inhibiteur de la voie MEK, abrégé PD03), permettant le maintien de l’expression des gènes de pluripotence et l’inhibition de l’expression des gènes de différenciation3,5 (Figure 1).
L’isolation des mESCs et leur culture in vitro représente un outil puissant dans l’étude de l’état de pluripotence des cellules à l’origine de tout un organisme. Leur caractérisation a d’ailleurs permis de mettre en lumière la complexité et l’importance de la régulation fine de l’expression des gènes dans le contexte de pluripotence, pour son maintien mais aussi pour, plus tard, garantir la bonne différenciation des cellules et la viabilité de l’embryon en formation.
1.2. Régulation du réseau de gènes de la pluripotence des ESCs
L’identité des ESCs souris et humaines est maintenue par un vaste réseau de gènes dont la régulation spatio-temporelle est orchestrée par un ensemble de gènes clefs : Oct4 (Pou5f1), Sox2 et Nanog. Ces gènes encodent des facteurs de transcription régulant leur propre expression et celles des
gènes de pluripotence/de différenciation annexes, faisant d’eux des effecteurs situés en aval des voies de signalisation BMP, WNT et LIF6.
Dans les conditions de culture in vitro, le LIF est le ligand du récepteur membranaire hétérodimérique de LIFR-gp130, qui une fois activé va provoquer la phosphorylation de la protéine STAT3 (Signal Transducer & Activator of Transcription-3), un transducteur du signal vers le noyau où il joue un rôle de facteur de transcription afin de promouvoir l’auto-renouvellement des mESCs (Figure 1.B). L’expression de STAT3 est d’ailleurs régulée par la liaison d’OCT4 au promoteur de son gène, un des facteurs de transcription au centre de la pluripotence cité précédemment3,7.
D’autre part, l’activation de la voie WNT par sa liaison au récepteur membranaire Frizzled entraine l’inhibition de la glycogene-synthase kinase-3 (GSK3) provoquant l’accumulation de β-caténine dans le noyau, où elle joue le rôle de facteur de transcription pour de nombreux gènes impliqués dans le maintien de la pluripotence. En culture, le CHIR mime l’activation de la voie WNT en inhibant GSK3 et permet le maintien du caractère indifférencié des ESCs en favorisant l’expression des gènes de pluripotence tels que Oct4, Rex1 et Nanog1,3 (Figure 1.B).
Il est clair que le maintien de l’identité des cellules souches embryonnaires se joue dans le noyau grâce aux facteurs de transcription activés en aval des protéines de signalisation mentionnées plus haut. Même si, seul, il ne peut empêcher la différenciation des cellules souches en absence de LIF8,
OCT4 est un facteur de transcription au centre de la pluripotence. En effet, il est exprimé à différents niveaux dans l’ensemble des cellules pluripotentes ou indifférenciées9,10 (Figure 1.C). En association
avec SOX2, OCT4 est essentiel au maintien de l’état pluripotent des ESCs. Néanmoins, lorsqu’il est surexprimé, ce facteur de transcription est nécessaire à la différenciation vers le mésendoderme. A l’inverse, lorsqu’OCT4 est sous-exprimé, les ESCs entrent préférentiellement dans le processus de différenciation vers le trophectoderme (Figure 1.A).
SOX2 est également impliqué dans ces processus et plus particulièrement dans la différenciation du neuroectoderme puisqu’il est à la fois exprimé dans les ESCs et les cellules souches neurales. En formant un complexe ternaire avec OCT4, il active ses gènes cibles tels que Fgf411, un gène impliqué
dans la survie et la croissance de la masse cellulaire interne de l’embryon, mais aussi lors de la différenciation des cellules souches neurales.
D’autre part, NANOG est requis pour la formation de l’épiblaste et son expression constitutive in vitro permet le renouvellement des cellules souches de l’embryon de manière LIF-indépendante. De plus il partage la majorité de ses cibles avec OCT4 et SOX212.
Ainsi l’état pluripotent des cellules souches embryonnaires est fragile et nécessite une régulation fine de son réseau de gènes. Ce réseau implique de multiples acteurs, des protéines membranaires et cytoplasmiques impliquées dans la transduction du signal ainsi que des facteurs de transcription nucléaires.
1.3. Les cellules souches pluripotentes induites (iPSCs)
L’étude des cellules souches ouvre la voie à la médecine régénératrice et personnalisée, au criblage de drogues mais aussi à la modélisation de maladies. Leur capacité d’auto-renouvellement permet de les maintenir facilement en culture in vitro et leur potentiel de différentiation sont des caractéristiques exploitables pour la régénération de tissus voir même un jour d’organes entiers. De plus, depuis 2006 il est possible de générer des cellules souches pluripotentes induites (iPSCs) à partir de cellules somatiques différenciées par la surexpression de quatre facteurs de transcription au cœur de la pluripotence : OCT4, SOX2, c-MYC et KLF413. Cependant c-MYC et KLF4 ne sont pas
indispensables à la reprogrammation cellulaire alors que les facteurs OCT4 et SOX2 sont absolument nécessaires ce qui fait d’eux les deux principaux facteurs, au centre de la pluripotence14,15. Ce
processus affecte l’identité cellulaire, de l’ADN au phénotype macroscopique des cellules. La surexpression de ces facteurs de transcription va provoquer le remodelage de la chromatine et des marques épigénétiques impactant le transcriptome, le traductome et les voies de signalisations, mimant ceux des cellules souches pluripotentes de la masse cellulaire interne de l’embryon.Même si quelques singularités existent entre ESCs et iPSCs16,17, ces dernières peuvent substituer la masse
cellulaire interne du blastocyste et donner naissance à la progéniture qui aura le même patrimoine génétique que l’individu dont les cellules différenciées sont issues18.
Dans le contexte de l’étude des maladies génétiques, la reprogrammation de cellules différenciées de patients vers le stade iPSCs facilite l’étude des pathologies de façon personnalisée. Étant donné les caractéristiques très proches des iPSCs avec les ESCs, elles sont faciles à cultiver, prolifèrent rapidement et peuvent également se différencier vers les 3 feuillets embryonnaires. La différenciation des iPSCs générées à partir des cellules des patients vers le type cellulaire affecté par les maladies développementales/génétiques permet ainsi de les modéliser in vitro de manière non invasive. Ainsi, tout en s’affranchissant de prélèvements répétés, il est possible d’effectuer des criblages pour de multiples drogues mais également de procéder à une thérapie génique. Au stade iPSCs, les cellules sont hautement prolifératives et leur génome est facilement manipulable/modifiable, du fait de l’état « ouvert » de leur chromatine17,19,20. Il serait donc possible d’effectuer une « réparation génétique »
peuvent alors être différenciées vers le(s) type(s) cellulaire(s) initialement défectueux et réinjectées chez les patients21,22. Cette approche représente une voie de traitement prometteuse, s’affranchissant
des rejets et réactions immunes rencontrées lors de greffes par exemple, car seules les cellules du « soi » sont utilisées ce qui garantit une haute compatibilité.
2. Les ARNs non-codants dans la régulation génique
Les ARNs ne possédant aucun ORF fonctionnel et ne codant donc pour aucune protéine ont longtemps été considérés comme du « Junk DNA » ou « bruit » de transcription23. De nos jours, il est
maintenant établi que certaines catégories de ces poly-ribonucléotides sont en réalité des transcrits fonctionnels24.
2.1. Les microARNs
Parmi les transcrits non-codants, les microARNs ont été les premiers à être étudiés. Ce sont de petits transcrits de 20-25 nucléotides faisant partie de la catégorie des « petits ARNs » ayant un rôle essentiel et largement documenté dans la régulation post-transcriptionnelle25. Ces petits ARNs
non-codants sont le résultat de la prise en charge et de la maturation de structures d’ARN double brin en « épingle » pour donner un microARN mature simple brin complémentaire à la séquence de leurs ARNm cibles. L’association d’un microARN couplé à une protéine argonaute AGO, complexe appelé RISC (RNA-Induced Silencing Complex), avec sa cible peut entrainer soit la dégradation de celle-ci via l’activité ribo-endonucléase du domaine PIWI de AGO, soit l’inhibition de sa traduction en bloquant l’association des sous-unités ribosomales ou en perturbant la phase d’élongation de la synthèse du polypeptide26. Ainsi, ces transcrits sont essentiels à la régulation génique et interviennent
dans de nombreux évènements liés aux processus développementaux2527. Les ARNs lin28 et let-7 en
sont de bons exemples puisqu’ils s’antagonisent dépendamment du stade cellulaire, si bien que lorsque lin28 est exprimé (activation par le complexe OCT4-SOX2-NANOG) dans les cellules pluripotentes, la protéine LIN28 se lie au pré-microARN let-7, ce qui empêche sa prise en charge par le complexe de maturation des microARNs28,29. Let-7 ne peut ainsi promouvoir ni la dégradation de
lin28 ni celles de ses autres ARNs cibles tels que Myc (proto-oncogène essentiel dans la prolifération des cellules souches) ou Sall4 (impliqué dans le maintien et l’auto-renouvellement des cellules souches embryonnaires et hématopoïétiques). Par opposition dans les cellules différenciées, le promoteur de lin28 n’est plus activé et let-7 est pris en charge par le complexe de maturation des microARNs, occasionnant ainsi la dégradation de ses ARNs cibles incluant l’ARN lin2828,29.
Ainsi les ARNs non-codants, ici les microARNs, sont essentiels à la régulation de l’expression génique en intervenant à différents niveaux de régulation : traduction, maturation ou stabilité d’autres transcrits.
Plus récemment, une autre catégorie de transcrits non-codants a été identifiée et associée à de nombreuses fonctions dans la cellule. Ces ARNs de plus de 200 nucléotides transcrits par l’ARN polymérase II, ont pour la plupart la particularité de subir l’épissage, d’être coiffés en 5’ et polyadénylés en 3’ de la même manière que les ARNm30,31. Ces « longs » ARNs (en comparaison
avec les microARNs) sont aujourd’hui appelés longs ARNs non-codants, « long non-coding RNAs » (lncRNAs). Enfin, même s’ils partagent certaines caractéristiques avec les ARNm, ils sont moins exprimés que ces derniers et sont souvent tissus-spécifiques32. Toutefois, ils interviennent à plusieurs
niveaux dans de nombreux processus cellulaire, notamment au cours du développement, faisant d’eux de potentielles cibles thérapeutiques.
2.2. Les longs ARN non-codants (lncRNAs)
Le terme « lncRNAs » représente en réalité une vaste famille de transcrits au sein de laquelle ils sont généralement associés à un des multiples sous-groupes32. Parmi eux se retrouvent, les long
ARNs non-codants intergéniques (lincRNAs), dont la séquence génomique ne se superpose avec celle d’aucun autre gène; les lncRNAs antisens, qui comme suggère leur nom, sont transcrits à partir du brin d’ADN complémentaire à un autre gène où la juxtaposition pourra être complète, partielle ou à cheval entre deux gènes; les pseudogènes, résultats de l’évolution du génome provoquant la perte de la capacité de certains ARNs à être traduits, transcrits à partir de gènes autrefois codants; les lncRNAs introniques, ARNs transcrits à partir d’introns de gènes codants; les ARNs associés aux promoteurs et aux activateurs (« enhancers »), représentent une classe à part puisque leur taille varie de 20 à plus de 2000 nucléotides et ne sont pas transcrits à partir de gènes « conventionnels » mais de régions promotrices de la transcription sur laquelle ils auraient un rôle régulateur23.
Les lncRNAs auxquels nous nous intéresserons dans le contexte des objectifs du laboratoire précisés plus bas, sont de plus en plus étudiés et de nombreuses fonctions leur sont associées33, notamment au
niveau de la régulation génique. Cependant les mécanismes moléculaires de nombre d’entre eux restent encore largement méconnus.
2.3. Fonctions des lncRNAs dans la régulation génique
directement avec la chromatine ou pour former des domaines nucléaires, mais aussi dans le cytoplasme pour affecter la stabilité et/ou la traduction de certains ARNm23.
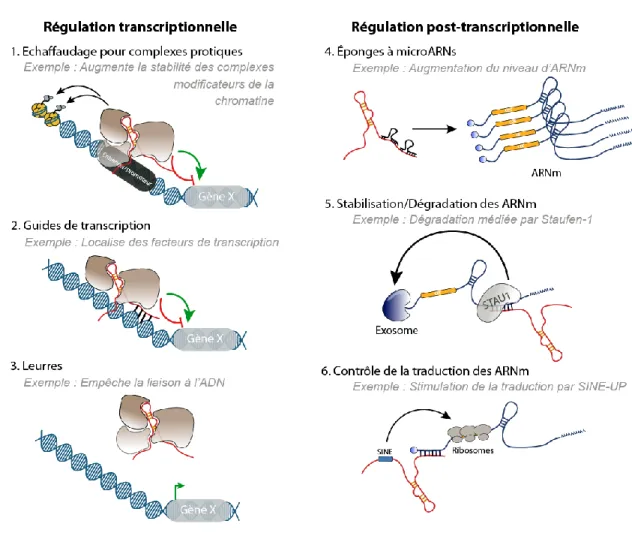
Dans le noyau, certains lncRNAs jouent le rôle d’échafaudage pour des complexes protéiques comme les unités de remodelage de la chromatine (Figure 2.1), qui apposent des modifications post-traductionnelles sur les « queues » des histones formant les nucléosomes, modulant ainsi leur accessibilité aux facteurs d’initiation de la transcription. Par exemple, l’ARN HOTTIP recrute le complexe MLL1 à proximité des promoteurs des gènes du domaine HOXA (intervient lors de la segmentation le long de l’axe antéro-postérieur) et agit en cis pour apposer les marques H3K4me3 activant la transcription34. D’autres lncRNAs nucléaires servent de guides pour des
activateurs/inhibiteurs de la transcription ou titrent des protéines régulatrices de la transcription32,33
(Figure 2.2 et 2.3). Par exemple, dans les cellules souches de l’embryon, les lncRNAs ES1 et ES2 se lient à la protéine SOX2, un facteur de transcription clef de la pluripotence, et se localisent aux promoteurs de gènes cibles impliqués dans le maintien de la pluripotence. La baisse de la transcription de ces deux lncRNAs entraine d’ailleurs la différenciation des cellules souches humaines de l’embryon35. Enfin, certains autres lncRNAs permettent la nucléation de domaines sub-nucléaires
nécessaires à la régulation de certains ARNm/de certaines protéines, en fonction des conditions de stress, grâce à leur structures secondaire et leurs partenaires d’interaction. Par exemple, les lncRNAs Neat1 et Neat2, vont participer à la formation et au maintien de l’architecture des Paraspeckles36, qui
interviennent entre autres sur la transcription et sur l’épissage des ARNs nouvellement synthétisés, modulant leur localisation et leur devenir dans la cellule37,38.
D’autre part, les lncRNAs localisés au cytoplasme peuvent avoir un rôle de régulation post-transcriptionnelle ou traductionnel des ARNm. Au niveau post-transcriptionnel certains lncRNAs vont jouer le rôle d’éponge à microARNs (Figure 2.4), affectant positivement la stabilité de leurs cibles. Lorsque ces lncRNAs ne sont plus exprimés, les ARNm sont alors dégradés par la voie des microARNs. Ceci a pour effet de diminuer indirectement la quantité de protéines produites, affectant l’ensemble des processus cellulaires dans lesquels elles étaient impliquées. Par exemple, le lncRNA H19 est impliqué dans la différenciation musculaire chez l’humain. En effet, lorsque H19 est sous-exprimé, il ne titre plus les microARN de la famille let-7 et provoque l’entrée précoce des cellules (en culture) en myogenèse, ce qui est également observé lorsque le microARN let-7 est surexprimé39.
Les lncRNAs cytoplasmiques n’interagissent pas seulement avec des microARNs mais aussi avec des protéines pour affecter la stabilité et la traduction des ARNm via de multiples mécanismes32,36. Par
exemple, le lincRNA p21 se lie aux ARNm JUNB (codant pour un facteur de transcription) et CTNNB1 (codant pour la β-caténine, agit en aval de la voie WNT mentionnée précédemment) via son
domaine Rck ce qui inhibe leur traduction. En revanche, lorsque la protéine HUR est exprimée et se lie à p21, ceci active sa dégradation par le complexe RISC chargé du microARN let-7. Dans ce cas de figure, les ARNm JUNB et CTNNB1 sont stabilisés et traduits32,40.
Les lncRNAs sont également impliqués dans la régulation de la stabilité des ARNm (Figure 2.5 et
2.6). Parmi eux, certains lncRNAs contiennent des séquences SINE/ALU capables de s’hybrider avec
des séquences complémentaires antisens présentes dans les 3’-UTR des ARNm. Cette structure ARN-ARN double brin est reconnue par la protéine STAU1, à l’origine de la voie de dégradation des ARNm médié par Staufen1 (SMD)41,42. Cependant STAU1 n’est pas toujours associée à la
déstabilisation des ARNm lorsqu’elle est liée à un lncRNA. En effet, lorsqu’elle est liée au lncRNA TINCR, lui-même associé aux ARNm de promotion de la différenciation épidermique, le complexe ainsi formé induit leur stabilisation et promeut leur traduction32,43.
A travers les différentes fonctions et exemples de lncRNAs, il paraît clairement qu’ils représentent des acteurs majeurs et essentiels dans la régulation de l’expression des gènes, et ce, à tous les niveaux. Nous avons également vu qu’ils affectaient tous types de gènes notamment impliqués dans la différenciation et le développement, et qu’ils interviennent donc dans le destin/la définition de l’identité cellulaire, qui requière un contrôle spatio-temporel fin et précis de l’expression génique.
2.4. Les lncRNAs dans le contrôle du destin cellulaire
Le développement embryonnaire assure la formation d’un organisme par la régulation spatio-temporelle de l’expression d’un réseau complexe et spécifique de gènes afin de garantir la bonne différenciation des cellules et la formation d’un embryon sain. Nous avons vu à travers les exemples précédents, que les lncRNAs sont impliqués dans la régulation génique nécessaire aux mécanismes de différenciation lors du développement. En effet, ils agissent aussi bien sur l’état de la chromatine que sur les protéines et les ARNs nécessaires au maintien des cellules, et ce, quel que soit leur stade de différenciation. À la base du développement se trouve les cellules souches pluripotentes de la masse cellulaire interne, générant l’intégralité des types cellulaires de l’organisme en formation. Par exemple, le lncRNA Fendrr est essentiel au développement embryonnaire et cardiaque chez la souris via son interaction avec les complexes de remodelage des marques épigénétiques de la chromatine PRC2 et TxrG/MLL. Ce complexe cible le réseau de gènes codants pour des facteurs de transcription, tels que Foxf1/Gata4/Gata6/Nkx2-5, impliqués dans la différenciation du mésoderme latéral, compromise lorsque Fendrr n’est plus exprimé44.
Les lncRNAs sont également impliqué dans le maintien de l’identité de ces cellules et ont un rôle essentiel dans le maintien des ESCs comme nous l’avons vu avec les ARNs ES1 et ES2 qui promeuvent la pluripotence. Dans un autre exemple, l’ARN linc-RoR est également un acteur important dans le maintien et la transition de d’un état cellulaire à un autre. En effet, dans les ESCs, linc-RoR titre le microARN mir-145 dont les cibles sont Oct4, Sox2 et Nanog, les « gardiens » de la pluripotence. Ainsi, lorsqu’il est exprimé il agit pour maintenir le caractère pluripotent des cellules. En revanche lors de la différenciation des cellules, le niveau d’expression de linc-RoR baisse et permet à mir-145 de cibler les ARNs essentiels au maintien de la pluripotence, qui sont alors dégradés par la voie des microARNs32,45,46. Indirectement, linc-RoR permet la différenciation des cellules souches par
la baisse de son expression.
Les lncRNAs sont donc bien des régulateurs essentiels de l’expression génique et permettent le contrôle du destin cellulaire.
3. La conservation des lncRNAs
Les lncRNAs sont souvent décrits comme peu conservés à travers l’évolution33 mais sont
généralement comparés aux ARNm qui eux présentent un haut taux de conservation que ce soit au niveau génomique, transcriptomique ou protéique. Cependant de récentes études montrent qu’au sein de la superfamille des lncRNAs se démarquent certains ARNs qui présentent une conservation particulièrement haute qu’elle soit synténique, en séquence, en structure et/ou en fonction47,48.
3.1. La conservation synténique
La conservation synténique ne fait intervenir ni la séquence, ni la structure, ni même la fonction du transcrit en tant que tel. Elle se réfère au simple mécanisme de transcription ayant lieu à un point précis du génome et pouvant être observé chez plusieurs espèces. Dans ce cas de figure, l’acte de transcription en lui-même, au locus génomique d’un lncRNA sur un chromosome donné, peut affecter l’expression de gènes voisins, orthologues d’une espèce à l’autre49. Par exemple, le
lncRNA HOTAIR (humain) et Hotair (murin) ne partagent que très peu d’homologie que ce soit au niveau de la structure de leur gènes respectifs, de leur séquence ou même de leur fonction. En effet, chez les deux espèces le gène encodant ce lncRNA se situe dans le locus du groupe de gènes HOXC (impliqués dans la segmentation et la définition de l’axe antéro-postérieur) mais ne présente pas le même nombre d’exons, soit 6 exons chez l’humain et 2 chez la souris. De plus, contrairement à ce qui est retrouvé chez l’humain, Hotair ne possède pas les régions de liaison aux complexes de remodelage de la chromatine LSD1 (déméthylation des lysines H3K4/K9me ou H3K4/K9me2) et
PRC2 (apposition de la marque H3K27me3), participant à la régulation en cis de la transcription des gènes du groupe HOXC50. En revanche, le Knock-Out (KO) de Hotair dans un modèle de souris
entraine la malformation du squelette. Cette observation est causée par la perturbation de l’expression des gènes du groupe HOXD (impliqués dans le développement des membres) engendrée par la perte des marques H3K4me3 et H3K27me3, ceci malgré le fait que Hotair ne possède pas les régions de liaisons aux complexes de remodelage de la chromatine responsables de ces modifications d’histones47,50,51. De manière intéressante, les gènes affectés par le KO de Hotair font également partie
des cibles de HOTAIR, sur lesquelles il agit en trans. En considérant l’absence des sites de liaison à LSD1 et PRC2 chez Hotair et l’effet conservé chez l’humain et la souris, la fonction de ce lncRNA serait très probablement une conséquence de sa simple transcription, conservée et affectant un même groupe de gènes d’une espèce à l’autre.
3.2. La conservation en séquence
Bien que les lncRNAs soient généralement décrits comme peu conservés en séquence, ils sont comparés aux ARNm dont la conservation principalement concentrée au niveau de leur région codante, garantissant la conservation en acides-aminés et ainsi celle de la fonction protéique. Cependant, comme les ARNm, certains lncRNAs se démarquent par une haute conservation régionalisée au niveau de portions de séquences spécifiques, la plupart du temps liées à leur mécanisme47. Parmi ces régions se retrouvent notamment des motifs médiant la liaison avec différents
types d’interacteurs, ARN, ADN ou protéiques23. Par exemple, Xist/XIST (X-inactive specific
transcript) est un lncRNA nucléaire longuement étudié, responsable de l’inactivation d’un des deux chromosomes X dans les cellules femelles52. Outre la conservation globale de son architecture, il
possède de nombreuses régions conservées chez l’humain, la souris et le campagnol (rongeur) sous la forme de répétition en tandem53. L’expression de variants de Xist et XIST, dans lesquels ces régions
ont été modifiées ou exprimées individuellement, a montré quelles étaient essentielles aux différentes fonctions de l’ARN dans l’inactivation du chromosome X52. La région répétée et conservée RepA en
5’ de Xist est, par exemple, responsable du recrutement du complexe PRC2 qui médie l’inactivation du chromosome X via remodelage de la chromatine (apposition de marques épigénétiques H3K27me3 réprimant la transcription) dans les diverses espèces où il est exprimé54,55.
Ainsi, même si les lncRNAs ne présentent pas une conservation en séquence aussi étendue que celle des ARNm, elle n’est pas négligeable et renseigne sur les mécanismes moléculaires dans lesquels ils sont impliqués, et plus généralement sur leur fonction.
3.3. La conservation en structure secondaire
Tous les lncRNAs ne présentent pas de régions à haute conservation en séquence. La conservation de l’arrangement spatial de la chaîne nucléotidique ne dépend pas uniquement de la conservation en séquence mais essentiellement de l’appariement base à base pour former les structures secondaires. En effet, plus de 90% de ces structures se retrouvent en dehors des éventuelles régions conservées en séquence des lncRNAs47,56. Ainsi, la divergence en séquence des lncRNAs
n’implique pas toujours celle de leur structure ni, par association, de leur(s) fonction(s).
Par exemple, l’ARN SRA (Steriod Receptor Activator) est un lncRNA impliqué dans de nombreux complexes ARN-protéines comme « échafaudage » et intervient ainsi dans de nombreux processus cellulaires. En effet, ses structures secondaires lui permettent d’interagir avec des facteurs de transcription activateur ou répresseurs, contrôlant ainsi l’expression des gènes cibles. Par exemple, il interagit avec le facteur MyoD via sa structure STR7 pour induire la transcription des gènes impliqués dans la différenciation musculaire des cellules myogéniques57. De la même manière il interagit avec
de nombreuses autres protéines/facteurs de transcription et est ainsi impliqué dans la prolifération/cycle cellulaire (cellule cancéreuses et adipocytes), la signalisation de l’insuline (adipocytes et hépatocytes), la signalisation via NOTCH (cellules cancéreuses), la signalisation par TNFa (adipocytes) et la signalisation par p38MAPK/JNK (adipocytes)58,59. Bien que cet ARN soit un
excellent exemple de l’implication des lncRNAs dans la régulation génique et le contrôle du destin cellulaire, il a la particularité de remplir sa fonction grâce aux différentes structures secondaire qu’il adopte. De plus, les 4 grands groupes structuraux (déterminés par SHAPE, DMS, RNase V1 et In-Line Probing) qui le compose sont conservés chez les mammifères malgré le fait qu’il présente une faible conservation en séquence. Les analyses de covariance ont également montré que la divergence en séquence n’affectait pas la similarité en structure, ce qui garantit la conservation des interactions avec les différents partenaires de l’ARN SRA60.
3.4. La conservation en fonction
Les exemples précédents dissèquent les types de conservation des lncRNA un à un. Cependant, il est aisé de remarquer que souvent ils s’associent et sont rarement exclusifs. De plus, ces exemples montrent bien que la conservation a un impact direct sur la conséquence de l’expression du lncRNA, voir la fonction du transcrit lui-même. Pour reprendre l’exemple de Xist, la conservation de certaines régions de sa séquence, fonctionnelles, est associée à la conservation de sa fonction d’une espèce à l’autre, celle d’inactiver le chromosome X lors du développement embryonnaire. De même,
l’ARN SRA, possède une haute conservation de ses différentes structures secondaires ce qui lui permet de conserver son association à ses différents partenaires, comme des facteurs de transcription, qui pourront jouer leur rôle dans l’ensemble des espèces où l’ARN SRA est exprimé.
Cependant, même si de plus en plus de travaux s’intéressent à la fonction des lncRNAs, peu d’entre-eux effectuent des études comparatives inter-espèces afin d’en déterminer la conservation. Cette constatation pourrait expliquer pourquoi il est encore difficile d’estimer la proportion de lncRNAs dont la fonction est véritablement conservée.
4. Le potentiel codant des lncRNAs
Comme nous l’avons souligné précédemment les lncRNAs sont généralement reconnus pour leur incapacité à coder pour des protéines. Or, nous avons également constaté que certains lncRNAs cytoplasmiques pouvaient s’associer à des protéines et même avoir un impact direct sur la traduction des ARNm23. Étant donné la nature ribonucléoprotéique des ribosomes, il serait donc étonnant
qu’aucun lncRNA n’y soit associé.
4.1. De nombreux lncRNAs sont associés aux ribosomes
Il paraît établi que les lncRNAs ne codent pour aucune protéine. Or, certains lncRNAs présentent des ORFs putatifs30,61 . De plus, près de 50% d’entre eux sont cytoplasmiques dont 70%
sont retrouvés dans les fractions ribonucléiques à l’issue d’analyses par Ribosome Footprint62,63
(empreinte de ribosome). Cette technique consiste à isoler les fragments d’ARNs « chargés » par les ribosomes et protégés du traitement à la RNase I (ribo-endonucléase capable de digérer uniquement les molécules d’ARNs simple brin) pour ensuite les séquencer.
La présence de ces petits ORFs pourraient servir de leurres aux ribosomes dans le but de réguler leur disponibilité et ainsi contrôler la traduction des ARNm64. Cependant, contrairement aux ARNm, ni
la longueur de l’ORF ni la couverture de celui-ci ne peut expliquer le recrutement des ribosomes. Malgré cela, certains lncRNAs sont retrouvés dans les fractions polysomales légères et les fractions lourdes, qui correspondent généralement aux ARNm activement traduits65. Une autre hypothèse pour
expliquer l’association de ces lncRNAs aux ribosomes, repose sur la nature de leur « 5’-UTR », dont la composition en bases G et C, la faible abondance en éléments répétés et la longueur seraient caractéristiques des transcrits non-codants associés aux ribosomes, comparés aux lncRNAs « libres » dans le cytoplasme.
Enfin de manière systématique, les transcrits associés aux ribosomes sont coiffés en 5’ de la même manière que les ARNm. Ainsi, les conditions nécessaires à l’association de lncRNAs aux ribosomes résideraient probablement dans la séquence en amont du potentiel ATG (codon START)62.
Le devenir des lncRNAs associés aux ribosomes est encore nébuleux, mais cette association médierait une stabilisation transitoire après la « pause » des ribosomes au codon STOP au niveau duquel les ribosomes « stagnent », habituellement pour marquer la fin du processus de traduction. Les transcrits seraient par la suite dégradés par les voies de régulation de la stabilité des ARNm : déadénylation, décoiffage et clivage par endonucléases. En effet, ces lnRNAs possèdent de petits ORFs et présentent un codon STOP, ils pourraient donc être dégradés par cette voie. Cependant, la majorité de ces lncRNAs ne présentent pas d’ORF canoniques, il est donc fort probable que la terminaison de leur traduction soit considérée comme prématurée par la cellule (c.à.d présence d’une jonction exon-exon en aval du codon STOP), engendrant la pause précoce du ribosome, et induisant la dégradation du transcrit par NMD (Non-sense Mediated Decay)32.
Nous avons vu que la cartographie des empreintes de ribosomes montre que non seulement certains lncRNAs sont effectivement associés aux ribosomes mais également que, dans certains cas, ces derniers sont activement « lus » par la machinerie de traduction. De plus, même si la plupart de ces transcrits sont dégradés à la suite de leur association aux ribosomes, ces observations n’excluent pas la possibilité d’une traduction de certains d’entre eux.
4.2. Certains lncRNAs encodent pour des peptides fonctionnels
Même si la majorité des lncRNAs sont dégradés après s’être associés aux ribosomes, il a été démontré que certains transcrits étaient bels et bien traduits et généraient des « peptides » fonctionnels66. En effet, l’étude initiale de ceux-ci utilisait les critères de caractérisation basés sur
ceux de gènes connus, tels que la conservation en séquence, la présence d’une séquence KOZAK forte, la nature du 5’-UTR ou encore la longueur de l’ORF, ne permettant pas une identification adéquate du potentiel codant de ces nouveaux transcrits67,68. La prédominance de certains lncRNAs
dans les fractions ribosomales a alors été le point de départ d’une étude approfondie de leur association aux ribosomes (comme vu plus haut) et de leur pouvoir codant, faisant appel à des techniques de pointe telles que le profilage de ribosomes et la spectrométrie de masse69,70.
L’intégration de ces données a permis d’amorcer la caractérisation en profondeur de cette nouvelle catégorie d’ARNs et de celle des peptides qu’ils encodent.
Parmi la quinzaine de polypeptides fonctionnels identifiés à ce jour, toutes espèces confondues, SPAR est encodé par LINC00961 et est conservé entre l’humain et la souris71. Il intervient dans le contrôle
de la régénération musculaire par son rôle inhibiteur du complexe mTORC1. En effet, SPAR affecte directement la capacité des muscles squelettiques à se régénérer après une blessure, rôle qui a été démontré par le KO dans des souris C57BL/6J (édition du génome par CRISPR/Cas9), ce qui a eu pour effet l’amélioration régénération musculaire en comparaison avec les souris sauvages. L’effet négatif sur la régénération musculaire due à l’expression de SPAR est d’ailleurs comparable à celui de l’inhibition de l’activité de mTORC1 par la rapamycine. SPAR agit au niveau du complexe membranaire lysosomale v-ATPase auquel il s’associe pour empêcher le relargage du régulateur et activateur du complexe mTORC1 à la suite d’une stimulation aux acides-aminés71.
L’ARN HOXB-AS3 est un autre bon exemple de transcrit initialement considéré comme « non-codant », puisqu’il encode en réalité un peptide 53 acides-aminés impliqué dans l’épissage alternatif de l’ARNm codant pour les protéines PKM1 et PKM2 (Pyruvate Kinase M1 et M2)72. En effet,
lorsque le peptide HOXB-AS3 est exprimé dans les cellules saines, il s’associe au facteur d’épissage hnRNP A1 au niveau des résidus arginine de son motif RGG. Cette association entre en compétition avec le site de liaison de hnRNP A1 avec l’exon 9 du pre-mRNA PKM (avant épissage), permettant l’expression finale de la protéine PKM1, saine. Dans les cellules cancéreuses du colon, la faible expression de HOXB-AS3 et donc du peptide qu’il lui est associé, ne permet plus de réguler l’association de hnRNP A1 à l’exon 9 de PKM ce qui entraine son épissage alternatif et la production de PKM2. Cette dernière se retrouve sous forme dimérique dans les cellules hautement prolifératives comme les cellules cancéreuses. Elle favorise l’accumulation d’intermédiaires métaboliques de la glycolyse qui empreintes alors des voies métaboliques alternatives, favorables aux cellules cancéreuses. Son expression entraine une reprogrammation métabolique des cellules cancéreuses du colon, favorisant leur prolifération, un mauvais pronostique pour la survie du patient72.
4.3. La multifonctionnalité des lncRNAs
Les précédents exemples ne font mention que de la fonction des transcrits ou des peptides dont ils sont à l’origine, de manière individuelle. Cependant, il est possible que ces deux entités aient leur fonction propre, attribuant ainsi un double rôle au gène correspondant, codant et non-codant. Comme nous avons pu le remarquer avec l’exemple de la conservation en structure secondaire des lncRNAs, il est clair que l’ARN SRA possède un rôle essentiel dans la régulation génique en tant que transcrit. Or, cet ARN possède de nombreux variants, résultats de l’épissage alternatif du transcrit
produisent la protéine SRAP57. En effet, ces évènements d’épissage restaurent le cadre de lecture avec
le codon STOP se trouvant dans le dernier exon de l’ARN. La particularité de SRAP est d’être capable de s’associer aux différents transcrits de SRA au niveau de la structure secondaire STR7 et ainsi d’entrer en compétition avec le facteur de transcription MyoD, essentiel pour la régulation de l’expression des gènes impliqués dans la myogenèse. De cette manière, SRAP régule la fonction non-codante du gène dont elle est issue57.
5. Tuna : un lncRNA conservé à deux visages
5.1. Tuna est conservé chez les vertébrés
Tuna (Tcl1 Upstream Neuron-Associated lncRNA) ou Tunar ou encore Megamind (linc-birc6) est un lncRNA identifié par Ulitsky et al. en 201149 chez le poisson zèbre (zebrafish). Ces
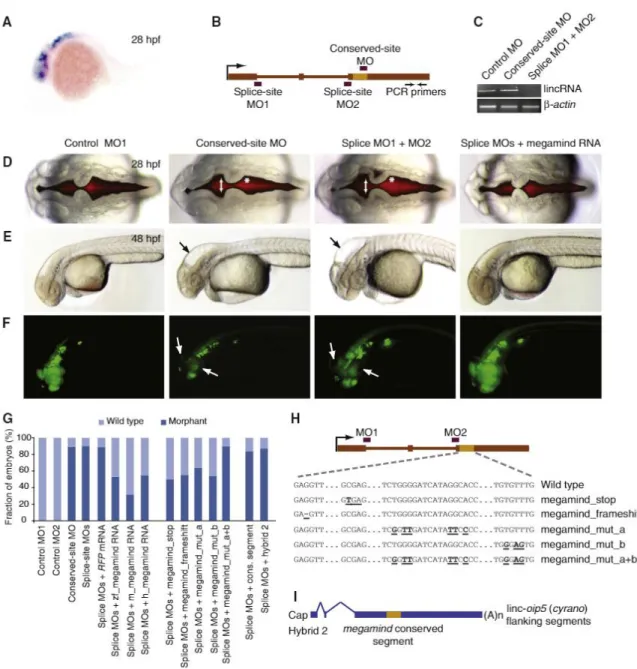
travaux visaient à identifier de nouveaux lncRNAs impliqués dans le développement embryonnaire et dont la fonction est conservée chez les vertébrés. Pour cela, Ulitsky et al. ont procédé à un filtrage des candidats en partant des régions riches en marques d’histones H3K4me3 (site d’initiation de la transcription) et H3K36me3 (élongation de la transcription) cartographiées par ChIP-Seq (séquençage des régions de chromatine associées aux histones isolées par immunoprécipitation) dans les embryons de poisson zèbre âgés de 24, 72heures et chez des individus adultes. Ces marques sont indicatrices de la transcription active des gènes, puisqu’elles permettent respectivement l’initiation de la transcription et marquent la progression de l’ARN polymérase II le long du brin d’ADN. Ces résultats ont été corrélés avec ceux obtenus à l’issue de 3P-Seq (Poly(A)-Position Profiling), méthode utilisée pour cartographier les sites de terminaison de la transcription. Ensemble, ces résultats ont été implémentés dans le processus de filtrage présenté en Figure 3 (extrait de Ulitsky et al., 2011). Ainsi, plus de 560 lncRNAs candidats, dont la séquence montre une certaine similarité avec l’humain et la souris, ont été identifiés49.
Parmi eux, Megamind (linc-birc6) est un transcrit de 2,4kb et 3 exons en antisens du gène birc6 chez le poisson zèbre. Au total, 75 séquences qui lui sont homologues ont été identifiés chez 45 vertébrés partageant une vaste région conservée à plus de 90%, de 97nt en 5’ du dernier exon. Bien que 3 homologues aient été détectés dans le poisson zèbre (dans l’intron du gène birc6 en antisens, dans la région intergénique des homologues bdkrb1/2 et près de homologues hhipl1), le lncRNA situé au locus du gène birc6 est le plus exprimé et a été choisi par Ulitsky et al. pour la suite des travaux, présentés ci-dessous49.
5.2. Tuna : un lncRNA impliqué dans l’embryogenèse
Suite à l’identification de Megamind (linc-birc6) dans les embryons de poisson zèbre, Ulitsky et al. ont poursuivi l’investigation sur le plan fonctionnel avec l’utilisation de MOs (Morpholinos) afin de perturber l’épissage de ce lncRNA lors du développement des yeux et du cerveau, où il est normalement exprimé. Cette technique se base sur l’utilisation d’oligonucléotides modifiés (cycles morpholines et liaisons phosphorodiamidates au lieu de désoxyriboses et liaisons phosphates) de 25 bases, complémentaires à leur cibles ARN. Ces molécules agissent par encombrement stérique et entre en compétition avec les différents interacteurs de leur ARN cible. Ils sont généralement utilisés in vivo par injection.
L’injection de 3 différents MOs, dirigés vers les sites d’épissage de l’exon 1 et 3 ainsi que la région conservée identifiée précédemment, a provoqué l’apparition de défauts chez les embryons, au niveau de la morphologie du ventricule, une expansion du mésencéphale, une disparition de la région charnière, la contraction du ventricule pro-encéphalique ainsi que des défauts oculaires notamment au niveau de la rétine. En revanche, les 2 MOs contrôles, comportant des mutations ou dirigé vers une région non-conservée de Megamind, n’ont pas affecté le développement des embryons. Le sauvetage par co-injection, avec les MOs, de Megamind (transcrit in vitro) du poisson zèbre, de la souris et de l’humain a partiellement rétabli le phénotype embryonnaire sauvage (50-70% des embryons sont normaux), prouvant l’importance du maintien de la structure exonique et de la conservation dans la fonction de ce lncRNA (Figure 4). Ils montrent également que la perturbation d’un potentiel ORF pour un peptide de 49 acides-aminés, présent dans la région la plus conservée (introduction d’un codon STOP prématuré, frameshift - délétion d’un nucléotide afin de décaler un ORF) n’affecte pas le sauvetage observé par la co-injection de MOs et de Megamind, et concluent que la probabilité pour que ce peptide soit exprimé est très faible au cours du développement neuronal du poisson zèbre. Cependant, la mutation de 6 acides-aminés conservés du potentiel peptide, aboli le sauvetage du phénotype sauvage, indiquant que la conservation en séquence de Megamind est essentielle à sa fonction (Figure 4). Finalement, l’injection de la région conservée ou d’un hybride d’ARN contenant la région conservée ne permet pas le sauvetage, démontrant que l’intégrité de ce lncRNA est également nécessaire à son rôle dans le développement du cerveau pendant l’embryogenèse (Figure 4).
5.3. Tuna : un lncRNA impliqué dans le contrôle de la pluripotence
(Megamind) qu’ils renomment Tuna (Tcl1 Upstream Neuron-Associated lncRNA) parmi les 20 candidats requis pour le maintien de la pluripotence des mESCs, qu’ils identifient suite à un criblage par shRNAs (short hairpin RNAs) et tests phénotypiques (aspect des cellules, expression du reporteur GFP (Green Fluorescent Protein), activité de la phosphatase alcaline). Les cellules souches embryonnaires utilisées pour ces tests possèdent un transgène comportant le promoteur du gène Oct4 et le gène codant pour le protéine fluorescente GFP, qui n’est alors exprimée que lorsque les cellules expriment le facteur de transcription OCT4, c.à.d. lorsqu’elles sont pluripotentes. La perte de l’expression de la protéine reportrice indique la perte du phénotype pluripotent des cellules.
Ainsi, l’équipe de Lin et al. identifie les 2 isoformes murines de Tuna, actuellement référencées dans la base de données Ensembl Genome Browser, grâce aux techniques de RACE (Rapid Amplification of cDNA Ends) et Northen Blot (identification de transcrits sur gel par amorces complémentaires radiomarquées). D’autre part, ils localisent Tuna à la fois dans le noyau et dans le cytoplasme par qRT-PCR dans les fractions cellulaires de mESCs. Toutefois, ils confirment le résultat de Ulitsky et al. (2011) et démontrent, par insertion de la séquence de eGFP dans l’ORF le plus probable de Tuna et par observation de la fluorescence (verte) au microscope, que Tuna ne possède pas de potentiel codant. Enfin, il est important de noter que l’ORF étudié par Lin et al. ne se situe que partiellement dans la région conservé précédemment mentionnée. Ainsi, il se pourrait qu’un autre ORF, fonctionnel, se trouve dans cette région hautement conservée.
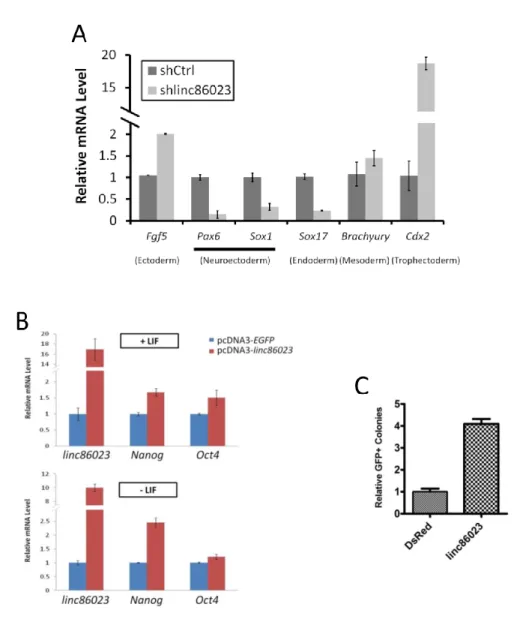
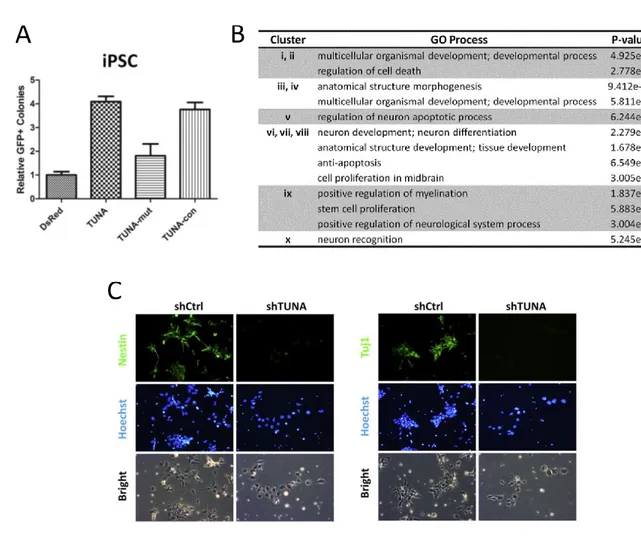
D’un point de vue fonctionnel, Lin et al. montrent que la perturbation de l’expression de Tuna affecte les marqueurs de différenciation du neuro-ectoderme (déplétion par shRNA) (Figure 5.A) et les niveaux de Nanog et Oct4 (surexpression ectopique) (Figure 5.B). Ces résultats sont cohérents avec les travaux de Ulitsky et al. (2011), mais Lin et al. démontrent également une autre facette de ce lncRNA, dans le contexte de la pluripotence des cellules souches. En effet, Tuna augmente également le nombre de cellules reprogrammées (iPSCs) à partir de MEFs (Mouse Embryonic Fibroblasts) lorsqu’il est surexprimé aux côtés des facteurs de reprogrammation (Figure 5.C).
Finalement, Lin et al. remarquent également l’étonnante conservation (souris, humain et poisson zèbre) de Tuna au début du 3ème exon de l’ARN sur une région qu’ils étendent à 200nt. Ils montrent
que cette région est suffisante pour récapituler l’effet de Tuna pleine longueur lors de la reprogrammation (Figure 6.A), démontrant une nouvelle fois l’importance de cette séquence dans la biologie de ce lncRNA.
5.4.
Tuna : un lncRNA impliqué dans la différenciation neuronale
De la même manière que les travaux de 2011, Lin et al. documentent la forte l’expression de Tuna dans le système nerveux central (CNS) des embryons de souris, conservée chez le poisson zèbre et chez l’humain. Consistants avec les résultats de Ulitsky et al., ils montrent que Tuna est spécifiquement exprimé pendant la différenciation neuronale de mESCs et que la déplétion de Tuna affecte négativement l’expression des gènes propres aux neuro-progéniteurs. Plus généralement, ils montrent par immunofluorescence que la déplétion de Tuna empêche la différentiation des mESCs (Figure 4.B) et par séquençage d’ARN qu’elle impacte de nombreux processus cellulaires impliqués dans la différenciation neurale (Figure 4.C). Ces résultats démontrent que Tuna possèdent deux rôles distincts et antagonistes, dépendants du type cellulaire.
Finalement, Lin et al. proposent un mécanisme d’action dans les mESCs, selon lequel Tuna interagirait avec les RBPs (RNA Binding Proteins) NCL, hnRNP-K et PTBP1 (expériences de co-immunoprécipitations) ciblant les promoteurs soit des gènes de pluripotence soit des gènes de différenciation neuronale pour activer leur transcription. Enfin, ils montrent la corrélation inversement proportionnelle entre les niveaux d’expression de TUNA et les grades de la maladie de Huntington (pathologie neurodégénérative impliquant la mort des cellules neuronales), faisant de ce lncRNA une potentielle cible thérapeutique.
6. Objectifs de recherche
Le laboratoire du Dr. Samer HUSSEIN vise à comprendre les fonctions et les mécanismes moléculaires de longs ARNs non-codants (lncRNAs) impliqués dans la régulation génique lors des changements d’états cellulaires. Pour ce faire, le laboratoire s’intéresse aux différentes interactions dans lesquelles peuvent être impliqués les lncRNAs, à savoir : ADN, ARN et ARN-protéines.
Les résultats présentés s’inscrivent dans le projet de recherche s’intéressant à l’ARN Tuna. En effet, celui-ci a la particularité d’intervenir dans deux processus cellulaires contradictoires, le maintien de la pluripotence des cellules souches de l’embryon et leur différenciation vers le phénotype neuronal. Si tel est bien le cas, cela suggèrerait que ce lncRNA possède deux mécanismes moléculaires distincts et dépendants du type cellulaire, impliquant probablement des partenaires d’interaction, protéines, ARN et/ou ADN et des cibles différentes. De plus, l’expression de Tuna est conservée chez les vertébrés dans le SNC (système nerveux central) et dans les ESCs, au moins chez l’humain et la
l’ensemble des vertébrés. En considérant les travaux de Ultisky et al. (2011) et Lin et al. (2014), et le fait que ce type de conservation est généralement lié à la fonction des lncRNAs, cette région représente certainement une caractéristique liée au mécanisme moléculaire de Tuna.
De ce fait, les objectifs du projet de recherche portant sur l’ARN Tuna visent à :
- Caractériser les séquences conservées de Tuna chez les ESCs et les neurones dérivés d’ESCs - Identifier le rôle fonctionnel des régions conservées de Tuna dans le contexte de la
pluripotence
A long terme, ce projet s’intéressera à identifier et caractériser d’autres lncRNAs impliqués dans les mêmes processus cellulaires que Tuna et d’en évaluer l’intérêt dans certains contextes physiologiques et pathologiques, tels que les maladies neurodégénératives, les désordres du développement et/ou les cancers du cerveau (glioblastomes par exemple).