Pépite | Diversité du récepteur au froid et au menthol, TRPM8 : du gène aux fonctions
Texte intégral
(2) HDR de Gabriel Bidaux, Lille 1, 2015. © 2015 Tous droits réservés.. doc.univ-lille1.fr.
(3) HDR de Gabriel Bidaux, Lille 1, 2015. . © 2015 Tous droits réservés.. doc.univ-lille1.fr.
(4) HDR de Gabriel Bidaux, Lille 1, 2015. © 2015 Tous droits réservés.. doc.univ-lille1.fr.
(5) HDR de Gabriel Bidaux, Lille 1, 2015. Année Universitaire: 2014-2015. N° d’ordre : 41754. HABILITATION A DIRIGER DES RECHERCHES Discipline: Sciences de la vie et de la santé. DIVERSITÉ DU RÉCEPTEUR AU FROID ET AU MENTHOL, TRPM8 : DU GÈNE AUX FONCTIONS Présentée publiquement le 18 juin 2015 à Lille par,. Dr. Gabriel BIDAUX Laboratoire de Physique des Lasers, Atomes et Molécules. Devant la commission d’examen: Directeur de Recherche Dr. Morad ROUDBARAKI, Professeur de l’université Lille1, INSERM U1003, Lille Rapporteurs Dr. Olivier BENSAUDE, Directeur de Recherche INSERM, UMR8197, IBENS, Paris Dr. Fabien VAN COPPENOLLE, Professeur de l’université Lyon 1, INSERM U1060, Lyon Dr. Thierry CAPIOD, Chargé de Recherche INSERM, HDR, INSERM U1151– INEM, Paris Examinateurs Dr. Natalia PREVARSKAYA, Professeur de l’université Lille1, INSERM U1003, Lille Dr. Marc LEFRANC, Professeur de l’université Lille1, PHLAM, UMR8523, Lille Dr. Laurent HÉLIOT, Ingénieur de recherche CNRS, HDR, PHLAM, UMR8523, Lille. © 2015 Tous droits réservés.. doc.univ-lille1.fr.
(6) HDR de Gabriel Bidaux, Lille 1, 2015. © 2015 Tous droits réservés.. doc.univ-lille1.fr.
(7) HDR de Gabriel Bidaux, Lille 1, 2015. . À ma femme, Anne-Sophie, À mes enfants, Charly et Camille,. Habilitation à Diriger des Recherches – Gabriel Bidaux © 2015 Tous droits réservés.. doc.univ-lille1.fr.
(8) HDR de Gabriel Bidaux, Lille 1, 2015. © 2015 Tous droits réservés.. doc.univ-lille1.fr.
(9) HDR de Gabriel Bidaux, Lille 1, 2015. . REMERCIEMENTS Je remercie tout d’abord le Prof. Natalia Prevarskaya pour m’avoir accueilli dans son laboratoire de 2002 à 2013 et m’avoir toujours soutenu dans mes recherches en trouvant ou m’aidant à trouver des financements pour les réaliser. Je remercie le Prof. Morad Roudbaraki pour ses conseils, sa patience et l’amitié qu’il m’a témoigné durant ces années. Merci aussi à toi d’avoir accepté d’être le directeur de recherche de cette HDR et d’y avoir consacré du temps. Je remercie le Dr. Laurent Héliot pour m’avoir intégré à son équipe pendant 2 années et m’avoir laisser une grande liberté pour développer nos travaux dans le cadre fixé par le projet. Je le remercie aussi de m’avoir laissé continuer mon travailler sur TRPM8 et m’avoir permis de réaliser de nombreuses collaboration. Pour terminer je le remercie pour son amitié et ses précieux conseils. Je remercie mes collègues les Drs. Olivier Bensaude, Thierry Capiod et le Prof. Fabien Van Coppenolle pour avoir accepté d’être les rapporteurs de cette HDR. Je remercie le Prof Marc Lefranc, spécialiste de la physique non linéaire des systèmes biologiques, d’avoir accepté de participer à ce jury. Merci à tous les collègues et les étudiants avec qui j’ai pu collaborer pendant toutes ces années, que ce soit au sein du laboratoire PHYCELL ou BCF, mais aussi à Brest, Rennes, Clermont-Ferrand, Paris, Dublin, Londres, Kiev… Enfin, je remercie chaleureusement les Profs Fabien Van Coppenolle et Michel Ovize, pour leur confiance et leur accueil au sein de l’équipe cardioprotection du laboratoire CarMeN à Lyon.. Habilitation à Diriger des Recherches – Gabriel Bidaux © 2015 Tous droits réservés.. doc.univ-lille1.fr.
(10) HDR de Gabriel Bidaux, Lille 1, 2015. © 2015 Tous droits réservés.. doc.univ-lille1.fr.
(11) HDR de Gabriel Bidaux, Lille 1, 2015. . RÉSUMÉ Le récepteur au froid et au menthol, TRPM8, est une protéine-canal fonctionnant comme un senseur thermodynamique et est responsable de la détection du froid de part son expression dans les neurones innervant l’épiderme. Après avoir cloné 35 ARN alternatifs et ARN épissés issus du gène trpm8, nous avons réalisé un travail de caractérisation moléculaire, biophysique et cellulaire de ces variantes du récepteur au froid. Nous avons, en parallèle, essayé de comprendre leurs rôles dans les grandes fonctions physiologiques comme l’homéostasies de l’épiderme, la réponse au froid dans des cellules germinales ou encore le métabolisme. Le problème intrinsèque des tentatives d’association des niveaux moléculaires et cellulaires avec la physiologie est qu’il est facile de trouver des corrélations mais techniquement beaucoup plus complexe de démontrer des liens de causalité. Or, la compréhension des mécanismes moléculaires, hormis la beauté de la compréhension de l’infiniment petit, n’a de sens en biologie que lorsqu’on arrive à expliquer les effets macroscopiques. C’est dans le respect de cette démarche, que ces 2 dernières années, j’ai acquis des compétences en biophysique et en microscopie photonique avancée. En effet, le développement des outils de fluorescence permet maintenant d’envisager l’observation quantitative de mécanismes moléculaire en cellule vivante unitaire, mais aussi dans l’organisme. L’association du génie génétique, de la photonique et des modèles animaux permet donc d’envisager le développement de la physiologie moléculaire dans les prochaines années.. Habilitation à Diriger des Recherches – Gabriel Bidaux © 2015 Tous droits réservés.. doc.univ-lille1.fr.
(12) HDR de Gabriel Bidaux, Lille 1, 2015. Habilitation à Diriger des Recherches – Gabriel Bidaux © 2015 Tous droits réservés.. doc.univ-lille1.fr.
(13) HDR de Gabriel Bidaux, Lille 1, 2015. . TABLE DES MATIÈRES . Habilitation à Diriger des Recherches – Gabriel Bidaux © 2015 Tous droits réservés.. doc.univ-lille1.fr.
(14) HDR de Gabriel Bidaux, Lille 1, 2015. TABLE DES MATIERES GLOSSAIRE ................................................................................................................................................ 1 CURRICULUM VITAE I. CURRICULUM VITAE -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 6 DIPLOMES -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 6 EXPERIENCE PROFESSIONNELLE -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 7 COMPÉTENCES TECHNIQUES -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 8 II. PUBLICATIONS -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 9 . ! ! !. ARTICLES ET REVUES -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 9 CHAPITRES DE LIVRES -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 13 III. COMMUNICATION -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 14 . ! !. ACTIVITÉS DE STRUCTURATION DE LA RECHERCHE I. ENSEIGNEMENT -‐ FORMATION -‐ ENCADREMENT -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 18 I.1. ENSEIGNEMENT -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 18 I.2. FORMATION -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 18 I.3. ENCADREMENT -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 18 II. SUBVENTIONS -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 19 III. TÂCHES COLLECTIVES -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 21 III.1. SOCIÉTÉS SAVANTES -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 21 III.2. PEER-‐REVIEWER -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 21 III.3. ÉVALUATEUR DE PROJETS SCIENTIFIQUES -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 21 III.4. GESTION DE BUDGETS -‐ COMMANDES -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 21 III.5. ORGANISATION – DÉVELOPPEMENT DE PLATEAUX DE BIOLOGIE MOLÉCULAIRE -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 21 ACTIVITÉS DE RECHERCHE I. AVANT-‐PROPOS -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 24 II. GÉNÉRALITÉS -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 25 II.1. LES CANAUX TRP ET LE’ION CALCIQUE -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 25 II.1.1. LE CALCIUM -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 25 Compartimentation de l’ion calcique -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 25 Homéostasie et signal calcique -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 25 Calciosome -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 25 II.1.2. LES CANAUX TRP, thermo-‐senseurs et le récepteur au froid : TRPM8 -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 26 . ! ! !. Historique : de la drosophile à l’homme -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 26 Classification et structures communes -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 26 Thermorécepteurs -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 28 II.2. P-‐TEFb ET LA RÉGULATION DE LA TRANSCRIPTION. -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 32 . ! ! !. II.2.1. SÉQUENCE DE LA TRANSCRIPTION -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 32 II.2.2. P-‐TEFb -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 33 II.2.3. DYNAMIQUE DES PROTÉINES NUCLÉAIRES -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 34 . © 2015 Tous droits réservés.. doc.univ-lille1.fr.
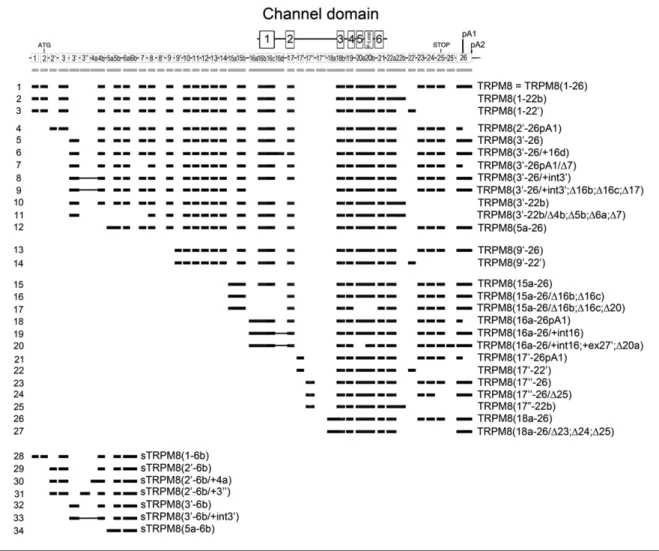
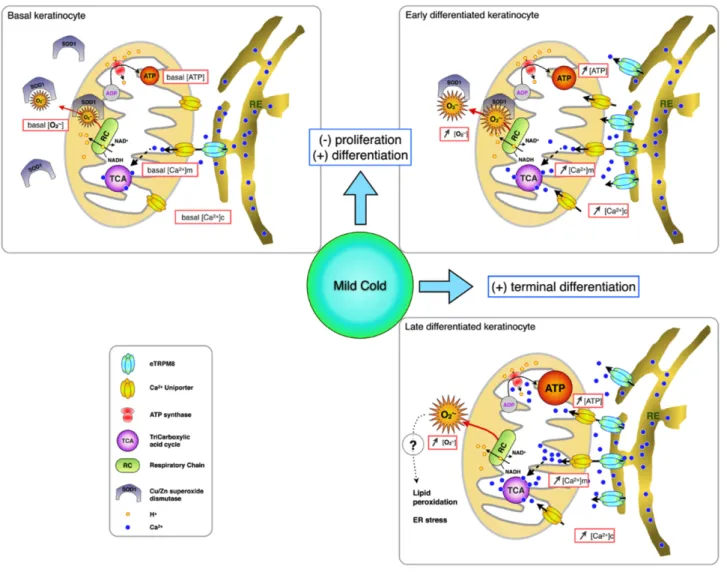
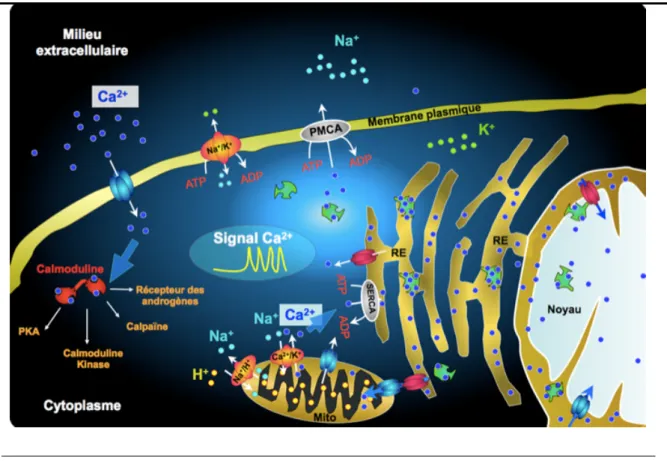
(15) HDR de Gabriel Bidaux, Lille 1, 2015. TABLE DES MATIERES II.3. INTRODUCTION AUX TECHNIQUES DE PHOTONIQUES AVANCÉES -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 35 II.3.1. TIME DOMAIN – FLUORESCENCE LIFETIME IMAGING MICROSCOPY (TD-‐FLIM) -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 35 II.3.1. FÖRSTER RESONNANCE ENERGY TRANSFERT (FRET) -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 36 II.3.2. FLUCTUATIONS (FLUORESCENCE) CORRELATION SPECTROSCOPY (FCS) -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 37 II.3.1. IMAGE CORRELATION SPECTROSCOPY (ICS) -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 38 III. RÉSULTATS -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 39 III.1. BIOLOGIE. Le récepteur au froid, TRPM8 : du gène à l’organisme. -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 39 III.1.1. TRPM8: ÉVOLUTION & DIVERSITÉ -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 39 III.1.2. TRPM8 : RELATION STRUCTURE-‐FONCTION DU PORE IONIQUE -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 47 III.1.3. DIVERSITÉ DES ISOFORMES DE TRPM8 DANS LA PROSTATE -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 50 RÉSUMÉ DES TRAVAUX DE THÈSE ET DE POST-‐DOCTORAT (2003-‐2009) -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 51 PETITES ISOFORMES DE TRPM8 -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 52 TRPM8-‐36 & MAM -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 56 III.1.4. LE RÉCEPTEUR AU FROID MODULE L’HOMEOSTASIE DE L’ÉPIDERME -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 58 . ! ! !. III.1.5. TRPM8 PRÉVIENT L’OXYDATION DES CELLULES GERMINALES INDUITE PAR LE FROID -‐-‐-‐-‐-‐-‐-‐-‐-‐ 63 III.1.6. ISOFORMES DE TRPM8 & DÉPENSE ÉNERGÉTIQUE -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 66 III.2. INGENIERIE : Développement de méthodes d’analyse d’image pour la biologie quantitative. -‐-‐ 71 III.2.1. CORRÉLATION TEMPORELLE DE DÉTÉCTION DE PHOTONS : CARACTÉRISATION DE L’OLIGOMERISATION DE PROTÉINES FLUORESCENCES PAR FCS -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 71 III.2.2. OPTIMISATION DU COUPLE DONNEUR-‐ACCEPTEUR POUR LE FRET -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 79 III.2.3. SEGMENTATION D’IMAGE ET CORRÉLATION D’IMAGE POUR LA CARACTÉRISATION DE NANODOMAINES DE FRET -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 83 CONCLUSION & PERSPECTIVES I. CONCLUSION -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 88 II. PERSPECTIVES : ICHÉMIE-‐REPERFUSION CARDIAQUE -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 89 II.1. RÔLE DES NANODOMAINS RE-‐MITOCHONDRIE DANS L’IRC -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 89 II.2. NANOSCOPY, MICROSCOPIE 3D ET ENDOSCOPIE -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 90 RÉFÉRENCES BIBLIOGRAPHIQUES ............................................................................................................ 93 ANNEXES I. ARTICLE 3 -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 102 II. ARTICLE 12 -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 122 III. ARTICLE 13 -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 143 IV. ARTICLE 15 -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 165 V. ARTICLE 16 -‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐-‐ 199 . Habilitation à Diriger des Recherches – Gabriel Bidaux © 2015 Tous droits réservés.. doc.univ-lille1.fr.
(16) HDR de Gabriel Bidaux, Lille 1, 2015. . © 2015 Tous droits réservés.. doc.univ-lille1.fr.
(17) HDR de Gabriel Bidaux, Lille 1, 2015. GLOSSAIRE . Habilitation à Diriger des Recherches – Gabriel Bidaux © 2015 Tous droits réservés.. 1 doc.univ-lille1.fr.
(18) HDR de Gabriel Bidaux, Lille 1, 2015. GLOSSAIRE AA : acides aminés APD : Avalanche Photo Diode ARN POL II : ARN polymérase II BAT : Brown Adipocyte Tissu Ca2+ : ion calcique cMOS : complementary Metal Oxide Semi-conductor CspA : Cyclosporine A CT1 : Cycline T1 CTD : Carboxy-terminal Domain E(FRET) : Efficacité de FRET. ε : brillance moléculaire apparente (nombre de photons détectés par seconde et par molécule) Ea : Énergie d’activation EMCCD : Electron Multiplying Charge Couplde Device eTRPM8 : epidermal TRPM8 FCCS : Fluorescence Cross Correlation Spectroscopy FCS : Fluorescence Correlation Spectroscopy FRET : Förster Resonnance Energy Transfert GPx : Glutathione Peroxidase Gτ : fonction d’autocorrélation Hexim : Hexamethylene-bis-acetamide-inducible HSF1 : Heat Shock Factor 1 HSPA2 : Heat Shock Protein A2 ICS : Image Correlation Spectroscopy IDH : Isocitrate Dehydrogenase INV : Involucrin IR : Ischémie-Reperfusion IRC : Ischémie- Reperfusion Cardiaque IRF : Instrument Response Function K10 : Keratin 10 KO : Knock-Out Larp7 : La RNA-binding domain of La-Related protein 7 Ma : Million d’années MAM : Mitochondria Associated Membranes MAM : Mitochondria-Associated Membranes mEGFP : monomeric Enhanced Green Fluorescent Protein mTurq2 ou mT2 : monomeric Turquoise protein, seconde version NELF : Positive Elongation Factor P-loop : hélice alpha formant la paroi interne du pore ionique d’un canal TRP 2 © 2015 Tous droits réservés.. doc.univ-lille1.fr.
(19) HDR de Gabriel Bidaux, Lille 1, 2015. GLOSSAIRE P-TEFb : Positive Transcription Elongation Facteur b P-TEFb : Positive Transcription Elongation Factor PALM : Photo-Activated Localization Microscopy PB : Photon Burst (bouffé de photons) PCH : Photon Counting Histogramm PCR : Polymerase Chain Reaction PMT : PhotonMultiplier Tube PO : probabilité d’ouverture d’un canal ionique PSA : Prostate Specifique Antigene PSF : Point Spread Function PTP : Pore de perméabilité de transition Q(10) : mesure la variation de vitesse d’un réaction chimique suite à une variation de 10°C de la température environnante. R0 : rayon de Förster d’un couple donneur-accepteur de fluorescence RACE : Rapide Amplification of cDNA-ends RE : Réticulum Endoplasmique RER : Respiratory Exchange Ratio ROS : Reactive Oxygen Species Rpb1 : large sous-unité de l’ARN polymérase II SO : SuperOxide SOD : SuperOxide Dismutase SPAD : Single Photon Avalanche Photo Diode STED : STimulated Emisson Depletion SYFP2 : Super Yellow Fluorescent Protein, version 2 TBP : TATA-Binding Protein TCSPC : Time Correlated Single Photon Counting TD-FLIM : Time Domain – Fluorescence Lifetime Imaging Microscopy TFII (A/D) : Transcription Factor II sous unité A ou D TG : TransGlutaminase TRP : Transient Receptor Potential UCP1,2,3 : UnCoupling Proteins 1,2 et 3 UTR : UnTranslated Region Vf : volume focal WAT : White Adipocyte Tissu. Habilitation à Diriger des Recherches – Gabriel Bidaux © 2015 Tous droits réservés.. 3 doc.univ-lille1.fr.
(20) HDR de Gabriel Bidaux, Lille 1, 2015. . 4 © 2015 Tous droits réservés.. doc.univ-lille1.fr.
(21) HDR de Gabriel Bidaux, Lille 1, 2015. CURRICULUM VITAE . Habilitation à Diriger des Recherches – Gabriel Bidaux © 2015 Tous droits réservés.. 5 doc.univ-lille1.fr.
(22) HDR de Gabriel Bidaux, Lille 1, 2015. CURRICULUM VITAE I.. CURRICULUM VITAE . Gabriel BIDAUX, Ph.D. Chercheur en Biologie moléculaire, cellulaire & Biophysique. Marié, 2 enfants Adresse personnelle : 1, rue Pierre de Coubertin 59310 Orchies Situation professionnelle : Chercheur associé (CDD Université Lille1) dans l’équipe Biphotonique Fonctionnelle Cellulaire Laboratoire de Physiques des Lasers, Atomes et Molécules (PHLAM) Equipe Biphotonique Fonctionnelle Cellulaire UMR8523 CNRS, Parc de la Haute Borne 50 av de Hallay BP70478 59658 Villeneuve d’Ascq, France gabriel.bidaux@univ-lille1.fr gabriel.bidaux@gmail.com. DIPLOMES 2003 -‐ 2006 Doctorat en science de la vie et de la santé, spécialité biologie moléculaire et cellulaire. Titre de la thèse : rôle du canal TRPM8 dans la physiopathologie de la prostate humaine. Tuteur: Pr N. Prevarskaya. (Laboratoire de physiologie cellulaire, INSERM U800, Villeneuve d’Ascq, France).. 2002 -‐ 2003 Master recherche (DEA) science de la vie et de la santé Option biologie cellulaire et moléculaire ; Université de Lille I (USTL) Sujet : rôle du canal TRPM8 dans la physiopathologie de la prostate humaine. Tuteur: Pr N. Prevarskaya et Pr M. Roudbaraki. (Laboratoire de physiologie cellulaire, INSERM U800, Villeneuve d’Ascq, France).. 2001 -‐ 2002 Maîtrise de Biologie Cellulaire et moléculaire, Université de Lille I (USTL) 6 © 2015 Tous droits réservés.. doc.univ-lille1.fr.
(23) HDR de Gabriel Bidaux, Lille 1, 2015. CURRICULUM VITAE EXPERIENCE PROFESSIONNELLE Juillet 2015 -‐ Chercheur associé. Laboratoire INSERM U1060, CARMEN; équipe 5: cardioprotection. Projet: Les jonctions ER-mitochondrie des cardiomyocytes et leurs modifications lors de l’ischémiereperfusion. http://carmen.univ-lyon1.fr http://carmen.univ-lyon1.fr/eq5/equipe5_1.php. 2013 -‐2015 Chercheur associé. Université Lille 1, Equipe Biphotonique Fonctionnelle Cellulaire, Laboratoire de Physique des Lasers, Atomes et Molécules (PhLAM). Projet: Dynamique d’assemblage du complexe P-TEFb avec le complexe d’élongation de la transcription, RNA POL II, en cellules vivantes.. http://www.phlam.univ-lille1.fr http://nonlineaire.univ-lille1.fr/SNL/. 2010 -‐ 2013 Chercheur associé. Université Lille 1, Laboratoire de physiologie cellulaire, INSERM U1003. Projet 1: Rôle des isoformes du récepteur au froid TRPM8 dans l’homéostasie de l’épiderme. Projet 2: Contrôle du métabolisme et de la thermogenèse par les isoformes du canal TRPM8 chez la souris. http://www.phycell.univ-lille1.fr/. 2007 -‐ 2009 Chercheur Post-doctorant. Financé par un projet blanc de l’Agence Nationale de Recherche Française (ANR). Université Lille 1, Laboratoire de physiologie cellulaire, INSERM U1003. Projet: Rôle du canal TRPM8 dans la prostate et les testicules implication dans la fertilité? Responsable scientifique: Pr Natalia. PREVARSKAYA. http://www.phycell.univ-lille1.fr/. Janvier 2003 -‐ février 2006 Chercheur Doctorant. Université Lille 1, Laboratoire de physiologie cellulaire, INSERM U1003. Projet: Rôle du canal TRPM8 dans la physiopathologie de la prostate. Responsable scientifique: Pr Natalia. PREVARSKAYA. http://www.phycell.univ-lille1.fr/ Habilitation à Diriger des Recherches – Gabriel Bidaux © 2015 Tous droits réservés.. 7 doc.univ-lille1.fr.
(24) HDR de Gabriel Bidaux, Lille 1, 2015. CURRICULUM VITAE COMPÉTENCES TECHNIQUES Techniques de biologie moléculaire: TA Cloning, protéines de fusion (pour FRET, étude de localisation), design de siRNA, vecteur shRNA, RTPCR, Real time PCR, Single Cell RT-PCR, RACE-PCR, mutagenèse dirigée, test d’activité promotrice, base en mRNAseq sur system SOLID 3D, formation sur les modifications du génome par TALEN, ZFN et CRISP9. Techniques de biologie cellulaire: Culture cellulaire : lignées cellulaires tumorales et culture primaires (épithéliales et musculaires lisse de la prostate, kératinocytes, hépatocytes et épithéliales de vessie). Transfection (phosphate de calcium, lipofection, nucleofector). Génération de lignée cellulaire stable ou inductible. Techniques de Biochimie: Western-blot, immunocytofluorescence, immunohistochimie et immunohistofluorescence (quadruple marquage) sur coupes congelées ou paraffinées, immunoprécipitation, cross-linking, test d'activité enzymatique (PLA2, Caspases, Déshydrogénases, ATP), biotinylation des protéines de surface, purification de protéines de surface , extraction de protéines recombinantes soluble ou membranaires à partir de modèle procaryote ou eucaryote, purification de microdomaines sur gradient d’iodoxanol (MAM, cavéoles). Imagerie: Microscopie photonique: « Spining Disk » et « Laser Scanning Confocal », multiphotonique (Zeiss LSM510, LSM700 et LSM780; Leica SP5). FRET: FRAP, sensitized emission, lambda FRET, TD FLIM (TCSPC) et FD FLIM. Imagerie de fluorescence du Calcium : Fluo-4, Rhod-2, Mag-fluo, biosenseurs Cameleon adressés dans les mitochondries, le RE, le Golgi ou le cytosol. Imagerie de fluorescence des DRO (dérivés actifs de l’oxygène): (Mitosox, BODPY C11, CellRox). Spectroscopie corrélative de fluorescence: FCS, FCCS, bases pour l’ICS et le TICS. Modèles Animaux Xénogreffes sous-cutanées de cellules tumorales, puis mesures de la croissance tumorale. Phénotypage de lignées de souris invalidées (KO). Bioinformatique. Maîtrise de la suite Office (Excel, Word, Powerpoint). Maitrise de solutions statistiques et graphiques (Graphpad, Origin, PRISM). Maîtrise de logiciels pour la biologie moléculaire (Vector NTI, SMART, Blast, Clustalw, APE, Snapgene...). Maîtrise des logiciels analyse d’image (ImageJ), Icy. Analyse de temps de vie : Symphotime (Picoquant), MAPI. Programmation : bases Matlabs (écriture de scripts simples, utilisation de scripts pré-écris) 8 © 2015 Tous droits réservés.. doc.univ-lille1.fr.
(25) HDR de Gabriel Bidaux, Lille 1, 2015. CURRICULUM VITAE PUBLICATIONS . II.. http://publicationslist.org/gabriel.bidaux https://www.researchgate.net/profile/Gabriel_Bidaux/ 24 articles, 3 revues, 1 chapitre de livre et 3 procédés de conférence publiés. 10 articles en révision/soumission/préparation h-index: 16 *: co-premier auteur. ¶: auteur référant.. ARTICLES ET REVUES 2015 1) Anne-sophie Borowiec, Benoit Sion, Frédéric Chalmel, Antoine Rolland, Loïc Lemonnier, Tatiana De Clerck, Alexandre Bokhobza, Sandra Derouiche, Etienne Dewailly, Christian Slomianny, Claire Mauduit, Mohamed Benhamed, Morad Roudbaraki, Bernard Jégou, Natalia Prevarskaya, and Gabriel Bidaux. Cold/menthol channels initiate the cold shock response and protect germ cells from cold shock mediated oxydation. In preparation. 2) Laurent Héliot, Mariano Gonzalez-Pisfil, Mélanie Henry, Corentin Le Nezet, Olivier Bensaude, Bernard Vandenbunder, and Gabriel Bidaux. A 2-steps model for the dynamic binding of PTEFb on the transcriptional paused complex. In preparation. 3) Samuel Blanquart, Anne-sophie Borowiec, Philippe Delcourt, Martin Figeac, Morad Roudbaraki, Natalia Prevarskaya and Gabriel Bidaux. Evolution of human TRPM8 isoforms. In preparation. 4) Charlotte Dubois, Artem Kondratsky, Gabriel Bidaux, Gilbert Lepage, Robert-Allain Toillon, Christian Slomianny, Eric Vancauwenberghe, Pascal Mariot, Morad Roudbaraki, Dominique Tierny, Jesper Vuust Moller, Natalia Prevarskaya, Guido Kroemer and Fabien Vanden Abeele. Dissecting Apoptosis Mediated by Mitochondria Calcium Overload Reveals a Tight Control of Mitochondrial Depolarization and Fission by Autophagy critical for Priming Cancer Cells to Chemotherapy. Soumis CELL. 5) Gabriel Bidaux*, Miriam Sgobba*, Loic Lemonnier, Mark SP Sansom, Alexander V Zholos, Shozeb Haider. Functional and Modeling Studies of the Transmembrane Region of TRPM8 channel. Minor revision in Biophysical Journal. 6) Gabriel Bidaux¶, Anne-Sophie Borowiec, Dmitri Gordienko, George Shapovalov, Benjamin Beck, Loïc Lemonnier, Matthieu Flourakis, Matthieu Vandenberghe, Christian Slomianny, Etienne Dewailly, Philippe Delcourt, Emilie Desruelles, Abigaël Ritaine, Renata Polakowska, Jean Lesage, Mounia Chami, Roman Skryma and Natalia Prevarskaya¶. Molecular identity of the Habilitation à Diriger des Recherches – Gabriel Bidaux © 2015 Tous droits réservés.. 9 doc.univ-lille1.fr.
(26) HDR de Gabriel Bidaux, Lille 1, 2015. CURRICULUM VITAE mild cold-transducer in epidermal homeostasis. Minor revision in PNAS. 7) Gabriel Bidaux¶, Loïc Lemonnier, Anne-Sophie Borowiec, Fabien Vanden Abeele, Gilbert Lepage, Etienne Dewailly, Christian Slomianny, Bonnal Jean-Louis, Mauroy Brigitte and Natalia Prevarskaya. Targeting TRPM8 regulatory subunits in prostate cancer: towards a new drug target? In preparation for submission in Cancer Research. 8) Gabriel Bidaux*,¶, Dmitri Gordienko*, Georges Shapovalov, Anne-sophie Borowiec, Matthieu Flourakis, Etienne Dewailly, Christian Slomianny and Natalia Prevarskaya¶. Novel archetype of TRPM8 channel characterized in mitochondria-associated membranes of prostate cells. Submitted in Journal of Cell Biology. 9) George Shapovalov, Gabriel Bidaux, Abigaël Ritaine, Christian Slomianny, Anne-Sophie Borowiec, Roman Skryma, Natalia Prevarskaya. Patch clamping ion channels in native subcellular membranes. Submitted in Nature methods. 10) Sandra Derouiche, Pascal Mariot, Marine Warnier, Gabriel Bidaux, Eric Vancauwenberghe, Pierre Gosset,Brigitte Mauroy, Jean-Louis Bonnal, Christian Slomianny, Philippe Delcourt, Gilbert Lepage, Natalia Prevarskaya and Morad Roudbaraki. Antibacterial agent Triclosan promotes prostate cancer progression via TRPA1 channel activation in stromal cells and VEGF secretion. In revision in Cancer Prevention Research.. 2014 11) D. Gkika, L Lemonnier, G Shapovalov, D Gordienko, C Poux, M Bernardini, A Bokhobza, G Bidaux, C Degerny, K Verreman, B Guarmit, M Benahmed, Y de Launoit, R Bindels, A Fiorio Pla and N Prevarskaya. TRPM8 channel-associated factors are a novel protein family that regulates TRPM8 trafficking and activity. Journal of Cell Biology, accepted 12-01-2014 (ISI 5year Impact Factor : 10.123) 12) C Dubois, F Vanden Abeel, V Lehen'kyi, D Gkika, B Guarmit, G Lepage, C Slomianny, AS Borowiec, G Bidaux, M Benahmed, Y Shuba, N Prevarskaya. Remodeling of Channel-Forming ORAI Proteins Determines an Oncogenic Switch in Prostate Cancer. Cancer cell. 2014 Jul 14;26(1):19-32. (ISI 2012, 5-year Impact Factor : 27,059). 2013 13) Anne-Sophie Borowiec, Gabriel Bidaux, Natascha Pigat, Vincent Goffin, Sophie Bernichtein, and Thierry Capiod. Calcium channels, external calcium concentration and cell proliferation. Review. European Journal of Pharmacology, accepted 11-20-2013. (ISI 2012, 5-year Impact Factor : 2,778) 14) Anne-Sophie Borowiec, Gabriel Bidaux, Rachida Tacine, Pauline Dubar, Philippe Delcourt, Olivier Mignen, and Thierry Capiod. Are Orai1 and Orai3 channels more important than calcium influx for cell proliferation? BBA- Molecular Cell Research, accepted 11-27-13. (ISI 2012, 5-year Impact Factor : 4,947) 15) Anne-sophie Borowiec, Philippe Delcourt, Etienne Dewailly, and Gabriel Bidaux¶. Multifactorial optimization of in vitro keratinocytes induction of differentiation. PLos One. 2013 10 © 2015 Tous droits réservés.. doc.univ-lille1.fr.
(27) HDR de Gabriel Bidaux, Lille 1, 2015. CURRICULUM VITAE Oct 7;8(10):e77507. (ISI 2012, 5-year Impact Factor : 4,244). 2012 16) Gabriel Bidaux*,¶, Benjamin Beck, Alexander Zholos, Dmitri Gordienko, Loic Lemonnier, Matthieu Flourakis, Morad Roudbaraki, Anne-Sophie Borowiec, Jose Fernández, Philippe Delcourt,. Gilbert. Lepage,. Yaroslav. Shuba,. Roman. Skryma,. Natalia. Prevarskaya. (2011) Regulation of transient receptor potential melastatin 8 (TRPM8) channel activity by its short isoforms. J. Biol. Chem. Accepted for publication 22th November. (ISI 5-year Impact Factor : 5,498) 17) Jose A Fernández, Roman Skryma, Gabriel Bidaux, Karl L Magleby, C Norman Scholfield, J Graham McGeown, Natalia Prevarskaya, Alexander V Zholos (2011). Short isoforms of the cold receptor TRPM8 inhibit channel gating by mimicking heat action rather than chemical inhibitors. J. Biol. Chem. Accepted for publication 22th November. (ISI 5-year Impact Factor : 5,498). 2011 18) J A Fernández, R Skryma, G Bidaux, K L Magleby, C N Scholfield, J G McGeown, N Prevarskaya, A V Zholos (2011). Voltage- and cold-dependent gating of single TRPM8 ion channels. J Gen Physiol. 137: 2. 173-95 Feb. (ISI 5-year Impact Factor : 4,712). 2010 19) A Bavencoffe, D Gkika, A Kondratskyi, B Beck, A S Borowiec, G Bidaux, J Busserolles, A Eschalier, Y Shuba, R Skryma, N Prevarskaya (2010) The transient receptor potential channel TRPM8 is inhibited via the alpha 2A adrenoreceptor signaling pathway. J Biol Chem. 285: 13. 9410-9 Mar. (ISI 5-year Impact Factor : 5,498) 20) M Monet, V Lehen'kyi, F Gackiere, V Firlej, M Vandenberghe, M Roudbaraki, D Gkika, A Pourtier, G Bidaux, C Slomianny, P Delcourt, F Rassendren, J P Bergerat, J Ceraline, F Cabon, S Humez, N Prevarskaya (2010). Role of Cationic Channel TRPV2 in Promoting Prostate Cancer Migration and Progression to Androgen Resistance. Cancer Res. 70: 3. 1225-35 Feb. (ISI 5-year Impact Factor : 8,438) 21) C Lagadec, R Romon, C Tastet, S Meignan, E Com, A Page, G Bidaux, H Hondermarck, X. Le Bourhis (2010) Ku86 is important for TrkA overexpression-induced breast cancer cell invasion. Proteomics Clin Appl. 4: 6-7. 580-90 Jul. (ISI 5-year Impact Factor : 1,912). 2009 22) Lallet-Daher, Roudbaraki, Bavencoffe, Mariot, Gackière, Bidaux, Urbain, Gosset, Delcourt, Fleurisse, Slomianny, Dewailly, Mauroy, Bonnal, Skryma, Prevarskaya (2009). Intermediateconductance Ca(2+)-activated K(+) channels (IK(Ca1)) regulate human prostate cancer cell proliferation through a close control of calcium entry. Oncogene. 28: 15. 1792-806 Mar. (ISI 5year Impact Factor : 7,109) 23) Michaël Monet, Dimitra Gkika, V'yacheslav Lehen'kyi, Albin Pourtier, Fabien Vanden Abeele, Gabriel Bidaux, Véronique Juvin, François Rassendren, Sandrine Humez, Natalia Prevarsakaya Habilitation à Diriger des Recherches – Gabriel Bidaux © 2015 Tous droits réservés.. 11 doc.univ-lille1.fr.
Figure
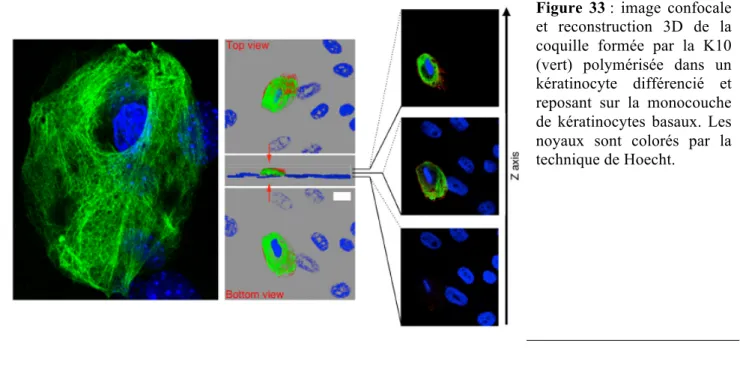
![Figure 9 : représentation schématique de la structure du noyau des cellules eucaryotes (adapté de [35]).](https://thumb-eu.123doks.com/thumbv2/123doknet/3517372.102872/50.892.359.793.370.755/figure-représentation-schématique-structure-noyau-cellules-eucaryotes-adapté.webp)
Documents relatifs
For example, Jackson's methodology derives an architecture from the data structure, and Struc- tured Design derives an architecture from data flow graphs.. The
En effet, en invitant à suivre un cheminement qui, de Tchernobyl, nous a conduit à La Hague, nous pourrions faire l’objet d’une double suspicion : celle, d’abord, de
Tinker- HP: a Massively Parallel Molecular Dynamics Package for Multiscale Simulations of Large Complex Systems with Advanced Point Dipole Polarizable Force Fields... Tinker-HP:
a,b) Experimental results for multiplexed concentration enhanced aptamer affinity probe electrophoresis assay for detecting IgE in buffer solution after 30 minutes of concentration,
Annexe# 1# :# Questionnaire# concernant# «# la# situation# actuelle#de#la#génération#Y#»#
Dans un premier temps, notre regard sera porté sur les nouvelles attitudes qui existent vis à vis du standard national de la langue russe (le russe normé, littéraire, la
These changed stasis and growth elements were calculated from the impact of herbivory (caged vs. control effect), and depended on whether absolute height growth rate was
Je quitterai la France, Ses villes et ses gratte-ciel, Ses cabarets et le pain rance Pour la jungle équatoriale, Le soleil et le miel.. Je vivrai loin de