Caractérisation du domaine de liaison à TARN de p54nrb
Mémoire présenté
à la Faculté des études supérieures de l'Université Laval
dans le cadre du programme de maîtrise en biologie cellulaire et moléculaire pour l'obtention du grade de Maître es Sciences (M. Se.)
FACULTE DE MEDECINE UNrVERSITÉ LAVAL
QUÉBEC
2011
The important thing is not to stop questioning. Albert Einstein
REMERCIEMENTS
J'aimerais d'abord remercier mon directeur de recherche Michel Vincent pour son support, son ouverture et son aide. De plus, j'aimerais le remercier de m'avoir donné l'opportunité d'intégrer le monde de la recherche assez tôt dans mon cheminement académique. Michel, je garderai toujours d'excellents souvenirs de mes premières expériences de recherche en milieu universitaire.
Je tiens à remercier Céline Bruelle, qui a été un mentor exceptionnel avec qui j'ai eu beaucoup de plaisir à travailler et Martin Gauthier pour son aide tout au long du projet. J'aimerais remercier mon co-directeur Stéphane Gagné de m'avoir donné l'opportunité de m'initier à la biologie structurale.
J'exprime toute ma gratitude aux membres du laboratoire de Stéphane Gagné, Sébastien Morin, Jean-Baptiste Duvignaud et Jérémie Leclerc dont l'aide a été essentielle à la réalisation de mon projet. Je vous en suis extrêmement reconnaissant.
Je voudrais remercier tous les membres du laboratoire du Dr Robert Tanguay, Marie, Geneviève et Diana pour les nombreux dîners passés ensemble à discuter de tout et de rien. Ce fut des moments forts agréables.
Je remercie Normand Voyer, Michel Auger et Jacques Lapointe de m'avoir permis d'utiliser leur appareil de CD, DLS et ITC.
Ce projet n'aurait pu avoir lieu sans l'appui du CRNSG et de PROTEO. Je remercie aussi le CRSNG, le FQRNT et Hydro-Québec de m'avoir appuyé financièrement tout au long du projet.
Je voudrais remercier tous les membres de ma famille et mes amis pour le soutien qu'ils m'ont accordé tout au long de ce projet. J'aimerais aussi les remercier pour l'intérêt et la curiosité qu'ils ont manifestés face à ce projet, j'espère avoir réussi à vous apprendre quelques notions de science en répondant à vos diverses questions ...
Finalement, j'adresse mes derniers remerciements à Élise et Alexandre pour leur amitié, leur aide, leur support et toutes les discussions scientifiques ou pas que nous avons eus. Merci pour tous les moments de folie (5 à 7 du pavillon) ou les moments plus sérieux que j'ai pu vivre avec vous pendant ces deux dernières années. Je m'estime chanceux de vous
RÉSUMÉ
p54nrb est une protéine de liaison à l'ARN nucléaire impliquée dans plusieurs processus cellulaires tels que la transcription, la maturation des ARNm et la rétention des ARNs hyper-édités. Cette protéine multifonctionnelle fait partie de la machinerie d'épissage et participe à ce processus en liant directement le site d'épissage en 5' du pré-ARNm. De plus, p54nrb se concentre dans un corps nucléaire nommé le paraspeckle en liant une fraction riche en G de l'ARNnc NEAT1. Récemment, nous avons démontré que la phosphorylation mitotique de la threonine 15 de p54nrb, située en N-terminal de deux RRMs en tandem, régule négativement sa capacité de liaison aux ARNs excepté pour ceux riches en G comprenant l'ARNnc NEAT1 (Bruelle et al., sous presse, annexe A).
Afin de caractériser la liaison des différents RRMs de p54nrb à l'ARN, une dissection moléculaire de son domaine de liaison à l'ARN (DLA) a été réalisée. Cette section contient les deux RRMs de la protéine précédés d'une région riche en H,Q, et P contenant le résidu T15 phosphorylable. Des tests de liaison in vitro à l'ARN du site d'épissage en 5' (5'SS, 11 nucleotides) et à des ARNs de polyguanosines ont permis de démontrer que le RRM1 de p54nrb est responsable de l'affinité de la protéine pour ces ligands. De plus, la cartographie des sites d'interaction du RRM1 avec le 5'SS et avec un ARN de polyguanosines (polyG, 11 nucleotides) a été réalisée par RMN et a révélé un site de liaison unique pour chacune de ces molécules. En effet, nous avons démontré que le RRM1 de p54nrb lie l'ARN polyG par un site de liaison non classique différent du site de liaison classique utilisé par la protéine pour lier le 5'SS. Ces expériences ont aussi permis de voir que le RRM1 de p54nrb avait plus d'affinité pour le polyG que pour le 5'SS.
Les résultats obtenus démontrent pour la première fois, à notre connaissance, la possibilité pour un seul RRM de posséder deux sites distincts de liaison à l'ARN. De plus, cette liaison non classique du RRM1 de p54nrb aux ARNs de polyguanosines pourrait potentiellement expliquer pourquoi la liaison de la protéine à ce type d'ARN n'est pas affectée par sa phosphorylation.
TABLE DES MATIÈRES
REMERCIEMENT I I
RÉSUMÉ I I I TABLE DES MATIÈRES I V
LISTE DES ABRÉVIATIONS V I I
LISTE DES FIGURES X
CHAPITRE I - INTRODUCTION 1
1.1 LE NOYAU CELLULAIRE EUCARYOTE 1
1.1.1 ORGANISATION DU NO Y AU 1 1.1.2 EXPRESSION GÉNIQUE 3
1.1.2.1 Couplage transcription et maturation des pré-ARNm 4
1.1.2.1.1 Épissage des pré-ARNm 6
1.2p54nrb 7
1.2.1 L'ANTICORPS PHOSPHO-DÉPENDANT CC-3 7
1.2.2 DÉCOUVERTE 8
1.2.2.1 Domaine de liaison à l'ARN de p54nrb 10
1.2.3 LOCALISATION CELLULAIRE DE P54NRB 12
1.2.3.1 Le paraspeckle 13 1.2.4 ÉDITION ET RÉTENTION DES A R N S HYPER-ÉDITÉS 1 5
1.2.5 IMPLICATION DE P54NRB DANS L'ÉPISSAGE DES PRÉ-ARNM 16 1.3HYPOTHÈSES ET OBJECTD7S DE RECHERCHE 18
CHAPITRE II - MATÉRIELS ET MÉTHODES 20
2.1 CLONAGES, EXPRESSION ET PURDTCATION DES SEGMENTS PROTÉIQUES
DU DLA DE P54NRB 2 0 2.1.1 CLONAGES 20 2.1.2 EXPRESSION ET PURIFICATION 20
2.4 SPECTROSCOPIE RMN 22 2.4.1 CONDITIONS D'ACQUISITION 22 2.4.2 EXPÉRIENCES DE TITRATION 23 2.4.3 ÉVALUATION DES CONSTANTES DE DISSOCIATION 2 3
2.5 TESTS DE LIAISON À L'ARN IN VITRO 2 4 2.6 ÉLECTROPHORÈSE ET IMMUNOBUVARDAGE 2 5
2.7 CALCUL DU POTENTIEL ÉLECTROSTATIQUE DE SURFACE 2 5
CHAPITRE III - RÉSULTATS 26
3.1 DISSECTION MOLÉCULAIRE DU DLA DE P54RNB : PURD7ICATION ET MISES
AU POINT POUR ÉTUDES RMN 2 6 3.1.1 PRÉSENTATION DES SEGMENTS PROTÉIQUES DU DLA DE P54RNB GÉNÉRÉS ET PURIFIÉS
POUR L'ÉTUDE 26
3.1.2 MISES AU POINT DES CONDITIONS POUR ÉTUDES PAR RMN 28 3.2 CARACTÉRISATION DE LA LIAISON DU DLA DE P54NRB À LA SÉQUENCE DU
SITE D'ÉPISSAGE EN 5' 3 9
3.2.1 LE RRM2 DE P54NRB SEUL EST INCAPABLE DE LIER LE 5'SS IN VITRO 39 3.2.2 LE RRM1 EST RESPONSABLE DE L'INTERACTION DE P54NRB AVEC LE 5'SS 42 3.2.3 LE RRM1 DE P54NRB INTERAGIT AVEC LE 5 'SS VIA DES RÉSIDUS BASIQUES ET
AROMATIQUES À LA SURFACE DE SON FEUILLET-B 45
3.3 CARACTÉRISATION DE LA LIAISON DU DLA DE P54NRB À UN ARN DE
POLYGUANOSINES 52
3.3.1 LE RRM2 DE P54NRB SEUL EST INCAPABLE DE LIER DES HOMORIBOPOLYMÈRES DE
GUANOSINE IN VITRO 52 3.3.2 LE RRM1 DE P54NRB EST RESPONSABLE DE SON INTERACTION AVEC UN ARN DE
POLYGUANOSINES 54 3.3.3 LE RRM1 DE P54NRB INTERAGIT AVEC LE POLYG VIA UN SITE DE LIAISONNON
CLASSIQUE DISTINCT DE CELUI UTILISÉ POUR LIER LE 5'SS 57
CHAPITRE IV - DISCUSSION ET CONCLUSION 62
4.1.1 LES SEGMENTS P R 0 T É I Q U E S N - R R M 1 / 2 ET RRM2 SONT DES PROTÉINES INSTABLES 62
4.1.2 LE N-TERMINAL DE P54NRB EST NON-REPLIÉ ET NE SEMBLE PAS PERTURBER LA
STRUCTURE DU RRM1 63 4.1.3 LE SEGMENT PROTÉIQUE RRM2 EST REPLIÉ ET COMPORTE DES STRUCTURES D'HÉLICE-A
ET DE FEUILLET-B 64
4.2 CARACTÉRISATION DE LA LIAISON DES SEGMENTS PROTÉIQUES DU DLA
DE P54NRB À L'ARN 65
4.2.1 LE RRM1 EST LE MOTIF DE P54NRB RESPONSABLE DE SA LIAISON À L'ARN 65 4.2.2 LE RRMl DE P54NRB LIE LE 5'SS ET LE POLYG PAR DEUX SITES DE LIAISON DISTINCTS 67
4.3 EFFET DE LA PHOSPHORYLATION DE LA THREONINE 15 DE P54NRB SUR SA
CAPACITÉ DE LIAISON À L'ARN 72
4.5 CONCLUSION 73
BIBLIOGRAPHIE 75
ANNEXE A 84
ANNEXE B 118
ANNEXE C 119
ANNEXED 121
ANNEXEE 123
ANNEXE F 124
Liste des abréviations
a-
Anti-A Anti-Adenosine 5'SS Site d'épissage en 5'
ADARS Adenosines deaminases qui agissent sur l'ARN ADN Acide désoxyribonucléotide
ARN Acide ribonucléotidique ARNr ARNs ribosomaux ARNm ARNs messagers ARNt ARNs de transfert
BSA Albumine de sérum de bœuf C Cytosine
CAT2 Cationic amino acid transporter 2 CDC Changement de déplacement chimique DC Dichroïsme circulaire
CDK1 Cyclin-Dependent Kinase 1 CHO Chinese hamster ovary CTD Domaine carboxy terminal
DAPI 4'6-diamidino-2-phenylindol db Double brin
DBHS Drosophila Behavior Human Splicing DLA Domaine de liaison à l'ARN
DLS Dynamic light scattering D.O. Densité optique
DTT Dithiothreitol EGF Epidermal growth factor
FISH Fluorescence in situ hybridization
FLIP Fluorescence loss induced by photobleaching FPLC Fast protein liquid chromatography
GAM Goat-a-mouse
hnRNPs Heterogeneous nuclear ribonucleoproteins hnRNAs Heterogeneous nuclear RNAs
HPLC High-performance liquid chromatography HSQC Heteronuclears single quantum coherence HTH Hélice Tour Hélice
IPTG Isopropyl-P-D-thiolgalactopyranoside IRAlus Inverted repeated Alu elements
Kd Constante de dissociation LB Luria-Bertani broth Nc non-coding
NEAT1 Nuclear Enriched Autosomal non-coding Transcipts 1
NonO non-POU domain containing, octamer-binding domain protein N-WASP Neuronal Wiskott-Aldrich syndrome protein
p54nrb nucleo ribo binding protein, 54kDa PCR Réaction de polymérisation en chaîne pdb Protein data bank
P.I. Point isoélectrique Pol ARN polymerase I Pol II ARN polymerase II Pol III ARN polymerase III
Pol HA Isoforme hypophosphorylé de l'ARN polymerase II Pol HO Isoforme hyperphosphorylé de l'ARN polymerase II polyG Ligand l'ARN compose de 11 guanosines
PML Promyelocitic Leukemia protein
PSF Polypyrimidine tract-binding protein-associated Splicing Factor PSP1 Paraspeckle protein 1
PTB Polypyrimidine tract-binding protein RMN Résonance magnétique nucléaire RNP Ribonucleoprotein
SDS-PAGE sodium dodecyl sulfate polyacrylamide gel electrophoresis snRNA Small nuclear RNA
snRNPs Small nuclear Ribonucleoproteins TBS Tris-buffered saline
TFE 2,2,2-trifluoroéthanol TGF-p Transforming growth factor (} U Uracile
UTR Untranslated region
Liste des figures
Figure 1. Fluorescence par hybridation in situ des chromosomes d'un fibroblaste humain
en GO 1 Figure 2. Microphotographie obtenue par microscopie confocale montrant différentes
structures nucléaires de cellules mammifères 3 Figure 3. Couplage de la transcription et de la maturation du pré-ARNm orchestré par le
CTD de la Pol II 5 Figure 4. Les différentes étapes du processus d'épissage du pré-ARNm 7
Figure 5. Patron d'immunoréactivité de l'anticorps phosphodépendant CC-3 8 Figure 6. Représentation schématique des protéines DBHS chez les mammifères 10
Figure 7. Structure tridimensionnelle des RRMs de p54nrb 12 Figure 8. NEAT1 et p54nrb se colocalisent auxparaspeckles dans les cellules HeLa... 14
Figure 9. Représentation schématique de l'édition et de la rétention des ARNm
hyper-édités aux paraspeckles par p54nrb 16 Figure 10. Modèle schématisant le complexe transcription/épissage 17
Figure 11. Présentation des trois segments protéiques du DLA de p54nrb utilisés pour
l'étude 27
Figure 12. Spectre 'H-15N HSQC de la protéine N-RRM1/2 30
Figure 13. Spectre 'H-^N HSQC du segment protéique N-RRM1 31 Figure 14. Le RRM2 de p54nrb agrège à faible force ionique et à haute température.... 33
Figure 15. L'augmentation de la force ionique et la baisse de la température sont les
facteurs clés pour stabiliser l'agrégation du RRM2 35
Figure 17. Spectre 3D HNCO du RRM2 de p54nrb 38 Figure 18. Le RRM2 de p54nrb seul est incapable de lier le 5'SS 41
Figure 19. Le RRM1 de p54nrb est responsable de son interaction avec le site 5'SS 44 Figure 20. Le RRM1 de p54nrb interagit avec le 5'SS via des résidus basiques et
aromatiques à la surface de son feuillet-P 50 Figure 21. La surface du feuillet-P du RRM2 de p54nrb est plus électronégative que celle
duRRMl 51 Figure 22. Le RRM2 de p54nrb seul est incapable de lier des homoribopolymères de G.
53 Figure 23. Le RRMl de p54nrb est responsable de son interaction avec le polyG 56 Figure 24. Le RRMl de p54nrb interagit avec le polyG via un site de liaison non
classique 61 Figure 25. Comparaison des sites de liaison du RRMl de p54nrb pour le 5'SS et du
1.1 Le noyau cellulaire eu c a n o t e 1.1.1 Organisation du noyau
Le noyau cellulaire est une organelle complexe contenant l'information et la machinerie nécessaires à l'expression génique. Ce compartiment est délimité par une double membrane appelée la membrane nucléaire qui contient des pores permettant des échanges constants avec le cytosol (Stoffler et al., 1999). Bien qu'il ait été longtemps cru que le noyau ne possédait pas d'organisation précise, de plus en plus de recherches contemporaines démontrent le contraire. En effet, chaque chromosome occupe un territoire spécifique au sein du noyau (Schardin et al., 1985). Les gènes actifs, contenus dans l'euchromatine, se localisent préférentiellement entre deux territoires chromatiniens où les différents facteurs impliqués dans l'expression génique y ont plus facilement accès. D'ailleurs, des études de localisation cellulaire réalisées sur différentes protéines nucléaires et ARNs ont permis de constater la présence de plusieurs compartiments nucléaires spécialisés localisés dans l'espace interchromatinien (Lamond and Earnshaw,
1998).
B
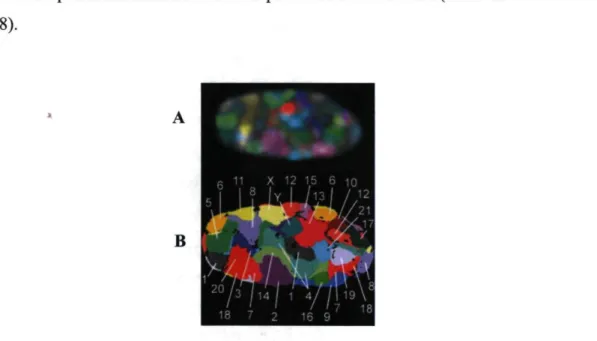
Figure 1. Fluorescence par hybridation in situ de tous les chromosomes (24-color 3D FISH) d'un fibroblaste humain en GO montrant les territoires chromatiniens (adaptée de Bolzer et al., 2005). Image réelle en A et représentation schématisé en B.
les corps de Cajal, les speckles et les paraspeckles. Le nucléole contient l'ADN ribosomal et est le site de synthèse des ARNs ribosomiaux par l'ARN polymerase I. De plus, il est le site d'assemblage des ribosomes et de certains autres complexes ribonucléoprotéiques comme les snRNPs (small nuclear ribonucleoproteiri) U5 et U6 (Carmo-Fonseca et al., 2000; Scheer and Hock, 1999). Finalement, le nucléole peut aussi servir de compartiment de séquestration pour empêcher l'action de certains facteurs protéiques (Ganot et al, 1999; Jady and Kiss, 2001; Lange and Gerbi, 2000). Les PMLs, typiquement présents au noyau en 10 à 30 copies, sont impliqués dans plusieurs processus. Entre autres, ils jouent un rôle dans la différenciation cellulaire terminale, la régulation transcriptionnelle, le contrôle de la croissance cellulaire et l'apoptose (Maul,
1998; Quignon et al., 1998; Ruggero et al., 2000). Les corps de Cajal, normalement présents en 1 à 5 copies, sont impliqués dans la biogenèse des différents snRNPs. On y retrouve notamment les snRNPs spliceosomales Ul, U2, U4/6 et U5 avant leur transit aux speckles, un compartiment riche en facteurs d'épissages (Spector, 2001). En effet, les speckles constituent un réservoir de facteurs d'épissage très dynamique dont les différentes composantes sont recrutées au site de transcription permettant la maturation des pré-ARNm. D'ailleurs, il est intéressant de noter que les speckles se localisent dans l'espace interchromatinien à proximité des sites actifs de transcription (Spector, 2001). Finalement, un compartiment appelé le paraspeckle, localisé en périphérie des speckles, a été découvert il y a quelques années (Fox et al., 2002). La formation de ce corps nucléaire est dépendante de deux isoformes de l'ARN non codant (ARNnc) NEAT-1 (Clemson et al., 2009; Hutchinson et al., 2007; Sasaki et al., 2009). Bien que ses fonctions demeurent encore vagues, il a été démontré que les paraspeckles participent à la séquestration des ARNs hyper-édités par les enzymes ADARs (dsRNA dependent adenosine deaminase). Cette séquestration se fait par l'intermédiaire d'une des protéines au paraspeckle, p54nrb, qui a de l'affinité pour ces ARNs modifiés (Chen and Carmichael, 2009; Zhang and Carmichael, 2001).
..
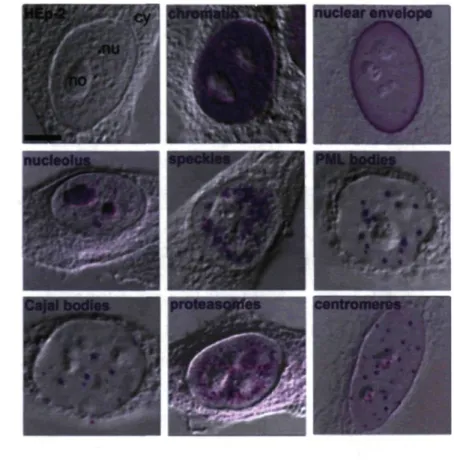
Figure 2. Microphotographie obtenue par microscopie confocale montrant différentes structures nucléaires de cellules mammifères marquées par immunofluorescence (http://www.fli-leibniz.de/groups/emmerich_structure_en.php). Cy: cytoplasme; Nu: nucléoplasme; No : nucléole; Échelle : 5 uM.
1.1.2 Expression génique
L'expression génique est le processus par lequel l'information génétique de la cellule est sélectivement lue pour mener à la synthèse de molécules d'ARNs et de protéines qui auront des fonctions spécifiques. Selon l'environnement dans lequel elle se trouve, la cellule exprimera un ensemble de gènes précis qui dicteront si elle doit se différencier, proliférer, entrer en quiescence ou entrer en apoptose. Par exemple, chez l'humain, des signaux générés par certains facteurs de croissance comme l'EGF {epidermal growth factor) stimuleront de façon générale les gènes pro-prolifératifs de la cellule alors que d'autres facteurs de croissance comme le TGF-P (transforming growth factor p) stimuleront les gènes antiprolifératifs. Dans les organismes pluricellulaires, l'expression
La transcription est la première étape de l'expression génique et consiste au processus par lequel la région d'ADN codante d'un gène sera copiée sous la forme d'une molécule d'ARN. La régulation de ce processus est complexe et implique plusieurs acteurs protéiques comprenant les activateurs et les répresseurs de la transcription. La liaison d'activateurs sur des séquences d'ADN bien précises appelées enhancers augmentera la transcription des gènes cibles alors que la liaison de répresseurs sur des séquences d'ADN silencers la diminuera. La transcription est catalysée par des complexes enzymatiques multi-protéiques connus sous le nom d'ARN polymerases (Koleske and Young, 1995). Il existe trois ARN polymerases (Pol I, Pol II, Pol III) au sein de la cellule eucaryote. La Pol I est responsable de la transcription des longs ARNs ribosomiaux 28S, 18S et 5,8S. La Pol II est responsable de la transcription des ARNm et la Pol III orchestre la synthèse des ARNs de transfert ainsi que de certains ARNs nucléaires (Sentenac, 1985). Pendant leur synthèse par la Pol II, les ARNm doivent subir plusieurs modifications moléculaires avant d'être exportés au cytosol où ils enclencheront la production de protéines.
1.1.2.1 Couplage transcription et maturation des pré-ARNm
Comme mentionné ci-haut, la transcription des pré-ARNm à partir de l'ADN contenue dans l'euchromatine s'effectue par la Pol II. Avant d'être exporté au cytoplasme pour être traduit en protéine, la plupart des pré-ARNm naissants subissent trois modifications post-transcriptionnelles: 1) l'ajout de la coiffe en 5', 2) l'épissage et 3) le clivage et la polyadénylation. La transcription et la maturation du pré-ARNm sont deux processus intimement liés dans le temps et l'espace grâce au domaine carboxy-terminal (CTD) de la Pol II qui agit comme plateforme de recrutement pour les facteurs de maturation des pré-ARNm (Proudfoot, 2000). Le CTD de la Pol II contient 52 répétitions du motif YSPTSPS dont les résidus S2 et S5 sont phosphorylables (Allison et al., 1985; Corden et al., 1985; Dahmus, 1996). La Pol II existe donc sous deux forme distinctes : la forme hypophosphorylée (Pol IIA) prédominante lors de la formation du complexe d'initiation
phosphorylation différentielle du CTD permet de coordonner le recrutement des différents facteurs de maturation requis pour opérer les trois modifications post-transcriptionnelles du pré-ARNm (Proudfoot, 2000).
Nuclear export
Figure 3. Couplage de la transcription et de la maturation du pré-ARNm orchestré par le CTD de la Pol II (Proudfoot, 2000).
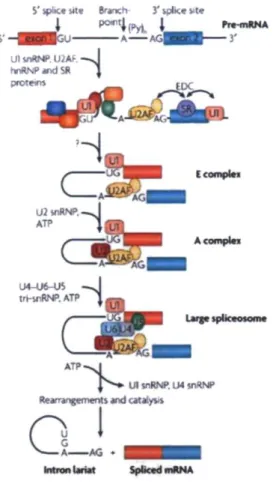
fait partie du processus de maturation des pré-ARNm. Chez les eucaryotes, la plupart des gènes contiennent des séquences codantes appelées exons et des séquences non-codantes appelées introns. Les introns contenus dans le pré-ARNm doivent être excisés et les exons joints afin de permettre la production d'une protéine fonctionnelle. Ce processus se fait par deux réactions de transesterification simples et est catalysé par un méga-complexe protéique appelé le spliceosome qui regroupe plus de 150 protéines et 5 ARNs. Les acteurs principaux du spliceosome sont les snRNPs Ul, U2, U4, U5 et U6 qui sont en fait des complexes protéines/ARNs (Jurica and Moore, 2003). Trois séquences consensus conservées sont nécessaires à la réalisation de ce processus par le spliceosome. La jonction exon/intron en 5' de l'intron, nommée le site d'épissage en 5' (5'SS), contient la
séquence GU alors que la jonction intron/exon en 3' de l'intron, appelée le site d'épissage en 3' (3'SS), doit contenir la séquence AG. La troisième séquence consensus nécessaire au processus se nomme le site de branchement et consiste en une adenosine conservée contenue dans l'intron suivie d'une séquence de polypyrimidines. Les rôles des snRNPs sont de reconnaître et de lier le 5'SS et le point de branchement par des interactions ARN/ARN, d'approcher ces deux sites dans l'espace et de catalyser la réaction de clivage de l'ARN et la fusion des exons. D'autres protéines du spliceosome doivent lier le 3'SS afin que le processus d'épissage puisse s'opérer (Li et al., 2007).
U r g e spUccosonw
Introo ItfMt Spliced mnNA
Nature Reviews Naureacwnc*
Figure 4. Les différentes étapes du processus d'épissage du pré-ARNm (Li et al., 2007). Les snRNPs vont se lier aux différentes séquences conservées du pré-ARNm pour permettre l'assemblage du spliceosome et la catalyse de l'excision de l'intron.
1.2 p54nrb
1.2.1 L'anticorps phospho-dépendant CC-3
À la fin des années 80, les recherches du laboratoire du Dr Vincent visent le développement d'anticorps monoclonaux capables d'identifier certaines structures spécifiques de l'embryon de poulet dans les différents stades de l'embryogenèse (Thibodeau et al., 1989). Un des anticorps produits retient l'attention puisqu'il est capable d'identifier spécifiquement les cellules mitotiques (Thibodeau and Vincent, 1991). Alors qu'il se lie seulement à la Pol II et à la protéine Spt5 en interphase, cet anticorps reconnaît une multitude de protéines phosphorylées sur des serines ou des
threonines lors de la mitose. Parmi ces protéines, on retrouve le facteur d'épissage SAP-155, la protéine associée au microtubule MAP-ID, les ADN topoisomérases II a et P, l'antigène Ki-67 ainsi que la protéine multifonctionnelle p54nrb (Albert et al., 2004). Comme le rôle de la phosphorylation mitotique de p54nrb n'est pas connu à ce moment-là, un des objectifs du laboratoire du Dr Vincent devient d'élucider la fonction de cette modification post-traductionnelle. 255 — 240 — 220 — 94 — 68 •* p54n r l — 42
Figure 5. Patron d'immunoréactivité de l'anticorps phosphodépendant CC-3 sur un extrait de cellules CHO interphasiques (A) et mitotiques (B) (Thibodeau et al., 1989). p54nrb interagit avec CC-3 seulement dans l'extrait mitotique.
1.2.2 Découverte
La protéine p54nrb fut découverte par hasard alors qu'un groupe de recherche tentait d'identifier un homologue du facteur d'épissage de levure PRP18 chez l'humain (Dong et al., 1993). En effet, une immunoprécipitation faite avec un anticorps contre PRP18 dans un extrait de cellules HeLa a mené à la purification d'un polypeptide de 54kDa correspondant à p54nrb. Malgré cette réaction croisée avec l'anticorps a-PRP18, p54nrb ne partage pas d'homologie de séquence avec PRP-18. Par contre, une analyse de la séquence peptidique de p54nrb révèle un très grand niveau d'identité de 71% avec la protéine humaine PSF (Dong et al., 1993). Parmi les 320 acides aminés partagés par ces
intéressant de noter que les deux protéines possèdent des domaines riches en prolines et glutamines dans leur extrémité N-terminale. Bien que ce type de domaine soit souvent présent dans les hnRNPs et les snRNPs, leur rôle reste à éclaircir. Certaines études proposent que les segments protéiques riches en Q et P soient impliqués dans des interactions protéine/protéine nécessaires au bon fonctionnement du spliceosome (Patton et al., 1993). En plus de posséder deux RRMs, p54nrb possède un domaine hélice-tour-hélice (HTH) suivi d'une région de résidus acides et basiques qui ensemble constituent un domaine de liaison à l'ADN. C'est la présence de ces différents domaines qui confère à p54nrb ses propriétés de protéine multifonctionnelle au noyau cellulaire. En effet, ces différents motifs lui permettent d'interagir avec l'ARN, l'ADN simple brin et l'ADN double brin (Shav-Tal and Zipori, 2002). Jusqu'à maintenant les processus nucléaires identifiés impliquant p54nrb regroupent : le contrôle de la transcription, l'épissage des pré-ARNm, le clivage en 3' des ARNm, la réparation et recombinaison de l'ADN et la rétention des ARNs hyper-édités au noyau.
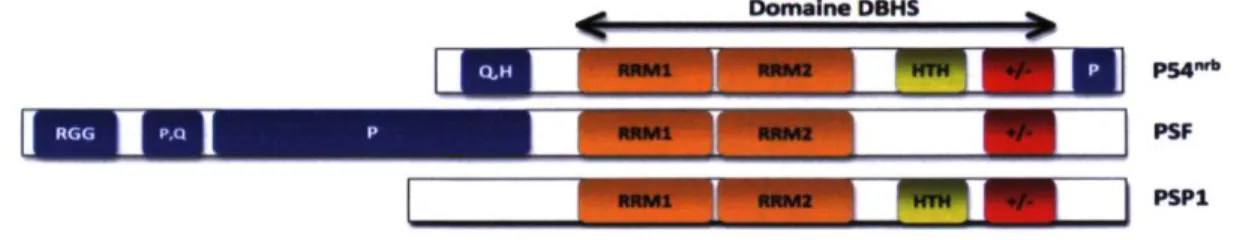
Il est intéressant de noter que l'homologie de séquence partagée entre p54nrb et PSF existe aussi avec la protéine de drosophile NONA/BJ6. Tout comme PSF et p54nrb, BJ6 semble être impliquée dans la maturation des pré-ARNm du fait qu'elle se localise dans les puffs transcriptionnellement actifs des chromosomes de la drosophile (Frasch and Saumweber, 1989). La conservation de cette séquence de 320 acides aminés, depuis la divergence de la drosophile et de l'humain jusqu'à maintenant, suggère qu'il y ait eu préservation de la fonction de ce segment protéique dans le temps. D'ailleurs, ce domaine phylogénétiquement conservé se nomme le domaine DBHS (Drosophila Behavior and Human Splicing, figure 6) (Dong et al., 1993).
Depuis la découverte du domaine conservé DBHS, deux nouvelles protéines ont été identifiées dans cette famille. L'une d'elle est la protéine du diptère Chironomus tentans Hrp65. Cette protéine, qui possède la capacité d'oligomériser, serait une composante des Connecting Fibers (CFs), une structure responsable du transport des pré-mRNPs
(Complexes de ribonucléoprotéines regroupant le pré-ARNm et les hnRNP) vers l'enveloppe nucléaire (Kiesler et al., 2003; Miralles et al., 2000). La seconde protéine, PSP1 (paraspeckle protein 1), fut identifiée grâce à des études de caractérisation du protéome du nucléole humain par spectrométrie de masse (Andersen et al., 2002). Des éludes de localisation de cette protéine ont permis la découverte d'un nouveau compartiment nucléaire nommé le paraspeckle (Fox et al., 2002). Étonnamment, les homologues humains de PSP1, p54nrb et PSF, ont aussi montré une localisation dans les paraspeckles.
Domaine DBHS
Figure 6. Représentation schématique des protéines DBHS chez les mammifères. Q,H domaine riche en glutamines et en histidines; RRM : motif de liaison à l'ARN; HTH domaine hélice-tour-hélice; +/- : domaine riche en acides aminés acides ou basiques; P domaine riche en prolines. RGG : domaine riche en glycines et arginines; P,Q : domaine riche en glutamines et prolines.
1.2.2.1 Domaine de liaison à l'ARN de p54nrb
Comme mentionné ci-dessus, p54nrb possède deux RRMs dans sa fraction N-terminale. Ce motif protéique, présent dans environ 2% des gènes humains, est celui le plus abondant chez les eucaryotes supérieurs et c'est pour cette raison qu'il est aussi le plus étudié (Maris et al., 2005). On retrouve les RRMs dans tous les règnes y compris chez les prokaryotes et les virus (Maris et al., 2005). La topologie typique de ce motif d'environs 90 acides aminés, Pia^Pso^P-*, mène à la formation d'un feuillet-P collé à deux hélices-a (figue 6). En général, les RRMs interagissent seulement avec de l'ARN simple brin via des résidus aromatiques et basiques conservés présents à la surface du feuillet-p. De façon classique, les résidus aromatiques génèrent des interactions d'empilement avec les
sucres de l'ARN alors que les résidus basiques du RRM stabiliseront le caractère acide de l'acide nucléique. En général, deux séquences consensus d'acides aminés nommées RNP1 (sur le brin P3) et RNP2 (sur le brin Pi) comportent ces résidus conservés. On retrouve typiquement un résidu F ou Y en position 3 et 5 du RNP1 et en position 2 du RNP2 ainsi que la présence d'un résidu K ou R en position 1 du RNP2. Même si ces caractéristiques sont présentes sur bien des RRMs, plusieurs de ces motifs présentent d'autres propriétés uniques leur permettant de lier de l'ADN ou encore des protéines. Les RRMs sont des motifs extrêmement plastiques et c'est pour cette raison que plusieurs groupes de recherche mobilisent leurs efforts afin d'approfondir les connaissances sur ce motif ubiquitaire.
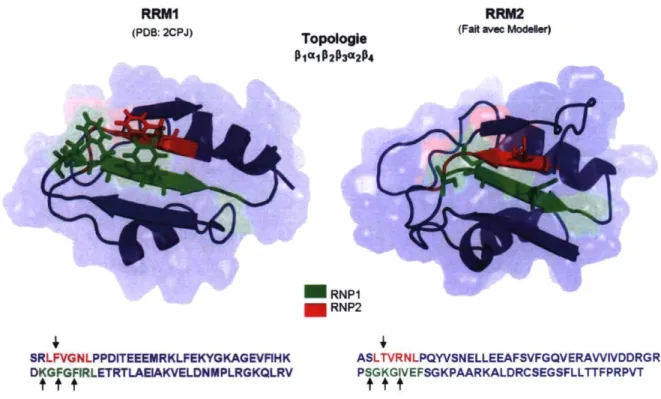
La structure du RRMl de p54nrb (pdb : 2CPJ) a été réalisée par résonance magnétique nucléaire (RMN) et révèle un RRM classique (figure 7). Une analyse de la structure primaire du RRMl permet de voir que les résidus conservés se retrouvent aux positions attendues des RNPs. De plus, la structure tridimensionelle du RRMl montre que ces résidus sont présents en surface du feuillet-P et qu'ils sont accessibles pour interagir avec l'ARN (figure 7). Pour sa part, la structure du RRM2 de p54nrb n'a pas encore été déterminée. Par contre, un modèle par homologie de ce motif, réalisé par Sami Aziz au laboratoire du Dr Stéphane Gagné, révèle qu'il possède la topologie et la structure classique d'un RRM mais l'analyse de sa structure primaire montre que les résidus normalement conservés des RNPs ne sont pas présents. Cela suggère que le RRM2 de p54nrb utilise un moyen non classique pour interagir avec l'ARN ou qu'il n'est pas impliqué dans ce type d'interaction.
RRM1 RRM2 (PDB: 2CPJ) Topologie
p1a1p2P3a2P4
(Fait avec Modeller)
IRNP1 RNP2 SRLFVGNLPPDITEEEMRKLFEKYGKAGEVFIHK DKGFGFIRLETRTLAEIAKVELDNMPLRGKQLRV t t t ASLTVRNLPQYVSNELLEEAFSVFGQVERAWIVDDRGR PSGKGIVEFSGKPAARKALDRCSEGSFLLTTFPRPVT t t T
Figure 7. Structure tridimensionnelle des RRMs de p54nrb. Les séquences consensus RNP1 (vert) et RNP2 (rouge) sont indiquées sur les structures et sur les séquences primaires des RRMs. Les résidus conservés normalement impliqués dans la liaison à l'ARN sont pointés par des flèches et sont en bâtonnets sur les structures. Le RRMl possède les résidus aromatiques normalement impliqués dans la liaison à l'ARN contrairement au RRM2.
1.2.3 Localisation cellulaire de p54nrb
En interphase, p54nrb est localisée de façon diffuse dans le nucléoplasme, exclue du nucléole et se concentre dans les paraspeckles (Fox et al., 2002; Fox et al., 2005). Bien que des études protéomiques du nucléole humain ont révélé la présence de p54nrb dans ce compartiment, la protéine n'y est pas détectable par imunofiuorescence (Andersen et al., 2002; Fox et al., 2002). Par contre, des expériences de perte de fluorescence après photoblanchiment (FLIP) réalisées sur la protéine des paraspeckles PSP1 révèlent que cette protéine oscille constamment entre les paraspeckles et le nucléole (Fox and Lamond, 2010). Comme les trois membres de la famille des DBHS sont capables d'hétérodimériser, ces résultats pourraient expliquer pourquoi ces protéines ont été identifiées dans le protéome du nucléole. Un autre fait intéressant est que lorsque des
cellules sont traitées avec l'actinomycine D et que la transcription est réprimée, les trois protéines de la famille des DBHS se relocalisent aux caps périnucléolaires (Fox et al., 2005; Fox et al., 2002). La localisation cellulaire de p54nrb et de ses deux homologues semble donc dépendante de la transcription.
1.2.3.1 Le paraspeckle
Le paraspeckle est un méga complexe ribonucléoprotéique de 0,5 à 1,0 um facilement détectable par immunofluorescence avec un anticorps contre une des protéines de la famille des DBHS (p54nrb, PSP1 et PSF). Même si les trois protéines des DBHS constituent le cœur du paraspeckle, plusieurs autres protéines ont montré des patrons de localisation dans ce compartiment (Fox and Lamond, 2010). On dénombre normalement entre 10 et 20 paraspeckles par cellule dans l'espace interchromatinien du noyau. Des études récentes ont démontré qu'un long ARNnc nommé NEAT1 est essentiel à la formation et au maintien des paraspeckles. En effet, une baisse de l'expression de cet ARNnc mène à la disruption du compartiment nucléaire (Clemson et al., 2009; Sasaki et al., 2009; Sunwoo et al., 2009). Cette observation est le premier exemple d'implication d'un ARNnc dans la biogenèse d'un corps nucléaire. NEAT1 existe sous deux isoformes, NEATvl (3,7 kb) et NEAT_v2 (22,7kb), transcrits à partir du même promoteur (Guru et al., 1997). Récemment, des expériences de microscopie électronique et d'hybridation in situ réalisées sur les deux isoformes de NEAT1 ont permis de découvrir que ces deux ARNs sont hautement structurés au sein du paraspeckle (Souquere et al., 2010). En périphérie de cette organelle, on retrouve NEATvl ainsi que les extrémités 3' et 5' de NEAT_v2 alors que la portion centrale de NEAT_v2 se situe dans le corps du paraspeckles. Malgré ces découvertes, il a été montré que l'ARNnc NEAT1 n'est pas la
seule molécule essentielle au maintien des paraspeckles. Des transfections d'ARNs interférents dirigées contre les ARNm des protéines des DBHS ont été réalisées dans des cellules HeLa afin d'en voir l'effet sur les paraspeckles (Sasaki et al., 2009). De ces trois protéines, la baisse de p54nrb et de PSF a mené à la perte des paraspeckles suggérant un rôle important de ces protéines dans l'intégrité de l'organelle. Les sites d'interaction de p54nrb avec VINC (l'équivalent murin de NEAT1) ont été identifiés (Murthy and Rangarajan, 2010). Un de ces sites situé en 5' de VINC, nommé PIR-1 (120 bases),
possède 70% d'identité avec la séquence de NEAT1. C'est probablement en liant ce site extrêmement riche en G de NEAT1 (plus de 40% de G) que p54nrb contribue à maintenir l'intégrité des paraspeckles.
Jusqu'à tout récemment, le rôle des paraspeckles était inconnu. Par contre, il avait été démontré que la protéine p54nrb est impliquée dans la rétention des ARNs hyper-édités au noyau (Zhang and Carmichael, 2001). Comme p54nrb est une des protéines des paraspeckles, l'hypothèse prédominante était que les paraspeckles étaient impliqués dans
ce processus. En effet, en 2009, une équipe de chercheurs a démontré que des cellules humaines dans lesquelles NEAT1 est sous exprimé (sans paraspeckles) sont incapables de séquestrer des ARNm hyper-édités au noyau (Chen and Carmichael, 2009). Ils ont aussi montré que les cellules souches embryonnaires humaines sont naturellement dépourvues de paraspeckles et sont incapables de retenir les ARNm hyper-édités au noyau. hNEATI * • • p54"rb • • Merge + DAPI hNEATI * • • p54"rb • • • • • + DAPI hNEATI * • • p54"rb • • • • • ^^^~ p54"rb • • • • •
Figure 8. NEAT1 et p54nrb se colocalisent aux paraspeckles dans les cellules HeLa (Chen and Carmichael, 2009). Vert: hybridation in situ de NEAT1; Rouge: immunomarquage de p54nrb; Bleu: noyaux colorés au DAPI.
1.2.4 Édition et rétention des ARNs hyper-édités
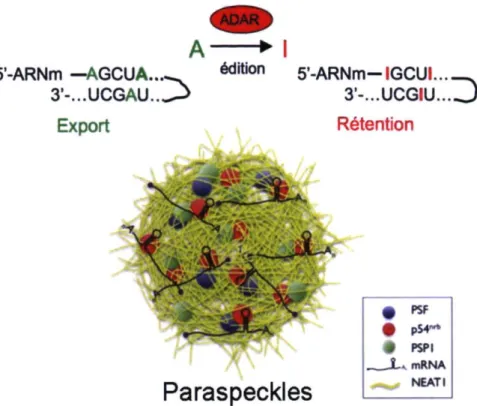
Dans le noyau cellulaire, les ARN double brin (ARNdb) sont souvent édités par les enzymes de type ADAR. L'édition est une modification post-transcriptionnelle des ARNs et il en existe plusieurs types. La déamination des adenosines en inosines est le type d'édition catalysé par les enzymes ADARs. Ces protéines peuvent éditer les ARNdb de deux façons différentes. D'abord, il y a l'édition de sites spécifiques de certains ARNm qui possèdent une structure secondaire créant un duplex d'ARN court. En général, pour ce type d'édition, une seule base d'un exon est modifiée changeant la signification du codon touché. Par la suite, il peut y avoir de l'édition aléatoire de long fragment d'ARNdb de plus de 25 paires de base dans lequel jusqu'à 50 % des A peuvent être changés en I (on parle alors d'hyper-édition) (Bass, 2002). Plus de 90% de ce type d'édition se produit dans les régions répétées inversées Alu (IRAlus) capables de former de longs duplexes d'ARN. De plus, 333 ARNm comportant ce type de séquence dans leur 3'UTR ont été identifiés (Chen and Carmichael, 2008). Comme mentionné dans la section précédente, il a été démontré que ces ARNm hyper-édités sont potentiellement liés par la protéine des paraspeckles p54nrb pour être retenus au noyau (Chen et al., 2008; Prasanth et al., 2005; Zhang and Carmichael, 2001). Ce processus permettrait à la cellule de faire un contrôle de qualité des ARNs et d'empêcher l'export de certains ARNm hyper-édités au cytosol. De plus, certaines études ont démontré que la rétention des ARNs hyper-édités pouvait permettre une certaine forme de régulation de l'expression génique. Par exemple, une des isoformes de l'ARNm de la protéine murine CAT2 (cationic amino acid transporter 2) contient une séquence IRAlu dans son 3'UTR. Sous condition de stress, cet ARN qui est normalement retenu aux paraspeckles peut être clivé en 3' permettant son export au cytoplasme et par conséquent une réponse rapide au stress cellulaire (Prasanth et al., 2005).
5'-ARNm —AGCUA. 3'-...UCGAU..^ Export ► I édition S'-ARNm-IGCUl 3'-...UCGIU...O Rétention
Paraspeckles
Figure 9. Représentation schématique de l'édition et de la rétention des ARNm hyper-édités aux paraspeckles par p54nrb (adaptée de Scadden, 2009)
1.2.5 Implication de p54nrb dans l'épissage des pré-ARNm
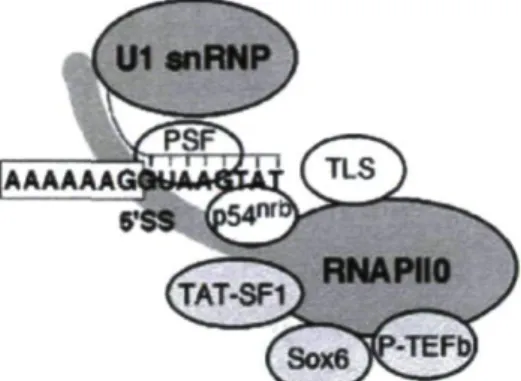
De plus en plus d'études tendent à démontrer que la transcription et l'épissage des ARNm sont deux processus spatio-temporellement couplés. En effet, le recrutement de facteurs d'épissage aux sites transcriptionnellement actifs par le CTD de la Pol II serait le moyen utilisé par la cellule pour lier ces deux processus. Des études ont démontré que la protéine multi-fonctionnelle p54nrb est capable de lier le CTD de la Pol II sans perdre ses capacités de liaison à l'ARN (Emili et al., 2002). D'autres études ont démontré que cette protéine multi-fonctionnelle est capable de lier directement le 5'SS des pré-ARNm et une séquence d'ARN contenue dans le snRNP U5 (Kameoka et al., 2004; Peng et al., 2002). De plus, des analyses de sédimentation ont montré que p54nrb interagit avec les complexes de snRNPs U4/U6.U5 (Peng et al., 2002). Le fait que p54nrb puisse interagir à la fois avec le CTD de la Pol II, avec le pré-ARNm et avec certains snRNPs démontre l'implication directe de cette protéine dans le couplage spatio-temporel de la transcription et de l'épissage.
Figure 10. Modèle schématisant le complexe transcription/épissage (Kameoka et al., 2004) . Ce modèle montre la liaison de p54nrb au domaine CTD de la Pol II ainsi qu'au site d'épissage en 5'.
1.2.6. Rôle de la phosphorylation mitotique de p54nrb
Comme mentionné plus tôt, suite à la découverte de la phosphorylation mitotique de p54nrb, le laboratoire du Dr. Vincent se penche sur la caractérisation de la fonction de cette modification post-traductionnelle. Il est alors démontré que p54nrb est multi-phosphorylée par CDK1 sur trois threonines en C-terminal (T412, T430 et T452) et sur une threonine en N-terminal (T15) par une kinase inconnue lors de la mitose (Proteau et al., 2005). Il est bien connu que l'expression génique de la cellule est fortement réprimée lors de la division cellulaire. La phosphorylation de facteurs de transcription et de maturation des pré-ARNm par des kinases mitotiques est un des moyens utilisés par la cellule pour arrêter l'expression génique lors de la division cellulaire (Blencowe, 2003; Colgan et al., 1996; Gottesfeld and Forbes, 1997). Comme il a été discuté plus tôt, l'implication de p54nrb dans le couplage de la transcription et de l'épissage a été documentée à plusieurs reprises dans les dernières années. A la lumière de ces constatations, l'hypothèse de recherche qui est émise au laboratoire est que la phosphorylation de p54nrb pourrait participer à l'arrêt de la transcription et de l'épissage en mitose en inhibant sa capacité de liaison à l'ARN ou à certaines protéines.
Les travaux menés par Céline Bruelle, auxquels j'ai aussi activement participé, ont permis de répondre partiellement à ces questions. Dans le manuscrit en annexe (Bruelle et al., sous presse, voir annexe A), récemment accepté pour publication dans Biochemistry and cell biology, nous démontrons à l'aide d'expériences in vivo et in vitro que la phosphorylation mitotique de p54nrb n'a pas d'effet sur son interaction avec ses partenaires protéiques majeurs (annexe A, figures 2 et 3). Par contre, il y est démontré que la protéine est capable de lier un vaste éventail d'ARNs et que la phosphorylation de sa threonine 15 inhibe sa capacité générale de liaison aux ARNs excepté pour les ARNs riches en G (annexe A, figures 4, 5, 6 et 7). En effet, des expériences in vitro ont permis de constater que la protéine phosphorylée perd son affinité pour la séquence d'ARN du 5'SS alors qu'elle reste associée à la séquence riche en G de PIR-1 (le fragment de NEAT1 lié par p54nrb). Des résultats in vivo ont aussi confirmé le maintien de l'association de p54nrb et de NEAT1 à travers le cycle cellulaire. Il semblerait donc que la phosphorylation mitotique de p54nrb puisse moduler ses fonctions au niveau de la transcription et de l'épissage sans nécessairement affecter son association avec les paraspeckles. D'ailleurs, bien que cette affirmation soit contreversée, plusieurs études tendent à démontrer que les paraspeckles persistent tout au long du cycle cellulaire. Cette régulation différentielle de la liaison de p54nrb aux ARNs soulève plusieurs questions tant au niveau biologique que structural. D'un point de vue structural, on pourrait se demander qu'est-ce qui permet à p54nrb d'avoir cette spécificité pour les ARNs riches en G ? Quelles sont les affinités de la protéine pour les différents types d'ARN. La caractérisation de la liaison des RRMs de p54nrb à l'ARN n'a jamais été effectuée et pourrait bien nous révéler des nouvelles caractéristiques jamais répertoriées auparavant pour ce motif ubiquitaire.
1.3 Hypothèses et objectifs de recherche
Comme mentionné ci-dessus, la phosphorylation mitotique de la threonine 15 de p54nrb, située en amont de deux RRMs, module la liaison de cette protéine aux ARNs. Alors que cette modification post-traductionnelle de p54nrb inhibe sa liaison générale aux ARNs, sa liaison aux ARNs riches en G persiste. La caractérisation de la liaison des RRMs de p54nrb à l'ARN n'a jamais été faite. Comme mentionné plus tôt, le RRMl de p54nrb
possède toutes les caractéristiques classiques d'un RRM capable d'interagir avec l'ARN. Par contre, le RRM2 de cette protéine ne possède aucun résidu aromatique à la surface de son feuillet-P et ne semble donc pas être en mesure d'interagir avec de l'ARN de façon classique. Sur la base de ces résultats et observations, nous avons émis l'hypothèse initiale que l'interaction de p54nrb avec les ARNs riches en G pourrait présenter des caractéristiques différentes de son interaction générale à l'ARN. De plus, nous croyons que le RRMl de p54nrb est celui responsable de son interaction avec l'ARN.
Afin de vérifier ces hypothèses, une dissection moléculaire du domaine de liaison à l'ARN (DLA) de p54nrb, comprenant le N-terminal et les deux RRMs, a été réalisée. Le rôle des RRMs de p54nrb dans la liaison à différents types d'ARN a été vérifié par des tests de liaison in vitro et par résonance magnétique nucléaire (RMN). De plus, des expériences de titration par RMN des différents ligands d'ARN ont permis de cartographier les sites d'interaction de p54nrb avec ces molécules et d'en évaluer les affinités respectives.
C h a p i t r e II - Matériels et méthodes
2.1 Clonages, expression et purification des segments protéiques du DLA de p54nrb
2.1.1 Clonages
Une construction plasmidique contenant l'ADNc de p54nrb murin (NonO, annexe B) dans le vecteur pGEX-4Tl a été gracieusement fournie par F. Moreau-Gachelin (Hallier et al., 1996). La construction plasmidique du N-RRM1/2, fut générée par PCR sur l'ADNc de NonO en utilisant les amorces F (5'-CAT ATG CAG AGC AAT AAA GCC TTT AAC TTG-3') et R (5'-GAA TTC ACT CTT CAT CAT CTA ACT GGT CCA-3'). Le produit PCR fut inséré dans le plasmide pDRIVE (Qiagen) avant d'être digéré par Ndel et EcoRI et inséré par les mêmes sites dans le vecteur pET-30a (Novagen). La construction du N-RRM1 fut générée de la même façon en utilisant les amorces F (5'-CAT ATG CAG AGC AAT AAA GCC TTT AAC TTG-3') et R (5'-GAA TTC ATG CAC TGT GAC AGG CAA AGC GCA-3'). Une PCR sur l'ADNc de NonO par les amorces F (5'-CAT ATG CAC AGT GCA TCC CTT ACA GTC CGC-3') et R (5'-CTC GAG CTC TTC ATC ATC TAA CTG GTC CAT-3') fut réalisée pour générer la construction plasmidique du RRM2. Le produit PCR fut inséré dans pDRIVE avant d'être digéré par Ndel et Xhol et inséré par les mêmes sites dans le vecteur pET-30a.
2.1.2 Expression et purification
Les constructions plasmidiques des fragments protéiques du DLA de p54nrb ont été transformées dans les bactéries compétentes Rosetta-pLysS (Novagen) Escherichia coll. Pour l'expression des protéines non-marquées, les bactéries ont été incubées dans le milieu LB (Luria-Bertani broth) jusqu'à une densité optique (D.O.) de 0,4 à 600 nm avant d'être induites à 0,4 mM d'IPTG 16 h à 29 °C et 150 rpm. Pour les protéines N-RRM1 et RRM2 marquées au 15N ou au 13C, les bactéries furent incubées dans le milieu
minimal M9 contenant du 15NFLtCl ou du 15NH4C1 et du 13C6Hi206 jusqu'à une D.O. de
0,7 à 600 nm avant d'être induites à 0,4 mM IPTG 16 h à 29 °C à 250 rpm. Les bactéries ont ensuite été culotées et congelées 30 minutes à - 80 °C avant d'être resuspendues dans
le tampon de purification (500 mM NaCl, 20 mM PO4 pH 7,4, 5 mM imidazole), supplémenté du mélange d'inhibiteur de protease complete (Roche), et lysées à l'aide de l'EmulsiFlex-C3 (Avestin). Les fractions solubles des lysats furent filtrées (0,45 uM) et les protéines solubles purifiées par FPLC à l'aide de la colone His Trap 1ml (GE Healthcare). Des étapes successives d'élution à différents pourcentages de tampon d'élution (500 mM NaCl, 20 mM PO4 pH 7,4, 500 mM imidazole) ont été réalisées pour récolter les différentes protéines (N-RRM1/2 : 6 %, 30 % et 75 %; N-RRM1 : 12,5 %, 25 %, 60 %; RRM2 : 10 %, 30% et 75 %). Les fractions pures des protéines N-RRM1/2 et N-RRMl furent dialysées toute la nuit dans le tampon RMN1 (100 mM NaCl, 10 mM P04 pH 7) et les fractions du RRM2 dans le tampon RMN2 (300 mM NaCl, 10 mM P04
pH7) avant d'être concentrées avec un filtre Amicon Ultra (Millipore) et lyophilisées. Les échantillons protéiques lyophilisés furent repris dans les volumes appropriés afin d'obtenir les concentrations de sel désirées pour les différentes études.
2.2 Expériences de diffusion dynamique de la lumière sur le RRM2
Les expériences de DLS (dynamic ligth scattering) furent utilisés pour évaluer l'agrégation du RRM2. La protéine a été concentrée à 2 mg/ml dans les tampons désirés, filtrée à 0,45 um et les mesures de DLS effectuées à l'aide du Zetasizer Nano ZS (Malvern) à 25 °C. Cet appareil fonctionne à une longueur d'onde de 633 nm avec une puissance de laser de 4 mW et un angle de diffusion de 173 "C. Les rayons hydrodynamiques, présentés sous forme de distribution par volume dans l'étude, ont été évalués à l'aide du logiciel Zetasizer 6,01 et sont tous représentatifs d'une seule expérience d'au minimum 11 cumulatifs. À noter que ces expériences n'ont pas été faites en triplicata et que les valeurs obtenues ont seulement été observées qualitativement pour détecter la présence d'agrégats protéiques.
2.3 Dichroïsme circulaire
Le dichroïsme circulaire (DC) a été utilisé afin de vérifier la présence des structures secondaires attendues pour le RRM2. La protéine a été concentrée à 2 mg/ml dans le tampon expérimental (300 mM NaCl, 10 mM Tris pH 7). Le spectre de DC a été enregistré à température pièce à l'aide du spectropolarimètre Jasco J-710 avec 20 balayages de 250 nm à 190 nm. Les données ont été corrigées pour le bruit de fond et sont exprimées en ellipticité molaire moyenne des résidus [0] (mdeg • cm2 • dmol"1). La
largeur de la cellule de quartz utilisée est de 0,01cm.
2.4 Spectroscopic RMN
Au cours du projet, la RMN fut principalement utilisée afin de détecter les interactions entre les segments protéiques N-RRM1 et RRM2 et différents ligands d'ARN. Les perturbations des résonances amides du squelette des protéines suite à l'ajout des ligands ont été utilisées pour cartographier les sites de liaison et ont permis d'évaluer les affinités entre les molécules.
2.4.1 Conditions d'acquisition
Tous les spectres 2D ont été enregistrés avec un spectromètre Varian INOVA de 600 MHz muni d'une sonde à température ambiante à quatres canaux (*H, 2H, 15N, 13C). Les
spectres 'H-15N HSQC du N-RRM1 ont été enregistré à 25 °C avec des échantillons
contenant 0.2 mM 15N N-RRM1, 10 mM P04 pH 7, 0,02 % NaN3, 1 mM DTT, 3 mM
imidazole, 10 % D2O et dans certains cas le ligand désiré. Les spectres !H-15N HSQC du
RRM2 utilisés pour vérifier l'interaction de la protéine avec l'ARN ont été enregistrés à 15 °C sur des échantillons contenant 0.13 mM ,5N RRM2, 150 mM NaCl, 10 mM P04
pH 7, 3 mM imidazole, 10 % D2O et le ligand désiré. Les spectres 3D du RRM2 ont été enregistrés avec un spectromètre Varian ENOVA 500 Hz (McGill) munit d'une sonde cryogénique à quatres canaux sur un échantillon comportant 0,5 mM 15N 13C RRM2, 300
mM NaCl, 10 mM P04 pH 7, 3 mM imidazole et 10 % D20. Toutes les données de
RMN ont été converties avec NMRPipe (Delaglio et al., 1995) et visualisées et analysées avec NMRView (Jonhson, 1994)
2.4.2 Expériences de titration
L'ARN du site d'épissage en 5' (5'SS: 5'-AAAAAGGUAAG-3) et l'ARN de polyguanosines (polyG : 5'-GGGGGGGGGGG-3') ont été synthétisés chez Sigma-Aldrich et purifiés par HPLC avant d'être lyophilisé. Pour la titration du 5'SS, l'ARN a été resuspendu à 3 mM dans une solution contenant 0.2 mM 15N N-RRM1, 10 mM P04
pH 7, 0,02 % NaN3, 1 mM DTT, 3 mM imidazole, 10 % D20 et a été ajouté
graduellement à un échantillon RMN contenant 0.2 mM 15N N-RRM afin d'obtenir des
ratio 5'SS/N-RRMl de 0,25, 0,5, 0,75, 1, 1,5 et 2,5 et un spectre 'H-^N HSQC a été enregistré pour chaque ratio. Pour la titration du poly G, l'ARN a été resuspendu à 0,5 mM dans la même solution que pour le 5'SS et dilué graduellement à l'aide d'un échantillon RMN contenant 0.2 mM 15N N-RRM1 afin d'enregistrer des !H-15N HSQC à
des ratios polyG/N-RRMl de 0,25, 0,5, 0,75 et 1.
2.4.3 Évaluation des constantes de dissociation
Pour chaque titration, les changements de déplacements chimiques (CDC) des résonances amides attribuées du RRMl de p54nrb ont été calculés et normalisés selon l'équation suivante : AO(15N+!H) = (Ô!H2 + (Ô15N/6.5)2)1/2. Les CDCs plus grand que la moyenne des
CDCs plus l'écart-type et qui ne présentaient pas de chevauchement de pic lors des titrations ont été utilisés pour déterminer l'affinité du motif pour les différents ligands. Les CDCs des résonances séléctionées ont été portées sur un graphique en fonction du ratio ARN/protéine et les données ont été ajustés non linéairement selon l'équation d'un modèle de liaison à deux états:
A<50bs = A<5r
(Kd+(l+r)[P]0) - y(^+(l+/-)[P]o)2-4[P]5/-}
2[P]0
Le Aôobs dénote le CDC observé, Aômax le CDC maximal, [P]0 la concentration totale
de la protéine, r le ratio ARN/protéine et Kd la constante de dissociation du complex. L'ajustement des données a été réalisé à l'aide du logiciel GRACE et à permis d'évaluer
le Aômax et la Kd. Les Kd calculés pour chaque résidus, pour un ligand donné, ont été soumises à un test de Q (Dean and Dixon, 1951) pour éliminer les Kd écartées avec un niveau de confiance de 95 %.
2.5 Expériences de liaison à l'ARN in vitro
Pour les expériences de liaison réalisés avec la séquence du site d'épissage en 5' (5'SS, 5'-AAAAAGGUAAG-3'), des billes d'agarose-Streptavidine (Sigma-Aldrich) ont été incubées 1 h dans le tampon de blocage (TB : 20 mM Hepes pH7,4, 10 % glycerol, 100 mM KC1, 0,2 mM EDTA , 0,5 mM DTT , 5 % BSA) contenant le mélange d'inhibiteurs de proteases complete (Roche). Par la suite, les billes ont été rincées 1 fois avec 200 uL de tampon de liaison (TL : 20 mM Hepes pH7,4, 10 % glycerol, 100 mM KC1, 0,2 mM EDTA, 0,5 mM DTT, 0,5 % BSA) et 1,5 nmoles d'ARNs synthétiques du 5'SS biotynilés a été incubés avec 20 uL de billes pour chaque test 30 minutes à 4 °C. Les billes couplées à l'ARN ont été rincées 3 fois avec 150 uL de TL et 5 ug de protéines ont été incubées avec les billes dans un volume final de 50 uL de TL pendant 30 minutes à 4 °C. Après trois lavages de 150 uL de TL les billes ont été reprises dans 30 uL de tampon de migration (Laemmli IX : 62,5 mM Tris pH 6,8, 2,3 % SDS, 10 % glycerol, 5 % p-mercaptoéthanol, bleu de bromophénol) et les protéines liées ont été soumises à un immunobuvardage avec l'anticorps monoclonale a-His (Sigma-Aldrich).
Pour les tests de liaison réalisés avec l'homoribopolymère de G couplé à des billes d'agarose (Sigma-Aldrich), 5 uL de billes (3 ug d'ARNs) ont été incubées 1 h dans le tampon de blocage à 4 °C pour chaque test. Après 1 lavage de 200 uL de TL, 5 ug de protéine ont été incubées avec les billes 30 minutes à 4 °C dans un volume final de 50 uL de TL. Après 3 lavages de 150 uL de TL, les billes ont été resuspendues dans 30 uL de tampon Laemmli IX et les protéines liées ont été soumises à un immunobuvardage contre l'anticorps a-His.
2.6 Electrophorèse et immunobuvardage
Les différents échantillons protéiques ont été repris dans le tampon Lammli IX avant d'être résolus par SDS-PAGE sur des gels de 12,5 ou de 15 % (W/V, acrylamide : bis = 30 : 0,15) à un voltage constant de 150 V. Après migration, les protéines ont été colorées au bleu de Coomassie ou éléctrotransférées sur des membranes de nitrocellulose pendant lh à un voltage constant de 100 V. Les membranes ont été bloquées dans le tampon 7ns-bufferred saline (TBS : 10 mM Tris pH 7,6, 150 mM NaCl) contenant 5 % de lait en poudre à température pièce pendant 30 minutes avant d'être incubées dans les mêmes conditions 1 h avec l'anticorps monoclonal de souris a-His (Sigma-Aldrich) dilué à 1 :
1000. Après lavage de 15 minutes dans le tampon TBS-T (TBS + 0,1 % Tween) les membranes ont été incubées dans du TBS contenant l'anticorps secondaire GAM (goat a-mouse, Jackson Laboratories) dilué à 1 : 5000 pendant 45 minutes à température ambiante. Après 3 lavages de 10 minutes dans le TBS-T, la révélation des protéines a été réalisée par chemiluminescence (Roche).
2.7 Calcul du potentiel électrostatique de surface
Les potentiels électrostatiques de surface des RRMs de p54nrb ont été calculés avec l'outil APBS implémenté dans PYMOL et les figures visualisées à l'aide de PYMOL. Les régions électronégatives (-6 kT) sont colorées en rouge alors que les régions électropositives (+6 kT) sont en bleu.
C h a p i t r e III - Résultats
3.1 Dissection moléculaire du DLA de p54rnb : purification et mises au point pour études RMN
3.1.1 Présentation des segments protéiques du DLA de p54rnb générés et purifiés pour l'étude
Comme il a été mentionné plus tôt, la caractérisation structurale de la liaison des différents RRMs de p54nrb à l'ARN n'a jamais été réalisée. Afin d'élucider la fonction de ces motifs, une dissection moléculaire du DLA de la protéine a été réalisée. Trois constructions plasmidiques ont été effectuées afin de permettre l'expression bactérienne de différentes portions du DLA de p54nrb (figure 11 A). La construction N-RRM1/2 code pour le N-terminal (en amont du RRMl) et les deux RRMs de la protéine pour composer le DLA complet. La construction N-RRM1 comporte le N-terminal et le RRMl de p54nrb alors que la construction RRM2 contient seulement la séquence du RRM2. Le domaine endogène riche en histidines et en glutamines de p54nrb a été utilisé pour purifier les protéines N-RRMl/2 et N-RRMl sur colonne de nickel alors qu'une étiquette de 6 histidines a dû être ajoutée au RRM2 pour permettre sa purification par la même méthode. Les segments protéiques du DLA de p54nrb ont donc pu être purifiés par FPLC (Fast Protein Liquid Chromatography) en une seule étape sur colonne de nickel (figure 11B).
p54nrb (1-473) N-RRM1/2 (1-236) N-RRM1 (1-146) RRM2 (148-236)
B
kDa < N-RRM1/2 < N-RRM1 < RRM2Figure 11. Présentation des trois segments protéiques du DLA de p54nrb utilisés pour l'étude. A) Représentation schématique des trois segments protéiques utilisés dans l'étude. Les résidus de p54nrb contenus pour chacune des fractions ainsi que leur poids moléculaires respectifs en kDa sont écrits. La séquence de la région riche en histidines et en glutamines de p54nrb utilisée pour la purification sur colonne de nickel a été mise en évidence. B) Les différentes fractions du DLA purifiées ont été résolues sur gel SDS-PAGE et colorées au bleu de Coomassie.
3.1.2 Mises au point des conditions pour études par RMN
Avant d'entreprendre des études structurales sur le DLA de p54nrb, les conditions optimales nécessaires à l'acquisition de bons spectres RMN ont dû être déterminées. D'abord, la fraction protéique N-RRM1/2 ne s'est pas avérée une protéine idéale pour la RMN pour plusieurs raisons. En premier lieu, ce polypeptide précipite à très faible concentration (moins de 0,lmM) et il devient donc difficile d'en obtenir des échantillons concentrés pour permettre des études par RMN. Par la suite, les spectres !H-15N HSQC
enregistrés avec cette protéine donnent peu de signal et montrent très peu de dispersion (figure 12) suggérant que la protéine est agrégée ou dégradée dans les conditions utilisées. Finalement, comme N-RRM1/2 est une protéine de 236 acides aminés (27 kDa) , elle est un peu grosse pour donner un bon signal en RMN à sa concentration permissive (~ 0.1 mM). À la lumière de ces constatations, le fragment protéique N-RRM1/2 ne fut pas utilisé dans les différentes études réalisées par RMN.
Contrairement au N-RRM1/2, le segment protéique N-RRM1 est très soluble et donne un bon signal RMN. Des conditions similaires aux conditions utilisées pour la détermination de la structure du RRMl (pdb :2CPJ) ont été utilisées pour nos études par RMN sur ce polypeptide (voir matériels et méthodes). Dans ces conditions, le spectre 'H-15N HSQC
du N-RRMl montre une belle dispersion des pics (~ 3 ppm, figure 13) nous indiquant que la protéine est repliée. De plus, les déplacements chimiques de la structure du RRMl (PDB :2CPJ), généreusement fournis par le Dr Takashi Nagata, ont permis l'identification des résonances amides de la plupart des résidus du RRMl nous confirmant par le fait même le bon repliement de la protéine. La faible dispersion des pics non attribués correspondant à la portion N-terminale de la protéine sur le 'H-15N HSQC
(figure 13) nous indique que cette fraction ne semble pas repliée. De plus, bien que le N-terminal comporte 71 résidus dont les résonances amides sont non attribuées, il est seulement possible de compter une quarantaine de pics sans étiquette sur le 'H-^N HSQC (excluant les résonances de la région des chaînes latérales). Le fait que le N-terminal ne semble pas replié et qu'il soit riche en glutamines (19) et en histidines (8) cause probablement beaucoup de chevauchement des pics dans le spectre expliquant
pourquoi on ne voit pas tous les pics attendus. De plus, le N-terminal contient 7 prolines ne donnant pas de signal dans ce type de spectre.
1(14 106 108 110 112 114 D. 2 118 m r i 120 122 124 126 128 130 « « •
gpjr * -fir
•• • • • •*. j f r *
o • . ♦ 9.7 9.5 9.3 9.1 8.9 8.7 8.5 8.3 8.1 7.9 7.7 7.5 7.3 7.1 6.9 6.7 6.5 6 3 lH (ppm) I n 15KFigure 12. Spectre 'H- N HSQC de la protéine N-RRM1/2. Conditions utilisées : ~ 0.1 mM N-RRM1/2, 100 mM NaCl, 25 mM tampon P04 pH 7,2, 3 mM imidazole, 10% D20.
103 105 107 109 d14 0100 103 105 107 109 r at on ribué attribué d14 0100 103 105 107 109 r at d14 0100 103 105 107 109
H ' 1
d14 0100 103 105 107 109 d14 0100 111 « d ^ *A*
113 •8 1V
^r.
115 © " 5p l i a i t
0 •01 2 8• i
E C lz
n H 117 119 121 ©"• # * ^ 1 6 • ^ ^ 1 3 0 Wc
©87 123 125 127 0137 © A T I • ©102 OW «80 1" 0106 w 0.7 129 131 «0» 133 . « 9.7 9.5 93 9.1 8.9 8.7 8.5 8.3 8.1 7.9 7.7 75 13 7.1 6.9 6.7 65 *H (ppm)Figure 13. Spectre !H-15N HSQC du segment protéique N-RRM1. Conditions utilisées :
0.2 mM N-RRM1, 100 mM NaCl, 10 mM P04 pH 7, 0.02% NaN3, 1 mM DTT, 3 mM imidazole
et 10 % D20. Enregistré à 25 °C. Les pics attribués grâce aux données du Dr Nagata sont
étiquetés selon leur numéro de résidu par rapport à la séquence de p54nrb. Le spectre comprend un schéma des segments du N-RRM1 attribués et non attribués.
Pour ce qui est du RRM2, les premières expériences 'H-15N HSQC réalisées sur la
protéine ont généré de beaux spectres montrant une belle dispersion des pics (~ 3ppm) et un bon signal RMN. Par contre, dans les conditions utilisées (100 mM NaCl, 10 mM PO4 (pH 7) enregistré à 25 °C), le signal était presque totalement perdu après 21 heures (figure 14A). Comme l'attribution du RRM2 n'a pas été réalisée jusqu'à maintenant, il était essentiel avant de commencer l'étude, de trouver des conditions qui prolongeraient la durée du signal RMN du motif afin de permettre l'acquisition de bons spectres 3D pour l'attribution. D'abord, afin de déterminer si la perte du signal RMN était causée par de la dégradation protéique ou de l'agrégation, des échantillons du RRM2 ont été incubés 24 h dans les mêmes conditions de sel, de température et de pH que celles utilisées pour la RMN (100 mM NaCl, 25 °C, pH 7). La migration des échantillons avant et après incubation sur gel SDS-PAGE, suivie d'une coloration au bleu de Coomassie, a permis de voir que la dégradation n'était pas responsable de la perte du signal RMN (figure
14B). Par contre, l'étude de ces mêmes échantillons en DLS (Dynamic Ligth Scattering) a démontré que la protéine agrège en fonction du temps dans ces conditions (figure 14C). Il semblerait donc que l'agrégation du RRM2 soit responsable de la perte du signal RMN. Afin d'identifier les facteurs ayant le plus d'impact sur la vitesse d'agrégation de la protéine, des spectres 'H-^N HSQC du RRM2 ont été enregistrés à plusieurs temps pour différentes conditions de sels et de températures (toujours à pH 7). Les intensités de 21 pics de ces spectres, choisis aléatoirement, ont été suivies dans le temps et normalisées par rapport à leur intensité initiale pour toutes les conditions afin d'évaluer la vitesse d'agrégation de la protéine (figure 15A). Ces tests ont permis de voir que l'augmentation de la force ionique de l'échantillon et la baisse de la température d'acquisition étaient les facteurs clés pour stabiliser la forme non agrégée de la protéine (figure 15A, B). De plus, des échantillons du RRM2 incubés 24h dans 300 mM NaCl à 15 °C et suivis en DLS ont permis de confirmer que la protéine agrège très peu dans ces conditions (figure 15C).
■•* ■*: 90 * « t*. * * < : ..' 7J 7* -4 • : 7» * j M «4
'H(ppm)
B
Figure 14. Le R R M 2 de p54nrb agrège à faible force ionique et à haute température. A) Spectres 'H-15N HSQC du RRM2 à 0 h et 24 h. Conditions : 0.21 mM RRM2, 100 mM NaCl,
10 mM P04 pH 7, 3 mM imidazole, 10% D20. Enregistrés à 25 °C. B) Un échantillon du RRM2,
dans 100 mM NaCl, a été incubé à 25 °C 24h avant d'être résolu sur gel SDS-PAGE et coloré au bleu de Coomassie. Piste 1 : RRM2 initial (t : Oh); piste 2 : RRM2 après incubation à 25 °C (t : 24h). C) Donné de DLS obtenue sur les mêmes échantillons de RRM2 qu'en B. Rouge : RRM2 initial (t : Oh); Vert : RRM2 après incubation à 25 °C (t : 24h)
1 I I l l I l I l I I I 96 120 144 16S Time (h) 192 216 240 264
B
1
t:Oh
• -• •V *
• 3 «3 *.l t » 17 IS 13 I t 7* 77 75 73 71 *.* 47 *3 *J 'HJppm)t:90h
- a
• J M • 1 « I » 13 13 «I 7* 7 7 75 75 7 | 4* *7 *5 »5 •Hlpptn)Sis* Distribution by Volum*
? 10
> 5
Figure 15. L'augmentation de la force ionique et la baisse de la température sont les facteurs clés pour stabiliser l'agrégation du RRM2. A) Des spectres 'H-15N HSQC du
RRM2 ont été utilisés pour tester différentes conditions. Les spectres ont été enregistrés à différents temps pour chaque condition et les intensités de 21 pics, choisis aléatoirement, ont été divisées par leur intensité initiale pour tous les temps afin de quantifier la perte globale du signal RMN. Les valeurs moyennes des intensités relatives et leur écarts-types associés sont montrés en fonction du temps pour les différentes conditions. B) Spectres H- N HSQC du RRM2 dans 300 mM NaCl enregistrés à 15 °C à 0 h et 90 h montrant l'amélioration de la stabilité du signal RMN. Conditions : 0.21 mM RRM2, 300 mM NaCl, 10 mM P04 pH 7, 3 mM imidazole et 10% D20. C)
Un échantillon protéique du RRM2, dans 300 mM NaCl, a été incubé 24h à 15 °C avant d'être soumis à des expériences de DLS. Rouge: référence à 0 h. Vert: après incubation de 24 h.
Comme mentionné plus tôt, une analyse du spectre 'H-^N HSQC du RRM2 nous permet de constater une belle dispersion des pics d'environ 3 ppm suggérant que la protéine est bien repliée. Un spectre de dichroïsme circulaire (DC) réalisé sur ce polypeptide (figure
16) présente un patron typique des protéines comportant des structures secondaires d'hélice-a et de feuillet-P séparées avec un minimum prédominant dans les environs de 208 nm et un minimum moins important autour de 222 nm (Manavalan and Johnson, 1983). En effet, ce type de spectre de DC est très caractéristique des RRMs et confirme donc le bon repliement de la protéine (Lyon et al., 2009; Manival et al., 2001). Il est intéressant de noter qu'environ 110 et 120 pics sont respectivement observés pour les spectres *H-15N HSQC et 3D HNCO (figure 17) du RRM2 alors qu'il devrait
théoriquement y avoir seulement 102 pics (incluant les chaînes latérales) présents pour chacun de ces spectres. Ce nombre de pics légèrement trop élevé pourrait être dû à un échange conformationnel lent entre la forme agrégée et la forme soluble du motif, mais cela reste à confirmer. Certains spectres 3D nécessaires à l'attribution du squelette du RRM2 ont été enregistrés (HNCO, HNCA, HNCOCA et CBCACONH) et pourront donner plus d'informations sur la nature de ces pics supplémentaires.
200 210 220 230
Wavelength (nm) 240 250
Figure 16. La protéine RRM2 est bien repliée. Spectre de dichroïsme circulaire généré par le RRM2 de p54nrb. Conditions : RRM2 2 mg/ml, 300 mM NaCl, 10 mM Tris pH 7. Enregistré à température pièce.
170
0
0
171 172 f 173 174 E 175 a c.•••t
M l * «
•m
t
aa.
*4 0 0 • «i l * • »
is
0 ©
177n ^ d
178 » • " •t
1791
•v •
<? 180 IB1 • • 9 9.5 9.3 9.1 8.9 8.7 8.5 8.3 8.1 7.9 7.7 7.5 7.3 7.1 6.9 6.7 6.5 *H (ppm)Figure 17. Spectre 3D HNCO du RRM2 de p54nrb. Spectre 3D HNCO du RRM2 montrant une superposition de tous les plans de l'axe de l'azote (Z). Conditions : 0.5 mM RRM2, 300 mM NaCl, 10 mM P04 pH 7, 3 mM imidazole, 10% D20. Enregistré à 15 °C.
3.2 Caractérisation de la liaison du DLA de p54nrb à la séquence du site d'épissage en 5'
Comme mentionné plus tôt, p54nrb est impliquée dans le couplage spatio-temporel de la transcription et de l'épissage. C'est en liant directement le CTD de la Pol II, le site d'épissage en 5' (5'SS) et certains facteurs du spliceosome que p54nrb contribuerait au couplage de ces deux processus (Emili et al., 2002; Kameoka et al, 2004; Peng et al., 2002). De plus, lors de la mitose, cette protéine devient soudainement phosphorylée et perd son affinité générale pour les ARNs excepté pour ceux riches en G. En effet, des études de liaison in vitro, réalisées au laboratoire, ont permis de constater que la phosphorylation N-terminale de p54nrb régulait négativement sa liaison à la séquence du 5'SS alors que cette modification n'avait pas d'impact sur sa liaison à l'ARN riche en G NEAT1 ou à un homoribopolymère de G (Bruelle et al. (2011) sous presse, voir annexe A). À la lumière de ces résultats, nous avons émis l'hypothèse que l'interaction de p54nrb avec les ARNs riches en G pourrait présenter des caractéristiques différentes de son interaction générale à TARN. La prochaine section porte sur la caractérisation de la liaison du DLA de p54nrb au 5'SS.
3.2.1 Le RRM2 de p54nrb seul est incapable de lier le 5'SS in vitro
Le RRM2 de p54nrb ne possède pas les résidus aromatiques et basiques normalement conservés pour interagir avec l'ARN contrairement au RRMl qui est un motif classique. Notre hypothèse initiale était donc que le RRMl de p54nrb est le seul motif du DLA responsable de son interaction avec l'ARN. Afin de vérifier cette hypothèse, les différents segments protéiques du DLA de p54nrb ont été incubés avec des billes d'agarose couplées ou non à un ARN représentant la séquence conservée du 5'SS (10 nucleotides, voir matériels et méthodes) et les protéines ayant résisté à une série de lavages ont été soumises à un immunobuvardage avec l'anticorps a-His (figure 18). En accord avec notre hypothèse, les résultats montrent que contrairement aux constructions RRMl/2 et N-RRM1, le RRM2 seul est incapable de lier le 5'SS in vitro. Il semblerait donc que le RRMl de p54nrb soit le motif responsable de sa liaison au 5'SS. Par contre, comme le polypeptide N-RRM1 contient aussi la portion N-terminale de la protéine, des études par
RMN sont requises afin d'identifier les résidus du N-RRM1 impliqués dans l'interaction et de vérifier si le RRMl est réellement le motif responsable de l'interaction.