Université de Montréal.
Evolution des réponses CD4 et CD$ dirigées contre le VIH au cours de l’infection.
Par
Airne-Elen Kernaleguen
Département de microbiologie et immunologie Faculté de médecine
Mémoire présenté à la faculté des études supérieures en vue de l’obtention du grade de
Maître ès sciences (M.Sc) en microbiologie et immunologie
Juin 2005
(
[j
U5
o
ZÏCN
V.
icDirection des bibliothèques
AVIS
L’auteur a autorisé l’Université de Montréal à reproduire et diffuser, en totalité ou en partie, pat quelque moyen que ce soit et sur quelque support que ce soit, et exclusivement à des fins non lucratives d’enseignement et de
recherche, des copies de ce mémoire ou de cettethèse.
L’auteur et les coauteurs le cas échéant conservent la propriété du droit
d’auteur et des droits moraux qui protègent ce document. Ni la thèse ou le mémoire, ni des extraits substantiels de ce document, ne doivent être imprimés ou autrement reproduits sans l’autorisation de l’auteur.
Afin de se conformer à la Loi canadienne sur la protection des renseignements personnels, quelques formulaires secondaires, coordonnées
ou signatures intégrées au texte ont pu être enlevés de ce document. Bien
que cela ait pu affecter la pagination, il n’y a aucun contenu manquant.
NOTICE
The author of this thesis or dissertation has granted a nonexclusive license allowing Université de Montréal to reproduce and publish the document, in part or in whole, and in any format, solely for noncommercial educational and
research purposes.
The author and co-authors if applicable retain copyright ownership and moral rights in this document. Neither the whole thesis or dissertation, nor
substantial extracts from it, may be printed or otherwise reproduced without the author’s permission.
In compliance with the Canadian Privacy Act some supporting forms, contact information or signatures may haie been removed from the document. While this may affect the document page count, it does flot represent any loss of
Ce mémoire intitulé:
Evolution des réponses CD4 et CD8 dirigées contre le VIH au cours de l’infection.
présenté par:
Anne-Elen Kemaleguen
a été évalué par un jury composé des personnes suivantes:
Dr Hugo Soudeyns président-rapporteur
Dr Rafick-Piene Sékaly directeur de recherche Dr Bader Yassine-Diab co directeur
Dr Nicole Bernard membre du jury
Afin d’étudier l’impact des mutations du VIH sur le développement des réponses T CD4 et CD$ dans une étude longitudinale, nous avons dans un premier temps caractérisé les épitopes spécifiques au VIH chez des patients en primo infection en mesurant la réponse proliférative de ces cellules à l’aide de la méthode du CFSE.
En utilisant une matrice de 147 pools de 10 à 12 peptides couvrant la totalité du génome consensus de la clade B du VIH-1, nous avons pu mettre en évidence
l’existence d’un grand nombre d’épitopes capables d’induire une réponse
proliférative des cellules CD4 et/ou CD$ en primo infection. Nos résultats ont également permis de mettre en évidence la présence d’une corrélation entre le nombre d’épitopcs capables d’induire une réponse proliférative des cellules T et la durée de d’infection.
Les épitopes “partagés’t (i.e. provoquant à la fois une réponse des cellules
CD4 et CD8) n’avaient à notre connaissance jamais été décrits auparavant et
semblent contribuer à l’élimination de la réponse proliférative des cellules CD4
spécifiques du VIH in vitro. On constate en effet une augmentation du pourcentage
de réponse des cellules CD4 lorsqu’elles sont stimulées en absence des cellules
CD8.
La diminution du nombre d’épitopes induisant une réponse proliférative de même que la disparition des réponses CD4 au cours du temps semblent donc être en partie dues à l’influence des cellules T CD8 spécifiques du VIH sur les cellules
CD4.
ABSTRACT
Our objective is to detenriine how mutations in HIV affect the generation of
CD4 and CD8 T celi responses in a longitudinal study. To achieve this, we studied
virus-specific T celi responses in PBMCs from acutely infected patients using a
CFSE proliferation assay.
Using a matrix of 147 pools of 10 to 12 overlapping peptides spanning the whole HIV-1 clade B consensus genome, we have defined a panel ofepitopes which are able to induce both CD4 and/or CD8 proliferative response in acutely infected untreated patients. Our results also suggest the presence of a correlation between the number of HW-specific epitopes recognized by CD4 and CD8 T cells and the onset of infection.
To our knowledge, the TTshared epitopes” (i.e. the ones that induce both a CD4 and a CD8 response) have neyer been described and interestingly seem to be involved in the elirnination of the HIV specific CD4 proliferative response. Indeed,
in vitro stimulation of CD4 T celis by these “shared epitopes” following the removal
ofthe CD8 cornpartment enabled an increase in the CD4 proliferation.
Therefore, our data show that the HIV specific CD$ T ceils seem to have a strong influence on CD4 T celis responses. Indeed, they play an important role in the reduction in the number of epitopes able to induce a proliferative response as well as the disappearance of CD4 responses.
TABLE DES MATIÈRES
PAGE DE TITRE j
PAGE D’IDENTIFICATION DU JURY ii
RESUME iii
ABSTRACT iv
TABLE DES MATIÈRES y
LISTE DES TABLEAUX ix
LISTE DES FIGURES x
LISTE DES ANNEXES xii
LISTE DES ABRÉVIATIONS xiii
REMERCIEMENTS x vii
INTRODUCTION 1
1. Activation des lymphocytes T 1
1.1. Généralités sur le système immunitaire 1
1.2. Lyn;phocytes T 2
1.3. Présentation antigénique 6
1.3.1. Voie de présentation par le CMH de classe I 6
1.3.2. Voie de présentation par le CMH de classe II 8
1.4. Le récepteur des cellules T 9
1.4.1. Organisation génétique du TCR 10
1.4.2. Recombinaison cLf3 11
1.4.3. Polymorphisme 13
1.4.4. Structure du TCR 14
1.4.5. Interaction du TCR avec le complexe CMH-peptide 17
1.5. Activation des cellules T 19
1.5.2. Différenciation et prolifération des cellules T 22
1.6. La réponse des cellules T CD4 23
1.6.1. Expansion 23 1.6.1.1 Activation 23 1.6.1.2 Prolifération 24 1.6.1.3 Différenciation 24 1.6.2. Contraction 27 1.6.3. Mémoire 29
1.7. Importance de la réponse CD4 lors d’une infection virale 31
1.8. Réponse CD4 dans le cas de l’infection par le VIH 33
2. Le virus de I’immunodéficience humaine de type I 36
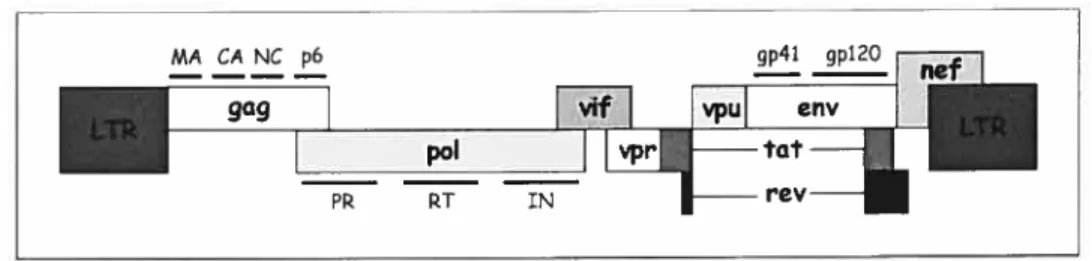
2.1. Généralités et organisation génétique 36
2.1.1. Généralités 36
2.1.2. Organisation génétique duHIV-1 36
2.2. Cycle de réplication du virus 32
2.3. Variabilité génétique 39
2.4. Tropisme cellulaire 39
2.5. Evolution naturelle de l’infection 41
2.6. Pathogénèse virale 44
2.7. Stratégies d’évasion virale 46
2.7.1. Mécanismes de sabotage 47
2.7.2. Mécanismes d’exploitation 48
2.7.3. Mécanismes de camouflage 48
INTRODUCTION AU TRAVAIL ET BUT DU PROJET 50
CHOIX DE LA MÉTHODE UTILISÉE POUR LE CRIBLAGE DES
MATERIEL ET METHODES .60
1. Patients 60
2. Peptides 61
4. Isolation des cellules mononuclées du sang périphérique 63
5. Déplétion autoMACS 63
6. Coloration CFSE 64
7. Anticorps 65
8. Coloration de surface 65
9. Coloration intracellulaire 66
10. CBA (Cytornetric Bead Array) 67
RESULTATS 6$
1. Choix des patients 68
2. Stimulation des cellules 70
2.1. Influence du nombre de peptides constituant un pool, sur la réponse
proliférative observée 70
2.2. Réponses prolifératives après stimulation par un pool de peptides 72
2.3. Caractérisation des peptides immunogènes 72
3. Répertoire des cellules T CD4 et CD8 spécifiques du VIH 73
3.1. Seuil de positivité des réponses prolifératives 73
3.2. Variation du nombre d’épitopes reconnus en fonction du temps 75
3.3. Distribution des réponses dans les compartiments CD4 et CD8 77
3.4. Pourcentage des réponses prolifératives observées pour les différentes
protéines du VIH 80
4. Reconnaissance de peptides individuels par les cellules T CD4 et CD8
spécifiques du VN 81
DISCUSSION $6
1. Choix des patients. 86
2. Séquence des peptides utilisés pour les stimulations 87
+ +
3. Repertoire des cellules T CD4 et CD8 specifiques du VIH 89 3.1. Evolution des réponses prolifératives spécifiques au VIH au cours du
temps 89
3.2. Existence de régions immunodominantes à l’intérieur de certaines
protéines 92
4. Reconnaissance de peptides individuels par les cellules T CD4 et CD8
spécifiques du VIH 94
5. Evolution des réponses prolifératives CD4 et CD8 spécifiques du VIH 95
CONCLUSION ET PERSPECTIVES 97
REFERENCES 101
LISTE DES TABLEAUX
Tableau I Famille des gènes du TCR chez les humains 10
Tableau II Caractéristiques des patients 67
LISTE DES FIGURES
Figure 1 Rôle des lymphocytes T CD4 dans la réponse immunitaire 5
Fïgure 2 Organisation des gènes du TCR 11
Figure 3 Structure d’un TCR ctf3 16
Figure 4 A) Structure du complexe TCR HA-HA1.7/DR1 1$
B) Orientation du TCR lors de l’interaction avec le complexe CMH-peptide 18
Figure 5 Cascade de signalisation menant à l’activation de la cellule T suite à
l’engagement du TCR par un complexe CMH-peptide 21
Figure 6 Modèles de différenciation des cellules mémoires 29
Figure 7 Organisation du génome du VIII-1 37
Figure 8 Représentation schématique de l’évolution naturelle du taux de CD4 et de la charge virale au cours de l’infection par le VIH-1 chez un progresseur typique.. 43
Figure 9 Principe de la méthode du CBA 54
Figure 10 Comparaison réponses détectées par différentes méthodes : (A) CFSE,
(B) ICS ou (C) CBA 56
Figure 11 Cinétique d’expression des différentes cytokines dosées par CBA 57
Figure 12 Variabilité intra-essai de la méthode du CFSE 59
Figure 13 Inter-variabilité de la méthode du CfSE 59
Figure 14 Stimulations peptidiques réalisées suivant une méthode de matrice 62
Figure 15 Réponses prolifératives observées en fonction de la taille des pools 71
Figure 16 Réponses prolifératives des cellules CD4 et CD8, observées après une
stimulation par un pooi de peptides du VIH 71
Figure 17 Exemple de réponses observées lors d’une stimulation suivant une
méthode de matrice 72
Figure 1$ Exemples de réponses observées lors d’une stimulation peptidique 74
Figure 19 Diminution du nombre d’épitopes provoquant une réponse spécifique du
VIH au cours du temps 76
Figure 21 Distribution des réponses dans les compartiments CD4 et/ou CD8 au
cours du temps 79
Figure 22 Potircentage des réponses prolifératives observées pour les différentes
protéines du VIH 79
Figure 23 Réponses prolifératives des cellules CD4 et CD8, observées après une
stimulation par un peptide individuel 81
figure 24 Impact des épitopes provoquant à la fois une réponse CD4 et CD8, sur le
développement d’une réponse des cellules T CD4 83
Figure25 Evolution des réponses prolifératives CD4 et CD8 au cours du temps... 85
LISTE DES ANNEXES
Annexe 1 Séquence des peptides Env consensus clade B xviii
Annexe 2 Séquence des peptides Gag consensus clade B xx
Annexe 3 Séquence des peptides Nef consensus clade B xxi
Annexe 4 Séquence des peptides Pol consensus clade B xxii
Annexe 5 Séquence des peptides Rev consensus cladeB xxiii
Annexe 6 Séquence des peptides Tat consensus clade B xxiv
Annexe 7 Séquence des peptides Vif consensus clade B xxv
Annexe 8 Séquence des peptides Vpr consensus clade B xxvi
Annexe 9 Séquence des peptides Vpu consensus clade B xxvii
LISTE DES ABRÉVIATIONS
ADCC : Antibody Dependent Celi Cytotoxicity ADN : Acide désoxyribonucléique
APC : Allophycocyanine
ARN t Acide Ribonucléique
ARNm : Acide Ribonucléique messager ASK1: Apoptosis Signal-regulating Kinase 1
f32m : f32 microglobuline
BD t Becton Dickinson
BSA s Bovine Serum Albumin CA s p24 capsid
CBA: Cytornetric Bead Array CD4 s Cluster of Differenciation 4
CDR: Complementarity Determining Region CCR5 s chemokine (C-C motif) Receptor 5 CD40L: Cluster of Differenciation 40 Ligand
CFSE: 5-6-Carboxyfluorescein diacetate Succinimidyl Ester CLIP s Class II associated Invariant chain Peptide
CMH s Complexe Majeur d’Histocornpatibilité CMV: Cytomegalovirus
CPA: Cellule Présentatrice d’Antigènes CTL: Lymphocytes T Cytotoxiques
CTLA-4 : Cytotoxic T-Lyrnphocyte-Associated protein 4 CXCR4: chemokine (C-X-C motif) Receptor 4
cyt c : cytochrome e DAG : Diacylglycérol
DC-SIGN : Dendritic Celi -Specific ICAM3 -Grabbing Non-integrin Degré Celsius
DO : Densité Optique EBV : Epstein-Barr virus
EDTA : Disodium Éthylènediarninetétra-acétate dihydrate ELISA : Enzyrne-Linked Immunosorbent Assay
Env : Enveloppe
FACS ® :fluorescence-Activated Celi Sorter
GM-CSF: Granulocyte-monocyte colony-stimulating factor Gag: Group spécifie antigens
HA : Hemaggiutinine A
HLA : Hurnan Leucocyte Antigen
HPLC : High Performance Liquid Chromatography HSP : Heat-Shock Proteins
HSV : herpes simplex virus ICS : IntracelÏular staining IFN-y : Interféron-y
1g : Immunoglobulines
IL-2 : Interleukine-2
IN: Intégrase
1P3 : Inositol- 1 ,4,5-triphosphate
ITAM: liTimunoréceptor Tyrosine-based Activation Motif
KIR : Killer Inhibitory Receptors LAT : Linker for Activation of T ceils
LCMV : Lyrnphocytic Choriorneningitis Virus LFA-1 : Leucocyte function-associated Antigen-1 LMP : Low Molecular Protein
LSRII : Live Science Research II
LTNP : Long Term Non Progressor (non progresseurs à long terme) LTR: Long Terminal Repeats
MA: p17 matrix
MAP : kinasesMitogen-Activated Protein Kinase microlitre
n1 : millilitre mM : millimolaire NC : nucleocapsid Nef: negative effector
NF-AT : Nuclear Factor of Activated T celi NFicB : Nuclear f actor KB
ng : nanogramme NK : Natural Killer
ORF : Open Reading Frame (cadres de lecture ouverts) PBMC : Peripheral Blood Mononuclear Celis
PBS : Phosphate Buffered Saline PE : Phycoérythrine
PerCP : Peridin Chlorophyll Protein PIP2 : Phosphoinositol-4,5-bisphosphate PKC : Protéine Kinase C PLCyl : Phospholipase C‘ I Pol : Polymérase PR : Protéase PTK : Protéines Tyrosine-Kinases RAG : Recombination Activating Genes RE : Réticulum Endoplasmique
Rev : regulation of expression of viral proteins Rpm : rotation par minute
RRE : Rev-responsive element RSS : Recognition Signal Sequence RT : Reverse Transcriptase
SEM: Staphylococcal Enterotoxin Mix SH2 : Src Homology 2
SDA: Syndrome de 1’Immunodéficience Acquise
SW : Virus de l’Irnrnunodéficience Simienne
S/L$ : Sensitive/Less Sensitive
STAT1 : Signal Transducer and Activator of Transcription 1 STI: Stntctured Treatment Interruption
SU: surface (ou gpl2O)
TAP : Transporter associated with Antigen Processing TAR: Trans-Activating Response elernent
Tat : Transactivator of Transcription TCM : cellules T Centrales Mémoires TCR: T Celi Receptor
TdT: Terminal deoxynucteotidyl Transferase TEM: cellules T Effecteurs Mémoires
Th: T helper (auxilliaire)
TM : transmembrane (ou gp4l) TNF-u : tumor necrosis factor-u. TT : Tetanus Toxoïd
Vi: Visite 1
V, D, J : Variable, Diversity, and Joining gene segments VHC : Virus de l’Hépatite C
Vif: Viral infectivity factor
VIH-1 : Virus de l’Irnmunodéflcience Humaine de type 1
Vpr: Viral protein r
Vpu : Viral protein u
REMERCIEMENTS
Je remercie te Dr Rafick-Pierre SÉKAL Y, mon directeur de recherche, de ni ‘avoir
accueillie dans son laboratoire, me donnant ainsi Ï ‘occasion de participer à un
projet aussi intéressant.
Je remercie le Dr Bader YASSINE-DIAB, mon co-directeur de recherche, de
in ‘avoir intégrée dans son équtpe et de in ‘avoir permis de profiter de son expérience
et de son enthousiasme.
Je remercie également tous les membres du laboratoire passés et présents, pour toute 1 ‘ciide qu ‘ils ont pu ni ‘apportel- ainsi que pour tous tes bons moments passés ensemble!
Plus particulièrement je remercie Olivier Chambenoit, GaêlÏe Breton, Sandrine Crabé, catherine Rion, Lina Kalfayan, Nicolas chomont Karine Maisnier-Patin et
Lydie Trautntann merci potcr les discussions du midi, les barbecues à la belle
étoile, les éclats de rire Merci surtout pour toute t ‘aide et tout le soutien que vous
1. Activation des lymphocytes T
1.1. Généralités sur le système immunitaire
Le système immunitaire est composé d’un ensemble de cellules et molécules dont le rôle est de défendre l’organisme contre les infections. La première ligne de défense de l’organisme est constituée par les barrières de surface physiques et chimiques formées par la peau et les membranes muqueuses. Pour pouvoir établir une infection, les pathogènes devront donc traverser ces barrières et créer une brèche dans l’ectoderme. Ils seront alors confrontés aux réponses immunitaires innée et acquise.
[1]
L’immunité innée (ou naturelle) regroupe tous les mécanismes de défense présents avant une infection. Ces mécanismes sont capables de fournir une réponse rapide, mais sont peu spécifiques et dépourvus de mémoire immunologique. Par conséquent, une réponse innée présentera toujours le même profil (qualitatif et quantitatif) quel que soit le nombre de fois où l’antigène aura été rencontré. [1]
Les composantes moléculaires de l’immunité innée comprennent les leucotriènes, les prostaglandines, certaines chernokines, les protéines de la phase aiguè (telles que la protéine C-réactive) ainsi que les éléments de la voie alterne d’activation du complément. [1] Au niveau des composantes cellulaires, on retrouve à la fois des cellules phagocytaires (tels que neutrophiles et macrophages), des cellules sécrétant des facteurs d’inflammation (tels qu’éosinophiles et basophiles) et les cellules NK (Natural Killer). Ces dernières sont spécialisées dans la lyse de cellules tumorales ou infectées par un virus, et ayant down-régulé le complexe majeur d’histocompatibilite de classe I (CMH I). Leur activité cytotoxique est en effet abolie à la suite de la reconnaissance du CMH de classe I par des récepteurs inhibiteurs présents à la
Les réponses acquises (ou spécifiques) sont plus lentes à apparaître. Elles sont provoquées par tine cascade d’évènements initiés suite à la rencontre d’un
antigène et seront dirigées spécifiquement contre cet antigène. Une des
caractéristiques principales de l’immunité acquise est sa capacité à établir une mémoire immunologique à l’issue de la première rencontre avec un pathogène. Grâce à cette mémoire, un deuxième contact avec le même antigène provoquera une réponse plus rapide et plus forte. [1]
L’immunité acquise peut générer deux types de réponses une réponse à médiation
humorale, qui fait intervenir les lymphocytes B et une réponse à médiation cellulaire, qui fait intervenir les lymphocytes T. Les lymphocytes B reconnaissent principalement des antigènes particulaires et sont spécialisés dans l’élimination des antigènes solubles. Les lymphocytes T reconnaissent quant à eux des peptides linéaires (issus de la protéolyse intracellulaire d’antigènes) présentés dans le contexte d’une molécule de CMH. [2]
1.2. Lymphocytes T
Les lymphocytes T jouent un rôle central dans l’immunité acquise. Il existe deux classes de cellules T, identifiables par la nature du TCR (T Cell Receptor)
qu’elles expriment à leur surface : ou ‘yi3. Les lymphocytes T sont localisés
dans les tissus épithéliaux. Les antigènes qu’ils reconnaissent ne sont pas encore bien caractérisés, mais on peut citer parmi eux les antigènes de classe lb, les HSP (Heat Shock Proteins) ou certains antigènes de nature bactérienne. En outre, la reconnaissance de ces antigènes n’est pas restreinte par le CMH. [3-5] Les lymphocytes cLf3 reconnaissent des antigènes de nature peptidique présentés par une
CPA (Cellule Présentatrice d’Antigènes) dans le contexte du CMH de classe I ou II.
bien définies les cellules T cytotoxiques, qui possèdent le co-récepteur CD8, et les
cellules T auxiliaires, qui expriment le co-récepteur CD4.
Les lymphocytes T CD$ reconnaissent des peptides dérivés de protéines cytosoliques (le plus souvent synthétisées de façon endogène) dans le contexte du CMH de classe I. Ils reconnaissent donc des cellules présentant des peptides du “soi
modifié” (i.e. cellules infectées par un virus ou cellules tumorales). Ils sont
également impliqués dans les réactions de rejet de greffe. [l;21 Suite à leur activation, ils se différencient en lymphocytes T cytotoxiques (CTL) et expriment diverses cytokines (interféron (IFN)-y, interleukine (IL)-2 et turnor necrosis factor (TNF)-a). Les CTL sont également capables de lyser leurs cellules cibles (i.e. celles exprimant le complexe CMH-peptide ayant déclenché leur activation) de façon
spécifique par un mécanisme contact-dépendant. Pour ce faire, ils sécrètent des
granules cytotoxiques contenant de la perforine et des granzymes. La perforine est une protéine libérée sous forme monomérique qui polymérise au niveau de la membrane de la cellule cible pour fonrier des pores. Les granzymes A et B sont des sérine-protéases qui, une fois entrés à l’intérieur de la cellule cible, vont induire son apoptose par une voie indépendante des caspases. [6;7J
Les lymphocytes T CD4 reconnaissent des peptides dérivés principalement de protéines extracellulaires ayant été intemalisées et dégradées par une CPA. Ces peptides sont alors présentés dans le contexte du CMH de classe II. Les cellules T
CD4 effectrices, ou T auxiliaires (Th), jouent un rôle fondamental dans l’activation
des cellules B, des autres cellules T, ainsi que des autres types cellulaires participant
à la réponse immunitaire. Ils sont en outre capables de stimuler l’expansion des
progéniteurs de la moelle osseuse en sécrétant de l’IL-3 et du GM-CSF
(Granulocyte-Monocyte Colony-Stirnulating Factor). Il existe deux sous-populations de cellules T auxiliaires, ThI et Th2, qui se distinguent par le type de cytokines qu’elles produisent et le type de réponse qu’elles provoquent.
Les Thi sont responsables de l’activation des fonctions régulées par l’immunité à médiation cellulaire comme l’activation des CTL. Ils produisent de
l’IFN-y qui agit d’une part comme un inhibiteur de la différenciation en Th2 et
provoque d’autre part l’activation des macrophages. Une fois activés, les
macrophages sécrètent de l’IL-12 qui agit sur les T CD4 en entraînant leur différenciation en Thi. La polarisation de la réponse s’en trouve de ce fait augmentée. Les Thi sont également producteurs d’IL-2 qui agit comme un facteur de croissance autocnne et dont l’action, couplée à celle de l’IFN-7, provoque la différenciation des CD$ et CTL. Les Thi sont donc particulièrement importants dans la réponse immunitaire dirigée contre les infections virales et les pathogènes intracellulaires.
Les Th2 sont plus impliqués dans la branche humorale de la réponse immunitaire. Ils induisent l’activation des éosinophiles en produisant de l’IL-5. Ils sécrètent aussi de l’IL-4 qui stimule le phénomène de commutation de classe chez les lymphocytes B pour induire la production d’immunoglobulines (Ig)G et d’IgE. De plus, l’IL-4 stimule l’activation des mastocytes. Les Th2 produisent également de t’IL-lO et de l’IL-13 qui agissent avec l’IL-4 pour augmenter la différenciation Th2 et inhiber l’action des macrophages et des Thi. Les Th2 sont donc plutôt impliqués dans les réponses aux antigènes solubles ainsi que dans les réactions allergiques. [2;$-12]
Figure 1 Rôle des lymphocytes TcD1 dans la réponse immunitaire
D’après Delves, P.J. et Roitt, I.M., The Immune System - Second of Two
Parts.NEngtJMed2000. 343: 10$-117.
Les lymphocytes T CD4 contrôlent virtuellement toutes les réponses
immunitaires face aux antigènes protéiques. Ils ont donc un rôle déterminant dans le
système immunitaire. CPA Cellule T CD4” naïve Cellule Lyse des — cellules infectées IFN-y Ccl1ulesT’
Activation des macrophages IFN-v
jouet différenciation
Activationdes éosinophiles
Activation des mastoes
Cellules Th2
1g-G 1g-E Cellule B Planocyte
1.3. Présentation antigénïque
Les lymphocytes T ne reconnaissent un antigène que s’il est présenté sous la forme d’un peptide complexé à une molécule de CMH. Les cellules CD4 et CD8 ne reconnaissent toutefois pas le même type d’antigènes. En effet, alors que les cellules T CD4 reconnaissent des antigènes extracellulaires (exogènes), les cellules
T C D8sont s timulées p ar d es protéines d ‘origine intracellulaire (endogènes). C es
deux types d’antigènes suivront donc deux voies de dégradation et de présentation différentes. Les antigènes endogènes seront dégradés par la voie e ytosolique pour être présentés dans le contexte du CMH de classe I. Les antigènes exogènes, eux, emprunteront la voie endocytaire et seront présentés par le CMH de classe II. [13]
1.3.1. Voie de présentation par le CMH de classe I
Le CMH de classe I est un hétérodimère constitué par l’association non
covalente entre une chaîne lourde polymoiphique (chaîne u) et la f32 microglobuline
(J32m). Les molécules de CMH de classe I présentent quatre domaines : trois formés
par la chaîne u et un par la f32m. La niche peptidique, située entre les segments u1 et
u2 de la chaîne lourde, est fermée à ses deux extrémités et ne peut donc fixer que des
peptides de petite taille n’excédant pas 8 à 11 acides-aminés. [14] Chez l’humain, les
gènes du CMH se trouvent tous sur le bras courtdu chromosome 6 (6p21.3). La
chaîne u du CMH de classe I est codée par trois gènes (Human Leucocyte Antigen (HLA)-A, -B et —C) hautement polyrnorphiques. Pour chaque gène, deux allèles sont exprimés de façon co-dominante. La f32m, quant à elle, est codée par un gène situé sur le chromosome 15. Le CMH de classe I est exprimé de façon constitutive à la stirface de toutes les cellules nucléées. Son expression peut toutefois être modulée sous l’action de certaines cytokines (tel que l’IFN-y, qui l’augmente). [13]
Dans la plupart des cellules, le CMH de classe I s’associe exclusivement avec des peptides dérivés de protéines cytoplasmiques endogènes telles que les
protéines codées par des gènes viraux, lors d’une infection, ou des antigènes tumoraux. Il e xiste cependant d es cas où I e C MH de classe I p eut présenter des antigènes exogènes. Dans ce cas précis, l’antigène sera dégradé en suivant la voie endocytaire. [15] Ce processus appelé ‘Tcross-présentation” n’a toutefois été décrit que dans le cas de cellules phagocytaires (cellules dendritiques et macrophages principalement).
De façon classique, les protéines cytoplasmiques sont ubiquitinylées et dirigées vers le protéasome où elles seront dégradées. Le protéasome est un complexe multirnérique constitué de sept sous-unités, dont trois comportant des sites catalytiques. Sous l’action de l’IFN-y, ces trois sous-unités seront remplacées par LMP (Low Molecular Protein)2, LMP7 et LMP1O, sous-unités codées par des gènes se trouvant au niveau des gènes du CMH et ayant des spécificités différentes. Le protéasorne ainsi modifié est appelé immunoprotéasome. [16] Les peptides issus de
la dégradation des antigènes cytosoliques sont ensuite dirigés vers le réticulum
endoplasmique (RE).
Les deux chaînes polypeptidiques formant la molécule de CMH de classe I
sont synthétisées séparément au niveau du RE. La chaîne Œ et la f32m vont ensuite
s’assembler et se replier dans la lumière du RE sous l’action de deux chaperonnes la calnexjne et la calréticuline. Le dirnère a/J32m ainsi formé va ensuite s’associer à une petite protéine, la tapasine, qui catalyse le chargement de la molécule de CMH de classe I par un peptide, en permettant l’interaction du CMH avec TAP (Transporter associated with Antigen Processing) au niveau de la membrane du RE. TAP est un tranporteur de peptides constitué de deux sous-unités: TAPi et TAP2, qui interagissent pour former un canal dans la membrane du RE. Les peptides sortant du protéasorne vont se lier à la partie cytosolique de TAP et traverser le canal pour parvenir dans la lumière du RE où ils se fixent sur les molécules de CMH de classe T. La liaison d’un peptide stabilise l’interaction entre la chaîne Œ et la f32m. Les
molécules de CMH de classe I chargées avec un peptide se séparent alors du complexe TAP/tapasine et transitent par l’appareil de Golgi pour être exporté vers la surface de la cellule où elles seront présentées aux lymphocytes T CD$. [17-19]
1.3.2. Voie de présentation par le CMH de classe II
A l’instar des molécules de CMH de classe I, les molécules de CMH de classe II forment une famille de protéines hétérodirnériques polymorphiques. Il existe trois isofornies de molécules de CMH de classe II (HLA-DR, -DP et -DQ),
dont les gènes sont également situés sur le bras court du chromosome 6. Chaque
molécule est constituée de deux chaînes polypeptidiques la chaîne u et la chaîne
f3.
Chacune de ces chaînes est polymorphique (sauf dans le cas de la chaîne u du HLA DR), et on observe l’expression de deux allèles de façon codominante. En conséquence, le nombre de molécules de CMH de classe II pouvant être exprimées chez un individu hétérozygote est de 10 à 20. Les molécules de classe II sont
formées p ar q uatre domaines t u1, u2, f3i, et f32. La niche p eptidique est située au
niveau du site d’interaction entre les chaînes u et
f3.
au niveau des domaines ai et f3i.Contrairement à celle de la molécule de CMH de classe I, la niche peptidique de la molécule de classe II est ouverte à ses deux extrémités, ce qui lui pemet de fixer des peptides dont la taille peut varier de 13 à 22 acides-aminés. [16] Le CMH de classe II n’est exprimé qu’à la surface des CPA dites “professionnelles” (soit cellules dendritiques, macrophages et lymphocytes B) et sur les cellules T activées. Son expression peut être modulée par certaines cytokines comme l’IFN-y. [13]
Les peptides présentés par le CMH de classe II sont des peptides issus de la dégradation de protéines extracellulaires. Ces antigènes exogènes sont intemalisés par endocytose ou par phagocytose. Les vésicules ainsi formées sont ensuite dirigées vers le compartiment endosomal où les protéines seront dégradées par voie enzymatique.
Les chaînes u et
f3
du CMH de classe II sont synthétisées au niveau du RE oùelles s’assemblent. Pour éviter que la molécule de CMH de classe II ne soit chargée
avec un peptide à ce stade, la chaîne invariante (Ii) bloque la niche peptidique de façon temporaire. Elle a également pour rôle de diriger le complexe ainsi formé vers
le compartiment endosomal. La chaîne invariante est alors dégradée par des
protéases telles que les cathepsines, jusqu’à ce qu’il ne reste qu’un petit peptide de 24 acides-aminés appelé CLIP (class II associated invariant chain peptide) qui
Q
bloque la niche peptidique. [20] Celui-ci sera enlevé par la molécule de CMH declasse II non classique HLA-DM, qui favorise son remplacement par un peptide antigénique. L’action d’HLA-DM est alors inhibée par HLA-DO. Les molécules de CMH de classe II stabilisées par la présence d’un peptide sont alors transportées vers la surface de la cellule pour être présentées aux lymphocytes T CD4. [13;19;21-23]La dégradation des antigènes par ces deux voies aboutit à la présentation d’une grande diversité de peptides par les molécules de CMH de classe I ou de classe II aux lymphocytes T. Ces complexes CMH-peptide vont interagir avec les cellules T par l’intermédiaire du TCR et du co-récepteur (CD4 ou CD8) présents à leur surface. A la suite de la reconnaissance par le TCR, une réponse immunitaire sera initiée.
1.4. Le récepteur des cellules T
Le récepteur des cellules T (TCR) est une molécule hétérodimérique
exprimée à la surface des cellules T, qui est responsable de la reconnaissance du
complexe CMH-peptide présenté par les CPA. Deux types de TCR ont été identifiés.
Ils sont constitués par l’association d’une chaîne Œ avec une chaîne
f3,
ou d’unechaîne y avec une chaîne & Chaque cellule T n’exprime qu’un seul type de TCR:
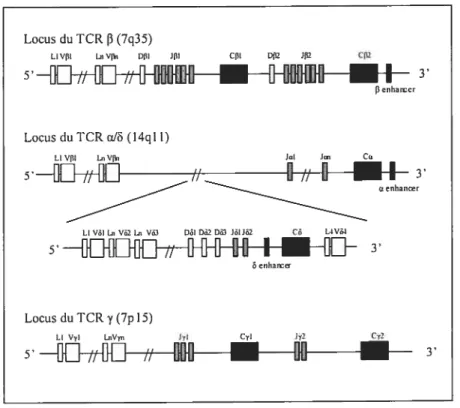
1.4.1. Organisation génétique du TCR
Les quatre familles de gènes codant pour les monomères du TCR (cx,
f3,
‘ et)
sont situées à trois loci différents (cx/ ,f3
et y) et ne s’expriment que dans lescellules T. Le locus a/3 est situé sur le chromosome 14 (14q11), le locus étant
inclus dans le locus cx. Les loci
f3
et y, quant à eux, se trouvent sur chacun des brasdu chromosome 7 (respectivement 7q35 et 7pl5).[24] Les chaînes polypeptidiques
composant le TCR comportent deux régions différentes une région variable
pouvant reconnaître des motifs peptidiques spécifiques et une région constante (C) ancrée dans la membrane de la cellule T. Chaque locus comporte donc différents éléments codant pour les différentes régions des monomères. La région variable est
codée par les éléments V (variable), D (diversité) et J (jonction) pour les chaînes
f3
ety, et par les éléments V et J dans le cas des chaînes cx et & Au cours de la maturation
des cellules T, il y a réarrangement des gènes et jonction des différents éléments pour former un TCR mature. t251 Le nombre de segments disponibles pour la recombinaison varie cependant en fonction de la chaîne considérée [26]. (Tableau I)
Nombre de segments Gène V D J C Chaîne e 51(112,) - 61(63) 1(1) Chaîne 3 (6) 3 (3) 4 (4) 1 (1) Chaîne f3 67 (/15) 2 (3) 14 (/6) 2 (4) Chaîne y 14 (22) - 5 (6) 2 (7,)
Tableau I famille des gènes du TCR chez les humains.
Nombre de segments de gènes ‘et d’aÏlèÏes,) répertoriés chez l’humain d’après les données venant du “International ImMunoGeneTics information system®” (http ://imgt. cines. fr), M-P Lefranc.
Chaque segment est flanqué par des séquences permettant la recombinaison: les RSS (Recognition Signal Sequence). [24;27j
G
1.4.2. Recombinaison ut3La recombinaison des gènes du 1CR a lieu lors de la maturation des
lymphocytes T dans le thymus (Figure 2). Le processus est similaire au
réarrangement des gènes des 1g dans les lymphocytes B et génère un vaste répertoire
de récepteurs reconnaissant undéterminant antigénique de manière spécifique.
——I{HHIHI — u KIWI
Jul J,,,
I H . h — .
Figure 2 Organisation des gènes dit TCR.
Les segments de gènes sont indiqués comme suit: L, région leader ; V,
variable ; D, diversité ; J, jonction ; C, constant
D’après Moss,P.A.. Rosenberg,W.M., et Bell,J.I., The hurnan T ceil receptor in health and disease. Anntt.Rev.ImmunoÏ 1992. 10: 7 1-96.
Le premier réarrangement a lieu au niveau du Iocus des gènes
f3
et provoquela fusion du segment DJ3j avec l’un des segments J531 ou du segment DJ32 avec un
segment ‘f32. On assiste ensuite à la recombinaison de la région Vf3 avec le DJf3 ainsi
Locus du TCRf3(7q35) LI VI Ln Vru D51 J51 -, •, n ) II U buwuju CI 052 J[12 C2 — n — I — I cnhaœer Locus du TCR aI(14q11) LI V51 Lu V6, LI V6I Ln V62 Lu V3 061 062 D63 261262 C6 L4U64 5,—3, 5cnhaœer C» — I -—. 3 u enhancer Locus du TCRy (7p 15)
LI Vyl Ln’ju Jyl
5
Cyl Jy2 Cy2 — An —
formé, pour donner un réarrangement VDIj3 mature. Il y a ensuite transcription du brin réarrangé à partir du promoteur et de la séquence leader situés en amont du segment Vf3. La jonction avec l’un des deux segments C13 a lieu par épissage de
l’ARN. Le réarrangement de la chaîne
f3
est fonctionnel si la traduction de cet ARNmature donne un produit VDI-Cf3 en phase. La recombinaison des gènes du locus a commence alors, et suit le même schéma. Cependant, puisque le locus c ne contient pas de segment D, le réarrangement consiste simplement à joindre un segment Va
avec un segment la. Lorsque cette fusion a lieu, le locus est alors délété, c ‘est
pourquoi la majorité des lymphocytes T expriment un récepteur af3.
Si la recombinaison des gènes sur le premier chromosome ne donne pas
naissance à un réarrangement fonctionnel, un second phénomène de recombinaison
peut alors avoir lieu sur le deuxième chromosome. Cependant, dès qu’un
réarrangement productif a eu lieu, le réarrangement des autres allèles est inhibé. Ce phénomène est appelé exclusion allélique. [24;271
La recombinaison des gènes du TCR implique la reconnaissance de séquences spécifiques, les RSS (Recombination Signal Sequence). Ces motifs sont
localisés en 3’ de tous les segments V, en 5’ de tous les segments I et flanquent
chaque segment D. Ils sont constitués de deux séquences hautement conservées un
heptamère et un nonarnère, séparées par une séquence “spacerT’ de 12 ou 23
nucléotides (correspondant à un ou detix tours de l’hélice d’ADN). La
recombinaison a lieu entre un RSS ayant un spacer de 12 nucléotides, et un autre
dont le spacer est de 23 nucléotides. La localisation des différents RSS permet d’éviter qu’il ne puisse s’effectuer une recombinaison entre deux segments de même famille. {25;28]
Les RSS sont reconnus par deux enzymes exprimées exclusivement dans les lymphocytes B et T immatures RAG (Recombination Activating Genes)- 1 et RAG
2. Cette reconnaissance provoque la liaison des séquences complémentaires et la
formation d’une boucle qui sera excisée ensuite par une endonucléase. Après ajout par la terminal deoxynucleotidyl transferase (TdT) d’un nombre aléatoire de
nucléotides au niveau des extrémités, les deux extrémités sont fusionnées sous l’action d’une ligase, complétant ainsi le phénomène de recombinaison. [28;29]
1.4.3. Polymorphisme
C’est pendant la recombinaison somatique des gènes du TCR qu’est générée la diversité du répertoire des cellules T. Le système immunitaire a donc évolué de manière à développer plusieurs mécanismes permettant d’augmenter cette diversité.
- Polymorphisme génétique: le premier mécanisme contribuant à
augmenter la diversité des TCR est constittié par la variété des segments V, D et J. (cf. tableau I)
- Diversité combinatoire: le phénomène de recombinaison permettant de synthétiser
les monomères Œ,
t3,
‘y et couplé au polymorphisme des différentes familles degènes augmente également de façon importante la diversité des TCR. En effet, au vu du nombre de segments participant à la recombinaison, il peut y avoir formation
d’environ 1000 chaînes
F
et de plus de 2500 chaînes u différentes. Le nombre dechaînes ‘y et différentes pouvant être formées est toutefois moins important. [25]
- Diversité des jonctions : ce mécanisme est le résultat de l’action des enzymes
prenant part à la recombinaison des gènes.
• Régions P : l’action de l’endonucléase qui clive la boucle formée lors de la recombinaison des régions V, D et J provoque la formation de courtes séquences paÏindromiques formant les régions P. [29]
• Régions N: ces régions sont dues à l’addition de nucléotides par une enzyme: la TdT. Celle-ci peut en effet ajouter de façon aléatoire entre 1 et 20 nucléotides au niveau des jonctions D-J et V-DJ. [27;29]
- Diversité du segment D : les deux segments Df3 sont formés par de
petites séquences de 9 à 16 nucléotides. [25] Il a été montré que ces segments D pouvaient être traduits dans les trois cadres de lecture avec une fréquence à
peu près équivalente. [27] Ce phénomène participe également à
l’augmentation de la diversité des TCR, bien que la petite taille de la séquence des régions D et sa faible complexité n’en limitent la contribution.
Ces différents mécanismes peuvent entraîner, en théorie, la formation d’environ i’ TCR o43 et participent ainsi à la création d’un répertoire très diversifié. Ce nombre théorique est cependant surestimé: le phénomène de sélection thymique diminuera en effet le nombre de TCR étant effectivement exportés en
périphérie. La population totale de cellules T a été estimée à 1012 cellules dans le
corps humain. Une étude a montré que la diversité des 1CR se rapprochait plus de
106 1CR différents. [30] Chaque cellule T ayant réarrangé un TCR uJ3 particulier
donnera ensuite naissance, si elle est activée, à une série de clones exprimant le même 1CR.
1.4.4. Structure du 1CR
Le complexe du TCR est une structure multimodulaire, comprenant le TCR et le CD3, présente à la surface des cellules T. Il est responsable de la reconnaissance d’un complexe CMH-peptide et de la transduction du signal qui en découle. [31]
Le 1CR a13 est un hétérodimère constitué de deux chaînes polypeptidiques trans-membranaires d’environ 45 kD, liées de façon covalente par un pont disulfure.
Chacune des chaînes u et
f3
comporte un court domaine cytoplasmique, une régiontrans-membranaire hydrophobe et deux domaines extracellulaires: un domaine constant (C) et un domaine variable (V). La structure de ces deux derniers domaines se rapproche de celle du domaine constant ou variable des 1g. [27] A l’intérieur du domaine variable, le polymorphisme est concentré dans trois régions : CDR (Complementary Determining Region)l, CDR2 et CDR3. [32] Alors que la
variabilité des domaines CDR1 et CDR2 est codée par les gènes V, celle de CDR3
apparaît lors du réarrangement V-J dans la chaîne a, ou V-DJ pour la chaîne
f3.
[33]Ce sont ces trois régions qui sont responsables de la spécificité de reconnaissance du complexe CMH-peptide (Figure 3).
Figure 3 Structure d’un TC’R a,B.
Le TCR est une glycoprotéine trans-membranaire constituée d’une chaîne c et d’une chaîne
J3,
liées de façon covalente par un pont disulfure. Chacune des chaînes a etf3
comporte tin court domaine cytoplasmique. une région trans membranaire hydrophobe et deux domaines extracellulaires : un domaine constant (C) et tin domaine variable (V). Les régions responsables de la spécificité de reconnaissance dci complexe CMH-peptide sont situées au mveau du domaine V des chaînes Œ etf3
et sont au nombre de trois: CDR1, CDR2 etCDR3.
Tiré de Garda, K.C., Degano, M.. Sianfield. R.. L.. Brunmark, A.. Jackson, M.R., Peterson, P.A., Teyton, L. et Wilson, 1. A. 1996. An Œf3 T Cdl Receptor Structure at 2.5
À
and Its Orientation in the TCR-MHC complex. Science 274:209-219.Cp
k -r T1Ç% * kVf3
Va
\
CDR3 CDR1 CDR1 CDR21.4.5. Interaction du 1CR avec le complexe CMH-peptide
La reconnaissance par le TCR d’un complexe CMH-peptide est un préalable au déclenchement d’une réponse immunitaire spécifique. Le peptide est présenté au TCR à l’intérieur de la niche peptidique d’une molécule de CMH. Celle-ci est délimitée sur les côtés par deux hélices u : les domaines u et u2 dans le cas du CMH de classe I, et les domaines u1 et f31 dans le cas du CMH de classe II. Les chaînes latérales des résidus acides-aminés du peptide sont dirigées soit vers le plancher de la niche, où ils s’insèrent dans des poches spécifiques, soit vers l’extérieur de la niche. Ils seront alors reconnus par le TCR. [34;35J.
Le complexe trimoléculaire formé par TCR, peptide et CMH a été cristallisé. Dans cette structure, le 1CR est positionné en diagonale au-dessus du CMH. Il interagit avec le complexe CMH-peptide par l’intermédiaire des domaines variables des chaînes u et
f3.
De façon plus précise. les régions CDR1 des chaînes u etf3
reconnaissent chacune une des extrémités du peptide (respectivement N-terminale et C-terminale) et les régions CDR2 interagissent avec le CMH au niveau des hélices u qui bordent la niche peptidique. Les régions CDR3, quant à elles, forment une poche pouvant reconnaître la plupart des résidus du peptide pointant hors de la niche(Figure 4). [36;37JM
TCR HA 1.7
peptide HA306318
Figure 44) Structure dit complexe TCR HA-HA]. 7/DR].
Le complexe trimoléculaire est formé par l’interaction d’un TCR avec un complexe CMH-peptide. Le TCR MAl .7 est présenté au-dessus de la molécule HLA-DRI contenant le peptide HA306318 dans sa niche peptidique.
B) Orientation du TCR lors de l’interaction avec le complexe CMH-peptide. Le TCR se positionne en diagonale au-dessus de la molécule de CMH présentant un peptide dans sa niche peptidique.
Tiré de Heimecke, J., Carfi, A. et Wiley, D.C.. 2000. Structure of a covalently stabilized complex of a human alphabeta T-cell receptor, influenza HA peptide and MMC class II molecule, HLA-DR1. EMBO J 19:5611-5624 (A) et de Klein, J. et Sato, A.. 2000. 1M Engl. JMed. 343. 702-709 (B)
Peptide Molécule HLA chaîne11 — duTCR chaîne [3 — duTCR Niche cOo-peptidique
1.5. Activation des cellules T
L’activation de la cellule T est initiée suite à la reconnaissance par le TCR d’un complexe CMH-peptide présenté à la surface d’tine CPA. C’est le complexe CD3 associé au TCR qui est responsable de la transduction du signal : la liaison des domaines V du TCR induit un changement de conformation des domaines C qui modifie l’interaction avec le CD3 associé au TCR. Il y aura alors transduction du signal à travers le complexe CD3, ce qui aboutira à l’activation de la cellule T. [37]
D’autres molécules participent également à l’activation de la cellule T: il s’agit des molécules d’adhésion (comme LFA-1 (Leucocyte function-associated Antigen-1)), des molécules de costimulation (tel que le CD28) et des co-récepteurs CD4 ou CD8. Toutes ces molécules sont retrouvées au niveau du point de contact entre le TCR et le complexe CMH-peptide et forment la synapse immunologique. Ces interactions moléculaires sont essentielles pour obtenir une activation optimale de la cellule T et, par conséquent, l’initiation et la régulation de la réponse immunitaire. [38;39]
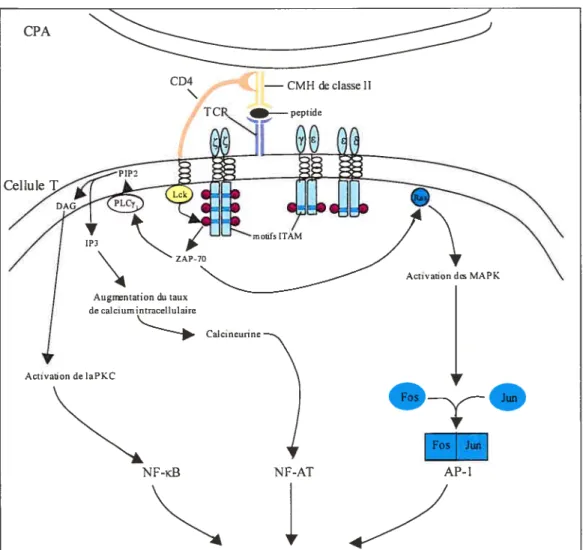
1.5.1. Signalisation via le complexe CD3
Le complexe CD3 est une structure hautement conservée constitué de trois chaînes invariantes : CD3-y, CD3- et CD3- (qui forment à elles trois un “sous complexe”) et un homodimère de deux chaînes CD3-Ç liées par un pont disulfure au niveau de leur domaine trans-membranaire. [40-42] Les chaînes du complexe CD3 interagissent de façon non covalente avec les chaînes Œ ou 3 du TCR au niveau
trans-membranaire par l’intermédiaire d’un résidu aspartate chargé négativement. La signalisation a lieu au niveau cytoplasmique et implique des protéines tyrosine kinases (PTK) (Figure 5). Les chaînes du complexe CD3 n’ont pas d’activité
enzymatique intrinsèque, mai s possèdent des motifs consensus par l’intermédiaire desquels elles peuvent interagir avec les PTK. Ces motifs ITAM (Immunoréceptor Tyrosine-based A ctivation M otif) s ont e xprimés e n u n e xemplaire sur J e d omaine
cytoplasmique des chaînes CD3-y, -, et -, et en trois exemplaires sur le domaine
cytoplasmique des chaînes . Le motif consensus est composé de deux copies de la
séquence tyrosine-XX-leucine ou isoleucine [Yxx(L/T)], séparées par 6 à 8 acides aminés. [41;42] Suite à l’engagement du TCR, ces motifs sont phosphorylés par des protéines kinases de la famille src : p56’< (associée au co-récepteur CD4 ou CD8) et
59fn [43] Les ITAMs phosphorylés recrutent alors une autre protéine tyrosine
kinase: ZAP-70, qui possède un domaine SH2 (Src Homology 2). Une fois active, ZAP-70 phosphoryle à son tour d’autres substrats tels que LAT (Linker for
Activation of T cells)[44] et SLP-76 (SH2 domains containing Leukocyte
Protein)[45], et transmet ainsi le signal en menant à l’activation de la phospholipase
C ‘yl (PLCyI) et de p21ras une petite protéine G. [46;47j
La PLC’yl activée hydrolyse le phosphoinositol-4,5-bisphosphate (PIP2) pour
générer de l’inosotol-1,4,5-triphosphate (1P3) et du diacylglycerol (DAG). Ces deux
molécules induisent respectivement une augmentation du taux de calcium
intracellulaire et l’activation de la protéine kinase C (PKC). L’élévation du calcium
cellulaire provoque l’activation de la calcineurine qui active à son tour NF-AT
(Nuclear Factor of Activated T cell) en le déphosphorylant. La PKC, elle, phosphoryle différents substrats et induit l’activation de N}icB cNuclear Factor KB).
La p21ras quant à elle, initie le déclenchement de la cascade de phosphorylation des MAP kinases (mitogen-activated protein kinase) qui aboutit à la dimérisation des membres de la famille Fos et Jun pour former le facteur de
transcription APi. [46-48]
Ces trois facteurs de transcription (NF-AT, NFKB et APi) induisent
l’expression de différents gènes responsables de la différenciation et la prolifération
Figure 5 Cascade de signalisation menant à Ï ‘activation de la cellule T suite à 1 ‘engagement du TCR par un complexe CMH-peptide.
D’après Leo, A. et Schraven, B. Adapters in lymphocyte signalling. Curr. Opin. Immunol. 13 : 307-3 16. [49) CPA CD4 - CMHdeclassell -peptide Cellule Activationde MAPK Augnntation du taux de calciurnintracellulaire Calcinetinne Activation de IaPKC NF-KB NF-AT os AP-1
1.5.2. Différenciation et prolifération des cellules T
L’activation de cellules T naïves (i.e. n’ayant jamais été stimulées par leur ligand) mène à leur division et leur différenciation en deux sous-populations. On retrouve d’une part les cellules effectrices (cellules T auxiliaires dans le cas des lymphocytes T CD4, et CTL dans celui des lymphocytes T CD8j, qui prolifèrent alors par expansion clonale et sécrètent les cytokines nécessaires à leurs fonctions. La seconde sous-population est représentée par les cellules mémoires. Ces cellules sont les artisans de la mémoire immunologique qui fera qu’une deuxième rencontre avec le même antigène donnera naissance à une réponse plus rapide et plus efficace.[50;51] On distingue deux types de cellules mémoires d’une part les cellules “centrales mémoires” situées préférentiellement au niveau des organes lymphoïdes secondaires, et d’autre part les cellules “effecteurs mémoires”, qui seraient responsables de la surveillance des tissus périphériques.[52-55]
Les méthodes d’étude de la prolifération cellulaire sont nombreuses. Celles ci incluent l’emploi de colorants tel qtie le carboxyfluorescein diacetate succinimidyl ester (CFSE), qui permet d’apprécier le nombre de divisions effectuées par une cellule. Couplée à l’utilisation de tétramères de molécules de CMH, cette technique permet d’identifier des populations spécifiques ayant proliféré en réponse à un antigène donné. Il est également possible d’utiliser les propriétés de la cytométrie en flux pour phénotyper des populations cellulaires à l’aide de marqueurs extra cellulaires et caractériser leur fonction en utilisant des marqueurs intracellulaires pci-mettant de mesurer les différentes cytokines qu’elles produisent.
1.6. La réponse des cellules T CD4
La réponse des cellules T peut être divisée en trois étapes expansion, contraction et mémoire. La première étape est initiée dans les tissus lymphoïdes suite à la rencontre d’un antigène et d’une cellule T et mène, dans la plupart des cas, à l’élimination de ce pathogène dans les jours qui suivent. Elle est suivie d’une phase de contraction lors de laquelle la grande majorité (plus de 90 %) des cellules effectrices générées lors de la phase d’expansion vont mourir. Les cellules ayant survécu participent alors à la formation d’un pool de cellules dites mémoires qui seront maintenues au cours du temps.
1.6.1. Expansion
Après stimulation antigénique, les cellules T CD4 naïves vont proliférer et se différencier en cellules effectrices. Activation, prolifération et différenciation sont cependant des évènements distincts qui n’ont pas nécessairement lieu simultanément.
1.6.1.1 Activation
Pour être activées et entrer en cycle cellulaire, les cellules CD4 naïves nécessitent un minimum de 6 heures de stimulation antigénique par une CPA. En l’absence des molécules de costimulation appropriées, la durée de stimulation nécessaire à l’activation des cellules augmente sensiblement (plus de 20 heures). [56] L’importance de la costimulation diminue cependant si la dose d’antigène est augmentée. [56]
Les cellules CD4 effectrices, quant à elles, sont activées après seulement une heure d’exposition à l’antigène et leur activation n’est que peu affectée par l’absence
de molécules costimulatrices. Cependant, une stimulation antigénique prolongée au delà de 12 heures provoque leur mort. [56]
1 .6.1 .2 Prolifération
La prolifération cellulaire n’intervient pas directement après l’activation. Il existe en effet un intervalle de 24 heures avant que l’on ne puisse observer la première division cellulaire. Durant cette période, on assiste à une augmentation de la taille des cellules et plusieurs gènes nécessaires à la réplication de l’ADN ou en relation avec la préparation de la prolifération sont exprimés. [50;57] Après ce laps de temps, les cellules entrent en division avec un temps de génération d’environ 10 heures. [50;58] L’ampleur de la prolifération cellulaire varie en fonction de la dose d’antigène initialement présente. On estime que lors d’une infection virale par LCMV (lyrnphocytic choriorneningitis virus), les cellules T CD4 effectueront environ 9 divisions. [58]
C
1.6.1.3 Différenciation
Si la première division peut être observée au bout de 24 heures d’activation, la différenciation des cellules T CD4 naïves en Thi ou Th2 ne débutera pas avant 48 heures de stimulation antigénique continue. La différenciation est un phénomène impliquant différents signaux tels que le type de cytokines présentes dans le milieu, la durée de l’exposition à l’antigène, la force du signal transmise par l’intermédiaire du TCR ou encore la dose d’antigène présent dans le milieu.[50;59;60]
- Cytokines
L’environnement en cytokines joue un rôle important dans la différenciation des lymphocytes T CD4. Il existe en effet des cytokines favorisant une réponse de type Thi, alors que d’autres induiront préférentiellement une réponse Th2.
Q
sont principalement l’IFN-y et 1’IL-12. L’IFN-y agit essentiellement comme unLes cytokines provoquant la différenciation des lymphocytes en cellules Thi inhibiteur de la différenciation en Th2. L’IL-12 à i’opposé, entraîne la différenciation des lymphocytes T CD4 en cellules Thi mais n’a aucun effet sur le développement des cellules Th2.La différenciation des lymphocytes T CD4 en cellules Th2, quant à elle, est dépendante de la présence d’IL-4 et d’IL-10. Si 1’IL-lO joue un rôle plutôt secondaire dans la différenciation des Th2, elle permet surtout d’inhiber l’action des macrophages et des cellules Thi. L’IL-4, au contraire, est absolument indispensable au développement des cellules Th2. [61]
- Durée de l’exposition à l’antigène
A l’issue de 48 heures de stimulation, 1 a majorité des cellules restent non polarisées si ces cellules possèdent la capacité de produire de l’IL-2, elles ne sécrètent, par contre, que peu o u pas d ‘IfN-y ou d ‘IL-4. [59;62] Il a e n e ffet é té prouvé que, en l’absence de cytokines exogènes, la polarisation des cellules n’était obtenue qu’après plusieurs cycles de division. [59;63] Certaines études ont ainsi montré que, pour générer des cellules effectrices activées de manière optimale, les lymphocytes T CD4 nécessitaient une activation peptidique d’une durée minimale de 2 jours. Cette première phase est ensuite suivie d’une phase d’expansion, également de 2 jours, dépendante de la présence d’IL-2. [64]
Des disparités ont en outre été mises en évidence dans la vitesse de différenciation des cellules Thi et Th2. L’expression des fonctions effectrices des cellules différenciées est en effet acquise en fonction du nombre de divisions effectuées t la production d’IFN-y est ainsi retrouvée dans des cellules ayant effectué
au moins un cycle, alors que celle d’IL-4 n’apparaît qu’au bout de quatre cycles de division. [59;65;66] La différenciation en Th2 nécessite donc une stimulation antigénique plus longue.
Enfin, il a également été démontré que le potentiel de différenciation des cellules diminuait avec le nombre de divisions t les cellules ayant effectué moins de
d’un phénotype Thi à un phénotype Th2, à l’inverse des cellules ayant accompli
plus de quatre divisions. [63;65-67]
Il a cependant été rapporté que, dans le cas des Thi, prolifération et
différenciation étaient deux phénomènes pouvant être séparés. En effet, il a été
montré qu’une exposition transitoire à un antigène pouvait à elle seule induire la
prolifération des cellules (jusqu’à 6 ou 7 cycles de division en absence de contact répété). Néanmoins, la différenciation en Thi nécessitera un réengagement du TCR en présence de cytokines polarisantes (IFN-’y et IL-12), tandis qu’il est possible d’obtenir des cellules Th2 différenciées sans que le contact avec l’antigène ne soit maintenu (si toutefois il y a présence d’IL-4 dans le milieu). [64;68]
- force du signal
il a été montré que la force du signal propagé par l’intermédiaire du 1CR
pouvait favoriser une voie de différenciation plutôt que l’autre. Une stimulation des cellules exercée par un ligand de faible affinité pour le TCR ou en présence des
molécules de costimulation appropriées induira par exemple de manière
préférentielle une réponse Th2. A l’opposé, des cellules stimulées par des ligands de haute affinité ou en l’absence de molécules de costimulation se différencieront plutôt
en cellules Thi. [59;69;70]
- Dose et nature de l’antigène
L’effet de la dose d’antigène sur le contrôle de la différenciation des cellules
T CD4 n’est pas clairement défini. La littérature traitant de ce sujet, si elle est
abondante, est aussi fortement contradictoire. Plusieurs études suggèrent en effet que l’activation des cellules par de fortes doses d’antigène induirait préférentiellement une réponse de type Th2, alors que l’utilisation de doses plus faibles favoriserait le développement d’une réponse Thi. [7 1-73] Cependant, d’autres études ont démontré que la situation inverse pouvait également se produire. [74-77] Il semblerait dès lors que ce ne soit pas tant la dose d’antigène, mais plutôt la nature même de l’antigène
utilisé qui détermine le type de réponse induite. En effet, dans la plupart des études
l’antigène utilisé était de nature parasitaire tandis que les études obtenant les résultats inverses utilisaient plutôt des antigènes de nature protéique. La réponse induite par un parasite implique de nombreux acteurs (NK, cellules T CD8...) et pourrait de ce fait influer sur la différenciation des cellules T CD4 en induisant un environnement particulier, qui ne serait pas observé lors de l’utilisation de faibles doses d e p rotéines. Les différences o bservées p ourraient également être dues à 1 a
présentation d e 1 ‘antigène. E n e ffet, I e ni ode d e d égradation et d ‘apprêtement d es
antigènes varie en fonction de la nature initiale de l’antigène [78] et de son niveau de glycosylation. [79;80] De ce fait, il est difficile de prédire la corrélation existant entre la dose d’antigène utilisée et sa densité finale à la surface des CPA sous forme
de complexes CMH II- peptides.
C’est l’intégration de ces différents signaux par le lymphocyte T CD4 qui permet de définir le type de différenciation qu’il subira.
1.6.2. Contraction
Dans la plupart des infections, le pathogène est éliminé à l’issue de la phase effectrice. Il devient alors nécessaire de limiter l’expansion massive des cellules T. Celle-ci risquerait en effet, si elle n’était pas contrôlée, de créer un déséquilibre au niveau du système immunitaire. Les lymphocytes T activés ayant proliféré subissent donc une phase de contraction à l’issue de laquelle la majorité d’entre eux sera éliminée. Cette phase de contraction intervient de façon plus précoce dans le cas des lymphocytes CD8 que dans le cas des lymphocytes CD4. Les mécanismes responsables de l’élimination des cellules effectrices ne sont pas encore totalement définis, niais il semble qu’ils impliquent plusieurs facteurs et soient différents pour les cellules CD4 et CD8.
Dans le cas des c ellules CD8, on observe une phase de contraction assez rapide (1 à 2 semaines) qui est indépendante de la magnitude de la phase d’expansion, de la dose d’antigène et de la durée de l’infection. [81] Il semble en effet, qu’à elle seule, l’influence inhibitrice de certaines cytokines telles que l’LFN-y soit suffisante pour induire l’élimination des cellules effectrices. La perforine jouerait également un rôle secondaire en agissant comme un régulateur négatif de la
phase d’expansion, en permettant probablement l’élimination des CPAs. [$2-24]
Dans 1 e c as des c ellules T C D4, I a c ontraction e st p lus Î ente (environ 5 0 jours) et subit une régulation plus importante impliquant la collaboration de plusieurs mécanismes. [58;85] La séquence des évènements menant à l’élimination des cellules effectrices est encore mal connue, mais semble mettre en jeu différents mécanismes tels qu’une signalisation négative par CTLA-4 [$6], l’activation de la voie Fas [87-89], ainsi que l’influence de différentes cytokines (dont IL-2, IFN-y, et
TNF). [90-92] Il existerait également un mécanisme de nature plus passive qui provoquerait la mort des cellules effectrices à la suite de la perte de contact entre le CD4OL sur les cellules T et le CD4O présent sur les CPAs. [93] Enfin, il semblerait que la régulation de la contraction des cellules T CD4 implique aussi les molécules de la famille bd, telles que Bd-2 et Bcl-x[, qui pennettraient d’augmenter la survie des cellules. [87;94]
A l’issue de la phase de contraction, la faible proportion de lymphocytes ayant survécu forme une population de cellules possédant une longue durée de vie qui va participer à la formation d’un pool de cellules dites mémoires. Ces cellules répondront plus rapidement et plus efficacement lors de toute nouvelle rencontre avec le pathogène ayant induit leur activation.
1.6.3. Mémoire
Il existe plusieurs modèles de différenciation des cellules mémoires et la
littérature traitant de ce sujet est donc assez contradictoire. On retrouve
principalement (Figure 6):
- un modèle dichotomique”, dans lequel les cellules naïves
donneraient naissance sinutttanément aux cellules effectrices et aux celLules
mémoires. Dans ce cas de figure, ces dernières n’acquerraient jamais de fonction
effectrices.
- un modèle “linéaire” pour lequel les cellules mémoires
seraient les descendantes directes des cellules effectrices. Les cellules naïves se différencieraient alors tout d’abord en cellules effectrices dans une première étape, elles-mêmes se différenciant par la suite en cellules mémoires dans une deuxième étape. [50;95]
Modêle dichotomique lêIe linéaire
ree
Cellule narve Cellule narve Cellule effec,rrce
Cellule nmnmrre
Cellule mémoire
Figure 6 Modè les de différenciation des cellules mémoires.
Tiré de Kaech S.M., Wherry E.J. et Ahmed R. 2002. Effector and memory T cell differentiation: implications for vaccine development. Nat Rev Imnninol. 2
Dans tous les cas de figures, on obtient à l’issue de la réponse primaire un pool de cellules spécifiques d’un antigène donné, dont la fréquence est supérieure à celle que l’on pouvait observer chez les cellules naïves [51;96-98], et qui présentent des caractéristiques fonctionnelles et phénotypiques particulières. La différenciation en cellule mémoire implique en effet un remaniement du profil d’expression de certains gènes (tels que le granzyrne B ou la perforine, par exemple, dans le cas des cellules T CD$). Ceux-ci seront alors exprimés de manière constitutive au niveau des cellules mémoires alors qu’ils ne l’étaient pas dans les cellules naïves. [99;l00] Les cellules mémoires expriment en outre un ensemble de molécules de surface qui
permettent de les classer en deux types d’une part les cellules “centrales mémoires”
(TCM) situées préférentiellement au niveau des organes lymphoïdes secondaires, et
d’autre part les cellules “effecteurs mémoires” (TEM), qui seraient responsables de la
surveillance des tissus périphériques. Ces deux types de cellules jouent un rôle
complémentaire t dans le cas d’une nouvelle infection, les TCM permettraient ainsi la
génération rapide de nouvelles cellules effectrices spécifiques au niveau des
ç
ganglions, tandis que les TEM pouffaient proliférer rapidement en périphérie, limitantde cette façon la dissémination du pathogène.[52-55]
Ces cellules mémoires sont maintenues au cours du temps et subissent un renouvellement lent mais constant grâce à l’influence de différents signaux. [101] Ceux-ci comprennent notamment une réexposition périodique à l’antigène par les CPAs présentes (en particulier les cellules dendritiques) [102], et d’autre part la présence certaines cytokines (principalement IL-7 et IL-15). [103] Il semblerait toutefois que, dans le cas des lymphocytes T CD4, le nombre de cellules mémoires présentes dans l’organisme ait tendance à diminuer avec le temps (contrairement aux lymphocytes T CD8 mémoires dont la population conserve l’importance qu’elle avait à l’issue de la phase de contraction). [5$;85]
Le nombre plus important de cellules spécifiques obtenues à l’issue de la réponse p rimaire, 1 eurs 1 ocalisations, 1 eurs fonctions mobilisables p lus r apidement ainsi que l’augmentation de leur durée de vie rendent les cellules mémoires aptes à
assurer une protection efficace de l’organisme dans le cas d’une nouvelle infection par le même pathogène.
1.7. Importance de la réponse CU4 lors d’une infection vïrale
Un certain nombre de virus, tels CMV (cytornegalovirus) ou EBV (Epstein Barr virus), ne sont pas totalement éliminés de l’organisme, mais persistent sous la forme d’une infection chronique contrôlée par une réponse immunitaire efficace. Ce contrôle est g énéralement le résultat d’une c ollaboration étroite entre les réponses des lymphocytes T CD4, des lymphocytes T CD8, ainsi que de e ontacts directs entre cellules. Les lymphocytes T CD4 sont donc indispensables au développement d’une réponse antivirale efficace [104].
Pour étudier les relations entre ces différents acteurs, de nombreuses études
ont utilisé le modèle murin d’infection par le LCMV. Ce virus provoque une
infection ayant peu d’effets cytopathiques et pouvant être résolue rapidement par une réponse des cellules CD8. [105] La réponse CD4 est cependant nécessaire au contrôle de la réplication virale lors de la phase chronique de l’infection. II a en effet été montré qu’en l’absence de cellules T CD4+, la réponse CD8 avait tendance à s’affaiblir avec le temps et que les fonctions effectrices des cellules T CD8+ subsistant étaient altérées. [106-108] De plus, une déplétion transitoire en cellules T CD4+ au moment de l’infection, conduira au développement d’une infection chronique plutôt qu’à la résolution de l’infection [106;109j. Enfin, des études vaccinales ont démontré qu’une vaccination peptidique était nettement moins efficace pour provoquer une réponse CD8 chez des souris ne possédant pas de cellules CD4. [110;lll]
Plusieurs études utilisant un modèle murin ont permis de démontrer que les cellules T CD4+ pouvaient aider au développement des réponses CD8 par



![Figure 44) Structure dit complexe TCR HA-HA]. 7/DR].](https://thumb-eu.123doks.com/thumbv2/123doknet/2057474.5831/37.918.172.782.125.576/figure-structure-dit-complexe-tcr-ha-ha-dr.webp)