T
T
H
H
È
È
S
S
E
E
En vue de l'obtention du
D
D
O
O
C
C
T
T
O
O
R
R
A
A
T
T
D
D
E
E
L
L
’
’
U
U
N
N
I
I
V
V
E
E
R
R
S
S
I
I
T
T
É
É
D
D
E
E
T
T
O
O
U
U
L
L
O
O
U
U
S
S
E
E
Délivré par l'Université Toulouse III - Paul Sabatier Discipline ou spécialité : Biologie Moléculaire
JURY
Dr Catherine Tomasetto, IGBMC, Illkirch, rapporteur Dr François Dautry, Institut André Lwoff, Villejuif, rapporteur Dr Florence Cabon, Institut André Lwoff, Villejuif, examinateur
Pr Gilles Favre, INSERM U563, Toulouse, président du jury Dr Stéphan Vagner, INSERM U563, Toulouse, directeur de thèse
Ecole doctorale : Biologie-Santé-Biotechnologies de Toulouse
Unité de recherche : INSERM U563, Centre de Physiopathologie de Toulouse-Purpan, Institut Claudius Regaud
Directeur de Thèse : Dr Stéphan Vagner
Présentée et soutenue par Gérard MAILLOT Le 23 octobre 2009
Titre :
Contribution à l’étude de l’expression et des fonctions
des microRNAs dans les cancers du sein.
Les personnes qui m’ont côtoyé me diront « toi Gérard tu écris des remerciements, toi avec ton caractère, pppffffff », mais du fond du cœur ces mots me viennent confus pour s’immortaliser sur ces pages.
Je remercie les professeurs Georges Delsol et Gilles Favre pour m’avoir accueilli au sein du centre de Physiopathologie de Toulouse-Purpan dans l’unité INSERM U563 à l’Institut claudius Regaud. J’ai bénéficié d’un environnement agréable et propice à la science. Merci.
Je tiens à remercier les membres du jury et notamment le Dr. François Dautry, le Dr. Catherine Tomasetto et le Dr. Florence Cabon qui m’ont fait l’honneur de lire et d’évaluer mon manuscrit et de participer à mon jury de thèse. Merci.
Stéphan, ah Stéphan. Que suis-je censé dire ? Mon encadrement n’a pas dû être une simple partie de plaisir. Mon impatience à tout, à une recherche rapide, dynamique et des fois « une recherche à ma façon » a dû être canalisée pour aboutir. Tu t’y es employé, en posant des garde-fous, en me faisant bénéficier de ton expérience. Tu as su me faire participer à la vie d’un laboratoire en me proposant d’encadrer Jacobo, Valérie, Nathalie et Margaux, signe je pense d’une confiance en moi. Mon exigence à avoir accès à des outils de pointe a été abreuvée par ton dévouement à faire vivre l’équipe. Ton pointilleux allait même à nous former à la bonne gestion du matériel et consommables. J’ai senti des fois où nous pouvions échanger d’égal à égal, la frontière de l’encadrement s’effritant peu à peu. Signe d’une perspicacité et d’un sens de l’observation digne d’un bon chercheur, tu as été le seul a remarqué sur une vidéo personnelle l’envol fortuit d’un groupe d’oiseaux pendant une scène chère à mes proches et à moi-même. Tu fais partie des personnes qui ont su me faire progresser, m’apporter énormément. Je crois te rendre hommage en te faisant part de mes phrases favorites si souvent répétées à la paillasse : « arrêtons d’e…… les mouches car la science dans tout ça », « cool calme zen, lexomil® ». Merci de tout cœur.
Stefania, tu as su me donner du courage quand j’en ai eu besoin « ma que ». Professionnellement mais aussi dans ma vie privée, ton soutien a été et je pense est indéfectible « mais qu’est ce que tu souhaites faire, tu veux que je réponde à ta place, non, donc… ». Les discussions scientifiques s’imprègnent d’une simplicité au détour de ta voix. Tu as su me guider dans mon apprentissage à la présentation orale, à la maîtrise d’un sujet
scientifique complexe. Tu m’as apporté énormément, je t’en suis reconnaissant. Mon seul regret est de ne pas t’avoir entendu chanter en italien au labo. Merci de tout cœur.
Un grand merci à Piou, Zad, Sandra (P et BF), Sophie, Loubna, Fred pour avoir été mes acolytes de bureau ou de paillasse. Nous nous sommes connus en tant que collègues et nous nous quittons en tant qu’amis, que dire de plus…ah oui :
Piou, nous avons fait et défait la science autour d’innombrables tasses de café, génial.
Zad, le plus grand mérite en ta faveur c’est d’avoir fait croire à Stefania et à Stéphan que tu es un étudiant de thèse. Chapeau, et merci pour toutes ces discussions scientifiques parfois houleuses mais constructives.
Sandra P, ton implication dans mes projets, ton envie de réaliser de façon très pointilleuse des expériences même les plus banales t’honore. Que de bons moments.
Sophie, je n’ai jamais entendu cela « poulait ». Tu as su te montrer d’une aide précieuse à l’avancée de nos projets. Pour une fois j’ai trouvé quelqu’un qui souhaite organiser et ranger plus que moi, c’est impressionnant.
Loubna, ou plutôt Loubni, tes chansons raisonneront longtemps dans ma tête. Sandra BF, ton sens commun et ta gentillesse font de toi quelqu’un d’inoubliable.
Fred, que de bons moments avec un aussi fin connaisseur des saveurs des îles, « un ‘ti punch ».
Magali ou « l’âne à pattes », « j’ai pas le temps là parce que je dois sauver des vies ». D’un professionnalisme, d’une gaieté de cœur, tu es généreuse. Nous avons mené à bâton rompu nos projets. Ton implication m’a été bénéfique. Merci.
Valérie, « ah non j’ai pas le temps j’suis pressée, après peut être ».Tu as un volume de travail impressionnant. Ta progression est à ton honneur et j’en suis content. Merci.
Anne C, « mon train est à 18h52, j’arrive à 19h12 et prends la voiture à 19h14 », ton sens de la précision et ta gestion du temps sont incomparables. On se verra peut être sur le morceau de caillou que nous partageons dans nos racines.
Anne C bis, nous n’avons pas encore eu le temps de bien discuter réellement mais ta présence a été agréable.
La place me manque, je ne peux donc qu’écourter mes remerciements pour l’ensemble des membres de l’U563 actuels ou qui sont vers d’autres horizons maintenant. Un grand merci à vous qui m’ont permis de travailler dans un cadre agréable (secteur culture Caro, Anne,
Florence, Lourdès, Christiane ; secteur laverie Marie-Ange, Adeline, Isa ; le secrétariat toujours au top : Jeannine ; la radioc : Anne, Jean-Charles, Isa). Sans vous, mon travail n’aurait pas été possible. Merci.
Un merci à Nico, Marine, Lolo, Guigui, Flo, Emilie (les deux), Leïla, Cyril ….. qui m’ont fait apprécier la vie de thésard.
Je remercie mes amis, Bruno (les deux), Ludo, Ti Bil. Dès le début, vous avez cru en moi et vous m’avez encouragé à me lancer dans de grandes études. Merci les gars.
Et c’est ici que mes remerciements sont autant de coup de poignard envers vous à qui je dois tout. En effet, mère et père, les quelques mots couchés ici sont bien insuffisants et ne seront d’ailleurs qu’un pâle reflet de toute la gratitude que je vous exprime. Vous m’avez donné envie de décrocher la lune, vous avez juste eu l’audace de croire en moi. A mes sœurs, à mon frère qui sont de solides piliers sur lesquels il fait bon s’appuyer. Vous, membres de ma famille, votre présence est réconfortante et m’est indispensable. Merci.
A la génération qui vient et qui souhaite prendre tonton Gé comme exemple car il a quitté sa campagne réunionnaise pour la grande aventure métropolitaine, courage les enfants.
A mes grands-parents qui je pense aurait été fiers de me voir soutenir un des diplômes les plus prestigieux de France.
A ma belle famille dont le sacrifice aura été grand. Vous m’avez permis de connaître votre fille, la plus belle aventure de toute ma vie. Votre soutien est sans faille.
Enfin, je tiens à dédier cette thèse à Aurélie pour avoir su me guider, être ma muse tout au long de mes études. Merci de m’avoir donné ce qu’un père peut rêver de mieux, un joli petit enfant qui me comble de bonheur. Du plus profond de mon cœur, merci.
ABREVIATIONS
ABL1 c-abl oncogene 1, receptor tyrosine kinase ADN acide désoxyribonucléique
Af4 (AFF1) AF4/FMR2 family, member 1
Ago argonaute
AIB1 amplified in breast cancer-1
All1 (MLL) myeloid/lymphoid or mixed-lineage leukemia (trithorax homolog,
Drosophila)
ANXA1 annexin A1
APAF-1 apoptotic peptidase activating factor 1 API5 apoptosis inhibitor 5
ARN acide ribonucléique
ARNm acide ribonucléique messager BAK1 BCL2-antagonist/killer 1
BCL2 B-cell CLL/lymphoma 2
BCL-W (BCL2L2) BCL2-like 2
BMPR2 bone morphogenetic protein receptor, type II (serine/threonine kinase)
CCND1 cyclin D1
CCNE1 cyclin E1
CD71 (TFRC) transferrin receptor (p90, CD71) CDC34 cell division cycle 34
CDK6 cyclin-dependent kinase 6
CLOCK circadian locomoter output cycles kaput protein CRK v-crk sarcoma virus CT10 oncogene homolog (avian)
CTSD cathepsin D
DAXX death-domain associated protein
DCL1 Dicer-Like 1
DCP1 decapping protein 1 DCP2 decapping protein 2
DGCR8 DiGeorge syndrome critical region gene 8 dsRBD double stranded RNA Binding domain
E2 17-β-œstradiol
E2F3 E2F transcription factor 3 EFNA3 ephrin-A3
EGFR epidermal growth factor receptor eIF4E eukaryotic initiation factor 4E
EMT epithelial mesenchymal transition
ERBB2 (HER2) v-erb-b2 erythroblastic leukemia viral oncogene homolog 2, neuro/glioblastoma derived oncogene homolog (avian) ERK extracellular-regulated kinase
ESR1 estrogen receptor 1
EWS ewing sarcoma
exp5 exportine-5
FOXO3 forkhead box O3
FZD3 frizzled homolog 3 (Drosophila) GAX growth arrest-specific homeo box GLS glutaminase
GW182 (TNRC6A) glycine-tryptophan protein of 182 kDa; trinucleotide repeat-containing gene 6A
HBV Hepatitis B Virus
HCV Hepatitis C Vivus
HES1 hairy and enhancer of split 1, (Drosophila)
HER3 (ERBB3) v-erb-b2 erythroblastic leukemia viral oncogene homolog 3 HIV-1 Human immunodeficiency virus type 1
HMGA2 high mobility group AT-hook 2 hnRNP A1 heterogeneous ribonucleoprotein A1 hnRNP K heterogeneous ribonucleoprotein K
HOXA5 homeobox A5
HOXD10 homeobox D10
HSPB1 heat shock 27kDa protein 1 HYL1 Hyponastic leaves 1 ITGA5 integrin, alpha 5 ITGB3 integrin beta 3
IRS1 insulin receptor substrate 1
JMY junction mediating and regulatory protein, p53 cofactor
KRT19 keratin 19
KSRP KH-type splicing regulatory protein LATS2 large tumor suppressor, homolog 2 lin-4 cell lineage-4 (C. elegans)
lin-14 cell lineage-14 (C. elegans)
lin-28 cell lineage-28 homolog (C. elegans)
LNA locked nucleic acid
Loqs loquacious
Lsm1-7 LSM1 homolog, U6 small nuclear RNA associated (S. cerevisiae) 1 à 7 Mcl1 myeloid cell leukemia sequence 1 (BCL2-related)
MERTK c-mer proto-oncogene tyrosine kinase
miRNA microRNA
miRISC miRNA-RNA-Induced Silencing Complex miRNP miRNA-ribonucleoproteins
MITF microphthalmia-associated transcription factor MMP16 matrix metallopeptidase 16
MOV10 Moloney leukemia virus 10, homolog (mouse)
MYB v-myb myeloblastosis viral oncogene homolog (avian)
MYCN v-myc myelocytomatosis viral related oncogene, neuroblastoma derived (avian)
NF45 (ILF2) interleukin enhancer binding factor 2, 45kDa NF90 (ILF3) interleukin enhancer binding factor 3, 90kDa
NTR non traduit (région 5’ ou 3’ non traduite d’un ARNm)
ORF Open Reading Frame
P-bodies Processing bodies
P27kip1 (CDKN1B) cyclin-dependent kinase inhibitor 1B
p57kip2 (CDKN1C) cyclin-dependent kinase inhibitor 1C (p57, Kip2) p68 (DDX5) DEAD (Asp-Glu-Ala-Asp) box polypeptide 5 p72 (DDX17) DEAD (Asp-Glu-Ala-Asp) box polypeptide 17
PACT (PRKRA) protein kinase, interferon-inducible double stranded RNA dependent activator
PDCD4 Programmed cell death protein 4
PEI polyethylenimine
PGR progesterone receptor
PHB prohibitin
PIK3R2 phosphoinositide-3-kinase, regulatory subunit 2 (beta) PPIF peptidylprolyl isomerase F
pré-miRNA précurseur de miRNA
pri-miRNA primary transcript of miRNA PTEN phosphatase and tensin homolog
PTMA prothymosin, alpha
PTPRN2 protein tyrosine phosphatase, receptor type, N polypeptide 2 R2D2 dsRNA-binding domains (R2) and is associated with DCR-2 (D2) RARA retinoic acid receptor, alpha
RBP RNA Binding protein
RCK/p54 (DDX6) DEAD box-6; DEAD/H (Asp-Glu-Ala-Asp/His) box polypeptide 6 (RNA helicase, 54kD)
RdRp RNA-dependent RNA polymerase
RDX radixin
RE récepteur des œstrogènes
RECK reversion-inducing-cysteine-rich protein with kazal motifs RHA ATP-dependent RNA helicase A
RISC RNA-induced silencing complex
RNAse ribonucléase
RNAi RNA interference
RSV virus respiratoire syncytial
RTKN rhotekin
shRNA short interfering RNA
SILAC stable-isotope labeling by amino acids in cultured cells siRNAs small interfering RNAs
SIRT1 sirtuin 1
SNALP stable nucleic acid lipid particles SNIP1 Smad nuclear interacting protein 1 SOX4 SRY (sex determining region Y)-box 4 SUFU suppressor of fused homolog (Drosophila)
TAF15 TAF15 RNA polymerase II, TATA box binding protein (TBP)-associated factor, 68kDa
TFF1 trefoil factor 1
TGFBR2 transforming growth factor, beta receptor II (70/80kDa) TGIF2 TGFB-induced factor homeobox 2
TIMP3 tissue inhibitor of metalloproteinase 3
TLS/FUS fusion (involved in t(12;16) in malignant liposarcoma) TOPORS topoisomerase I binding, arginine/serine-rich
TP53BP2 tumor protein p53 binding protein, 2
TP53INP1 tumor protein p53 inducible nuclear protein 1 TP73L (TP63) tumor protein 63
TPM1 tropomyosin 1
TRBP TAR (HIV) RNA binding protein TRIM25 tripartite motif-containing 25
TRKC (NTRK3) neurotrophic tyrosine kinase, receptor, type 3
TUSC2 tumor suppressor candidate 2
UBC9 (UBE2I) ubiquitin-conjugating enzyme E2I (UBC9 homolog, yeast) VEGF vascular epithelial growth factor
Xrn1 5'-3' exoribonuclease 1
ZEB1 zinc finger E-box binding homeobox 1 ZEB2 zinc finger E-box binding homeobox 2
SOMMAIRE
INTRODUCTION...15
I- Généralités sur les miRNAs...17
I-1- Mise en évidence des miRNAs...19
I-1-1- mise en évidence chez le nématode...19
I-1-2- caractérisation des miRNAs dans de nombreuses espèces et de nombreux génomes...20
I-1-3- conservations, origines et implications potentielles des miRNAs au cours de l’Evolution...21
I-2- La nomenclature des miRNAs...22
I-3- Les similitudes et les différences entre les miRNAs et les siRNAs dans le processus de l’interférence à l’ARN ou RNAi...25
II- Mode de synthèse et régulations de l’expression des miRNAs...29
II-1- Localisation génomique des transcrits contenant les miRNAs...29
II-2- transcription des transcrits primaires de miRNAs...31
II-3- Maturation des transcrits ARN contenant les miRNAs...33
II-3-1- libération du pré-miRNA à partir du transcrit primaire dans le noyau...33
II-3-1-1- libération du pré-miRNA par la voie canonique du « microprocessor » Drosha/DGCR8...33
II-3-1-2- libération du pré-miRNA par la voie non canonique des mirtrons...37
II-3-2- export du pré-miRNA vers le cytoplasme...37
II-3-3- excision du miRNA mature du pré-miRNA au niveau du cytoplasme...38
II-3-4- Chargement du miRNA mature dans le complexe RISC...41
III- Les mécanismes de régulation de l’expression post-transcriptionnelle des gènes par les miRNAs...46
III-1- Reconnaissance des ARNm ciblés par RISC, importance de la complémentarité de séquence miRNA::ARNm...46
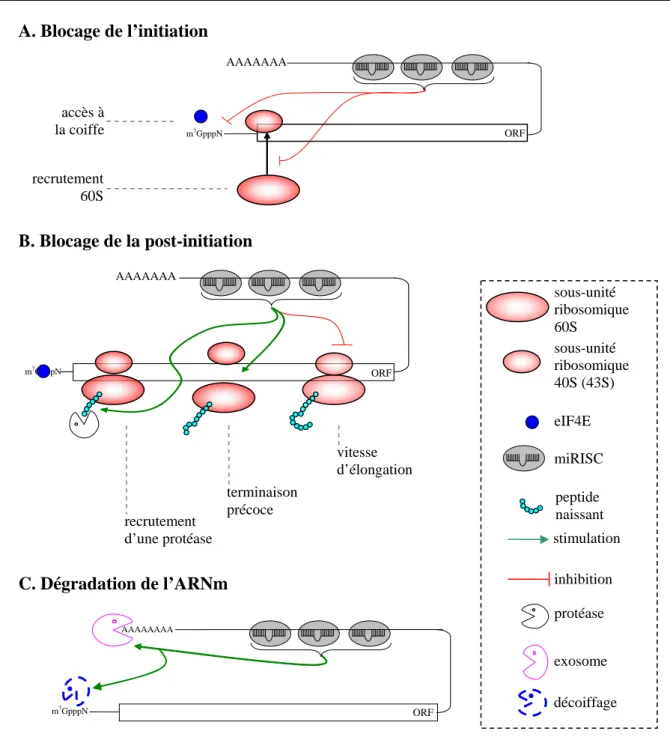
III-2- inhibition de la traduction et/ou dégradation des ARNm par les miRNAs...50
III-2-1- répression de l’initiation de la traduction par les miRISC...51
III-2-2- inhibition de la post-initiation de la traduction...53
III-2-3- cas particulier de l’activation de la traduction par les miRNAs...54
III-2-4- régulation de la dégradation des ARNm ciblés par miRISC...55
IV- miRNAs et cancers...57
IV-1- Perturbations de l’expression des miRNAs dans les cancers...57
IV-1-2- Perturbations d’expression des miRNAs par réarrangement
chromosomique...58
IV-1-3- Perturbations d’expression des miRNAs par régulation transcriptionnelle...59
IV-1-4- Perturbations d’expression des miRNAs par des défauts d’expression des facteurs de maturation des pri/pré-miRNAs...60
IV-1-5- Perturbations d’expression des miRNAs par des polymorphismes de nucléotides...60
IV-1-6- Expression des miRNAs comme outils de pronostic des cancers...61
IV-2- Dérégulation de la liaison miRNA::ARNm cible...61
IV-2-1- Dérégulations liées aux réarrangements chromosomiques...61
IV-2-2- Dérégulations liées aux polymorphismes sur les ARNm...62
IV-2-3- Dérégulations liées aux polymorphismes sur les miRNAs...62
IV-3- Fonction des miRNAs dans le cancer...63
IV-3-1- Les miRNAs oncogènes...63
IV-3-2- Les miRNAs suppresseurs de tumeur...64
IV-3-3- Les miRNAs régulateurs de la prolifération, de l’apoptose et du cycle cellulaire...65
IV-3-4- Les miRNAs régulateurs de l’angiogenèse...67
IV-3-5- les miRNAs régulateurs des processus métastatiques...67
PRESENTATION ET OBJECTIF DE L’ETUDE...87
RESULTATS...89
I- Régulation œstrogénique de l’expression et des fonctions des miRNAs...90
I-1- Introduction...90
I-1-1- Les récepteurs des œstrogènes...90
I-1-2- Mécanismes d’action des RE...91
I-1-3- Les gènes cibles régulés par les RE...94
I-1-4- Les RE dans les cancers du sein...95
I-2- La répression œstrogéno-induite de l’expression de miRNAs est impliquée dans la prolifération des cellules tumorales mammaires...97
I-2-1- stratégie expérimentale...98
I-2-1-1- étude des variations d’expression des miRNAs sous E2...98
I-2-1-2- étude des fonctions des miRNAs candidats dans la prolifération œstrogéno-induite des MCF-7...99
I-2-1-3- Etude in vivo des effets de l’œstradiol sur l’expression des miRNAs dans les cancers du sein...99
I-3- Article…...99
I-4- Conclusion/Discussion...145
II- Expression et fonctions miRNAs dans la progression métastatique des cancers du sein...149
II-1- Introduction...149
II-2- Matériel et méthode...150
II-2-1- Culture cellulaire...150
II-2-2- Cellules et infection virale...151
II-2-3- Formation de tumeurs et de métastases chez la souris...151
II-2-4- Analyse de l’expression des miRNAs par puces à miRNAs...152
II-2-5- Test de migration et d’invasion...152
II-2-6- Caractéristiques des tumeurs de patientes...153
II-3- Résultats...154
II-3-1- Caractérisation de l’expression des miRNAs dans les tumeurs primaires du modèle 4T1...154
II-3-2- Confirmation des données d’expression des miRNAs issus des cribles à grande échelle...154
II-3-3- Effets de l’expression de miR-31, -125b ou -223 sur la migration et l’invasion des cellules 67NR...155
II-3-4-Effets de la sous-expression de miR-31 ou -125b sur la colonisation métastatique pulmonaire chez la souris...156
II-3-5- Analyse du taux d’expression de miR-31 et -125b dans des tumeurs primaires mammaires de patientes atteintes de métastases...156
II-4- Discussion...157
DISCUSSION ET PERSPECTIVES...167
I- Identification des ARNm cibles de miRNAs...169
II- Régulation de l’expression des miRNAs par les exosomes...171
III- Expression des miRNAs comme outils diagnostic/pronostic potentiel des cancers...174
IV- Utilisation d’oligonucléotides comme outils thérapeutiques potentiels des pathologies humaines...176
REFERENCES...181
ANNEXES...213
ANNEXES PROTOCOLES...216
FIGURES
Figure 1 : maturation des miRNAs dans les cellules animales...18
Figure 2 : exemple de nomenclature de la famille miR-181 (humain)...24
Figure 3 : similitudes et différences entre les miRNAs et les siRNAs...26
Figure 4 : localisation génomique des miRNAs...30
Figure 5 : reconnaissance des tige-boucles par le « microprocessor »...34
Figure 6 : clivage des pré-miRNAs par Dicer...40
Figure 7 : Etapes de la formation du complexe RISC chez l’homme...42
Figure 8 : Asymétrie dans la sélection d’un des deux brins d’un double brin de miRNA en tant que miRNA mature...43
Figure 9 : interaction des miRNAs avec les ARNm dans les cellules animales...47
Figure 10 : mécanismes moléculaires de la régulation de la traduction et/ou de la dégradation des ARNm…....52
Figure 11 : domaines fonctionnels des récepteurs des œstrogènes REα et β...91
Figure 12 : modes d’action des récepteurs des œstrogènes...93
Figure 13 : Effet du 17-β-œstradiol sur l’expression d’un gène rapporteur sous contrôle du promoteur du miR-21...146
Figure 14 : Analyse des effets du 17-β-œstradiol sur le recrutement des ARNm au niveau des polysomes dans les cellules MCF-7...148
Figure 15 : caractérisation de l’expression des miRNAs dans le modèle murin 4T1...160
Figure 16 : Analyse supervisée de l’expression des miRNAs dans le modèle murin 4T1...161
Figure 17 : l’expression de miR-31, 125b ou 223 corrèle avec les capacités métastatiques de lignées cancéreuses mammaires...163
Figure 18 : miR-31 et 125b auraient des propriétés pro-métastatiques dans les cellules cancéreuses mammaires...164
Figure 19 : expression des miR-31 et -125b dans une série de tumeurs primaires du sein...165
Figure 20 : effet de miR-31 et 125b sur la migration et l’invasion des cellules MCF-7...166
Figure 21 : effet des exosomes dans la maturation des pri-miRNAs chez Arabidopsis thaliana...173
Annexe figure 1 : modèle d’ARNm bicistronique...214
Annexe figure 2 : profil polysomique des cellules MCF-7...215
TABLEAUX
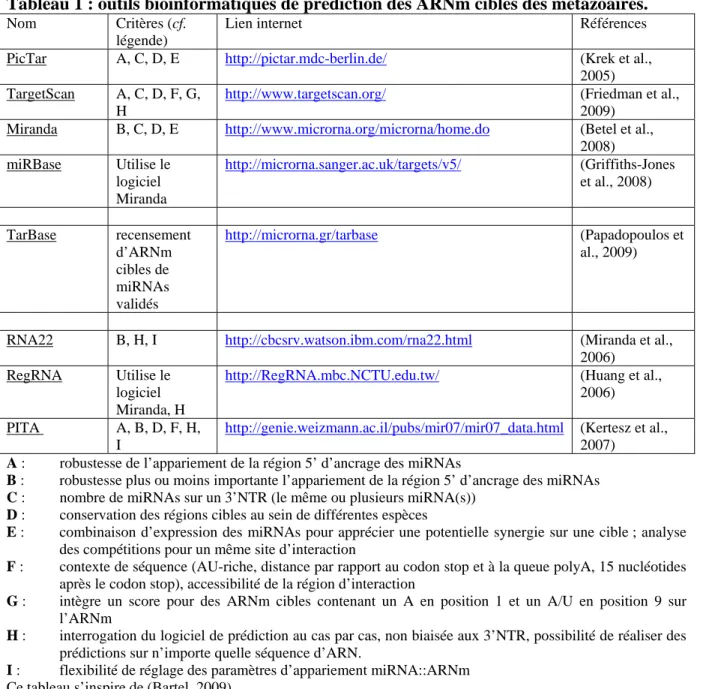
Tableau 1 : outils bioinformatiques de prédiction des ARNm cibles des métazoaires...49Tableau 2 : Différences d’expression des miRNAs dans les cancers...70
Chez les eucaryotes, le contrôle dynamique de l’expression des gènes permet de générer une diversité protéique et fonctionnelle nécessaire à la survie cellulaire et à l’adaptation à l’environnement. Jusqu’aux années 1980, l’idée prévalait que la majorité des régulations des gènes se faisait à l’étape transcriptionnelle. Récemment, d’autres niveaux de régulation ont été mis en évidence au cours d’évènements post-transcriptionnels. Pour être fonctionnels, les ARNm eucaryotes doivent contenir, en plus d'un cadre de lecture, des informations qui définissent leur maturation, leur export nucléaire, leur localisation, leur traduction et leur stabilité. Une notion apparue ces dernières années est que la plupart de ces informations est portée par des protéines de liaison aux ARNm et des microRNAs (miRNAs). Ces molécules s'associent aux transcrits pour former des complexes ribonucléoprotéiques dynamiques qui influencent leur fonction et devenir à travers le contrôle qu'elles exercent sur des étapes spécifiques de l'expression post-transcriptionnelle des gènes.
Dans cette introduction, nous présenterons dans une première partie les généralités sur les miRNAs, leur appartenance au processus d’interférence à l’ARN, leurs mises en évidence dans les génomes, leurs degrés de conservation ainsi que la nomenclature appliquée. Dans une deuxième partie nous détaillerons la localisation, l’organisation des miRNAs sur les génomes et verrons comment ils sont exprimés et maturés. Dans une troisième partie, nous décrirons les mécanismes mis en jeu par les miRNAs pour contrôler l’expression post-transcriptionnelle des gènes. Enfin, dans une quatrième partie, nous nous intéresserons à l’implication des miRNAs dans les cancers.
I- Généralités sur les miRNAs
L’intérêt que porte la recherche scientifique sur les miRNAs a connu un essor à partir des années 2000. Une simple recherche bibliographique pour le terme « microRNA » montre une apparition quasi exponentielle de publications scientifiques entre 2000 et 2008.
Les miRNAs sont des petits ARN non codant d’environ 22 nucléotides répandus dans de très nombreux génomes (référencés dans miRBase, (Griffiths-Jones et al., 2006)). En régulant l’expression post-transcriptionnelle des gènes, les miRNAs sont impliqués dans de très nombreux processus physio(patho)logiques tels que le contrôle du développement des organismes, la régulation de la prolifération, l’apoptose, la différentiation cellulaire ou encore dans divers processus oncogéniques (Kim, 2005). Trois étapes clés aboutissent à la production d’un miRNA mature dans les cellules animales (Bartel, 2004; Bartel, 2009; Filipowicz et al., 2008; Winter et al., 2009) (figure 1) :
(i) l’excision d’une tige-boucle de 60~70 nucléotides d’un transcrit primaire d’ARN
(ou pri-miRNA) par la RNAse III Drosha associée à son cofacteur DGCR8. Il est à noter qu’il existe également une production minoritaire de miRNAs par la machinerie d’épissage où l’intron excisé constitue le miRNA (appelé pré-mirtron)
(ii) l’export de la tige-boucle (ou pré-miRNA) vers le cytoplasme par l’exportine-5
0 200 400 600 800 1000 1200 1400 1600 1800 2000 2000 2001 2002 2003 2004 2005 2006 2007 2008 années n o mbr e de pu bl ic at io n s
Figure 1
Figure 1 : maturation des miRNAs dans les cellules animales.
Drosha DGCR8 ADN transcription m7GpppN AAAAAA exportine-5 noyau cytoplasme // // transcription pri-miRNA pré-mirtron miRISC ou miRNP
(sélection d’un des brins pour incorporation à RISC cf. figure 7) Ago1-4 // // machinerie d’épissage Dicer pré-miRNA
exon exon intron
double brin de miRNA
Le schéma indique les deux voies de production des miRNAs dans les cellules animales. Le panel de gauche représente la voie de production majoritaire des miRNAs. Dans cette voie, les tige-boucles des transcrits primaires (pri-miRNAs) sont excisées par le complexe Drosha/DGCR8. Drosha, une RNAse III, est recrutée par la protéine de liaison aux ARN DGCR8 aux structures en tige-boucle et clive celle-ci, libérant alors le pré-miRNA. Le panel de droite représente la voie de production minoritaire des miRNAs. Dans cette voie, la machinerie d’épissage libère l’intron qui se structure en tige-boucle pour former le mitron. Les pré-miRNAs/pré-mirtrons sont alors exportés par l’exportine-5 (vert), maturés au niveau du cytoplasme par Dicer (violet) pour fournir un double brin de miRNA. L’un des deux brins est alors incorporé dans RISC (RNA-induced silencing complex). Dans notre cas, le brin (en rouge) constitue le miRNA mature et interagit directement avec une des quatre protéines Argonaute (Ago1-4, en jaune) pour former le cœur minimaliste de RISC (miRISC : miRNA-RISC, miRNP : miRNA-ribonucleoprotein). m7GpppN : coiffe, avec N pour un des quatre nucléotides (A, T, C, G), m : méthyl ; AAAAA : queue polyA.
(iii) l’excision de la boucle par la RNAse III Dicer et le choix d’un des deux brins de la tige pour constituer le miRNA mature.
Chez les plantes, cette maturation diffère en partie. En effet, le pré-miRNA généralement plus long (90~120 nucléotides) est maturé dans le noyau ; le double brin de miRNA généré est alors exporté vers le cytoplasme où l’un des deux brins est choisi pour former le miRNA mature (Shabalina and Koonin, 2008).
Les miRNAs matures sont alors incorporés au sein de complexes ribonucléoprotéiques appelés RISC (RNA-Induced Silencing Complex). Au travers de ce complexe ils interagissent avec leurs ARNm cibles, la plupart du temps dans les régions 3’ non traduites (3’NTR), et entraînent l’inhibition de la traduction et/ou de la dégradation des ARNm. Une estimation chez l’homme suggère que les miRNAs cibleraient environ 30% des gènes du génome (Lewis et al., 2005). Les protéines de la famille Argonaute (Ago), qui interagissent directement avec les miRNAs, sont l’une des composantes majeures de la répression exercée par RISC. Il existe quatre protéines Ago encodés par quatre gènes distincts dans le génome humain (Ago1-4), 26 chez Caenorhabditis elegans, deux chez Drosophila melanogaster, dix chez Arabidopsis
thaliana (Hutvagner and Simard, 2008).
I-1- Mise en évidence des miRNAs
I-1-1- mise en évidence chez le nématode
Plusieurs éléments vont amener à la caractérisation des miRNAs, le premier découvert étant lin-4 :
(i) le gène lin-4 fait partie de la famille des gènes hétérochroniques qui contrôlent la coordination temporelle du développement larvaire du nématode (division, différentiation, migration et mort cellulaires) (Ambros, 1989). Le produit du gène est requis dans presque toutes les transitions des différents stades larvaires (Ambros, 1989; Chalfie et al., 1981), et notamment il est requis pour la sous-expression de la protéine lin-14 à partir du milieu du stade larvaire L1 et subséquemment (Ruvkun et al., 1991)
(ii) l’(es) élément(s) régulateur(s) négatif(s) nécessaire(s) à la répression temporelle de la protéine lin-14 au cours du développement se situe dans le 3’NTR de l’ARNm
(iii) il a été suggéré que lin-4 puisse être l’élément en trans capable de se lier à la région régulatrice du 3’NTR de lin-14 (Ruvkun et al., 1991).
Ainsi, en 1993, le groupe d’Ambros détermine le produit du gène lin-4 non pas comme une protéine mais comme étant compris de deux petits ARNs : lin-4S le plus abondant d’une longueur de 22 nucléotides, et lin-4L de 61 nucléotides (Lee et al., 1993). Les auteurs suggèrent que lin-4S soit le produit final provenant de lin-4L, et que, lin-4L formerait une tige-boucle avec lin-4S dans l’un des bras de la tige. Cet article caractérise pour la première fois ce que l’on appellera plus tard les précurseurs de miRNAs (l’ARN de 61 nucléotides) ainsi que le miRNA mature (22 nucléotides). Ces deux petits ARN contiennent des séquences complémentaires à des éléments répétés situés dans le 3’NTR de l’ARNm lin-14 (spécifiquement dans la région régulatrice mise en évidence par (Ruvkun et al., 1991; Wightman et al., 1991)). Leur modèle propose alors une régulation de la traduction de lin-14 par lin-4 via une interaction ARN-ARN antisens. Parallèlement, un autre article démontre aussi que le 3’NTR de l’ARNm lin-14 est nécessaire et suffisant pour conférer la régulation de lin-14 par lin-4 par des expériences de gènes rapporteurs (Wightman et al., 1993). Ils proposent également un mécanisme par lequel lin-4 interagit par appariement complémentaire sur le 3’NTR de l’ARNm lin-14 pour en inhiber sa traduction. Ce modèle de régulation de la traduction sera en partie validé en 1999 (Olsen and Ambros, 1999).
La découverte de lin-4 chez le ver fut considéré comme une singularité jusqu’en 2000 avant la découverte d’un second ARN de 21 nucléotides, let-7 (Reinhart et al., 2000). Ce petit ARN, tout comme lin-4 (i) fut mis en évidence par crible génétique en réalisant des mutations du génome de C. elegans, (ii) est un petit ARN régulé de manière temporelle et dont la fonction est essentielle au cours du développement, (iii) intervient dans la transition du stade larvaire L3/L4 vers les cellules adultes en partie en réprimant l’expression de lin-41 (Reinhart et al., 2000).
I-1-2- caractérisation des miRNAs dans de nombreuses espèces et de nombreux génomes
L’observation que la séquence de let-7 soit conservée au cours de l’évolution amène à la découverte de ce petit ARN dans plusieurs tissus humains mais aussi chez Drosophila
melanogaster (Pasquinelli et al., 2000). Plusieurs laboratoires vont par la suite décrire une
multitude de petits ARNs dans des lignées cellulaires humaines, chez la mouche, le ver, les poissons, les plantes (Ambros et al., 2003; Aravin et al., 2003; Dostie et al., 2003; Grad et al., 2003; Houbaviy et al., 2003; Quintana et al., 2001; Quintana et al., 2003;
Lagos-Quintana et al., 2002; Lau et al., 2001; Lee and Ambros, 2001; Lim et al., 2003a; Michael et al., 2003; Mourelatos et al., 2002). Si en 2000, lin-4 et let-7 furent appelés stRNA (small temporal RNA ; (Pasquinelli et al., 2000)) car ils sont régulés au cours du développement, ils sont renommés miRNAs en 2001 car nombre d’entre eux sont exprimés sans relation avec les étapes du développement (Lagos-Quintana et al., 2001). La plupart des miRNAs décrits proviennent de techniques de séquençage par clonage préalable. Des approches informatiques se basant sur (i) la détection des tige-boucles de 60~70 nucléotides caractéristique des pré-miRNAs (Lim et al., 2003a), (ii) une conservation des pré-miRNAs au sein des espèces ont également été utilisées pour définir de nouveaux miRNAs. Il est à noter qu’à leur mise en place, les logiciels de prédiction ont démontré leur efficacité chez les animaux mais une inefficacité de détection des miRNAs chez les plantes car les structures en tige-boucles de leurs précurseurs sont plus longues (Lim et al., 2003a). Par la suite, des logiciels plus perfectionnés ont permis de détecter ~97% des pré-miRNAs de la plante après « entraînement » sur le génome humain (Kadri et al., 2009). En 2009, la base de données miRBase ((Griffiths-Jones, 2004), http://www.sanger.ac.uk/Software/Rfam/mirna/) recense 885 miRNAs dans le génome humain, 689 chez la souris, 153 miRNAs chez Drosophila
melanogaster, 207 miRNAs chez Arabidopsis thaliana. Pas moins de 105 génomes
(comprenant plus de 8000 miRNAs) répartis entre Métazoaires, Mycétozoaires, Viridiplantae et Virus possèdent au moins un miRNA. De manière intéressante et mis à part les virus, les miRNAs se retrouvent dans des génomes d’organismes multicellulaires. Un des seuls exemples connus maintenant d’organisme unicellulaire possédant des miRNAs est
Chlamydomonas reinhardtii (Zhao et al., 2007). Aucune littérature ne fait état de la présence
de miRNA chez des bactéries ou chez les eucaryotes inférieurs tels que Saccharomyces
cerevisiae.
I-1-3- conservations, origines et implications potentielles des miRNAs au cours de l’Evolution
Chez les animaux comme chez les plantes, les miRNAs semblent exister depuis plus de 400 millions d’années (Floyd and Bowman, 2004; Pasquinelli et al., 2000). Chez les plantes, il a été montré que de nombreuses familles de miRNAs sont conservées chez les mousses, gymnospermes, monocotylédones et eucotylédones (Zhang et al., 2006a). De même chez les animaux, de nombreux miRNAs sont conservés entre espèces apparentées et certains le sont même entre invertébrés et vertébrés (Hertel et al., 2006; Lau et al., 2001; Lee and
Ambros, 2001; Pasquinelli et al., 2003; Pasquinelli et al., 2000). S’il apparaît que les miRNAs soient conservés au sein des deux règnes, il existe très peu de conservation croisée entre les deux. Une seule étude fait état d’une conservation entre les miR854 et 855 de la plante (Arabidopsis thaliana) et des animaux (homme, souris, nématode) (Arteaga-Vazquez et al., 2006). Du fait de cette conservation de miR854 et 855, et, du fait que les miRNAs soient retrouvés chez les plantes mais aussi chez les métazoaires, il a été suggéré que ces petits ARNs étaient déjà présents chez leur ancêtre commun (Bartel, 2004). Cependant, on ne peut exclure une apparition indépendante des miRNAs au sein des deux règnes car de nombreuses différences structurales ainsi qu’une différence de maturation sont observées entre les plantes et les animaux (pour revue (Shabalina and Koonin, 2008)). L’origine des miRNAs reste encore hypothétique ; néanmoins, il a été suggéré que les miRNAs dérivent d’éléments transposables (Piriyapongsa and Jordan, 2007; Piriyapongsa and Jordan, 2008; Piriyapongsa et al., 2007; Shabalina and Koonin, 2008).
Plusieurs études ont fait ressortir une implication potentielle des miRNAs dans la complexification morphologique des organismes. Ces observations se basent sur le fait que l’augmentation du nombre de gènes codant des protéines (par duplication) soit impliquée dans les processus de complexification des organismes, mais elle ne peut à elle seule expliquer ces phénomènes, notamment dans l’acquisition de nouvelles morphologies et de nouveaux caractères (Donoghue and Purnell, 2005; Heimberg et al., 2008). De manière intéressante, il a été observé que plusieurs évènements d’acquisition de nouveaux miRNAs dans les génomes de certaines espèces corrèlent avec des innovations du développement (Hertel et al., 2006; Prochnik et al., 2007; Zhang et al., 2008b). De plus, trois publications ont suggéré que l’innovation et l’ajout de nouveaux miRNAs est un processus en cours dans les génomes de métazoaires, et, lorsqu’ils sont intégrés à un réseau de gènes subiraient peu de mutations et seraient rarement perdus suite à leurs acquisitions (Heimberg et al., 2008; Hertel et al., 2006; Wheeler et al., 2009).
I-2- La nomenclature des miRNAs
Devant le nombre croissant de miRNAs découverts, la base de données miRBase a été créée pour les recenser et uniformiser leurs nomenclatures ((Jones, 2004; Griffiths-Jones et al., 2006)).
Seuls les miRNAs qui font l’objet d’une publication scientifique sont nommés, à savoir démonstration par clonage, par étude d’expression et/ou de maturation. Il est impératif que le
miRNA mature soit retrouvé dans une structure en tige-boucle (5’ et/ou 3’ du bras de la tige). Les miRNAs détectés par homologie de séquence peuvent être nommés sans évidences expérimentales mais en respectant le repliement en tige-boucle sur le génome détecté (Griffiths-Jones et al., 2006).
La nomenclature des miRNAs se fait selon plusieurs critères. Historiquement, le produit mature des gènes codant les miRNAs est écrit « miR » alors que les précurseurs ou les gènes eux-mêmes sont écrits « mir » (Griffiths-Jones et al., 2006). Le préfixe des miRNAs se constitue de trois lettres indiquant l’organisme d’origine (hsa-miR-21 : homo sapiens ; mmu-miR-24 : mus musculus) et est suivi d’un suffixe chiffré. Un pré-miRNA est capable de donner en théorie deux miRNAs matures qui constituent soit le bras 5’ (5p) de la tige soit le bras 3’ (3p) de la tige. Une sélection est réalisée entre les deux bras de la tige pour n’en garder qu’un seul. Si un pré-miRNA ne fournit qu’un seul miRNA mature il sera nommé miR-X, X étant le chiffre. Si le pré-miRNA fournit deux miRNAs matures, la nomenclature devient :
- miR-X-5p miR-X-3p (exemple : miR-17-5p et miR-17-3p), ou,
- miR-X et miR-X* (exemple : miR-17 et miR-17*). La nomenclature (*) représente le miRNA le moins abondant par rapport aux deux brins de la tige
- miR-X-s (bras 5’) ou miR-X-as (bras 3’) (Griffiths-Jones, 2004).
Il est suggéré de favoriser la nomenclature miR-X-5p ou miR-X-3p pour un pré-miRNA fournissant deux pré-miRNAs matures jusqu’à ce que les données soient suffisantes pour utiliser la nomenclature miR-X et miR-X* relatif à l’abondance des deux brins (Griffiths-Jones, 2004). Une étude a cependant révélé que certains miR-X* sont retrouvés en plus grande quantité que le X correspondant, complexifiant cette appellation X et miR-X* (Okamura et al., 2008).
On retrouve également des familles de miRNAs matures, à savoir possédant une homologie de séquence. Ce sont des miRNAs qui diffèrent de un à trois nucléotides. Leur nomenclature est alors par exemple miR-181a et miR-181b, a et b marquant leurs différences (figure 2). Des loci différents qui fournissent un même miRNA mature sont indiqués par un suffixe chiffré. La séquence du miRNA mature doit être identique. La nomenclature est par exemple miR-181a-1 et miR-181a-2, 1 et 2 soulignés indiquant la provenance de deux régions chromosomiques différentes (figure 2).
Nous devons noter que miR-181a-1* et a-2* sont relativement proches, avec trois bases différentes. Cependant, il est difficile de les classer en famille car même s’ils ont trois bases de différences, deux d’entre elles se situent dans la région 5’ du miRNA (figure 2). Cette
Figure 2
Figure 2 : exemple de nomenclature de la famille miR-181 (humain)
5’ 3’
5’
3’
issu du chromosome 1 issu du chromosome 9
pri-mir-181-2 pri-mir-181-1
indication de deux chromosomes différents
miR-181a 5’ AACAUUCAACGCUGUCGGUGAGU 3’ (issu des chromosomes 1 et 9)
miR-181b 5’ AACAUUCAUU
La famille des miR-181 est encodée sur deux loci distincts qui produisent potentiellement deux pri-miRNAs, le pri-mir-181-1 (chromosome 1) et le pri-mir-181-2 (chromosome). La distinction chromosomique est marquée par les -1 ou -2 soulignés. Quatre miRNAs peuvent être produits à partir de ces loci, 181a (rouge), miR-181b (vert), miR-181a-1* (rose) et miR-181a-2* (bleu). * indique le miRNA le moins abondant par rapport aux deux brins de la tige. Trois bases différencient miR-181a de miR-181b ou miR-181a-1* de miR-181a-2* (bases soulignées). Néanmoins seuls les miR-181a et -181b sont considérés comme faisant partie d’une même famille car les bases différentes se situent hors de la région 5’ d’ancrage des miRNAs (nucléotides 2-7), région nécessaire pour la reconnaissance des ARNm cibles.
région 5’ est cruciale pour la reconnaissance des ARNm cibles (cf. § III-1), et, dans le cas de ces deux miRNAs il s’avère que les cibles seraient trop différentes. Ainsi, la notion de famille entre miRNAs tient de plus en plus compte de l’ensemble commun d’ARNm cibles que ces derniers pourraient réguler.
Lors de la mise en place du consensus, il a été proposé que les noms des miRNAs ne devaient porter que peu d’informations (homologie/paralogie/orthologie, localisation chromosomique) (Griffiths-Jones et al., 2006). Au cas par cas, l’attribution du nom lorsqu’il s’avère complexe (5p, 3p, *) est laissé à l’appréciation de l’auteur de la soumission. Aussi, cette nomenclature
GCUGUCGGUGGGU 3’ (issu des chromosomes 1 et 9)
5’ ACCAUCGACCGUUGAUUGUACC 3’ (issu du chromosome 1) 5’ ACCACUGACCGUUGACUGUACC 3’ (issu du chromosome 9) miR-181a-1*
miR-181a-2*
est susceptible d’être modifiée, notamment pour le degré de conservation des miRNAs entre eux (Griffiths-Jones et al., 2006).
Il est à noter qu’il existe quelques entorses aux règles pour certains miRNAs, par exemple pour let-7, lin-4 ou bantam. En effet, leurs noms sont de type hsa-let-7, cel-lin-4 ou encore dme-bantam.
I-3- Les similitudes et les différences entre les miRNAs et les siRNAs dans le processus de l’interférence à l’ARN ou RNAi
Les miRNAs font partie de la famille des petits ARN impliqués dans le processus de l’interférence à l’ARN, appelé RNAi (RNA interference). Ce mécanisme est conservé au cours de l’évolution et se retrouve dans divers organismes uni- et pluricellulaires (Shabalina and Koonin, 2008). Il est impliqué dans les défenses contre les virus, les éléments transposables mais également dans la régulation post-transcriptionnelle de l’expression des gènes (pour revue (Bartel and Bartel, 2003; Carthew and Sontheimer, 2009; Umbach and Cullen, 2009)).
Le RNAi est activé par la reconnaissance d’ARN double brin exogènes (virus) ou endogènes (pré-miRNAs, éléments transposables, transcription d’ARN antisens) qui sont maturés en petits ARN de 20~25 nucléotides par Dicer afin d’être incorporé à RISC (Bernstein et al., 2001; Fire et al., 1998; Hamilton and Baulcombe, 1999; Hammond et al., 2000) (figure 3). Il est à noter que chez les plantes, le ver ou certaines levures, les RdRp (RNA-dependent RNA polymerase) peuvent transformer des ARN simple brin (considérés comme aberrants) en ARN double brin pour amplifier le RNAi et réguler l’ARN amplifié (Petersen and Albrechtsen, 2005; Sijen et al., 2001; Wassenegger and Krczal, 2006). Au sein de RISC, le petit ARN assure la spécificité de l’ARN à cibler par complémentarité de séquence et permet ainsi à Argonaute d’amorcer la répression de l’ARN ciblé. Deux classes principales de petits ARN provenant d’ARN double brin, les miRNAs et les siRNAs (small interfering RNAs), interviennent dans le RNAi.
Les siRNAs et miRNAs sont relativement similaires (pour revue (Adams et al., 2007; Bartel, 2004; Carthew and Sontheimer, 2009; Tang, 2005), figure 3) :
- ils dérivent d’ARN double brin et font à peu près 21 nucléotides - ils peuvent être produits de façon endogène
- ils sont maturés par Dicer et utilisent RISC pour induire la répression de leurs cibles.
Figure 3
Mécanismes d’action Cibles ARNs
Fonctions long double brin d’ARN
double brin de siRNA
5’ 5’ 5’ 5’ 5’ 5’ siRISC siRISC infection virale, transposon,
transcrits d’ARN endogènes complémentaires (transcription antisens)
ARN viraux de transposons
de transcriptions antisens
dégradation endonucléolytique
défense antivirale, inhibition de la propagation des éléments transposables, régulation génique (par autoinhibition du gène produisant le siRNA) Dicer 5’ pri-miRNA pré-miRNA 5’
double brin de miRNA
miRISC / miRNP gène contenant un miRNA
ARNm
- inhibition de la traduction
- dégradation exonucléolytique voire endonucléolytique
inhibition post-transcriptionnelle de l’expression génique (autre que le locus produisant le miRNA lui-même)
5’
5’
Figure 3 : similitudes et différences entre les miRNAs et les siRNAs
Le panel de gauche indique la production de siRNAs, provenant soit du génome (transcription antisens ou transposons) soit d’infections virales, à partir de longs doubles brins d’ARN parfaitement appariés. Dans le cas présenté, deux doubles brins des siRNAs peuvent être produits (en vert/bleu et rouge/violet). Le panel de droite indique la production de miRNAs à partir de pri-miRNAs. Seul un seul double brin de miRNA (vert pâle/orange) est produit à partir de la tige-boucle de pré-miRNA (cf. figure 1). Les deux voies de maturation convergent vers la protéine Dicer qui réalise le clivage de doubles brins d’ARN. Les siRNAs ou miRNAs sont alors chargés avec une protéine Ago (en jaune) pour former des siRISC (siRNA-RISC) ou des miRISC/miRNP. Les cibles respectives, les mécanismes d’action mis en jeu par les siRISC et miRISC ainsi que les fonctions des siRNAs et des miRNAs sont indiqués. RISC : RNA-Induced Silencing Complex ; RNP :ribonucleoproteins.
Quelques différences permettent néanmoins de les caractériser.
- les miRNAs sont conservés au cours de l’évolution alors que les siRNAs ne le sont pas ou moins.
- les siRNAs sont issus de longs ARN double brin parfaitement appariés pouvant être endogènes ou exogènes (issus de la transcription antisens (Watanabe et al., 2008; Yelin et al., 2003), d’éléments transposables, de virus, de synthèse d’ARN double brin par les RdRp à partir de matrice simple brin (Petersen and Albrechtsen, 2005; Sijen et al., 2001; Wassenegger and Krczal, 2006)). Les miRNAs, étant exclusivement produits façon endogène, sont issus d’ARN double brin se repliant en tige-boucle (pré-miRNAs) et l’appariement dans la tige est en général imparfait.
- Un seul double d’ARN est généré à partir de pré-miRNAs alors qu’une multitude de siRNAs sont générés à partir des précurseurs.
- les siRNAs régulent les gènes qui les produisent alors que les miRNAs sont impliqués dans le contrôle post-transcriptionnel de gènes autres que les loci les produisant.
- les siRNAs s’hybrident parfaitement à leurs cibles et induisent la dégradation de l’ARN ciblé par clivage endonucléolytique. Néanmoins, chez les mammifères, le clivage endonucléolytique n’est possible que si les siRNAs sont associés à Ago2 car elle seule possède cette activité (Liu et al., 2004). Les miRNAs en fonction du degré d’appariement ARN cible::miRNA induisent une répression traductionnelle et/ou une dégradation de l’ARNm ciblé. Chez les plantes la distinction entre siRNAs et miRNAs dans le mécanisme de régulation de l’ARN ciblé est minime. En effet, ces miRNAs s’hybrident de façon quasi-parfaite à l’ARNm cible pour le dégrader (Bartel, 2004). Toutefois, il a été montré que les miRNAs de plantes peuvent également réguler la traduction (Brodersen and Voinnet, 2009).
- la fonction des siRNAs est dévolue au maintien de l’intégrité des génomes contre des acides nucléiques invasifs (suppression des virus, des transposons) mais également dans la régulation post-transcriptionnelle de l’expression des gènes (pour la transcription antisens). Les miRNAs sont plutôt spécialisés dans la gestion du transcriptome et du protéome d’une cellule (Carthew and Sontheimer, 2009).
Récemment une autre classe de petits ARN de 25~30 nucléotides, les piRNAs (piwiRNAs), ont été découverts chez les animaux et interviennent dans le processus du RNAi. Ils ne sont
pas produits à partir d’ARN double brin. Leur maturation n’est pas totalement comprise mais certains éléments semblent indiquer qu’ils dérivent de précurseurs simples brins. Les piRNAs sont exprimés dans les lignées germinales. Ils se lient à une famille d’Ago ne pouvant lier que les piRNAs, au nombre de quatre dans le génome humain, et différent du complexe Ago-siRNAs ou Ago-miRNAs. Au travers des piRISC, les piRNAs servent à la mise en silence d’éléments transposables afin de préserver l’intégrité du génome et éviter la prolifération des transposons dans les cellules germinales (pour revue (Malone and Hannon, 2009)).
II- Mode de synthèse et régulations de l’expression des miRNAs
II-1- Localisation génomique des transcrits contenant les miRNAs
Le clonage extensif des miRNAs ainsi que leur positionnement sur divers génomes étudiés, en particulier chez l’homme, nous révèlent que les unités géniques contenant les miRNAs se situent soit dans des régions intergéniques indépendantes (figure 4A, cas n°1) soit dans des unités géniques codant des pré-ARNm (figure 4A, cas n°2). Dans les deux cas, les miRNAs peuvent se regrouper sur certains transcrits primaires en « cluster » de pré-miRNAs, suggérant pour ces derniers une possible unité transcriptionnelle polycistronique (figure 4A). En général, les miRNAs issus de polycistrons ont une expression similaire (Liang et al., 2007; Ozsolak et al., 2008), mais des stratégies peuvent exister pour découpler l’expression des miRNAs d’un même « cluster » (Sun et al., 2009).
Une estimation faite via la base de données miRBase (706 pré-miRNAs sur le génome humain, en avril 2009), suggère qu’il y aurait autant de miRNAs dans des gènes codant des protéines que dans des unités géniques indépendantes chez l’homme (341 dans des introns et/ou exons alors que 365 sur 706 sont dans des unités géniques indépendantes ; données personnelles et (Saini et al., 2008)). Chez Drosophila melanogaster 65% (99 sur 152) seraient dans des unités géniques indépendantes (données personnelles, miRBase). Il est à noter que chez les plantes, la majorité des miRNAs seraient dans des unités géniques indépendantes (Shabalina and Koonin, 2008).
Un faible pourcentage des miRNAs situés dans des unités géniques indépendantes se retrouve dans une orientation antisens par rapport à un gène codant une protéine (14% pour la drosophile, 13% pour l’homme et aucun pour Arabidopsis). Parmi ces miRNAs, quelques uns peuvent être retrouvés en antisens d’un exon d’un gène et pourraient dégrader l’ARNm par effet siRNA (figure 4B). Par exemple, il a été montré que miR-136 et miR-127 étant orientés en antisens du rétrotransposon Rtl1 entraînent le clivage de l’ARN de ce dernier (Seitz et al., 2003). Il en serait de même pour miR-220a et LOC402422, miR-220b et TUBB4, miR-662 et
MSLNL, miR-1538 et NFAT5, ou encore miR-1184 et F8A1 (données personnelles).
Chez l’homme, parmi les miRNAs situés dans des unités géniques codant des protéines, 88% des miRNAs seraient dans des introns, 10% dans des exons (dont 12% dans les régions 5’ non traduites (5’NTR), 52% dans les cadres de lecture ouvertes (ORF : Open
Figure 4
A
cas n°2 ADN pré-ARNm pri-miRNAB
AAAA m7GpppN cas n°2cas n°1 unité génique indépendante unité génique codant des pré-ARNm pré-miRNAs exons clivage de l’ARNm ADN pré-ARNm / pri-miRNA
C
88% dans des introns 10% dans des exons 2% sur des introns/exons chez l’homme +1 ADN pré-TMEM49 / pri-mir-21D
/ / +1pri-mir-21 +1 point +1 de transcription
ADN m7 GpppN coiffe en 5’ +1 pri-miRNA « cluster » de pri-miRNA +1 cas n°1 pré-ARNm / pri-miRNA pré-ARNm / « cluster » de pri-miRNA +1 +1 +1 +1 +1 introns
Figure 4 : localisation génomique des miRNAs
(A) Les miRNAs peuvent être codés dans des unités géniques indépendantes (cas n°1) soit sur des transcrits ne possédant qu’une seule tige-boucle soit sous forme de « cluster » (plusieurs tige-boucles). Ils peuvent se retrouver dans des unités géniques codant des pré-ARNm (cas n°2) sous forme de cluster ou non. (B) Dans certains cas, en particulier chez l’homme, certains pré-miRNAs (vert) se retrouvent en antisens d’un exon (rectangle gris) d’un pré-ARNm, et suite aux différentes maturations, le miRNA pourra alors reconnaître l’ARNm et le cliver. (C) Répartition des pré-miRNAs situés dans des unités géniques codant des pré-ARNm chez l’homme. (D) Les miRNAs peuvent être localisés dans des unités géniques codant des pré-ARNm mais sont transcrits par leurs propres promoteurs. Par exemple, miR-21 peut dériver de sa propre unité transcriptionnelle (pri-mir-21) mais également du pré-ARNm TMEM49 (pré-TMEM49 / pri-mir-21).
Reading Frame) et 36% dans des 3’NTR) et 2% à cheval sur des introns-exons (données personnelles, miRBase) (figure 4C). Chez Drosophila melanogaster la quasi-totalité des miRNAs colocalisés avec des gènes codant sont dans des introns (données personnelles, miRBase). Si les miRNAs situés dans des introns de pré-ARNm n’affecteraient pas l’intégrité du futur ARNm, ceux localisés sur des exons pourraient détruire ce dernier lors de la maturation de la tige-boucle. Cette compétition potentielle entre production d’ARNm et de miRNAs pourrait constituer un autre niveau de régulation de l’expression de ces gènes.
Les miRNAs situés dans des gènes codant des protéines, dans les introns des pré-ARNm en particulier, suivent en général le même patron d’expression que le gène qui le porte (Baskerville and Bartel, 2005; Saini et al., 2008). Néanmoins, un tiers des miRNAs colocalisés dans des gènes codant des protéines serait transcrit par leur propre promoteur et leur expression pourrait être découplée du pré-ARNm (Ozsolak et al., 2008). C’est le cas de miR-21 qui est transcrit par son propre promoteur situé dans le dernier intron du gène
TMEM49 ((Cai et al., 2004), (figure 4D)). L’expression de miR-21 pourrait être la résultante
de deux transcrits d’ARN, le pri-miR-21 mais également celui du pré-ARNm TMEM49.
Il est à noter que trois tige-boucles capables de fournir des pré-miRNAs à partir du génome nucléaire humain se retrouvent sur le génome mitochondrial (hsa-miR-1974, 1977 et 1978 ; observation sur miRBase ; (Schotte et al., 2009)). Il reste maintenant à définir si à partir du génome mitochondrial un miRNA peut être produit ou si c’est une coïncidence sur la conservation de séquences, ce qui pourrait potentiellement aider à la compréhension de l’origine et de l’évolution des miRNAs.
II-2- transcription des transcrits primaires de miRNAs
Une récente étude à grande échelle a permis de caractériser à 150 bases près le point +1 de transcription de 175 miRNAs humains, et, combinée à des expériences d’immunoprécipitation de la chromatine suggère que la transcription par l’ARN polymérase II soit majoritaire (Ozsolak et al., 2008). Des études plus ciblées révèlent aussi que les pri-miRNAs portent en général les marques d’une transcription par l’ARN polymérase II (Bracht et al., 2004; Cai et al., 2004; Houbaviy et al., 2005; Lee et al., 2004). Ces ARNs relativement longs (plus de 1000 bases) ont une coiffe en 5’ avec la structure caractéristique m7G(5’)ppp(5’)N (N étant un des quatre nucléotides) et une extrémité 3’ polyadénylée. Une expérience de rétention des ARN coiffés montre que sept pri-miRNAs choisis de manière
aléatoire se fixent à la protéine se liant à la coiffe (eIF4E) alors que les pré-miRNAs et miRNAs respectifs ne sont pas retenus (Lee et al., 2004). Il a également été montré que le pri-miR-290~295 murin est coiffé, épissé et polyadénylé (Houbaviy et al., 2005). Plusieurs promoteurs de miRNAs, clonés dans un plasmide en amont de gènes rapporteurs (luciferase ou gfp), sont capables de les exprimer, suggérant une activité de l’ARN polymérase II. C’est le cas pour les promoteurs des pri-miR-21, pri-miR-17~92 (humain), pri-miR-290~295 (murin), pri-lsy-6 et pri-let-7 (nématode) (Cai et al., 2004; Houbaviy et al., 2005; Johnson et al., 2003; Johnston and Hobert, 2003; Ozsolak et al., 2008). L’ARN polymérase II est capable de s’associer physiquement au promoteur du pri-miR-23a~24-2 ; de plus, l’expression de ce dernier est diminuée par de faibles concentrations d’α-amanitine, inhibant spécifiquement l’ARN polymérase II (Lee et al., 2004).
Il a aussi été montré que plusieurs facteurs de transcription, régulant généralement l’ARN polymérase II, sont capables de réguler un grand nombre de miRNAs. Une surexpression de c-Myc, entraîne la répression de nombreux miRNAs ; cette diminution d’expression est corrélée à un enrichissement de la fixation de c-Myc aux promoteurs de ces pri-miRNAs (Chang et al., 2008). De plus, c-Myc est capable d’activer l’expression du « cluster » pri-17~92 contenant sept miRNAs (O'Donnell et al., 2005) ou de réprimer l’expression des miR-23a et 23b (Gao et al., 2009). Le facteur de transcription p53 régule également de nombreux miRNAs dont miR-34a (Chang et al., 2007; Raver-Shapira et al., 2007). Twist, un facteur de transcription impliqué dans le développement est capable de contrôler l’expression de miR-10b chez la souris (Ma et al., 2007).
Certaines études ont aussi montré que les pri-miRNAs peuvent être transcrits par l’ARN polymérase III. En effet, chez l’homme il a été proposé qu’environ 20% des miRNAs seraient transcrits par cet ARN polymérase, environ la moitié se situant dans le « cluster » C19MC (chromosome 19 miRNA cluster) (Borchert et al., 2006). Néanmoins, il a récemment été montré que ce « cluster » serait plutôt transcrit par l’ARN polymérase II (Bortolin-Cavaille et al., 2009). Plusieurs miRNAs du génome γ-herpes-virus 68 murin sont transcrits par l’ARN polymérase III (Pfeffer et al., 2005). Enfin, il a été montré que l’ARN polymérase III peut être recruté sur au moins 11 promoteurs de miRNAs ; cependant, 8 de ces 11 promoteurs recrutent les deux ARN polymérase II et III mais leur transcription serait plutôt lié à l’ARN polymérase II (Ozsolak et al., 2008).
II-3- Maturation des transcrits ARN contenant les miRNAs
II-3-1- libération du pré-miRNA à partir du transcrit primaire dans le noyau
Jusqu’à présent, deux voies alternatives de production des miRNAs sont décrites chez les animaux (figure 1). La voie canonique fait intervenir le complexe Drosha-DGCR8 alors que la voie minoritaire utilise la machinerie d’épissage.
II-3-1-1- libération du pré-miRNA par la voie canonique du « microprocessor » Drosha/DGCR8
A partir de l’observation de plusieurs transcrits primaires de miRNAs, trois éléments de structure ont pu être caractérisés et sont plus ou moins essentiels à l’étape de clivage et de libération des pré-miRNAs à partir des pri-miRNAs (figure 5) (Han et al., 2006) :
(i) une boucle terminale qui ne semble a priori pas nécessaire au clivage bien qu’une petite boucle diminue la réaction de coupure
(ii) une tige d’environ trois tours d’hélices (~33 pb) avec quelques mésappariements composée d’une partie basse et d’une partie haute
(iii) des séquences flanquantes non appariés essentielles au clivage de la tige.
Le « microprocessor », un complexe protéique composé de Drosha et DGCR8 (appelé également pasha, partner of Drosha) reconnaît ces structures et réalise l’étape de clivage (Gregory et al., 2004). Drosha est une RNAse contenant deux domaines RNAse de type III qui clive de manière asymétrique les deux brins d’ARN de la tige, provoquant la libération de la tige-boucle du transcrit primaire (Lee et al., 2003). Cette asymétrie de clivage propre aux RNAse III résulte en la présence de deux nucléotides non appariés à l’extrémité 3’ de la tige-boucle ainsi qu’un phosphate en 5’ (figure 5). Drosha, bien que possédant un domaine de liaison aux ARNs double brin en extrémité C-terminale, ne se lie pas directement aux tige-boucles. En effet, c’est DGCR8 (ayant deux domaines de liaison aux ARNs double brin) qui reconnaît la base de la tige et les séquences flanquantes non appariées, et qui « mesurerait » la distance de clivage pour Drosha à partir de ces dernières (coupure vers 11 pb dans l’hélice, figure 5) (Han et al., 2006). Il a été observé que la fixation de l’hème à DGCR8 augmente les capacités de clivage du « microprocessor » (Faller et al., 2007).
Figure 5 5’ 3’ 5’-P 3’-OH séquence flanquante partie basse ~11 pb partie haute ~22 pb boucle terminale Drosha DGCR8 tige ~33 pb
Figure 5 : reconnaissance des tige-boucles par le « microprocessor ».
Un pri-miRNA (chez les animaux) se compose de séquences flanquantes non appariées, d’une tige d’environ 33 paires de bases (pb) se décomposant d’une partie basse et d’une partie haute et d’une boucle terminale de 6-10 nucléotides. DGCR8 (marron) reconnaît la partie basse de la tige et positionne Drosha (vert) pour induire un clivage asymétrique de la tige (deux nucléotides non appariés en 3’-OH du produit). Dans notre cas, le miRNA (rouge) est positionné en 5’ de la tige, par conséquent, Drosha définit son extrémité 5’. 3’-OH : 3’-hydroxyl 5’-P : 5’-phosphate. Adapté de (Han et al., 2006).
L’étape de libération du pré-miRNA par le « microprocessor » est cruciale car elle détermine au nucléotide près l’extrémité 5’ et/ou 3’ du miRNA mature (figure 5). Si le miRNA se situe sur le bras 5’ de la tige, l’extrémité 5’ du miRNA sera définie par le « microprocessor » et inversement pour le bras 3’. Le miRNA guidant le miRISC sur ses ARNm cibles, un mauvais site de clivage de Drosha pourrait redéfinir un nouvel ensemble d’ARNm cibles par excision erronée de la tige-boucle. Cette étape nécessite ainsi un contrôle particulier mais aussi une régulation pour fournir in fine un miRNA mature correct. Il a été observé que les deux protéines du « microprocessor » sont capables de s’autoréguler pour permettre un contrôle précis de la maturation des miRNAs (Han et al., 2009). La liaison de DGCR8 à Drosha stabilise la protéine Drosha, et, Drosha peut détruire l’ARNm de DGCR8
en clivant deux structures en tige-boucle situées dans le 5’NTR et l’ORF permettant ainsi la mise en place d’une boucle d’autorégulation.
Plusieurs éléments suggèrent que les étapes de production et de maturation des pri-miRNAs sont coordonnées.
En effet, pendant la synthèse du pri-miRNA par l’ARN polymérase, Drosha est recrutée au niveau de la tige-boucle. L’excision provoquée par Drosha permet le recrutement d’exonucléases 5’-3’ et 3’-5’, entraînant la dégradation des séquences flanquantes, et, dans le cas d’un intron de pré-ARNm la dégradation de l’intron avant même son épissage (Kim and Kim, 2007; Morlando et al., 2008). Il est intéressant de noter qu’un pri-miRNA retenu à son site de transcription par défaut de maturation en 3’ (clivage et/ou polyadénylation) produit plus efficacement un pré-miRNA. Les exons, qui augmentent la rétention des pré-ARNm aux sites de transcription, contribuent également à augmenter la maturation des pré-miRNAs introniques (Pawlicki and Steitz, 2008). Plusieurs modes de recrutement du complexe de maturation peuvent être envisagés.
Le « microprocessor » pourrait interagir de façon directe ou indirecte avec l’extrémité C-terminale de l’ARN polymérase II, comme c’est le cas pour les protéines de maturation des pré-ARNm (pour revue (de Almeida and Carmo-Fonseca, 2008; Pandit et al., 2008)). L’ARN polymérase II, portant les protéines régulatrices, agit comme « transporteur de cargo » et permettrait le dépôt de ces régulateurs à l’endroit de leur fixation sur l’ARN naissant. De manière intéressante, plusieurs facteurs régulant et coordonnant la transcription et la maturation des pré-ARNm s’associent avec Drosha. En effet, en plus d’interagir avec DGCR8, Drosha est également capable de s’associer avec deux ARN hélicases (p68, p72) et plusieurs des protéines de liaison aux ARNs (RBP : RNA Binding protein) (TLS/FUS, EWS et TAF15) (Gregory et al., 2004). Les protéines p68, p72 peuvent augmenter la maturation de pri-miRNAs spécifiques (Fukuda et al., 2007), être recrutées au niveau de certains promoteurs (Bates et al., 2005; Watanabe et al., 2001)), interagir avec certains co-activateurs de la transcription et avec l’ARN polymérase II (Rossow and Janknecht, 2003). Les RBP
TLS/FUS, EWS et TAF15 sont connues pour s’associer à l’ARN polymérase II, réguler la
transcription et coordonner cette dernière à la maturation des pré-ARNm (Law et al., 2006). Ces facteurs, en interagissant avec Drosha, pourraient ainsi servir de lien entre la transcription et la maturation des pri-miRNAs (Pawlicki and Steitz, 2009). Enfin, les protéines oncogéniques issues des fusions de gènes All1/Af4(Af9) en s’associant à l’ADN permettraient





![Figure 9 ORF [ ] AAAA miRNA 1 8 13 16 région 5’ d’ancrage complémentarité 3’ 5’-P 3’-OH A [ ] AAAA miRNA 5’-P 3’-OH B [ ] 1 8 13 16 NNNNNNNNNN NNNNNNNNNN NNNNNNNNNNNNNNNNNNNNN NNNNNNNNNNNNNNNNNNNNN NNNNNNNA NNNNNNNN A U NAgo2 ARNm](https://thumb-eu.123doks.com/thumbv2/123doknet/2259335.19552/47.892.109.787.384.812/figure-complementarite-nnnnnnnnnn-nnnnnnnnnn-nnnnnnnnnnnnnnnnnnnnn-nnnnnnnnnnnnnnnnnnnnn-nnnnnnna-nnnnnnnn.webp)