HAL Id: hal-02810416
https://hal.inrae.fr/hal-02810416
Submitted on 6 Jun 2020HAL is a multi-disciplinary open access archive for the deposit and dissemination of sci-entific research documents, whether they are
pub-L’archive ouverte pluridisciplinaire HAL, est destinée au dépôt et à la diffusion de documents scientifiques de niveau recherche, publiés ou non,
de Botrytis cinerea dans l’air, variables climatiques
locales et mouvements des masses d’air
Ludovic Lovet
To cite this version:
Ludovic Lovet. Evaluer le lien statistique entre concentration de spores de Botrytis cinerea dans l’air, variables climatiques locales et mouvements des masses d’air. [Stage] Université de la Méditerranée (Aix Marseille 2), Marseille, FRA. 2011, 40 p. �hal-02810416�
Evaluer le lien statistique entre concentration de spores de Botrytis cinerea dans l’air, variables climatiques locales et mouvements des masses d’air de Lovet Ludovic est mis à
Ludovic LOVET
Licence Professionnelle Gestion et Traitement
Statistique de Bases de Données
Evaluer le lien statistique entre concentration
de spores de Botrytis cinerea dans l’air,
variables climatiques locales et mouvements
des masses d’air
Année universitaire 2010-2011
Maîtres de stage : Christel Leyronas
Samuel Soubeyrand
Unité de Pathologie Végétale, UR PV – 407,
Domaine Saint- Maurice, Avignon
Unité de Biostatistique et Processus Spatiaux, UR BioSP – 546,
Domaine Saint Paul, Avignon
REMERCIEMENTS
J’adresse mes remerciements au centre INRA PACA d’Avignon pour m’avoir permis d’effectuer mon stage au sein des unités de Pathologie Végétale et de Biostatistique et Processus Spatiaux. Je remercie plus particulièrement :
Madame Cindy Morris, directrice de l’unité de Pathologie Végétale et Monsieur Denis Allard, directeur de l’unité de Biostatistique et Processus Spatiaux pour m’avoir acceuilli dans leurs unités de recherche.
Je remercie également Madame Christel Leyronas et Monsieur Samuel Soubeyrand, mes maîtres de stage, pour m’avoir confié cette mission et fait confiance dans la réalisation de ce projet.
Enfin, je remercie l’ensemble du personnel des deux unités avec qui j’ai été amené à travailler, pour avoir fait preuve de disponibilité et d’attention à mon égard tout au long de mon stage et qui ont concouru à rendre ce passage en entreprise agréable.
Ce stage a été effectué dans la cadre des projets « Dispersion Groupée » et « Aérobiologie » financés par le département Santé des Plantes et Environnement de l’INRA.
SOMMAIRE
Introduction ________________________________________________________ 5
I. L’INRA __________________________________________________________ 6
1. L’Institut National de la Recherche Agronomique ____________________________________ 6 2. Le centre de recherche d’Avignon ________________________________________________ 6 3. L’unité de Pathologie Végétale ___________________________________________________ 6 4. L’unité de Biostatistique et Processus Spatiaux ______________________________________ 7
II. Matériel et méthodes _______________________________________________ 8
1. L’objet biologique étudié _______________________________________________________ 8 2. Les données _________________________________________________________________ 10 3. La base de données ___________________________________________________________ 12 4. Méthode statistique ___________________________________________________________ 14
III. Résultats _______________________________________________________ 20
IV. Analyse, synthèse ________________________________________________ 27
Conclusion ________________________________________________________ 29
Références bibliographiques __________________________________________ 30
INTRODUCTION
Du 4 Avril au 12 Août 2011, j’ai effectué un stage au sein de l’Institut National de la Recherche Agronomique situé à Avignon. Au cours de ce stage dans les unités de Pathologie Végétale et de Biostatistique et Processus Spatiaux, j’ai pu m’intéresser au domaine de la biostatistique.
Plus largement, ce stage a été l’opportunité pour moi d’appréhender comment mettre en pratique mes connaissances et mes savoirs dans le domaine professionnel.
Au-delà d’enrichir mes connaissances en statistique et en informatique, ce stage m’a permis de comprendre dans quelle mesure ces deux domaines peuvent être mis à la disposition du monde du travail.
Mon stage dans ces deux unités s’est axé sur deux disciplines, la statistique et les bases de données, pour tenter de résoudre un problème concernant le champignon Botrytis cinerea.
La quantité de spores de Botrytis cinerea dans l’air, à proximité de tunnels maraîchers, a été mesurée pendant 84 journées réparties sur 3 années sur le site d’Avignon (Vaucluse), et pendant 22 journées réparties sur 10 mois sur le site d’Alénya (Pyrénées Orientales).
Lors de ce stage, une base de données incluant ces données biologiques ainsi que des données climatiques locales et distantes a été créée, dans le but de déterminer les relations entre la présence de spores de B. cinerea et les paramètres climatiques des zones considérées.
L’élaboration de ce rapport a pour principale source les différents enseignements tirés de la pratique journalière des tâches auxquelles j’étais affecté. Enfin, les nombreux entretiens que j’ai pu avoir avec les agents des deux unités m’ont permis de donner une cohérence à ce rapport.
En vue de rendre compte de manière fidèle et analytique des 19 semaines passées au sein du centre INRA d’Avignon, il apparaît logique de présenter à titre préalable le cadre du stage : l’INRA. Ensuite, il sera précisé les différentes missions et tâches que j’ai pu effectuer au sein du centre de recherche.
I. L’INRA
1. L’Institut National de la Recherche Agronomique
C’est dans un contexte de pénurie alimentaire, suite à la seconde Guerre Mondiale, que L’Institut National de la Recherche agronomique (INRA) est fondé en 1946. Il acquiert son statut d’établissement public à caractère scientifique en 1984, et est aujourd’hui sous la double tutelle du ministère de l’Enseignement supérieur et de la recherche et du ministère de l’agriculture et de la pêche. Ses recherches sont tournées vers l’agriculture, l’alimentation et l’environnement, dans un contexte de développement durable. C’est grâce à ses nombreux contrats avec l’Etat, l’Europe, les collectivités territoriales, le secteur privé et les organismes de recherche que l’INRA finance ses projets. Il possède également des partenariats avec certaines universités. Il représente aujourd’hui le premier institut européen de recherche agronomique, le deuxième dans le monde. Il est divisé en 14 départements scientifiques, 21 centres de recherche régionaux et possèdent plus de 150 implantations.
2. Le centre de recherche d’Avignon
C’est en 1953 que le centre de recherche d’Avignon est fondé. Il compte actuellement 27 unités, dont celles de Pathologie Végétale et de Biostatistique et Processus Spatiaux, et est implanté sur 8 sites, principalement localisés en Région Provence-Alpes-Côte d’Azur mais également en Rhône Alpes et en Languedoc-Roussillon. Il est important de par sa taille et par ses thématiques de recherche.
Le centre d’Avignon s’organise autour de trois pôles de compétences : - Le pôle production horticole intégrée
- Le pôle adaptation au changement global - Le pôle santé des plantes
3. L’unité de Pathologie Végétale
L’unité de Pathologie Végétale étudie les maladies des cultures maraîchères méditerranéennes, la maladie affectant le platane (arbre régional) et les viroses des cultures florales. Cette unité est répartie en trois équipes de recherche : la virologie, la bactériologie et la mycologie. Cette dernière a pour objectif l’étude de l’épidémiologie de la pourriture grise de la tomate sous serre, avec la quantification du développement spatio-temporel des épidémies, l’étude de l’origine et de la dissémination de l’inoculum et la caractérisation de la diversité de l’évolution des populations de
Botrytis cinerea. L’équipe de mycologie travaille également sur la protection intégrée de la tomate
sous serre, avec le développement de méthodes de lutte biologique contre B. cinerea et l’étude de la durabilité de méthodes de lutte non chimique.
4. l’Unité de Biostatistique et Processus Spatiaux
L'unité de Biostatistique et Processus Spatiaux (BioSP) conduit des recherches en statistiques et en modélisation spatiales et spatio-temporelles, à la fois théoriques et appliquées, avec un intérêt particulier pour les applications relevant de l'environnement, de l'écologie, de l'épidémiologie et de la biologie des populations.
Dans ce cadre, la mission de l'unité est double :
Mener des recherches méthodologiques dans les domaines de la modélisation spatiale, pour leur intérêt propre, ou pour l'intérêt de leur application dans les champs de recherche de l'institut. En particulier, l'unité mène des recherches disciplinaires sur les champs aléatoires, les statistiques spatiales (géostatistique, processus ponctuels, champs de Markov), les modèles de dispersion et les équations aux dérivées partielles (équations de réaction/diffusion).
Participer avec les départements et les unités partenaires à des recherches pluridisciplinaires visant à améliorer la prise en compte de la dimension spatiale et/ou spatio-temporelle des phénomènes agronomiques, écologiques et biologiques.
II. MATERIEL ET METHODES
1. L’objet biologique étudié
Le sujet de mon travail au cours de ce stage fut axé sur la dissémination des spores de Botrytis
cinerea dans l’air.
Botrytis cinerea, agent de la pourriture grise, est un champignon polyphage et saprophyte,
c'est-à-dire qu'il se développe sur de nombreuses plantes (plus de 200 espèces) et peut vivre sur des tissus morts. Il attaque notamment des plantes d’intérêt agronomique comme la vigne, la tomate et la laitue entre autres.
Le développement de la maladie peut être explosif si les conditions météorologiques lui sont favorables, comme par exemple sur le raisin lorsque les baies sont réceptives.
Sur tomate, la maladie peut entrainer la mort des plantes et des taches sur fruits. La maladie peut rendre non commercialisables les laitues.
Ainsi, sur de nombreuses espèces, cultivées en plein champ ou sous abris, la pourriture grise engendre des dégâts quantitatifs (pertes de récolte) et qualitatifs (produits non commercialisables, altération des arômes et de la couleur pour le raisin).
Lutte contre la pourriture grise
La lutte chimique contre la pourriture grise est possible. Cependant, d’une part B. cinerea devient résistant à certains fongicides et d’autre part afin de faire évoluer l’agriculture vers une agriculture plus respectueuse de l’environnement il convient d’aller vers des pratiques de lutte moins consommatrices en pesticides. La protection intégrée des cultures est un pas dans ce sens.
La protection intégrée utilise différents moyens de lutte et ne se sert de produits chimiques que lorsqu’aucune autre solution n’est possible. Ces différents moyens sont par exemple la lutte biologique, en utilisant des moyens « naturels » de protection des cultures, ou encore en agissant sur les facteurs climatiques en serre, en adaptant les techniques culturales. Cette protection permet d’une part de limiter l’apparition de résistance aux produits chimiques, d’autre part de diminuer les doses de produits chimiques utilisés et permet donc de diminuer les résidus de produits sur les aliments (Decognet et al., 2000). Elle s’accorde avec la notion de lutte raisonnée, qui consiste à utiliser de manière réfléchie les produits chimiques et ne pas faire une application systématique et non adaptée.
Dans le cas de la pourriture grise, différents facteurs entrent en jeu pour la mise en place d’une protection intégrée. L’un de ces facteurs est l’inoculum primaire (propagules qui déclenche l’apparition de la maladie). En fonction de son origine, la protection devra être adaptée. Par exemple dans une serre, l’inoculum peut être d’origine intérieure (produit par les plantes à l’intérieur de la serre) ou extérieure (amené par le jeu des déplacements d’air avec une source proche ou distante de plusieurs kilomètres). Dans le premier cas on pourra être amené à mieux gérer le climat à l’intérieur de la serre ou traiter les plantes avec un fongicide. Dans le second cas il conviendra plutôt de gérer les ouvertures de la serre pour en protéger l’intérieur.
L’inoculum aérien de B. cinerea est généralement composé de spores.
Les spores de B. cinerea
B. cinerea peut produire 2 types de spores pouvant potentiellement constituer un inoculum aérien.
Les conidies sont des spores issues de la reproduction asexuée, portées par des structures arborescentes (conidiophores) présentes sur les lésions des plantes (chancres ou taches) (cf. annexe 1, figure 1 et 2). En conditions sèches, les conidiophores s’enroulent sur eux-mêmes et libèrent les conidies. Celles-ci sont alors disséminées dans l’air et dans l’eau et se déposent ensuite sur les plantes. Si les tissus végétaux sont blessés ou sénescents, la germination des conidies est facilitée. Un nouveau mycélium se développe, pénètre et colonise les tissus de l’hôte. Des conidiophores sont formés, produisant à leur tour des conidies. Il y a sporulation et dispersion des spores. Le champignon peut accomplir plusieurs fois son cycle de multiplication asexuée si les conditions climatiques lui sont favorables (Decognet et al., 2004). Les conditions climatiques favorables au développement de B. cinerea sont une humidité relative d’au moins 95% et une température comprise entre 17 et 23°C (Eden et al., 1996).
Dans des conditions défavorables, des organes de conservation de plus grande taille peuvent se former, les sclérotes, sortes de filaments entrelacés de forme cylindrique et de couleur noire. Ces sclérotes peuvent alors produire des spores issues de la reproduction sexuée : les ascospores. L’espèce cinerea est hétérothallique, c’est-à-dire que sa reproduction sexuée nécessite la rencontre de deux mycéliums ayant des polarités différentes (l’un +, l’autre -). Les sclérotes ne sont vraisemblablement pas responsables des dégâts causés aux plantes car la forme sexuée Botryotinia
fuckeliana n’a jamais été observée sous serre (Nicot et al., 2004).
Dissémination des conidies
Les conidies de B. cinerea peuvent être disséminées par la pluie, le vent, et les courants d’air. Cela dépend également de la température et de l’humidité. En effet, le déclin de l’humidité associé à l’augmentation de la température en début de matinée provoque la torsion et l’assèchement des conidiophores qui éjectent les conidies dans les courants d’air. La sporulation peut également se faire environ 48 heures après une période de pluie ou après une chute de l’hygrométrie d’au moins 15%. Les gouttes d’eau favorisent la dispersion des conidies mais ne sont pas le mode de transport le plus important. La dissémination peut encore se faire de proche en proche car plus les plants sont rapprochés les uns des autres, plus le risque de contamination est élevé (Williamson et al., 2007).
Problématique
Peut-on prédire le moment et les quantités de spores de Botrytis cinerea arrivant en un lieu donné de façon à pouvoir protéger les cultures à l’avance et ainsi éviter leur contamination ?
Pour aborder cette problématique, j’ai tenté de faire le lien entre données de concentrations de spores et données météorologiques.
2. Les données
Les concentrations de spores dans l’air
Les spores en suspension dans l’air ont été collectées à l’aide d’échantillonneurs d’air. L’un était placé sur le site de l’INRA d’Avignon (lat. 43.95, long. 4.81), unité de Pathologie Végétale responsable des échantillonnages, et l’autre sur le site de l’unité expérimentale INRA d’Alénya (lat. 42.63, long. 2.98).
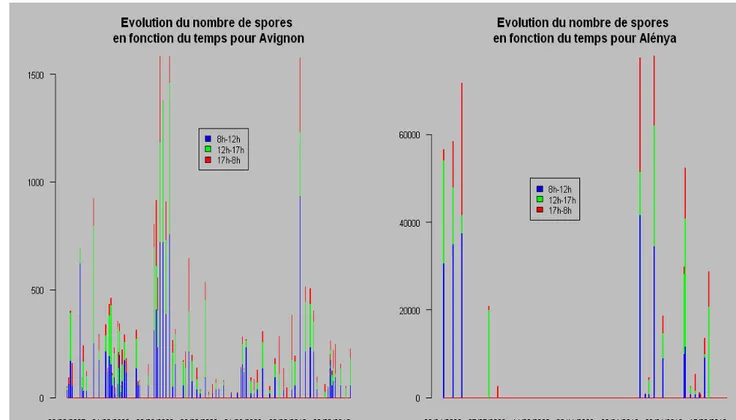
Pour Avignon l’échantillonnage a été réalisé pendant 84 périodes de 24h réparties entre le 20 Septembre 2007 et le 6 Décembre 2010 et pour Alénya pendant 22 jours répartis entre le 29 Avril 2009 et le 13 Juillet 2010 (cf. figure 1).
Les journées de collecte ont été découpées en tranches horaires pour voir s’il y avait une influence du moment de la journée sur le nombre de spores récoltées.
Pour Alénya le découpage était 8h/14h, 14h/20h, 20h/8h et pour Avignon 8h/12h, 12h/17h, 17h/8h.
L’air, ainsi que les particules en suspension dans l’air, ont été aspirés dans les échantillonneurs d’air (débit 500L/min à Avignon et 20L/min à Alénya) puis les particules ont été déposées sur un milieu de culture semi-sélectif permettant de mettre en évidence les spores viables de B. cinerea. Les boites de Petri contenant le milieu et les particules collectées on été mises à incuber en laboratoire. Au bout de deux semaines les colonies de B. cinerea qui se sont développées ont été comptées. Le nombre de spores viables de B. cinerea par mètre cube d’air a été déduit du nombre de colonies, du débit d’aspiration et du temps de collecte.
Données météorologiques de la base de données « Climatik »
Les données météorologiques ont été collectées sur la plateforme « Climatik » pour les dates où des prélèvements avaient été effectués, pour les deux sites de prélèvements.
La plateforme « Climatik » est une plateforme en ligne gérée par l’INRA, qui permet de récupérer différentes variables météorologiques, pour différentes stations météorologiques existantes (47 sites dans l’hexagone et 4 sites dans les DOM-TOM).
Les variables considérées pour les 2 sites sont :
le rayonnement global journalier (joules/cm²), la hauteur de précipitations journalière (millimètres), la température moyenne (degrés Celsius), la température minimale (degrés Celsius), la température maximale (degrés Celsius), l’humidité moyenne (%), l’humidité minimale (%), l’humidité maximale (%), la vitesse moyenne du vent (m/s), la vitesse maximale du vent (m/s).
Données concernant les masses d’air :
Concernant les données des masses d’air je me suis servi de la plateforme en ligne de l’ARL, Air Ressources Laboratory, où j’ai utilisé le modèle Hysplit (Hybrid Single Particule Lagrangian Integrated Trajectory Model), qui permet d’avoir accès à des données météorologiques des rétro-trajectoires des masses d’air. Ainsi on peut savoir d’où provient la masse d’air qui est arrivée à l’endroit et au moment où l’on a échantillonné, et quel parcours elle a emprunté. En plus de la trajectoire de la masse d'air, Hysplit fournit des caractéristiques météorologiques de la masse d'air. Hysplit peut être utilisé pour la reconstruction des trajectoires passées (en se fondant sur la base de données GDAS) ou pour la prévision des trajectoires futures (en se fondant sur la base de données GFS). L’objectif étant l'utilisation des rétro-trajectoires des masses d’air, j’ai choisi le modèle se fondant sur GDAS.
Hysplit fournit une rétro-trajectoire pour chaque altitude d'arrivée de la masse d'air. Pour effectuer de longues distances les particules en suspension dans l’air doivent voyager à des altitudes où les masses d’air ne sont pas trop gênées par le relief. J’ai ainsi opté pour considérer les rétro-trajectoires des masses d'air arrivant aux altitudes 500m, 1500m et 2500m.
J’ai récupérer les données pour chaque date de prélèvement sur les deux sites (Avignon et Alénya). Pour chaque date de prélèvement, il fallait récupérer les données en faisant partir la masse d’air à 3 heures différentes : 11h, 15h, 00h. Chacune de ces heures se trouve dans un intervalle de prélèvement (8h/14h, 14h/20h, 20h/8h pour Alénya, 8h/12h, 12h/17h, 17h/8h pour Avignon). J’ai exécuté les rétro-trajectoires sur 96 heures.
Les variables fournies par Hysplit sont la date, l’heure, la latitude, la longitude, l’altitude (mètres), la pression atmosphérique (Hectopascal), la température (degrés Celsius), la hauteur des précipitations (millimètres), l’humidité (%) et le rayonnement solaire (joules/cm²).
3. La base de données
Une fois les données récoltées, une des missions qui m’a été confiée était de créer une base de données relationnelle pour pouvoir les stocker.
Le système de gestion de bases de données utilisé est PostgreSQL 9.0, car c’est un logiciel libre et qu’il possède une API (Application Programming Interface) ODBC (Open Database Connectivity) permettant à n'importe quelle application supportant ce type d'interface d'accéder à des bases de données de type PostgreSQL, en l’occurrence le logiciel de statistique R. J’ai aussi utilisé pgAdmin III qui est un outil d’administration graphique de bases de données PostgreSQL.
Pour pouvoir envoyer les données collectées dans la base de données, je me suis servi du logiciel de statistique R. En effet, j’ai utilisé un package R nommé RpgSQL qui permet de faire la connexion entre le logiciel R et une base de données PostgreSQL locale ou distante pour lancer des requêtes SQL et ainsi pouvoir créer la base de données.
Avant de me lancer dans la création de la base de données, j’ai construit un modèle relationnel, pour modéliser les différentes relations existantes entre les données de la base (cf. annexe 2).
Pour créer la base de données j’ai utilisé du langage SQL dans le logiciel R, grâce à des commandes spécifiques au package.
Pour créer les tables de la base de données, je n’ai pas créé les différentes tables à l’aide de requêtes SQL, mais j’ai directement importé dans PostgreSQL les tableaux de données que j’avais récupérés.
Cependant, je n’ai pas directement importé ces fichiers tels quels, j’ai préalablement fait un travail de rangement sur ces données, pour pouvoir faire un travail statistique par la suite sur celles-ci.
Pour les données météorologiques de la base « Climatik » (cf. annexe 3), il a fallu faire quelques modifications sur les jeux de données. J’ai rajouté pour chaque date où l’on a fait des prélèvements, les variables météorologiques concernant les 9 jours précédents cette date. C’est-à-dire que pour un jour de prélèvement j, j’ai rajouté les données météorologiques de j-1 jusqu'à j-9. Bien sûr, la quantité de spores pour ces jours est celle du jour de prélèvement.
J’ai aussi décidé de rajouter 2 champs date, un correspondant à la date d’arrivée de la masse d’air c’est-à-dire la date du jour j, et un autre correspondant à la date du jour j-i pour i allant de 1 à 9. De plus j’ai ajouté une colonne identifiant, qui est un identifiant unique pour chaque ligne de données.
En ce qui concerne les données des masses d’air de « Hysplit » (cf. annexe 4), j’ai ajouté : - un identifiant unique pour chaque ligne de données
- une colonne référence altitude correspondant à l’altitude d’arrivée de la masse d’air ( 1 500m, 2 1500m, 3 2500m)
- 2 champs de dates comme pour les données de « Climatik », c'est-à-dire un champ date d’arrivée qui correspond à la date d’arrivée de la masse d’air et un autre correspondant à la
date du jour j-i, i allant de 1 à 4, puisque l’on effectue les rétro-trajectoires sur 96 heures.
- un champ heure d’arrivée qui correspond aux trois heures d’arrivée des masses d’air (11h, 15h, 00h)
- un champ heure qui correspond à chaque heure de relevé de mesure au cours du déplacement de la masse d’air
- un champ h allant de 0 à 96 pour chaque heure d’arrivée vu que les rétro-trajectoires étaient effectuées sur 96 heures
Une fois les données correctement ordonnées, j’ai pu les importer dans la base de données.
Pour cela, une fois la connexion avec la base établie, j’ai importé mes fichiers au format CSV dans la base grâce à une commande spécifique, en indiquant le nom que je voulais donner à la table, ce qui fait que les tables étaient directement créées.
Une fois tous les fichiers importés, il ne me restait plus qu’à faire des changements de types de variables et d’ajouter les clés primaires et étrangères.
Quand tout cela fut exécuté, j’ai testé différentes requêtes SQL de consultation de la base pour voir s’il n’y avait pas de problème particulier.
4. Méthode statistique
Vers une approche basée sur un GLM
L’objectif du stage est de construire un outil statistique permettant d’explorer le lien entre les concentrations de spores de Botrytis cinerea dans l’air, les variables climatiques locales, les rétro-trajectoires et leurs caractéristiques climatiques.
Un modèle général de la forme suivante pourrait être construit :
Y(x,t) = F({ z(x,h,t-s) : h>0, s>0 } , v(x,t))
où Y(x,t) représente la concentration de Botrytis en x (Avignon ou Alénya) au temps t ; z(x,h,t-s)
représente les coordonnées au temps t-s de la masse d’air située, au temps t, aux coordonnées x et à la hauteur h ; v(x,t) représente les variables climatiques en x au temps t.
Cependant, construire un tel modèle s’avère complexe. Par exemple, dans la fonction F, chaque z(x, h, t-s) doit être modulé par la force de la source en z(x, h, t-s), par les conditions climatiques associées à la masse d'air entre t-s et t.
Le modèle est difficile à construire car il y a beaucoup de processus et de connaissances à intégrer, et donc non adapté à une analyse exploratoire (préliminaire) du lien statistique entre concentration de Botrytis (Y), rétro-trajectoires (z) et variables climatiques locales (v).
L'approche choisie pour construire un outil d'analyse du lien statistique entre concentration de Botrytis, rétro-trajectoires et variables climatiques a consisté
- à utiliser un modèle linéaire généralisé (GLM) plutôt qu'un modèle mécaniste et
- à réduire l'information contenue dans les données de masses d'air (en considérant des
données d'angles de provenances et en agrégeant les données à travers des moyennes temporelles par exemple).
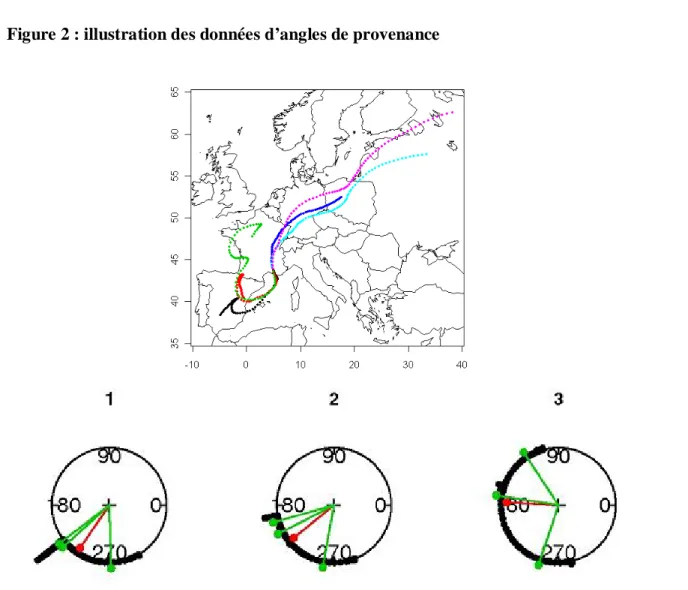
Figure 2 : illustration des données d’angles de provenance
Cette figure montre comment sont résumées les données angulaires pour chaque trajectoire de masse d’air.
Le cercle trigonométrique numéro 1 représente la trajectoire noire, le numéro 2 la trajectoire rouge et le numéro 3 la trajectoire verte.
Sur chaque cercle, le point rouge représente l’angle moyen de la trajectoire.
Ainsi j’obtiens des variables résumées du genre : angle moyen de la trajectoire arrivée en x à la hauteur h et au temps t, angle moyen d’une trajectoire sur les dernières 24 heures, sur j-1, sur j-2. Ce seront les variables explicatives.
Variable réponse et variables explicatives
Le but est d’essayer de prédire la concentration de spores de Botrytis cinerea en fonction des différentes variables météorologiques.
La variable réponse est donc la concentration de spores de Botrytis cinerea dans l’air. Afin d'homogénéiser les mesures, j'ai ramené toutes les mesures à un nombre de spores pour 1000m3 d'air.
Les variables explicatives sont les données météorologiques de Climatik et les données concernant les masses d’air issues de Hysplit.
Du fait principalement de l'utilisation d'Hysplit, le nombre de variables explicatives possibles est très grand (de l'ordre du millier). De plus, les données Hysplit qui sont des données horaires contiennent de fortes dépendances. Afin de réduire le nombre de variables explicatives et les dépendances, j’ai construit des variables agrégées temporellement (ex: moyenne journalière). Cette procédure pouvant être menée de manières différentes, j'ai formé plusieurs jeux de variables explicatives et évalué le pouvoir explicatif de chacun de ces jeux.
1er jeu de variables explicatives (table très agrégée, 21 variables) : les moyennes sur 4 jours des variables de « Hysplit » triées par date de prélèvement et heure d’arrivée des masses d’air ; les moyennes journalières des données de Climatik correspondant aux jours de prélèvement de spores (cf. annexe 5).
2ème jeu de variables explicatives (table avec interactions, 274 variables) : Le même type que la table très agrégée mais en ayant ajouté les interactions, c'est-à-dire que l’on a multiplié les variables deux à deux (cf. annexe 6).
3ème jeu de variables explicatives (table peu agrégée, 199 variables) : Les moyennes journalières des variables de « Hysplit » triées par référence altitude et tranches horaires ainsi que les moyennes journalières des variables de « Climatik » pour les jours de prélèvement de spores et les 9 jours précédant le prélèvement. Ces données sont aussi rangées par date de prélèvement et heure d’arrivée des masses d’air (cf. annexe 7).
Modèles Modèle poissonnien :
La variable explicative (nombre de spores dans 1000m3) étant vue comme une variable de comptage, j'ai essayé d'ajuster aux données un GLM avec la loi de Poisson :
Y | X ~ Poisson (exp (β’X))
où β est un vecteur de paramètres et X est une matrice de variables explicatives. Sous ce modèle,
E (Y | X) = Var (Y | X) = exp (β’X)
Cependant, je me suis aperçu que le nombre de spores était fortement surdispersé (Var (Y | X) >> E (Y | X)), quelque soit la matrice de variables explicatives utilisée.
La présence de surdispersion dans le modèle poissonnien conduit à envisager différentes extensions de ce modèle, comme les modèles négatif-binomial et quasi-poissonnien qui sont des modèles surdispersés.
Modèle négatif-binomial :
J'ai essayé d'ajuster aux données un GLM avec la loi Négative-Binomiale (McCullagh and Nelder, 1989, chap. 6) :
Y | X ~ Négative-Binomiale (exp (β’X) , θ)
sous lequel θ > 1 et
E (Y | X) = exp (β’X) = µ
Var (Y | X) = exp (β’X) * θ = µθ.
Dans ce modèle, une variable latente Z suit une loi Gamma de moyenne µ=exp (β’X) et de variance µ*(θ-1) ; et, sachant Z, Y suit une loi de Poisson de moyenne Z.
Modèle quasi-poissonnien :
Le modèle négatif-binomial, bien qu'offrant plus de flexibilité que le modèle poissonnien, ne permet de représenter qu'une forme spécifique de surdispersion. Une représentation plus flexible des variables surdispersées a été proposée: le modèle quasi-poisson. Sous ce modèle, comme pour le modèle négatif-binomial,
E (Y | X) = exp (β’X) = µ
mais contrairement au modèle négatif-binomial, β est estimé comme sous le modèle poissonnien (maximum de quasi-vraisemblance) et θ est estimé par la méthode des moments (cf. McCullagh and Nelder, 1989, chap. 4 et 9). La différence avec le modèle poissonnien est que les tests faits sur β (tests de significativités) sont corrigés par l'estimation de θ.
Sélection de variables explicatives
Etant donné le nombre important de variables explicatives présentes dans les jeux de données, il me fallait déterminer quelles étaient les variables explicatives que je devais choisir pour expliquer au mieux les données. Pour cela j’ai utilisé une méthode de sélection de variables.
La sélection de variables est un processus très important qui permet de chercher les variables les plus pertinentes pour expliquer et prédire les valeurs prises par la variable à prédire. Cette technique permet de réduire le nombre de variables à recueillir et souvent elle améliore la qualité de la prédiction.
Pour effectuer la sélection de variables, plusieurs méthodes ont été envisagées (Burnham and Anderson, 2002; cours de l'ISPED (http://campus.isped.ubordeaux2.fr/PLEIADE/PV3/ASPX/ PV2_VPS.aspx?3792)) :
Méthode d'élimination progressive ou « backward selection »:
La procédure démarre en estimant les paramètres du modèle complet incluant toutes les variables explicatives que l'on a sélectionnées et jugées pertinentes à introduire. A chaque étape, la variable associée à la plus grande p-value (du test de F partiel ou de Student) est éliminée du modèle, si cette valeur est supérieure au seuil fixé a priori (en général 10% ou 5%). La procédure s'arrête lorsque les variables restant dans le modèle ont toutes une p-value plus petite que le seuil.
Méthode d'introduction progressive ou « forward selection »:
Il faut choisir au départ les variables que l'on juge comme pouvant appartenir au modèle. A chaque étape de la procédure, une variable est ajoutée en commençant par la variable la plus fortement associée à Y (plus petite p-value obtenue en réalisant l'ensemble des modèles de régression linéaire simple). Ensuite, on évalue l'apport spécifique de chacune des variables non encore introduites dans le modèle qui contient déjà la ou les variable(s) retenue(s) dans les étapes précédentes et on introduit la variable dont l'apport spécifique est le plus important. L'introduction d'une nouvelle variable dans le modèle ne se fait que si la p-value correspondante est inférieure à un seuil fixé a priori (en général 10% ou 5%). La procédure s'arrête lorsque toutes les variables sont introduites ou lorsqu'on ne peut plus introduire de nouvelles variables selon le critère choisi (plus petite p-value des variables restantes supérieure au seuil).
Méthode de régression pas à pas ou « stepwise regression » :
Il s'agit d'une amélioration de la méthode d'introduction progressive. A chaque étape de la procédure, on examine à la fois si une nouvelle variable doit être ajoutée selon un seuil d'entrée fixé, et si une des variables déjà incluses doit être éliminée selon un seuil de sortie fixé. Cette méthode permet de retirer du modèle d'éventuelles variables qui seraient devenues moins indispensables du fait de la présence de celles nouvellement introduites. La procédure s'arrête lorsqu’aucune variable ne peut être rajoutée ou retirée du modèle selon les critères choisis.
La méthode « stepwise » a conduit à des erreurs de prédiction moindres (cf. les explications ci-dessous). Les résultats présentés ci-dessous ont été obtenus avec cette méthode.
Critère utilisé pour la procédure « stepwise »
Il est d'usage d'utiliser l’AIC (Akaike Information Criterion ; Burnham and Anderson, 2002) qui est un indicateur polyvalent qui permet d’évaluer la bonne adéquation d’un modèle et surtout de comparer plusieurs modèles entre eux.
Ce critère permet par exemple d’évaluer des régressions multiples (à l’instar du R² ajusté), des prévisions sur séries chronologiques ou encore des régressions logistiques.
L’AIC utilise le maximum de vraisemblance et pénalise les modèles comportant trop de paramètres:
AIC = -2 ln L(θ) + 2k
où L est la vraisemblance du modèle et k est le nombre de paramètres.
L'AIC ne peut pas être utilisé pour le modèle quasi-poisson puisqu'on ne peut pas écrire de vraisemblance pour ce modèle. J’ai essayé d'utiliser le QAIC (quasi-AIC, Burnham and Anderson, 2002) mais les résultats n'étaient pas satisfaisants.
Puisque notre objectif est de prédire les concentrations de B. cinerea, j’ai opté pour un critère de prédiction calculé par validation croisée.
Soit Yij le nombre de spores obtenu le jour i durant la tranche horaire j.
Soit pred(Yij) le nombre prédit de spores pour le jour i durant la tranche horaire j par le GLM ajusté
à toutes les données sauf celles du jour i.
Le critère de prédiction que j'ai cherché à minimiser est :
(1/N) Σij {Yij - pred(Yij)}²
où N est le nombre de données total. Le terme validation croisée se réfère au fait que l’on n’utilise pas les données du jour i pour les prédire.
III. RESULTATS
Sélection des variables explicatives
J'ai appliqué la procédure de sélection stepwise avec le modèle quasi-poisson en prenant comme variable réponse la concentration de spores à Avignon ou Alénya et comme variables explicatives possibles celles issues de la table très agrégée, de la table avec interactions ou de la table peu agrégée. Dans chacun des cas, la procédure stepwise a permis de sélectionner un certain nombre de variables explicatives et a abouti à une valeur plus ou moins grande du critère de prédiction (cf. Tableau 1). On peut voir avec le tableau 1 que le critère de prédiction est le plus faible pour les données de la table peu agrégée, que ce soit pour Avignon ou pour Alénya. Cela montre que la prédiction est meilleure lorsque l'on sélectionne les variables explicatives dans la table peu agrégée (199 variables).
Tableau 1 : Valeurs du critère de prédiction par validation croisée en fonction du type de données.
Localité Table très agrégée Table avec interactions Table peu agrégée
Avignon 17.8 x 103 11.7 x 103 9.2 x 103
Alénya 109 x 106 55 x 106 32 x 106
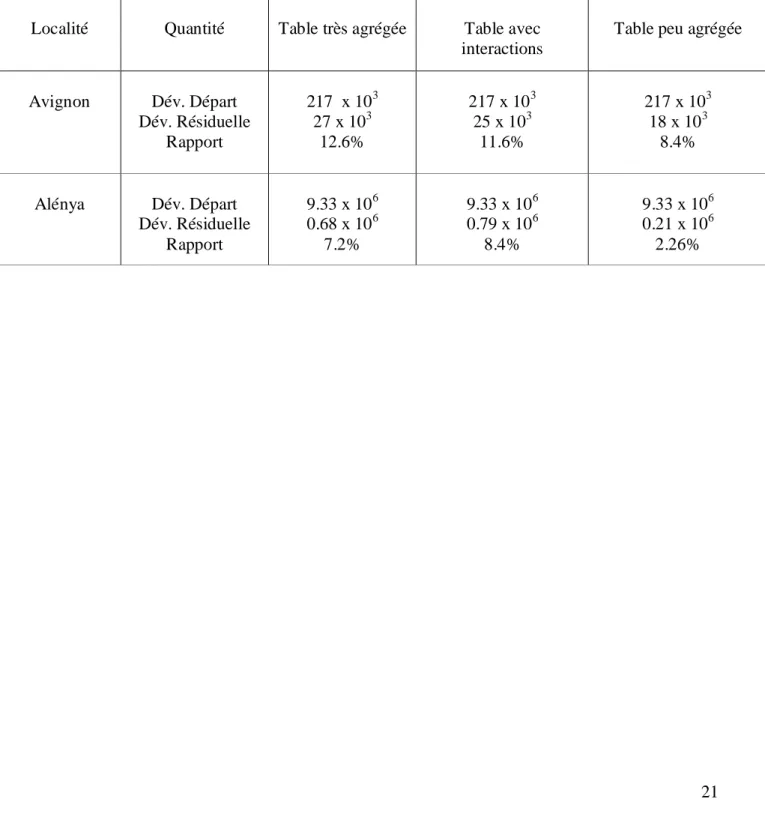
L'intérêt d'utiliser la table peu agrégée pour sélectionner les variables explicatives est confirmé par les rapports entre déviances résiduelles et déviances nulles calculées pour chacun des modèles sélectionnés (cf. Tableau 2) et par les graphiques de la figure 3 montrant les valeurs prédites des concentrations de spores en fonction des concentrations observées.
Tableau 2 : Valeurs des déviances nulles et résiduelles et valeurs du rapport des deux déviances en fonction du type de données. Déviance nulle : Σij 2*{Yij log(Yij/m)-Yij+m} où m
est la moyenne empirique des Yij. Déviance résiduelle : Σij 2*{Yij log(Yij/pred(Yij
))-Yij+pred(Yij)} où pred(Yij) est la valeur prédite par le modèle de Yij.
Localité Quantité Table très agrégée Table avec
interactions
Table peu agrégée
Avignon Dév. Départ Dév. Résiduelle Rapport 217 x 103 27 x 103 12.6% 217 x 103 25 x 103 11.6% 217 x 103 18 x 103 8.4% Alénya Dév. Départ Dév. Résiduelle Rapport 9.33 x 106 0.68 x 106 7.2% 9.33 x 106 0.79 x 106 8.4% 9.33 x 106 0.21 x 106 2.26%
Analyse des sorties des GLM
Dans ce paragraphe, je présente les résultats obtenus après sélection des variables explicatives dans la table peu agrégée qui mène à la meilleure prédiction (au sens du critère choisi).
Les sorties des GLM pour Avignon et Alénya sont données dans les Tableaux 3 et 4. Pour Avignon, des variables fournies par Hysplit et Climatik ont été sélectionnés tandis que pour Alénya seulement des variables fournies par Hysplit l'ont été (l'annexe 8 fournit les significations des variables). Ainsi, dans chacun des cas, il semble qu'il y ait une information contenue dans les caractéristiques de la masse d'air qui s'est déplacée à haute altitude (au delà de 500m). Ce résultat incite à rejeter l'hypothèse selon laquelle les spores produites localement contribuent presque exclusivement aux concentrations de spores mesurées.
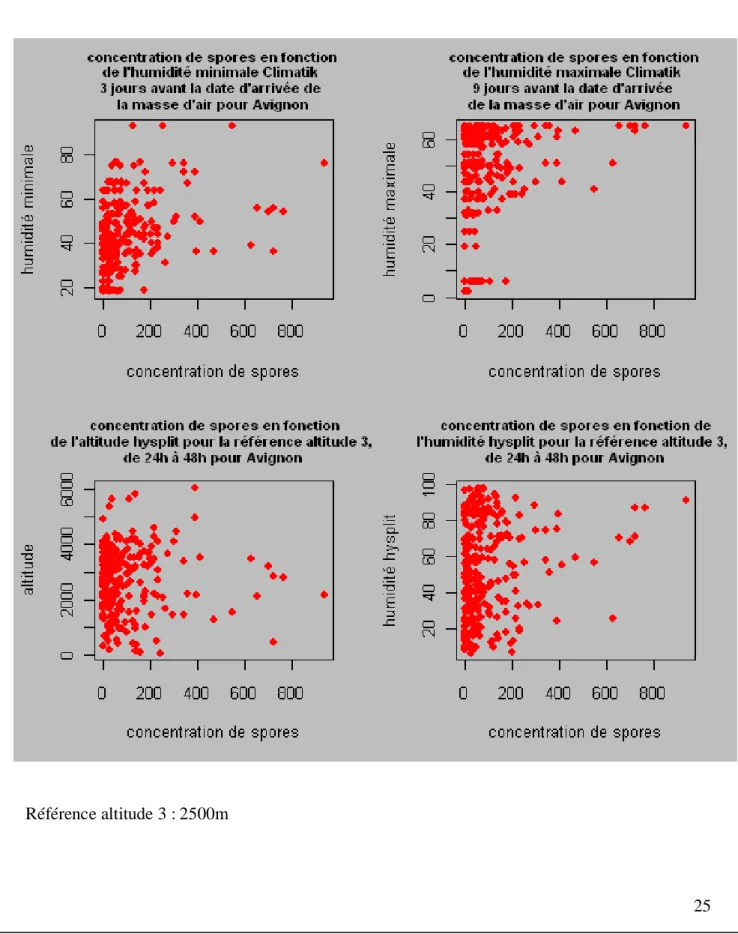
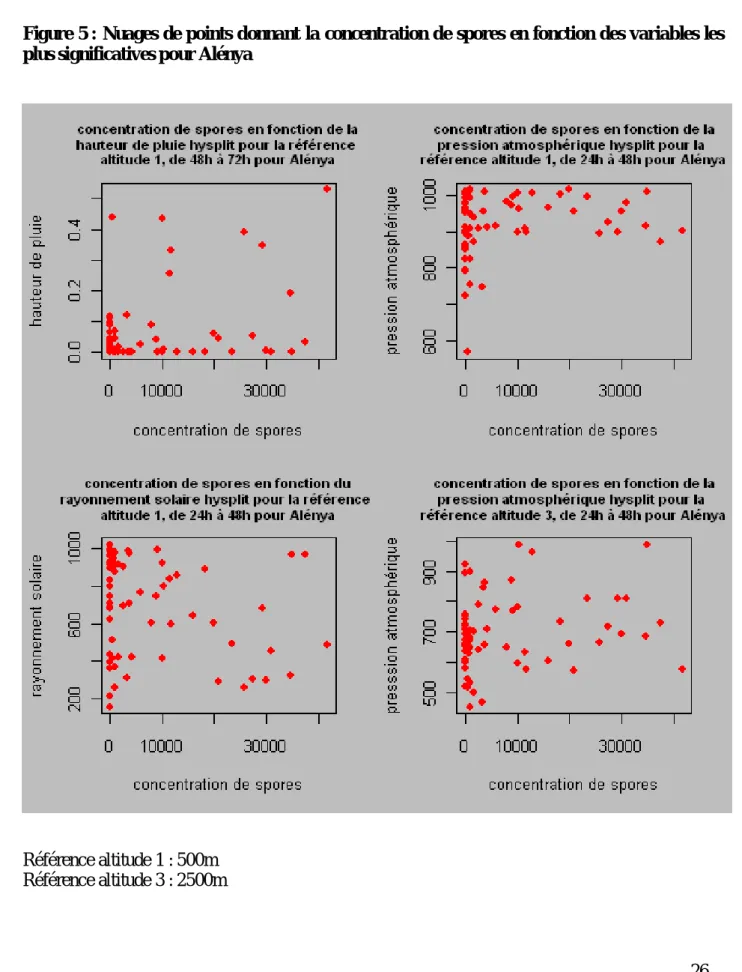
Dans les deux sorties de GLM, on voit que seulement une partie des variables explicatives ont un effet significatif (les p-valeurs fournies sont corrigées dans le cadre du modèle quasi-poissonnien en tenant compte de la surdispersion des données). Ceci est dû au fait que les variables ont été sélectionnées en fonction de leurs aptitudes à prédire les concentrations de spores et pas en fonction de la significativité de leurs effets. J'ai représenté pour Avignon et Alénya les nuages de points donnant la concentration de spores en fonction des variables les plus significatives (cf. figures 4 et 5).
Les variables qui semblent être les plus annonciatrices d’arrivée de spores sont pour Avignon : - l’humidité minimale climatik (données climatiques locales) 3 jours avant la date de
prélèvement qui semble avoir un effet positif
- l’humidité maximale climatik 9 jours avant la date de prélèvement qui semble avoir un effet positif
- l’altitude hysplit (données météorologiques lointaines) pour la référence altitude 3 (2500m), de 24h à 48h avant la date de prélèvement qui semble ne pas avoir d’effet visible - l’humidité hysplit pour la référence altitude 3 (2500m), de 24h à 48h avant la date de prélèvement qui semble avoir un effet positif
et pour Alénya :
- la hauteur de pluie hysplit pour la référence altitude 1 (500m), de 48h à 72h avant la date de prélèvement qui semble avoir un effet positif
- la pression atmosphérique hysplit pour la référence altitude 1 (500m), de 24h à 48h avant la date de prélèvement qui semble avoir un effet positif
- le rayonnement solaire hysplit pour la référence altitude 1 (500m), de 24h à 48h avant la date de prélèvement qui semble ne pas avoir d’effet visible
- la pression atmosphérique hysplit pour la référence altitude 3 (2500m), de 24h à 48h avant la date de prélèvement qui semble ne pas avoir d’effet visible
Tableaux 3 et 4 : sorties des GLM. Ces tableaux représentent les sorties des GLM pour Avignon (tableau 3) et Alénya (tableau 4). Les variables les plus significatives sont celles qui possèdent 3 étoiles sur la droite.
Tableau 3 Tableau 4
Les colonnes importantes de ces 2 tableaux sont les colonnes « Pr(>|t|) » qui est la p-valeur de chaque variable et qui correspond à la significativité de chaque variable, et la colonne « Estimate » qui correspond au coefficient estimé de chaque variable.
Ainsi le lien positif ou négatif de chaque variable par rapport au nombre de spores de Botrytis
cinerea peut être observé.
Cependant il faut noter que ce lien se caractérise par des combinaisons de variables explicatives et non pas par chaque variable, ce qui fait que l’on peut avoir du mal à observer ce lien.
Figure 4 : Nuages de points donnant la concentration de spores en fonction des variables les plus significatives pour Avignon
Figure 5 : Nuages de points donnant la concentration de spores en fonction des variables les plus significatives pour Alénya
Référence altitude 1 : 500m Référence altitude 3 : 2500m
IV. Analyse , synthèse
Au cours de ce stage j’ai beaucoup appris. Les apports que j’ai tiré de cette expérience professionnelle peuvent être regroupés selon trois axes: les apports scientifiques, les apports sur le plan personnel et les apports sur le plan relationnel.
Sur le plan scientifique
Durant ce stage, j’ai pu mettre en place une base de données qui pourra être utilisée par mes encadrants pour la suite de l'étude.
J’ai aussi réalisé les premières analyses statistiques multivariées des données (des analyses univariées ont été réalisées par le passé). Ces analyses sont un point de départ pour la suite du travail.
Dans les données originales, on a des nombres de spores collectées par des échantillonneurs d'air. On a donc une variable de comptage. Mais ces données de comptage sont hétérogènes du fait de l'hétérogénéité des durées de collecte et de l'hétérogénéité des débits des échantillonneurs d'air. L'approche choisie durant mon stage afin de simplifier les analyses a consisté à ramener toutes les mesures à un nombre de spores pour 1000m3. Dans le futur, la variable réponse du GLM sera le nombre réellement observé de spores et la moyenne de ce nombre sera proportionnelle à la durée de collecte et au débit de l'échantillonneur d'air. Bien que cette option de modélisation soit plus rigoureuse, nous ne nous attendons pas à des résultats très différents.
De plus, la capacité de prédiction du GLM qui a été construit est relativement grande (cf. Tableau 1). Nous pouvons donc espérer dans le futur prédire, avec une incertitude assez réduite, les concentrations de spores en utilisant des prévisions des variables climatiques. Ceci dit, la prédiction dépendra alors de la qualité des prévisions climatiques locales et des prévisions climatiques des masses d'air.
Sur le plan personnel
Ce stage a été pour moi l’opportunité de pouvoir mettre en œuvre mes connaissances dans un domaine professionnel, ce que l’on ne perçoit pas totalement avec la théorie en classe.
Cela m’a permis de comprendre comment tout ce que j’ai appris pendant cette année pouvait être appliqué à un domaine particulier, celui de la biostatistique.
Pendant ce stage, j’ai appris à gérer des tâches que l’on m’avait confiées, tant au niveau de la gestion du contenu que de la gestion du temps. Cela m’a permis de me responsabiliser et de me mettre en situation dans l’entreprise comme un vrai salarié.
Je me suis aussi fait confiance dans mes choix et mes idées pour mener à bien mon stage.
Cette période m’a permis de découvrir un secteur d’activité professionnel, celui de la recherche, et plus particulièrement de découvrir la discipline de la biologie végétale.
Ce stage m’a permis de voir comment je pouvais mener à bien un travail en autonomie, et comment je pouvais m’adapter au monde du travail.
Sur le plan relationnel
Mon stage au sein du centre INRA d’Avignon a été très instructif. Au cours de ces 19 semaines, j’ai ainsi pu observer le fonctionnement d’une unité de recherche. Par ailleurs, les relations humaines entre les différents employés de l’unité, indépendamment de l’activité exercée par chacun d’eux, m’a appris sur le comportement à avoir en toute circonstance.
Au-delà du fonctionnement de l’unité, j’ai pu ressentir une vraie cohésion entre les salariés.
En effet, l’atmosphère au sein de l’unité était très agréable. J’ai ainsi constaté que la hiérarchie des fonctions était peu marquée dans les rapports entre les employés, favorisant par là la bonne entente entre les différents salariés.
Au travers de cette convivialité, j’ai pu comprendre que l’activité d’une société est plus performante dans une atmosphère chaleureuse et bienveillante.
La circulation de l’information est ainsi un des points forts que j’ai retenu de cette unité, tant au niveau du travail collaboratif, que dans l’implication de tous dans le bon fonctionnement de l’unité.
CONCLUSION
Ce stage de 19 semaines au sein du centre de recherche INRA PACA à Avignon a été une expérience très enrichissante pour ma connaissance du monde des statistiques et du monde de la biologie végétale qui était nouveau pour moi.
J’ai pu réinvestir et approfondir mes connaissances et compétences dans l’analyse de données. Tout au long de ce stage, la communication avec le personnel des deux unités, ainsi que l’analyse du projet ont été des composantes essentielles pour le bon déroulement du stage. Cela a permis de bien comprendre les besoins, d’être plus réactif, et de mieux répondre aux attentes.
Les difficultés majeures de ce stage ont été la mise en forme et la récupération des données, car ce sont ces tâches qui m’ont pris le plus de temps.
Au terme de ce stage, j’ai eu la satisfaction d’avoir réalisé une analyse de données statistiques sur un problème d’actualité. Nous avons réussi à faire ressortir quelques variables qui semblent être corrélées à l’arrivée de spores de B. cinerea dans l’air pour les sites d’Avignon et d’Alénya. De plus, les résultats encouragent à rejeter l’hypothèse selon laquelle les spores produites localement contribuent presque exclusivement aux concentrations de spores mesurées. On peut donc penser qu’il reste à creuser les relations entre les variables explicatives pour expliquer le mieux possible la quantité de spores récoltée, puis déterminer comment ces variables pourraient être intégrées dans un modèle prédictif accessible à la Profession (agriculture, recherche).
Mais plus que cette satisfaction, j’ai eu le plaisir de travailler dans un domaine pour lequel je manifeste un réel intérêt.
En effet, ce stage m’a permis non seulement d’approfondir mes connaissances en statistique et en informatique mais aussi d’acquérir une expérience extrêmement valorisante d’un point de vue personnel.
Dans la mesure où il reflète parfaitement le domaine dans lequel j’aimerais exercer ma profession, j’estime être heureux d’avoir pu effectuer ce stage entouré de personnes compétentes qui ont su me guider dans mes démarches tout en me laissant une certaine autonomie.
Par la suite, cette étude que j’ai commencé servira de point de départ à l’équipe de l’unité de Biostatistique et Processus Spatiaux qui approfondira la modélisation du problème, et ainsi, on l’espère, permettra de pouvoir prévoir de l’arrivée du champignon Botrytis cinerea, et donc d’adopter une gestion des cultures sensibles afin d’éviter le développement d’épidémies de pourriture grise.
REFERENCES BIBLIOGRAPHIQUES
Burnham, K. P. & D. R., Anderson, 2002. Model selection and multimodel inference: a practical
information-theoretic approach. 2nd Edition. Springer-Verlag, New York, New York, USA.
Decognet, V., Bardin, M., Romiti, C., Trottin-Caudal, Y., Fournier, C., Leyre, J.M., Nicot, P.C., 2000. Lutte biologique contre l'oïdium et la pourriture grise sous serre de tomate. Journées Jean Chevaugeon Aussois (FRA)
Decognet, V., Bardin, M. & Nicot, P., 2004. Dynamique spatio-temporelle d’une épidémie de Botrytis sur tomate sous serre : importance pour la protection intégrée. PHM Revue Horticole 461
Eden, M.A., Hill, R.A., Beresford, R., Stewart, A., 1996. The influence of inoculum
concentration, relative humidity, and temperature on infection of greenhouse tomatoes by Botrytis
cinerea. Plant Pathology 45: 795-806
McCullagh, P. & J. A., Nelder, 1989. Generalized Linear Models. Chapman and Hall: London
Nicot, P., Decognet V. & Bardin M., 2004. Dynamique spatio-temporelle d’une épidémie de Botrytis sur tomate sous serre : importance pour la protection intégrée. PHM Revue Horticole 461
Williamson, B., Tudzynski, B., Tudzynski, P. & Van Kan, J. A. L., 2007. Botrytis cinerea: the
Annexe 1
Figure 1 : conidiophores de B. cinerea dressés sur une tige de tomate.
conidies conidiophores
local_alenya date nb_spore_m3_air h8_h14 h14_h20 h20_h8 rayonnement_global hauteur_pluie temperature_moy temperature_min temperature_max humidite_moy humidite_min humidite_max vitesse_moy vitesse_max ... date decimal decimal decimal decimal decimal decimal decimal decimal decimal decimal decimal decimal decimal decimal <pk> date_arrivee date nb_spore_m3_air h8_h14 h14_h20 h20_h8 rayonnement_global hauteur_pluie temperature_moy temperature_min temperature_max humidite_moy humidite_min humidite_max vitesse_moy vitesse_max ... date date decimal decimal decimal decimal decimal decimal decimal decimal decimal decimal decimal decimal decimal decimal <pk> <pk> hysplit_alenya id ref_altitude date_arrivee date heure_arrivee heure loc_date h nb_spore_m3_air latitude longitude altitude pression temperature hauteur_pluie humidite rayonnement_solaire ... integer integer date date integer integer date integer decimal decimal decimal decimal decimal decimal decimal decimal decimal <pk> <pk,fk1> <pk,fk1> <pk> <pk> <fk2> local_avignon date nb_spore_m3_air h8_h12 h12_h17 h17_h8 vitesse_moy temperature_moy humidite_moy ... date decimal decimal decimal decimal decimal decimal decimal <pk> climatik_avignon id date_arrivee date nb_spore_m3_air h8_h12 h12_h17 h17_h8 rayonnement_global hauteur_pluie temperature_moy temperature_min temperature_max humidite_moy humidite_min humidite_max vitesse_moy integer date date decimal decimal decimal decimal decimal decimal decimal decimal decimal decimal decimal decimal decimal <pk> <pk> hysplit_avignon id ref_alti tude date_arrivee date heure_arrivee heure loc_date h nb_spore_m3_air latitude longitude altitude pression temperature hauteur_pluie integer integer date date integer integer date integer decimal decimal decimal decimal decimal decimal decimal <pk> <pk,fk1> <pk,fk1> <pk> <pk> <fk2> 1 1 1 1 1 1 1 3 3
Annexe 3
Exemple de données météorologiques de la base « Climatik » pour Alénya pour le 29 avril, 14 mai et 28 mai 2009 :
Annexe 4
Annexe 5
1er jeu de variables explicatives :
3
Annexe 6
2ème jeu de variables explicatives :
Je n’ai pas pu mettre tout le jeu de données mais que quelques morceaux car il a une taille trop importante.
3
Annexe 7
3ème jeu de variables explicatives :
Ici aussi je n’ai pas pu mettre tout le jeu de données mais que quelques morceaux car il a une taille trop importante.
Annexe 8
Signification des variables des GLM :
Pour Avignon :
Chum_min_j3 : humidité minimale climatik 3 jours avant la date de prélèvement Chum_max_j9 : humidité maximale climatik 9 jours avant la date de prélèvement Ctemp_moy_j8: température moyenne climatik 8 jours avant la date de prélèvement Ctemp_moy_j9: température moyenne climatik 9 jours avant la date de prélèvement Hhum_3_24 : humidité hysplit pour la référence altitude 3, de 24h à 48h avant la date de
prélèvement
Ctemp_min : température minimale climatik
Halt_3_24 : altitude hysplit pour la référence altitude 3, de 24h à 48h avant la date de prélèvement Ctemp_max : température maximale climatik
Hpluie_1_48 : hauteur de pluie hysplit pour la référence altitude 1, de 48h à 72h avant la date de
prélèvement
Cpluie_j1 : hauteur de pluie climatik 1 jour avant la date de prélèvement
Hhum_3_72 : humidité hysplit pour la référence altitude 3, de 72h à 96h avant la date de
prélèvement
Chum_min_j2 : humidité minimale climatik 2 jours avant la date de prélèvement Ctemp_min_j2 : température minimale climatik 2 jours avant la date de prélèvement
Hpluie_1_24 : hauteur de pluie hysplit pour la référence altitude 1, de 24h à 48h avant la date de
prélèvement
Hcos_1_0 : cosinus de l’angle moyen de la trajectoire hysplit pour la référence altitude 1, de 1h à
24h avant la date de prélèvement
Cray : rayonnement solaire climatik le jour du prélèvement
Ctemp_moy_j1 : température moyenne climatik 1 jour avent la date de prélèvement
Hcos_1_72 : cosinus de l’angle moyen de la trajectoire hysplit pour la référence altitude 1, de 72h à
96h avant la date de prélèvement
Ctemp_max_j7 : température maximale climatik 7 jours avant la date de prélèvement Cvent_max_j3 : vitesse maximale du vent climatik 3 jours avant la date de prélèvement
Hpluie_3_48 : hauteur de pluie hysplit pour la référence altitude 3, de 48h à 72h avant la date de
prélèvement
Ctemp_max_j4 : température maximale climatik 4 jours avant la date de prélèvement
Hray_3_24 : rayonnement solaire hysplit pour la référence altitude 3, de 24h à 48h avant la date de
Pour Alénya :
Hpluie_1_48 : hauteur de pluie hysplit pour la référence altitude 1, de 48h à 72h avant la date de
prélèvement
Hhum_2_24 : humidité hysplit pour la référence altitude 2, de 24h à 48h avant la date de
prélèvement
Hpress_3_0 : pression atmosphérique hysplit pour la référence altitude 3, de 1h à 24h avant la date
de prélèvement
Hpress_1_24 : pression atmosphérique hysplit pour la référence altitude 1, de 24h à 48h avant la
date de prélèvement
Hray_1_48 : rayonnement solaire hysplit pour la référence altitude 1, de 48h à 72h avant la date de
prélèvement
Hpress_3_24 : pression atmosphérique hysplit pour la référence altitude 3, de 24h à 48h avant la
date de prélèvement
Hpluie_3_48 : hauteur de pluie hysplit pour la référence altitude 3, de 48h à 72h avant la date de
prélèvement
Hray_1_24 : rayonnement solaire hysplit pour la référence altitude 1, de 24h à 48h avant la date de
prélèvement
Hcos_3_48 : cosinus de l’angle moyen de la trajectoire hysplit pour la référence altitude 3, de 48h à
72h avant la date de prélèvement
Htemp_1_48 : température hysplit pour la référence altitude 1, de 48h à 72h avant la date de
prélèvement
Hpluie_1_0 : hauteur de pluie hysplit pour la référence altitude 1, de 1h à 24h avant la date de
prélèvement
Halt_1_48 : altitude hysplit pour la référence altitude 1, de 48h à 72h avant la date de prélèvement Hpress_2_48 : pression atmosphérique hysplit pour la référence altitude 2, de 48h à 72h avant la
date de prélèvement
Hpluie_2_0 : hauteur de pluie hysplit pour la référence altitude 2, de 1h à 24h avant la date de
prélèvement
Hpluie_1_72 : hauteur de pluie hysplit pour la référence altitude 1, de 72h à 96h avant la date de
prélèvement
Hhum_1_24 : humidité hysplit pour la référence altitude 1, de 24h à 48h avant la date de
prélèvement
Hpress_1_72 : pression atmosphérique hysplit pour la référence altitude 1, de 72h à 96h avant la