Université de Sherbrooke
Etude de la recombinaison intermoléculaire dans les cellules somatiques de mammifères
par
Suzanne Br ouillette
Département de microbiologie
Thèse présentée à la faculté de médecine en vue de 1 'obtention du grade de
ma~tre es sc i ences CM.Sc.)
Permission bas been granted to the National Library of Canada to microfilm this thesis and to lend or sell copies of the film.
The author (copyright owner) bas reserved other publication rights, and neither the thesis nor extensive extracts from it may be printed or otherwise reproduced without his/her written permission.
L'autorisation a été accordée
à
la Bibliothèque nationaledu Canada de microfilmer cette thèse et de prêter ou de vendre des exemplaires du
film.
L'auteur (titulaire du droit d'auteur) se réserve les autres droits de publication: ni la thèse ni de longs extraits de celle-ci ne doivent être imprimés ou autrement reproduits sans son autorisation écrite.
Madame Maryse Courtemanche Bibliothèque générale
Univ. de Sherbrooke 2500 boul. Université Sherbrooke, J1K 2R1
3131 est, rue Sherbrooke Montréal, Québec, Canada H1W 182
Téléphone: (514) 527-1501 Services transfusionnels Centre de Montréal
Montréal, le 23 mars 88
OBJET: Microfilmage du mémoire de Suzanne Brouillette Matricule 80138579
Madame,
Je consens à ce que Suzanne Brouillette inclut dans
son mémoire de ma~trise la publication suivante:
"Intermolecular recombination assay for mammalian cells that produces recombinants carrying bath homologous
and nonhomologous junctions. Mol.Cell.Biol.
Z:
2248-2255."J'autorise que cette publication soit microfilmée avec son mémoire.
Veuillez agréer, madame, l'expression de mes sentiments les meilleurs.
A mes parents
TABLE DES MATIERES
TABLE DES MATIERES... ii
LISTE DES ILLUSTRATIONS •••••••••••••••••••••• ~ •••• iii LISTE DES SIGLES ET ABREVIATIONS •••••••••••••••••• SOMMAI RE • ••••••••••••••••••••••••••••••••••••••••• INTRODUCTION •••••••••••••••••••••••••••••••••••••• MATERIEL ET METHODES •••••••••••••••••••••••••••••• I. Cellules et culture ••••••••••••••••••••••• II. Préparation de RmI et construction des iv V 1 7 7 vecteurs plasmidiques.~... 7
III. Transfection et extraction de l'ADN extrachromosomique... 9
IV. Transformation bactérienne... 10
V.
Identification des plasmides recombinants. 10 VI. Analyse des plasmides recombinants... 11RESULTATS... 12
I. Description de l'essai •••••••••••••••.••.. 12
II. Analyse des plasmides recombinants... 16
III. Effet de la longueur de l'homologie... 20
IV. Etude de la réplication... 25
V. Effet de la linéarisation... 31
VI. Recombinaison intramoléculaire... 36
DISCUSSION... 45
I. Essai ....•...•....•••.•...•...•....•.. 45
II. Etendue de l'homologie... 46
III. Recombinaison non-homologue... 47
IV. Linéarisation... 48
V. Modèle de recombinaison... 51
REMERCIEMENTS... 57
i i i LISTE DES ILLUSTRATIONS
FIGURES
Figure 1. Molécules parentales... 8
Figure 2. Description de l'essai de Figure 3.
Figure 4. Figure 5.
recombinaison... 14
Cartographie des jonctions homologues 17
Jonctions homologues de pSBD176... 21
Régions délétées des recombinants
homologues entre p2313 et RmI... 26
Figure 6. Cartographie physique du recombinant
fl~l3~4-. • • • • • • • • • • • • • • • • • • • • • • • • • • • • • • ~EJ
Figure 7. Cartographie physique du recombinant
pSBD176... 43
Figure 8. Modèle de recombinaison homologue... 54
TABLEAUX
Tableau 1. Essais de recombinaison
intermoléculaire... 23
Tableau 2. Essais de recombinaison en absence
de réplication... 28
Tableau 3. Essais de recombinaison
ADN ccc DEAE ~-coli EDTA Kpb. pb. P.M. [32p] Py Rec A-tr i s ug ul um
LISTE DES SIGLES ET ABREVIATIONS
acide désoxyribonucléique ADN superhélicoidal
diéthyl aminoéthyl Escherichia coli
Ethylène diamine tëtraacétate kilopaires de bases
paire de bases poid moléculaire
radioisotope 32 du phosphate
virus du Polyome
phénotype recombinase A déficient
hydroxyméthyl-aminométhane microgramme
microlitre micromètre
SOMMAIRE
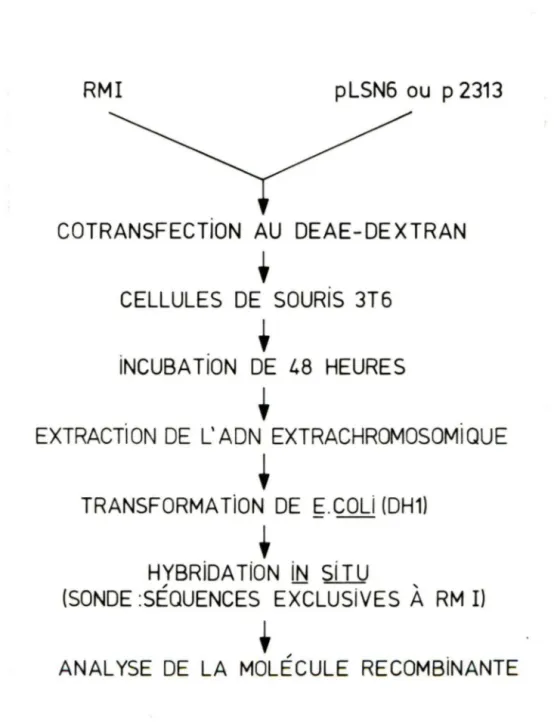
La plupart des essais de recombinai5on dans les cellules de mammifères sont basés sur la reconstitution d'un marqueur de sélection. Nous avons élaboré un essai intermoléculaire ou les recombinants sont isolés sur la base de la présence de séquences provenant des deux molécules parentales.
Nous étudions la recombinaison intermol*culaire entre un vecteur procaryotique et un vecteur eucaryotique dans des cellules de souris 3T6. Après cotransfection au DEAE-dextran, l'ADN extrachromosomique est extrait des cellules et les molécules plasmidiques sont isolées par transformation bactérienne. Les plasmides recombinants sont alors identifiés par hybridation in situ avec une sonde provenant de séquences exclusives au vecteur eucaryotique.
Les deux vecteurs ont des séquences en commun. Ainsi, notre essai permet de comparer directement les événements de recombinaison intermoléculaire homologue et non-homologue en absence de sélection pour un mécanisme particulier de recombinaison. Nos résultats démontrent que la recombinaison intermoléculaire non-homologue est le mécanisme prépondérant dans les cellules même lorsque les partenaires partagent d'importantes étendues de séquences. De plus, nous démontrons que la fréquence relative des deux types d ' événements de recombinaison est influencée par l'étendue de l'homologie entre les deux molécules, par l'état réplicatif du vecteur eucaryotique mais pas par la linéarisation comme telle.
vi En effet, la linéarisation du vecteur procaryotiqua ou des deux molécules parentales à des sites analogues dans l'homologie n'a pas influencé la fréquence relative fréquence rapport des de des non-homologues linéarisation
deux mécanismes de recombinaison ni la recombinaison totale. Par contre, le recombinaisons homologues versus a augmenté significativement après du vecteur eucaryotique ou des deux partenaires à des sites asymétriques dans l'homologie.
Les recombinants intermoléculaires homolog~es isolés ne possèdent pas les deux jonctions générées lors d'un seul événement de recombinaison réciproque. Dans presque tous les cas, ils possèdent plutôt une jonction homologue et une jonction non-homologue. Nous proposons un modèle de recombinaison décrivant les mécanismes
modèle, la chevauchement
menant à leur formation. est Selon générée notre par le jonction homologue
des extrémités homologues permettant l'appariement des séquences entre les deux partenaires. Une ligation bout-à-bout des extrémités libres non-homologues génère la jonction non-homologue.
INTRODUCTION La recombinaison cellulaire organismes. fondamental Dans est génétique contribuant les cellules un mécanisme à la diversité des somatiques . de mammifères, on a récemment associé la recombinaison à
des phénomènes tels le réarrangement des gènes des immunoglobulines lors de la formation d'anticorps <Alt
&
Baltimore 1982; Hedrick et al 1984; Honjo 1983>,l'activation d'oncogènes cellulaires par translocation, et l'intégration de virus tumorigènes <Bird et al 1979; Botchan et
êl.
1976; Ketner&
Kelly jr 1976). En introduisant du nouveau matériel génétique dans une cellule,thérapie corriger
on peut envisager la régulation génique ou la génique en permettant à une cellule de l'information existante et d'acquérir de nouveaux caractères.
Trois mécanismes distincts de recombinaison ont été décrits jusqu'à présent dans les cellules somatiques. La recombinaison dite homologue s'effectue entre des séquences homologues d'ADN de deux partenaires. Au niveau chromosomique, c'est le cas d es échanges entre chromatides-soeurs, de la production de certaines délétions et de la formation de certaines molécules libres d'ADN dans les cellules CKrolewski et al 1984; Schmookler Reis et al 1983). L'ADN exogène introduit dans une cellule peut également se recombiner de façon homologue avec l e chromosome <Folger et al 1984; Kata et al 1986; Lin et~ 1984a; Smithies et al 1985).
Il peut aussi participer à des événements de recombinaison homologues épisomiques CAyares et al 1986; Chakrabarti
&
Siedman 1986; Folger et al 1985;2
Kucherlapati ~
êl.
1984J Miller&
Temin 1983J Rauth ~ iù._ 1986; Rubnitz&
Subramani 1985; Small&
Scangos1983).
Le mécanisme d• recombinaison sites-spécifiques fait intervenir des séquences spécifiques d'un fragment donneur ou des fragments donneur et receveur. . Ce mécanisme ne requiert aucune homologie de séquences entre les deux partenaires. L'intégration des rétrovirus <Temin 1980; Weinberg 1980>, la transposition d'éléments génétiques et possiblement des séquences répétitives CJeline~
&
Schmid 1982>, le réarrangement des gènes des immunoglobulines <Alt&
Baltimore 1982; Hedrick etè!.
1984; Honjo 1983) en sont des exemples.Enfin, événements
la recombinaison illégitime définie les de recomb inaison entre deux séquences d'ADN spécificité ni homologie importante où ni
n'interviennent. C'est le mode par lequel s'intègrent les génomes des virus tumorigènes à ADN CBirg et al 1979; Botchan et al 1976; Bullock
&
Botchan 1982; Ketner&
Kelly jr 1976). Ce mécanisme est aussi observé lors de l ' introduction de molécules d'ADN exogènes danschromosome Cde
des cellules eucaryotes entre épisome et Saint Vincent et ~ 1981; Lin et al
et al 1981; Wallenburg et al 1984) ou 1985; entre 1985; 1980). à peu Sc anges
épisomes CChakrabarti et al 1985; Roth
&
Wilson Subramani&
Berg 1983; Winocour&
Keshet Chez les procaryotes, ce type de mécanisme est près inexistant alors que la recombinaison homologue est de loin leur mécanisme prédominant.Afin d'étudier les divers mécanismes de recombinaison dans les cellules somatiques de mammifères, plusieurs essais ont récemment été élaborés
<présentés plus est introduit mammifères en suivantesz la
<Graham van
bas>. Dans la majorité dea cas, l'ADN dans des cellules somatiques de culture par l'une des méthodes transfection par précipité au CaCl2 der Eb 1973> ou au DEAE-dextran <McCutchan
&
Pagano 1968>, la micro-injection <Anderson et al 1980; Capecchi 1980>, l'infection <Shimotohno&
Temin 1981>, 1 'électroporation CNeuman
lt1.
al 1982; Patter@!.
~ 1984>, la fusion de protoplastesCSchaffner 1980). Par ailleurs, certains es~ais
utilisent des extraits cellulaires <Darby
&
Blattner 1984; KucherlapatiK.:t. êl.
1985). Ces diverses approches ont permis d'étudier la recombinaison épisomique intramoléculaire et intermoléculaire, la recombinaison entre un épisome et le génome ainsi qu ' au niveau intrachromosomique.La plupart des essais ont mis en évidence la recombinaison homologue intramoléculaire ou intermoléculaire en sélectionnant pour la reconstitution d ' un marqueur génétique <Anderson et ~
1984; Ayares et al 1985; Bandyopadhyay et ~ 1984; Brenner et a l 1985; de Saint Vincent
&
Wahl 1983; Folger et ~ 1982; Jackson 1980; Kucherlapati et al 1984; Lin et al 1984b; Lin&
Sternberg 1984; Liskay&
Stachelek 1983; Miller&
Temin 1983; Pomerantz etal 1983; Rubnitz
&
Subramani 1984, 1985; Shapira et al 1983; Small&
Scangos 1983; Stringer et al 1985; Subramani&
Berg 1983; Subramani&
Rubnitz 1985; Upcroft et al 1980; Vogel et a l 1977; Wake&
Wilson 1979). Sous ces conditions, on a démontré que l'efficacité de la recombinaison augmente avec la longueur d ' homologie entre les séquences participantes <Bandyopadhyay et al 1984; Brenner et al 1985; Kovar&
Wintersberger 1985J4
Lin ~
ê.l
1984b; Rubnitz&
Subramani 1984; Shapiraf!1.
~ 1983; Subr•mani&
Berg1983). On a aussi démontré que l'efficacité de l'év9nement homologue augmente de 10 à 300 fois après coupure double-brin dans ou près de l'homologie CAyares et al 1985; Brenner
&
Camerini-Ot•ro 1985; Brenner ~ al 1985; Chakrabarti et al 1985; Folger ~ ~ 1985J Kucherl•pati et ~ 1984J Lin et al 1984b; Rubnitz&
Subramani 1985; Song ~ê.l.
1985; Wake et al 1985; Wong&
Capecchi 1986>.Deux modèles récents de recombinaison homologue chez les eucaryotes découlent de tels essais et proposent une série d'événements initiés par une coupure double-brin <Lin et al 1984b, 1987; Szostak et
ël.
1983). Cette coupure est suivie d ' une digestion exonucléasique libérant des extrémités simple-brin. Szostak et al (1983 ) ont élaboré un modèle suite à des travaux chez la levure. Selon eux, un événement de recombinaison homologue réciproque s'effectue par invasion du simple-brin dans le duplex homologue et par synthèse des brins complémentaires. Le recombinant homologue résultant possède deux jonctions homologues et a conservé les séquences des deux parents. Chez les mammifères, 1 'équipe de Sternberg ont proposé un modèle basé sur celui de Szostak etël.
(1983) en proposant que suite à la linéarisation initiale, une activité exonucléasique plus importante libère deux fragments homologues simple- brin. L'alignement homologue s ' effectue alors entre ces brins homologues complémentaires et résulte en une structure intermédiaire. Sa résolution génère une jonction homologue (Lin et al 1984b, 1987). Ce modèle ne prédit cependant pas ce qui advient du reste de la molécule,5
plu& particulièrement de l'autre jonction générée lors d'un tel événement de recombinaison.
Certains essais ont mesuré le& événement& illégitimes <Anderson
l!1.
Al_ 1984; Chakrabarti et al 1985; Dorsett etAl.
1983' Grossmanl!1.
AL 1984; Kretschmer etA!..
1981; Miller&
Temin 1983; Roth&
Wilson 1985; Roth etA!..
1985J Wilsonl!1.
al 1982; Winocour&
Keshet 1980>. Dans ces cas, l'événement semble essentiellement une ligation bout-à-bout entre des extrémités libres de fragments linéaires (Chakrabarti ~ al 1985; Folger etA!...
1982; Miller&
Temin 1983; Roth et al 1985; Wake et al 1984; Wilson et al 1982). Roth et al (1985) ont élaboré un modèle décrivant de telles ligations.Récemment, des essais ont permis de comparer les illégitimes au niveau mécanismes homologues et
intramoléculaire <Chakrabarti et al 1985; Roth
&
Wilson 1985; Stringer 1985; Subramani
&
Berg 1983). Selon ces travaux, les deux mécanismes s'effectuent àdes taux comparables dans les cellules.
Jusqu'à présent, aucun essai ne nous permettait de comparer directement les rapports des mécanismes homologues et non-homologues entre deux molécules d'ADN ou d'étudier les paramètres impliqués dans les fréquences
élaboré un
relatives des deux événements. Nous avons essai de transfection afin d'étudier la recombinaison homologue et non-homologue entre deux molécules. Notre système nous permet de calculer les fréquences des deux mécanismes et de les comparer dans un même essai. Nous pouvons varier certains paramètres tels: la longueur de l'homologie, l'état réplicatif des molécules, la linéarisation d'une ou des deux molécules et les positions relatives des coupures
6
double-brin. Nous pouvons ainsi étudier l'effet de ces
paramètres sur les fréquences des événements homologues et non-homologues.
Durant nos travaux, d'autres ont élaboré des essais
s'y rapprochant CAyares et al 1985; Rubnitz
&
Subramani 1984, 1985>. Notre système demeure unique du fait que les molécules recombinantes sont récupérées sans sélectionner pour un mécanisme particulier ou un
type de produit de recombinaison. Cette
caractéristique nous permet d'analyser les deux
jonctions présentes sur une même molécule
recombinante. Nous croyons que de telles molécules·
n'ont pas pu être observées par d'autres lors d'essais basés sur une sélection pour un mécanisme particulier
de recombinaison. Enfin, nous présentons un modèle de
recombinaison permettant d'expliquer la formation de ces molécules recombinantes.
7 MATERIEL ET METHODES
I. Cellules et culture
Les cellules d'embryons de souris Swiss albinos 3T6
<Todaro
&
Green 1963) ont été cultivées à 37oc (10Y.C02> dans du DMEM <Dulbecco's Modified Eagle's
Medium: Flow> enrichi de L-glutamine <Sigma: 5,84 x
10-4 g/ml>, de 5% de sérum de veau <Flow> et de 50
ug/ml de sulfate de gentamycine <United States
Biochemical).
II. Préparation de RmI et construction des vecteurs
plasmidigues
RmI est une molécule recombinante naturelle
contenant 1,03 copies du génome de Py interrompues par
une insertion cellulaire d'ADN de souris (fig.1>.
C'est un produit d'excision de C12a1 <Sylla et
2.1
1980>, une lignée de cellules de souris transformée par
un mutant thermosensible de Py: tsP 155 <Eckhart
1974). La mutation de tsP155 est contenue dans la
portion du génome codant pour l'antigène grand T, et rend la molécule RmI thermosensible pour la réplication <Bourgaux et al 1982>. RmI peut se répliquer librement dans des
le faire C12a1, RmI
cellules de souris
à 39oc. Produit
est obtenu par
à 33oc mais ne peut pas
d'excision des cellules induction de cette lignée par e x traction et purification d'ADN extrachromosomique
8
FIGURE 1. MOLECULES PARENTALES
pLSN6 dérive du plasmide pAT153. Il contient un
fragment Bell de 191 pb. de séquences cellulaires de
souris de RmI inséré à son site unique BamHI
<gracieuseté de P. Bourgaux>. Ori est l'origine de
réplication procaryotique ColE1 et ampr correspond au
segment codant pour le gène de résistance à
l'ampicilline.
p2313 est aussi dérivé de pAT153 par insertion à
son site unique Ball d'un fragment HincII de 2313 pb.
provenant de RmI. Ce fragment inclut l'insertion
cellulaire et les s é quences virales flanquantes. Les sites uniques SalI et !!gl_II correspondent aux sites utilisés lors d'essais de linéarisation de p2313.
RmI est une molécule recombinante naturelle
contenant 1,03 copies du génome de tsP155, un mutant de
Py thermosensible pour l a réplication. Le Py est
interrompu dans sa région tardive par une insertion
d'ADN cellulaire de souris de 1628 pb. Les sites
uniques SalI et !!gl_II ont été utilisés lors d'essais de linéarisation de RmI. Ori est l'origine de réplication
de Py. Les O, 30, 70 et 100 correspondent à la carte
physique du génome du virus du Py.
Le trait simple correspond aux séquences
plasmidiques. Le trait noir indique les séquences de
,/
f
pLSN6 l3848pb) '...--1/"Q / Sail ( p2313 ~. l5970pb)\
0/100 Figure 19
Le plasmide pLSN6 nous a été généreusement fourni
par le Dr P.Bourgaux. Il contient un fragment BclI
cellulaire de RmI (191 pb.> inséré au site unique BamH1 de pAT153.
Le plasmide p2313 provient du clonage d'un fragment
HincII de RmI <2313 pb.) dans le site unique BalI de
pAT153. Ce fragment comprend la totalité des 1628 pb.
de l'insertion cellulaire de RmI ainsi que les
séquences de Polyome flanquant de part et d'autre la
région cellulaire. Les deux plasmides ont été isolâs
en présence d'ampicilline. Les fragments clonés de 191 pb. et 2313 pb. respectivement, représente la seule homologie existant entre ces plasmides et RmI.
III. Transfection et extraction de l'ADN e xtrachromosomigue
Les cellules de souris 3T6 ont été ensemencées 24
heures avant la transfection à une confluence de 1,0 x
106 cellules par bo~te de pétri de 90 mm. Chaque
pétri a été cotransfecté par 0,5 ug chacun de RmI et de pLSN6 ou p2313 sous formes circulaires ou linéaires par la méthode de transfection au DEAE-dextran (Sigma: P.M. 500,000> de McCutchan et Pagano (1968) avec les modifications de Sussman et Milman (1984). Les essais
ont été effectués à la température permissive pour la
réplication de RmI C330C) pendant les 48 heures
d'incubation sauf lorsque contre-indiqué dans le texte. L'ADN extrachromosomique a été isolé des cellules 3T6 par la méthode de Hirt (1967), 48 heures après transfection dans tous les cas. Les échantillons ont
alors été extraits au phénol, précipités à l'éthanol et
resuspendus dans un volume de 20 ul de 10 mM Tris.HCl <pH 7,5>, 1,0 mM EDTA par pétri transfecté.
10 IV. Transformation bactérienne
Les extraits purifiés de Hirt ont été utilisés pour transformer directement la bactérie DH1, une souche Rec
A- de ~.coli tel que décrit par Hanahan C1983> avec
modifications mineures. La sélection était basée sur
l'acquisition
ug/ml> dont
plasmidique. l'équivalent
de la résistance à l'ampicilline C35
le gène est porté par le vecteur
Par tube d'essai, 4 ul du Hirt ou
de 20ï. d'un pétri transfecté a été
utilisé. Hanahan
Dans notre laboratoire, la technique donne une efficacité reproductible de
de 107 transformants/ug d'ADN.
V. Identif i cation des p lasmides recombina nts
Les colonies transformantes sur bo~tes de pétris
ont été dupliquées et transférées sur membranes de nitrocellulose CAmersham, 0,45 CMillipore, um> par la 0,45 um> méthode ou de nylon de transfert de colonies décrite par Grunstein et Hogness C1975). Les
colonies recombinante s ont été identifiées par
hybridation in
du Polyome
nick-translation
situ à l'aide de sondes d'ADN du virus
tsP155 marqué au [32p] par
selon Rigby et al <1977>. Pour les essais avec RmI et pLSN6, le génome complet de tsP155 a
été utilisé comme sonde. Dans les cas d'essais avec
RmI et p2313 cependant, seulement la portion virale de
RmI non incluse dans p2313 a été utilisée.
L ' hybridation a été effectuée selon la technique de van der Ploeg et Flavell C19BO> avec les modifications de
11 filtres hybridés ont été développés sur films Kodak
XAR-5 ou Fuji XRP-1 après une exposition de 2 à 20
heures à -70oC avec écrans intensifiants.
VI. Analyse des plasmides recombinants
Les colonies ayant hybridées ont été repiquées, et l'ADN plasmidique a été extrait par lyse alcaline
<Maniatis et al 1982>. L'analyse subséquente par
enzymes de restriction a été effectuée selon les
recommandations des fabricants <Amersham Corp.,
Boehringer Mannhein Biochemicals, New England Biolabs Inc., Pharmacia Inc.> en présence de 5X excès d'enzyme
ou avec modifications mineures . Les fragments d'ADN
ont été séparés par électrophorèse sur gels hori z ontaux
d'agarose de 0,7/. à 1,2/. dans du 40 mM trizma, 10 mM
acétate de sodium, 1,0 mM EDTA, pH 7,9 et visualisés par fluorescence au bromure d ' éthidium.
RESULTATS
I. Description de l'essai
Nous avons cotransfecté les molécules RmI et pLSN6
ou p2313 (fig.1) circulaires ou linéaires dans des
cellules de souris 3T6 à 33 ou 390C (fig.2>. La
molécule eucaryotique RmI est un variant naturel du virus du Py contenant en plus d'un génome viral complet, des séquences cellulaires de souris. Comme le Py, RmI peut se répliquer dans les cellules de souris. A 33oc, la réplication de la molécule RmI est possible alors qu'à
39oc, la mutation de Polyome présente dans le RmI
empêche sa réplication, tel que décrit par Bourgaux et al
( 1982). Nous avons choisi la température de 33oc pour
la plupart des essais de façon à permettre
l'amplification des molécules recombinantes entre RmI et les vecteurs plasmidiques. La molécule procaryotique est le plasmide pAT153 contenant des longueurs définies de
séquences cellulaires homologues à RmI. Les molécules
ont donc le potentiel de se recombiner de façon homologue
ou non-homologue. De plus, une molécule recombinante
homologue ou non-homologue issue d'un événement entre RmI et le plasmide sera un vecteur navette puisqu'elle pourra être répliquée autant chez la souris que dans une cellule bactérienne.
L'ADN extrachromosomique fut extrait des cellules
3T6 selon Hirt après une incubation de 48 heures
Ce temps d'incubation a été choisi parmi des de 24, 48 et 72 heures parmi lesquels une (fig.2).
essais
récupération maximale d'ADN a été obtenue après 48 heures (essais non montrés>. Nous croyons que l'incubation doit
être assez longue pour permettre la recombinaison et
l'amplification des molécules recombinantes dans la
cellule sans toutefois que les molécules d'ADN aient le
temps d'être dégradées de façon appréciable par la
cellule ni que d'importantes morts cellulaire s puissent occasionner la perte de matériel génétique.
utilisé A- de
L'ADN extrait des cellules transfectées a été pour
~.coli
transformer DH1, une souche bactérienne Rec
(fig.2>. L'extrait contenait en plus des
deux molécul es cotransfectées au départ et nonrépliquées, des copies répliquées de la molécule Rm1 ainsi que des formes recombinantes entre Rm1 et le plasmide introduit. Or, les molécules RmI non recombinées ne contenant pas d'origine de réplication procaryotique, n'ont pu être récupérées lors de la transformation bactérienne et ont ainsi été éliminées. Le plasmide non recombiné et toute
forme recombinante contenant au moins l'origine de
réplication procaryotique et les séquences codant pour la
résistance à l'ampicilline ont pu être récupérés lors de
la transformation bactérienne. Nous croyons de plus que la présence de séquences de RmI permettant la réplication dss formes recombinantes dans la cellule de souris a
conféré un avantage. A l'appui, la grande majorité des
recombinants présentés au tableau 1 possèdent l'origine de réplication de Py. Cette caractéristique de vecteur
navette est à la base même de la récupération des
recombinants par notre essai.
Après étalement sur pétris, nous avons procédé à
une hybridation in situ des colonies transformées en utilisant comme sonde radioactive l'ADN du virus du Py
non contenu dans le vecteur plasmidique. Nous avons
ainsi identifié les plasmides recombinants contenant des
FIGURE 2. DESCRIPTION DE L'ESSAI DE RECOMBINAISON RmI
technique
et de
pLSN6 ou p2313 sont cotransfectés par la transfection au DEAE-dextran dans des cellules de souris 3T6 en culture. Après une incubation de 48 heures, utilisé pour l'ADN extrachromosomique transformer ~.coli. est extrait et Les plasmides recombinants sont identifiés par hybridation in situ avec
une sonde provenant de séquences exclusives à la molécule
RmI. Les molécules recombinantes sont extraites et
Figure 2
RMI
pLSN6 ou p 2313
COTRANSFECTION AU DEAE-DEXTRAN
•
CELLULES DE SOURIS 3T6
.
INCUBATION DE 48 HEURES
. t
t
EXTRACTÎON DE L' ADN EXTRACHROMOSOMÏQUE
+
TRANSFORMATiON DE
~.COLi
(DH1)
t
HYBRiDA TiON iN Si TU
(SONDE :SÉQUENCES EXCLUSiVES
À
RM
Il
t
~ .molécules recombinantes ont été purifiées, extraites et cartographiées par une variété d'enzymes de restriction.
Nous avons comptabilisé comme étant des molécules recombinantes issues d'événements indépendants de recombinaison que celles présentant des patrons de restriction distincts. Or, certaines molécules recombinantes présentaient le même patron de restriction. Ces molécules au patron de restriction identique ont été considéré des formes amplifiées d'un même recombinant de départ. Le nombre de formes amplifiées d'un même recombinant variait d'un patron de restriction à un autre. En e f fet, nous avons isolé jusqu'à 21 copies d'un même recombinant alors que d'autres n'ont été iso l és qu'en une seule copie. L'amplification dans les bactéries ne réussit pas à elle seule à expliquer cette variabilité dans le nombre de formes amplifiées de chaque recombinant ni le fait que certains recombinants soient amplifiés un grand nombre de fois. Dans le cas où l'événement de recombinaison aurait eu lieu dans les bactéries, on se serait plutôt attendu à peu ou pas de formes amplifiées <correspondant seulement aux divisions bactériennes lors de la transformation avant étalement sur pétri). L'amplification observée suggère plutôt qu'après recombinaison, les molécules recombinantes se répliquaient un certain nombre de fois dans les cellules de souris 3T6. On pourrait ainsi expliquer la variabilité dans le nombre de formes amplifiées de chaque recombinant de deux façons: soit !)que les recombinants ne se répliquent pas tous aussi bien les uns que les autres dans les cellules de souris ou 2>que les événements distincts de recombinaison ont lieu à différents moments après transfection. · Néanmoins, afin de s'assurer q~e la recombinaison entre les deux
16 molécules de départ s'effectuait dans les cellules de souris et non pas lors de la transformation bactérienne subséquente, nous avons transformé directement l'hôte Rec
A- d'~.coli avec les différentes paires de molécules
parentales présentées au tableau 1. Tel qu'attendu, aucune des colonies obtenues lors de ces essais n'hybridait avec la sonde Polyome <tableau 1, contrôles>.
II. Analyse des plasmides recombinants
Afin de déterminer la nature du mécanisme à la base de la formation des molécules recombinantes, nous avons effectué une série de digestions par des enzymes de restriction et cart ographié les recombinants. Plus spécifiquement, il s'agissait de mettre en évidence les fragments de restriction contenant les régions de
jonction entre les deux molécules de départ: jonctions pLSN6-Rm1 ou p2313-Rm1 Cfig.3>.
A la fig.3, la région d'homologie entre les deux molécules pLSN6 et Rm1 est illustrée (région hachurée>. Cette région de 191 pb. correspond à la seule région d'homologie entre les deux molécules parentales. Un événement intermoléculaire homologue réciproque entre les deux partenaires générerait deux jonctions homologues sur un même molésule recombinante.
Dans notre essai, une jonction est définie somme homologue si elle contient une copie complète de la région d'homologie parentale et que cette région est bordée d'une part par des séquences de RmI et d'autre part par des séquences plasmidiques. Ces jonctions homologues peuvent ètre identifiées par des fragments de restriction de longueur précise générés à la suite de l'événement de recombinaison.
FIGURE 3. CARTOGRAPHIE DES JONCTIONS HOMOLOGUES
Cartographie physique des régions d'homologie
entre les paires de molécules parentales: pLSN6-RmI et
p2313-Rml (gauche). Les jonctions homologues générées
par un événement intermoléculaire homologue réciproque entre les partenaires sont aussi illustrées (droite>. On appelle j et J les jonctions homologues issues d'un événement homologue entre p2313 et RmI. Des événements non-homologues entre les paires de molécules parentales génèrent des fragments de jonction non-homologues de longueurs variables.
les séquences
Les traits simples représentent
plasmidiques de pLSN6 ou de p2313. représentent l'ADN du virus du Py.
Les traits noirs Les traits clairs
correspondent à l'insertion de séquences cellulaires de
souris.
Les régions hachurées représentent la seule
homologie existant entre les partenaires et ainsi la
région à l'intérieur de laquelle s'effectue l'événement
de recombinaison homologue.
A: Aval, B: BamH1, C: HincII, E: EcoR1, G:
~I I , H: Hi nd I I I , I : ê.Q.l.I , P: Pst 1 , S: Sal 1 , V: Pvu! I.
Figure 3 REGIONS HOMOLOGUES PARENTALES EVENEMENT INTERMOLECULAIRE ANALYSE DES JONCTIONS EH 0 1
PL
1
e V H B [p^ I I c/s I I I , P ///*6 VH B'///, H C ///, // / ///C/S P P I 1/I////I I I I r///j| NOM\hom
E H V 6 V H B I l I I Ii
É
c/s I NON-NOM - Absence de jonctions homologues - Présence de jonctions non-homologues 0] c/s p TTTTf / / / n VG V H B 2313 pb. aEn molécule
plus d'un recombinante
événement homologue, une même peut participer à d'autres événements non-homologues tels qu'observés dans le cas de 39 des 40 recombinants homologues obtenus entre p2313 et Rm1 (présentés plus bas>. Ainsi, il se peut qu'en plus des séquences homologues impliquées dans l'événement de recombinaison homologue, une molécule recombinante ait subi d'importants réarrangements. Il est donc essentiel que les fragments de jonctions homologues servant à
l'identification des recombinants homologues bordent le plus près possible chaque côté de la région d'homologie. A cet effet, plusieurs enzymes et plusieurs combinaisons différentes de digestions enzymatiques ont été utilisés pour les essais de recombinaison entre pLSN6 et Rm1, dont certains sont illustrés à la figure 3.
La présence d'une des deux jonctions homologues est suffisante pour qu'un recombinant soit considéré un recombinant homologue. Comme nous le verrons plus loin, aucune jonction homologue n'a pu être mise en évidence lors des essais entre pLSN6 et Rm1. En effet, tous les recombinants entre ces deux molécules semblent issus d'événements non-homologues et ont été identifiés comme étant des recombinants non-homolgoues selon les critères suivants. Le premier est l'absence de jonctions homologues chez ces recombinants. Le second est la présence des régions homologues parentales intactes de pLSN6 et/ou de RmI, suggérant que les séquences homologues n'aient pas participer à l'événement de recombinaison. Ces régions homologues parentales n'étaient pas toujours présentes cependant, puisque certains recombinants non-homologues très réarrangés avaient subi de grandes délétions incluant l'homologie. Le dernier est la mise en évidence de fragments de
restriction correspondant à des jonctions non-homologues entre les deux molécules confirmant que
l'événement de recombinaison a eu lieu à l'extérieur de
1 'homologie.
Les recombinants entre les molécules p2313 et
Rml ont été analysés de façon analogue à ceux obtenus
entre pLSN6 et RmI (fig.3>. La région hachurée de 2313 pb. représente l'unique segment d'homologie entre les
deux molécules parentales. Des digestions par
multiples enzymes de restriction ont permis de
cartographier les recombinants. Entre autres, les deux
enzymes: ~I et HincII ont été particulièrement
utiles dans l'analyse des jonctions des recombinants et la recherche des jonctions homologues. Ils permettent de générer les fragments de jonctions les plus courts possibles, pour les mêmes raisons que celles citées plus haut pour les essais entre pLSN6 et RmI. La digestion double d'un recombinant entre p2313 et RmI
par ~I et HincII génère un certain nombre de
fragments. Si la molécule recombinante est issue d'un
seul événement de recombinaison homologue réciproque,
l~s fragments de jonction seront ceux illustrés à la
fig.3 et à la fig.4, appelés j et
J.
Par exemple, un recombinant intermoléculaire
homologue réciproque entre p2313 et RmI aurait la même
carte de restriction que celle illustrée pour le
recombinant pSBD176 (fig.4, en II>. Les deux régions de 2313 pb. correspondent aux régions d'homologie entre
RmI et p2313 avant la recombinaison <référer à la
fig. 1). Dans la molécule pSBD176, elles sont toutes
deux bordées par des séquences de p2313 d'une part et
par des séquences de RmI d'autre part. Après dig~stion
20 mettre en évidence les deux fragments de jonctions de la figure 3 (j et J). Ces deux fragments ne comigrent pas avec des fragments uniques à p2313 ou à RmI (fig.4, en I, piste A>. J, de 3650 pb. et j, de 2590 pb. sont confirmés comme étant les deux jonctions homologues par une coupure supplémentaire par l'enzyme êgj_II (fig.4, en I, piste B>. J est alors scindé en J/1 et en J/2, de 2368 et 1282 pb. et j en j/1 et j/2, de 1555 et 1095 pb. respectivement. Il devient alors fort improbable que la position des deux êgj_II soit une coincidence et que les
jonctions digestions effectuées
fragments puissent être autre chose que les homologues elles-mêmes. De multiples supplémentaires ont quand même été afin de cartographier plus précisément chaque recombinant analysé <non montrées>.
Pour tout recombinant quelconque, la confirmation de la présence de J et/ou de j lors de son analyse permet de le classifier comme recombinant homologue. Le patron de restriction d'un recombinant non-homologue coupé par les mêmes enzymes ne présente ni l'une montré>. jonctions croyons récupérés analyse inaperçu,
ni l'autre des deux jonctions homologues <non Il possède cependant des fragments de non-homolgoues de longueurs variables. Nous que le nombre de recombinants homologues par notre système et qui échappe à notre est négligeable. La raison est que pour passer un recombinant devra être modifié au point où ni l'une ni l'autre de ses deux jonctions homologues ne puissent être détectées par l'analyse de restriction.
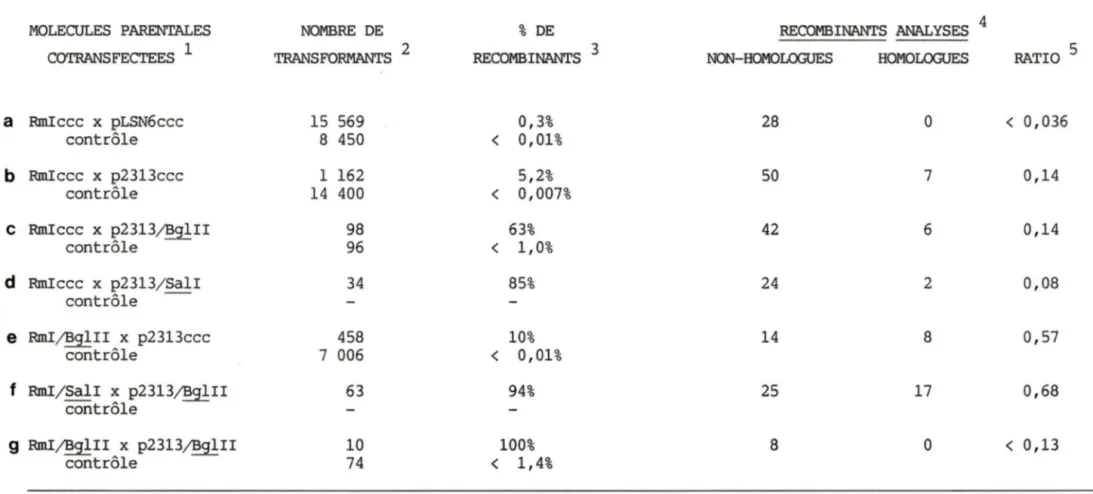
III. Effet de la longueur de l'homologie
21
FIGURE 4. JONCTIONS HOMOLOGUES DE pSBD176
pSBD176 est un recombinant homologue réciproque entre p2313 et RmI.
1·
Gel de digestion enzymatique illustrant les fragments de jonctions homologues de pSBD176.A> Digestion double ~1 et HincII. J et j représentent les fragments de jonctions homologues. Les fragments A et C correspondent à des fragments de RmI. Q est un fragment de p2313.
B> Digestion triple ~1, HincII et ~II. Les fragments de jonctions homologues J et j sont scindés par ggJ_II en J/1, J/2 et j/1, j/2 respectivements.
11·
Carte de la molécule pSBD176Les sites de coupure pour ~1, HincII et full.II ainsi que les fragments de restriction générés: A, C,
Q, J et j sont montrés. Les deux segments de 2313 pb. représentent l'homologie partagée par les molécules parentales p2313 et RmI. J et j sont les fragments de restriction ggJ_1 et HincII correspondant aux jonctions homologues. Les traits simples sont les séquences plasmidiques de p2313. Les traits noirs représentent l'ADN viral du Py. Les traits clairs correspondent à
la numérotation de la carte physique de Py. Ori des séquences virales est l'origine de réplication de Py. Ori et ampr des séquences plasmidiques représentent respectivement l'origine de réplication procaryotique ColE1 et le gène de résistance à l ' ampicilline.
A B
]:3650.
1
j
:2590 ...
1 1
Figure 4- ]/1
- j /
1
- J
/2
- j /
2
22 cotransfectés sous forme circulaire <tableau 1, en a>. Cinq cotransfections indépendantes ont été effectuées
et l'ADN récupéré a été utilisé dans 19 essais
distincts de transformation bactérienne <non montrés).
La proportion des 15 569 colonies transformantes
isolées ayant hybridées avec la sonde Polyome est de
0,3/.. D'un total de 44 signaux positifs obtenus lors
de l'hybridation, 28 présentaient des patrons de
restriction distincts, les 16 autres étant des formes
amplifiées des premiers. Après analyse détaillée par
enzymes de restriction, aucun des 28 recombinants
obtenus ne possédait de jonctions homologues. Ainsi,
aucun recombinant ne provenait d'une recombinaison
homologue entre les 191 paires de bases d'homologie partagée par les deux molécules parentales. En effet, ils possédaient tous des jonctions non-homologues entre RmI et pLSN6 et ont été identifiés comme étant des recombinants intermoléculaires non-homologues. Le taux
d'événements de recombinaison homologue versus
non-homologue était donc inférieur à 0,036.
U!::mr-= d::f l urn yap11 1111ui:I onmaii,, maIU.S5 ~ ~
sous forme <tableau 1, circulaire en b). les Elles molécules RmI et p2313 par tagent 2313 paires de bases d'homologie, soit 12,1 fois plus qu'entre RmI et
pLSN6. A partir des Hirt de trois cotransfections
distinctes, 14 transformations bactériennes ont été
effectuées. Des 1162 colonies transformantes obtenues,
5,2ï. hybridaient avec la sonde Py.
Cette fréquence de recombinaison est 17 fois plus élevé e que celle observée entre RmI et pLSN6
<tableau 1, en a>. Pourtant, l ' augmentation de la
fréquence de recombinaison homologue d'un facteur de
a
bc
de
f MOLECULES PARENTALES COTRANSFECTEES l Rmiccc x pLSN6ccc contrôle Rmiccc x p2313ccc contrôle Rmiccc x p2313/BglII contrôle Rmiccc x p2313/ SalI contrôle RmI/BglII x p2313ccc contrôle RmI/SalI X p2313/BglII contrôleTABLEAU 1. ESSAIS DE RECOMBINAISON INTERMOLECULAIRE NOMBRE DE TRANSFORMANTS 2 15 569 8 450 1 162 14 400 98 96 34 458 7 006 63 % DE RECOMBINANTS 3 0,3%
<
0,01% 5,2%<
0,007% 63%<
1,0% 85% 10%<
0,01% 94% RECOMBINANTS ANALYSES 4 NON-HOMOLcx;UES HOMOLcx;UES 28 0 50 7 42 6 24 2 14 8 25 17 RATIO 5<
0,036 0,14 0,14 0,08 0,57 0,689
RmI/BglII X p2313/BglII 10 100% 8 0<
0,13 contrôle 74<
1,4%1) ccc indique que les molécules d 'ADN cotransfectées étaient sous forme superhélico!dale. RmI/BglII, RmI/ SalI, p2313/BglII et p2313/SalI sont RmI ou p2313 linéarisés à leurs sites uniques ,BglII ou SalI respectivement. Les contrôles sont des essais témoins lors desquels les mêmes molécules ont été utilisees pour transformer directement la souche DHl de E. coli sans transfection dans des cellules 3T6.
2) Nombre de colonies bactériennes analysées.
3) Pourcentage des transformants ayant hybridé avec la sonde Polyome (reférer à Matériel et méthodes).
4) Nombre de formes recombinantes analysées qui présentaient des patrons de restriction distincts l'un de l'autre. 5) Rapport des recombinants homologues versus non-homologues.
cette hausse.
24 Effectivement, nous n'avons aucune cette valeur reflète une augmentation fréquence de recombinaison comme telle. indication que
réelle de la
De plus, nous obtenons transformants par essai et p2313ccc qu'entre dernière observation
en moyenne 18 fois moins de de transformation entre Rm!ccc Rm!ccc et pLSN6ccc. Cette suggérerait plutôt que les différentes
pLSN6ccc et différents
fréquences de recombinaison observées entre p2313ccc représentent principalement les taux avec lesquels les deux molécules persistent dans les cellules de mammifères.
Sur 57 recombinants RmI-p2313 de formes sept possédaient une jonction homologue distinctes,
p2313-RmI homologues
issue d'une recombinaison entre les séquences des deux molécules. Les 50 autres recombinants ne possédaient que des jonctions non-homologues entre les deux partenaires. Donc, le rapport des recombinants homologues versus non-homologues était de sept sur 50, soit de 0,14. Avec une homologie de 2313 pb. versus les 191 pb. entre pLSN6 et RmI, la fréquence de recombinaison homologue versus non-homologue a au moins quadruplé et atteint un niveau assez élevé pour que les deux mécanismes puissent être détectés dans un mème essai. La longueur de l'homologie affecte donc le type de mécanisme de recombinaison d'une cellule. Notre essai intermoléculaire est ainsi en accord avec les résultats déjà publiés sur la recombinaison intramoléculaire où une augmentation de la longueur d'homologie augmente la fréquence de recombinaison homologue <voir
introduction). Il est à noter que malgré les 2313 pb. d'homologie entre RmI et p2313, le mécanisme de recombinaison dominant demeure néanmoins non-homologue.
entre en A. Six des p2313ccc Ils 25 sept recombinants homologues obtenus
et Rmiccc sont illustrés à la figure 5
possèdent tous une délétion plus ou moins
importante si on les compare à un recombinant homologue
réciproque <pSBD176> issu d'un seul événement de
recombinaison réciproque entre p2313 et RmI. De plus,
ils ne possèdent qu'une seule jonction homologue
intacte. Le septième recombinant a été omis puisque sa
formation impliqua en plus d'une délétion, une
insertion de séquences supplémentaires de RmI. - Sa structure en est par le fait même plus complexe. Une étude comparative détaillée des recombinants homologues issus de tous les essais entre RmI et p2313 sera élaborée plus loin (voir discussion).
IV. Etude de la réplication
La molécule RmI contient un génome mutant de Py
qui la
<Bourg aux
rend thermosensible pour la réplication
et al 1982>. La température de 33°C est
permissive pour la réplication de RmI. L'amplification de toute molécule recombinante entre RmI et un plasmide
est donc possible à cette température à condition que
la molécule possède l'origine de réplication de Polyome
provenant de RmI. A 39oC cependant, RmI ne peut se
répliquer et par conséquent les formes recombinantes issues d'un événement entre RmI et pLSN6 ou p2313 ne
s'amplifieront pas. Un certain nombre d'essais de
cotransfection ont été tentés à 39oc, selon deux
conditions expérimentales différentes. En un premier
temps, la cotransfection a été effectuée à 39oc et
les cellules maintenues à cette température pour les 24
26
FIGURE 5. REGIONS DELETEES DES RECOMBINANTS HOMOLOGUES ENTRE p2313 ET RmI
Les cartes circulaires représentent un
recombinant intermoléculaire homologue réciproque entre
p2313 et RmI. HOM est l'homologie de 2313 pb. part~gée
par les deux molécules parentales. Les traits simples correspondent aux séquences plasmidiques, les traits noirs aux séquences de Py et les traits clairs aux
séquences cellulaires de souris. Ori des séquences
virales représente l'origine de réplication de Py. Ori et ampr des
réplication
résistance à
séquences plasmidiques sont l'origine de
procaryotique ColE1 et le gène de
l'ampicilline, respectivement. ê..91..II et
Sal1 sont les sites de restriction utilisés pour
linéariser les molécules parentales avant 1 'essai de recombinaison.
Pour chaque recombinant, seules sont illustrées
les séquences qui lui sont délétées par rapport à un
recombinant homologue réciproque. Les traits
pointillés représentent les régions d'incertitude des délétions.
En D, 0176 est le recombinant pSBD176 n'ayant
subie aucune délétion. A> p2313ccc et Rmiccc, B>
p2313/fül.!_II et Rmiccc, C> p2313/Sal1 et Rmiccc, D>
_,..,.~o.~- ~oil
B
D
E
26
33oC pour 24 heures subséquentes.
27 Selon un second protocole, la transfection a aussi été effectuée à
39oC mais les cellules ont été maintenues à cette température pendant une période de cinq heures après transfection suivie d'une incubation subséquente de 43 heures à 33oC. Les extractions selon Hirt ont été effectuées après 48 heures d'incubation dans les deux cas, tout comme pour les essais effectués exclusivement à 33oC.
pLSN6 et RmI ont été cotransfectés selon les conditions d'incubation de 24 heures â 39oc suivies de 24 heures â 33oc <tableau 2, en a>. Les extractions de l'ADN extrachromosomique de deux cotransfections indépendantes ont été utilisées dans six transformations bactériennes. Les 5530 colonies transformantes obtenues ont été hybridées dans les mêmes conditions que celles décrites précédemment pour les essais entre pLSN6 et RmI à 33oc du tableau 1
<repris au obtenues à Py, d'où 39oc est une baisse moins 15 Elle peut
tableau 2, en b>. Aucune des colonies 39oc n'a cependant hybridé avec la sonde la fréquence de recombinaison observée à
inférieure à 0,02ï.. Cette valeur représente dans l'efficacité de recombinaison d'au fois si on la compare au même essai à 33oc.
s'expliquer de deux façons: soit que l'amplification est nécessaire à la récupération des recombinants ou qu'en absence de réplication, RmI lui-même est moins disponible à la recombinaison. Ce point sera discuté plus loin avec RmI et p2313.
Des essais entre RmI et p2313 ont été effectués
à 39oc pour les mêmes raisons que celles citées pour RmI et pLSN6. Dans un premier temps, p2313 et RmI ont été cotransfectés selon une première période
29
d'incubation de 24 heures à 39oc suivie de 24 heures
à 33oc <tableau 2, en c>. A partir de trois essais distincts de cotransfections, 14 transformations bactériennes ont permis d'obtenir 1700 colonies transformantes. Un certain nombre d'entre elles ont donné un signal positif lors de l'hybridation quoique le taux de recombinaison de 0,176/. obtenu à 39oc est presque 30 fois inférieur au 5,2ï. obtenu lors d'essais analogues à 33oC (données du tableau 1 reprises au tableau 2, en e>.
Dans un second temps, les molécules RmI et p2313 ont été cotransfectées à 39oc et les cellules maintenues à cette température pendant cinq heures après transfection et incubées pendant 43 heures subséquentes à 33oc <tableau 2, end). Cinq essais de transformation bactérienne ont été réalisés dans ces conditions à partir de deux cotransfections distinctes et 1370 colonies transformantes ont été obtenues. Le pourcentage de recombinants donnant un signal positif lors de l'hybridation était de 5,1ï., valeur comparable au 5,2ï. obtenu lors d'essais à 330C pour l'étendue du 48 heures d'incubation après transfection.
Dans le cas des essais entre pLSN6 et RmI à 39oc et à 33oc, le nombre de colonies transformantes récupérées par essai n'a pas diminué en absence de réplication de RmI: 922 et 819 respectivement <tableau 2, en a et b). Dans le cas des deux protocoles de cotransfections de p2313 et RmI à
39oc, le nombre de transformants obtenus par transformation bactérienne est même plus élevé qu'à
330~ soit de 121 C39oC pendant 24 heures> et de
274 C39oC pendant cinq heures> versus 83 colonies par essai à 33oC pendant 48 heures <tableau 2, en c,d et
TABLEAU 2. ESSAIS DE RECOMBINAISON EN ABSENCE DE REPLICATION ESSAIS a b 1 c d 1 e MOLECULES COTRANSFECTEES 3 Rmiccc x pLSN6ccc id Rmiccc x p2313ccc id id TEMPS D'INCUBATION 3 39°c - 33°c 24 h
-
24 h 0 h-
48 h 24 h-
24 h 5 h - 43 h 0 h - 48 h1) Ces essais sont aussi représentés au Tableau 1
NOMBRE COLONIES D'ESSAIS 4 TRANSFORMANTES 5 6 5 530 19 15 569 14 1 700 5 1 370 14 1 162 TRANSFORMANTS PAR ESSAI 922 819 121 274 83 % DE RECOMBINANTS 6
<
0,02% 0,3% 0,176% 5,1% 5,2%2) ccc indique l'ADN sous forme superhélicoldale.
3) Les 48 heures d'incubation après transfection ont été réparties selon une première incubation à 39°c, suivie d'une incubation subséquente
à
33°c.4) Nombre de transformations bactériennes. 5) Nombre de colonies bactériennes analysées.
6) Pourcentage de transformants ayant hybridé avec la sonde Polyome.
1\)
30
e). Ainsi une diminution dans la stabilité des plasmides pLSN6 et
pas un facteur
p2313 dans les cellules 3T6 n'est pouvant expliquer la baisse l'efficacité de recombinaison à significative de
39oC.
Les variations d'efficacité de recombinaison observées dans les essais de 39oc pendant 24 heures suivis de 24 heures à 330C, autant pour p2313-RmI que pLSN6-RmI plus haut, peuvent être causées par le fait que la réplication de la molécule RmI à 33oc est intimement liée à sa recombinaison. Nous et d'autres avons déjà démontré que la réplication influence la nature
substrat <Dailey
et la fréquence de recombinaison en générant un préférentiel pour la recombinaison illégitime et al 1984; Della-Valle et al 1981; Dorsett et al 1985; Muller et al 1983; Wallenburg et al 1984). Ceci peut effectivement être le cas puisqu'une incubation de 43 ou 48 heures à 33oc <tableau 2, en d et e) résulte en des efficacités de recombinaison similaires. Une baisse significative est cependant observée dès que les cellules sont incubées à 33oc pendant seulement 24 heures, indépendamment des molécules utilisées (tableau 2, en a ou c).
Cependant, il est aussi possible que la variabilité observée dans les taux de recombinaison à
différentes températures ne soit que la conséquence de l'amplification des molécules recombinantes dans les cellules de mammifères. Or effectivement les recombinants formés à 39oc ne peuvent être amplifiés dans les
incubations ainsi passés essais faute
cellules de souris que lors de leurs subséquentes à 33oc. Ils pourraient sous les limites de détection de nos d'amplification suffisante. A l'appui,
31 certains recombinants présentés dans ce manuscript ont été amplifiés un très grand nombre de fois, puisque
nous avons récupéré jusqu'à 21 copies d'un même
recombinant. A cause du rôle déterminant que joue
l'amplification dans la récupération des recombinants dans notre essai, nous n'avons pas de preuve que la fréquence de recombinaison elle-mèfne diminue. en absence de réplication de RmI à 39oc.
V.
Effet de la linéarisationIl a été démontré que la linéarisation dans des
séquences homologues augmente de façon appréciable la recombinaison homologue de 10 jusqu'à 300 fois selon
les auteurs <voir introduction>. Mais quel effet
aurait la linéarisation sur la nature des mécanismes, i.e. le rapport des recombinaisons homologues versus
non-homologues entre deux molécules? Une série
d'essais ont été effectués lors desquels une ou les deux molécules de départ ont été linéarisées avant transfection.
Lors d'un premier essai de linéarisation, nous
avons cotransfecté à 33oC la molécule RmI circulaire
et le plasmide p2313 linéarisé dans 1 'homologie à son
site unique ~II (fig.1>.
A
partir d'ADN obtenu dec i nq cotransfections indépendantes, 30 transformations
bactériennes ont été effectuées. Seulement 98 colonies
transformantes ont été obtenues <tableau 1, en c>. Par
contre, 63% d'entre elles hybridaient avec la sonde
radioactive.
Des 62 recombinants analysés, 48
restriction distincts ont été obtenus,
patrons
dont 42
de ne
étaient donc issus
32 d'événements de recombinaison
non-homologue. Six contenaient une seule des deux
attendues d'une
jonctions homologues RmI-p2313
recombinaison homologue réciproque entre les deux
molécules, l'autre jonction ayant subie une délétion plus ou moins importante (fig.5, en B>. Tout comme lors des essais entre RmI et p2313 circulaires, aucun
recombinant homologue n'était le résultat d'un
événement de recombinaison homologue réciproque entre les deux
homologues
molécules. La proportion des recombinants
versus non-homologues est demeurée à 0,14,
valeur identique à celle obtenue lors de l'essai où les
deux molécules cotransfectées étaient circulaires.
Donc la linéarisation de p2313 dans l'homologie avant
transfection n'a eu aucun effet sur la fréquence
relative des mécanismes de recombinaison.
Cependant, le pourcentage des recombinants
totaux obtenus entre Rmiccc et p2313/êfl.!_II était
beaucoup plus élevé qu'entre Rmiccc et p2313ccc: 63/. versus 5,2ï. <tableau 1). Cette hausse importante dans le pourcentage des plasmides recombinants récupérés peut s'expliquer par des facteurs autres que l'effet de la linéarisation sur la recombinaison. En effet, un
plasmide linéaire ne transforme pas efficacement ~.coli
<Cohen et al 1972). Selon cette hypothèse, le nombre de transformants obtenus par essai devrait chuter. Nous avons effectivement observé une telle baisse de plus de 10 fois pour tous les essais où le plasmide est linéaire au moment de la transfection. De plus, comme nous le verrons plus loin en linéarisant RmI plutôt que
p2313, le pourcentage des plasmides recombinants
récupérés est rétabli à une valeur comparable à celle
33 Nous avons effectué un essai où RmI circulaire est cotransfecté à 33oc avec p2313 linéarisé à son site unique SalI, à 795 pb. à l'extérieur de l'homologie Cfig.1>. Il s'agissait ainsi de vérifier l'effet sur la recombinaison d'une coupure double-brin
à l'extérieur de l'homologie. Dans ce cas, l'homologie de 2313 pb. demeure ininterrompue et les extrémités libres générées n'ont pas d'homologie avec RmI. De trois cotransfections distinctes, 15 transformations bactériennes ont été effectuées et 34 colonies transformantes ont été obtenues <tableau 1, en d). De ces transformants, 29 étaient des recombinants, dont 26 présentaient des patrons de restriction distincts. Le pourcentage de recombinaison mesuré est de 85%, valeur du même ordre de grandeur que celle obtenue lors de la linéarisation de p2313 par ê.9.!_II dans l'homologie. Seulement deux recombinants sur les 26 possédaient une jonction homologue entre RmI et p2313, la seconde jonction de ces molécules ayant une fois de plus subie une délétion Cfig.5, en C>. Les 24 autres recombinants ne possédaient que des jonctions non-h o mologues. Le rapport des mécanismes homologues versus non-homologues était de 0,08, ce qui est un peu plus faible que la valeur 0,14 obtenue lorque p2313 était linéarisé dans l ' homologie ou que les deux molécules utilisées étaient circulaires.
Nous pouvons donc conclure que la linéarisation du plasmide dans l'homologie ou à l'extérieur de l'homologie ne résulte pas en une augmentation significative du rapport des recombinaisons homologues versus non-homologues dans nos essais.
Un essai réciproque a été effectué dans lequel p2313 circulaire a été cotransfecté avec RmI linéarisé
34
dans l'homologie à son site unique ~II, analogue au
site ~II de p2313 à 33oC Cfig.1>. L'ADN extrait de
deux transfections indépendantes a été utilisé dans 13
transformations bactériennes et 458 colonies
transformantes ont été obtenues et analysées <tableau 1, en e). Le nombre de transformants hybridant avec la sonde Py était de 10,5Y.. Cette valeur est un peu plus élevée que le 5,2% obtenu lorsque les deux molécules cotransfectées étaient circulaires.
Des 46 recombinants obtenus, 22 présentaient des patrons · distincts dont 14 ne contenaient que des
jonctions RmI-p2313 non-homologues. Sept des huit
autres recombinants possédaient une des deux jonctions homologues entre RmI et p2313, l'autre jonction ayant
subie une délétion plus ou moins grande. Un
recombinant possédait les deux jonctions homologues et la structure attendue d'une recombinaison homologue réciproque entre
et fig. 5, en type isolé lors manuscript.
les deux partenaires: pSBD176 (fig.4
D>. Il est le seul recombinant de ce
des nombreux essais présentés dans ce La linéarisation de RmI n'a pas eu d'effet important sur la fréquence totale de recombinaison dans
cet essai. Cependant, le rapport de la recombinaison
homologue versus non-homologue a quadruplé, passant de 0,14 dans le cas où p2313 seul était linéarisé ou que
les deux molécules étaient circulaires à 0,57 lorsque
RmI était linéarisé dans l'homologie. Puisque cette hausse n'avait pas été observée lorsque p2313 était
linéarisé, nous ne pouvons pas associer cet effet à la
linéarisation comme telle de 1 'une des deux molécules. Cependant, une fois linéarisée, la réplication de RmI
35 l'état réplicatif de RmI et non sa linéarisation comme
telle qui modifie le rapport des recombinaisons
homologues versus non-homologues dans cet essai. Il reste que malgré la hausse du rapport des événements
homologues versus non-homologues, la recombinaison
non-homologue demeure toutefois le mécanisme dominant dans cet essai.
Lors de l'essai suivant, RmI et p2313 ont tous
deux été linéarisés à des sites asymétriques: RmI à
son site unique SalI et p2313 à son site unique l!gl_II.
Le SalI de RmI est à la limite du 2313 pb. d'homologie
entre les · deux molécules alors que le ~II de p2313
est situé dans l'homologie <fig.U. Une fois
linéarisées, les deux molécules partagent 1278 pb.
d'homologie aux extrémités. A partir de trois
cotransfections indépendantes, 63 transformants ont été isolés;
en f). patrons
94/. hybridaient avec la sonde Py <tableau 1, Ces 59 plasmides recombinants représentaient 42
de restriction distincts, dont 25 ne
possédaient que des jonctions non-homologues. Les 17 autres possédaient l'une des deux jonctions homologues, l'autre jonction ayant subi des délétions plus ou moins
importantes. Le rapport des recombinaisons homologues
versus non-homologues était de 0,68, valeur similaire
au 0,57 observé lorsque RmI seul avait été linéarisé à
ê.g!_I I. La linéarisation des deux partenaires n'a donc
pas modifié davantage le rapport des deux mécanismes de
recombinaison. Il est intéressant de noter que tous
les recombinants homologues isolés possédaient la même
jonction homologue. De plus, la jonction réciproque
avait délété au moins les séquences homologues
s'étendant de ê.g!_II à SalI dans tous les cas. A