TWEAK
Un nouveau régulateur de la ribonucléase III Dicer
Mémoire
Raphaël Matusiak
Maîtrise en biologie cellulaire et moléculaire
Maître ès sciences (M.Sc.)
Québec, Canada
III
Résumé
La répression de la traduction des ARN messagers (ARNm) par les microARN est un processus qui régule environ 60% de tous les gènes chez l’humain et module la quasi-totalité des processus biologiques cellulaires. La ribonucléase Dicer étant l’enzyme responsable de la conversion des précurseurs de microARN en microARN matures, nous nous attendons à ce que le maintien de l’homéostasie cellulaire dépende d’une régulation fine des niveaux d’expression et de l’activité enzymatique de Dicer.
L’utilisation du double-hybride chez la levure nous a permis d’identifier la protéine Tumor necrosis factor (TNF)-like weak inducer of apoptosis (TWEAK), comme un partenaire potentiel de la forme humaine de Dicer. TWEAK est un membre de la famille des TNF qui a été découvert récemment et qui est impliqué dans de nombreux processus biologiques, dont l’inflammation. Produit majoritairement par les leucocytes, son expression protéique est altérée dans de nombreuses maladies, dont l’arthrite rhumatoïde.
L’objectif de notre étude vise donc à déterminer la nature, l’implication et le rôle de l’interaction entre Dicer et TWEAK dans le processus inflammatoire. Nous avons confirmé l’interaction Dicer-TWEAK par co-immunoprécipitation et leur colocalisation par immunofluorescence dans la lignée cellulaire HEK293. Nous avons également documenté un effet inhibiteur de TWEAK sur la capacité enzymatique de Dicer à produire des microARN matures in vitro et dans les cellules HEK293.
Cette étude permet de mieux comprendre les mécanismes régulatoires de l’activité de Dicer et de l’expression des microARN dans le processus inflammatoire.
V
Table des matières
Résumé ... III Abréviations ... VII Liste des figures ... IX Remerciements ... XI Avant-propos – rôles et contributions ... XIII
1. Introduction ... 1
1.1. Les microARN ... 1
1.1.1.Découverte des microARN ... 1
1.1.2.Biogenèse des microARN ... 1
1.1.2.1. La transcription des gènes de microARN ... 1
1.1.2.2. Conversion des pri-microARN en pré-microARN ... 2
1.1.2.3. Conversion des pré-microARN en microARN matures ... 2
1.1.2.4. Le complexe miRNP ... 6
1.1.3.Fonction des microARN ... 7
1.1.3.1. Liaison du microARN à l’ARNm cible ... 7
1.1.3.2. Répression et dégradation de l’ARNm ... 8
1.1.4.Régulation des microARN ... 9
1.1.4.1. Régulation du complexe Microprocesseur Drosha-DGCR8 ... 9
1.1.4.2. Régulation du complexe de maturation Dicer-TRBP ... 11
1.2. La protéine TWEAK ... 12 1.2.1.La superfamille des TNF ... 12 1.2.1.1. Les ligands ... 12 1.2.1.2. Les récepteurs ... 13 1.2.1.3. Rôle des TNF ... 14 1.2.2.TWEAK ... 15 1.2.2.1. Caractéristiques ... 15
1.2.2.2. Expression et synthèse de TWEAK ... 15
1.2.3.Fonction de TWEAK ... 16
VI
1.2.3.2. L’action de TWEAK via CD163 ... 17
2. Raisonnement ... 19
3. Hypothèses & objectifs ... 21
4. Matériel et méthodes ... 23
4.1. Culture cellulaire ... 23
4.2. Isolation et différenciation de monocytes humains ... 23
4.3. Extraction protéique ... 23
4.4. Co-immunoprécipitation ... 24
4.5. Immunobuvardage ... 24
4.6. Immunofluorescence ... 25
4.7. Essai Dicer ... 25
4.8. Essai de gènes rapporteurs ... 26
4.9. Détection et quantification des microARN par qPCR ... 26
4.10. Extraction d’ARN et migration sur gel d’agarose ... 27
5. Résultats ... 29
5.1. Dicer et TWEAK font partie du même complexe protéique ... 29
5.2. Les protéines Dicer et TWEAK colocalisent dans les cellules humaines ... 31
5.3. Effet de TWEAK sur l’activité enzymatique de Dicer ... 32
5.4. Effet de TWEAK sur le fonctionnement de la voie des microARN ... 34
5.5. Effet de TWEAK sur les niveaux de miR-223 ... 35
5.6. La protéine TWEAK soluble est internalisée par les macrophages humains ... 37
5.7. La protéine TWEAK soluble augmente l’expression de miR-146a dans les cellules THP-1. ... 38
5.8. La protéine TWEAK soluble régule l’expression du TNF-α ... 41
6. Discussion ... 43
VII
Abréviations
5LO 5-Lipoxygénase
ADN Acide désoxyribonucléique
ADNc ADN codant
Ago Argonaute
ARE Adenylate-uridylate-rich elements
ARN Acide ribonucléique
ARNdb Acide ribonucléique double brin
ARNm ARN messager
C. elegans Caenorhabditis elegans
CCR4 C-C chemokine receptor type 4 CD Cluster of differentiation
CHUQ CHU de Québec
CLIMP-63 Cytoskeleton-linking membrane protein of 63 kDa CRCHUQ Centre de recherche du CHU de Québec
DCP1/DCP2 mRNA-decapping enzyme 1 and 2 DGCR8 DiGeorge syndrome critical region 8 DMEM Dulbecco's modified Eagle medium dsRBD Double stranded RNA binding domain DUF283 Domain of unknown function 283
EGF Epidermal growth factor
ERK Extracellular signal-regulated kinases Fn14 Fibroblast growth factor-inducible 14
HEK Human Embryonic Kidney
hnRNP Heterogeneous nuclear ribonucleoproteins
HSP Heat Shock Protein
IFN Interferon
IL Interleukine
IP Immunoprécipitation
JNK c-Jun N-terminal kinases
KSRP KH-type splicing regulatory protein
LPS Lipopolysaccharide
LT-α Lymphotoxin-alpha
MAPK Mitogen-activated protein kinase NF-κB Nuclear factor-kappa B
NK Natural Killer
NLS Nuclear Localization signal
VIII
Nt Nucléotides
NTR Non traduite
PABP 1 Poly(A)-binding protein PACT Protein kinase RNA activator
PAGE Polyacrylamide Gel Electrophoresis
PAZ Piwi/Argonaute/Zwille
p-bodies Processing bodies
PBS Phosphate buffered saline PCR Polymerase chain reaction
PFA Paraformaldéhyde
PMA Phorbol myristate acetate Pré-microARN Précurseur de microARN Pri-microARN MicroARN primaire PVDF Polyvinyl difluoride
RANKL Receptor activator of nuclear factor kappa-B ligand RISC RNA-induced silencing complex
RPMI Roswell Park Memorial institute medium S. pombe Schizosaccharomyces pombe
SDS Sodium Dodecyl Sulfate
TBS Tris-buffered saline
TDP-43 TAR DNA-binding protein 43
TNF Tumor necrosis factor
TNF-R Tumor necrosis factor receptor
TNFSF12 Tumor necrosis factor ligand superfamily member 12 TNRC 6A Trinucleotide repeat containing protein 6A
TRAIL Tumor-necrosis-factor related apoptosis inducing ligand
TRBP TAR RNA Binding protein
TWEAK Tumor necrosis factor-like weak inducer of apoptosis VEGF Vascular endothelial growth factor
IX
Liste des figures
Figure 1. Biogenèse des microARN ... 3
Figure 2. Structure et domaines de la ribonucléase Dicer.. ... 6
Figure 3. Liaison d’un microARN à un ARNm cible ... 8
Figure 4. GW182 contrôle la répression et la dégradation des ARNm ... 10
Figure 5. Régulation de la maturation des microARN primaires (pri-microARN) ... 11
Figure 6. Ligands et récepteurs de la superfamille des TNF. ... 14
Figure 7. Voies de signalisation induitent par TWEAK/Fn14 ... 17
Figure 8. La protéine TWEAK interagit avec le dsRBD de Dicer ... 19
Figure 9. Dicer et TWEAK font partie d’un même complexe protéique ... 30
Figure 10. Dicer et TWEAK colocalisent dans la région périnucléaire des cellules humaines ... 31
Figure 11. FLAG-TWEAK inhibe la capacité de synthèse de microARN matures par Dicer. ... 33
Figure 12. L’expression de FLAG-TWEAK altère le fonctionnement de la voie des microARN ... 35
Figure 13. L’expression de FLAG-TWEAK diminue la synthèse de miR-223 mature ... 36
Figure 14. La protéine sTWEAK est internalisée par les macrophages primaires humains. 38 Figure 15. La protéine sTWEAK altère l’expression de miR-146a et miR-132 ... 40
XI
Remerciements
Tout d’abord, je tiens à remercier le Dr Patrick Provost de m’avoir accueilli dans son laboratoire et de m’avoir ainsi permis de mener à bien ce projet. Je tiens à le remercier également de ses conseils et de son temps dans la rédaction de ce mémoire.
Je tiens à remercier tous les membres du laboratoire. Merci à Geneviève Pépin, de m’avoir aidé, encadré et conseillé. Merci pour toutes nos conversations, qui malgré nos désaccords, ont toujours été constructives. Sa patience, son écoute et son ouverture d’esprit m’aura permis d’apprendre énormément de la vie, scientifique ou non. Merci à Patricia Landry, dont la patience et la connaissance m’ont permis de réaliser beaucoup de choses au laboratoire. Merci pour tes conseils et nos moments « geek », qui me manquent déjà beaucoup. Je tiens aussi à remercier Aurélie Corduan, pour ses conseils, sa compréhension et sa bonne humeur communicative. Je te souhaite beaucoup de bonheur pour l’heureux évènement qui arrive (je parie sur une fille). Merci aussi à Benoit Laffont, dont les conseils ont été précieux mais également pour toutes les franches rigolades. Merci pour tous ces bons moments autour de Homeland, Breaking Bad et Dexter. Enfin, merci à Hélène Plé pour toutes nos discussions et rigolades. Votre bonne humeur et enthousiasme à tous ont été communicatifs et je vous souhaite plein de réussite dans vos projets futurs
Je tiens également à remercier William Sibran, Mickaël Di-Luoffo et Caroline Daems pour tous les moments partagés et avec qui j’ai découvert le monde canadien.
Merci à Olivia Gavard, avec qui j’ai partagé cette expérience à l’étranger, qui m’a aidé et conseillé, et sans qui tout ceci n’aurait jamais été possible.
Enfin, merci à mes parents, qui ont toujours été là, malgré la distance et m’ont toujours épaulé et encouragé dans mes projets.
XIII
Avant-propos – rôles et contributions
Les résultats présentés dans ce mémoire ont été obtenus grâce à la collaboration de plusieurs membres de l’équipe du Dr. Patrick Provost, dont le rôle est précisé à l’intérieur de cette section.
Dr. Oscar Peralta-Zaragoza (stagiaire post-doctoral) :
- Figure 8: Analyse du criblage de la banque d’ADNc par double hybride Geneviève Pépin (étudiante au doctorat) :
- Figure 9: Elaboration et optimisation du protocole de co-immunoprécipitation - Figure 10: Réalisation de la microscopie par immunofluorescence
- Figure 12: Réalisation de l’essai de gène rapporteur Luciférase - Coordination et supervision globale du projet
Patricia Landry (professionnelle de recherche) :
1
1. Introduction
1.1. Les microARN
Les microARN sont de petits ARN, de 19 à 24 nucléotides (nt), qui se lient de manière partiellement complémentaire à un ARN messager (ARNm) cible. Le plus souvent, le microARN se lie à un site de liaison situé dans la région 3’ non-traduite (NTR), induisant la répression de la traduction de l’ARNm [1].
1.1.1. Découverte des microARN
C’est en 1993 que le premier microARN est découvert, chez le nématode Caenorhabditis elegans (C. elegans), par l’équipe du Dr Victor Ambros. Son équipe a identifié un ARN non-codant long de 22 nt, appelé Lin-4, capable de se lier sur la région 3’ NTR de l’ARNm Lin-14 [2]. Il a été découvert que l’implication de ce microARN dans le développement du nématode, par la régulation négative du gène Lin-14, est essentielle pour permettre le passage du stade larvaire L1 au stade L2 [2]. Il est ensuite apparu que les microARN étaient présents chez tous les métazoaires, notamment chez l’humain. Par exemple, miR-15 et miR-16 sont détectés chez l’humain, la souris, le poisson et faiblement la grenouille [3]. On estime que les microARN seraient impliqués dans la régulation d’environ 60% des gènes chez l’humain, suggérant un lien étroit entre la dérégulation des niveaux et/ou de la fonction des microARN et les maladies humaines [2-4].
1.1.2. Biogenèse des microARN
1.1.2.1. La transcription des gènes de microARN
L’information contenue dans les microARN est emmagasinée dans le génome de la cellule hôte sous forme de gènes de microARN. Plusieurs étapes de maturation sont nécessaires pour l’obtention de microARN matures et fonctionnels. Le processus débute par la transcription de ces gènes de microARN, qui possèdent leur propre région promotrice et séquence de terminaison. Ce mécanisme est régulé par des facteurs de transcription, de la même manière que les gènes codants pour des protéines. Ces gènes de microARN sont transcrits principalement par l’ARN polymérase II en microARN primaires (pri-microARN) et possèdent ainsi une coiffe 7-méthyl-guanosine en 5’ et une queue polyadénylée en 3’ (Figure 1) [5]. Ces microARN primaires, dont la taille peut atteindre
2
plusieurs milliers de nucléotides, peuvent également provenir du clivage d’introns résultants de l’épissage des ARNm [6]. Ces introns, présentant une structure en tige-boucle, subissent une première étape de maturation dans le noyau de la cellule (Figure 1). Il est intéressant de noter que certains microARN peuvent provenir d’introns très courts possédant les caractéristiques structurales des précurseurs de microARN (pré-microARN) [7]. Dans ce cas, ils sont exportés vers le cytoplasme via l’Exportin-5, puis incorporés au complexe de maturation contenant la ribonucléase Dicer. Quelque soit leur origine, les pri-microARN subissent deux étapes de maturation : la conversion des pri-pri-microARN en pré-microARN, puis la conversion des pré-microARN en microARN matures.
1.1.2.2. Conversion des pri-microARN en pré-microARN
Les pri-microARN possèdent une structure en tige-boucle qui est reconnue par le complexe Microprocesseur (Figure 1). Ce complexe est composé de la ribonucléase III Drosha et de la protéine DiGeorge syndrome critical region gene 8 (DGCR8) qui agit comme cofacteur [8, 9]. DGCR8 se fixe à la base d’une tige-boucle du pri-microARN et, grâce à ses deux domaines de liaisons à l’ARNdb, aide Drosha à se positionner sur son substrat. Drosha va cliver la tige d’ARN double brin au niveau du 11ème nucléotide à partir de la base de la tige-boucle, laissant une protubérance de 2 nt à l’extrémité 3’. Il en résulte alors une structure en tige-boucle d’ARN, d’environ 60-70 nt, appelé pré-microARN (Figure 1). Celui-ci s’associe au complexe Exportin-5-Ran•GTP et est exporté vers le cytoplasme, où il est pris en charge par le complexe de maturation des pré-microARN [10, 11].
1.1.2.3. Conversion des pré-microARN en microARN matures
Dans le cytoplasme, le pré-microARN est ensuite incorporé au complexe de maturation des pré-microARN, composé de la ribonucléase III Dicer et de son cofacteur, soit TAR RNA Binding protein (TRBP) ou protein kinase RNA activator (PACT) [12]. Au sein de ce complexe, Dicer clive le pré-microARN à la base de la boucle pour donner naissance à un duplexe de microARN (Figure 1) [13].
3 Figure 1. Biogenèse des microARN. Les gènes de microARN sont transcrits par l’ARN
polymérase II ou III (RNA pol II ou III) en microARN primaire (microARN). Les pri-microARN peuvent aussi provenir des introns (sens ou antisens) d’un ARNm. Le pri-pri-microARN est ensuite clivé par le complexe Microprocesseur, composé de la ribonucléase III Drosha et de la protéine DiGeorge syndrome critical region 8 (DGCR8), pour générer un précurseur de microARN (pré-microARN). Ce précurseur est exporté vers le cytoplasme par l’Exportin-5, puis clivé par la ribonucléase III Dicer, qui est assistée de son cofacteur TAR RNA binding protein (TRBP). Le clivage du pré-microARN par Dicer résulte en un duplexe de microARN matures, composé de deux brins asymétriques nommés 5p et 3p. Le duplexe de microARN mature est ensuite incorporé dans un complexe ribonucléoprotéique effecteur, au sein duquel un brin du duplexe de microARN sera clivé et dégradé. Ce complexe contient la protéine Argonaute 2 (Ago2), qui est responsable de l’activité catalytique du complexe. Guidé par le microARN mature, le complexe effecteur se lie aux ARNm cibles, principalement pour réprimer leur traduction. Illustration tirée de Winter J, et al, 2009 [14].
4
1.1.2.3.1. La ribonucléase III Dicer
Dicer est une endoribonucléase d’environ 217 kDa que l’on retrouve chez de nombreuses espèces, et dont la forme humaine a été identifiée et caractérisée en 2002 [4, 15].
1.1.2.3.1.1. Structure de Dicer
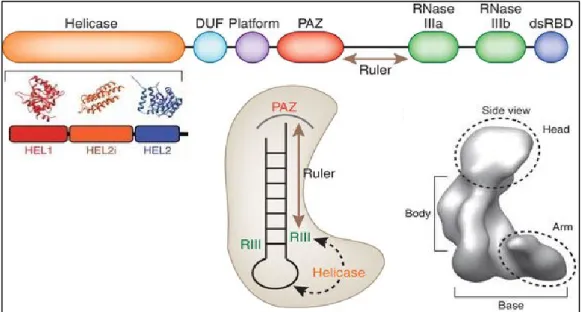
Cette enzyme présente une structure complexe capable d’ancrer et de cliver le pré-microARN grâce à la participation de plusieurs domaines (Figure 2). De l’extrémité N-terminale à C-N-terminale, on retrouve :
Un domaine hélicase, qui permet l’interaction de Dicer avec certains cofacteurs, comme TRBP et PACT (Figure 2) [16]. De plus, ce domaine intervient dans la sélection du brin guide et dans l’auto-inhibition de Dicer [17] ;
Un domaine, dont la fonction est encore inconnue, appelé domain of unknown function 283 (DUF283) (Figure 2). Ce domaine possède des similitudes structurales avec les domaines de liaison à l’ARN double brin [18] ;
Un domaine platform, qui oriente et facilite l’ancrage du pré-microARN grâce à sa teneur élevée en résidus chargés positivement (Figure 2) [19] ;
Un domaine PIWI/Argonaute/Zwille (PAZ), qui lie l’extrémité protubérante de 2 nt à l’extrémité 3’ des pré-microARN et contribue ainsi au positionnement du centre catalytique, formé d’un dimère intramoléculaire des domaines RNase IIIa et IIIb C-terminaux, sur le pré-microARN (Figure 2) [19] ;
Deux domaines RNases (IIIa et IIIb), qui se positionnent à ~22 nucléotides du domaine PAZ et clivent le pré-microARN à la base de la boucle (Figure 2). Le domaine RNase IIIa clive le brin 3p, alors que le domaine RNase IIIb clive le brin 5p [13] ;
Un domaine double stranded RNA-binding protein (dsRBD), en position C-terminale, qui renforce l’ancrage du précurseur à la ribonucléase pendant le clivage (Figure 2). Des études ont montré que, sans ce domaine, la quantité de pré-microARN clivé était inférieure [17, 18]. Au sein du laboratoire, une étude a montré que la protéine 5-Lipoxygenase (5LO) pouvait interagir avec le dsRBD
5 de Dicer et réguler son activité. Toutefois, le rôle exact du dsRBD demeure obscur.
Des analyses structurales de la protéine Dicer ont permis d’élucider les mécanismes de reconnaissance et de clivage des pré-microARN en microARN matures. Ces études ont révélées que le site de clivage du précurseur est déterminé par la distance entre le domaine PAZ et le domaine catalytique de la ribonucléase, lui-même formé par la dimérisation de ses deux domaines RNases [19]. C’est ainsi que la taille des microARN variera entre les espèces, selon la structure de l’enzyme Dicer et la configuration du domaine PAZ [19]. La protubérance de 2 nt à l’extrémité 3’ du précurseur va s’ancrer dans une pochette formée par le domaine PAZ de Dicer. Chez l’humain, le duplexe de microARN généré par Dicer, partiellement complémentaire et d’environ 22 nt, est ensuite transféré au complexe microRNA-containing ribonucleoprotein complex (miRNP).
1.1.2.3.1.2. Rôle et fonction de Dicer
Bien que le rôle principal de Dicer soit le clivage des pré-microARN en microARN matures, d’autres fonctions ont également été rapportées. Chez Schizosaccharomyces pombe (S. pombe), Dicer (Dcr1) est retrouvé dans le noyau et est impliqué dans la formation de l’hétérochromatine [20]. En effet, la ribonucléase va cliver les ARNdb provenant des séquences répétitives centromériques en small interfering RNA (siRNA) qui, une fois associés à un complexe contenant Ago1, s’appareillent à la chromatine et influencent sa conformation [20]. Chez la drosophile, la ribonucléase Dcr2 a également été localisée au niveau du noyau, où elle interagit avec l’euchromatine et peut réguler le complexe de transcription [21]. En effet, il a été montré que la perte de fonction de Dcr2 induit des erreurs dans le positionnement de l’ARN polymérase II sur les régions promotrices de gènes et perturbe la transcription [21]. Chez l’homme, des études ont montrés que Dicer pouvait être présente dans le noyau des cellules et lier l’ADN ribosomal [22]. Toutefois, le rôle de Dicer dans ce phénomène reste obscur et demande des études plus approfondies [22]. Egalement, il a été montré que Dicer pouvait cliver des ARN double brin aboutissant dans le noyau dans les cellules HEK293 [23]. De récentes études ont montré que le dsRBD de Dicer contenait un nuclear localization signal (NLS), pouvant avoir un rôle dans l’importation de Dicer au noyau via des importines [24]. Ces données
6
corroborent celles d’une étude chez S. pombe montrant que le dsRBD serait impliqué dans la séquestration de Dcr1 dans le noyau [25].
Figure 2. Structure et domaines de la ribonucléase Dicer. Dicer est composé de plusieurs
domaines : (1) Un domaine hélicase, qui se lie à la boucle du précurseur et intervient dans la sélection du brin guide ; (2) un domain of unknown fonction 283 (DUF 283) dont la fonction demeure méconnue ; (3) un domaine platform, qui va guider et orienter l’ancrage du précurseur ; (4) un domaine PIWI/Argonaute/Zwille (PAZ), qui lie l’extrémité 3’ protubérante du précurseur ; (5) deux domaines RNases, qui vont cliver le précurseur suite à leur dimérisation ; (6) un domaine
double stranded RNA binding domain (dsRBD), qui maintient le précurseur au sein de la protéine
Dicer. La distance entre le domaine PAZ et les domaines RNases, appelé ruler dans la figure ci-dessus, permet de déterminer le site de clivage et influe sur la longueur des microARN matures synthétisés. Illustration adaptée de Lau P.W, et al, 2012 et Sawh A.N, et al, 2012 [19, 26].
1.1.2.4. Le complexe miRNP
Le clivage du pré-microARN par la ribonucléase Dicer mène à la formation d’un duplexe de microARN, dont un des brins est sélectivement incorporé à la protéine Argonaute (Ago), pour former un complexe miRNP. La structure de la protéine Ago, qui est cœur du complexe effecteur, est composée de 3 domaines : (1) un domaine N-terminal PAZ, capable de lier la protubérance de 2 nt à l’extrémité 3’ d’un microARN ; (2) un domaine central MID, qui lie l’extrémité 5’ du microARN ; (3) et un domaine C-terminal PIWI, de type RNase, responsable de l’activité endonucléasique de la protéine [27, 28]. Chez les mammifères, on compte 4 protéines Ago, parmi lesquelles on distingue Ago2,
7 dont le domaine endonucléase est fonctionnel, contrairement aux autres membres de cette famille.
C’est TRBP, le cofacteur de Dicer, qui va recruter une protéine Ago au sein de laquelle sera incorporé un brin du duplexe de microARN [29]. La sélection préférentielle du brin est déterminée par la stabilité thermodynamique de son extrémité 5’ [30]. Ainsi, Dicer et son cofacteur mesurent la stabilité des extrémités du duplexe de microARN, sélectionnent le microARN dont l’extrémité 5’ est la moins stable et déterminent l’identité du brin sélectionné (brin guide) et du brin passager. Bien qu’il soit encore méconnu, ce processus de sélection des brins fait intervenir des protéines chaperonnes, tels que les Heat Shock protein (HSP), et dépend de plusieurs paramètres, comme le type cellulaire, l’expression de TRBP et la complémentarité du duplexe [12, 31, 32]. Le brin guide est fermement ancré dans la protéine Ago, tandis que le brin passager est soit clivé et dégradé, ou dans une moindre mesure, incorporé au sein d’un complexe effecteur [33]. Dans la littérature, on distingue dorénavant les 2 microARN d’un duplexe selon leur origine et position sur le précurseur d’origine ; on parlera alors de brin 5p ou de brin 3p.
1.1.3. Fonction des microARN
La fonction principale connue des microARN est de réprimer la traduction des ARNm endogènes. Le complexe miRNP est guidé par un microARN mature pour s’assembler à un ARNm cible, où des facteurs modulent la répression, soit en séquestrant et/ou en dégradant le messager.
1.1.3.1. Liaison du microARN à l’ARNm cible
Les microARN se lient, le plus souvent, à la région 3’ NTR des ARNm cibles. Les sites de liaison sont généralement conservés entre les espèces et sont situés dans des régions peu structurées [34]. Ces sites sont souvent situés à proximité les uns des autres, et de domaines riches en A et en U [34]. Enfin, ils doivent être situés à au moins 15 nucléotides du codon STOP de la séquence codante [34]. Chez les plantes, la liaison est souvent parfaitement complémentaire, permettant ainsi le clivage du messager par Ago2. Cependant, chez les mammifères, il n’y a que très peu d’exemples de complémentarité parfaite et, le plus souvent, celle-ci est partielle (Figure 3). Le 1er nucléotide, situé en 5’ du microARN, reste ancré dans la protéine Ago et n’intervient pas dans la reconnaissance de
8
l’ARNm cible (Figure 3). Les nucléotides 2 à 8 sont, le plus souvent, parfaitement complémentaires et forment la région seed (Figure 3). Généralement, les nucléotides 9 à 11 du microARN ne sont pas complémentaires à l’ARNm cible, évitant ainsi le clivage par Ago2. L’appariement des nucléotides restant ne suit pas de règles particulières, bien qu’il permette de renforcer la liaison entre le complexe miRNP et l’ARNm cible.
Figure 3. Liaison d’un microARN à un ARNm cible. Le microARN ancré dans la protéine Ago
se lie à l’ARNm cible au niveau de l’extrémité 3’ NTR. Le premier nucléotide n’intervient pas dans l’appariement et maintient l’ancrage du microARN au complexe. Les nucléotides 2 à 8 font partie de la région seed et sont parfaitement complémentaire à l’ARNm cible. Les nucléotides 9 à 11 ne sont pas complémentaires et forment un renflement (bulge) qui prévient un clivage par Ago2. L’appariement des nucléotides suivants est variable et permet de stabiliser (ou non) la liaison entre le microARN et son ARNm cible. Les sites de liaison des microARN sont situés généralement à plus de 15 nucléotides de la séquence codante. Illustration tirée de Filipowicz W, et al, 2008 [35].
1.1.3.2. Répression et dégradation de l’ARNm
Après la liaison à l’ARNm cible, le complexe miRNP, et plus particulièrement la protéine Ago, va recruter la protéine GW repeats proteins of 182 kDA (GW182) (aussi connu sous le nom de trinucleotide containing 6A (TNRC6A)) [36]. Cette protéine exerce un rôle primordial dans la répression et la dégradation de l’ARNm (Figure 4).
La circularisation de l’ARNm assure l’initiation de sa traduction, tout en le protégeant de la dégradation. Ce lien, reliant la coiffe en 5’ et la queue poly(A) en 3’, a lieu via l’association entre le facteur d’initiation eIF4F et la protéine Poly(A) binding protein 1 (PABP1) [37]. La protéine GW182 recrutée par Ago interagit avec PABP1 de manière compétitive, réprimant la circularisation et, par conséquent, l’initiation de la traduction (Figure 4). En plus de sa fonction dans la répression traductionnelle, GW182 est impliquée dans le recrutement de complexes protéiques impliqués dans la dégradation de la queue poly(A) et dans le phénomène de decapping. Le complexe de déadénylation CCR4-Not1 va
9 dégrader la queue poly(A) de l’ARNm cible via l’action d’exonucléases, alors que la coiffe sera dégradée par le complexe enzymatique DCP1/DCP2 (Figure 4) [38, 39].
Ainsi, les ARNm cibles sont réprimés et/ou déstabilisés via des mécanismes enzymatiques activés par GW182. De récentes études ont montrés que ces deux phénomènes pourraient être consécutifs ; la répression précédant la dégradation. Ces deux phénomènes semblent se dérouler dans les processing bodies (p-bodies) impliqués dans la séquestration d’ARNm. Ces granules de nature ribonucléoprotéique colocalisent avec les corps GW contenant GW182 et contiennent les enzymes responsables du decapping, de la déadénylation et de la dégradation des ARNm [40].
1.1.4. Régulation des microARN
Les différentes étapes de maturation des microARN font intervenir plusieurs complexes protéiques qui médient la biogenèse des microARN en régulant leur maturation. Cette section décrit brièvement les protéines impliquées dans la régulation du complexe Microprocesseur Drosha-DGCR8 et, plus en détails, la régulation du complexe de maturation Dicer-TRBP.
1.1.4.1. Régulation du complexe Microprocesseur Drosha-DGCR8
Plusieurs protéines peuvent s’associer au complexe Microprocesseur Drosha-DGCR8 et ainsi influencer la maturation des pri-microARN. C’est le cas des hélicases p68 et p72, qui facilitent la maturation de certains microARN (Figure 5) [41]. Il y a également la protéine p53 qui, en s’associant à p68, agit sur la maturation de certains microARN suite à des dommages à l’ADN [14]. De la même manière, le complexe SMAD-p68 facilite la maturation de certains microARN, comme pri-miR-21 (Figure 5) [42]. Enfin, d’autres protéines interagissent, non pas avec le complexe, mais avec la boucle de certains pri-microARN en reconnaissant des motifs spécifiques. C’est le cas notamment des protéines KH-type splicing regulatory protein (KSRP) et heterogeneous nuclear ribonucleoproteins A1 (hnRNP A1), qui agissent de manière compétitive sur la maturation de pri-let-7a (Figure 5) [43].
10
Figure 4. GW182 contrôle la répression et la dégradation des ARNm. La protéine GW182
recrutée par Ago empêche la circularisation de l’ARNm et réprime ainsi la traduction des ARNm. GW182 peut également recruter des complexes enzymatiques qui participent au decapping, à la déadénylation et au clivage des ARNm cibles. Illustration tirée de Huntzinger E, et al, 2011 [44].
11 Figure 5. Régulation de la maturation des microARN primaires (pri-microARN). (a) Le
complexe Microprocesseur est composé au moins des protéines Drosha et DiGeorge syndrome
critical region 8 (DGCR8). (b) Les hélicases p68 et p72 peuvent lier la ribonucléase et son
cofacteur afin de faciliter la maturation du pri-microARN. (c) La protéine hnRNP A1 peut lier la boucle de certains pri-microARN sur une séquence spécifique et faciliter leur maturation comme, par exemple, pri-miR-18a. (d) L’hélicase p68 peut également s’associer avec la protéine SMAD, contribuant ainsi à la maturation de pri-miR-21. Illustration adaptée de Nature Cell Biology [14].
1.1.4.2. Régulation du complexe de maturation Dicer-TRBP
Parmi les protéines capables d’interagir avec le complexe de maturation contenant Dicer, on observe notamment TRBP et PACT. TRBP possède deux domaines dsRBD et un domaine medipal à l’extrémité C-terminale impliqué dans l’association avec Dicer [16]. TRBP se lie au domaine hélicase de Dicer et influence sa cinétique de clivage, en augmentant l’affinité de Dicer pour son substrat, en limitant l’auto-inhibition de la ribonucléase et en facilitant la libération du duplexe produit [17]. Il a également été montré que TRBP agit dans la sélection du brin guide et stabiliserait l’interaction du complexe de maturation avec les protéines Ago [30]. PACT ressemble beaucoup à TRBP, possède le même nombre de domaine que TRBP et agit selon le même schéma. TRBP et PACT agissent sur la cinétique de maturation des pré-microARN en microARN matures par Dicer
12
[45]. Bien que ces deux cofacteurs ne soient pas indispensables au clivage du précurseur, leur absence réduit considérablement l’activité enzymatique de Dicer. La ribonucléase Dicer peut également interagir avec la protéine cytoskeleton-linking endoplasmic reticulum (ER) membrane protein of 63 kDa (CLIMP-63) à l’intérieur du réticulum endoplasmique [46]. CLIMP-63 stabilise les niveaux d’expression de Dicer et pourrait permettre sa sécrétion à l’extérieur des cellules humaines [46]. De la même manière que pour le complexe Microprocesseur, les protéines KSRP et hnRNP A1 peuvent intervenir dans la maturation de certains microARN en se liant à la boucle du précurseur. Enfin, on peut également citer la protéine TAR DNA-binding protein-43 (TDP-43), qui peut se lier à la boucle du pri-microARN ou du pré-microARN pour réguler la maturation de certains microARN, tel que miR-132 [47].
La régulation du complexe de maturation des microARN fait l’objet de nombreuses études. Au laboratoire, le Dr Oscar Peralta-Zaragoza s’est intéressé à identifier de nouveaux partenaires protéiques de la forme humaine de Dicer. Cherchant à découvrir de nouveaux régulateurs de la biogenèse des microARN, il réalisa un criblage d’une banque d’ADNc par double hybride chez la levure à l’aide d’une forme tronquée de Dicer comme appât. C’est ainsi que tumor necrosis factor-like weak inducer of apoptosis (TWEAK), a été identifié comme partenaire protéique potentiel de Dicer. Cette cytokine, découverte dans les années ’90, fait partie de la famille des Tumor Necrosis Factor (TNF).
1.2. La protéine TWEAK
1.2.1. La superfamille des TNF
La superfamille des TNF peut être divisée en deux groupes : les ligands et les récepteurs. Les deux groupes sont retrouvés dans les cellules du système immunitaire et exercent un rôle primordial dans l’organisation et la fonction du système immunitaire [48].
1.2.1.1. Les ligands
Les ligands TNF sont au nombre de 19 et sont codés par un total de 18 gènes. Ils partagent un motif structurel trimérique commun, le « TNF homology domain », qui va permettre la liaison aux récepteurs TNF sur un domaine riche en cystéine [49]. Les ligands sont majoritairement des protéines transmembranaires de type II, c’est-à-dire ayant
13 l’extrémité N-terminale du côté intracellulaire et l’extrémité C-terminale du côté extracellulaire [50]. Ces ligands peuvent être produits autant par des cellules immunitaires, telles que les macrophages, les mastocytes, les Natural Killer (NK) et les granulocytes, que par des cellules non-immunitaires, comme les cellules endothéliales, les ostéoblastes, les fibroblastes ou les cellules musculaires lisses [51]. Les ligands de la famille des TNF possèdent une forme soluble, qui peut être obtenue par clivage protéolytique des formes membranaires. Ces formes tronquées sont relâchées dans la circulation et le milieu extracellulaire, et peuvent ainsi exercer des effets à distance sur d’autres cellules. Les ligands peuvent aussi conservés leur forme membranaire et exercer leur rôle biologique lors de contact cellule-cellule ; on parle alors de liaison juxtacrine. Parmi les ligands TNF les plus connus, notons le TNF-α, tumor-necrosis-factor related apoptosis inducing ligand (TRAIL), apoptosis-inducing ligand (APRIL), receptor activator of nuclear factor kappa-B ligand (RANKL) et TWEAK (Figure 6) [49].
1.2.1.2. Les récepteurs
Le groupe des récepteurs TNF compte 29 protéines membranaires de Type I ou III [49, 51]. La présence de domaines riche en cystéine sur la portion extracellulaire est la caractéristique principale de ces récepteurs. Le nombre de ces domaines varie de 1 à 4, et ils sont généralement composés de 6 résidus cystéine formant 3 ponts disulfures [49]. Ces récepteurs sont présents sur la majorité des cellules immunitaires et ils sont capables de générer une forme soluble par clivage protéolytique. Ces formes solubles vont être relâchées dans le sang, où ils iront se lier à leur ligand afin de moduler leur activité [49, 52]. Parmi ces récepteurs, on retrouve TNF-Receptor 1 et 2 (TNF-R1, TNF-R2), Fibroblast growth factor-inducible 14 (Fn14), Fas et CD40 (Figure 6) [49].
14
Figure 6. Ligands (en haut) et récepteurs (en bas) de la superfamille des TNF. Les ligands sont
des protéines transmembranaires de type II qui possèdent un TNF homology domain (ici en vert). Les flèches présentes sur la structure des ligands indiquent la localisation du clivage protéolytique générant la forme soluble. Les récepteurs sont des protéines transmembranaires de type I ou III. La longueur des domaines intracellulaires est indiquée pour les ligands et les récepteurs, et les rectangles rouges indiquent les Death Domain. Illustration tirée de Bodmer J.L, et al, 2002 [49].
1.2.1.3. Rôle des TNF
Les membres de la famille des TNF contrôlent et orchestrent la réponse immunitaire et l’inflammation. Le TNF-α, RANKL ou la lymphotoxin-alpha (LTα) jouent un rôle important dans la morphogenèse des organes lymphoïdes secondaires [53]. Dans la régulation de la réponse immunitaire, le CD40L et BAFF activent les lymphocytes B [54, 55] ; OX40L et CD27L activent les lymphocytes T [56] ; et RANKL active les cellules dendritiques [57]. D’autres études montrent l’implication des membres la famille des TNF dans la différentiation cellulaire et l’apoptose [51, 58]. Il va sans dire qu’une meilleure
15 compréhension des effets exercés par les TNF permettra de mieux conceptualiser la réponse immunitaire et d’identifier de nouvelles cibles thérapeutiques potentielles.
1.2.2. TWEAK
1.2.2.1. Caractéristiques
TWEAK a été découvert en 1997 par Yves Chicheportiche [50]. Alors que l’équipe travaillait sur les fonctions de l’érythropoïétine, les chercheurs ont identifié une protéine qui présentait de fortes homologies structurales avec les membres de la famille des TNF, notamment le TNF Homology Domain. Le nom Tumor necrosis factor-like weak inducer of apoptosis (TWEAK) provient de la capacité de la protéine à induire faiblement l’apoptose dans la lignée de carcinome de colon HT29 [50].
TWEAK appartient à la famille des ligands du TNF. Connu également sous les termes Tumor necrosis factor ligand superfamily member 12 (TNFSF12), d’APO3 Ligand (APO3L) ou CD255, TWEAK est codé par le gène TNFSF12, situé sur la région p13 du chromosome 17. Comme pour de nombreux membres de la famille des TNF, la séquence de l’ARNm de TWEAK contient une région riche en adénine-uracile (AU-rich element ; ARE) au niveau de sa région 3’ NTR [59]. Cette région est reconnue par des protéines pouvant lier l’ARN et dont la fonction est de promouvoir la stabilité ou la dégradation de l’ARNm [59]. Le gène TNFSF12 code pour une protéine de 249 acides aminés et l’analyse de cette séquence protéique a permis d’identifier TWEAK comme une protéine membranaire de type II. TWEAK est composé d’une séquence N-terminale hydrophile intracellulaire, d’une partie transmembranaire et d’un long domaine C-terminal extracellulaire, mesurant respectivement 18, 25 et 206 acides aminés [60]. Un site de N-glycosylation a été répertorié sur la partie extracellulaire, au niveau de l’asparagine 139 [50]. TWEAK peut subir un clivage protéolytique par l’action de la Furin, une sérine protéase qui coupe la partie C-terminale au niveau de l’arginine 93 et induit la libération de la forme soluble [61]. A ce jour, deux récepteurs sont connus pour être fixés par la cytokine, soit Fn14 et CD163 [62, 63].
1.2.2.2. Expression et synthèse de TWEAK
L’ARNm de TWEAK est exprimé et retrouvé dans de nombreux types de cellules : les cellules immunitaires, telles que les monocytes/macrophages, les cellules dendritiques,
16
les lymphocytes et les NK, mais également au sein de cellules non-immunitaires, telles que les astrocytes ou les cellules endothéliales [50, 64, 65]. Cependant, la protéine TWEAK est principalement exprimée dans les leucocytes, notamment les monocytes/macrophages, les NK et les cellules dendritiques [66, 67]. La synthèse de TWEAK dans les lymphocytes T et B ne semble pas être ubiquitaire et n’a été décelée que pendant des maladies, telles que le lupus et la sclérose en plaques [68, 69]. Bien que la régulation de l’expression de TWEAK reste encore mal connue, il semblerait que son expression soit augmentée par l’IFN-γ et le PMA, mais non par le LPS ou l’IFN-α [66, 70].
1.2.3. Fonction de TWEAK
La fonction biologique de TWEAK peut s’exercer via deux récepteurs : Fn14 ou CD163.
1.2.3.1. L’action de TWEAK via Fn14
Fn14 est le plus petit des récepteurs de la famille des TNF et représente le récepteur principal de TWEAK. C’est une protéine transmembranaire de type I de 129 acides aminés [71]. L’affinité de TWEAK pour Fn14 étant plus grande que pour CD163, la cytokine se fixera préférentiellement sur Fn14 si les deux récepteurs sont présents. On retrouve Fn14 à la surface des cellules endothéliales, des cellules épithéliales et de certaines cellules progénitrices [58, 72]. Les niveaux d’expression du récepteur Fn14 restent faibles dans les tissus sains, mais peuvent être augmentés, d’une part, par différents facteurs de croissance, comme l’epidermal growth factor (EGF) ou le vascular endothelial growth factor (VEGF), et d’autre part, par des cytokines, comme le TNF-α, l’IL-1β ou l’IFN-γ [58].
La liaison de TWEAK à Fn14 va entrainer l’homotrimérisation du récepteur. Ce signal facilite l’association de protéines TRAF (1, 2, 3, 5) avec le domaine cytoplasmique de Fn14 et, plus particulièrement, sur un motif de liaison PIEET (Figure 7) [67]. Cette voie de signalisation mène à l’activation du facteur de transcription NF-κB (Figure 7). Cette activation peut s’effectuer via la voie canonique NF-κB1, caractérisée par la phosphorylation de IκB et la translocation de l’hétérodimère p50/p65 (NF-κB1) dans le noyau, ou la voie non-canonique, caractérisée par la stabilisation de NIK et la translocation vers le noyau de l’hétérodimère P52/RelB (NF-κB2) (Figure 7) [67]. Des études ont également montrées que la voie de signalisation MAPK pouvait être stimulée, via
17 l’activation des voies ERK, JNK et/ou p38. Cette activation des voies de signalisation NF-κB ou MAPK implique de nombreux phénomènes biologiques, comme la prolifération cellulaire, la régénération tissulaire, la migration cellulaire, la survie ou encore la mort cellulaire (Figure 7) [73-77].
Figure 7. Voies de signalisation induitent par TWEAK/Fn14. TWEAK se fixe au récepteur Fn14
et induit son homotrimérisation. Cette modification structurale permet la liaison des protéines TRAF (1, 2, 3, 5) sur des motifs PIEET présents dans le domaine intracellulaire de Fn14. Cette liaison active le facteur de transcription NF-κB via la voie canonique (NF-κB1) et/ou la voie non-canonique (NF-κB2). La voie non-canonique est caractérisée par la phosphorylation d’IκB et la migration vers le noyau de p50/p65. La voie non-canonique est caractérisée par la stabilisation de
NF-κB Inducing Kinase (NIK) et la migration vers le noyau de p52/RelB. Illustration adaptée de
Burkly L.C, et al, 2011 [67].
1.2.3.2. L’action de TWEAK via CD163
CD163 est une protéine transmembranaire de type I de 1 076 acides aminés et qui appartient à la famille des scavenger receptor [78]. Tout comme son ligand, ce récepteur existe sous deux formes, une forme ancrée à la membrane et une forme clivée, soluble. Son appartenance à la famille des scavenger receptor a été établie grâce à la présence de neuf
18
domaines riches en cystéine. La localisation extracellulaire et la structure de ces domaines font de CD163 un scavenger receptor du groupe B [79]. Il est spécifique des monocytes/macrophages et sa fonction première est de lier le complexe hémoglobine-haptoglobine [80-82]. En effet, lors d’une hémolyse importante, une forte quantité d’hémoglobine peut être relarguée. Dans ce cas, cette hémoglobine sera fixée par l’haptoglobine et ce complexe sera fixé par CD163. Ainsi, en éliminant ce complexe de la circulation, les macrophages contribuent à prévenir le stress oxydatif ou l’inflammation dû au métabolisme de l’hémoglobine [83].
De récentes études ont montré que TWEAK pouvait se lier spécifiquement à CD163, plus précisément à son domaine extracellulaire. Les domaines d’interaction ont été identifiés sur huit domaines riches en cystéines [63]. Il a été montré que cette interaction TWEAK-CD163 donne lieu à une internalisation de TWEAK [84]. Cependant, le rôle de la forme internalisée de TWEAK reste encore méconnu. Certains auteurs ont émis l’hypothèse que ce mécanisme pourrait contribuer à la séquestration de TWEAK et à la diminution concomitante de ses effets biologiques dans la circulation [63]. Une autre équipe a montrée que sTWEAK pouvait colocaliser avec la protéine GSK-3β dans le noyau de certaines lignées cellulaires, bien que les implications de cette colocalisation n’aient pas été élucidé [85].
19
2. Raisonnement
TWEAK a été identifiée comme une protéine pouvant interagir avec Dicer lors d’un criblage par double-hybride chez la levure d’une librairie d’ADNc de poumon humain, en utilisant la portion C-terminale (1772-1912) de la protéine Dicer humaine (Figure 8).
Figure 8. La protéine TWEAK interagit avec le dsRBD de Dicer. Le criblage d’une librairie
d’ADNc par double hybride chez la levure, en utilisant différentes formes de Dicer comme appât, a permis d’identifier des partenaires potentiels de Dicer, notamment la protéine TWEAK. L’activité des gènes rapporteurs de l’adénine (Ade), histidine (His) et de la β-galactosidase (LacZ) ont été testés.
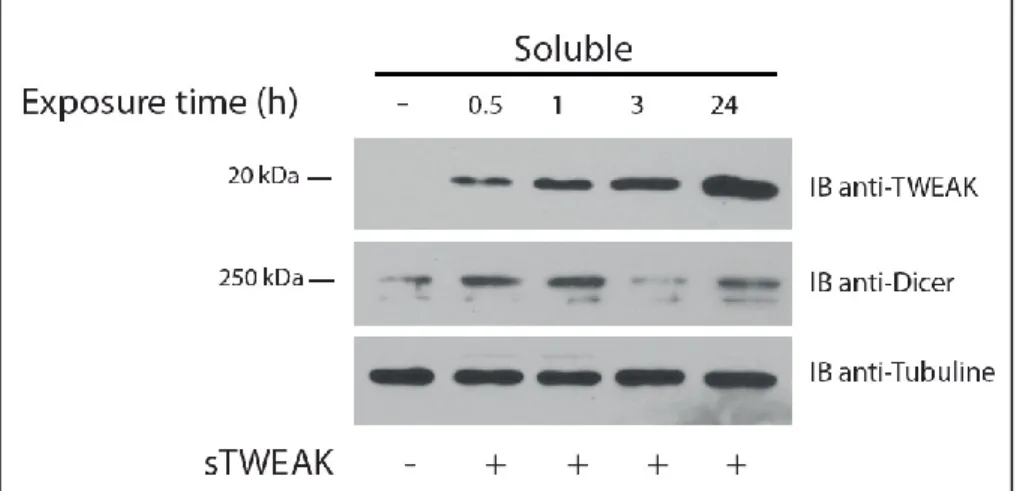
Les données obtenues suggèrent que la protéine Dicer complète pourrait ne pas interagir avec TWEAK, ce qui laisse penser que l’interaction avec TWEAK requiert un changement conformationnel de la protéine Dicer afin que le domaine dsRBD soit disponible. Durant l’étude de cette interaction, nous avons travaillé avec les cellules HEK293 et sur des macrophages humains. Toutefois, les limitations pour l’obtention de monocytes humains intègres et fonctionnels, due aux variations saisonnières observées nous ont poussé à utiliser la lignée cellulaire THP-1 dans l’étude de l’internalisation de sTWEAK. Cependant, il semblerait que ces cellules ne soient pas capables d’internaliser la cytokine en deçà d’une exposition à 500 ng/ml, une quantité bien éloignée des conditions physiologiques. Au cours de ces expériences, nous avons réalisé des PCR quantitatives et observé une altération de l’expression de certains microARN alors que TWEAK n’était pas internalisé. L’objectif a donc été, en plus d’étudier l’interaction Dicer-TWEAK, de comprendre l’origine de cette modification d’expression qui semblerait avoir lieu via le récepteur Fn14.
21
3. Hypothèses & objectifs
Les résultats préliminaires et observations nous ont amenés à nous interroger sur les conditions d’interaction et l’impact fonctionnel de cette interaction sur Dicer. La protéine TWEAK est une cytokine impliquée dans l’inflammation et sa régulation. Ainsi, l’objectif de mon projet est de caractériser l’interaction entre Dicer et TWEAK dans le but de comprendre son rôle, sa nature et sa fonction dans le contexte de maladies inflammatoires. Dans le but de vérifier notre hypothèse, nous avons adopté une approche binaire. D’abord, nous avons tenté de confirmer l’interaction entre Dicer et TWEAK dans les cellules HEK293. Puis, nous avons analysé l’impact de TWEAK sur l’activité de Dicer et le fonctionnement de la voie des microARN. L’objectif visé ici était de comprendre les conditions et les conséquences de cette interaction dans une lignée de cellules humaines pour ensuite se rapprocher d’avantage des conditions physiologiques et confirmer cette interaction dans les cellules primaires. Dans notre seconde approche, nous avons abordé un aspect encore non documenté dans la littérature scientifique, l’impact de la voie TWEAK/Fn14 sur l’expression des microARN pouvant réguler l'expression de gènes impliqués dans l'inflammation. Pour ce faire, nous avons d’abord confirmé l’altération d’expression de microARN spécifiques de l’inflammation. Puis nous avons analysé la fonctionnalité des ces microARN en analysant leurs ARNm cibles. Enfin, nous avons vérifié que cette modification dépend bien de la voie TWEAK/Fn14. Etant donné les limitations pour l’obtention de monocytes humains intègres et fonctionnels, due aux variations saisonnières observées, nous avons travaillé sur la lignée cellulaire THP-1.
23
4. Matériel et méthodes
4.1. Culture cellulaire
Les cellules humaines embryonnaires de rein HEK293 (ATCC CRL-1573) et les cellules humaines d’adénocarcinome humain HeLa (ATCC, CCL-2) ont été maintenues en culture dans du milieu Dulbecco’s modified Eagle’s medium (DMEM). La lignée de monocytes THP-1 (ATCC TIB-202) a été cultivée dans du milieu Roswell Park Memorial Institute medium 1640 (RPMI 1640) 1640. Ces deux milieux sont supplémentés avec 10% de sérum de veau fœtal (FBS), 1 mM de sodium pyruvate, 100 U/ml de penicilline, 100 µg/ml de streptomycine et 2 mM de L-glutamine. Les cellules ont été incubées à 37°C sous 5% de CO2.
Pour la différentiation des THP-1, les cellules sont incubées en présence de 100 nM de phorbol 12-myristate 13-acetate (PMA) pendant 48h. Le TWEAK recombinant est utilisé à 100 ng/ml selon les recommandations du fournisseur (Peprotech).
4.2. Isolation et différenciation de monocytes humains
Les monocytes ont été récupérés à partir du sang de donneurs sains et isoler à l’aide d’un gradient de Ficoll selon le protocole fourni par Lise-Andrée Gobeil, du laboratoire du Dr Michel J. Tremblay (CRCHUQ/CHUQ, Université Laval). D’abord repris dans du milieu RPMI 1640 (supplémenté avec 100 U/ml de penicilline, 100 µg/ml de streptomycine) pendant 2 h, les monocytes sont ensuite incubés avec du RPMI 1640 complet supplémenté avec 25 ng/ml de M-CSF et 15 ng/ml d’IL-4 pendant 7 jours. Une fois différenciés, ils sont décollés à l’aide d’un traitement à l’accutase (Gibco) pendant 45 min, puis utilisés dans les expériences.
4.3. Extraction protéique
Les protéines sont extraites à partir de cellules en culture, lysées avec un tampon de lyse (50 mM Tris–HCl, 137 mM NaCl, 1% Triton X-100, 1 mM PMSF, 1X de cocktail d’inhibiteurs de protéases sans EDTA, pH 8.0). Les lysats sont incubés pendant 15 min sur
24
glace puis centrifugés à 16 000 g pendant 15 min à 4°C. La concentration protéique est déterminée par dosage Bradford, en utilisant l’albumine de sérum bovin comme standard.
4.4. Co-immunoprécipitation
Des cellules HEK293 sont lysées, selon le protocole décrit à la section 3.3 dans 500 µl de tampon de lyse complet. Des billes magnétiques (Invitrogen, Dynabeads®) sont préalablement lavées au PBS 1X puis incubées avec des anticorps anti-FLAG (0.5 µg/échantillons) sous rotation pendant 30 min à 4°C. Par la suite, 450 µl du lysat est ajouté aux billes et l’incubation poursuivie sous rotation pendant 1h30 à 4°C. Après 3 lavages de 10 min avec du tampon de lyse complet, les billes sont reprises dans 20 µl de tampon (7 M Urée, 2 M Thiourée, 3% v/v 3-[(3-Cholamidopropyl)dimethylammonio]-1-propanesulfonate (CHAPS), 20 mM dithiothreitol (DTT), 5 mM tris, (2-carboxyethyl)phosphine (TCEP), 1 mM phenylmethylsulfonyl fluoride (PMSF), 1x Roche Complete antiprotease cocktail
ethylenediaminetetraacetic acid (EDTA) free ) et 10 µl de bleu de Laemmli. Après chauffage à 95°C pendant 8min, les 30 µl sont déposés sur un gel dénaturant SDS-PAGE afin d’être analyser par immunobuvardage. Dans le cas de traitement aux RNases T1 et V1 (Ambion), celles-ci ont été ajoutées au 450 µl de lysat pendant l’incubation avec les billes magnétiques. Le lysat des échantillons traités est conservé pour récupéré l’ARN et analyser sa dégradation sur gel d’agarose.
4.5. Immunobuvardage
Après dosage du lysat cellulaire, 50 µg de protéines sont chauffés et déposés sur un gel dénaturant SDS-PAGE de 7% et 12%. Suite à la séparation des protéines par électrophorèse à 25 mA pendant 3 h, celles-ci sont transférées sur une membrane de polyvinylidène fluoride (PVDF). Les membranes sont incubées toute la nuit dans une solution de TBS-Tween 0.1% + lait 5% avec des anticorps primaires : anti-Dicer de souris [4] (Abcam, 1/5 000), anti-FLAG de souris (Sigma, 1/5 000), anti-TWEAK de lapin (Cell signaling, 1/5 000) ou anti-tubuline de souris (DSHB, 1/10 000). Les membranes sont lavées 2 x 5min et 3 x 10 min au TBS-Tween 0.1%, puis incubées avec des anticorps secondaires anti-souris (Sigma, 1/10 000) ou anti-lapin (Sigma, 1/10 000) pendant 1 h. Après une dernière étape de lavage 2 x 5 min et 3 x 15 min au TBS-Tween 0.1%,
25 l’expression protéique sur les membranes est révélé à l’aide d’un kit enhanced chemilumescent (ECL) prime (Amersham Biosiences).
4.6. Immunofluorescence
Des cellules HeLa ont été transfectées avec deux constructions : un vecteur codant pour FLAG-TWEAK et un autre codant pour la protéine Dicer. Après 18 h, les cellules sont lavées 3 fois au PBS 1X, puis fixées pendant 20 min avec une solution de paraformaldéhyde (PFA) 4%. Afin de perméabiliser les cellules, une solution de Triton X-100 (0.1%) dans du PBS 1X est appliquée pendant 15 min, avant de bloquer les cellules avec une solution de FBS 10% dans du PBS 1X pendant 30 min. Des anticorps primaires anti-FLAG de lapin (Santa Cruz, 1/500) et anti-Dicer de souris (Abcam, 1/1 000) ont été rajoutés aux cellules et incubés dans une chambre humide pendant 1 h à température ambiante. Puis, après 8 lavages au PBS 1X, des anticorps secondaires anti-IgG de lapin (couplé à un fluorophore Alexafluor-546 (Life Technologies)) et anti-IgG de souris (couplé à un fluorophore Alexafluor-488 (Life Technologies)) ont été ajoutés et incubés dans une chambre humide pendant 1h à l’obscurité. Les noyaux ont ensuite été marqués au DAPI (Life Technologies). Les cellules marquées ont finalement été évaluées par microscopie confocale à fluorescence, en utilisant un objectif 63X, par tranche de 0.5 μm. Les images ont été acquises par un microscope Quorum Spinning Disc Wave Fx (Quorum Technologies), appartenant au Dr Maria Fernandes (CRCHUQ/CHUL, Université Laval), et analysées par le logiciel Velocity 4.
4.7. Essai Dicer
Des cellules HEK293 ont été transfectées avec différentes quantités de vecteurs pcDNA3.1-FLAG-TWEAK (5-20 μg), puis lysées et centrifugées à 10 000 g pendant 10 min afin d’obtenir un lysat dépourvu de débris cellulaires. Les pré-microARN humains hpre-miR-223 (ref miRbase : MI0000300) ou hpre-let-7c (ref miRbase : MI0018703), marqués au 32P en 5’, ont été incubés avec 50 μg de l’extrait protéique à 37°C pendant 60 min. La réaction a été stoppée en ajoutant 0.5 mg/ml de protéinase K et en incubant à 50°C pendant 20 min. Après une extraction au phénol/chloroforme, l’ARN a été précipité à l’éthanol et resuspendu dans l’eau. La formation de microARN matures a été analysée par
26
électrophorèse sur gel de polyacrylamide dénaturant (PAGE) de 12% et visualisée par autoradiographie [86].
4.8. Essai de gènes rapporteurs
Des cellules HEK293 ont été ensemencées dans une plaque à 6 puits et co-transfectées, par la méthode au phosphate de calcium, avec : (i) 5 à 25 ng d’ADN d’un vecteur exprimant pré-miR-328a (pGeneClip ; Promega), (ii) 100 ng d’ADN d’un vecteur psiCHECK-2 (Promega) contenant un gène rapporteur Renilla Luciferase (Rluc), dans lequel a été inséré 3 sites de liaison de miR-328a dans la région 3’ NTR, et un gène de normalisation firefly Luciferase (Fluc), et (iii) un vecteur pcDNA3.1 codant pour pcDNA3.1 FLAG-TWEAK ou uniquement l’épitope FLAG. L’expression de Rluc et Fluc a été mesurée à l’aide du système Dual Luciferase Reporter Assay (Promega). Pour mesurer la régulation du gène rapporteur par le microARN d’intérêt, le rapport Rluc/Fluc obtenu pour chaque concentration de vecteurs pGeneClip exprimant le pré-microARN a été normalisé avec celui obtenu avec le contrôle négatif à la même concentration.
4.9. Détection et quantification des microARN par qPCR
Des cellules HEK293, préalablement ensemencées dans une plaque 6 puits, ont été lysées dans 500 µl d’une solution de TRIzol® (Life Technologies). Après extraction de l’ARN, selon la méthode du fabriquant, 20 µl d’eau traité au diéthyl dicarbonate (DEPC) a été ajouté, puis l’ARN a été dosé à l’aide d’un spectrophotomètre Nanodrop (Thermo Scientific). Après le traitement de 1 µg d’ARN à la DNase, (Ambion), une reverse transcriptase a été réalisée à l’aide du kit miScript RT kit (Qiagen). L’utilisation du tampon HiFlex permet la détection de petits ARN et d’ARNm à partir d’un même échantillon. La quantification des ARN par qPCR a été réalisée grâce aux amorces et au système miScript PCR system (Qiagen). Le protocole a été réalisé selon les recommandations du fournisseur. La quantification relative des microARN a été réalisée selon le méthode 2-ΔΔCt et, pour chaque échantillon, les résultats obtenus ont été rapportés sur le gène de référence RNU6, et normalisés par rapport aux résultats obtenus avec des échantillons non-stimulés ou transfectés.
27
4.10. Extraction d’ARN et migration sur gel d’agarose
L’extraction d’ARN a été réalisée selon la méthode phénol-chloroforme acide. Les ARN ont été extraits d’un lysat protéique par l’ajout d’un volume équivalent de phénol-chloroforme acide (Ambion). La solution a ensuite été vortexée pendant 20 s et centrifugée à 16 000 g pour séparer les phases acqueuses et organiques. La phase aqueuse supérieure a été récupérée et les ARN précipités, par ajout de 0.1 volume d’acétate de sodium (3 M, pH 5,2), 2,5 volumes d’éthanol et 20 μg de glycogène (Life Technologies) ; puis incubés pendant au moins 1 h à -80°C. Par la suite, une centrifugation à 16 000 g pendant 30 min à 4°C a permis de sédimenter les ARN et l’excès de sel a été éliminé par lavage à l’éthanol 70%. Enfin, le culot, séché à l’air, a été repris dans de l’eau DEPC, et 5 µl a été déposé sur un gel d’agarose. Après migration pendant 1 h à 90 V, la présence d’ARN est analysée par
29
5. Résultats
5.1. Dicer et TWEAK font partie du même complexe protéique
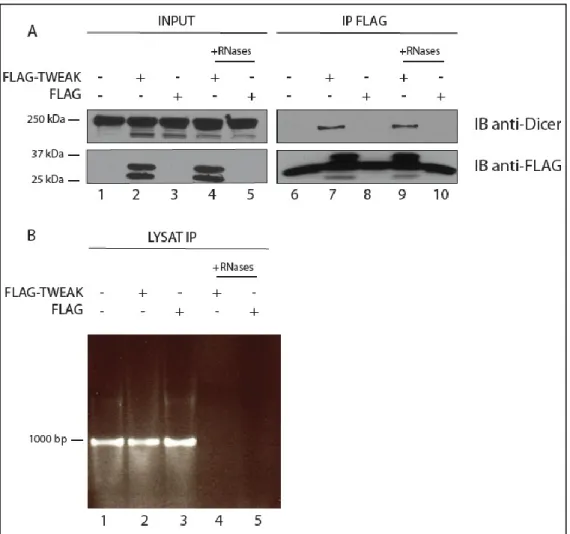
Les résultats préliminaires obtenus à l’aide du double hybride nous indiquaient une interaction potentielle entre Dicer et TWEAK, qui n’était décelé qu’avec la protéine Dicer tronquée (Figure 9). Notre premier objectif a été de confirmer cette interaction, notamment en employant les protéines complètes. Nous avons donc transfecté des cellules Human Embryonic Kidney 293 (HEK293) avec un vecteur codant pour la protéine TWEAK, marquée en N-terminal avec un épitope FLAG, ou avec un vecteur codant uniquement pour l’épitope FLAG. Après 18 h, nous avons lysé les cellules et réalisé une co-immunoprécipitation à l’aide d’un anticorps anti-FLAG, afin de déterminer si Dicer et TWEAK faisait partie d’un même complexe protéique.
Une analyse de l’extrait par immunobuvardage a tout d’abord confirmé l’expression des protéines Dicer et FLAG-TWEAK dans les cellules HEK293 (Figure 9A, puits 2). Comme on peut le voir à la Figure 9A (puits 1 à 5), Dicer est exprimé à des niveaux comparables dans tous les échantillons analysés. Par la suite, nous avons réalisé une co-immunoprécipitation en utilisant des anticorps anti-FLAG conjugués avec des billes magnétiques, puis analysé la présence de FLAG-TWEAK et de Dicer dans l’immunoprécipitat. Etant donné que la protéine TWEAK est liée à l’épitope FLAG, il était important de confirmer l’absence de Dicer dans l’immunoprécipitat anti-FLAG, préparé à partir de cellules surexprimant uniquement l’épitope FLAG (Figure 9A, puits 8). Cependant, on remarque que la protéine Dicer est immunoprécipitée par l’anti-FLAG, à partir de cellules surexprimant FLAG-TWEAK, indiquant que Dicer et TWEAK font partie d’un même complexe protéique (Figure 9A, puits 7).
Etant donné que Dicer lie et clive les précurseurs de microARN, et que ceux-ci peuvent être liés par des protéines, nous avons réalisé une co-immunoprécipitation en présence de RNases afin de vérifier si l’interaction entre nos deux protéines d’intérêts nécessite la présence d’un ARN. L’efficacité des RNases à dégrader les ARN a été confirmée par précipitation, puis migration de l’ARN sur un gel d’agarose. Les échantillons
30
traités avec des RNases présentent une dégradation marquée de l’ARN (Figure 9B, puits 4 et 5 versus puits 1 à 3). L’analyse des immunoprécipitats traités avec les RNases a révélé que Dicer est toujours présente dans le complexe protéique précipité, suggérant que la formation du complexe protéique Dicer-TWEAK ne nécessite pas de l’ARN.
Figure 9. Dicer et TWEAK font partie d’un même complexe protéique. (A-B) Les cellules
HEK293 ont été transfectées avec un vecteur pcDNA3.1 codant pour FLAG-TWEAK ou un vecteur pcDNA3.1 codant uniquement pour l’épitope FLAG. Après la lyse des cellules dans 500 µl de tampon de lyse, 450 µl ont été incubé avec un anticorps anti-FLAG conjugué à des billes pendant 90 min. Les billes ont ensuite été lavées 3 fois avec du tampon de lyse complet. Certains échantillons ont été traités aux RNases A/T1/V1 pendant l’incubation. Les 50 µl restants du lysat ont été analysés par immunobuvardage. Les billes ont ensuite été resuspendues dans 20 µl de tampon T8 et 10 µl de bleu de Laemmli. Après chauffage à 95°C pendant 8 min, les extraits protéiques (30 µl) ont été analysés par électrophorèse sur gel de polyacrylamide dénaturant (SDS-PAGE) (7% et 12%) et immunobuvardage avec des anticorps primaires anti-Dicer de souris (Abcam, 1/5000) ou un anti-FLAG de souris (Sigma, 1/5000). L’expression des protéines a été révélée à l’aide d’un kit ECL. Les lysats protéiques incubés avec les billes conjuguées ont été récupérés, l’ARN précipité et chargé sur un gel d’agarose afin de vérifier l’efficacité du traitement aux RNases.
31
5.2. Les protéines Dicer et TWEAK colocalisent dans les cellules
humaines
Afin de valider cette interaction, nous avons analysé la localisation intracellulaire de Dicer et TWEAK en microscopie confocale par immunofluorescence dans des cellules humaines HeLa afin de déceler une éventuelle colocalisation. Pour ce faire, nous avons transfecté les vecteurs exprimant Dicer et FLAG-TWEAK dans les cellules HeLa, puis révélé leur localisation avec des anticorps marqués avec des fluorochromes. La Figure 10 (3ème panneau) montre que la protéine Dicer est majoritairement présente dans le cytoplasme ; on peut également la déceler dans le noyau, mais en quantité beaucoup plus faible. TWEAK, quant à elle, est présente très majoritairement dans le cytoplasme des cellules et semble localisée dans la région périnucléaire (Figure 10, 4ème panneau). On peut noter que, dans les cellules exprimant nos deux protéines d’intérêt, Dicer et TWEAK colocalisent dans le cytoplasme des cellules, près du noyau (Figure 10, 5ème panneau). Ces résultats renforcent l’existence d’un complexe Dicer-TWEAK dans les cellules humaines.
Figure 10. Dicer et TWEAK colocalisent dans la région périnucléaire des cellules humaines.
Les cellules HeLa ont été transfectées avec un vecteur codant pour FLAG-TWEAK et un vecteur codant pour Dicer. Le lendemain, après 3 lavages avec du tampon PBS, les cellules ont été fixées au paraformaldéhyde (PFA) pendant 20 min, puis perméabilisées avec du Triton X-100 (0.1%) dans du PBS pendant 15 min à température ambiante. Des anticorps primaires anti-FLAG de lapin (Santa Cruz, 1/500) et anti-Dicer de souris (Abcam, 1/1000) ont été ajoutés aux cellules, suivi des anticorps secondaires, soit un IgG de lapin couplé au fluorophore Alexafluor-546 ou un anti-IgG de souris couplé à au fluorophore Alexafluor-488, respectivement. Le noyau des cellules a été marqués avec l’agent intercalant 4',6'-diamidino-2-phénylindole (DAPI). La fluorescence émise a été captée par microscopie confocale à fluorescence en utilisant un objectif 63X, par tranche de 0.5 μm. DIC, differential interference contrast.
32
5.3. Effet de TWEAK sur l’activité enzymatique de Dicer
Après l’obtention de ces premiers résultats, nous avons étudié l’impact de cette interaction sur la capacité enzymatique de Dicer à cliver son substrat (pré-microARN).
Pour y arriver, nous avons réalisé un essai enzymatique Dicer, au cours duquel la capacité de la ribonucléase Dicer à cliver un pré-microARN, présent dans un extrait protéique de cellules humaines, est analysée. Les cellules HEK293, exprimant ou non FLAG-TWEAK à différentes concentrations, ont été lysées et incubées avec deux pré-microARN différents marqués au phosphore-32 (32P) à l’extrémité 5’. Ainsi, si Dicer clive correctement le pré-microARN, l’analyse sur gel dénaturant devrait révéler un microARN mature d’environ 20 nucléotides marqué. Si le clivage du pré-microARN est altéré, on observera une quantité plus faible de microARN mature synthétisé, jumelé une accumulation du précurseur.
On observe tout d’abord que, dans les cellules transfectées uniquement avec l’épitope FLAG, Dicer est capable de synthétisé des microARN matures (Figure 11A, puits 1 et 6). De plus, on peut noter que les pré-microARN incubés seuls ne sont pas convertis en microARN matures (Figure 11A, puits 5 et 10). Dans les cellules transfectées avec FLAG-TWEAK, la synthèse de microARN matures semble diminuer, suggérant que l’activité enzymatique de Dicer est altérée (Figure 11A, puits 2 à 4 et 7 à 9). Cette observation est commune pour les deux pré-microARN utilisés, soit hpre-miR-223 et hpre-let-7c (Figure 11A, puits 2 à 4 et 7 à 9, respectivement). L’analyse du lysat protéique par immunobuvardage montre que Dicer est présent en quantité comparable dans les cellules, sauf dans le puits 3 (Figure 11B, puits 3). On peut supposer que la quantité plus faible de Dicer dans le puits 3 pourrait être responsable de cette diminution de synthèse. Cependant, dans les puits 2 et 4, l’expression de Dicer est stable et la quantité de microARN mature synthétisé diminue tout de même (Figure 11A et 4B). Les analyses par densitométrie confirment que la quantité de microARN matures synthétisés varie de manière inversement proportionnelle à la quantité de FLAG-TWEAK transfecté (Figure 11C et 11D).