HAL Id: hal-02595603
https://hal.inrae.fr/hal-02595603
Submitted on 15 May 2020HAL is a multi-disciplinary open access archive for the deposit and dissemination of sci-entific research documents, whether they are pub-lished or not. The documents may come from teaching and research institutions in France or abroad, or from public or private research centers.
L’archive ouverte pluridisciplinaire HAL, est destinée au dépôt et à la diffusion de documents scientifiques de niveau recherche, publiés ou non, émanant des établissements d’enseignement et de recherche français ou étrangers, des laboratoires publics ou privés.
Quantification et diversité des micro-organismes
méthanogènes et suffato-réducteurs dans les réacteurs de
digestion anaérobies
M. Ballouche
To cite this version:
M. Ballouche. Quantification et diversité des micro-organismes méthanogènes et suffato-réducteurs dans les réacteurs de digestion anaérobies. Sciences de l’environnement. 2008. �hal-02595603�
QUANTIFICATION ET DIVERSITE DES
MICRO-ORGANISMES METHANOGENES
ET SULFATO-REDUCTEURS DANS LES
REACTEURS
DE
DIGESTION
ANAEROBIES.
Mathieu BALLOUCHE
M1BCGMP
Rapport de stage de M1
Encadré par M. Peu
CemOA
: archive
ouverte
d'Irstea
CemOA
: archive
ouverte
d'Irstea
TABLE DES MATIERES
I Introduction
II Matériels et méthodes
-A) Echantillons
-B) Extraction de l’ADN génomique
-C) Choix des amorces de PCR
-D) Amplification des gènes de fonction par PCR
-E) Clonage
-F) Quantification par PCR en temps réel
-G) Digestion enzymatique
-H) T-RFLP
-I) Séquençage et analyse phylogénétique
III Résultats et discussion
-A) Extraction des ADN
-B) Amplification des gènes de fonction
-C) Quantification des microorganismes par PCR quantitative
-D) T-RFLP
CemOA : archive ouverte d'Irstea / CemagrefIV Conclusion
CemOA : archive ouverte d'Irstea / CemagrefCemOA
: archive
ouverte
d'Irstea
1
I Introduction
La digestion anaérobie ou méthanisation est un processus fermentaire de dégradation de la matière organique qui conduit à la production de biogaz, mélange gazeux de méthane et de dioxyde de carbone qui peut être valorisé comme une énergie renouvelable. En réacteur, ce procédé peut s’appliquer de façon intéressante au traitement de nombreux substrats organiques tels que les déjections animales ou les déchets de collectivité ou d’entreprises. Pour produire ce biogaz, une chaine trophique implique différents acteurs microbiens qui vont hydrolyser en anaérobiose la matière organique des effluents pour produire des substrats utilisables par des micro-organismes méthanogènes. Néanmoins le rendement des procédés biologiques de méthanisation est très sensible à la présence de certains inhibiteurs (bactériostatiques ou bactéricides) et à la présence de groupes microbiens compétiteurs utilisant les mêmes substrats que les archaea méthanogènes (acétate, H2…), en particulier les bactéries sulfato-réductrices (BSR). Certains effluents traités dans les réacteurs anaérobies sont riches en sulfate, comme ceux provenant des papèteries qui utilisent de grandes quantités d’acide sulfurique. Le sulfate n’est pas un produit directement polluant, car il est chimiquement inerte et non toxique. En revanche, la réduction dissimilatrice du sulfate réalisée en anaérobiose par les BSR produit du sulfide, un produit hautement réactif, corrosif et très toxique, qui pose donc des problèmes environnementaux pour le traitement des déchets riches en sulfate. C’est pourquoi la désulfatation de ces déchets est souvent requise avant leur passage dans les réacteurs.
Mon stage s’inscrit dans le cadre d’une thèse réalisée par M. Peu au Cemagref de Rennes, étudiant les processus de sulfato-réduction et de méthanogénèse dans les digesteurs anaérobies destinés au traitement des déchets agricoles. Ce stage a pour objectifs la mise au point d’une méthode de quantification et d’étude de la diversité des microorganismes méthanogènes et sulfato-réducteurs. Ces microorganismes sont anaérobies stricts et sont donc difficilement cultivables. De plus la mise en culture introduit un biais important dans l’étude de la diversité, car seule une partie minoritaire des bactéries et des archaea sont cultivables actuellement. Par conséquent, nous avons opté pour des méthodes moléculaires basées sur l’amplification de gènes spécifiques des groupes d’organismes ciblés. Les techniques moléculaires ciblent souvent le gène de l’ARN 16S des procaryotes, car ce gène comporte des parties plus ou moins variables qui permettent de
CemOA
: archive
ouverte
d'Irstea
2 CemOA : archive ouverte d'Irstea / Cemagref
3 cibler des groupes phylogénétiques plus ou moins larges. En raison de la diversité phylogénétique importante des BSR (Fareleira et al. 2003), nous avons décidé de cibler des gènes codant pour des protéines intervenant dans le processus de la sulfato-réduction et de la méthanogénèse, et donc présents chez tous les microorganismes d’intérêt et uniquement chez eux. Les gènes cibles sont le gène mcrA pour les méthanogènes, codant pour la sous-unité α de la Methyl-Coenzyme Reductase, et le gène dsrAB pour les sulfato-réducteurs, codant pour les sous unités α et β de la disulfite réductase. Ces microorganismes ont été caractérisés au niveau de l’abondance et de la diversité, dans des échantillons pilotes. Par la suite, la méthode mise au point servira à étudier ces mêmes microorganismes dans différents réacteurs pilotes, afin d’optimiser la méthanisation.
II Matériels et méthodes.
A) Echantillons
Les échantillons étudiés proviennent de deux installations agricoles suisses utilisant des réacteurs de digestion anaérobie pour produire du biogaz. Les échantillons B proviennent de l’installation de Bösingen (Canton de Fribourg). Cette installation traite 2000m3 de lisier porcin et 700m3 de lisier bovin par an, pour une production de biogaz équivalente à 106 kWh/an. L’échantillon B1 correspond au mélange entrant dans le digesteur, l’échantillon B2 au digestat sortant du digesteur et l’échantillon B3 au digestat sortant du post fermenteur (pas de séparation de phases).
Les échantillons S proviennent de l’installation des Saugealles (Canton de Vaud). Cette installation traite 1200 tonnes de purin et 700 tonnes de lisier bovin par an, pour une production de 1,32E+06kWh/an. L’échantillon S1 correspond au mélange liquide de la préfosse entrant dans le digesteur. L’échantillon S2 correspond au mélange de lavures et d’huiles entrant dans le digesteur comme co-substrat. L’échantillon S3 correspond au digestat sortant du digesteur avant la séparation des phases solides et liquides, et l’échantillon S4 correspond à la partie liquide après la séparation de phase. Ces échantillons ont été collectés et envoyés par la société Erep de traitement et de valorisation de déchets et d’effluents organiques (EREP s.a. Aclens, Suisse).
Un échantillon de lisier brut provenant d’un élevage porcin des Côtes d’Armor a également été utilisé pour le test des différentes amorces.
CemOA
: archive
ouverte
d'Irstea
4 CemOA : archive ouverte d'Irstea / Cemagref
5
B) Extraction de l’ADN génomique
Une extraction d’ADN a été réalisée sur 250mg de chaque échantillon, en utilisant le kit d’extraction QIAamp DNA Stool Mini (Quiagen, Hilden, Allemagne). Les échantillons sont mélangés à une solution de lyse contenant des détergents et des inhibiteurs des réactions enzymatiques. Les cellules sont lysées par chauffage à 95°C et action des détergents. Les débris cellulaires sont éliminés par centrifugation et les protéines par traitement à la protéinase K. Les acides nucléiques sont ensuite purifiés par chromatographie sur une mini colonne d’hydroxy-apatithe. Après purification, les acides nucléiques sont élués avec de l’eau Ppi (Prête pour injection) dans un microtube à vis de 2ml. Deux élutions sont réalisées afin de récupérer le maximum d’acide nucléique. Une électrophorèse sur gel d’agarose 1,5% est ensuite réalisée afin de vérifier la quantité et la qualité de l’ADN extrait. L’ADN est marqué avec du gel red, un équivalent du BET (Waring. 1965), qui permet la révélation des bandes d’ADN après migration.
C) Choix des amorces de PCR
Dans cette étude, la quantité et la diversité des microorganismes sulfato-réducteurs et méthanogènes sera étudiée en amplifiant les gènes de fonction dsrAB et mcrA. Afin d’utiliser les amorces les plus efficaces, une étude bio-informatique est réalisée avec le logiciel ARB (Ludwig et al. 2004).Ce logiciel permet d’aligner un ensemble de séquences nucléotidiques puis d’étudier l’hybridation d’un couple d’amorces donné sur ces séquences. A partir des séquences des gènes dsrAB et mcrA disponibles sur GenBank (Benson et al. 2008), des alignements ont été réalisés et différents couples d’amorces ont été testés pour chaque gène. Les amorces donnant les meilleurs résultats ont été sélectionnées et commandées auprès de Eurogentec (Eurogentec S.A. Liège, Belgique). Les séquences de ces amorces sont présentées en annexe (Tableau 1)
D) Amplification des gènes de fonction par PCR
Les réactions de PCR sont effectuées dans des microtubes de 200µl contenant le mélange réactionnel suivant : 14µl d’eau ppi, 2,5µl de tampon Red Taq 10X, 2µl de dNTP, 2,5µl de chaque amorce, 2U de Red Taq Polymerase et 1µl d’ADN matrice, pour un total de 25µl. Les amorces utilisées sont le couple MLf/MLr (Luton et al. 2002) pour l’amplification du gène mcrA. Pour l’amplification du gène dsrAB, un mix d’amorces « reverse » DSR4Rmix (Zverlov et al. 2005) a été testé avec deux amorces « forward », DSR1F (Wagner et al. 1998) et DSR1F+ (Kondo et al, 2004). Les réactions de PCR ont été réalisées sur un
CemOA
: archive
ouverte
d'Irstea
6 Thermocycleur MJ mini (BioRad, Hercules, USA) avec les cycles suivants : pour mcrA, 5min de dénaturation initiale à 95°C, puis 35 cycles de 45s à 95°C, 45s à 56°C et 1min30 à 72°C,
CemOA
: archive
ouverte
d'Irstea
7 avec une étape d’élongation finale de 7min à 72°C ; pour dsrAB, 20s de dénaturation initiale à 95°C, puis 30 cycles de 15s à 94°C, 20s à 55°C et 2min à 72°C, avec une étape d’élongation finale d’1min à 72°C. La taille des am plicons obtenus est vérifiée par électrophorèse sur gel d’agarose 1,5% (taille attendue : 1,9kb pour dsrAB, 460-490pb pour
mcrA).
E) Clonage
Les amplicons obtenus sont insérés dans le vecteur de clonage pSC-A du kit de clonage StrataClone PCR Cloning Kit (Stratagene). Ce vecteur est ensuite utilisé pour transformer des Escherichia coli compétentes (Strataclone SoloPack, Stratagene) qui sont sélectionnées sur des boites de milieu LB-Agar contenant de l’ampicilline et du X-Gal. La taille des fragments d’ADN portés par le plasmide de ces bactéries est vérifiée par une PCR avec les amorces T7/P13.
F) Quantification par PCR en temps réel
A partir d’un clone contenant transformé par le vecteur pSC-A et contenant un amplicon du gène mcrA, une extraction de plasmide est réalisée avec le kit Wizard Plus SV Minipreps (Promega). Le clone sélectionné est tout d’abord remis en culture en bouillon durant une nuit, puis 10ml de cette culture sont centrifugés. Le culot est repris dans une solution de resuspension puis lysé. Après un traitement à la protéase alcaline, le lysat bactérien est placé sur une mini colonne permettant de purifier l’ADN plasmidique.
La concentration de la solution d’ADN obtenue est mesurée par spectroscopie UV à l’aide d’un Nanodrop (Labtech France) puis une gamme de concentration de plasmide est réalisée par dilutions en série. Cette gamme sert de gamme étalon à la réaction de PCR quantitative, réalisée sur un réacteur de PCR quantitative Chromo4 (BioRad). Le mélange réactionnel est constitué de 12,5µl de Mix SybrGreen, 5,5µl d’eau ppi, 500nM de chaque amorce et 2µl d’ADN matrice. Pour les échantillons d’ADN extraits, une dilution au 1/10 est utilisée. Les cycles de la PCR quantitative sont les mêmes que ceux décrits précedemment. Chaque échantillon est analysé en triplicat. Pour chaque échantillon, un CT (Threshold Cycle) est déterminé, défini comme le nombre de cycle nécessaire pour qu’une valeur significative de fluorescence soit détectée. Grâce à la gamme étalon, ce CT est ensuite rapporté à un nombre de copies du gène ciblé dans l’ADN matrice.
CemOA
: archive
ouverte
d'Irstea
8
G) T-RFLP : Digestion enzymatique
A partir des séquences des gènes mcrA et dsrAB disponibles dans les bases de données GenBank, des digestions in silico sont réalisées afin de déterminer les enzymes les
CemOA
: archive
ouverte
d'Irstea
9 plus efficaces dans le cadre de la TRFLP. Plusieurs enzymes sont sélectionnées et testées in vitro. Pour mcrA, on teste les enzymes Acc65I et TaqI. Pour dsrAB, on teste les enzymes RsaI, MspI et AluI.
Les digestions in vitro sont réalisées sur les produits de PCR amplifiant le gène ciblé, selon les instructions du fournisseur de l’enzyme (Promega). L’ADN digéré est analysé par électrophorèse sur un gel d’agarose 3%. Les enzymes donnant des résultats satisfaisant sont ensuite utilisées pour digérer des amplicons issus de l’amplification des gènes de fonction sur les banques de clones ou sur les échantillons environnementaux avec les amorces DSR1F-6-FAM pour dsrAB, et MLr-6-FAM pour mcrA. Ces amorces sont les mêmes que celles utilisées précedemment, mais elles sont marquées avec un fluorophore qui permet la détection des fragments d’ADN en T-RFLP.
H) T-RFLP : Analyse des fragments
Les fragments d’ADN marqués obtenus après digestion sont analysés sur un séquenceur ABI 310 (Applied BioSystems) par électrophorèse sur un capillaire. Ce capillaire est rempli de POP4, un polymère permettant la migration des fragments d’ADN en condition dénaturantes. Un marqueur de taille 400 HD-ROX est utilisé pour déterminer la taille des fragments d’ADN. Les profils obtenus sont ensuite traités et analysés grâce au logiciel GeneScan 3.1 (Applied Biosystems).
III Résultats et discussion
A) Extraction des ADN
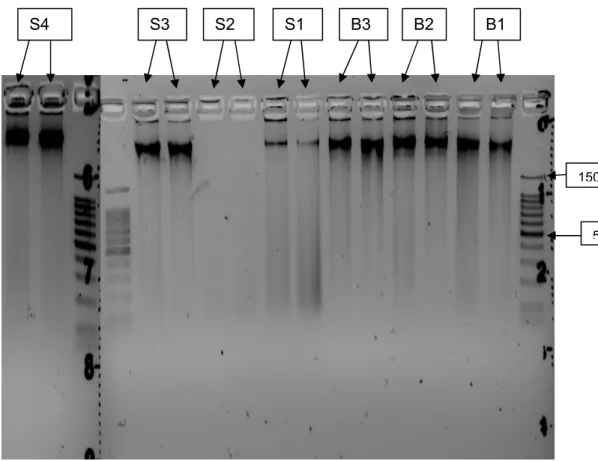
Une extraction d’ADN a été réalisée sur les échantillons B1 à B3 et S1 à S4. La figure 1 présente les résultats de ces extractions. Pour les échantillons B1, B2, B3, S3 et S4, les bandes observées sont de forte intensité, indiquant une concentration d’ADN élevée. Les bandes correspondant à la première et à la deuxième élution sont d’intensité comparable pour tous les échantillons. Les deux élutions pourront donc être utilisées par la suite. La présence d’un smire de faible intensité indique que l’ADN n’a pas été dégradé de façon trop importante. Pour l’échantillon S1, on observe des bandes d’intensité beaucoup plus faible, indiquant une concentration d’ADN inférieure. Pour l’échantillon S2, aucune bande n’est détectée, indiquant l’absence d’ADN. Cet échantillon correspond au mélange de lavures et
CemOA
: archive
ouverte
d'Irstea
10 d’huiles entrant dans le digesteur comme co-substrat, et contient donc peu de microorganismes.
Figure 1 : Electrophorèse de l'ADN extrait des échantillons B1 à S4. Pour chaque échantillon, une première élution est réalisée (colonne de droite), puis une seconde (colonne de gauche)
B1 B2 B3 S1 S2 S3 S4 ADN pur ADN 1/10 ADN 1/100 Témoin négatif 500pb 1500pb CemOA : archive ouverte d'Irstea / Cemagref
11
Figure 2 : Amplification du gène mcrA sur un échantillon de lisier brut
CemOA
: archive
ouverte
d'Irstea
12 Il est donc cohérent de n’extraire aucun ADN de cet échantillon. Cet échantillon ne sera donc pas analysé, par la suite.
Les extractions ont été réalisées selon le même protocole, et les quantités d’ADN extraites peuvent donc donner une première indication sur la quantité de micro-organismes présents dans chaque échantillon. Pour les échantillons B, la quantité d’ADN extraite n’est pas sensiblement différente sur le gel d’agarose. En revanche, l’échantillon S1 semble contenir moins d’ADN que les échantillons S3 et S4.
B)Amplification des gènes de fonction
Afin de tester in vitro l’efficacité des amorces choisies, une PCR est réalisée sur un échantillon d’ADN extrait de lisier brut à différentes dilutions pour les amorces MLf/MLr (Figure 2). Avec l’ADN non dilué, aucune bande n’est détectable. En effet la solution d’ADN contient des inhibiteurs qui perturbent le bon déroulement de l’amplification. Les dilutions au 10ème et au 100ème sont préférables pour observer une bonne réaction en diminuant la quantité d’inhibiteurs présents. La bande observée a une taille de 500pb, ce qui correspond à la taille attendue des fragments amplifiés par les amorces MLf/MLr.
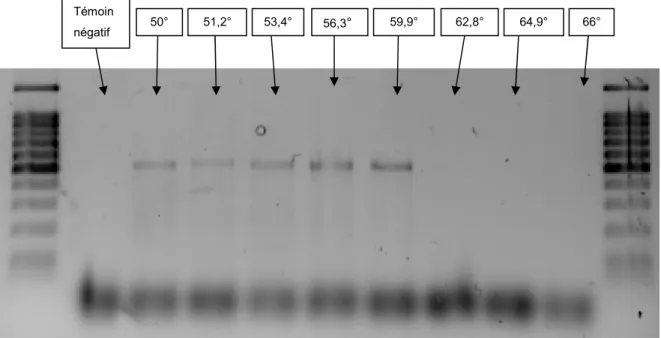
Afin d’optimiser notre protocole, une nouvelle PCR est réalisée sur le même ADN avec un gradient de Tm (Température d’hybridation). Cette expérience permet d’optimiser l’étape d’hybridation et donc l’amplification. Dans la bibliographie, différents cycles de PCR sont utilisés, avec différents Tm : 56°C (Vianna et al. 2006) ou 55°C (Luton el al. 2002 ; Juottonen et al. 2006). Nous avons donc réalisé un gradient de Tm de 50°C à 66°C. Les résultats de cette PCR sont présentés dans la figure 3. Pour un Tm supérieur à 60°C on observe aucune bande, en effet avec un Tm trop élevé, l’hybridation des amorces avec l’ADN cible est impossible. Les bandes les plus intenses sont celles correspondant à des Tm de 59,9°C et 56,3°C. Pour éviter tout problème d’hy bridation lié à un Tm trop élevé et pour rester proche des températures indiquées dans les autres études utilisant les amorces MLf/MLr le Tm sera fixé à 56°C pour le reste de not re étude.
Les mêmes expériences ont été conduites avec les amorces destinées à amplifier le gène dsrAB, sur le même ADN extrait de lisier brut. La première PCR réalisée avec les amorces DSR1F+/DSR4Rmix n’ayant donné aucun résultat, l’hypothèse d’une absence de micro-organismes sulfato-réducteurs dans l’échantillon a été posée. Par conséquent une deuxième PCR a été réalisée sur l’ADN extrait de l’échantillon B2. En effet dans le digesteur auquel correspond cet échantillon, une désulfuration est réalisée, ce qui indique la présence de sulfure et donc de sulfato-réducteurs. Cette deuxième PCR ne donne aucun résultat.
CemOA
: archive
ouverte
d'Irstea
13
Figure 3 : Gradient de Tm pour les amorces MLf/MLr
Figure 4 : Gradient de Tm pour les amorces DSR1F/DSR4R
66° 64,9° 62,8° 59,9° 56,3° 53,4° 51,2° 50° Témoin négatif 59,9° 61° 57,8° 54,9° 51,3° 48,3° 46,2° 45° CemOA : archive ouverte d'Irstea / Cemagref
14 Pour identifier les raisons de cette absence de résultat, nous avons pensé à utiliser un témoin positif. Comme les sulfato-réducteurs sont des bactéries anaérobies très difficilement cultivables, nous avons à des collègues (à l’équipe du professeur Wagner, qui à conçu les amorces DSR1F et DSR4R) de nous envoyer un plasmide lyophilisé contenant le gène
dsrAB. L’envoi par la poste prenant du temps, nous avons étudié les autres paramètres
pouvant être à l’origine de l’absence de résultats. Après avoir modifié la concentration des amorces, les cycles de PCR et la polymérase utilisée, n’obtenant toujours aucun résultat, nous avons envisagé un problème au niveau des amorces. Nous avons tout d’abord remplacé le mix d’amorces reverse par DSR4R uniquement, puis devant l’absence de résultats, l’amorce forward DSR1F+ a été remplacée par DSR1F. Le couple DSR1F/DSR4R donnait de moins bons résultats lors de l’analyse bioinformatique préalable, car certaines bactéries sulfato-réductrices n’étaient pas reconnues, mais ce couple est souvent utilisé dans les autres études sur la diversité des sulfato-réducteurs (Wagner et al. 1998 ; Leloup et
al. 2003 et 2005). L’utilisation de ce couple d’amorces sur les ADN extraits de lisier brut et de
l’échantillon B2 donne une bande d’une taille d’à peu près 1,9kb qui correspond à la taille de fragment attendue. Le problème semble donc venir des amorces DSR1F+ et du mix d’amorces. C’est donc le couple d’amorces DSR1F/DSR4R qui a été utilisé dans la suite de notre étude. Comme pour les amorces MLf/MLr nous avons réalisé un gradient de Tm afin d’optimiser les cycles de PCR utilisés par la suite (Figure 4). Pour un Tm supérieur à 60° on n’observe aucune amplification. En revanche, pour un Tm inférieur à 52°, on observe une deuxième bande d’une taille de 500pb. Cette bande indique un phénomène d’hybridation aspécifique pour un Tm trop bas. En effet un faible Tm facilite l’hybridation des amorces avec des séquences non spécifiques sur l’ADN matrice et l’amplification de fragments de tailles différentes de celle attendue. Pour la suite de notre étude, le Tm sera donc fixé à 54°C, ce qui correspond au Tm utilisé dans les étud es précédemment citées.
C)Quantification des microorganismes par PCR
quantitative
Après avoir vérifié le bon fonctionnement des amorces choisies, et optimisé leur utilisation, la quantification des microorganismes sulfato-réducteurs et méthanogènes a été réalisée par PCR quantitative. Cette méthode implique la conception d’une gamme étalon qui permet de faire correspondre le CT mesuré à un nombre de copies du gène ciblé. Il faut donc une gamme de concentrations connues du gène en question. Nous avons réalisé cette gamme en insérant le fragment amplifié par nos amorces dans un plasmide, puis en réalisant un clonage avec ce plasmide. Une fois que les bactéries transformées se sont
CemOA
: archive
ouverte
d'Irstea
15 ‘
Figure 5 : PCR sur colonie sur des E.coli transformées par le plasmide contenant le fragment du gène
mcrA amplifié par les amorces MLf/MLr
Figure 6 : CT en fonction du nombre de copies initial du gène ciblé. Le CT représente le nombre de cycles nécessaire pour que la fluorescence atteigne un niveau seuil
développées, le plasmide a été extrait suivant le protocole décrit dans la partie « Matériel et Méthodes ».La concentration de plasmide obtenue a été mesurée et une gamme étalon a été réalisée par des dilutions au 1/10 en série. Cette méthode est plus longue que l’utilisation de dilutions d’un produit de PCR purifié comme gamme étalon, mais les bactéries transformées peuvent être conservées indéfiniment, ce qui permet ensuite de reproduire rapidement une gamme étalon pour de futures manipulations. Le clonage a été réalisé sur l’ADN extrait de lisier brut. Avant l’extraction de plasmide, une PCR sur colonies a été réalisée pour vérifier la taille de l’insert, et ainsi s’assurer que le plasmide extrait contient bien le fragment du gène d’intérêt. La figure 5 présente le résultat de cette PCR pour le gène
mcrA. Un clone donnant une bande de la taille attendue est ensuite sélectionné et le
CemOA
: archive
ouverte
d'Irstea
16 plasmide est extrait. Une première PCRQ est réalisée pour vérifier que la gamme étalon est exploitable. Pour cela, la courbe représentant le CT mesuré en fonction du nombre initial de copies du gène (Figure 6) doit être linéaire. La courbe obtenue est linéaire sur 10log et le seuil de détection de la fluorescence permettrait de détecter jusqu’à un minimum de 10 copies du gène cible.
Une fois que la gamme étalon exploitable est obtenue, la PCRQ est réalisée simultanément sur cette gamme et sur les échantillons en triplicat. Le CT mesuré pour chaque échantillon permet grâce à la gamme étalon de déduire un nombre de copies du gène cible par échantillon. Les microorganismes ciblés possèdent généralement une seule copie des gènes dsrAB (Leloup et al. 2003) et mcrA (Inagaki et al. 2004), ce qui permet de considérer que le nombre de copies du gène par échantillon correspond au nombre de microorganismes présents. Les figures 7 et 8 présentent les résultats de la quantification respectivement des archaea méthanogènes et des microorganismes sulfato-réducteurs. Pour les méthanogènes, on observe pour les deux séries d’échantillons une augmentation significative de la quantité de microorganismes après passage dans le digesteur (7,77E+08 dans l’échantillon B1 contre 1,07e+09 dans l’échantillon B2 ; 2,79e+08 dans l’échantillon S1 contre 2,26 e+09 dans l’échantillon S3). Pour la série d’échantillons B, cette quantité augmente également après passage dans le post fermenteur. En revanche la séparation de phase qui a lieu entre les échantillons S3 et S4 ne semble pas influencer la quantité de méthanogènes. La majorité des méthanogènes se retrouve donc dans la phase liquide. L’augmentation de la quantité de méthanogènes semble la plus importante entre les échantillons S1 et S3. Cependant, l’extraction d’ADN de l’échantillon S1 avait fourni une faible concentration d’acides nucléiques, qui peut être due à une quantité plus faible de microorganismes, à un problème d’échantillonnage ou à un problème lors de l’extraction, ce qui pourrait entrainer une sous-estimation de la quantité de microorganismes dans cet échantillon. CemOA : archive ouverte d'Irstea / Cemagref
17
Figure 7 : Quantification des Archaea méthanogènes par PCRQ. La quantité est exprimée en cellules/ml
Figure 8 : Quantification des microorganismes sulfato-réducteurs. La quantité est exprimée en cellules/ml
Pour les sulfato-réducteurs, la variation de la quantité de microorganismes semble moins importante à l’intérieur des séries d’échantillons. Compte tenu des écarts types, les
CemOA
: archive
ouverte
d'Irstea
18 quantités de sulfato-réducteurs mesurées ne sont pas significativement différentes entre les différents échantillons B (6,22e+07 à 1,11e+08), et entre les différents échantillons S (2,70e+07 à 2,98e+07). En revanche, la quantité de sulfato-réducteurs semble plus importante dans les échantillons B que dans les échantillons S. Cette constatation peu s’expliquer par la concentration en sulfate qui diffère dans les deux lots d’échantillons. A partir des analyses physico-chimiques réalisées sur ces échantillons, présentées en annexe (Tableaux 2 à 8), on remarque que l’échantillon B1 contient plus de sulfates (51mg/L) que l’échantillon S1 (20mg/L). Or il a été démontré que la concentration en sulfates influençait le développement des sulfato-réducteurs, de hautes concentrations favorisant leur développement (Fareleira et al, 2003). Les différences entre la quantité de sulfato-réducteurs entre les deux lots d’échantillon seraient donc dues à la plus faible concentration en sulfates des échantillons S.
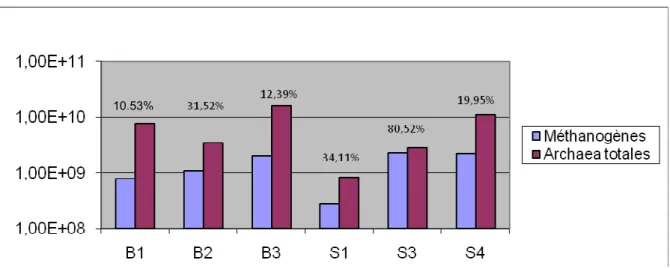
En parallèle de cette étude, des expériences de PCR quantitative ont été menées sur les mêmes échantillons afin de quantifier la totalité des bactéries et des archaea présentes, dont les résultats sont présentés en annexe (Figures 15 et 16). A partir de ces mesures, nous avons tout d’abord calculé le rapport entre la quantité de méthanogènes et la quantité totale d’archaea (Figure 9). D’après ces mesures, la proportion de méthanogènes varie de 10,13% dans l’échantillon B1 à 80,52% dans l’échantillon S3. Pour les deux lots d’échantillon, cette proportion augmente après le passage dans le digesteur, puis diminue en raison de la forte concentration d’archaea mesurée dans les échantillons B3 et S4. Ces proportions semblent faibles par rapport à celles rapportées dans d’autres études sur les
archaea méthanogènes des réacteurs de digestion anaérobies, où les méthanogènes
représentaient la quasi-totalité des archaea présentes (Montero et al, 2008). La quantité de méthanogènes mesurée par PCR quantitative a été confirmée par des expériences de microscopie réalisées par une autre stagiaire. Par conséquent la faible proportion de méthanogènes peut venir d’une surestimation de la quantité d’archaea totale. En effet la quantification des archaea a été réalisée en amplifiant le gène de l’ARN 16S, avec des amorces ciblant la région V3. L’analyse de ces amorces avec le logiciel ARB a montré qu’elles n’étaient pas tout à fait spécifiques des archaea, s’hybridant avec certaines séquences d’ADNr 16S de bactéries. L’ADN de certaines bactéries a donc pu être amplifié par ces amorces, ce qui aurait conduit à une surestimation de l’abondance des archaea totales. De la même façon, la quantité totale de bactéries a été mesurée par PCR quantitative, permettant d’établir la proportion de bactéries sulfato-réductrices (Figure 10).
CemOA
: archive
ouverte
d'Irstea
19
Figure 9 : Rapport entre la quantité de méthanogènes et la quantité totale d'archaea mesurée. Les quantités sont exprimées en cellules/ml
Figure 10 : Rapport entre la quantité de sulfato-réducteurs et la quantité totale de bactéries mesurée. Les quantités sont exprimées en
cellules/ml
Figure 11 : Rapport entre les quantités de sulfato-réducteurs et de méthanogènes. Les quantités sont exprimées en cellules/ml 10,53% CemOA : archive ouverte d'Irstea / Cemagref
20 Cette proportion est très faible (de 0,35% pour l’échantillon S3 à 1,50% pour l’échantillon B3). Les mesures physico-chimiques indiquent que les concentrations en sulfate des échantillons B2-B3 sont très faibles (4 et 2 mg/L) par rapport à la concentration de l’échantillon B1. La situation est la même pour les échantillons S. Le sulfate présent est donc rapidement consommé, et devient donc limitant pour la croissance des sulfato-réducteurs, ce qui explique la faible part qu’ils représentent sur l’ensemble des bactéries.
Nous avons également calculé le rapport entre la quantité de sulfato-réducteurs et de méthanogènes (Figure 11), car un des principaux intérêts de ces mesures est la compréhension de la compétition entre ces deux groupes d’organismes. Pour les échantillons B comme pour les échantillons S, les sulfato-réducteurs sont présents en quantité beaucoup plus faible que les méthanogènes. De plus, ce rapport diminue au cours du processus de digestion (8,01% pour B1 contre 5,57% pour B3 ; 10,67% pour S1 contre 1,23% pour S4), indiquant que dans des milieux pauvres en sulfate comme le sont les réacteurs étudiés, les sulfato-réducteurs ne sont pas des compétiteurs efficaces des méthanogènes.
D) T-RFLP
Afin d’étudier la diversité des microorganismes quantifiés, des expériences de T-RFLP ont été réalisées sur les échantillons B. La première étape a été le choix des enzymes de restriction à utiliser. Il faut en effet que ces enzymes digèrent toutes les séquences possibles pour les amplicons de mcrA et dsrAB, et qu’elles fassent apparaitre le polymorphisme présent entre les différentes espèces en formant des fragments de tailles différentes. De plus, il faut que les fragments terminaux analysés en T-RFLP ne dépassent pas 500 nucléotides, car le marqueur de taille et le séquenceur utilisés ne permettent pas d’analyser des morceaux plus longs. En couplant la recherche bibliographique et la digestion
in silico, nous avons sélectionné l’enzyme TaqI pour la digestion de mcrA et RsaI pour la
digestion de dsrAB. Pour les deux gènes, un clonage a été réalisé à partir de l’amplification de l’ADN de l’échantillon B2, afin de réaliser une banque de clones. A partir de 20 clones contenant un amplicon de mcrA, une PCR sur colonie a été réalisée afin de réamplifier le gène de fonction, puis les produits de cette PCR ont été digérés par l’enzyme TaqI selon les instructions du fournisseur. Les produits de digestion ont été analysés par électrophorèse sur un gel d’agarose à 3% (Figure 12). On remarque plusieurs profils différents, ce qui indique que la digestion par TaqI permet de différencier les clones possédant des séquences différentes de mcrA. L’enzyme peut donc être utilisée pour la T-RFLP. Une PCR sur colonie est de nouveau réalisée sur 40 clones, avec les amorces fluorescentes, ainsi que sur les
CemOA
: archive
ouverte
d'Irstea
21
ADN extraits des échantillons B.
CemOA
: archive
ouverte
d'Irstea
22
Figure 12 : Digestion des amplicons de mcrA des clones 1 à 20 de l'échantillon B2
B1 B2 B3 29 10 8 7 15 14 1 2 3 4 5 6 7 89 10 11 12 13
Figure 13 : Profils de fluorescence pour le gène mcrA
20 pb 200pb CemOA : archive ouverte d'Irstea / Cemagref
23 Les produits de PCR marqués sont digérés par TaqI, puis analysés par le séquenceur. Pour chaque produit de PCR, on obtient un profil de fluorescence (Figure 13). Chaque pic du profil correspond à une taille de fragment marqué différente. Comme l’amorce marquée est MLr, les fragments d’ADN marqué sont les extrémités 5’ du gène mcrA. Les profils des échantillons B comportent plusieurs pics, dont chacun correspond à au moins une séquence différente du gène de fonction. Les profils des clones ne comportent qu’un seul pic car l’ADN amplifié provient d’une colonie et ne contient donc qu’une seule séquence. Le but de l’analyse des profils est de faire correspondre au moins un clone à chaque pic observé pour les échantillons. Les clones seront ensuite séquencés permettant de faire correspondre une séquence puis une espèce de méthanogène à chaque pic, et donc d’obtenir une image de la diversité des méthanogènes présents dans l’échantillon analysé.
Les profils de T-RFLP obtenus pour le gène mcrA indiquent que la diversité de l’échantillon B1 diffère de celle des échantillons B2 et B3. En effet les on retrouve les mêmes pics (1 à 5 et 8 à 13) dans les échantillons B2 et B3, mais pas pour l’échantillon B1 (absence des pics 10 à 13). Cela indique une évolution des populations méthanogènes entre le lisier avant digestion et après le passage par le digesteur. La diversité de la population semble augmenter, avec l’apparition de nouvelles espèces (pics 10 à 13) et la disparition d’autres espèces (pics 6 et 7) au cours du processus de digestion. La mise en parallèle des profils des échantillons B et des clones permet d’identifier les clones correspondant aux pics observés. Les 6 clones dont les profils sont présentés correspondent chacun à un pic observé sur les profils des échantillons. Ces clones ont été envoyés pour séquençage au Génopôle Ouest (Roscoff, France). Les séquences obtenues seront ensuite comparées aux séquence connues du gène mcrA grâce au logiciel ARB (déjà utilisé pour le choix des amorces), afin de déterminer les espèces de méthanogènes correspondantes. Sur les 40 clones analysés, plusieurs présentent le même pic et donc une taille de fragment 5’ identique. Jusqu’à 5 clones par pic ont été envoyés pour séquençage, afin de vérifier le pouvoir discriminant de notre technique et surtout de l’enzyme utilisée TaqI. Certains pics observés dans les profils des échantillons ne correspondent à aucun des clones que nous avons analysé (1, 2, 4, 5, 6, 7 et 8). Les pics 1 et 2 sont faibles par rapport aux autres pics de B1, comme les pics 4 et 5 qui sont plus importants, pourtant aucun clone ne leur correspond. Cela peut venir d’un problème au niveau du clonage. Certaines séquences sont plus difficiles à insérer dans le vecteur que d’autres, ce qui peut créer un biais. Pour les pics 6 et 7, ils ne sont présents que pour l’échantillon B1. Comme le clonage n’a été réalisé qu’avec l’ADN de l’échantillon B2, il est logique de ne pas retrouver de clones correspondant à ces pics. CemOA : archive ouverte d'Irstea / Cemagref
24 B1 B2 B3 1 4 1 2 3 4 5 6 7 8 9 10 11 12 13
Figure 14 : Profils de fluorescence pour le gène dsrAB
20pb 200pb CemOA : archive ouverte d'Irstea / Cemagref
25 La même analyse a été réalisée pour le gène dsrAB (Figure 14). Comme pour les méthanogènes, on remarque que les profils des échantillons B2 et B3 sont très proches mais que le profil de l’échantillon B1 est différent. Ainsi, le pic 4 n’est présent que pour B1, tandis que les pics 8 à 13 sont présents uniquement pour B2 et B3. Au cours du processus de digestion, il y’a donc apparition de nouveaux pics, et disparition du pic 4. De plus, pour l’échantillon B1, le pic 7 est dominant, alors que pour B2 et B3, le pic 5 est aussi important que le 7. Deux clones (1 et 4) correspondant aux pics 5 et 6 ont été isolés, mais la plupart des pics des échantillons ne correspondent à aucun clone analysé. Cela est du au faible rendement du clonage pour le gène dsrAB. En effet, seuls 16 clones ont pu être sélectionnés et analysés. La grande taille du fragment à insérer dans le plasmide (1,9kb) explique peut-être le faible rendement. Il est également possible que certains pics soient des artefacts de fluorescence, ne correspondant à aucun fragment d’ADN. Un nouveau clonage va être réalisé afin d’analyser plus de clones, pour pouvoir identifier le maximum de pics présents sur les profils.
IV Conclusion
Les différentes expériences réalisées au cours de ce stage m’ont permis de prendre en main des outils d’analyse de l’abondance et de la diversité des microorganismes méthanogènes et sulfato-réducteurs. Certaines manipulations peuvent encore être optimisées, comme le clonage du gène dsrAB dont le rendement est trop faible. Mon stage se poursuit jusqu’au 31 juillet, et d’autres expériences vont donc être menées : l’analyse phylogénétique des séquences, l’analyse par notre méthode de différents échantillons de lisiers, et la mise en place d’un réacteur de digestion expérimental afin d’étudier l’influence de différents paramètres sur les microorganismes d’intérêt.
Je voudrais remercier M. Peu et tout le personnel du Cemagref pour leur accueil, et pour ce stage qui m’a permis de découvrir de nombreuses techniques, mais aussi différents aspects du travail de chercheur, du laboratoire à la conception de méthodes d’étude et à l’analyse des résultats.
CemOA
: archive
ouverte
d'Irstea
CemOA
: archive
ouverte
d'Irstea
V Bibliographie
-
Benson D.A., I. Karsch-Mizrachi, D.J. Lipman, J.Ostell, D.L. Wheeler. GenBank. Nucleic Acid Res. (2008) 36, D25-30- Fareleira P., B.S. Santos, C. Antonio et al. Response of a strict anaerobe to oxygen : survival strategies in Desulfovibrio gigas. Microbiolog-SCM (2003) 149, 1513-22
- Inagaki F., U. Tsunogai, M. Suzuki, A. Kosaka, H. Masiyama, K. Takai, T. Nunoura, K.H. Nealson, K. Horikoshi. Characterization of C1-Metabolizing
Prokaryotic Communities in Methane Seep Habitats at the Kuroshima Knoll, Southern Ryukyu Arc, by Analyzing pmoA, mmoX, mxaF, mcrA, and 16S rRNA Genes. Appl. Environ. Microbiol. (2004), 70, 7445-7455
- Juottonen H., P.E. Galand, K. Yrjälä. Detection of methanogenic Archaea in peat : comparison of PCR primers targeting the mcrA gene. Research in
Microbiology (2006), 157, 914-921
-
Kondo J.,
D.B. Nedwell, K.J. Purdy, S. de Queiroz Silva. Detection and Enumeration of Sulphate-Reducing Bacteria in Estuarine Sediments by Competitive PCR. Geomicrobiology Journal. (2004), 24, 145-157-
Leloup J., L. Quillet, C. Oger, D. Boust, F. Petit. Molecular quantification of sulfate-reducing microorganisms (carrying dsrAB genes) by competitive PCR in estuarine sediments. FEMS Microbiology Ecology. (2003), 47, 207-214-
Leloup J., L. Quillet, T. Berthe, F. Petit. Diversity of the dsrAB (dissimilatory sulfite reductase) gene sequences retrieved from two contrasting mudflats of the Seine estuary, France. FEMS Microbiology Ecology. (2005), 55, 230-238-
Ludwig W., O. Strunk, R. Westram, L. Richter, H. Meier, Yadhukumar, A. Buchner, T. Lai, S. Steppi, G. Jobb, W. Förster, I. Brettske, S. Gerber, A. W. Ginhart, O. Gross, S. Grumann, S. Hermann, R. Jost, A. König, T. Liss, R. Lüßmann, M. May, B. Nonhoff, B. Reichel, R. Strehlow, A. Stamatakis, N. Stuckmann, A. Vilbig, M. Lenke, T. Ludwig, A. Bode, and K.-H. Schleifer. ARB: a software environment for sequence data. Nucleic Acids Res. (2004) 32, 1363-1371- Luton P.E., J.M. Wayne, R.J. Sharp, P.W. Riley. The mcrA gene as an alternative to 16S rRNA in the phylogenetic analysis of methanogen populations in landfill. Microbiology (2002), 148, 3521-3530. CemOA : archive ouverte d'Irstea / Cemagref
- Montero B., J.L. Garcia-Morales, D.Sales, R. Solera. Evolution of microorganisms in thermophilic-dry anaerobic digestion. Bioresource Technology (2008), 99, 3233-3243 CemOA : archive ouverte d'Irstea / Cemagref
- Vianna M.E., G. Conrads, B.P.F.A. Gomes, H.P. Horz. Identification and Quantification of Archaea Involved in Primary Endodontic Infections. Journal of
Clinical MicrobiologyI (2006), 44, 1274-1282
- Waring M.J. Complex formation between ethidium bromide and nucleic acids. Journal of Molecular Biology (1965), 13, 269-282.
- Wagner M., A.J. Roger, J.L. Flax, G.A. Brusseau, D.A. Stahl. Phylogeny of dissimilatory sulfite reductase supports an early origin of sulfate respiration.
J.Bacteriol (1998), 180, 2975-2982
- Zverlov V., M. Klein, S. Lücker, M.W. Friedrich, J. Kellerman, David A. Stahl, A. Loy, M. Wagner. Lateral gene transfert of dissimilatory (Bi)Sulfite reductase revisited. J.Bacteriol. (2005), 187, 2203-2208 CemOA : archive ouverte d'Irstea / Cemagref
CemOA
: archive
ouverte
d'Irstea
Annexes
Nom de l'amorce
Type de
l'amorce Séquence Reference
MLf Forward GGTGGTGTMGGATTCACACARTAYGCWACA GC MLr Reverse TTCATTGCRTAGTTWGGRTAGTT Luton et al. 2002 MLr-6-FAM Reverse
marquée TTCATTGCRTAGTTWGGRTAGTT Cette étude
DSR1F+ Forward ACSCACTGGAAGCACGGCGG Kondo et al. 2004
DSR1F Forward ACSCACTGGAAGCACG Wagner et al. 1998
DSR1F-6-FAM
Forward
marquée ACSCACTGGAAGCACG Cette étude
DSR4R Reverse GTGTAGCAGTTACCGCA Wagner et al. 1998
DSR4Ra Reverse GTGTAACAGTTTCCACA
DSR4Rb Reverse GTGTAACAGTTACCGCA
DSR4Rc Reverse GTGTAGCAGTTKCCGCA
Loy et al. 2004
DSR4Rd Reverse GTGTAGCAGTTACCACA
DSR4Re Reverse GTGTAACAGTTACCACA Zverlov et al. 2005
Tableau 1 : Amorces utilisées au cours de cette étude. Les amorces DSR4R à DSR4Re forment le mélange d’amorce DSR4Rmix. Le marquage 6-FAM correspond à un marquage par la 6-Carboxyfluoresceine
CemOA
: archive
ouverte
d'Irstea
UR GERE Rennes
Rapport d'essai
Réf : EN RR 001 Version : 1 Date: 28/11/06 Page 1/1Projet :
Thèse PP
Type
déjection :
Entrée réacteur
Bosingen
Date prélèvement
:
9 avril 2008
Origine :
Suisse
Opérateur :
P PEU
Animal :
Intervalle de
confiance
Moyenne
Ecart type
CV %
Méthode
utilisée
pH
6,24
MS g/kg
118,53
4,44
3,7
MA RO 004MO g/kg
101,95
4,45
4,4
MA RO 004MES g/kg
108,16
-
-
MA RO 003MVS g/kg
98,55
-
-
MA RO 003NTK gN/kg
4,77
0,29
6,1
MA RA 001NH4
+gN/kg
1,65
0,09
5,3
MA RA 001DCO totale gO
2/kg
84,74
5,34
6,3
MA RO 008DCO soluble gO2/kg
57,89
2,23
3,9
MA RO 008AGV totaux g/L
4474
-
-
MA RC 002AGV totaux gC/L
1915
-
-
MA RC 002Acide Acétique g/L
3017
-
-
MA RC 002Acide Propionique
g/L
1430
-
-
MA RC 002Acide Isobutyrique
g/L
nd
-
-
MA RC 002Acide Butyrique g/L
27
-
-
MA RC 002Acide Isovalérique
g/L
nd
-
-
MA RC 002Acide Valérique g/L
nd
-
-
MA RC 002Chlorure mg/L
2600
-
-
MA RI 001Phosphate mg P /L
262
-
-
MA RI 001Sulfate mg S /L
51
-
-
MA RI 001Tableau 2 : Rapport des analyses physico-chimiques réalisées sur l’échantillon B1
CemOA
: archive
ouverte
d'Irstea
UR GERE Rennes
Rapport d'essai
Réf : EN RR 001 Version : 1 Date: 28/11/06 Page 1/1Projet :
Thèse PP
Type
déjection :
Sortie réacteur
Bosingen
Date
prélèvement :
9 avril 2008
Origine :
Suisse
Opérateur :
P PEU
Animal :
Intervalle de
confiance
Moyenne
Ecart type
CV %
Méthode
utilisée
pH
8,06
MS g/kg
58,36
1,82
3,1
MA RO 004MO g/kg
44,78
1,69
3,8
MA RO 004MES g/kg
45,78
-
-
MA RO 003MVS g/kg
39,75
-
-
MA RO 003NTK gN/kg
4,27
0,07
1,6
MA RA 001NH4
+gN/kg
2,23
0,15
6,7
MA RA 001DCO totale gO2/kg
110,48
4,34
3,9
MA RO 008DCO soluble
gO
2/kg
17,13
0,42
2,5
MA RO 008AGV totaux g/L
608
-
-
MA RC 002AGV totaux gC/L
260
-
-
MA RC 002Acide Acétique g/L
412
-
-
MA RC 002Acide Propionique
g/L
196
-
-
MA RC 002Acide Isobutyrique
g/L
nd
-
-
MA RC 002Acide Butyrique
g/L
nd
-
-
MA RC 002Acide Isovalérique
g/L
nd
-
-
MA RC 002Acide Valérique g/L
nd
-
-
MA RC 002Chlorure mg/L
1559
-
-
MA RI 001Phosphate mg P /L
18
-
-
MA RI 001 CemOA : archive ouverte d'Irstea / CemagrefSulfate mg S /L
4
-
-
MA RI 001Tableau 3 : Rapport des analyses physico-chimiques réalisées sur l’échantillon B2
UR GERE Rennes
Rapport d'essai
Réf : EN RR 001 Version : 1 Date: 28/11/06 Page 1/1Projet :
Thèse PP
Type
déjection :
Sortie
post-fermenteur
Bosingen
Date prélèvement
:
9 avril 2008
Origine :
Suisse
Opérateur :
P PEU
Animal :
Intervalle de
confiance
Moyenne
Ecart type
CV %
Méthode
utilisée
pH
7,73
MS g/kg
49,00
1,21
2,5
MA RO 004MO g/kg
36,75
1,29
3,5
MA RO 004MES g/kg
30,10
-
-
MA RO 003MVS g/kg
24,08
-
-
MA RO 003NTK gN/kg
4,07
0,06
1,4
MA RA 001NH4
+gN/kg
2,29
0,08
3,4
MA RA 001DCO totale gO2/kg
66,97
2,29
3,4
MA RO 008DCO soluble gO2/kg
16,85
0,02
0,1
MA RO 008AGV totaux g/L
231
-
-
MA RC 002AGV totaux gC/L
92
-
-
MA RC 002Acide Acétique g/L
231
-
-
MA RC 002Acide Propionique
g/L
nd
-
-
MA RC 002Acide Isobutyrique
g/L
nd
-
-
MA RC 002Acide Butyrique g/L
nd
-
-
MA RC 002Acide Isovalérique
g/L
nd
-
-
MA RC 002Acide Valérique g/L
nd
-
-
MA RC 002Chlorure mg/L
1531
-
-
MA RI 001Phosphate mg P /L
25
-
-
MA RI 001Sulfate mg S /L
2
-
-
MA RI 001 CemOA : archive ouverte d'Irstea / CemagrefTableau 4 : Rapport des analyses physico-chimiques réalisées sur l’échantillon B3 UR GERE Rennes
Rapport d'essai
Réf : EN RR 001 Version : 1 Date: 28/11/06 Page 1/1Projet :
Thèse PP
Type
déjection :
Entrée réacteur
Saugealles
Date prélèvement
:
9 avril 2008
Origine :
Suisse
Opérateur :
P PEU
Animal :
Intervalle de
confiance
Moyenne
Ecart type
CV %
Méthode
utilisée
pH
7,23
MS g/kg
24,22
1,53
6,3
MA RO 004MO g/kg
19,60
0,92
4,7
MA RO 004MES g/kg
19,22
-
-
MA RO 003MVS g/kg
16,66
-
-
MA RO 003NTK gN/kg
1,38
0,02
1,7
MA RA 001NH4
+gN/kg
0,73
0,01
1,0
MA RA 001DCO totale gO2/kg
62,59
4,15
6,6
MA RO 008DCO soluble gO
2/kg
57,89
0,39
0,7
MA RO 008AGV totaux g/L
2036
-
-
MA RC 002AGV totaux gC/L
887
-
-
MA RC 002Acide Acétique g/L
1336
-
-
MA RC 002Acide Propionique
g/L
474
-
-
MA RC 002Acide Isobutyrique
g/L
nd
-
-
MA RC 002Acide Butyrique g/L
226
-
-
MA RC 002Acide Isovalérique
g/L
nd
-
-
MA RC 002Acide Valérique g/L
nd
-
-
MA RC 002Chlorure mg/L
250
-
-
MA RI 001 CemOA : archive ouverte d'Irstea / CemagrefPhosphate mg P /L
21
-
-
MA RI 001Sulfate mg S /L
20
-
-
MA RI 001Tableau 5 : Rapport des analyses physico-chimiques réalisées sur l’échantillon S1
UR GERE Rennes
Rapport d'essai
Réf : EN RR 001 Version : 1 Date: 28/11/06 Page 1/1Projet :
Thèse PP
Type
déjection :
Entrée co-substrat
(huile) réacteur
Saugealles
Date prélèvement
:
9 avril 2008
Origine :
Suisse
Opérateur :
P PEU
Animal :
Intervalle de
confiance
Moyenne
Ecart type
CV %
Méthode
utilisée
pH
4,08
MS g/kg
358,50
1,21
0,3
MA RO 004MO g/kg
347,21
1,29
0,4
MA RO 004MES g/kg
148,75
-
-
MA RO 003MVS g/kg
139,85
-
-
MA RO 003NTK gN/kg
5,58
0,06
1,0
MA RA 001NH4
+gN/kg
0,12
0,00
3,5
MA RA 001DCO totale gO2/kg
-
-
-
MA RO 008DCO soluble
gO
2/kg
-
-
-
MA RO 008AGV totaux g/L
-
-
-
MA RC 002AGV totaux gC/L
-
-
-
MA RC 002Acide Acétique g/L
-
-
-
MA RC 002Acide Propionique
g/L
-
-
-
MA RC 002Acide Isobutyrique
g/L
-
-
-
MA RC 002Acide Butyrique g/L
-
-
-
MA RC 002Acide Isovalérique
g/L
-
-
-
MA RC 002Acide Valérique g/L
-
-
-
MA RC 002 CemOA : archive ouverte d'Irstea / CemagrefChlorure mg/L
-
-
-
MA RI 001Phosphate mg P /L
-
-
-
MA RI 001Sulfate mg S /L
-
-
-
MA RI 001Tableau 6 : Rapport des analyses physico-chimiques réalisées sur l’échantillon S2
UR GERE Rennes
Rapport d'essai
Réf : EN RR 001 Version : 1 Date: 28/11/06 Page 1/1Projet :
Thèse PP
Type
déjection :
Sortie
post-fermenteur
Saugealles
Date prélèvement
:
9 avril 2008
Origine :
Suisse
Opérateur :
P PEU
Animal :
Intervalle de
confiance
Moyenne
Ecart type
CV %
Méthode
utilisée
pH
7,54
MS g/kg
82,56
0,56
1
MA RO 004MO g/kg
64,44
0,52
1
MA RO 004MES g/kg
66,21
-
-
MA RO 003MVS g/kg
53,48
-
-
MA RO 003NTK gN/kg
3,17
0,11
3
MA RA 001NH4
+gN/kg
1,14
0,08
7
MA RA 001DCO totale gO2/kg
121,79
-
-
MA RO 008DCO soluble gO
2/kg
23,95
0,92
4
MA RO 008AGV totaux g/L
nd
-
-
MA RC 002AGV totaux gC/L
nd
-
-
MA RC 002Acide Acétique g/L
nd
-
-
MA RC 002Acide Propionique
g/L
nd
-
-
MA RC 002Acide Isobutyrique
g/L
nd
-
-
MA RC 002Acide Butyrique g/L
nd
-
-
MA RC 002Acide Isovalérique
g/L
nd
-
-
MA RC 002 CemOA : archive ouverte d'Irstea / CemagrefAcide Valérique g/L
nd
-
-
MA RC 002Chlorure mg/L
940
-
-
MA RI 001Phosphate mg P /L
40
-
-
MA RI 001Sulfate mg S /L
3
-
-
MA RI 001Tableau 7 : Rapport des analyses physico-chimiques réalisées sur l’échantillon S3
UR GERE Rennes
Rapport d'essai
Réf : EN RR 001 Version : 1 Date: 28/11/06 Page 1/1Projet :
Thèse PP
Type
déjection :
Sortie post-fermenteur Saugealles après séparationde phase
Date prélèvement
:
9 avril 2008
Origine :
Suisse
Opérateur :
P PEU
Animal :
Intervalle de
confiance
Moyenne
Ecart type
CV %
Méthode
utilisée
pH
7,93
MS g/kg
48,65
1,06
2
MA RO 004MO g/kg
33,40
0,26
1
MA RO 004MES g/kg
31,68
-
-
MA RO 003MVS g/kg
22,81
-
-
MA RO 003NTK gN/kg
2,72
0,02
1
MA RA 001NH
4+gN/kg
1,14
0,08
7
MA RA 001DCO totale gO
2/kg
67,46
2,29
3
MA RO 008DCO soluble gO
2/kg
22,11
0,02
0
MA RO 008AGV totaux g/L
nd
-
-
MA RC 002AGV totaux gC/L
nd
-
-
MA RC 002Acide Acétique g/L
nd
-
-
MA RC 002Acide Propionique
g/L
nd
-
-
MA RC 002Acide Isobutyrique
g/L
nd
-
-
MA RC 002Acide Butyrique g/L
nd
-
-
MA RC 002Acide Isovalérique
nd
-
-
MA RC 002 CemOA : archive ouverte d'Irstea / Cemagrefg/L
Acide Valérique g/L
nd
-
-
MA RC 002Chlorure mg/L
735
-
-
MA RI 001Phosphate mg P /L
26
-
-
MA RI 001Sulfate mg S /L
4
-
-
MA RI 001Tableau 8 : Rapport des analyses physico-chimiques réalisées sur l’échantillon S4
Figure 15 : Quantification des archaea totales. Les quantités sont exprimées en cellules/ml
Figure 16 : Quantification des bactéries totales. Les quantités sont exprimées en cellules/ml
CemOA
: archive
ouverte
d'Irstea