HAL Id: tel-01674168
https://tel.archives-ouvertes.fr/tel-01674168
Submitted on 2 Jan 2018HAL is a multi-disciplinary open access archive for the deposit and dissemination of sci-entific research documents, whether they are pub-lished or not. The documents may come from teaching and research institutions in France or abroad, or from public or private research centers.
L’archive ouverte pluridisciplinaire HAL, est destinée au dépôt et à la diffusion de documents scientifiques de niveau recherche, publiés ou non, émanant des établissements d’enseignement et de recherche français ou étrangers, des laboratoires publics ou privés.
surface de nano-objets pour des applications biologiques
Laurent Adumeau
To cite this version:
Laurent Adumeau. Développement de stratégies de biofonctionnalisation de surface de nano-objets pour des applications biologiques. Matériaux. Université de Bordeaux, 2015. Français. �NNT : 2015BORD0425�. �tel-01674168�
THÈSE PRÉSENTÉE POUR OBTENIR LE GRADE DE
DOCTEUR DE
L’UNIVERSITÉ DE BORDEAUX
ÉCOLE DOCTORALE DES SCIENCES CHIMIQUES SPÉCIALITÉ : PHYSICOCHIMIE DE LA MATIÈRE CONDENSÉE
Par Laurent ADUMEAU
DEVELOPPEMENT DE STRATEGIES DE BIOFONCTIONNALISATION
DE SURFACE DE NANO-OBJETS POUR DES APPLICATIONS
BIOLOGIQUES
Sous la direction de : M. Stéphane MORNET
Soutenue le 9 décembre 2015 Membres du jury :
M. MAGLIONE, Mario Directeur de recherche CNRS Président
Mme GAZEAU, Florence Directrice de recherche CNRS Rapporteur
M. TILLEMENT, Olivier Professeur Université Claude Bernard Lyon 1 Rapporteur
M. RICHARD, Cyrille Chargé de recherche HDR CNRS Examinateur
M. BRISSON, Alain Professeur Université de Bordeaux Examinateur M. MORNET, Stéphane Chargé de recherche HDR CNRS Directeur de thèse
Parce qu’une thèse est en réalité le résultat d’un travail collectif, j’ai tenu à écrire les quelques lignes suivantes pour remercier les différentes personnes qui ont contribué à ce résultat.
Les travaux de thèse décrits dans ce manuscrit ont principalement été réalisés au sein de l’Institut de Chimie de la Matière Condensée de Bordeaux (ICMCB). Ainsi, je remercie Claude Delmas, ancien directeur, ainsi que Mario Maglione, directeur actuel, pour m’avoir accueilli au sein de l’institut. Merci également à Etienne Duguet, responsable du groupe « Chimie des nanomatériaux » pour son accueil.
J’adresse mes plus sincères remerciements à Mme Florence Gazeau et M. Olivier Tillement pour le temps qu’ils ont consacré à rapporter ce travail, ainsi qu’à M. Cyrille Richard et M. Alain Brisson pour avoir accepté d’examiner cette thèse, et enfin, à Mario Maglione pour avoir présidé ce jury de thèse. Je remercie tous les membres du jury pour l’intérêt qu’ils ont porté à ce travail et pour les discussions constructives lors de ma soutenance.
Je tiens à exprimer ma profonde gratitude à mon directeur de thèse, Stéphane Mornet, pour de nombreuses raisons. Tout d’abord, merci d’avoir fait confiance au « biologiste » et de m’avoir permis de faire cette thèse en chimie. Merci également de ne pas avoir hésité à m’impliquer dans plusieurs projets et de m’avoir fait rencontrer de nombreux chercheurs bordelais aux cours des réunions avec lesquels j’ai toujours eu la sensation d’être écouté et considéré durant mes prises de paroles. Merci pour la grande liberté que tu m’as laissée tout en maintenant un échange permanent. Merci pour ta disponibilité, je savais que je pouvais venir te voir et repartir avec de nouvelles idées. Enfin, je te suis redevable pour le temps que tu as consacré à me conseiller et à corriger ce manuscrit de thèse. Merci pour tous ces points formateur.
Je remercie, à l’ICMCB, les personnes qui ont contribué à cette thèse, au travers de discussions scientifiques ou en me donnant accès à différentes caractérisations : Patrick Rosa, Olivier Toulemonde, Thierry Cardinal, Alexandre Fargue, Olivier Nguyen, Laetitia Etienne, Dominique Denux, Etienne Durant, et Mathieu Duttine. Merci également aux services collectifs (accueil, administration et infrasctructure) qui nous apportent un soutien quotidien et nous permettent de travailler dans de bonnes conditions et notamment à Stéphane Toulin de la Bibliothèque et Carole Malburet du service mission.
Cette thèse étant le fruit de différentes collaborations, j’ai été amené à interagir avec plusieurs laboratoires bordelais : l’institut de Chimie et Biologique des Membranes et des Nano-objets (CBMN), le laboratoire de Résonance Magnétique des Systèmes Biologiques (RMSB) et le Laboratoire de Chimie des Polymères Organiques (LCPO).
Au CBMN, je souhaite adresser mes sincères remerciements à Alain Brisson pour l’accueil dans son équipe, pour les discussions scientifiques et pour les conseils qu’il m’a donnés, à Céline Gounou pour son efficacité, ainsi qu’à Romain Linares, Nicolas Arraud, et Sisareuth Tan. Merci également à Olivier Lambert pour les discussions scientifiques.
Au sein du RMSB, je voudrais remercier Gisèle Clofent-Sanchez pour l’accueil dans son équipe, Marie-Josée Jacobin Valat et Jeanny Laroche-Traineau pour leur bonne humeur inoxydable, ainsi que Mélusine Larivière, Audrey Hemadou et Cyril Lorenzato. Merci également à Franck Couillaud pour les éclaircissements sur les tumeurs, à Coralie Germain-Genevois, ainsi qu’à Claire Marozzo et Cyril Masante
Au LCPO, merci à Olivier Sandre et Christophe Schatz pour les discussions scientifiques, Anne-Laure Wirotius pour la RMN et Gauvin Hemery pour l’accès aux mesures de DLS.
Je remercie Geoffrey Prévot, Romain Melich, Kadiali Bodiang et Marine Delgado, les différents stagiaires que j’ai eu le plaisir d’encadrer, pour le travail de qualité qu’ils ont fourni, ainsi que pour les petites expériences de dépannage sur la fin. J’en profite pour leur souhaiter une bonne continuation.
Bien entendu, je n’oublie pas les membres du groupe V pour les discussions scientifiques, l’accueil et la bonne humeur. Ils ont contribué à faire de ces 3 années un moment inoubliable. Ma reconnaissance va à Marie-Hélène Delville, Graziella Goglio (que devient Jamal ?), Jérome Majimel, Mona Treguer-Delapierre, et Lydia Roudier. Merci à Nora avec qui j’ai fait mon stage de Master 2. Je remercie les post-docs du groupe pour les discussions et leurs conseils : Hélène, Noelia et Sergio. Merci aux anciens : Hakim, Thomas, Aurélie et Quentin. Merci également à Cyril et Stéphanie avec qui j’ai commencé et fini cette thèse, je repense à la bonne ambiance et l’entraide qu’il y avait entre nous. Je voudrais également souhaiter bon courage aux « thésards » actuels : Uli, Arnaud, Magali, Alexandra, Véronique, Bai, Weiya et Cécilia.
Merci également à mes amis et, en particulier, à Cécile à qui j’exprime ma profonde reconnaissance pour le grand soutien qu’elle m’a apporté tout au long de cette thèse. Je voudrais également marquer le courage dont mes colocs ont dû faire preuve pour me supporter au quotidien, notamment David, avec qui, je partage une vision différente des programmes TV de l’été, mais également à Alex et Salla, arrivés pendant la période de rédaction.
Enfin, je remercie mes parents pour m’avoir transmis cet esprit de curiosité et m’avoir toujours donné les moyens de faire les études de mon choix. Merci également à mon « p’tit » frère d’avoir, en quelque sorte, ouvert le chemin.
LISTE DES ABREVIATIONS ... V
INTRODUCTION GENERALE ... 1
RÉFÉRENCES ... 5
CHAPITRE I. GESTION DES INTERACTIONS ENTRE LA SURFACE DES NANOPARTICULES ET LEUR MILIEU BIOLOGIQUE D’APPLICATION ... 7
A. INTERACTIONS ENTRE LES NANOPARTICULES ET L'ENVIRONNEMENT BIOLOGIQUE ... 10
A.1 Stabilité colloïdale des nanoparticules dans un milieu simple ... 11 A.2 Comportement des nanoparticules dans un milieu biologique ... 12 A.2.1 Mécanisme de formation de la couronne biomoléculaire ... 12 A.2.2 Dynamique de la couronne biomoléculaire ... 13 A.2.3 Interactions entre le complexe nanoparticule‐couronne biomoléculaire et le milieu biologique. ... 15 A.2.4 Conclusion ... 17 A.3 Gestion des interactions non spécifiques ... 18 A.3.1 Les polymères anti‐adsorption ... 18 A.3.2 Etude conformationnelle des chaines linéaires de PEG à la surface d’une nanoparticule ... 22 A.4 Conclusion ... 26 B. BIOFONCTIONNALISATION DE NANOPARTICULES PAR CONJUGAISON DE BIOMOLECULES. ... 26 B.1 Modification chimique de la surface des nanoparticules ... 27 B.2 Stratégies de bioconjugaison ... 30 B.2.1 Bioconjugaison par liaison chimique ... 31 B.2.2 Bioconjugaison par reconnaissance spécifique entre deux biomolécules ... 35 B.3 Conclusion ... 36 C. DESCRIPTION DU PROJET D’ETUDE ... 37 C.1 Extraction de vésicules extracellulaires d’un fluide biologique ... 37 C.2 Diagnostic de l’athérosclérose par imagerie par résonance magnétique ... 40 C.3 Marquage in vivo du cancer de la prostate pour l’imagerie de fluorescence dans le proche infrarouge. ... 45 C.4 Stratégie générale ... 47 REFERENCES BIBLIOGRAPHIQUES ... 49 CHAPITRE II. DEVELOPPEMENT D’UNE STRATEGIE DE PEGYLATION COVALENTE DE SURFACES DE NANOPARTICULES D’OXYDES METALLIQUES ... 55
A. FONCTIONNALISATION DE LA SURFACE DE NANOPARTICULES PAR DES MACROMOLECULES DE PEG ... 58
A.1 Voie directe : Modification chimique de la surface de nanoparticules de silice par des agents de couplage silanique portant un bras macromoléculaire de PEG ... 58
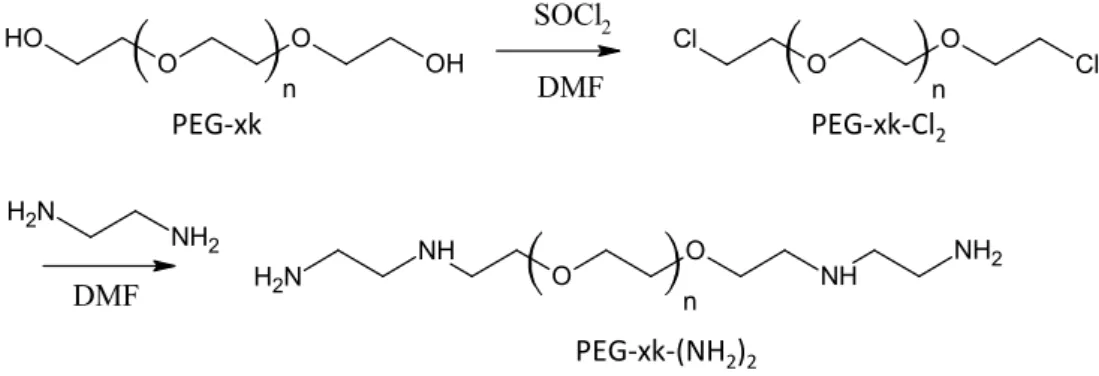
A.1.2 Caractérisation de la surface des nanoparticules de silice modifiées par le mPEG‐ silane ... 61 A.1.3 Conclusion ... 64 A.2 Voie indirecte : Greffage covalent de macromolécules de PEG par ancrage sur des sites fonctionnels présents à la surface de la nanoparticule. ... 65 A.1.1 Modification chimique de la surface de nanoparticules de silice avec un agent de couplage silanique aminé ... 66 A.1.1.1 Greffage de l’aminosilane à la surface des nanoparticules de silice ... 66 A.2.1.1 Caractérisations de la modification de surface ... 67 A.2.1.2 Conclusion ... 71 A.2.2 Conversion des extrémités du PEG en fonctions aldéhyde ... 71 A.2.2.1 Synthèse de PEG‐al par oxydation de Pfitzner‐Moffatt ... 71 A.2.2.2 Caractérisation des macromolécules modifiées ... 72 A.2.3 PEGylation de surfaces aminés de nanoparticules de silice ... 76 A.2.3.1 Greffage covalent du PEG‐al sur les nanoparticules aminées ... 77 A.2.3.2 Extension du procédé de greffage du PEG‐al sur les nanoparticules aminées ... 83 A.2.4 Discussion ... 86 A.3 Etude de la conformation des macromolécules de PEG greffées à la surface des nanoparticules et estimation de l’épaisseur de la couronne polymère ... 87 A.4 Conclusion ... 89 B. FONCTIONNALISATION DE LA COURONNE DE PEG ... 90 B.1 Amination de macromolécules de PEG libres ... 91 B.1.1 Synthèse du PEG‐4k‐Cl2 ... 91 B.1.2 Synthèse du PEG‐4k‐(NH2)2 ... 94 B.2 Application à l'amination de la couronne polymère greffée autour des nanoparticules de silice ... 96 B.3 Conclusion ... 99 C. ETUDE DES PROPRIETES DES NANOPARTICULES CŒUR‐COURONNE ... 100 C.1 Stabilité colloïdale des nanoparticules ... 100 C.2 Propriété antiadhésive envers les protéines ... 100 D. CONCLUSION ... 102 REFERENCES BIBLIOGRAPHIQUES ... 104 CHAPITRE III. SYNTHESE ET BIOFONCTIONNALISATION DE NANOPARTICULES INORGANIQUES DESTINEES A DES APPLICATIONS BIOTECHNOLOGIQUES OU BIOMEDICALES... 107
A. SYNTHESE ET BIOFONCTIONNALISATION DE SUPRAPARTICULES COLLOÏDALES DESTINEES A L’EXTRACTION PAR MAGNETOPHORESE DE VESICULES EXTRACELLULAIRES CIRCULANTES ... 109
A.1 L’extraction par magnétophorèse d’objets biologiques ... 109
A.2.1.1 Formulation d’un ferrofluide hydrophobe ... 115 A.2.1.2 Assemblage des nanoparticules magnétiques ... 117 A.2.2 Consolidation des SPC50 dans une matrice de silice ... 121 A.2.2.1 Transfert des SPC50 en milieu hydro‐alcoolique ... 121 A.2.2.2 Encapsulation dans une matrice de silice par voie sol‐gel ... 125 A.2.3 Caractérisation magnétique des SPC ... 130 A.2.4 Conclusion ... 131
A.3 Biofonctionnalisation des SPC@SiO2 par une protéine de reconnaissance et de capture des vésicules lipidiques ... 132
A.3.1 Formation d’une couronne de macromolécules de PEG à la surface des SPC fonctionnalisées par des amines ... 133
A.3.2 Greffage de l’annexine A5 ... 135
A.4 Conclusion ... 139
B. MARQUAGE PAR DES SONDES FLUORESCENTES DANS LE PIR ET BIOFONCTIONNALISATION D’UN AGENT DE CONTRASTE DESTINE AU DIAGNOSTIC DE L’ATHEROSCLEROSE PAR IRM. ... 140
B.1 Marquage de l’agent de contraste avec un fluorophore dans le proche infrarouge . ... 140 B.1.1 Fonctionnalisation de l’agent de contraste par des fonctions thiol ... 141 B.1.2 Greffage du fluorophore ... 143 B.2 Biofonctionnalisation de l’agent de contraste par greffage régiosélectif d’un fragment d’anticorps ... 145 B.2.1 Activation des fragments d’anticorps ... 145 B.2.2 Greffage des fragments d’anticorps ... 147 B.3 Conclusion ... 149 C. ELABORATION ET CHIMIE DE SURFACE DE NANOPARTICULES DE SILICE FLUORESCENTES DESTINEES AU MARQUAGE IN VIVO DE TUMEURS. ... 151 C.1 Synthèse des nanoparticules bi‐fluorescentes ... 151 C.2 Conclusion ... 161 D. CONCLUSION ... 162 REFERENCES BIBLIOGRAPHIQUES ... 163 CHAPITRE IV. EVALUATION DES NANO‐OBJETS DANS LE CADRE DE LEUR APPLICATION BIOLOGIQUE ... 165 A. EXTRACTION DE VESICULES EXTRACELLULAIRES PAR MAGNETOPHORESE ... 167 A.1 Etude des interactions entre les SPC biofonctionnalisées et les bicouches lipidiques par QCM‐D ... 167 A.2 Tests d’extraction d’objets membranaires ... 176 A.2.1 Tests préliminaires d’extraction de vésicules lipidiques fluorescentes ... 178 A.2.2 Extraction d’objets membranaires d’origine biologique ... 180 A.3 Conclusion ... 183
B.1 Marquage de plaques d’athérome vulnérables ... 184 B.1.1 Etude des propriétés de reconnaissance des VUSPIO dirigés contre la cible intégrine αIIbβ3 ... 184 B.1.1.1 Etude de la reconnaissance biomoléculaire par résonance plasmonique de surface ... 184 B.1.1.2 Mise en évidence du marquage spécifique des zones pathologiques par immunohistochimie ... 189 B.1.1.3 Conclusion ... 190 B.1.2 Etude in vivo du marquage des plaques d’athérome ... 190 B.2 Test de ciblage in vivo de tumeurs avec des VUSPIO fonctionnalisées par des ligands acides foliques. ... 192 B.3 Conclusion ... 194
C. MARQUAGE DE TUMEURS PAR DES NANOPARTICULES HYBRIDES DE SILICE FLUORESCENTES PEGYLEES. ... ... 194
D. CONCLUSION ... 197
REFERENCES BIBLIOGRAPHIQUES ... 199
CONCLUSION GENERALE ET PERSPECTIVES ... 201
ANNEXES ... I ANNEXE NO 1 : STABILITE COLLOÏDALE DES NANOPARTICULES DANS UN MILIEU SIMPLE ... III
ANNEXE NO 2 : SYNTHESE DE NANOPARTICULES DE SILICE MODELES ...X
ANNEXE NO 3 : DETERMINATION DE LA DENSITE SURFACIQUE DES MOLECULES GREFFEES ... XVII ANNEXE NO 4 : SYNTHESE ET CARACTERISATION DE NANOPARTICULES D’OXYDE DE FER
SUPERPARAMAGNETIQUES PAR CO‐PRECIPITATION ... XX ANNEXE NO 5 : DOSAGE SPECTROPHOTOMETRIQUE DES IONS FE3+ DANS LES FERROFLUIDES PAR
COMPLEXATION AVEC LE THIOCYANATE DE POTASSIUM ... XXIV ANNEXE NO 6 : MISE AU POINT DES CONDITIONS DE BIOCONJUGAISON SUR LES VUSPIO AVEC UNE PROTEINE MODELE (ANX‐A5) ... XXVI ANNEXE NO 7 : LA MICROBALANCE A CRISTAL DE QUARTZ AVEC MESURE DE DISSIPATION (QCM‐D) ... XXIX
APTES (3-aminopropyl)triéthoxysilane
AcOH Acide acétique
Anx-A5 Annexine-A5
ARN Acide ribonucléique
ARNm ARN messagé
ARNmi micro ARN interferant
ATG Analyse thermogravimétrique
BSA Bovine Serum Albumine
CBMN Laboratoire de Chimie et de Biologie des Membranes et des Nano-objets de Bordeaux
CDCl3 Chloroforme deutéré
CTAB Bromure de cetyltriméthylammonium Cy 7.5 Cyanine 7.5
DCC N,N'-Dicyclohexylcarbodiimide DCM Dichlorométhane
DLS Dynamic Light Scattering DMF Diméthylformamide DMSO Diméthylsulfoxyde
DOPC Dioléoylphosphatidylcholine DOPS Dioléoylphosphatidylsérine
DRIFT Diffuse reflectance infrared fourier transform
DTNB Dithionitrobenzoic acid; 5,5'-Dithiobis(2-nitrobenzoic acid) EDC 1-éthyl-3-(3-diméthylaminopropyl) carbodiimide EDPS N-[3-(Trimethoxysilyl)propyl]ethylenediamine EDTA Ethylène Diamine Tétra Acétique
EPR Enhanced Permeability and Retention
ET3N Triéthylamine
EtOH Ethanol
FDA Food Drug Administration FITC Isothiocyanate de fluoescéine FRET Förster Resonance Energy Transfer
GNP Nanoparticule d'or
HEPES 4-(2-hydroxyethyl)-1-piperazineethanesulfonic acid ICMCB Institut de Chimie de la Matière Condensée de Bordeaux ICP Inductive Coupled Plasma
IECB Institut Européen de Chimie et de Biologie Ig Immunoglobuline
IR Infrarouge
IRM Imagerie par Résonance Magnétique KSCN Thiocyanate de potassium
LDL Low Density Lipoprotein
MeOH Méthanol
MET Microscopie électronique à transmission
MLV MultiLamelar Vesicle
NHS N-Hydroxysuccinimide NP Nanoparticule
NTA Acide nitrilotriacétique
OHP Plan externe de Helmholtz PC Phosphatidylcholine PCB Poly(méthacrylate de carboxybetaïne) PDI Indice de polydispersité
PEG Poly(éthylène glycol)
PIR Proche InfraRouge
POE Poly(oxyde d'éthylène)
PS Phosphatidylsérine PVP Polyvinylpyrrolidone Py Pyridine
QCM-D Microbalance à Cristal de Quartz avec mesure de Dissipation
QD Quantum dot
RMN Résonance Magnétique Nucléaire
RMSB Centre de Résonance Magnétique des Systèmes Biologiques
SAM Monocouche auto-assemblée
scFv Single chain Fragment variable
SDS Sodium dodecylsulfate
SOCl2 Chlorure de thionyle
SPC Supraparticule colloïdale
SPIO SuperParamagnetic Iron Oxide SPR Surface plasmon resonance
SRE Système Réticulo-Endothélial
SUV Small Unilamelar Vesicle
TCEP Tris(2-carboxyethyl)phosphine hydrochloride
TE Température d'ébullition
TEOS Tetraethylorthosilicate TFAA Acide trifluoroacetique THF Tétrahydrofurane
TNB 3-thio-6-nitrobenzoic acid
USPIO Ultrasmall Superparamagnetic Iron Oxide vdW van der Waals
VE Vésicule extracellulaire
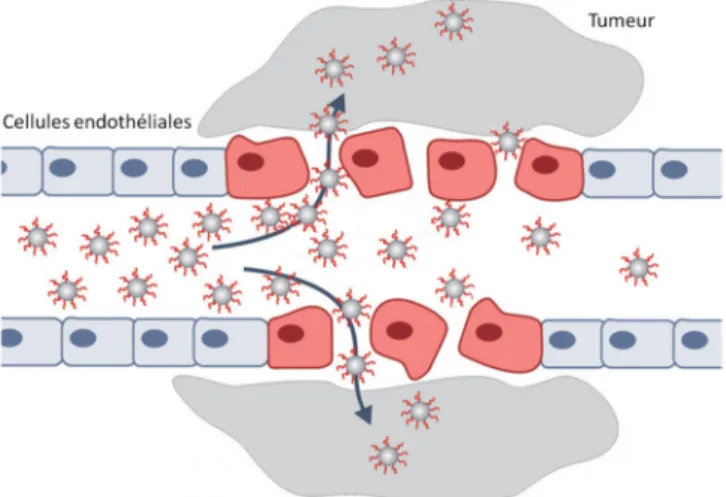
Le développement de nano-objets pour des applications biomédicales constitue un domaine de recherche qui a connu depuis les deux dernières décennies un fort essor. Cette recherche intensive a fait naitre de nombreuses plateformes pour la délivrance de médicament,1 le marquage en bio-imagerie,2,3 ou encore, la bio-détection.4–6 Ce grand intérêt pour les nanomatériaux en biologie s’explique par les nouvelles propriétés physiques que présentent les matériaux divisés à l’échelle nanoscopique, telles que des propriétés magnétiques, ou optiques. Sous cette forme, la matière présente une surface développée considérable. La notion de gestion de l’interface devient donc un domaine d’étude à part entière, tant elle est importante pour ces applications. La surface est le lieu de rencontre entre le matériau et l’environnement biologique complexe, et les propriétés physico-chimiques de cette interface vont diriger le comportement des nano-objets. Selon les spécificités de l’application en biologie et de la nature chimique du matériau, des stratégies de modifications chimiques sont mises en places. Celles-ci ont pour but principalement d’adresser la problématique de la gestion des interactions entre les nanoparticules et leur environnement. Un des principaux problèmes à traiter concerne l’adsorption spontanée des biomolécules à leur surface via des interactions multiples (van der Waals, électrostatiques…), pouvant provoquer la perte du contrôle des propriétés recherchées des nanoparticules dans leur milieu d’application, et donc de potentiels problèmes.7,8 La maitrise de la chimie de surface élargit, également, les champs d’applications potentiels en biologie, en rendant possible le couplage entre les propriétés physiques intrinsèques des nanoparticules avec des fonctions biologiques apportées par la conjugaison de biomolécules.9 La biofonctionnalisation fait appel à des stratégies complexes prenant en compte les spécificités de la biomolécule ainsi que celles de l’application (Figure 1). Le greffage de la biomolécule ainsi que la conservation de son activité biologique ne peuvent ainsi être réalisés que si la chimie, ainsi que les propriétés physico-chimiques de surface sont parfaitement maîtrisées.
Figure 1 : Schéma résumant les principaux paramètres de surface à considérer pour la fonctionnalisation de nanoparticules par des molécules biologiques.
Les travaux présentés dans ce mémoire sont focalisés sur la mise en place de nano-systèmes biofonctionnalisés développée, en étroite collaboration avec des équipes de biologistes, dans le cadre des applications suivantes :
- L’extraction magnétique de vésicules extracellulaires d’un fluide biologique. - Le diagnostic de l’athérosclérose par imagerie in vivo.
- Le marquage de tumeurs pour l’imagerie in vivo dans le proche infrarouge.
Ces applications, qui seront détaillées au cours de ce manuscrit, semblent certes à première vue différentes, mais présentent toutefois une problématique commune du point de vue de la chimie de surface des nano-objets. De plus, elles possèdent un objectif commun portant sur l’adressage de nano-objets par reconnaissance moléculaire en milieu biologique que ce soit pour cibler sélectivement une zone pathologique ou pour extraire des objets biologiques.
Dans le but de développer les systèmes dans le cadre de ces applications biologiques et de répondre à cette problématique commune, les objectifs de l’étude étaient de :
1) Développer les nano-objets portant la propriété physique adaptée à l’application ; 2) Gérer la stabilité colloïdale de ces nano-objets à chaque étape de leur élaboration
jusqu’au milieu biologique d’application ;
3) Contrôler leur propriétés physico-chimiques de surface afin de limiter l’adsorption non spécifique de biomolécules ;
4) Contrôler la chimie de surface pour mieux maîtriser les étapes ultérieures de greffage des éléments de ciblage tout en conservant la fonction de reconnaissance biomoléculaire ;
5) Promouvoir la propriété de reconnaissance moléculaire en cherchant à apporter un grand degré de liberté à la biomolécule greffée, en réalisant des greffages régiosélectifs et en contrôlant le nombre de biomolécules greffées par nano-objet. Ce mémoire est divisé en quatre chapitres :
Le premier présente la problématique générale de l’étude. L’accent est principalement mis sur la description des interactions entre les nanoparticules et l’environnement biologique ainsi que sur les moyens disponibles pour gérer ces interactions. Les problématiques propres aux différentes applications seront par la suite présentées.
La seconde partie est consacrée au développement de greffages covalents haute densité de macromolécules de poly(oxyde d’éthylène) sur des surfaces de silice
nanométriques. Les nanoparticules cœur-couronne ainsi obtenues servent, par la suite, de base pour l’étude de bioconjugaison.
Le troisième expose la construction des différents nano-objets au regard de leur application biologique.
Enfin le quatrième chapitre présente les premiers résultats biologiques obtenus avec les nano-systèmes développés au cours du chapitre précédent dans le cadre de leur application respective.
Références
(1) Li, Z.; Barnes, J. C.; Bosoy, A.; Stoddart, J. F.; Zink, J. I. Chem. Soc. Rev. 2012, 41 (7), 2590–2605.
(2) De, M.; Ghosh, P. S.; Rotello, V. M. Adv. Mater. 2008, 20 (22), 4225–4241.
(3) Lee, D.-E.; Koo, H.; Sun, I.-C.; Ryu, J. H.; Kim, K.; Kwon, I. C. Chem. Soc. Rev. 2012, 41 (7), 2656–2672.
(4) Swierczewska, M.; Liu, G.; Lee, S.; Chen, X. Chem. Soc. Rev. 2012, 41 (7), 2641– 2655.
(5) Saha, K.; Agasti, S. S.; Kim, C.; Li, X.; Rotello, V. M. Chem. Rev. 2012, 112 (5), 2739–2779.
(6) Jans, H.; Huo, Q. Chem. Soc. Rev. 2012, 41 (7), 2849–2866.
(7) Monopoli, M. P.; Åberg, C.; Salvati, A.; Dawson, K. A. Nat. Nanotechnol. 2012, 7 (12), 779–786.
(8) Walkey, C. D.; Chan, W. C. W. Chem. Soc. Rev. 2012, 41 (7), 2780–2799.
(9) Sapsford, K. E.; Algar, W. R.; Berti, L.; Gemmill, K. B.; Casey, B. J.; Oh, E.; Stewart, M. H.; Medintz, I. L. Chem. Rev. 2013, 113 (3), 1904–2074.
Chapitre I. Gestion des interactions
entre la surface des nanoparticules et
Les nanoparticules trouvent actuellement de nombreuses applications en biologie, que cela soit dans le domaine biomédical, ou biotechnologique. Cela s'explique par les propriétés physiques singulières remarquables qu'elles présentent à l’échelle nanométrique (1-100 nm) par rapport au matériau massif. La matière ainsi finement divisée possède également une surface spécifique très élevée. Ce domaine de recherche, actuellement très actif, fait régulièrement l'objet de revues dans lesquelles les différentes propriétés des nanoparticules sont abordées ainsi que les applications pour lesquelles elles sont envisagées.1–5 Depuis plus de vingt ans, les chimistes développent de nouvelles synthèses de nanoparticules afin d’améliorer le contrôle de la morphologie et des propriétés. En dehors de leurs propriétés physiques qui ont fait l’objet de nombreuses publications et ouvrages, il est également pertinent, dans le contexte biotechnologique de s’intéresser au comportement des nanoparticules dans l’environnement biologique. L’interface nanoparticule-milieu biologique est le lieu où se concentrent toutes les interactions qui conditionneront le rôle que peuvent jouer les nanoparticules dans le cadre d’applications en biologie. Du fait de la surface spécifique élevée développée par les nanoparticules, la gestion de ces interactions se résume en un travail d’ingénierie de surface de matériaux divisés et dispersés en milieu liquide.
L’environnement biologique au sens large est un système complexe, que cela soit à l’échelle d’un organisme entier ou à l’échelle d’un fluide biologique. C’est un milieu caractérisé par une très grande diversité de molécules, allant de simples sels, à des (macro)molécules complexes tels que les protéines, les acides nucléiques ou encore les lipides. Certaines de ces (macro)molécules sont-elles-mêmes capables de s’auto-assembler. C’est un milieu dynamique au sein duquel des réactions chimiques s’y produisent constamment, et où la matière est en mouvement. Les organismes vivants sont compartimentés et hiérarchisés par des barrières biologiques et donnent lieu à des compositions variables entre les compartiments.
Dans le cadre d’applications biologiques, la notion d’adressage est très importante. En effet, pour présenter un rôle biologique, une nanoparticule doit avoir un impact sur une cible particulaire (molécules, cellule, etc…). Il est donc nécessaire que la nanoparticule puisse reconnaitre sa cible dans cet environnement complexe. Pour intégrer cette notion de ciblage, la nanoparticule doit répondre idéalement aux deux critères suivants : une grande affinité et qu’une grande spécificité pour sa cible. Alors que l’affinité traduit la force avec laquelle se forme un complexe ligand-cible, la spécificité représente quant à elle la capacité du ligand à se lier préférentiellement à sa cible plutôt qu’à d’autres objets. Afin d’optimiser ses deux
propriétés, il est capital de connaitre et de comprendre le comportement des nanoparticules dans un milieu biologique.
A. Interactions entre les nanoparticules et l'environnement
biologique
Le comportement des nanoparticules dans un milieu biologique est fonction d’une multitude de facteurs, tels que leur taille, leur forme, ou la nature de leur surface (charge, balance hydrophile/hydrophobe), mais également de la composition en biomolécule et de l’environnement physiologique (Figure I.1).6
Figure I.1 : Schématisation des différents facteurs physico‐chimiques influençant les interactions entre les nanoparticules et les biomolécules. Adaptée de la référence.6
Cette multitude de paramètres physico-chimiques explique la grande difficulté pour prédire avec précision le comportement des nanoparticules dans un milieu biologique. Il est cependant actuellement possible de décrire les comportements généraux.
Avant de nous intéresser aux interactions des nanoparticules dans le milieu d’application réel, nous allons dans un premier temps poser les bases de la stabilisation colloïdale décrivant leur comportement dans un milieu simple.
A.1 Stabilité colloïdale des nanoparticules dans un milieu simple
Une suspension colloïdale est, par définition, un système composé d’au moins une phase dispersée (colloïde) dans une phase dispersante. La gravité a un effet négligeable sur les colloïdes par rapport à ceux de l’agitation thermique. Le système ne sédimente généralement donc pas sur des temps courts, et de nouvelles forces gouvernent la stabilité colloïdale. La notion de stabilité colloïdale permet de décrire la capacité des nanoparticules à rester ou non en suspension dans leur environnement.
Une des principales caractéristiques des nanoparticules est leur très grande surface spécifique comparée au matériau massif. Ceci a pour conséquence d’augmenter l’interface, ce qui nécessite de l’énergie. En effet, une interface correspond à une discontinuité entre deux milieux, et donc à des liaisons chimiques non compensées. Un nanomatériau de grande surface spécifique est donc souvent moins stable que le même matériau l’échelle macroscopique, ce qui peut entrainer la coalescence des nanoparticules pour minimiser cette énergie de surface. Cette perte de compensation de liaisons chimiques se retrouve également au niveau du milieu dispersant. Lorsque des nanoparticules sont dispersées dans un solvant, les molécules de celui-ci entrent en interaction avec la surface des nanoparticules, or, si cette interaction perturbe la structure du solvant, alors l’énergie libre de Gibbs augmente. En effet, le gain d’entropie engendré par la dispersion des nanoparticules au sein du milieu ne suffit pas à compenser la perte d’entropie subie par les molécules de solvant. Ainsi le système est moins stable à l’état dispersé qu’agrégé.7 Les nanoparticules entrent en permanence en collision à cause du mouvement brownien, ce qui aboutit dans ces conditions à la déstabilisation de la dispersion. Il existe également des forces attractives entres les nanoparticules, contribuant à la formation d’agrégats. Ce sont les forces de van der Waals. D'autres forces attractives peuvent également entrer en jeu, comme par exemple les forces dipolaire magnétiques, ou électrostatiques dans le cas d'un système hétérogène. Une dispersion colloïdale stable n’est donc possible uniquement que s’il existe des forces de répulsions entre les nanoparticules afin de contrebalancer les contributions présentées précédemment.
Le choix ici a été de se focaliser sur le comportement des dispersions de nanoparticules placées dans l’environnement biologique. Toutes les notions de stabilité en milieu simple, certes essentielles, mais très basiques sont présentées en annexe no 1.
A.2 Comportement des nanoparticules dans un milieu biologique
Lorsque les nanoparticules entrent dans un milieu physiologique, celles-ci se trouvent dans un environnement très concentré en molécules variées. Par exemple, le plasma sanguin est composé de plus de 3 000 protéines différentes8 représentant une concentration globale de 60 g/l.9 La surface de la nanoparticule est alors rapidement saturée par l’adsorption non spécifique de biomolécules provoquée par différents effets (interactions de vdW, interactions électrostatiques, effet hydrophobe, par exemple).10–12 Cette couche se formant à la surface des nanoparticules est nommée « couronne biomoléculaire ». Elle est composée principalement de protéines, de lipides et de glucides.10 C’est finalement cette couronne qui forme l’interface entre la nanoparticule et l’environnement biologique.
A.2.1
Mécanisme de formation de la couronne biomoléculaire
Pour que l’adsorption d’une biomolécule à la surface d’une nanoparticule soit spontanée, celle-ci doit être thermodynamiquement favorable. Cela veut dire que :
∆ ∆ ∆ 0
où ΔGads, ΔHads, et ΔSads sont respectivement les variations de l’énergie libre de Gibbs,
de l’enthalpie et de l’entropie et T la température.13
La perte en énergie du système peut provenir de différents paramètres tels que la formation de nouvelles liaisons non covalentes, de la libération des molécules d’eau interfaciales ou encore d’un changement de conformation des biomolécules.
La stabilité du complexe biomolécule-nanoparticule est déterminée par ΔGads. Dans le
cas où cette valeur est élevée alors la probabilité de rupture du complexe est faible, et inversement. Les surfaces chargées promouvant la formation de liaisons électrostatiques formeront des complexes plus stables avec les biomolécules que les particules neutres. Il en est de même pour les nanoparticules hydrophobes car l’adsorption de biomolécules à leur surface libère les molécules d’eau à l’interface. Des études avec des nanoparticules présentant des surfaces différentes ont montré que la composition de la couronne est fortement influencée par les propriétés de surface des nanoparticules.14–17
Il est possible qu’une protéine s’adsorbant à la surface de la nanoparticule subisse des changements conformationnels si ces changements conformationnels permettent l’exposition d’une séquence d’acide aminé enfouie hydrophobe ou chargée pour former de nouvelles liaisons.18 Etant donné que les charges d’une protéine sont généralement en surface, c’est avec les surfaces hydrophobes que cet effet est le plus important.19
Les propriétés physicochimiques de la surface ne sont pas les seules à présenter un impact sur la composition de la couronne, le rayon de courbure est également un paramètre à prendre en compte. Par exemple, Tenzer et al. ont observé des différences de composition de la couronne entre des nanoparticules de silice de 8 nm, 20 nm et 125 nm.20 En effet, le rayon de courbure impacte la surface de contact entre la nanoparticule et la biomolécule, ce qui induit des différences d'affinité. Le rayon de courbure impacte également le niveau de modifications conformationnelles des protéines adsorbées.13
Les biomolécules constituant la couronne peuvent être classées en deux groupes différents. D’un point de vue général, les protéines s’adsorbent soit fortement, soit faiblement à la surface de la nanoparticule.16,21 Le premier groupe constitue ce qui est nommé « couronne dure » alors que le second groupe est décrit par le terme de « couronne molle ». 11,13 La couronne dure serait composée de biomolécules directement et fortement liées à la surface, alors que la couronne molle serait composée de biomolécules, soit faiblement liée à la surface de la nanoparticule soit à la couronne dure via des interactions faibles (Figure I.2).
Figure I.2 : Représentation schématique des couronnes dure et molle.
A.2.2
Dynamique de la couronne biomoléculaire
Bien que l’environnement soit très riche en biomolécules, toutes ne participent pas à la formation de la couronne. Lorsque les particules sont mises en contact avec le plasma sanguin, seulement quelques dizaines de protéines sont détectées dans la couronne. Par exemple, Tenzer et al ont détecté 125 protéines à la surface de nanoparticules de silice après un contact avec le plasma.20 De plus, ils observent que l’abondance des protéines présentes dans la couronne n’est pas directement dictée par l’abondance dans le plasma. En effet, comme il a été abordé auparavant, la liaison de certaines biomolécules peut être favorisée par rapport à d’autres. La couronne biomoléculaire doit également être vue comme une interface dynamique et non figée dans l’état le plus rapidement atteint, c’est-à-dire une couronne composée par les biomolécules les plus abondantes.
L’adsorption de biomolécules et leur désorption se produisent en permanence. Les vitesses d’association et dissociation sont définies par les constantes kon et koff respectivement
et la constante d'équilibre de dissociation KD est définie par :
A l’heure actuelle, cette valeur, déterminée pour quelques couples nanoparticules-protéines, varie entre 10-4 M et 10-9 M.22–25
Bien qu’une biomolécule soit en grande concentration et s’adsorbe rapidement avec un kon élevé, si celle-ci se désorbe également rapidement (koff élevé), alors, elle sera remplacée au
cours du temps par une biomolécule possédant un KD plus faible (Figure I.3).
Figure I.3 : Représentation schématique de la dynamique de la couronne biomoléculaire. Les biomolécules sont donc en compétition permanente. Les différences entre leur koff
font que les biomolécules se désorbant le plus rapidement sont remplacées par les biomolécules présentant des cinétiques de désorption plus lentes. La concentration de chaque biomolécule joue également un rôle. Par exemple, lorsque des nanoparticules sont injectées dans le sang, rapidement une couronne composée par l’albumine ou le fibrinogène, deux biomolécules très présentes dans le plasma, sera constituée. Ces protéines seront par la suite échangées par des protéines possédant une plus grande affinité, comme certaines apolipoprotéines dans le cas de nanoparticules hydrophobes.16 A l’opposée, quand les nanoparticules se retrouvent dans un environnement pauvre en biomolécules, alors même celles présentant une faible affinité resteront adsorbées sur la particule. Ces cinétiques sont fortement dépendantes des types de surfaces mis en jeu et ne peuvent pas être généralisées car dans le cas où les biomolécules concentrées présentent également une affinité élevée, le second processus n'a pas lieu d'être.
La première adsorption se produit très rapidement et permet dans certains cas de stabiliser les nanoparticules d'un point de vue colloïdal. Les fluides biologiques présentent une force ionique équivalente à 150 mM de NaCl, or comme il a été présenté auparavant, dans
le cas de nanoparticules chargées, l'augmentation de la force ionique comprime la couche de contre-ions, ce qui diminue les forces de répulsion et provoque l'agrégation des nanoparticules. Lorsque la couronne se forme suffisamment rapidement, celle-ci apporte une stabilité stérique aux nanoparticules. Ce phénomène a été observé par exemple pour des particules d'or citratées ainsi que des nanoparticules de silice et d’argent après ajout dans du plasma.26–28 A l'opposée, cette couronne peut être à l'origine de formation d'agrégats si celle-ci forme un pont entre plusieurs particules.
Le phénomène d'échange et d'atteinte de l'équilibre sont quant à eux plus long, pouvant courir sur plusieurs heures.29 Les durées sont telles que dans le cas où la nanoparticule est dans un milieu biologique dynamique, comme un organisme, alors la couronne n’atteindra jamais l’équilibre. En effet, le temps que l’équilibre soit atteint, la particule aura changé de compartiment. La couronne sera alors influencée par la nouvelle composition du compartiment. De plus, lorsque les nanoparticules changent de compartiment biologique, ou traversent des barrières, comme les cinétiques de désorption des biomolécules de la couronne dure sont lentes, alors celles-ci ne seront que partiellement remplacées. Cette couronne représente alors l’historique de la nanoparticule. Par exemple, des nanoparticules inhalées atteignent le sang en emmenant également des protéines pulmonaires, ce qui peut engendrer une réponse physiologique totalement différente de celle obtenue avec des nanoparticules directement injectées en intraveineuse.30
A.2.3
Interactions entre le complexe nanoparticule-couronne
biomoléculaire et le milieu biologique.
Il est communément admis, étant donné les différences de cinétiques entre la couronne dure et la couronne molle, que c’est la couronne dure qui définit l’identité biologique des nanoparticules.10,13
Si la couronne biomoléculaire est reconnue par des récepteurs biologiques, que cela soit par des interactions spécifiques ou non, celle-ci sera à l’origine du déclanchement d’un processus physiologique.
Pour les applications biomédicales in vivo, la problématique de l’élimination des nanoparticules par le système immunitaire doit être prise en compte. Lorsqu’une nanoparticule est injectée chez un animal, celle-ci est considérée comme un objet étranger à l’organisme. Les opsonines sont des biomolécules plasmatiques ayant pour fonction d'induire la phagocytose des antigènes auxquels elles se fixent par les macrophages. L'un des membres
les plus présents est la protéine du complément C3. Cette protéine peut dans certains cas correspondre à plus de 30 % en masse de la totalité des protéines adsorbées sur les nanoparticules, ce qui déclenche la phagocytose des nanoparticules par les macrophages.31 Les immunoglobulines G (IgG) font également partie des opsonines et sont présentes en grande quantité dans le plasma. Lorsque celles-ci sont liées à l'antigène, ils activent également la phagocytose de l'antigène. L'orientation de l'anticorps adsorbé est importante car pour qu'il puisse promouvoir la phagocytose, sa région Fc doit rester accessible. La dénaturation de l’albumine provoquée par son adsorption à la surface de nanoparticules de silice fait apparaitre un épitope normalement absent. Cet épitope reconnu par les phagocytes mononucléaires favorise l’élimination des nanoparticules.32 D’un point de vue général, les opsonines adsorbées à la surface des nanoparticules sont reconnues par les cellules du système réticulo-endothélial (SRE) et les nanoparticules sont éliminées de la circulation sanguine. Le SRE est composé de macrophages, libres ou fixés, répartie dans de nombreux tissus, dont le foie qui contient les cellules de Kupffer, représentant 90 % des cellules macrophagiques de l’organisme.33 Sans contrôle des interactions entre la surface de la nanoparticule et le milieu biologique, les nanoparticules sont rapidement captées par le foie.
La couronne biomoléculaire n’a pas seulement un rôle dans l’opsonisation des nanoparticules. Par exemple, Kreuter et al. suggèrent que l’adsorption d’apolipoprotéines à la surface de nanoparticules facilite le passage de la barrière hémato-encéphalique par imitation des lipoprotéines.34 La couronne biomoléculaire est également capable d’interagir avec les processus de signalisation. Deng et al. ont montré que l'adsorption du fibrinogène à la surface de nanoparticules d'or modifiées par des macromolécules de poly(acide acrylique) provoque une modification conformationnelle de la protéine. Cette dénaturation fait apparaitre un nouvel épitope reconnu par l'intégrine Mac-1, ce qui a pour conséquence d'amplifier la voie de signalisation NF-κB, et donc de déclencher le processus d'inflammation.35
Un autre phénomène plus inquiétant lié à la dénaturation des protéines s'adsorbant a également été observé. Certaines nanoparticules augmentent le taux de nucléation de la fibrillation de protéines.36,37 La fibrillation de protéines est impliquée dans des maladies telles que la maladie d'Alzheimer, la maladie de Creutzfeld-Jacob, ou autres maladies neurodégénératives à plaques amyloïdes.
Les cellules peuvent interagir avec les nanoparticules via la reconnaissance de biomolécules présentes dans la couronne, où directement par reconnaissance de la surface de la nanoparticule. Dans le second cas, les mécanismes d’internalisation peuvent être variés,
phénomène de reconnaissance est plus probable dans le cas d’études in vitro sur des cultures cellulaires sans plasma, car in vivo la surface de la nanoparticule n’est généralement pas accessible. La capture est alors dite « sérum-indépendante ». Cependant ces mécanismes sont également observés en présence de plasma lorsque la surface n’adsorbe que très faiblement les biomolécules.31 Les nanoparticules peuvent interagir directement avec la membrane plasmique. Par exemple, il a été montré que les nanoparticules de silice au contact d’une membrane lipidique provoque sa courbure par formation d’interactions favorables débouchant sur l’internalisation de la nanoparticule (Figure I.4).38
Figure I.4 : Reconstruction en tomographie électronique et micrographie cryo‐électronique d'une nanoparticule de silice internalisée dans un liposome. Barre d’échelle : 50 nm. Adapté de 38
La surface de la nanoparticule ainsi que la couronne biomoléculaire adsorbée ne sont pas les seuls paramètres influençant le devenir des nanoparticules dans un milieu biologique. Les biomolécules volontairement greffées à la surface pour apporter la fonction biologique à la nanoparticule peuvent également avoir un impact. Beduneau et al. ont montré que le greffage d’IgG à la surface de nanoparticules d’oxydes de fer permettait d’augmenter leur capture par les macrophages après reconnaissance de la région Fc des IgG.39 Dans cette étude, les macrophages sont les cellules ciblées, donc l’effet est bénéfique, cependant les IgG sont également intéressantes pour leur capacité à reconnaitre spécifiquement un antigène, et sont utilisées en tant que fonction de ciblage. Dans ce cas, la reconnaissance des nanoparticules par les macrophages est à éviter.
A.2.4
Conclusion
Cette partie permet d’apprécier l’étendue de la complexité des interactions entre les nanoparticules et l’environnement biologique. La grande complexité du système d’étude rend celles-ci délicates et difficilement comparables. De plus, le manque de connaissance dans ce domaine rend difficile la prédiction du devenir ainsi que de l’impact des nanoparticules dans le milieu biologique. Les nanoparticules peuvent simplement ne pas remplir leur rôle car elles sont mises hors service via de nombreux mécanismes possibles ou induire une modification
du milieu biologique en provoquant la dénaturation de protéines par exemple, ou en initiant des processus débouchant sur des pathologies (inflammations, plaques amyloïdes…).
A.3 Gestion des interactions non spécifiques
Apporter une fonction biologique contrôlée à une nanoparticule consiste à la fois à promouvoir les interactions désirées et limiter les interactions non souhaitée avec l’environnement (passivation des surfaces d’un point de vue biologique).
A notre connaissance, il existe deux grandes stratégies afin de limiter l’adsorption non désirée de biomolécules aux surfaces. La première stratégie consiste à former une interface basée sur des biomolécules spécifiques. La formation d’une couronne fixe composée de biomolécules choisies permet de limiter l’adsorption d’autres biomolécules par gêne stérique. Par exemple, Peng et al. montrent qu’en formant ex vivo une couronne d’albumine à la surface de nanoparticules, le temps de circulation dans le système sanguin est augmenté par rapport à des liposomes nus.40 Les auteurs expliquent cette observation par une diminution de la liaison des opsonines. Dans la même idée, Nooney et al. augmentent la stabilité colloïdale de nanoparticules de silice en adsorbant de la BSA à leur surface.41 Cette stratégie nécessite une purification de l’objet biologique désiré. Or la purification de biomolécules peut provoquer leur dénaturation partielle, ce qui nous le rappelons peut être à l’origine d’interactions non spécifiques.32,35 Afin de s’affranchir de cette étape de purification, une solution consiste à développer des surfaces de nanoparticules présentant une grande affinité pour une protéine cible présente dans le milieu biologique d’étude. La surface de la nanoparticule est alors spontanément recouverte par une couronne composée de la biomolécule désirée.42 L’adsorption spontanée de la protéine d’intérêt sur la nanoparticule nécessite un contrôle élevé de la surface ainsi que de l’origine des interactions mises en jeu, afin d’éviter les adsorptions non spécifiques.
La seconde stratégie, bien plus commune, pour limiter les interactions non spécifiques consiste à former une barrière énergétique artificielle grâce à la présence de macromolécules polymère fortement hydrophiles.
A.3.1
Les polymères anti-adsorption
La stratégie la plus répandue pour empêcher les interactions non spécifiques consiste à greffer des macromolécules polymères hydrophiles électriquement neutres sur la surface des nanoparticules.13,43,44 L’origine principale de l’effet antiadhésif réside dans la capacité de la
macromolécule à générer des répulsions stériques vis-à-vis des biomolécules rendant leur rapprochement de la surface énergétiquement défavorable (voir également l’annexe no 1 à propos de la répulsion stérique).45,46
Les macromolécules polymères peuvent être soit simplement adsorbées47 soit greffées de façon covalente à la surface,48 mais dans les deux cas celle-ci doivent former des boucles ou des queues afin d’apporter la répulsion stérique (Figure I.5).
Figure I.5 : Représentation schématique de macromolécules adsorbé ou greffées de façon covalente à une surface apportant de la répulsion stérique.
Pour les applications en biologie, il est préférable que les macromolécules polymères soient greffées de façon covalente car les macromolécules adsorbées risquent de se désorber en milieu physiologique, 13 par compétition avec des biomolécules par exemple. L’obtention de surfaces recouvertes de polymères greffés covalemment peut être obtenue via deux voies :44,48
- « Grafting to » : les chaines de polymères pré-synthétisées sont greffées à la surface par des points d’ancrage.
- « Grafting from » : la polymérisation est initiée à partir des fonctions chimiques portées par la surface.
Le poly(oxyde d’éthylène) (POE) également nommé poly(éthylène glycol) (PEG) (formule en Figure I.6) est considéré comme le « gold standard » des polymères destinés à prévenir l’adsorption des biomolécules.44 La modification de la surface d’un objet par des macromolécules de PEG est désignée par le terme « PEGylation ». Il a été reconnu dans de nombreuses études que la PEGylation de nanoparticules permet de diminuer l’adsorption de protéines et d’augmenter leur temps de circulation.49–52 Le premier système de délivrance de principes actifs, basé sur des liposomes PEGylés encapsulant la doxorubicine, date de 1995.53 L’utilisation du PEG s’est depuis largement répandue.
O H
O OH
n
Figure I.6 : formule du poly(oxyde d'éthylène).
Le PEG présente plusieurs avantages expliquant son grand intérêt. Il est très soluble dans l’eau, contrairement au poly(méthylène glycol) et au poly(propylène glycol). Cela s’explique par le fait que dans sa conformation gauche, il offre deux accepteurs de liaisons hydrogène avec une seule molécule d’eau (Figure I.7). Cette conformation est à l’origine de sa propension à former des boucles.54 Le PEG est également soluble dans de nombreux solvants organiques, ce qui facilite la modification chimique au niveau de ses fonctions alcool.
Figure I.7 : Structure 3D du PEG en conformation gauche, et liaisons hydrogènes avec une molécule d’eau. Structure réalisée avec le logiciel libre Avogadro 1.1.0,55 et le champ de force
MMFF94s. Blanc : hydrogène ; Gris : carbone ; Rouge : oxygène.
Bien que la PEGylation réduise l’adsorption des biomolécules à la surface des nanoparticules, elle ne permet pas de la supprimer en totalité, même avec des densités de greffage élevées.31 Ceci est probablement dû à l’adsorption non spécifique des biomolécules directement sur les macromolécules de PEG.56 De plus, les nanomatériaux PEGylés pourraient induire une réponse immunitaire.57 Il a notamment communément été observé une diminution du temps de circulation de nanoparticules PEGylées à la seconde injection.58 Ce phénomène est accompagné de la présence d’IgM anti-PEG dans le sérum. Cependant cette réponse immunitaire semble très dépendante de la composition de l’objet PEGylé.59
Afin de pallier les lacunes du PEG, d’autres polymères ont récemment été étudiés pour leur capacité à prévenir l’adsorption de biomolécules.44 Parmi les potentielles alternatives à l’utilisation du PEG il existe notamment les poly(2-oxazoline), les peptides et peptoïdes (polymères synthétique mimétique des peptides), ainsi que les polymères zwitterioniques. Ces derniers sont électriquement neutres, mais l’unité monomère possède à la fois une fonction chargée positivement et une fonction chargée négativement. Ces polymères sont fortement hydratés, ce qui peut être à l’origine de leur efficacité dans la répulsion des biomolécules.
Parmi cette famille de polymère, le poly(méthacrylate de carboxybetaïne) (PCB) est très prometteur (formule chimique en Figure I.8).
O O N+ O -C H3 CH3 O n Figure I.8 : Formule du poly(méthacrylate de carboxybétaïne).
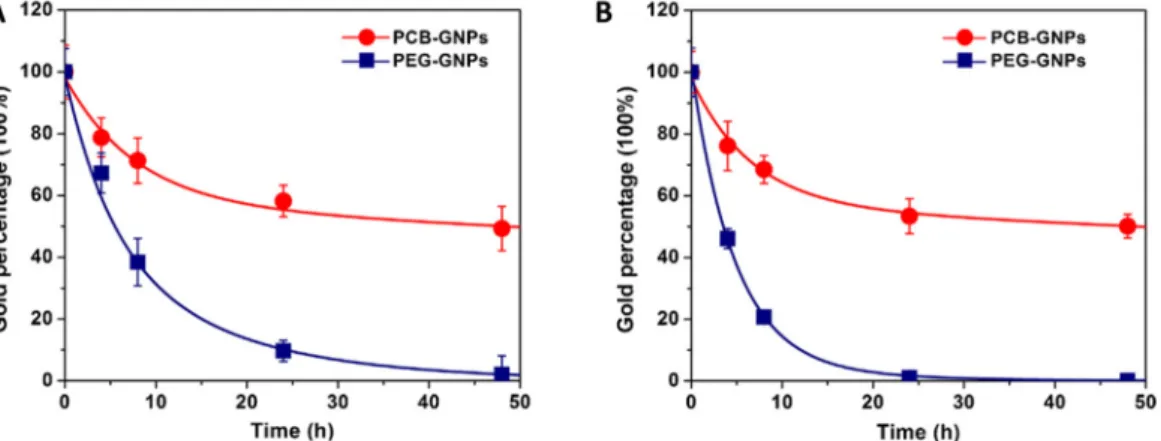
En 2008, Ladd et al. montrent sur un substrat d’or que le PCB présente une capacité à prévenir l’adsorption de protéines supérieure au PEG, alors même que l’épaisseur de la couche polymère est plus faible.60 Récemment, l’étude réalisée avec des nanoparticules d’or (GNP) modifiées par des macromolécules de PEG ou de PCB par Yang et ces collaborateurs montre que les GNP-PCB présentent un temps de demi-vie de circulation (t1/2) de 56 h contre
un t1/2 d’environ 9 h pour les GNP-PEG (Figure I.9).61 De plus ce temps de demi-vie est
identique à la seconde injection pour les GNP-PCB, alors qu’il chute à environ 5 h avec les particules PEGylées comme il a déjà été observé.58
Figure I.9 : Suivi de la quantité de nanoparticules dans le sang après la première (A) et la seconde injection (B). Adapté de la réf.61
Au vu des résultats, l’utilisation des alternatives aux dérivés du poly(oxyde d’éthylène) devrait se développer dans les années à venir. Le PEG reste cependant actuellement le plus employé grâce à sa facilité de production, mais également du fait des savoir-faire développés au cours des décennies précédentes. De plus, ce polymère est intégré dans des nanomatériaux approuvés par la FDA.52 C’est pourquoi nous allons plus particulièrement nous intéresser aux surfaces PEGylées.
A.3.2
Etude conformationnelle des chaines linéaires de PEG à la
surface d’une nanoparticule
Pour que la couche de PEG présente la capacité à prévenir l’adsorption des biomolécules, il a été montré, dans un premier temps d’un point de vue théorique, que la densité de greffage ainsi que la longueur du polymère étaient deux paramètres importants.45,46,62 Quatre modes d’adsorption différents sont proposés (Figure I.10):
1) La biomolécule diffuse au sein de la couche polymère et s’adsorbe directement à la surface du matériau. Ce cas peut se produire quand l’espace entre les polymères est supérieur aux dimensions de la biomolécule.
2) La biomolécule s’adsorbe à la surface de la couche de polymère du fait de l’établissement d’interactions de vdW ou électrostatiques avec la surface.
3) La biomolécule comprime la couche de polymère. Ce cas peut se produire si la compression des macromolécules ne contrebalance pas les interactions à longues portées (vdW, électrostatiques).
4) La biomolécule s’adsorbe sur le polymère par des interactions favorables entre les deux.
Figure I.10 : Modes d'adsorption de biomolécules à une surface PEGylée : 1) Adsorption à la surface par diffusion, 2) Adsorption par interactions à longue portées, 3) Adsorption par compression, 4) adsorption par interactions entre la biomolécule et le PEG.
Une densité de macromolécule polymère greffée élevée permet de diminuer l’espace disponible entre les macromolécules, limitant de ce fait la possibilité de diffusion des biomolécules au sein de la couche de polymère. Cette densité permet également d’impacter la compressibilité de la couche polymère. En effet, la compression des chaines de PEG provoque une élévation de la concentration en motif monomère et donc une élévation de la pression osmotique, phénomène qui est d’autant plus important pour une couche dense en macromolécules. L’épaisseur de la couche permet, quant à elle, de maintenir à distance les
protéines de la surface et de diminuer ainsi la contribution des interactions de vdW et électrostatiques.
La conformation des macromolécules polymères greffées à la surface d’une nanoparticule est fonction de la densité de greffage à la surface et de la taille des macromolécules greffées, caractérisée par le rayon de Flory RF.
Avec :
a : longueur d’une unité monomère, a = 0,358 nm pour le PEG. 63
N : degré de polymérisation
La distance moyenne D entre deux points d’ancrage peut être exprimée par la relation suivante : 64,65
2 Équation I.1
Avec : σ la densité de greffage (molécules/nm).
En fonction du rapport entre D et RF il est possible de distinguer différents régimes
(Figure I.11) :66
1) Pour les faibles densités de surface où ⁄ 2, les chaines sont suffisamment espacées pour ne pas rentrer en interaction. Les macromolécules adoptent alors une conformation en pelote. C’est le régime en « champignon ». 2) Pour les fortes densités de surface où ⁄ 1, la distance moyenne entre
deux points d’ancrage étant inférieur au rayon de Flory, les macromolécules polymères développent de fortes interactions entre elles. Un équilibre se crée entre la pression osmotique, tendant à étirer les polymères, et l’élasticité des chaines qui s’oppose à cet étirement. Les chaines sont sous une conformation étirée, c’est le régime en « brosse ».
3) Si 1 ⁄ 2, c’est un régime intermédiaire nommé « champignons en interaction »
En régime « champignon », l’épaisseur de la couche de polymère L correspond au rayon de Flory du polymère. Cette conformation ne permet pas de limiter l'adsorption des biomolécules à la surface car celle-ci reste accessible. En régime « brosse », la répulsion mutuelle des polymères les force à s’étirer perpendiculairement à la surface. Sur une surface plane, Lp est défini par l’expression :67
∗
Avec :
σ* : densité de greffage sans dimension ; ∗ . où s est la surface projetée d’une
unité monomère. / avec v le volume occupé par une unité monomère. s = 0,236 nm2 pour le PEG
Cependant le rayon de courbure des nanoparticules étant élevé, il n’est pas possible de le négliger. En effet sur une surface courbe, la densité de monomères décroit au fur et à mesure que l’on s’éloigne de la surface. La force des interactions diminue donc avec la distance par rapport à la surface. Les segments de polymères loin de la surface ne sont donc pas autant étirés que ceux proches de celle-ci (Figure I.12).
Figure I.12 : Représentation schématique du modèle de Daoud et Cotton. Adapté de la réf.68
Daoud et Cotton ont développé un modèle permettant de prédire le rayon d’un polymère en étoile.68 Cette théorie a été étendue pour des polymères greffés sur des particules par Vagberg et al.. L’épaisseur Ls de la couronne de polymère en régime brosse sur une
particule de rayon R est approchée par la relation :69 8
3 4
Équation I.2 où Nk est le nombre de segments de Kuhn dans la macromolécule ; ν, l’exposant de
Flory (ν = 3/5 en condition de bon solvant) ; f, le nombre de chaines greffées sur la nanoparticule, et Lk, la longueur de Kuhn.
4
où c∞ est le ratio caractéristique (4,1 pour le PEG d’après le Polymer Handbook) ; l0,
la longueur moyenne d’une liaison (0,147 nm pour le PEG), et n, le nombre de liaisons dans la macromolécule (3 × degré de polymérisation pour le PEG)
Des modèles plus fins ont été développés afin de décrire plus précisément la conformation des macromolécules de polymère sur des surfaces sphériques. Ces modèles prennent en compte deux sous régimes différents au sein du régime en brosse (B) : un sous régime semi-dilué (BSD), et un sous régime concentré (BC).70,71 La transition entre le régime BC et le régime BSD est définie par le rayon critique rc. Au sein du régime BC, les
interactions entre les chaines polymères sont très fortes, ce qui fait que celles-ci sont fortement étirées contrairement au régime BSD (Figure I.13). Ces modèles intègrent donc différents régimes en même temps.
Figure I.13 : Représentation schématique de la conformation des macromolécules de polymère sur une nanoparticule en fonction de la densité de greffage.
Expérimentalement, pour des masses de macromolécules de PEG variant de 750 g.mol-1 à plus de 20 kg.mol-1, des densités de greffages comprises entre 0,1 macromolécules/nm2 ou moins et 1 macromolécules/nm2 ont été obtenues.31,65,72–75 Ces valeurs peuvent largement fluctuer en fonction de la taille des macromolécules employées ainsi que de la stratégie de greffage mise en place. Ces études confirment la théorie en montrant que la conformation brosse des macromolécules linéaires de PEG présente les meilleures propriétés anti-adsorption. A titre d’exemple, Walkey et al. montrent que l'adsorption de protéines est minimale sur des nanoparticules d'or de 15 nm à partir de 0,5 macromolécules/nm2 pour des PEG de 5 000 g.mol-1.31 Dans une autre étude, Gref et al. ont observé que l’adsorption de protéines sur une surface PEGylée avec des macromolécules de 5 000 g/mol était réduite de plus de 50 % par rapport à celle mesurée avec des chaines linéaires de 2 000 g/mol.73 Puis, au-delà de 5 000 g/mol, l’effet de l’augmentation de la masse
moléculaire des macromolécules sur la diminution de l’adsorption des protéines est fortement réduit.
Pour résumer, l'épaisseur de la couronne polymère permet de diminuer la contribution des interactions à longues portées. La densité de greffage conditionnera la possibilité de diffusion des biomolécules au sein de la couronne ainsi que l'épaisseur de la brosse et la compressibilité de celle-ci. Enfin, pour une densité de greffage donnée, la longueur de la macromolécule linéaire jouera sur l'épaisseur de la brosse. Une faible densité de greffage peut donc être compensée par l’utilisation d’une longue macromolécule.
A.4 Conclusion
A l’heure actuelle, la prédiction exacte du comportement des nanoparticules ainsi que leurs conséquences dans un milieu biologique (échantillon ou organisme) est impossible du fait de la complexité du système d’étude. D’un point de vue général, les nanoparticules sont rapidement recouvertes par une couronne biomoléculaire qui lui apporte son identité biologique. Le manque de contrôle des interactions à l’origine de la formation de cette couronne biomoléculaire peut engendrer la dégradation de l’échantillon biologique alors que pour les applications biomédicales in vivo, cette couronne est principalement responsable de l’élimination des nano-objets par le système réticulo-endothéliale.
La passivation de la surface des nanoparticules consiste, idéalement, à supprimer toute sorte d’interaction entre la nanoparticule et les biomolécules. Cette passivation peut être obtenue en modifiant la surface avec des macromolécules polymère hydrophiles. Dans le cas de l’utilisation de chaines linéaires de PEG, pour que la réduction de l’adsorption des biomolécules à la surface soit optimale, les macromolécules doivent adopter une conformation en brosse, et la couronne polymère formée doit être suffisamment épaisse pour écranter les interactions à longue portée.
Une fois la surface passivée, il est possible d’apporter les fonctions souhaitées à la nanoparticule.
B. Biofonctionnalisation de nanoparticules par conjugaison de
biomolécules.
la biochimie.76 Le contrôle de la fonctionnalisation d’une nanoparticule par bioconjugaison fait appel à différentes notions :
- Il doit être possible de former un lien entre la surface de la nanoparticule et la biomolécule. Cette notion évidente sous-entend de maitriser la chimie de surface de la nanoparticule.
- La mobilité de la biomolécule liée à la nanoparticule ainsi que son orientation doivent être prise en compte. Par exemple la mauvaise orientation d'une protéine peut bloquer l'accès à son site de fixation (site actif, paratope, etc…). - La fonctionnalité de la biomolécule doit être conservée après conjugaison. La
fonction d’une macromolécule biologique (peptides, protéines, acides nucléiques…) est fortement liée à sa structure. Cette structure est maintenue par la présence de liaisons faibles (interactions de vdW, interactions électrostatiques, liaisons hydrogènes) au sein de la molécule. Elle peut donc facilement être perturbée (dénaturation de la biomolécule) dans le cas où la surface de la nanoparticule n’est pas correctement préparée pour son accueil. La stratégie de bioconjugaison est dictée par de nombreux facteurs tels que la chimie de surface de la nanoparticule, le type de biomolécule, ou encore, l’application finale. Une vision très détaillée du domaine est rapportée par Sapsford et al.,76 ainsi que dans l’ouvrage Bioconjugate Technique.77 Nous allons, ici, nous attacher à donner une vision d’ensemble de cette vaste discipline en présentant dans un premier temps différentes modifications chimiques de surface à l’origine du lien entre la nanoparticule et la biomolécules. Nous nous intéresserons ensuite aux différentes voies de biocongugaison à proprement parler.
B.1 Modification chimique de la surface des nanoparticules
La fonctionnalisation d’une nanoparticule peut être réalisée à l’aide deux grandes classes de molécules : les ligands qui sont des molécules possédant une ou des fonctions chimiques présentant une affinité pour la surface (ancre), et les agents de couplage silaniques organofonctionnels.
a. Le cas des ligands
Les nanoparticules métalliques, telles que les nanoparticules d’or, sont très étudiées et employées pour leurs propriétés physiques remarquables, notamment en optique. La surface