HAL Id: tel-00799083
https://tel.archives-ouvertes.fr/tel-00799083
Submitted on 11 Mar 2013
HAL is a multi-disciplinary open access archive for the deposit and dissemination of sci-entific research documents, whether they are pub-lished or not. The documents may come from teaching and research institutions in France or abroad, or from public or private research centers.
L’archive ouverte pluridisciplinaire HAL, est destinée au dépôt et à la diffusion de documents scientifiques de niveau recherche, publiés ou non, émanant des établissements d’enseignement et de recherche français ou étrangers, des laboratoires publics ou privés.
Etude biochimique d’un cytochrome P450 de cerveau
humain : le CYP2U1
Lionel Ducassou
To cite this version:
Lionel Ducassou. Etude biochimique d’un cytochrome P450 de cerveau humain : le CYP2U1. Médecine humaine et pathologie. Université René Descartes - Paris V, 2012. Français. �NNT : 2012PA05P642�. �tel-00799083�
ÉCOLE DOCTORALE MÉDICAMENT TOXICOLOGIE CHIMIE ENVIRONNEMENT
Doctorat
Chimie Biologique
Etude biochimique d’un cytochrome
P450 de cerveau humain : le CYP2U1
D
UCASSOULionel
Thèse dirigée par
BOUCHER Jean-Luc Soutenue le 09 novembre 2012
Jury :
CORCOS Laurent DR INSERM rapporteur
CRESTEIL Thierry DR CNRS rapporteur
BOUCHER Jean-Luc DR CNRS directeur de thèse
ANDRE François CEA Saclay
ETCHEBEST Catherine Prof. Université Paris Diderot GARBAY Christiane Prof. Université Paris Descartes
LOMBARD Murielle CR CNRS
INTRODUCTION
1
Cette thèse a été préparée au :
UMR8601
Laboratoire de Chimie et Biochimie Pharmacologiques et Toxicologiques 45 rue des Saints Pères
75006 Cedex
01 42 86 21 69
INTRODUCTION
2
Résumé :
Parmi les 57 cytochromes P450 identifiés lors du séquençage complet du génome humain, on en dénombre environ 15 dont on ne connaît pratiquement rien de leurs rôles physiologiques, de leurs substrats, et de leurs structures, d’où le nom de «P450 orphelins». Le CYP2U1 est l’un des cytochromes P450 les plus fortement exprimé au niveau du cerveau et du cervelet mais c’est aussi l’un des plus conservé parmi les différentes espèces du règne animal.
Ce travail de thèse a tout d’abord consisté à optimiser les conditions d’expression du CYP2U1 sous une forme active. Un premier système d’expression dans la levure Saccharomyces Cerevisiae a permis une production d’un complexe CYP2U1-P450 réductase catalytiquement actif permettant des études de recherche de substrat. Un second système d’expression dans Escherichia Coli devrait permettre d’obtenir de plus grandes quantités d’enzyme soluble destinée à des études structurales.
Dans un second temps, une recherche de substrats a été effectuée à l’aide d’analyse d’incubats par chromatographie liquide couplée à une détection par spectrométrie de masse. A ce jour, un screening dirigé de plus de soixante-dix molécules, substrats de P450s de la famille 2, a permis d’identifier les premiers substrats exogènes du CYP2U1, les analogues de terfénadone et la débrisoquine.
D’autre part, une étude par modélisation moléculaire de la structure du CYP2U1 a été effectuée. Cette étude montre que le CYP2U1 diffère de tous les autres P450s par la présence d’un
insert très spécifique dans son domaine N-terminal. Des modèles par homologie basés sur les
structures cristallographiques des P450s de la famille 2 ont été construits. Ces modèles ont été validés par dynamique moléculaire et ont permis de proposer un mode d’interaction avec la membrane, d’identifier la position des canaux d’accès ainsi que de déterminer la topologie du site actif. Enfin, un docking des premiers substrats exogènes au sein du site actif du CYP2U1 a permis de confirmer la régiosélectivité des hydroxylations catalysées par le CYP2U1.
INTRODUCTION
3
Sommaire
CHAPITRE I. INTRODUCTION ... 7
I.1 Les cytochromes P450 ... 8
I.1.1 Présentation générale... 8
I.1.2 Propriétés catalytiques... 12
I.1.3 Caractéristiques structurales ... 21
I.1.4 Les cytochromes P450 humains ... 35
I.1.5 Les cytochromes P450 du cerveau ... 38
I.2 Les cytochromes P450 orphelins chez l’homme ... 39
I.2.1 Déorphelinisation en cours ... 39
I.2.2 Stratégies de « déorphelinisation »... 42
I.3 Le cytochrome P450 2U1 ... 44
I.3.1 Caractérisation génétique ... 44
I.3.2 Répartition dans les tissus ... 47
I.3.3 Caractérisation biologique... 48
I.3.4 CYP2U1 et cancer ... 48
I.4 Objectifs de la thèse ... 49
CHAPITRE II. LE CYP2U1, UNE STRUCTURE SINGULIERE ... 50
II.1 Alignements multiples de séquences ... 51
II.1.1 Alignement des séquences de P450s de la famille 2 ... 51
II.1.2 Deux inserts spécifiques du CYP2U1 ... 52
II.2 Construction de modèles par homologie ... 53
II.2.1 Choix des domaines à modéliser ... 53
II.2.2 Construction, évaluation et comparaison des différents modèles ... 54
INTRODUCTION
4
II.2.4 Optimisation des deux modèles de CYP2U1 (modèles 1 & 2) ... 61
II.2.5 Description et comparaison des différents modèles ... 65
II.3 Interaction du CYP2U1 avec la membrane ... 67
II.3.1 Prédiction de l’orientation par rapport à une membrane cellulaire ... 67
II.3.2 Dynamique de 100 ns de modèles complets (CYP + membrane) ... 71
II.3.3 Interaction de l’insert avec la membrane ... 78
II.4 Etude de la topologie du site actif ... 81
II.4.1 Calcul Voidoo du volume de la cavité du site actif ... 81
II.4.2 Résidus tapissant la cavité du CYP2U1, description de la cavité ... 86
II.5 Evaluation des canaux d’accès au site actif ... 88
II.6 Conclusion ... 94
CHAPITRE III. PRODUCTION DU CYP2U1 DANS UN SYSTEME HETEROLOGUE ... 96
III.1 Choix des systèmes de production ... 97
III.1.1 Un système eucaryote adapté aux études enzymatiques, la levure Saccharomyces cerevisiae 97 III.2 Caractérisation biochimique du cytochrome P450 2U1 ... 99
III.2.1 Dosage spectroscopique ... 99
III.2.2 Immunodétection du CYP2U1 par Western-Blot ... 100
III.3 Conclusions ... 102
CHAPITRE IV. VERS DE NOUVEAUX SUBSTRATS DU CYP2U1 ... 103
IV.1 Quels substrats pour un CYP orphelin ? ... 104
IV.1.1 Méthodologie ... 104
IV.1.2 Liste des molécules testées ... 104
IV.2 Métabolisme de la débrisoquine par le CYP2U1 ... 107
IV.2.1 Régiosélectivité d’oxydation par le CYP2U1 ... 110
INTRODUCTION
5
IV.2.3 Activités comparées de divers P450 humains ... 118
IV.2.4 Arrimage de la débrisoquine dans le site actif du CYP2U1 ... 121
IV.3 Métabolisme des dérivés de la terfénadone par le CYP2U1. ... 129
IV.3.1 Présentation de la famille de substrats ... 129
IV.3.2 Régioselectivité d’hydroxylation des analogues de terfénadone par le CYP2U1 .. 130
IV.3.3 Cinétique d’hydroxylation des analogues de terfénadone par le CYP2U1 ... 133
IV.3.4 Arrimage de l’ethylterfénadone dans le site actif de l’enzyme ... 135
IV.4 Conclusions ... 144
CHAPITRE V. CONCLUSIONS ET PERSPECTIVES ... 146
CHAPITRE VI. PROTOCOLES EXPERIMENTAUX ... 149
VI.1 Méthodes Bioinformatiques ... 150
VI.1.1 Génération de modèles par homologie et leur évaluation ... 150
VI.1.2 Recuits simulés sous Rosetta ... 151
VI.1.3 Dynamiques moléculaires sous AMBER ... 151
VI.1.4 Dynamiques moléculaires sous CHARMM ... 152
VI.1.5 Analyse des fichiers de dynamique ... 154
VI.1.6 Evaluation des canaux d’accès des substrats ... 155
VI.1.7 Evaluation du volume du site actif ... 157
VI.1.8 Docking de petites molécules dans le site actif des cytochromes P450 ... 158
VI.2 Méthodes Biochimiques et Analytiques ... 160
VI.2.1 Milieux de culture et tampons ... 160
VI.2.2 Expression de la protéine recombinante dans Saccharomyces cerevisae ... 160
VI.2.3 Expression de la protéine recombinante dans Escherichia coli ... 164
VI.2.4 Microsomes commerciaux exprimant les cytochromes P450 humains ... 167
INTRODUCTION
6
VI.2.6 Origine des molécules testées ... 167
VI.2.7 Conditions d’incubation pour les études métaboliques ... 167
VI.2.8 CLHP-SM ... 168
VI.2.9 Détection par fluorimétrie des produits d’hydroxylation de la débrisoquine . Erreur ! Signet non défini. VI.2.10 Analyse des fractions microsomales ... 169
VI.2.11 Spectroscopie RMN ... 171
CHAPITRE VII. ANNEXES ... 172
VII.1 Alignements ... 173
VII.1.1 Alignement des 16 P450s humains de la famille 2 ... 173
VII.1.2 Alignement pour Modeller ... 175
VII.2 Scripts et données bioinformatiques ... 176
VII.2.1 Script Modeller ... 176
VII.2.2 Fichiers de topologie pour l’hème et le résidu CYQ ... 177
VII.3 Un système procaryote adapté aux études structurales, la bactérie Escherichia coli .... 179
VII.3.1 Modifications du gène du CYP2U1... 179
VII.3.2 Un isoforme sensible ... 179
VII.3.3 Caractérisation du CYP2U1 exprimée dans E. coli ... 180
VII.4 Gradients CLHP ... 181
VII.4.1 Analogues de terfénadone ... 181
VII.4.2 Débrisoquine et ses analogues ... 181
VII.4.3 Autres molécules testées ... 182
INTRODUCTION
7
INTRODUCTION
8
I.1
Les cytochromes P450
I.1.1 Présentation générale
I.1.1.1 Genèse du métabolisme des xénobiotiques
La découverte des cytochromes P450 s’est faite dans la continuité des études menées sur la détoxification de composés exogènes, les xénobiotiques. Ces études ont débuté chez le chien dès 1824 puis chez l’homme à partir de 1841 (Wöhler 1824; Ure 1841; Li, Baudry et al. 2004).
La première réaction caractérisée fut la conjugaison de la glycine à l’acide benzoïque. En 1841 et 1842, deux scientifiques ont ingéré de l’acide benzoïque et ont observé l’excrétion en grande quantité dans leurs urines d’un produit inconnu (Ure 1841; Keller 1842; Conti and Bickel 1977). Ce produit est similaire à l’acide benzoïque mais contient aussi de l’azote. L’identification de cette molécule inconnue comme étant l’acide hippurique (voir Figure I.1), formée par conjugaison de l’acide benzoïque avec un équivalent de glycine endogène est établie en 1845 par un scientifique français, Victor Dessaignes (Dessaignes 1845). De ces expériences, bien que rudimentaires, a émergé un paradigme fondateur pour le métabolisme : l’organisme est capable de prendre en charge des molécules exogènes, de leur faire subir des réactions chimiques puis de les excréter via les urines sous une forme chimiquement altérée.
Figure I.1 : détoxification de l’acide benzoïque chez l’homme
Lors des décennies suivantes, de nombreuses nouvelles réactions du métabolisme ont été découvertes et ce, principalement par des scientifiques et laboratoires allemands à la pointe de la chimie dans cette deuxième partie du XIXème siècle. Ainsi Wöhler et Frerichs (1948) ont montré que le benzaldéhyde était métabolisé en acide benzoïque chez le chien (Conti, Harwood et al. 1977), la première méthylation fut décrite en 1887 par His (His 1887) qui, après avoir fait ingéré de l’acétate de pyridine à un chien, a isolé un métabolite N-méthylé dans ses urines. Par la suite, en 1879, il fut
INTRODUCTION
9 démontré que le camphre était éliminé dans les urines sous forme d’un adduit glucuronique (Schmiedeberg 1879).
A la fin du XIXème siècle, pour chacune des principales voies de métabolisme, un ou plusieurs exemples avaient été publiés. Cependant, les mécanismes chimiques mis en jeu pour ces transformations restaient inconnus.
Toutes ces observations ont mené Williams à énoncer les principes fondamentaux de la détoxification des xénobiotiques par les organismes vivants (Williams 1947). Celle-ci s’effectue en deux étapes (voir Figure I. 2) :
- La première phase de fonctionnalisation (ou phase I) du composé exogène permet d’introduire une fonction hydrophile. Cette fonction est ajoutée le plus souvent par oxydation. Dans certains cas, elle peut apparaître suite à une réduction ou une hydrolyse. Le composé exogène est donc transformé en une molécule plus hydrophile qui peut ainsi être prise en charge par les voies d’excrétion (urinaire ou biliaire).
- Si le métabolite de phase I n’est pas assez hydrophile pour être excrété, il va subir la seconde phase de détoxification dite phase de conjugaison (ou phase II) qui consiste à former un adduit avec un composé endogène très hydrophile comme l’ion sulfate, le glutathion ou l’acide glucuronique. Le composé conjugué, beaucoup plus hydrosoluble pourra être excrété.
Figure I. 2 : Les mécanismes de détoxification des xénobiotiques.
Ce n’est qu’à partir des années 1950 que débute les premières études biochimiques et mécanistiques du métabolisme. Elles s’orientent logiquement vers le foie qui, par sa position intermédiaire entre l’intestin et la circulation systémique, doit être impliqué dans le métabolisme de xénobiotiques. Dans cette optique, Mueller et Miller mettent en évidence in vitro l’implication des cellules hépatiques dans la biotransformation du diméthylaminobenzène chez le rat (Mueller and Miller 1949). Plus tard, J. Axelrod et B. Brodie découvrent que des enzymes hépatiques membranaires
INTRODUCTION
10 sont capables d’oxyder des composés exogènes en présence de NADPH et de dioxygène (Axelrod 1955; Brodie, Axelrod et al. 1955).
I.1.1.2 Découverte et identification des cytochromes P450
A la même époque, deux événements majeurs ont initié la recherche sur les cytochromes P450, à savoir l’isolation de la fraction microsomale de tissus animaux (Claude 1943; Palade and Siekevitz 1956) et la détection des premières activités enzymatique de type oxygénase pour lesquelles la source d’oxygène est le dioxygène (Ure 1841; Hayano, Lindberg et al. 1955).
En 1958, Klingenberg et Garfinkel mirent en évidence la présence d’un pigment dans les fractions microsomales hépatiques de rat et de cochon qui, réduit par du NADPH ou du dithionite et en présence de monoxyde de carbone présentait une absorption spécifique à 450 nm (Garfinkel 1958; Klingenberg 1958). Les travaux de Omura et Sato permirent d’identifier ces pigments, en montrant qu’il s’agissait d’hémoprotéines, qui seront appelés par la suite « cytochrome P450 » (Omura and Sato 1962), le P faisant référence à son cofacteur, la protoporphyrine IX et 450 à la longueur d’onde d’absorption du complexe FeII-CO. Toutefois, la nature du ligand axial coordinant l’hème reste un mystère : dans les hémoprotéines connues à cette époque, la protoporphyrine est toujours coordinée à un imidazole d’histidine en position axiale et présente un spectre d’absorption différentiel avec un pic à 420 nm. L’effet bathochrome observé indique donc l’existence d’un nouveau type de liaison au fer. Ce n’est qu’en 1974, que le rôle d’un ligand cystéinate a été établi (Stern and Peisach 1974).
Au cours des années 60, l’implication des cytochromes P450 a été démontrée dans la biotransformation des stéroïdes (Estabrook, Cooper et al. 1963) puis dans l’oxydation de composés exogènes (Cooper, Levin et al. 1965). En 1968, le premier P450 est purifié, il s’agit d’une protéine soluble issue de la bactérie Pseudomonas putida et catalysant l’hydroxylation du camphre en 5-exo-hydroxycamphre (Katagiri, Ganguli et al. 1968).
Enfin, il a fallu attendre les années 70-80 pour que soient élucidées les principales étapes du cycle catalytique des P450s et notamment les mécanismes d’activation de l’oxygène (Groves and Mcclusky 1976).
INTRODUCTION
11
I.1.1.3 Une grande diversité de gènes
L’optimisation des techniques de séquençage des génomes ces quinze dernières années a permis la découverte d’un nombre exponentiel de gènes codant pour des cytochromes P450. Ces données sont regroupées dans une base de données qui rassemble les différentes séquences de P450 (http://drnelson.uthsc.edu/CytochromeP450.html). En août 2009, 11294 différentes gènes codants pour des P450s étaient alors identifiés. Ils sont présents dans tout le règne du vivant : on retrouve ainsi des représentants de cette superfamille chez les animaux, les plantes, les champignons, les bactéries, les archaebactéries, les protistes et récemment, deux gènes de P450s ont même été découverts dans le génome d’un virus (Acanthamoeba polyphaga mimivirus)(Lamb, Lei et al. 2009).
Le nombre de gènes de P450 présents dans une espèce est quant à lui, très variable. Ainsi, l’homme possède 57 gènes tandis que l’on dénombre 246 gènes chez A. thaliana (Ortiz de Montellano 2005) et jusqu’à plus de 300 gènes dans le riz (Ingelman-Sundberg 2005).
Malgré cette grande diversité de gènes, la superfamille des cytochromes P450 est caractérisée par deux signatures dans la séquence protéique qui sont représentées en Figure I. 3.
La première est située du côté proximal de l’hème. Ce motif, FxxGx(R/H)CxG, appelé Cys-Pocket contient la cystéine coordinant l’hème et est particulièrement bien conservée aussi bien au niveau structural qu’au niveau de la séquence primaire car cette structure assure la stabilité du cystéinate liant l’hème par un jeu de liaisons hydrogènes entre résidus voisins. C’est aussi la signature historique des P450s, utilisée dans la base de données PROSITE (Sigrist, Cerutti et al. 2010) sous la forme : [FW]-[SGNH] -x-[GD]-{F}-[RKHPT]-{P}-C-[LIVMFAP]-[GAD].
La seconde séquence consensus, située du côté distal de l’hème, [G/A]Gx[E/D]Tx, est moins bien conservée que la première et joue un rôle au cours de l’activation du dioxygène dans le cycle catalytique de l’enzyme.
INTRODUCTION
12
I.1.1.4 Nomenclature
A l’origine, les P450s étaient désignés par leur principal substrat. Par exemple, le P450 ayant pour substrat le camphre était appelé P450CAM. Cette nomenclature était pertinente à une époque où peu de
P450 étaient caractérisés. Mais très vite, cette technique a atteint ses limites : deux P450s peuvent partager un même substrat ou un P450 peut ne pas avoir de substrat connu ; comment nommer ces nouvelles enzymes ?
De ce fait, une nomenclature alpha numérique basée sur l’identité de séquence a été progressivement définie (Nelson 1998). Ainsi, deux P450s sont de la même famille si leur identité de séquence est supérieure à 40% et de la même sous-famille pour une identité de séquence supérieure à 55%. Par exemple, le P450 dénommé CYP2U1 appartient à la famille 2 et à la sous-famille U et le CYP46A1 appartient à la famille 46 et à la sous-famille A.
I.1.2 Propriétés catalytiques
I.1.2.1 Réactions catalysées par les cytochromes P450
Les cytochromes P450 catalysent des réactions de mono-oxygénation, dont les plus connues sont des hydroxylations et des époxydations (Figure I. 4). Ces réactions nécessitent la présence de dioxygène ainsi que d’un cofacteur apportant les électrons nécessaires (le NADPH ou le NADH).
INTRODUCTION
13
RH +
O
2 + NADPH + H+R
O
H + H2
O
+ NADP
+Figure I. 4 : Principales réactions d’oxydation catalysées par les cytochromes P450. D’après Mansuy et Battioni (Mansuy and Battioni 2000)
Par ailleurs, diverses autres réactions comme des déshydratations, des isomérisations et des réductions ont aussi été observées (Ortiz de Montellano 2005).
Cette réactivité propre aux CYPs s’explique par la présence au sein du site actif d’un hème (protoporphyrine IX de Fer) relié à l’apoprotéine par un groupement cystéinate formant une liaison avec le fer (Figure I. 5)
INTRODUCTION
14
I.1.2.2 La chaine de transfert d’électrons
La transmission des électrons jusqu’à la protoporphyrine se fait via plusieurs protéines de transfert d’électrons telles que la cytochrome P450 réductase (CPR). Il existe dix classes de P450s selon le système de transfert d’électrons qu’ils utilisent (Hannemann, Bichet et al. 2007). Cependant, on distingue essentiellement deux classes de P450s (classe I et classe II) qui correspondent aux modes de transfert d’électrons les plus fréquemment rencontrés.
Les P450s de classe I, correspondent, sauf exceptions, aux enzymes bactériennes et mitochondriales. La chaine de transfert d’électrons est constituée de deux partenaires rédox : une protéine à centre fer-soufre (la ferrédoxine) et la ferrédoxine réductase. Les électrons sont d’abord prélevés par la ferrédoxine réductase via son cofacteur FAD puis transférés sur le centre fer-soufre de la ferrédoxine et enfin sur le P450 pour permettre la catalyse.
Les P450s de classe II, correspondent en partie aux enzymes microsomales et reçoivent leurs électrons d’un seul partenaire rédox, la cytochrome P450 réductase. Cette protéine ancrée comme le P450 à la membrane du réticulum endoplasmique possède deux cofacteurs (FMN et FAD) en charge du transfert d’électrons vers le P450. Un second système, non spécifique aux P450s, peut aussi participer au transfert d’électrons : le cytochrome b5 peut transférer des électrons du NADPH vers certains P450s (Schenkman and Jansson 1999).
Bien que les partenaires rédox des P450s soient généralement exprimés de façon indépendante, il existe, chez certaines bactéries et certains champignons, des systèmes de type P450 pour lesquels il y a eu fusion au cours de l’évolution des gènes de P450 avec celui (ceux) de partenaire(s) rédox. Cette
N N N N HOOC HOOC Fe SCys Hème Apoprotéine
INTRODUCTION
15 fusion d’un domaine oxygénase à un domaine réductase se retrouve aussi chez l’homme dans une famille enzymatique voisine : les NO synthases.
La protéine P450RhF issu de Rhodococcus sp est un exemple de protéine de fusion ; on peut la classer comme un P450 de classe I (Roberts, Grogan et al. 2002). Cette protéine possède un domaine réductase à flavine FMN, fusionné à un domaine à centre fer-soufre et à un domaine P450.
Il existe un autre type de protéine de fusion plus proche d’un système de classe II. Ainsi, le P450BM3 de Bacillus megaterium, qui catalyse l’hydroxylation en ω-2 d’acides gras et dont le domaine
P450 est fusionné en C-terminal à un domaine réductase à deux flavines (Munro, Leys et al. 2002). Les P450s catalysant des réactions d’isomérisation ou de déshydratation ne nécessitent pas l’apport d’électron via un système rédox et/ou ne nécessitent pas l’entrée de dioxygène au site actif. Les substrats transformés lors de ces réactions sont souvent riches en électrons, tels que des alkylhydroperoxydes ou des hydroperoxydes (Mansuy 1998). Ces enzymes sont impliquées dans la biosynthèse de molécules de voies de signalisation telles que les prostaglandines chez les mammifères. Bien qu’aucun système de transfert ne soit nécessaire à l’activité, on définit quand même une classe
III, qui englobe ces systèmes.
Enfin, un P450 qui reçoit directement ses électrons du NADPH a aussi été décrit et constitue à lui seul la classe IV. L’unique représentant actuel de cette classe est le P450NOR qui catalyse la
INTRODUCTION
16
I.1.2.3 Le cycle catalytique
Le cycle catalytique conduisant à l’oxydation d’un substrat se déroule en trois grandes étapes : fixation du substrat, fixation et activation du dioxygène et oxydation du substrat qui quitte alors le site actif du P450.
Au repos, le FerIII est en équilibre entre deux états : une forme haut-spin (S=5/2) pentacoordinée et une forme bas-spin (S=1/2) hexacoordinée (le sixième ligand étant une molécule d’eau). Le cycle catalytique est initié par l’entrée du substrat au sein du site actif (voir Figure I. 7 – (1)). Cette association avec le substrat modifie le potentiel rédox du Fer, ce qui permet le transfert
Figure I. 6: Les partenaires de transport d’électrons des P450s. La classe I comprend les P450 mitochondriaux (A), la
plus grande partie des systèmes solubles et certains systèmes fusionnés de type P450RhF (B). La classe II comprend les P450 microsomaux (C) et les systèmes fusionnés du type P450 BM3 (D). FxR : ferrédoxine réductase, Fx : ferrédoxine, CPR : cytochrome P450 réductase. Les flèches rouges correspondent au transfert d’électrons du cofacteur jusqu’au P450.
INTRODUCTION
17 d’un premier électron issu de la cytochrome P450 réductase (Guengerich and Johnson 1997). Cette réduction est suivie de la fixation de dioxygène donnant naissance au complexe (3). Le transfert d’un nouvel électron (4) depuis la cytochrome P450 réductase permet la formation d’un complexe de type péroxyferrique (Sansen, Yano et al. 2007) qui par protonation (5) conduit à l’hydropéroxyde [FeIII-OOH]. La deuxième protonation (6) mène à une rupture homolytique de la liaison O-O avec le départ d’une molécule d’eau et la formation d’un complexe à haut degré d’oxydation, le fer-oxo [FeV=O] qui a été mis en évidence pour la première fois par Rittle et Green en 2010 (Rittle and Green 2010). Cette entité transfère alors son oxygène au substrat (6). Enfin, le métabolite (produit d’oxydation) est libéré (Bosch, Dinklage et al.) ; l’hème retrouve son état initial.
INTRODUCTION
18
Figure I. 7: Le cycle catalytique des P450s. Les voies abortives, non productives (découplage) sont indiquées en rouge, le cycle
court (en bleu) est obtenu par addition d’espèces riches en oxygène, la voie réductase est indiquée en marron. D’après la thèse de P. Lafite (Lafite 2007)
Une étude de l’époxydation des oléfines a aussi démontré que dans certains cas, l’oxydation du substrat peut se faire à partir de deux espèces électrophiles : le fer-oxo [FeV=O] mais aussi l’espèce hydropéroxyde [FeIII-OOH] (Vaz, McGinnity et al. 1998).
L’utilisation d’agents oxydants donneurs d’atomes d’oxygène (H2O2, hydroperoxydes,
peracides,…) permet d’obtenir directement le Fer-oxo à partir de l’état natif (en bleu sur la Figure I. 7).
INTRODUCTION
19 Ce cycle catalytique idéal aboutit à la formation d’un métabolite pour chaque molécule de substrat et de NAD(P)H entrant dans le cycle. Toutefois, il arrive que ce couplage ne soit pas efficace et dans ce cas, bien qu’il y ait consommation de NAD(P)H, le substrat n’est pas oxydé (voies en rouge sur la Figure I. 8). On parle alors de voies abortives ou non productives (découplage). Ceci se traduit par un retour à l’état natif du complexe et par la production d’espèces réactives de l’oxygène. Ce découplage entre le transfert d’électrons et le transfert d’atomes peut avoir lieu au niveau de plusieurs étapes du cycle catalytique :
• Le complexe ferreux − (étape 3) peut se décomposer, l’ion superoxyde ainsi généré pourra se dismuter et former du péroxyde d’hydrogène.
− → + .
2 . + 2 → +
• L’addition d’un proton sur l’oxygène proximal coordinant le fer du complexe ferrique − (étape 4) ou de son équivalent hydropéroxo (étape 5), va conduire à la formation d’un équivalent de péroxyde d’hydrogène.
− − − − +
− − − − → +
• L’espèce fer-oxo (étape 6) peut elle aussi redonner l’état natif en consommant deux protons et deux électrons.
= + 2 + 2 → +
I.1.2.4 Caractérisation spectrale de l’interaction entre l’hème et un composé fixé au site actif
A l’état natif, le complexe de FeIII est en équilibre entre ses deux formes : un état haut spin pentacoordiné dans lequel le fer est en dehors du plan de l’hème et qui possède un maximum d’absorption aux environs de 390 nm et un état bas spin hexacoordiné absorbant à 420 nm dans lequel le fer est dans le plan de l’hème (voir Figure I. 8).
Lors de l’entrée d’un composé qui se fixe au site actif, la signature spectrale dans le domaine UV-visible du complexe va être modifiée. On distingue trois cas de figure (Schenkman, Sligar et al. 1981) :
INTRODUCTION
20
• Le composé hydrophobe entre dans le site actif et chasse la molécule d’eau qui coordine le fer. Le complexe se retrouve alors majoritairement sous forme pentacoordinée, avec un maximum d’absorption à 390 nm. On observe alors un spectre de différence (par rapport à l’état natif) avec un pic à 390 nm et une vallée à 420 nm. On parle alors d’interaction de
type I qui est l’interaction la plus fréquemment rencontrée.
• Le composé hydrophobe porte cette fois un atome d’oxygène capable de coordiner le fer de l’hème et qui se substitue à la molécule d’eau comme sixième ligand du fer. Cette espèce est FeIII bas spin avec un pic de Soret à 420 nm. On observe donc un spectre de différence résultant avec un pic à 420 nm et une vallée à 390 nm. On parle alors d’interaction de type
I inversé.
• Le composé hydrophobe porte cette fois un atome d’azote ou de soufre accessible capable de coordiner le fer de l’hème et qui se substitue à la molécule d’eau comme sixième ligand du fer. Cette espèce est FeIII bas spin avec un pic de Soret entre 425 et 435 nm. On observe alors un spectre de différence résultant avec un pic entre 425 et 435 nm et une vallée entre 390 et 410 nm. On parle alors d’interaction de type II.
INTRODUCTION
21
Figure I. 8 : Différentes interactions structurales observées lors de la fixation d’un composé au site actif d’un P450. (Schenkman, Sligar et al. 1981; Mansuy, Battioni et al. 1989). BS : bas spin, HS : haut spin
I.1.3 Caractéristiques structurales
I.1.3.1 Un nombre de structures connues en expansion
Depuis le dépôt de la première structure de cytochrome P450 bactérien en 1985 (P450CAM
renommé CYP101 ) (Poulos, Finzel et al. 1985), le nombre de structures déposées sur la Protein Data Bank (PDB - accessible à l’adresse http://www.rcsb.org) n’a cessé de croître de façon exponentielle (voir Figure I. 9) et présente aujourd’hui1 391 structures différentes de P450s. La majorité des structures résolues sont issues de bactéries (270 entrées dans la PDB). On compte 112 structures de protéines d’eucaryotes (85 de mammifères, 15 de champignons, 10 de plantes et 2 de poisson) et 9 structures de P450 d’archaebactéries.
INTRODUCTION
22
Figure I. 9 : Evolution du nombre de structures de CYPs déposée à la Protein Data Bank.
Le premier P450 cristallisé dont la structure haute définition aux rayons X a été résolue est le cytochrome P450CAM (CYP101), issu de Pseudomonas putida, et qui catalyse l’hydroxylation régio-
et stéréo- spécifique du camphre en 5-exo-hydroxycamphre (Poulos, Finzel et al. 1987). Cette structure d’un P450 soluble est restée longtemps un paradigme pour l’étude des relations structure-fonction des P450s, jusqu’à la résolution d’autres structures de P450s solubles au cours de la décennie suivante.
Les structures des P450s membranaires de mammifères sont quant à elles restées longtemps inconnues, la cristallisation de domaines membranaires s’avérant extrêmement difficile. Il a donc fallut attendre 2000 pour que soit déposée la première structure de P450 de mammifère (Williams, Cosme et al. 2000). Cette structure du CYP2C5, une isoforme de lapin, a été résolue après plusieurs mutations de sa séquence et surtout la troncature de l’unique domaine transmembranaire (en position N-terminale) qui a été remplacé par un peptide hydrophile, rendant la protéine soluble. Il est accepté que ces modifications faites aux protéines membranaires n’affectent en rien leur repliement, ni leur fonction, dans la mesure où celle-ci conservent leurs activités enzymatiques (Cosme and Johnson 2000; Pan, Abd-Rashid et al. 2011). Cette technique a été ensuite largement adaptée à la résolution de structures de P450 membranaires. Le premier P450 humain, le CYP2C8, a ainsi été cristallisé en 2004 (Schoch, Yano et al. 2004) et, à ce jour 21 structures de P450s de mammifères différents ont été résolues (80 structures déposées à la PDB), dont les principales sont répertoriées dans le Tableau I. 1. On notera que la famille 2 a été la plus étudiée avec 10 des 16 isoformes présentes chez l’homme dont la structure a été résolue (voir aussi Tableau I. 3).
0 100 200 300 400 19 88 19 90 19 92 19 94 19 96 19 98 20 00 20 02 20 04 20 06 20 08 20 10 20 12 Nombre de structures déposées Date de dépôt bactérie mammifère archaébactérie champignon plante poisson
INTRODUCTION
23
CYP Entrées PDB Résolution (Å) Source Référence
CYP1A2 2HI4 1.95 Homo sapiens (Sansen, Yano et al. 2007)
CYP1B1 3PM0 2.70 Homo sapiens (Wang, Savas et al. 2011)
CYP2A13 (2 structures) Homo sapiens
dont 2P85 2.35 (Smith, Sanders et al. 2007)
CYP2A6 (12 structures) Homo sapiens
dont 2PG5 1.95 (Sansen, Hsu et al. 2007)
CYP2B4 (12 structures) Oryctolagus cuniculus
dont 3MVR 1.76 (Wilderman, Shah et al. 2010)
CYP2B6 (3 structures) Homo sapiens
dont 3IBD 2.00 (Gay, Shah et al. 2010)
CYP2C5 (2 structures) Oryctolagus cuniculus
dont 1NR6 2.10 (Wester, Johnson et al. 2003)
CYP2C8 (5 structures) Homo sapiens
dont 2NNH 2.60 (Schoch, Yano et al. 2008)
CYP2C9 (3 structures) Homo sapiens
dont 1R9O 2.00 (Wester, Yano et al. 2004)
CYP2D6 (2 structures) Homo sapiens
dont 3QM4 2.85 (Wang, Savas et al. 2012)
CYP2E1 (6 structures) Homo sapiens
dont 3T3Z 2.35 (Devore, Meneely et al. 2011)
CYP2R1 (3 structures) Homo sapiens
dont 3CZH 2.30 (Strushkevich, Usanov et al. 2008)
CYP3A4 (9 structures) Homo sapiens
dont 3NXU 2.00 (Sevrioukova and Poulos 2010)
CYP7A1 (2 structures) Homo sapiens
dont 3SN5 2.75 n/p
CYP8A1 (2 structures) Homo sapiens
dont 3B6H 1.62 (Li, Chiang et al. 2008)
CYP19A1 3EQM 2.90 Homo sapiens (Ghosh, Griswold et al. 2009)
CYP17A1 (2 structures) Homo sapiens
dont 3SWZ 2.40 (DeVore and Scott 2012)
CYP21A2 3QZ1 3.00 Bos taurus n/p
CYP24A1 (2 structures) Rattus norvegicus
dont 3K9V 2.50 (Annalora, Goodin et al. 2010)
CYP46A1 (6 structures) Homo sapiens
dont 3MDM 1.60 (Mast, White et al. 2008)
CYP51A1 (3 structures) Homo sapiens
dont 3I3K 2.80 (Strushkevich, Usanov et al. 2010)
Tableau I. 1: Les 80 structures des P450s de mammifères déposés à la Protéine Data Bank. Structures déposées avant le
08/03/12. n/p : non publié
I.1.3.2 Un repliement très conservé
Pour tous les P450s, qu’ils soient solubles ou membranaires, de classe I, II, III ou IV, et malgré de faibles pourcentages d’identité entre leurs séquences protéiques primaires (le P450CAM bactérien et le
CYP3A4 humain ne partagent que 23 % d’identité de séquence protéique) le repliement général et la topologie de tous les P450s cristallisés sont identiques. Toutes les structures cristallographiques de
INTRODUCTION
24 P450s ont une forme de prisme à base triangulaire dont la hauteur, pour la partie non membranaire, mesure environ 70Å pour une base de 40Å (voir Figure I. 10). Ce repliement est caractéristique des P450s et n’a été, à ce jour, retrouvé pour aucune autre protéine n’appartenant pas à la super-famille de P450.
Figure I. 10 : Comparaison d’une structure de P450 bactérien avec une de P450 de mammifère. Le CYP101 (code PDB :
2CCP) et le CYP3A4 (code PDB ITQN) sont représentés selon la même orientation. Les hélices sont colorées en rouge, les feuillets β en mauve, l’hème est représenté en bâtons roses. Le reste de la protéine est en gris
La nomenclature utilisée pour les éléments de structures secondaires est celle définie par Poulos en 1985 (Poulos, Finzel et al. 1985) lors de la publication de la toute première structure de P450, le P450CAM . Les 12 hélices α sont identifiées par des lettres majuscules (de A à L). Les feuillets β sont
eux numérotés de 1 à 5. Cette nomenclature a ensuite été enrichie au fur et à mesure de la découverte de nouvelles structures secondaires. De courtes hélices additionnelles ont été identifiées dans les structures résolues postérieurement. Elles ont été nommées B’, F’,… en fonction du nom de l’hélice qui les précède.
INTRODUCTION
25
Figure I. 11: (A) Topologie et éléments de structures secondaires du cytochrome P450 BM-3 d’après Graham et Peterson (Graham and Peterson 1999) Les hélices sont représentées en barres rouges de longueur proportionnelle à la taille de l’hélice, les brins
β par des flèches bleues et l’hème par un carré orange. (B) Structure du cytochrome P450 BM-3 annotée (code PDB 2HPD). Les hélices C’, J’, K’ et K’’ ainsi que les feuillets 2,4 et 5 ne sont pas indiqués par souci de visibilité.
On distingue deux régions dans la structure des P450s : un domaine α riche en hélices α bien structuré et un domaine β composé essentiellement de feuillets et de boucles qui présente une flexibilité plus importante (Figure I. 11).
INTRODUCTION
26 Les régions les plus conservées d’un point de vue structural sont celles qui participent à l’activation du dioxygène par le complexe hème-thiolate. Cet ensemble est composé de trois hélices parallèles (les hélices D, L et I) ainsi que de l’hélice E. De plus, afin d’assurer la stabilité du ligand cystéinate coordinant l’hème, une structuration particulière de la boucle contenant la cystéine est observée dans toutes les structures de P450s. Cet arrangement, appelé Cys-pocket correspond à la formation de deux liaisons hydrogènes entre la cystéine et deux amides du squelette peptidique.
Les régions les plus variables, quant à elles, correspondent à celles qui forment la cavité distale au- dessus de l’hème comprenant le site actif ainsi que les canaux d’accès des substrats et du solvant, elles correspondent aux hélices B’, F, G et à la boucle F-G (qui comprend les hélices F’ et G’ dans les protéines membranaires.
Les homologies structurales sont encore plus fortes lorsque l’on compare les P450s de mammifères (Otyepka, Skopalik et al. 2007). Les structures sont aisément superposables, avec une bonne conservation du positionnement de l’hème et de la position des canaux d’accès au site actif comme on peut le voir sur la Figure I. 12.
Figure I. 12: Superposition de deux structures de P450 de la famille 2, le CYP2D6 (code PDB : 2F9Q) et le CYP2C5 (code PDB : 1DT6) Les hélices sont colorées en rouge, les feuillets β en mauve, l’hème est représenté en bâtons. Le reste de la protéine est en gris. RMSD entre les deux structures 0.844Å.
INTRODUCTION
27
I.1.3.3 Topologie du site actif
Le site actif des P450s est une cavité hydrophobe située en position distale par rapport à l’hème. Il est délimité par les hélices F, G et I, les boucles B-C, C-terminale et K-K’. Cette topologie générale présente, mis à part le positionnement de l’hélice I, des disparités selon la nature (soluble ou membranaire) du P450, illustrées sur la Figure I. 13. Ainsi, la boucle F-G des P450s solubles qui forme le toit du site actif est remplacée par les hélices F’ et G’ des P450s membranaires, une grande variabilité est aussi observée au niveau de la boucle B-C et de l’hélice B’ et l’arrangement spatial des hélices F et G diffère selon le type de P450.
Figure I. 13: Superposition des structures secondaires bordant la cavité du P450CAM bactérien (en vert, code PDB : 1AKD)
et du CYP2R1 humain (en jaune, code PDB : 3CZH). L’hème est représenté en stick, le fer en sphère de Van Der Waals. Les deux
vues correspondent à une rotation de 90° de la structure.
En 1992, Gotoh a publié une carte des sites de reconnaissance de substrats (SRS ou substrate Recognition Site) des P450s de mammifères de la famille 2 (Gotoh 1992). Ces données obtenues par un simple alignement manuel des séquences avec celle du P450CAM ont permis de délimiter 6
domaines (notés SRS1 à SRS6), répartis sur tout le long de la séquence protéique. Ils définissent les sites de reconnaissance des substrats avec l’apoprotéine. Ces domaines forment des blocs homogènes qui présentent une forte variabilité de séquence ; cette variabilité ainsi que le positionnement des éléments structuraux du site actif peut donc expliquer la grande diversité de substrats pris en charge par les P450s.
INTRODUCTION
28 En 2011, une mise à jour de ce travail a été effectuée à partir des structures cristallographiques et d’expériences d’arrimage moléculaire (docking) (Zawaira, Ching et al. 2011). La seule différence majeure par rapport à la cartographie présentée par Gotoh est l’apparition de deux nouvelles régions d’interaction substrat/protéine (SRS1’a et SRS1’b) en amont du SRS1 (Figure I. 14).
Figure I. 14: Illustration de différents SRS obtenus par alignement de séquence (en jaune) ou par comparaison de structures (en vert). [A] : les deux nouveaux domaines identifiés en 2011, [B] : un domaine SRS similaire, le SRS1 identifié par Gotoh en 1992. Les SRS sont projetés sur un alignement des séquences de CYP2C9, CYP3A4 humain et CYP2B4 de lapin, d’après (Zawaira,
Ching et al. 2011)
I.1.3.4 Interaction avec la membrane
Contrairement aux P450 procaryotes, les P450 d’eucaryotes sont généralement des protéines membranaires. La plupart sont localisés au niveau de la membrane du réticulum endoplasmique. Toutefois quelques P450s de mammifères impliqués dans la biosynthèse de stérols, ou de stéroïdes, comme le CYP27B1 par exemple, sont situés sur la membrane interne des mitochondries (Paulson and DeLuca 1985). L’adressage de la protéine à la mitochondrie est rendu possible par la présence d’un peptide signal de 30 à 50 acides aminés qui guide la translocation de la protéine. Un fois le transfert du P450 mitochondrial effectué, le peptide signal est clivé (Omura and Ito 1991).
On peut supposer que l’interaction de la membrane avec le domaine catalytique du P450 facilite l’entrée de substrats hydrophobes depuis la membrane et oriente le P450 de façon à garantir une
INTRODUCTION
29 interaction optimale avec ses partenaires rédox (cytochrome P450 réductase, cytochrome b5). Il est toutefois accepté que l’absence du domaine N-terminal n’affecte en rien l’activité catalytique du P450, l’expression d’enzymes tronquées et actives ayant été effectuée pour de nombreux isoformes (von Wachenfeldt, Richardson et al. 1997; Cosme and Johnson 2000; Strushkevich, Usanov et al. 2008).
L’ancrage principal des P450s à la membrane se fait par un domaine prédit comme transmembranaire en partie N-terminale. Cette région contient successivement une vingtaine de résidus apolaires (partie enfouie) suivie d’un motif conservé riche en proline, comprenant des résidus chargés positivement (en interaction avec les têtes polaires des phospholipides) comme représenté sur la Figure I. 15.
Figure I. 15: alignement des séquences N-terminales de P450s humains. Alignement effectué avec MUSCLE1 (Edgar 2004)
L’étude de l’association de P450 avec une monocouche de phospholipides de type Langmuir-Blodget a montré que l’aire déplacée par le P450 lors de son association avec la membrane est supérieure à l’aire d’une simple hélice transmembranaire (Shank-Retzlaff, Raner et al. 1998). Il existerait donc d’autres motifs structuraux en interaction avec la membrane.
Une des particularités des structures de P450s eucaryotes est la présence de nouvelles hélices entre F et G : soit deux courtes hélices F’ et G’ (comme dans le CYP2C5 ou le CYP2R1) soit une seule hélice plus longue F’ (comme dans le CYP2B4). Cette région est composée majoritairement de résidus hydrophobes et est localisée au voisinage immédiat du domaine N-terminal. Il a donc été envisagé que ces hélices soient en interaction avec la membrane ou même qu’elles soient enfouies dans celle-ci. Cette suggestion a été confirmée par l’étude des épitopes du CYP2B4 (De Lemos-Chiarandini, Frey et al. 1987), des études de fluorescence sur le CYP11A1 de bovin (Headlam, Wilce et al. 2003), des
INTRODUCTION
30 expériences de mutagénèse (Williams, Cosme et al. 2000; Williams, Cosme et al. 2003) et des expériences de lyse trypsique (Pikuleva, Mast et al. 2008; Mast, Liao et al. 2009).
De même, l’étude par fluorescence des tryptophanes du CYP2C2 de lapin (Ozalp, Szczesna-Skorupa et al. 2006) montre que des tryptophanes de l’hélice A et du brin β2-2 voient leur fluorescence diminuée en présence de phospholipides modifiés afin de quencher l’émission de rayonnements. Ces deux éléments de structure secondaire sont donc aussi potentiellement en interaction avec la membrane.
Deux autres paramètres caractérisant l’interaction protéine / membrane ont été étudiés :
• L’orientation de l’hème (et donc de la protéine) par rapport à la membrane a pu être estimée entre 50° et 70° pour les CYP17 et CYP21 en étudiant la décroissance de l’absorption anisotrope consécutive à la dissociation des complexes [FeII-CO] (Ohta, Kawato et al. 1992).
• Une étude de microscopie à force atomique a aussi permis de déterminer que le P450 affleurait à une hauteur de 35 à 45 nm au-dessus du plan supérieur de la membrane (Bayburt and Sligar 2002).
Grâce à tous ces éléments, la position et l’orientation des P450s mammifères a pu être déterminée et la construction de modèles d’assemblage en membrane des CYP2C5 (Williams, Cosme et al. 2000), 2B4 (Zhao, White et al. 2006) et 2C9 (Berka, Hendrychova et al. 2011; Cojocaru, Balali-Mood et al. 2011) a démontré l’importance de cette interaction notamment pour l’incorporation de substrats hydrophobes au site actif de l’enzyme.
I.1.3.5 Interaction avec la cytochrome P450 réductase chez les eucaryotes
Structure des P450 réductases.
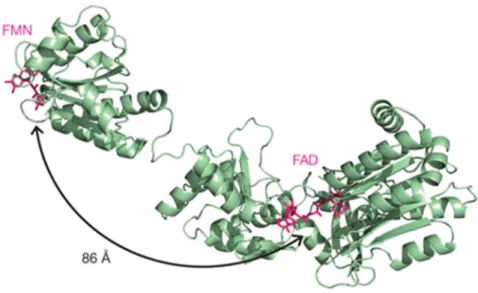
En parallèle à l’étude structurale des P450s, la cristallisation de son principal partenaire rédox, la P450 réductase, a fait l’objet d’efforts soutenus. En 1997, la première structure de P450 réductase (de rat) a été déposée à la PDB (Wang, Roberts et al. 1997). Aujourd’hui, on compte une vingtaine de structures déposées dont celle de la P450 réductase humaine (codes PDB : 3QE2, 2QFC et 3QFR) (Xia, Hamdane et al. 2011). Comme pour les P450s, la réductase est ancrée à la membrane par une hélice N-terminale transmembranaire ; cette partie a été tronquée afin de pouvoir produire une protéine soluble compatible avec les conditions de cristallisation. Les structures publiées ont permis de distinguer 4 éléments structuraux caractéristiques des P450 réductases : un domaine N-terminal
INTRODUCTION
31 comprenant un cofacteur FMN, un domaine de liaison au P450, un domaine à flavine FAD et un domaine C-terminal de liaison au NADPH (voir Figure I. 16).
Figure I. 16 : Les quatre domaines structuraux de la P450 réductase humaine (code PDB : 3QE2, protéine tronquée de 63 résidus en partie N-terminale). La protéine est représentée en mode cartoon, les cofacteurs en bâton. Le domaine N-terminal à FMN
est coloré en rose, le domaine de liaison au P450 en bleu, le domaine à flavine FAD en vert et le domaine de liaison au NADPH en orange.
Interaction et transfert d’électrons au P450.
Contrairement aux P450s tronqués qui sont actifs malgré la délétion de leur hélice transmembranaire, les formes solubles de réductase de mammifère ne sont pas capables de transférer des électrons aux P450s. L’ancrage de ces enzymes à la membrane semble être nécessaire pour assurer une bonne orientation spatiale des centres rédox et ainsi assurer un transfert d’électron efficace (Gideon, Kumari et al. 2012).
L’étude de ces assemblages (P450, P450 réductase, bicouche lipidique) reste complexe et l’orientation relative de la réductase par rapport au P450 non totalement élucidée. La composition
INTRODUCTION
32 lipidique de la membrane affecte en effet l’interaction entre P450 et réductase (Ingelman-Sundberg, Haaparanta et al. 1981; Ingelman-(Ingelman-Sundberg, Blanck et al. 1983).
Cependant des expériences d’arrimage moléculaire (Wang, Roberts et al. 1997) et de mutagénèse dirigée (Zhao, Modi et al. 1999) ont montré que la principale zone d’interaction de la réductase avec le P450 était située au niveau du domaine à FMN via une interaction principalement électrostatique accompagnée d’une modification conformationnelle, la P450 réductase adoptant alors une conformation ouverte (Figure I. 17) propice au transfert d’électrons.
Figure I. 17 : Structure ouverte d’une P450 réductase. Protéine chimérique humaine / de levure. D’après L. Aigrain (Aigrain,
Pompon et al. 2009)
I.1.3.6 Les canaux d’accès au site actif
Le site actif des P450s étant profondément enfoui au cœur de la protéine et à proximité du complexe hème-thiolate, donc complètement isolé du milieu extérieur, l’identification des chemins d’entrée et de sorties du solvant et des substrats est un enjeu crucial afin, notamment, de comprendre les spécificités de substrat observées. L’ouverture et la fermeture de canaux d’entrée de substrats ont tout d’abord été observés et caractérisés par des expériences de dynamique moléculaire sur des structures de P450 bactériens au cours desquelles la sortie sous contrainte du substrat du site actif de l’enzyme permettait de définir les canaux d’accès (Ludemann, Lounnas et al. 2000). L’étude s’est ensuite élargie aux structures de P450s de mammifères (Otyepka, Skopalik et al. 2007). Les expériences de dynamique moléculaire ont été abandonnées au profit d’algorithmes - CAVER (Petrek, Otyepka et al. 2006) puis MOLE (Petrek, Kosinova et al. 2007) - explorant les cavités de la protéine avec une sonde afin de trouver des voies d’accès au site actif (Cojocaru, Winn et al. 2007; Winn,
INTRODUCTION
33 Cojocaru et al. 2007). Une nomenclature de ces canaux d’accès, bien qu’historiquement plus adaptée aux structures bactériennes, est aujourd’hui utilisée pour caractériser les canaux d’accès au site actif aussi bien pour les P450s bactériens que pour les P450s de mammifère. Elle est présentée dans la Figure I. 18 et le Tableau I. 2.
Le canal 1 est un canal rare qui n’a été observé que sur le P450CAM. Les canaux de la famille 2 sont
tous localisés au voisinage de la boucle B-C et de l’hélice B’. C’est une région très variable aussi bien en termes de séquence qu’en termes de structure et qui conditionne donc la spécificité de substrat de chaque isoforme. La boucle B-C a d’ailleurs été identifiée comme l’un des six sites de reconnaissance de substrat (SRS) des P450s de la famille 2 (Gotoh 1992). Les canaux 3, 4 et 5 sont, au même titre que le canal 1, rarement observés, les canaux S et W servent quant à eux à la diffusion de l’eau du fait de leurs orientations et de leurs largeurs non compatibles avec le passage d’un substrat (Poulos, Finzel et al. 1987). Il existe cependant des exceptions puisqu’un canal S d’un rayon minimal de 2,2Å (suffisamment large pour le passage d’un substrat) a été observé sur le CYP2D6 (Rowland, Blaney et al. 2006). Il est donc possible que le canal S soit aussi une voie d’entrée des substrats au site actif de l’enzyme.
INTRODUCTION
34
Figure I. 18 : Représentation des différents canaux d’accès des P450s (Winn, Cojocaru et al. 2007). Les différents canaux sont
visualisés sur la protéine P450CAM (code PDB : 1AKD). La protéine est représentée en cartoon gris, l’hème en bâtons. Les deux vues A et B (reliées par une rotation de 90°) sont nécessaires à la visualisation de tous les canaux. La boucle B-C est colorée en magenta, la boucle F-G en vert. Les ouvertures des canaux sont représentées par des cercles positionnés en surface de la protéine. Les canaux identifiés par une même couleur sont liés par (i) un même mode d’ouverture (2a, 2b, 2c, 2ac, 2f en rouge et 4, 2c, 2e en vert), (ii) leur fonction (canaux solvant-S et water-W en bleu) ou (iii) ceux identifiés plus récemment (canal 5 en violet)
INTRODUCTION
35
canal Localisation Commentaire
1 entre les hélices C/C' et H ou L, proche de la boucle G-H et du
feuillet β2 Canal rare - identifié sur P450CAM
2a entre la boucle F-G, l'hélice B', la boucle B-B' et le feuillet β1
2b entre la boucle B-B' et les feuillets β1 et β3
2c entre les hélices B', G, I et la boucle B-C
2ac entre l'extrémité de la boucle B-C et l'hélice G canal situé entre les canaux 2a et 2c
2e à travers la boucle B-C
2d proche du canal 2a. Sortie du canal entre les hélices A et A'
2f proche du canal 2a. Sortie du canal entre l'hélice F et la boucle F-G
3 entre les hélices F et G ou au niveau de la boucle E-F canal rare
4 à travers la boucle F-G canal rare – ex : CYP6B (Li, Baudry et al.
2004)
5 entre les hélices K et K' canal rare – ex : CYP2A6
Solvant (S) entre les hélices E, F, I et le feuillet β5
Water (w) sortie du canal à la base de la boucle B-C à proximité de l'extrémité C-terminale de l'hélice B, canal proximal entrée de la molécule d'eau entrant dans le cycle catalytique du P450 Tableau I. 2 : Localisation des canaux d’accès au site actif des P450s (Winn, Cojocaru et al. 2007)
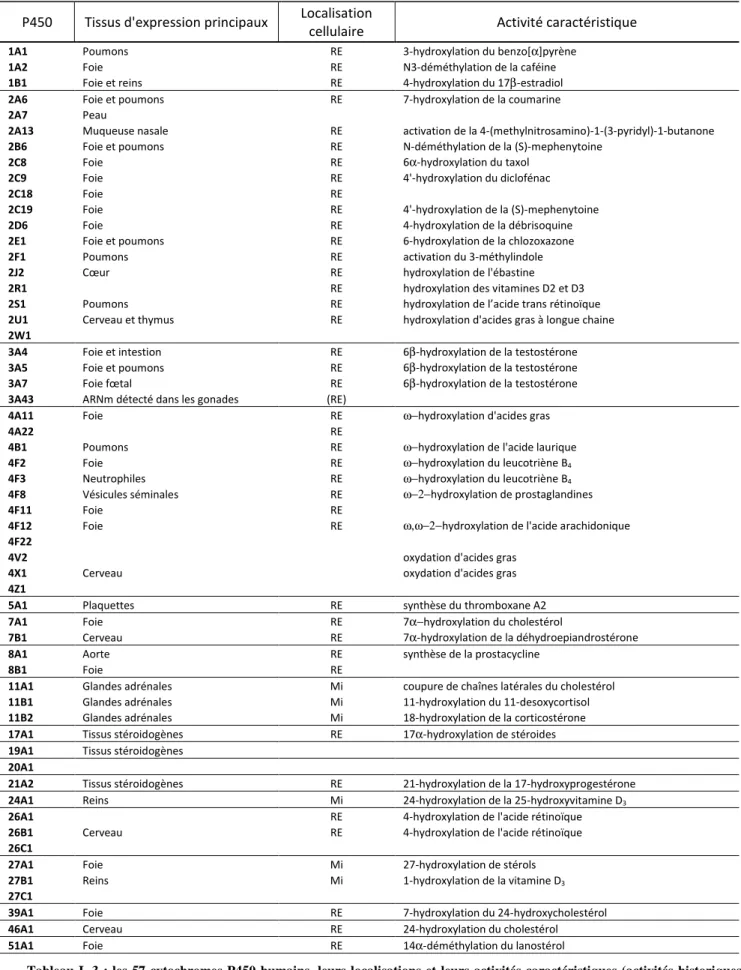
I.1.4 Les cytochromes P450 humains
Le séquençage du génome humain (achevé en 2003) a montré qu’il existe 57 gènes codant pour des cytochromes P450. Ces enzymes sont membranaires, certaines ancrées aux membranes du réticulum endoplasmique, d’autres aux membranes des mitochondries.
INTRODUCTION
36
P450 Tissus d'expression principaux Localisation
cellulaire Activité caractéristique
1A1 Poumons RE 3-hydroxylation du benzo[α]pyrène
1A2 Foie RE N3-déméthylation de la caféine
1B1 Foie et reins RE 4-hydroxylation du 17β-estradiol
2A6 Foie et poumons RE 7-hydroxylation de la coumarine
2A7 Peau
2A13 Muqueuse nasale RE activation de la 4-(methylnitrosamino)-1-(3-pyridyl)-1-butanone
2B6 Foie et poumons RE N-déméthylation de la (S)-mephenytoine
2C8 Foie RE 6α-hydroxylation du taxol
2C9 Foie RE 4'-hydroxylation du diclofénac
2C18 Foie RE
2C19 Foie RE 4'-hydroxylation de la (S)-mephenytoine
2D6 Foie RE 4-hydroxylation de la débrisoquine
2E1 Foie et poumons RE 6-hydroxylation de la chlozoxazone
2F1 Poumons RE activation du 3-méthylindole
2J2 Cœur RE hydroxylation de l'ébastine
2R1 RE hydroxylation des vitamines D2 et D3
2S1 Poumons RE hydroxylation de l’acide trans rétinoïque
2U1 Cerveau et thymus RE hydroxylation d'acides gras à longue chaine
2W1
3A4 Foie et intestion RE 6β-hydroxylation de la testostérone
3A5 Foie et poumons RE 6β-hydroxylation de la testostérone
3A7 Foie fœtal RE 6β-hydroxylation de la testostérone
3A43 ARNm détecté dans les gonades (RE)
4A11 Foie RE ω−hydroxylation d'acides gras
4A22 RE
4B1 Poumons RE ω−hydroxylation de l'acide laurique
4F2 Foie RE ω−hydroxylation du leucotriène B4
4F3 Neutrophiles RE ω−hydroxylation du leucotriène B4 4F8 Vésicules séminales RE ω−2−hydroxylation de prostaglandines
4F11 Foie RE
4F12 Foie RE ω,ω−2−hydroxylation de l'acide arachidonique
4F22
4V2 oxydation d'acides gras
4X1 Cerveau oxydation d'acides gras
4Z1
5A1 Plaquettes RE synthèse du thromboxane A2
7A1 Foie RE 7α−hydroxylation du cholestérol
7B1 Cerveau RE 7α-hydroxylation de la déhydroepiandrostérone
8A1 Aorte RE synthèse de la prostacycline
8B1 Foie RE
11A1 Glandes adrénales Mi coupure de chaînes latérales du cholestérol
11B1 Glandes adrénales Mi 11-hydroxylation du 11-desoxycortisol
11B2 Glandes adrénales Mi 18-hydroxylation de la corticostérone
17A1 Tissus stéroidogènes RE 17α-hydroxylation de stéroides
19A1 Tissus stéroidogènes
20A1
21A2 Tissus stéroidogènes RE 21-hydroxylation de la 17-hydroxyprogestérone
24A1 Reins Mi 24-hydroxylation de la 25-hydroxyvitamine D3 26A1 RE 4-hydroxylation de l'acide rétinoïque
26B1 Cerveau RE 4-hydroxylation de l'acide rétinoïque
26C1
27A1 Foie Mi 27-hydroxylation de stérols
27B1 Reins Mi 1-hydroxylation de la vitamine D3 27C1
39A1 Foie RE 7-hydroxylation du 24-hydroxycholestérol
46A1 Cerveau RE 24-hydroxylation du cholestérol
51A1 Foie RE 14α-déméthylation du lanostérol
Tableau I. 3 : les 57 cytochromes P450 humains, leurs localisations et leurs activités caractéristiques (activités historiques qui ont permis de détecter le P450). RE : Reticulum endoplasmique, Mi : Mitochondrie, d'après (Guengerich, 2005)
INTRODUCTION
37 Parmi ces 57 P450s humains, quinze environ sont connus pour être impliqués dans la phase I du métabolisme des xénobiotiques (voir Tableau I. 4). Ils participent ainsi à l’élimination des composés exogènes comme les médicaments, les toxines ou les polluants. On dénombre aussi environ 20 P450s impliqués dans la biosynthèse et la dégradation des stérols, vitamines et eïcosanoïdes. Parmi les P450s dont le rôle biologique est encore incertain ou inconnu, on peut distinguer ceux qui semblent être impliqués dans la dégradation des eïcosanoïdes ou dans la mono-oxygénation des acides gras. Enfin les 15 P450s dont l’existence est connue depuis le séquençage du génome humain mais dont l’étude du rôle biologique est incomplète ou inexistante sont appelés « P450s orphelins ».
Métabolisation Rôle endogène Rôle biologique incertain ou inconnu des xénobiotiques Stérols Eïcosanoïdes Vitamines Acides gras Eïcosanoïdes Orphelins
1A1 1B1 5A1 24A1 2J2 4F2 2A7
1A2 7A1 8A1 26A1 4A11 4F3 2R1
2A6 7B1 26B1 4B1 4F8 2S1 2B6 8B1 27B1 4F12 2U1 2C8 11A1 2W1 2C9 11B1 3A43 2C18 11B2 4A22 2C19 17A1 4F11 2D6 19A1 4F22 2E1 21A2 4V2 2F1 27A1 4X1
3A4 39A1 20A1
3A5 46A1 26C1
3A7 51A1 27C1
Tableau I. 4 : Classification des P450s humains basée sur leur principal substrat (Guengerich, Wu et al. 2005) Cette
INTRODUCTION
38
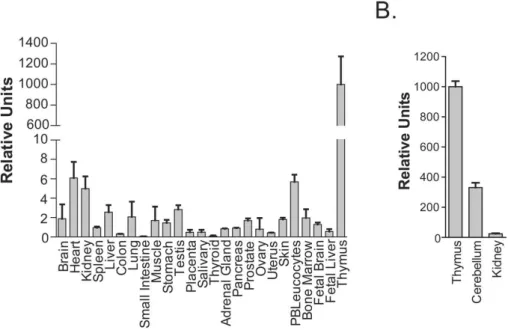
I.1.5 Les cytochromes P450 du cerveau
Le taux d’expression de P450 dans le cerveau a été pour la première fois évalué chez le rat à environ 30 pmol/mg de protéine ce qui correspond à 3% du niveau atteint dans le foie (Sasame, Ames et al. 1977). Chez l’homme, selon les auteurs, ces même taux correspondraient à 1 à 10 % de ceux relevés au niveau hépatique. Toutefois, ces résultats ne prennent pas en compte une distribution non uniforme des P450s au sein de l’organe. En effet, la quantité et la nature des P450s diffèrent selon la région de cerveau considérée (voir Tableau I. 5).
Par exemple la proportion de P450s présents dans le cervelet est la plus élevée (Warner, Kohler et al. 1988). Il existe aussi quelques P450s exprimés de façon prédominante dans le cerveau : le CYP26B1 présent dans le cervelet et le pont de Varole, ou le CYP46A1 présent dans l’ensemble de l’organe, en sont des exemples. Ces enzymes exprimées spécifiquement dans le cerveau ne sont pas impliquées dans le métabolisme de xénobiotiques mais possèdent des fonctions endogènes majeures comme la régulation de la biosynthèse du cholestérol ou le métabolisme de la vitamine A (Karlgren, Miura et al. 2005).
Plus récemment, des techniques plus sensibles que le Western Blot, comme la RT-PCR, le Northern Blot ou l’hybridation in situ, ont permis de détecter d’autres P450s dans le cerveau : ainsi, des P450s de la famille 4 ont été identifiés dans le cortex, le cervelet et le tronc cérébral (Bylund, Zhang et al. 2002). A l’heure actuelle, on ne connaît pas de fonction physiologique ni d’activité enzymatique précise pour ces P450s.
Le fait que le taux de certains P450s dans le cerveau soit faible ne signifie pas qu’ils n’aient pas une importance capitale dans certaines fonctions physiologiques. Ainsi le CYP19, également appelé aromatase, catalyse la conversion d’androgènes en œstrogènes dans une zone très particulière du cerveau et joue un rôle physiologique capital (Roselli, Liu et al. 2009).
INTRODUCTION
39
Cortex Cervelet Ganglions Hippocampe Substance Bulbe Pont
de la base noire rachidien de Varole
CYP1A1 + + + + + - + CYA1A2 + + + ND + - + CYP1B1 + + + + + + - CYP2C8 + + + ND ND - - CYP2D6 + + + + + - + CYP2E1 + + + + + + + CYP2U1 ND + ND ND ND ND ND CYP3A4 + - + ND ND - - CYP3A5 + - + ND ND - - CYP26B1 + + - ND ND + + CYP46A1 + - + + + - ND
Tableau I. 5 : Présence des ARNm de quelques P450s dans différentes régions du cerveau humain (Dutheil, Beaune et al. 2008) ND : non déterminé
I.2
Les cytochromes P450 orphelins chez l’homme
I.2.1 Déorphelinisation en cours
Lors de la publication de la liste des P450s humains orphelins en 2005 (voir Tableau I. 4), on ne connaissait que très peu de chose sur ces protéines. Souvent, seule l’existence du gène était connue. Depuis, de nombreuses équipes se sont penchées sur le rôle biologique de cette quinzaine d’isoformes. Sont regroupées, ci-dessous, les principales découvertes faites pour chaque P450 orphelin ces dernières années:
CYP2A7 : Très proche du CYP2A6 avec 96% d’identité de séquence nucléotidique, l’expression
de cet isoforme a été étudiée ainsi que le polymorphisme génétique qui lui est associé. Il existe aussi sous la forme d’un variant d’épissage présent dans les fibroblastes de peau. Aucun substrat n’est encore connu. (Ding, Lake et al. 1995; Fukami, Nakajima et al. 2006).
CYP2R1 : Le rôle biologique du CYP2R1 a été élucidé en 2004 avec la découverte de sa capacité
à hydroxyler en position 25, les deux formes de la vitamine D : les vitamines D2 et D3 (Cheng, Motola et al. 2003). Cristallisé en complexe avec les vitamines D3 puis D2, sa structure tridimensionnelle est déposée à la PDB depuis février 2008 (Strushkevich, Usanov et al. 2008).
INTRODUCTION
40
CYP2S1 : Cette isoforme est fortement exprimée dans des tissus extrahépatiques (trachée,
poumons, estomac, intestin grêle, rate,…) (Rylander, Neve et al. 2001). L’expression du CYP2S1 chez l’homme est inductible par un certain nombre de molécules cancérigènes telles que la dioxine (Rivera, Saarikoski et al. 2002) et par les UV (Smith, Wolf et al. 2003). Enfin, deux activités enzymatiques ont été découvertes : le CYP2S1 catalyse l’hydroxylation de l’acide trans rétinoïque (Smith, Wolf et al. 2003) et, surexprimé dans la levure, il transforme le naphtalène en deux métabolites non identifiés à ce jour (Karlgren, Miura et al. 2005).
CYP2U1 : détaillé infra (paragraphe I.3)
CYP2W1 : La recherche par Northern Blot du CYP2W1 dans différents tissus est longtemps
restée infructueuse, les taux d’ARNm détectés étant très bas et la protéine non détectable en Western Blot (Karlgren, Miura et al. 2005). L’expression de cette enzyme a ensuite été établie et ce, à de hauts niveaux d’expression, dans certaines tumeurs (Karlgren, Gomez et al. 2006; Gomez, Nekvindova et al. 2010). Surexprimé dans E. coli, le CYP2W1 montre une activité hydroxylase sur l’indole, la benzphetamine, le 3-methylindole et la chlorzoxazone (5-chloro-3H-benzoxazol-2-one). (Wu, Sohl et al. 2006; Yoshioka, Kasai et al. 2006). D’un point de vue structural, bien que cette isoforme n’ait pas été cristallisée, la topologie du site actif a été déterminée avec la construction d’un modèle par homologie (Li, Tang et al. 2009).
CYP3A43 : Quatrième membre de la sous-famille 3A, le CYP3A43 possède de 71 à 76%
d’identité de séquence avec les autres CYP3A. Bien que détecté dans le foie, les reins, le pancréas et la prostate, les taux d’expression du CYP43A3 sont très faibles (moins de 0.1% des taux observés pour le CYP3A4). Au contraire, dans le cerveau, son taux d’expression est 10 fois supérieur à celui du CYP3A4. La protéine recombinante métabolise aussi l’alprazolam (Agarwal, Kommaddi et al. 2008) ainsi que la testostérone (Domanski, Finta et al. 2001). Son orthologue chez le macaque a été surexprimé dans E. coli et présente une activité d’hydroxylation du midazolam en position 1 (Uno, Matsuno et al. 2009).
CYP4A22 : On connaît très peu de chose sur cette isoforme. Longtemps confondu avec le
CYP4A11 avec lequel il partage 95% d’identité de séquence, il est plus faiblement exprimé que celui-ci aussi bien dans le foie que dans les reins (Bellamine, Wang et al. 2003). Le polymorphisme génétique a été étudié sur différentes populations montrant que cette protéine est faiblement conservée avec plus de cinq allèles différents du gène découverts (Hiratsuka, Nozawa et al. 2006; Lino Cardenas,