U.F.R SVT
T H E S E
Pour obtenir le grade de
DOCTEUR DE L’UNIVERSITE TOULOUSE III
Discipline : Biosciences végétales
Spécialité : Phytopathologie Cellulaire et Moléculaire
Présentée par
Seyed Kazem SABBAGH
Soutenue le 17 Janvier 2008 à 14 heures
Salle de Séminaire de l’IFR40
Pôle de Biotechnologie Végétale,
Chemin de Borde Rouge, 31326 Castanet-Tolosan
Titre :
Adaptation à la pénétration racinaire de deux Ustilaginaceae parasites du maïs : Ustilago
maydis et Sporisorium reilianum – Analyse microscopique et transcriptomique
JURY
Pr Patrice REY, ENITA de Bordeaux
Rapporteur
Dr Philippe REIGNAULT, Université Littoral Côte d'Opale
Rapporteur
Pr. Grégory DECHAMP-GUILLAUME, ENSA de Toulouse
Examinateur
Pr Philippe DELAVAULT, Université de Nantes
Examinateur
U.F.R SVT
T H E S E
Pour obtenir le grade de
DOCTEUR DE L’UNIVERSITE TOULOUSE III
Discipline : Biosciences végétales
Spécialité : Phytopathologie Cellulaire et Moléculaire
Présentée par
Seyed Kazem SABBAGH
Le 17 Janvier 2008
Titre :
Adaptation à la pénétration racinaire de deux Ustilaginaceae parasites du maïs : Ustilago
maydis et Sporisorium reilianum – Analyse microscopique et transcriptomique
JURY
Pr Patrice REY, ENITA de Bordeaux
Rapporteur
Dr Philippe REIGNAULT, Université Littoral Côte d'Opale
Rapporteur
Pr. Grégory DECHAMP-GUILLAUME, ENSA de Toulouse
Examinateur
Pr Philippe DELAVAULT, Université de Nantes
Examinateur
Remerciements
Je tiens tout d’abord spécialement exprimer ma gratitude de mon responsable de thèse,
Monsieur le Professeur Christophe ROUX pour le confiance qu’il ma témoignée en me
proposant ce sujet, pour sa disponibilité, sa gentillesse et son ouverture d’esprit tout au long
de cette thèse.
Je remercie à Professeur Guillaume Bécard de m’avoir accueilli au sein de laboratoire et
dans son équipe.
Je remercie également à Dr. Nathalie Séjalon-Delmase pour ses conseils scientifique et sa
disponibilité.
Je tiens à remercier Messieurs Patrice REY et Philippe REIGNAULT, Philippe DELAVAULT,
Grégory DECHAMP-GUILLAUME ont accepté de participer au jury de thèse. Je les en
remercie.
Je tiens à remercier également professeur Ahmad SARRAFI pour son aide précieuse sur le
plan professionnel et également Yves Martinez pour la découverte en toute décontraction
de la microscopie.
Je remercie chaleureusement toute l’équipe du « Symbiose Endomycorhizienne et
Signalisation chez les végétaux » (les chercheurs et les techniciens) ainsi que tous les
personnel de laboratoire
En fine, un grand merci à ma femme, Dr. Mahta MAZAHERI et ma petite fille Sahel pour leur
soutien chaleureux ainsi qu’à nos familles en IRAN.
Cette thèse a été réalisée dans le cadre d’une bourse accordé par le Ministre de l’Education
National et la Recherche en Iran. J’exprime ici mon remerciement pour le financement de
mes études en France ainsi qu’au service de l’Education de l’Ambassade d’ Iran en France.
Table des matières
Table des matières ____________________________________________________________ 1
Résumé _____________________________________________________________________ 4
Summary ____________________________________________________________________ 5
Préambule ___________________________________________________________________ 6
Introduction générale __________________________________________________________ 7
Le monde des champignons : de l’observation microscopique à l’observation des génomes _______ 7 Les types trophiques des champignons _________________________________________________ 9 Les Ustilaginaceae : des basidiomycètes parasites biotrophes facultatifs ______________________ 12 Les Ustilaginaceae au sein des Basidiomycotina _______________________________________ 12 Diversité des Ustilaginaceae ______________________________________________________ 14 Divergence de Ustilaginaceae et domestication des plantes _____________________________ 15 Le cycle des Ustilaginaceae _______________________________________________________ 16 Cycle typique et variations _______________________________________________________ 16 Mating-type chez les Ustilaginaceae ________________________________________________ 21 Solopathogénie chez les Ustilaginaceae _____________________________________________ 23 Le maïs, plante modèle en pathologie végétale ? ________________________________________ 24 Le maïs _______________________________________________________________________ 24 Résistance du maïs aux charbons __________________________________________________ 25 La rhizosphère ___________________________________________________________________ 26 Le sol, une matrice entre les organismes du sol et les plantes ____________________________ 26 Les exsudats racinaires __________________________________________________________ 26 Rôle des exsudats racinaires dans les étapes précoces des interactions racinaires ____________ 27 Les strigolactones ______________________________________________________________ 28 Objectifs de la thèse _______________________________________________________________ 30 1-Quel est le taux de souches solopathogènes formées par différentes Ustilaginaceae ? _______ 30 2-Ustilago maydis est il un agent pathogène racinaire ? _________________________________ 30 3-Quelle est la réponse des Ustilaginaceae aux strigolactones ? __________________________ 31CHAPITRE I – Les formes solopathogènes des Ustilaginaceae : une adaptation au mode de
dissémination ? ______________________________________________________________ 33
Avant Propos _____________________________________________________________________ 33
1-2 Introduction___________________________________________________________________ 34
1-3 Materials and Methods__________________________________________________________ 36 Fungal material _________________________________________________________________________ 36 Protocole of obtention of solopathogenic strains of S. reilianum __________________________________ 36 Comparison of fuzzy strain production _______________________________________________________ 37 Plant infection test ______________________________________________________________________ 37 Microscopic observations _________________________________________________________________ 37 Molecular analysis. ______________________________________________________________________ 37 1-4 RESULTS ______________________________________________________________________ 39 Isolation of fuzzy strains of S. reilianum ______________________________________________________ 39 Stable fuzzy strains of S. reilianum are monocaryotic and diploid __________________________________ 40 Monocaryotic diploid fuzzy strains of S. reilianum are solopathogen, but do not form sori ______________ 41 Frequency of production of solopathogenic strains differs according to Ustilaginaceae species __________ 41 1-5 Conclusion ____________________________________________________________________ 42
1-6 References ____________________________________________________________________ 44
CHAPITRE II - Ustilago maydis : un nouveau modèle pour l’étude des infections racinaires chez
les plantes hôte et non-hôte ____________________________________________________ 46
Avant Propos _____________________________________________________________________ 46II-1 Abstract ______________________________________________________________________ 47
II-2 Introduction __________________________________________________________________ 47
II-3 Materials and methods __________________________________________________________ 49 Fungal strains and culture condition _________________________________________________________ 49 Plants and culture conditions ______________________________________________________________ 49 Microscopic observations _________________________________________________________________ 49 PCR detection __________________________________________________________________________ 50 Effect of GR24 on cell respiration ___________________________________________________________ 50 RNA extraction and RT-PCR ________________________________________________________________ 51 Primer design and qRT-PCR ________________________________________________________________ 51 II-4 Results _______________________________________________________________________ 53 Ustilago maydis infects maize roots _________________________________________________________ 53 Maize root infection by Ustilago maydis didn’t cause symptoms___________________________________ 55 Ustilago maydis can infect Medicago roots ___________________________________________________ 55 A strigolactone analogue stimulates U.maydis cell respiration ____________________________________ 57
II-5 Discussion ____________________________________________________________________ 58
II-6 References ____________________________________________________________________ 60
CHAPITRE III - Analyse du transcriptome de Sporisorium reilianum en réponse au GR24, un
analogue de synthèse de strigolactone ___________________________________________ 63
Avant propos _____________________________________________________________________ 63
III-1 Summary ____________________________________________________________________ 65
III-2 Introduction __________________________________________________________________ 65
III-3Experimental procedures ________________________________________________________ 66 Fungal strain and cultural condition _________________________________________________________ 66 GR24 induction _________________________________________________________________________ 66 Cell respiration _________________________________________________________________________ 67 RNA isolation and Reverse Transcriptase-Polymerase Chain Reaction (RT-PCR) _______________________ 67 Suppressive Subtractive Hybridization (SSH) __________________________________________________ 68 Probe labelling _________________________________________________________________________ 68 cDNA array and hybridization ______________________________________________________________ 69 Detection and quantification of hybridization signals ___________________________________________ 69 Sequence analysis and annotation __________________________________________________________ 70 Primers design __________________________________________________________________________ 71 RT-qPCR _______________________________________________________________________________ 71 III-4 Results ______________________________________________________________________ 71 GR24 increases cell respiration of S. reilianum _________________________________________________ 71 Construction of SSH cDNA libraries and macroarray design _______________________________________ 72 Hybridization of macroarray _______________________________________________________________ 73 cDNA sequence analysis __________________________________________________________________ 73 RT-q PCR ______________________________________________________________________________ 75 III-5 Discussion ____________________________________________________________________ 77
III-6 References ___________________________________________________________________ 78
Supplementary data _______________________________________________________________ 80
Discussion et perspectives _____________________________________________________ 85
Les Ustilaginaceae : modèles fongiques d’étude des interactions biotrophes parasites racinaires _ 85Les Ustilaginaceae, modèles d’étude pour la perception des strigolactones ___________________ 88
Résumé
Les Ustilaginaceae constituent une famille de champignons basidiomycètes parasitant essentiellement les Poaceae (graminées). Les 33 genres constituant cette famille induisent sur leur hôte une pathologie appelée charbon, du fait de la formation de sores remplis de millions de spores noirâtres à paroi épaisse : les téliospores. Une espèce représentative de cette famille, Ustilago maydis, responsable du charbon commun du maïs, s’est imposée au cours des 3 dernières décennies comme un modèle majeur en pathologie végétale pour 3 raisons essentielles : i) l’induction de la physiologie parasitaire, de même que sa morphologie (transition levure-mycélium) sont contrôlées par les gènes de compatibilité sexuelle, ce qui a permis d’en étudier les mécanismes de transduction de signaux ; ii) les nombreux outils moléculaires disponibles (vecteurs de transformation, gènes de sélection, promoteurs inductibles…) en font un modèle de génie génétique ; iii) le génome de cette espèce, mis à disposition de la communauté scientifique en avril 2004, permet désormais d’envisager des études globales s’appuyant sur des ressources génomiques exhaustives. Si cette espèce constitue un formidable outil de laboratoire, les connaissances sur sa biologie restent encore incomplètes. Mon travail de thèse a consisté à établir les capacités d’infection racinaire d’Ustilago maydis en établissant un comparatif avec une espèce très proche phylogénétiquement : Sporisorium reilianum f.sp. zeae. Cette dernière espèce est responsable du charbon des inflorescences du maïs. S. reilianum infecte uniquement via les racines, croît in planta vers le méristème caulinaire apical et sporule lors de la transition florale, après une vie biotrophe endophyte de plusieurs semaines. Dans une première partie de mes travaux, j’ai étudié la capacité de trois espèces d’Ustilaginaceae à produire des souches solopathogènes, souches infectieuses en absence de croisement avec une souche compatible. Il apparaît une corrélation entre la fréquence de production de telles cellules et le mode de dissémination : fréquente chez l’espèce disséminée de façon aérienne et rare chez l’espèce tellurique. La capacité d’Ustilago maydis est intermédiaire, laissant supposer que la voie de pénétration racinaire pourrait intervenir dans l’étiologie de la maladie. J’ai ensuite suivi par différentes approches microscopiques et par PCR les processus de la pénétration racinaire du maïs par Ustilago maydis. Après pénétration, le champignon se développe majoritairement de façon intracellulaire et envahit les racines. Nos analyses de diagnostic PCR montrent que le champignon peut atteindre les tissus végétatifs aériens. La formation de sore n’a toutefois pu être observée sur la variété testée. Ainsi la mise en évidence de la capacité d’Ustilago maydis à pénétrer au niveau racinaire fait de cet agent pathogène, modèle génétique, une espèce de choix pour étudier les mécanismes d’infection racinaire des plantes, particulièrement en comparaison avec des symbiotes racinaires biotrophes. Dans une dernière partie de mon travail, j’ai étudié la perception par les Ustilaginaceae de molécules présentes dans les exsudats racinaires : les strigolactones. Ces composés induisent la respiration cellulaire d’U. maydis et de S. reilianum. Une analyse du transcriptome par construction d’une banque SSH de S. reilianum en réponse à un analogue de strigolactones a révélé une induction rapide de gènes de la respiration et du développement cellulaire. Ces molécules, déjà identifiées pour leur rôle signalétique dans l’interaction plante parasite-plante hôte et dans l’interaction endomycorhizienne, pourraient ainsi avoir une incidence plus large dans l’organisation de la flore rhizosphérique.
Summary
Ustilaginaceae are a family of basidiomycete fungi infecting grass plants (Poaceae). All species of the 33 genera belonging to this family are responsive of a smut disease, due to the presence of sori full of black encysted spores: teliospores. The representative species of this family, Ustilago maydis, the causal agent of the common smut of maize, became since the 3 last decades a major model in plant pathology. The discovery that mating type genes control parasitism induction and morphology (yeast-hypha transition) allowed investigating of cell transduction mechanism. Second, this species is a model in genetic analysis as many molecular tools were developed (transformation vector, selectable marker genes, inducible promoters …). Third its genome is available since April 2004, and it is now possible to perform global genetic studies based on complete genomic resources. Although this species is a powerful tool for laboratory uses, some knowledge on its biology are still missing. My thesis work consisted in evaluating the ability of two phylogenetically related Ustilaginaceae, U. maydis and S.
reilianum f.sp. zeae, to interact with plant roots. S. reilianum f.sp. zeae causes maize head smut. This
species infects maize roots, grows up to the caulinar apex as a biotrophic endophyte during several weeks, then sporulates when floral transition is induced. In a first part, I compared the ability of three species of Ustilaginaceae to produce solopathogen strains –i.e. infectious strains in absence of mating. I observed a correlation between the frequency of formation of solopathogenic form and the dispersal way of each species: from a high frequency for airborne species to low frequency for soil-borne one. U.
maydis present an intermediate behavior, suggesting that root infection could participate in the etiology
of this disease. Root infection process was investigated in a second part by using different microscopic techniques and PCR detection. After penetration, the fungus grows mostly intracellularly and invades root tissue. PCR detection revealed that the fungus could migrate systemically up to the aerial tissue. However we didn’t observe formation of sori in the maize variety used. As conclusion, this result makes
U. maydis as a paradigm to study mechanism of root infection by a biotrophe pathogen. In a third part
of my works, I investigated the perception by U. maydis and S. reilianum of root exuded molecules: strigolactones. These molecules induce the cell respiration on the two species. A transcriptomic SSH-based analysis of a S. reilianum strain in presence of a strigolactone analogue revealed a rapid induction of genes involved in cell respiration and in cell division. These molecules, already described in plant-parasitic weeds and plant-arbuscular mycorrhizal fungi interactions, could be involved in a larger extend in the establishment of the rhizospheric flora.
Préambule
Ce travail constitue la conclusion de quatre années de travaux au laboratoire Surface Cellulaire et Signalisation chez les Végétaux, UMR Université Toulouse III-CNRS, dans l’équipe actuellement nommée Symbiose Endomycorhizienne et Signalisation Cellulaire dirigée par le Pr Guillaume Bécard. Ces travaux s’inscrivent dans le projet d’équipe qui porte actuellement sur l’étude de la perception de molécules impliqués dans les phases précoces d’interaction plante-champignon endomycorhizien, et à moyen terme sur les mécanismes mis en jeu dans de développement biotrophe in planta de ces champignons. Ces champignons étant inaptes à toute manipulation génétique (absence de système de transformation donc aucun promoteur validé ou système de sélection connus ; sexualité inconnue ; ploïdie et taille du génome controversées ; tentative de séquençage du génome en cours depuis 4 ans) une stratégie d’étude consisterait à utiliser un organisme «de validation des hypothèses» permettant l’expression hétérologue de gènes identifiés sur les champignons endomycorhiziens par des techniques transcriptomiques ou génomiques. Les mots clés pour obtenir de tels organismes sont « infection racinaire » et « biotrophie ». C’est dans ce contexte que s’insère désormais ce travail, initié en 2002 après qu’il eut été observé une induction de la respiration cellulaire par les exsudats racinaires sur un champignon du sol, Sporisorium reilianum.
Introduction générale
Le monde des champignons : de l’observation microscopique à l’observation des génomes
Les champignons constituent un phylum à part entière parmi les eucaryotes. Plus de 80000 espèces sont actuellement décrites et nommées (Hawksworth DL. 2001). Comparé au vertigineux travail des entomologistes qui ont proposé près de 1 million de binômes taxonomiques et autant de descriptions, on pourrait croire à la faible diversité du groupe des champignons. Cette image trompeuse peut tenir à la discrétion et la difficulté d’observation des organismes microscopiques. Prenant en compte les espèces collectées mais non décrites, le nombre d’espèces associées au groupe des insectes, les régions du globe qui restent encore à explorer par les mycologues, que ce
soit les zones de forte diversité comme les forêts équatoriales, mais aussi les zones où la curiosité humaine s’est peu exercée comme en milieu marin, certains auteurs ont estimé à plus de 1,5 million le nombre d’espèces fongiques (Hawksworth, 2001). Au rythme actuel des descriptions, de l’ordre de quelques centaines d’espèces nouvelles par an, il faudra quelques milliers d’années pour recenser la diversité des espèces existantes actuellement, et il est probable que nombre d’entre elles auront disparus avec la régression des zones écologiquement fragiles.
La systématique de ce vaste groupe d’organismes présente bien des difficultés. Principales raisons à cela, la rareté des caractères ontogéniques (bien peu de caractères ancestraux et dérivés peuvent être déduits des processus de développement chez les champignons) et la quasi-absence de données paléontologiques. Certes quelques célèbres échantillons reliques sont disponibles, inclus dans l’ambre fossile (Gewin, 2007; Poinar Jr & Buckley, 2007) ou observés dans des tissus végétaux– cas de structure rappelant les structures arbusculaires des actuels champignons endomycorhiziens (Remy et
al., 1994). Mais ces échantillons sont rares, les champignons sont des organismes à l’appareil végétatif
microscopique et se conservant peu. Ainsi ne sont disponibles pour tenter d’établir une classification Figure 1 : L'arbre du vivant.
naturelle des champignons que les seuls caractères morphologiques. Pour aussi perspicaces que furent les mycologues dans leurs descriptions et leurs comparaisons, ils ne pouvaient échapper aux nombreux pièges de la convergence morphologique que leur tendaient ces espèces aux caractères parfois très discrets. L’utilisation des données nucléotidiques dans les phylogénies moléculaires a débuté en milieu des années 70. Pour des raisons techniques, ces premières analyses ont été réalisées sur des ARN ribosomiques, abondants dans la cellule végétale et pouvant être séparés par centrifugation. Ainsi dès 1984 était proposé une première brève révision de la classification des Ustilaginales par comparaison des séquences ARN 5S chez 10 espèces d’Ustilaginaceae, isolées par centrifugation sur chlorure de césium puis séquencées selon la technique de Sanger (Blanz & Gottschalk, 1984). Mais il faut attendre l’avènement de l’amplification génique par PCR en 1986 (Mullis et al., 1986) et surtout dans sa forme actuelle en 1988 (utilisation de DNA polymérase thermostable, Saiki et al., 1988) pour voir ces approches se démocratiser dans les laboratoires. L’application en phylogénie moléculaire est immédiate chez les organismes dont la différenciation sur la base des critères morphologiques était difficile. Le gène, avec l’évolution graduelle de séquence au cours des temps, devient la donnée paléontologique qui faisait défaut. En mycologie, la publication d’un grand choix d’amorces ribosomiques dites universelles (Bruns et al., 1991; Gardes & Bruns, 1993; White et al., 1990) permettra très rapidement la multiplicité des études. Des avancées considérables ont été réalisées ces dernières années sur la classification des champignons. La classification des champignons élaborée depuis deux cents ans a ainsi été profondément remaniée, aussi bien pour certaines grandes lignes (qui prétendrait encore que les Oomycota, ex phycomycètes, sont encore des champignons ?) que dans le détail des différents taxons.
L’interrogation de banques de données génétiques (Genbank, EMBL) au 8/12/2007 révèle que 68166 séquences ribosomiques fongiques de type ITS1-5.8S-ITS2, 67070 séquences 18S et 47792 séquences 28S sont présentes. Il ne faut pas pour autant en conclure que la quasi-totalité des espèces identifiées à ce jour (70 000) ont été analysées au niveau moléculaire, de nombreuses redondances sont présentes, et l’interrogation taxonomique « ne » recense « que » 12155 binômes d'espèces fongiques. Mais les méthodologies et les concepts sont établis et les grandes lignes taxonomiques sont proposées avec une plus grande robustesse des caractères. Le monde des champignons s’ouvre à nous avec une nouvelle cohérence.
Ce recensement de la diversité et des ressources naturelles n’en est qu’à ses débuts que déjà se profile une nouvelle révolution des connaissances. Genbank recense 10 génomes fongiques complets, 65 en cours d’assemblage, et 46 en cours de séquençage, soit 121 génomes fongiques allant de 2,5 Mb pour
Pneumocystis murina à 81.5 Mb pour Puccinia graminis (pour 193 génomes animaux - allant de 104 Mb
pour un nématode à 3800 Mb pour le kangourou Macropus eugenii - et 46 génomes végétaux - allant de 212,5 Mb pour Ostreococcus tauri à 2365 Mb pour le maïs). Si l’on prend en considération que GenBank ne recense pas l’ensemble des génomes disponibles ou en cours de séquençage, on comprend que des
analyses comparatives tant sur la synténie des génomes que sur leur séquence aient déjà commencé (Kuramae et al., 2006). Une étude récente effectuée sur 33 génomes fongiques (Kuramae et al., 2007) a évalué la pertinence de 4852 gènes orthologues pour définir des phylogénies, ainsi que les algorithmes applicables pour de telles analyses multiples. A n’en pas douter, le temps de la métagénomique fongique est arrivé.
A travers ce bref aperçu nous mesurons l’effort consenti en systématique et en génomique fongique depuis 15 ans.
Les types trophiques des champignons
La grande majorité des champignons sont saprotrophes, c'est-à-dire proliférant sur de la matière inerte, approximativement 10% de toutes les espèces connues peuvent causer des maladies de la plante (Hibbett et al., 2007). Qui des saprophytes ou des parasites seraient apparus les premiers ? Il n’est pas certain que cette question se pose en ces termes simplistes, mais elle révèle notre Il n’est pas certain que cette question se pose en ces termes simplistes, mais elle révèle notre méconnaissance actuelle de l’évolution globale des champignons. Des approches de phylogénie globale basée sur la comparaison de plusieurs gènes (James et al., 2006) n’ont pu apporter de réponse formelle. Tout au plus ces auteurs proposent que les premiers basidiomycètes furent parasites. En effet les rouilles (Urédiniomycètes) et charbons (Ustilaginomycètes) sont en position basale de cette branche (figure 2). Rien ne permet de certifier toutefois que l’ancêtre des espèces actuelles de ces groupes fongiques n’était pas saprotrophe. La métagénomique apportera peut être de nouvelles révélations sur ces rapports entre clades, mais ne répondra pas à toutes les interrogations.
Figure 2 : phylogénie des champignons.
Les mécanismes de parasitisme chez les champignons sont diversifiés. Certains de ces champignons sont des parasites obligatoires, associés à leur plante hôte tout au long de leur cycle de vie, tandis que d’autres parasites facultatifs n’infectent les plantes que pour accomplir une partie de leur cycle, généralement la reproduction sexuée.
De nombreux parasites sont des biotrophes. Ce terme dans son acception stricte signifie que le champignon établie avec la plante une zone d’interface. Parmi ces parasites, certains sont des parasites biotrophes obligatoires (les agents des rouilles, de l’oïdium) d’autres des parasites biotrophes facultatifs (les agents des charbons). Une interface membrane à membrane s’effectue entre cellule fongique et cellule végétale. Une régulation fine de la signalisation cellulaire et de l’expression génique, encore à découvrir, permet au champignon de se fournir en nutriments auprès de la cellule végétale. Ce type d’interaction, apparue très tôt dans les interactions plante-champignon avec la mise en place de l’interaction mutualiste endomycorhizienne, a conduit M. Parniske à proposer le néologisme « symbiosome » pour décrire cette zone d’interface (figure 3 ; Parniske, 2000). Quoiqu’il en soit, ce procédé d’interaction semble avoir connu un certain succès dans les différentes branches évolutives des champignons.
Enfin nombreux champignons sont nécrotrophes, tuant les
cellules hôtes grâce à leur batterie de gènes d’agressivité : toxines spécifiques ou non spécifiques, sécrétion d’enzymes lytiques, protéases et inhibiteurs de protéases. Ils peuvent ainsi récupérer les nutriments sur les ruines de la structure végétale.
Cette différence d’interaction entraine des stratégies différentes de contournement des réactions de défense de l’hôte. Les champignons biotrophes obligatoires, tels que les agents des rouilles et les oïdiums, établissent un long contact intime avec leurs plants hôtes, retardant leur mort et limitant les réponses de défense de cellules, du moins durant les phases précoces de l’infection. Le séquençage du génome d’Ustilago maydis vient de révéler la présence de clusters de gènes codant pour de nombreux petits peptides de fonction inconnue (Kamper et al., 2006). La délétion de ces clusters de gènes ne permet plus à l’espèce d’établir une interaction biotrophe avec la cellule hôte et une hypothèse serait que ces peptides ont un rôle de confusion des réactions de défense de la cellule hôte. De leur côté les agents pathogènes nécrotrophes sont moins précautionneux avec les tissus infectés : ils peuvent causer la nécrose avant la pénétration et ainsi croître dans rencontrer de difficultés.
Figure 3 : interaction biotrophes.
(a) Haustoria de Peronospora (Oomycota) dans une cellule foliaire de mésophylle (b) Haustorium de Blumeria graminis dans une cellule foliaire épidermique. (c) Arbuscule de Glomus sp. Dans une cellule racinaire corticale.
Les Ustilaginaceae : des basidiomycètes parasites biotrophes facultatifs
Les Ustilaginaceae constituent une famille de champignons pathogènes responsables de maladie des Poaceae appelées charbon, en raison de la formation de sores remplis de spores noirâtres. Le spectre d’hôte de ces agents pathogènes est toujours très étroit. Cette particularité peut être corrélée à l’interaction biotrophe que toutes ces espèces établissent avec leur hôte. Cette interaction nécessite une « collaboration » physiologique et génétique entre les cellules des deux organismes, et l’on peut admettre que ceci résulte d’une coévolution étroite entre les deux partenaires. Le parasitisme biotrophe sur les Poaceae n’est pas une spécificité des Ustilaginaceae : les agents des rouilles et de l’oïdium par exemple sont des parasites biotrophes majeurs en phytopathologie. Une distinction toutefois est que ces derniers sont des parasites obligatoires, ne pouvant se maintenir que sur les cultures ou sur les plantes adventices. Les Ustilaginaceae sont pour leur part des parasites biotrophes facultatifs, que l’on peut maintenir en culture sur un milieu gélosé riche. Il faut toutefois noter que dans de telles conditions, on peut induire les premières phases de la sexualité (fusion de souches compatibles), mais cette fusion ne se poursuit jamais par la caryogamie et l’induction de la méiose.
Les Ustilaginaceae infectent souvent les pièces florales et l’impact sur la production agronomique peut être élevé (Agrios, 1988). La gestion des tolérances et le traitement des semences ont contribué à limiter fortement l’incidence de ces maladies. Malgré tout, certains de ces agents pathogènes sont très présents sur les cultures. Tel est le cas d’Ustilago maydis, présent dans toutes les zones maïscicoles, induisant des pertes pouvant atteindre sur variétés sensibles 30 % de la production et nécessitant un suivi strict dans le cas de production de maïs semence. Par ailleurs, le développement de pratiques culturales plus respectueuses pour l’environnement, limitant l’utilisation de certaines molécules antifongiques notamment dans le traitement de semence, remet sous les feux de l’actualité phytopathologique plusieurs de ces espèces spécialisées dans l’infection des jeunes plantules en germination.
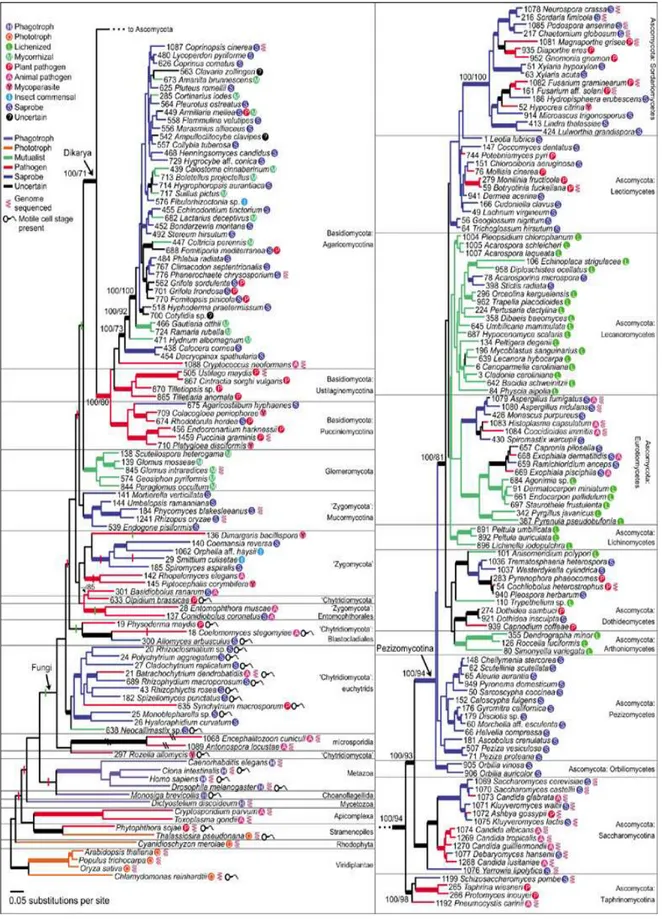
Les Ustilaginaceae au sein des Basidiomycotina
Les basidiomycètes (Basidiomycotina) constituent une division des champignons qui regroupe des espèces caractérisées par des méiospores formées de façon exogène à partir de cellules spécialisées, les basides. Cette division est formée d’environ 22.300 espèces distinctes, soit plus d’un quart des champignons décrits actuellement. Morphologiquement, les Basidiomycotina ont été traditionnellement divisés en Homobasidiomycètes (les champignons à carpophore) et les Heterobasidiomycètes parmi lesquels les téliomycètes (basides formées à partir de spores disséminées : agents responsables des rouilles et des charbons). Mais cette dichotomie a été invalidée par les analyses moléculaires. Désormais, sur la base de caractères morphologiques et moléculaires (gènes ribosomiques
18S, ITS, 28S) trois groupes majeurs sont différenciés : Agaricomycotina, Ustilaginomycotina et Pucciniomycotina (Bauer, 1997; Begerow et al., 1997).
Figure 4 : Phylogénie des Basidiomycètes.
Réalisée par comparaison des gènes ribosomiques LSU + SSU (Bauer et al., 2006)
La classe des Ustilaginomycotina regroupe tout au plus 70 genres, avec environ 1400 espèces. Ce sont tous des parasites des phanérogames. La question de la monophylie des espèces responsables de charbons s’est posée. Les relations phylogénétiques entre ces espèces fongiques traditionnellement
regroupées dans la famille des Ustilaginales (genres Microbotryum, Sphacelotheca, Ustilago, Sporisorium) ont été précisées par différentes analyses par comparaison des gènes ribosomiques (Begerow et al., 1997; Roux et al., 1998). Ces analyses appuient la division des charbons en deux groupes : les Ustilaginaceae s. str. regroupent les espèces responsables des charbons sur inflorescences des Poaceae et des Juncaceae, et les Microbotryaceae regroupent les espèces responsables de charbon sur différentes Dicotylédones (souvent au niveau des étamines), et sont inclus dans les Pucciniomycotina.
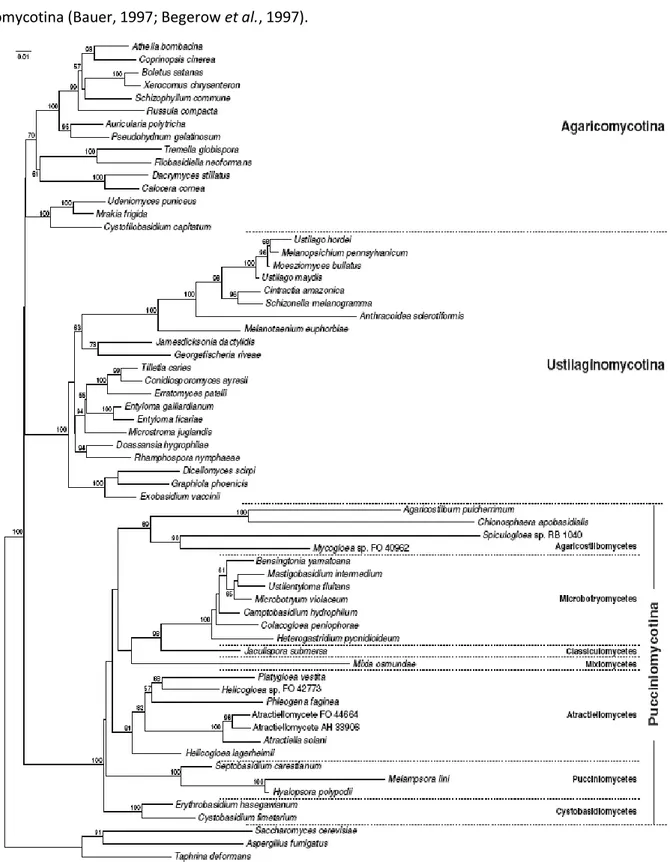
Diversité des Ustilaginaceae
Les Ustilaginaceae constituent une famille dont la taxinomie a été fortement remaniée ces dernières années.
Figure 5 : Phylogénie des Ustilaginaceae.
Réalisée par comparaison des régions ribosomiques ITS. La position relative d’U. maydis et de S. reilianum est surlignée en rouge (Roux, C – non publié)
Cette famille comprend 600 espèces connues à ce jour (Piepenbring, 2003), toutes parasites des Poaceae. Deux genres majeurs constituent actuellement cette famille : Sporisorium et Ustilago. La distinction entre ces deux genres n’est pas clairement établie tant par les caractères moléculaires que par les caractères morphologiques. Les analyses basées au niveau des régions ITS (internal transcribed spacer, ITS1-5.8S-ITS2) révèle que l’espèce Ustilago maydis particulièrement est un taxon intermédiaire entre les Ustilago typiques et les Sporisorium (Stoll et al., 2005; Stoll et al., 2003).
Divergence de Ustilaginaceae et domestication des plantes
Hormis des relevés sur les ancêtres sauvages du maïs, les teosintes Zea sp., Ustilago maydis est une espèce décrite comme uniquement parasite du maïs Zea mays L.. Sporisorium reilianum est une espèce parasite sur maïs mais aussi sur différentes espèces de sorgho. Il a été défini que des populations différentes infectent les 2 plantes, formant ainsi deux formes spéciales : S. reilianum f.sp. zeae parasite du maïs, S. reilianum f.sp. reilianum parasite du sorgho. Frederiksen (Frederiksen, 1977) a montré par des infections croisées que cette distinction est justifiée, hormis pour certaines variétés de maïs doux qui se montrent hôte de S. reilianum f.sp. reilianum. Etant donnée la proximité phylogénétique entre ces deux espèces, certains auteurs ont cherché à définir si cette distinction entre ces deux espèces fongiques et entre formes spéciales de S. reilianum pouvait être concomitante à la différenciation des espèces végétales hôtes (Munkacsi et al., 2007). Au-delà de ces deux espèces, la question porte sur l’incidence de la domestication des espèces végétales cultivées sur la séparation génétique de populations d’agents pathogènes, et donc sur les processus de spéciation.
Munkacsi et al. (2007) ont ainsi choisi quatre espèces responsables de charbon : U. maydis parasite du maïs, S. reilianum parasite du maïs et du sorgho, S. sorghi parasite du sorgho, S.
scitamineum parasite de la canne à sucre. Par une analyse de vitesse de substitution au niveau de
plusieurs gènes fongiques (calculs d’horloge moléculaires), et en prenant pour date de domestication des espèces végétales -10.000 ans, les auteurs concluent à une divergence des espèces fongiques avant le développement de l’agriculture du fait d’une séparation des espèces fongiques datant de plusieurs millions d’années. La domestication des espèces végétales n’est pas à une échelle de temps susceptible d’être à l’origine des divergences actuelles des espèces responsables de charbon des Poaceae, du moins ces isolements génétiques ne sont pas encore perceptibles. Les auteurs analysent ensuite à partir de données historiques sur les civilisations andines la séparation entre formes spéciales de S. reilianum. Le fait que S. reilianum n’était pas référencé sur le maïs avant l’introduction du sorgho au XVIIème siècle dans le Nouveau Monde souligne que l’impact de l’Homme et de la domestication des espèces végétales sur l’apparition de ces formes spéciales tiendrait dans ce cas à l’introduction d’une espèce hôte nouvelle et à une adaptation de souches préexistantes à ce nouvel hôte, et non à une divergence ancienne de cet agent pathogène.
Le cycle des Ustilaginaceae
La sexualité des Ustilaginaceae est à l’origine de l’intérêt porté à ce groupe de champignons. Deux raisons à cela : i) ces champignons se développent in vitro sous une forme levure, facile à manipuler ; ii) ce sont des champignons dimorphiques passant d’une forme levure haploïde saprotrophe à une forme filamenteuse dicaryotique parasite. Ainsi dès le début du XXème siècle (Hanna, 1929; Kniep, 1926), il a été observé la possibilité d’effectuer des croisements intra et interspécifiques qui ont pour effet de générer des structures filamenteuses. Ces travaux, initialement très descriptifs se sont ensuite orientés vers l’héritabilité des facteurs de compatibilité et ont participé à l’élaboration des bases génétiques de la compatibilité fongique par Holliday (Holliday, 1974).
Les Ustilaginaceae sont le plus souvent hétérothalliques, avec un système de compatibilité monogénique-bipolaire (Ustilago hordei) ou digénique-tétrapolaire (Ustilago maydis). Comme la plupart des Basidiomycètes, une longue phase dicaryotique est observée chez les Ustilaginaceae, mais celle-ci n’est observée qu’in planta.
Le parasitisme des Ustilaginaceae comprend généralement trois phases :
1) Une phase saprotrophe où le champignon est présent sous forme levure, peut croître à la surface des tissus ou dans le sol. La fusion de deux souches compatibles génère une souche dicaryotique infectieuse.
2) Une phase biotrophie où le champignon se développe dans les tissus infectés sous forme filamenteuse de façon le plus souvent intracellulaire. Ce développement se caractérise par un comportement biotrophe de l’infection : le champignon traverse les cellules, entouré d’une membrane de biotrophie. Cette phase du parasitisme, qui correspond à une phase de croissance systémique plus ou moins longue selon les espèces, induit une très faible symptomatologie.
3) Une phase nécrotrophe, observée à la fin du cycle de développement lorsque la sporogénèse du champignon est induite.
Cycle typique et variations
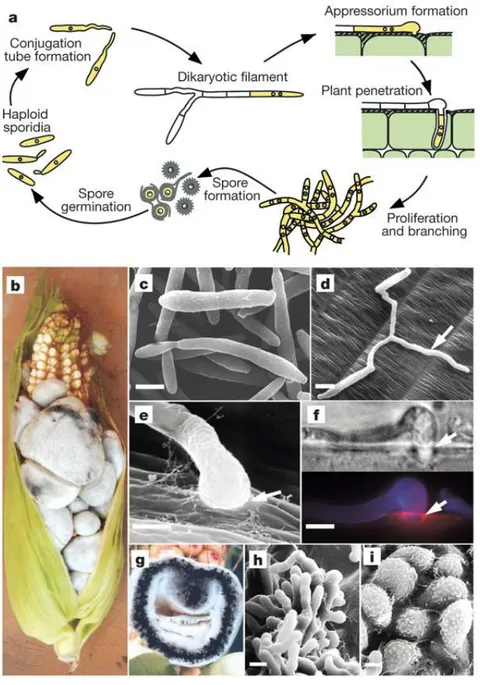
Le cycle de vie typique généralement proposé pour les Ustilaginaceae est le cycle d’Ustilago maydis
Figure 6 : illustration du cycle d’U. maydis (Kamper 2006)
La figure 6a résume ce cycle illustré dans les photographies suivantes. Comme toutes les Ustilaginaceae,
U. maydis produit dans des sores (Fig 6-b, g) des spores enkystées dicaryotiques qui assurent la
dissémination de l’espèce : les téliospores (Fig 6-i). Celles-ci sont disséminées par le vent et restent au niveau du sol. Passée la mauvaise saison, la caryogamie s’effectue au niveau des téliospores, puis une baside se forme, produisant des basidiospores levuriformes haploïdes saprotrophes (Fig6-c). Chaque téliospores produit 4 types de cellules compatibles deux à deux. Lors de la conjugaison cellulaire, les cellules arrêtent transitoirement leur cycle cellulaire en phase G2 (Garcia-Muse et al., 2003), forment des tubes de conjugaison (Snetselaar & Mims, 1992; Spellig et al., 1994) et génèrent un filament dicaryotique (figure 6-d). Ce filament va former une structure de type appressorium, légèrement renflée (fig6-e, f), qui permettra l’infection de l’hôte, le maïs. Seules les parties aériennes semblent être infectées par cette espèce. Cette infection génère la formation d’une galle : sores formés par des
cellules hypertrophiées de l’hôte (déformation des organes) dans lesquels le champignon sporule (fig 6- g, h, i), détruisant les structures végétales.
Plusieurs aspects de ce cycle ont été décrits en détail.
Lors de la formation de l'appressorium un matériel fibreux se produit entre l'appressorium et le paroi de cellule hôte qui pourraient indiquer la production d'adhésines (Snetselaar & Mims, 1993a). Les appressoria constitués par U. maydis ne sont pas les structures très proéminentes mais apparaissent seulement comme des gonflements en bout d’hyphe, ils ne sont pas isolés des hyphes par un septum avant la pénétration, et il n'y a aucune évidence de mélanisation. A ces égards, l'appressorium d’U
maydis diffère sensiblement des appressoria produits par Magnaporthe grisea et Colletotrichum graminicola où l'entrée se produit par la force mécanique par mélanisation de l’appressorium et
création d’une forte pression de turgescence (Bechinger et al., 1999; de Jong et al., 1997).
Plus récemment, en utilisant des souches transformantes GFP, il a été montré que la pénétration peut précéder la formation de l'appressorium. Ceci a laissé suggérer un mode de pénétration principalement basé sur la libération d’enzymes lytiques (Snetselaar & Mims, 1992; Snetselaar & Mims, 1993). La question qui reste à adresser est comment le champignon parvient à arrêter et réorienter sa croissance d'un angle de 90 degrés à ce stade, comment des enzymes de dégradation de la paroi cellulaire végétale sont excrétées avec précision au point d'entrée. Comme par ailleurs il n'a pas été possible de produire jusqu’ici des structures d'infection in vitro, cela indiquerait le besoin de certains signaux de la plante impliqués dans le déclenchement des mécanismes de pénétration, et accessoirement dans la différentiation d’appressorium.
Bien que les parois cellulaires de la plantes hôtes soient traversées laissant passer les hyphes, le plasmalemme de la cellule hôte reste intact et s’invagine autour des hyphes intracellulaires (Snetselaar & Mims, 1992). Plus précisément, il s’agit de pseudo hyphes : seules les parties apicales sont viables. Les parties plus anciennes dégénèrent, se vident de leur cytoplasme, sont deviennent isolées et s'effondrent. Entre 3 et 4 jours après l’infection, les hyphes commencent à brancher et à former des téliospores. Les cellules végétales deviennent alors nécrosées. Les facteurs de contrôle qui déclenchent cette transition, susceptible d'impliquer des divisions mitotiques, sont actuellement inconnus. Ce changement dramatique du mode de croissance coïncide avec le commencement du développement de tumeur autour de 5 jours après l’infection. 9 jours après l’infection, les structures fongiques sont entourés par un mucilage dont l’origine n’est pas encore identifiée (polysaccharides exsudés par le champignon, produits de dégradation du contenu cellulaire).
Sporisorium reilianum f.sp. zeae
Appartenant à la même famille qu’U. maydis et parasitant la même espèce végétale, S. reilianum
la première fois par Kühn en 1875, qui le nomma Ustilago reiliana en hommage au Dr Reil qui le découvrit en Egypte en 1868. Ce champignon a été depuis observé dans toutes les régions du monde. Sa taxonomie actuelle est Sporisorium reilianum (Langdon & Fullerton, 1975).
Son cycle de développement est comparable à celui d’U. maydis. Il convient toutefois d’en souligner les différences.
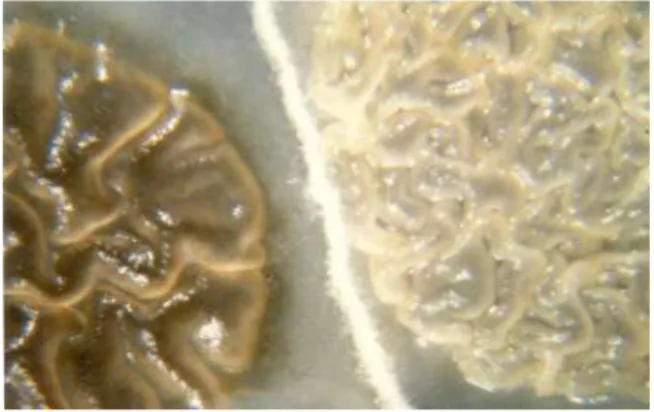
La symptomatologie entre ces deux charbons diffère. D’une part les sores formés par S. reilianum n’apparaissent qu’au niveau des inflorescences, il n’y a jamais de galle sur l’appareil végétatif (d’où le nom donné à la maladie : charbon des inflorescences). Les sores correspondent à la formation d’amas noirâtre initialement entourée d’un périderme d’origine fongique mais formé en dehors des tissus de la plante : il n’y a pas formation de galle. Enfin le sore, formé le plus souvent au niveau de l’épi, correspond à une transformation totale de l’épi où seuls subsistent les faisceaux libéro-ligneux du rachis et les glumes. Il persiste rarement des grains à l’intérieur. Cette disparition totale des grains de l’épi explique que cette pathologie ait une incidence forte sur les rendements : un
champ infecté peut entrainer une perte de 80 % de la production. Dans une moindre mesure, le sore peut se former au niveau de la panicule.
Sur l’appareil végétatif, un nanisme peut être observé chez certaines lignées de maïs sensibles, mais ce symptôme est rare chez les hybrides commercialisés (Christensen, 1963).
Par ailleurs des taches chlorotiques peuvent apparaitre au niveau foliaire, correspondant au développement du champignon dans les tissus (figure 8 ; Martinez et al., 1999).Ce symptôme discret est difficile à observer au champ.
Figure 7 : formation de sores sur panicule (haut) et épi (bas) par S. reilianum (gauche) et U. maydis (droite)
Figure 8 : taches foliaires dues à la présence de S. reilianum dans les feuilles de maïs
S. reilianum est un champignon du sol infectant le maïs uniquement au niveau racinaire (Martinez et al., 2002). De fait l’interaction avec la plante se caractérise par une longue phase biotrophe, c’est à dire un comportement de parasitisme aux effets atténués. De même que pour U.
maydis, le champignon vient puiser ses nutriments au contact des cellules
hôtes, sans pour cela traverser leur plasmalemme et nuire durablement à l’intégrité de ces cellules et de la plante. Cette phase de biotrophie est très longue puisque après infection racinaire au stade plantule, le champignon croît vers le méristème caulinaire où il reste à l’état biotrophe durant toute la durée de croissance végétative du maïs (Martinez et al., 2002). Ce n’est que lors de l’induction florale que le champignon va développer une physiologie nécrotrophe, détruisant les prémices des pièces florales et formant le sore (figure 9). Il s’agit donc d’un cycle hémi-biotrophie, comme
U. maydis, mais fortement décalé dans le temps (quelques jours pour U. maydis, plusieurs semaines pour S. reilianum). Cette caractéristique en fait
un modèle de choix pour l’étude de la biotrophie chez les champignons phytopathogènes. Le cycle complet est illustré dans la figure 10.
Figure 9 : croissance biotrophe (haut) et nécrotrophe (bas) de S. reilianum selon le stade de développement du maïs
Mating-type chez les Ustilaginaceae
Chez les Ustilaginaceae, la reproduction sexuée implique des étapes séparées dans le temps : i) La fusion de cellules haploïdes formant des hyphes dicaryotiques, ii) la caryogamie qui s’effectue dans les téliospores iii) la méiose qui s’effectue lors de la germination de la baside avec migration des 4 noyaux haploïdes, puis sporulation des cellules de la baside.
La fusion cellulaire est donc une étape importante de la sexualité. Elle est gérée par des gènes de compatibilité cellulaire ou mating type (facteurs d’accouplement) : les gènes MAT. La fusion s’effectue entre types cellulaires de compatibilité différente. Il a été mis en évidence par des expériences de croisement (figure 11) qu’un nombre limité de gènes contrôlent la compatibilité. Selon les espèces, un ou deux gènes interviennent avec une variabilité allélique qui diffère selon les espèces et les gènes.
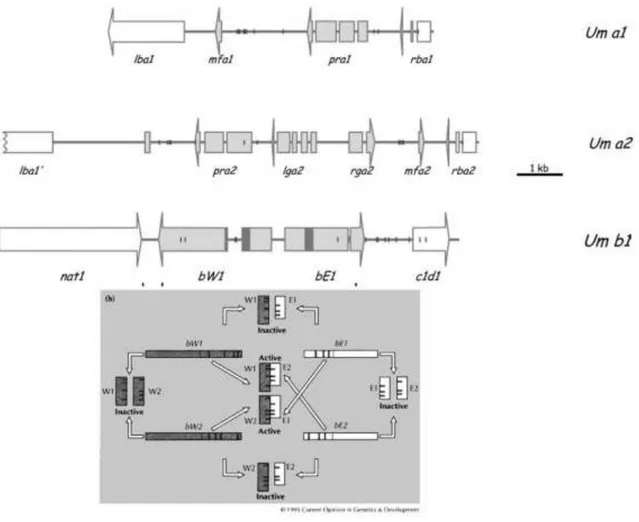
Ainsi chez U. hordei, agent du charbon de l’orge, deux types cellulaires sont formés lors de la germination des téliospores, correspondant à deux allèles d’un même gène (MATA1, MATA2) (bakkeren, 1993). Ce système est appelé bipolaire car une téliospore va générer 2 types de cellules (a1 ou a2), compatibles 2 à 2. Chez U. maydis, deux gènes sont impliqués (MATA et MATB), avec deux allèles pour le gène MATA (MATA1 et MATA2) et plus de 40 allèles pour le gène MATB (Holliday, 1974). Ce système de compatibilité est appelé tétrapolaire car une téliospore va générer 4 types de cellules (a1b1, a1b2, a2b1, a2b2), compatibles 2 à 2. Les études moléculaires menées dans les années 90 ont permis de mieux comprendre ces mécanismes. MAT A et MATB constituent des loci sur lesquels sont présents plusieurs gènes. Les allèles du locus MATA (figure 12) sont idiomorphes (séquences différentes sur le même locus), constitué notamment d’un gène mfa1-2 codant pour une phéromone de type lipopeptide qui sera perçue par un récepteur produit par le gène pra1-2 présent sur le locus complémentaire.
Figure 11 : formation d'une zone de confrontation filamenteuse entre deux colonies levuriformes compatibles
Figure 12 : organisation des loci MATA, MATB et schéma d’interaction des protéines codées par les gènes BE et BW chez U. maydis
La cytoduction est contrôlée par le locus MATA, mais l’induction de la croissance filamenteuse et l’entrée en phase parasitaire nécessite la présence d’allèles MATB différents (Kahmann et al., 2004). Le locus MATB code entre autres pour deux facteurs de transcription avec des homéodomaines très conservés dans leur région N-terminale (exons IIbE et IIbW), mais variables dans leur région C-terminale (exons IbW et IbE). Les protéines bW et bE forment des dimères, mais uniquement par des combinaisons provenant de locus b différents : protéines bW1 et bE2, et entre bW2 et bE1 (Kahmann et
al., 1995). L’hétérodimère ainsi formé à un rôle de facteur de transcription permettant l’expression de
gènes impliqués dans les processus de filamentisation et de pathogénie. L’identification des gènes cibles des facteurs de transcription hétérodimérique a débuté (Brachmann et al., 2001), mais une avancée importante est attendue avec l’utilisation de puces Affymetrix représentatives du génome d’U. maydis (6200 gènes estimés, Kamper et al., 2006). En effet, depuis 2003, le génome de cette espèce a été séquencé et mis à disposition de la communauté scientifique (http://www.broad.mit.edu/annotation/genome/ustilago_maydis/Home.html ). L’objectif est alors de mettre en évidence les réseaux d’expression de gènes. Le défi sera de prolonger ces études aux étapes où le champignon se développe à l'intérieur du tissu végétal.
Ces études sur U. maydis ont permis de définir plus précisément les gènes de mating sur d’autres Ustilaginaceae. Dans un premier temps, il a été défini que des régions les loci a et b hybridaient avec des ADN de nombreuses espèces d’Ustilaginaceae (Bakkeren et al., 1992), y compris les espèces bipolaires. D’autres études ont permis de définir que les espèces bipolaires comme U. hordei présentait bien des loci MATB en plus du locus MATA (Bakkeren & Kronstad, 1993), mais les 2 loci étant liés, ils co-ségrégent pour ne laisser apparaitre que deux types de compatibilité (Anderson et al., 1999). Plus récemment, une étude à permis de décrire le système de compatibilité chez S. reilianum zeae. Cette espèce est tétrapolaire comme U. maydis est l’organisation des gènes est très comparables dans les 2 loci MATA et MATB, a tel point que des complémentations fonctionnelles ont pu être réalisées (Schirawski et al., 2005). Curieusement, l’analyse de souches collectées dans différentes zones du globe a révélé qu’il existait 3 allèles pour le locus A (5 allèles pour le locus B).
Solopathogénie chez les Ustilaginaceae
Certaines souches d’Ustilaginaceae peuvent infecter la plante hôte sans nécessité de croiser avec une souche compatible pour être pathogène, de telles souches sont dites solopathogènes. Il s’agit de souches infectieuses stables (les souches dicaryotiques ou diploïdes étant normalement instables chez les Ustilaginales). Plusieurs types de souches solopathogènes ont été obtenus chez U. maydis ou d’autres Ustilaginaceae.
Les souches solopathogènes naturelles.
Chez les Ustilaginales, la solopathogenie a été observée dans les essais d’isolement à partir de téliospores en germination. Chez Tilletia caries, certaines souches issues d’isolements monospores ne fusionnent avec aucune souches haploïde de cette espèce normalement hétérothallique (Kendrick E.L., 1960, 1975). Chez Ustilago maydis la solopathogénie serait assez fréquente (de l’ordre de 1%,Holliday, 1974). Ces lignées monospores conservent leur capacité parasitaire pendant 30 repiquages, alors que d’autres sont redevenues non pathogènes après quelques mois. De telles souches seraient monocaryotiques (Chilton S.P., 1938, 1943; Christensen J.J., 1931; Eddins A.H., 1929). L’existence des souches solopathogènes a été interprétée par la présence d’un facteur héréditaire dont l’action se manifesterait par des accidents méiotiques (Chilton S.P., 1938, 1943; Christensen J.J., 1931). A vrai dire l’origine et le rôle de telles souches sont totalement méconnus, et hormis des travaux anciens, il n’a guère été porté d’intérêt sur ces souches solopathogènes naturelles.
Souches solopathogènes artificielles.
Parmi les premiers travaux de génétiques réalisés sur U. maydis, l’intérêt s’est porté initialement sur le contrôle de la filamentisation. Suite aux travaux de Holliday (1974), plusieurs publications ont porté sur la recherche de mutants EMS ou UV dérégulés pour leur filamentisation. Flora Banuett et Ira Herskowitz (Banuett & Herskowitz, 1988) ont généré des souches solopathogènes stables par
croisement de souches auxotrophes compatibles obtenues par mutations chimiques sur les souches FBD1 et FBD2 isolées par Holliday. Après croisement et sélection sur milieu carencé, les souches ayant complémenté sont solopathogènes. Ces souches (FBD11, FBD 12) sont toujours utilisées dans les travaux récents pour valider l’expression de gènes, 20 ans après avoir été obtenues.
Souches solopathogènes haploïdes.
La mise en évidence du rôle du locus MATB et des hétérodimères bEx et bWy dans l’induction de
la filamentisation et du parasitisme a entraîné la mise en place de stratégie d’obtention de souches haploïdes infectieuses par transformation. Cette stratégie a consisté à transformer des cellules d’un type de compatibilité donnée avec une séquence codant pour l’allèle complémentaire. Ainsi la souche CL13 par exemple (a1 bE1 bW2) a-t-elle été générée (Bolker et al., 1995). L'expression à un niveau élevé du complexe bE/bW dans des cellules haploïdes transformantes est suffisante pour l'induction de la croissance filamenteuse. Ces souches présentes un grand intérêt dans les études génétiques : l’utilisation de souches haploïdes permet de réaliser les expérimentations de délétion de gènes impliqués dans l’infection ou de complémentation fonctionnelle bien plus aisément que sur des souches diploïdes. Les validations d’infection sont plus rapides à réaliser car ne nécessitent par de croisement préalable. Une limitation est évidement que ces infections ne permettent pas une sporulation, la méiose ne pouvant être induite sur une souche haploïde.
Le maïs, plante modèle en pathologie végétale ?
L’espèce végétale hôte d’Ustilago maydis et de Sporisorium reilianum étant le maïs cette introduction décrit quelques points particuliers de cette espèce et de son interaction avec des agents pathogènes fongiques.
Le maïs
Le maïs (Zea mays L.ssp.mys) est une graminée de la famille des Poceae, sous – famille de Panicoideae. Le maïs, originaire d’Amérique centrale était la culture dominante chez les Incas, les Mayas, les Aztèques ainsi que chez certains peuples du sud-ouest des Etats-Unis. Le maïs constituait la base de l’alimentation de toutes la civilisation précolombienne et à ce titre était vénéré comme l’un des principaux dieux chez les Aztèques et les Mayas. Les premiers maïs découverts dans cette région datent de 7000 ans et seraient le résultat de la domestication d’une variété mutante de téosinte par les civilisations amérindiennes (Kato, 1984). Certains auteurs proposent que l’ancêtre du maïs moderne puisse être une variété de téosinte diploïde (n=10), Zea mays ssp. Parviglumis , originaire des vallées du sud-ouest de Mexico (Matsuoka et al., 2002). Les maïs cultivés de nos jours présents encore 78% de la diversité des mutations silencieuses observées chez Zea mays ssp. Parviglumis (Rafalski & Morgante, 2004). Si les premiers maïs furent importés en Europe au début du XVIème siècle, le développement de cette culture date surtout des années 1950 avec l’importation des premiers hybrides en provenance des
USA (Meyers et al., 2001). Ces hybrides ont de multiples avantages agronomiques, le haut niveau de production, mais aussi une meilleure résistance aux agents pathogènes que leur confère la vigueur hybride.
S’il a un grand intérêt agronomique (encore accru par la volonté politique de transformer une partie de la production en bioéthanol), si de nombreuses pathologies de cette plante sont étudiées, et si la première plante génétiquement modifiée commercialisée est un maïs (maïs Bt), le maïs n’est pas encore un grand modèle génétique pour la phytopathologie. Il possède au moins deux caractéristiques défavorables : l’énorme taille de son génome (2400 Mb – 3000 Mb pour Homo sapiens) et un très fort contenu en séquences répétées (80-85% du génome) impliquant massivement des éléments mobiles. Le mais est considéré par certains comme le plus grand « cimetière » de rétrotransposons qui constituent entre 65 et 70 % de son génome (Meyers et al., 2001). Malgré cela le séquençage de son génomes est en cours et de nombreuses ressources génomiques annotées sont déjà disponibles (http://maize.tigr.org/ , www.maizegdb.org/map.php ).
Résistance du maïs aux charbons
Les charbons du maïs étant des pathologies très présentes, plusieurs travaux ont porté sur l’analyse de la résistance à cette maladie. Un premier point à souligner est qu’il n’a été identifié aucune résistance totale chez le maïs à ces agents pathogènes. Il ne s’agit pas d’une impossibilité intrinsèque liée au mécanisme d’infection très particulier des agents pathogènes responsables de charbon (biotrophes induisant peu de symptômes avant la sporulation), un gène d’avirulence Avrh1 correspondant au gène de résistance Ruh1 ayant été identifié sur Ustilago hordei (Linning et al., 2004). Les recherches n’ont permis à l’heure actuelle que d’identifier des résistances partielles. Des croisements entre des variétés plus ou moins tolérantes à U. maydis montrent que la résistance est polygénique. Selon les génotypes et les souches utilisées, certaines études suggèrent la présence de QTL (Quantitative trait Loci) contribuant fortement à la résistance (Lubberstedt et al., 1999; Parisseaux & Bernardo, 2004), d’autres indiquent la présence de nombreux gènes mineurs (Kerns et al., 1999; Lubberstedt et al., 1998). Par ailleurs, les facteurs environnementaux affectent la résistance à U.maydis. Les résultats obtenus récemment montrent que plusieurs QTLs contribuent à la résistance dans différents tissus du maïs. Ainsi certains loci contribuent préférentiellement à la résistance au niveau des épis et des inflorescences (Baumgarten et al., 2007).
Divers génotypes tolérants à S. reilianum ont été identifiés. Certains des QTL identifiés sont majeurs, et présentent surtout un effet additif, mais ils ne sont que très partiellement communs à ceux définis sur U. maydis (Lu & Brewbaker, 1999; Lubberstedt et al., 1999).
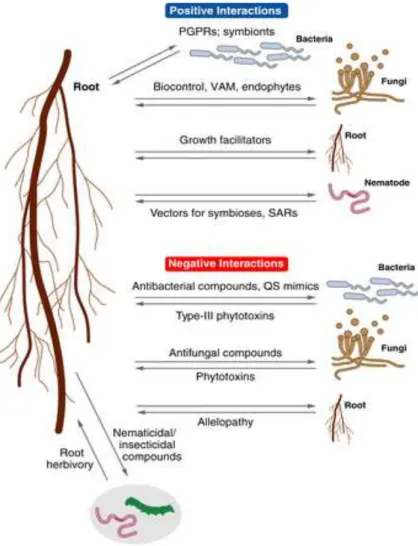
La rhizosphère
Le sol, une matrice entre les organismes du sol et les plantes La rhizosphère constitue une
zone de sol un espace autour des racines des plantes définissant des microrégions du sol en contact direct avec les racines des plantes supérieures. Le sol rhizosphérique est notamment caractérisé par une flore fongique et bactérienne très spécifique (Walker et al., 2003b). A l’origine de cela, les abondants exsudats racinaires émis par les plantes, dont on différencie les rhizo-dépôts -formés de composés organiques issus du métabolisme primaire et exsudés en quantité appréciable (sucres, acides aminés…)- des autres nombreuses molécules issues du métabolisme secondaires, exsudées en quantité souvent infinitésimale. Ces molécules exsudées constituent un vecteur de dialogue
moléculaire entre la plante et les organismes de la rhizosphère (figure 13).
Les exsudats racinaires
les racines de plantes sécrètent continuellement des composés dans le sol (Bais et al., 2001; Gleba et al., 1999). Ces exsudats racinaires inclus la sécrétion d’ions, d'oxygène, d’eau libre, d’enzymes, de mucilage, et de molécules carbonées variées, métabolites primaires et secondaires (Bertin et al., 2003; Uren, 2000). Si une grande partie des métabolites exsudés sont considérés comme des déchets métaboliques, cette vision est plus liée à la méconnaissance du rôle de ces molécules. Pour d’autres composés, leur rôle dans la lubrification ou la défense contre les agents pathogènes a pu être démontrée (Bais et al., 2004; Uren, 2000).
L’exsudation de molécules peut être un mécanisme actif. Les molécules exsudées sont transportées à travers la membrane cellulaire vers l’environnement (la rhizosphère). Des exsudats sont aussi libérés par
Figure 13 : dialogues moléculaires au niveau de la rhizosphère (Biais et al 2006)
des cellules de la coiffe, qui se détachent ensuite mais demeurent viables quelque temps dans l’environnement rhizosphérique.
On différencie souvent deux classes de composés parmi les exsudats racinaires : i) Les molécules en de faible poids moléculaires tels les acides aminés, organiques acides, les sucres, phénoliques, et les autres métaboliques secondaires représentent une grande partie de la diversité des exsudats racinaires ii) les molécules, ou macromolécules de haut poids moléculaire, tels le mucilage (polysaccharides) et les protéines, moins diversifiés mais composant une grande proportion des exsudats racinaires en masse (de l’ordre de 90 % du carbone exsudé chez le maïs). Les exsudats racinaires représentent un coût non négligeable pour la plante en terme de carbone relargué (Marschner, 1995), mais l'importance des photosynthétats sécrétés dans les exsudats de racine est variable selon le type du sol, l'âge, l'état physiologique de la plante et la disponibilité de matière nutritive (Brady & RR., 1999; Brimecombe et al., 2001). Les implications des molécules exsudées dans des processus biologiques ont été définies pour certaines d’entre elles, mais pour beaucoup ce rôle reste à découvrir (Bais et al., 2001; Bais et al., 2003a; Bais et al., 2003b; Kneer et al., 1999).
Rôle des exsudats racinaires dans les étapes précoces des interactions racinaires De nombreux travaux ont porté sur le rôle biologique des métabolites secondaires émis par les plantes (Walker et al., 2003a). Il y a des exemples célèbres d’action de ces métabolites secondaires dans les interactions plantes-microorganismes : implication des flavonoïdes dans l’activation des gènes NOD dans la symbiose Rhizobium/Légumineuse ou encore activation par l’acetosyringone des gènes VIR d’
Agrobacterium tumefaciens. La rhizosphère constitue en fait un milieu dynamique dans lequel
interviennent des microbes, des invertébrés, d’autres plantes qui sont autant d’organismes compétiteurs ou interagissant (Hirsch et al., 2003). En particulier, parmi les centaines de molécules présentes, certaines constituent des signaux chimiques intervenant dans les étapes précoces des interactions entre la plante et les micro-organismes du sol. La signalisation chimique entre les racines et des organismes du sol, y compris les racines des plantes voisines, est souvent basée sur des molécules issues de la racine en très faible quantité. Par ailleurs les mêmes signaux chimiques peuvent provoquer des réponses différentes selon le destinataire, repoussant un organisme et attirant un autre, ou deux organismes très différents peuvent être stimulés avec des conséquences différentes à la plante. Un exemple concret des divers sens pour un signal chimique est la sécrétion des isoflavones par les racines de soja, qui attirent une bactérie symbiotique (Bradyrhizobium japonicum) mais aussi un Oomycète pathogène (Phytophthora sojae ; Morris et al., 1998). Un exemple plus récent constitue le cas de l’effet des strigolactones sur les plantes parasites et sur les champignons endomycorhiziens.