Prédiction des pré-miARN basée sur la conservation de structure dans les pri-miARN
par
Chabane Tibiche
Département d informatique et de recherche opérationnelle Faculté des arts et des sciences
Mémoire présenté à la Faculté des études supérieures en vue de l’obtention du grade de Maître ès sciences (IVI.Sc.)
en informatique
Juillet, 2005
n
Z cX’%
Université
dl1
de Montréal
Direction des biblïothèques
AVIS
L’auteur a autorisé l’Université de Montréal à reproduire et diffuser, en totalité ou en partie, par quelque moyen que ce soit et sur quelque support que ce soit, et exclusivement à des fins non lucratives d’enseignement et de recherche, des copies de ce mémoire ou de cette thèse.
L’auteur et les coauteurs le cas échéant conservent la propriété du droit d’auteur et des droits moraux qui protègent ce document. Ni la thèse ou le mémoire, ni des extraits substantiels de ce document, ne doivent être imprimés ou autrement reproduits sans l’autorisation de l’auteur.
Afin de se conformer à la Loi canadienne sur la protection des renseignements personnels, quelques formulaires secondaires, coordonnées ou signatures intégrées au texte ont pu être enlevés de ce document. Bien que cela ait pu affecter la pagination, il n’y a aucun contenu manquant. NOTICE
The author of this thesis or dissertation has granted a nonexclusive Iicense allowing Université de Montréal to reproduce and publish the document, in part or in whole, and in any format, solely for noncommercial educational and research purposes.
The author and co-authors if applicable retain copyright ownership and moral rights in this document. Neither the whole thesis or dissertation, nor substantial extracts from it, may be printed or otherwise reproduced without the authot’s permission.
In compliance with the Canadian Privacy Act some supporting forms, contact information or signatures may have been removed from the document. While this may affect the document page count, it does flot represent any Ioss of content from the document.
Faculté des études supérieures
Ce mémoire intitulé:
Prédiction des pré-miARN basée sur la conservation de structure dans
les pri-miARN
présenté par: Chahane Tibiche
a été évalué par un jury composé des personnes suivantes: Sylvie Harnel président-rapporteur François Major directeur de recherche Julie Vachon membre du jury
Les niicroARN sont de petites séquences d’ARN d’environ 22 nucléotides. Leur principal rôle est la régulation génétique en bloquant la traduction de certains gènes en protéines. L’objectif de ce travail est d’identifier des séquences génomiques susceptibles d’être porteuses de microARN. Afin d’y arriver, nous nous sommes basé sur les séquences de microARN connus desquels nous avons extrait des ca
ractéristiques structurelles et séquentielles que nous utiliserons pour établir les
différents filtres qui composent notre méthode. Les filtres ont été choisis de manière à tenir compte de la séquence et de la structure. Les caractéristiques retenues sont celles relatives à la composition nucléotidique, l’énergie libre de repliement en
structure secondaire, la différence d’énergie libre de repliement de la séquence na
tive avec des séquences aléatoires de même composition nucléotidique globale et la conservation de la structure secondaire le long du processus de biosynthèse. Une at
tention particulière est accordée à la conservation de la structure secondaire. Cette
dernière nous donne des résultats encourageants vu que nous avons considéré les séquences de manière à tenir compte de la composition nucléotidique aux voisinages immédiats des séquences porteuses de microARN ce que les travaux antérieurs ne considéraient pas. Au regard des résultats obtenus, cette caractéristique possède un
important potentiel de discrimination et mérite une exploration plus approfondie.
Mots Clés : Micro-ARN, Régulation génétique, ARN non codant, interférence
MicroRNAs are short endogenous sequences of about 22 nucleotides that are used to target messenger RNA for transiational repression. This work is intended to
predict genomic sequences of 65 ntds that may be putative microARN predursors.
To do so, based on the known miRNA precursor sequences we derived some stru
cutural and sequencial features. These features are then used to buiÏd a prediction
pipeline. The main carateristics considered are the secondary structure free folding
energy, the differential free folding energy which is the difference between the free folding energy of the wild sequence ami the mean of the free folding energies of the
sequeces resulting from the shuffling of the wild sequence, the nucleic composition
and the conservation of the secondary structure of the predursor. A great atteil tion is paid for the conservation of the secondary structure and the resuits show
that it is worth to be considered since almost ail of the known pre-miRNA have
their structures well conserved. This lias neyer been considered in previous miRNA
prediction works eventhought it takes into account the nucleic composition in the neiglihorhood of the candidate pre-miRNA which influences the resulting secondary
structure of the candidate precursor.
Keywords: Micro-RNA prediction, Gene regulation, post-transcriptional regula
RÉSUMÉ
ABSTRACT iv
TABLE DES MATIÈRES y
LISTE DES TABLEAUX viii
LISTE DES FIGURES ix
NOTATION x
DÉDICACE xi
REMERCIEMENTS xii
CHAPITRE 1: INTRODUCTION 1
CHAPITRE 2: ÉTAT DE L’ART SUR LES ACIDES NUCLÉIQUES Z
2.1 Introduction 7
2.2 Les acides désoxyribonucléiques (ADN) 8
2.3 Les acides ribonucléiques (ARN) 9
2.3.1 Les ARN codants 10
2.3.2 Les ARN non-codants 12
2.3.3 Structures des acides ribonucléiques 14
CHAPITRE 3: MICROARN : BIOSYNTHÈSE ET MODALITÉS
3.1 Introduction 16
3.2 Biosynthèse des microARN 18
3.2.1 Localisation 19 3.2.2 Transcription 20 3.2.3 Éboutage 22 3.2.4 Exportation 26 3.2.5 Maturation 27 3.3 Modalité d’action 29
3.3.1 Formation du complexe actif 29
3.3.2 Ciblage des ARN messagers 32
3.4 Conclusion 34
CHAPITRE 4: LE PROBLÈME ET LES TRAVAUX ANTÉRIEURS 35
4.1 Introduction 35
4.2 Définition du problème 35
4.3 Survol des travaux antérieurs 37
4.3.1 Chez les métazoaires 37
4.3.2 Chez les plantes 39
CHAPITRE 5: PRÉSENTATION DE LA MÉTHODE DE PRÉDICTION
DES PRÉ-MI-ARN 42
5.1 Introduction 42
5.2 Bases de données 43
5.3 Méthode et application 45
5.3.1 Énergie libre de repliement 47
5.3.2 Composition nucléotidique des séquences 49
5.3.4 Conservation de structure 55
5.4 Validation 61
5.5 En définitive 61
CHAPITRE 6: RÉSULTATS ET DISCUSSION 63
6.1 Résultats 63
6.2 Discussion 65
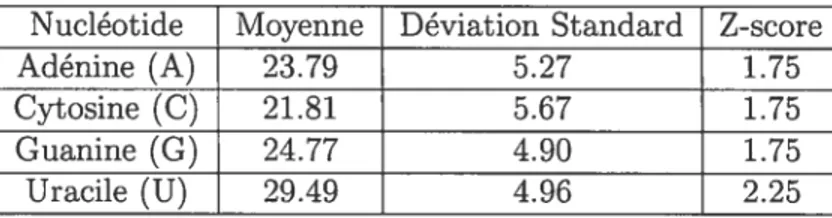
5.1 Z-score limite pour chacun des nucléotides A, C, G et U 52 6.1 Liste des 50 séquences présentant les meilleurs scores 65
2.1 Constituants des acides nucléiques 9
2.2 Structure secondaire d’une séquence d’ARN 15
3.1 L’ARN polymérase III Drosha 24
3.2 Sites de clivage de Drosha 24
3.3 Distribution des nucléotides le long des pré-miARN 25
3.4 L’ARN polymérase III Dicer 29
5.1 Séquences de pré-m1ARN 44
5.2 Homogénéisation des pré-miARN 45
5.3 Distribution le l’énergie libre de repliement des pré-miARN 48 5.4 Taux de filtrage des pré-miARN par le filtre de l’énergie 49 5.5 Distribution des compositions nucléiques globales des pré-miARN . 50 5.6 Taux de filtrage des pré-miARN en utilisant les filtres nucléiques 52 5.7 Influence de la longueur de la fenêtre sur le signal de spf 54 5.8 Taux de filtrage des pré-miARN selon le filtre de la différence d’énergie 56 5.9 Distribution de la conservation des pré-miARN 59 5.10 Taux de filtrage par conservation de la structure 62
A : Adélline
ADN $ Acide DésoxyriboNucléique
ARN Acide RiboNucléique C $ Cytosine
G $ Guanine
T : Thymine
U $ Uracile
UTR $ UnTranslated Region
RISC : RNA Induced Silencing Complex RLC : RISC Loading Complex
À
mes deux enfants, lanis et Rayan, qui ont in sisté pour être partie prenanti en me rappelant que la seule façon de décompresser c’est de jouer.Je tiens, avant tout, à remercier mon directeur, Dr. François Major, pour m’avoir accepté dans son laboratoire et pour son aide, ses orientations et conseils qui m’ont grandement aidés pour réaliser ce travail. Mes remerciements à tous les membres du LBIT qui m’ont facilité la tâche en répondant à mes nombreuses questions.
INTRODUCTION
Pendant longtemps, la biologie moléculaire faisait du caractère unidirectionnel de l’information génétique son dogme central soit l’ADN fait l’ARN et l’ARN fait la protéine. Le processus de synthèse va de l’ADN, support physique et stable de cette information, donc des gènes, vers les protéines, entités moléculaires a.ssu rant les réactions enzymatiques et autres fonctions régulatrices et/ou structurales indispensables à la vie de la cellule et donc des organismes vivants. Dans ce para digme, les différents types d’acides ribonucléiques soient ARN messager (ARNm), l’ARN ribosomal (ARNr) et l’ARN de transfert (ARNt) sont perçus comme de simples intermédiaires impliqués dans le transport et le décodage de cette informa tion génétique afin de produire des protéines.
Cette vision, centrée autour d’un rôle prépondérant des protéines, est en train de changer suite notamment à l’identification, il y une quinzaine d’années, d’un nombre croissant de gènes dits non codants [LFA93, CVO1]. En effet, de tels gènes ne génèrent pas de l’ARN conventionnel mais plutôt de petits ARN, appelés mi croARN. Ces derniers, de tailles variables, assurent des fonctions cellulaires d’une extrême importance, notamment celles reliées au développement de certains orga nismes, sans pour autant être traduits en protéines [BHSO3,MLA97].
Le processus régissant la biosynthèse et le mode d’action de ces petits ARN, dont le recensement ne fait que débuter, s’avère être très minutieux et complexe [BarO4]. En ne regardant que les étapes inhérentes à ce processus, on peut être tenté de dire
que cela semble être simple. Toutefois, les aspects temporels et fonctionnels sont loin de l’être. Pour preuve, plus d’un millier de microARN ont été identifiés [EJO4] et de ce nombre seule une infime partie a sa fonction et son expression tempo relle connues. Cette difficulté s’explique, entre autres, par l’incapacité des procédés biochimiques actuels à prendre en charge des expériences à l’échelle génomique à laquelle s’ajoute la nouveauté du phénomène de régulation génétique par des petits ARN endogènes [CVOÏ].
Les premiers balbutiements de la régulation génétique par l’ARN endogène re montent aux débuts des années 90 quand Arsu et ses collègues ont démontré, pour la première fois en 1991 dans le ver Caenorhabditis elegans, l’implication du gène lin-4 dans la répression du gène lin-14 [AWR91]. Il a été constaté, quelques années auparavant, que le gène lin-14 est impliqué dans le développement du ver dans ses premiers stades larvaires [AH8Z].
En 1993, Ambros et ses collègues réussissent à isoler le gène lin-14 [LFA93] d’une longueur d’environ 22 nucléotides à son stade final de biosynthèse. La mutation ou la suppression des régions des gènes cibles, lin-14 et lin-28, complémentaires au miARN lin-4 mène à la perte de la fonction de régulation. Cette perte de la fonc tion de régulation se traduit par une stagnation du développement au même stade larvaire alors que la larve est sensée passer à un stade supérieur {0A99, WHR93, LFA93]. Ces études suggèrent que le gène lin-4 réprime les gènes lin-14 et lin-28 en se liant, avec une certaine imperfection, à des régions localisées dans les régions non traduites du côté 3’ ou 3’UTR (3’ UnTransÏated Regions) de ces gènes. Les ARN messagers réprimés ne perdent pas leur stabilité
[0A991
ce qui veut dire que la répression se fait par arrêt de la traduction et non par la destruction des ARNmcibles comme il a été déjà observé dans l’action des petits ARN exogènes [FXM98].
L’existence du petit ARN lin-4 dans le C. elegans était, au départ, perçue comme une caractéristique propre à ce ver. Cette perception a radicalement changé avec la découverte d’un autre gène de même nature, soit le gène let-Z
[PRS+OO].
Contrai rement à lin-4, let-Z n’est pas propre au C. elegans mais se retrouve chez plusieurs autres espèces y compris l’humain, ce qui suggère que ce type de régulation est com mun à un certain nombre d’organismes vivants. Cette suggestion a été confirmée par d’autres études qui ont révélé, plus tard, que ce phénomène est assez généralisé et qu’il est assuré par plusieurs petits ARN [LQRLTO1, LAO1, LLWBO1] connus alors sous le nom de petits ARN temporels conséquemment à leurs tailles et modes d’expression mais qu’on appelle maintenant microARN ou miARN. Beaucoup de ces miARN sont conservés à travers le temps [0U05, T$04j et les études confirment que la régulation génétique par de petits ARN endogènes est commune à plusieurs espèces vivantes.Les plantes ne sont pas en reste, d’autres miARN y seront découverts [LKRCO2, MvdWMMO2, JonO2, RWRO2]. Si les étapes de la biosynthèse des miARN chez les plantes sont semblables à ce qui a été observé chez les animaux, le mode d’action, lui, est différent. En effet, chez les plantes une complémentarité parfaite ou presque entre le miARN et sa région cible dans l’ARNm est nécessaire pour qu’il y ait cata lyse [MWO5, HWR96]. Cette catalyse se solde par la destruction de l’ARNm ce qui n’est pas le cas chez les animaux. Chez ces derniers, une complémentarité imparfaite particulière est suffisante pour arrêter la traduction alors qu’une complémentarité parfaite mène à la dégradation de la cible.
Les miARN ont été initialement associés au développement de certains orga nismes vivants [MLA9Z,WHR93,FA99, 0A991 mais d’autres fonctions s’y ajouteront
au fur et à mesure que les recherches avancent. Parmi ces fonctions, on retrouve la prolifération cellulaire [XGHO4, XVGHO3, BH$O3]. Ces nouvelles fonctions sont d’une importance capitale vu qu’elles peilvent être en relation avec certaines ma ladies tel que le cancer. Des signaux confirmant cette hypothèse ont commencé à apparaître il y trois années environ. Dans une première étude allant dans ce sens, Calin et ses collègues ont identifié un lien entre les miARN et la leucémie [CSDO4]. En effet, le gène codant pour les miARN miR-15a et miR-16a est inexistant dans les cellules atteintes de leucémie. Ces résultats suggèrent que la leucémie est peut-être due à l’absence de ce gène et donc des miRNA régulateurs qu’il contient. La ou les ARNm cibles de ces deux miARN ne sont pas encore identifiés mais il est fort pro bable que cette ou ces cibles soient impliquées dans la maladie. Dans une autre étude plus exhaustive, Calin et ses collègues sont arrivés à la conclusion que les miARN sont issus, de façon générale, des régions fragiles du génome notamment celles iden tifiées comme étant reliées au cancer [C$D+04]. Des résultats d’autres études vont de même sens [LGMO5,TKYO4,KTOO5]. Les miARN sont aussi impliqués dans le système de défense des cellules en ciblant certains rétrovirus [LDAO5].
Les miARN sont vulnérables. Dans une étude récente [LCO4], Lu et Cullen ont montré que les miARN peuvent être la cible de certains adénovirus. Ils ont constaté que l’adénovirus VAl agit sur l’exportation des pré-miARN vers le cytoplasme ainsi que sur l’activité de l’enzyme responsable de la maturation des miARN.
La compréhension plus approfondie de la régulation post-transcriptionnelle et de son niveau d’implication dans la vie des organismes vivants passent par l’identi
fication des miARN, de leurs cibles ainsi que le degré d’implication de ces dernières dans la vie cellulaire. Aucune de ces trois tâches n’est aisée, toutefois, toute avancée, aussi petite soit elle, peut être d’une grande importance. C’est dans ce cadre que s’inscrit ce travail dans lequel nous essayons d’explorer d’autres avenues pour pou voir identifier de nouveaux gènes de miARN.
La principale caractéristique bien intégrée dans les différents outils de prédiction de pré-miARN est la structure secondaire en forme d’épingle à cheveux com mune à tous les pré-miARN déjà identifiés [EJO4]. Cette structure est un motif structural duquel on ne peut pas se passer. La principale raison est que c’est justement cette structure qui fait que le transcrit primaire est reconnu et cata lysé [LAHO3,HLYO4,ZYCO5]. Ce motif est aussi important pour l’agent d’expor tation qui exporte le pré-miARN du noyau vers le cytoplasme [ZCO4]. La seconde raison est l’absence d’autres signaux perceptibles propres aux pri-miARN ou aux pré-miARN ou encore aux miARN matures en mesure de nous aider à classer les candidats.
La structure de tige-boucle, même commune à tous les pré-miARN, n’est pas capable à elle seule de filtrer les bons candidats. Il est donc impératif de la jumeler à d’autres critères et c’est que nous avons fait dans ce travail. La méthode que nous proposons consiste, par une approche statistique, à attribuer un score aux
séquences analysées. Ce score tient compte de la composition nucléotidique, de l’énergie libre de repliement, de la différence d’énergie libre de repliement de la séquence à analyser et du degré de conservation de la structure secondaire de cette dernière dans la structure secondaire d’une séquence de 500 nucléotides. Pour chacune de ses caractéristiques un score limite est calculé en se basant sur les pré miARN connus. Le calcul de ses scores limites s’est fait à partir des précurseurs des
miARN connus. Les séquences qui n’auront pas surpassé un de ces scores limites seront rejetées. La nouveauté dans cette méthode est la prise en compte du transcrit primaire du miARN. Le transcrit primaire est simulé en prenant une séquence de 500 nucléotides ayant en son milieu le précurseur. Le repliement de cette longue séquence nous donne une multitude de structures sous-optimales et c’est à travers ces structures que la conservation de la structure du précurseur est recherchée. Cette methode nous donne une liste de séquences classées de la plus probable à la moins probable et nous estimons que les séquences ayant les meilleurs scores représentent sont des candidats miARN potentiels.
Pour faciliter la compréhension de ce travail, nous avons jugé bon d’expliquer ce qui gravite autour de la régulation génétique. Dans le premier chapitre de mémoire, nous nous sommes penchés sur la biologie cellulaire. Nous avons introduit les no tions de base nécessaires à la compréhension de la biosynthèse des miARN. Dans le second chapitre, nous avons fait une revue, assez détaillée, de ce qui a été publié en relation avec la biosynthèse et le mode d’action des miARN. Ces connaissances sont nécessaires pour bien situer ce travail. Dans le 3ème chapitre, nous survolons les travaux qui ont été déjà réalisés dans le domaine de la prédiction des pré-miARN. Le 4ème chapitre est consacré à l’explication de la méthode et des différents pa ramètres que nous avons utilisés. Le dernier chapitre résume les calculs effectués, les résultats obtenus et leur analyse et nous finissons ce mémoire par une conclusion.
ÉTAT DE L’ART SUR LES ACIDES NUCLÉIQUES
2.1 Introduction
Les acides nucléiques ont été à l’origine isolés dans le noyau cellulaire et sont communs à tous les organismes vivants. Ils existent sous deux formes les acides désoxyribonucléiques (ADN) et les acides ribonucléiques (ARN). L’ADN est une macromolécule biologique qui se trouve dans le noyau cellulaire. Elle est formée par des millions, voir des milliards, de nucléotides selon les espèces. Les nucléotides sont des monomères composés essentiellement d’un acide phosphorique, d’un sucre et d’une base azotée [LewO4] (voir figure 2.1).
Les nucléotides sont au nombre de cinq et forment deux classes selon la base azotée qu’ils contiennent. La classe des nucléotides à bases pyrimidiques composée de la Cytosine (C), la Thymine (T) et l’Uracile (U) et la classe des nucléotides à bases puriques composée de l’Adénine (A) et de la Guanine (G) [EpsO3j. Contrai rement aux autres nucléotides, l’Uracile ne se trouve que dans les ARN à la place de la Thymine qui, elle, ne se trouve que dans l’ADN. Les nucléotides sont des molécules polarisées et donc en mesure de former des liaisons hydrogènes lorsqu’ils sont mis en contact. L’Adénine et l’Uracile dans le cas de l’ARN, ou la Thymine dans le cas de l’ADN, se lient par deux liaisons hydrogènes. La Cytosine et la Gua nine s’apparient par trois liaisons hydrogènes {LewO4}. Les nucléotides en mesure de s’apparier sont dits complémentaires.
nucléiques appelées brin d’ARN ou d’ADN . Ces liaisons, appelées ponts phospho
diesters, se produisent entre l’acide phosphorique du nucléotide i+1 et un des car bones du sucre du nucléotide i.
À
cause de la polarité de ses constituants, les brins polynucléotidiques d’ADN s’associent, selon les règles énoncées plus haut, pour for mer de l’ADN double brin. L’ADN double brin, tel qu’il est trouvé dans la cellule de la majorité des espèces, est formé de deux brins parfaitement complémentaires et antiparallèles appelés brin sens et brin antisens. Le sens de lecture de l’ADN est de l’extrémité 5’ vers l’extrémité 3’. L’appellation 5’ et 3’ des extrémités d’une séquence d’acides nucléiques est due à une convention. Par cette convention, les cinq carbones du sucre sont indexés de 1’ à 5’ dans le sens des aiguilles d’une montre (voir figure 2.1). De cette numérotation est issue l’appellation des extrémités de la séquence d’ARN ou d’ADN. L’extrémité 5’ se trouve du côté du carbone 5’ du sucre et l’extrémité 3’ du côté du carbone 3’. L’ADN double brin adopte une conformation spatiale hélicoïdale [WC53].2.2 Les acides désoxyribonucléiques (ADN)
Hormis quelques virus pour lesquels l’ARN est le support génétique, l’ADN est le dépositaire de l’information génétique de presque toutes les espèces vivantes. Cette information génétique est nécessaire àtout organisme vivant du plus simple au plus complexe. Elle préserve la lignée des espèces comme elle constitue un moule pour la production de protéines et autres molécules dont les cellules vivantes ont be soin. On parle d’information génétique car les génomes, constitués à base d’ADN, ne jouent aucun rôle actif dans la formation des organismes vivants mais four nissent plutôt des copies des séquences nucléotidiques nécessaires à la synthèse des protéines et autres enzymes. Chaque cellule de l’organisme est en possession de tous les gènes [LewO4]. Cet aspect d’équité est assuré par la réplication de l’ADN. La
NU O O NU2 H II /C\ \ /C\
\
/C\ /CI /C\ N N C N C N C N C I II I II I II II C /C \ /C /C\ /C C\ /C /C\ /C/
O N O N N NU2 N N I I I ICytosine X Uracile X Thymine X Adenine X
/C\ /C
/
I
II
NH2 N N O-5H o Guanine Phosphate Sucre Hr,
I2H OH OH NucleofideFigure 2.1 — Constituants des acides nucléiques.
réplication est un processus par lequel l’ADN est dupliqué dans la cellule parentale avant d’être transmise aux cellules filles lors la division cellulaire. La réplication de l’ADN n’est pas parfaite, des mutations se produisent parfois.
Physiquement, L’ADN peut être vu comme un ensemble de macromolécules, appelées chromosomes et, fonctionnellement, il peut être vu comme un ensemble d’unités fonctionnelles appelées gènes. Ces macromolécules se trouvent dans le noyau cellulaire. Chez l’humain, le génome est composé de 22 paires d’autosomes et de 2 chromosomes sexuels.
2.3 Les acides ribonucléiques (ARN)
L’ARN est une molécule polynucléotidiques mono-brin. Contrairement à l’ADN qui n’est qu’un support d’information, l’ARN est destiné à la formation d’unités actives
ou de complexes actifs formés par l’association de plusieurs de ces unités. Certains ARN, dits ARN codants, subissent des transformations avant d’acquérir la capacité d’être actifs tel que l’ARN messager qui est préalablement traduit en protéine. D’autres sont intégrés en tant que chaînes nucléotidiques dans des complexes tels que l’ARN ribosomal, l’ARN de transfert et d’autres petits ARN et sont appelés ARN non-codants.
2.3.1 Les ARN codants
Les ARN codants sont formés d’un seul type d’ARN soit l’ARN messager. Comme son nom l’indique, cet ARN a pour mission de transporter l’information nécessaire à la synthèse de protéines du noyau vers le cytoplasme ou la machinerie dédiée à la traduction y est domiciliée. Les transcrits primaires sont issus de l’ADN par un processus appelé transcription. Ces derniers sont épissés dans le noyau puis exportés vers le cytoplasme où ils seront traduits. Ci-dessous une description sommaire des étapes de biosynthèse des protéines.
2.3.1.1 Transcription
La transcription est un processus qui consiste à produire une copie d’une région de l’ADN. Seules certaines portions de l’ADN sont transcrites et elles sont ap pelées gènes. Les portions à transcrire présentent des signaux en amont, appelés promoteurs, et en aval qui signalent, respectivement, le début et la fin de la portion à transcrire [Rot93]. La duplication de la région à transcrire est réalisée par une enzyme appelée une polymérase d’ARN. La transcription passe par trois étapes appelées initiation, élongation et terminaison [LewO4l.
1. Lors de l’initiation, le complexe enzymatique responsable de la transcription écarte les deux brins de l’ADN et via des protéines appelées facteurs de
transcription, reconnaît le promoteur et s’y fixe.
2. Lors de l’élongation, la polymérase d’ARN forme le brin d’ARN messager en appariant les nucléotides complémentaires au brin d’ADN à transcrire. Ces nucléotides se trouvent dans le nucléoplasme. Le double brin d’ADN se referme après le passage du complexe.
3. L’élongation s’arrête à la rencontre du signal de terminaison. L’ARN messager est libéré et les brins de l’ADN se referment.
Une modification est apportée aux transcrits originaux soit l’ajout d’une coiffe à l’extrémité 5’ et d’une séquence polyadenine à l’extrémité 3’ [EpsO3]. Ces ajouts préviennent la dégradation du pré-ARNm par les exonuléases.
2.3.1.2 Epissage
Les transcrits primaires ne codent pas dans leur totalité pour des protéines. Ils contiennent des régions codantes, appelées exons, et des régions non-codantes, ap pelées introns, parsemées le long de la séquence [LewO4]. Ils contiennent aussi d’autres régions non-codantes appelées régions non-traduites aux extrémités 5’ (5’UTR UnTranstated Regioris) et 3’ (3’UTR). Lors de l’épissage, les introns sont excisés et les exons recollés entre eux. Cette tâche est réalisée par des molécules appelées ribonucléoprotéines nucléaires. Les introns possèdent des signaux que le complexe d’épissage reconnaît. L’excision doit se faire à des endroits très précis et toute erreur fera perdre à l’ARNm final son sens. Une fois que l’épissage est réalisé, la séquence résultante est exportée du noyau vers le cytoplasme [Rot93].
2.3.1.3 Traduction
Cette étape consiste à traduire la séquence nucléique en une séquence polypep tidique. Cette opération est réalisée par une machinerie composée d’ARN ribo
somaux, d’ARN de transfert et de quelques protéines. La traduction se fait par groupes de trois nucléotides appelés codons. En dehors des codons d’arrêt qui sont des signaux, chaque codon est traduit en un acide aminé selon un code universel. Comme il n’existe que 20 acides aminés naturels et qu’avec quatre nucléotides on peut former 43 soit 64 combinaisons de trois nucléotides, chaque acide aminé peut provenir de plus d’un codon. La traduction passe par trois étapes
1. La première étape, appelée initiation, consiste à mettre en place le complexe traducteur. Cette mise en place fait intervenir certaines protéines appelées facteurs protéiques d’initiation. Ces facteurs reconnaissent le codon initiateur qui est presque toujours AUG.
2. La seconde étape consiste à lire les codons, à apporter les acides aminés correspondant et à les fixer à la chaîne polypeptidique en formation. L’apport des acides aminé est du ressort de l’ARN de transfert.
3.
À
la rencontre d’un des codons stop, la traduction s’arrête et la protéine est libérée.Contrairement a codon initiateur qui code pour une Méthionine, les codons d’arrêt ne codent pour aucun acide aminé. Lors de la traduction, l’ARNm n’est pas détruit et peut donc servir à la synthèse d’une ou de plusieurs autres protéines.
2.3.2 Les ARN non-codants
Les ARN non-codants, comme le nom l’indique, ne codent pas pour des protéines mais sont plutôt intégrés en tant que chaînes nucléotidiques dans des complexes actifs. Les premiers à être caractérisés sont l’ARN ribosomal et l’ARN de transfert.
2.3.2.1 ARN ribosomal et ARN de transfert
L’ARN ribosomal ainsi que l’ARN de transfert sont, eux aussi, transcrits à par tir de l’ADN dans le noyau cellulaire. Ils sont ensuite exportés vers le cytoplasme. L’ARN ribosomal, avec un certain type de protéines appelées protéines ribosomales forment le ribosome. Le ribosome représente l’ullité de traduction des ARN mes sagers en protéines.
Les ARN de transfert, d’une centaine de nucléotides environ, sont eux aussi impliqués dans le processus de traduction. Leur rôle est de reconnaître le codon en instance de lecture et d’apporter l’acide aminé correspondant vers la chaîne poly peptidique. Les ARNt présentent une structure spatiale en forme de trèfle [Rot93]. Les ARNt possèdent deux sites d’une importance capitale, soient l’extrémité 3’ et l’anticodon situé sur une des boucles. L’extrémité 3’ présente trois nucléotides ca ractéristiques soient CCA-3’-OH [Rot93]. C’est sur ce site que se fixe l’acide aminé qui sera présenté au ribosome. L’anticodon doit s’apparier parfaitement avec le codon lu de l’ARN messager. L’appariement se fait par des liaisons hydrogènes et les triplets sont disposés de manière antiparallèle. Le code génétique de l’acide aminé que l’ARNt fixe à son extrémité 3’ correspond au codon de l’ARN messager complémentaire à l’anticodon de cet ARNt.
2.3.2.2 Les petits ARN non-codants
On retrouve aussi d’autres petits ARN non-codants dans le noyau et le cytoplasme cellulaires. Certains de ces petits ARN ont été caractérisés, parmi lesquels on re trouve les microARN. Contrairement aux ARN précédents qui interviennent posi tivement dans le processus de synthèse des protéines, les microARN sont impliqués dans la répression de la traduction des ARNm qui est aussi connue comme la
régulation de l’expression génétique [LFA93]. D’autres outils sont connus pour être des régulateurs de l’expression génétique tels les promoteurs qui indiquent à l’ARN polymérase la région de l’ADN à transcrire [LewO4]. Les promoteurs interviennent dans le noyau pour empêcher la transcription d’une région de l’ADN alors que les microARN interviennent dans le cytoplasme pour arrêter la traduction.
La synthèse des microARN ressemble à celle des autres ARN. La transcription se fait à partir de l’ADN en des séquences de quelques centaines ou quelques mil liers de nucléotides. Cette longue séquence nucléotidique et ensuite excisée pour ne laisser qu’une séquence d’environ 80 nucléotides de long structurée en épingle à cheveux. La prochaine étape est de l’exporter vers le cytoplasme où elle sera clivée une seconde fois pour donner naissance à un duplex d’environ 22 paires de bases. Ce duplex sera ensuite défait et un des deux brins sera recruté par un complexe actif de répression et il sera utilisé comme appât pour reconnaître les messagers à réprimer [3ar04].
2.3.3 Structures des acides ribonucléiques
Les nucléotides sont des molécules polarisées donc en mesures de former des liasons lorqu’ils sont mis en contact. L’Uracile et la l’Adenine se lient par deux ponts hy drogènes, la Guanine et la Cytosine se lient par trois ponts hydrogènes. Ces liaisons sont appelées liaisons Watson-Crick [Sae83]. La Guanine peut se lier à l’Uracile avec deux ponts hydrogènes et cette liaison est appelée liaison Wobble [$ae831.
Pour une séquence d’ARN plusieurs structures secondaires sont possibles et à chaque structure correspond une énergie libre. La structure la plus stable est celle qui minimise l’énergie libre. L’energie libre d’une structure et fonction des paires de bases et des boucles qui la composent 2.2. Les bases appariées favorisent la stabilité de la structure tandis que les boucles et les autres bases non appriées la
défavorisent. Les énergies libres des types de paires de bases (A U, G :C, G :U) sont calculées en utilisant la thermodynamique et dependent principalement des liaisons hydrogènes dont est formée la paire de bases ainsi que des paires de bases voi sines [KWT96, KBT99, XJB98]. Certains algorithmes de prédiction de structures secondaires des ARN se basent sur la minimisation de l’énergie libre [ZukO3, PL88].
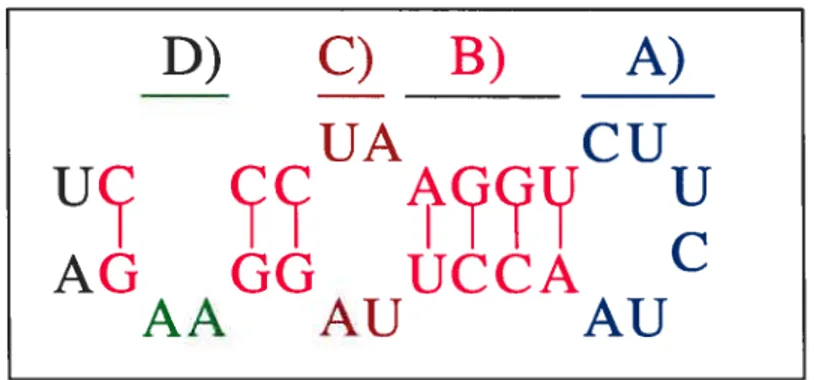
D)
C)B)
A)
UA
CU
uc
u
AG
GG
UCCA
C
AA
AU
AU
Figure 2.2 —Structure secondaire d’une séquence d’ARN. L ‘énergie
libre d’une structure secondaire est ta somme des énergies d’appa riement des paires de bases de t’hetice (B). La boucte terminate (A) , ta boucle interne (C) et te butge (D) défavorisent ta stabi
MICROARN : BIOSYNTHÈSE ET MODALITÉS D’ACTION
3.1 Introduction
La régulation de l’expression des gènes est indispensable pour le bon fonctionne ment cellulaire ainsi qu’au développement et au maintien de tout organisme vivant pluricellulaire. Des mécanismes génétiques, prévus à cet effet, permettent d’activer ou de réprimer l’expression d’un ou de plusieurs gènes sans toutefois altérer le sup port de l’information génétique. On peut distinguer quatre niveaux de régulation génétique
— Pré-Transcriptionnel : Ce mode de régulation se fait par la désactivation du promoteur de transcription. Les promoteurs jouent le rôle d’indicateurs à l’ARN polymérase. En absence de ces promoteurs actifs sur l’ADN, cette enzyme n’est plus en mesure de reconnaître la région à transcrire [LewO4]. C’est ce qui est utilisé dans la spécialisation cellulaire car même si chaque cellule contient une copie complète du génome, et donc de tous les gènes, seule une partie de ces gènes a la possibilité de s’exprimer, ce qui donne une identité ou une spécialité à la cellule.
— Post-Transcriptionnel Dans ce mode de régulation c’est la traduction qui est bloquée. Cette régulation est assurée par des miARN endogènes ou des petits ARN d’interférence (siARN) exogènes. Ces petits ARN, d’une longueur d’environ 22 nucléotides, s’intègrent dans des complexes ribonucléoprotéiques actifs qui les utilisent comme outils de reconnaissance des ARN messagers à réprimer. La régulation se manifeste soit par un simple arrêt de la traduction, sans effet sur la stabilité de la séquence du messager, ou par la destruction
de ce dernier.
— Pré-Traductionnel Ce mode de régulation existe principalement chez les procaryotes. Chez ces organismes, en pius de codon de d’initiation de tra duction soit le AUG, les ARN messagers sont dotés, en amont de ce dernier, d’une autre séquence AGGAGG appelée séquence de $hine-Dalgarno. L’ab sence de cette séquence affecte la traduction du messager [MAKO2] indiquant sa probable implication dans la régulation de l’expression génétique.
— Post-Traductionnel : La régulation post-traductionnelle se fait sur les protéines. L’action est d’empêcher les protéines d’être activées en jouant sur la structure de ces dernières. C’est sur ce mode que se base la conception de la plupart des médicaments [EpsO3l.
La régulation génétique post-transcriptionnelle était encore inconnue il y a une quinzaine d’années [CVOÏ]. C’est plus précisément en 1993 que Ambros et ses collègues ont pu isoler, dans le ver Caenorhabditis elegans, une séquence nucléotidique de 21 nucléotides, le microARN 4, responsable de la répression du gène lin-14 [LFA93I. Cette découverte a ouvert les portes à un grand champ d’exploration et plusieurs chercheurs s’intéressent de près à ce phénomène.
Les avancées, quoi que lentes, démontrent à quel point ce type de régulation est important car il est lié, entre autres, au développement des organismes. En effet, à titre d’illustration, le développement du ver C.elegans passe par trois stades larvaires avant d’atteindre le stade adulte. Le passage d’un stade à l’autre est assuré par un certain nombre de protéines dont fait partie lin-14 et lin-28, un autre gène régulé par lin-4 [MLA9Z]. L’expression de ces deux gènes se fait selon le stade dans lequel se trouve la larve. Au l stade, les protéines nécessaires à
la vie du ver dans les autres stades ainsi que celles nécessaires au changement de stade larvaire sont inhibées, ne laissant que celles nécessaires au développement de l’organisme dans son premier stade. Au moment de passer du premier au second stade larvaire, il y a extinction des protéines utiles au premier stade pour laisser place à celles nécessaires au changement de stade. Cette activation et désactivation de protéines se fait par lin-4 à travers les gènes cibles lin-14 et lin-28
[MLA9Z].
La perturbation de ce processus de régulation de l’expression des gènes lin-14 et lin-28, par la mutation des sites d’intérêt du miARN lin-4 ou par l’inhibition de celui-ci, mène à la stagnation du développement du ver.
3.2 Biosynthèse des microARN
Succinctement, la biosynthèse des miARN est un processus minutieux qui peut être scindé en trois étapes. La première étape est la transcription. Elle consiste à donner naissance au transcrit initial du miARN appelé microARN primaire ou pri-miARN. Cette étape est semblable à la transcription de l’ARN messager. La seconde étape consiste à extraire, de cette séquence initiale, une sous séquence d’environ 80 nucléotides dont la seule spécificité avérée se trouve être la conservation de la structure secondaire en épingle à cheveux. Cette étape est analogue à l’épissage du pré-ARNm. Ces deux étapes, pour les deux types d’ARN, se déroulent dans le noyau de la cellule. La séquence de 80 nucléotides, appelée précurseur ou pré miARN, est alors exportée vers le cytoplasme. Elle y sera clivée et le résultat sera un duplex d’ARN d’une longueur d’environ 22 paires de bases. De ce duplex, un des deux brins sera choisi et deviendra un miARN mature prêt à l’emploi.
3.2.1 Localisation
Les miARN peuvent être issus de toutes les régions du génome à des proportions différentes. La majorité des transcrits primaires sont situés dans les régions intro-niques des gènes codants pour des protéines (ARNc) ou dans de longs transcrits d’ARN non-codants (ARNnc).
Rodriguez et al. ont constaté que sur 232 miARN de mammifères analysés, 117 sont localisés dans les régions nommées plus haut [RGJABO4I. Approximativement 40% (90 miRNA) se trouvent dans des régions introlliques des ARNc tels que mir-25, mir-93 et miR-Ï06b qui sont localisés dans le gène hôte de MCM7, un gène codant pour une protéine impliquée dans la réplication de l’ADN [RGJABO4J. En viron 10% (27 miARN) se trouvent dans les régions introniques des ARNnc tel que miR-155 qui se trouve dans BIC, un transcrit précédemment identifié comme étant un ARNnc [TamOli. On les retrouve toutefois dans d’autres régions du génome mais à de moindres proportions. Quelques uns se trouvent soient dans les introns ou dans les exons selon l’épissage alternatif du transcrit primaire du messager, d’autres sont localisés à cheval avec les régions exoniques des ARNnc [RGJABO4].
Dans leur travaux, Altuvia Y. et al. se sont intéressés, entre autres, aux dis tances entre les pré-miARN [ALL’OS]. Ils ont pu constater que les miARN sont situés dans des agrégats. Sur les 207 miARN humains analysés, 37% sont situés dans des agrégats d’au moins deux éléments dont la distance est d’au plus 3 000 nucléotides. Sur ce point, nous considérons que prendre une longueur unique pour définir un agrégat pour tous les chromosomes est un peu subjectif. D’une part, parce que les chromosomes ne sont pas de même longueur, d’autre part, la densité de miARN varie d’un chromosome à l’autre.
Un autre facteur plus important dans la définition des agrégats est la gran deur des transcrits primaires générés pas l’ARN polymérase II. Car si ces agrégats existent, c’est, peut-être, pour que tous les rniARN s’y trouvant soient transcrits au même temps et, dans ce cas, peut-on toujours parler d’agrégat si l’ARN polymérase II n’est pas en mesure de le transcrire en entier? Une distance nucléique est plus en mesure de situer les miARN dans les chromosomes que de nous aider à définir des unités pouvant être transcrites.
3.2.2 Transcription
La transcription est la première étape dans la biosynthèse des miARN et elle a lieu dans le noyau cellulaire. Les transcrits primaires sont de longueurs variables allant jusqu’à quelques milliers de nucléotides et peuvent contenir un ou plusieurs miARN. Le processus de transcription des miARN est de mieux en mieux élucidé.
Ambros et ses collègues, dans leur travaux, ont constaté que le gène hôte du pre mier miARN connu, lin-4, est d’une longueur de 693 paires de bases [LFA93]. Lee Y. et al. [LKH+04] ont pu établir clairement, dans leur travaux, que les transcrits primaires des miARN sont des séquences de quelques centaines de nucléotides qui sont dotés d’une coiffe à l’extrémité 5’ et d’une queue poly(A) à l’extrémité 3’. Ces caractéristiques indiquent que ces transcrits sont des produits de l’ARN polymérase II, une enzyme qui est aussi impliquée dans la transcription des ARN messagers. D’autres résultats d’expériences obtenus par la même équipe l’ont confirmé. En effet, le traitement de cellules humaines par c-amanitin, connu pour l’inhibition de l’ARN polymérase II, a conduit à la diminution de la concentration de pré-miARN dans les cellules traitées. Ceci met en évidence l’implication de l’ARN polymérase II
dans la transcription des miARN primaires. D’autres résultats vont dans le même sens [CHBO4].
Kurihara et al. ont pu constater que chez les plantes, plus précisément chez l’Arabidopsis Thatiana, la reconnaissance du gène pri-miR-163 est rendue relati vement facile par l’existence d’une boîte ou cassette TATA, semblable à ce que l’on trouve dans des ARN messagers [KWO4I. Ces boîtes sont reconnues par l’ARN polymérase II lors de la transcription de ces derniers et que les deux pri-miR-163 obtenus par une amplification rapide de l’ADNc renferment un signal de polyadeny lation. La présence de signaux semblables a été aussi observée dans le transcription des pri-miARN humain [CHBO4]. Cette étude révèle l’existence d’un promoteur de transcription à l’extrémité amont et une polyadenylation à l’extrémité aval du transcrit primaire.
Ces évidences n’ont pas encore été explorées de manière plus approfondie sur les autres miARN connus mais il apparaît clairement que les transcrits primaires dédiés aux miARN sont de quelques centaines à quelques milliers de nucléotides et qu’il y aurait des signaux qui serviraient de guide à l’ARN polymérase II. Il n’est pas exclu que d’autres enzymes, telle que l’ARN polymérase III, soient impliquées notamment dans les pri-miARN originaires des régions introniques des ARN messagers
[BarO4l.
Car s’il est connu que l’ARN polymérase II est impliqué dans des transcriptions issues de l’ADN, des cas de transcription de miARN à partir d’un messager par l’ARN polymérase II n’ont pas encore été observés. Deux hypothèses émergent
1. La première voudrait que le transcrit primaire soit destiné à devenir un ARN messager. Dans ce cas l’ARN polymérase II se charge de la transcription et lors de l’épissage certains des introns générés, de par leur structure, sont
prédestinés à devenir des miARN et seront traités comme tels en suivant le processus approprié de biosynthèse.
2. La seconde voudrait plutôt que le transcrit primaire soit destiné à donner naissance à des miARN. Dans ce cas aucun épissage n’a lieu et dans cette étape le flou reste entier. Car un transcrit primaire avec deux précurseurs ou plus est probablement soumis à une étape intermédiaire qui consisterait à découper la séquence initiale en petites séquences contenant chacune un précurseur, en quelque sorte une étape qui se substituerait à l’épissage. C’est dans ce cas que l’intervention d’autres ARN polymérases est envisageable.
3.2.3 Éboutage
Les transcrits primaires sont dans un premier temps éboutés pour n’en laisser qu’une séquence d’environ 70 nucléotides. La structure secondaire de la séquence résultante est une tige boucle et elle est conservée dans toutes les espèces {EJO4]. La tâche d’éboutage du pri-miARN incombe à Drosha, une enzyme de la famille des
ARN polymérase III [LAH03]. Cette famille peut être scindée en trois classes selon leurs compositions et l’organisation de leurs différentes composantes [HLY+04].
— La classe I, composée d’un domaine RNaseIII et d’une protéine liaison avec l’ARN double brin. On la retrouve dans les levures et les bactéries.
— La classe II, dont fait partie Drosha, est composée de deux domaines RNaseIII et d’un domaine de liaison avec l’ARN double brin. Cette classe ne se trouve que dans les animaux.
— La classe III renferme les homologues de Dicer, une enzyme impliquée dans la maturation des miARN.
Drosha, d’une grandeur de 130 à 160 kDa, présente à son N terminal une région riche en Proline précédée d’une autre région riche en Serine/Arginine, comme illustré dans la figure 3.1. Han et ses collègues ont pu mettre en évidence qu’en plus de la nécessité de la présence de toutes ses composantes, Drosha doit interagir avec une autre protéine, DGCR8, pour former un complexe fonctionnel capable de cliver la séquence primaire [HLY 04].
Si le niveau d’activité du complexe n’est pas affecté par la suppression de toute la région riche en Proline jumelée à la suppression d’une partie de la région riche en Serine/Argenine, la suppression totale de ces régions particulières du N termi nal altère l’association et le clivage. Les auteurs ont aussi confirmé que DGCR8 est indispensable à l’activité du complexe {HLY04]. Son rôle serait de stabiliser la liaison entre le double brin du transcrit primaire et le complexe comme il est possible que cette protéine joue un rôle dans l’orientation de la tige-boucle sur le complexe. Cette hypothèse est plus que probable puisqu’il est clairement établi que chacun des domaines RNaseIII clive un brin de la tige du pri-miRNA [HLY04]. Plus précisément R1I1Da s’occupe du brin 3’ alors que RIIIDb agit sur le brin 5’. Donc il est évident qu’un ajustement doit être fait avant que la tige-boucle ne soit intégrée dans le complexe.
Zeng et ses collègues se sont intéressés à l’interaction de Drosha avec les pri miRNA [ZYCO5]. Ils ont confirmé que Drocha agit en un seul site et le résultat est une tige-boucle avec un débordement de 2 ou 3 nucléotides à l’extrémité 3’ [LAH03, HLY04, ZKJ04a].
Il a été aussi constaté que le site de clivage de Drosha coïncide avec une des extrémités du miARN mature [LAW103, HLY04, ZYCO5]. Si le miARN mature
est localisé sur le brin 5. de la tige, le clivage se fera à l’extrémité 5’ du miARN mature et le brin 3’ sera coupé à deux nucléotides en aval du vis-à-vis du site de clivage du brin 5. Si par contre la séquence mature est située sur le brin 3’. le clivage du brin 3’ se fait juste après le dernier nucléotides du miARN mature, le site de clivage du brin 5’ est à deux nucléotides en aval. (voir la figure 3.2).
FE
D
C
B
Figure 3.1 — L’ARN polymérase III Drosha. Drosha est composé
(A) d’un domaine de liaison avec l’ARN doubte brin. (B) et (C) de deux domaines RNase II], R1I1Da et RIIIDb, (D) d’une séquence protéique intermédiaire, (E), d’une région riche en Se rine/Arginine et (F) d’une région riche en Protine
Figure 3.2 — Sites de clivage de Drosha. (A) : Le miRNA mature
est situé sur te brin 3’. (B) : Le miRNA mature est situé sur te brin 5’.
Les opérations de clivage, quoi que assez caractérisées dans l’espace. ne le sont pas dans le temps donc on ne sait pas encore quel est le brin qui est traité en premier.
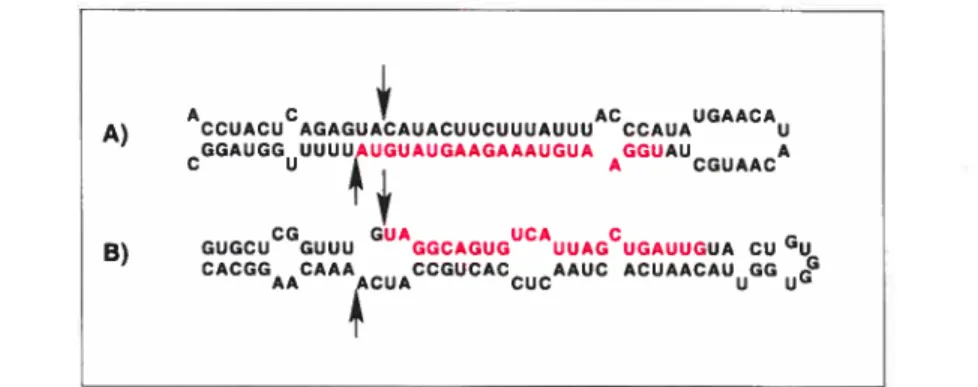
A
A C AC UGAACA
A) CCUACU AGAGUACAUACUUCUUUAUUU CCAUA U GGAUGG UUUUAUGUAUGAAGAAAUGUA GGUAU A
C U A CGUAAC
CG GUA UCA C
B) GUGCU GUUU GGCAGUG UUAG UGAUUGUA cii Gu CACGG CAAA CCGUCAC AAUC ACUAACAU GG
AA ACUA CUC U U
Cette étape du processus ne manque pas de soulever des questionnements sur ce que reconnaît Drosha dans la séquence mature. On sait que le site de clivage de Drosha coïncide avec une des extrémités du miARN mature mais on ne sait pas encore quel est le signal qui indiqilerait ces extrémités. Car hormis d’être un peut plus riches en U comparativement aux autres régions, comme illustré dans la figure 3.3, les séquences matures ne recèlent pas d’autres caractéristiques notables. Considérant la minutie avec laquelle ces tâches doivent être exécutées, on ne peut pas dire que ces choix sont aléatoires. L’hypothèse qui nous vient est la possibilité de formation de liaisons hydrogènes entre les premiers nucléotides du miARN et un site du complexe et ces liaisons seraient favorisées par la présence de U compa rativement aux autres nucléotides.
o o o E o o o I I I I I I
Figure 3.3 — Distribution des nucléotides le long des pré-miARN.
Les régions povieuses des miRNA matures, régions proches de t’extrémité 5’ du pré-miARN, sont plus riches en U (courbe noire) comparativement aux autres nuctéotides.
0 10 20 30 40 50 60
Zeng et son équipe ont étudié l’effet de la longueur de la boucle terminale sur
l’activité de Drosha. Il a été constaté qu’une boucle de moins de 9 nucléotides
affecte considérablement l’activité de Drosha alors que des structures secondaires avec des boucles de 10 nucléotides ou plus sont bien transformées.
Comme Drosha traite des milliers, peut être des millions, de pri-miARN de
compositions nucléotidiques différentes, il doit bien y avoir un signal que ce dernier reconnaît pour déterminer, avec une telle exactitude, son site de clivage. La présence
d’une boucle commune à tous les miARN suggère une particularité fonctionnelle
ayant un rôle important dans la catalyse qui a lieu à environ 22 nucléotides de cette
dernière {ZYCO5].
3.2.4 Exportation
Le transcrit primaire, une fois clivé dans le noyau par Drosha, est exporté vers le
cytoplasme cellulaire. Cette tâche est assurée par Exportin-5 (Exp5) via le com
plexe du port nucléaire (NPC). Ces agents de transport sont des assemblages de macromolécules d’une grandeur variant de 50 kDa dans les levures à 160 kDa chez les mammifères [BCGO4].
Exp5 se lie aux pré-miARN indépendamment de leurs séquences [BCGO4I mais la structure semble jouer un rôle primordiale dans cette étape de la biosynthèse. En effet, si une absence de débordement la base de la tige est tolérée, un débordement
à l’extrémité 5’ de la tige nuit considérablement à l’exportation du précurseur en
inhibant la liaison de ce dernier à l’agent de transport [ZCO4I.
D’autres détails structuraux affectent l’exportation telles que la longueur de la
tures secondaires avec des tiges courtes se lient moins bien à Exp5 et par conséquent s’exportent moins bien. Une fois dans le cytoplasme, ces structures restent stables impliquant que le processus de maturation n’est pas immédiat [BCGO4]. En plus d’assurer le transport du noyau vers le cytoplasme, l’exportin-5 protège les miARN d’être altérés à leur arrivée dans le cytoplasme [ZCO4J.
3.2.5 Maturation
La maturation est la dernière étape dans le processus de biosynthèse des miARN. Elle consiste à cliver la tige-boucle à quelques paires de bases de la boucle terminale et d’en libérer un duplex d’ARN d’environ 22 paires de bases avec des débordements de 2 nucléotides aux deux extrémités 3’ des deux brins du duplex. Cette tâche est dévolue à Dicer, une ARN polymérase III de la classe III.
Dicer est constitué de plusieurs domaines. En plus des deux domaines d’ARNase III a et b et du domaine de liaison avec l’ARN double brin, on y trouve un domaine PAZ (Piwi/Argonaute/Zwille), d’une protéine appelée DUF283, dont la fonction demeure incollnue, et d’un domaine ATPase/helicase [ZKJ+04b] (voir figure 3.4). PAZ est un complexe protéique propre à Dicer et à la famille Argonaute et est très conservé à travers les espèces. D’après des expériences réalisées avec des petits ARN d’interférence exogènes (siRNA), le domaine PAZ reconnaît le débordement de 2 nucléotides de l’extrémités 3’ de la tige vue que ce domaine est connu pour son affinité pour de l’ARN simple brin.
Les deux nucléotides du débordement sont attirés vers une cuvette où ils sont emprisonnés. Les deux nucléotides suivants sont attirés vers un site de la protéine riche en résidus aromatiques et hydrophobes où des liens hydrogènes se forment
entre les acides aminés de ce site de PAZ et les oxygènes des liens phosphodiester
du siARN [MYPO4]. Il est fort probable que c’est ce domaine, PAZ, qui reconnaît les produits de Drosha, vu son affinité pour le débordement à l’extrémité 3’ de ces derniers. En effet, le domaine de liaison reconnaît les ARN double brin via les
débordements et s’y lie. Les ARNaselli a et b s’accrochent chacun à un brin pour
former un complexe stable capable de réaliser l’excision qui se fait à environ 22 nucléotides de la base de la tige.
S’il est connu, chez Drosha, que chacun des domaines a et b s’occupe d’un brin on ne sait pas encore s’il existe, chez Dicer, une quelconque affinité entre un brin
particulier avec un domaine particulier. Si c’est le cas, il serait plus que probable
que le duplex résultant de Dicer subisse une autre opération avant de se retrouver dans le complexe actif de répression. Cette opération intermédiaire serait dédiée à la séparation du duplex avant qu’un des deux brins ne soit recruté. Dans l’absence de cette étape intermédiaire, Dicer doit être en possession de deux domaines qui se chargeraient de vérifier la stabilité de chacune des extrémités du duplex pour pouvoir en sélectionner un brin.
Au niveau de cette étape, une divergence apparaît entre l’humain et la droso phile. Cette dernière possède deux complexes Dicer soient DCR1 et DCR2. L’un est dédié au clivage des siARN tandis que l’autre s’occupe des miARN. Chez l’humain, et les mammifères en général, un seul complexe, Dicer, prend en charge les deux
types d’ARNnc. Ce dernier semble avoir une affinité pour les ARN double brin avec
un débordement localisé à l’extrémité 3’ et son efficacité dépend de la composition de ce débordement.
Vermeulan et al. ont constaté que, dans les siARN, la composition nucléotidique des débordements n’affecte pas la longueur des résidus de clivage mais elle agit gran dement sur l’efficacité de Dicer [VBRO5]. Les ARN double brin sont efficacement traités s’ils ont des débordements se terminant avec un A ou ayant un C à l’avant dernière position. Un C à la position terminale ou un A à l’avant dernière position réduisent l’efficacité de Dicer [VBRO5].
F
D
C
D
B
Figure 3.4 — L’ARN polymérase III Dicer. Dicer est composé : (A)
d’une protéine de liaison avec l’ARN doubte brin, (B) et (C) de deux domaines RNaseIII R11IDa et RIIIDb, (D) d’un domaine PAZ, (E) de ta protéine DUF283 et (F) et de ATPase/helicase
3.3 Modalité d’action
Le résidu de Dicer, comme mentionné plus haut, est un duplex d’ARN avec des débordements aux deux extrémités 3’ des brins. Ce duplex entrera dans une autre étape qui consiste à le défaire, de sélectionner un des deux brins et de l’intégrer dans un complexe ribonucléoprotéique actif qui a pour tâche de cibler les ARN messagers pour arrêter la traduction ou tout simplement pour les détruire. L’une ou l’autre des actions dépend du niveau de complémentarité du brin choisi et la région de l’ARN messager cible avec laquelle ce brin se lie.
3.3.1 Formation du complexe actif
Cette étape est bien modélisée chez la drosophile mais pas encore chez les mam mifères. Elle consiste à prendre le duplex résultant de l’action de Dicer pour former
un complexe actif. En effet, l’activité des petits ARN d’interférence, siARN, passe par cette étape où le duplex est chargé dans un complexe intermédiaire appelé RLC (RISC Loading Complexe) pour finir dans un complexe de répression appelé RISC (RNA-Indnced Sitencing Complexe). Ce chargement se fait principalement via une interaction entre le duplex et R2D2, qui est une protéine avec deux domaines de liai son avec l’ARN double brin, qui interagit dans DCR2 lors du clivage. DCR2 est un des deux complexes Dicer dédié principalement au clivage des siARN double brin. L’autre complexe, DCR1, est dédié au clivage des pré-miARN [Tan05,LNP04]. Rien n’indique que ce même procédé sera suivit par les miARN car ces derniers interagissent plutôt avec DCRÏ, qui ne possède qu’un seul domaine de liaison avec l’ARN double brin.
Le complexe de chargement des ARN d’interférence, s’initie par une interac tion entre le domaine R2D2 est le duplex d’ARN. Les détails de cette interaction ne sont pas bien cernés, mais nous savons que R2D2 privilégie l’extrémité la plus stable [LNP04], laissant la moins stable libre pour faciliter la séparation des brins du duplex. Cette asymétrie dans la sélection des brins caractérise aussi les com plexes des miARN ou le brin choisi est celui dont l’extrémité 5’ est la moins stable. Une fois dans le RLC, le duplex est défait et un des deux brins est sélectionné. Cette tâche, normalement dévolue pour une ARN helicase, semble faire appel à un ou plusieurs autres catalyseurs car cette opération ne se produit pas in vitro. Ce processus n’est donc pas a.ussi simple qu’il a été imaginé préalablement [TanO5].
Le complexe ainsi formé par le brin du siARN et R2D2, et probablement d’autres agents, est initialement inactif. Pour devenir actif, il s’associe avec une protéine de la famille des Argonautes qui a la capacité de cliver les ARN messa
gers [Tan05,
MLP+04].
Cette protéine, que l’on retrouve dans presque toutes les espèces, est souvent localisée dans le complexe RISC laissant sous-entendre un rôle dans le ciblage des ARNm.Nous avons vu que c’est le domaine PAZ qui reconnaît les débordements de 2 nucléotides à l’extrémité 3’ de la tige du précurseur et que ce domaine est composé de 3 protéines Piwi, Argonailte et Zwille. Des résultats récents ont montré que l’extrémité 5’ du brin choisi est emprisonnée dans une cuvette très conservée de la protéine Piwi [MYM+05j. Ce qui demeure méconnu c’est le comment et le quand de cette interaction. Piwi interagit dans une première étape avec le phosphate du bout 5’. En effet, pendant que le phosphate terminal est attiré dans la cuvette, les 4 ou 5 nucléotides suivants se lient à la protéine via les oxygènes du pont phos phodiester ayant la capacité de le faire. Cela concorde avec le calcul des surfaces enterrées de la protéine, où le brin actif couvre 1 500 A2 tandis que le brin passif (ARNm) ne couvre que 350 À2 Comme les séquences de la protéine Piwi de Ar chaeogÏo bus fuÏgidus, sur lequel ces tests ont été réalisés, et celles des Argonautes 1 et 2 humaines présentent des points de ressemblance, d’autres tests ont été réalisés en utilisant Argonaute2 humaine [MYM05]. Une réduction de l’activité de clivage se fait sentir avec une mutation d’ull acide aminé au niveau du site d’interaction entre Argonante2 humaine et le brin d’ARN et elle est plus prononcée avec une mutation de deux acides aminés ce qui sous entend l’importance de ce site et de cette liaison.
L’association, chez l’humain entre Argonaute 2, la protéine d’excision, un brin du miARN, le brin directeur, et probablement d’autres protéines forme le complexe ribonucléoprotéique RISC prêt à agir est cibler les ARN messagers dans ses régions non-traduites.
3.3.2 Ciblage des ARN messagers
Le complexe actif ainsi formé cible les ARN messagers. L’action résultante dépend du degré de complémentarité entre le brin du miARN recruté par le complexe de répression et le messager ciblé. Une complémentarité parfaite conduit à la des truction de ce dernier alors que dans le cas d’une complémentarité imparfaite, le site actif ne pouvant probablement pas atteindre le brin du messager suite à une géométrie défavorable, ne fait qu’arrêter le processus de traduction de ce der nier [HZO2, MYM05, $JAO3j.
Dans le cas d’une complémentarité imparfaite, le duplex formé par le petit brin et l’ARN messager doit satisfaire certaines conditions. La principale condition est l’appariement des 5 premiers nucléotides du côté 5’ du miARN. Ce sont ces derniers qui sont présentés dans un premier lieu au messager. En présence d’un duplex instable, i.e un duplex qui n’a pas assez d’appariements entre ces premiers nucléotides éclaireurs, ce dernier se défait probablement tandis qu’en présence d’un duplex stable l’opération d’appariement a la possibilité d’aller de l’avant. Comme les 5 premiers nucléotides du miARN mature sont liés à Piwi [MYM05], ils sont donc stables et adoptent probablement une conformation leur permettant de se lier, par des ponts hydrogènes, à l’ARN messager. C’est probablement la stabilité de ces premiers nucléotides qui favorise la formation d’autres appariements en aval. L’appariement du premier nucléotide n’est pas nécessaire et peut être même im possible à réaliser vu la conformation qui lui est imposée par l’emprisonnement du phosphate terminal dans la cuvette de la protéine Piwi.
Cette activité de clivage ou de répression sous-entend une accessibilité du miARN au messager [B CR05, OAFO5]. En absence de cette accessibilité, une diminution
drastique de l’activité du siARN a été constatée laissant croire que la complémentarité parfaite et globale, ou locale du côté 5’, n’offre aucune certitude que le messager soit la cible du miARN ou siARN considéré. Cette complémentarité doit être prise dans un contexte plus globale en essayant d’intégrer le plus possible des éléments du messager cible, dont l’accessibilité. Dans le cas d’une complémentarité parfaite et d’un messager accessible ce dernier est dégradé par un clivage au niveau dil 10cme
nucléotide {EMP’j.
Un certain ifou règne encore quant à l’agent responsable de ce clivage. L’ab sence de tous les membres de la famille Argonaute mène à une absence totale de clivage, indiqilant que c’est un membre de cette famille qui est responsable du clivage. D’autres possibilités commencent à émerger voulant qu’il y ait deux com plexes RISC, un avec des capacités de clivage et l’autre sans. Le complexe doté d’une capacité de clivage dégrade le messager dans le cas d’une complémentarité parfaite et inhibe la traduction dans le cas d’une complémentarité imparfaite. Le complexe dépourvu de la faculté de clivage ne peut qu’arrêter la traduction même dans le cas d’une complémentarité parfaite [TanO5].
Cette possibilité de deux complexes soupçonnées chez la drosophile peut ne pas exister chez l’humain puisque on a constaté une divergence au niveau de la maturation. Cette divergence peut être le point de départ de deux voies, l’une pour les siARN avec complexe RISC pourvu d’une capacité de clivage et une autre pour les miARN sans cette dernière. Puisque chez les mammifères le clivage des siARN et miARN est réalisé par un seul complexe, Dicer, il est moins probable qu’il existe deux complexes RISC d’autant plus qu’aucune étape intermédiaire n’est encore mise en évidence.
Le site de clivage pour les siARN se situe entre les 10eme et 11eme nucléotides
indépendamment de la longueur du siARN mature. Cela indique que le site de clivage dépend plus de la structure du complexe RISC dans l’espace que de la longueur ou la composition de siARN mature {EMP+].
3.4 Conclusion
Des différents résultats de recherche, nous avons pu constater l’importance de la structure secondaire des pré-miARN pour qu’ils soient recrutés par les différents complexes actifs. La structure seule semble expliquer la formation des complexes qui interviennent dans la biosynthèse des miARN.
Le premier motif structural est la boucle de l’épingle à cheveux. L’activité de Drosha dépend de la longueur de la boucle qui affecte aussi l’activité de l’agent d’ex portation. Cela montre la concordance entre ces deux acteurs. Les débordements de 2 nucléotides à l’extrémité 3’ de la structure sont aussi des motifs structuraux reconnus par l’agent de transport. La longueur de la tige affecte aussi l’efficacité de Exp5 où les tiges courtes sont moins bien exportées. Dicer, aussi, semble reconnaître les produits de Drosha par leurs débordements de 2 nucléotides à l’extrémité 3’. Pour le moment, cela semble être le seul motif notable en commun entre Drosha, Exp5 et Dicer, mais il n’est pas impossible que d’autres motifs structuraux locaux apparaîtront. Des motifs de séquence semblent aussi être utilisés par Drosha no tamment pour la reconnaissance des extrémités du miARN mature lors du clivage du pri-miARN.
LE PROBLÈME ET LES TRAVAUX ANTÉRIEURS
4.1 Introduction
Les pré-miARN sont connus pour se replier en tige-boucle imparfaite [EJO4].Cette caractéristique, commune aux pré-miARN, est le point de départ dans la prédiction de ces derniers.
L’étape initiale d’identification de candidats potentiels par des outils bioin formatiques est nécessaire vu que des expériences biochimiques à la grandeur du génome avec les moyens actuels est impensable. En s’aidant de ce moyen, les cher cheurs font des gains énormes en temps et en ressources. Toutefois les outils bio-informatiques ne peuvent pas, à eux seuls, nous prédire avec certitude ce genre de gènes ce qui rend indispensable une validation biochimique des candidats identifiés.
4.2 Définition du problème
Le problème est de trouver des séquences d’une centaine de nucléotides ayant des structures secondaires en forme d’épingle à cheveux susceptibles d’être des candi dats potentiels pour devenir des gènes de miARN. Si trouver des séquences avec ce genre de structures secondaires est relativement facile et peu coûteux en temps, les filtrer et classer les candidats ne l’est pas. Nous voyons deux raisons à cela
1. La première réside dans la divergence entre les compositions nucléotidiques de ces gènes. En effet, les alignements multiples sont inefficaces à produire une séquence consensus indiquant que la composition nucléotidique des précurseurs
n’est peut-être pas un critère important.
2. La seconde réside dans la divergence entre les structures. Les structures des pré-miARN ne présentent pas de motifs structuraux locaux notables en de hors de la structure globale en forme de tige-boucle.
Il est vrai que les outils actuels de prédiction de structures secondaires ne sont pas très fiables. Suite aux expériences qu’ils ont réalisés [KSWO4Ï, Krol et aï. ont constaté ces différences. Sur un ensemble de dix séquences, 8 montrent un cer tain nombre de différences de structure avec les prédictions de mfoïd [ZukO3]. Ces différences se trouvent surtout dans les boucles terminales et dans le nombre, la localisation et la longueur des bulges et des boucles internes [K$WO4]. Cela peut être d’une importance capitale sachant que la boucle terminale figure parmi les motifs structuraux très importants dans la biosynthèse des miARN [ZYCO5, ZCO4]. Il est possible qu’il y ait d’autres sites d’intérêt pour les enzymes et autres complexes intervenant dans la biosynthèse des miARN. C’est le cas pour la boucle terminale. Néanmoins d’autres motifs, même communs à un certain nombre de précurseurs, ne sont d’aucune utilité pour les complexes actifs et par conséquent inutiles pour la prédiction. L’identification des motifs structuraux importants ne peut pas se faire avec certitude au stade actuel de la recherche. Cela pousse la communauté scientifique à combiner les caractéristiques extraites des séquences de pré-miARN connus. La pondération de ces différentes caractéristiques se base prin cipalement sur la prépondérance de ces dernières plutôt que sur leur apport dans le processus de biosynthèse. Cet apport ne peut être vérifié que par des expériences impliquant des délais et des ressources supplémentaires.