FRÉDÉRIC TREMPE
Étude du rôle de la protéine U94 de l'herpèsvirus humain
de type 6 dans le processus de l’intégration
chromosomique.
Mémoire présenté
à la Faculté des études supérieures et postdoctorales de l’Université Laval dans le cadre du programme de maîtrise en microbiologie-immunologie
pour l’obtention du grade de maître ès sciences (M.Sc.)
DÉPARTEMENT DE MICROBIOLOGIE-IMMUNOLOGIE FACULTÉ DE MÉDECINE UNIVERSITÉ LAVAL QUÉBEC 2013 © Frédéric Trempe, 2013
Résumé
L’herpèsvirus humain de type 6 (HHV-6) infecte les enfants en bas âge et sa prévalence est estimée à près de 95% dans la population mondiale. Ce virus se distingue des autres membres de la famille des herperviridae par sa capacité à s’intégrer aux chromosomes cellulaires. On estime qu’environ 1% de la population mondiale serait porteuse d’une copie du génome du HHV-6 par cellule (52, 73, 100, 119, 131). Le mécanisme de cette intégration est toujours inconnu.
Nous pensons que la protéine U94 du HHV-6 joue un rôle important dans l'intégration chromosomique du génome viral. Elle serait nécessaire à l'éxécution d'une recombinaison homologue entre les séquences télomériques cellulaires et virales (motif TTAGGG). U94 a une homologie de séquence de 24% avec la protéine Rep68 responsable de l'intégration du génome de l’adeno-associated virus 2 (AAV-2) au chromosome 19 (123). Pour ce faire, quatre activités intrinsèques à la protéine Rep68 lui sont nécessaires : la liaison à l'ADN simple et double brin, l'ATPase, l'hélicase et l'endonucléase (54, 97).
Le but de ce projet de recherche était de caractériser les activitités biochimiques de la protéine U94. Tout d’abord, nous avons démontré que la protéine U94A est localisée au noyau en accord avec les résultats obtenus par le laboratoire du Dr. Mori (87). Pour réaliser nos études, nous avons exprimé et purifié U94 chez E. coli en fusion avec la protéine
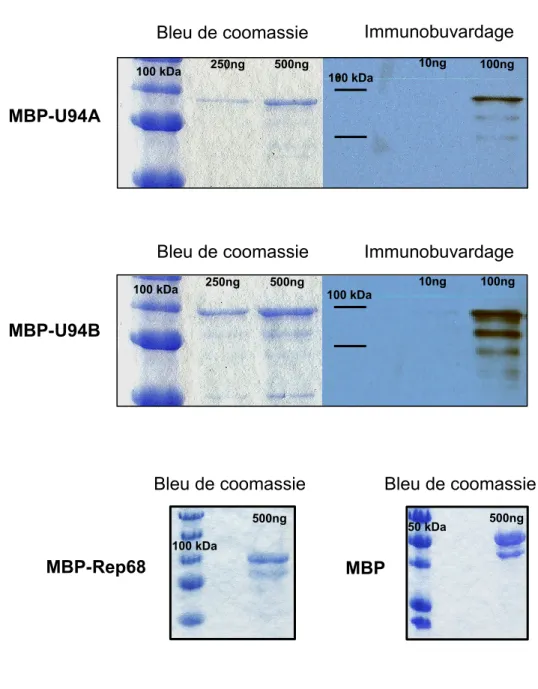
maltose-binding protein (MPB). Nos résultats démontrent que les protéines MBP-U94A et
MBP-U94B sont capables de lier préférentiellement l'ADN simple brin avec un motif CCCTAA (complément du motif télomérique TTAGGG). L’essai de liaison sur le Proteon™ XPR36 démontre également que la protéine MBP-U94B lie un ADN double brin ayant le motif télomérique cellulaire. Nos résultats nous ont amenés à vérifier si l'ARN de la télomérase, qui contient un motif CCCTAA, pouvait aussi être lié par les protéines MBP-U94A et MBP-U94B. Les résultats obtenus indiquent que MBP-U94 ne lie pas l'ARN, ni la séquence équivalente en ADN ce qui suggère que la liaison de U94 requiert plus d’une répétition du motif CCCTAA. En se basant sur des études de mutagenèse réalisées sur la protéine Rep68 (128, 129), nous avons générés des mutants U94A et U94B. Les résultats
démontrent que la mutation K395A affecte négativement la capacité de liaison à l’ADN de U94.
Les essais d'ATPase indiquent que MBP-U94A et MBP-U94B hydrolysent l'ATP en ADP et AMP en présence d'un ADN simple brin ou d’un ADN double brin. Divers mutants des activités d’ATPase/Hélicase et d’endonucléase présumées furent générés et devront être caractérisés.
Ces résultats suggèrent que U94 possède les activités biochimiques nécessaires à l'intégration du génome viral aux chromosomes cellulaires.
Abstract
Human herpesvirus 6 infects young children with an estimated prevalence of 95% in the world population. It differs from the other members of the herperviridae family by its capacity to integrate cell's chromosomes. It is estimated that approximately 1% of the world population carries a copy of the HHV-6 genome per cell (52, 73, 100, 119, 131). The chromosomal integration mechanisms used by HHV-6 are currently unknown.
Our hypothesis is that the HHV-6 U94 protein plays an important role in chromosomal integration that we suspect occur through homologous recombination between cellular and viral telomeric sequences (TTAGGG). The U94 gene product shares 24% sequence homology with Rep68, a responsible for the genomic integration of adeno-associated virus 2 (AAV-2) (123). To promote integration, Rep68 relies on four intrinsic activities: binding to single and double stranded DNA, ATPase activity, helicase and endonuclease (54, 97).
The goal of this research project is to characterize the biochemical properties of U94 and determine whether it posseses activities similar to Rep68. First, we confirmed the results of Dr. Mori's laboratory by showing that U94 is localized in the nucleus (87).Next, to conduct our studies, we’ve expressed and purified maltose-binding-U94 recombinant proteins (MBP-U94) in E. coli. Our results suggest that MBP-U94A and MBP-U94B preferentially bind single-strandred DNA containing the CCCTAA motif (complement to the TTAGGG telomeric motif). Surface plasmon resonance (SPR) experiments also indicate that MBP-U94B binds double-stranded DNA containing telomeric motifs. Since the telomerease RNA component TERC contains the CCCTAA motif, we investigated whether MBP-U94 could bind a single-stranded RNA molecule containing the CCCTAA motif. SPR analysis clearly indicates that MBP-U94 does not bind such RNA nor a single-stranded DNA molecule having a single CCCTAA motif, suggesting that more than one motif is required for proper binding. Based on published work on Rep68 (128, 129), we generated specific U94 mutants. Our results indicate that the K395A mutation greatly diminishes U94 binding to DNA pointing out the importance of this residue.
ATPase assays were also performed and indicate that both MBP-U94A and MBP-U94B possess the ability to hydrolyze ATP into ADP and AMP when incubated in the presence of DNA. Several other mutants targeting the helicase and endonuclease activities were generated and will be tested in the near future.
Altogether these results suggest that U94 has biological properties that are consistent with a role for this protein in the process of chromosomal integration of the HHV-6 genome into the host chromosomes.
Avant-Propos
Je tiens à remercier toutes les personnes qui m'ont aidé à l'accomplissement de ce projet. Je remercie d'abord le Dr. Louis Flamand de m'avoir accueilli dans son laboratoire et offert un projet aussi intéressant. Il a su partager ses connaissances avec moi et m'enseigner l'art de travailler en laboratoire. À cet effet, les Drs Annie Gravel et Guillaume Morissette ont aussi été d'une aide plus que précieuse tout au long de mes deux ans de maîtrise. Un grand merci à ces trois, sans qui ce projet ne serait pas ce qu'il est maintenant. Je veux aussi remercier Mathieu Iampietro, Isabelle Dubuc et à nouveau Guillaume Morissette pour ces bons moments passés au bureau, ces échanges sur la politique et les bons coups sur les marchés boursiers, sans quoi les journées auraient été beaucoup plus longues. Un grand merci à Tania Lévesque qui fut d'une aide précieuse pour les expériences au HPLC. Enfin, je veux remercier ma copine, Amélie Veillette pour son support constant tout au long de ces deux belles années de maîtrise.
Table des matières
RÉSUMÉ... ii
ABSTRACT... iv
AVANT-PROPOS... vi
TABLE DES MATIÈRES... vii
LISTE DES TABLEAUX... x
LISTE DES FIGURES... xi
LISTE DES ABBRÉVIATIONS... xiii
INTRODUCTION... 1
I. LA FAMILLE DES HERPESVIRIDAE... 1
1.1 Contexte... 1
1.2 Les trois sous-familles... 1
II. GENRE DES ROSEOLOVIRUS... 3
2.1 Historique... 3
2.2 Deux espèces du HHV-6... 3
2.3 Épidémiologie... 4
2.4 Pathologies associées... 4
2.5 Structure du virus... 6
2.5.1 Le génome... 6
2.5.2 Nucléocapside, tégument et enveloppe... 9
2.6 Cycles viraux... 10
2.6.1 Cycle lytique... 11
III. INTÉGRATION CHROMOSOMIQUE... 13
3.1 Découverte... 13
3.2 Intégration de novo et transmission héréditaire... 14
3.3 Virus latent ou mort? ... 15
3.4 Répercussions cliniques... 16
3.5 Mécanisme de l'intégration... 17
3.5.1 La recombinaison homologue... 17
3.5.2 Les télomères... 17
3.5.3 Virus de Marek (MDV)... 18
3.5.4 Hypothèses pour HHV-6... 18
IV. LA PROTÉINE REP68 DU AAV-2... 19
4.1 Contexte... 19
4.2 Rôle de la protéine Rep du AAV-2... 19
4.3 Domaine d'interaction avec l'origine et hélicase SF3.... 21
V. PROTÉINE U94 DU HHV-6... 22
5.1 Unique à HHV-6... 22
5.2 U94 et Rep68/78... 23
5.3 Latence et transactivation... 26
VI. HYPOTHÈSE ET OBJECTIFS DE RECHERCHE... 26
6.1 Hypothèse... 26
6.2 Objectif principal... 27
MATÉRIEL ET MÉTHODES...
29
RÉSULTATS...
41
DISCUSSION...
71
CONCLUSION ET PERSPECTIVES...
79
Liste des tableaux
Liste des figures
Figure 1. Schéma simplifié du génome viral du HHV-6... 7
Figure 2. Génome de l’herpèsvirus humain de type 6 (HHV-6)... 8
Figure 3. Cycle réplicatif du HHV-6... 11
Figure 4. Domaines de la protéine Rep78... 21
Figure 5. Transcrits de l’ORF U94 du HHV-6... 22
Figure 6. Alignement des séquences en acides aminés de Rep68 et U94... 25
Figure 7. Localisation cellulaire de la protéine de fusion Flag-U94A... 42
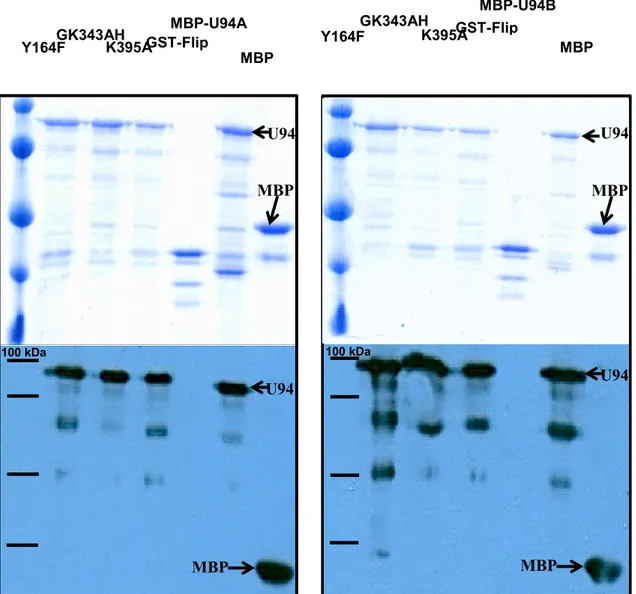
Figure 8. Immunobuvardage et bleu de coomassie des protéines purifiées 44 Figure 9. Immunobuvardage et bleu de coomassie des protéines mutées... 46
Figure 10. Retard sur gel en présence d'ADN simple brin riche en G et riche en C... 48
Figure 11. Retard sur gel de la protéine U94 sur ADN simple brin... 50
Figure 12. Retard sur gel de la protéine U94 sur ADN double brin... 52
Figure 13. Schéma de la puce du Proteon™ XPR36... 53
Figure 14. Expérience de mise au point de la liaison de la protéine MBP à l'ADN simple et double brin au Proteon™ XPR36... 55
Figure 15. Expérience de mise au point de la liaison de la protéine MBP- U94B à l'ADN simple et double brin au Proteon™ XPR36... 56
Figure 16. Témoins négatif et positif du test de liaison à l'ADN simple et double brin au Proteon™ XPR36... 57
Figure 17. Test de liaison de la protéine MBP-U94A à l'ADN simple et double brin au Proteon™ XPR36... 59
Figure 18. Test de liaison de la protéine U94B à l'ADN simple et double brin au Proteon™ XPR36... 60
Figure 19. Test de liaison de la protéine MBP-U94A mutée à l'ADN
simple et double brin au Proteon™ XPR36... 62
Figure 20. Test de liaison de la protéine MBP-U94B mutée à l'ADN simple et double brin au Proteon™ XPR36... 63
Figure 21. Essais d'ATPase (HPLC) avec oligonucléotide simple brin... 65
Figure 22. Essai d'ATPase (HPLC) avec ADN double brin... 67
Liste des abréviations
A260/280 (Absorbance à 260 nm/280 nm)
AAV-2 (virus associé aux adénovirus de type 2) ADP (adénosine diphosphate)
ALT (Alternative lengthening of telomeres) AMP (adénosine monophosphate)
ATP (adénosine triphosphate)
BCA (essai à l’acide bicinchoninique)
CBMC (cellules mononucléées du sang de cordon) ChIP (Chromatin Immunoprecipitation)
ciHHV-6 (Intégration chromosomique de l'herpèsvirus humain de type 6) CMV (Cytomégalovirus)
cpm (comptes par minute) DR (directement répété) EBV (virus Epstein-Barr) E. coli (Escherichia coli)
EDTA (Acide éthylène diamine tétraacétique) EMSA (Electrophoretic mobility shift assay) HBLV (Human B-lymphotropic lymphocyte virus) HHV (Herpèsvirus humain)
HSCT (Hematopoietic stem cell transplantation)
HPLC (High-performance liquid chromatography) HSV1 (Herpès simplex de type 1)
HSV2 (Herpès simplex de type 2) hTBP (Human TATA-binding protein) IE (région précoce-immédiate)
IPTG (Isopropyl β-D-1-thiogalactopyranoside)
kpb (kilo paires de bases)
KSHV (virus associé au sarcome de Kaposi)
MBP (protéine qui lie le maltose)
MDV (Marek disease virus) NTP (nucléoside triphosphate) OBP (Origin binding protein) ORF (cadre de lecture ouvert)
PBMC (cellules mononucléées du sang périphérique) PBS (Phosphate Buffered Saline)
PBST (Phosphate buffered Saline Tween) POT1 (Protection of telomeres 1)
RAP1 (Ras-related protein 1)
RBE (site de liaison de Rep)
RH (recombinaison homologue) Rmax (réponse maximale)
RU (response unit) SF3 (super famille III)
SIDA (syndrome de l’immunodéficience acquise)
SNC (système nerveux central)
SOT (Solid organ transplant) SP (Sclérose en plaques)
SSC (solution de citrate de sodium salin)
TAE (Tris-acetate-EDTA buffer)
TBE (Tris-borate-EDTA buffer)
TBST (Tris-buffered saline tween buffer) TERC (Telomerase RNA component) TIN2 (TRF1 interacting nuclear factor 2)
Tm (Melting temperature) TP (Température de la pièce) TPP1 (Tripeptidyl peptidase 1)
TRS (Séquences télomériques répétées) TRF1 (Telomeric repeat-binding factor 1) TRF2 (Telomeric repeat-binding factor 2) trs (Site de résolution terminale)
UL (section interne unique longue)
US (section courte unique)
VIH (Virus de l’immunodéficience humaine) VZV (Virus de la varicelle et du zona)
Introduction
1. La famille des Herpesviridae
1.1 Contexte
Les herpèsvirus infectent une grande variété d’organismes vivants. Chez l’humain, on compte neuf virus de la famille des Herpesviridae caractérisés par leurs structures et leur mode d’infection à deux vitesses. Le génome de ces virus est constitué d'un ADN linéaire double brin, empaqueté dans une capside de forme icosaédrique, recouverte d’un tégument et d’une enveloppe composée de glycoprotéines (138). Ces virus sont parmi les parasites intracellulaires les mieux adaptés à leur hôte. En état de latence, leurs génomes adoptent une forme épisomale où ils expriment un minimum de gènes nécessaires à leur maintien ce qui diminue la reconnaissance de la cellule infectée par le système immunitaire. Lorsque les défenses de l’hôte s’affaiblissent, le virus se réactive et engendre une infection lytique de type cytocide. Le virus peut ainsi se multiplier et se transmettre tout en conservant une certaine pérennité de l’hôte.
1.2 Les trois sous-familles
Basé sur des critères biologiques et plus récemment sur l’analyse de la glycoprotéine conservée gH, on distingue 3 sous-familles d’herpèsvirus humain (HHV). Le tableau 1.1 en fait le résumé et présente les caractéristiques propres à chaque virus (68). La sous-famille des alphaherpesvirinae regroupe l’herpès simplex 1 et 2 (HSV-1 et -2) et le virus de la varicelle et du zona (VZV). Ce sont des virus neurotropiques à cycle réplicatif court. Les sites de latence de ces virus sont les ganglions neurosensitifs (1). Les événements de réactivation sont fréquents pour ces virus. Pour HSV-1 et 2 les épisodes de réactivation provoquent des symptômes cutanés sur les muqueuses près des ganglions touchés. Quant au VZV, la réactivation qu’on appelle le zona se traduit par une dermatose localisée près des nerfs liés aux ganglions infectés. La deuxième sous-famille, les gammaherpesvirinae, est constituée du virus Epstein-Barr (EBV) et de l’herpèsvirus de type 8. Ces virus sont lymphotropiques et ciblent les lymphocytes B et les cellules épithéliales. Ce sont les seuls herpèsvirus humains oncogènes, donc capables d’induire une transformation cellulaire.
Finalement, on trouve chez les betaherpesvirinae le cytomégalovirus (CMV), les herpèsvirus humains 6A et 6B ainsi que le HHV-7. En bref, ce sont des virus lymphotropiques à cycle réplicatif long. Leurs sites de latence sont : les glandes sécrétoires, le système lymphoréticulaire et les cellules épithéliales (1). Le CMV est associé tout comme l’EBV à la mononucléose infectieuse, mais aussi à des infections congénitales graves, surtout retrouvées dans les pays industrialisés. Le HHV-6B et le HHV-7 sont les agents étiologiques de la roséole, une maladie qui touche les jeunes enfants. Quant au HHV-6A, on ne connaît pas encore les conséquences de son infection, mais cette espèce a un tropisme marqué pour les cellules neuronales (88).
Tableau 1 : Les herpèsvirus humains et leurs caractéristiques
Nom Nom usuel Sous-famille Pathologies
associées
HHV-1 Herpès simplex 1 Alphaherpesvirinae Herpès buccal HHV-2 Herpès simplex 2 Alphaherpesvirinae Herpès génital HHV-3 Virus de la varicelle et du zona Alphaherpesvirinae Varicelle
Zona HHV-5 Cytomégalovirus Betaherpesvirinae Mononucléose et
infections congénitales HHV-6A Espèce A de l'herpèsvirus humain
de type 6
Betaherpesvirinae Aucune associée HHV-6B Espèce B de l'herpèsvirus humain
de type 6
Betaherpesvirinae Roséole HHV-7 Herpèsvirus humain de type 7 Betaherpesvirinae Roséole HHV-4 Virus Epstein-Barr Gammaherpesvirinae Mononucléose HHV-8 Herpèsvirus associé au sarcome de
Kaposi
Gammaherpesvirinae Sarcome de Kaposi
2. Genre des roseolovirus
2.1 Historique
Avant la découverte du HHV-6, le dernier herpèsvirus à avoir fait l’objet d’une découverte remontait aux années soixante, il s’agit du EBV. Près de 20 ans plus tard, grâce au développement des méthodes de culture cellulaire et l’engouement de la recherche pour le virus de l'immunodéficience humaine (VIH), une nouvelle espèce d’herpèsvirus humain fut identifiée. Le virus fut isolé à partir de cellules mononucléées du sang périphérique (PBMC) de patients atteints du VIH et de syndromes lymphoprolifératifs (110). Il fut d’abord nommé Human B Lymphotropic Virus (HBLV) à cause de son tropisme pour les lymphocytes B (59). Puis, les expériences subséquentes menées sur différents types cellulaires ont démontré que le virus infecte un large spectre de cellules et préférentiellement les lymphocytes T. Depuis, le HBLV a été renommé HHV-6, selon la nomenclature moderne.
2.2 Deux espèces du HHV-6
La souche GS provenant des États-Unis fut le premier isolat du HHV-6 à être découvert. Rapidement, d'autres souches, d’origines africaines, furent isolées de patients atteints du VIH. Les souches Z29 et U1102 proviennent respectivement de la république démocratique du Congo, anciennement appelée le Zaïre (77) et de l’Ouganda (36). En culture cellulaire, ces deux souches infectent les PBMC et les cellules mononucléées du sang de cordon (CBMC), mais n’infectent pas les mêmes lignées cellulaires (14, 36, 134). On découvre dans les années 1990 que les souches Z29 et U1102 représentent des virus proches, mais différents. En effet, une première équipe a démontré que la souche Z29 n’infecte pas les lignées cellulaires HSB2 et JJHAN pourtant infectées par le virus U1102 (3, 11, 74, 114, 134). De plus, des expériences menées avec des anticorps monoclonaux et des enzymes de restriction ont révélé que les deux souches étaient de types différents (134,10). Par la suite, d'autres expériences vinrent appuyer l'existance de deux sous-types de HHV-6, les types A et B (10). De fait, les souches U1102 et GS sont de type A, tandis que la souche Z29 est du type B. En général, ces deux variants partagent une homologie de séquence nucléotidique
de 90%. Certaines régions du génome, plus variables, semblent être responsables des divergences entre les deux variants. Ces divergences sont d’ordre : biologiques, immunologiques, épidémiologiques et moléculaires. Elles seront davantage décrites dans les prochaines sections. Dernièrement, la nomenclature a changée, il n’est plus question de variants, mais bien de deux espèces à part entière puisque les différences entre les deux virus son assez significatives (4).
2.3 Épidémiologie
HHV-6 est ubiquitaire et sa prévalence est de plus de 95%. On estime qu’avant l’âge de 6 ans, plus de 90% des jeunes enfants auront déjà des anticorps dirigés contre ce virus, indiquant qu’ils ont contracté le HHV-6 (50, 94, 113).La propagation du virus est associée au fait que les glandes salivaires sont un site de latence important pour le HHV-6B (38). Le relâchement du virus dans la salive, lors d’évènements de réactivation lui permettrait de se transmettre efficacement des parents à l’enfant (34). De façon beaucoup moins importante, la transmission pourrait se faire par infection congénitale chez 1% des naissances et elle serait asymptomatique (42). On connaît moins bien l’épidémiologie et la pathogénèse du HHV-6A. On sait toutefois que cette espèce serait davantage associée à l’infection du système nerveux central (SNC) (44). Par ailleurs, un mode de transmission vertical, c’est-à-dire de la mère à l’enfant, unique au HHV-6, serait en cause dans la majorité des transmissions congénitales. La section sur le HHV-6 intégré (ciHHV-6) en fera la description de façon détaillée.
2.4 Pathologies associées
Le HHV-6 est un pathogène opportuniste. L’infection primaire à HHV-6B touche habituellement les enfants avant l’âge de 2 ans. Le pic de l’infection est observé entre le 6ième et 9ième mois de vie, suite à la chute des anticorps maternels acquis à la naissance (45). La majorité des enfants qui sont touchés par le virus développent une maladie fébrile aigüe de courte durée. Le tier d’entre-eux développeront l’exanthème subit, mieux connue sous le nom de roséole ou sixième maladie. Cette maladie est caractérisée par une forte fièvre de 3 à 5 jours, suivi par une éruption cutanée localisée au niveau du tronc et du visage. Dans
moins de 10% des cas, la maladie peut entrainer des convulsions fébriles qui se résorbent rapidement (9). C’est en 1988 que la roséole a été associée pour la première fois au HHV-6 (135). Rappelons que la très grande majorité des cas de roséole sont dus à une infection par le HHV-6B bien que quelques cas semblent attribuables à une infection par le HHV-7. Dans un second temps, il est rare d’observer une infection primaire à HHV-6 chez l’adulte puisque dans plus de 90% des cas, l’infection a lieu en bas âge (50, 94, 113). De plus, comme tout les herpèsvirus le HHV-6 entre en latence après la primo-infection et persiste au sain de l’hôte tout au long de sa vie. Advenant une infection durant l’enfance, l’adulte pourrait être sujet à un évènement de réactivation du virus. Dans la majorité des cas, celui-ci se réactive généralement en période d’immunosuppression de l’hôte. Plus rarement, le virus peut se réactiver chez un hôte relativement immunocompétent, tel que chez les femmes enceintes où les défenses immunitaires sont moins efficaces (25). Les individus immunodéprimés les plus souvent touchés par la réactivation du virus sont ceux atteints par le VIH, ceux sous traitements de chimiothérapie pour combattre un cancer ou ceux ayant subit une transplantation d’organe. Principalement chez les transplantés de moelle osseuse, la réactivation du HHV-6 est fréquente et peut être très problématique. En effet, près de 50% des individus qui subissent une transplantation de möelle osseuse et de 20 à 30% des transplantations d’organes solides seront touchés par l’infection ou la réactivation du HHV-6, et ce, dans les 2 à 4 semaines après l’opération (137). L’infection d’un receveur à partir des tissus d’un donneur infecté est également possible. Autrement dit, un receveur de greffe séropositif pour HHV-6 peut être superinfecté par une souche distincte provenant du donneur (71). Les symptômes pouvant être associés sont : fièvre, éruptions cutanées, pneumonies, encéphalites et même le rejet de la greffe (28). Dans un autre ordre d’idée, on a associé plus rarement le HHV-6 à des maladies telles que le syndrome de l’immunodéficience acquise (SIDA) et la sclérose en plaques (SP). En 1988, des expériences ont démontré que le HHV-6 infecte les lymphocytes T CD4+, tout comme le VIH (63, 81). Depuis, le HHV-6 est mis en cause dans certaines infections opportunistes chez les patients atteints du VIH. Selon certains, il pourrait même contribuer à la progression du virus, mais plusieurs expériences semblent démontrer le contraire (2). Quant à l’association entre le HHV-6 et la sclérose en plaques (SP), elle est encore très controversée. La présence d’ADN viral dans le noyau d’oligodendrocytes près des plaques
de démyélinisation responsable du SP a créé un réel engouement pour cette piste de recherche (21). De plus, on associe surtout l’espèce A du HHV-6 à la sclérose en plaques (SP) (5), mais la controverse est encore vive et davantage de preuves expérimentales seront nécessaires. En bref, il n’est pas facile de déterminer les pathologies associées à HHV-6A et HHV-6B. Puisque ces virus sont ubiquitaires et qu’ils se réactivent, il devient très difficile de déterminer si les virus sont responsables des symptômes reliés à la maladie ou s’ils ne sont pas plutôt une conséquence d’une maladie déjà en place.
2.5 Structure du virus
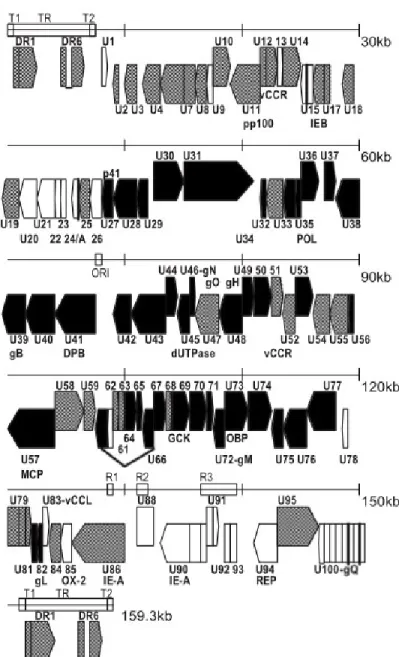
2.5.1 Le génomeLe génome viral est un ADN double brin linéaire de 160 kilo paires de bases (kpb) à 162 kpb selon la souche en question. Il est composé d’une section interne unique longue (UL) de 143 à 145 kpb, entrecoupée de 3 répétitions intermédiaires (R1-R3) contenant, entre autres, la région précoce-immédiate A (IE-A). La région unique est flanquée à chaque extrémité par les séquences directement répétées (DR) d’une longueur approximative de 9 kpb (10, 12, 136). Les séquences DR sont à leur tour flanquées de séquences télomériques répétées (TRS), l’une dite homologue (GGGTTA)n et l’autre dite hétérologue het(GGGTTA)n qui consistent en un patron de séquences télomériques irrégulières (93). Ces dernières auraient un rôle lors de la réplication virale et dans le maintien du génome sous la forme épisomale durant la latence (126). Dans le contexte de l’intégration chromosomique du HHV-6, ces séquences seraient nécessaires à l’insertion du génome viral dans les chromosomes cellulaires via la recombinaison homologue; mais ce sujet sera développé de façon plus approfondie dans la section sur l’intégration (138). Pour compléter la séquence génomique virale, on retrouve aux extrémités des séquences télomériques les séquences Pac1 et Pac2 nécessaires à l’empaquetage du génome viral dans la capside (41). La figure 1 ci-bas représente un schéma de la structure simplifiée du génome viral.
Figure 1: Schéma simplifié du génome viral du HHV-6.
Tirée de la revue Trempe et coll., 2011 (39)
D’un point de vue transcriptionnel, le HHV-6B compte 119 cadres de lecture ouverts (ORF), c’est neuf de plus que le HHV6-A; ils sont identifiés B1-B9 (10). Les séquences codantes peuvent être sur l’un des deux brins et sont situées dans la section UL (U1-U100) et dans les séquences DR (DR1-DR7). Certains gènes peuvent être épissés, mais en général la séquence génomique et l’ARNm sont identiques. Un schéma des différents ORF est représenté en figure 2.
Figure 2 : Génome de l’herpèsvirus humain de type 6 (HHV-6).
Tirée de Gompels et coll., 2006 (40)
Les membres de la famille des Herpesviridae ont sept blocs de gènes conservés codants principalement pour des protéines de structures, des enzymes du métabolisme des acides nucléiques et des protéines impliquées dans la réplication de l’ADN viral. La figure 2 représente en noir les gènes qui sont conservés chez les Herpesviridae. La configuration et l’ordre de ces blocs sont très variables d’une espèce à l’autre. Par exemple, HHV-6 et HHV-7, tous deux du genre roseolovirus, ont conservé la même organisation de leurs blocs géniques (106). Par ailleurs, un huitième bloc contenant les gènes de la famille US22 est
présent seulement chez les quatre membres des β-herpèsvirus humains et leurs fonctions seraient diverses. Ces gènes sont marqués en gris sur la figure 2. Enfin, il existe sept gènes spécifiques à HHV-6: U1, U9, U22, U61, U78, U83 et U94. Tandis que certains d’entres eux sont de petits gènes dont nous n’avons aucune évidence de leur expression, les transcrits U22, U83 et U94 sont impliqués dans les processus biologiques importants du virus. U22 code pour une glycoprotéine de l’enveloppe virale et U83 pour une chimiokine utile dans le recrutement des monocytes (3). Quant au gène U94, il est d’un grand intérêt pour ce projet, il sera décrit plus en details dans les prochaines sections.
Si la majorité des loci génomiques sont conservée à 95%, d’autres le sont beaucoup moins et permettent de différencier les espèces A et B (10). La région IE-1 a été décrite comme étant la plus variable entre les deux espèces, avec une homologie de séquence nucléique de 31% (37, 88). D’ailleurs, les sites d’épissage et de transcription des gènes dans le temps seraient différents et auraient un effet marqué sur la biologie du virus (10). Une autre partie du génome diffère entre les deux espèces, il s’agit de la section génique U97 à U100 qui code pour le complexe glycoprotéique à la surface de l’enveloppe virale et qui affecte donc le tropisme cellulaire. Dans leur ensemble, ces facteurs contribuent aux divergences biologiques entre les deux virus.
2.5.2 Nucléocapside, tégument et enveloppe
La structure des Herpesviridae est caractéristique de cette famille. En effet, malgré les différences biologiques, génomiques et pathologiques de ces virus, la structure du virion est très conservée d’une espèce à l’autre. On distingue 4 structures essentielles: le génome, la capside, le tégument et l’enveloppe. La capside consiste en une structure protéique de 162 capsomères de forme icosahédrique mesurant près de 90 à 110 nm de diamètre (12). Elle renferme le génome viral et le protège de l’environnement extérieur jusqu’au moment où le génome viral est relâché dans le cytoplasme de la cellule.
Autour de la capside se trouve le tégument, une structure diffuse de protéines cellulaires et virales acquises par la nucléocapside dès la sortie du noyau cellulaire. Lors de l’infection, le tégument contient les premières protéines effectrices. Par conséquent, ces protéines ne
dépendent pas de la transcription/traduction. Cette structure renferme plus de 40% de la masse protéique totale du virus, ce qui témoigne de l’importance structurelle du tégument (41).
Enfin, l’enveloppe représente la structure extérieure du virion. Elle est formée à partir de la membrane nucléaire de la cellule et d’autres structures membranaires intracellulaires. Elle inclut les protéines du complexe glycoprotéique souvent impliquées dans le tropisme cellulaire et l’infection virale.
2.6 Cycles viraux
Le HHV-6 infecte préférentiellement les lymphocytes T CD4+ activés, mais peut également infecter les cellules épithéliales et endothéliales, les astrocytes et les lymphocytes B (37). In vitro, la propagation virale se fait surtout grâce à des lignées de cellules T immortalisées choisies selon une infection à HHV-6A ou HHV-6B. Les cellules subissent un changement morphologique caractéristique qui consiste en leur gonflement et à l’apparition de cellules géantes multinucléées. Le mécanisme exact par lequel le virus pénètre dans la cellule reste incompris, mais certaines pistes sont suggérées. L’infection des cellules par le HHV-6 se fait via le CD46; un récepteur présent sur toutes les cellules nucléées (112). Ce récepteur est un modulateur de l’activité cellulaire lors de l’infection et est impliqué dans la voie du complément. Il est donc important dans la réponse de l’hôte face à l’infection (108). À la surface du virion, quatre glycoprotéines ont un rôle prédominant dans l’attachement du virion à la cellule: gH (U48), gL (U82), gQ1 et gQ2 (U100). L’ensemble de ces quatre glycoprotéines constitue le complexe viral nécessaire à l’interaction avec le CD46 (89). Il semble que la présence de ce récepteur à la surface des cellules soit insuffisante pour permettre l’infection. En ce sens, des expériences menées avec des anticorps bloquants pour les glycoprotéines gH et gB (U39) ont démontré leur importance dans le processus de fusion membranaire (37). Plusieurs données récentes révèlent que certains virus peuvent interagir avec les radeaux lipidiques lors de l’infection. Ce pourrait être le cas de l’infection à HHV-6, puisque l’attachement du virus au CD46 entraîne un déplacement du récepteur vers ces microdomaines, ce qui pourrait faciliter l’infection (120). Après l’attachement du virus à la surface cellulaire, un changement de
conformation s’opère et le virion entre dans la cellule par endocytose. Par comparaison avec le cytomégalovirus (CMV), on suppose que le virus pourrait traverser le cytoplasme via le réseau de microtubules et rejoindre les complexes du nucléopores afin que le génome soit libéré dans le noyau (28). À ce niveau, on distingue deux modes d’infections.
2.6.1 Cycle lytique
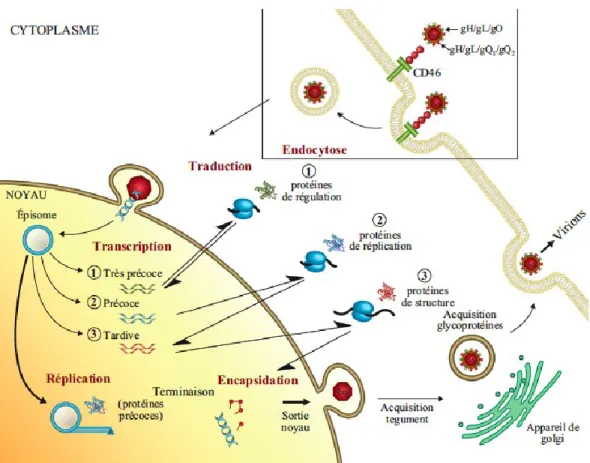
Après l’entrée au noyau, le génome viral prend la forme d’un épisome. Dès lors, il exprime ses gènes de façon séquentielle (α, β, γ) pour répliquer son génome et former de nouveaux virions. La figure 3 illustre le cycle réplicatif du HHV-6.
Figure 3: Cycle réplicatif du HHV-6
Pour ce faire, le virus utilise la machinerie cellulaire de transcription et de traduction, mais possède sa propre ADN polymérase. Les protéines très précoces (α) sont des facteurs de transcription ou des protéines régulatrices. Elles ont donc une influence importante sur la transcription séquentielle des protéines, mais aussi au niveau de la régulation de la réactivation et sur l’évasion du système immunitaire (121). Les protéines précoces (β) sont quant à elles nécessaires au métabolisme de l’ADN et à la réplication. Enfin, les protéines tardives (γ) sont des protéines structurales nécessaires à l’assemblage de la capside. Elles sont dépendantes de la polymérase virale (95).
La réplication du génome viral se fait selon la méthode du cercle roulant et fait appel à sept facteurs viraux différents. Une première protéine, Origin Binding Protein (OBP) lie l’origine de réplication et provoque l’ouverture de l’épisome (32, 55, 56). Un complexe protéique hélicase/primase tient cette ouverture stable et produit des séquences d’ARN guides pour la synthèse du brin lent (92). Pendant que l’ADN polymérase virale (pU38) synthétise l’ADN, la protéine majeure de liaison à l’ADN (U41) stabilise le simple brin (122). Du même coup, un facteur de processivité (U27) s’associe à l’ADN polymérase (75). Au fur et à mesure de la réplication, des concatémères se forment. Ceux-ci sont ensuite clivés via les sites Pac1 et Pac2 situés aux extrémités du génome et empaquetés (31). La nucléocapside maintenant mature migre vers le cytoplasme en empruntant une enveloppe temporaire à la membrane interne du noyau (126). Puis, elle perd cette première enveloppe et acquiert à l’appareil de golgi ou aux lamelles annelées le tégument et l’enveloppe parsemée de glycoprotéines virales. Par la suite, le virus rejoint la membrane plasmique via des vésicules de transport et sort par exocytose; de nouveaux virions sont formés et peuvent infecter à leur tour d’autres cellules. Le cycle réplicatif prend en moyenne 72 heures (13).
2.6.2 Cycle latent
L’une des caractéristiques des herpèsvirus humains est leur capacité à entrer en état de latence. Le HHV-6 diminue son expression génique au minimum, afin de réduire sa détection par le système immunitaire de l’hôte. On différencie deux types d’infection latente: la réplication chronique de faible niveau et l’infection latente vrai. La première se ferait surtout au niveau des glandes salivaires et du cerveau; elle serait impliquée dans la
transmission du virus via la salive (22, 38). La seconde, dite «vrai», n’implique pas de réplication à faible niveau. Elle consiste plutôt en un foyer viral qui peut se réactiver et produire une infection lytique dans un moment de vie où l’hôte subit un stress important ou se trouve dans un état d’immunosupression. Les cellules cibles sont les monocytes/macrophages (64) et les cellules progénitrices de la moëlle (78). Par ailleurs, certains rapportent avoir détecté de l’ADN viral à faible niveau dans les PBMC d’adultes sains (107) et dans plusieurs tissus (23); mais aucune preuve n’a permis de démontrer qu’il s’agissait de réactivation.
De la même façon que le CMV, HHV-6 exprime des transcrits de latence qui proviennent de la région IE-A du génome (66). Ceux-ci fortement exprimés avant la réactivation in vivo et in vitro seraient un élément déclencheur de la réactivation et de la transcription des gènes très précoces (65).
3. Intégration chromosomique
3.1 Découverte
L’intégration chromosomique de certains virus est un phénomène connu depuis bien longtemps. En effet, elle a d’abord été décrite chez les familles des rétrovirus et des virus associés aux adénovirus. Chez les herpèsvirus, le phénomène est beaucoup moins commun et sa découverte chez quelques espèces est assez récente. L’herpèsvirus de poulet responsable de la maladie de Marek (MDV) est en mesure de s’intégrer aux chromosomes des cellules de volailles et provoquer des lymphomes. Chez les herpèsvirus humain, le phénomène est plutôt rare. L’observation de l’intégration du virus Epstein-Barr (EBV) a déjà été réalisée dans des lignées de cellules transformées d’un lymphome de Burkitt, mais semble être pour l'instant que marginal (103). De façon beaucoup plus importante, il a été démontré dans les dernières années que le HHV-6 pouvait s’intégrer au génome cellulaire. La première observation provient du laboratoire du Dr. Luppi, qui démontra, dans les années 90, la présence d’ADN du HHV-6 intégré dans le génome cellulaire de trois patients atteints de trois maladies différentes : la maladie de Burkitt, le lymphome non-Hodgkinien et la sclérose en plaques (80). Depuis, de nombreux cas d’intégration chromosomique par
HHV-6 (ciHHV-6) ont été reportés. Les travaux de plusieurs laboratoires permettent maintenant d’estimer sa prévalence à 1% dans la population mondiale (52, 73, 100, 119, 131). Les espèces A et B du virus peuvent tous deux s’intégrer, mais les cas rapportés permettent d'estimer que la prévalence du ciHHV-6B est trois fois plus importante que celle du ciHHV-6A (42). Par conséquent, on attribue maintenant à HHV-6 la capacité indéniable de s’intégrer dans le génome cellulaire, et ce, avec la plus haute prévalence de tout les herpèsvirus humain. Enfin, la particularité de l’intégration à HHV-6 réside dans le fait que toutes les cellules d’un individu contiennent une copie du génome viral (130). Il advient alors, que la transmission du ciHHV-6 se fait par l’hérédité, selon les lois de Mendel.
3.2 Intégration de novo et transmission héréditaire
Tous les cas d’intégration observés jusqu’à maintenant sont la conséquence d’une transmission verticale (héréditaire). L’intégration de novo est très difficile à démontrer in
vivo car le nombre de cellules contenant le génome viral intégré est faible et donc trop
restreint pour être détectable par les méthodes diagnostiques. Ce qui laisse croire que ce type d’intégration est possible, provient d’une expérience menée par Arbuckle et son équipe, où ils ont démontré que le virus HHV-6 était sous forme intégrée après avoir infecté des cellules JJhan et HEK-293 (7). Ces auteurs ont d’ailleurs vérifié qu’il ne s’agissait pas de latence en s’assurant de l’absence de forme épisomale (7). Par contre, de plus amples recherches devront être faites à ce sujet pour démontrer véritablement la capacité du HHV-6 à intégrer le génome humain de novo. Malgré tout, la vraisemblance de l’intégration de novo va de soi puisque malgré le fait qu’on observe toujours la présence de la forme intégrée de par sa transmission verticale, il a bien fallu au départ qu’une cellule germinale soit permissive à l’intégration.
Aujourd’hui, même si la transmission germinale est bien acceptée par la communauté scientifique, ce concept n’a pas toujours fait l’unanimité. En premier, Daibata et coll.,1988 (27) ont démontré que le site d'intégration du HHV-6 était identique chez trois individus de la même famille. Contesté par certains, une intégration préférentielle sur certains loci spécifiques fut proposée comme explication aux observations (79). En 1999, une observation du laboratoire du Dr. Daibata vint appuyer fortement le concept de la
transmission héréditaire. Il observa chez un enfant dont les deux parents étaient intégrés par le HHV-6, que les sites d’intégrations (22q13 et 1q44) étaient d’une part celui de sa mère et d’autre part celui de son père (26). Quelques-uns ont réfuté cette observation en affirmant qu’il était possible qu’une infection active provenant des parents est eu lieu durant l’embryogenèse ou à la naissance de l’enfant (46). Pourtant, peu de doute persiste aujourd’hui quant au fait que le HHV-6 puisse se transmettre de manière héréditaire puisque plusieurs expériences ont appuyé ce concept (53, 130). Enfin, basé sur les expériences et observations faites jusqu’à maintenant, on suppose que la transmission peut s’effectuer sur au moins 3 générations (27).
3.3 Virus latent ou mort?
La fonction de l’intégration chromosomique est très controversée. Certains parlent d’une autre forme de latence, tandis que d’autres sont plus prudents et n’écartent pas la possibilité de la «mort» virale. La latence se veut un état de dormance où le virus minimise son expression génique et passe inaperçue face au système immunitaire. Après la latence, le virus doit aussi pouvoir en sortir et être en mesure de se répliquer. Or, très peu d'éléments nous permettent de conclure que l'intégration du génome est complète et que le virus peut se réactiver. L’équipe du Dr. Arbuckle a séquencée les jonctions virus-cellule d’un des sites d’intégration pour se rendre compte qu’une partie du DR droit était délétée de façon variable d’un individu à l’autre (7). Quant à l’autre extrémité, aucun résultat n’a été dévoilé. Par conséquent, le virus peut-il se réactiver même si lors de l’intégration il perd des bouts de séquence? Peut-être que les séquences perdues ne sont pas nécessaires au bon fonctionnement du virus? Par ailleurs, aucune preuve de réactivation in vivo n’a été publiée jusqu’à maintenant. En effet, démontrer que la réactivation provient bien de cellules intégrées et que le virus infectieux est le même que celui qui était intégré n’est pas une tâche facile. La réactivation a été observée en condition in vitro une seule fois. Pour ce faire, ils ont utilisé un agent chimique, la trichostatine A (inhibiteur de l’histone déacétylase) qui a permis d’augmenter le nombre de copies d’ADN du HHV-6A dans les lymphocytes T de porteurs du ciHHV-6. Ensuite, le virus aurait été transmis à des cellules MOLT-3 (7). Néanmoins, ce papier s’est attiré plusieurs critiques quant au choix des cellules à infecter et au gène utilisé pour associer le virus infectieux à celui intégré (90). En
effet, l'espèce virale utilisée pour cette étude n'est pas connue pour infecter les cellules MOLT-3. Puis, le gène U94 utilisé pour associer le virus intégré au virus infectieux est beaucoup trop conservé chez le HHV-6, le choix d'un gène plus variable aurait appuyé davantage les résultats.
3.4 Répercussions cliniques
Puisque la réactivation à partir de l’état latent n’a pas encore été démontrée, il est très difficile d’associer l’intégration chromosomique à une quelconque maladie. Par contre, il est facile d’imaginer qu’une telle insertion dans notre code génétique pourrait causer des tords. En ce sens, on trouve un taux plus élevé de ciHHV-6 chez des individus atteints de maladies diverses (~2%) que chez des donneurs de sang sains (0,85%) (98). Un des groupes de patients majoritairement touchés par le ciHHV-6 est celui des greffés de cellules hématopoïétiques (HSCT) et d’organes solides (SOT) (58, 60, 100). Une récente étude réalisée chez des transplantés de foie démontre qu’un taux plus élevé d’infections bactériennes et de rejet de greffe est associé aux patients intégrés par le ciHHV-6 (72). Dans un autre ordre d’idée, il est difficile de différencier l’infection active à l’intégration chromosomique, ce qui dans certains cas peut engendrer un mauvais diagnostique. Un individu intégré dans toutes les cellules de son corps obtient des valeurs très élevées d’ADN viral lorsque ses prélèvements sont soumis à des tests quantitatifs de polymérisation en chaine (qPCR). Les résultats peuvent laisser croire que l’individu souffre d’une infection active par le HHV-6. Or, l’administration d’antiviraux doit être fait seulement par nécessité, puisque leur administration peut être dangereuse chez des patients déjà durement atteints. En fait, chez des patients atteints du ciHHV-6, les antiviraux n’auront aucun effet, sauf celui de détériorer l'état du patient (51). Enfin, on en connait peu sur les réels enjeux du ciHHV-6 et de ses répercussions cliniques, d'autres études seront nécessaires afin de cerner le problème.
3.5 Mécanisme de l’intégration
Le mécanisme de l’intégration du HHV-6 est inconnu à ce jour. Évidemment on peut en apprendre beaucoup en étudiant d’autres virus qui peuvent s’intégrer. Avant de faire des analogies entre le virus responsable de la maladie de Marek (MDV) et le HHV-6, quelques concepts se doivent d’être détaillés. Il sera donc question de la recombinaison homologue et des séquences télomériques.
3.5.1 La recombinaison homologue
La recombinaison homologue (RH) consiste en un échange entre deux ADN de séquences en partie équivalentes. Cette permutation peut être réciproque ou non et dépend absolument d’une recombinase. En contexte cellulaire, la RH sert à maintenir l’intégrité du génome, mais peut être impliquée dans le remaniement des chromosomes en période de stress cellulaire. Par conséquent, elle est régulée par plusieurs facteurs cellulaires puisqu’elle peut entrainer des conséquences graves pour la cellule. En génie génétique elle est utilisée pour remplacer ou déléter un gène. D’un point de vue moléculaire, la RH se fait à partir d’une ouverture simple brin dans un des ADN. Cette ouverture est causée par un bris ou par une protéine à activité endonucléase. La recombinase, une protéine habilité à faire la recombinaison homologue, peut ainsi lier l’extrémité simple brin et s’apparier sur la séquence homologue de l’autre séquence d'ADN. S’ensuit un échange de séquences entre les deux ADN (118).
3.5.2 Les télomères
Les télomères sont les séquences aux extrémités des chromosomes. Ils consistent en une série de répétitions (TTAGGG)n d’une longueur variant de 5 à 15 kb dans les cellules somatiques (91). Ces différences sont observables entre les chromosomes et même entre les allèles. Au bout, le télomère est simple brin, riche en guanosines et est associé à plusieurs complexes de six protéines (TRF1, TRF2, POT1, TIN2, TPP1, RAP1). Les fonctions de ces protéines sont de contrôler la structure adoptée par l’extrémité des télomères, participer à la formation d’une boucle en T et de contrôler l’élongation par la télomérase (29). La télomérase est une ribonucléoprotéine qui allonge les télomères grâce à son activité de
transcription inverse et sa composante ribonucléique qui lui sert d'amorce (15). Dans une cellule différenciée l’activité télomérase est nulle et la longueur des télomères diminue à chaque division cellulaire (104). Lorsque la grandeur atteint un seuil critique, la cellule entre en sénescence ou meurt par apoptose. À l’inverse, dans 85% des cas de cancers, les cellules se protègent du raccourcissement des télomères en favorisant l'activité de la télomérase (115). L'autre 15% des cancers contourneraient le raccourcissement via le système alternatif d'élongation des télomères (ALT) qui dépend de la recombinaison homologue (20).
3.5.3 Virus de la maladie de Marek (MDV)
Le virus de la maladie de Marek est un alphaherpesvirus oncogène qui infecte les poulets. Sa forme intégrée aux chromosomes cellulaires est à l’origine de lymphome des cellules T. Par conséquent, il cause de gros problèmes en production avicole et nécessite que les élevages soient vaccinés. La structure de son génome est analogue aux herpèsvirus humains 1 et 2. Elle consiste en une section longue unique (UL) et une section courte unique (US), séparées par deux séquences internes répétées (IRL et IRS) et flanquées à leurs extrémités par des séquences répétées terminales (TRL et TRS) (19). De plus, chaque TR contient en son extrémité une séquence télomérique parfaite (CCCTAA)n, tandis qu’une séquence imparfaite de même nature cloisonne les deux séquences IR (62). Les séquences télomériques répétées du MDV sont homologues à celles des télomères cellulaires et pourraient être impliquées dans l’intégration virale. En fait, l’intégration se fait indépendamment du chromosome et le génome viral est inséré en entier (30). Par contre, il est préférentiellement localisé dans les régions télomériques des chromosomes (105). Une récente étude confirme qu’un délétant de séquences télomériques du MDV s’intègre moins fréquemment et ne cible plus de région en particulier (61).
Hypothèses pour HHV-6
Le MDV et le HHV-6 ont en commun quelques caractéristiques. D’abord, ils se distinguent des autres herpèsvirus par leur capacité à s’intégrer aux chromosomes cellulaires. Puis, tous deux ont des séquences télomériques dans leur génome. Certains ont émis l’hypothèse que le MDV pourrait s’intégrer grâce à la recombinaison homologue via les séquences
télomériques. Étant donné ces ressemblances, il est probable que le HHV-6 s’intègre via les séquences télomériques de son génome. Cette hypothèse est en partie soutenue par l’observation des cas reportés dans la littérature, où on observe que les sites d’intégration du HHV-6 se trouvent dans les régions télomériques (39).
4. La protéine Rep68 du AAV-2
4.1 Contexte
Selon notre hypothèse, le HHV-6 pourrait s’intégrer aux chromosomes cellulaires par recombinaison homologue via les séquences télomériques. Pour que la recombinaison se fasse, il est nécessaire d’avoir une recombinase. Quant aux virus HHV-6 et MDV, on a des preuves que l’intégration est possible. Il y a donc deux possibilités: soit que le virus code pour sa propre recombinase, soit il utilise la machinerie cellulaire. Mais la nature fait bien les choses et par conséquent l’existence du HHV-7 nous permet d’aller un peu plus loin dans cette réflexion. En effet, le HHV-7 est un très proche voisin du HHV-6, avec une homologie de séquence nucléique de 85% (28). Comme le HHV-6, il contient dans ses DR, des séquences télomériques. Pourtant, le HHV-7 est incapable, jusqu’à preuve du contraire, de s’intégrer aux chromosomes des cellules (43). Alors, pour que le HHV-6 puisse s’intégrer, il doit nécessairement coder pour une ou des protéines qui ont certaines activités biochimiques des recombinases. Le HHV-6 a la particularité d’avoir dans son génome l’origine de réplication fonctionnelle (ORF) U94, qu’il ne partage pas avec le HHV-7 (85). Ce pourrait-il que cet ORF soit un élément clé de l’intégration chromosomique? Des éléments de cette protéine laisse croire que ce serait possible. En effet, la protéine U94 du HHV-6 partage une homologie de séquence en acide aminés de 24% avec la protéine Rep du virus associé aux adénovirus de type 2 (AAV-2) (123).
4.2 Rôle de la protéine Rep du AAV-2
Le AAV-2 est un parvovirus qui nécessite la présence d’un adénovirus pour infecter les cellules. En son absence, le AAV-2 entre en latence en s’intégrant au génome de la cellule et se réactive lors d’une infection par le virus auxilliaire (49). Il est associé à aucune maladie chez l’être humain. Son génome est un ADN simple brin de 4700 nucléotides qui
contient l’ORF Rep et Cap. Rep est associé à la réplication virale et Cap est une protéine de structure. Le génome est aussi flanqué par des TR composés de séquences palindromiques répétées. Ces séquences lui permettent de former à ses extrémités une structure en T nécessaire à sa réplication et à la transcription de ses gènes. Le fonctionnement de la réplication et le rôle joué par la protéine Rep sont bien connus. D’abord, le site de liaison de Rep (RBE) est essentiel dans ce processus, il consiste en 3 répétitions du motif (GAGC) (83, 109). La liaison de Rep à ce site provoque le réarrangement spatial du génome et libère le site trs, endroit où Rep clive le génome grâce à son activité endonucléasique intrinsèque (54). La protéine se lie ensuite à la nouvelle extrémité 5’ ainsi créée et la réplication se fait grâce à la machinerie cellulaire à partir de l’extrémité 3’ (117). Comprendre le rôle de Rep dans la réplication du AAV-2 permet de jeter les bases du mécanisme de l’intégration. En ce sens, l’analyse des sites d’intégration du AAV-2 permet de conclure que le virus s’intègre préférentiellement à un locus bien précis du chromosome 19 (111). En effet, ce site est désigné AAVS1 et consiste en quatre répétitions du même motif retrouvé dans le site RBE des TR du virus (67). Or, la protéine Rep pourrait se lier simultanément au site situé sur le chromosome cellulaire et au RBE viral pour ainsi les rapprocher et permettre la réplication au niveau du chromosome (84). Les dernières étapes du procédé sont encore inconnues, mais on sait que le génome viral s’intègre sous la forme de concatémères et persiste dans la cellule hôte jusqu’à la réactivation.
D’un point de vue mécanistique, la protéine Rep semble avoir besoin de quatre activités pour permettre l’intégration du AAV-2 : la liaison à l’ADN simple et double brin et des activités hélicase et endonuléase ATP dépendantes (54, 97).Voici une brève description des domaines nécessaires à ces différentes activités.
4.3 Domaine d’interaction avec l’origine et hélicase SF3
Figure 4 : Domaines de la protéine Rep78
Adaptée de la revue Hickman et coll., 2005 (47)
L’ORF REP code pour 4 isoformes de la protéine Rep nommés selon leur poids moléculaire: Rep40, Rep52, Rep68, Rep78 (76). Les protéines Rep68/78 sont nécessaires à la réplication du génome de AAV-2 (16). La figure 4 illustre l’arrangement des domaines de la protéine Rep78. Elle se distingue de la protéine Rep68, par son domaine composé de doigts de zinc qui lui permet d’interagir avec des facteurs cellulaires (82). Dans un premier temps, le domaine d’interaction avec l’origine (en jaune) est responsable des activités de liaison à l’ADN simple brin et endonucléase de la protéine. La liaison à l’ADN est indépendante de l’ATP et se fait préférentiellement aux sites RBE des TR viraux ou sur le locus AAVS1 situé sur le chromosome 19 de l’humain. Par contre, l’apport en ATP semblerait stimuler la liaison à l’ADN par la protéine Rep68 (69, 96). Quant à l’activité endonucléasique, elle est ATP dépendante et coupe obligatoirement sur un ADN simple brin et à un site très particulier ; le site de résolution terminale (trs). Dans un second temps, le domaine responsable de l’activité hélicase (en vert pâle) est caractéristique de la super famille III (SF3). En effet, il existe 3 grandes familles d’hélicases: SF1, SF2 et SF3. La famille SF3 est unique aux petits virus à ARN ou ADN. Ces hélicases sont caractérisées par 4 motifs : Walker A, Walker B, B’ et C. Les deux premiers sont conservés dans les trois familles d’hélicases. Ces motifs sont responsables de l’activité ATPase de la protéine nécessaire à sa translocation sur l’ADN double brin (116). Quant au motif C, il est unique à cette famille. Il serait essentiel au mouvement de la protéine le long de l’ADN, puisqu’il contient une arginine qui capte le signal de phosphorylation du domaine A et engendre le changement de conformation de la protéine (47). Les connaissances actuelles sur la
structure quaternaire des hélicases SF3 suggèrent fortement la formation de complexes protéiques pour leur bon fonctionnement. Pour la Rep68/78, il a été démontré in vitro qu’il pouvait y avoir formation de complexes dépendant de la structure de l’ADN (82). Enfin, une autre particularité des hélicases SF3 est leur capacité à accomplir plusieurs tâches telles que le remodelage de l'origine de réplication et la réplication.
5. Protéine U94 du HHV-6
5.1 Unique à HHV-6
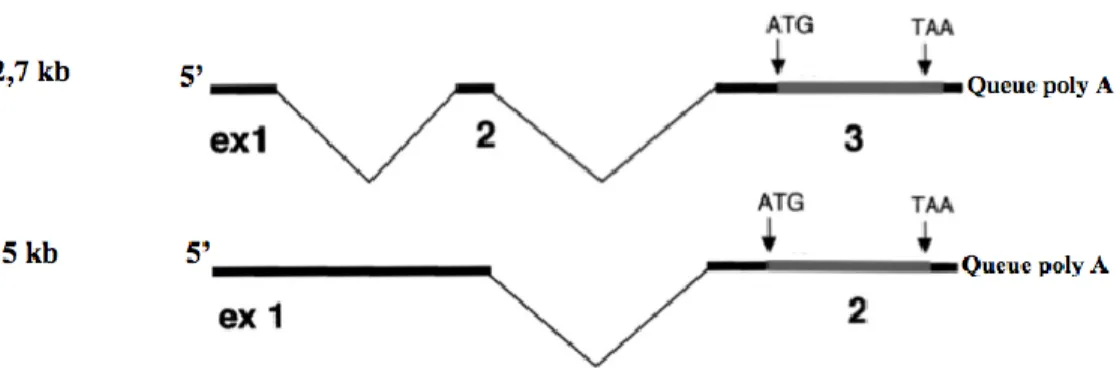
Le gène U94 est unique à HHV-6. Tel que mentionné, l’ORF U94 est même absent du génome du HHV-7, l’espèce la plus rapprochée du HHV-6 (85). Brièvement, ce gène est situé à l’extrémité droite de la région UL du génome du HHV-6 et il est codé par 1473 pb sur le brin complémentaire. Lors de la transcription, on détecte 2 transcrits émanant de cette région : l’un de 5 kb beaucoup moins fréquent et l’autre de 2,7 kb qui représente la très grande majorité des transcrits produits (87). Tel que représenté à la figure 5, l’ARNm le plus fréquent (2,7 kb) contient 3 exons et deux introns épissés (102). Les deux premiers exons ne codent pour aucune séquence protéique, tandis que l’exon 3 code pour la protéine U94 de 490 acides aminés; celle décrite dans la littérature et dont il sera question ici (87).
Figure 5 : Transcrits de l’ORF U94 du HHV-6
Figure adaptée de Mori et coll., 2000 (87)
isolats. En effet, la séquence génomique et protéique de U94 seraient identique à près de 97% entre le HHV-6A et HHV-6B (35, 57). De plus, la divergence entre les souches de même espèce serait de moins de 0,2 à 0,6 % ce qui suggère que cette protéine joue un rôle biologique prédominent (102). L’équipe du Dr. Mori, qui a identifiée les transcrits, a aussi vérifié la localisation cellulaire de la protéine U94. 24 heures après l’infection de cellules MT-4 par le HHV-6B, la protéine U94 est localisée au niveau du noyau. Avec le temps, soit 48 heures après l’infection, la protéine est cytoplasmique (87). Quant à son type temporel, les résultats diffèrent selon la technique utilisée. Certains affirme qu’elle est une protéine très précoce (86, 95), tandis que d’autres concluent qu’elle est précoce (127).
5.2 U94 et Rep68/78
Les herpèsvirus peuvent jouer le rôle de virus auxilliaires pour permettre l’infection par le AAV-2 (48). En ce sens, le Dr. Thomson affirmait en 1991 que la protéine U94 serait le résultat d’un transfert direct entre le AAV-2 et le HHV-6 (123). Selon lui, l’homologie de séquence en acides aminés de 24% avec la protéine Rep68/78 suggère que ce gain de gène n’est pas le résultat d’un ancêtre commun ou d’une convergence lors de l’évolution. Ce serait plutôt l’infection d’une même cellule par les deux virus qui aurait permis au HHV-6 d’obtenir par recombinaison non-homologue le gène Rep. Par la suite, l’une de ses expériences vint appuyer fortement sa proposition lorsqu’il démontra qu’un mutant du AAV-2 délétant du gène Rep pouvait, grâce à l’apport de U94 en trans, regagner la capacité de se répliquer (124).
La comparaison des séquences homologues entre ces deux protéines révèle l’importance des domaines et acides aminés conservées. Tel que mentionné, la protéine Rep68/78 possède quatre activités nécessaires à la réplication et par conséquent à l’intégration chromosomique : la liaison à l’ADN simple et double brin, l’activité ATPase, l’activité hélicase et l’activité endonucléase spécifique. Des expériences de mutagenèse sur la protéine Rep ont permis d’identifier les domaines et résidus nécessaires aux activités (128, 129). Par conséquent, nous avons aligné les séquences des protéines Rep68, U94A et U94B (figure 6) et observé les homologies. On s’aperçoit assez rapidement que U94 possède théoriquement les domaines nécessaires pour avoir les mêmes activités biochimiques que la
protéine Rep68 du AAV-2. D’ailleurs, il a été démontré que la protéine U94 est en mesure de lier l’ADN simple brin et que cette activité serait plus évidente avec l’ajout d’extrait cellulaire à la réaction in vitro (33).
Figure 6 : Alignement des séquences en acides aminés de Rep68 et U94A
Alignement des séquences en acides aminés des protéines Rep68 du AAV-2, U94A et U94B du HHV-6. Les acides aminés nécessaires aux activités biochimiques de la protéine Rep68 (liaison à l'ADN, ATPase, hélicase et endonucléase) sont en rouge. Les rectangles noir délimitent les différents domaines responsables de l'activité hélicase de la protéine Rep68. L'arginine du site actif de l'activité hélicase est en bleu. Les acides aminés retrouvés dans la séquence des trois protéines sont marqués d'un astérisque (*), les acides aminés conservés, d'un deux points (:) et les semi-conservés, d'un point (.).
Y164F Mutant endonucléase GK343AH Mutant hélicase K395A Mutant de liaison
5.3 Latence et transactivation
Mis à part le rôle potentiel de la protéine U94 dans l’intégration chromosomique, on lui reconnait déjà un rôle dans la latence virale. Ainsi, lors de l'infection d'une cellule in vitro l'expression de transcrits d'U94 pendant le cycle lytique est très faible (102). À l'inverse, seuls les transcrits de l'ORF U94 sont présents lors d'une infection latente de PBMC d’individus sains (107). La réplication du HHV-6 et des autres β-herpèsvirus est inhibée en transfectant des cellules avec un vecteur qui exprime U94 (17). Pour mieux comprendre l’effet de la protéine U94 sur la réplication virale, le laboratoire du Dr. Di luca a infecté une lignée de cellules lymphoïdes exprimant de façon stable la protéine U94. Les cellules permissives à l’infection n’ont révélé aucun effet cytopathique (107).
Bref, la protéine U94 joue un rôle important dans la latence et semble être impliquée dans la régulation de la réplication virale. Ceci suggère que U94 serait un transactivateur important chez le HHV-6. En effet, dans des cultures in vitro de fibroblastes la protéine U94 peut lier le promoteur LTR du virus de l’immunodéficience humaine de type 1 et l’activer (124). U94 influence également différents éléments cellulaires puisqu'il a été démontré que la protéine U94 est capable de lier le facteur de transcription humain TATA
Binding Protein (hTBP) pour ainsi moduler la réponse cellulaire (87) et supprimer la
transformation cellulaire causée par l’oncogène H-ras (6).
6. Hypothèse et objectifs de recherche
6.1 Hypothèse
Il a été observé depuis plus d’une vingtaine d’années que le HHV-6 est en mesure de s’intégrer dans les chromosomes cellulaires. Depuis, plusieurs cas raportés permettent d’affirmer que l’intégration s’effectue dans les régions subtélomériques/télomériques des chromosomes. Le virus de la maladie de Marek, un α-herpèsvirus chez le poulet, s’intègre lui aussi au niveau des télomères probablement via les séquences télomériques répétées de son génome. Ces mêmes séquences sont présentes dans les régions directement répétées du génome du HHV-6. Le AAV-2, quant à lui, adopte la forme d’un provirus en absence de virus auxilliaire nécessaire pour le déclenchement de son cycle lytique. Il partage avec le
HHV-6 une homologie de séquence en acides aminés de 24% pour la protéine Rep/U94. Cette protéine, qui est associée à l’intégration du AAV-2, a la capacité de cibler le locus de l’insertion via une recombinaison non-homologue. À partir de ces connaissances et du rôle que joue U94 dans la latence du HHV-6, nous émettons l’hypothèse que la protéine U94 du HHV-6 est impliquée dans le processus de l’intégration chromosomique. La démonstration que la protéine U94 partage les mêmes activités biochimiques que la protéine Rep du AAV-2 permettrait d’appuyer fortement cette hypothèse. Éventuellement des tests d’intégration in vitro à l’aide d’un mutant délétant de U94 seraient nécessaires afin de bien cerner la problématique. Enfin, ces observations serviraient à mieux comprendre l’intégration chromosomique du HHV-6.
6.2 Objectif principal
L’objectif du projet est de vérifier si la protéine U94 possède les activités biochimiques de la protéine Rep essentielles à l’intégration du AAV-2. Ces activités sont la liaison à l’ADN simple brin et double brin, l’ATPase, l’hélicase et l’endonucléase. Pour ce faire, des tests in
vitro seront faits dans un premier temps en radioactivité, puis dans un second temps à l’aide
d’appareils spécialisés.
6.3 Objectifs spécifiques
Les objectifs spécifiques sont:
1- De produire la protéine U94 en fusion avec une étiquette immunogénique (Flag) grâce à une construction dans un vecteur eucaryote pour permettre son expression dans des lignées cellulaires.
2- Produire une protéine de fusion entre la protéine qui lie le maltose (MBP) et U94 pour obtenir une protéine soluble dans un système bactérien et faciliter sa purification.
3- Élaborer des mutants de la protéine U94 par mutagenèse dirigée pour les quatre activités biochimiques.
4- Déterminer la localisation cellulaire de la protéine U94 par immunofluorescence.
5- Vérifier l’activité de liaison à l’ADN simple et double brin par des essais de retards sur gels. Étudier la spécificité de la liaison pour un type d'ADN avec le Proteon™ XPR36.
6- Effectuer des tests d’ATPase pour vérifier la capacité de U94 à hydrolyser l'ATP.
7- Immunoprécipiter la protéine FLAG-U94 pour de futurs essais d'immunoprécipitation sur la chromatine et de co-immunoprécipitation avec des protéines partenaires potentiels d'U94.
8- Séquencer et comparer le gène U94 de souches virales de laboratoire et de souches virales intégrées.