HAL Id: hal-02823020
https://hal.inrae.fr/hal-02823020
Submitted on 6 Jun 2020
HAL is a multi-disciplinary open access
archive for the deposit and dissemination of sci-entific research documents, whether they are pub-lished or not. The documents may come from teaching and research institutions in France or abroad, or from public or private research centers.
L’archive ouverte pluridisciplinaire HAL, est destinée au dépôt et à la diffusion de documents scientifiques de niveau recherche, publiés ou non, émanant des établissements d’enseignement et de recherche français ou étrangers, des laboratoires publics ou privés.
L’évolution du pouvoir pathogène de la bactérie
Pseudomonas syringae en présence de microalgues
Valentin Debreyne
To cite this version:
Valentin Debreyne. L’évolution du pouvoir pathogène de la bactérie Pseudomonas syringae en présence de microalgues. [Stage] Institut Supérieur d’Agriculture de Lille (Groupe ISA), Lille, FRA. 2010, 52 p. �hal-02823020�
Institut Supérieur d’Agriculture 48 Boulevard Vauban
59046 Lille Cedex
Institut National de Recherche Agronomique Unité de Pathologie Végétale – BP 94
Domaine Saint Maurice 84143 Montfavet Cedex
L’évolution du pouvoir pathogène de la bactérie
Pseudomonas syringae en présence de microalgues
Rapport de Stage Assistant Ingénieur
DEBREYNE Valentin
Novembre 2010
4
èmeannée
RÉSUMÉ
Résumé :
Située au cœur du bassin français le plus productif de melons, l’équipe de bactériologie de l’INRA d’Avignon a découvert, que le cycle de vie de Pseudomonas syringae, bactérie responsable d’une sévère bactériose sur cette plante, se déroulait aussi en dehors des agro-systèmes. Ces études ont montré que les populations de P. syringae se retrouvent tout au long du cycle de l’eau douce, y compris dans les biofilms se développant dans les cours d’eau. Or, les microalgues dont certaines sont relativement proches des plantes, sont l’une des composantes majoritaires de ces biofilms : P. syringae étant une bactérie phytopathogène au large spectre d’hôte, il pourrait exister des interactions entre les deux types de microorganismes, bactéries/microalgues. Ces interactions pourraient jouer un rôle dans l’épidémiologie des maladies causées par cette bactérie.
Mais y a t-il réellement des interactions entre P. syringae et certaines microalgues au sein des biofilms ? Si c’est le cas, les caractères pathogènes de ces bactéries jouent-ils un rôle dans ces interactions ?
La présente étude a eu pour but d’apporter une première réponse à ces interrogations en postulant que, si les interactions existent, elles se traduiront par une modification de la croissance microbienne en présence du partenaire d’interaction. Pour cela, un protocole a été mis en place afin de suivre puis de comparer les évolutions des populations de bactéries et de microalgues au sein de culture pure et de co-culture : les populations microbiennes sont dénombrées au début puis à la fin de la culture grâce à la technique de suspension-dilution- étalement pour les bactéries et, grâce à un compteur de cellules pour les microalgues. Ce protocole a ensuite été testé sur une trentaine de souches de P. syringae et sur la souche modèle 137 de Chlamydomonas reinhardtii. Un accroissement des populations bactériennes plus important au sein des co-cultures qu’au sein des cultures pures a été observé. Inversement, au contact des bactéries, les populations de microalgues n’ont pas atteint les niveaux des cultures pures. Pour finir, les caractères pathogènes étudiés ne semblent pas jouer un rôle dans ces interactions.
Mots clés : Pseudomonas syringae, biofilm, microalgue, Chlamydomonas reinhardtii, interaction, phytopathogène.
REMERCIEMENTS
Je tiens à remercier tout particulièrement Odile Berge qui m’a consacré beaucoup de temps durant ce stage et avec qui j’ai énormément appris tant d’un point de vue technique que d’un point de vue humain.
Je remercie également Catherine Glaux ainsi que Caroline Monteil pour leur disponibilité ainsi que pour toutes les explications qu’elles m’ont fournies sur leur travail.
Merci également à Caroline Guilbaud pour l’aide qu’elle m’a apportée tant au niveau de la préparation du matériel que du nettoyage de celui-ci.
De manière plus générale, merci à toute l’équipe de pathologie végétale pour leur accueil, leur gentillesse et leur disponibilité qui ont permis de rendre ce stage aussi bien intéressant qu’enrichissant.
SOMMAIRE
INTRODUCTION...1
LE CADRE DE L’ETUDE ... 2
1. L’INSTITUT NATIONAL DE RECHERCHE AGRONOMIQUE (INRA)... 2
a) Historique...2
b) Missions actuellement menées... 2
c) Quelques chiffres ... 3
2. LE CENTRE INRA D’AVIGNON ... 3
3. L’UNITE DE PATHOLOGIE VEGETALE... 4
4. L’EQUIPE DE BACTERIOLOGIE... 4
LE CONTEXTE ET LA PROBLEMATIQUE... 5
1. PSEUDOMONAS SYRINGAE : UNE BACTERIE PHYTOPATHOGENE... 6
2. PSEUDOMONAS SYRINGAE : UNE BACTERIE DE L’ENVIRONNEMENT... 7
3. LES INTERACTIONS DANS LES BIOFILMS... 8
MATERIELS ET METHODES ... 10
1. CHOIX DES SOUCHES... 10
a) Les souches bactériennes ... 10
b) La souche de microalgue ... 11
2. LA CULTURE ET LE DENOMBREMENT DES MICROORGANISMES ... 11
a) Milieux de culture... 11
b) Dénombrements « classiques » des microorganismes ... 12
c) Dénombrement des microorganismes au compteur de cellules... 13
3. DETERMINATION DU PROTOCOLE DE CO-CULTURE BACTERIES / MICROALGUES... 14
a) Détermination des paramètres de croissance ...14
b) Protocole du test de co-culture... 15
c) Plan d’expérience 16 4. TRAITEMENT DES DONNEES ...16
PRESENTATION ET ANALYSE DES RESULTATS... 17
1. MISE AU POINT D’UN PROTOCOLE D’UTILISATION DU COMPTEUR DE CELLULES... 17
a) Le dénombrement des microalgues ... 17
b) Le dénombrement des bactéries... 18
c) Protocole retenu ... 18
2. L’INFLUENCE DES BACTERIES SUR LES POPULATIONS D’ALGUES... 20
a) Au niveau du nombre de microalgues ...20
b) Au niveau du diamètre des microalgues...21
3. L’INFLUENCE DES MICROALGUES SUR LE NIVEAU DES POPULATIONS BACTERIENNES ... 22
4. COMPARAISON DES SOUCHES BACTERIENNES... 25
a) Homogénéité des manipulations ...25
b) Variations de la vitesse de croissance des bactéries... 26
DISCUSSION ... 36
CONCLUSION ET PERSPECTIVES... 38
RÉFÉRENCES BIBLIOGRAPHIQUES ... 39
GLOSSAIRE ... 41
LISTE DES ANNEXES ... 42
TABLE DES FIGURES... 47
INTRODUCTION
En agriculture, les pertes de récolte dues aux ravageurs ont toujours été importantes : globalement en 2009, l’organisation des Nations Unies pour l’alimentation et l’agriculture (FAO) les estime à plus de cinquante pour cent de la production agricole potentielle. Toujours selon ce même organisme, les insectes nuisibles seraient responsables de la destruction de quinze pour cent des cultures, les agents pathogènes et les adventices de treize pour cent chacun, et les infestations après récolte de dix pour cent.
Causant ainsi près du quart des pertes, les agents pathogènes (virus, champignons et bactéries) ont été fortement étudiés afin de contrôler voir d’éviter certaines épidémies. Jusqu’à très récemment, les études portant sur ces agents pathogènes ont été le plus souvent focalisées sur un contexte agricole alors qu’en ce qui concerne les pathogènes humains, les biologistes ont depuis longtemps reconnu que certains agents pouvaient vivre et prospérer dans des habitats non hôtes. Ces « pathogènes environnementaux » évoluent et se diversifient dans des milieux très divers indépendamment de leurs activités de pathogènes chez l’homme. Ceci a conduit à des pratiques telles que l’assainissement des eaux, la surveillance des populations d’animaux sauvages ou le nettoyage des systèmes de ventilation… Ce n’est que depuis quelques années qu’il a été montré que des agents phytopathogènes peuvent aussi résider et donc évoluer dans des niches écologiques en dehors de l’agriculture.
Pseudomonas syringae, une bactérie phytopathogène, représente bien cette situation. En effet, elle a été très étudiée en agriculture puisqu’elle cause des maladies sur une grande gamme de plantes : des arbres fruitiers (le chancre du marronnier ou du cerisier), des plantes maraîchères (tomate, concombre, haricot, melon…) et également des cultures de céréales. Les études concernant la biologie, l’écologie et la génétique de cette bactérie sont donc nombreuses mais pour la plupart en relation avec les plantes cultivées malades alors que Pseudomonas syringae est présente dans des habitats divers : le sol, l’air, les eaux douces et même dans les nuages.
Cette bactérie se retrouve donc dans de nombreux habitats naturels du cycle de l’eau et, en particulier, dans les biofilms de rivière. Un biofilm est une communauté de microorganismes qui se développe à la surface d’un solide dans une matrice. Ces biofilms sont le lieu d’importantes interactions entre les différents organismes présents et, notamment entre Pseudomonas syringae et des microorganismes qui se retrouvent très fréquemment dans les biofilms de rivière : les microalgues.
Mais y a-t-il réellement des interactions entre les bactéries Pseudomonas syringae et certaines microalgues au sein des biofilms ? Et si c’est le cas, les caractères pathogènes de ces bactéries jouent-ils un rôle dans ces interactions ?
Pour répondre à ces interrogations, nous verrons tout d’abord le lieu où s’est déroulée cette étude. Puis, nous expliciterons clairement la problématique et le contexte dans lequel ce travail a été initié. Nous passerons ensuite à une présentation du matériel et des méthodes utilisés avant d’exposer les résultats obtenus ainsi que leur analyse. Dans un dernier temps, nous verrons comment ces résultats peuvent être utilisés dans un cadre plus large que celui de l’étude.
LE CADRE DE L’ETUDE
1. L’Institut National de Recherche Agronomique (INRA)
a) Historique
Créé en 1946, dans le contexte de pénurie alimentaire de l’après-guerre, l’INRA avait pour mission initiale de favoriser l’essor de l’agriculture française afin que notre pays devienne autosuffisant d’un point de vue alimentaire. Pour cela, l’INRA a participé à la révolution agricole française : en mettant la science et la technologie au service de l’agriculture, l’Institut National de Recherche Agronomique a permis, parmi d’autres organismes de recherche, d’améliorer les techniques de production ainsi que la sélection animale et végétale. Ceci a aboutit à une hausse de la productivité : la France est devenue
Source : www.INRA.fr
autosuffisante à la fin des années 1960.
Dans les années 70, notre pays devient excédentaire et va donc exporter des produits issus de l’agriculture y compris des aliments. Les recherches de l’INRA vont donc s’élargir à la transformation des matières premières agricoles en aliments. Celles-ci vont aboutir à l’élaboration de différents procédés agroalimentaires comme l’ultrafiltration du lait par exemple. Grâce à ces recherches, la France deviendra le premier pays exportateur de produits agroalimentaires.
Les années 1980-1990 voient émerger de nouvelles demandes. On passe d’un système où l’on veut produire en quantité à un système où l’on veut des produits de qualité. Dans cette perspective, la santé humaine entre au cœur des enjeux : les problématiques de recherche concernent désormais l’alimentation et non plus uniquement les aliments eux-mêmes. L’INRA participe alors à la recherche sur des variétés adaptées aux demandes des consommateurs mais aussi à des techniques de lutte contre
certaines contaminations microbiennes. De plus, avec le développement du mouvement écologiste, l’environnement devient un nouvel enjeu : on veut continuer à produire mais en préservant l’environnement c’est-à-dire en ayant le minimum d’impacts sur celui-ci. C’est pourquoi des problématiques de recherche environnementales voient le jour dans ces années là.
Durant les années 2000, l’INRA développe des Source : www.INRA.fr
recherches interdisciplinaires qui permettent d’intégrer des critères de durabilité. Les enjeux sont de produire une alimentation saine et équilibrée tout en préservant l’environnement.
Mais quelles sont les missions actuelles de l’Institut National de Recherche Agronomique ?
b) Missions actuellement menées
Certaines missions menées actuellement par l’INRA restent les mêmes que lors de sa création au lendemain de la seconde guerre mondiale, d’autres ont émergé plus récemment.
En tant qu’établissement de recherche public, la mission principale de l’INRA est de produire et diffuser des connaissances scientifiques. Dans cette perspective, l’institut doit innover et proposer de nouveaux savoir-faire afin de répondre à de nouvelles problématiques sociétales concernant l’agriculture, l’environnement, l’agroalimentaire et le développement durable.
- la sécurité alimentaire mondiale : mettre fin aux problèmes de crises alimentaires
- la sûreté sanitaire des aliments : produire des aliments sains pour l’homme
- la localisation des activités agricoles face à la mobilité économique
- la gestion des ressources naturelles et la préservation de l’environnement
- les projets et les modèles de développement économique et social.
Pour cela, l’institut souhaite continuer à participer à l’évolution et au renforcement du dispositif de recherche français et européen qui permet d’harmoniser la recherche entre les différents pays.
c) Quelques chiffres
La stratégie menée au fil des années par l’INRA lui a permis d’aboutir à des résultats concrets :
En 2009, l’Institut National de Recherche Agronomique (INRA) est :
• 1er institut de recherche européen dans les domaines de l’agriculture, de l’alimentation
et de l’environnement
• 2ème institut mondial pour ses publications scientifiques en sciences agricoles, sciences
des plantes et de l’animal
• 2ème institut de recherche publique française
Parallèlement aux missions menées depuis sa création, l’INRA s’est développé dans toute la France. On compte aujourd’hui 21 centres régionaux et plus de 150 sites répartis en France métropolitaine et outre-mer. Ce développement a permis à chaque centre de se préoccuper de problématiques régionales, donc d’être au cœur des enjeux et au plus près des personnes concernées par les recherches. Par exemple, l’un des axes de recherche de l’INRA Bordeaux concerne la vigne et le vin, l’INRA Nancy se préoccupe des forêts, l’INRA Avignon axe ses recherches sur le maraîchage…
C’est d’ailleurs dans ce dernier centre, au cœur de la région Provence-Alpes Côte d’Azur, que l’étude qui sera décrite dans la suite de ce rapport a été effectuée.
2. Le centre INRA d’Avignon
Fondé en 1953, ce centre compte actuellement 580 agents permanents et 400 agents temporaires par an (dont 100 doctorants) répartis en 27 unités. Les moyens financiers s’élèvent à 35 millions d’euros salaires compris.
Les recherches du centre d’Avignon s’effectuent au sein de trois pôles :
- la production horticole intégrée
- l’adaptation aux changements climatiques
- la santé des plantes.
C’est sur cette dernière thématique que nous nous intéresserons plus particulièrement dans ce rapport, les recherches ayant été faites au sein de l’unité de pathologie végétale.
3. L’unité de pathologie végétale
L’unité de recherche de pathologie végétale (UR 407) est implantée dans le domaine de Saint-Maurice à Montfavet (en bordure d’Avignon). Elle comprend 42 agents permanents organisés en trois équipes de recherche : bactériologie, mycologie et virologie.
Tout comme le centre INRA d’Avignon, l’unité a développé ses compétences autour des principales productions de la région puisqu’elle s’intéresse aux maladies des cultures maraîchères méditerranéennes. Le principal objectif de cette unité est de développer des méthodes de luttes raisonnées et efficaces dans un contexte de production agronomique durable et de qualité. Les méthodes de lutte préventives définies par l’unité s’appuient sur :
- un diagnostic rapide, simple et fiable
- la gestion durable des résistances variétales
- l’utilisation d’agents de lutte biologique
- la prévision des maladies par la compréhension de la biologie et l’évolution des
pathogènes ainsi que de l’épidémiologie des maladies.
C’est principalement sur cette dernière thématique que travaille l’une des trois équipes de l’unité : l’équipe de bactériologie dans laquelle des recherches permettant de répondre à la problématique de ce rapport ont été effectuées.
4. L’équipe de bactériologie
Après de sévères épidémies bactériennes sur le melon, un groupe de travail national est créé en 1997. L’équipe de bactériologie de l’INRA d’Avignon en fait partie, elle va s’intéresser à cette maladie et identifier Pseudomonas syringae comme l’agent responsable de la bactériose. Depuis, l’équipe étudie exclusivement cette bactérie : elle propose un cycle de vie de ce microorganisme phytopathogène en interaction avec le cycle de l’eau et cela, aussi bien dans des écosystèmes agricoles que non agricoles. L’objectif à long terme est de connaître toute la gamme de résistance au stress de la bactérie étudiée, ceci afin de lutter contre des maladies telles que la bactériose du melon.
LE CONTEXTE ET LA PROBLEMATIQUE
En 2009, 301 724 tonnes de melon ont été produites en France. Cette production est répartie sur 14 749 hectares en 2010 et provient principalement de trois secteurs : le Centre- ouest (Pays de la Loire et Poitou-Charentes), le Sud-ouest (Aquitaine et Midi-Pyrénées) et le Sud-est (Languedoc-Roussillon et Provence-Côte d’Azur) qui représentaient à eux seuls 93,1% de la production française en 2009 (données Agreste 2009-2010).
Or depuis le début des années 1990, ces différentes régions de production sont régulièrement touchées par une épidémie plus ou moins sévère selon les années. La maladie a d’abord touché les régions de production du Centre-ouest et du Sud-ouest, notamment lors des campagnes 1995, 1996 et 1997, si bien qu’en 1997, les conseils régionaux du Tarn-et- Garonne et du Lot ont déclaré officiellement l’état de catastrophe naturelle pour les parcelles malades. L’épidémie s’est ensuite propagée dans tous les bassins de production français sans exception (Mention et al., 2004).
Situé au cœur du bassin de production de melons le plus productif au niveau national (le Sud-Est représentait plus de 40% de la production française en 2009), l’INRA Avignon s’est intéressé à cette maladie. En collaboration avec le Service Régional de la Protection des Végétaux (S.R.P.V.), l’INRA a identifié l’agent pathogène de cette bactériose comme étant Pseudomonas syringae.
Dans un premier temps, les recherches ont porté sur la bactérie présente sur les plants de culture malades mais l’étude des pathogènes dans cet unique contexte permet d’aborder seulement une partie du cycle de vie de ces organismes. Ces études ont toutefois permis d’avancer dans la compréhension des maladies et de ce qui les détermine mais des inconnues demeurent. Pour P. syringae, l’étude des souches pathogènes isolées de plantes malades ne suffit pas à expliquer ce qui détermine qu’une souche va attaquer une plante donnée. Il reste donc des facteurs à élucider (Guttman et al., 2006).
Pour la plupart des pathogènes humains, les études ont déjà porté sur leur écologie en milieu naturel. Ceci afin de comprendre certains de leurs caractères utiles à la pathologie humaine (Cangelosi et al., 2004). Les études sur Vibrio cholerae, par exemple, ont montré que les protéines en jeu dans l’interaction avec la chitine support et source de nutriments principal de cette bactérie dans les océans sont les mêmes que celles qui lui permettent d’adhérer aux parois intestinales chez l’homme (Pruzzo et al., 2008). D’autre part, les épidémies causées par cette bactérie sont reliées directement aux changements climatiques, vents et courants à l’échelle de la planète (Mourino-Pérez, 1998).
Dans le domaine de la pathologie végétale, cette nouvelle dimension est à peine naissante.
Que nous ont appris les études essentiellement agronomiques sur le pouvoir pathogène de Pseudomonas Syringae ?
La « vie sauvage » de cette bactérie influence-t-elle l’évolution de ce pouvoir pathogène ?
1. Pseudomonas syringae : une bactérie phytopathogène
P. syringae est un bacille Gram négatif d’une grande importance agronomique car il est capable d’attaquer une gamme très étendue de plantes. Il est retrouvé seul, en petits groupements ou en biofilms. La bactérie est aérobie stricte, chimio-hétérotrophe. Certains caractères biochimiques permettent son identification : production d’un pigment fluorescent aux UV (pyoverdine) sur le milieu B. de King, test oxydase négatif, absence de cytochrome C.
Cette bactérie est capable de survivre à la surface des feuilles (c’est une bactérie épiphyte) sans provoquer de maladies. Durant cette phase, P. syringae est capable de se développer en biofilm, ce qui représente 15 à 40% de la population totale (Morris et al., 1998). Quand les conditions sont plus favorables pour elle, la bactérie pénètre dans la plante via les stomates ou des blessures et prolifère dans les espaces intercellulaires (Hirano & Hupper, 2000). P. syringae est un agent nécrogène qui se développe lentement et qui s’attaque aux tissus parenchymateux des plantes. Les feuilles, les tiges et les fruits ainsi attaqués prennent un aspect huileux et sont gorgés d’eau, ils brunissent rapidement et peuvent être confondus avec des dégâts ayant une autre origine (fongique, par exemple). Seuls des tests d’identification peuvent confirmer la présence de la bactérie.
Cinquante deux pathovars (sous-groupes) de cette espèce ont été recensés sur la base de la spécificité d’hôte. Trois groupes monophylétiques ont été dégagés (Sawada et al., 1999). En effet, alors que P. syringae possède collectivement une large gamme d’hôte, les souches individuellement ne sont virulentes que sur un panel très restreint de plantes (Gaignard et al., 1993).
Les recherches portant sur les mécanismes de virulence de la bactérie sont nombreuses. On sait maintenant que P. syringae injecte directement des « protéines effectrices » à l’intérieur du cytoplasme des cellules eucaryotes. Pour cela, ces bactéries possèdent le système de sécrétion de type III qui est comparable à une seringue moléculaire. Les gènes codant pour ce système de type III sont situés à l’intérieur d’un îlot de pathogénicité et ont été acquis par divers transferts entre pathogènes de plantes, d’animaux et de symbiotes appartenant aux espèces telles que Shigella, Salmonella, Yersinia, Pseudomonas, Xanthomonas, Erwinia, et Rhizobium (He et al., 2004 ; Journet et al., 2005). Actuellement, plus de 300 gènes bactériens associés à la virulence ont été identifiés chez P. syringae dont plus de 50 codent pour des protéines effectrices transmises à la plante par le système de sécrétion de type III (Sarkar et al., 2006).
Cependant ce système à lui seul ne permet pas une attaque efficace. D’autres facteurs sont entrent en jeu notamment ceux relatifs à l’acquisition du fer ou de nutriments, à l’osmoprotection, à la tolérance aux UV et au stress oxydant ou à la formation de biofilm (Morris & Monier, 2003). La production de toxines est aussi considérée comme un facteur de virulence important.
Pour finir, P. syringae est dotée d’une activité glaçogène. Elle produit sur sa membrane externe une protéine permettant la fixation de molécules d’eau et leur orientation en cristaux de glace et est utilisée dans la production commerciale de neige (Rixen et al., 2003). Cette propriété n’est pas un facteur de virulence même si elle entraîne de graves dégâts sur les plantes (dégâts dus au gel).
2. Pseudomonas syringae : une bactérie de l’environnement
Cette bactérie est très présente dans les zones agricoles (Morris et al., 2007) : notamment dans les tissus et les débris végétaux, les équipements et structures agricoles, les eaux d’irrigation… Suite au labour, les populations de P. syringae décroissent rapidement dans le sol. Cependant, elle semble être capable de coloniser les graines et les racines (Tornero & Dangl, 2001) notamment les racines d’A. thaliana (Bais et al., 2004).
Des souches de P. syringae ont aussi été isolées dans des régions éloignées des zones agricoles (Morris et al., 2007), cet aspect n’ayant pendant longtemps pas été pris en compte. Récemment, l’équipe de Cindy E. Morris a montré la présence de cette bactérie au sein de substrats variés : dans des biofilms épilithes (situés à la surface des roches), ainsi que dans des réservoirs alpins tels que : neige, pluies, torrents, lacs mais aussi sur des plantes sauvages (Morris et al., 2007). Dans ces réservoirs, les souches de P. syringae retrouvées peuvent être virulentes pour différentes cultures ou totalement avirulentes. Les propriétés liés à la virulence semblent dépendre du substrat dans lequel on retrouve la bactérie : la production de toxine ou le pouvoir glaçogène semblent, par exemple, moins fréquents dans les populations des biofilms épilithes (Morris et al., 2008).
Depuis quelques années, la présence de P. syringae au sein des nuages a été montrée (Sands et al., 1982). Ceci rattaché au fait que P. syringae est dotée d’une activité glaçogène suggère que la bactérie joue un rôle dans les processus liés à la formation des précipitations (Morris et al. 2004, Morris et al., 2008). Cet élément permettrait à la bactérie de coloniser une large gamme de substrats non agricoles et de trouver des niches écologiques dans lesquelles elle peut survivre.
Les connaissances actuelles sur P. syringae montrent que cette bactérie a la capacité de survivre à la dissémination à grande distance et de s’adapter à divers habitats. La nature de ces habitats suggère un parcours en milieu aquatique qui suit le cycle des eaux douces. Ce cycle de vie en liaison avec celui de l’eau est représenté sur le schéma de la figure 1 :
Figure 1 : Schéma des habitats et des flux possibles de P. syringae dans l’environnement. Chute de neige ou de pluie contenant P. syringae (1). La neige habitat potentiel de prolifération ou de stress (2). La neige et la pluie contribuent aux eaux de montagnes, vecteurs et habitats de P. syringae (3). La bactérie est entraînée vers les plantes sauvages où elle va proliférer. Survie et multiplication de variants de P. syringae (4). Le ruissellement et la percolation des eaux souterraines sélectionnent des variants de P.
syringae qui alimentent
les rivières (5). L’irrigation porte la bactérie vers les plantes cultivées et vers les équipements agricoles (6). Apport direct aux cultures de P. syringae par la pluie, la dissémination aérienne et des sources du système
agricole. Certains variants de P. syringae sont mieux adaptés à l’agro-écosystème (7). La bactérie en phase épiphyte sur les plantes devient un composant des aérosols et elle est transportée vers les nuages (8). Les nuages pourraient porter P. syringae vers d’autres bassins versants (9). Morris et al. (2008).
3. Les interactions dans les biofilms
Comme il a été dit précédemment, P. syringae est capable de se développer en biofilm, ce qui représente 15 à 40% de la population totale (Morris et al., 1998). Cet élément, ajouté au fait que le cycle de vie de la bactérie est rattaché au cycle de l’eau, offre la possibilité d’étudier P. syringae en dehors des zones agricoles : au sein de biofilms en milieu aquatique.
L’ensemble des milieux aquatiques naturels sont concernés par la formation de biofilms. La plupart du temps cette formation se fait au fond des cours d’eau à la surface des galets : on parle de biofilms épilithes. Ces derniers représentent un habitat important pour P. syringae : il serait intéressant d’y connaître la biologie des populations de cette bactérie ainsi que leur pouvoir pathogène ce qui serait un plus pour lutter contre ce microorganisme en milieu agricole. En effet, les biofilms abritent un ensemble de communautés diverses qui cohabitent, interagissent et évoluent ensemble : ils y subissent une diversification phénotypique afin de s’adapter y compris P. syringae (Little et al., 2008).
Ces communautés sont diverses. Elles sont constituées d’un mélange d’espèces microbiennes, procaryotes et eucaryotes, le tout étant dans une matrice constituée de polysaccharides, protéines, acides nucléiques, lipides et autres molécules. Les biofilms épilithes sont composés d’algues (unicellulaires ou pluricellulaires), de bactéries, de champignons, de nématodes, de protozoaires, de petits invertébrés et de détritus organiques ou inorganiques, le tout englobé là encore dans une matrice de polysaccharides.
Les microalgues sont une des composantes importantes des biofilms épilithes. Ce terme de « microalgues » désigne l’ensemble des microorganismes photosynthétiques y compris les cyanobactéries. Les interactions algues-bactéries ont jusqu’à présent été assez peu étudiées. Certaines algues auraient un effet positif sur la croissance des populations bactériennes, d’autres un effet négatif. A l’inverse, des bactéries favoriseraient la croissance des populations algales tandis que d’autres influeraient négativement sur ces mêmes populations. Les mécanismes peuvent être directs : production de toxines, d’antibiotiques ou interactions trophiques (Giroldo et al., 2007), mais aussi indirects comme l’effet de protection en biofilm bactérien, ou l’effet sur d’autres populations.
Dans le cas de P. syringae, rien n’était connu sur cette interaction avant une étude préliminaire effectuée par le laboratoire de Cindy E. Morris montrant clairement l’effet de trois souches de P. syringae sur la croissance de la diatomée Nitzschia palea (Laussel, 2007). Quelle que soit la souche bactérienne testée, la croissance de l’algue est retardée quand elle est co-cultivée avec P. syringae (Laussel, 2007). Cette interaction est donc à explorer davantage et constitue une première étape dans la connaissance des relations entre organismes vivants dans les biofilms épilithes.
L’hypothèse principale est donc qu’il existe une interaction entre les populations de P. syringae et les microalgues aquatiques Cette interaction exercerait une pression de sélection pour la population bactérienne et son pouvoir pathogène. En d’autres termes, nous voulons montrer que le contact des populations de P. syringae avec les microalgues d’eaux douces affecte les caractères pathogènes de ces populations.
Ces hypothèses soulèvent certaines questions :
Y a-t-il réellement des interactions entre les bactéries P. syringae et certaines microalgues au sein des biofilms ?
Si c’est le cas, les caractères pathogènes de ces bactéries jouent-ils un rôle dans ces interactions ?
Pour répondre à ces interrogations, les objectifs du stage sont :
- de mettre en place un test simple de co-culture microalgues / bactéries. Ceci
afin de montrer si la croissance bactérienne est affectée par la présence d’une microalgue ou si la présence de la bactérie modifie la croissance de la microalgue
- de cribler la diversité des souches de P. syringae présentes dans la collection
du laboratoire. Ceci afin d’apporter une première réponse sur le rôle des caractères pathogènes des bactéries dans les interactions avec les microalgues.
MATERIELS ET METHODES
1. Choix des souches
a) Les souches bactériennes
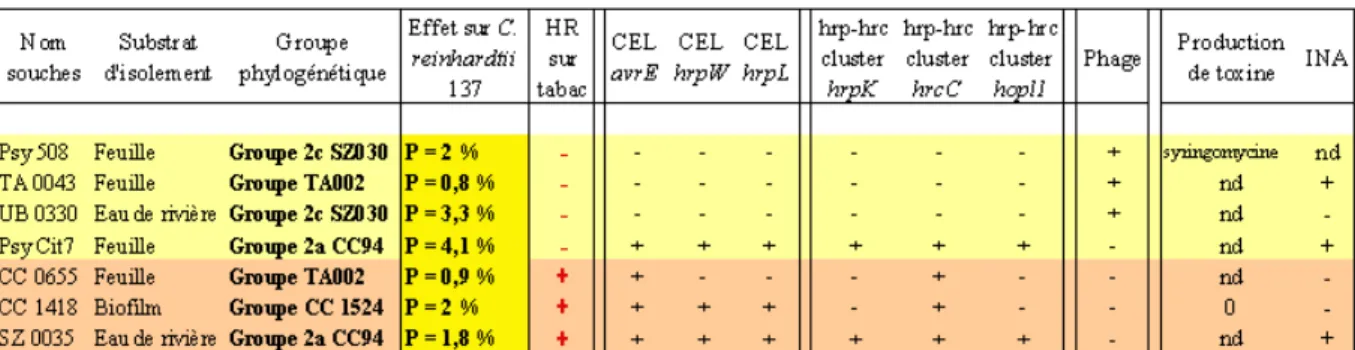
La collection de souches de P. syringae du laboratoire de pathologie végétale est unique au monde, car elle contient non seulement des souches issues de plantes malades comme dans la plupart des autres collections, mais aussi des souches issues de l’environnement (Morris et al., 2008). Le laboratoire possède donc une très grande diversité de souches de P. syringae qui peuvent avoir des propriétés différentes selon les pressions de sélection qu’elles ont rencontrées (Morris et al., 2010). L’objectif général de cette étude est de balayer cette diversité en choisissant des souches différentes, afin de les tester dans leurs relations aux microalgues d’eau douce, habitat partagé puisque les populations de P. syringae suivent le cycle de l’eau. Pour cette étude, il fallait mettre au point un test de co-culture destiné à mesurer l’effet de souches de P. syringae sur la croissance d’une microalgue, et réciproquement, l’effet de la microalgue sur la croissance bactérienne. Parmi les différentes propriétés des souches bactériennes susceptibles d’avoir un effet sur les microalgues, celles qui intéressent le plus le laboratoire sont celles qui sont liées au pouvoir pathogène. Une série de souches récemment bien caractérisées génétiquement au laboratoire (Demba Diallo, 2010) a été choisie pour la mise au point de ce test. Elles figurent dans l’annexe 1 (annexe 1 : les différentes souches bactériennes étudiées et certaines de leurs propriétés, p.43) et ont été choisies pour leur variabilité pour les critères suivants :
- la réaction d’hypersensibilité du tabac qui détermine si la souche est pathogène
ou non
- la présence de certains gènes codant pour des protéines effectrices (injectées
dans les cellules eucaryotes lors de l’interaction avec la plante) : avrE, hrpW, hrpL
- la présence de certains gènes de structure du système de sécrétion de type III,
sorte de seringue moléculaire qui sert à l’injection des effecteurs : hrpK, hrcC, hopl1
- la présence d’un phage proche de l’îlot de pathogénicité dans le génome
bactérien. Cet îlot est une région physique qui contient les gènes principaux impliqués dans le pouvoir pathogène dont ceux cités au-dessus.
- la production de toxine qui est un auxiliaire au pouvoir pathogène
- la présence de la protéine permettant l’activité glaçogène (test INA : ice
nucleation activity).
L’intérêt étant ici de voir si un ou plusieurs de ces critères engendrent un effet lors de la co-culture bactéries/algues, il convient de cribler un maximum de souches bactériennes ayant des combinaisons différentes de ces différents critères. Cependant, le temps étant limité, 28 souches de P. syringae ont été choisies, chacune étant différente pour au moins un des critères décrits précédemment. Par ailleurs, trois autres souches de bactéries (phytopathogènes ou non) autres que des P. syringae ont aussi été sélectionnées comme des « témoins » afin de vérifier si les effets observés sont spécifiques à l’espèce ou peuvent être communs à d’autres bactéries, même éloignées.
b) La souche de microalgue
Dans le projet global d’étude des interactions entre les populations de P. syringae et celles des microalgues des biofilms d’eau douce, le choix des microalgues est orienté vers plusieurs groupes d’organismes et en particulier le groupe des diatomées très abondant dans ces biofilms, et le groupe des microalgues vertes, plus proche des plantes au niveau du fonctionnement de la photosynthèse.
La microalgue sélectionnée dans la présente étude est la souche modèle Chlamydomonas reinhardtii 137. Il s’agit d’une microalgue verte, bien connue par les scientifiques : son génome entier a été décrypté récemment. Elle a été sélectionnée pour sa facilité de culture au laboratoire, pour l’absence d’agrégats de cellules qui gênent les dénombrements, et pour le fait qu’elle existe en culture pure, ce qui n’est que rarement le cas des microalgues. En effet la plupart des cultures de microalgues en laboratoire contiennent des bactéries associées qu’il est difficile voire impossible d’éliminer : ces dernières pourraient interagir avec P. syringae dans les tests ce qui n’est pas souhaitable dans notre cas. De plus, C. reinhardtii possède 35% de ses gènes en commun avec les plantes à fleur (Merchant et al., 2007), or, s’il existe des interactions entre P. syringae et des microalgues, on peut s’attendre à ce qu’il y en ait d’autant plus que les microalgues sont proches de la cible « habituelle » des bactéries, c’est-à-dire proches des plantes : d’où le choix de C. reinhardtii.
Remarque : Par la suite, il est prévu que l’équipe de bactériologie fasse les expériences menées ici avec d’autres microalgues et d’autres souches bactériennes. Cette étude préliminaire a pour but de mieux cerner les conditions de ces tests de croissance avec une microalgue et une première série de souches, pour ensuite les effectuer en batterie sur un grand nombre de souches.
2. La culture et le dénombrement des microorganismes
a) Milieux de culture
Différents types de milieu ont été utilisés pour cultiver les microorganismes :
- le TSB/10e (voir l’annexe 2 : la composition des milieux de culture utilisés,
p.44): les souches bactériennes sont cultivées en routine sur ce bouillon nutritif courant et riche (même s’il est dilué). Les souches de P . syringae atteignent
108 à 109 cellules par mL en 48 h à 20 °C dans ce type de milieu. Ce milieu
peut être gélosé (15g/L d’agar) pour les cultures en boite de Petri (il est alors
appelé TSA/10e).
- le TAT (voir l’annexe 2 : la composition des milieux de culture utilisés, p.44) :
ce milieu est utilisé pour les cultures de C. reinhardtii et peut être utilisé avec une source de carbone comme l’acétate (il est alors appelé milieu TAP) ou sans source de carbone. Dans ce dernier cas, seule la photosynthèse fourni le carbone et l’énergie aux cellules de microalgues qui peuvent atteindre une
densité autour de 106 cellules/mL.
Le milieu choisi pour les co-cultures est le milieu TAT, sans source de carbone, qui convient à la croissance de l’algue et dans lequel P. syringae survit et peut même avoir une croissance toutefois limitée. La source de carbone pour les bactéries sera, (en dehors des traces de carbone organique présentes dans le milieu TAT) issue du métabolisme des algues et donc indirectement du dioxyde de carbone fixé par la photosynthèse de C. reinhardtii 137.
b) Dénombrements « classiques » des microorganismes
• Les microalgues
La méthode classique de dénombrement des microalgues consiste à compter directement les cellules au microscope à l’aide d’une lame de comptage spéciale : la cellule de Malassez. Il s’agit d’une lame en verre avec un quadrillage correspondant à un volume donné : en remplissant la chambre de comptage, on peut compter le nombre de cellules sur ce quadrillage. On obtient la concentration en cellules de la suspension.
Remarque : Cette méthode est adaptée aux cellules ayant un volume important (microalgues, levures…). Un autre type de lame (lame Thoma) est adapté au dénombrement des bactéries, mais n’a pas été utilisé dans cette étude.
• Les bactéries
Pour dénombrer approximativement et directement le nombre de bactéries au sein d’une suspension, on peut utiliser un spectrophotomètre. En effet, une culture bactérienne absorbe et réfléchit la lumière passant à travers elle. Or, dans une certaine gamme de
concentrations bactériennes (5.107 à 5.109 cellules / mL), la lumière absorbée ou réfléchie par
la suspension bactérienne est directement proportionnelle à la concentration de cellules dans la culture pour une longueur d’onde donnée. Ainsi, en mesurant l’absorbance d’une suspension bactérienne, on peut estimer le nombre de cellules au sein de cette suspension après calibrage.
Pour dénombrer plus précisément le nombre de bactéries au sein d’une suspension, il existe une autre technique : la dilution puis l’étalement sur milieu solide. Le principe est le suivant : au laboratoire, le niveau de la population bactérienne dans les suspensions est déjà connu approximativement (grâce à l’absorbance ou en prédisant l’évolution de la culture en
un temps donné). Après deux jours de pré-culture en milieu TSB/10e, la population est située
autour de 108 cellules / mL et, au bout de six jours de culture en milieu TAT, elle se situe
autour de 106 cellules / mL.
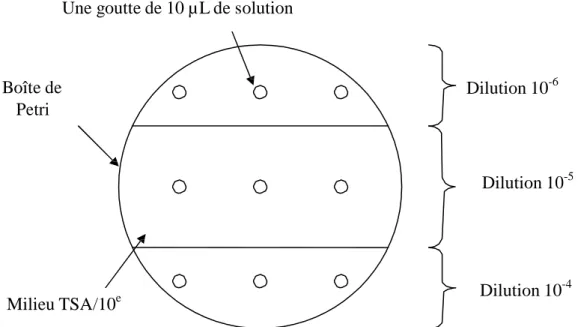
Le comptage sur milieu solide TSA/10e nécessite de diluer les suspensions de départ pour
avoir entre 5 et 50 colonies par goutte de 10 µ L ensemencée afin de pouvoir dénombrer. Pour
les bactéries issues d’une suspension évaluée à 108 cellules / mL par exemple, nous diluerons
jusqu’à 10-6 et nous ensemencerons trois gouttes de 10 µ L de la dilution 10-6, 10-5 et 10-4
Une goutte de 10 µ L de solution
Boîte de Petri
Dilution 10-6
Dilution 10-5
Milieu TSA/10e Dilution 10
-4
Figure 2 : Schéma représentant un exemple de dénombrement bactérien sur milieu solide en boîte de Petri Après avoir ensemencé les bactéries sur milieu solide, les boîtes de Petri sont placées à l’étuve à 20°C pendant deux jours, puis les colonies sont dénombrées et la concentration bactérienne dans la solution initiale est calculée en tenant compte des différentes dilutions
utilisées. Par exemple, pour la dilution 10-6, si on trouve 12 colonies dans les 3 gouttes, soit en
moyenne 4 colonies/10 µ L, soit 400 colonies/mL alors le nombre de bactéries par mL dans la
suspension de départ est de 400 x 106 = 4,0.108 bactéries par mL. En faisant la moyenne du
nombre de bactéries trouvées pour les dilutions où le comptage est possible, on obtient le nombre de bactéries/mL pour un tube donné.
Remarque : Cette méthode a l’avantage de nous indiquer quand il y a eu contamination de la suspension lorsque l’on observe des colonies de types différents.
c) Dénombrement des microorganismes au compteur de cellules
Dans nos expériences comprenant beaucoup de traitements (nous voulions tester de nombreuses souches), il fallait allier la rapidité de la technique et la possibilité de compter dans la même suspension des cellules de deux types différents. C’est pourquoi l’unité a récemment eu accès à un compteur de cellules de type MS3 (de marque Beckman). Cet appareil permet quasi instantanément (< 1 minute) de compter et de mesurer la taille (le volume) de toutes sortes de particules en suspension dans un liquide suivant le principe de Coulter expliqué en annexe 3 (annexe 3 : principe de fonctionnement du compteur de particules Beckman Coulter Multisizer 3, p.45)
L’une des missions du stage était de comparer les résultats des techniques « classiques » de dénombrement des microorganismes avec les mesures effectuées par le compteur de cellules, ceci afin de fixer les différents paramètres permettant d’effectuer les mesures grâce à la machine. Ces éléments étant issus de résultats de manipulations, ils seront détaillés dans la partie « Présentation et analyse des résultats ».
lo g ( n o m b re d e b a c té ri e s / m L ) lo g ( n o m b re d e m ic ro a lg u e s / m L )
3. Détermination du protocole de co-culture bactéries / microalgues
a) Détermination des paramètres de croissance
La croissance d’une population microbienne dans un milieu de culture adapté suit une cinétique bien connue et décrite en quatre phases :
- une phase de latence durant laquelle la population ne croît pas ou très peu, elle
s’adapte
- une phase exponentielle : les cellules se divisent activement, il y a
augmentation exponentielle du nombre de cellules au cours du temps
- une phase stationnaire au cours de laquelle la population se stabilise autour
d’une valeur donnée car un des facteurs de croissance devient limitant
- une phase de déclin : les cellules ne se divisent plus et meurent, il y a alors
diminution de la population au cours du temps.
Dans notre cas, nous voulons comparer l’effet de la présence d’une population bactérienne (respectivement la population algale) sur la croissance de la souche de microalgue (respectivement de bactéries). Pour cela, nous choisissons d’utiliser comme variable, le niveau de la phase stationnaire atteint par les populations de manière à ce que les valeurs soient plus facilement comparables.
Dans une expérience pilote, nous avons déterminé en combien de temps cette phase stationnaire est atteinte par un suivi la croissance d’une souche représentative des P. syringae (CC094) avec et sans microalgues ainsi que la croissance de C. reinhardtii 137 avec et sans bactéries.
Les conditions de l’expérience avaient été fixées par des essais antérieurs du laboratoire énumérés plus bas Pour réduire la phase stationnaire, les ensemencements sont
faits avec des cultures jeunes à t0 à partir de pré-cultures en phase exponentielle. Nos mesures
montrent que pour les deux types d’organismes, la phase stationnaire est atteinte au bout du sixième jour (figure 3) ce qui nous a permis d’élaborer le protocole définitif du test.
A B 8 7 7 6 6 5 5 4 CC94 seules 4 CC94 avec 137 3 3 137 seules 137 avec cc94 2 2 1 1 0 0 2 4 6 8 10
Nombre de jours en culture
0
0 2 4 6 8 10 12 14
Nom bre de jours e n cult ure
Figure 3 : Evolution des populations microbiennes en fonction du temps. A : population de P. syringae CC94 en présence ou en absence de C. reinhardtii 137 B : population de C. reinhardtii 137 en présence ou en absence de P. syringae CC94
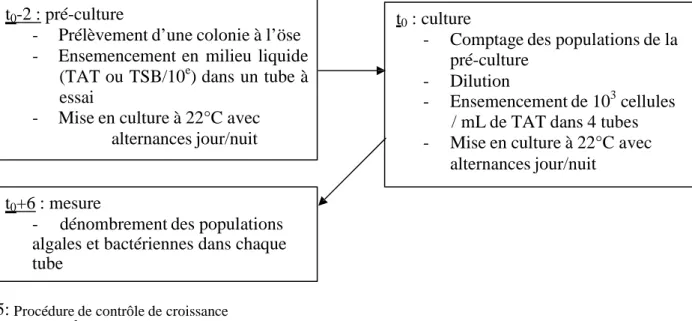
b) Protocole du test de co-culture
Toutes les cultures sont effectuées dans des tubes à essai en verre contenant 5 ml de milieu, fermés avec des bouchons non étanches aux gaz, inclinés de façon à exposer le liquide à la lumière, et placés en chambre de culture en conditions contrôlées (figure 4), sans agitation continue, à des températures et une photopériode spécifique : 14 heures de jour à 22 °C et 10 heures de nuit à 20 °C.
- Pré-culture : t0-2j
Les souches bactériennes de la collection du laboratoire sont fournies sur milieu solide. Une colonie est prélevée à
l’öse et ensemencée sur milieu liquide TSB/10e. C.
reinahrdtii 137 est maintenue sur milieu TAT liquide au laboratoire. Du milieu TAT frais est ensemencé avec 50 µ L de culture. Les bouillons sont incubés dans la chambre de culture.
- Mise en culture : t0
À t0, les populations d’algues et de bactéries sont
dénombrées et environ 103 cellules/mL (valeurs proches
de ce que l’on retrouve dans les cours d’eau à l’état Figure 4 : Intérieur de la chambre de culture naturel) sont inoculées à du milieu TAT stérile.
où les co-cultures sont effectuées en conditions contrôlées
- Incubation : de t0 à t0+6j
Les tubes sont placés dans la chambre de culture sans agitation, excepté une agitation quotidienne à la main, pour assurer une homogénéisation des cultures, et un renouvellement des gaz.
- Dénombrements : t0+6j
Les microorganismes sont dénombrés grâce à l’une des techniques décrites précédemment.
Les différentes étapes sont résumées sur la figure 5 :
t0-2 : pré-culture
- Prélèvement d’une colonie à l’öse - Ensemencement en milieu liquide
(TAT ou TSB/10e) dans un tube à
essai
- Mise en culture à 22°C avec alternances jour/nuit
t0 : culture
- Comptage des populations de la
pré-culture
- Dilution
- Ensemencement de 103 cellules
/ mL de TAT dans 4 tubes
- Mise en culture à 22°C avec
alternances jour/nuit
t0+6 : mesure
- dénombrement des populations
algales et bactériennes dans chaque tube
Figure 5: Procédure de contrôle de croissance
c) Plan d’expérience
Selon le temps restant après la détermination des paramètres liés au compteur de cellules, un plan d’expérience a été construit, basé sur une série de neuf manipulations avec dans chacune, 3 à 6 souches bactériennes et pour chaque traitement deux répétitions (le nom des souches correspondant à chaque manipulation se trouvent en annexe 4 : correspondance souches/manipulations, p.46).
Dans chaque expérience il y a:
- deux tubes de C. reinhardtii 137 seules
- deux tubes de chaque souche bactérienne
- deux tubes de chaque souche bactérienne en co-culture avec C. reinhardtii 137
4. Traitement des données
Un test de Student a été utilisé pour comparer deux à deux les populations microbiennes seules ou en co-culture ainsi que la taille des microalgues, seule ou en co- culture pour chaque souche bactérienne. L’ANOVA à un facteur a permis de comparer les populations d’algues dans les différentes expériences et l’ANOVA à deux facteurs de comparer les effets de la co-culture et de la souche bactérienne sur la croissance de l’algue. Ces tests ont été appliqués grâce au logiciel Statistica.
Les boîtes à moustaches, les histogrammes et autres courbes ont été obtenus grâce à ce même logiciel ainsi que grâce à Excel.
lo g (v a le u rs r e le v é e s a u c o m p te u r d e c e ll u le s )
PRESENTATION ET ANALYSE DES RESULTATS
1. Mise au point d’un protocole d’utilisation du compteur de cellules
La taille des bactéries étant bien distincte de celle des microalgues, le comptage simultané des deux types d’organismes est théoriquement possible avec cette technologie qui a l’avantage d’être bien plus rapide que les dénombrements classiques de microorganismes.
a) Le dénombrement des microalgues
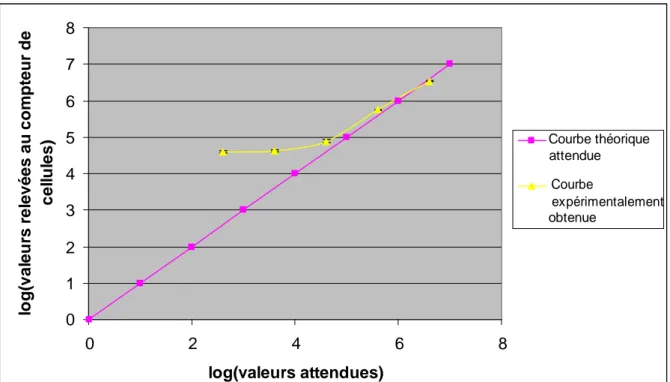
Pour mettre au point un protocole d’utilisation du compteur de cellules, il faut tout d’abord vérifier que celui-ci compte de manière exacte. Pour cela, nous avons comparé les valeurs obtenues par le dénombrement classique (cellules de Malassez) aux valeurs obtenues grâce à l’appareil. Les résultats sont donnés dans la figure 6 (les écarts-types des différentes valeurs ne sont pas visibles ici car très faibles).
8 7 6 5 Courbe théorique attendue 4 Courbe expérimentalement 3 obtenue 2 1 0 0 2 4 6 8 log(valeurs attendues)
Figure 6 : Dénombrements de C. reinhardtii 137 au compteur de cellules en fonction des valeurs attendues calculées à partir d’une estimation par cellule de Malassez sur la suspension dense
On remarque que la courbe obtenue est différente de la courbe attendue pour les valeurs les plus faibles. Le compteur de cellules n’est donc pas fiable pour des populations
basses : son seuil de sensibilité est approximativement de 105 cellules/mL. Cela signifie qu’en
deçà de cette valeur, les mesures effectuées par l’appareil ne sont pas utilisables.
Or que ce soit en fin de pré-culture ou au bout de six jours d’expérimentation, les
populations de microalgues devraient toutes atteindre et dépasser les 105 cellules/mL : il sera
donc possible de dénombrer les C. reinhardtii grâce au compteur de cellules.
Dans la suite de ce rapport, nous garderons toujours en tête que si des populations de microalgues sont inférieures au seuil de sensibilité de la machine, il ne faudra pas prendre en compte les valeurs trouvées. De même, si des valeurs supérieures à celles testées ici (donc supérieures à un logarithme de 6,6) sont trouvées, nous ne les prendrons pas en compte dans
lo g ( v a le u rs r e le v é e s a u c o m p te u r d e c e ll u le s )
la suite de l’analyse car nous ne savons pas si le compteur de cellules reste fiable au-delà d’un logarithme de 6,6.
b) Le dénombrement des bactéries
La même manipulation a été effectuée avec des suspensions bactériennes. Le dénombrement de la solution mère a cette fois été réalisé par suspension-dilution-étalement : les valeurs attendues sont donc les valeurs correspondant aux calculs liés aux dilutions de cette solution mère. Les résultats sont donnés figure 7 (encore une fois les écarts-types sont trop faibles pour être visibles).
E. Coli 9 8 7 6 5 4 3 2 1 0 0 2 4 6 8 10
log (valeurs attendues)
Courbe
expérimentalement obtenue
Courbe théorique attendue
Figure 7 : Dénombrements d’E. coli au compteur de cellules en fonction des valeurs attendues calculées à partir d’une estimation par suspension-dilution-étalement
Cette fois à aucun moment les courbes ne se confondent. Il n’y a donc pas de seuil de sensibilité du compteur de cellules à E. Coli, et plus généralement, aux bactéries (l’expérience ayant été réalisée avec d’autres bactéries et n’étant pas plus concluante). En d’autres termes, il ne sera pas possible de dénombrer les bactéries grâce à l’appareil. Ceci est sans doute dû au fait que le compteur de cellules utilisé durant les manipulations est doté d’un tube ayant un orifice de 100 µm, non adapté pour de petites particules (inférieures à 3 µm).
En l’absence de tube à orifice plus petit (50 ou 30 µm), les méthodes de dénombrements bactériens adoptés par la suite seront donc les méthodes classiques (absorbance et suspension-dilution-étalement).
c) Protocole retenu
Les résultats exposés précédemment ont permis de déterminer les techniques de comptage des microorganismes. Pour les bactéries, les méthodes classiques seront privilégiées. Elles ont l’inconvénient d’être plus lourde que le comptage de cellules au MS3, de donner les résultats après deux jours d’incubation et non pas instantanément, mais elles sont moins délicates (présence de poussières gênantes au MS3) et permettent de repérer des contaminants éventuels. Ceci a d’ailleurs été le cas dans ces expériences. En effet, sur les boîtes de comptage des deux dernières manipulations (15 et 16 septembre), un contaminant bactérien est apparu très tardivement (après plus d’une semaine sur milieu solide). Après contrôle, il s’avère que le milieu TAT était contaminé. Cette contamination ne semble pas
avoir perturbé les mesures que nous avons faites (voir analyses statistiques) et nous avons considéré que ces résultats sont fiables. Toutefois, il sera plus prudent de les vérifier dans une nouvelle expérience.
Pour les microalgues, nous utiliserons le compteur de cellules. Il s’agit d’une technologie très robuste avec laquelle il faut toutefois faire extrêmement attention à ne pas avoir de contaminants (particules de même taille que les microalgues) dans la verrerie utilisée. Dans les premiers essais sans rinçages de la verrerie nous comptions plus de 70 000 particules par mL dans de l’eau distillée représentant les poussières et autres contaminants atmosphériques. Un processus spécial de nettoyage du matériel utilisé a donc été instauré : toute la verrerie est rincée deux fois à l’eau osmosée, une fois à l’eau distillée puis rebouchée soigneusement pour éviter les contaminants atmosphériques. Par ailleurs, les acuvettes (petits béchers en plastique) dans lesquelles les mesures sont effectuées sont rincées avant chaque mesure à l’eau distillée filtrée (filtres en nitrocellulose de porosité 0,22 µm). La solution de NaCl (0,9 %) utilisée dans l’appareil et lors des mesures est, elle aussi filtrée (0,22 µm).
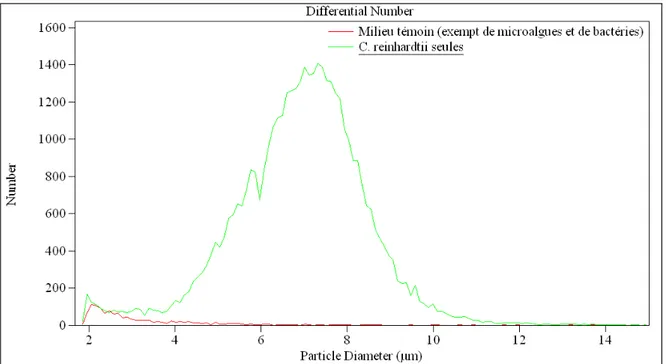
Lors de la mesure (voir annexe 3 : principe de fonctionnement du compteur de particules Beckman Coulter Multisizer 3, p.45), 400 µ L de la suspension à analyser sont ajoutés dans l’acuvette contenant 15 mL de NaCl (0,9 %) filtré, fermée par un bouchon étanche, Après agitation la mesure est effectuée sur un volume que nous avons fixé à 500 µ L (volume ni trop élevé pour privilégier des mesures rapides, ni trop faible pour assurer la fiabilité des mesures). Nous obtenons en moins de 12 secondes une courbe de répartition des effectifs en fonction de leur taille illustré figure 8.
Figure 8 : Nombre de particules dans 500 µ L de suspension en fonction de leur « diamètre équivalent » qui indique celui de la sphère de même volume que celui de la particule quelle que soit la forme de la particule. En rouge, témoin TAT dans du NaCl 0,9 % filtré (0,22 µ M), en vert, suspension de C. reinhardtii 137 dans le
même soluté.
On remarque que malgré les précautions prises, des poussières restent présentes à de faibles diamètres (inférieurs à 3,5 µm) puisqu’un pic de particules est observé pour les deux courbes autour de 2 µm. Ceci est d’ailleurs une autre difficulté rencontrée lors du dénombrement des bactéries par le compteur de cellules.
lo g ( n o m b re d e c e ll u le s / m L )
Dans cet exemple représentatif de nos conditions, la population de C. reinhardtii 137 contient donc des cellules dans une fourchette de tailles entre 3,5 et 12 µm. Le nombre de particules totales situées dans cet intervalle dans le témoin est très faible (<50) : le nombre obtenu lors de la mesure entre 3,5 et 12 µm sera donc assimilé à des algues. La conversion de ce nombre de particules comptées en concentration dans la suspension de départ se fait par simple calcul.
Par exemple si le résultat donné par le compteur est de 32 573 particules. Cela signifie que nous avons 32 573 particules dans 500 µ L de la suspension de l’acuvette donc 65 146
particules / mL dans l’acuvette. Soit en tout 65 146 x 15,4, (1,0.106 particules) dans les 15,4
mL de l’acuvette. Nous avions donc apporté 1,0.106 cellules dans 400 µL provenant de la
suspension de départ, qui était à une concentration de 1,0.106 x 1000 / 400 = 2,5.106 cellules /
mL. Pour chaque suspension, un ensemble de trois mesures sont réalisées dans la même acuvette. La moyenne de ces trois mesures est utilisée pour calculer la concentration initiale.
2. L’influence des bactéries sur les populations d’algues
a) Au niveau du nombre de microalgues
Au terme des expériences menées, nous sommes arrivés à différents résultats
bruts concernant le niveau des populations algales : le nombre de cellules à t0, le nombre de
cellules à t0+6 en présence et en absence de bactéries.
Grâce à ces résultats, on a pu remarquer que dans tous les cas (cultures simples et co-
cultures), la population d’algues atteint et dépasse 106 cellules/mL au bout de six jours
d’expérimentation. De plus, dans 27 cas sur les 32 testés, le niveau des populations d’algues en co-culture est inférieur au niveau de ces mêmes populations en culture pure.
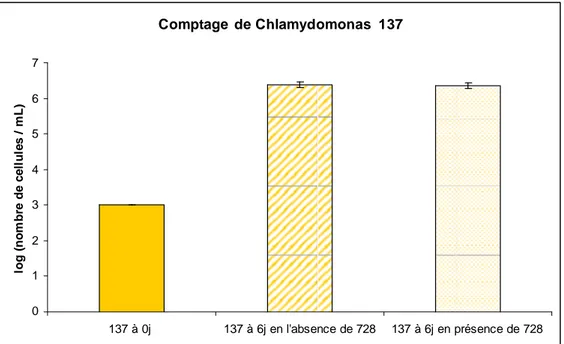
L’effet de la souche P. syringae 728 sur C. reinhardtii 137, représenté figure 9, est représentatif de l’effet de la plupart des autres souches bactériennes.
Comptage de Chlamydomonas 137 7 6 5 4 3 2 1 0 137 à 0j 137 à 6j en l'absence de 728 137 à 6j en présence de 728
Figure 9 : Evolution de la population de C. reinhardtii 137 en fonction de la présence ou de l’absence de P. syringae B728a dans le milieu de culture
Que les algues soient cultivées avec ou sans bactéries, la population inoculée à t0 reste
la même (autour de 103 cellules/mL) : c’est pourquoi une seule barre est représentée à t0. Par
ailleurs, au bout de six jours, la population atteint un logarithme supérieur à 6,3, ceci qu’il y ait ou non des bactéries Ps. 728 dans le milieu.
Pour finir, les niveaux des populations à t0+6j (en présence ou non de bactéries), bien
que très proches, ne sont pas égaux. Ceci est valable pour toutes les souches. Pour savoir, si la différence entre les algues cultivées seules ou en compagnie de bactéries est significative, nous avons appliqué un test de Student pour chaque souche bactérienne. Ce dernier montre une différence significative avec un risque d’erreur de 5% (p<5%) pour sept souches. Grâce au tableau 1, voyons si les différences observées chez ces dernières peuvent être reliées à l’un des caractères étudiés.
Tableau 1 : Les caractères pathogènes des souches bactériennes ayant un effet significatif négatif sur les populations de C. reinhardtii 137
Tout d’abord, on remarque que quelle que soit la souche bactérienne, on a toujours un effet négatif sur le nombre d’algues présents dans le milieu. De plus, les souches bactériennes qui engendrent un effet significatif sur l’algue ne partagent pas un caractère étudié en commun. Il n’y a donc pas de corrélation entre les effets des bactéries sur la croissance de C. reinhardtii et les propriétés testées.
b) Au niveau du diamètre des microalgues
Comme il a été expliqué précédemment, le compteur de cellules est capable de donner, pour chaque particule, un « diamètre équivalent » qui indique celui de la sphère de même volume que celui de la particule quelle que soit la forme de cette dernière. Ainsi, pour chaque
mesure effectuée à t0+6j, nous avons relevé le diamètre moyen de la sphère équivalente aux
algues.
L’ensemble des résultats montre des diamètres moyens variant de 5,45 µ m à 6,64 µm. Mais les valeurs ne permettent pas de conclure quant à un effet global particulier des bactéries sur le diamètre des algues.
Là encore un test de Student a été appliqué sur les valeurs pour chaque souche afin de
voir si le diamètre trouvé à t0+6 est significativement différent selon que l’algue est en
présence ou non de bactéries. Les résultats de ce test montrent des valeurs significativement différentes avec un risque d’erreur de 5% entre les algues en culture pure et celles en co- culture pour cinq souches.
Les diamètres des algues cultivées seules ou en présence de P. syringae Psy642, représentés sur la figure 10, sont représentatifs de ces cinq valeurs.
D ia m è tr e d e s C . re in h a rd ti i 1 3 7 a p rè s 6 j d e c u lt u re ( e n µ m ) 6,6 6,5 6,4 6,3 6,2 6,1 6 5,9 5,8 137 Psy642+137
Figure 10 : Evolution du diamètre de C. reinhardtii 137 en fonction de la présence ou de l’absence de P. syringae Psy642 dans le milieu de culture
On remarque que les algues cultivées seules ont un diamètre moyen qui est supérieur à celui des algues en co-culture avec des P. syringae Psy642. Cette observation peut être généralisée à l’ensemble des résultats pour lesquels la différence de diamètre entre la culture pure et la co-culture est significative avec un risque d’erreur de 5%.
Grâce au tableau 2, voyons si ces différences peuvent être reliées à l’un des caractères étudiés.
Tableau 2 : les caractères pathogènes des souches bactériennes ayant un effet significatif sur le diamètre de C. reinhardtii 137
Dans le tableau 2, on observe que les microalgues pour lesquelles la différence est significative étaient en présence de bactéries issues de plantes ou de biofilms mais jamais de bactéries provenant d’eau de rivière. Par ailleurs, les P. syringae engendrant un effet sur le diamètre des C. reinhardtii sont toutes des souches négatives au test d’hyperréactivité sur tabac.
3. L’influence des microalgues sur le niveau des populations bactériennes
Les résultats bruts obtenus concernant les bactéries sont de trois types :- nombre de bactéries par millilitre de solution à t0 (ce résultat est identique que
la bactérie soit en co-culture ou pas)