Université de Sherbrooke
L’étude de la relation phage-hôte chez Clostridium difficile
Par
Ognjen Sekulović
Département de microbiologie et d’infectiologie
Thèse présentée à la Faculté de médecine et des sciences de la santé en vue de l’obtention du grade de philosophiae doctor (Ph.D.) en microbiologie
Sherbrooke, Québec, Canada Mai, 2015
Membres du jury d’évaluation
Pr Louis-Charles Fortier, Microbiologie et Infectiologie, directeur de thèse Pr Brendan Bell, Microbiologie et Infectiologie, directeur du programme Pr Vincent Burrus, Biologie, évaluateur externe du programme
Pr Roger C. Lévesque, Microbiologie-infectiologie et d'immunologie, Faculté de médecine, Université Laval, évaluateur externe à l’Université
R
ÉSUMÉ
L’étude de la relation phage-hôte chez Clostridium difficile
Par
Ognjen Sekulović Programme de microbiologie
Thèse présentée à la Faculté de médecine et des sciences de la santé en vue de l’obtention du diplôme de philosophiae doctor (Ph.D.) en microbiologie, Faculté de médecine et des
sciences de la santé, Université de Sherbrooke, Sherbrooke, Québec, Canada, J1H 5N4 De nos jours, les bactériophages (c.-à-d. des virus bactériens, ou phages) sont reconnus comme un des principaux facteurs qui influencent l’évolution et la biologie bactérienne. De plus, la nature dynamique des relations phage-hôte engendre des adaptations mutuelles au niveau des stratégies d’infection et de défense, phénomène communément appelé « course à l’armement ». Malgré une importance démontrée chez de nombreuses espèces bactériennes, l’étude du rôle des phages dans la biologie du pathogène Clostridium difficile est demeurée très limitée. Or, les infections à C. difficile sont considérées comme étant la principale cause des diarrhées associées à la prise d’antibiotiques. Alors, l’objectif de la présente étude avait pour but de mieux caractériser l’implication des phages dans la biologie de C. difficile. Des travaux préalables ont montré que la lysogénisation par le phage tempéré φCD38-2 pouvait mener à une augmentation de la production de toxines chez certaines souches de C. difficile suggérant une implication des phages dans la virulence bactérienne. En utilisant cette étude comme point de départ, nous avons évalué l’influence de la lysogénisation du phage φCD38-2 sur le transcriptome global d’une souche de C. difficile d’importance clinique. Ainsi, nous avons montré que la lysogénisation par le phage φCD38-2 a un impact significatif sur la transcription de 39 gènes bactériens dont près de la moitié encodent des protéines reliées au métabolisme des sucres, suggérant une implication du phage dans les processus métaboliques de l’hôte. Cependant, le gène présentant la plus grande altération transcriptionnelle encode une protéine de surface nommée CwpV. À partir de sa localisation sur la surface bactérienne, nous avons démontré que son expression a un effet protecteur sur les cellules face aux infections par les phages. Les expériences subséquentes ont permis de lier l’activité antiphage au domaine carboxy-terminale variable de la protéine. Étant donné que l’adsorption virale n’est pas affectée par la présence de CwpV, nous avons établi que le mode d’action du système consiste à bloquer l’injection d’ADN virale dans la cellule bactérienne. De plus, l’effet antiphage est plus prononcé envers les siphophages comparé aux myophages suggérant un mode d’action dépendant de la morphologie virale. Finalement, les expériences préliminaires suggèrent que les cellules qui expriment la CwpV ont un avantage sélectif par rapport aux cellules qui ne l’expriment pas dans un essai de co-culture soumise à une infection virale.
S
UMMARY
Phage-host interactions in Clostridium difficile
By
Ognjen Sekulović Microbiology Program
Thesis presented at the Faculty of medicine and health sciences for the obtention of Doctor degree diploma philosophiae doctor (Ph.D.) in microbiology, Faculty of medicine
and health sciences, Université de Sherbrooke, Sherbrooke, Québec, Canada, J1H 5N4 Bacteriophages (or simply phages) are viruses that specifically infect and kill bacteria. They are omnipresent in every niche where bacteria thrive and as such are considered as the most abundant biological entities in the biosphere. Their massive impact on bacterial biology has incited scientific community to consider the phages as the major driving force in bacterial evolution. Nowadays, it is also assumed that phages act as the principal vectors for horizontal transfer of genetic information among prokaryotes. Moreover, highly dynamic nature of phage host relationships usually results in mutual adaptations that effectively stimulates acquisition of new offensive and defensive strategies. This phenomenon is generally described as the “phage-host arms race”. Despite their obvious importance, the contribution of phages to the biology of Clostridium difficile, the main cause of nosocomial infectious diarrhea, has not been extensively explored. Thus, the main objective of this study was to assess the overall impact of phages to C. difficile lifestyle. Our previous work has revealed the potential of a specific C. difficile phage termed φCD38-2 to stimulate the production of bacterial toxins. Based on those results, we have performed a global study of the impact of the φCD38-2 lysogeny on the bacterial transcriptome. Thus, we have found a total of 39 genes whose expression was altered during the lysogeny of φCD38-2 with near half of them encoding proteins implicated in bacterial sugar metabolism. This suggests phage implication in the regulation of bacterial utilization of carbon sources. However, the largest transcriptional alteration has been observed for cwpV which encodes a phase-variable surface-anchored protein. Owing to its variable nature, we have hypothesized that CwpV might play a role in phage infection and indeed, we have shown that CwpV expression protects bacterial cells from phage infection. Moreover, variable C-terminal domain of CwpV was found to be essential for antiphage phenotype since its deletion restored bacterial susceptibility to infection. Additionally, CwpV did not significantly affect phage adsorption, but phage DNA replication was prevented suggesting that CwpV act as a superinfection exclusion system. Interestingly, the antiphage effect was more pronounced against phages from Siphoviridae family compared to phages from Myoviridae family suggesting that structural differences are important for the antiphage phenotype. Finally, our preliminary data suggest that CwpV expression confers selective advantage when mixed co-cultures are challenged by phage infection.
T
ABLE DES MATIÈRES
Résumé ... ii
Summary ... iv
Table des matières ... v
Liste des figures ... ix
Liste des tableaux ... x
Liste des abréviations ... xi
Chapitre I ... 13
Introduction ... 13
Nous ne sommes pas seuls ... 13
Les bactériophages ... 14
Partenaire ou adversaire? ... 15
Les phages strictement lytiques ... 16
Les phages tempérés ... 16
Phage lambda (λ) ... 17
Le choix entre le cycle lytique ou lysogénique chez λ ... 18
La lysogénie représente plutôt la règle que l’exception ... 20
Coévolution des phages tempérés avec leurs hôtes bactériens ... 20
Régulation de l’expression génique durant le cycle lytique et lysogénique ... 22
Impact du cycle lytique et lysogénique sur le transcriptome de l’hôte ... 24
Induction spontanée et son rôle dans la biologie de l’hôte ... 28
« La course à l’armement » ... 29
L’adsorption ... 30
L’injection d’ADN... 31
Interférence au niveau des étapes tardives du cycle lytique ... 33
Clostridium difficile ... 36
Facteurs de virulence ... 37
Épidémiologie et évolution de C. difficile ... 39
Les objectifs de la présente étude ... 44 Chapitre II ... 46 Article 1 ... 46 Avant-propos ... 46 Résumé : ... 47 Abstract ... 49 Introduction ... 49
Materials and Methods ... 51
Bacterial strains and growth conditions. ... 51
cDNA library construction and RNA sequencing ... 53
Alignment of sequenced reads and bioinformatics analyses ... 54
Validation of RNA-seq expression data by RT-qPCR ... 55
Pulsed-Field Gel Electrophoresis and Southern blotting... 55
Cell surface protein extraction and SDS-PAGE ... 56
Immunofluorescence detection of CwpV ... 56
Quantitative PCR analysis of the ON/OFF cwpV genetic switch ... 56
Gene inactivation using the ClosTron system ... 57
Results ... 57
Creation of a R20291 lysogen carrying the ϕCD38-2 prophage ... 57
Overview of the transcriptomic data in R20291 and R20291LYS ... 58
Transcriptome of the ϕCD38-2 prophage ... 61
Transcriptome and re-annotation of the endogenous phi-027 prophage... 62
Interference of ϕCD38-2 with transcription of host genes ... 65
The cell wall protein CwpV is upregulated in the R20291LYS ... 67
Discussion ... 71
Effect of lysogeny on bacterial gene expression ... 73
Effect of lysogeny on cwpV expression ... 74
Conclusion ... 75 Acknowledgements ... 75 References ... 76 Chapitre III ... 83 Article 2 ... 83 Avant-propos ... 83
Résumé ... 84
Abstract ... 86
Author summary (200 words) ... 87
Introduction ... 88
Methods ... 90
Bacterial strains, bacteriophages and plasmids ... 90
Determination of phage titers and efficiency of plaquing (EOP) ... 92
Isolation of R20291OFF and R20291ON clones ... 92
Cloning and expression of CwpV-related constructions ... 93
Immunoblotting for detection of CwpV ... 94
Bacterial survival assays ... 95
Phage adsorption assays ... 95
Detection of phage DNA replication ... 95
Results ... 96
CwpV protects against phage infection ... 96
CwpV protection is highly selective toward Siphoviridae phages ... 102
The C-terminal domain of CwpV carries the antiphage activity ... 104
CwpV functions as a superinfection exclusion (Sie) system ... 105
Discussion ... 109 Concluding remarks ... 113 Authors' contributions ... 114 Acknowledgements ... 114 References ... 114 Chapitre IV ... 123 Discussion ... 123
Rappel des objectifs du projet ... 123
Les considérations préliminaires de l’étude ... 124
Le phage φCD38-2 dans son contexte biologique ... 124
R20291 dans son contexte biologique ... 125
Le choix de la technique ... 125
L’analyse du transcriptome viral ... 126
Le profil transcriptomique du prophage phi-027 ... 126
L’analyse du transcriptome bactérien ... 133
Observations générales ... 133
Analyse des fonctions spécifiques ... 134
Le rôle biologique de CwpV ... 141
Le rôle de CwpV dans l’infection virale ... 142
La protection est sélective envers les siphophages ... 142
La protection agit au niveau de l’injection d’ADN ... 144
Les hypothèses concernant le mécanisme d’inhibition d’injection d’ADN ... 144
Autres fonctions de CwpV ... 147 CONCLUSION... 148 REMERCIEMENTS ... 150 ANNEXE I ... 151 ANNEXE II ... 161 ANNEXE III ... 172
L
ISTE DES FIGURES
CHAPITRE I Figure 1………15 Figure 2………18 Figure 3………30 Figure 4………35 Figure 5………41 Figure 6………42 CHAPITRE II Figure 1………59 Figure 2………61 Figure 3………67 Figure 4………68 Figure 5………69 Figure S1………..58 Figure S2………..63 CHAPITRE III Figure 1………..100 Figure 2………..101 Figure 3………..105 Figure 4………..106 Figure 5………..107 Figure S1………..……98 Figure S2………..………99 CHAPITRE IV Figure 7………..131 Figure 8………..146L
ISTE DES TABLEAUX
CHAPITRE II Table 1………..59 Table 2………..66 Table S4………65 Table S5………53 CHAPITRE III Table 1.………..91 Table 2……….103 Table S1………93L
ISTE DES ABRÉVIATIONS
ABC ATP-binding cassette ADN Acide désoxyribonucléique ARN Acide ribonucléique
ARNm Acide ribonucléique messager ARNr Acide ribonucléique ribosomal ARNt Acide ribonucléique de transfert BHI Brain Heart Infusion
BLAST Basic Local Alignment Search Tool cAMP cyclic Adenosine Monophosphate CDAD Clostridium difficile associated disease cDNA Complementary deoxyribonucleic acid CDT Clostridium difficile toxin
CRISPRs Clustered regularly interspaced short palindromic repeats CWB2 Cell_wall_binding_2
CwpV Cell Wall Protein Variable dNTP Déoxyribonucléotides
DO600 Densité Optique à la longueur d’onde 600 nanomètres
EDTA Acide éthylène-diamine-tetraacétique EHEC Escherichia coli entérohémorragique FbpA Fibronectin-binding protein A Fis Factor for inversion stimulation
H-NS Heat-stable nucleoid-structuring protein HRP Horseradish Peroxydase
HTH Helix-turn-helix
ICD Infections à Clostridium difficile IHF Integration host factor
kDa Kilodaltons
LC-MS/MS Liquid Chromatography-tandem Mass Spectrometry LSS Leucine rich repeats
M Molaire
mM Millimolaires
MOI Multiplicity of Infection NAP North America Pulse field
NCBI National Center for Biotechnology Information
ng Nanogrammes
NGS Next-generation sequencing ORF Open reading frame
PaLoc Pathogenicity locus pb Paires de base
PBS Phosphate-Buffered Saline
PCR Polymerase Chain Reaction PFGE Pulsed-field gel electrophoresis PFU/ml Plaque-forming units per millilitre pH Potentiel hydrogen
ppGpp Guanosine tétraphosphate PTS Phosphotransférase system
qRT-PCR Quantitative Reverse-transcriptase Polymerase Chain Reaction RM Restriction-modification
RPKM Reads per kilobase of transcript per million reads mapped RPM Rotation Par Minute
RT-PCR Reverse-transcriptase Polymerase Chain Reaction Sie Superinfection exclusion
SDS Sodium Dodecyl Sulfate
SDS-PAGE Sodium Dodecyl Sulfate Polyacrylamide Gel Electrophoresis SLP Surface Layer Protein
TAE Tris-acétate EDTA T/AT Toxin/Antitoxin
TEM Transmission Electron Microscopy TMP Tape Measure Protein
TTSS Type three secretion system TY Tryptose-Yeast extract UA Uranyl-acétate
ufp/ml Unité formant des plages par millilitre w/v Rapport poids/volume
C
HAPITRE
I
I
NTRODUCTION
Nous ne sommes pas seuls
La coévolution des espèces est un concept clé qui a défini de nombreux aspects de l’existence des êtres vivants. Ce concept, hautement dynamique et très commun au sein de la biosphère, repose sur l’influence réciproque entre deux ou plusieurs espèces se traduisant par une évolution antagoniste ou mutualiste commune. Le corps humain, en tant que système biologique extrêmement complexe, n’échappe pas à la règle. Nous sommes habités par un nombre impressionnant de microorganismes qui, en réalité, dépasse le nombre de nos propres cellules dans un ratio 10:1 (Savage, 1977). Ce nombre titanesque est également accompagné par une extraordinaire diversité qui englobe surtout les bactéries, mais aussi des archéobactéries, fungi et virus (Human Microbiome Project, 2012). On estime que le tractus gastro-intestinal abrite à lui seul quelques centaines d’espèces bactériennes communément appelées le microbiote (Sperandio, 2012). En parallèle, il héberge une diversité étonnante de virus qui se chiffre à environ 1500 virotypes communément appelés le virome (Breitbart et al., 2003; Reyes et al., 2010). La très grande majorité de cette diversité virale est constituée de bactériophages (ou simplement phages), c’est-à-dire des virus capables d’infecter uniquement les bactéries. Le microbiote et le virome évoluent ensemble selon une dynamique précise et avec une certaine conséquence pour l’homme, cependant cette dépendance n’est pas exclusive aux espèces bactériennes commensales. Ainsi, de nombreux pathogènes ont évolué de pair avec leurs phages spécifiques selon un mode mutualiste qui assure les bénéfices pour les deux parties concernées au détriment de l’hôte. La colonisation de ce dernier par un pathogène opportuniste (processus appelé infection), se caractérise généralement par une disparition de la diversité générale du microbiote, apparition de dommages au site de l’infection et parfois la mort de l’hôte. On sait maintenant que les bactériophages sont intrinsèquement impliqués dans la biologie et la virulence d’un grand nombre de pathogènes et peuvent fortement influencer l’issue d’une infection. À cet égard,
les relations phages-bactéries vont bien au-delà de la simple interaction prédateur-proie et représentent un monde évolutif unique, fascinant et ayant des implications majeures pour la santé de l’humain. Pourtant, malgré l’importance du sujet, notre compréhension de cette relation est loin d’être complète.
Les bactériophages
Depuis leur découverte par Frederick Twort et Félix d’Hérelle en 1915 et 1917 respectivement, les phages ont continué de fasciner la communauté scientifique. Imaginé initialement par d’Hérelle comme un traitement aux infections bactériennes, l’intérêt pour les phages a diminué suite à la découverte et l’exploitation des antibiotiques. Toutefois, les phages reprennent un rôle central en définissant les grands paramètres et les premières découvertes en biologie moléculaire. Dû à la simplicité de leurs génomes et la facilité de manipulation, ils furent utilisés comme modèles pour les découvertes majeures tels que la nature du code génétique, la notion du gène ou encore la réplication et transcription du matériel génétique. Un siècle après leur découverte, nous en savons infiniment plus sur la biologie et l’évolution des phages, pourtant nous sommes loin d’en comprendre toutes les subtilités.
Ce qui est certain de nos jours, c’est la distribution ubiquitaire des phages. Ils sont retrouvés partout où les procaryotes prospèrent et dépassent typiquement le nombre de leurs hôtes par un facteur 10. Cette caractéristique leur vaut l’épithète de l’entité biologique la plus répandue sur Terre avec une estimation qui dépasse 1030 particules virales pour l’ensemble
de la biosphère (Chibani-Chennoufi et al., 2004; Mann, 2005). Morphologiquement, les phages se présentent sous toutes les formes imaginables, cependant la très grande majorité (96%) possède une structure commune caractérisée par l’absence d’une membrane périphérique et la présence d’une capside et d’une queue (Fokine et Rossmann, 2014). Ces phages font partie d’une des trois grandes familles soit Myoviridae (queue contractile), Siphoviridae (longue queue non contractile), ou Podoviridae (courte queue non contractile) regroupées dans le grand ordre des Caudovirales (Ackermann, 1998). Le génome d’un phage est typiquement modulaire et présente un regroupement localisé des gènes ayant une fonction similaire.
Partenaire ou adversaire?
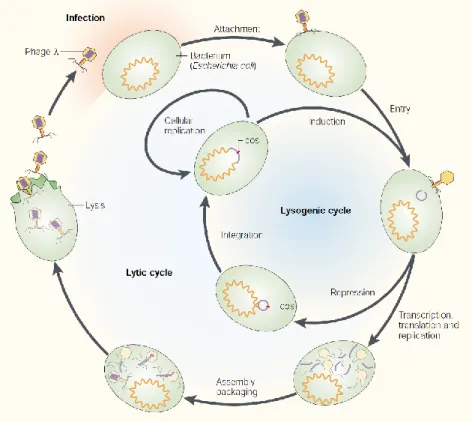
Ultimement, un phage peut être perçu comme un parasite égoïste ayant pour but unique la multiplication massive destinée à éviter l’extinction de la lignée. Après tout il ne s’agit que du matériel génétique (génome) protégé par un emballage protéique (capside). Pourtant, au fil du temps, les phages ont pu interagir, s’adapter et s’imposer pour finalement évoluer avec les hôtes présents dans leur environnement. Afin de bien saisir l’ampleur de cette relation, il est nécessaire de comprendre les fondements derrière le processus d’infection. En tenant compte du mode réplicatif des phages, il nous est possible de les séparer en deux grandes catégories : les phages strictement lytiques et les phages tempérés (ou à cycle lysogénique) (Figure 1, Chapitre I).
Figure 1 Le cycle lytique et lysogénique du coliphage λ. L’adsorption du phage est
suivie rapidement par l’injection du génome viral dans la cellule bactérienne. Dans le cas du cycle lysogénique, le génome viral est intégré dans le génome bactérien et se réplique en même temps que celui-ci. Dans le cas du cycle lytique, les composantes structurelles des phages sont produites et la cellule hôte est lysée pour libérer la progéniture virale. Reproduit avec l’autorisation de (Campbell, 2003)
Les phages strictement lytiques
L’infection par un phage strictement lytique entraine obligatoirement la mort de la cellule hôte. Dans cette optique, un phage lytique se comporte comme un prédateur hautement sophistiqué qui, suite à l’infection, détourne le système cellulaire en son avantage pour assurer la production de la progéniture virale. Cette prise d’assaut de la machinerie cellulaire est brutale et extrêmement rapide, de telle sorte que le destin d’une cellule infectée, la mort ou la survie, se décide typiquement dans les minutes suivant l’infection. Le résultat final est le relâchement de la progéniture virale qui peut infecter d’autres cellules avoisinantes et le cycle recommence. Ce mode de multiplication assure une augmentation locale de la population virale dans un temps minimal. Par conséquent, la multiplication d’un phage corrèle positivement avec la capacité reproductive de l’hôte dans un milieu donné (Riemann et al., 2000; Sandaa et al., 2009). La conséquence d’une multiplication virale rapide réside dans la disparition tout aussi rapide de l’hôte nécessaire pour l’infection. Cependant, une lyse importante de la population bactérienne entraine un relâchement de nutriments essentiels (carbone, azote, phosphate) sous forme de métabolites cellulaires tels que les sucres, acides aminés et acides nucléiques. Ce flux nutritif peut être considérable (Riemann et al., 2009) au point d’alimenter la même population bactérienne donnant lieu à un autre cycle de croissance bactérienne et de prédation par les phages. Cette dynamique relationnelle est omniprésente dans l’environnement marin résultant en 1023 infections par seconde et donnant lieu au
processus crucial du cycle de carbone (Fuhrman, 1999; Suttle, 2005). Ainsi, les phages strictement lytiques sont considérés comme les principaux régulateurs de la biomasse bactérienne dans l’environnement marin (Suttle, 2007), mais aussi des joueurs clés dans le contrôle des populations microbiennes dans les sols (Allen et al., 2010; Srinivasiah et al., 2008).
Les phages tempérés
Le mode réplicatif des phages tempérés diffère passablement avec celui des phages strictement lytiques. Certes, les phages tempérés peuvent suivre la voie lytique classique et donner lieu à une multiplication locale relativement rapide. Cependant, ils ont la possibilité de suivre une voie alternative appelée voie lysogénique qui assure la survie de l’espèce sans le génocide microbiologique propre aux phages lytiques.
Le mode de vie lysogénique repose sur le principe d’une coexistence passive du génome viral avec le génome bactérien. En d’autres termes, l’infection d’une cellule bactérienne par un phage tempéré peut se solder soit par la fusion des deux génomes, c’est-à-dire l’intégration du génome du phage dans celui de la bactérie, soit par une existence épisomale (à la manière d’un plasmide) du phage en question. Typiquement, le génome du phage est soumis aux mêmes contraintes que le génome bactérien et se réplique avec celui-ci de telle sorte que le matériel génétique viral est transmis efficacement à chaque génération de division bactérienne. Ce mode de réplication est nettement moins brutal que la voie lytique, mais il a le désavantage d’entièrement dépendre de l’hôte. Néanmoins, cette voie passive peut être interrompue en tout temps suite aux signaux internes ou externes, permettant au phage résidant de passer en mode lytique. Ce phénomène, appelé induction, entraine la mort de la cellule hôte, mais permet un relâchement de la progéniture virale qui peut infecter d’autres cellules avoisinantes. Ainsi, la régulation du cycle lytique et lysogénique est d’une importance capitale pour un phage.
Phage lambda (λ)
Assurément, le phage tempéré le plus connu et le plus étudié est le coliphage λ. Décrit pour la première fois en 1953 par Esther Lederberg (Lederberg et Lederberg, 1953), il a servi comme outil génétique durant plus d’un demi-siècle. De nos jours, λ est considéré comme l’archétype du phage tempéré. Il est en mesure d’infecter son hôte, Escherichia coli, de façon lytique ce qui résulte au relâchement d’une progéniture d’environ 150 phages à partir d’une seule cellule bactérienne infectée (Shao et Wang, 2009). Typiquement, ce type d’infection est préféré lorsque l’hôte prospère dans un milieu nutritif riche, et donc une augmentation de la population de l’hôte se traduit par une augmentation proportionnelle de la charge virale. À l’inverse, dans les conditions de limitation nutritive, λ préfère la voie lysogénique qui assure la protection du génome du phage à l’intérieur de la cellule bactérienne. Cette observation implique que le phage tempéré est en mesure de percevoir l’état physiologique de la cellule lors de l’infection et de s’ajuster en conséquence. Les effecteurs moléculaires du phage λ, intrinsèquement reliés dans un réseau génétique complexe, sont maintenant bien connus.
Le choix entre le cycle lytique ou lysogénique chez λ
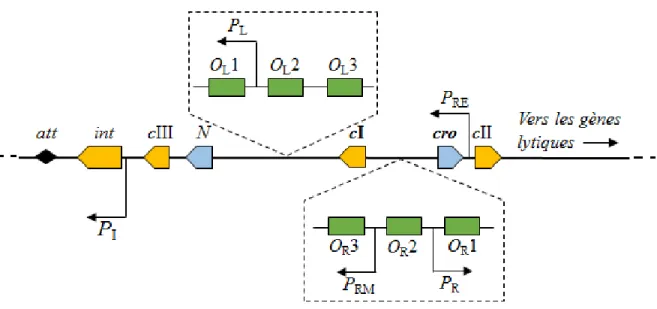
La régulation du cycle lytique-lysogénique chez λ est centrée autour de deux régulateurs transcriptionnels, CI et Cro, confinés dans une région précise du génome du phage illustrée à la Figure 2, Chapitre I.
Figure 2 La représentation schématique des régions du génome du coliphage λ impliquées
dans la prise de décision entre le cycle lytique et le cycle lysogénique. La figure n’est pas à l’échelle et la distance entre les gènes n’est pas représentative des distances réelles dans le génome. Les rectangles pointus représentent des gènes impliqués dans la lysogénie (orange) ou le cycle lytique (bleu). Tous les gènes impliqués dans ces processus ne sont pas représentés. Les rectangles verts illustrent les opérateurs. Les flèches noires sont indicatives des promoteurs. Modifié à partir de (Calendar, 2006)
Ces deux régulateurs sont antagonistes de sorte que l’expression du CI assure la voie lysogénique et inversement, l’expression du Cro assure la voie lytique. Cependant, l’équilibre entre l’expression et l’action de chacun des régulateurs est finement contrôlé via les promoteurs / opérateurs situés à proximité. Tout d’abord, une première région régulatrice se trouve entre les deux gènes cI et cro. Elle comporte deux promoteurs en direction opposée, PRM et PR, avec en plus trois opérateurs nommés OR1, OR2 et OR3. En principe, les deux
Par exemple, Cro lie les régions OR3-OR2-OR1 avec une affinité décroissante. De telle
manière, à des niveaux d’expression modérée, Cro lie tout d’abord la région OR3 bloquant
par le fait même l’expression du CI à partir du promoteur PRM. Ceci a pour effet de stimuler
la voie lytique. Or, avec les quantités croissantes de Cro, les sites OR2 et OR1 deviennent
occupés bloquant progressivement l’expression du Cro à partir du promoteur PR créant ainsi
une boucle d’autorégulation négative.
Le cas du CI est plus complexe. Par exemple, CI est en mesure de lier l’opérateur OR1
avec une grande affinité et les deux autres sites (OR2 et OR3) avec une faible affinité.
Cependant, cette liaison est coopérative, c’est-à-dire que la liaison sur le premier site (OR1)
stimule d’environ 200 fois la liaison sur le site suivant (OR2). Cette double liaison a deux
conséquences : premièrement, cela mène à la répression du promoteur PR (Cro réprimé) et
deuxièmement, cela active le promoteur PRM (CI activé). Le tout mène à une production en
continu du CI et une répression constante du Cro, avec comme conséquence finale la mise en place du cycle lysogénique. De plus, durant la lysogénie, CI agit sur la deuxième région régulatrice contenant trois opérateurs nommés OL1, OL2 et OL3. L’occupation des opérateurs
OL assure une répression du promoteur PL qui régule l’expression d’un autre facteur lytique
appelé N. Ainsi, CI et Cro sont au cœur même de cette régulation transcriptionnelle.
Cependant, lorsque le phage λ infecte une cellule, ni CI ni Cro ne sont pas présents d’emblée. Alors, comment la prise de décision entre le cycle lytique et le cycle lysogénique se fait-elle? La réponse se trouve dans la capacité du phage à percevoir l’état physiologique de la cellule et à réagir en conséquence. Les deux régulateurs CI et Cro seront exprimés peu après l’injection de l’ADN du phage dans la cellule bactérienne. L’expression du Cro est assurée par son propre promoteur fort PR. À l’inverse, le promoteur PRM du CI est
relativement faible en absence du CI et donc l’expression du CI est assurée par un troisième promoteur appelé PRE. En principe, la fluctuation entre les niveaux d’expression du Cro et
CI décideront de la voie à suivre. Cro étant exprimé en continu, c’est le CI qui a le potentiel de pencher l’équilibre du côté du cycle lysogénique. Or, l’expression du CI à partir du PRE
est stimulée de manière directe par un troisième facteur clé appelé CII qui a son tour est sous la régulation complexe par le facteur CIII et la protéase de l’hôte FtsH. Ainsi, CII est dégradé rapidement par la protéase FtsH, mais l’action de la protéase bactérienne est régulée par CIII qui agit comme un inhibiteur compétitif. Également, l’activité protéolytique de FtsH est
inhibée par la présence de l’AMP cyclique (cAMP) et de la guanosine tétraphosphate (ppGpp), deux messagers qui sont synthétisés lorsque la cellule est en carence de carbone et d’acides aminés.
Ainsi, la décision d’un phage tempéré à établir le cycle lysogénique plutôt que le cycle lytique est multifactorielle et implique une cascade interactive et une relation complexe avec les facteurs de l’hôte. Cependant, la dépendance envers l’hôte dans la prise de décision lyse-lysogénie n’est pas un aspect isolé. La nature même de la lyse-lysogénie, c’est-à-dire la coexistence du génome viral et du génome bactérien, peut avoir des répercussions immenses sur l’hôte bactérien.
La lysogénie représente plutôt la règle que l’exception
Avec l’arrivée des techniques de séquençage à haut débit, le nombre de génomes bactériens séquencés a augmenté de manière exponentielle. L’analyse de cette quantité phénoménale d’informations génomique a permis de constater que la distribution des prophages est beaucoup plus grande que ce qui était soupçonné auparavant (Canchaya et al., 2003). Ainsi, la lysogénie est un phénomène commun au sein des espèces bactériennes. De plus, plusieurs espèces bactériennes peuvent contenir plus d’un phage dans leurs génomes donnant lieu au phénomène de la polylysogénie. Par exemple, certaines souches de Streptococcus pyogenes possèdent de 4 à 6 prophages dans leur génome, représentant de 12-16% d’ADN génomique total (Aziz et al., 2005; Ferretti et al., 2001; Smoot et al., 2002). Un autre cas extrême est décrit chez Escherichia coli O157:H7 ou on dénombre jusqu’à 18 prophages pouvant atteindre 16% d’ADN génomique total (Hayashi et al., 2001; Ohnishi et al., 2001). Néanmoins,
cette distribution presque ubiquitaire des prophages dans les génomes bactériens s’explique partiellement par une coévolution mutualiste alimentée par les avantages multiples que vont procurer les phages tempérés à leurs hôtes.
Coévolution des phages tempérés avec leurs hôtes bactériens
Notre intérêt pour les bactéries provient en grande partie de l’impact qu’elles ont sur la santé humaine. Ainsi, il n’est pas surprenant de constater qu’un effort particulier a été naturellement dédié aux études portant sur les bactéries pathogènes. Assez tôt dans cette exploration globale, les phages tempérés ont été mis en évidence comme des facteurs
importants et même déterminants dans la virulence de certains pathogènes. Par exemple, dès 1927, il a été remarqué que la capacité toxinogène des streptocoques était transférable via les surnageant des cultures. Aujourd’hui, on sait que ce phénomène de transfert génique (appelé transduction) était causé par les phages encodant les toxines érythrogènes (Johnson et al., 1986). Également, dès le début des années 1960, il est devenu évidant que la toxine diphtérique, facteur de virulence principal chez Corynebacterium diphtheriae, est encodée par un prophage (Barksdale et al., 1960). Suite à ces observations initiales, de nombreux phages tempérés ont été identifiés comme véhicules de toxines puissantes chez de nombreux pathogènes notoires tels que Clostridium botulinum (toxine C1), Streptococcus pyogenes (toxines type A et C), Staphylococcus aureus (Enterotoxin A et C), Vibrio cholerae (Cholera Toxin), Pseudomonas aeruginosa (Cytotoxine) et Escherichia coli (Shiga Toxines 1 et 2) (Brussow et al., 2004). Dès lors, il devint évident que les phages tempérés, malgré leur nature potentiellement lytique, pouvaient être bénéfiques pour leurs hôtes bactériens, renversant du coup le dogme qui illustrait les relations phage-hôte comme une simple extension du concept prédateur-proie. De nos jours, les phages tempérés sont perçus comme un véritable réservoir environnemental de toxines ayant la capacité de mobilité et de dissémination (Casas et al., 2006). Concrètement, les facteurs de virulence encodés sur les phages, comme les toxines, donnent la possibilité aux bactéries hôtes d’exploiter une nouvelle niche ou de mieux utiliser une niche existante. Cet avantage peut devenir crucial dans la mesure où la bactérie-hôte évolue dans un environnement nécessitant une compétition directe avec une flore existante; d’ailleurs, c’est le cas pour la plupart des infections opportunistes chez l’Homme.
Ultimement, l’avantage procuré par l’acquisition d’un facteur de virulence via un phage tempéré sera bénéfique pour la bactérie hôte, mais également pour le phage qui l’encode. Cet aspect est lié à l’interdépendance phage-hôte : une meilleure propagation de l’hôte dans un environnement donné favorisera également une meilleure propagation des phages spécifiques à cet hôte. Cependant, seulement une minorité des phages tempérés caractérisés jusqu’à présent semblent encoder dans leurs génomes des facteurs de virulence démontrés. Certes, cette proportion peut augmenter avec la découverte de nouveaux phages ou encore la caractérisation des nouvelles protéines encodées par les phages, néanmoins il semblerait qu’une relation phage-hôte plus subtile est de mise dans la majorité des cas.
Régulation de l’expression génique durant le cycle lytique et lysogénique
La relation interdépendante phage-hôte a été extensivement explorée au cours des dernières années. Beaucoup de ces études ont visé à comprendre le développement du cycle lytique chez les coliphages modèles tels que le phage T4, T7 et λ (Roucourt et Lavigne, 2009). Typiquement, le transcriptome d’un phage lytique (ou d’un phage tempéré en mode lytique) va suivre une activation temporelle qui consiste à exprimer simultanément des groupes de gènes associés à une fonction précise. Ainsi, les premiers gènes exprimés durant un cycle lytique (gènes précoces) servent généralement à détourner la machinerie cellulaire de l’hôte dans le but de servir uniquement l’infection en cours et à protéger l’ADN viral des nucléases bactériennes. Ce processus est habituellement atteint par l’expression de facteurs sigma alternatifs et des protéines qui déstabilisent ou modifient les ARN polymérases de l’hôte. La deuxième étape consiste en l’expression des gènes médians qui assurent la réplication efficace et rapide du génome viral. La dernière série de gènes (gènes tardifs) est constituée des composantes structurales des particules virales (capside, queue) ainsi que les gènes nécessaires à la lyse bactérienne et la libération de la progéniture. Cette régulation transcriptomique temporelle durant un cycle lytique est globalement conservée parmi les différents phages. Par exemple, les phages T4 et λ infectant E. coli (Luke et al., 2002; Osterhout et al., 2007), phage TP901-1 infectant Lactococcus lactis (Madsen et Hammer, 1998), phages DT1 et 2972 infectant Streptococcus thermophilus (Duplessis et al., 2005), phage LUZ19 infectant P. aeruginosa (Lavigne et al., 2013) et phage Giles infectant Mycobacterium smegmatis (Dedrick et al., 2013) ont tous un profil d’expression similaire durant leur cycle lytique.
Contrairement au cycle lytique, la lysogénie est un mode qui ne nécessite aucune régulation temporelle. Typiquement, un phage en mode lysogénie est transcriptionellement silencieux. Les seuls gènes exprimés sont ceux nécessaires au maintien de la lysogénie tels que les répresseurs et parfois les unités transcriptionnelles indépendantes qui constituent les gènes de conversion lysogénique. Par exemple, les expériences d’hybridation de type Northern ont permis de limiter l’activité transcriptomique des prophages résidant dans diverses espèces de Lactobacillus principalement à proximité du répresseur (Ventura et al., 2006; Ventura et al., 2004). De façon similaire, le profil transcriptomique a été restreint à seulement deux régions dans les prophages Sfi21 et O1205 du S. thermophilus. La première
région englobait une section du module de la lysogénie contenant le répresseur et la deuxième région comprenait un groupe de gènes sans fonction identifiable à la fin du module de lyse. De par leur position génomique et du fait qu’ils soient transcrits durant la lysogénie, ces gènes peuvent être potentiellement impliqués dans la biologie de l’hôte (Ventura et al., 2002). De la même manière, l’activité transcriptomique du prophage Bbr-1 résidant dans le génome de Bifidobacterium breve UCC 2003 a été confinée surtout au niveau du répresseur. Cependant, un transcrit correspondant au gène encodant une β-glucosidase a également été détecté. Encore une fois, l’hypothèse retenue est que gène peut potentiellement jouer un rôle dans le métabolisme des sucres chez l’hôte.
À l’opposé de ces études qui ont utilisé l’hybridation de type Northern, des nouvelles techniques ayant une meilleure résolution ont été mises à profit récemment pour la détermination du profil transcriptomique des prophages. Ainsi, le transcriptome du coliphage λ à l’état du prophage a été déterminé en utilisant les micropuces à ADN (Osterhout et al., 2007). Tel qu’attendu, le répresseur cI constituait le gène le plus exprimé. Cependant, sept autres transcrits ont été détectés incluant deux gènes (bet et xis) impliqués dans la recombinaison et l’excision reflétant probablement le phénomène de l’induction spontané. De plus, les gènes rexAB encodant le système d’immunité ont été exprimés presque aussi fortement que le répresseur cI. Finalement, deux composantes de la queue du phage (lambdap14 et lambdap18) ont été détectées ainsi qu’un gène codant pour une protéine sans fonction connue (orf63).
RNA-seq est une technique récente et relativement puissante pour les études transcriptomiques. Contrairement aux méthodes d’hybridation (Northern et micropuces à ADN), cette technique est basée sur les récents développements au niveau du séquençage à haut débit. Techniquement, l’ARN total est isolé dans les conditions voulues, rétrotranscrit en ADN complémentaire (ADNc) pour ensuite être séquencé et aligné sur le génome référence. De telle manière, on obtient une image globale, sensible et très précise de la transcription à un moment donné (Garber et al., 2011; Marioni et al., 2008; Mortazavi et al., 2008). Cette technique a été utilisée pour déterminer le transcriptome du phage Giles chez M. smegmatis (Dedrick et al., 2013). Comme prévu, l’expression du répresseur (gp46) a été détectée, avec cependant une amplitude dépassant largement celle décrite pour le phage λ (Ptashne et al., 1980). Mis à part le répresseur, peu de gènes avaient une expression
significative. En effet, une expression très faible a été détectée pour seulement 6 autres gènes, dont trois potentiellement impliqué dans le maintien de la lysogénie (gènes 44-46) et trois autres sans fonction connue (2-4). Cependant, une région particulière d’environ 100 pb a été très fortement exprimée sans toutefois couvrir un cadre de lecture ouvert suggérant qu’il s’agit d’un petit ARN non-codant. Le rôle de ce dernier n’est pas connu, cependant sa délétion n’a pas d’impact ni au niveau du cycle lytique ni au niveau du cycle lysogénique supportant encore une fois l’idée d’un rôle chez l’hôte.
En résumé, les deux modes de vie des phages sont caractérisés par des profils transcriptomiques différents et uniques. Ainsi, le cycle lytique implique une expression séquentielle massive, mais finement contrôlée qui permet une régulation temporelle et spatiale des gènes requis pour l’accomplissement du cycle. À l’inverse, le cycle lysogénique est caractérisé par une expression très limitée et étroitement liée aux répresseurs et gènes similaires nécessaires au maintien de la lysogénie. Cependant, durant la lysogénie, des gènes de conversion lysogénique peuvent être exprimés indépendamment du reste du génome du phage. Cette expression différentielle est possiblement sous régulation des facteurs de transcription de l’hôte.
Impact du cycle lytique et lysogénique sur le transcriptome de l’hôte
Inévitablement, la conséquence ultime d’un cycle lytique est la mort de la cellule infectée. Cependant, il est intéressant de constater que la réponse transcriptomique de l’hôte face à une infection lytique va varier considérablement en fonction du phage qui infecte la cellule. Par exemple, l’infection lytique par deux phages tempérés infectant L. lactis, Tuc2009 et c2, provoque un changement relativement modeste sur le transcriptome bactérien, représentant respectivement 5.5% et 7.5% du génome de l’hôte (Ainsworth et al., 2013). Entre autres, l’altération transcriptomique présentait un enrichissement des gènes impliqués dans le métabolisme des polysaccharides, de l’azote, des acides aminés et de la séquestration et de l’utilisation des purines et pyrimidines. Une situation relativement similaire a été observée lors de l’infection d’E. coli par le phage lytique PRD1 (Poranen et al., 2006) et du phage PRR1 chez P. aeruginosa (Ravantti et al., 2008). Dans ces cas, il s’agit de simplement rediriger les processus métaboliques de l’hôte afin d’assurer un apport énergétique suffisant. En opposé, l’infection d’E. coli par la série-T des phages lytiques (T2,
T4, T5 et T7) provoque un arrêt presque instantané de la plupart des fonctions cellulaires, accompagné peu après par la dégradation complète du génome bactérien (Hesselbach et Nakada, 1977; Koerner et Snustad, 1979; Sadowski, 1971; Warner et al., 1975). Dans ces cas, la cellule entière est séquestrée et les composantes structurelles (ex. les acides nucléiques dérivés de la dégradation du génome de l’hôte) servent directement comme ressource énergétique. Malgré cette différence dans la subversion de l’hôte, le but ultime est le même et il consiste à assurer un flux énergétique suffisant pour soutenir la production de la progéniture virale.
À l’inverse du cycle lytique, l’altération du transcriptome de l’hôte durant le cycle lysogénique est moins dramatique, néanmoins elle peut avoir des conséquences très importantes. Récemment, un exemple fascinant de la relation phage-hôte a été décrit chez E. coli (Wang et al., 2010). En plus de contenir plusieurs prophages fonctionnels, la souche modèle d’E. coli K12 contient également neuf prophages défectifs (CP4-6, DLP12, e14, rac, Qin, CP4-44, CPS-53, CPZ-55 et CP4-57). Ces prophages sont généralement caractérisés par l’incapacité de la production des particules virales, formation de plages de lyses ou de la lyse cellulaire. Ces défauts proviennent de multiples réarrangements chromosomiques qui ont amené à une perte de fonction et une désintégration de la structure génomique des prophages. Ces prophages défectifs (ou cryptiques) ont longtemps été considérés comme superflus, c’est-à-dire sans aucune utilité pour la bactérie hôte. Cependant, lorsque l’ensemble des neuf prophages cryptique est enlevé du génome bactérien, la souche ainsi générée démontre plusieurs altérations phénotypiques dont un ralentissement de croissance, un défaut de formation de biofilm ainsi qu’une plus faible capacité à résister à certains antibiotiques (quinolones et β-lactames) et aux stress environnementaux (pH, température, stress osmotique et oxydatif). En combinant les mutants de délétion, les micropuces à ADN et le PCR quantitatif en temps réel (qRT-PCR), les auteurs ont réussi à identifier les gènes responsables de cette altération phénotypique. La résistance accrue aux quinolones et β-lactames est en relation directe avec l’expression de deux gènes, dicB et kilR, situés respectivement dans les prophages cryptiques rac et Qin. De manière similaire, trois gènes de fonction inconnue, yfdK, yfdO and yfdS situés dans le prophage cryptique CPS-53 sont responsables de la résistance au stress oxydatif. Finalement, l’altération au niveau de la
production du biofilm peut être attribuée aux prophages cryptiques e14 (gènes intE and ymfD) et rac (gènes intR, stfR, ydaF, ydaS and ydaW).
De la même manière, un exemple intéressant a récemment été décrit chez Bacillus anthracis, agent causal de la maladie de l’anthrax (Schuch et Fischetti, 2009). Dans cette étude, les auteurs ont comparé l’effet phénotypique qu’avait l’introduction de certains phages tempérés isolés dans la nature (ex. Wip1, Wip2, Wip4, Wip5, Frp1, Frp2, Slp1, Bcp1 et Wβ) sur une souche modèle dépourvue de tout prophage (B. anthracis ΔStern). De façon intéressante, la lysogénisation de la souche ΔStern avait des effets sévères sur plusieurs processus bactériens importants. Par exemple, l’introduction des phages Bcp1, Wip1, Wip4 et Frp2 avait un effet drastique sur la capacité de la bactérie à produire un biofilm robuste et viable. De la même manière, la capacité de sporulation a été affectée à la hausse via l’introduction des phages Wip4, Wip5 ou Frp1 ou à la baisse via l’introduction des phages Wβ, Wip1, Wip2 ou Frp2. Cette altération de la production de biofilm et de la sporulation avait un impact considérable quant à la capacité de la bactérie à survivre dans le sol pendant une période prolongée. Ultimement, en couplant une librairie d’expression d’ADN complémentaire pour les phages Bcp1 et Wip4 aux tests phénotypiques, les auteurs ont réussi à identifier deux régions spécifiques (bcp25-26 et wip48-49) codant pour des facteurs sigma alternatifs. Ainsi, il a été proposé que les phages Bcp1 et Wip4 détournent la régulation transcriptionnelle de l’hôte avec une reprogrammation des ARN polymérases via les facteurs sigma alternatifs.
Ces deux exemples démontrent bien la complexité et l’enchevêtrement des régulations transcriptomiques entre les phages tempérés et leurs hôtes bactériens. Cependant, dans les deux cas, un effet phénotypique a été facilement observable, ce qui malheureusement n’est pas applicable dans toutes les situations. Très souvent, des conditions particulières sont nécessaires afin d’observer un impact significatif et ainsi certaines interactions phage-hôte passent sous le radar. Afin de contourner ce problème, il est possible d’utiliser une approche globale basée sur l’analyse du transcriptome entier de l’hôte en présence ou en absence d’un phage afin de décrypter les relations phage-hôte qui ne résultent pas nécessairement en un phénotype facilement observable. Toutefois, cette manière de procéder n’a pas été appliquée systématiquement et seulement quelques exemples sont disponibles actuellement.
Ainsi, la confirmation que le cycle lysogénique est moins perturbateur pour l’hôte que le cycle lytique a été obtenue en examinant l’impact de la lysogénisation du phage λ sur le transcriptome d’E. coli (Chen et al., 2005). Le niveau d’expression de seulement 8 gènes bactériens a été altéré dans le lysogène comparativement à la souche sauvage. Ces changements transcriptomiques concernaient pour la plupart des gènes impliqués dans des processus métaboliques (pckA, phosphoenolpyruvate carboxykinase; nagA, N-acetylglucosamine-6-phosphate deacetylase) transport (b0572 et b0574, transporteurs de cuivre) et les prophages résidants (b2002, prophage CP4-44; b0557 prophage DLP12). Des résultats similaires ont été obtenus dans une étude subséquente portant sur la relation lysogénique entre λ et une autre souche d’E. coli (Osterhout et al., 2007). Dans cette étude, le niveau d’expression de 18 gènes bactériens a été altéré suite à la lysogénisation par le phage λ. Il s’agissait principalement de gènes codant pour des protéines de transport impliqué dans le métabolisme cellulaire tels que proWX (proline), pstB (phosphate), potCB (spermidine), lldPR (L-lactate). Finalement, mis à part le couple classique λ-E. coli, la relation prophage-hôte a été étudiée seulement chez L. lactis UC509 (Ainsworth et al., 2013). La transcription de 44 gènes a été altérée (9 à la hausse et 35 à la baisse) suite à la réintroduction du phage Tuc2009 dans une souche de L. lactis préalablement curée (UC509.9). Parmi les gènes régulés à la baisse, on dénote des gènes codant pour les protéines impliqués dans synthèse des nucléotides (ntd, dukA, nudH), métabolisme des acides aminés (hisB, trpAD), respiration (menE, hemH) ainsi qu’une série de régulateurs transcriptionnels de type MarR (uc509_0317 et uc509_0706), lysR (uc509_0396), MerR (uc509_1191), TetR (uc509_1462) et de type HTH (uc509_2032). Parmi les gènes régulés à la hausse, on remarque des gènes codant pour des transporteurs dont glycérol (glpF2), cadmium et ammonium (cadA et amtB), lipides (uc509_2209) et le fer (uc509_1435 et uc509_1436).
En résumé, l’ensemble de ces études nous montre que malgré une différence dans les espèces bactériennes ciblées, certains thèmes communs émergent dans les interactions phage-hôte. Par exemple, la nature du mode lytique requiert une quantité d’énergie considérable afin de soutenir la production massive de la progéniture virale. Donc, il est commun d’observer une subversion des fonctions métaboliques bactériennes suite au développement d’un cycle lytique. Ce détournement peut être relativement modeste, dans les cas où les phages exploitent la machinerie cellulaire existante, mais parfois on observe une
séquestration totale des ressources cellulaires menant, entre autres, à une dégradation du génome bactérien. À l’opposé, la lysogénie représente un mode de vie beaucoup plus passif. L’expression de la majorité des gènes du phage est réduite par les répresseurs responsables du maintien de la lysogénie. Toutefois, les études démontrent que l’interaction avec le transcriptome bactérien n’est pas nulle. Encore une fois, le thème commun est centré autour des gènes métaboliques même si les raisons derrière cette altération ne sont pas bien comprises. De plus, cette interaction avec l’hôte semble reposer sur les régulateurs transcriptionnels encodés par les phages tels que les répresseurs (Chen et al., 2005) ou les facteurs sigma alternatifs (Schuch et Fischetti, 2009).
Induction spontanée et son rôle dans la biologie de l’hôte
Comme mentionnée précédemment, la lysogénie est un état très stable, néanmoins ce cycle peut être rompu sous certaines conditions et donner lieu au phénomène d’induction, caractérisé par le développement lytique du prophage résidant. Habituellement, l’induction d’un prophage est dépendante de la réponse SOS de l’hôte. Ce dernier est généralement activé soit suite à l’accumulation des dommages à l’ADN causés par la lumière UV ou les agents mutagènes (induction provoquée) soit par des aberrations dans la réplication du génome bactérien (induction spontanée). Dans le cas du prophage λ, l’activation du système SOS entraine l’activation de la protéine RecA qui à son tour stimule l’activité autoprotéolytique du répresseur CI. La diminution du niveau du régulateur transcriptionnel CI lève la répression des promoteurs lytiques (dont le PR, voir Figure 2, Chapitre I) permettant ainsi l’expression
du Cro et l’établissement du cycle lytique. Cependant, tous les prophages ne sont pas activés de la même manière. Dans le cas du coliphage 186, l’induction est dépendante du système SOS, mais indépendante de la protéine RecA. En effet, une autre composante du système SOS (protéine LexA) est responsable de l’activation d’un antirépresseur viral (protéine Tum) qui à son tour interagit directement sur CI pour lever l’inhibition des promoteurs lytiques (Lamont et al., 1989; Shearwin et al., 1998). D’autres phages (N15 d’E.coli, Fels-2 de Salmonella enterica et CTXΦ de V. cholerae) présentent un mécanisme d’activation similaire, dépendant de LexA plutôt que RecA (Bunny et al., 2002; Mardanov et Ravin, 2007; Quinones et al., 2005).
Ainsi, même à l’état de prophage, la perception de l’état physiologique de l’hôte est d’une grande importance pour les phages tempérés. Lorsque les conditions deviennent critiques (accumulation importantes de dommages, activation du système SOS), le cycle lytique est enclenché et le virus s’échappe pour éventuellement infecter d’autres hôtes. Alors, en principe, un prophage peut être considéré comme une bombe à retardement et présente un danger perpétuel pour l’hôte bactérien. Cependant, les bactéries ont évolué de manière à tirer un certain profit de cette situation. Par exemple, le relâchement de certaines toxines encodées par les prophages tels que la toxine Shiga Stx1 ou la toxine diphtérique est possible grâce à la lyse bactérienne provoquée par l’induction des prophages (Barksdale et al., 1960; Wagner et al., 2002). De plus, il semblerait que le relâchement de la toxine Shiga Stx2 chez E. coli entérohémorragique (EHEC), causé par l’induction spontanée des prophages, a pour effet de reloger certains récepteurs sur la surface des cellules épithéliales du tractus gastro-intestinal ce qui augmente l’adhésion bactérienne (Los et al., 2012; Xu et al., 2012). Également, la formation de biofilm par plusieurs espèces bactériennes est soutenue par le relâchement d’ADN chromosomique suite à la lyse cellulaire causée par l’induction des prophages (Carrolo et al., 2010; Godeke et al., 2011; Petrova et al., 2011).
En résumé, les divers exemples exposés dans les sections précédentes illustrent bien les aspects de l’interdépendance et de la coévolution qui semblent façonner les relations phages-bactéries. Toutefois, la nature de cette relation présente une certaine dualité. Certes, les phages peuvent être extrêmement bénéfiques pour leurs hôtes bactériens, mais ils sont avant tout un danger perpétuel et omniprésent, et donc cette relation peut être également qualifiée d’antagoniste. Alors, il n’est pas surprenant de constater qu’au fil du temps, les bactéries ont développé un arsenal impressionnant de stratégies de défense qui permettent de limiter ou contrôler les interactions avec les phages et ainsi maintenir un équilibre relatif entre les bénéfices et les dangers que présentent ces relations.
« La course à l’armement »
Les interactions compétitives dans la nature, telle que celle décrite précédemment entre les phages et les bactéries, mènent invariablement à une transformation continuelle qui se traduit par l’adaptation de l’hôte (bactérie) et la contre-adaptation du parasite (phage) (Stern et Sorek, 2011). Cette relation constitue la base du concept de « la course à l’armement »
(phage-host arms race), ce qui implique qu’une exposition continuelle aux phages va mener à l’émergence des mutants bactériens, résistants à l’infection. Cette adaptation peut être active via l’acquisition ou la modification des systèmes antiphages ou encore passive via les mutations ponctuelles ou modification des composantes intrinsèques. Dans les deux cas, le temps requis pour l’adaptation et la contre-adaptation est extrêmement court ce qui stimule la coévolution des deux entités et l’apparition de mutants fonctionnels. Les stratégies évasives employées par les hôtes sont variées et peuvent intervenir à n’importe quel stade d’infection, soit au niveau de l’adsorption, de l’injection d’ADN viral ou encore les étapes subséquentes telles que la réplication ou la transcription des gènes viraux.
Ces stratégies défensives bactériennes et les contre-adaptations des phages seront brièvement discutées dans les sections qui suivent.
L’adsorption
La première étape d’un cycle infectieux, qu’il soit lytique ou tempéré, consiste en adsorption de la particule virale sur la cellule hôte. Cette étape présente une difficulté considérable pour les phages due à l’immense variabilité présente au niveau des surfaces cellulaires bactériennes. Le processus d’adsorption viral se fait typiquement en deux étapes. Une première étape réversible implique une reconnaissance d’une structure protéique ou glycoprotéique commune (récepteur primaire) sur la surface cellulaire. Suite à cette première reconnaissance positive, un deuxième contact irréversible est établi avec un constituant spécifique (récepteur secondaire) de la paroi cellulaire ce qui assure un ancrage permanent du phage à la surface (Figure 3, Chapitre I).
Un mécanisme commun employé par les bactéries consiste à muter ou à masquer les récepteurs reconnus par les phages. Par exemple, de multiples mutants spontanés du gène csaB chez B. anthracis ont été isolés et positivement corrélés à une résistance à l’infection par le phage lytique AP50c (Chen et al., 2010). CsaB est une protéine ancrée sur la surface bactérienne et impliquée dans la maturation de la paroi cellulaire (S-layer) (Mesnage et al., 2000). Les mutations identifiées sont délétères de telle sorte qu’une protéine fonctionnelle n’est pas exprimée et des défauts majeurs dans la maturation de la paroi cellulaire sont observés (Bishop-Lilly et al., 2012). Une situation similaire est observée chez certains coliphages qui nécessitent les constituants internes du cœur lipopolysaccharidique (LPS) de la surface bactérienne pour l’infection. Lorsque le LPS de S. enterica est modifié grâce aux antigènes O de nature polysaccharidique, l’adsorption du phage P1 est abolie (Ho et Waldor, 2007; Ornellas et Stocker, 1974). À l’inverse, certains phages ont évolué à reconnaitre spécifiquement les antigènes O (Xu et al., 2013) (Shin et al., 2012; Steinbacher et al., 1997) (Pajunen et al., 2000; Perry et al., 2009). De plus, la production d’une matrice extracellulaire peut empêcher les phages à accéder à leur récepteur spécifique (Hanlon et al., 2001; Scholl et al., 2005). À l’inverse, les phages peuvent acquérir des enzymes capables de dégrader ces structures cellulaires. D’autres mécanismes qui préviennent l’adsorption des phages ont été décrits, comme la production d’inhibiteurs qui masquent spécifiquement les récepteurs cellulaires reconnus par les phages (Nordstrom et Forsgren, 1974; Pedruzzi et al., 1998).
L’injection d’ADN
La reconnaissance d’un récepteur spécifique sur la surface cellulaire lors de l’étape de l’adsorption stimule une série d’évènements irréversibles qui mènent à l’injection d’ADN viral dans la cellule hôte (Kutter et Sulakvelidze, 2005). Le mécanisme moléculaire derrière
Figure 3 Différentes étapes de l’adsorption du phage p2 infectant L. lactis. A) Les phages p2
se retrouvent à proximité de la cellule bactérienne. B) Une première interaction implique la reconnaissance des structures communes de la surface bactérienne. Cette interaction est faible, mais suffisante pour retenir le phage sur la paroi bactérienne. C) Une interaction spécifique assure l’ancrage permanent du phage suivit de D) l’activation de la plaque basale et d’un changement conformationnel des protéines de structure ce qui va ultimement provoquer l’injection du génome viral dans le cytoplasme bactérien. Reproduit avec autorisation de (Bebeacua et al., 2013)
ce processus est variable d’un phage à un autre. Pour certains phages (ex. coliphage λ), l’ADN viral, compacté sous haute pression dans la capside, est simplement éjecté suite aux changements conformationnels des composantes de la queue du phage dû à l’interaction avec le récepteur LamB (Evilevitch et al., 2003). Par contre, pour d’autres phages (ex. coliphage T7), le processus d’injection est dépendant de la transcription bactérienne (Kemp et al., 2005; Molineux, 2001).
Divers systèmes qui permettent un blocage de l’injection d’ADN viral ont été décrits, mais on connait encore très peu les mécanismes moléculaires sous-jacents. Autre fait intéressant, tous les systèmes qui bloquent le transfert d’ADN viral dans la cellule bactérienne sont encodés par les phages et actuellement des systèmes bactériens n’ont pas encore été décrits. Le nom utilisé pour décrire l’ensemble des systèmes qui agissent au niveau de l’injection d’ADN est superinfection exclusion ou simplement Sie.
Le coliphage lytique T4 possède deux systèmes Sie, encodé par les gènes imm et sp. Les deux systèmes sont exprimés peu après l’infection d’une cellule par le phage T4 et préviennent une surinfection par d’autres phages similaires. Cependant, ils agissent séparément et leur mode d’action est différent. Par exemple, le système Imm est localisé au niveau de la membrane interne chez E.coli et possiblement associé au récepteur. Cette association altère le site d’injection et l’ADN entrant est dévié dans l’espace périplasmique au lieu de pénétrer dans le cytosol de la cellule (Lu et al., 1993). Le système Sp est également situé au niveau de la membrane, mais son mécanisme d’action est différent. Ce système agit comme inhibiteur de l’activité lysozyme encodée par gp5 sur le génome du phage T4 et ainsi prévient la dégradation du péptidoglycan et l’injection subséquente de l’ADN viral (Lu et Henning, 1994). D’autres systèmes Sie ont été identifiés chez diverses espèces d’Enterobacteriaceae, cependant le mécanisme précis n’a pas été décrit. Par exemple, le système Sim encodé par le phage P1 prévient la surinfection sans toutefois affecter l’adsorption des particules virales ni la réplication d’ADN du phage si celui-ci est transféré par transfection (Kliem et Dreiseikelmann, 1989). Également, le système SieA encodé par le phage lysogénique P22 infectant S. enterica agit également au niveau de la membrane et prévient l’infection par les phages L, MG148 et MG40 (Ebel-Tsipis et Botstein, 1971; Hofer et al., 1995; Susskind et al., 1971). Quelques systèmes Sie ont été décrits chez les bactéries à Gram positif. Le premier système décrit, nommé Sie2009, est encodé sur le génome du phage
tempéré Tuc2009 qui infecte une série de souches de L. lactis (McGrath et al., 2002). Tout comme pour les systèmes décrits plus haut, Sie2009 empêche l’infection par un groupe de
phages génétiquement différents du Tuc2009 sans toutefois affecter l’adsorption des particules virales. Subséquemment, trois autres systèmes semblables ont été identifiés sur des prophages présents dans trois différentes souches de L. lactis, mais aucun mécanisme moléculaire n’a été décrit (Mahony et al., 2008). À l’inverse, le système Sie encodé par le phage TP-J34 infectant Streptococcus thermophilus a été étudié plus en détail. Tout d’abord, il a été montré qu’une lipoprotéine encodée par le gène ltp sur le génome du TP-J34 est en mesure d’interférer avec l’infection de certains phages (Sun et al., 2006). Par la suite, cet effet a été attribué à une interaction directe de LtpTP-J34 avec les composantes de la queue du
phage ce qui empêchait son bon positionnement au niveau de la membrane. Des mutants de phages, partiellement insensibles à l’effet de LtpTP-J34, ont été isolés et leur caractérisation
par la microscopie électronique et le séquençage de génomes entiers a montré une altération au niveau de la tail tape measure protein (TMP), une composante essentielle de la queue du phage. La structure tridimensionnelle de la LtpTP-J34 a été obtenue et des prédictions
bio-informatiques in silico ont montré une interaction possible entre les charges négatives de la LtpTP-J34 et les charges positives de la TMP (Bebeacua et al., 2013).
Interférence au niveau des étapes tardives du cycle lytique
Les dernières étapes dans un cycle lytique englobent la réplication d’ADN viral, la production des composantes structurales (queue, capside) et la lyse de la cellule infectée. La restriction d’ADN viral avant sa réplication via un système de restriction-modification (RM) est un mécanisme très commun présent sur le génome de divers genres bactériens. Les systèmes RM peuvent être classifiés en quatre grands types (type I à IV), mais le mécanisme général est relativement similaire. Typiquement, ces systèmes englobent une composante « restrictive » qui est responsable du clivage du nouvel ADN entrant dans la cellule via la reconnaissance de certains sites spécifiques. À l’inverse, le génome bactérien est protégé par la méthylation des sites sensibles sur le génome par la composante « protectrice » du système RM. Ce système de protection étant quasi universel, il n’est pas surprenant de constater que beaucoup de phages ont évolué des mécanismes leur permettant de résister aux enzymes restrictives. Les stratégies telles que la mutation des sites reconnus par les nucléases (Kruger