RÉGULATION DES GÈNES DE L’HÔTE PAR LES
MICROARN DÉRIVÉS DE L’ÉLÉMENT TAR DU VIH-1
Mémoire présenté
à la Faculté des études supérieures et postdoctorales de l’Université Laval dans le cadre du programme de maîtrise en Biologie cellulaire et moléculaire
pour l’obtention du grade de Maître ès sciences (M.Sc.)
FACULTÉ DE MÉDECINE UNIVERSITÉ LAVAL
QUÉBEC
2013
I. Résumé
Les microARN (miARN) sont des acides ribonucléiques (ARN) endogènes, d’environ 19 à 24 nucléotides (nt), produits lors du clivage d’une structure d’ARN en forme de tige-boucle par la ribonucléase III (ARNase III) Dicer. Il a été rapporté que le virus de l’immunodéficience humaine de type 1 (VIH-1), en période de latence dans les lymphocytes T CD4+, produit un court transcrit d’ARN appelé « Trans-Activation Response
element » (TAR) et que celui-ci est sujet au clivage par Dicer, ce qui génère deux miARN fonctionnels, soit miR-TAR-5p et miR-TAR-3p. Il a donc été suggéré que les miARN dérivés de TAR pourraient jouer un rôle dans la latence du VIH-1. L’objectif, au cours de ma maîtrise, était de déterminer le rôle potentiel de ces miARN dans la régulation de l’expression des gènes de l’hôte en utilisant les cellules en culture J-lat et Jurkat exprimant miR-TAR-5p et miR-TAR-3p de manière stable. Suite à cela, une analyse protéomique grande échelle iTRAQ a été effectuée pour tenter de faire la lumière sur le rôle potentiel des miARN dérivés de l’ARN TAR du VIH-1.
En conclusion, il a été montré que la majorité des protéines d’intérêts sont réfractaires à une régulation génique par les miARN. Par contre, ceci ne supporte pas l’idée que ces miARN ne possèdent aucun rôle, d’où l’importance des résultats protéomiques qui ont montré que plusieurs protéines sont potentiellement régulées par les miARN viraux.
II. Avant-propos
J’aimerais remercier les membres de ma famille qui m’ont toujours supporté au cours de ma vie et dans les divers choix que j’ai pu effectuer. Je remercie tout particulièrement mon père Romulus qui m’a inculqué les précieuses valeurs que je possède aujourd’hui et qui font l’homme que je suis. Merci Mulus ! Je remercie aussi tous mes précieux amis qui m’ont toujours encouragé : Simon, Guillaume, Anthony, Marc-André, Charles, Emy, Bertrand et Édith.
Je remercie aussi la gang de karaté Shotokan Sainte-Foy et Gilles : vous êtes pour moi une deuxième famille. Peu importe l’endroit où je serai plus tard, vous serez toujours dans mon cœur.
Finalement je remercie toute l’équipe du laboratoire avec qui j’ai travaillé : Patrick, de m’avoir accueilli dans ton laboratoire; Patricia, Hélène, Aurélie, Geneviève et Marjorie, vous m’avez écouté, conseillé, consolé, encouragé et… botté le cul ! Je remercie aussi Dominique Ouellet pour ses précieux conseils et pour le matériel qu’elle a préparé au cours de son doctorat, ainsi que Lise-Andrée Gobeil et Jésus Jérôme Lacroix qui, par leurs expérimentations, ont permis l’élaboration de mon projet de maîtrise.
Vitam Impendere Vero
III. Table des matières
I. Résumé ... i
II. Avant-propos ...ii
III. Table des matières... iv
IV. Liste des figures et tableau ... v
V. Abréviations ...vii
Chapitre 1... 1
Introduction ... 1
1.1 La voie des microARN ... 1
1.1.1 Historique et découverte... 1
1.1.2 Définition et caractéristiques des miARN... 2
1.1.3 La voie des miARN ... 4
1.1.3.1 La transcription des gènes de miARN ... 5
1.1.3.2 Le complexe Microprocesseur... 5
1.1.3.2.1 La ribonucléase III Drosha ... 6
1.1.3.2.2 Le cofacteur DiGeorge syndrome Chromosomal Region 8 (DGCR8) ... 7
1.1.3.3 L’export du pré-miARN au cytoplasme par l’Exportin-5 ... 8
1.1.3.4 La ribonucléase III Dicer ... 9
1.1.3.5 Le cofacteur « TAR RNA-Binding Protein » (TRBP) ... 11
1.1.3.6 Le cofacteur « Fragile X Mental Retardation Protein » (FMRP) ... 12
1.1.3.7 Le complexe effecteur ... 13
1.1.3.7.1 Les protéines Argonautes (Ago) ... 13
1.1.3.7.2 Les complexes ribonucléoprotéiques contenant des miARN (miRNP) . 14 1.2 Les miARN et les virus ... 15
1.2.1 Aperçu général ... 15
1.2.1.1 Les virus à ADN et leurs miARN ... 16
1.2.2 Le virus de l’immunodéficience humaine de type 1 (VIH-1) ... 16
1.2.2.1 La biologie du VIH-1 ... 16
1.2.2.2 Le cycle réplicatif du VIH-1 ... 18
1.2.2.2.1 La primo-infection ... 18
1.2.2.2.2 La phase de latence clinique ... 18
1.2.2.2.3 La phase SIDA ... 19
1.2.3 Le VIH-1 et la voie des miARN ... 19
1.2.3.1 Changement du profil d’expression des miARN cellulaires relié à l’infection au VIH-1 ... 19
1.2.3.2 Le ciblage du VIH-1 par des siARN exogènes ... 21
1.2.3.3 Le ciblage du VIH-1 par la voie des microARN ... 22
1.2.3.5 Les miARN de TAR... 26
1.3.1 Hypothèse ... 27
1.3.2 Objectif de recherche ... 27
Chapitre 2... 29
Régulation des gènes de l’hôte par les miARN dérivés de l’élément TAR du VIH-1 ... 29
2.1 Matériels et méthodes ... 29
2.1.1 Amplification des gènes par PCR ... 29
2.1.2 Clonage moléculaire ... 29
2.1.3 Transformation des bactéries compétentes ... 31
2.1.4 Amplification de l’ADN plasmidique ... 31
2.1.5 Analyse de restriction et séquençage ... 32
2.1.6 Maintien en culture des cellules HEK 293 et Jurkat ... 32
2.1.7 Transfection des cellules HEK 293 par précipitation au phosphate de calcium ... 33
2.1.8 Essais des gènes rapporteurs ... 34
2.1.9 Lyse des cellules Jurkat et détection des protéines d’intérêt par immunobuvardage ... 34
2.1.10 Essai de protection aux ARNases («RNase protection assay»;RPA) ... 36
2.1.11 Analyses protéomiques par spectrométrie de masse ... 37
2.2 Résultats... 38
2.2.1 Essais de régulation génique par les miARN dérivés de l’élément TAR du VIH-1 ... 43
2.2.2 Induction à la Prostratine des cellules J-lat et immunobuvardage ... 48
2.2.3 Analyse des lignées de cellules Jurkat exprimant le shTAR de manière stable ... 50
2.2.3.1 Détection des miARN libérés dans les cellules Jurkat-shTAR ... 50
2.2.3.2 Analyses par immunobuvardage de type western ... 52
2.2.3.3 Analyses protéomiques iTRAQ des Jurkats stables ... 54
2.2.3.3.1 Résultats de l’analyse protéomique iTRAQ ... 54
2.2.3.3.2 Analyses bio-informatiques des nouvelles cibles potentielles ... 58
Chapitre 3... 61
Discussion générale ... 61
Chapitre 4... 75
Conclusion et perspectives ... 75
Bibliographie ... 78
IV. Liste des figures et tableau
Figure 1. Représentation schématique de la voie des miARN ... 5Figure 2. Structure des domaines de la ribonucléase de classe II Drosha ... 7 Figure 3. Structure des domaines de la ribonucléase de classe III Dicer... 10 Figure 4. Modèle du clivage catalytique des précurseurs de miARN par Dicer ... 10 Figure 5. Présentation schématique de l’emplacement des différents miARN dérivés du génome du VIH-1 ... 24 Figure 6. Séquence et conformation de l’ARN TAR du VIH-1 ... 26 Figure 7. Schéma de la voie de l’apoptose dans les cellules humaines ... 40 Figure 8. Sites de liaison potentiels pour les miARN miR-TAR-5p et miR-TAR-3p dans les ARNm potentiellement régulés, en relation avec l’analyse protéomique iTRAQ ... 43 Figure 9. Validation fonctionnelle des sites de liaison aux miARN de l’ARN TAR du VIH-1 dans les ARNm de facteurs pro-apoptotiques ... 44 Figure 10. Validation fonctionnelle des sites de liaison aux miARN de l’ARN TAR du VIH-1 présents dans le 3’UTR de l’ARNm de BBC3/Puma ... 46 Figure 11. Validation des sites de liaison potentiels (BS) aux miARN de TAR dans le 3’UTR de Procaspase-8 ... 47 Figure 13. Expression protéique des différentes cibles potentielles de miR-TAR-5p/3p dans les cellules exprimant l’ARN TAR et induites à la Prostratine ... 50 Figure 14. Détection des miARN libérés dans les Jurkat-shTAR ... 51 Figure 15. Expression protéique des différentes cibles potentielles de miR-TAR-5p/3p dans les cellules Jurkat exprimant le shTAR de manière stable ... 53 Figure 16. Résultats de l’analyse protéomique iTRAQ des cellules Jurkat-shTAR cl.1 ... 54 Figure 17. Résultats de l’analyse protéomique iTRAQ des cellules Jurkat-shTAR cl.3 ... 57 Figure 18. Résultats de l’analyse protéomique iTRAQ des cellules Jurkat-shTAR cl.4 ... 58 Figure 19. Sites de liaison des miARN miR-TAR-5p et miR-TAR-3p dans les ARNm étant potentiellement régulés, en relation à l’analyse protéomique iTRAQ... 60
Tableau 1. Séquence des différents oligonucléotides d’ADN utilisés pour le clonage moléculaire des cibles de miR-TAR-5p/3p ... 30
V. Abréviations
ADN Acide désoxyribonucléique
Ago « Argonaute »
ARN Acide ribonucléique
ARNm ARN messager
ARNdb ARN double brin
ARNsb ARN simple brin
ARNt ARN de transfert
A. thaliana Arabidopsis thaliana
C. elegans Caenorhabditis elegans
CHS « Chalcone synthase »
DGCR8 «DiGeorge syndrome critical region 8»
DGS «Di George syndrome»
DUF «Domain of unknown function»
DsRBD «Double-stranded RNA binding domain»
EBV «Epstein-Barr Virus»
HSV-1 «Herpes simplex virus 1»
HTLV-1 «Human T Lymphotropic Virus 1»
iARN Interférence à l’ARN
kDa Kilodalton
LTR «Long terminal repeat»
mg Milligramme
miARN MicroARN
miRNP «MicroRNA ribonucleoprotein complex»
ng Nanogramme
nt Nucléotides
PAZ PIWI-Argonaute-Zwille
Pré-miARN Pré-microARN
PC8 Pro-Caspase-8
PCR «Polymerase chain reaction»
PRR «Proline-rich region»
PTGS «Post-transcriptional gene silencing »
RAKE «RNA-primed Array-based Klenow Enzyme»
RISC «RNA-induced silencing complex»
RLUC «Renilla Luciferase»
RNase Ribonucléase
RPM Révolutions par minute
shRNA «Short hairpin RNA»
siRNA «Small interfering RNA»
TAR «Trans-activating response»
μg Microgramme
UTR «Untranslated region»
Chapitre 1.
Introduction
1.1 La voie des microARN
1.1.1 Historique et découverte
Ce fut en 1990 qu’un premier cas d’interférence à l’ARN a été découvert, et ce sans même le savoir encore. Les travaux de Napoli et al. [1] consistaient à étudier la biosynthèse des anthocyanes, un pigment naturel produit chez les feuilles du pétunia leur conférant une couleur pourpre. Les chercheurs ont démontré que la chalcone synthase (CHS) est l’enzyme limitante dans la formation des anthocyanes du maïs [1]. L’équipe de Napoli a décidé de surexprimer le gène CHS chez le pétunia afin de déterminer son importance dans la synthèse biochimique de cette fameuse couleur pourpre. Les chercheurs ont été stupéfaits de voir que l’expression du transgène ne donnait pas une couleur pourpre comme attendu, mais plutôt une couleur blanche caractéristique d’une absence d’anthocyane. Les analyses effectuées sur ces plantes ont montré qu’il y avait une réduction de 50 fois du niveau des ARN messagers (ARNm) du transgène. Ces résultats démontraient que l’introduction d’un transgène chez la plante engendre à la fois l’inhibition de l’expression du gène endogène et du transgène; les chercheurs ont nommé ce phénomène « co-suppression » [2, 3].
En 1991, l’équipe de Wightman et al. [4] ont découvert un élément de régulation dans la région 3’ non-traduite (3’UTR) de l’ARNm de lin-14 (un gène important dans le développement de C. elegans) et que l’élimination de celui-ci engendre une augmentation du niveau de la protéine lin-14p [4]. En fait, cette régulation est médiée par lin-4, un court
transcrit d’ARN ciblant le 3’UTR de l’ARNm lin-14 via sept sites de liaison différents, qui engendre une diminution de la traduction de cette protéine d’un facteur de 10 alors que le niveau de son ARNm reste toujours stable [5, 6].
L’équipe de Fire et al. (1998) ont ensuite déterminé qu’un ARN double brin est beaucoup plus efficace pour médier l’interférence à l’ARN que les brins sens et anti-sens pris individuellement ou en combinaison. Ils ont aussi démontré que le phénomène d’interférence à l’ARN est un processus s’effectuant de manière post-transcriptionnelle [7, 8]. L’année suivante, Hamilton et Baulcombe (1999) ont découvert que l’agent effecteur clé de la régulation post-transcriptionnelle (« posttranscriptional gene silencing », PTGS ) est un court transcrit d’ARN d’environ 25 nt et que celui-ci assure la spécificité de la régulation par un appariement ARN anti-sens : ARNm [9].
C’est en octobre 2001, lorsque trois équipes de recherche indépendantes ont publié leurs travaux sur les petits ARN régulateurs endogènes dans la revue Science, qu’il y eu un consensus pour nommer ces petits ARN interférant endogènes, d’une longueur de 19-24 nt, microARN (miARN) [10-12].
Dix ans plus tard, la recherche sur les miARN est toujours aussi actuelle, que ce soit sur les mécanismes par lesquels ils médient leurs actions, leurs rôles dans le développement, leurs liens avec les maladies humaines ou leurs diverses interactions avec les virus. C’est pourquoi j’ai consacré mes travaux de maîtrise à l’étude des interactions entre ceux-ci et les virus, plus particulièrement avec le Virus de l’Immunodéficience Humaine de type 1 (VIH-1).
Les miARN sont reconnus pour jouer un rôle important dans la régulation des gènes chez les plantes et les animaux. Tous les différents miARN identifiés à ce jour sont répertoriés dans miRBase (site web : http://www.mirbase.org). La plus récente version du site (miRBase version 19.0, août 2012) montre l’existence de plus de 2042 miARN chez l’humain, de 1281 miARN chez la souris, de 368 miARN chez C. elegans et de 338 miARN chez A. thaliania [13-15].
Les miARN sont de petits ARN non-codants, d’une longueur de 19 à 24 nt, qui sont reconnus pour jouer un rôle crucial dans la régulation d’une multitude de processus cellulaires, notamment l’apoptose, la différentiation neuronale, l’hématopoïèse, la prolifération cellulaire et la tumorigénèse [16].
Les miARN sont exprimés de différentes manières, soit constitutives, temporelles et spatiales. Par exemple, chez C. elegans, les miARN miR-13b-1 et miR-13b-2 sont identiques, mais sont exprimés dans le système nerveux et dans le groupe muscles/intestins respectivement. Tout au long du développement de C. elegans, les membres de la famille let-7 sont exprimés de manières différentes. Leur expression temporelle particulière joue d’ailleurs un rôle crucial dans chacun des stades du développement du nématode [16-18].
Une particularité intéressante des miARN est leurs différents niveaux d’expression au sein des cellules, qui peuvent varier de 1 000 molécules/cellule jusqu’à 50 000 molécules/cellule [19]. Il est à noter que les miARN peuvent réguler de manière synergique les ARNm, c’est-à-dire qu’ils peuvent lier un ARNm pour inhiber sa traduction. Un seul miARN peut réguler jusqu’à quelques centaines d’ARNm différents [20, 21]. Un ARNm donné peut être régulé par plusieurs microARN différents, ce qui permet une régulation fine et complexe de l’expression génique. L’importance accordée aux miARN provient de leur
abondance, leur diversité et leur fort pouvoir de régulation sur l’ensemble du transcriptome au sein des cellules du corps humain.
1.1.3 La voie des miARN
La formation des miARN matures (19-24 nt) nécessite plusieurs étapes successives. Elles consistent (i) en la transcription des gènes de miARN, principalement par l’ARN polymerase II, (ii) au clivage de ces ARN structurés en tige-boucle par l’ARNase III Drosha, (iii) à l’export vers le cytoplasme par l’Exportin-5, (iv) au clivage par l’ARNase III Dicer, et (v) à l’insertion des miARN matures dans des complexes effecteurs contenant des protéines de la famille Ago qui permettent la régulation des ARNm (Figure 1). Ces principales étapes sont présentées ici, dans le détail, pour bien comprendre cette voie de régulation génique.
1.1.3.1 La transcription des gènes de miARN
Plusieurs modèles ont été proposés pour décrire la transcription des gènes de miARN. Premièrement, il a été soupçonné que l’ARN polymérase III est l’enzyme qui génère les miARN primaires, car elle médie également la synthèse d’autres petits ARN non-codants, tels les ARN de transfert (ou transfert RNA; tRNA) et les petits ARN nucléaires U6 (ou small nuclear RNA; snARN). D’ailleurs, certains miARN se retrouvent à l’intérieur de gènes contenant des régions riches en séquence Alu et transcrits par l’ARN polymérase III [22]. Cependant, il a été démontré plus tard que la majorité des pri-miARN sont engendrés par l’ARN polymérase II. C’est pourquoi les pri-miARN portent la même signature que les ARNm de la cellule, soit une extrémité 5’ 7-méthyl-guanosine et une
Figure 1. Représentation schématique de la voie des miARN. Les gènes de miARN sont
transcrits par l’ARN polymérase II pour former des miARN primaires (pri-miARN). Situé au noyau, le complexe microprocesseur composé de Drosha et DGCR8 clive ces pri-miARN précurseurs de pri-miARN (pré-pri-miARN). Ces pré-pri-miARN sont ensuite transportés au cytoplasme par l’Exportin-5 pour être clivés par un complexe formé de Dicer et TRBP et ainsi former un duplex miARN:miARN*. Après un processus de sélection du brin effecteur, le miARN mature est incorporé dans un complexe contenant des ribonucléoprotéines (miRNP) afin de réprimer l’ARNm. Figure tirée intégralement de Ouellet et al.[23]
queue poly (A) en 3’ [24, 25]. Certains Pri-miARN sont transcrits à l’intérieur d’un regroupement de miARN (« cluster ») qui, lors du clivage par Drosha, engendre plusieurs miARN matures différents. Les miARN peuvent aussi se retrouver à l’intérieur d’introns, où ils sont appelés « mirtrons ». Ceux-ci sont excisés par le spliceosome, tout comme les introns des ARNm codants, pour engendrer directement un pré-miARN qui ne nécessite pas de clivage préalable par la ribonucléase III Drosha [26, 27].
Constitué de l’ARNase III Drosha et de son cofacteur DGCR8, le Microprocesseur assure le clivage initial du pri-miARN et engendre le précurseur de miARN (pré-miARN), le substrat reconnu par Dicer pour former un miARN mature.
1.1.3.2.1 La ribonucléase III Drosha
L’ARNase Drosha est une endoribonucléase III de classe II qui a initialement été clonée et caractérisée par l’équipe de Wu et al. [28]. Ceux-ci ont étudié cette protéine pour son importance dans la biosynthèse des précurseurs des ARN ribosomaux (pré-ARNr). Ils ont établi que Drosha est une protéine se situant principalement dans la fraction nucléaire des cellules et qui possède un poids moléculaire de 160 kDa [28]. Drosha est constituée de plusieurs domaines : deux domaines RNase III (RIIIDa et RIIIDb), un domaine de liaison de l’ARNdb (dsRBD) (à l’extrémité C-terminale), un domaine riche en proline (PRR) et un domaine riche en arginine/sérine (RS) (à l’extrémité N-terminale) [29] (Figure 2). Drosha initie la première étape de la maturation des pri-miARN en miARN fonctionnels en convertissant les pri-miARN en précurseurs de miARN (pré-miARN). Lee et al. [30] ont observé qu’en diminuant l’expression de Drosha par iARN, il y a une accumulation de pri-miARN au noyau et une diminution des niveaux de pré-pri-miARN et pri-miARN matures. Des études ont démontré que Drosha est incorporée dans trois différents complexes protéiques, soit un complexe de très haut poids moléculaire possédant une activité RNase non-spécifique, un complexe de ~440 kDa possédant une faible activité endoribonucléasique et un complexe de ~650 kDa, dont DGCR8 fait partie, qui possède la plus grande activité catalytique dans la formation de pré-miARN [31, 32]. Il est à noter que seules Drosha et
DGCR8 se retrouvent dans le complexe de 650 kDa et qu’elles seules sont nécessaires au bon fonctionnement du Microprocesseur.
Du côté structural, en raison de la structure en forme d’hélice de l’ARN double-brin, le domaine « RNase III Domain a » (RIIIDa) clive le pri-miARN sur le brin 3’, tandis que le domaine « RNase III Domain b » (RIIIDb) clive à l’extrémité 5’, ce qui engendre une structure en tige-boucle ayant un bout protubérant de 2 nt en 3’, ce qui est caractéristique des coupures médiées par les RNases III) [32].
Figure 2. Structure des domaines de la ribonucléase de classe II Drosha. Drosha
possède deux domaines d’interactions protéine-protéine en position N-terminale, soit un domaine riche en Proline ainsi qu’un domaine Sérine-Arginine (RS). En C-terminale, il y a deux domaines catalytiques RNase III (RIIIa & RIIIb) ainsi qu’un domaine de liaison à l’ARNdb (dsRBD). Tirée d’un article de revue publié par Carmel et Hannon [29].
1.1.3.2.2 Le cofacteur DiGeorge syndrome Chromosomal Region 8
(DGCR8)
DGCR8 a tout d’abord été étudiée pour son rôle dans la maladie qui lui a valu son nom, le syndrome de DiGeorge. Dans cette maladie, la plupart des patients montrent une délétion dans le chromosome 22q11 pouvant aller jusqu’à 1.5 Mégabase (Mb) et qui est appelée DGCR (DiGeorge syndrome Chromosomal Region). Les chercheurs ont identifié les gènes présents dans ce segment délété-- qui engendre les phénotypes documentés chez les patients atteints de la maladie --, notamment le gène codant pour la protéine DGCR8.
Cette protéine possède deux domaines de liaisons à l’ARN double-brin (« double strand RNA Binding Domain »; dsRBD) ainsi qu’un domaine WW, qui médie les interactions protéine-protéine en se liant à un domaine riche en proline (« Proline Rich
Region »; PRR), comme celui que possède Drosha (Figure 1) [33]. DGCR8 a plus tard été étudiée pour son importance dans la biogenèse des miARN : elle agit comme cofacteur de Drosha pour le clivage des pri-miARN [32, 34]. Celle-ci reconnaît la jonction d’ARN simple-brin et double-brin du transcrit primaire et sert d’ancrage moléculaire permettant à Drosha d’exercer son activité catalytique à environ deux tours d’hélice alpha de la jonction ARNsb-ARNdb [35].
1.1.3.3 L’export du pré-miARN au cytoplasme par l’Exportin-5
Une fois le travail de Drosha ayant formé le pré-miARN, celui-ci est dirigé vers le cytoplasme, où il est pris en charge par la ARNase III Dicer. Pour ce faire, la cellule a recourt au transport actif utilisant le gradient de Ran-GTP qui est présent entre le noyau et le cytoplasme. L’Exportin-5, qui est un membre de la famille des transporteurs nucléaires Karyopherine β, a été démontré comme étant l’unique transporteur des pré-miARN vers le cytoplasme [36, 37]. Exportin-5 est nécessaire à la régulation initiée par les pré-miARN endogènes et par les ARN en tige-boucle (ou short hairpin RNAs; shRNAs) exogènes, mais non pour les siARN, qui, eux, ne nécessitent pas de transport actif vers le cytoplasme pour médier leurs effets [37].
L’export médié par l’Exportin-5 est considéré comme étant l’étape limitante dans la biosynthèse des miARN, car son niveau d’expression influence directement la quantité de miARN matures générés au cytoplasme. En effet, lorsque les cellules HeLa sont traitées avec des siARN ciblant Exportin-5, il y a une baisse générale d’environ 40-60% des miARN matures dans la cellule ainsi qu’une accumulation des pré-miARN au noyau [38]. Inversement, l’efficacité des miARN est grandement augmentée lorsqu’il y a une
surexpression de ce transporteur nucléaire au sein des cellules [39]. Il est à noter que la voie de l’Exportin-5 est saturable, c’est-à-dire qu’en transfectant un plasmide surexprimant des shARN, ceux-ci entreront en compétition avec l’export des pré-miARN endogènes, diminuant du coup la formation des miARN matures endogènes [39].
1.1.3.4 La ribonucléase III Dicer
La forme humaine de la ribonucléase III Dicer a été découverte grâce à des expériences de double hybride chez la levure visant à identifier les principales protéines interagissant avec l’enzyme 5-lipoxygénase (5LO). Un ADNc ayant beaucoup d’homologie à l’ARNm codant pour la protéine K12H4.8 de C. elegans, qui possède des motifs ARNase III et un domaine de liaison à l’ARNdb, a été identifié [40] et nommé Dicer en raison de son homologie avec la protéine du même nom identifiée chez la drosophile [41]. La protéine Dicer contient 1912 acides aminés et a un poids moléculaire de 218 kDa. Celle-ci a la capacité de lier l’ADNdb et de le cliver pour générer de petits fragments d’ARNdb [41-43].
Dicer possède plusieurs domaines fonctionnels, soit un domaine ATPase/hélicase avec un motif DExH dans la portion N-terminale de la protéine, un « Domain of Unknown Function 283 » (DUF283), un domaine PIWI-Ago-Zwille (PAZ) et deux domaines ARNase III couplés à un domaine « dsRBD » dans sa portion C-terminale [29, 42, 43] (Figure 3).
Figure 3. Structure des domaines de la ribonucléase de classe III Dicer. Dicer possède
un domaine hélicase et un domaine de fonction inconnue (Domain of Unknown Function 283 ; DUF283) en position N-Terminale ainsi qu’un domaine central PIWI-Argonaute-Zwille (PAZ). En C-terminale, Dicer possède deux domaines RNase III, dont un domaine non fonctionnel indiqué avec un X, ainsi qu’un domaine de liaison à l’ARNdb. Tirée intégralement d’un article de revue publié par Carmel et Hannon [29].
Il a été démontré que le domaine PAZ de Dicer reconnaît les pré-miARN via son interaction avec les 2 nt protubérants en 3’ sur l’ARNdb générés par Drosha. Une dimérisation intramoléculaire de Dicer regroupe ensuite ses deux domaines ARNase III à proximité l’un de l’autre afin de cliver l’ARN deux tours d’hélice α plus loin. Ce clivage donne naissance à un ARNdb, d’environ 21 à 23 paires de bases, montrant une extrémité phosphate en 5’ ainsi qu’une extrémité protubérante de 2 nt hydroxylée en 3’ [44, 45] (Figure 4).
Figure 4. Modèle du clivage catalytique des précurseurs de miARN par Dicer. Le
domaine PAZ de la ribonucléase III Dicer reconnaît l’extrémité 5’-phosphate ainsi que l’extrémité 3’-hydroxyle exhibant 2 nt protubérants. L’architecture entre les domaines catalytiques et le domaine PAZ serait la cause de la formation d’ARNdb de 22 nucléotides. Tirée intégralement d’un article de revue publié par Carmel et Hannon [29].
Il a été montré par des études génétiques que Dicer est cruciale au développement chez les mammifères, ce qui a été démontré à l’aide d’expériences effectuées sur des souris déficiences en Dicer qui décédaient au stade embryonnaire [46, 47]. Des cellules souches embryonnaires de souris dont le gène dcr-1 avait été supprimé demeuraient toujours viables. Après analyses, ces cellules ont montré une forte déficience au sein de la biogénèse des miARN, ce qui engendre une altération majeure de la différenciation cellulaire. Celle-ci peut être rétablie par une ré-expression de Dicer, sous forme recombinante, dans ces cellules [46]. Il est aussi rapporté que plusieurs cas de cancer proviennent d’une dérégulation du niveau d’expression de Dicer : une diminution de son expression dans les carcinomes du poumon n’étant pas à petites cellules [48], ainsi qu’une augmentation de son expression dans les adénocarcinomes de prostate et dans les lésions précurseurs d’adénocarcinomes de poumons [49, 50]. Ceci montre l’importance d’une fine régulation de l’expression de Dicer pour conserver l’homéostasie cellulaire.
1.1.3.5 Le cofacteur « TAR RNA-Binding Protein » (TRBP)
Le cofacteur TRBP a été caractérisé en 1991 par l’équipe de Gatignol et al.[51]. Ils ont montré que celui-ci agit de manière synergique avec la protéine virale Tat en se liant à l’élément TAR du VIH-1 afin de transactiver le promoteur « Long terminal repeat » (LTR) et d’accroître la transcription du génome viral. TRBP possède trois domaines de liaisons à l’ARNdb (dsRDB) et est présent sous deux isoformes dans les cellules, soit : TRBP1 et TRBP2, qui a 21 acides aminés de plus que l’isoforme 1. Présentement, TRBP a été montrée comme jouant un rôle cellulaire via au moins trois différents mécanismes : par l’augmentation de la transcription des ARN contenant l’ARN viral TAR, et ce,
indépendamment de l’inhibition de PKR [52]; par la formation d’un hétérocomplexe avec la protéine PKR empêchant son autophosphorylation et ainsi la phosphorylation du facteur de transcription « eukaryotic Initiation Factor 2 alpha » (eIF2α) par celle-ci [53, 54]; et par son interaction avec Dicer comme cofacteur dans la voie de l’iARN [55, 56]. De plus, une étude protéomique récente a montré que TRBP interagit avec 160 protéines intracellulaires jouant des rôles dans la synthèse protéique, la transcription de l’ADN ainsi que la prolifération cellulaire [57].
TRBP a été identifiée, par spectrométrie de masse, par son interaction avec la ribonucléase III Dicer via son troisième domaine (dsRBD) en C-terminal [55]. Il a été montré que le complexe TRBP•Dicer interagit avec la protéine Argonaute 2 (Ago2) et que TRBP est un cofacteur requis dans le processus de transfert d’un siARN de Ago2 vers Dicer pour son clivage protéolytique [55].
1.1.3.6 Le cofacteur « Fragile X Mental Retardation Protein » (FMRP)
Une perte d’expression du gène FMR1 (« Fragile Mental Retardation 1 »), qui engendre une perte d’expression de la protéine FMRP (« Fragile X Mental Retardation Protein »), est l’agent étiologique menant au syndrome du X fragile, cause la plus fréquente de retards mentaux non liés à un héritage génétique [58-60]. FMRP est une protéine associée aux polyribosomes possédant un domaine de liaison à l’ARN et est reconnue comme étant un régulateur négatif de la traduction des ARNm [58, 61]. Dernièrement, notre équipe a découvert un rôle de FMRP au sein de la voie de l’iARN. Il a été montré que FMRP est capable de prendre en charge des miARN issus du clivage de Dicer, ce qui facilite la formation du complexe miARN:ARNm cible [62], et que la forme phosphoryléede FRMP se dissocie de Dicer, ce qui suggère un mécanisme moléculaire de l’assemblage des miARN sur leurs cibles [63].
1.1.3.7 Le complexe effecteur
1.1.3.7.1 Les protéines Argonautes (Ago)
Durant les dernières années, les protéines Argonaute ont été largement étudiées chez différents organismes, dont S. pombe, C. elegans, D. melanogaster et certains mammifères [64]. Chez l’humain, il existe huit membres de la famille AGO séparés dans deux sous-familles, quatre PIWI et quatre « eukaryotic Initiation Factor 2C /Argonaute» (eIFC2/AGO). Tous les membres de la famille AGO possèdent une même structure en domaines, soit un motif PAZ dans le centre de la protéine et un motif PIWI à l’extrémité C-terminale. Les gènes Ago sont situés sur différents chromosomes et sont exprimés différemment au sein de l’organisme : les quatre protéines Ago (Ago1-2-3-4) sont exprimées de manière ubiquitaire dans les différents tissus adultes, alors que les protéines Piwi se concentrent pour la plupart au niveau des testicules [65]. Il a été démontré que les quatre protéines Ago sont en mesure de lier les miARN et les siARN, mais que seul Ago2 possède une activité catalytique. Des analyses cristallographiques chez Pyrococcus furiosus ont démontré que le domaine Piwi d’Ago2 se replie pour former une structure analogue au domaine catalytique de l’ARNase H, lui conférant ainsi son activité catalytique de clivage [66, 67]. Le domaine PAZ, quant à lui, jouerait le même rôle que pour Dicer, c’est-à-dire qu’il reconnaîtrait les 2 nt protubérants à l’extrémité 3’ des pré-miARN pour les incorporer au complexe effecteur. Des études ont montré qu’Ago2 seule est nécessaire à la répression
d’un ARNm et que les miARN ne servent que de guide pour apporter le complexe répresseur vers l’ARNm cible [68].
1.1.3.7.2 Les complexes ribonucléoprotéiques contenant des miARN
(miRNP)
Une fois que la ribonucléase III Dicer a effectué son activité catalytique de clivage et formé un duplex de miARN, il se forme un complexe effecteur de nature ribonucléoprotéique autour de ce duplex : les complexes ribonucléoprotéiques contenant des miARN (miRNP). Les rôles fonctionnels de ce complexe sont de recevoir le duplex miARN : miARN*; de déterminer le brin effecteur (miARN mature) ainsi que le brin passager (miARN*); de reconnaître l’ARNm à réguler avec le brin effecteur et d’inhiber sa traduction ou le diriger vers les « processing-bodies » (p-bodies) (foci cytoplasmiques où se trouvent l’activité ribonucléasique de Ago2 ainsi que des ARNm non traduits [69]), où il sera stocké et, éventuellement, dégradé [16]. La ségrégation des deux brins de miARN est de nature thermodynamique, c’est-à-dire que le brin dont l’énergie d’appariement avec son complément est la plus faible en 5’ est sauvegardé, tandis que l’autre brin est dégradé [70]. Chez les mammifères, la majorité des complexes miRNP engendre une répression traductionnelle de l’ARNm cible. Du moins, c’est ce que l’on croyait initialement : des études de biopuces à ADN ont montré que certains miARN peuvent réduire considérablement le niveau de leurs ARNm cibles [71]. C’est le cas, notamment, de miR-196, qui se lie avec sa cible HOXB8 de manière parfaitement complémentaire (à l’exception d’un G :U) et engendre sa dégradation [72].
La régulation d’un ARNm par un miARN nécessite un appariement parfait dans la partie 5’ du miARN avec sa cible. Cette région est appelée « seed » et est constituée des nucléotides 2-7 [73].
Une question persiste toujours : quelle est la composition protéique exacte de ce complexe effecteur ? Un complexe de ~150 kDa à 500 kDa possédant une activité nucléase catalytique a été isolé à partir de cellules de Drosophile et est appelé RISC [74]. Quant au complexe miRNP, il partage plusieurs caractéristiques avec le complexe RISC [75], notamment la présence d’un petit ARN non-codant guide (miARN), la ribonucléase III Dicer et Ago2.
1.2 Les miARN et les virus
1.2.1 Aperçu général
La première interaction ayant été reconnue entre les miARN et les virus a été découverte chez les plantes. Celles-ci utilisent la voie de l’interférence à l’ARN (iARN) afin de se protéger contre les virus en ciblant leur génome et en engendrant leur dégradation [9]. Ce système de défense serait aux plantes ce que le système immunitaire est aux humains, c’est-à-dire que les plantes auraient évolué en produisant ces petits ARN afin de protéger leur génome de tout remaniement possible de la part des virus. Par contre, l’évolution étant ce qu’elle est, certains virus ont développé des stratégies afin de contrer l’iARN, et ce, par l’entremise de protéines virales suppressives de l’iARN [76-78].
Il a été montré que certains génomes viraux sont la cible de miARN endogènes et que, vice-versa, certains virus produisent des miARN aptes à cibler différents gènes de l’hôte. De plus, certains virus produisent des miARN qui ciblent leur propre génome, ce qui
régule leur réplication au sein de la cellule hôte. Différents exemples seront présentés dans les prochaines sections.
1.2.1.1 Les virus à ADN et leurs miARN
Le virus Epstein-Barr (EBV) est un virus Herpes oncogène infectant l’humain et présent dans ~15% des lymphomes à grandes cellules B [79]. Il a été rapporté initialement que le virus EBV exprimait cinq miARN à partir de deux régions différentes de son génome [80]. Par contre, les cellules utilisées lors de ces études étaient infectées avec la souche B95-8, qui contient une délétion de 12 kb dans la région BamH1. Celle-ci a été rapportée, plus tard, comme étant une région contenant la majorité des microARN d’EBV [79]. Depuis, plus de 140 miARN ont été découverts chez les virus Herpes, dont 25 chez le virus EBV [81, 82].
Quant au virus herpès humain de type 8 « Kaposi's sarcoma-associated herpesvirus » (KHSV), il semble capable de produire 12 pré-miARN pouvant former un total de 17 miARN matures différents [83]. Il a été montré que les miARN produits chez HSV1-2 et MDV1-2, membres de la famille des herpèsvirus, sont les seuls ARN produits au cours de la latence et servent au maintien de celle-ci dans les cellules infectées [84].
1.2.2 Le virus de l’immunodéficience humaine de type 1 (VIH-1)
1.2.2.1 La biologie du VIH-1
En 1981, il a été rapporté que cinq individus montrant des syndromes de pneumonie combinés à une déplétion de lymphocyte T se sont présentés dans des hôpitaux de Los-Angeles afin d’y recevoir des soins appropriés. Plusieurs groupes de recherche se sont
intéressés à ces patients, car il avait été découvert qu’un rétrovirus, le « Human T Lymphotrophic Virus 1 » (HTLV-1), était en mesure d’infecter les cellules T et qu’un rétrovirus présent chez les chats (FeLV) induisait, quant à lui, une immunodéficience similaire à celle observée chez ces patients. Il était soupçonné qu’un rétrovirus de la famille HTLV, encore inconnu de la communauté scientifique, pouvait être impliqué [85].
C’est en 1983 que l’agent étiologique du SIDA a été isolé, à partir d’un ganglion lymphatique, par le Dr. Luc Montagnier et qu’il a été analysé par microscopie électronique par la Dre. Françoise Barré-Sinoussi, qui se virent décerner le prix Nobel de médecine pour leur découverte du virus de l’immunodéficience humaine de type 1 (VIH-1) [85, 86]. Il a été montré que les cellules permissives à l’infection in vivo sont principalement les cellules exprimant à leur surface la molécule CD4, tels les lymphocytes T CD4+, les monocytes et les macrophages. Il existe aussi différents réservoirs mineurs, notamment les cellules CD8+, les cellules B, les cellules « Natural Killer » (NK) et les cellules dendritiques (pour une revue détaillée, voir [87]).
Pour ce qui est de l’épidémiologie du VIH-1, en 2009, on estimait que le nombre de personnes nouvellement infectées dans l’année se situait à 2,6 millions, pour un total de 33,3 millions de personnes vivant avec le VIH sur la planète. Le rapport 2010 d’ONUSIDA dresse un bilan plus positif que par les années antérieures :
« La croissance globale de l’épidémie mondiale de SIDA semble s’être stabilisée. Le nombre annuel de nouvelles infections du VIH recule régulièrement depuis la fin des années 1990 et l’on note une diminution du nombre de décès liés au SIDA grâce à un élargissement et à une intensification significatifs de l’accès au traitement antirétroviral au cours des dernières années. Bien que le nombre de nouvelles infections ait diminué, leurs niveaux généraux demeurent élevés et le nombre de personnes vivant avec le VIH augmente dans le monde du fait de la réduction significative de la mortalité. »
(Tiré du Rapport ONUSIDA 2010)
http://www.unaids.org/globalreport/Global_report_fr.htm)
1.2.2.2 Le cycle réplicatif du VIH-1
1.2.2.2.1 La primo-infection
Le mode d’infection du VIH-1 le plus fréquent est par transmission sexuelle via les muqueuses génitales. Le virus se lie aux cellules de Langerhans (cellules dendritiques), présentes au niveau de l’épithélium urogénital, via la lectine DC-SIGN qui lie de manière spécifique la protéine virale gp120. Cette interaction est utilisée comme vecteur par le virus afin de le rapprocher des cellules CD4+ présentes au niveau des ganglions lymphatiques et de les infecter [88].
Une fois le virus pénétré à l’intérieur des cellules et intégré à leur génome, une réplication massive de celui-ci produit une multitude de virions pouvant, à leur tour, infecter d’autres cellules. Au pic de la production, on dénombre un niveau d’ARN viral en circulation sanguine pouvant aller jusqu’à 5 millions de copies par ml [89]. Cette grande production de virus a pour conséquence, directe ou indirecte, de diminuer les lymphocytes T CD4+ dans l’organisme [89]. Après deux à quatresemaines de production virale intense, il y a un retour spontané du niveau de production virale dû à la réponse immunitaire primaire au VIH-1. Pendant tous les stages de l’infection, le virus procède à un taux de réplication minimal et il est possible de trouver le matériel génétique du virus dans les cellules T CD4+ en tout temps.
Intervient ensuite la phase de latence clinique, ou asymptomatique, qui se traduit par une charge virale plasmatique à son minimum et correspondant, après qu’il ait infecté une multitude de lymphocytes CD4+, à l’entrée du virus dans son cycle de latence. Au cours de cette phase, qui peut durer plusieurs années, il y a une diminution progressive du nombre de lymphocytes T CD4+ circulant qui est inversement proportionnelle à la charge virale plasmatique. Il existe donc un équilibre dynamique entre la mort cellulaire des cellules infectées, le taux de renouvellement de ces cellules et la charge virale [89, 90].
1.2.2.2.3 La phase SIDA
Le stade clinique de la phase SIDA est atteint lorsque la concentration des lymphocytes CD4+ circulants est inférieure à 200 cellules/µl. Cette perte de cellules immunitaires est due à l’intensification de la réplication virale dans l’organisme, ce qui engendre la mort des cellules infectées et favorise l’apparition de maladies opportunistes s’avérant, éventuellement, mortelles chez les individus infectés.
1.2.3 Le VIH-1 et la voie des miARN
1.2.3.1 Changement du profil d’expression des miARN cellulaires relié à
l’infection au VIH-1
Toute modification de l’environnement d’une cellule engendre une cascade signalétique menant à un remaniement génomique et à un changement du profil d’expression génique. C’est le cas du VIH-1 qui, lors de l’infection au sein des cellules hôtes, les reprogramme à son avantage. Étudiant cette question à l’aide de leur méthode « RNA-primed Array-based Klenow Enzyme » (RAKE), l’équipe de Kuan-Teh Jeang a
observé une baisse importante des miARN dans les cellules HeLa transfectées avec le plasmide infectieux pNL4-3 [91], ce qui supporte l’idée que le VIH-1 affecte considérablement le profil d’expression génique des cellules qu’il infecte. L’équipe a obtenu des résultats semblables en faisant appel à des sujets humains infectés à divers degrés, c’est-à-dire une diminution majeure des miARN, et ce, peu importe le stade de l’infection virale [92].
Des analyses plus détaillées ont permis d’identifier une baisse des niveaux d’expression du regroupement, ou « cluster », de miARN miR-17/92 chez les cellules infectées par le VIH-1. Ce regroupement de miARN est connu pour réguler l’expression de « P300/CBP-associated factor » (PCAF), une protéine cellulaire agissant comme cofacteur dans la transactivation du VIH-1 par la protéine virale tat. Donc, lorsque le virus infecte une cellule, il y a une diminution du regroupement de miARN miR-17/92 qui engendre, du même coup, une augmentation du facteur PCAF favorisant la réplication virale [93].
Lors de l’infection par le VIH-1, une quantité considérable de protéines virales Tat est relâchée par les cellules infectées et circule librement dans le sang. Celle-ci est capable de passer la barrière hémato-encéphalique pour ainsi atteindre les neurones et diminuer l’expression du miARN miR-128a. Ce miARN est responsable de la régulation de « Synaptosomal-associated protein 25 » (SNAP25), une protéine nécessaire aux échanges vésiculaires entre les neurones. Cette dérégulation des échanges entre les neurones serait l’une des causes de la dégénération neuronale observée chez les personnes infectées par le VIH-1 [94]. D’ailleurs, les patients atteints du VIH peuvent développer, au cours du stade final d’infection, divers troubles neurologiques pouvant mener à la démence. En effet, des neurotoxines libérées par le virus et une réponse immunitaire soutenue des macrophages et
des cellules microgliales au cours de l’infection peuvent entraîner une encéphalite menant à la démence. Il a été montré chez ces patients qu’une multitude de miARN subissent un changement d’expression [95], dont miR-21 [96] et miR-219 [95, 97] qui, paradoxalement, sont exprimés à la hausse en comparaison à des sujets sains.
Le miARN miR-21 est induit dans les neurones par une stimulation continue du récepteur « N-methyl-D-aspartic acid » (NMDA), un procédé excitotoxique actif dans l’infection par le VIH-1 ainsi que d’autres maladies neurodégénératives. L’équipe de Fox et al. [96] ont montré que l’ARNm codant pour la protéine « Myocyte-specific enhancer factor 2C » (MEF2C) est ciblé par miR-21, ce qui engendre une baisse d’expression dans les neurones. MEF2C est un facteur de transcription important dans le système nerveux central et sa perte serait l’une des causes des troubles neurodégénératifs, tels « HIV-associated dementia » (HAD) et « HIV-HIV-associated neurocognitive disorders » (HAND) [96]. Quant à miR-219, il semble médier les dysfonctions neurocomportementales de la schizophrénie, dans lesquelles les récepteurs NMDA jouent un rôle de médiation [97], et il est surexprimé chez les patients HIV/ « Major depressive disorder » (MDD) [95].
Pour finir, il a été montré qu’une fois les cellules dendritiques ayant été en contact avec des agents pathogènes tels que le VIH-1, il y a une augmentation importante du niveau de miR-155. Celui-ci diminue l’expression du facteur de transcription de la lectine « Dendritic Cells-specific intercellular adhesion molecule-3 grabbing non-integrin » (DC-SIGN), une protéine essentielle à l’adhésion de surface des pathogènes sur les cellules dendritiques, en régulant négativement l’expression du facteur de transcription PU.1 [98].
Se basant sur l’activité antivirale des plantes grâce aux siARN produits dans leurs cellules, plusieurs laboratoires se sont lancés dans la course thérapeutique contre le VIH à l’aide de siARN spécifiques. Ainsi, plusieurs siARN ont été produits afin de contrer l’infection au VIH-1 [99]. Les premières cibles ont été les gènes essentiels au VIH-1, soit : la protéine structurale gag [100], les facteurs d’infection vif et nef [101] ainsi que la protéine responsable de la régulation post-transcriptionnelle, rev [102]. En plus d’avoir ciblé d’autres gènes du VIH-1 (nef, tat, gag, vif, env) [99, 103, 104], certains groupes ont ciblé des protéines cellulaires importantes au cycle du 1. D’abord i) à l’entrée du VIH-1 dans les cellules via les co-récepteurs CCR5 et CXCR4 [VIH-105, VIH-106]; ensuite ii) à l’étape de pré-intégration au niveau du complexe Arp2/3, qui est responsable de la polymérisation de l’actine impliquée dans le transport du VIH-1 vers le noyau [107], au niveau de « poly (ADP-ribose) polymerase-1 » (PARP-1), enzyme nucléaire requis pour l’intégration du VIH-1[108]; puis iii) au niveau transcriptionnel, en ciblant les protéines essentielles à la transcription du LTR par l’ARN polymérase II, soit cycline T1, CDK-9 et SPT5 [109, 110] Cependant, il reste encore beaucoup de chemin à parcourir afin d’augmenter l’efficacité de tels traitements, les siARN n’étant que transitoires chez les cellules humaines et leurs effets n’étant que temporaires. De plus, le virus est capable d’échapper à la voie d’interférence à l’ARN par divers moyens (pour références, voir [111] ), rendant ainsi la thérapie encore plus compliquée.
1.2.3.3 Le ciblage du VIH-1 par la voie des microARN
La première évidence du ciblage du VIH-1 par la voie des microARN vient d’une étude effectuée en 2005 par l’équipe de Hariharan et al. [112]. Des analyses
bio-informatiques ont identifié 5 microARN pouvant cibler une région conservée dans le génome du VIH-1. Elles ont confirmé la présence de ces miARN au sein des lymphocytes T par biopuces [112]. Deux de ces miARN, soit miR-29a et miR-29, ont été confirmés comme pouvant moduler le VIH-1 en régulant l’expression du gène Nef [113, 114]. La protéine virale Nef est essentielle à la progression de l’infection virale du VIH-1, car celle-ci est exprimée précocement dans le cycle viral et déclenche le relâchement des virions qui engendrent l’infection persistante du VIH-1 [115].
En 2007, l’équipe de Huang et al. [116] ont montré que des microARN cellulaires pouvaient inhiber la réplication virale latente au sein des lymphocytes CD4+ primaires. Un regroupement de cinq miARN cellulaires incluant miR-150, miR-223, miR-28, miR-125b et miR-382, a été montré comme pouvant cibler l’extrémité 3’ des ARNm viraux. Les chercheurs ont découvert que ces miARN, en comparaison aux cellules en réplication virale dites activées, sont enrichis dans les cellules CD4+ latentes, ce qui suggère un rôle majeur
dans le maintien de la latence du VIH-1 [116, 117]. Les transcrits viraux ciblés par la voie des microARN sont amenés vers les p-bodies pour y être éventuellement dégradés. Ceci a été démontré en immunoprécipitant les complexes de répression et en examinant les différents ARNm qui y étaient associés [116]. D’ailleurs, l’inhibition de la formation des p-bodies, réalisée à l’aide de siARN régulant à la baisse l’expression des protéines qui les composent, est associée à une recrudescence de la réplication virale [114, 118].
Il a été montré auparavant que les monocytes sont des cellules aptes à l’infection du VIH-1, mais que celui-ci ne peut s’y répliquer qu’une fois ces cellules différenciées en macrophages par l’augmentation de la cycline T1 [119]. L’équipe de P. Rice et al. [119] ont montré que le microARN miR-198 régule à la baisse l’expression de la cycline T1 dans les
monocytes. Or, une fois que les monocytes se différencient en macrophages, il y a une diminution de moitié de l’expression de miR-198, ce qui engendre une forte augmentation de la cycline T1; c’est à partir de ce moment que le virus commence à produire des virions. La réplication virale du VIH-1 est fortement dépendante de l’expression de la cycline T1, celle-ci étant un cofacteur essentiel de la protéine trans-activatrice tat. Leur étude suggère donc un rôle de miR-198 dans le contrôle de la réplication virale dans les monocytes par l’entremise de la répression de la cycline T1 [119].
1.2.3.4 Les miARN dérivés du VIH-1
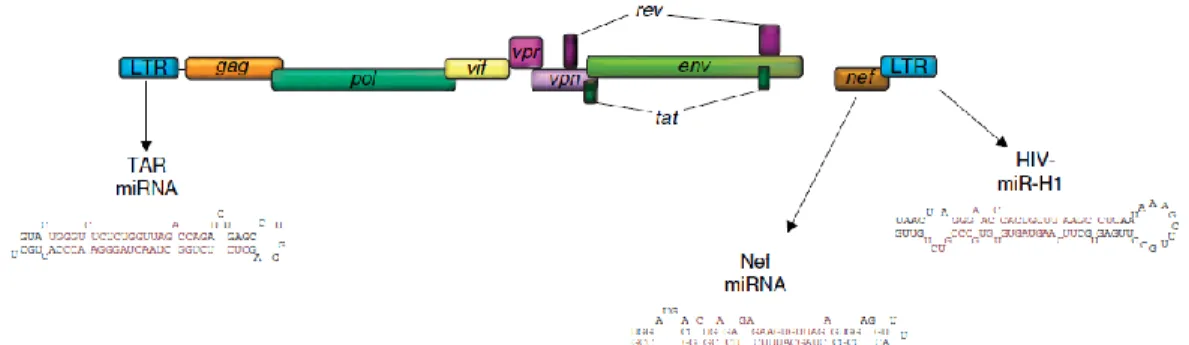
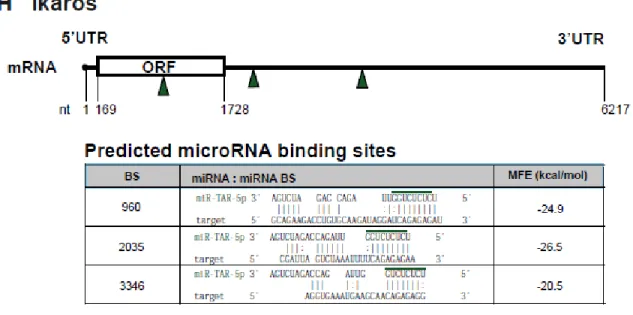
Tout a commencé en 2004, lorsque l’équipe de Bennasser et al. [120] ont démontré que le génome du VIH-1 pouvait être une source de miARN. Ceux-ci ont identifié cinq structures en tige-boucle au sein du génome du VIH-1 pouvant engendrer une dizaine de miARN [120]. Jusqu’à maintenant, des miARN de trois de ces structures ont été confirmés (Figure 5).
Figure 5. Présentation schématique de l’emplacement des différents miARN dérivés du génome du VIH-1. Le génome du VIH-1 contient trois structures arborant des
tiges-boucles pouvant être clivées par Dicer pour engendrer des miARN, i.e. les miARN de TAR (miR-TAR-5p et miR-TAR-3p), le miARN de NEF (miR-N367) ainsi que le miARN HIV-miR-H1 présent dans la région 3’-« long terminal repeat » (LTR). Tirée intégralement de Narayanan et al. [121].
En 2002, l’équipe d’Omoto et al. [122] ont montré qu’une portion d’environ 500 nt du gène de Nef interfère avec la transcription du VIH-1. Deux ans plus tard, ceux-ci ont découvert que ce phénomène était dû au miARN miR-N367, découvert chez des cellules T MT-4 infectées de manière permanente par la souche HIV IIIB. De plus, ils ont montré que miR-N367 diminue la transcription virale à des niveaux différents : soit au niveau post-transcriptionnel, en régulant à la baisse l’expression de la protéine accessoire Nef [123]; et au niveau transcriptionnel, en interférant avec le promoteur via le NRE de la région U3 du 5’LTR, ce qui empêche l’ARN polymérase II de transcrire cette région [124].
Le miARN viral HIV1-miR-H1 provient d’une structure en tige-boucle de 81 nt située en amont des deux sites « Nuclear Factor κB » (NF-κB) dans le 3’LTR du VIH-1. Celui-ci engendre le clivage du 12ième exon du gène « Apoptosis Antagonizing Transcription Factor » (AATF), causant ainsi la dégradation de son produit. Une perte d’expression de ce facteur est accompagnée d’une réduction de la viabilité des cellules, ainsi que d’une diminution de certaines protéines, telles Dicer, « B-cell lymphoma 2 » (Bcl-2), c-myc et « Prostate apoptosis response-4 » (Par-4) [125]. Ce miARN aurait donc un effet antagoniste à celui exercé par les miARN anti-apoptotiques de TAR [121]. Il est à noter que le HIV-miR-H1 pourrait diminuer l’expression du miARN cellulaire miR-149, qui cible le gène viral Vpr, favorisant ainsi l’expression de ce dernier. De plus, une étude récente a démontré que, parmi un groupe de sujets étudié [126], ce miARN montre une grande diversité en termes de séquence. Des mutations ou délétions dans son pré-miARN pourraient donc engendrer différents miARN pouvant être responsables, du moins en partie, de la diversité de la pathogénicité du VIH-1 dans la population [126].
1.2.3.5 Les miARN de TAR
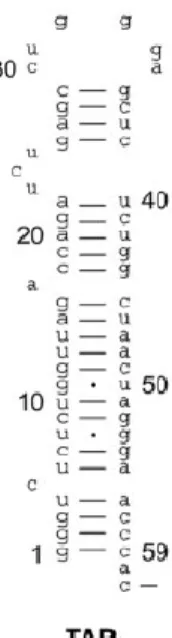
Des analyses de délétions de la région LTR (Long Terminal Repeat) du VIH-1 ont permis d’identifier la région d’ARN structuré TAR comme étant la séquence régulatrice responsable de l’activité de la protéine virale tat. Cet élément structuré est situé en aval du site d’initiation de la transcription (nucléotides +1 à +59) et est présent en 5’ de tous les transcrits du VIH-1 (Figure 6). En l’absence de tat, il y a un avortement de la transcription par l’ARN polymérase II, engendrant la formation de courts transcrits viraux, tandis qu’en présence de tat, il y a un recrutement de divers facteurs d’élongation, telles la kinase « Cyclin-Dependent Kinase 9 » (CDK-9) et la cycline T1, permettant la transcription de long-transcrits viraux essentiels pour la réplication du VIH-1 [127].
Figure 6. Séquence et conformation de l’ARN TAR du VIH-1. Le court transcrit de
TAR, position +1 à +59, se replie sur lui-même pour former une structure en tige-boucle qui est sujette au clivage de la ribonucléase III Dicer. Tirée intégralement de Ouellet et al.[128]
Plus récemment, notre équipe a démontré que l’ARN en tige-boucle TAR, dont la structure est très similaire aux précurseurs de miARN, pouvait être reconnu par la ribonucléase III Dicer et clivé en miARN fonctionnels [128, 129]. Il a été démontré que les miARN de TAR peuvent jouer un rôle dans le remodelage de la chromatine au niveau du LTR du VIH-1 en recrutant une histone déacétylase HDAC-1, ce qui suggère un rôle de ces miARN dans la régulation de la transcription du VIH-1 [129]. De plus, il a été découvert que ces miARN régulent l’expression de certaines protéines de la voie de l’apoptose, telles « Excision repair cross-complementing rodent repair deficiency, complementation group 1 » (ERCC1) et « Radiation-inducible immediate-early gene IEX-1 » (IER3), afin de protéger les cellules infectées de la mort cellulaire programmée [130].
1.3 Hypothèse et objectif de recherche
1.3.1 Hypothèse
Les résultats présentés dans ce mémoire ont été obtenus dans le cadre d’un projet de recherche déjà en cours au laboratoire. Sachant que le VIH-1 possède une région d’ARN structuré appelée TAR, qui est présente à l’extrémité 5’ de tous les transcrits viraux, et que celle-ci a été montrée comme précurseur de la ribonucléase III Dicer dans la formation de miARN viraux, l’hypothèse de recherche de mes travaux de maîtrise était la suivante :
L’ARN TAR du VIH-1 libère des miARN qui peuvent réguler une multitude d’ARNm de la cellule hôte via la reconnaissance de sites de liaison situés dans la région 3’ non-codante de ces messagers.
Lors de la dernière décennie, il a été montré que plusieurs virus produisent des miARN (voir section 1.2). Cependant, il reste encore beaucoup à faire avant de pouvoir apprécier leur juste contribution au cours de l’infection du VIH-1. L’objectif de mes travaux de maîtrise était donc d’identifier les ARNm des cellules hôtes pouvant être
ciblés par les miARN dérivés de l’élément TAR du VIH-1.
Afin d’y parvenir, j’ai effectué i) des essais de régulation génique utilisant des gènes rapporteurs luciférases, ii) des immunobuvardages de type Western sur des cellules J-lat ou transfectées, ainsi qu’ iii) une analyse protéomique à grande échelle de cellules Jurkat exprimant l’élément TAR du VIH-1.
Chapitre 2.
Régulation des gènes de l’hôte par les miARN dérivés de
l’élément TAR du VIH-1
2.1 Matériels et méthodes
2.1.1 Amplification des gènes par PCR
Les réactions PCR ont été préparées en mélangeant 150 ng d’ADNc, amplifiés à partir d’ARN total isolé de cellules Jurkat, 50 pmol de chaque oligonucléotides, 2.5 mM de chaque nucléotide, 4 µl de tampon concentré 5X, 2.0 µl de la polymérase Phusion (New England Biolabs) et de l’eau milliQ à 50 µl. Les réactions ont été soumises aux cycles « Polymerase Chain Reaction » (PCR) suivants : un cycle de dénaturation initial de 30 s à 98oC; 35 cycles d’amplification de 10 s à 98oC, 30 s à 550C et de 30 s à 72oC; un cycle final de 10 min à 72oC. Les oligonucléotides utilisés pour l’amplification de la région 3’UTR des ARNm Procaspase-8 (PC8) et de Puma sont présentés dans le Tableau 1.
2.1.2 Clonage moléculaire
Les vecteurs d’expression psiSTRIKE et psiCHECK ont été obtenus chez Promega. La séquence de l’ARN TAR a été clonée dans le vecteur psiSTRIKE selon le protocole de la compagnie.
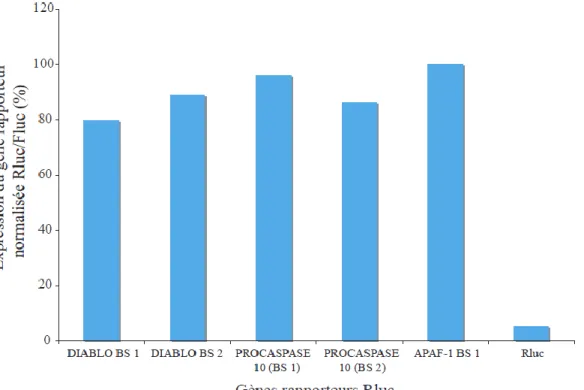
Les régions 3’UTR de Procaspase-8 et de Puma, amplifiées par PCR, ont été clonées dans les sites de restrictions Xho1 et Not1 (New England Biolabs) en aval du gène rapporteur Rluc dans le vecteur psiCHECK. De plus, des gènes rapporteurs ont été créés
dans le vecteur psiCHECK, où des sites de liaisons (binding sites; BS) aux miARN issus de l’ARN de TAR ont été insérés, en 3 copies contiguës (3xBS), en aval du gène rapporteur Rluc.
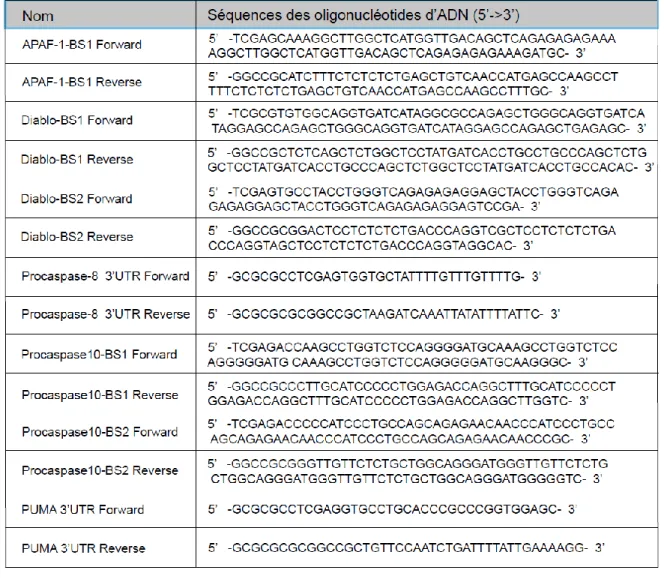
Tableau 1. Séquence des différents oligonucléotides d’ADN utilisés pour le clonage moléculaire des cibles de miR-TAR-5p/3p. Les oligonucléotides pour PUMA et
Procaspase-8 ont été synthétisés à la plate-forme de séquençage du CRCHUL par Ronald Maheux. Les oligonucléotides de Diablo et Procaspase-10 ont été synthétisés par Invitrogen et APAF-1 par IDT (Integrated DNA technologies). APAF-1, « Apoptotic Peptidase Activating Factor 1 »; BS, « Binding Site » (site de liaison) ; PUMA, « p53 Upregulated Modulator of Apoptosis »; UTR, « UnTranslated Region » (région non-traduite).
Les réactions de ligation ont été préparées en mélangeant 100 ng d’ADN du vecteur psiCHECK, digéré par les enzymes de restriction Xho1/Not1, avec un ratio molaire de 1:3
du produit PCR ou avec des oligonucléotides d’ADN appariés. Le mélange a été incubé pendant 5 min à 42oC, avant l’ajout d’un µl du tampon de réaction 10X (500 mM Tris-HCl, 100 mM MgCl2, 100 mM DTT, 10 mM ATP, pH 7,5) et d’un µl de l’enzyme « T4 DNA
ligase » (Roche). La réaction de ligation s’est déroulée pendant 3 h à TP.
2.1.3 Transformation des bactéries compétentes
Une fois la réaction de ligation complétée, 50 µl de bactéries E. coli DH5α (F-(80dlacZ M15) (lacZYA-argF) U169hsdR17(r-m+) recA1 enA1 relA1 deoR) compétentes ont été incubées avec 10 µl de la réaction de ligation pendant 20 min à 4oC, pendant 38 s à 42oC, puis pendant 2 min à 4oC. Un (1) ml de milieu Lysogeny broth (LB) a ensuite été ajouté. Ce mélange a été incubé pendant 60 min à 37oC sous une légère agitation de 250 révolutions par minute (RPM). Les bactéries ont ensuite été centrifugées pendant 3 min à 594 g, étalées sur un pétri contenant un milieu d’agar avec 50 µg/ml d’ampicilline et incubées 16 h à 37oC.
2.1.4 Amplification de l’ADN plasmidique
Les colonies ayant passé sous la sélection de l’ampicilline ont été piquées, inoculées dans 4 ml de milieu LB contenant 50 µg/ml d’ampicilline, et incubées à 37oC pendant 15 h sous agitation à 250 RPM. Au terme de cette incubation, les bactéries ont été recueillies par centrifugation afin d’en extraire l’ADN plasmidique à l’aide du « QIAGEN plasmid miniprep kit » (Qiagen®). Ce protocole se traduit en trois étapes, soit : i) la préparation du
lysat de bactéries dans un tampon alcalin; ii) l’adsorption de l’ADN sur la membrane Qiagen® de silice; iii) le lavage et l’élution de l’ADN plasmidique. La concentration
d’ADN purifié a été mesurée à l’aide d’un spectrophotomètre en prenant la mesure de l’absorbance à 260 nm.
2.1.5 Analyse de restriction et séquençage
Afin de vérifier l’insertion des inserts dans les vecteurs d’expression, des analyses de restrictions ont été réalisées. L’ADN des constructions a ainsi été soumis à une digestion par des enzymes de restriction, selon le même protocole qu’à la section 2.1.1, puis analysé par une électrophorèse sur gel d’agarose afin de déterminer le poids moléculaire des fragments de digestion. Par la suite, l’ADN des plasmides montrant le patron de digestion attendu a été séquencé par la plate-forme protéomique du Centre de génomique de Québec afin de vérifier l’intégrité des séquences d’ADN d’intérêt et leur insertion dans les vecteurs d’expression, assurant ainsi que ces vecteurs ne comportaient aucune mutation pouvant compromettre les expériences futures.
2.1.6 Maintien en culture des cellules HEK 293 et Jurkat
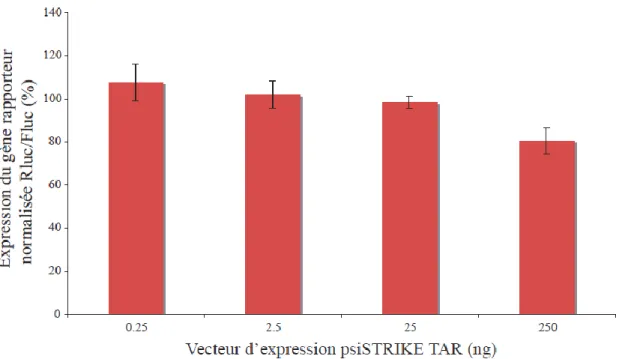
Les cellules HEK 293 ont été cultivées dans un milieu DMEM supplémenté avec 10% de sérum bovin fœtal (FBS) décomplémenté, 10 mM de pyruvate de sodium, 1mM de glutamine, 100 µg/ml de pénicilline et 100 µg/ml de streptomycine. Quant aux cellules Jurkat exprimant l’ARN TAR du VIH-1 du plasmide psiSTRIKE, elles ont été cultivées dans un milieu DMEM supplémenté avec 10% de FBS et 10 mM de pyruvate de sodium, puis maintenues sous sélection à l’antibiotique G418 (400 µg/ml).
2.1.7 Transfection des cellules HEK 293 par précipitation au phosphate
de calcium
Les transfections des cellules HEK 293 ont été effectuées selon un protocole élaboré au laboratoire. Un jour avant la transfection, les cellules ont été déposées dans des plaques à 24 puits pour atteindre une confluence de 80% ou moins au moment de la transfection. Tel que présenté à la section 2.1.6, 0.5 ml de milieu DMEM supplémenté a été ajouté pour éviter que les cellules sèchent et, 1 heure avant la transfection, le milieu a été changé pour du frais. En même temps, dans des tubes eppendorf stériles, ont été mélangés 2,5 µl de CaCl2 (2,5 M), 100 ng d’ADN de vecteur psiCHECK contenant notre gène rapporteur
d’intérêt, ainsi qu’une quantité croissante (0,25 ng 250 ng) d’ADN de vecteur psiSTRIKE contenant des séquences en tige-boucle Neg, TAR ou Rluc; le volume étant ajusté à 25 µl avec de l’eau MilliQ. Toujours en même temps, dans d’autres tubes eppendorf, a été mélangé 25 µl du tampon HEPES 2X (HEPES 50 mM, NaCl 140 mM, Na2HPO4 1,5 mM, pH 7,05 ajusté avec du NaOH 2 N et complété avec de l’eau MilliQ).
Après une incubation d’une (1) min, la solution d’ADN-CaCl2 a été ajoutée à la solution
d’HEPES goutte à goutte tout en insufflant, avec l’embout d’une pipette de verre, des bulles d’air pour favoriser la formation de précipités. Après une attente d’une (1) min, 50 µl du mélange a été ajouté doucement sur le dessus des cellules et la plaque a été agitée avec précaution afin de mélanger le milieu et l’ADN précipité (T0 de transfection). Les cellules
ont ensuite été placées dans un incubateur, dont l’atmosphère était humidifiée à 37oC avec 5% de dioxyde de carbone (CO2), pour une durée de 6 h. Ensuite, le milieu avec l’ADN
l’incubateur pour une durée totale de transfection de 24 h ou de 48 h (T24 ou T48 de
transfection).
2.1.8 Essais des gènes rapporteurs
Les cellules ont ensuite été récoltées aux fins d’analyses et lavées avec 1 ml de PBS (Phosphate Buffered Saline) stérile (137 mM NaCl, 2,7 mM KCl, 10 mM Na2HPO4 • 2
H2O, 1,76 mM KH2PO4, pH ajusté à 7,4 avec HCl 1 N et complété avec de l’eau MilliQ).
Puis, les cellules ont été lysées dans 100 µl de tampon de lyse 1X (Passive lysis buffer) (Promega) et la plaque a été agitée pendant 15 min à la température de la pièce (TP) afin de bien compléter leur lyse . Le lysat des cellules a ensuite été homogénéisé à la pipette et 15 µl ont été prélevés et déposés dans une plaque à 96 puits pour les essais enzymatiques luciférase. Ces derniers se sont déroulés comme suit : la plaque est entrée dans l’appareil « Microliter plate luminometer » (Dynex) où 2 produits ont été ajoutés à un intervalle fixe de 10 sec dans les puits, soit : 100 µl de « Luciferase Assay Reagent II » (LARII) (Promega) et 100 µl de solution « Stop and Glow » (Promega). Après ce processus, les résultats ont été affichés, ainsi que les différents ratios luciférase/luciférine. Les résultats ont ensuite été analysés statistiquement par un test de t de Student's, et les différences ayant une probabilité inférieure à 5% (i.e. une valeur de p<0,05) ont été considérées comme significatives.