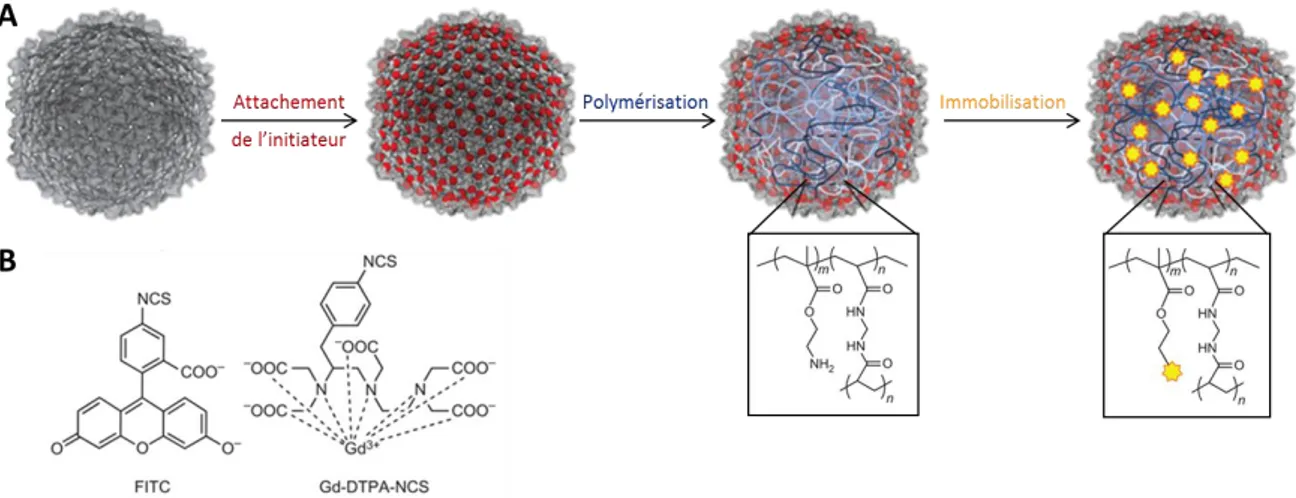
HAL Id: tel-01500099
https://tel.archives-ouvertes.fr/tel-01500099
Submitted on 2 Apr 2017HAL is a multi-disciplinary open access
archive for the deposit and dissemination of sci-entific research documents, whether they are pub-lished or not. The documents may come from teaching and research institutions in France or abroad, or from public or private research centers.
L’archive ouverte pluridisciplinaire HAL, est destinée au dépôt et à la diffusion de documents scientifiques de niveau recherche, publiés ou non, émanant des établissements d’enseignement et de recherche français ou étrangers, des laboratoires publics ou privés.
Maturation de la capside du bactériophage T5 : étude
structurale et fonctionnelle de la protéine de décoration
pb10
Emeline Vernhes
To cite this version:
Emeline Vernhes. Maturation de la capside du bactériophage T5 : étude structurale et fonctionnelle de la protéine de décoration pb10. Biochimie [q-bio.BM]. Université Paris-Saclay, 2016. Français. �NNT : 2016SACLS465�. �tel-01500099�
NNT : 2016SACLS465
T
HESE DE DOCTORAT
DE
L’U
NIVERSITE
P
ARIS
-S
ACLAY
PREPAREE A
L’U
NIVERSITE
P
ARIS
-S
UD
E
COLED
OCTORALE N°569
Innovation thérapeutique : du fondamental à l’appliqué
Pôle Ingénierie des protéines et cibles thérapeutiques
Spécialité de doctorat : Biochimie et biologie structurale
Par
Mme Emeline VERNHES
Maturation de la capside du bactériophage T5 :
étude structurale et fonctionnelle de la protéine de décoration pb10
Thèse présentée et soutenue à Orsay, le 30/11/2016 :
Composition du Jury :
Prof. Philippe MINARD Professeur, I2BC (CEA/CNRS/U. Paris-Sud) Président
Dr. Mark VAN RAAIJ Dir. de recherche, Centro Nacional de Biotecnología Rapporteur Dr. Jean-Pierre SIMORRE Dir. de recherche, IBS (CEA/CNRS/U. Grenoble-Alpes) Rapporteur Dr. Paulo TAVARES Dir. de recherche, I2BC (CEA/CNRS/U. Paris-Sud) Examinateur
Dr. David BIKARD Chargé de recherche, Institut Pasteur Examinateur
Dr. Bertrand RAYNAL Ingénieur de recherche, Institut Pasteur Examinateur
Dr. Pascale BOULANGER Dir. de recherche, I2BC (CEA/CNRS/U. Paris-Sud) Directrice de thèse Dr. Sophie ZINN-JUSTIN Dir. de recherche, I2BC (CEA/CNRS/U. Paris-Sud) Invitée
I Ces travaux ont été initiés au sein de l’équipe Virologie Bactérienne dirigée par Pascale Boulanger
dans l’Institut de Biochimie et Biophysique Moléculaire et Cellulaire (IBBMC) au bâtiment 430 du campus scientifique d’Orsay. L’IBBMC (UMR 8619 CNRS / Université Paris-Sud), dirigé par Michel Desmadril, a fait l’objet d’un regroupement avec d’autres unités de recherche et a fermé le 31
décembre 2014.
Au 1er janvier 2015, l’équipe de Pascale Boulanger est devenue l’équipe Bactériophage T5 dans le
Département de Virologie de l’Institut de Biologie Intégrative de la Cellule (I2BC). Le Département de Virologie est dirigé par Yves Gaudin et l’I2BC (UMR 9198 CEA / CNRS / Université Paris-Sud) par
II
Remerciements
Je souhaite tout d’abord remercier ma directrice de thèse, Pascale Boulanger, pour avoir eu confiance en moi dès le début pour réaliser ce projet, pour m’avoir soutenue et encouragée tout au long de ces trois années, pour m’avoir permis de participer à des congrès internationaux et de rencontrer de nombreux scientifiques du domaine des phages, pour avoir toujours tenu compte de mes souhaits et de mes contraintes personnelles.
Je souhaite également remercier Sophie Zinn-Justin qui m’a encadrée pour les expériences de RMN, qui a pris le temps de m’expliquer et me réexpliquer ce que je ne comprenais pas. Je remercie également Bernard Gilquin qui a fait les modélisations de structure des domaines de pb10 isolés, et
Philippe Cuniasse pour son travail sur la protéine pb10 entière.
Un grand merci à Jean-Pierre Simorre et Mark van Raaij qui ont accepté d’être rapporteurs pour ce manuscrit, ainsi qu’à David Bikard, Philippe Minard, Bertrand Raynal et Paulo Tavares qui ont accepté de juger mon travail en tant qu’examinateurs.
Mes remerciements vont ensuite à Madalena Renouard qui a fait les premières expériences sur pb10, qui m’a encadrée au début de ma thèse pour la plupart des expériences et m’a appris de nombreuses choses, qui m’a souvent été d’une grande aide lorsque j’avais des problèmes de manipulations, et qui a toujours fait preuve d’une grande patience et d’une grande générosité à mon égard.
Je remercie toutes les personnes qui m’ont encadrée ou aidée pour certaines techniques : - Dominique Durand et Cécile Mérigoux pour les expériences de SAXS et de DLS
- Patrick England et Sylviane Hoos de l’Institut Pasteur pour la SPR
- Magali Aumont-Nicaise pour le DSC, et pour sa constante jovialité
- Christophe Velours pour les premières expériences de CD et pour l’ultracentrifugation
analytique
- Luc Bousset pour les expériences de CD qui ont suivi
- Eric Jacquet et Naima Nhiri de l’ICSN pour les expériences de thermo-fluorescence
- Franck Wien pour les expériences de CD au synchrotron Soleil
- Jéril Degrouard et Claire Boulogne pour la microscopie électronique
- Inès Gallay pour les tentatives de cristallisation de pb10
Je remercie à nouveau Dominique Durand pour les nombreuses discussions informelles sur mes résultats et les idées qu’elle a apportées pour faire progresser mon travail.
III Merci à Vladimir Ksenzenko et Anatoly Glukhov de l’Institut de Recherche des Protéines à Pushchino (Russie) pour les nombreux phages, mutants ou non, qu’ils ont fourni et pour leurs expériences sur la stabilité des phages en présence et absence de pb10.
Je remercie Alexis Huet de l’Université de Pittsburgh (Etats-Unis) pour les reconstructions de cryo-EM sur différentes formes de la capside de T5 (avec ou sans ADN, avec ou sans pb10) et pour les discussions sur l’interaction de pb10 avec la capside de T5.
Je remercie à nouveau Paulo Tavares pour les nombreuses discussions scientifiques qui ont permis de faire avancer mon projet. Merci également aux autres membres de l’équipe SPP1 : Karima
Djacem, avec qui j’ai apprécié passer du temps lors des formations et des congrès scientifiques, Isabelle Auzat et Sandrine Brasilès qui ont en partie encadré les étudiants de M2 pour leurs TP sur la
mutagénèse aléatoire du gène de pb10, et qui m’ont également beaucoup appris.
Je remercie Karim Benihoud et Linda Larbi-Cherif de l’Institut Gustave Roussy qui se sont impliqués dans la valorisation des capsides de T5 pour une utilisation médicale et qui ont pris en charge l’étude des réponses immunitaires induites par les capsides fonctionnalisées.
Mes remerciements vont également à Jeannine Drouin-Whabi, qui a partagé son bureau avec moi pendant un an et demi, qui a fait des préparations de phages pour moi, et avec qui j’ai passé de très bons moments.
Je remercie Thibaut Besnier qui a optimisé les conditions de production de la protéine p144 au cours de son stage de L2.
Je remercie à nouveau Pascale Boulanger, Madalena Renouard, Dominique Durand, Cécile
Mérigoux, ainsi que Léo Zangelmi, Catherine Berrier, Alexandre Pozza, Marie-Ange Richard, Patrice Vachette, Ombeline Rossier et Luis Ramirez pour les nombreux repas et goûters que nous avons pris
ensemble ainsi que pour tous les moments que nous avons partagés dans la convivialité.
Je remercie aussi les doc-postdocs pour les discussions enrichissantes et pour les bons moments que j’ai passés avec eux.
Enfin je remercie toutes les personnes de l’ancien IBBMC ainsi que les gens du département de
IV
Table des matières
Remerciements ... II
Table des matières ... IV
Abréviations ... VIII
Partie I : Introduction ... 1
I.1. Les Bactériophages ... 3
I.1.1. Histoire : découverte des bactériophages ... 3
I.1.2. Utilisation des bactériophages en médecine : la phagothérapie ... 3
I.1.3. Les bactériophages et la biologie moléculaire ... 4
I.1.4. Morphologies et classification des bactériophages ... 5
I.2. Structure du bactériophage T5 ... 9
I.2.1. Structure de la tête de T5 ... 10
I.2.1.1. Les capsides icosaédriques ... 10
I.2.1.2. La capside de T5 ... 12
I.2.1.3. L’ADN de T5 ... 13
I.2.2. Structure de la queue de T5 ... 15
I.3. Le cycle infectieux du bactériophage T5 ... 16
I.3.1. L’infection ... 16
I.3.1.1. L’adsorption ... 16
I.3.1.2. Le transfert d’ADN dans la cellule hôte ... 16
I.3.1.2.1. Passage à travers la paroi bactérienne ... 16
I.3.1.2.2. Transfert de l’ADN en deux étapes ... 17
I.3.2. La production de nouveaux virions ... 17
I.3.2.1. La réplication de l’ADN phagique ... 17
I.3.2.2. L’assemblage de la tête ... 18
I.3.2.2.1. Assemblage de la procapside ... 18
I.3.2.2.2. Encapsidation de l’ADN et expansion ... 18
I.3.2.3. L’assemblage de la queue... 20
I.3.3. La lyse cellulaire ... 21
I.4. Les protéines auxiliaires de capside ... 22
I.4.1. Les protéines-ciment ... 22
I.4.2. Les protéines de décoration ... 24
I.4.2.1. Structures connues des protéines de décoration ... 24
I.4.2.2. Fonctions des protéines de décoration ... 26
I.4.2.2.1. Cas de la double fonction de Psu : stabilisation de la capside et suppression de polarité 26 I.4.2.2.2. Adhésion réversible à la surface des bactéries grâce aux domaines Ig-like ... 27
V
I.5. Utilisation de capsides modifiées : « capsid engineering » ... 29
I.5.1. Fonctionnalisation interne et externe de la capside du phage P22 ... 30
I.5.1.1. Internalisation de composés d’intérêt ... 31
I.5.1.1.1. Internalisation de protéines par fusion à la protéine d’échafaudage ... 31
I.5.1.1.2. Internalisation de composés chimiques ... 32
I.5.1.2. Fonctionnalisation de la surface externe de la capside de P22 ... 33
I.5.2. Exemple de fonctionnalisation multiple de la capside de T4 ... 34
I.5.3. Immobilisation d’antigènes sur des capsides de phage ... 35
I.6. Sujet de thèse : étude de la protéine de décoration pb10 et de la protéine de fermeture p144 du bactériophage T5 ... 39
I.6.1. Capsides vides expansées pour des études in vitro ... 39
I.6.2. Travaux préliminaires sur la protéine de décoration pb10 ... 41
I.6.2.1. pb10 est une protéine monomérique à deux domaines ... 41
I.6.2.2. pb10 se fixe à la capside par son domaine N-terminal ... 42
I.6.3. La protéine de fermeture p144 ... 43
I.6.4. Objectifs de thèse ... 45
Partie II : Résultats ... 47
II.1. Structure de pb10 ... 49
II.1.1. Tentatives de cristallisation de pb10 ... 49
II.1.2. Résolution de la structure de pb10 en solution par résonance magnétique nucléaire (RMN) ... 50
II.1.2.1. Structures des domaines N- et C-terminaux isolés... 52
II.1.2.2. Structure de la protéine pb10 entière ... 57
II.1.3. Comparaison avec les autres protéines de décoration des phages de la famille de T5 61 II.1.4. Homologies avec des protéines de structure connue ... 63
II.2. Interaction pb10 – capside ... 65
II.2.1. La protéine de décoration pb10 se fixe sur les capsides de T5 avec une très forte affinité ... 66
II.2.1.1. Expériences de retard sur gel (EMSA) ... 66
II.2.1.2. Détermination de l’affinité par résonance plasmonique de surface (SPR) ... 68
II.2.1.2.1. Préparation de la surface et du matériel ... 68
II.2.1.2.2. Les cycles de SPR ... 69
II.2.1.2.3. Régénération de la surface ... 71
II.2.1.2.4. Analyse qualitative des courbes d’association et dissociation ... 71
II.2.1.2.5. Analyse cinétique ... 76
II.2.1.3. Etude de l’effet coopératif de la décoration des capsides de T5 ... 79
II.2.2. Exploration de l’interface d’interaction entre pb10 et la capside de T5 ... 81
II.2.2.1. Interaction pb10-capside en présence de fortes concentrations en NaCl ... 85
II.2.2.2. Affinité des mutants pour la capside de T5 ... 86
II.2.2.3. Vérification du repliement correct des mutants ... 90
VI
II.2.2.5. Bilan des mutations ... 96
II.3. Fonction de pb10 ... 99
II.3.1. Rôle dans l’adsorption sur l’hôte ... 99
II.3.2. Stabilisation de la capside ... 101
II.3.2.1. Stabilisation des capsides vides : expériences de DSC ... 101
II.3.2.2. Influence de pb10 sur la résistance des phages T5 à la chaleur : mesure de la perte d'ADN par fluorescence. ... 108
II.3.2.3. Mécanisme de sortie de l’ADN des phages chauffés ... 112
II.4. Applications : « capsid engineering » ... 117
II.4.1. La fusion n’affecte pas le repliement ... 118
II.4.2. La fusion n’affecte pas l’affinité pour la capside ... 120
II.5. Production et purification de la protéine de fermeture p144 ... 123
II.5.1. Clonages avec HisTag et site de protéolyse ... 123
II.5.2. Optimisation des conditions de production ... 124
II.5.3. Optimisation des conditions de purification ... 126
Conclusions ... 129
Perspectives ... 135
Partie III : Matériels et Méthodes ... 141
III.1. Bactéries et phages utilisés ... 143
III.1.1. Souches bactériennes d’Escherichia coli utilisées ... 143
III.1.2. Phages T5 utilisés ... 143
III.1.3. Mutations ambre et souches suppressives ... 143
III.2. Tampons ... 144
III.3. Clonages et mutagenèses ... 144
III.3.1. Clonage conventionnel avec enzymes de restriction : p144 ... 144
III.3.2. Clonage par PCR (« FastCloning ») : protéines de fusion avec pb10 ... 144
III.3.3. Mutagenèse dirigée ... 144
III.3.4. Mutagenèse aléatoire ... 145
III.4. Surproduction de protéines ... 145
III.4.1. Culture en milieu riche ... 145
III.4.2. Culture en milieu minimum ... 145
III.5. Purification de protéines ... 145
III.5.1. Cassage des bactéries ... 145
III.5.2. Chromatographie d’affinité sur colonne de métal immobilisé (IMAC) ... 146
III.5.3. Chromatographie d’échange d’ion ... 146
VII
III.6. Production et purification de phages et de particules phagiques ... 147
III.6.1. Production de phages : infection à faible multiplicité d’infection ... 147
III.6.2. Production de particules phagiques : infection à forte MOI ... 147
III.6.3. Purification des phages et particules phagiques ... 147
III.6.3.1. Purification des phages et capsides pleines d’ADN sur gradient de CsCl ... 147
III.6.3.2. Purification des procapsides vides ... 148
III.6.3.3. Expansion des capsides vides ... 148
III.7. Décoration de capsides in vitro ... 149
III.8. Diffusion des rayons X aux petits angles (SAXS) ... 149
III.9. Résonance Magnétique Nucléaire (RMN) ... 150
III.10. Calorimétrie Différentielle à balayage (DSC) ... 150
III.11. Dichroïsme circulaire (CD) ... 150
III.12. Résonance plasmonique de surface (SPR) ... 150
III.13. Microscopie électronique à coloration négative ... 151
III.14. Tests d’adsorption ... 151
III.15. Thermo-fluorescence (FTSA) ... 151
III.16. Ultracentrifugation analytique (UCA) ... 152
Annexes ... 153
A. Séquences nucléotidiques et protéiques ... 155
A.1. Le plasmide pET28 (Novagen) ... 155
A.2. Constructions de pb10 et des domaines N- et C-terminaux ... 156
A.2.1. Plasmide pET28b-pb10 ... 156
A.2.2. Séquences protéiques de pb10 entière et des domaines N- et C-terminaux ... 156
A.3. Protéines de fusion pb10 + mCherry ... 157
A.4. Constructions de p144... 157
B. Propriétés des protéines et capsides utilisées ... 158
B.1. Propriétés des protéines utilisées ... 158
B.2. Masses molaires des capsides vides et pleines ... 158
C. Propriétés des tampons utilisés ... 159
D. Article ... 159
VIII
Abréviations
2xTY : milieu de culture 2xTY (Yeast extract Tryptone)ADN : Acide DésoxyriboNucléique ARN : Acide RiboNucléique ARNt : ARN de transfert
AU, mAU : unités arbitraires (en anglais : arbitrary units), millièmes d’unités arbitraires (milli-AU) bp, kbp : paires de bases (en anglais : base pairs), milliers de paires de bases (kilo-bp)
BSA : albumine de sérum bovin (en anglais : Bovine Serum Albumin) CD : Dichroïsme Circulaire (en anglais : Circular Dichroism)
CsCl : chlorure de césium
C-ter : extrémité carboxy-terminale (ou C-terminale) d’une protéine Da, kDa, MDa : Daltons, kilo-Daltons, méga-Daltons
DLS : Diffusion Dynamique de la Lumière (en anglais : Dynamic Light Scattering) DOxxx : densité optique à xxx nm de longueur d’onde
DSC : Calorimétrie Différentielle à Balayage (en anglais : Differential Scanning Calorimetry)
E. coli : bactérie Escherichia coli
EDTA : Ethylène Diamine Tétra-Acétique
EM, cryo-EM : microscopie électronique (en anglais : Electron Microscopy), cryo-microscopie électronique
EMSA : retard sur gel (en anglais : Electrophoretic Mobility Shift Assay)
FST : premier fragment d’ADN de T5 à être éjecté dans la cellule hôte (en anglais : First Step Transfer) et comprenant les gènes précoces
FTSA : thermo-fluorescence (en anglais : Fluorescence-based Thermal Shift Assay) GFP : protéine fluorescente verte (en anglais : Green Fluorescent Protein)
His-tag : étiquette Histidine (en anglais : Histidine tag), généralement 6 histidines
HSQC : spectre de RMN faisant apparaître les liaisons N-H (Heteronuclear Single Quantum Coherence) I2BC : Institut de Biologie Intégrative de la Cellule
IBBMC : Institut de Biochimie et Biophysique Moléculaire et Cellulaire ICSN : Institut de Chimie des Substances Naturelles
Ig-like : de type immunoglobuline
IMAC : chromatographie d’affinité sur colonne de métal immobilisé (en anglais : Immobilized Metal Affinity Chromatography)
IPTG : IsoPropyl β-D-1-ThioGalactopyranoside Kan : Kanamycine
LB : milieu de culture Luria-Bertani LPS : LipoPolySaccharide
MALS : diffusion de lumière multi-angle (en anglais : Multi-Angle Light Scattering)
IX NHS-EDC : N-hydroxysuccinimide et 1-ethyl-3-(3-dimethylaminopropyl)-carbodiimide
NOESY : spectre RMN faisant apparaître les atomes d’hydrogène spatialement proches (Nuclear Overhauser Effect SpectroscopY)
NTA : acide nitrilotriacétique
N-ter : extrémité amine (ou N-terminale) d’une protéine O/N : sur la nuit (en anglais : overnight)
PBS : Tampon Phosphate Salin (en anglais : Phosphate-Buffered Saline)
PCR : Réaction de polymérisation en Chaîne (en anglais : Polymerization Chain Reaction) pI : point isoélectrique d’une protéine (pH auquel la charge globale de la protéine est nulle) pmC : protéine de fusion pb10-mCherry
pNmC : protéine de fusion pb10Nter-mCherry
RBP : protéine de fixation au récepteur (en anglais : Receptor Binding Protein) située à l’extrémité de la queue des phages caudés
RBS : site de fixation du ribosome (en anglais : Ribosome Binding Site) RMN : Résonance Magnétique Nucléaire
RMSD : distance moyenne entre les atomes de protéines superposées (en anglais : Root-Mean-Square Deviation)
RU : unités de résonance en SPR (en anglais : Resonance Units)
SAXS : Diffusion des Rayons X aux Petits Angles (en anglais : Small-Angle X-ray Scattering) SDS : Sodium Dodécyl Sulfate
SDS-PAGE : Electrophorèse sur gel de polyacrylamide en présence de SDS (en anglais : PolyAcrylamide Gel Electrophoresis)
SEC : chromatographie d’exclusion de taille (en anglais : Size Exclusion Chromatography) SPR : Résonance Plasmonique de Surface (en anglais : Surface Plasmon Resonance) TerL : grande sous-unité de la terminase (en anglais : Terminase Large sub-unit) TerS : petite sous-unité de la terminase (en anglais : Terminase Small sub-unit) TEV : protéase du virus de la gravure du tabac (en anglais : Tobacco Etch Virus)
TMP : protéine déterminant la longueur de la queue des phages caudés (en anglais : Tape-Measure Protein)
TOCSY : spectre RMN faisant apparaître les atomes d’hydrogène séparés par 3 liaisons au maximum (TOtal Correlation SpectroscopY)
Tris : Trishydroxyméthylaminométhane UCA : UltraCentrifugation Analytique UMR : Unité Mixte de Recherche
VLP : particules similaires aux virus (terme anglais : Virus-Like Particle) WB : Western Blot
1
Partie I : Introduction
3
I.1.
Les Bactériophages
Les bactériophages, aussi appelés phages, sont les virus infectant les bactéries. Comme tous les virus, ce sont des parasites obligatoires qui se multiplient aux dépens d’une cellule hôte. Les phages sont les entités biologiques les plus répandues sur Terre : leur nombre est estimé à 1030 – 1032, soit au
moins 10 fois plus que le nombre de bactéries. Ils sont présents dans tous les écosystèmes colonisés par les bactéries, y compris dans les milieux extrêmes [1]–[3]. Par le nombre d'infections lytiques qu'ils produisent (estimé à environ 1023 par seconde), ils sont responsables d'une part importante du
recyclage de la matière organique et influent donc largement sur les cycles biogéochimiques [4], [5]. Les phages tempérés sont des acteurs majeurs du transfert horizontal de gènes chez les bactéries et de ce fait jouent un rôle écologique primordial dans la dynamique et l'évolution des populations bactériennes [1], [2].
I.1.1. Histoire : découverte des bactériophages
Les bactériophages ont été découverts indépendamment par deux chercheurs : Frederick Twort de la Brown Institution à Londres, et Félix d’Hérelle de l’Institut Pasteur à Paris. En 1915, Frederick Twort a observé des « colonies vitreuses » sur boîtes de Pétri. Ces « colonies » étaient capables de se répandre et de se multiplier indéfiniment dans ses cultures. Elles ont par la suite été attribuées à la présence de phages. De son côté, Félix d’Hérelle a observé en 1917 un micro-organisme provoquant la lyse bactérienne. Il pensait déjà à l’époque qu’il s’agissait d’un virus et lui a donné le nom de bactériophage. Certains phénomènes observés précédemment sont aujourd’hui également attribués aux bactériophages. Par exemple, plusieurs scientifiques ont reporté à la fin du XIXe siècle un effet antiseptique des eaux du Gange et de la Yamuna en Inde contre certaines bactéries, comme Vibrio
cholerae, qui était probablement dû à des bactériophages [6], [7].
Après leur découverte au début du XXe siècle, les bactériophages ont fait l’objet de nombreux débats quant à leur nature. Certains scientifiques pensaient qu’il s’agissait d’enzymes bactériennes autolytiques induites par des facteurs extérieurs alors que d’Hérelle soutenait que les bactériophages étaient des particules indépendantes proches des virus. C’est finalement avec l’avènement de la microscopie électronique que les bactériophages ont pu être observés pour la première fois en 1940. Leur nature particulaire a alors été avérée, mettant ainsi fin au débat [6].
I.1.2. Utilisation des bactériophages en médecine : la phagothérapie
Dès 1919, F. d’Hérelle a voulu développer l’utilisation des phages pour soigner les infections bactériennes. Il a obtenu des résultats très encourageants contre la typhose aviaire des poulets, la septicémie hémorragique bovine, puis la peste humaine [6].
Dans les années 1920 – 1930, plusieurs expériences de phagothérapie, appelée aussi thérapie phagique, ont été menées pour lutter contre les épidémies, notamment contre le choléra. Malgré des résultats très prometteurs, certains scientifiques et le grand public sont restés septiques quant à
4
l’efficacité de ce mode de traitement. En effet, les expériences menées manquaient souvent de contrôles adéquats (comme des groupes témoins non traités ou traités avec des méthodes conventionnelles) prouvant que les effets observés étaient dus aux phages. Dans certains cas, l’utilisation de bactériophages n’a pas permis de soigner les patients, et a parfois même empiré leur état. On pense aujourd’hui que ces effets négatifs étaient probablement dus à des toxines bactériennes présentes dans les préparations de phages qui étaient administrées. De plus, comme la nature exacte des phages n’était pas connue, les conditions de préparation et de conservation des cocktails de phages étaient souvent inadaptées, conduisant à des préparations peu efficaces [7], [8].
Au cours de la deuxième moitié du XXe siècle, l’intérêt pour les phages a fortement diminué avec la découverte et le développement des antibiotiques. En effet, les phages sont très spécifiques et ne ciblent souvent qu’une seule souche bactérienne, alors que les antibiotiques permettent d’éliminer un large spectre de bactéries. Cependant, la phagothérapie a continué à être développée dans l’ex-URSS où l’accès aux antibiotiques était limité. Aujourd’hui encore, l’Institut Eliava (du nom de son fondateur George Eliava) situé en Géorgie est une référence mondiale en thérapie phagique [7]–[9].
De nos jours, il y a un regain d’intérêt pour l’utilisation des phages en médecine principalement à cause de l’émergence de souches de bactéries pathogènes multi-résistantes aux antibiotiques. Un des intérêts majeurs des phages par rapport aux antibiotiques est leur capacité à co-évoluer avec leurs hôtes : lorsque les bactéries développent des résistances aux phages, ces derniers peuvent rapidement évoluer pour contourner ces résistances. De plus, la spécificité des phages permet de combattre un pathogène ciblé sans affecter les autres bactéries qui participent au bon fonctionnement du corps humain, contrairement aux antibiotiques qui ont un effet délétère sur la flore intestinale par exemple [8], [10]. Ainsi des groupes de recherche de plus en plus nombreux travaillent aujourd’hui sur la phagothérapie qui apparaît comme une bonne alternative à l’utilisation des antibiotiques pour le traitement de certaines infections. Il reste cependant des freins d’ordre législatif dans de nombreux pays, dont la France : les bactériophages ont un mode d’action très différent des autres substances thérapeutiques actuellement utilisées et autorisées. Les règlementations doivent donc être adaptées pour que la phagothérapie puisse être correctement développée [11], [12].
I.1.3. Les bactériophages et la biologie moléculaire
Les bactériophages sont des systèmes simples comparés à la cellule, et se reproduisent très rapidement. Ils sont donc un modèle de choix pour l’étude des mécanismes du vivant. En effet, c’est en étudiant les bactériophages que les chercheurs Max Delbrück, Alfred Hershey et Salvador Luria, récompensés du prix Nobel de physiologie ou médecine en 1969, ont fait des découvertes majeures dans le domaine de la biologie moléculaire. Parmi ces découvertes, le phénomène de recombinaison a été mis en évidence à la fin des années 1940 : la co-infection d’une bactérie par deux phages différents a donné lieu à des phages hybrides, et des phages endommagés par des radiations ont pu être « réparés » grâce à un échange génétique avec d’autres phages infectant la même bactérie. Hershey a également démontré en 1952 que l’ADN est le support de l’information génétique : en marquant séparément l’ADN et les protéines, il a observé que seul l’ADN des phages pénètre dans la
5 cellule hôte et qu’il suffit à déclencher la production de nouveaux phages, cette expérience est connue sous le nom de l’expérience de Hershey-Chase [13]. Le rôle des ARN comme intermédiaires entre ADN et protéines a également été découvert par des études sur les phages : les compositions des ARN (adénine, uracil, cytosine et guanine) dans une bactérie non infectée ou dans des bactéries infectées par les phages T2 ou T7 sont différentes, et dans le cas des bactéries infectées cette composition correspond à la composition de l’ADN du phage [14].
Le système de restriction / modification, mécanisme de défense des bactéries contre les bactériophages, a été découvert dans les années 1960. Cette découverte a, elle aussi, fait l’objet d’un prix Nobel en physiologie ou médecine décerné en 1978 à Werner Arber, Dan Nathans et Hamilton Smith. Arber a découvert les enzymes de restriction capables de dégrader l’ADN en reconnaissant des séquences précises, procurant à la bactérie un moyen de défense contre l’ADN étranger, ainsi que le système de modification permettant de protéger l’ADN contre les enzymes de restriction. Smith a confirmé cette découverte en purifiant une enzyme de restriction et démontrant qu’elle coupe l’ADN au milieu d’une séquence symétrique spécifique. Enfin, Nathans a été le premier à envisager les applications possibles de ce système, notamment pour établir des cartes génétiques [15].
D’autres systèmes dérivés des bactériophages sont très utilisés en biologie moléculaire. Notamment, l’ADN ligase du phage T4 est utilisée pour lier des molécules d’ADN séparées. Le système enzyme de restriction + ADN ligase de T4 est un outil très pratique et encore couramment utilisé pour manipuler l’ADN, principalement pour le clonage de gènes. Le phage lambda a été développé comme vecteur de clonage et est utilisé pour injecter des grands fragments d’ADN dans des bactéries. L’ADN polymérase du phage T7 a été utilisée pour des séquençages, et l’ARN polymérase de T7 est utilisée pour l’expression de gènes sous contrôle du promoteur T7 [16].
Plus récemment, le système CRISPR-Cas, procurant à la bactérie une immunité adaptative contre l’ADN étranger dont les ADN de phages, a été découvert en 2007. Ce système repose sur l’enzyme Cas qui fait une coupure double brin au niveau d’une séquence d’ADN reconnue spécifiquement par un ARN guide. La séquence reconnue a été « mémorisée » lors d’une précédente rencontre. En utilisant des ARN guides conçus pour reconnaître une séquence cible, on peut utiliser le système CRISPR-Cas pour couper un ADN en un point précis et générer des mutations particulières par recombinaison homologue. Des enzymes Cas mutées ayant perdu leur activité nucléase peuvent également être utilisées pour réprimer ou activer l’expression d’un gène en se fixant sur une séquence opératrice [16]. L’étude des phages et de leurs interactions avec leurs hôtes a donc permis de nombreuses avancées dans la compréhension du vivant et a fourni divers outils largement utilisés en biologie moléculaire. Des systèmes dérivés des phages sont également utilisés dans de nombreux domaines d’application, dont certains sont discutés dans la partie I.5 page 29.
I.1.4. Morphologies et classification des bactériophages et virus d’archées
Les bactériophages, comme les virus d’archées, infectent des organismes unicellulaires. Pour cette raison ils sont souvent regroupés dans une même classification. Ces virus présentent des
6
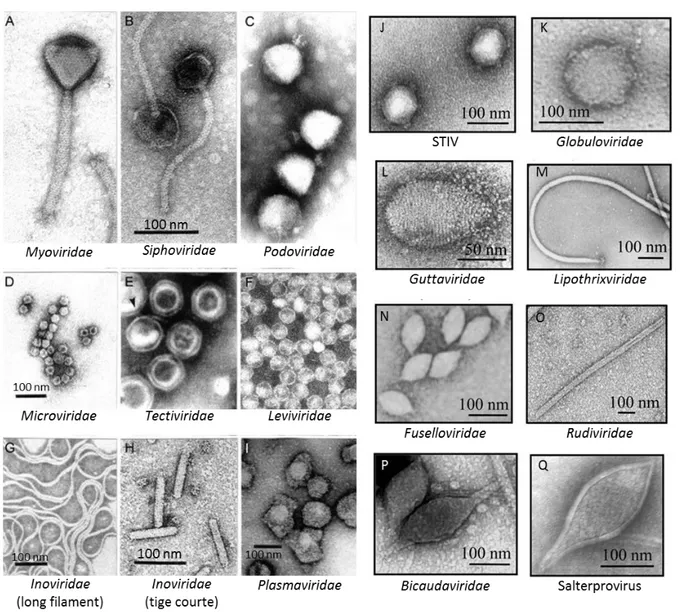
morphologies très diverses (Figure 1, Figure 2 page 8) : formes polyédriques plus ou moins allongées, avec ou sans queue, avec ou sans enveloppe lipidique, formes filamenteuses, pléomorphes. De même, les génomes de ces virus sont de nature variée : ADN ou ARN, simple ou double brin, linéaire ou circulaire [1], [17].
Figure 1 : Morphologie des bactériophages et virus d’archées.
Les virus d’archées sont identifiés par un astérisque. Figure adaptée de [17].
La classification évolue en fonction des nouveaux phages et virus d’archées qui sont découverts chaque année. On compte aujourd’hui 15 familles de bactériophages et virus d’archées et 5 groupes viraux supplémentaires qui ne sont pas encore classés (Figure 1). Ces familles peuvent être regroupées en 4 catégories basées sur la forme des virions [1], [17]–[19]:
Les phages à queue (phages caudés)
Cette catégorie de phages correspond à l’ordre des Caudovirales, l’un des 7 ordres de virus établis par le Comité International de Taxonomie des Virus [19]. Ce sont les phages les plus représentés : 96 % des phages observés jusqu’à aujourd’hui sont des phages caudés [20]. Ils sont composés d’une capside de forme icosaédrique renfermant un ADN double brin linéaire et d’une
queue plus ou moins longue. Il en existe 3 familles:
- Myoviridae (Figure 2-A): phages possédant une queue contractile
- Siphoviridae (Figure 2-B): phages possédant une longue queue non-contractile
7 Les phages de forme polyédrique
Ces phages possèdent une capside de forme icosaédrique et leur génome est constitué d’ADN simple ou double brin. Ils sont répartis en 5 familles et 2 groupes non classés :
- Microviridae (Figure 2-D): phages possédant un ADN simple brin circulaire
- Corticoviridae : phages possédant un ADN double brin circulaire, leur capside est composée de
deux enveloppes protéiques séparées par une bicouche lipidique
- Tectiviridae (Figure 2-E): phages possédant un ADN double brin linéaire dont la capside contient
une vésicule lipoprotéique capable de prendre la forme d’un tube au moment de l’injection de l’ADN dans la cellule hôte
- Leviviridae (Figure 2-F): phages possédant un ARN simple brin linéaire et similaires aux poliovirus
- Cystoviridae : phages possédant 3 molécules d’ARN double brin linéaire et dont la capside est
entourée d’une enveloppe lipidique
- Groupe SHI : phages possédant un ADN double brin linéaire dont la capside contient une vésicule lipidique
- Groupe STV1 : phages possédant un ADN double brin circulaire dont la capside arbore des protrusions
Les phages et virus d’archées filamenteux
Il existe 3 familles et groupes de phages et virus d’archées de forme filamenteuse :
- Inoviridae (Figure 2-G et H) : phages en forme de long filament ou de tige courte possédant un
ADN simple brin circulaire et dont les virions nouvellement formés sont libérés de la cellule hôte par extrusion
- groupe Lipothrixviridae : virus d’archées possédant un ADN double brin linéaire formant un complexe de type nucléosome et dont la capside est entourée d’une enveloppe lipoprotéique - groupe Rudiviridae : virus d’archées en forme de tige rigide possédant un ADN double brin linéaire
et présentant des structures de fixation sur une extrémité Les phages et virus d’archées pléomorphes
Ces virus sont classés en 6 familles et un groupe en attente de classification :
- Plasmaviridae (Figure 2-I): phages possédant un ADN double brin circulaire formant une granule
nucléoprotéique entourée d’une enveloppe lipidique
- Fuselloviridae : virus d’archées en forme de fuseau possédant un ADN double brin circulaire dont
l’enveloppe est composée de protéines et de lipides et dont les virions nouvellement formés sont libérés de la cellule hôte par extrusion
8
- Guttaviridae : virus d’archées en forme de goutte possédant un ADN double brin circulaire
- Ampullaviridae : virus d’archées en forme de bouteille possédant un ADN double brin linéaire
- Bicaudaviridae : virus d’archées possédant un ADN double brin circulaire et 2 queues
- Globuloviridae : virus d’archées possédant un ADN double brin linéaire
Figure 2 : Images de microscopie électronique représentant plusieurs familles de phages (A-I) et virus d’archées (J-Q). A. Myoviridae : φBC6 infectant Bacillus cereus. B. Siphoviridae : γ infectant Bacillus anthracis. C. Podoviridae : P22 infectant
Salmonella typhimurium. D. Microviridae : φX174 infectant Escherichia coli. E. Tectiviridae : 37-14 infectant Thermus thermophilus. F. Leviviridae : MS2 infectant E. coli. G. Inoviridae en forme de long filament : X infectant E. coli. H. Inoviridae
en forme de tige courte : MVL51 infectant Acholeplasma laidlawii. I. Plasmaviridae : L2 infectant A. laidlawii. J. STIV :
Sulfolobus turreted icosahedral virus. K. Globuloviridae : Pyrobaculum spherical virus (PSV)-like particle. L. Guttaviridae : Sulfolobus neozealandicus droplet-shaped virus (SNDV). M. Lipothrixviridae : Sulfolobus islandicus filamentous virus (SIFV). N. Fuselloviridae : Sulfolobus spindle-shaped virus (SSV1). O. Rudiviridae : Sulfolobus islandicus rod-shaped virus (SIRV). P. Bicaudaviridae : Acidianus two-tailed virus (ATV)-like particle. Q. Salterprovirus : Sulfolobus tengchongensis spindle-shaped
9
I.2.
Structure du bactériophage T5
Le laboratoire dans lequel j’ai effectué ma thèse étudie le bactériophage T5 (Figure 3) qui infecte la bactérie à Gram négatif Escherichia coli. Ce phage appartient à l’ordre des Caudovirales et à la famille des Siphoviridae (voir section I.1.3 page 4). Il est composé d’une capside de forme icosaédrique de grande taille (90 nm de diamètre) renfermant un ADN double brin linéaire de 121 752 paires de bases et d’une longue queue non contractile. Les protéines de structure de T5 sont dénommées selon leur position sur gel de polyacrylamide. Les lettres « pb » (pour « protein band ») sont suivies d’un numéro allant de 1 à 11 de la protéine la plus grande à la plus petite [23].
Figure 3 : Le bactériophage T5.
A. Image de microscopie électronique. B. Reconstruction de la particule virale à partir de données de cryo-microscopie électronique. La tête et la queue sont respectivement à 19 Å et à 30 Å de résolution. Les parties non résolues sont symbolisées en jaune (figure adaptée de [24]).
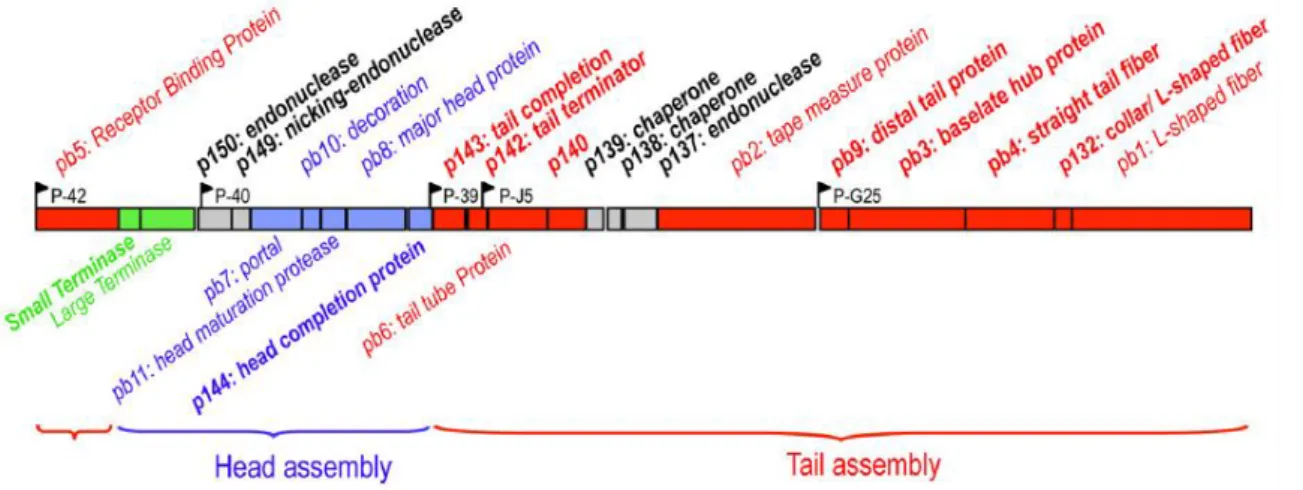
Les 16 gènes connus codant pour les protéines de structure sont regroupés dans un même module sur le génome de T5 (Figure 4) [25].
Figure 4 : Gènes codant pour les protéines de structure du phage T5.
Les gènes des protéines de capside sont colorés en bleu, ceux des protéines de queue en rouge, ceux de l’encapsidation de l’ADN en vert et les autres gènes sont en gris. Figure reproduite de [25].
10
I.2.1. Structure de la tête de T5
La tête d’un bactériophage à queue désigne la capside protéique avec son ADN.
I.2.1.1.
Les capsides icosaédriques
La capside est la coquille protéique qui protège le génome du virus. Comme celle de nombreux autres virus, procaryotes et eucaryotes, la capside de T5 est icosaédrique. Un icosaèdre est un polyèdre à 20 faces triangulaires et 12 sommets (Figure 5-A) et comporte 3 sortes d’axes de symétrie : les axes d’ordre 5 passant au niveau des sommets, les axes d’ordre 3 passant au centre des faces, et les axes d’ordre 2 passant au milieu des arrêtes. En faisant tourner l’icosaèdre de (360/5)°, (360/3)° et (360/2)° respectivement selon ces axes, on retrouve la même image (Figure 5-C). Dans une symétrie icosaédrique, on peut placer 60 sous-unités ayant des positions équivalentes (symbolisées par une apostrophe noire sur la Figure 5-B) [26].
Figure 5 : La symétrie icosaédrique.
A. Un icosaèdre dont les arêtes visibles sont représentées en trait plein et les arrêtes situées de l’autre côté de la figure sont en pointillés. B. Un icosaèdre dont les positions équivalentes sont représentées par une apostrophe noire. C. Vues d’un icosaèdre selon les axes d’ordre 5, 3 et 2. Les axes de symétrie d’ordres 5, 3 et 2 sont représentés respectivement par un pentagone, un triangle et un ovale. Figures B et C adaptées de [26].
Les capsides icosaédriques sont généralement constituées de multiples copies d’une ou deux protéines principales. Il existe des virus de petite taille ayant des capsides formées uniquement de 60 copies d’une protéine majeure de capside formant 12 pentamères, mais la plupart des capsides sont composées d’un plus grand nombre de sous-unités. Les protéines sont alors généralement assemblées en capsomères : hexamères sur les faces de l’icosaèdre et pentamères sur les sommets (Figure 6-A). Les sous-unités n’occupent plus des positions équivalentes car elles ont des interactions différentes avec les sous-unités voisines en fonction de leur position. On parle alors de « quasi-équivalence » : sur la Figure 6-B, les sous-unités occupent 4 positions quasi-équivalentes représentées par 4 couleurs différentes, les sous-unités d’une même couleur occupent des position équivalentes [26]. Le nombre de triangulation T a été introduit par Caspar et Klug en 1962 [27] pour décrire ce type de capside dont les faces sont divisées en plusieurs triangles plus petits. Le nombre de triangulation est un indicateur de la taille de la capside et se calcule à partir du nombre de capsomères selon la formule suivante :
11 T = h² + k² + h x k
où h et k sont les nombres de capsomères séparant deux pentamères proches (voir Figure 6-C).
Figure 6 : Organisation des capsides.
A. Carte de densité électronique de la capside du phage HK97 à 3.65 Å de résolution. Les hexamères sont représentés en jaune et bleu et les pentamères en rouge. Les structures d’un hexamère et d’un pentamère sont représentées sur la droite. Figure reproduite de [28]. B. Un icosaèdre de taille T=4 (H=2, K=0) composé de 240 sous-unités représentées par des apostrophes colorées selon leur position : les sous-unités ayant des positions équivalentes sont colorées d’une même couleur. Les centres des pentamères et des hexamères sont représentés respectivement par des pentagones et des hexagones. Figure reproduite de [26]. C. Icosaèdres avec différents nombres de triangulation. Les hexamères sont représentés en blanc et les pentamères en noir. Figure reproduite de [29].
Dans les capsides ayant un nombre de triangulation supérieur à 1, il existe des axes de symétrie locaux : sur la Figure 6-B il y a des axes de quasi-symétrie d’ordre 3 au centre de chaque triangle, mais seuls les triangles noirs possèdent de véritables axes de symétrie 3. De même, il y a des axes de quasi-symétrie d’ordre 2 entre deux triangles adjacents [26].
12
I.2.1.2.
La capside de T5
La capside du bactériophage T5 (Figure 7) a un nombre de triangulation T = 13, soit 120
hexamères sur les faces et 11 pentamères sur 11 des 12 sommets. Elle mesure environ 90 nm de diamètre [30]. Elle est composée des protéines suivantes :
- 775 copies de la protéine majeure de capside pb8 assemblées en hexamères et pentamères et formant la structure de la capside (en bleu sur la Figure 7)
- 120 copies de la protéine de décoration pb10 (en rouge sur la Figure 7) situées au centre de chaque hexamère et dont la fonction est inconnue
- 12 copies de la protéine portale pb7 assemblées en anneau sur un sommet unique et formant un pore (ou canal) d’entrée et de sortie pour l’ADN
- la protéine p144 formant un bouchon qui ferme le pore et permet de connecter la queue, le nombre de copies n’est pas connu mais il est probable qu’elle s’assemble en dodécamère comme c'est le cas pour d'autres phages [31].
Figure 7 : Reconstruction à 19 Å de résolution de la capside du phage T5 à partir d'images de microscopie électronique. La protéine majeure de capside pb8 est représentée en bleu et la protéine de décoration pb10 en rouge.
La protéine majeure de capside pb8 adopte un repliement de type HK97 (du nom de la première structure de protéine de capside résolue : protéine gp5 du phage HK97 [32]). Ce repliement est très répandu pour les protéines de capside icosaédrique. Un modèle de la structure de pb8 basé sur la structure de gp5 est présenté sur la Figure 8 [33]. Le domaine A (pour « axial ») est situé au centre des capsomères et le domaine P (pour « périphérique ») permet des contacts entre capsomères (Figure 8-D).
13 Figure 8 : pb8 a un repliement de type HK97.
A. Structure de la protéine de capside gp5 du phage HK97. Figure adaptée de [28]. B. Modèle de structure de la protéine pb8 de T5 obtenu par homologie avec la protéine gp5 de HK97 et généré à partir du serveur PHYRE. C. Alignement des séquences protéiques de pb8 et gp5. Les éléments de structure secondaire de gp5 sont indiqués par des flèches (brins bêta) et des cylindres (hélices alpha). Figures B et C adaptées de [33]. D. Modèle d’un hexamère de pb8 de T5, une sous-unité est colorée en rouge.
I.2.1.3.
L’ADN de T5
Le génome de T5 est formé d’une unique molécule d’ADN double brin linéaire de 121 752 bp [34] enroulée dans la capside en couches successives espacées d’environ 24.4 Å [24] (Figure 9). L’ADN est fortement compacté dans la capside (à environ 500 mg/mL [35]) et exerce une pression interne de plusieurs Mégapascals (MPa). La force interne générée par l’ADN compacté dans la capside a été déterminée chez le phage φ29 par Smith et al. [36]. Lorsque le génome complet de φ29 est dans la capside, la force interne est d’environ 57 picoNewtons (pN). La pression interne qui en résulte est estimée à environ 6 MPa [36].
Figure 9 : Image de cryo-EM en coupe de la tête de T5 montrant les couches successives d’ADN à l’intérieur de la capside. Figure adaptée de [24].
Les gènes de T5 sont classés en 3 groupes en fonction du moment auquel ils sont exprimés au cours du cycle viral : les gènes très précoces (« pre-early » en anglais) sont exprimés au tout début de l’infection, les gènes précoces (« early » en anglais) sont exprimés après la prise de contrôle des machineries de biosynthèse bactériennes et permettent, entre autres, la réplication de l’ADN phagique
14
et les gènes tardifs (« late » en anglais) sont exprimés en fin de cycle et codent principalement pour les protéines de structure. La portion d’ADN codant pour les gènes précoces (environ 10 kbp, soit 8 % du génome) est répétée aux deux extrémités de la molécule d’ADN. La majorité des gènes de T5 est codée sur le brin anti-sens.
L’ADN de T5 est interrompu en plusieurs positions sur le brin sens uniquement. Certaines de ces interruptions sont présentes sur le génome de chaque phage T5, alors que d’autres ne sont présentes que dans l’ADN de certains phages. Toutes ces interruptions sont précédées d’une courte séquence riche en cytosines et guanines qui semble être le signal reconnu par l’endonucléase responsable de ces coupures, qui a été récemment identifiée [25], [37]. Cette nucléase est codée par un gène localisé dans la région du génome codant pour les protéines de structure et d'assemblage de la capside de T5 (gène p149 : « nicking-endonuclease » sur la Figure 4 page 9 et la Figure 11-A page 19). La présence de ces interruptions n’est pas indispensable au phage car certains mutants de T5 qui sont dépourvus de cette nucléase sont infectieux. Malgré de nombreuses études menées dans les années 1970-1980, le rôle de ces interruptions, ou l'avantage qu'elles pourraient apporter au phage n’est toujours pas connu [37].
Le génome de T5 contient des gènes codant pour 24 ARN de transfert qui peuvent être délétés sans affecter le cycle viral dans des conditions de laboratoire (mutant T5st0).
15
I.2.2. Structure de la queue de T5
La queue permet la reconnaissance de la cellule hôte et le transfert de l’ADN phagique dans le cytoplasme de la bactérie. Chez T5, la queue mesure environ 200 nm et est constituée de 10 protéines différentes (Figure 10) [25]. 120 copies de la protéine de tube pb6 forment la structure principale de la queue. La « tape-measure protein » (TMP) pb2 fixe la longueur de la queue et est située dans le tube. 9 copies de la protéine pb1 forment 3 fibres longues trimériques permettant l’adsorption du phage à la surface de certaines souches de E. coli, par fixation réversible de pb1 au lipopolysaccharide (LPS) de la membrane externe [38], [39]. La protéine p132 est associée aux fibres longues et pourrait former le collier d’ancrage de ces fibres à la base du tube. La structure cristallographique de la protéine
distale pb9 a été résolue. Pb9 s'assemble en un anneau hexamèrique qui fait la jonction entre le tube
et la partie conique [40]. La protéine pb3 forme la partie supérieure du cône, et la protéine pb4 constitue la fibre droite à l’extrémité distale de la queue. Le nombre de copies pour chacune de ces deux protéines est estimé à 3. Une seule copie de pb5, la protéine de fixation au récepteur (RBP : « receptor binding protein » en anglais) reconnaît spécifiquement le récepteur FhuA à la surface de la bactérie hôte. Deux protéines p142 et p143 sont situées à l’extrémité proximale de la queue qui fait la jonction avec la capside. Par analogie avec des protéines équivalentes caractérisées chez d’autres phages comme la protéine gpU du phage lambda (λ) [41], on peut prédire qu’elles sont présentes au nombre de 6 copies et forment un anneau hexamérique.
16
I.3.
Le cycle infectieux du bactériophage T5
Il existe chez les phages deux types de cycles infectieux :
- le cycle lytique : après infection de la cellule hôte, de nouveaux virions sont assemblés et libérés rapidement par lyse de la cellule hôte.
- le cycle lysogénique : après infection, le génome du phage peut s’insérer dans le chromosome bactérien (formant ainsi un prophage) ou former un plasmide, se répliquant en même temps que l’ADN bactérien et se propageant dans les cellules filles. Le phage peut ainsi rester en dormance pendant des années jusqu’à ce que des conditions particulières le fassent entrer en cycle lytique.
Les phages virulents ne peuvent infecter que par la voie lytique alors que les phages tempérés peuvent emprunter les deux voies.
T5 est un phage virulent incapable d’entrer en lysogénie. Son cycle infectieux comprend
l’infection (reconnaissance de la bactérie hôte, injection de l’ADN dans le cytoplasme et détournement des machineries de biosynthèse), la production de nouveaux virions (réplication de l’ADN phagique, production des protéines de structure, assemblage des têtes et des queues) et la lyse
cellulaire (éclatement de la bactérie hôte pour libérer les virions nouvellement formés).
I.3.1. L’infection
I.3.1.1.
L’adsorption
L’adsorption désigne la fixation du phage sur sa cellule hôte. Dans un premier temps, T5 se lie de manière réversible sur le lipopolysaccharide (LPS) de la bactérie E. coli via ses fibres longues (voir section I.2.2 page 15) [42]. L'extrémité distale des fibres formées par la protéine pb1 interagit spécifiquement avec la partie antigène O du LPS de certaines souches d’E. coli, permettant ainsi au phage de se déplacer sur la surface de la cellule jusqu’à ce qu’il entre en contact avec son récepteur FhuA. FhuA est une protéine de la membrane externe d’Escherichia coli structurée en tonneau β. C’est un transporteur des ions Fe3+ complexés au Ferrichrome [43], [44]. La protéine pb5,
située à l’extrémité de la fibre centrale de la queue de T5 (Figure 10 page 15) reconnait spécifiquement la protéine FhuA et s’y fixe de manière irréversible [45].
I.3.1.2.
Le transfert d’ADN dans la cellule hôte
I.3.1.2.1. Passage à travers la paroi bactérienne
Après s’être fixée sur le récepteur FhuA, la protéine pb5 subit un changement conformationnel servant de signal pour l’éjection de l’ADN [46]. Le signal est propagé le long de la queue jusqu’au connecteur tête-queue. Celui-ci s’ouvre permettant d’initier l’éjection de l’ADN sous l’effet de la pression interne de la tête du phage. Les mécanismes moléculaires permettant le passage de l’ADN à
17 travers la paroi bactérienne ne sont pas connus. Toutefois, l’interaction pb5 – FhuA provoque l’ouverture du cône et l’éjection de la « tape measure protein » pb2 (voir section I.2.2 page 15). Celle-ci, avec la protéine de la fibre droite pb4, pourrait être responsable de la perforation de la paroi bactérienne. [25], [47], [48].
I.3.1.2.2. Transfert de l’ADN en deux étapes
Le phage T5 a la particularité de transférer son ADN dans la bactérie hôte en deux étapes [49]. La première partie de l’ADN à être transférée est l'ADN FST (pour « first step transfer ») qui comprend les gènes très précoces et représente environ 8 % du génome de T5. Lorsque l'ADN FST est entré dans le cytoplasme de l’hôte, le transfert est interrompu et le reste de la molécule d’ADN reste dans le phage encore adsorbé sur la cellule. Cette interruption dure quelques minutes pendant lesquelles les gènes très précoces codés par l’ADN FST sont exprimés. Les produits de ces gènes prennent le contrôle des fonctions cellulaires de l'hôte en détournant les machineries de biosynthèse et en neutralisant les mécanismes de défenses : l'ADN bactérien est massivement dégradé, la transcription des gènes bactériens est inactivée et l’ARN polymérase est redirigée vers la transcription des gènes viraux, les systèmes de restriction/modification et de réparation de l'ADN sont inactivés. Deux gènes précoces, A1 et A2, sont requis pour la reprise du transfert de l'ADN permettant le déroulement complet du processus infectieux : réplication de l'ADN viral, synthèse des protéines de structure et assemblage de nouveaux virus, puis lyse de la cellule hôte. Les mécanismes moléculaires qui contrôlent l’arrêt et la reprise du transfert de l’ADN ne sont pas connus [49], [50].
I.3.2. La production de nouveaux virions
Lorsque l’intégralité du génome est entrée dans la bactérie hôte, les gènes précoces (souvent appelés gènes intermédiaires chez les autres phages) sont exprimés pendant 8 minutes environ. Ceux-ci sont prinCeux-cipalement responsables de la réplication de l’ADN viral. Enfin, les gènes tardifs, codant principalement pour les protéines de structure, sont exprimés environ 12 minutes après l’infection [50]. La tête et la queue sont assemblées séparément. Les différentes étapes d’assemblage de la tête ont été décrites par Huet et al. [33], mais le processus d’assemblage de la queue est encore mal compris.
I.3.2.1.
La réplication de l’ADN phagique
L’ADN de T5 est répliqué par une ADN polymérase codée par un gène précoce [34] mais le mécanisme de réplication n’a pas encore été élucidé. La présence de concatémères formés de plusieurs unités de génome répétées dans une même molécule d’ADN a été démontrée [51]. Cependant la voie de formation de ces concatémères n’est pas connue. In vitro, l’ADN polymérase de T5 est capable de répliquer à la fois l’ADN circulaire par « rolling circle » et l’ADN linéaire en structures branchées en utilisant des interruption simple brin comme point de départ, l’ADN en amont de l’interruption servant d’amorce [37]. In vivo, les deux formes d’ADN, linéaire et circulaire, ont été
18
observées à différentes étapes du cycle de T5. Les deux voies de réplications sont donc possibles, et aucune n’a pu être écartée [52].
I.3.2.2.
L’assemblage de la tête
Le processus d’assemblage de la tête de T5 est similaire à celui de nombreux autres phages et virus eucaryotes de type herpès. Il consiste en l’assemblage d’une procapside compacte vide qui est ensuite remplie avec l’ADN viral, ce qui provoque l’expansion de la capside [53].
I.3.2.2.1. Assemblage de la procapside
Le processus d’assemblage de la tête de T5 est illustré par la Figure 11 (page 19) [33]. La première étape est l’assemblage d’une capside compacte appelée procapside, ou « prohead ». Cet assemblage implique trois protéines : la protéine portale pb7 (représentée en bleu sur la Figure 11), la protéine majeure de capside sous sa forme précurseur pb8p (en violet) et la protéase de maturation pb11 (en jaune). La protéine portale pb7 s’assemble en dodécamère et forme un anneau. La protéine pb8p s’auto-assemble grâce à son domaine d’échafaudage, aussi appelé « Δ-domain » (symbolisé par un triangle gris). Chez certains autres phages (par exemple P22, T7 ou φ29) et virus eucaryotes (HSV-1), c’est une protéine d’échafaudage indépendante qui permet l’assemblage de la protéine majeure de capside. Ce premier intermédiaire d’assemblage est appelé « prohead I ». La protéase de maturation pb11 se clive elle-même et clive le domaine d’échafaudage de pb8p libérant l’espace intérieur. La régulation de cette maturation protéolytique n'est pas connue, ni le nombre de copies de pb11 impliquées, mais on sait que quelques copies de pb11 clivée à ses deux extrémités restent associées à la capside. Cette procapside vide est appelée « prohead II ».
I.3.2.2.2. Encapsidation de l’ADN et expansion
L’ADN est encapsidé dans la prohead II à travers le canal formé par la protéine portale grâce à une enzyme appelée terminase (en vert sur la Figure 11). Le processus d’encapsidation est commun aux phages caudés et aux herpesvirus. La machine d'encapsidation est composée de deux sous-unités de la terminase TerS et TerL qui s’assemblent en oligomères ainsi que du canal portal situé sur l’un des sommets de la procapside. La petite sous-unité TerS forme un anneau de 8 à 12 copies qui reconnaît l’ADN phagique répliqué sous forme de concatémères. La grande sous-unité TerL est pentamérique et a une double activité ATPase et DNAse. Elle permet la translocation de l’ADN dans la capside grâce à l’énergie d’hydrolyse de l’ATP. Dans le cas de T5, lorsqu’une unité de génome a été encapsidée, TerL coupe l’ADN au niveau d'un site spécifique pour le séparer du concatémère grâce à son activité endonucléase. Ainsi tous les virions produits ont exactement la même séquence d’ADN. Ce mode d’encapsidation s’oppose à l’encapsidation dite de « tête pleine » décrite pour d’autres phages (SPP1, T4, lambda par exemple) et pour laquelle la coupure est non spécifique et intervient lorsque la tête est remplie. Le processus de tête pleine conduit à l’encapsidation d’environ 105 % du génome. Il y a alors une fraction d’ADN qui est répétée, mais cette fraction varie d’un virion à l’autre [54]–[58].
19
La capside s’expanse au cours de l’encapsidation, probablement sous l’effet de la pression
exercée par l’ADN compacté et/ou d’interactions électrostatiques entre l’ADN et la protéine pb8. Toutes les sous-unités de la protéine majeure de capside subissent simultanément un réarrangement conformationnel irréversible permettant de doubler le volume de la capside qui peut ainsi contenir la totalité du génome. Le diamètre moyen de la capside augmente ainsi de 70 nm (prohead II) à 90 nm (capside expansée) [30], [33]. Lorsque la capside est expansée, la protéine de décoration pb10 (en rouge sur la Figure 11) se fixe sur la surface externe de la capside, au centre de chacun des hexamères de pb8. L’assemblage de la tête est achevé par la fixation de la protéine de fermeture p144 qui ferme le canal portal pour empêcher la sortie de l’ADN encapsidé. La protéine de fermeture associée à la portale forme le connecteur qui se fixe à la queue assemblée séparément [33]. Chez la plupart des autres phages dont la structure a été caractérisée, le connecteur est composé de deux protéines : un adaptateur qui prolonge le canal portal, et un stoppeur qui ferme le pore [31]. T5 présente donc un modèle de connecteur différent, qui n'a pas encore été décrit sur le plan structural.
Figure 11 : Assemblage de la tête du bactériophage T5.
A. Schéma des gènes codant pour l’assemblage de la tête. B. Schéma du procédé d’assemblage de la tête. C. En haut, un schéma d’une capside ayant un nombre de triangulation T = 13 sur lequel les hexamères sont symbolisés par le chiffre 6 et les pentamères par le chiffre 5. En bas, un modèle de la capside de T5 reconstruit à partir de données de cryo-microscopie électronique. Figure adaptée de [33].
20
I.3.2.3.
L’assemblage de la queue
Les différentes étapes de l’assemblage de la queue de T5 ne sont pas connues, mais le procédé est probablement similaire à celui de l’assemblage de la queue du phage lambda (illustré en Figure 12), qui appartient également à la famille Siphoviridae et infecte E. coli. Dans le cas de lambda, la première étape consiste en l’assemblage de 3 copies de la protéine gpJ de fixation au récepteur. Ensuite les protéines gpI, gpL puis gpK, qui font également partie de l’extrémité distale de la queue, s’assemblent autour de gpJ. Puis la « tape-measure protein » gpH se positionne sur le complexe initial pour former la partie centrale de la queue. Elle sert de support pour la polymérisation de la protéine majeure de queue gpV qui s’assemble autour de gpH avec le concours des protéines chaperonnes gpG et gpGT. La taille de la queue est ainsi déterminée par la longueur de gpH. Enfin la protéine de terminaison de la queue gpU coiffe l'extrémité de la queue pour permettre l’attachement de la tête en présence de la protéine gpZ [59].
21
I.3.3. La lyse cellulaire
Le procédé de lyse de la cellule hôte est commun à de nombreux phages et fait appel principalement à deux types de protéines : les holines et les endolysines (appelées aussi lysozymes). Les holines sont de petites protéines membranaires qui perméabilisent la membrane interne pour permettre aux endolysines d’accéder au peptidoglycane. Ces dernières dégradent le peptidoglycane ce qui provoque la lyse de la bactérie et la libération des virions nouvellement formés. Les holines et endolysines sont produites assez tôt dans le cycle viral et s’accumulent dans la cellule. L’action des holines est fortement minutée : la perméabilisation de la membrane ne survient qu’à un moment précis. En absence d’holines, la bactérie hôte n’est pas lysée et la synthèse de nouveaux virus continue de manière linéaire pendant des durées étendues. L’avantage procuré par une lyse programmée est de permettre une succession de cycles infectieux dans une population bactérienne conduisant à une production de phages exponentielle. Le minutage de la lyse bactérienne peut être régulé par les holines en fonction des conditions. Par exemple, la lyse peut être prématurée si la bactérie hôte subit une deuxième infection par un autre phage. Ainsi ce deuxième phage ne peut pas se multiplier. Au contraire, la lyse peut intervenir plus tard si des signaux indiquent qu’il n’y a pas d’autres bactéries hôtes dans l’environnement [60], [61].
Dans des conditions optimales, la bactérie hôte est lysée 35 à 50 minutes après l’infection par T5 et libère environ 200 nouveaux phages [37].
22
I.4.
Les protéines auxiliaires de capside
Les protéines auxiliaires se fixent à la surface des capsides icosaédriques de certains bactériophages et virus eucaryotes dans les dernières étapes d’assemblage (après l’expansion). L’absence de fixation des protéines auxiliaires avant l’expansion indique que les sites de fixation sont formés (ou rendus accessibles) par le changement de conformation des protéines majeures de capsides au moment de l’expansion. Ces protéines auxiliaires peuvent être classées en deux catégories : les protéines-ciment, qui lient les capsomères entre eux et stabilisent la capside, et les
protéines de décoration, qui se fixent au centre des hexamères et dont la fonction est plus floue.
Certains phages n’ont aucune protéine auxiliaire (par exemple HK97), certains présentent l’un ou l’autre des deux types de protéines, et d’autres possèdent les deux (par exemple T4 avec ses protéines Soc et Hoc).
I.4.1. Les protéines-ciment
La capside de certains phages et virus eucaryotes (herpès et adénovirus) est renforcée par une ou plusieurs protéines-ciment [62]. Par exemple, la protéine gpD du phage λ s’assemble en trimères au niveau des jonctions entre 3 capsomères de manière à les stabiliser (Figure 13-A) [62]–[64]. Le
phage L, apparenté au phage P22, possède une protéine-ciment, Dec, qui s’assemble en trimères et
qui se fixe sur deux types de sites entre les capsomères : au niveau des axes de quasi-symétrie d’ordre 3 et au niveau des véritables axes de symétrie 3 (Figure 13-B)[65]. La protéine Soc (pour « small outer capsid prtotein») du phage T4 apporte une double stabilisation : une copie de Soc lie 2 capsomères adjacents et Soc trimérise au niveau de l’interface entre 3 hexamères (Figure 13-C, Figure 15-A) [66]– [68]. La protéine gp10 du phage ε15 forme des dimères jouant un rôle d’agrafes entre les capsomères [69]. Le virus herpès HSV-1 possède plusieurs protéines ciment : les protéines triplex VP19C et VP23 assemblées en hétéro-trimères servent de protéines d’échafaudage externes pendant l’assemblage de la capside, puis servent d’attaches entre 3 capsomères adjacents dans la capside mature, et la protéine UL25 forme des hétéro-dimères avec la protéine UL17 et se fixe autour des pentamères situés aux sommets de la capside apportant une stabilisation supplémentaire (Figure 13-D) [62], [70], [71]. La protéine IX confère aux adénovirus une meilleure résistance à la chaleur en stabilisant les interfaces entre les hexamères [62], [72], [73].
23 Figure 13 : Différentes stratégies de stabilisation des capsides.
A. La protéine-ciment gpD du phage lambda. Haut : densités électroniques correspondant à gpD. Bas : densité électronique de gpD superposée à la structure cristalline de la capside du phage HK97 qui est analogue à celle de lambda. Figure adaptée de [63]. B. La protéine-ciment Dec du phage L. Haut : soustraction de la carte de cryo-EM du phage P22 (ne possédant pas de ciment) à celle du phage L. Bas : grossissement sur deux trimères de Dec. Figure adaptée de [65]. C. La protéine-ciment Soc du phage T4. Haut : représentation de Soc en rouge, les pentamères et le complexe portal sont représentés en magenta et vert respectivement. Bas : un hexamère (bleu) entouré de 6 trimères de Soc (rouge), les hexamères voisins sont représentés en vert, noir et magenta. Figure adaptée de [67]. D. Les protéines-ciment VP19C, VP23 et UL25 du virus herpès HSV-1. Haut : coupe d’une reconstruction de cryo-EM de la capside de HSV-1, l’ADN est représenté en vert et la protéine UL25 en orange. Bas : un pentamère entouré de 5 protéines UL25 (en orange), les protéines triplex VP23 et VP19C stabilisant les capsomères sont représentées en bleu. Figure adaptée de [62] E. Les liaisons inter-capsomère du phage P22. Haut : cryo-reconstruction de la capside du phage P22 expansée artificiellement par la chaleur (les pentamères sont perdus au cours de ce procédé, voir section I.5.1 page 30) avec des modèles d’hexamères de 3 couleurs différentes. Les arrêtes de l’icosaèdre sont représentées par des lignes noires. Bas : grossissement de la zone entourée en pointillés, les boucles P (« P-loops », voir Figure 8) impliquées dans la stabilisation sont représentées en noir. Figure adaptée de [74]. F. Les pontages inter-capsomères du phage HK97. Haut : représentation des régions impliquées dans la formation d’un réseau de type cotte de maille. Bas : pontage (en magenta) liant 3 hexamères adjacents. Figure adaptée de [63].
![Figure 12 : Schéma du procédé d'assemblage de la queue du phage lambda. Figure reproduite de [59]](https://thumb-eu.123doks.com/thumbv2/123doknet/12888105.370468/35.892.188.733.450.670/figure-schéma-procédé-assemblage-queue-lambda-figure-reproduite.webp)