Dynamique d’infection des sous-populations
lymphocytaires T CD4 chez le macaque Rhésus infecté
par le Virus de l’Immunodéficience Simienne
Mémoire
Henintsoa RABEZANAHARY AINA
Maîtrise en microbiologie-immunologie
Maître ès sciences (M.Sc.)
Québec, Canada
iii
Résumé
Au cours de l’infection par le VIH, les réservoirs viraux s’établissent précocement, et représentent un problème majeur quant à leurs éradications. Comprendre les bases de cette dissémination virale, en particulier dans les tissus profonds comme la rate difficilement analysable chez l’Homme, et au cours de la phase précoce de l’infection, représente un enjeu de santé publique. Notre hypothèse de travail a été d’étudier une sous-population de lymphocytes T CD4 (Tfh, T follicular helper cells) qui joue un rôle essentiel dans l’établissement de la réponse humorale, et qui est prépondérante dans la rate. Mes résultats montrent chez le macaque Rhésus infecté par le virus de l’immunodéficience simienne (SIV) que durant la phase de primo-infection les cellules Tfh de la rate sont une cible du virus. Cette infection précoce de ces cellules pourrait donc contribuer à la dissémination de l’infection et rendre compte de l’absence de réponse humorale efficace contre ce virus.
v
Abstract
During HIV infection, viral reservoirs are established early, and represent a major problem regarding their eradication. Understanding the basis of viral dissemination, particularly in deep tissues such as the spleen, which is difficult to analyze in Human, particularly during the early phase of infection, is a challenge for public health. Our hypothesis was to determine the contribution of a subset of CD4 T cells namely Tfh (T follicular helper cells) that play a major role in the establishment of humoral response and which is preponderant in the spleen. My results showed in rhesus macaques infected with simian immunodeficiency virus (SIV) that during the acute phase of infection, splenic Tfh cells are a target of the virus. This early infection of these cells could therefore contribute to the spread of infection and account for the lack of effective antibody response against the virus.
vi
vii
Table des matières
Résumé ... iii
Abstract ... v
Table des matières ... vii
Liste des tableaux... ix
Liste des figures ... xi
Liste des abréviations ... xiii
Remerciements ... xv
Chapitre 1 : Introduction ... 1
1.1 Epidémiologie du Sida ... 1
1.2 Primate non humain et SIDA... 4
1.2.1 PNH-hôtes naturels ... 4
1.2.2 PNH-hôtes non naturels ... 4
1.3 Infection à VIH ... 5
1.3.1 Virus du Sida ... 5
1.3.1.1 Structure du virus ... 5
1.3.1.2 Etapes du cycle de réplication virale ... 8
1.3.1.2.1 Étapes précoces ... 8
1.3.1.2.2 Étapes tardives ... 10
1.3.2 Evolution de la pathogenèse ... 11
1.4 Cellules cibles ... 14
1.4.1 Cellules de la lignée myéloïde ... 14
1.4.2 Cellules de la lignée lymphoïde ... 15
1.4.2.1 Différenciation des lymphocytes T CD4 ... 15
1.4.2.2 Infection des sous-populations LT CD4 ... 18
Chapitre 2 : Hypothèse et objectifs ... 19
Chapitre 3 : Matériel et méthodes ... 21
3.1 Mise au point des techniques de qPCR ... 21
3.1.1 Infection des cellules CEMx174 ... 22
3.1.2 qPCR sur les CEMx174 infectées ... 22
3.1.2.1 Principe de la qPCR ... 22
3.1.2.2 Quantification relative des rétrotranscrits précoces et tardifs ... 23
3.1.2.3 Quantification de l’ADN viral ... 23
3.1.2.4 Quantification de la charge virale ... 24
3.2 Infection des macaques Rhésus par le VIS ... 25
3.2.1 Animaux, infection et prélèvements ... 26
3.2.2 Quantification de la charge virale plasmatique ... 26
3.2.3 Tri cellulaire ... 26
3.2.3.1 Préparation des échantillons ... 26
3.2.3.2 Tri des différentes sous-populations ... 27
3.2.3.3 Activation cellulaire ... 28
3.3 Analyse statistique ... 30
3.4 Ethique ... 30
Chapitre 4 : Résultats ... 31
4.1 Dynamique temporelle du cycle réplicatif dans les cellules CEMx174 infectées par le SIVmac251 31 4.2 Infection des macaques Rhésus ... 33
4.2.1 Évolution de la charge virale plasmatique ... 33
4.2.2 Dynamique d’infection des sous-populations LT CD4 ... 35
viii
4.2.2.1.1 Au niveau de la rate ... 35
4.2.2.1.2 Au niveau des ganglions axillaires et inguinaux... 37
4.2.2.1.3 Comparaison entre rate et ganglions ... 39
4.2.2.2 Dynamique du cycle réplicatif durant la phase chronique ... 43
4.2.2.2.1 Au niveau de la rate ... 43
4.2.2.2.2 Au niveau des ganglions axillaires et inguinaux... 44
4.2.2.2.3 Comparaison entre rate et ganglions ... 45
4.2.3 Capacité des cellules infectées à produire du virus infectieux ... 47
Chapitre 5 : Discussion ... 49
Chapitre 6 : Conclusions et perspectives ... 53
ix
Liste des tableaux
Tableau 1 : Fonction des protéines virales ... 7 Tableau 2 : Amorces et sondes utilisées pour les qPCR ... 25 Tableau 3 : Charge virale plasmatique aux jours d’euthanasie ... 33 Tableau 4 : Corrélation entre CV plasmatique et infection des lymphocytes T CD4 de la rate et des ganglions durant la phase aigüe ... 42 Tableau 5 : Corrélation entre CV plasmatique et infection des sous-populations lymphocytaires T CD4 de la rate et des ganglions durant la phase aigüe ... 42 Tableau 6 : Corrélation entre CV plasmatique et la somme de l’infection des sous-populations lymphocytaires T CD4 de la rate et des ganglions durant la phase chronique ... 47 Tableau 7 : Corrélation entre CV plasmatique et infection des sous-populations lymphocytaires T CD4 de la rate et des ganglions durant la phase chronique ... 47
xi
Liste des figures
Figure 1 : Origine des virus du Sida ... 2
Figure 2 : Prévalence mondiale du Sida en 2013 ... 3
Figure 3 : Structure du virus VIH-1 ... 6
Figure 4 : Organisation génomique du VIH/VIS... 7
Figure 5 : LTR du VIH ... 8
Figure 6 : Étapes du cycle de réplication du VIH ... 11
Figure 7 : Évolution typique de l’infection à VIH ... 12
Figure 8 : Différenciation phénotypique des populations lymphocytaires T CD4 ... 17
Figure 9 : Interaction entre Tfh et cellule B au niveau du centre germinatif ... 17
Figure 10 : Étapes du cycle de réplication virale et les cibles des qPCR ... 21
Figure 11 : Séparation des cellules par la technique des gradients de ficoll ... 27
Figure 12 : Stratégie de tri cellulaire ... 28
Figure 13 : Schéma expérimental ... 29
Figure 14 : qPCR effectuées sur des CEMx174 infectées par le VIS ... 32
Figure 15 : Évolution de la charge virale plasmatique chez 4 individus ayant présenté un profil d’évolution distinct ... 34
Figure 16 : Étapes de pré-intégration (rétrotranscrits précoces et tardifs), de l’ADN et de l’ARN viral dans les sous-populations lymphocytaires T CD4 de la rate durant la phase aigüe ... 36
Figure 17 : Schéma récapitulatif de l’infection des sous-populations lymphocytaires T CD4 au niveau de la rate durant la phase aigüe ... 37
Figure 18 : Étapes de pré-intégration (rétrotranscrits précoces et tardifs), de l’ADN et er de l’ARN viral dans les sous-populations lymphocytaires T CD4 des ganglions durant la phase aigüe ... 38
Figure 19 : Schéma récapitulatif de l’infection des sous-populations lymphocytaires T CD4 au niveau des ganglions durant la phase aigüe ... 39
Figure 20 : Moyenne des rétrotranscrits précoces et tardifs, de l’ADN et de l’ARN viral dans les sous-populations lymphocytaires T CD4 de la rate et des ganglions durant la phase aigüe ... 40
Figure 21 : Étapes de pré-intégration (rétrotranscrits précoces et tardifs), de l’ADN et de l’ARN viral dans les sous-populations lymphocytaires T CD4 de la rate durant la phase chronique ... 43
Figure 22 : Étapes de pré-intégration (rétrotranscrits précoces et tardifs), de l’ADN et de l’ARN viral dans les sous-populations lymphocytaires T CD4 des ganglions durant la phase chronique ... 45
Figure 23 : Moyenne des rétrotranscrits précoces et tardifs, de l’ADN et de l’ARN viral dans les sous-populations lymphocytaires T CD4 de la rate et des ganglions durant la phase chronique ... 46
Figure 24 : Activation in vitro des sous-populations lymphocytaires T CD4 ... 48
xiii
Liste des abréviations
ADN : Acide désoxyribonucléique AID : Animal infectious dose
APOBEC3G : Apolipoprotein B mRNA-editing enzyme, catalytic polypeptide-like 3G ARN : Acide ribonucléique
ATP : Adénosine triphosphate BCL-6 : B-Cell Lymphoma 6
Blimp-1 : B lymphocyte-induced maturation protein-1 BST-2 : Bone marrow stromal antigen 2
CA : Protéine de la capside CCR : C-C chemokine receptor CD : Clusters of differentiation ChemR23 : Chemerin Receptor 23
CMH : Complexe majeur d'histocompatibilité CO2 : Dioxyde de carbone
Ct : Threshold cycle CV : Charge virale
CXCL13 : C-X-C motif chemokine 13 CXCR4 : Chemokine receptor type 4 DCs : Dendritic cells
DC-SIGN : Dendritic Cell-Specific Intercellular adhesion molecule-3-Grabbing Non-integrin FBS : Fetal bovine serum
FOXO-1 : Forkhead box protein O1 Gp : glycoprotéine
GPR15 : G Protein-Coupled Receptor 15 HLA : Human leukocyte antigen
ICAM : InterCellular Adhesion Molecule ICOS : Inducible T-cell COStimulator IFN : Interferon
IL : Interleukine IN : Intégrase virale
KIF5B : Kinesin Family Member 5B KLF2 : Krüppel-like Factor 2
LFA : Lymphocyte function-associated antigen LT : Lymphocyte T
LTNP : Long-term nonprogressors LTR : Long terminal repeat MA : Protéine de la matrice NC : Nucléocapside
Nef : Negative enhancer factor NUP153 : Nucleoporin 153 PNH : Primate non humain PR : Protéase virale
PVVIH : Personne vivant avec le VIH
qPCR : Quantitative polymerase chain reaction Rev : Regulator of viral expression
RMs : Rhesus macaques RT : Reverse transcriptase
xiv
RT-qPCR : Quantitative reverse transcription PCR rRNA : Ribosomal ribonucleic acid
SAMHD1 : SAM domain and HD domain 1 SEM : Standard Error of the Mean
SIDA : Syndrome d'immunodéficience acquise
SIV (ou VIS) : Simian Immunodeficiency Virus (ou virus d’immunodéficience simienne) SRV-1 : Rétrovirus de type D
STLV-1 : Simian T Leukemia Virus de type-1
STRL33 : Seven transmembrane receptor-like from clone 33 TAR : Transactivating region
Tat : Transactivator of transcription TCM : Lymphocyte T central mémoire TDT : Lymphocyte T différencié terminal TEM : Lymphocyte T effecteur mémoire Tfh : T Follicular helper
TFG-β: Transforming growth factor beta Th1 : T helper cell type 1
TNF : Tumor necrosis factor TNP03 : Transportin 3
TRIM5α : Tripartite motif protein 5α tRNALys :Lysine transfer RNA Vif : Viral infectivity factor
VIH : Virus de l’Immunodéficience Humaine Vpu : Viral protein U
Vpx : Viral protein X Vpr : Viral protein R
xv
Remerciements
Je remercie le bon Dieu tout puissant qui m’a donné la force, le courage et la santé afin que je puisse accomplir dans la sérénité mes travaux de recherche.
Je tiens à remercier mon directeur de recherche, le Pr Jérôme Estaquier, qui m’a accepté dans son équipe de recherche. Merci pour vos conseils et vos encadrements. Vous m’avez permis de participer à ce projet qui me passionne énormément. Votre détermination et votre rigueur nous inspirent !
Je remercie le Pr Jacques Corbeil et le Pr Fawzi Aoudjit d’avoir accepté d’être membre du jury de mon mémoire.
Merci à mes anciens professeurs et encadreurs de stage à Madagascar et à l’Ile de La Réunion, je cite spécialement le Pr Rasamindrakotroka, le Pr Rapelanoro Rabenja, le Pr Rakotozandrindrainy, le Pr Rakoto Alson, le Dr Michault et le Dr Carod, qui m’ont formé et m’ont initié à la recherche et qui ne cessent de me soutenir dans mes formations universitaires.
Un grand merci à mon très cher mari Aina qui me soutient toujours et m’encourage à en faire davantage. Mais surtout, je dédie ce mémoire à ma petite fille adorée Antso pour sa patience et son amour, et qui reste des fois éveillée pour soutenir maman dans ses différentes occupations.
Merci à mes parents dadabe Rabezanahary et bebe Blandine, qui, malgré les éloignements, m’ont toujours soutenu durant ce long cursus.
A mon frère Tsiry, à mes sœurs Laingo et Salohy ainsi qu’à leurs époux (se), à tous mes neveux et nièces que j’aime tant et qui me manque des fois, je vous remercie car vous prenez une grande part dans ma motivation. Merci à ma belle-famille pour vos soutiens et vos conseils.
Sans oublier toute l’équipe du laboratoire, Lynda Robitaille, Gina Racine (belle Gigi), Guadalupé Andréani et mon ami Félicien Moukambi qui font partie de ma grande famille ici à Québec, avec qui je partage les journées d’euthanasie de singes, mes joies et ma réussite. Merci beaucoup à vous !
xvi
1
Chapitre 1 : Introduction
1.1 Epidémiologie du Sida
Le SIDA ou Syndrome d’Immunodéficience Acquise est un ensemble de symptômes associé au déficit immunitaire. Il est lié au lentivirus appelé VIH ou Virus de l’Immunodéficience Humaine, qui cible les cellules exprimant la molécule de surface CD4 comme les lymphocytes T CD4 et les macrophages. Le Sida se manifeste sous forme d’une lymphadénopathie généralisée. La phase ultime est la survenue des infections opportunistes qui est souvent responsable du décès du patient [1].
Le VIH appartient au genre lentivirus des Retroviridae. Il regroupe deux sous-types de virus, le 1 et le VIH-2. Le VIH-1 est composé par 4 groupes, M, O, N et P. Le groupe M cause le plus d’infection dans le monde. Le VIH-2 est composé de deux groupes, VIH-2A et VIH-2B [2].
Le transfert du virus de l’immunodéficience simienne appelé VIS à partir du primate non humain, hôte naturel au virus, chez l’Homme par anthopozoonose a été proposé [3]. D’autres souches de SIV ont été isolées ultérieurement chez les singes d’Afrique comme le Mandrill au Gabon [4]. Des analyses épidémiologiques et phylogénétiques ont mis en exergue que l’origine des souches VIH-1 et VIH-2/SIVmac résulte de multiples transmissions inter-espèce des souches de VIS. Les groupes M et N du VIH-1 proviennent des chimpanzés (SIVcpz) et les groupes P et O sont issus des gorilles (SIVgor) d’Afrique central. Le VIH-2 est issu du SIVsmm des Sooty mangabey de l’Afrique de l’ouest [2, 5-8] (Figure 1). Bien que le VIS (ou SIV en anglais) n’entraine pas le Sida chez son hôte naturel, son transfert chez un hôte non permissif comme le macaque (singe vivant en Asie) entraine chez ces derniers une maladie similaire à celle observée chez l’Homme au cours du Sida.
Les premiers cas de Sida chez l’Homme ont été rapportés aux Etats-Unis en Juin 1981 chez des homosexuels, souffrant d’une pneumonie à Pneumocystis carnii [9]. Deux ans plus tard, le VIH a été découvert par les équipes de l’Institut Pasteur [10]. D’autres épidémies se sont succédées par la suite. Actuellement, le Sida est devenu une pandémie à échelle mondiale. Des analyses rétrospectives ont été effectuées sur des échantillons humains de patients présentant le même syndrome afin de déterminer la date de début de cette pandémie. Cela a permis d’identifier le plus vieil échantillon connu positif au VIH qui date de 1959 [11]. Une approche statistique sur des séquences de VIH-1 en provenance d’Afrique centrale a montré que la souche à l’origine de l’épidémie du Sida provient de Kinshasa et date approximativement de 1920 [12].
2
Figure 1 : Origine des virus du Sida
Figure tirée de Sharp et al., 2011 [2]
Selon les données de l’ONUSIDA en 2013, 35 millions de patients vivaient avec le VIH (PVVIH). Trente-sept pour cent d’entre-deux étaient sous thérapie antirétrovirale. Les maladies liées au SIDA causent près de 2 millions de décès par année. La tuberculose en est la principale cause. Néanmoins, le taux de mortalité a diminué de 35% depuis 2005. Le nombre de personnes nouvellement infectées a également diminué de 38% depuis 2001 [13].
La répartition géographique de l’infection est très variable (Figure 2). Près de 70% des PVVIH vivent en Afrique Subsaharienne [14]. Les femmes sont majoritairement infectées. Les nouveaux cas sont estimés de 1-2 millions par année, dont 1-210000 cas chez les enfants. Au Moyen-Orient et en Afrique du Nord, la prévalence de l’infection à VIH est faible. Une augmentation de 7% de nouveaux cas et de 66% de décès liés au Sida ont été observés de 2005 à 2013. En Europe Occidentale-Centrale et en Amérique du Nord, 2.3 millions de PVVIH ont été recensés en 2013. Le taux de mortalité est faible (1.17%) et la couverture thérapeutique est plus élevée (51%) [13]. Au Canada, 71300 de PVVIH étaient recensés en 2011, dont 3175 nouveaux cas. Le
3 nombre annuel de nouveaux cas reste stable durant les dix dernières années. Le taux d’infection est plus élevé chez les Hommes, principalement chez les homosexuels [15].
Il existe différents modes de transmission du VIH. Le virus peut être transmis par voie sexuelle, sanguine ou verticale c’est-à-dire de mère à enfant. La transmission sexuelle est de loin la plus fréquente (80 %) [16]. Elle peut s’effectuer entre homosexuels ou hétérosexuels. La contamination par voie sanguine s’effectue par transfusion sanguine, par utilisation de matériel non ou mal stérilisé, ou bien par réutilisation d’objets souillés comme les seringues, observée fréquemment chez les utilisateurs de drogue injectable [17]. Le virus peut être transmis de mère à enfant in utero, au cours de l’accouchement ou durant l’allaitement. Le risque de transmission est variable selon les régions, allant de 13 à 52% [18]. Il est plus élevé durant un stade avancé de la maladie, surtout lorsque la charge virale (CV) plasmatique est élevée chez la mère [18]. En absence de traitement antirétroviral, 50% des enfants infectés progressent vers le SIDA et peuvent mourir au bout de 2 ans [19]. L’administration précoce d’antirétroviral aux enfants dans les heures suivant l’accouchement limite la morbidité et la mortalité liées au VIH. Néanmoins, elle n’empêche pas la dissémination virale précoce dans différents organes, comme le système nerveux central qui peut être infecté dès le 6ème jour de naissance [20-22].
Figure 2 : Prévalence mondiale du Sida en 2013
4
1.2 Primate non humain et SIDA
1.2.1 PNH-hôtes naturels
Comme mentionné précédemment, il existe plus d’une quarantaine d’espèces de singe naturellement infectées par différentes souches de SIV sur le continent Africain [24, 25].
L’infection des PNH-hôtes naturels par le SIV est caractérisée par l’absence d’évolution vers un Sida [2]. En effet, durant la phase aigüe de l’infection, l’évolution de la CV plasmatique, bien que comparable au hôte non-naturel comme le macaque, n’est pas associée à une déplétion des cellules CD4 ni à une évolution vers un Sida [26-29]. Après quelques semaines d’infection, la CV plasmatique diminue dans le sang périphérique, mais elle reste équivalente entre les deux groupes d’animaux [28, 30, 31]. Par contre, quelques différences ont été observées au niveau tissulaire. La CV tissulaire est rapidement contrôlée chez les hôtes naturels contrairement aux macaques [30]. On note un faible niveau d’apoptose, une faible expression des marqueurs d’activation et de prolifération des LT CD4 [32, 33], ce qui maintien stable la population lymphocytaire T CD4 circulante [24, 34, 35]. Le niveau de l’apoptose des polynucléaires neutrophiles reste stable, la morphologie des ganglions est conservée et le niveau d’inflammation est faible [30].
1.2.2 PNH-hôtes non naturels
À la différence des singes d’Afrique, les singes vivants en Asie, tel l’Inde où la Chine, sont indemnes de tout lentivirus. Toutefois, le transfert de VIS d’un singe africain chez un hôte non naturel, comme le macaque, conduit à une infection similaire à celle observée chez l’Homme au cours du Sida.
Il existe plusieurs espèces de macaque connues comme le macaque Rhésus ou RMs (Macaca mulatta), le cynomolgus (Macaca fascicularis) ou encore le pigtail (Macaca nemestrina) [36]. Le niveau de sensibilité à l'infection ainsi que la gravité de la maladie dépendent de l’espèce de macaque infecté et du type de virus utilisé.
L’infection des macaques d’origine Indienne présente toutefois quelques différences comparées à celle observée chez l’Homme au cours du Sida. La CV plasmatique est plus élevée, le niveau d’apoptose au niveau tissulaire est plus élevé et le délai de progression vers un Sida est beaucoup plus court [33]. On note une déplétion rapide et soutenue des lymphocytes T mémoires CD4+ CCR5+, constituant chez eux un bon marqueur prédictif de l’évolution de la maladie [37], différemment de ce qu’on observe chez l’Homme au cours
5 de l’infection à VIH, et chez les RMs d’origine chinoise infectés par le SIVmac [38, 39]. Chez ces derniers, ceux sont les niveaux d’activation du système immunitaire et de l’apoptose qui correspondent positivement à la progression de la maladie [40]. La dynamique virale, la réponse immune et la dynamique lymphocytaire observées chez l’Homme au cours d’une infection à VIH, est plus proche de celle observée chez les macaques Rhésus ce qui en fait un bon modèle d’étude [39, 41].
Les macaques cynomolgus peuvent être également infectés par le SIVmac [25]. Avec la même souche, la virémie et la pathogénicité sont moins importantes chez les cynomolgus [36, 42]. L'infection des RMs avec un isolat de SIVsmm conduit généralement au Sida [43]. Les macaques à queue de cochon (Pigtail) peuvent succomber rapidement d’un Sida suite à l'infection par le SIVsm et le SIVmac, et par certaines souches de SIVagm [44].
1.3 Infection à VIH
1.3.1 Virus du Sida
1.3.1.1 Structure du virus
Une particule virale mature se présente sous forme sphérique, d’environ 120 nm de diamètre [45]. Elle est enveloppée par une bicouche lipidique provenant de la membrane plasmique de la cellule hôte, que le virus acquiert durant le bourgeonnement. L’enveloppe virale est parsemée par des glycoprotéines virales, les gp120 qui sont liées aux protéines transmembranaires gp41 (Figure 3). Elle contient également d’autres protéines provenant de la cellule hôte comme le complexe majeur d’histocompatibilité (CMH), l’ICAM-1 et la LFA-1 [46, 47]. La face interne de l’enveloppe est tapissée par la matrice, constituée par 2000 copies de protéines de la matrice MA (p17). Elle contient la capside, de forme conique, composée par 2000 copies de protéine de la capside CA (p24). La capside contient à son tour 2 copies de génome viral, non épissé, stabilisé avec la protéine CA pour former le complexe ribonucléoprotéique. Elle contient également d’autres protéines essentielles à la réplication virale telle que la protéase (PR), la reverse transcriptase (RT), l’intégrase (IN) et les protéines accessoires Nef (Negative enhancer factor), Vif (Viral infectivity factor) et Vpr (Viral protein R) [48].
6
Figure 3 : Structure du virus VIH-1
Figure tirée de Gemrot et al., 2009 [49]
Le génome viral est constitué par deux ARN simple brin, de polarité positive. Il est composé par 9200 nucléotides, regroupant neuf gènes codant pour 15 protéines virales [50]. Les trois gènes majeurs dont gag,
pol et env codent pour les protéines structurales (MA, CA et la nucléocapside p6), pour les enzymes (PR, RT,
IN) et pour les protéines d’enveloppe (gp120, gp41). Suite à des épissages alternatifs, le reste du génome code pour deux groupes de protéines dont les protéines régulatrices Tat (Transactivator of transcription) et Rev (Regulator of viral expression) ; et les protéines accessoires Nef, Vif, Vpr et Vpu (Viral protein U), qui sont nécessaires pour la réplication virale et la pathogenèse [51] (Tableau 1). On note quand même une petite différence entre l’organisation génomique de certaines souches de VIH et VIS. Les souches VIH-2, SIVsm et SIVmac possèdent un gène supplémentaire appelé Vpx (Viral protein X) à la place du Vpu (Figure 4). Quel que soit la souche, le génome viral est délimité à chaque extrémité, par des séquences régulatrices non traduites appelées LTR ou « long terminal repeat » [50] (figure 4). Les séquences LTR contrôlent l’expression du génome viral. Elles interagissent avec les facteurs de transcription virale et cellulaire [52]. Chaque LTR est subdivisée en 3 régions dont U3, R et U5. La région U3 contient la plupart des promoteurs et des modulateurs de la transcription. La transcription débute au niveau de la région R où se trouve la séquence transactivatrice TAR. La région U5 se trouve entre R et gag. Elle n’a pas de fonction spécifique [52] (Figure 5).
7
Nom Fonction
p24 Protéine de la capside, fonction structurale
p17 Protéine de lamareice, fonction structurale
p7 Protéine de la nucléocapside, régule la condensation et l'encapsidation du génome viral
p6 Régulation du bourgeonnement
Transcriptase inverse Production d'ADN double-brin à partir du génome viral
Intégrase Insertion de l'ADN viral dans l'ADN chromosomique de la cellule cible
Protéase Clivage des polyprotéines Pr55Gag et Pr160Gag-Pol pour former le virion mature
gp120 Attachement viral
Liaison au récepteur et au co-récepteur
gp41 Peptide de fusion
Tat Augmente la transcription des gènes du VIH
Rev Export des ARNm non épissés
Nef Régulation négative du CD4 et du CMH-I de la surface cellulaire
Augmentation de l'infectivité virale Inhibition de la commutation de classe Effet cytopathique interne
Vpu Agit contre le facteur de restriction BST-2 (Tétherine)
Vif Agit contre le facteur de restriction APOBEC3G
Vpr Arrêt du cycle cellulaire en G1
Import nucléaire du complexe de pré-intégration
Vpx Agit contre le facteur de restriction SAMHD1
Tableau 1 : Fonction des protéines virales
Tableau modifié à partir de Gobeil et al., 2012 [53]
Figure modifiée à partir de Ayinde et al., 2010 et Beer et al., 2010 [54, 55]
8
Figure 5 : LTR du VIH
Figure tirée du web-books.com [56]
1.3.1.2 Etapes du cycle de réplication virale
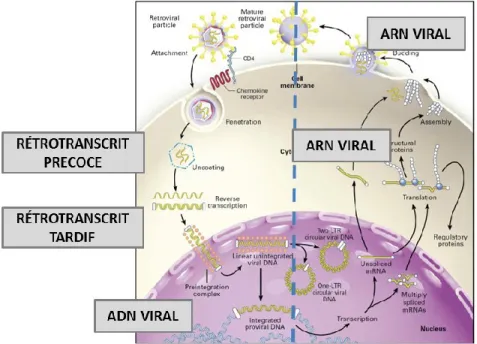
Pour se répliquer, le virus a besoin de la machinerie de la cellule hôte. Les cellules exprimant la molécule de surface CD4, comme les lymphocytes T CD4 et les macrophages constituent les principales cibles du VIH et du VIS, dans lesquels ils peuvent se répliquer ou rester en latence. Le cycle de réplication virale peut être subdivisé en deux grandes étapes : les étapes précoces qui correspondent à l’entrée du virus jusqu’à l’intégration de l’ADN viral dans le génome de la cellule hôte et les étapes tardives qui correspondent aux étapes de réplication virale proprement dite aboutissant à la production virale (Figure 6).
1.3.1.2.1 Étapes précoces
Entrée du virus : Le virus se fixe sur la molécule de surface CD4 de la cellule hôte via sa glycoprotéine d’enveloppe gp120. Cette première interaction n’est pas suffisante pour l’entrée du virus, car il faut un corécepteur. Les molécules CCR5 et CXCR4 constituent les principaux corécepteurs utilisés par le VIH. Le VIS n’utilise pas le CXCR4. D’autres corécepteurs peuvent être utilisés comme le CCR1, CCR2B, CCR3, CCR4, CCR8, CCR9, GPR1, GPR15, STRL33, V28, US28, ChemR23 et ApJ [57]. La liaison au corécepteur permet un changement de conformation du complexe protéique au niveau de l’enveloppe virale permettant l’exposition de la gp41, la fusion des membranes virales et cellulaires, et la libération du complexe nucléoprotéique viral qui est encapsidé dans le cytoplasme cellulaire [58].
9 Décapsidation : Cette étape correspond à la déstructuration de la capside et à la libération du complexe nucléoprotéique dans le cytoplasme cellulaire. Le déroulement de la décapsidation n’est pas encore bien décrit. Certaines protéines cytoplasmiques comme la dynéine et la kinesine KIF5B semblent faciliter cette étape [59]. Par contre, d’autres facteurs comme TRIM5α l’inhibent [60]. Le TRIM5α est un facteur de restriction de la réplication virale qui se trouve dans le cytoplasme de la cellule hôte. L’interaction directe du TRIM5α avec la capside virale favorise son ubiquitination et sa dégradation par le protéasome. Ce mécanisme pourrait être à l’origine de la perturbation structurale de la capside virale aboutissant à une décapsidation aberrante [61]. Chez les primates non humains, TRIM5α agit comme médiateur spécifique d’espèce permettant la résistance cellulaire innée au VIH, en modulant l’étape précoce de la décapsidation [62].
Rétrotranscription : Elle se déroule dans le cytosol sous l’action de la RT. L’ARN viral est rétrotranscrit, générant dans un premier temps un brin court d’ADN, appelé rétrotranscrit précoce (strong stop fragment), qui présente une extrémité 5’ libre permettant sa détection. Cette première étape est initiée par l’amorce tRNALys cellulaire et facilitée par la protéine accessoire vif et la protéine de la nucléocapside [48]. La seconde étape permet la synthèse d’un bout beaucoup plus long, incluant le rétrotranscrit précoce et une partie du gène gag, permettant ainsi une détection des rétrotranscrits tardifs (LTR-Gag fragment). Le tout aboutit à la synthèse d’ADN bicaténaire, délimité à chaque extrémité par un LTR. La rétrotranscription peut être inhibée par les facteurs de restriction cellulaire comme APOBEC3G et SAMDH1, mais qui peuvent être contrecarrés par les protéines virales accessoires, en particulier Vpx et Vif [63-65].
Import nucléaire : L’ADN bicaténaire nouvellement formé est transporté dans le noyau sous forme de complexe de préintegration, composé d’ADN bicaténaire avec diverses protéines comme l’IN, la MA, la RT, la vpr et la protéine HMG-I(Y) (high-mobility group DNA-binding protein) de la cellule hôte [48]. Différents facteurs semblent impliqués dans cette étape, comme l’interaction de l’IN avec les importines α/β et 7, l’import nucléaire à travers une voie nécessitant de l’ATP (Adénosine triphosphate), l’interaction entre MA et les protéines accessoires Vpr et Vpx, la participation de l’ADN viral appelé « central DNA flap » formé lors de la rétrotranscription et l’implication d’autres protéines d’import nucléaire comme la transportine 3 (TNPO3) et la nucléoporine NUP153 [66, 67].
Intégration : Suite à l’import nucléaire et sous l’activité catalytique de l’IN virale, l’ADN viral peut s’intégrer dans le génome de la cellule hôte. A partir de là, il est appelé provirus ou ADN proviral. Cette étape dépend généralement de l’état d’activation cellulaire et s’effectue au niveau des unités transcriptionnelles actives. Néanmoins, il a été rapporté que l’intégration d’ADN viral peut s’effectuer même chez les cellules naïves, mais avec une efficacité moindre [68]. Les sites d’intégration sont différents entre les cellules quiescentes et
10
activées [69]. Cette sélection au niveau du site d’intégration pourrait jouer un rôle dans la pathogénicité virale [70].
Le cycle de réplication peut s’arrêter à ce stade chez les cellules quiescentes. Il faut une réactivation cellulaire ultérieure pour qu’il y ait poursuite du cycle de réplication aux étapes tardives.
Une partie de l’ADN non intégré peut rester dans le noyau sous forme linéaire ou circulaire avec une ou deux LTR. Ces formes d’ADN non intégré pourraient contribuer au développement de la réinfection cellulaire [71].
1.3.1.2.2 Étapes tardives
Transcription et traduction : L’ADN proviral sert de matrice pour la synthèse d’ARNm, permettant la synthèse de nouveaux virions. Suite à une activation cellulaire via le complexe CD3 et la molécule coactivatrice CD28, la transcription de l’ADN proviral est initiée au niveau des séquences LTR par l’ARN polymérase II cellulaire. Dans un premier, elle va générer un court ARNm multi-épissé, qui va être traduit en protéines régulatrices Tat, Rev et Nef. La protéine Tat va activer la transcription de l’ADN proviral. Cela va générer un ARNm non épissé et partiellement épissé dont l’export nucléaire est facilité par la protéine Rev. Les ARNm non épissés vont servir d’une part de précurseur pour les polyprotéines Pr55Gag et Pr160Gag-Pol, et d’autre part, ils vont former le génome des nouveaux virions. Les ARNm partiellement épissés vont coder pour les protéines Vif, Vpu/Vpx, Vpr et pour la glycoprotéine précurseur Env (gp160). La gp160 va être clivée par la protéase cellulaire pour former les gp d’enveloppe gp120 et gp41, qui seront exportés à la membrane cellulaire [72]. La protéine Nef favorise la régulation négative de l’expression de CD4 et de CMH-I à la surface cellulaire [73], l’apoptose des cellules [74], ainsi que le déficit immunitaire et la progression de la maladie [75-77]. Assemblage, bourgeonnement et libération : Les polyprotéines, précurseurs de Gag et de Gag-Pol, vont s’assembler avec les copies de génome viral non épissé et les protéines accessoires, puis bourgeonner à la surface cellulaire pour former de nouveaux virions. Cette libération de particules virales est favorisée par la protéine Vpu. Les enzymes PR, RT et IN sont synthétisées à partir de la Pr160Gag-Pol. La Pr55Gag va être clivée par la PR virale pour former les protéines de structure MA, CA et NC. Cette étape de clivage est cruciale pour les nouveaux virions car elle permet la production de particules virales matures et infectieuses qui ne représentent qu’une partie des virions produits. Les virus nouvellement formés, libérés par la cellule peuvent infecter à nouveau d’autres cellules. Cela permet la continuité du cycle et la dissémination virale [72].
11
Figure 6 : Étapes du cycle de réplication du VIH
Figure modifiée à partir de Pasternak et al., 2013 [78]
1.3.2 Evolution de la pathogenèse
L’infection à VIH se déroule en deux grandes phases. La phase aigüe ou primo-infection, correspondant aux premiers mois de l’infection, puis la phase chronique qui se termine au stade de SIDA maladie.
Au cours de la primo-infection, les organes lymphoïdes comme la rate et les ganglions sont infectés dès les premiers jours de l’infection [79]. Le cerveau est infecté au bout d’une semaine [80, 81]. Un pic de CV plasmatique s’observe au bout de deux semaines d’infection. Elle peut atteindre jusqu’à des millions de copies d’ARN viral par millilitre de plasma. Cette augmentation de la CV plasmatique correspond à l’augmentation de l’infection et de l’apoptose au sein des tissus lymphoïdes qui atteint un niveau maximal au moment du pic de réplication virale. Le niveau d’apoptose au niveau des ganglions durant la phase précoce de l’infection détermine la progression ultérieure vers un Sida [33, 40, 82]. On observe une baisse du taux des lymphocytes T CD4 dans le sang périphérique, mais également au niveau des tissus muqueux et lymphoïdes [37]. La CV plasmatique est inversement proportionnelle au taux de LT CD4 dans le sang périphérique [83-85](Figure 9). Durant ce stade, on observe une augmentation de la sécrétion de certaines cytokines pro-inflammatoires comme l’IFN-α, l’IL-15, le TNF-α, l’IFN-γ et l’IL-12. Elle favorise l’amplification de la réplication virale et la dissémination du virus dans différents compartiments de l’organisme. La sécrétion d’IL-10 qui est un immunomodulateur est également augmentée. Cela pourrait favoriser l’établissement de la latence dans certaines cellules mémoires. Afin de contrôler la progression de l’infection, les lymphocytes T cytotoxiques CD8 se multiplient rapidement permettant la réduction de la CV plasmatique [86]. Malgré l’activation intense
12
des LT CD8 et l’augmentation de l’expression en CCR5, leur conférant une fonction effectrice optimale, celle-ci n’arrive pas à éradiquer le virus [87, 88]. Durant la primo-infection, le niveau d’activation des LT CD8 est plutôt associé à un mauvais pronostic d’évolution vers un Sida [87].
Au niveau de la réponse humorale, on observe une dérégulation physiologique et fonctionnelle des lymphocytes B. L’hyperactivation du système immunitaire favorise l’activation polyclonale des cellules B, l’hypergammaglobulinémie, la production d’autoanticorps et l’augmentation de la fréquence des lymphocytes B immatures circulants [89]. On observe aussi une perte des cellules B mémoires. Bien que la production d’anticorps spécifiques s’effectue au bout de quelques semaines, leur efficacité à neutraliser le virus est médiocre. Ainsi, chez la plupart des individus infectés par le VIH, nombreux sont les anticorps qui sont non spécifiques et non protecteurs [90]. La réponse anticorps à IgA spécifiques est faible au niveau des muqueuses, sites importantes de réplication virale. Cela permettrait la dissémination précoce du virus [89]. Un point d’équilibre virologique et un contrôle immunologique s’effectuent rapidement correspondant à la descente de la CV plasmatique et à la remontée du taux les LT CD4 [91]. Néanmoins, les dysfonctionnements des lymphocytes B peuvent persister jusqu’à la phase chronique, aboutissant à leurs épuisements [89, 92]. La primo-infection peut être asymptomatique. Sinon, elle peut se manifester sous forme de fièvre associée à une polyadénopathie, à des manifestations digestives et cutanéomuqueuses [93]. Des manifestations neurologiques peuvent être présentes chez 10 à 15 % des patients symptomatiques [94].
Figure 7 : Évolution typique de l’infection à VIH
13 Durant la phase chronique, le virus continue à se répliquer. Selon l’évolution typique de l’infection, la CV plasmatique peut rester stable durant plusieurs années, avec un taux plasmatique de LT CD4 supérieur à 500 LT CD4 / mm3. Le patient peut rester longtemps asymptomatique pendant la période dite de latence clinique, durant laquelle, les lymphocytes T CD8 sont toujours activés dans le sang et dans les ganglions. Bien que le taux des lymphocytes T CD4 activés reste stable, le taux des lymphocytes T CD8 activés augmente proportionnellement à la CV plasmatique [96].
L’immunodéficience peut se développer après plusieurs années d’évolution, malgré l'absence de manifestation clinique, qui peut aller de 3 à 20 ans [97]. Pendant la période précédant le Sida, le taux des lymphocytes T CD4 activés dans les ganglions augmente, différemment de ce qu’on observe dans le sang périphérique, pouvant conduire à un épuisement du système immunitaire [96]. L’état d’immunodéficience correspondant à une déplétion des LT CD4, avec un taux plasmatique inférieur à 200 LT CD4/mm3, qui est le plus souvent associée à une charge virale plasmatique élevée et une déstructuration des ganglions. Cet état rend les patients plus susceptibles aux infections opportunistes comme les mycoses, différentes pneumopathies bactériennes, entre autres qui pourraient causer leur mort. Des manifestations neurologiques sont également observées [94].
L’infection peut évoluer de différentes façons. Pour certains patients appelés « progresseurs rapides », le Sida peut survenir rapidement causant le décès au bout de 3 à 5 ans d’évolution. Un faible pourcentage de patients, appelés les « progresseurs à long terme » (long-term non-progressors ou LTNP), représentés par 2 à 5% des séropositifs, peuvent contrôler l’infection pendant des années en absence d’antirétroviraux. Ils sont souvent asymptomatiques pendant 10 à 20 ans d’infection à VIH, les taux des LT CD4 et LT CD8 restent supérieurs à 500 cellules/mm3, avec une CV plasmatique en dessous de 10000 copies/ml. Une sous-population des LTNP, appelés « elites controleurs », ont un taux plasmatique de LT CD4 supérieur à 500 cellules/mm3, mais la CV plasmatique peut être indétectable, c’est-à-dire inférieure à 50 copies par ml pendant des mois ou même des années durant l’évolution de leur maladie. Certains patients appelés « progresseurs lents », ont un taux plasmatique de LT CD4 inférieur à 500 cellules/mm3, mais avec une CV plasmatique légèrement supérieure à celle des LTNP [98].
Des études effectuées chez les macaques ont montré que les LT CD8 des animaux progresseurs expriment fortement du TGF-β (Transforming growth factor beta). Cette hyperexpression est retrouvée particulièrement au niveau des tissus intestinaux [88]. L’étude génotypique sur les allèles du complexe majeur d’histocompatibilité (CMH) a permis de montrer que les singes ayant l’allèle Mamu-A*01, infectés par le SIVmac251 sont capables de contrôler la réplication virale, et de préserver de façon significative le taux des LT CD4 [99, 100].
14
D’autres études effectuées chez l’Homme ont montré que l’allèle HLA-B57 est retrouvé chez la plupart des patients LTNP, permettant une bonne présentation des peptides antigéniques à la surface des lymphocytes T CD4 aux lymphocytes T cytotoxiques [98]. Une résistance totale ou partielle à l’infection est également observée chez les individus porteurs du gène CCR5-∆32, qui est une mutation du gène CCR5, ne permettant plus l’expression du récepteur CCR5 à la surface cellulaire [101-103].
1.4 Cellules cibles
Comme mentionné précédemment, le VIH et le VIS ciblent principalement les cellules exprimant la molécule de surface CD4, regroupant les cellules de la lignée myéloïde et lymphoïde.
1.4.1 Cellules de la lignée myéloïde
La lignée myéloïde regroupe différents types cellulaires. Parmi lesquels, les cellules dendritiques (DCs), les macrophages et les monocytes exprimant la molécule de surface CD4 sont les cibles du virus [104].
Les DCs constituent une des premières cibles du virus lors de l’infection mucosale. Elles véhiculent le VIH jusqu’au niveau des organes lymphoïdes secondaires, où elles interagissent avec les lymphocytes T CD4 (LT CD4). Durant cette migration, les DCs immatures subissent une étape de maturation permettant la présentation antigénique et la transmission efficace du virus. La transmission du virus par les DCs s’effectue de deux façons, par une trans-infection ou par une cis-infection. Lors de la trans-infection, il s’effectue une synapse infectieuse entre les deux types cellulaires via la DC-SIGN (Dendritic Cell-Specific Intercellular adhesion molecule-3-Grabbing Non-integrin), facilitant la transmission directe du virus. La transmission virale peut s’effectuer par la cis-infection, au cours de laquelle, les nouveaux virus produits par les DCs sont transmis aux LT CD4 à leur surface. Avec ce mode de transmission, les DCs peuvent produire du virus à long terme [105].
La réplication virale au sein des DCs est moins productive, comparée à celle dans les LT CD4. Une étude effectuée sur les cellules myéloïdes montre que seulement 1 à 3 % des DCs sont capables de produire du virus in vitro. La fréquence de l’infection des DCs est 10 à 100 fois moins que celle des LT CD4 [106].
15 Les monocytes et les macrophages font partie des cibles du virus durant la primo-infection. Au niveau du cerveau, elles sont infectées au bout d’une semaine d’inoculation [81]. Les monocytes circulant peuvent être infectés de façon latente et une infection productive peut être initiée durant leur différenciation en macrophage [107, 108]. La demi-vie de l’ADN viral dans les monocytes et les macrophages est beaucoup plus longue que celle dans les LT CD4 quiescents, suggérant leur importance dans la dissémination virale [109].
1.4.2 Cellules de la lignée lymphoïde
Les LT CD4 jouent un rôle central dans l’orchestration de la réponse immune adaptative. Ils sont parmi les cibles précoces du virus au cours de l’infection. Ils constituent les sites majeurs de réplication et de dissémination virale [110]. Les LT CD4 ont une capacité plus élevée de production virale comparés aux cellules de la lignée myéloïde [106]. Il a été montré que la CV plasmatique durant la primo-infection à VIH corrèle avec la fréquence des infections productives des lymphocytes T au niveau des organes lymphoïdes [111].
Parallèlement à l’infection des LT CD4, on note leur déplétion qui peut être observée précocement dans le sang périphérique et au niveau des tissus profonds comme la muqueuse intestinale, bien que l’infection directe des cellules ne soit pas le seul facteur qui y est impliqué. La mort cellulaire programmée ou apoptose induite ou non par l’activation du récepteur FAS/CD95 constitue une des principales cause de la déplétion des LT CD4 [112-115]. C’est un processus physiologique d’autodestruction cellulaire qui peut être dérégulé par le virus au niveau de différentes sous-populations cellulaires [116]. Elle pourrait constituer également un système de défense de l’organisme pour éliminer les cellules infectées [74]. L’apoptose peut toucher d’autres types cellulaires infectés ou non par le VIH, comme les cellules de l’immunité innée à savoir les monocytes, les DCs et les polynucléaires neutrophiles [33, 117, 118]. Au stade terminal de l’infection à VIH, on observe un effondrement des LT CD4 correspondant au déficit immunitaire [119-121].
1.4.2.1 Différenciation des lymphocytes T CD4
Les LT CD4 se développent à partir des thymocytes qui ont achevé le processus de maturation au niveau du thymus. Les cellules matures dites naïves quittent le thymus, au stade G0 (au repos) du cycle cellulaire. Elles suivent la circulation sanguine et lymphatique à la recherche d’antigène spécifique. Elles expriment fortement le « homing receptor » CD62L et le récepteur aux chimiokines CCR7 (C-C chemokine receptor type 7),
16
permettant leur migration au niveau des organes lymphoïdes secondaires, où s’effectue la présentation antigénique [122].
Suite à la présentation antigénique, les cellules naïves se différencient au cours du temps en centrale mémoire (TCM), effectrice mémoire (TEM) et différenciée terminale (TDT). Ces populations lymphocytaires sont identifiées sur la base de l’expression de différentes molécules de surface. Les cellules naïves et les TDT expriment la molécule CD45RA, contrairement aux cellules mémoires. À la différence des cellules naïves et des TCM qui expriment la molécule CD62L, les cellules TEM n’expriment ni CCR7 ni CD62L, ce qui leur permet de s’accumuler dans les tissus périphériques, où elles peuvent agir immédiatement en présence d’antigène. Comme les TCM expriment fortement le CCR7, elles se trouvent principalement dans la zone T des organes lymphoïdes secondaires, où elles sont prêtes à proliférer et à se différencier en TEM en réponse à une stimulation antigénique [122, 123].
En pratique, ces différents marqueurs de surface sont utilisés pour différencier les sous-populations lymphocytaires par cytométrie en flux. Cependant, il existe une sous-population lymphocytaire T CD4 appelée Tfh (T follicular helper), qui exprime fortement les molécules de surface PD1 (programmed cell death 1) et CXCR5 (chemokine C-X-C motif receptor 5) (Figure 7). Le CXCR5 est un récepteur pour la chimiokine CXCL13 (C-X-C motif chemokine 13). Son expression permet la migration cellulaire dans la zone folliculaire B des organes lymphoïdes secondaires et la formation de centres germinatifs (GC) [124-127]. Les cellules Tfh sont des cellules particulières car elles fournissent de l’aide aux lymphocytes B. Elles secrètent une quantité abondante d’IL-21 qui est nécessaire à la différenciation et la prolifération des cellules B, ainsi que pour la commutation de classe et l’hypermutation somatique des immunoglobulines [128]. Tout cela permet de générer des plasmocytes et des cellules B mémoires producteurs d’anticorps de haute affinité [125]. Les cellules Tfh sont également caractérisées par une forte expression d’ICOS (inducible co-stimulator) et de BCL-6 (B-Cell Lymphoma BCL-6). L’ICOS est un corécepteur puissant induit au niveau des cellules T activées. Sa stimulation permet l’inactivation du facteur de transcription FOXO-1 (Forkhead box protein O1), qui est un régulateur négatif de la BCL-6. L’activation du facteur de transcription BCL-6 permet à son tour la différenciation des cellules T activées en Tfh et au développement du GC [129-131]. Le maintien des cellules Tfh au sein des GC et leur différenciation terminale nécessitent la stimulation via le ligand ICOSL par les lymphocytes B [132] (Figure 8). D’autres facteurs de transcription peuvent inhiber la différenciation des lymphocytes T activés en Tfh, comme le facteur de transcription KLF2 (Krüppel-like Factor 2) qui induit l’expression du facteur de transcription Blimp-1 (B lymphocyte-induced maturation protein-1) qui est un répresseur de BCL-6 [133].
17
Figure modifiée à partir de [123]
Figure 9 : Interaction entre Tfh et cellule B au niveau du centre germinatif
Figure tirée de Nutt et al., 2011 [134]
18
1.4.2.2 Infection des sous-populations LT CD4
Parmi les LT CD4, les lymphocytes mémoires sont les cibles préférentielles du VIH et constituent les principaux réservoirs viraux [135-137]. Comparés aux cellules naïves, ils expriment fortement la molécule CCR5, ce qui les rend beaucoup plus susceptibles à l’infection [138-140].
Comme les lymphocytes mémoires sont composés de différentes sous-populations, des études effectuées au niveau du sang périphérique montrent que la population TCM est celle qui présente le taux le plus élevé d’ADN viral [37, 141]. Etant donné que les lymphocytes du sang périphérique ne représentent que 2% de tous les lymphocytes, la dynamique cellulaire observée dans le sang ne reflète pas toujours ce qui se passe réellement dans les organes profonds.
Au sein des tissus lymphoïdes périphériques et profonds, une perte importante des LT CD4 mémoires est observée durant la primo-infection et surtout au moment du pic de la CV plasmatique [37]. Comme ces cellules mémoires sont constituées par différentes sous-populations, quelques études se sont focalisées sur l’infection des différentes sous-populations lymphocytaires T CD4.
Une étude réalisée par Xu Y et al chez des macaques a montré que les cellules Tfh au niveau des ganglions sont infectées au même niveau que les cellules mémoires non Tfh durant les phases aigües et chroniques de l’infection. Dans cette étude, les niveaux d’expression de PD-1 et de CD127 ont été utilisés pour séparer les différentes sous-populations mémoires [142]. Une étude publiée par Perreau M et al, utilisant les marqueurs CXCR5, PD1 et BCL-6, a montré qu’au niveau des ganglions de patients en phase chronique d’infection, les cellules Tfh sont les sous-populations les plus infectées comparativement aux autres cellules mémoires [143]; ce qui pourrait favoriser leurs dysfonctionnements au cours de l’infection à VIH.
À côté des cellules mémoires, les cellules naïves constituent une proportion considérable de LT CD4 [37]. Il a été montré que même les LT CD4 naïves peuvent être infectées par le virus et intégrer l’ADN viral [141, 144]. Différemment des cellules mémoires, leurs chromatines sont moins décompactées, limitant l’intégration de l’ADN viral [145].
19
Chapitre 2 : Hypothèse et objectifs
Comme la majorité des études se sont focalisées sur l’analyse du compartiment sanguin et les ganglions périphériques, il est paradoxale qu’il n’existe pas d’étude approfondie de la rate, un organe majeur de la réponse humorale et qui représente un organe cible de l’infection à VIH. En outre si la majorité des études porte sur la phase chronique de l’infection, il existe très peu d’étude ayant analysé cette dynamique au cours de la phase de primo-infection, phase considérée aujourd’hui comme essentielle dans l’établissement de la pathogenèse et la progression vers un Sida. Ainsi, une infection précoce des cellules Tfh, en particulier au niveau de la rate, pourrait rendre compte de l’absence de réponse humorale efficace à contrôler le virus en particulier via la genèse de cellules B mémoires et la production d’anticorps neutralisant.
Ainsi, nous avons posé comme hypothèse que les cellules Tfh de la rate seraient une des cibles précoces du virus. Cette infection précoce pourrait contribuer à la dissémination de l’infection et à la différence d’évolution vers un Sida.
Pour vérifier cette hypothèse, et dans la mesure où les lymphocytes T CD4 sont constitués par différentes sous-populations, notre premier objectif a été d’étudier la dynamique d’infection des sous-populations lymphocytaires T CD4 par le VIS, et ce en comparant les différentes sous-populations lymphocytaires T CD4, à différents temps d’infection, et en comparant pour chacune d’elles les étapes du cycle viral. Cette première étude porte sur la comparaison entre les ganglions périphériques et la rate. En second lieu, nous avons étudié la capacité des différentes sous-populations lymphocytaires T CD4 à produire un virus infectieux.
21
Chapitre 3 : Matériel et méthodes
Afin de répondre aux objectifs mentionnés, j’ai mis au point dans le laboratoire des techniques de PCR quantitative ou qPCR pour étudier l’infection des sous-populations lymphocytaires T CD4 chez le macaque Rhésus infecté par le SIVmac251. Pour vérifier la propriété infectieuse des virions produits par chaque sous-population, les cellules ont été cultivées et activées in vitro.
3.1 Mise au point des techniques de qPCR
Pour évaluer les différentes étapes de la réplication virale, j’ai mis au point des tests ciblant chaque étape sur une lignée cellulaire, le CEMx174 que j’ai infecté par le SIVmac251. La lignée CEMx174 est une lignée lymphocytaire humaine, infectable par le VIS. Elle est composée de cellules hybrides, par fusion de T et de B. Elles expriment à leur surface le récepteur orphelin GPR15 qui est un des corécepteurs utilisés par les VIS [146].
L’étape de pré-intégration a été étudiée en quantifiant les « rétrotranscrit précoce » et « rétrotranscrit tardif ». L’étape d’intégration a été évaluée par une quantification absolue de l’ADN viral. L’ARN viral a été mesuré en intracellulaire, dans le plasma et dans le surnageant de culture cellulaire (Figure 10).
22
3.1.1 Infection des cellules CEMx174
Les CEMx174 ont été mises en culture dans du RPMI complété avec 10% de FBS, 1mM de Sodium Pyruvate et 1% de Penicillin-Streptomycin-Glutamine à 50 mg/ml à 37°C et à 5% de CO2.
Le SIVmac251 est un homogénat de virus, obtenu initialement à partir de macaque Rhésus Mm251-79 infecté par des tissus lymphomateux. Des stocks ont été effectués et titrés in vivo chez le macaque Rhésus par le Dr Anne Marie Aubertin. Le SIVmac251 fait partie des virus fréquemment utilisés dans les expériences avec les modèles animaux [147].
Au bout de deux passages cellulaires, les 5x106 cellules ont été infectées par du SIVmac251 contenant 5x108 copies d’ARN viral.
L’efficacité de l’infection a été vérifiée par la présence de syncytia dans la culture au bout de 72 heures d’infection correspondant à la fusion des glycoprotéines d’enveloppe gp120 exprimées à la surface des cellules infectées avec les molécules CD4 exprimées par les cellules non infectées.
Trois aliquotes contenant 105 cellules chacune et une aliquote de surnageant de culture ont été réalisées tous les jours pendant 7 jours afin d’étudier la dynamique temporelle de la réplication virale. Deux aliquotes cellulaires ont été gardées à sec pour les tests d’ADN viral. Une troisième aliquote a permis l’extraction de l’ARN viral par ajout de 500 ul de trizol.
3.1.2 qPCR sur les CEMx174 infectées
Pour chaque test, les échantillons ont été testés en duplicata. Cela permet de vérifier la reproductibilité du test. Les tests ont été effectués sur une faible quantité de cellules afin de vérifier leur sensibilité.
3.1.2.1 Principe de la qPCR
La PCR consistent à amplifier un fragment d’acide nucléique, en utilisant des amorces spécifiques et une substance fluorescente. On peut utiliser soit un agent intercalant comme le « sybr green » soit une sonde liée à un fluorochrome comme la sonde Taqman. Un signal fluorescent émis à la fin de chaque cycle est proportionnelle aux produits amplifiés [148]. L’efficacité de l’amplification est évaluée à la fin de chaque réaction. Elle doit être près de 1.
23 La quantification peut être absolue, en déterminant la quantité du produit amplifié par rapport à une courbe d’étalonnage d’un plasmide standard. Elle peut être relative en comparant le produit amplifié par rapport à un gène de référence comme l’ARN18S [149].
3.1.2.2 Quantification relative des rétrotranscrits précoces et tardifs
Ces tests ont été effectués à partir d’un culot sec contenant 105 cellules. L’ADN viral a été extrait avec 50ul de tampon de lyse composé de 10 mM de TRIS (pH = 8), 0.5 mM d’EDTA, 0.0001% de SDS et 0.001% de Triton [63]. Nous avons choisi cette technique d’extraction car elle évite les pertes en rétrotranscrit précoce, comparée à celle utilisant la colonne de purification.
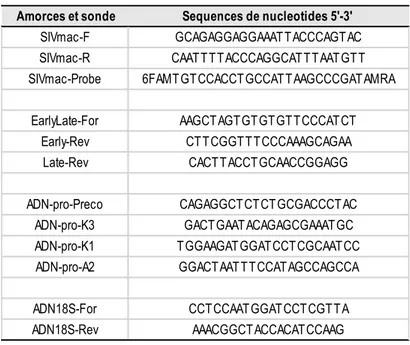
Cinq microlitres d’ADN a été amplifié par qPCR, dans un mix composé de 2XQuantitec Sybr Green, 250 nM de chaque amorce sens et antisens pour un volume final de 20 µl. Chaque échantillon a été testé avec les amorces ciblant le rétrotranscrit précoce, le rétrotranscrit tardif et l’ADN18S (Tableau 2).
L’amplification a été effectuée avec le thermocycleur Applied Biosystems 7500 Real-Time PCR System selon le protocole d’amplification ci-après [50°C pendant 2 minutes, 95°C pendant 15 minutes + 40 cycles (94°C pendant 15 secondes, 60°C pendant 30 secondes, 72°C pendant 35 secondes) + étape de dissociation]. Des cellules CEMx174 non infectées ont été utilisées comme contrôle négatif. Cela nous a permis d’obtenir des résultats semi-quantitatifs pour chacun des rétrotranscrits, exprimés en ∆Ct égal à [25 (correspondant au ∆Ct d’un échantillon négatif) - ∆Ct de l’échantillon (correspond à la différence entre le Ct du rétrotranscrit de l’échantillon et celui de l’ADN18S)].
3.1.2.3 Quantification de l’ADN viral
L’ADN a été extrait à partir du 2ème culot sec contenant 105 cellules, avec le kit Genomic DNA from Tissue (Macherey Nagel). De la RNAse A a été ajoutée durant l’étape de lyse afin d’éliminer les contaminations d’ARN. L’ADN a été élué dans 50 µl d’eau RNAse-free. Pour les étapes d’amplification, nous nous sommes basés sur le protocole de Khatissian E et de Monceaux V [82, 150]. Quelques modifications ont été apportées sur le protocole initial afin d’obtenir un résultat quantitatif exprimé en nombre de copie par cellule.
Cinq microlitres de l’extrait d’ADN a été amplifié par une PCR nichée utilisant deux couples d’amorces, ciblant la région nef. La 1ere série de PCR amplifie un fragment de 961 paires de bases. Pour cela, nous avons utilisé une mixture composée de tampon 10X PCR, 0.8 mM de DNTP, 2 mM de MgCl2, 1.25 U d’AmpliTaq, 100 nM
24
de chaque amorces preco et K3 (Tableau 2), pour un volume final de 50 µl. L’amplification a été effectuée sur le thermocycleur Biometra selon le protocole d’amplification suivant [95°C pendant 1 minute 45 secondes + 25 cycles (95°C pendant 30 secondes, 60°C pendant 30 secondes, 72°C pendant 1 minute 10 secondes) + 72°C pendant 6 minutes]. Un clone de cellule SIV-1C fourni par François Villinger, contenant 1 copie de provirus de SIVmac251 par cellule a été utilisé comme standard pour la quantification d’ADN viral. Les cellules SIV-1C ont été diluées en série avec des cellules CEMx174. L’ADN issu de ce mélange de cellules a été extrait puis amplifié avec les mêmes conditions que ceux des cellules infectées.
Cinq microlitres du produit amplifié, dilué au 1/8, a été ré-amplifié avec le deuxième couple d’amorces A2 et K1 [151] (Tableau 2), en utilisant la même concentration de mix et les mêmes conditions d’amplification pour les rétrotranscrits. Cela a permis d’obtenir le nombre de copie d’ADN viral dans 5 µl d’échantillon initial.
L’ADN18S a été amplifié à partir de 5 µl de l’échantillon initial, parallèlement à la 2ème série d’amplification d’ADN viral. Une dilution en série d’ADN18S obtenue à partir de cellules CEMx174 non infectées a été utilisée comme standard pour estimer le nombre de cellule dans chaque réaction.
Avec cette approche, nous avons pu exprimer nos résultats par nombre de copie d’ADN proviral de SIV sur 104 cellules.
3.1.2.4 Quantification de la charge virale
Au niveau cellulaire, l’extraction d’ARN a été effectuée à partir du 3ème aliquote de 100000 cellules gardées dans 500 µl de trizol, selon les instructions du fabricant (Invitrogen). Un traitement avec du Turbo-DNA free (Invitrogen) a été effectué à la fin de l’extraction. Cela permet d’éliminer toute contamination possible avec l’ADN génomique. Après resuspension dans 50 µl d’eau RNAse-free, la pureté des extraits d’ARN ainsi que leurs intégrités ont été vérifiées soit avec le Bioanalyser soit avec le NanoDrop.
Cinq microlitres d’extrait d’ARN ont été amplifiés par une PCR quantitative associée à la rétrotranscription (RT-qPCR). Le mix est composé de 4X TaqMan Fast Virus 1-Step Master Mix contenant l’enzyme la reverse transcriptase, 750 nM de chaque amorce SIVmac Sens et Antisens, 200 nM de sonde (Tableau 2) pour un volume final de 20 µl. Comme standard, nous avons utilisé un plasmide de SIVMac251 contenant le gène SIV
gag, dilué en série (au dixième) à partir de 109 copies/µl de SIV. L’amplification a été effectué avec le thermocycleur Applied Biosystems 7500 Real-Time PCR System selon les conditions d’Amplification ci-après [50°C pendant 5 minutes, 95°C pendant 20 secondes, 40 cycles (95°C pendant 15 secondes, 60°C pendant 1 minute)]. Cela a permis d’obtenir le nombre de copie d’ARN viral dans 5 µl d’échantillon.
25 L’ARN18S a été amplifié en parallèle à l’ARN viral intracellulaire. Pour cela, nous avons utilisé le mix Eukaryotic 18S rRNA Endogenous Control (Lifetechnologies). Une dilution en série d’ARN18S de CEMx174 a été utilisée comme standard pour estimer le nombre de cellule dans chaque réaction. Les résultats ainsi obtenus nous ont permis d’obtenir une estimation du nombre de copie de SIV par cellule.
Dans le surnageant de culture, l’extraction d’ARN a été effectuée sur 200 µl d’échantillon avec le kit PureLink Viral RNA/DNA (Invitrogen). L’ARN est elué dans 25 µl d’eau RNAse-free dont cinq microlitres sont amplifiés par RT-qPCR. Nous avons utilisé les mêmes conditions d’amplification pour l’ARN viral intracellulaire. Cela permet d’obtenir le nombre de copie d’ARN viral par millilitre de surnageant. Le résultat a été rapporté au nombre de cellules correspondant au surnageant. Cela a permis d’obtenir une estimation du nombre de copie de SIV produit par une cellule dans le surnageant de culture.
Amorces et sonde Sequences de nucleotides 5'-3'
SIVmac-F GCAGAGGAGGAAATTACCCAGTAC SIVmac-R CAATTTTACCCAGGCATTTAATGTT SIVmac-Probe 6FAMTGTCCACCTGCCATTAAGCCCGATAMRA EarlyLate-For AAGCTAGTGTGTGTTCCCATCT Early-Rev CTTCGGTTTCCCAAAGCAGAA Late-Rev CACTTACCTGCAACCGGAGG ADN-pro-Preco CAGAGGCTCTCTGCGACCCTAC ADN-pro-K3 GACTGAATACAGAGCGAAATGC ADN-pro-K1 TGGAAGATGGATCCTCGCAATCC ADN-pro-A2 GGACTAATTTCCATAGCCAGCCA ADN18S-For CCTCCAATGGATCCTCGTTA ADN18S-Rev AAACGGCTACCACATCCAAG
Tableau 2 : Amorces et sondes utilisées pour les qPCR
3.2 Infection des macaques Rhésus par le VIS
Les techniques de qPCR mises au point avec les cellules CEMx174 ont été utilisées sur des échantillons de macaques Rhésus infectés par le même virus SIVmac251 afin de répondre à la question de la dynamique d’infection des sous-populations lymphocytaires T CD4.
26
3.2.1 Animaux, infection et prélèvements
Nous avons utilisé vingt macaques Rhésus, d’origine indienne, séronégatifs pour STLV-1 (Simian T Leukemia Virus de type-1), SRV-1 (rétrovirus de type D), herpès virus de type B et SIVmac.
Dix-huit animaux ont été infectés par du SIVmac251 par voie intraveineuse, avec une dose de 20 AID50 (Dose Infectieuse pour 50% des animaux). Deux animaux ont été utilisés comme contrôle non infecté. Des prélèvements sanguins ont été effectués régulièrement pour le suivi des animaux. Pour chacun des prélèvements, un hémogramme sanguin et une quantification de la charge virale plasmatique ont été réalisés. Les animaux ont été euthanasiés à différents temps d’infection, durant la phase aigüe de l’infection (n=2 à J0, J4, J7, J11, J14, J18-21 et J30) et en chronique (n=1 à J46, J55J167, J194, J223 et J237). À chaque jour d’euthanasie, du sang périphérique et des organes lymphoïdes incluant la rate, les ganglions axillaires et inguinaux ont été prélevés pour chaque singe. Les cellules des organes lymphoïdes ont été triées et utilisées pour les études au niveau cellulaire.
3.2.2 Quantification de la charge virale plasmatique
Nous avons utilisé le même protocole pour la quantification de la CV dans le surnageant de culture. L’extraction d’ARN a été effectuée sur 200 µl de plasma avec le kit PureLink Viral RNA/DNA (Invitrogen). Les résultats ont été exprimés en nombre de copie d’ARN viral par millilitre de plasma.
3.2.3 Tri cellulaire
3.2.3.1 Préparation des échantillons
A chaque jour d’euthanasie, nous avons récupéré les cellules des organes lymphoïdes prélevés après dilacération, puis lavage avec du RPMI complet. Les cellules des ganglions axillaires et inguinaux ont été mélangées. Les cellules mononuclées de la rate ont été récupérées suivant la technique des gradients de ficoll. Cette technique permet de séparer les cellules selon leur densité par rapport à celle du ficoll (Figure 11).
27
Figure modifiée à partir de [152]
3.2.3.2 Tri des différentes sous-populations
Pour chaque organe rate et ganglions, nous avons réalisé un tri cellulaire à l’aide d’un trieur BD influx à partir d’environ 100 millions de cellules. Ceci a été réalisé sur la base de l’expression de marqueurs de surface à savoir CD3, CD8, CD4, PD-1, CXCR5, CD45RA et CCR7. Avec ce trieur, nous avons pu isoler simultanément 6 sous-populations dont 5 sous-populations de lymphocytes T CD4 et une population de lymphocytes T CD8 que nous avons utilisé comme contrôle négatif de l’infection.
Afin d’isoler les différentes sous-populations de lymphocytes T CD4, nous avons tout d’abord défini la région des lymphocytes T CD3+. A partir de cette première région, nous avons séparé les lymphocytes T CD4 des lymphocytes T CD8. A partir des lymphocytes T CD4, les 5 sous-populations ont été définies, d’une part les cellules Tfh exprimant fortement les molécules CXCR5 et PD1, puis les 4 autres sous-populations CD4 séparées selon l’expression des molécules CD45RA et CCR7, définissant les cellules naïve, centrale mémoire ou TCM, effectrice mémoire ou TEM, T folliculaire auxiliaires (helper) ou Tfh et différenciée terminale ou TDT (Figure 12).
Des aliquotes contenant 105 cellules de chacune des souspopulations cellulaires triées ont été conservées à -80°C pour des études de qPCR.
![Figure 1 : Origine des virus du Sida Figure tirée de Sharp et al., 2011 [2]](https://thumb-eu.123doks.com/thumbv2/123doknet/5586403.134142/18.918.104.764.96.639/figure-origine-des-virus-sida-figure-tirée-sharp.webp)
![Figure 2 : Prévalence mondiale du Sida en 2013 Figure tirée des données de Nations Unies sur Actualix.com [23]](https://thumb-eu.123doks.com/thumbv2/123doknet/5586403.134142/19.918.148.748.631.980/figure-prévalence-mondiale-figure-tirée-données-nations-actualix.webp)
![Figure 3 : Structure du virus VIH-1 Figure tirée de Gemrot et al., 2009 [49]](https://thumb-eu.123doks.com/thumbv2/123doknet/5586403.134142/22.918.115.781.127.479/figure-structure-virus-vih-figure-tirée-gemrot-al.webp)
![Tableau modifié à partir de Gobeil et al., 2012 [53]](https://thumb-eu.123doks.com/thumbv2/123doknet/5586403.134142/23.918.146.824.106.649/tableau-modifié-partir-gobeil-al.webp)

![Figure 6 : Étapes du cycle de réplication du VIH Figure modifiée à partir de Pasternak et al., 2013 [78]](https://thumb-eu.123doks.com/thumbv2/123doknet/5586403.134142/27.918.236.699.100.403/figure-étapes-cycle-réplication-figure-modifiée-partir-pasternak.webp)
![Figure 7 : Évolution typique de l’infection à VIH Figure tirée de [95]](https://thumb-eu.123doks.com/thumbv2/123doknet/5586403.134142/28.918.186.722.662.1011/figure-évolution-typique-l-infection-vih-figure-tirée.webp)
![Figure 9 : Interaction entre Tfh et cellule B au niveau du centre germinatif Figure tirée de Nutt et al., 2011 [134]](https://thumb-eu.123doks.com/thumbv2/123doknet/5586403.134142/33.918.151.801.137.491/figure-interaction-cellule-niveau-centre-germinatif-figure-tirée.webp)