Caractérisation des systèmes hôte-microbiote ichthyens dans
des contextes environnementaux contrastés du bassin
amazonien
Mémoire
François-Étienne Sylvain
Maîtrise en biologie
Maître ès sciences (M. Sc.)
Québec, Canada
© François-Étienne Sylvain, 2017
Résumé
Les microbiotes des Poissons téléostéens du bassin de l'Amazone - regroupant plus de 3000 espèces - ont reçu peu d'attention de la communauté scientifique (contrairement aux microbiotes des poissons des milieux tempérés), même si cet écosystème est reconnu comme étant le plus riche réservoir de poissons dulcicoles de la planète. Notre étude visait à répondre à cette lacune, en caractérisant les structures taxonomiques des microbiotes (du mucus cutané et des intestins) des Poissons d'Amazonie, et en identifiant certains des facteurs biotiques et abiotiques qui modulent la composition et la diversité de ces microbiotes. Nous avons échantillonné trois espèces de poissons ayant des régimes et des mode de vie contrastés, à sept sites d'échantillonnage dans le bassin de l'Amazone brésilien (10 individus / espèce / site) durant la saison sèche. Les sept sites d'échantillonnage regroupaient 5 sites d'eau blanche (eutrophique, pH neutre) et 2 sites d'eau noire (oligotrophique, pH acide), afin d'évaluer l'impact des paramètres physicochimiques sur la structure taxonomique des communautés microbiennes. Les microbiotes cutané et intestinaux de chaque individu ont été caractérisés par profilage d'amplicons du gène de l'ARNr 16S. Nos résultats du Chapitre 2 montrent que la structure taxonomique des deux microbiotes étudiés corroborent différents traits de l'écologie des espèces étudiées, tels leur régime alimentaire, leur mode de vie, leur histoire évolutive et la connectivité des habitats qu'elles exploitent. Les résultats du Chapitre 3 indiquent que la couleur de l'eau - blanche ou noire - joue un rôle majeur dans la structuration des deux microbiotes ichthyens étudiés. Les prochaines études devraient utiliser une approche de métagénomique fonctionnelle afin de vérifier si les divergences taxonomiques documentées impliquent aussi des différences fonctionnelles. De plus, les prochaines études devraient déterminer si l'importante saisonnalité du bassin de l'Amazone a un impact significatif sur les structures taxonomiques des microbiotes que nous avons documentés.
Abstract
Teleostean fishes, represented by more than 28 000 species, encompass a wide range of different physiologies, evolutionary histories and ecologies, and therefore embody a key group to study the nature and the factors driving vertebrate-associated microbiotas. In contrast to temperate fish microbiotas, Amazonian fishes' microbial assemblages have never been investigated, even if this ecosystem is widely recognized as Earth's richest freshwater fish reservoir. The main objective of our study was to fill this knowledge gap, by characterizing the taxonomic structures of Amazonian teleost-associated microbiotas (from skin mucus and gut), and by identifying precisely the biotic and abiotic factors shaping the composition and diversity of these microbiotas. We sampled three species of fish with contrasted diets and lifestyles, at seven sampling sites of the Brazilian Amazon (10 individuals/species/site) in November 2015 (dry season). The seven sampling sites included five white water sites (eutrophic, neutral pH), and 2 black water sites (oligotrophic, acidic pH), to assess the impact of water physicochemical parameters on the taxonomic structure of microbial communities. Cutaneous and gut microbiotas of each sampled fish were characterized by profiling libraries of 16S rRNA amplicons. Our results from Chapter 2 showed that the taxonomic structure of the two microbiotas investigated corroborated different ecological traits from the three species, including their diet, their lifestyle, their evolutive history and the connectivity of the habitats exploited by these species. The results from Chapter 3 show that water colour (white or black) play a major role in structuring both teleost-associated microbiotas. Future studies should use a functional metagenomic approach to assess if the reported taxonomic differences also represent significant functional divergences. Furthermore, future studies should examine the seasonal variation in the taxonomic patterns observed in our study.
Table des matières
Résumé ... iii
Abstract ... iv
Liste des tableaux ... vii
Liste des figures ... viii
Liste des abréviations et des sigles ... xi
Remerciements ... xii
Avant-propos ... xiv
Chapitre 1. Introduction ... 1
1.1 Bactéries symbiotiques ... 1
1.2 Holobionte et hologénome ... 2
1.3 Microbiote, microbiome et métagénome ... 4
1.4 Recrutement du microbiote: transmission verticale et horizontale ... 6
1.5 Facteurs influençant la structure taxonomique du microbiote ... 8
1.6 Les Téléostéens comme modèles d'étude ... 10
1.7 Les symbiontes bactériens des Poissons ... 11
1.8 Problématique et objectif principal ... 14
1.9 Milieu d'étude ... 16
1.10 Études antérieures et objectifs détaillés ... 17
1.11 Trois espèces ciblées ... 22
1.12 Résumé de la méthode ... 23
1.13 Importance de l'étude ... 24
Chapitre 2: Gut and skin microbiomes of wild Amazonian Teleosts reveal contrasted fingerprints of host ecology and ancestry ... 25
Résumé du chapitre 2 ... 25
Abstract ... 26
2.1 Introduction ... 27
2.2 Materials and Methods ... 29
2.2.1 Ethics statement ... 29
2.2.2 Fish sampling ... 29
2.2.3 Preparation of 16S amplicon libraries ... 30
2.2.4 Analysis of 16S sequences ... 30
2.3 Results and Discussion ... 33
2.3.2 Skin mucus and gut microbiomes both reflect host phylogeny ... 37
2.3.3 Species-specific inter-population microbiome divergence in both tissues ... 37
2.3.4 More potential ecological insights from the gut microbiota ... 39
2.5 Acknowledgements ... 43
Chapitre 3: Teleost microbial communities driven by hydrochemically contrasted black and white waters of the Amazon Basin ... 44
Résumé du chapitre 3 ... 44
Abstract ... 45
3.1 Introduction ... 46
3.2 Materials and Methods ... 46
3.2.1 Fish sampling ... 46
3.2.2 Preparation of 16S amplicon libraries ... 47
3.2.3 Analysis of 16S sequences ... 48
3.2.4 Water chemistry parameters ... 50
3.3 Results ... 51
3.4 Discussion ... 53
3.4.1 Impact of habitat type on microbial assemblages ... 53
3.4.2 Niche-specific response to different water colors ... 54
3.4.3 From water physicochemical parameters to microbial signatures ... 54
3.5 Acknowledgements ... 55
Chapitre 4. Conclusion ... 56
4.1 Synthèse des résultats majeurs ... 56
4.2 Points à considérer et direction des études futures ... 58
Liste des tableaux
Chapitre 2: Gut and skin microbiomes of wild Amazonian Teleosts reveal contrasted fingerprints of host ecology and ancestry
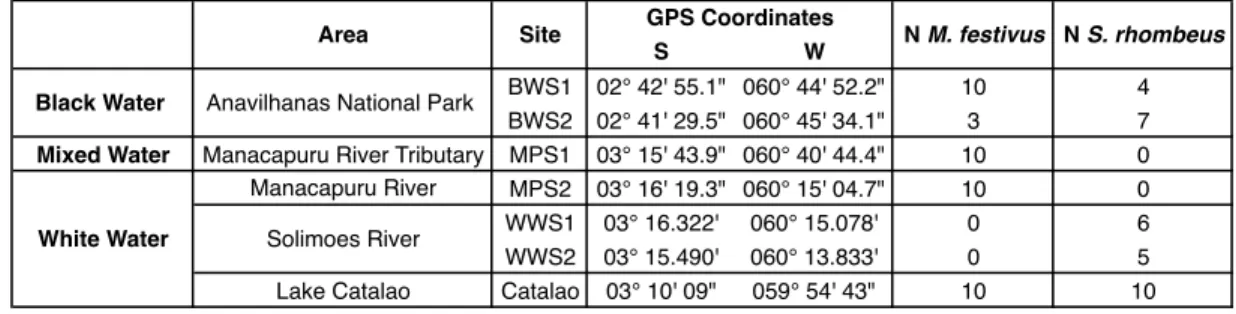
Table 1: GPS Coordinates and number of fish sampled at each location...32 Chapitre 3: Teleost microbial communities driven by hydrochemically contrasted black and white waters of the Amazon Basin
Table 1: GPS Coordinates and number of fish sampled at each location...51 Table 2: Water physicochemical parameters at each sampling location...51
Liste des figures
Chapitre 1: Introduction
Figure 1: Recrutement du microbiote aux différentes phases du cycle de vie des Téléostéens. Les numéros correspondent aux étapes du cycle de vie (en ordre croissant). Les cercles de différentes couleurs représentent différents taxa bactériens. Adaptation de la Figure 3 de Llewellyn et al. 2014...13
Chapitre 2: Gut and skin microbiomes of wild Amazonian Teleosts reveal contrasted fingerprints of host ecology and ancestry
Figure 1: (a,b,c): Stacked bar charts representing the mean taxonomic structure from each sampling location per species and per microbial niche. The relative abundance of the 10 most abundant bacterial classes was documented in skin mucus (a), in gut samples (b), and in environmental water (c). The legend for the colour's attribution in (a,b,c) can be found in (c). (d): Heatmaps representing the mean taxonomic structure per species and per microbial niche (generated with the PhyloSeq package on R). The relative abundance of the 10 most abundant bacterial phyla was documented in skin mucus, and in gut samples. (e, f): Non-parametric Shannon Diversity Indexes for each microbial niche, per species and per sampling location. Diversity Indexes were calculated using mothur. (e): diversity estimations for skin mucus samples, and (f): diversity estimations for gut samples. Different letters over two boxes of a same boxplot highlight significant differences (p-value < 0.05) between two sampling sites for one species...34 Figure 2: (a,b) Networks of samples per microbial niche per species and per sampling location. The Spearman correlations were calculated with R v.3.2.1 and were plotted with Cytoscape v.3.2.1. Significant correlation values used for the network construction had a Bonferroni corrected p-value ≤ 0.05. The nodes of the network represent a sample and the edges are attributed to significant Spearman correlations between nodes. The size of each node is proportional to the number of
connections (edges) it makes with other nodes. The shape of each node is related to the sampling site and the colour of the node is related to the species. Skin mucus samples are plotted in (a), and gut samples in (b). (c,f): PERMANOVAs were used to assess the influence of the microbial niche, the species and the sampling locations, on the microbiota taxonomic structure of skin mucus (c) and gut (f) samples. Green boxes mean significant p-values (< 0.05). Pictures of the three species in (c) were taken by F-ÉS and are free of rights. (d,e): Multi-Factorial Analysis (MFA) per microbial niche and per specie, generated with the R package FactomineR. Each data point represents a sample of skin mucus (d) or gut contents (e). Clusters which have non-overlapping confidence ellipses are significantly different (default confidence level of 0.95)...36 Figure 3: (a,b,c,d): Boxplots made with gut samples of each species representing: (a) the Firmicutes / Bacteroidetes ratio per specie, (b) the number of OTUs detected in the guts of each specie, (c) the Thetayc Dissimilarity Inter-Location of each specie, and (d) the Shannon Taxonomic Diversity between each specie. Different letters over two boxes of a same boxplot highlight significant differences (p-value < 0.05). ...41 Chapitre 3: Teleost microbial communities driven by hydrochemically contrasted black and white waters of the Amazon Basin
Figure 1: Alpha diversity and taxonomic structure is related to the water color and the microbial niche. (a) Non-parametric Shannon diversity index for the skin mucus and gut of M. festivus and S. rhombeus at different water colors. "White" refers to "White water", "Mixed" refers to "Mixed water (black and white)", and "Black" refers to "Black water". Significantly different groups (in each microbial niche) are defined by the significance letters above each box. The diversity of water samples is not shown, as only one sample of black water was successfully amplified and sequenced. Diversity Indexes were calculated using mothur. (b) Relative abundance (%) of the 20 most abundant bacterial genera in each microbial niche and in each
water color. For easier viewing, this bar chart is scaled at 60% of the total relative abundance of the microbial community – meaning only the 20 genera that together composed 60% of the total bacterial community are represented. (c,d,e,f) Multi-Factorial Analysis (MFA) generated with the R package FactomineR. Each data point represents a sample of skin mucus (c,d) or gut contents (e,f). Clusters which have non-overlapping confidence ellipses are considered to be significantly different (default confidence level of 0.95). This MFAs are based on the relative abundance of the 50 most abundant OTUs. Note: In a, b, d, f, there is no "Mixed water" samples for S. rhombeus, as no specimens of this species were fished at the "Mixed water" site. ...52 Figure 2: (a,d) Networks of microbial genera per microbial niche (skin mucus in (a) and gut (d)). The Spearman correlations were calculated with R v.3.2.1 and were plotted with Cytoscape v.3.2.1. Significant correlation values used for the network construction had a Bonferroni corrected p-value ≤ 0.05. The nodes of the network represent a sample and the edges are attributed to significant Spearman correlations between nodes. The size of each node is proportional to the number of connections (edges) it makes with other nodes. Orange nodes represent water physicochemical parameters. Blue nodes are microbial genera with connecting edges to water physicochemical parameters (orange nodes). Grey nodes are microbial genera with connecting edges only to other microbial genera (not to any water physicochemical parameters). (b, e) Bar charts showing the number of bacterial genera significantly interacting with each water physicochemical parameter in skin mucus (b) and gut (e) samples. (c, f) Bar charts showing the differential abundance between water color, of the 5 bacterial genera that were affected by the most number of water physicochemical parameters, in skin mucus (c) and in gut (f) samples. In (c, f), brown bars represent abundance in black water, whereas cream colour bars represent abundance in white water...53
Liste des abréviations et des sigles
INPA: «Instituto Nacional de Pesquisas da Amazonia», Institut National de recherche de l'Amazonie.
PCR: "Polymerase Chain Reaction", Réaction d'amplification en chaîne par polymérase. dNTPs: deoxynucleotide
OTU: "Operational Taxonomic Unit", Unité taxonomique opérationnelle.
PERMANOVA: "Permutational Analysis of Variance", Analyse de variances avec permutations.
Remerciements
Je débute d'abord par remercier sincèrement mon directeur de recherche, Nicolas Derome, qui a cru en moi et qui m'a donné les ailes pour amorcer ma carrière en science. Au cours des dernières années, Nicolas a non seulement été un professeur et un mentor hors pair, mais est aussi devenu un ami très cher. Merci à ton dévouement, ta patience, ta disponibilité et ton ouverture.
J'aimerais aussi remercier très chaleureusement mon co-directeur, Adalberto Luis Val de l'Institut National de recherche de l'Amazonie, pour nous avoir grand ouvert les portes de son laboratoire et pour nous avoir donné tout son support au cours des quatre dernières années. Adalberto, merci pour ton accueil et ta confiance.
Étant en milieux éloignés, les expéditions d'échantillonnage en Amazonie nécessitent une importante logistique. Conséquemment, elles ne pourraient pas être effectuées sans l'aide de précieux organisateurs/trices et de professionnels qualifiés sur le terrain. À cet effet, je tiens à remercier: Maria de Nazare Paula da Silva, Fernanda Dragan, Rogerio Pereira et Raquel Abecassis (organisation des expéditions); Thiago L. A. Nascimento et Derek Campos (techniciens sur le terrain); Fabiano Mota, Francisco Fonseca et Frank Queirzo Coelho (pêcheurs professionnels); Jansen Zuanon, Derek Campos, Dinho Heinrichs et Natalia Wagner (identification des poissons). Je tiens aussi à remercier tous ceux qui ont contribué à la réalisation de ce projet, notamment Pierre-Luc Mercier et Camille Desrosiers (manipulation des échantillons en laboratoire); Brian Boyle, Sidki Bouslama, Luciana Fé, Érica Souza et Daniel Fagundes (analyse moléculaire et support bioinformatique); et tous les membres des laboratoires Derome et Val.
Je remercie aussi tous ceux qui ont contribué financièrement et administrativement à ce projet, particulièrement: André Darveau, la faculté de sciences et génie de l'Université Laval, Ressources Aquatiques Québec, le Fonds de recherche du Québec - Nature et technologies (FRQNT), le Conseil de recherches en sciences naturelles et en génie du
Canada (CRSNG), le «Ministério da Ciência, Tecnologia, Inovações e Comunicações (MCTIC)», le «Conselho de Ciência e Tecnologia» ainsi que l'«Instituto Chico Mendes de Conservação da Biodiversidade» pour le support sur le terrain et pour l'octroi de permis de transport d'échantillons.
Avant-propos
Les objectifs principaux de ce projet étaient de caractériser les structures taxonomiques des microbiotes cutanés et intestinaux des Poissons d'Amazonie, ainsi que d'identifier certains des facteurs biotiques et abiotiques qui modulent la composition et la diversité de ces microbiotes. Nous avons échantillonné les microbiotes cutané et intestinal chez trois espèces de Poissons du bassin de l'Amazone, qui ont des écologies (régime alimentaire, mode de vie, histoire phylogénétique) contrastées et qui exploitent une vaste gamme d'habitats, incluant des cours d'eau blanche (eutrophe, riche en ions, bien oxygénée et de pH neutre) et d'eau noire (oligotrophe, pauvre en ions, en oxygène et de pH acide). Le microbiote des trois espèces a été caractérisé par une approche de profilage de librairies d'amplicons de la sous unité 16S du gène de l'ARNr, afin de mettre en évidence l'effet des variables suivantes sur la structure taxonomique des microbiotes cutané et intestinal: la phylogénie, le régime alimentaire, le mode de vie, la connectivité inter-habitats, différents sites d'études (variations inter-populations) et différents types d'eau (blanche versus noire). Ce mémoire comprend d'abord un premier chapitre d'introduction présentant les notions théoriques liées à cette étude. Le second chapitre est rédigé en anglais et comprend essentiellement un article scientifique explorant les effets de la phylogénie, du régime alimentaire, du mode de vie, de la connectivité inter-habitats et de différents sites d'études sur la structure taxonomique des microbiotes chez les trois espèces ciblées. Le troisième chapitre est aussi rédigé en anglais et représente un article court (sous forme de "Short Communication"), détaillant l'effet de différentes couleurs d'eau sur le microbiote de deux des trois espèces ciblées. Le quatrième chapitre est une conclusion faisant un retour sur les résultats principaux et présentant une ouverture sur des perspectives de recherche futures. Le texte formant le chapitre 2 du présent mémoire sera soumis au journal de l'International Society of Microbial Ecology (ISME Journal) prochainement. Les auteurs de ce manuscrit sont, dans l'ordre, François-Étienne Sylvain, Aleicia Holland, Émie Audet-Gilbert, Adalberto Luis Val et Nicolas Derome. Le manuscrit est retrouvé dans le mémoire tel qu'il
sera soumis à ISME excepté les modifications suivantes, pour des raisons de mise en page lors de l'insertion de l'article dans le mémoire:
• Modification de la police du document en Times New Roman
• Suppression des numéros de lignes et modification des numéros de pages • Suppression de la page titre
• Inclusion de la section "Supplementary Material" dans "Material and Methods". • Suppression des sections "Conflict of interest", "Figure legends", "Tables"
• Reformatage de la section "References" pour inclusion dans la section Bibliographie du mémoire
Le texte formant le chapitre 3 du présent mémoire a été soumis au journal ISME le 16 juin 2017 et est actuellement en revue. Les auteurs de ce manuscrit sont, dans l'ordre, François-Étienne Sylvain, Aleicia Holland, Émie Audet-Gilbert, Camille Lavoie, Adalberto Luis Val et Nicolas Derome. Le manuscrit est retrouvé dans le mémoire tel que soumis à ISME Journal, excepté les modifications suivantes, pour des raisons de mise en page lors de l'insertion de l'article dans le mémoire:
• Modification de la police du document en Times New Roman
• Suppression des numéros de lignes et modification des numéros de pages • Suppression de la page titre
• Inclusion de la section "Supplementary Material" dans "Material and Methods". • Suppression des sections "Conflict of interest", "Figure legends", "Tables"
• Reformatage de la section "References" pour inclusion dans la section Bibliographie du mémoire
Pour l'ensemble des deux articles: Nicolas Derome (directeur de recherche) et Adalberto Luis Val (co-directeur) ont supervisé toutes les étapes du projet, organisé et participé aux expéditions d'échantillonnage sur le terrain et ont participé activement à la rédaction des manuscrits. Aleicia Holland a participé aux échantillonnages sur le terrain et aux manipulations des échantillons en laboratoire. Émie Audet-Gilbert a contribué significativement aux manipulations des échantillons en laboratoire (extractions d'ADN et PCRs). Pour le premier article: Camille Lavoie a contribué à l'analyse statistique des
données par la conception des "Heatmaps" retrouvées dans la Figure 1d du premier article. Tous les auteurs ont révisé les versions finales des manuscrits soumis. De mon côté, je suis premier auteur des deux articles en raison de mon implication à toutes les étapes du projet: la conception de l'étude, l'échantillonnage sur le terrain, la construction des librairies d'amplicons en laboratoire, l'analyse statistique bioinformatique ainsi que la rédaction des manuscrits.
Chapitre 1. Introduction
1.1 Bactéries symbiotiques
Nous vivons dans un monde dominé par les bactéries (Whitman et al. 1998). Les Bactéries seraient apparues sur Terre 2 milliards d'années avant les premiers signes de vie eucaryote (Lee et Mazmanian 2010). Depuis plus de 500 millions d'années, les Bactéries co-évoluent en symbiose avec des organismes hôtes eucaryotes (Hickman 2005). Les interactions Bactéries-Eucaryotes ont potentiellement joué d'importants rôles dans l'histoire de la vie sur Terre: Margulis (1993) a documenté l'endosymbiose de Protéobactéries et de Cyanobactéries dans les cellules eucaryotes ayant respectivement mené aux mitochondries et aux chloroplastes. Ceci suggère que les Bactéries ont joué un rôle crucial dans l'évolution des Métazoaires (Lee et Mazmanian 2010). Les études menées par Alegado et al. (2012) suggèrent que les Bactéries ont même joué un rôle dans l'initiation de la multicellularité. Lederberg décrit aujourd'hui les Métazoaires en tant que super-organismes, composés d'une association de cellules microbiennes et de cellules de l'hôte, où la survie de chacun des partenaires dépend de la survie de l'autre (Lederberg 2000).
Cette co-évolution (Ley et al. 2006), co-adaptation et co-dépendance entre l'hôte et ses communautés de bactéries endogènes (Dethlefsen et al. 2007) ont permis aux Métazoaires d'exploiter des niches variées et d'élargir leur spectre alimentaire (Douglas 2010; Dubilier et al. 2008). Par exemple, les bactéries symbiotiques du tractus digestif jouent des rôles importants dans la nutrition (Floch et al. 1970). Ces bactéries sont essentielles pour la synthèse de vitamines, de facteurs de croissance (Teply et al. 1947; Uphill et al. 1977) et pour la bioconversion de composés toxiques (Chaucheyras et Durand 2010). De plus, des études menées sur des poissons gnotobiotiques (stériles) mettent en évidence le rôle joué par certaines bactéries gastro-intestinales dans la différenciation épithéliale et la maturation du tractus digestif (Bates et al. 2006). En plus d'être impliqué dans l'assimilation des nutriments et la différenciation épithéliale, les symbiotes bactériens des Poissons retrouvés dans l'intestin et le mucus cutané peuvent inhiber la prolifération d’agents pathogènes opportunistes (Boutin et al. 2013; Robertson et al. 2000; Sugita et al. 2002). Par exemple,
une espèce du genre Carnobacterium, isolée de l'intestin des Salmonidés, a la capacité d'inhiber plusieurs agents pathogènes comme Aeromonas hydrophila, Aeromonas salmonicida, Flavobacterium branchiophilum, Photobacterium damselae, Vibrio anguillarum et Streptococcus milleri (Robertson et al. 2000).
Récemment, des rôles joués par les Bactéries ont été mis en évidence dans le développement du cerveau, du comportement (Heijtza et al. 2011) et dans les préférences sexuelles de l'hôte (Sharon et al. 2010). De plus, il a été montré que les symbiontes bactériens intestinaux jouent des rôles importants dans la régulation des fonctions immunitaires chez les Vertébrés (Rawls et al. 2004). Par exemple, Lee et Mazmanian (2010) ont documenté le rôle joué par certaines bactéries dans la différenciation des cellules régulatrices T. Finalement, la composition des communautés de Bactéries dans le tractus digestif de certains poissons a récemment été associée à des taux de croissance différentiels, qui peuvent être affectés par des facteurs tels que le rapport entre les phyla Firmicutes et Bacteroidetes (Xuemei et al. 2013).
Ces différents exemples énumérés ci-haut n'illustrent qu'une petite partie des fonctions bénéfiques qui ont été attribuées aux Bactéries. De nombreux autres rôles potentiellement bénéfiques joués par les communautés de Bactéries pour remédier à des situations de désordre métabolique de leur hôte, constituent aujourd'hui le sujet de nombreux projets de recherche, notamment sur : la maladie de Crohn (Sokol et al. 2008), le cancer (Tlaskalova-Hogenova et al. 2011), l'autisme (Cryan et al. 2012), l'obésité (Kalliomaki et al. 2008), le diabète (Amar et al. 2011) et les allergies (Kalliomaki et al. 2001).
1.2 Holobionte et hologénome
L'holobionte, décrit par Margulis (1993) et Rohwer (2002), caractérise l’association entre l'organisme hôte et l'ensemble de ses microorganismes symbiotiques. L'hologénome correspond à : l’ensemble du génome de toutes les bactéries vivant en association avec l'hôte et le génome de l’organisme hôte (Zilber-Rosenberg et Rosenberg 2008). Ainsi, le concept de l'hologénome est une vision holistique des organismes, considérant ces derniers comme des entités polygénomiques, résultant de l’interaction entre le génome de l'hôte et le
métagénome des microorganismes endogènes. La théorie de l'hologénome telle que décrite par Zilber-Rosenberg et Rosenberg (2008) repose sur trois piliers : (1) tous les Animaux et Plantes connus contiennent un microbiote spécifique; (2) le microbiote symbiotique affecte la valeur sélective de l'hôte; et (3) les microorganismes symbiotiques sont héritables (en partie).
Lorsque des variations environnementales affectent la diversité et la distribution d’abondance (c'est-à-dire la structure taxonomique) des taxons qui composent le microbiote de l'hôte, cela modifie l'hologénome. Ces variations de l'hologénome peuvent se traduire aussi par une variation du phénotype, sur lequel des forces évolutives telles la dérive génétique - la dérive est présente même s'il n'y a pas de modification du phénotype - et la sélection naturelle peuvent agir (Bordenstein et Theis 2015). Dans le contexte de l’hologénome, ces forces évolutives agissent à deux échelles : populationnelle et individuelle. Ainsi, la dérive génétique correspond à la fois à une fluctuation aléatoire, au cours des générations, des fréquences de génotypes de l’organisme hôte, ainsi que celui de sa communauté de bactéries symbiotiques (i.e. « métagénotype ») parmi les individus (échelle populationnelle) et sur la fluctuation aléatoire des fréquences des fonctions microbiennes (gènes, allèles) parmi les différents symbiontes bactériens, et ce, au cours d’une seule génération de l’organisme hôte (échelle individuelle). De même, la sélection naturelle correspond à la fois à la survie ou reproduction différentielle des différents phénotypes, incluant celui de la communauté de bactéries symbiotiques (i.e. « métaphénotype »), dans une population d’organismes hôtes (échelle populationnelle) ; et à la survie différentielle des différents symbiontes bactériens au cours d’une seule génération de l’organisme hôte (échelle individuelle). L’action de la sélection naturelle à l’échelle individuelle (e.g. la perte / gain de symbiotes bactériens clés) peut avoir un impact immédiat sur l’échelle populationnelle en diminuant / augmentant la valeur adaptative de l’holobionte. Par exemple, chez l’humain, il a été démontré que suite à l'utilisation d'antibiothérapies multiples, le phénotype de l'holobionte (via la perte de symbiontes bactériens importants), devient particulièrement susceptible à l'infection par Clostridium difficile, un agent pathogène opportuniste, potentiellement mortel dans ce contexte (Austin et al. 2014).
Le concept de l'hologénome est appuyé par les études récentes, qui mettent en évidence la communication hôte-bactérie et les interactions entre génome de l'hôte et l'ensemble des génomes bactériens, ce qui souligne l'action coordonnée et la co-dépendance de l'hôte et sa communauté de bactéries symbiotiques. En effet, les bactéries communiquent avec l'hôte par détection du quorum et par des molécules se trouvant à la surface cellulaire, ce qui affecte directement la réponse des cellules de l'hôte (Gonzalez et Venturi 2013; Hughes et Sperandio 2008; Karlsson et al. 2012; Stevens et al. 2012). Les interactions fonctionnelles entre le génome de l'hôte et l'ensemble du génome bactérien ont été mises en évidence par Backhed et al. (2004) et Rawls et al. (2006), en documentant le changement d'expression des gènes de l'hôte chez des animaux gnotobiotiques (dépourvus de microbiote; stériles) suite à l'inoculation avec un microbiote normal.
Récemment, de nouvelles avancées ont montré que les interactions entre le génome de l'hôte et du génome bactérien étaient encore plus complexes: l'hôte a la capacité de moduler les fonctions exprimées par son microbiote et vice-versa. Ce phénomène s'opère par l'entremise de microARNs, de courtes séquences d'ARN synthétisées par l'hôte ou son microbiote, qui ont la capacité de moduler l'expression d'un autre gène (Liu et al. 2016; Benmoussa et al. 2016). Chaque microARN synthétisé par le microbiote est constitué d'une séquence particulière qui lui permet de s'hybrider spécifiquement avec un brin d'ARNm synthétisé par l'hôte, ce qui permet d'inhiber la traduction de la protéine (si la séquence d'ARNm code une protéine) ou sa fonction (s'il s'agit d'un ARN ribosomique, par exemple). 1.3 Microbiote, microbiome et métagénome
Ces dernières années, l'explosion du nombre de recherches sur les dynamiques des communautés de bactéries a mené à l'apparition d'un nouveau domaine de recherche : l'écologie microbienne. Ce domaine, toujours en pleine expansion, se concentre principalement sur deux branches. Premièrement, une attention particulière est portée sur les rôles bénéfiques, commensaux ou pathogéniques joués par les communautés de bactéries environnementales ou associées à des hôtes. Deuxièmement, une emphase est aussi mise sur l'identification et l'étude des facteurs biotiques et abiotiques qui régulent la structure taxonomique et le profil fonctionnel des communautés de Bactéries. Ces dernières
années, de nouveaux termes spécifiques au domaine de l'écologie microbienne ont commencé à être utilisés : microbiote, microbiome et métagénome. Un microbiote correspond à une communauté écologique de microorganismes commensaux, symbiotiques et pathogéniques (Peterson et al. 2009). Ainsi, le microbiote ne fait aucune discrimination quant aux profils des associations des Bactéries envers les hôtes: autant les bactéries symbiotiques, que commensales et pathogènes sont inclues dans le microbiote. Lorsque mentionnée dans ce document, la structure taxonomique du microbiote réfère à l'abondance relative des différents taxa bactériens d'une communauté bactérienne. Le terme microbiome, quant à lui, réfère au génome collectif de tous les symbiontes bactériens vivant dans un certain environnement (Turnbaugh et al. 2007) ainsi qu'aux conditions abiotiques de l'environnement (e. g. type de tissu de l'organisme hôte étudié).
Bien qu'il peut souvent être utile de s'intéresser à un gène particulier d'une souche bactérienne (e.g. dans le cas d'un gène conférant une résistance aux antibiotiques chez une souche pathogénique de Clostridium difficile), l'écologie microbienne adopte souvent une approche plus holistique des microbiotes, en s'intéressant surtout à l'ensemble des gènes présents dans une communauté plutôt qu'à un gène particulier. Ainsi, l'ensemble des gènes d'une communauté bactérienne est définie par le terme «métagénome» - et son étude, par le terme «métagénomique».
L'étude de la structure taxonomique des microbiotes date de longtemps. Autrefois, les échantillons environnementaux étaient mis directement en culture, et les différentes souches qui arrivaient à croitre étaient caractérisées par leur morphologie et leurs capacités physiologiques (revue dans Amann et al. 1995). Plus tard, la technique du "Denaturating Gradient Gel Electrophoresis" - plus souvent appelée par son acronyme : DGGE - (Muyzer et al. 1993) a été développée, qui ne nécessitait pas de mise en culture, mais qui reposait sur un profil d'électrophorèse de gènes bactériens tels que le gène de la sous-unité 16S de l'ARN ribosomique. Semi-quantitative, cette technique ne pouvait donner qu'une approximation de la structure taxonomique des échantillons. Aujourd'hui, avec le développement d'outils de séquençage puissants, la méthode la plus efficace pour caractériser les communautés bactériennes est le séquençage de métagénomes. Deux
approches sont possibles: (1) le séquençage "shotgun" qui séquence l'ensemble des gènes bactériens, et qui permet donc de caractériser à la fois la structure taxonomique et fonctionnelle des communautés bactériennes (Huson et al. 2007); et (2) le séquençage de librairies d'amplicons (ensemble de courtes séquences d'ADN amplifiées) du gène de la sous-unité 16S de l'ARN ribosomique, qui permet de caractériser la structure taxonomique des communautés bactériennes (Schloss et al. 2009).
Le gène de la sous-unité 16S de l'ARN ribosomique est spécifique aux Bactéries et Archées. Ce gène marqueur est particulièrement intéressant puisqu'il contient des régions conservées ainsi que certaines régions hypervariables. Les régions conservées facilitent la conception et l'hybridation d'amorces pour l'amplification par PCR. De plus, elles permettent d'obtenir une grande profondeur de séquençage, puisque la plupart des souches bactériennes pourront être documentées à partir d'une même paire d'amorces universelles. Quant à elles, les huit régions hypervariables (désignées par V1-V8) permettent de discriminer les séquences appartenant à différents genres bactériens (Ashelford et al. 2005). Les régions V3-V4 sont des régions hypervariables permettant une discrimination puissante des genres bactériens chez les microbiotes associés aux Vertébrés (Werner et al. 2012). Lorsque mentionnés plus loin dans ce document, les métagénomes caractérisés lors de notre expérience ont tous été obtenus par une approche par amplicons du gène de la sous-unité 16S de l'ARN ribosomique.
1.4 Recrutement du microbiote: transmission verticale et horizontale
La transmission verticale du microbiote correspond à la transmission de symbiontes bactériens de générations en générations. Elle a été documentée par Ley et al. (2005), qui ont montré que les Humains héritaient du microbiote de leur mère. Plus récemment, certaines observations chez le Discus (Symphysodon aequifasciata, Teleostei) suggèrent qu'il y a aussi transmission verticale de symbiontes bactériens chez les Poissons, et que certaines de ces souches jouent des rôles structuraux importants dans la communauté intestinale des juvéniles (Sylvain et Derome 2017). Malgré une transmission verticale
efficace, de la variation est nécessairement observée entre le microbiote parental et celui de la progéniture, dû à de nombreux facteurs environnementaux (e.g. structure taxonomique du bactérioplancton environnemental) qui influencent le recrutement des symbiontes intestinaux dans les premiers stades de vie des Poissons (Burns et al. 2016). Cette transmission de Bactéries retrouvées dans l'environnement (e.g. dans la nourriture ou dans l'eau) à des hôtes eucaryotes constitue une transmission horizontale de symbiontes.
Selon Rosenberg et al. (2010), les changements de l'hologénome se produisent lorsqu'il y a variation du génome de l'hôte ou du génome bactérien. Les variations du génome bactérien s'effectuent par l'acquisition de nouvelles lignées provenant de l'environnement, ce qui correspond à la transmission horizontale de symbiontes bactériens, qui s'effectue suite à la naissance. La transmission horizontale d'un type spécifique de symbionte est parfois vitale; certains animaux dépendent du recrutement d'une lignée de bactérie spécifique provenant de l'environnement pour leur survie. Par exemple, les Aphides sont des Insectes se nourrissant de la sève du phloème qu'ils ne peuvent assimiler que s'ils recrutent le symbionte Buchnera aphidicola (Brock 1967). D'autres études ont montré que la morphogenèse de l'organe lumineux dont dépend le Calmar Euprymna scolopes est induite par le recrutement de la bactérie Vibrio fischeri dans l'environnement (Jones et Nishiguchi 2004; Montgomery et McFall-Ngai 1994; Nyholm et McFall-Ngai 2004). Finalement, une équipe de l'Université de l'Oregon a montré qu'en l'absence de microbiote, le développement de l'intestin du Poisson zèbre Danio rerio s'arrête à certains stades de différenciation (Bates et al. 2006). Cependant, malgré l'exposition aux nombreuses lignées de Bactéries provenant de l'environnement, la diversité du microbiote est normalement limitée à seulement quelques phyla, dont les fonctions sont spécifiques à chaque espèce d’hôte, suite à des milliers d'années de co-évolution hôte-microbiote (Ley et al. 2008; Wang et al. 2015). Cette spécificité des interactions hôte-microbiote est potentiellement reliée à l'interdépendance entre les symbiontes acquis verticalement et horizontalement. En effet, le recrutement horizontal et la colonisation de certains symbiontes bactériens sont souvent dépendants de la composition du microbiote de l'hôte, qui est initialement acquis par transmission verticale (Kremer et al. 2011). Les symbiontes acquis verticalement «sélectionnent» les nouveaux symbiontes qui pourront rejoindre horizontalement le microbiote de l'hôte, par le phénomène de résistance à la colonisation (Vanderwaaij et
Vanderwaaij 1990), s'opérant par plusieurs mécanismes tels que la production d'antibiotiques par certains membres du microbiote endogène. Ainsi, la séquence d'assemblage et les interactions inter-symbiontes du microbiote de l'hôte influencent le recrutement des symbiontes environnementaux potentiels.
Lamarck affirmait que les changements du phénotype induits par l'environnement étaient héritables. Ainsi, le Lamarckisme stipule que les individus perdent les caractéristiques non utilisées et développent des caractères plus utiles pour exploiter leur environnement, ces derniers caractères devenant héritables. Bien qu'il ait été montré que l'évolution des organismes est mieux caractérisée par la théorie de la sélection naturelle du Darwinisme que par le Lamarckisme, la théorie de l’holobionte rejoint le Lamarckisme, car les symbiontes bactériens optimisant l'exploitation d'une niche particulière sont acquis puis transmis de génération en génération par transmission verticale (Palmer et al. 2007; Sylvain et Derome 2017). À ce titre, l'acquisition par transmission horizontale ou la perte d'un symbionte bactérien peut être interprétée comme une mutation, puisqu'il y a modification du métagénotype (i.e. génotype du microbiote) et, par extension, du génotype de l'hologénome (Bordenstein et Theis 2015).
1.5 Facteurs influençant la structure taxonomique du microbiote
Différents facteurs peuvent moduler la structure taxonomique du microbiote tout au long du cycle de vie. Chez les Humains, Dominguez-Belloa et al. (2010) ont montré que le mode d'accouchement affecte la composition du microbiote de nouveau-nés. Suite à la naissance, les différentes espèces de Bactéries qui constituent la communauté bactérienne du nouveau-né et celles qui sont recrutées de l'environnement plus tard dans la vie de l'enfant, interagissent de façon compétitive pour les nutriments et les sites d'attachement (Palmer et al. 2007). Ainsi, selon la théorie de l'holobionte, les communautés de bactéries et leur hôte peuvent être considérées en tant qu'un écosystème complexe et dynamique, répondant à une grande diversité de facteurs biotiques et abiotiques.
Les facteurs écologiques qui influencent le recrutement et la persistance d'un taxon dans un microbiome incluent : la communauté de bactéries environnementales potentiellement
colonisatrices, l'alimentation de l'hôte (agissant en tant que prébiotique) (Turnbaugh et al. 2009a,b; Sylvain et Derome 2017), les facteurs immunitaires modulant les interactions hôte-microbe (Macpherson et al. 2009) et les interactions inter-microorganismes du microbiote (Gall 1970). De plus, le niveau trophique (Wu et al. 2012), la saisonnalité (Gilbert et al. 2009), les interactions écologiques (Steele et al. 2011), le stade de développement (Bell et al. 1971; Hansen et Olafsen 1999; Stephens et al. 2016; Sylvain et Derome 2017), le stade de vie (Llewellyn et al. 2015) et la structure de l'intestin (Stephens et al. 2016) affectent aussi la composition du microbiote. Finalement, un facteur important qui contrôle aussi la structure taxonomique du microbiote est le génotype de l'hôte. Ceci a été démontré aussi bien pour le microbiote intestinal (Fraune et Bosch 2007; Khachatryan et al. 2008; Navarrete et al. 2012; Zhao et al. 2013) que le microbiote cutané (Boutin et al. 2014). De plus, il a été montré que l'histoire évolutive de l'hôte, faisant ici référence à la phylogénie de l'hôte, façonne aussi la composition taxonomique de son microbiote (Miyake et al. 2015). Par exemple, il a été montré que le microbiote intestinal de certains Mammifères ayant des régimes alimentaires atypiques pour leur clade, tel que le Panda (Zhu et al. 2011) et les Mammifères myrmécophages (Delsuc et al. 2013), ressemble davantage à celui d'espèces du même clade, qu'à celui d'autres espèces ayant un régime alimentaire similaire. Cette inertie phylogénique rappelle le rôle important du génome de l'hôte dans la structuration du microbiote intestinal (Soverini et al. 2016).
Spécifiquement chez les organismes aquatiques, d'autres facteurs biotiques et abiotiques modifiant la composition du microbiote ont été identifiés: la salinité (Lozupone et Knight 2007; Sullam et al. 2012 ; Llewellyn et al. 2015), le statut trophique du cours d'eau (Jones et al. 2009; Yannarell et al. 2003), la température (Thurber et al. 2009), le niveau d'oxygène dissout (Boutin et al. 2013), le pH (Chu et al. 2010; Fierer & Jackson 2006; Lindstrom et al. 2005; Sylvain et al. 2016; Thurber et al. 2009) et le niveau de certains contaminants dans l'eau (e.g. Cadmium) (Laplante et Derome 2011).
Comme il vient d'être abordé, la composition taxonomique du microbiote peut être influencée par une multitude de facteurs biotiques et abiotiques. Or, comme les Bactéries ont un temps de génération particulièrement court (quelques minutes) et qu’elles sont très sensibles aux variations environnementales, elles sont considérées comme des
biomarqueurs de choix (Shin et al. 2015). Ainsi, elles permettent de détecter une perturbation physiologique de l'hôte avant que celle-ci ne puisse être détectée par des marqueurs physiologiques provenant directement de l'hôte. Par conséquent, la caractérisation de microbiotes dans des conditions environnementales contrastées ou variables, permet d'identifier des souches microbiennes particulières dont la présence ou l’abondance varie selon telle ou telle condition. Ces biomarqueurs peuvent être spécifiques à un état de stress physiologique (Boutin et al. 2013; Sylvain et al. 2016), à un état sain, à un milieu pollué ou encore à un milieu intègre. Par exemple, Shin et al. (2015) ont identifié la surabondance du phylum Proteobacteria comme biomarqueur de dysbiose chez les humains atteints de désordres nutritionnels ou immunitaires. Chez le Tambaqui (Colossoma macropomum, Teleostei), Sylvain et al. (2016) ont identifié les genres Undibacterium et Duganella comme biomarqueurs de stress physiologiques lors d'une chute de pH.
1.6 Les Téléostéens comme modèles d'étude
Les nombreux facteurs énumérés ci-haut suggèrent que l'environnement local affecte de façon importante la composition du microbiote. Une connaissance plus approfondie de l'influence de l'environnement local sur le microbiote est néanmoins nécessaire, afin de répondre à des problématiques variées (e.g. en aquaculture, en santé humaine, en nutrition, etc.).
Afin de répondre à ce besoin, les Poissons téléostéens sont des modèles d'étude particulièrement intéressants. Les Téléostéens sont apparus il y a plus de 600 millions d'années et sont aujourd'hui représentés par plus de 28 000 espèces (Reis et al. 2004), soit près de la moitié des Vertébrés (Sullam et al. 2012). Ils englobent une large gamme de différentes physiologies, d'histoires évolutives et d'écologies (Wong et Rawls 2012). Il s'agit donc d'un groupe clé pour l'étude de la nature et de la grande variété des communautés microbiennes des Vertébrés (Nayak 2010). Les surfaces corporelles des Poissons sont colonisées par des communautés de Bactéries denses (Smriga et al. 2010; Llewellyn et al. 2015; van Kessel et al. 2011). De plus, ce groupe est intéressant puisque plusieurs niches distinctes (i.e. différents sites sur le corps) des Poissons peuvent être
échantillonnées et facilement documentées : les branchies (Llewellyn et al. 2014), le mucus cutané (Boutin et al. 2014) et les intestins (Bates et al. 2006; Rawls et al. 2006; Rawls et al. 2004). De plus, Sylvain et al. (2016), ont montré que ces différentes niches (mucus cutané et intestins) réagissaient de manière différentes à une perturbation des conditions environnementales. Cet aspect est important puisqu'il indique la possibilité d'évaluer les effets de perturbations dans des niches plus (intestins) ou moins (mucus cutané) dépendantes de l’état physiologique de l'hôte. De plus, les poissons ont l'avantage d'être facilement exposés à différentes conditions physicochimiques : ils vivent dans l'eau, qui, elle, peut être facilement modulée selon les buts de l'expérience. Finalement, la plupart des Poissons sont abondants et facilement échantillonnables en nature autant qu'en captivité, ce qui fait d'eux d'excellents sujets d'étude.
Dans un autre ordre d'idée, il est aussi important de s'intéresser aux Poissons en raison de leur grande importance dans le contexte socio-économique mondial. En effet, la FAO (2016) mentionne que 167,2 millions de tonnes de poissons sauvages ont été consommées en 2014 (données les plus récentes disponibles) sur l'ensemble de la planète, générant des revenus supérieurs à 160 milliards de dollars US. Avec l'accroissement de la population humaine, l'aquaculture devient de plus en plus nécessaire comme moyen de subsistance (FAO 2016). Il est donc crucial d'optimiser la production des systèmes aquacoles. Cette optimisation passe inévitablement par une connaissance plus approfondie des facteurs modulant le microbiote des poisons, puisque la composition du microbiote affecte le taux de croissance (Lara-Flores et al. 2003), la prise de poids (Suprayudi et al. 2017), l'assimilation de la nourriture (Bud et al. 2003) et la résistance aux maladies (Boutin et al. 2013).
1.7 Les symbiontes bactériens des Poissons
Les microbiotes chez les Poissons jouent des rôles très importants pour la survie de leurs hôtes. Le microbiote cutané est particulièrement impliqué dans la défense immunitaire (Boutin et al. 2013). En effet, le microbiote retrouvé dans le mucus cutané des Poissons
constitue la toute première ligne de défense contre les agents pathogènes opportunistes présents dans l'environnement (Trivedi 2012; Sugita et al. 2002; Boutin et al. 2013; Llewellyn et al. 2017). Quant à lui, le microbiote retrouvé dans le tractus digestif joue d'importants rôles nutritionnels. La composition du microbiote intestinal peut affecter la quantité d'énergie extraite de l'alimentation et peut jouer d'importants rôles dans le métabolisme de substrats alimentaires (Xia et al. 2014). Par exemple, le microbiote intestinal des Poissons est impliqué dans la régulation de l'absorption intestinale et du métabolisme des acides gras (Semova et al. 2012), ainsi que dans la glycolyse (Ni et al. 2014). De plus, le microbiote intestinal permet l'assimilation des nutriments, en produisant des vitamines, des acides aminés, des enzymes digestives et des métabolites essentiels (Cahill 1990; Sugita et al. 1990; Skrodenyte-Arbaeiauskiene 2000). Par exemple, certains genres bactériens tels que Brevundimonas, Aeromonas et Clostridium, sont reconnus pour dégrader la cellulose (Ley et al. 2006) et ont été documentés dans les intestins de carpes (Feng et al. 2008; Wu et al. 2012). Similairement, les genres Acinetobacter, Clostridium, Pseudomonas et Aeromonas sont des producteurs d'enzymes et ont aussi été isolés chez plusieurs espèces de Poissons (Sugita et al. 1997; Ray et al. 2012).
Selon la littérature, les symbiontes bactériens intestinaux chez les Poissons sont originellement recrutés horizontalement à partir de l'environnement (i.e. eau et nourriture) (revu par Llewellyn et al. 2014), bien que chez certaines espèces, un recrutement vertical à partir des parents est aussi observé (Sylvain et Derome 2017). Suite au recrutement des souches pionnières, des processus neutres tels la dérive et la dispersion régulent en grande partie la diversité microbienne chez les microbiotes de Poissons (Burns et al. 2016). Dans le même ordre d'idées, Stephens et al. (2016) ont montré que les communautés bactériennes associées au Danio zébré (Danio rerio) dans les premiers stades de développement de l'hôte, variaient faiblement en composition entre les individus. De plus, la composition des communautés bactériennes chez de jeunes individus était plus similaire à celle du bactérioplancton, qu'à celle du microbiote d’adultes (Stephens et al. 2016). Plus tard durant le développement, la structure taxonomique du microbiote intestinal chez D. rerio s'est différenciée du bactérioplancton et davantage de variation inter-individuelle a été observée entre les hôtes.
Transmission verticale Dérive et dispersion Transmission horizontale via la nourriture Transmission horizontale via le bactérioplancton Dérive et dispersion Maturation
Figure 1: Recrutement du microbiote aux différentes phases du cycle de vie des Téléostéens. Les numéros correspondent aux étapes du cycle de vie (en ordre croissant). Les cercles de différentes couleurs représentent différents taxa bactériens. Adaptation de la Figure 3 de Llewellyn et al. 2014.
Suite au recrutement initial du microbiote, de nombreux facteurs exogènes et endogènes, abiotiques et biotiques, peuvent affecter la structure taxonomique du microbiote intestinal des Téléostéens (revu dans Ghanbari et al. 2015; Miyake et al. 2015; Nayak et al. 2010; Sullam et al. 2012; Wong et Rawls 2012; Llewellyn et al. 2015; Sylvain et al. 2016, 2017): la phylogénie de l'hôte, l'espèce, le génotype, la structure de l'intestin, le régime alimentaire, le stade de développement, les paramètres physicochimiques de l'eau environnante et la composition du bactérioplancton environnemental. Dans le même ordre d'idées, il a été montré que certaines conditions provoquaient une dysbiose (i.e. un débalancement) du microbiote : un milieu anoxique (Boutin et al. 2013), l'exposition à des désinfectants dans l'eau (Mohammed et Arias 2015), une alimentation inappropriée (Green et al. 2013) et une baisse de pH (Sylvain et al. 2016). Ainsi, ces résultats suggèrent que la structure taxonomique complexe du microbiote des Poissons est affectée par une multitude de facteurs indépendants, de nature biotiques et abiotiques.
1.8 Problématique et objectif principal
Comme mentionné précédemment, les Poissons sont d'excellents modèles pour l'étude des facteurs influençant la structure taxonomique des microbiotes. De plus, il a aussi été noté qu'il est important de s'intéresser à la régulation du microbiote des Poissons en raison de leur grande importance socioéconomique. Bien que plusieurs des facteurs régulant le microbiote aient déjà été identifiés, très peu d'études se sont intéressées à quantifier simultanément l'influence de plusieurs paramètres physicochimiques sur la structure taxonomique des microbiotes associés aux Téléostéens. De plus, les quelques études réalisées jusqu'à maintenant se sont concentrées sur des espèces de milieux tempérés, particulièrement sur la Carpe (Li et al. 2013, 2015; Ye et al. 2014; Wu et al. 2013; Wu et al. 2012; van Kessel et al. 2011; Ni et al. 2014) et sur les Salmonidés (Wong et al. 2013; Ingerslev et al. 2014a, 2014b; Zarkasi et al. 2014; Boutin et al. 2013, 2014; Llewellyn et al. 2015, 2017; revu dans Llewellyn et al. 2014). Cependant, très peu d'études se sont intéressées au microbiote de la faune ichthyenne néotropicale, comprenant plus de 4500 espèces (Reis et al. 2004) et représentant l'assemblage de Poissons le plus diversifié (i.e. comprenant le plus grand nombre d'espèces) et le plus riche (i.e. représentant la plus importante biomasse ichthyenne) au monde.
Pour palier cette lacune, l'objectif principal de la recherche faisant l'objet de ce mémoire était de caractériser l'influence simultanée de plusieurs facteurs biotiques et abiotiques sur la structure taxonomique du microbiote de Téléostéens néotropicaux, en milieu naturel. Les résultats répondant à cet objectif ont diverses applications possibles, notamment en écologie, en aquaculture et en protection des stocks de Poissons en milieu naturel.
En utilisant certaines informations sur l'écologie des espèces étudiées (régime alimentaire, distribution des populations, mode de vie) qui est déjà présente dans la littérature, il sera possible de faire des liens/corrélations entre les structures taxonomiques des microbiotes documentées et certains traits écologiques spécifiques à ces Téléostéens. Cela permettra de
vérifier à quel point les données récoltées sur le microbiote peuvent prédire des traits écologiques d'un poisson. Par exemple, si des études antérieures ont montré qu'une espèce était sédentaire, nous pourrions inférer ce trait écologique à partir de nos données récoltées sur le microbiote si la variation inter-populationnelle de composition du microbiote observée chez cette espèce est plus élevée que celle d'espèces reconnues comme étant migratrices.
Les résultats obtenus permettront aussi d’identifier les paramètres de l'eau qui influencent le plus la structure des communautés bactériennes chez les Téléostéens. Cette information pourrait être utilisée pour cibler des paramètres clés de l'eau avec lesquels il faut intervenir pour optimiser / moduler les communautés de bactéries chez les poissons d'aquaculture. Par exemple, on pourrait vouloir modifier le ratio des bactéries appartenant aux phyla Firmicutes/Bacteroidetes, puisque ce ratio affecte directement le gain de poids des Poissons (Li et al. 2013). À cet effet, une étude a d'ailleurs montré qu'en modifiant le pH de l'eau, le ratio Firmicutes/Bacteroidetes augmente dans le microbiote intestinal du Tambaqui (Colossoma macropomum), un poisson d'aquaculture au Brésil (Sylvain et al. 2016), suggérant la possibilité d'utiliser certains paramètres physicochimiques comme modulateurs de la structure taxonomique du microbiote chez d'autres poissons d'aquaculture dans le futur.
De plus, les résultats nous donneront de l'information sur les genres bactériens (bénéfiques, commensaux et pathogènes) dont l'abondance est corrélée avec certains paramètres physicochimiques spécifiques. Cette information pourrait permettre de prédire l'effet de certaines perturbations sur l'état de santé général ou la résistance immunitaire des poissons -- qui sont tous les deux en lien avec l'homéostasie des microbiotes associés aux poissons (Boutin et al. 2013). Par exemple, lorsque le pH d'un cours d'eau diminue suite à une série de pluies acides, nous pourrions prédire quels seront les genres bactériens endogènes les plus affectés dans l'intestin de poissons exploitants ce cours d'eau. Nous pourrions, à partir des mêmes résultats, identifier certains taxa agissant en tant que biomarqueurs microbiens. Tel que spécifié précédemment, ces biomarqueurs peuvent être utilisés pour discriminer
l'état de santé des poissons (i.e. en santé/microbiote équilibré ou en dysbiose/microbiote déséquilibré) d'un certain type de cours d'eau (i.e. sain ou pollué).
1.9 Milieu d'étude
Notre objectif principal était de caractériser l'influence simultanée de plusieurs facteurs biotiques et abiotiques sur la structure taxonomique du microbiote de Téléostéens néotropicaux, en milieu naturel. Les facteurs biotiques sélectionnés comprenaient : l'espèce, le régime alimentaire, le mode de vie (sédentaire ou nomade). Les facteurs abiotiques comprenaient le site d'étude, ainsi que 10 paramètres physicochimiques de l'eau: pH, dureté, [Calcium], [Sodium], [Potassium], [Carbone organique dissout], [Chlore], Température, [Oxygène dissout] et [Magnésium]. Ainsi, nous avions besoin de trouver un milieu d'étude comprenant plusieurs sites en milieu tropical, nous permettant d’échantillonner plusieurs individus de plusieurs espèces de Téléostéens, ayant des régimes alimentaires contrastés, des modes de vie différents, et exploitant différents environnements adjacents. De plus, les différents sites devaient représenter une large gamme de variations au niveau des paramètres physicochimiques de l'eau. Finalement, les espèces échantillonnées devaient être retrouvées dans tous les environnements, quelque soit les paramètres physicochimiques de l'eau. Le milieu d'étude sélectionné pour notre projet de recherche est un des rares endroits au monde répondant à toutes ces conditions : le bassin de l'Amazone.
Le bassin de l'Amazone, avec une longueur de 6992 km et couvrant plus de sept millions de kilomètres carrés, est le plus grand bassin versant au monde. L'âge de cet écosystème, sa position près de l'équateur, sa grande diversité d'habitats et de niches écologiques en font l'écosystème avec la plus grande diversité de Poissons sur la planète (Junk et al. 2007). Aujourd'hui, plus de 3000 espèces - appartenant à plus de 60 familles (Reis et al. 2004) - ont été documentées dans l'Amazone et de nouvelles espèces sont découvertes chaque année. La diversité des habitats, les conditions extrêmes (hypoxie, température élevée, pH acide) et la compétition interspécifique ont poussé les espèces de l'Amazonie à développer
des adaptations particulières (Fink et Fink 1979; Val et Almeida-Val 1995). Ces espèces ont une plasticité phénotypique (capacité d'un génotype à produire différents phénotypes permettant aux individus de persister dans un écosystème en perpétuel changement (Price et al. 2003)) particulière qui leur permet de survivre dans des environnements très contrastés, notamment dans les deux principaux types d'eau pouvant être distingués en Amazonie: l'eau blanche et l'eau noire (Lima and Ribeiro 2011). L'eau blanche, eutrophique, est riche en sédiments et en ions, et a un pH neutre (Sioli 1984). Elle provient de l'écoulement des montagnes, où il y a beaucoup d'érosion. L'eau noire contient moins de sédiments, est pauvre en ions, mais est plus riche en acides humiques dissouts provenant de la décomposition des feuilles dans l'eau - relâchant une grande quantité de tannins et d'acides humiques - ce qui la rend plus acide (pH 3.8-5) (Furch et Junk 1997). Bien que ces deux types d'eau présentent des habitats très contrastés, quelques espèces de poissons, tels le Piranha noir (Serrasalmus rhombeus) et le Cichlidé drapeau (Mesonauta festivus) sont ubiquitaires dans les deux types d'eau (Pires et al. 2015).
1.10 Études antérieures et objectifs détaillés
Bien que des traits écologiques (mode de vie, régime alimentaire, distribution génétique des populations) de plusieurs espèces de Téléostéens du bassin de l'Amazone aient été abondamment documentés, les microbiotes qui leur sont associés n'ont jamais été étudiés. À notre connaissance, jusqu'à aujourd'hui aucune étude n'a été réalisée sur le microbiote de Téléostéens sauvages du bassin de l'Amazonie. Dans le domaine de l'écologie microbienne, seulement deux études utilisant une approche métagénomique ont été réalisées dans le bassin de l'Amazone. Celles-ci portaient sur la caractérisation du bactérioplancton (bactéries pélagiques) retrouvé dans les eaux blanches et les eaux noires (Ghai et al. 2011; Peixoto et al. 2011).
Tel qu'énoncé précédemment, l'objectif principal de notre étude était de caractériser l'influence simultanée de plusieurs facteurs biotiques et abiotiques sur la structure taxonomique du microbiote de Téléostéens néotropicaux, en milieu naturel. Cette étude a
été divisée en deux chapitres (qui font chacun l'objet d'article soumis à des journaux scientifiques):
1.10.1 Objectif général du chapitre 2: vérifier comment les structures taxonomiques des microbiotes cutanés et intestinaux de trois espèces de Poissons téléostéens du bassin de l'Amazone corroborent certains traits écologiques et l'histoire phylogénétique de leurs hôtes.
Mise en contexte de l'objectif général du chapitre 2 : Plusieurs études antérieures suggèrent que les structures taxonomiques des microbiotes sont corrélées avec les traits écologiques des Poissons. Les résultats obtenus par Ley et al. (2008) et Ward et al. (2009) suggèrent que la diversité de bactéries intestinales diffère significativement entre niveaux trophiques. Leurs résultats indiquent que la diversité est relativement faible chez les carnivores, plus élevée chez les omnivores et encore plus élevée chez les herbivores. En effet, le tube digestif des herbivores est généralement plus long et plus complexe que celui des carnivores, offrant potentiellement davantage de niches pour les symbiontes intestinaux et notamment pour ceux qui sont essentiels à la dégradation de la cellulose (Buddington et al. 1997). Concernant la variable «espèce», Ley et al. (2008) ont montré que la structure taxonomique du microbiote est relativement conservée au sein d'une espèce, avec des différences interindividuelles importantes. Les différences inter-individus sont telles que Tap et al. (2009) ont montré que chez l’Humain, 78.6% des unités taxonomiques opérationnelles (OTUs) étaient spécifiques à chaque individu. Une étude menée par Turnbaugh et al. (2009a) sur les microbiotes de jumeaux est arrivée à un résultat similaire, ce qui suggère que malgré l'influence du patrimoine génétique de l'hôte, une grande part du microbiote est acquise par transmission horizontale à partir des bactéries retrouvées dans l'environnement exploité par l'hôte. Ainsi, puisque l'environnement joue un rôle important dans la structuration des microbiotes, nous nous attendions à ce que la structure taxonomique des microbiotes cutané et intestinal chez les Poissons de l'Amazone corrobore les différents «modes de vie» : sédentaire (exploitation d'un seul habitat) ou nomade (exploitation de plusieurs habitats dans le temps).
1.10.1.1 Sous-objectif 2.1: caractériser l'impact de 3 variables biotiques (espèce, régime alimentaire et mode de vie) sur la structure taxonomique du microbiote cutané et intestinal de trois espèces de Poissons du bassin Amazonien.
Spécifications sur la méthode : Nous avons échantillonné et caractérisé les structures taxonomiques des microbiotes de trois espèces différentes de Poissons, ayant des régimes alimentaires et des mode de vie contrastés. Les trois espèces sont: le Cichlidé drapeau (Mesonauta festivus, détritivore et sédentaire), le Pacu (Mylossoma duriventre, planctivore et plutôt nomade) et le Piranha noir (Serrasalmus rhombeus, piscivore et nomade).
1.10.1.2 Sous-objectif 2.2: évaluer l'impact d'une variable abiotique (site d'étude) sur la structure taxonomique des microbiotes cutanés et intestinaux de trois espèces de Poissons du bassin Amazonien.
Spécifications sur la méthode: Nous avons échantillonné et caractérisé les structures taxonomiques des microbiotes des trois espèces de Poissons à cinq sites d'étude, tous des sites d'eau blanche situés à proximité de Manaus (AM), Brésil.
1.10.1.3 Sous-objectif 2.3: comparer les variations de composition inter-sites d'échantillonnage des deux niches microbiennes étudiées (mucus cutané versus intestins) chez trois espèces de Poissons du bassin Amazonien. Cela permet d’identifier les traits écologiques de l’organisme hôte qui peuvent être spécifiquement inférés à partir de chaque niche microbienne étudiée.
Spécifications sur la méthode : Sur chaque poisson pêché, le mucus cutané a été prélevé au site d'échantillonnage. Ensuite, tous les poissons ont été transportés sur glace jusqu'au laboratoire où leurs intestins complets ont été prélevés par dissection. Ainsi, les communautés de bactéries cutanées et intestinales ont toutes deux été échantillonnées et caractérisées sur chaque poisson. Des observations faites sur le Tambaqui (Colossoma macropomum), soumis à deux régimes de pH (Sylvain et al.