HAL Id: hal-02826684
https://hal.inrae.fr/hal-02826684
Submitted on 7 Jun 2020
HAL is a multi-disciplinary open access
archive for the deposit and dissemination of
sci-entific research documents, whether they are
pub-lished or not. The documents may come from
L’archive ouverte pluridisciplinaire HAL, est
destinée au dépôt et à la diffusion de documents
scientifiques de niveau recherche, publiés ou non,
émanant des établissements d’enseignement et de
Microbiologie prévisionnelle. Estimation du paramètre
de croissance maximum à partir des données de
l’appareillage BIOSCREEN
Jean-Pierre Gauchi
To cite this version:
Jean-Pierre Gauchi. Microbiologie prévisionnelle. Estimation du paramètre de croissance maximum
à partir des données de l’appareillage BIOSCREEN. [Rapport Technique] RT2001-5, 2001.
�hal-02826684�
Institut National de la Recherche Agronomique
Centre de Recherche de Jouy-en-Josas
Unité de Biométrie / Equipe MatRisq
Microbiologie prévisionnelle
Estimation du paramètre de croissance maximum
à partir des données de l’appareillage BIOSCREEN
Jean-Pierre Gauchi
Jean-Pierre.Gauchi@jouy.inra.fr
Rapport technique 2001-5, 185 pp.
Sommaire
Introduction
………...………..……...…p. 4
Première Partie : Dénombrements
……….…p. 6
I . Description des processus de dilution et du mode opératoire BIOSCREEN
……p. 7
II. Estimation des variances des différentes sources d’erreur
………….…………..………..p. 16
II.1 Estimation des variances d’erreur de pipetage
…….……….……….……….…p. 16
II.2 Estimation des variances d’erreurs de comptage
……...…….……….…………..……...…p. 19
II.3 Estimation des variances d’erreurs de manipulation
……...……….…p. 20
III. Calcul et estimation de l’espérance et de la variance du nombre et de la
concentration de bactéries dans un tube D
1
……….…….……..……p. 22
III.1 Quelques formules fondamentales utiles
………….………..…….…..….….…p. 23
III.2 Espérance et son estimation du nombre de bactéries dans un tube D
1
…………p. 26
III.3 Variance et son estimation du nombre de bactéries dans un tube D
1
…….…..…p. 31
III.4 Espérance et variance de la concentration en bactéries dans un tube D
1
………p. 36
IV. Etude de la sensibilité de la variance des estimations du nombre de bactéries
dans un tube D
1
.……….…p. 38
V. Calcul et estimation de l’espérance et de la variance du nombre et de la
concentration de bactéries dans les tubes D
2
à
D8
……….…….…….…p. 40
V.1 Tube D
2
………..…….….….…..p. 40
Deuxième Partie : Estimation du paramètre
µ
max
………...…….…p. 45
VI. Temps de détection
……….……….…p. 45
VI.1 Les courbes Bioscreen
………..…….…………..……….….…p. 45
VI.2 Calcul des temps de détection par régression inverse
………..………...…p. 46
VI.3 Variances, intervalles de confiance et poids des temps de détection
……….….…p. 47
VII. La méthode IDRLS
……….……….…p. 49
VII.1 Principe de la méthode IDRLS
………..……...……….….…p. 49
VII.2 Régression robuste avec l’algorithme IRLS
………..………...…p. 50
VII.3 Détails de la méthode IDRLS
………..……….…p. 53
VII.4 Algorithme de la méthode IDRLS
………. ……….….…p. 57
VII.5 Etude empirique de la robustesse de la méthode IDRLS
………. …….………….….…p. 58
VIII. Estimation du paramètre
µ
maxpar une méthode de moyenne des temps
de génération
……….……….…p. 66
Troisième Partie : Résultats
……….…p. 68
IX. Résultats en fonction du %NaCl
………..………p. 69
X. Résultats en fonction du pH
………….………..…………p. 80
Conclusion
………..………...………p. 83
Annexes
…..…………..……….……….………p. 86
Annexe 1 : Courbes BIOSCREEN %NaCl à partir des données E. Coli de l’ADRIA Quimper
Annexe 2 : Courbes BIOSCREEN %NaCl à partir des données Listeria de l’ADRIA Quimper
Annexe 3 : Courbes BIOSCREEN %NaCl à partir des données L.M 118 III et S. Enteridis
de l’ADRIA Quimper
Annexe 4 : Courbes BIOSCREEN %NaCl à partir des données Listeria de IPL
Annexe 5 : Courbes BIOSCREEN %NaCl à partir des données E. Coli ECF-187 de IPL
Annexe 6 : Courbes BIOSCREEN pH à partir des données E. Coli de l’ADRIA Quimper
Annexe 7 : Estimations des
µ
max
obtenues à partir des données de l’ADRIA Quimper
Annexe 8 : Estimations des
µ
max
obtenues à partir des données de l’IPL
Introduction
Ce rapport technique représente la contribution principale de l’équipe MatRisq de l’Unité de
Biométrie de Jouy-en-Josas au projet PREVIUS, sur l’année 2001. Il s’adresse principalement
aux microbiologistes de la cellule opérationnelle de ce projet. On rappelle que l’objectif majeur
de PREVIUS est la constitution d’une base de données sur la microbiologie prévisionnelle dans
le domaine des aliments. Cette base est renseignée à la fois par des données issues de la littérature
et des données issues d’expériences en laboratoire réalisées par les partenaires de PREVIUS
(ADRIA-Quimper, Institut PASTEUR de Lille, INRA de Lille, ENV de Maisons-Alfort, le
Groupe industriel Danone, la SOREDAB, le CTSCCV).
Le problème statistique principal qui nous intéresse ici est l’estimation (ponctuelle et par
intervalles de confiance) d’un paramètre particulier : « pente maximum de la courbe de
croissance bactérienne », paramètre noté
µ
max. Pour atteindre cet objectif il nous faut dans un
premier temps résoudre le problème de l’estimation de la variance du nombre de bactéries
présentes dans un tube d’une solution bactérienne diluée. Ce travail fait l’objet de la première
partie de ce rapport, il met en jeu des données issues de comptages de colonies sur boites de Pétri
fournies par IPL.
Dans la deuxième partie de ce rapport on utilise des courbes de cinétique de croissance obtenues
avec l’appareillage BIOSCREEN (fournies par IPL et ADRIA-Quimper). Celui-ci permet de
réaliser rapidement des cinétiques de croissance bactérienne en tubes dilués et sur lesquels
l’évolution de cette croissance est suivie quasiment en continu par la mesure de la densité
optique. On expose et on compare plusieurs méthodes de régression avant d’en proposer une bien
adaptée à l’estimation de
dans un tel contexte. La troisième partie est consacrée aux résultats
des méthodes exposées dans les parties I et II appliquées aux micro-organismes Listeria
Monocytogenes, Escherichia Coli et Salmonella Enteridis.
max
Un avantage majeur du BIOSCREEN est qu’il permet d’étudier des conditions
environnementales de pH et d’activité de l’eau Aw variables. Ainsi, la suite attendue de cette
étude est l’élaboration de modèles dits secondaires (non linéaires au sens de la régression)
représentant l’évolution du
en fonction de ces conditions environnementales. Ces modèles
secondaires sont caractérisés par des paramètres cardinaux : pH
min
, pH
max
, pH
opt
, Aw
min
,
Aw
max
, Aw
opt
, T
min
, T
max
, T
opt
dont les valeurs dépendent du micro-organisme étudié. Ce
travail fera l’objet d’un rapport ultérieur.
max
Première Partie : Dénombrements
L’objectif de cette partie est l’estimation (ponctuelle et par intervalles de confiance) du nombre et
de la concentration en bactéries dans un tube dilué, à partir de comptages de colonies sur boites
de Pétri. Les variances de ces nombres et concentrations nous permettront de disposer de poids à
affecter aux données intervenant dans les méthodes de régression de la deuxième partie.
La difficulté de ces estimations est due à l’existence de plusieurs sources d’erreur :
- erreurs de dilution (qu’on appellera aussi erreurs de pipetage),
- erreurs d’échantillonnage,
- erreurs de comptage des UFC (unités formant colonies) sur les boites de Pétri,
- erreurs humaines de manipulation, typiquement lors de l’étalement d’une petite quantité
de solution microbienne sur la boite de Pétri .
Les erreurs de dilution et d’échantillonnage jalonnent tout le processus de la préparation des
solutions bactériennes, depuis la préparation des solutions microbiennes primaires jusqu’à la
solution prête à étaler. Avant d’exposer les calculs statistiques (chap. III) il nous faut donc faire
une description détaillée du processus de dilution ainsi que du mode opératoire de l’appareillage
Bioscreen d’une part (chap. I), et expliquer comment obtenir des estimations des variances de ces
différents types d’erreur d’autre part (chap. II).
I . Description des processus de dilution et du mode opératoire Bioscreen
On décrit ci-après ces processus de dilution et ce mode opératoire à partir des informations
recueillies au sein du groupe de travail PREVIUS ; les aspects purement biologiques de l’étape 1
ne seront qu’évoqués. On introduit au fur et à mesure les notations pour les tubes et les volumes
qui nous seront utiles par la suite. Ce mode opératoire se décompose en sept étapes :
- étape 1 : préparation du tube primaire P,
- étape 2 : préparation du tube D
1,
- étape 3 : dénombrement dans le tube D
1,
- étape 4 : préparation des tubes D
2à D
8,
- étape 5 : ensemencement des microplaques,
- étape 6 : mesure de la densité optique en continu,
- étape 7 : calculs,
étapes que nous développons maintenant.
Etape 1 : préparation du tube primaire P
On part du stock d’une souche d’une espèce E (Listeria monocytogenes, E. Coli, …) conservé à –
80°C duquel on prélève une microbille d’inoculum que l’on place dans un tube rempli d’un
volume V
Pde milieu nutritionnel BHI adapté, à température ambiante. Ce tube est mis en
incubation à une température T dépendant de l’espèce E, pendant une durée ∆t. Ensuite un
repiquage est effectué à p% et ce deuxième tube est mis à incuber à une température T’ pendant
une durée ∆t’. On obtient ainsi un tube primaire P de concentration C
P, correspondant à un
Les valeurs usuelles pour les différentes grandeurs (mais dépendant de l’espèce E) sont :
- V
P= 10 ml,
- T = 30°C,
-
∆t = 8 heures,
- p% = 1% ou 0.1%,
- T’ = 30°C,
-
∆t = 16 ou 24 heures,
- C
P≈ 10
9bactéries/ml.
Remarque :
En réalité la valeur de C
Pest très mal connue, elle peut différer largement de 10
9bactéries/ml.
Etape 2 : préparation du tube D
1On remplit n tubes à essais avec V
1ml de BHI pour n
1d’entre eux (tubes T
1à Tn
1
) et V
2ml de
BHI pour les n
2autres (tubes Tn
1+1à
Tn
1+n
2). Ce faisant on fait des erreurs de pipetage quand on
place soit V
1ml, soit V
2ml dans les tubes. Ces erreurs, de nature aléatoire, sont évidemment
différentes selon les tubes mais leurs variances sont également différentes selon que l’on place V
1ou V
2: les volumes réels placés dans ces tubes lors d’une expérience donnée sont donc inconnus.
Ensuite, on pipette v
Pml dans le tube P que l’on place dans le tube T
1, le tube T
1devient le tube
: ce faisant on fait encore une
' 1
T
erreur de pipetage (on prélève en fait un volume inconnu proche
de v
Pml,) et une erreur d’échantillonnage (on prélève en fait un nombre de bactéries différent du
nombre attendu v
P/V
P). Notons d
P= v
P/(V
1+ v
P) le facteur de dilution. S’il y avait exactement N
Pbactéries
dans P et s’il n’y avait pas d’erreurs on aurait maintenant dans
T
1':
-
'=
N
Pv
P/V
P= C
Pv
Pbactéries,
1T
N
- un volume de solution égal à
'= V
1+ v
P,
1T
V
- une concentration théorique (nombre de bactéries par ml) de
'=
/
= C
Pd
P.
1 T
C
' 1 TN
' 1 TV
On opère ensuite par dilutions en cascade. Raisonnons avec la valeur usuelle de 4 pour n
1. A
partir de
on pipette v
Tque l’on place dans T
2où se trouvent déjà V
1ml de BHI, le tube T
2devient le tube
; puis de
on prélève v
Tque l’on place dans T
3où se trouvent déjà V
1ml de
BHI, le tube T
3devient le tube
, et enfin de
on prélève v
Tque l’on place dans T
4où se
trouvent déjà V
1ml de BHI, le tube dans T
4devient le tube
. En théorie
,
et
devraient
donc contenir maintenant un volume de BHI exactement égal à
= V
1+ v
T, (i = 2, 3, 4) en
négligeant le volume occupé par les bactéries dans les tubes, et de concentrations respectives
'2
T
C
,
e
. En réalité, on fait à chaque cascade des erreurs de pipetage et d’échantillonnage. La
concentration réelle dans le tube T n’est donc pas C . Pour évaluer cette concentration on opère
comme à l’étape 3 ci-dessous.
' 1
T
' 2T
T
2' ' 3T
T
3' ' 4T
T
2'T
3'T
4' ' i TV
' 3 TC
t
4 T4 ' 4 TC
Les valeurs usuelles pour les différentes grandeurs sont :
- n = 11,
- V
1= 9 ml,
- n
1= 4,
- V
2= 5 ml,
- n
2= 7,
- v
P= 1 ml,
- v
T= 1 ml (c’est-à-dire une dilution au dixième),
-
,
et
= 10
7,
10
6et 10
5bactéries/ml, respectivement, si on suppose 10
9bactéries/ml dans le tube P.
' 2 TC
' 3 TC
' 4 TC
Etape 3 : dénombrement dans le tube D
1On appelle maintenant le tube
le tube D
1, contenant un nombre inconnu de bactéries N
D1, un
volume inconnu V
D1=
et de concentration inconnue C
D1=
. Pour établir le dénombrement
N
D1on procède de la façon suivante. A partir de D
1on prélève v
1ml que l’on place dans un tube
D
1acontenant V
1ml de BHI, le tube D
1adevient le tube
. Puis à partir de
on prélève v
aml
que l’on place dans un tube D
1bcontenant V
1ml de BHI, le tube devient le tube
. La
concentration théorique dans le tube
est donc
= C
D1d
1d
1asi on note d
1et d
1ales facteurs
de dilution avec d
1= v
1/
(V
1+ v
1) et d
1a= v
a/(V
1+ v
a). Enfin, depuis
on prélève un volume v
bque l’on place, sans le diluer, dans un tube
(l’indice e pour ensemencement) duquel on prélève
indépendamment S fois v
bsml (s = 1 ,…, S) qui servent à ensemencer indépendamment S boites
de Pétri. On dépose donc en théorie
=
v
bsbactéries sur chacune des S boites b
1s(l’indice
1 de b
1sfait référence au tube D
1). En opérant ainsi le microbiologiste espère déposer environ une
centaine de bactéries sur une boite compte tenu de la valeur supposée de 10
5
pour C
D1. Au bout
de la durée adéquate il compte visuellement sur chaque boite b
1sun nombre n
1sde colonies. Si
celles-ci ne sont pas trop chevauchées et si on fait l’hypothèse que chaque bactérie déposée
conduit à une colonie (on parle alors d’unité formant colonie UFC) alors n
1sest égal au nombre
de bactéries dans le volume
déposé sur la boite b
1s. Pour remonter à la concentration C
D1qui
l’intéresse vraiment, le microbiologiste calcule d’abord la moyenne
' 4
T
' 4 TV
' 4 TC
' 1aD
D
1a' ' 1bD
' 1bD
' 1b DC
' 1bD
eD
' 1b Dn
' 1b DC
bsv
1n des comptages obtenus sur
les S boites puis obtient C
D1par C
D1=
n v d d
1 bs 1 1a. On dispose parfois de plusieurs vraies
répétitions de tubes D
1(pour une souche donnée), c’est-à-dire de tubes D
1préparés
indépendamment à partir de la microbille. On verra au chapitre III comment ces répétitions nous
seront utiles.
Remarque
En réalité, comme pour les étapes 1 et 2 des erreurs de pipetage et d’échantillonnage jalonnent
tout ce procédé de dilution et des erreurs de manipulation entachent la procédure
d’ensemencement qui se fait manuellement à l’aide d’un même râteau utilisé séquentiellement
pour étaler un volume
v
bssur une boites b
1s.
Les valeurs usuelles pour les différentes grandeurs sont :
- v
1= 1 ml,
- V
1= 9 ml,
- v
a= 1ml,
- v
b= 1 ml,
- v
bs= 0.1 ml,
- s = 1 à 5,
- n
1s
= on s’attend avec les valeurs précédentes à la valeur de 100 en supposant 10
9bactéries/ml dans le tube primaire P.
La figure 1 schématise cette méthode de dénombrement d’un tube D
1.
boite 1
boite 2
boite s
D1
(VD1 = V1+ vt)
D1a -> D'1a
(V1 -> V1+v1)
D1b -> D'1b
(V1 -> V1+va)
De
(0 -> vb)
v1
va
vb
vb1
vb2
vbs
Etape 4 : préparation des tubes D
2à D
8Pour obtenir les tubes D
2à D
8on opère en cascade comme suit. On prélève v
2ml dans le tube D
1que l’on place dans le tube T
5où se trouvent déjà V
2ml de BHI (voir étape 2) : le tube T
5devient
le tube D
2. Puis du tube D
2on prélève v
2ml que l’on place dans le tube T
6où se trouvent déjà V
2ml de BHI (voir étape 2) : le tube T
6devient le tube D
3et ainsi de suite jusqu’au tube D
8. Une
valeur usuelle pour v
2est 5 ml (dilution au demi).
Sans erreur de pipetage ni d’échantillonnage la concentration en bactéries dans le tube D
i, pour i
= 2 à 8, serait de
, en notant d = v
2/(V
2+ v
2) le facteur de dilution. En général d = 1/2
donc
, pour i = 2 à 8.
i D DC
d
C
i=
1 i D DC
C
i=
1(
1
/
2
)
Etape 5 : ensemencement des microplaques,
Une microplaque de BIOSCREEN est une plaque stérile en matière plastique et comportant 100
cupules (10 colonnes de 10). On dépose dans la première cupule de la première colonne v
cmicrolitres prélevés du tube D
1, puis v
cprélevés de D
2dans la deuxième cupule de cette première
colonne, etc … jusqu’à la huitième cupule de la première colonne où on dépose v
cprélevés de D
8.
Une valeur usuelle pour v
cest 350 µl (= 0.350 ml). Les deux dernières cupules de la colonne sont
remplies par un BHI non inoculé (témoin). On opère de la même façon pour les 9 autres
colonnes, sachant qu’il ainsi possible de faire varier une condition environnementale de colonne
en colonne. Par exemple, on peut imposer une variation du pH (ou de l’Aw) des solutions de la
colonne 1 à la colonne 10.
On peut placer deux microplaques dans l’appareil. On note que les microplaques vierges sont
rigoureusement identiques (même lot de fabrication très précise et de stérilisation commune) ce
qui fait évacuer l’hypothèse d’un effet de la plaque vierge, hypothèse qui ne sera plus toujours
valide quand on considérera plus loin les plaques ensemencées et en fin de croissance
bactérienne.
v2
D1
T5 -> D2
T11 -> D8
témoins
pH1 pH2
pH10
vc
vc
vc
v2
v2
Figure 2 : Schéma du mode opératoire de la préparation des dilutions pour une plaque de BIOSCREEN.
Etape 6 : mesure de la densité optique en continu
L’appareil BIOSCREEN est basé sur le principe que la turbidité d’une solution bactérienne
s’accentue quand la croissance bactérienne augmente au cours du temps. Corrélativement la
densité optique de cette solution évolue. Des études antérieures nombreuses ont montré une
relation linéaire de pente positive entre la densité optique et la masse bactérienne (vivante ou
morte, la distinction ne pouvant se faire dans ces conditions) dans la cupule. La relation linéaire
n’est valable que pour certaines gammes de concentration bactérienne, typiquement de 10
5à 10
9bactéries/ml ce qui est le cas des études réalisées ici.
Les paramètres usuels du BIOSCREEN sont : température de 30°C, régulée avec précision,
longueur d’onde de 600 nm, intervalle entre deux mesures optiques de 15 minutes. Avant la
Etape 7 : calculs
Dans ce rapport on part des deux méthodes de calcul utilisées couramment par les
microbiologistes que l’on compare et que l’on améliore grâce à un point de vue statistique que
l’on développe dans la deuxième partie. On se contente ici de donner le principe de ces deux
méthodes appelées méthode de la droite de régression ordinaire et méthode de la moyenne des
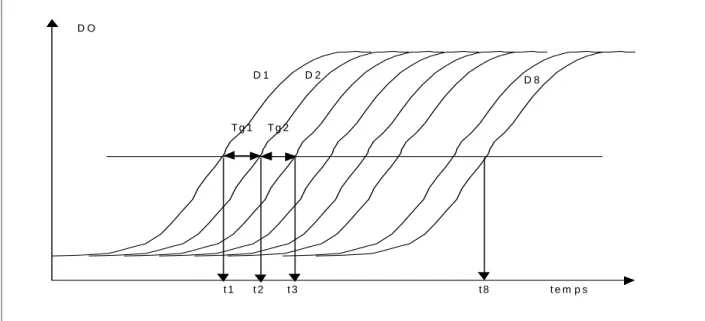
temps de génération (Tg).
méthode de la droite de régression
Le principe est de calculer une droite de régression aux moindres carrés ordinaires des
logarithmes de concentration en inoculum dans les huit tubes en fonction des huit temps de
détection repérés sur les courbes de densité optique fournies par le Bioscreen (voir Figure 3). La
pente de cette courbe fournit l’opposé du µ
maxcherché. Aucun intervalle de confiance n’est
fourni.
méthode de la moyenne des Tg
On détermine sept temps de génération pour huit courbes en calculant les différences entre deux
temps de détection. Comme on a la relation Tg = Log
e2
/
µ
maxon en déduit sept valeurs possibles
pour µ
maxdont on fait la moyenne par la suite. En réalité on verra que les valeurs aberrantes sont
souvent fréquentes parmi les huit valeurs et donc influencent fortement le µ
maxmoyen ainsi
t e m p s
t 1 t 2 t 3 t 8
T g 1 T g 2
D 1 D 2 D 8
D O
Figure 3 : Schéma de principe d’obtention des temps de détection t
iet des temps de génération Tgi sur les
courbes de densité optique en fonction du temps, issues du Bioscreen.
II . Estimation des variances des différentes sources d’erreurs
Pour mener à bien nos calculs statistiques ultérieurs nous aurons besoin d’estimation des
variances des erreurs des sources d’erreurs signalées plus haut. Ces estimations ont été possibles
grâce à des données expérimentales et des informations fournies par les microbiologistes. Les
erreurs d’échantillonnage liées à la nature de la loi de probabilité attachée au prélèvement (loi
binomiale) seront examinées au chapitre III.
II .1 Estimation des variances d’erreurs de pipetage
Dans le cadre de cette étude les microbiologistes de l’IPL nous ont fourni plusieurs répétitions de
pipetées pesées avec une balance de grande précision (tableau 1).
Opérateur 1
Pipetée de 0.1 ml
Pipetée de 1 ml
Pipetée de 5 ml
Pipetée de 9 ml
0.1000 0.98 5.02
8.97
0.0929 1.00 5.04
8.97
0.1019 0.98 5.00
8.97
0.0928 0.99 4.99
9.02
0.1000 1.00 4.98
9.00
0.1000 0.99 4.99
9.02
0.0971 1.00 5.00
9.03
0.0990 0.99 5.01
9.04
0.1051 1.00 4.98
9.01
0.0997 1.00 4.96
9.02
Moyenne = 0.0989
Variance = 1.4224×10
-5Moyenne = 0.993
Variance = 6.7778×10
-5Moyenne = 4.997
Variance = 5.1222×10
-4Moyenne = 9.005
Variance = 6.9444×10
-4Opérateur 2
0.1256 1.02 5.02
0.0982 1.01 4.99
0.0959 1.00 5.00
0.0848 1.01 5.02
0.1127 1.00 4.99
0.1017 1.01 4.99
0.1130 1.01 5.02
0.0826 1.00 5.00
0.1019 1.00 4.99
0.1082 1.00 5.00
Moyenne = 0.10246
Variance = 1.7225×10
-4Moyenne = 1.006
Variance = 4.8889×10
-5Moyenne = 5.002
Variance = 1.7333×10
-4Tableau 1 : pesées en grammes de différentes pipetées (les densités des solutions pipetées sont considérées
Ces données nous ont permis d’estimer des variances de pipetage comme on le montre
maintenant.
Pour les pipetées de 0.1, 1 et 5 ml nous avons testé au préalable l’égalité des variances des deux
échantillons (opérateurs 1 et 2) par un test usuel de Fisher-Snedecor (test F) au risque de première
espèce de 5%.
Estimation de la variance d’une pipetée d’un volume souhaité de 0.1 ml
Les variances des deux échantillons de pipetées de 0.1 ml, notées q
0.1, figurant au tableau 1, sont
déclarées significativement différentes au moyen du test F : les résultats de l’opérateur 2 sont plus
dispersés que ceux de l’opérateur 1. Mais comme dans la suite de l’expérience BIOSCREEN on
ne sait pas quel opérateur exécute telle ou telle pipetée (ce n’est pas forcément toujours le même
opérateur qui prépare toutes les solutions d’une expérience BIOSCREEN) on prendra comme
estimation de la variance de la pipetée q
0.1la valeur 9.18963×10
-5obtenue en calculant la
variance des données des deux échantillons réunis. On prendra comme estimation de l’espérance
la valeur cible de 0.1 ml.
Donc :
1
.
0
)
(
ˆ
1 . 0=
q
E
5 0.1ˆ (
)
9.18963 10
V q
=
×
−.
Estimation de la variance d’une pipetée d’un volume souhaité de 1 ml
Les variances des deux échantillons de pipetées q
1ne sont pas déclarées significativement
différentes par le test F : les résultats de l’opérateur 2 présentent une dispersion comparable à
ceux de l’opérateur 1. On choisira comme estimation de la variance de la pipetée de 1 ml la
valeur 9.9737×10
-5obtenue en calculant la variance des données des deux échantillons réunis.
On prendra comme estimation de l’espérance la valeur cible de 1 ml.
Donc :
Estimation de la variance d’une pipetée d’un volume souhaité de 5 ml
Les variances des deux échantillons de pipetées q
5ne sont pas déclarées significativement
différentes par le test F : les résultats de l’opérateur 2 présentent une dispersion comparable à
ceux de l’opérateur 1. On choisira comme estimation de la variance de la pipetée q
5la valeur
3.3132×10
-4obtenue en calculant la variance des données des deux échantillons réunis. On
prendra comme estimation de l’espérance la valeur cible de 5 ml.
Donc :
5
)
(
ˆ
5=
q
E
4 5ˆ( ) 3.3132 10
V q
=
×
−.
Estimation de la variance d’une pipetée d’un volume souhaité de 9 ml
On choisira comme estimation de la variance de la pipetée q
9la valeur 6.9444×10
-4. On prendra
comme estimation de l’espérance la valeur cible de 9 ml.
Donc :
9
)
(
ˆ
9=
q
E
4 9ˆ( ) 6.9444 10
V q
=
×
−.
Estimation de la variance d’une pipetée d’un volume souhaité de 350
µ
l
D’après les microbiologistes la pipetée de 350 µl est connue à ± 0.3125 % (valeur obtenue par
interpolation linéaire à partir de volumes encadrants) ce qui nous conduit à un intervalle de
confiance approximatif (à 95%), exprimé en ml, de [ 0.34891 ; 0.35109 ] soit une variance
estimée pour une pipetée de 0.350 ml de :
2 7 0.350
0.35109 0.34891
ˆ (
)
3.0927 10
2 1.96
V q
=
⎛
⎜
−
⎞
⎟
=
−×
⎝
⎠
×
On prendra comme estimation de l’espérance la valeur cible de 0.350 ml.
Donc :
35
.
0
)
(
ˆ
35 . 0=
q
E
7 0.35ˆ(
)
3.0927 10
V q
=
×
−.
On récapitule ces résultats dans le tableau 2.
Pipetée
E
ˆ
V
ˆ
0.1q
0.1
59.18963 10
×
− 1q
1
59.9737 10
×
− 5q
5
43.3132 10
×
− 9q
9
46.9444 10
×
− 0.35q
0.35
73.0927 10
×
−Tableau 2 : Estimations des espérances et des variances des pipetées.
II .2 Estimation des variances d’erreurs de comptage
L’erreur dont il est question ici est l’erreur faite en comptant les UFC sur la boite de Pétri. En
effet, les praticiens expérimentés de ces comptages recommandent que le nombre d’UFC sur une
boite de Pétri soit proche de 100 pour permettre un comptage facile et entaché de peu d’erreur. Il
arrive cependant que le nombre d’UFC sur le boite dépasse largement la valeur de 100 (par
exemple 200 ou 300) ce qui conduit au chevauchement plus ou moins partiel de quelques UFC et
donc à une erreur dans le comptage du nombre total d’UFC sur la boite.
estimation basée sur les informations fournies par ces praticiens, tout en envisageant trois cas
possibles :
- Dans la plage des 50-150 UFC on supposera une erreur de ±3 UFC correspondant à une
variance de comptage de 18 (estimation basée sur deux comptages, donc un seul degré
de liberté).
- Pour des nombres d’UFC supérieurs à 150 on supposera une erreur de ±6 UFC
correspondant à une variance de comptage de 72.
La variance d’erreur de comptage sera notée par la suite
V K% )
(
rsoù
K%
rsdésigne la variable
aléatoire associée au comptage sur une boite s ensemencée par une solution bactérienne préparée
à partir d’une répétition r , notée D
1r, d’un tube D
1.
II .3 Estimation des variances d’erreurs de manipulation
Ces erreurs, d’origine humaine, sont également très difficiles à apprécier. Elles proviennent
essentiellement du « geste » effectué pour étaler la solution sur la boite de Pétri : c’est
l’ensemencement. Ce geste peut être manuel, c’est la technique dite du râteau ; en outre, ce râteau
sert en général à ensemencer au moins deux boites ce qui peut propager encore une erreur sur le
comptage futur. Le geste peut également être effectué par un appareil qui dépose selon une
spirale la solution sur la boite de Pétri. A titre d’exemple, d’après les spécialistes, pour deux
boites ensemencées par étalement manuel à partir de solutions préparées selon le même protocole
(mais évidemment soumises à des erreurs de dilution et d’échantillonnage) on peut compter un
nombre d’UFC de 80 pour l’une et 120 pour l’autre. Pour les données disponibles on considérera
ainsi deux situations possibles :
- Dans la plage des 50-150 UFC on supposera une erreur de ±50 UFC correspondant à
une variance de comptage de 625 en supposant que les intervalles d’incertitude sont
assimilables à des intervalles de confiance à 95% et donc qu’ils recouvrent quatre
écart-types.
- Pour des nombres d’UFC supérieurs à 150 on supposera une erreur de ±62 UFC
correspondant à une variance de comptage d’environ 1000.
La variance d’erreur de comptage établie sur S boites sera notée par la suite
où
désigne la variable aléatoire associée aux comptages sur S boites chacune étant ensemencée par la
même solution bactérienne préparée à partir d’une répétition r d’un tube D
1. Cependant, les S
boites diffèrent également par les volumes
réalisations de la variable aléatoire
.
(
r)
V K%
K%
rbs
v
v
%
bsOn étudiera au chap. IV la sensibilité des résultats à la variation des variances de tous ces types
d’erreur.
III. Calcul et estimation de l’espérance et de la variance du nombre et de la
concentration de bactéries dans un tube D
1
Pour établir ces calculs et estimations il nous faut montrer :
-
comment les erreurs d’échantillonnage et de dilution se propagent depuis un tube D
1r,
une répétition d’un tube D
1, jusqu’au volume
v
bs,
- et comment prendre en compte les erreurs détaillées au chap. II.
Pour l’établissement des formules nous aurons besoin de formules fondamentales bien connues
rappelées au §III.1. Notre approche sera semi-analytique dans le sens où les calculs seront
essentiellement basés sur des formules analytiques mises à part quelques simulations rendues
nécessaires dans les cas d’absence de formules exactes ou d’approximation médiocre.
Pour l’application numérique on utilisera les hypothèses numériques de variances du chap. II et
les données de comptage sur boites de Pétri du tableau 3.
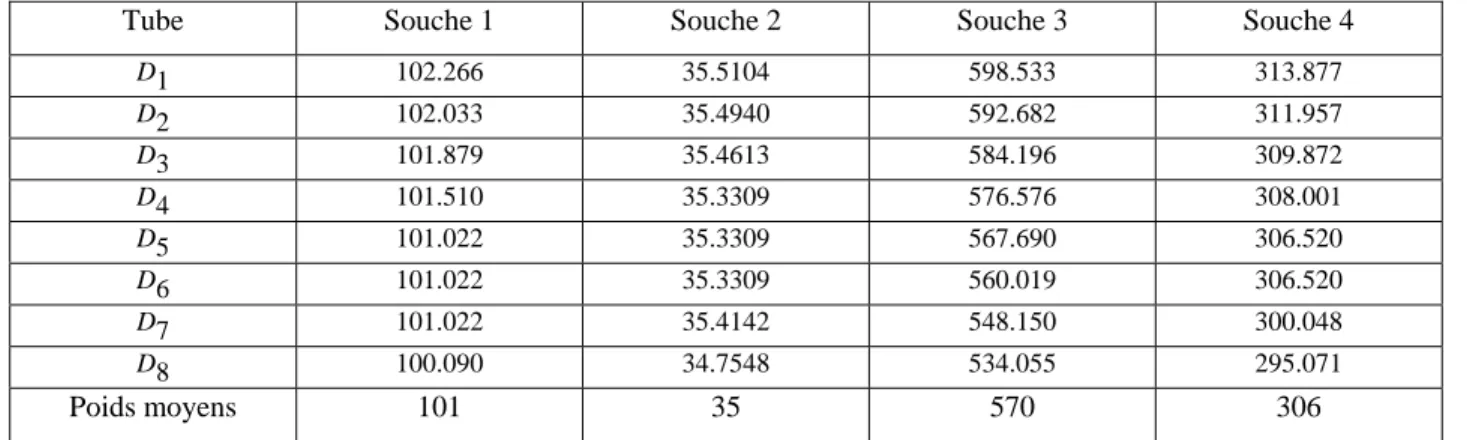
Répétitions
D
1rSouche 1
(E. Coli)
Souche 2
(E. Coli)
Souche 3
(L.M.)
Souche 4
(L.M.)
du tube
D
1(1.08×10
9) (1.26×10
9) 1.94×10
9) 1.41×10
9)
1 245 75
1150
(
?)
175
2 260
145
1210
(
?)
232
3 119
150 55
221
4 85
150 73
225
5 145
160
250
213
6 185
105
253
266
7 120 90
267
216
8 65
150
320
197
9 275
150
231
145
10 140 75 205 141
Moyenne 164
128
207
218
Ecart-type 74
33
94
27
Tableau 3 : Comptages moyens d’UFC (à partir de deux boites de Pétri) pour 4 souches et 10 répétitions de
tubes D
1par souche ; entre parenthèses apparaissent les concentrations supposées dans la microbille de la
souche, en bactéries/ml. NB : les valeurs 1150 et 1210 de la souche 3 étant suspectes ont été remplacées par la
moyenne des 8 autres valeurs.
Enfin, on supposera, comme habituellement dans ce type d’expérience, que les bactéries dans les
différents tubes sont en suspension et ne sont pas agrégées, hypothèse raisonnable si la dilution
dans un tube est assez grande.
III.1 Quelques formules fondamentales utiles
Espérance totale et variance totale
Pour une variable aléatoire Y conditionnée par les réalisations d’une variable aléatoire X
on rappelle que l’on a :
))
(
(
)
(
Y
E
E
Y
X
E
=
(1)
))
(
(
))
(
(
)
(
Y
V
E
Y
X
E
V
Y
X
V
=
+
. (2)
Espérance d’un quotient de variables aléatoires
Il est bien connu que l’espérance d’un quotient n’est pas égale, en général, au quotient des
espérances, tout particulièrement dans les cas où les variables aléatoires du numérateur et du
dénominateur suivent des lois uniformes sur [0 ;1] ou des gaussiennes centrées réduites.
Toutefois, dans les cas qui nous intéressent ici en supposant des lois gaussiennes de moyennes et
variances spécifiées, nous avons vérifié à chaque fois, par simulation, que l’approximation
suivante était tout à fait acceptable :
)
(
)
(
Y
E
X
E
Y
X
E
⎟
≈
⎠
⎞
⎜
⎝
⎛
. (3)
Variance d’un quotient de variables aléatoires
Pour deux variables aléatoires non indépendantes X et Y (Kendall & Stuart, 1976) on a:
(
)
3 4 2 2)
(
)
,
cov(
)
(
2
))
(
(
))
(
)(
(
))
(
(
)
(
Y
E
Y
X
X
E
Y
E
X
E
Y
V
Y
E
X
V
Y
X
V
⎟
≈
+
−
⎠
⎞
⎜
⎝
⎛
. (4)
Variance d’un produit de variables aléatoires indépendantes
peut écrire la variance
de leur
(5)
emarque :
obtient facilement la formule (5) en appliquant la définition de la variance sur le produit des h
(6)
(7)
←
ar exemple, pour deux variables aléatoires indépendantes X et Y on retombe sur la formule
2 2
Y
V
X
E
X
V
+
. (8)
Pour h variables aléatoires indépendantes X
i, i = 1,…, h on
V
Pproduit comme la somme de 2
h
- 1 termes. Les
C
1hpremiers termes sont constitués du produit de
la variance d’une variable aléatoire, notée
V , et du produit des carrés des espérances des h-1
iautres variables aléatoires, notées
E , les
i2
C
termes suivants sont constitués du produit de deux
variances et du produit des espérances carrées des h-2 variables aléatoires, etc… et enfin le
dernier terme est constitué du produit des variances des h variables aléatoires. Chaque terme de la
somme globale est constitué de h éléments. On écrit :
h
{
}
∑
{
∏
}
∏
∏
∑
∏
∑
= ∉ < < ∉ < ≠ =+
⎥
⎥
⎦
⎤
⎢
⎢
⎣
⎡
⎟
⎟
⎠
⎞
⎜
⎜
⎝
⎛
×
×
×
+
+
+
=
− − − h i i i i j j i i i i i i i i j j i i i i i j j h i i PV
E
V
V
E
V
V
V
E
V
V
h h h 1 , , 2 , 2 2 1 1 2 1 1 1 1 2 1 2 1 2 1 2 1 K KL
L
R
→
On
variables aléatoires, qui conduit à (6), qui elle-même se développe en (7), c’est-à-dire :
⎪⎭
⎪
⎬
⎫
⎪⎩
⎪
⎨
⎧
⎥
⎦
⎤
⎢
⎣
⎡
⎭
⎬
⎫
⎩
⎨
⎧
−
⎥
⎦
⎤
⎢
⎣
⎡
=
∏
∏
= = 2 1 1 h i i h i i PE
X
E
X
V
∏
∏
= =−
⎭
⎬
⎫
⎩
⎨
⎧
=
h i i h i iE
X
E
1 2 1 2{ }
∏
∏
= =−
=
h i i h i i PE
X
E
V
1 2 1 2∏
∏
= =−
+
=
h i i h i i iE
E
V
1 2 1 2)
(
P
exacte bien connue (Kendall & Stuart, 1976) :
(
(
)
(
)
(
)
(
XY
V
X
V
Y
E
Y
V
=
+
))
(
)
(
(
))
(
)
Variance d’un produit de variables aléatoires non indépendantes
man, 1962). On se contentera
On peut aussi établir une formule générale (voir par exemple Good
de donner ici la formule exacte pour deux variables aléatoires non indépendantes, formule qui
nous sera utile par la suite.
(
2 2)
2 2)
(
)
(
)
(
))
(
(
)
(
))
(
(
)
(
XY
E
Y
V
X
E
X
V
Y
E
X
Y
V
=
+
+
∆
∆
(
(
)(
)
)
2
(
)
(
(
)
(
)
)
)
(
2
E
X
E
∆
X
∆
Y
2+
E
Y
E
∆
X
2∆
Y
+
22 ( ) ( )
E X E Y Cov X Y
( , )
Cov X Y
( , )
+
−
(9)
vec :
Cov(X,Y) la covariance entre X et Y.
On
b
ppant les termes de :
a
-
∆
X
=
X
−
E
( X
)
,
-
∆
Y
=
Y
−
E
( X
)
,
-
éta lit facilement cette formule en dévelo
{
2}
))
(
(
)
(
XY
E
XY
E
XY
V
=
−
.
n remarque dans (9) la présence de moments mixtes centrés d’ordre 3 et 4.
a formule (9) se simplifie dans le cas où X et Y sont des variables gaussiennes. En effet, d’une
}
(
z
3E
z
3z
4E
z
4O
L
part les moments mixtes d’ordre 3 s’annulent et d’autre part, en dérivant la fonction
caractéristique d’une loi gaussienne conjointe de quatre variables aléatoires z
i, i = 1,…,4,
(Anderson, 1958), on a :
{
(
z
1E
(
z
1))(
z
2E
(
z
2))
E
−
(
))(
−
(
))
,
)
( ,
)
( ,
)
( ,
)
Cov z z Cov z z
Cov z z Cov z z
Cov z z Cov z z
=
+
+
ce qui conduit ici à :
)
−
−
1 2 3 4 1 3 2 4 1 4 3 4( ,
)
( ,
)
(
2 2 2(
) (
)
( ) ( )
2
( ,
E
⎡
⎣
∆
X
∆
Y
⎤
⎦
=
V X V Y
+
Cov X Y
onc finalement (9) se simplifie en
D
III.2. Calcul de l’espérance et de son estimation du nombre de bactéries dans un tube D
1n rappelle que l’on note D
1rune répétition r , r = 1,…, R, d’un tube D
1, R prenant la valeur 10
otons :
-
O
dans cette étude.
N
1r
D
N%
la variable aléatoire « nombre de bactéries dans un tube D
1r»,
et
-
1(
)
r DE N%
1(
)
r DV N%
son espérance et sa variance respectives,
1
~
D
N la variable aléatoire «
nombre de bactéries dans un tube
D
1», )
(
~
1 DN
E
et )
(
~
1 DN
V
son
espé
-
colonies sur la boite
»,
an
une répétition D
1rd’un
tub
1n s’intéresse donc dans ce paragraphe au calcul et à l’estimation de
rance et sa variance respectives,
1rs
n
% la variable aléatoire « nombre de
b
1rs-
n
%
1rla variable aléatoire « nombre de colonies correspond
t à
e D », E( n
% ) et V( n% ) son espérance et sa variance respectives,
1r 1rO
(
~
)
1 DN
E
. Pour y parvenir on
.
ompte tenu des hypothèses postulées précédemment, la probabilité
d’avoir un nombre
rob
les
= Prob
s’intéressera dans un premier temps au calcul et à l’estimation de
%
1(
)
r DE N
C
P
1rsn
1rsde bactéries dans le dernier volume prélevé v s’exprime à l’aide des p
abilités conditionnel
des étapes de dilution (deux dans notre situation) et de l’étape finale de prélèvement. En partant
du dernier prélèvement et en remontant jusqu’au premier on peut écrire :
bs
1rs
P
(
n
1rs∈
v n
bs 1rs∈ )×Prob(
v
bn
1rs∈
v n
b 1rs∈ )×Prob(
v
an
1rs∈
v n
a 1rs∈ )×Prob(
v
1n
1rs∈ )
v
1(
11)
Chaque volume apparaissant dans (11) , ayant été prélevé par pipetage, est entaché d’une erreur
expérimentale ; il sera donc considéré comme la réalisation d’une variable aléatoire et en
conséquence la probabilité
est une réalisation de la variable aléatoire
. Alors, la variable
aléatoire
suit une loi Binomiale de paramètres aléatoires
1rs
P
P%
1rs 1rsn
%
1r DN%
et
P%
1rs. On écrit :
1rsn
% ∼ B(
1r DN%
,
P%
1rs)
avec :
1rsP%
=
bs bv
v
×
%
%
~
1~
~
~
~
~
~
~
1 1 1 1 D a a bV
v
v
V
v
v
V
v
×
+
×
+
=
1 1 bs b a b Dv
v
d d
v
×
V
×
%
%
% %
%
%
(12)
où
1 a a av
d
V
v
=
+
%
%
%
%
et
1 1 1 1v
d
V
v
=
+
%
%
%
%
sont les facteurs (aléatoires) de dilution.
Les volumes pipetés sont surmontés d’un tilde pour souligner leur caractère aléatoire. Leurs
espérances et variances sont différentes mais restent constantes respectivement pour toutes les
répétitions D
1rdu tube D
1et toutes les souches. On rappelle (voir étape 3 du chap. I) que la
formation du volume
v~ n’amène à aucune dilution mais cette manipulation conduit à une
bnouvelle erreur de pipetage.
Comme la variable aléatoire
n
%
1rest conditionnée par
1rD
N%
et
P%
1rs, on peut écrire d’après (1) que :
E(
n
%
1r) =
E E n
( (
%
%
1rN
%
D1r,
P
%
1rs))
(13)
et d’après la définition de l’espérance d’une loi binomiale il vient :
1 1 1( (
,
))
r r D rsE E n
%
%
N
%
P
%
=
1 1(
r)
D rsE N
%
P
% (14)
Par ailleurs, on peut considérer que les deux variables aléatoires
1r DN%
et
sont indépendantes
puisque :
1rsP%
- d’une part le nombre de bactéries dans un tube D
1rdépend du nombre de bactéries
présentes dans la souche congelée et des erreurs de pipetage, indépendantes les unes des
autres, réalisées depuis le tube primaire jusqu’au tube D
1r,
Ainsi, on a pour chaque répétition D
1r:
1(
r)
E n
% =
( ) (
1r 1)
D rsE N
%
×
E P
%
(15)
=
( )
1r DE N
%
×
1 1 bs b a b Dv
v
E
d
v
V
⎛
⎞
×
× ×
⎜⎜
⎝
⎠
%
%
%
%
%
%
d
⎟⎟ (16)
=
( )
1r DE N
%
×
1 bs b b Dv
v
E
v
V
⎛
⎞
×
×
⎜
⎟
⎜
⎟
⎝
⎠
%
%
%
%
)
~
(
)
~
(
d
E
d
1E
a×
(17)
puisque que les trois variables aléatoires
1 bs b b D
v
v
v
V
⎛
⎞
×
⎜
⎟
⎜
⎟
⎝
⎠
%
%
%
%
,
d
a~
et
sont indépendantes, les volumes
ayant été pipetés indépendamment. On peut donc obtenir facilement une estimation de
l’espérance
à partir de (17) si on explicite les autres termes et que l’on dispose d’une
estimation de
.
1~
d
(
D1rE N%
)
1(
r)
E n
%
Explicitation du terme
1 bs b b Dv
v
E
v
V
⎛
⎞
×
⎜
⎟
⎜
⎟
⎝
⎠
%
%
%
%
Compte tenu du processus de dilution c’est la même réalisation de la variable aléatoire
qui
intervient deux fois mais il nous faut considérer cependant que deux variables aléatoires quotients
non indépendantes constitue ce terme pour ne pas perdre l’information liée à la variance de
.
Donc on écrit :
bv
%
bv
%
1 bs b b Dv
v
E
v
V
⎛
⎞
×
⎜
⎟
⎜
⎟
⎝
⎠
%
%
%
%
=
1 1cov
bs,
b bs b b D b Dv
v
v
v
E
E
v
V
v
V
⎛
⎞
⎛
⎞
⎛
+
×
⎜
⎟
⎜
⎟
⎜
⎜
⎟
⎝
⎠
⎜
⎝
⎠
⎝
%
%
%
%
%
%
%
⎞
⎟⎟
⎠
%
(18)
On peut approximer les espérances des deux quotients de (18) en utilisant (3) après avoir vérifié
l’approximation (3) par simulation de lois gaussiennes, soit :
1 bs b b D
v
v
E
v
V
⎛
⎞
×
⎜
⎟
⎜
⎟
⎝
⎠
%
%
%
%
=
1 1(
)
( )
cov
,
( )
(
)
bs b bs b b D b Dv
v
E v
E v
v
V
E v
E V
⎛
⎞
+
×
⎜
⎟
⎜
⎟
⎝
⎠
%
%
%
%
%
%
%
%
(19)
Pour l’estimation de cette espérance, on établira ci-dessous une estimation de la covariance par
simulation en supposant une distribution gaussienne pour les termes
v
%
bs,
v~ et
b1
~
D
V et en utilisant
les informations du tableau 2.
Application numérique :
En simulant pour les termes
v
%
bs,
v~ et
b 1~
D
V les lois gaussiennes respectives :
- N( moy = 0.1 ; var = 9.19×10
-5),
- N( moy = 1 ; var = 9.97×10
-5),
- N( moy = (9+1) ; var = (6.94×10
-4+ 9.97×10
-4)),
on obtient :
1ˆcov
bs,
b b Dv
v
v
V
⎛
⎞
⎜⎜
⎝
⎠
%
%
%
%
⎟⎟ = -1.0×10
-6
, ce qui correspond à une corrélation d’environ –0.41,
et donc à partir de (19) on a :
1ˆ
bs b b Dv
v
E
v
V
⎛
⎞
×
⎜⎜
⎝
⎠
%
%
%
%
⎟⎟ = (-1.0×10
-6)+(0.1/1)
× (1/10) ≈ 0.01 . (20)
Explicitation des termes
E
(
d
~
a)
et
E
(
d
~
1)
En simulant :
- pour
1~
V
la loi gaussienne : N( moy = 9 ; var = 6.94×10
-4),
- pour
~
v et
1v~ la loi gaussienne : N( moy = 1 ; var = 9.97×10
a-5
),
on obtient les estimations :
1







![Tableau 6 : Un exemple d’estimations et d’intervalles de confiance pour les temps de détection pour les huit courbes correspondant aux huit tubes de dilution D 1 à D 8 , pour la plage de DO de [0.35↔0.65], pour la plaque 1 de la page A1-1 de l’annexe A](https://thumb-eu.123doks.com/thumbv2/123doknet/12367654.329287/56.918.144.759.391.563/tableau-exemple-estimations-intervalles-confiance-détection-correspondant-dilution.webp)