UNIVERSITÉ DE SHERBROOKE
Faculté de génie
Département de génie électrique et de génie informatique
ÉTUDE DE PRÉCISION ET DE
PERFORMANCE DU PROCESSUS DE
CLASSIFICATION D’IMAGES DE
PHYTOPLANCTON À L’AIDE DE
MACHINES À VECTEURS DE SUPPORT
Mémoire de maîtrise
Spécialité : génie électrique
Eugène MORIN
Jury : Ruben GONZALEZ-RUBIO (directeur)
Daniel DALLE
Yannick HUOT
À ma conjointe Penny et mes deux enfants, Jeremy et Evelyn. Merci pour votre support et vos encouragements.
RÉSUMÉ
Ce projet de recherche cible l’étude et l’amélioration de la précision de la classification d’images de phytoplancton et la diminution du temps de traitement moyen requis par image. Deux solutions de classification sont proposées pour atteindre ces objectifs. La première solution vise à effectuer la classification d’images en passant par les phases de prétraitement, de discrimination et de classification, et la deuxième solution utilise uni-quement les phases de prétraitement et de classification.
En résumé, la phase de prétraitement manipule une image en vue de caractériser l’élément principal (le phytoplancton), la phase de discrimination utilise les arbres décisionnels à intervalles pour éliminer les catégories ayant peu ou pas de similitude avec l’image traitée et finalement, la phase de classification se sert de machines à vecteurs de support (SVM) pour prédire une catégorie d’appartenance à chaque image traitée.
À la base, il y a un appareil de capture automatisée d’images qui transmet celles-ci à un classificateur. Selon la vitesse de classification, une portion ou l’ensemble des images géné-rées seront classifiés. Donc, plus le nombre d’échantillons à classifier est grand, meilleure est l’approximation de la population de chaque groupe de phytoplanctons, à un temps donné. Le but étant d’obtenir une analyse qualitative, quantitative et temporelle plus précise de ce micro-organisme.
Pour permettre la classification de ce type d’image, un logiciel nommé Biotaxis a été développé. Celui-ci offre à l’utilisateur l’option de choisir parmis les deux solutions de classification proposées ci-haut. Toutes deux débutent par l’entraînement d’un groupe de classification, qui est composé de plusieurs catégories d’image, suivi par des tests de clas-sification, qui sont effectués sur ce groupe pour vérifier la précision de la classification des catégories d’image qui le compose. Pour entraîner et tester le classificateur du logi-ciel Biotaxis, deux ensembles d’images ont été employés. L’un d’eux sert uniquement à l’entrainement de groupes de classification et le second à tester ces derniers.
Les résultats obtenus dans ce projet de recherche ont permis de confirmer la validité des deux solutions proposées. Il fut possible d’atteindre une précision de la classification moyenne de 87 % et plus avec des groupes de classification de 13 catégories et moins. De plus, un temps de traitement moyen inférieur à 200 ms par image a été réalisé à partir de ces mêmes groupes de classification.
Le logiciel Biotaxis est proposé comme une nouvelle solution pour classifier rapidement des images de phytoplancton.
Mots-clés : Prétraitement, Discrimination, SVM, Précision, Temps de traitement,
REMERCIEMENTS
La réalisation d’une recherche de maîtrise nécessite beaucoup de travail. C’est grâce à l’aide, au support et au temps donnés par certaines personnes, que cette recherche a pu être complétée. J’aimerais donc en profiter pour remercier chacune de ces personnes. D’abord, je remercie M. Ruben Gonzalez-Rubio, mon directeur de recherche, qui a donné de son temps sans compter. Il m’a guidé tout au long de ma recherche et ramené à l’essentiel de la recherche lorsque je bifurquais. Merci également à M. Yannick Huot, membre du jury de cette recherche, qui a donné un thème aux images classifiées dans cette recherche et contribué à l’augmentation de mes connaissances sur les phytoplanctons. Je voudrais ensuite remercier M. Daniel Dalle, membre du jury de cette recherche, pour avoir aidé à améliorer la rédaction de ma définition du projet de recherche (DPR). Merci également à Mme Heidi Sosik, du Woods Hole Oceanographic Institution (WHOI) de Boston, pour avoir contribué à cette recherche en fournissant les groupes d’images d’entraînement et de test du classificateur.
Finalement, j’aimerais remercier la FQRNT (Fonds de recherche du Québec - Nature et technologies) pour m’avoir attribué une bourse de recherche. Ce support financier fut grandement apprécié.
TABLE DES MATIÈRES
1 INTRODUCTION 1
1.1 Mise en contexte et problématique . . . 1
1.2 Définition du projet de recherche . . . 2
1.3 Objectifs du projet de recherche . . . 2
1.4 Objectifs spécifiques . . . 3
1.5 Plan du mémoire de recherche . . . 4
2 CONCEPTS GÉNÉRAUX DE LA CLASSIFICATION D’IMAGES DE PHYTOPLANCTON 5 3 ÉTAT DE L’ART 9 3.1 Prétraitement . . . 11
3.1.1 Filtrage et détection de contour . . . 12
3.1.2 Seuillage d’image et retrait des particules distantes . . . 13
3.1.3 Tracé de contour de l’élément principal . . . 15
3.1.4 Acquisition des caractéristiques de l’élément principal . . . 16
3.2 Discrimination de catégories . . . 19
3.2.1 But recherché par la discrimination de catégories . . . 20
3.2.2 Calcul des limites d’une caractéristique . . . 20
3.2.3 Traitement de discrimination . . . 21
3.2.4 Arbre décisionnel à intervalles . . . 21
3.3 Classification . . . 23
3.3.1 Classification SVM linéaire . . . 24
3.3.2 Classification SVM non linéaire . . . 26
3.3.3 Traitement de classes multiples . . . 28
3.4 Précision et temps de traitement . . . 28
3.5 Résumé de l’état de l’art . . . 30
4 DÉVELOPPEMENT LOGICIEL 32 4.1 Analyse préliminaire . . . 32
4.1.1 Quelles images utiliser pour la classification ? . . . 33
4.1.2 Identification des étapes de traitement requises par le logiciel . . . . 35
4.1.3 Choix des langages de programmation et du logiciel de développe-ment . . . 39
4.2 Conception du logiciel Biotaxis . . . 41
4.2.1 Communication entre les langages Java et C++ . . . 41
4.2.2 Chargement d’images . . . 43
4.2.3 Prétraitement . . . 44
4.2.4 Discrimination . . . 48
4.2.5 Classification SVM . . . 50
4.2.7 Description des options de traitement du logiciel Biotaxis . . . . 55
4.3 Tests des différentes phases de classification . . . 63
4.4 Méthode de classification d’image avec le logiciel Biotaxis . . . . 64
4.5 Bilan du développement logiciel . . . 66
5 RÉSULTATS ET ANALYSE 67 5.1 Présentation des résultats obtenus . . . 67
5.1.1 Cadre expérimental . . . 67
5.1.2 Spécification du système informatique . . . 69
5.1.3 Solution 1 - Classification AVEC discrimination . . . 69
5.1.4 Solution 2 - Classification SANS discrimination . . . 70
5.2 Analyse des résultats . . . 71
5.2.1 Comparaison des résultats des deux solutions . . . 71
5.2.2 Interprétation des résultats . . . 73
6 DISCUSSION 75 6.1 Les bonnes décisions . . . 75
6.2 Les décisions à considérer . . . 76
7 CONCLUSION 78 7.1 Récapitulatif . . . 78 7.2 Contributions . . . 79 7.3 Travaux futurs . . . 80 A DONNÉES 81 B DONNÉES (Suite) 82
C Analyse des métriques du logiciel Biotaxis 83 LISTE DES RÉFÉRENCES 84
LISTE DES FIGURES
2.1 Schéma de la suite d’étapes de classification d’une image selon les solutions
1 et 2 proposées dans le cadre de cette recherche . . . 6
2.2 Schéma simplifié du processus de classification d’une image . . . 7
3.1 Kernel 3 x 3 du filtre Sobel . . . 13
3.2 Kernel 3 x 3 du filtre Prewitt . . . . 13
3.3 Séquence de seuillage de la méthode hystérésis . . . 14
3.4 Démonstration du calcul des Seuils Sb et Sh . . . 15
3.5 Méthode Convex Hull appliquée à une image de phytoplancton . . . . 16
3.6 Convex Hull et tracé de contour . . . . 17
3.7 Trouver la plus longue diagonale du Convex Hull . . . 17
3.8 Première caractérisation . . . 17
3.9 Étapes A et B de création d’un arbre décisionnel à intervalles . . . 22
3.10 Étape C de création d’un arbre décisionnel à intervalles . . . 22
3.11 Classificateur SVM avec hyperplan optimal et marge maximale . . . 25
3.12 Mappage d’un problème non linéaire vers espace supérieur . . . 26
3.13 Matrice de confusion et calcul de la précision . . . 29
4.1 Exemple d’images des 21 catégories de phytoplancton utilisées pour cette recherche . . . 33
4.2 Organisation des fichiers images en ensemble d’images de 21 catégories . . 35
4.3 Séquence d’étapes d’entraînement d’un ensemble d’images menant à la créa-tion d’un groupe de classificacréa-tion . . . 37
4.4 Séquence d’étapes de test d’un ensemble d’images et d’un groupe de clas-sification menant à la clasclas-sification d’images (comprend l’intégration de la séquence d’étapes selon les solutions 1 et 2). . . 39
4.5 Schéma de concept simplifié du logiciel Biotaxis . . . . 41
4.6 Interface après la sélection de l’onglet « Analysis » (logiciel Biotaxis) . . . 55
4.7 Interface après la sélection de l’onglet « Training » (logiciel Biotaxis) . . . 58
4.8 Interface après la sélection de l’onglet « Testing » (logiciel Biotaxis) . . . . 61
4.9 Exemple d’informations produites en console lors de la génération d’arbres décisionnels à intervalles (logiciel Eclipse) . . . . 64
5.1 Comparaison de la précision de la classification atteinte en fonction du nombre de catégories à classer (par solution et filtre) . . . 72
5.2 Comparaison du nombre d’images traitées par seconde en fonction du nombre de catégories à classer (par solution et filtre) . . . 72
LISTE DES TABLEAUX
2.1 Précision de la classification d’images de phytoplancton obtenue avec l’usage de la méthode SVM selon les articles à l’étude ayant obtenus plus de 70 % de précision de la classification . . . 6 3.1 Compilation des phases de prétraitement de deux articles à l’étude . . . 11 3.2 Phases de prétraitement et méthodes retenues pour cette recherche . . . . 12 3.3 Caractéristiques obtenues à l’aide du Convex Hull d’un objet . . . . 18 3.4 Discrimination de catégorie selon les limites d’une caractéristique . . . 19 3.5 Quelques kernels fréquemment utilisés . . . 27 5.1 Liste des catégories appartenant aux différents groupes de classification TEST 68 5.2 Résultats obtenus avec la solution 1 selon le filtre utilisé (précision et temps
de traitement) . . . 69 5.3 Résultats obtenus pour la solution 2 selon le filtre utilisé (précision et temps
de traitement) . . . 70 A.1 Resultats obtenus avec la solution 1 (AVEC discrimination) . . . 81 B.1 Resultats obtenus avec la solution 2 (SANS discrimination) . . . 82
LISTE DES SYMBOLES
Symbole Définition
Rn L’univers de données possibles de n dimension(s)
S Un échantillon de données de l’univers Rn
D Un groupe de formation préidentifié d’un classificateur SVM
f Une fonction cible d’un classificateur SVM
b
f Modèle d’approximation de la fonction f
* Exposant d’une variable lorsque sa valeur est optimale
~
x Représente un vecteur dans l’univers Rn
~
w Vecteur normal de l’hyperplan optimal
b Décalage entre l’hyperplan optimal et l’origine
sgn() Donne le signe de l’élément dans la parenthèse (soit -1, 0, +1) Φ Mappage non linéaire
Φ(~x) Application du mappage non linéaire sur le vecteur ~x α Multiplicateur de Lagrange
c Constante de coût du kernel polynomial non homogène
β Largeur de bande pour le kernel gaussien
σ Écart-type
LISTE DES ACRONYMES
Acronyme Définition
WHOI Woods Hole Oceanographic Institution IFCB Imaging FlowCytobot
k-NN k-nearest neighbor (k plus proches voisins) SVM Support Vector Machine
(Machines à vecteurs de support) CPU Central Processing Unit
GPU Graphics Processing Unit TP True Positive (Vrai Positif) TN True Negative (Vrai Négatif) FP False Positive (Faux Positif) FN False Negative (Faux Négatif) JNI Java Native Interface
JVM Java Virtual Machine ROI Region of Interest
MVC Modèle - Vue - Contrôleur
min Minimum
max Maximum
png Portable Network Graphics
csv Comma-Separated Values
CHAPITRE 1
INTRODUCTION
1.1
Mise en contexte et problématique
L’étude de la biodiversité aquatique présente encore à ce jour, son lot de questionnements. Les percées technologiques des dernières années ont permis l’avènement d’une multitude d’appareils conçus dans le but d’aider les chercheurs à résoudre ces interrogations. Parmi ces appareils, ceux dédiés à la capture automatisée d’images marines ont contribué à l’émergence de nouvelles approches de classification. Dans le cadre de cette recherche, ce sont les approches liées à la classification d’images de phytoplancton qui ont servi de base au développement de ce projet.
Un phytoplancton est un micro-organisme du plancton végétal que l’on retrouve dans les différentes étendues d’eau de notre planète [Falkowski et Raven, 2007]. On dénombre des milliers d’espèces, mais leur variété sur un site donné est fonction de leur habitacle (eau douce, salée, lac, rivière, etc.). Ils sont si petits qu’il est possible de dénombrer une centaine, voir des milliers de spécimens dans une seule gouttelette d’eau. Leur rôle est d’importance, car ils sont à la base de la chaîne alimentaire aquatique et parce qu’ils consomment plus de la moitié du CO2 rejeté dans l’atmosphère. Une des questions à
l’étude pour le phytoplancton est de connaître son évolution dans une étendue d’eau.
Il y a quelques années, la classification des images de phytoplancton était réalisée par un opérateur qui analysait et classait chacune de ces images. Mais la puissance toujours croissante de l’ordinateur combinée avec l’implémentation de modèles de classification a permis la création d’outils logiciels pouvant traiter automatiquement une grande quantité d’images. À partir de ces modèles, des groupes de chercheurs ont travaillé sur différentes approches afin d’avoir des outils leur permettant de faire une analyse qualitative, quan-titative et temporelle du phytoplancton. Plusieurs approches ont été essayées et testées, mais il reste encore place à améliorations du point de vue de la précision de la classification et du temps de traitement.
1.2
Définition du projet de recherche
En partant de ces observations, une analyse de la précision de la classification des diffé-rentes approches à l’étude a permis de déterminer que dans leur ensemble, les processus employés offrent la possibilité d’obtenir une précision de la classification de 87 %. L’analyse de ces études sera précisée au chapitre 2.
Ce nombre représentant la précision de la classification est très important, car plus il est grand, plus la classification des phytoplanctons par catégorie sera fiable. Ainsi, la projection sur un graphique de l’évolution du nombre de phytoplanctons par catégorie en fonction du temps donnera une courbe beaucoup plus représentative de la variation de la population. Permettant une meilleure compréhension de l’interaction entre ces micro-organismes et leur environnement. Cette projection en temps réel de l’évolution de la population sous forme graphique serait en soi un outil très utile pour la communauté scientifique travaillant sur le sujet.
Avant de pouvoir faire cette projection en temps réel, il est impératif de connaître le temps de traitement requis par le processus de classification. Les articles à l’étude traitant de la classification de phytoplancton n’ont pas permis d’identifier les temps de traitement requis. Une étude de ce temps et de la précision de la classification lors de l’usage de processus donnés permettrait de faire un choix de méthodes de traitement plus éclairé lorsque le niveau d’importance de l’un ou l’autre de ces deux aspects varie. C’est à partir de cette observation que la question de recherche suivante fut établie :
« Comment améliorer ou conserver la précision de classification actuelle des images de phytoplancton, tout en diminuant le temps de traitement requis ? »
1.3
Objectifs du projet de recherche
L’objectif global est donc d’étudier le temps de traitement requis, dépendamment des processus de classification utilisés, afin de présenter une base décisionnelle quant au bien-fondé de l’usage ou non de ces processus.
Pour atteindre cet objectif, le projet réalisé a d’abord nécessité l’identification de balises, qui ont ensuite été utilisées pour guider l’ensemble du processus de classification par catégorie des images de phytoplancton. Les différentes balises représentant les objectifs secondaires du projet sont :
- de viser un temps de traitement moyen de 200 ms ou moins par image, - de concevoir un logiciel de classification d’images.
Afin d’atteindre l’objectif principal et de respecter les objectifs secondaires, la recherche fut orientée de la façon suivante. D’abord, il convenait d’avoir un outil logiciel qui permettrait de comptabiliser les résultats obtenus et qui offrirait l’assurance du respect des objectifs secondaires. Le logiciel Biotaxis1 a donc été conçu dans cette optique. Soit d’établir la
précision de la classification d’images de phytoplancton selon différents processus employés, tout en générant des informations sur le temps de traitement requis pour chacun de ces processus.
Suite à cette approche, il convenait de déterminer les processus à utiliser pour faire la classification d’images et ensuite, de faire l’analyse des résultats obtenus. C’est en se fondant sur ces prémisses que le projet de recherche fut réalisé.
1.4
Objectifs spécifiques
Cette recherche proposée vise à la réalisation du logiciel Biotaxis. Ce logiciel offre la possibilité à l’utilisateur de choisir l’une parmi deux solutions proposées pour effectuer la classification d’images de phytoplancton. Les phases de traitements appliqués étant :
- Solution 1 : comprenant les phases de prétraitement, de discrimination de catégories et de classification.
- Solution 2 : comprenant les phases de prétraitement et de classification.
Au terme de la classification d’images, il est possible de compiler les résultats de la pré-cision de la classification et de réaliser une comparaison avec ceux générés par l’usage d’autres filtres, groupes d’images ou solution de traitement. Cette comparaison permet d’identifier la meilleure combinaison (images, filtres, solution) pour atteindre la précision de la classification ciblée.
En plus du logiciel Biotaxis, une autre contribution originale est la présentation d’une analyse comparative de la précision de la classification et du temps de traitement selon le nombre de catégories utilisé par groupe. Cette analyse permet d’établir la relation existant entre :
1Afin d’éviter toute ambiguïté entre la désignation des projets référés avec notre projet, le nom Biotaxis
sera utilisé. Ce nom réfère à la définition suivante : « la classification d’organisme vivant en fonction de leur caractéristique physique » [Encyclopedia, 2011].
- La précision de la classification versus le nombre de catégories à classifier.
- Le nombre d’images traitées par seconde versus le nombre de catégories à classifier.
1.5
Plan du mémoire de recherche
Ce mémoire de recherche a été organisé de la façon suivante. On retrouve tout d’abord le chapitre du « Concept généraux de la classification d’images de phytoplancton » , qui est une introduction aux différents concepts ayant été utilisés dans ce projet. Ensuite, on passe au chapitre de l’« État de l’art », qui est une synthèse des avancées recensées dans la littérature qui ont été utilisées dans ce projet. Par la suite vient le chapitre sur le « Développement logiciel » qui présente l’application des concepts théoriques de l’état de l’art ayant mené à la réalisation du logiciel Biotaxis. Le chapitre suivant est « Résultats et Analyse » où sont présentés les résultats obtenus dans cette recherche et l’interprétation de ceux-ci. Puis vient ensuite le chapitre de « Discussion » qui est une évaluation de certaines décisions prises en cours de projet. Finalement, le mémoire se termine par le chapitre de « Conclusion ».
CHAPITRE 2
CONCEPTS GÉNÉRAUX DE LA
CLASSIFICA-TION D’IMAGES DE PHYTOPLANCTON
Ce chapitre présente certains concepts nécessaires à la compréhension du sujet de re-cherche. Ce survol des idées maîtresses du projet de recherche, ainsi que leur interaction a donc pour but de permettre au lecteur d’avoir une idée globale du projet et de faciliter la lecture du chapitre suivant, traitant de l’état de l’art.
Ainsi, l’élément principal du projet Biotaxis est la classification d’images d’organismes vi-vants, soit les phytoplanctons. À ce jour, plusieurs méthodes sont disponibles pour classifier des images. Parmi celles-ci on retrouve la méthode des k plus proches voisins (k − N N ) [Cover et Hart, 1967], les réseaux de neurones [Rosenblatt, 1958] et les machines à vecteurs de support (SVM) [Vapnik, 1995], qui a été la méthode sélectionnée. Ce choix se fonde sur deux raisons majeures :
- Tous les articles de classification d’images de phytoplancton à l’étude pour cette recherche ont utilisé cette méthode.
- La qualité de la précision obtenue lors de son usage pour la classification d’images de phytoplancton (voir le tableau 2.1).
Pour illustrer cette qualité de la précision, voici un tableau énumérant les résultats qui ont été publiés dans des articles à l’étude. Les articles présentés ont été sélectionnés en fonction des détails donnés sur le nombre d’échantillons et de catégories utilisés, ainsi qu’au fait d’avoir obtenu une précision de la classification supérieure à 70 %.
Comme on peut le constater, les pourcentages de précision cités dans le tableau 2.1 sont très élevés. Il y a cependant certaines divergences dans les approches employées pour obtenir ces résultats. Voici une liste des divergences prédominantes ressorties lors de l’étude des articles du tableau 2.1 :
- Le type et le nombre de catégories taxonomiques1 utilisées.
- Le nombre d’échantillons employés pour entraîner le classificateur. 1Relatif à la taxonomie, science de la classification des êtres vivants.
Article Nb. Échantillons Nb. Catégories Précision Alvarez et al. [2012] 526 6 86 % Su et al. [2010] 1629 4 89 % Kang et al. [2010] 1300 35 86.59 % Sosik et Olson [2007] 6600 22 88 % Hu et Davis [2005] 20000 7 72 % Luo et Kramer [2004] 6000 5 75.57 %
Tableau 2.1 Précision de la classification d’images de phytoplancton obtenue avec l’usage de la méthode SVM selon les articles à l’étude ayant obtenus plus de 70 % de précision de la classification
- Le choix des méthodes utilisées pour le prétraitement des images.
- Le type et la quantité d’attributs de caractérisation des images employés.
À défaut de connaître l’impact des divergences énumérées ci-haut, une analyse des ap-proches de classification citées dans les articles listés dans le tableau 2.1 a été réalisée pour cette recherche et synthétisée par le schéma de la figure 2.1. Ce schéma présente les deux solutions de classification proposées, ainsi que les séquences d’opérations propres à chacune de ces solutions.
Figure 2.1 Schéma de la suite d’étapes de classification d’une image selon les solutions 1 et 2 proposées dans le cadre de cette recherche
Les trois étapes de classification citées dans la figure 2.1 se résument comme suit :
- Le prétraitement comprend plusieurs phases de traitement tel que : la réduction du bruit, l’extraction de la zone d’intérêt d’une image (conserver uniquement le phytoplancton) et la caractérisation de la zone d’intérêt.
- La discrimination de catégories ayant comme objectif : la réduction du nombre de catégories d’appartenance d’une image en fonction d’attributs spécifiques (tel que la grandeur et la forme de la zone d’intérêt). Le but étant de discriminer des catégories afin que la classification SVM se fasse uniquement sur les catégories restantes. Il est espéré que cette étape permettra de diminuer le temps de traitement.
- La classification attribue une catégorie à l’image traitée en fonction du résultat obtenu par la méthode SVM.
La figure 2.2 présente un exemple simplifié d’un classificateur d’images contenant une forme rectangulaire. Ce schéma donne une idée générale du fonctionnant d’un classificateur. Les traitements énoncés dans chacune des étapes du schéma sont ceux employés par les classificateurs utilisés dans cette recherche (solution 1 ou 2).
Si l’on se réfère au schéma de la figure 2.1, on y retrouve une échelle de temps comprenant deux jalons pour marquer la séquence temporelle à l’étude. Puisque l’un des objectifs du projet Biotaxis est d’obtenir un temps de traitement moyen de la classification de 200 ms. Alors le temps compris entre t0 et tf, se doit d’être quantifié pour être en mesure de
déterminer le temps de traitement moyen par image, par catégorie d’images et par groupe de classification (qui un ensemble de catégories).
En conclusion, ce chapitre avait pour but de présenter les concepts globaux de la clas-sification d’images de phytoplancton. Le choix de la méthode de clasclas-sification utilisée a été abordé (méthode SVM), ainsi qu’une présentation des résultats de la précision de la classification obtenus pour certains articles (ayant utilisé la méthode SVM). Ensuite, deux solutions de classification ont été proposées suivies par une description des différentes étapes de traitement requises par chacune d’elle. Ce chapitre avait donc pour objectif de faciliter la compréhension des concepts généraux de la classification, avant de passer au chapitre suivant (l’« État de l’art ») qui traite de façon détaillée, l’ensemble des concepts qui ont été abordés.
CHAPITRE 3
ÉTAT DE L’ART
Chaque jour, une grande quantité d’images sont générées quotidiennement par des appa-reils automatisés de capture d’images de phytoplancton. Il suffit de citer comme exemple le IFCB [Olson et Sosik, 2007], qui est un appareil pouvant capturer 10 000 images à l’heure (soit environ 240 000 images par jour), pour comprendre que la classification manuelle de l’ensemble de celles-ci représente une tâche presque impossible à réaliser, à défaut d’avoir un grand nombre de ressources humaines attitrées à cette tâche.
Puisque cette énorme quantité d’images reste inutilisable si aucune caractérisation et clas-sification de celles-ci n’est faite, des groupes de chercheurs ont donc travaillé sur des solu-tions possibles afin d’automatiser ce processus. L’analyse de quelques-unes des solusolu-tions avancées illustre la possibilité de combiner certaines des méthodes et approches proposées en vue de réaliser l’implémentation d’un logiciel de classification.
Cependant, avant de pouvoir réaliser un tel outil, il est important de cibler certains critères spécifiques permettant d’évaluer le logiciel. Parmi les critères possibles, deux d’entre-eux requièrent une attention particulière :
- Le premier est la précision de la classification, qui va de pair avec la qualité de l’analyse, car plus il y a d’images « bien » classées, plus l’évaluation de la variation de populations dans le temps est précise.
- Le deuxième est le temps de traitement. Ce paramètre détermine en combien de temps la classification d’une image est effectuée. Par exemple, si le logiciel peut classifier 1 000 images à l’heure, mais que 2 000 images sont générées au cours de cette même période de temps, alors seulement une fraction d’entre elles pourra être traitée à l’heure (50 % pour cet exemple).
Ces deux critères sont intimement liés, car l’usage d’une méthode ayant un impact sur la précision aura incidemment un impact sur le temps de traitement. Il convient alors de bien choisir les méthodes et approches utilisées en vue d’accroître la précision et de minimiser le temps d’opération. Certaines décisions pourraient ainsi mener à une légère perte de précision en vue d’obtenir un large gain en temps de traitement et la situation inverse est également possible.
Pour mener à bien ce projet, les deux critères cités ci-haut ont été pris en compte. La revue de littérature a été orientée vers les ouvrages traitant de méthodes de classification des phytoplanctons, utilisant les machines à vecteurs de support (SVM) comme modèle d’apprentissage supervisé. Parmi les ouvrages étudiés, seulement ceux ayant atteint 70 % et plus de précision de la classification ont été retenus comme référence pour cette recherche. Chacun d’eux a appliqué leurs traitements sur des images bidimensionnelles et a utilisé des métriques de discrimination taxonomique pour favoriser l’augmentation de la précision de leur processus de classification.
Les prochaines sections de ce chapitre traitent des différentes étapes du processus de classification (tel que défini à la figure 2.1 du chapitre 2). Les deux solutions considérées dans cette recherche et détaillées au chapitre 2, comportent la série d’étapes suivantes :
- Solution 1 (avec discrimination) : 1. Prétraitement
2. Discrimination 3. Classification
- Solution 2 (sans discrimination) : 1. Prétraitement
2. Classification
En se basant sur les étapes de la solution 1, cela permet de couvrir également les étapes de la solution 2. C’est pourquoi les sections suivantes de l’état de l’art présentent l’ensemble des étapes utilisées par la solution 1. Évidemment, peu importe la solution choisie, le but commun recherché est de prendre une image et de la manipuler en vue de déterminer sa catégorie d’appartenance.
La présentation des sections suivantes suit donc l’ordre d’opération de la solution 1. Les méthodes citées dans chacune de ces sections font référence à des ouvrages sur la classifi-cation de phytoplanctons, ainsi qu’à d’autres ouvrages ciblant un type de traitement bien spécifique. En résumé, le choix des méthodes, citées dans l’état de l’art, résulte de l’analyse et la comparaison de chacun des traitements utilisés dans les ouvrages sur le classement de phytoplanctons, avec des ouvrages dédiés spécifiquement au traitement comparé. La sélection finale du procédé ou de la méthode pour chaque traitement fut basée sur leur contribution à l’augmentation de la précision de la classification et/ou leur capacité à diminuer le temps de traitement.
3.1
Prétraitement
Pour bien définir les différents processus caractérisant cette étape, une analyse des articles à l’étude sur le sujet a été menée afin de cibler, puis de déterminer la suite logique des trai-tements nécessaires. Le passage par ces processus vise l’obtention de valeurs numériques permettant de caractériser les différents paramètres d’une image donnée. Le tableau 3.1 présente la séquence d’opérations de prétraitement citée dans deux des articles à l’étude ayant décrit l’ensemble de leurs procédés (certaines parties présentes également une réfé-rence).
Phases de prétraitement Articles de référence
Sosik et Olson [2007] Su et al. [2010] 1. Filtrage et détection de
contour
« Noisecompensated phase congruency » [Kovesi, 1999]
Méthode Canny de détection de contour [Canny, 1986] 2. Seuillage de l’image Seuillage (« thresholding »)
3. Retraits des particules distantes Opérateurs morphologiques (fermeture, dilatation et amincissement) Opérateurs morphologiques (dilatation, fermeture) et dé-tection de la région d’intérêt (ROI )
4. Tracé de contour de l’élément principal
Tracé des contours simpli-fiés de l’élément principal en fonction du premier 10 % du descripteur de Fourier [Gon-zalez et al., 2009]
5. Acquisition des carac-téristiques de l’élément principal
Caractérisation de l’élément principal (périmètre, aire, longueur, etc.)
Tableau 3.1 Compilation des phases de prétraitement de deux articles à l’étude
Note : Le choix des deux articles utilisés pour la création du tableau 3.1 est basé sur la description des méthodes employées lors de la phase de prétraitement. Tel que défini dans ce même tableau, l’article de Sosik et Olson [2007] est le plus exhaustif sur les méthodes utilisées lors des différentes phases de prétraitement.
Suite à l’analyse des différentes phases de prétraitement énumérées dans le tableau 3.1, l’étude subséquente fut de comparer dans la littérature les méthodes citées dans ce tableau versus d’autres méthodes au rendu similaire en fonction du temps de traitement requis. Cette étude sur le prétraitement des images a permis d’identifier et choisir les méthodes utilisées dans cette recherche. Le tableau 3.2 fait état de ces choix de méthodes selon les différentes phases de prétraitement.
Phases de prétraitement Méthodes retenues et références
1. Filtrage et détection de contour Prewitt et Sobel [Prewitt, 1970; Sobel et Feld-man, 1968]
2. Seuillage de l’image et retraits des particules distantes
Seuillage par hystérésis [Pridmore, 2002] 3. Tracé de contour de l’élément
principal
Méthode Convex Hull [de Berg et al., 2008] 4. Acquisition des caractéristiques
de l’élément principal
Caractérisation de l’élément principal selon le
Convex Hull (aire, périmètre, etc.) [Luo, 1998]
Tableau 3.2 Phases de prétraitement et méthodes retenues pour cette recherche
Les sous-sections suivantes de l’état de l’art présentent chacune des phases de prétrai-tement du tableau 3.2 ci-haut mentionnées, en respectant la séquence des traiprétrai-tements énumérée.
3.1.1
Filtrage et détection de contour
Cette étape vise d’abord à filtrer une image en vue d’enlever le bruit, puis de faire la détection des contours de l’élément principal de l’image (soit le phytoplancton). L’article de Sosik et Olson [2007] cite l’usage de la méthode « noise-compensated phase congruency » pour réaliser les deux processus cités précédemment. Cependant, il est fait mention que cette méthode est très intensive en calcul de processeur, c’est pourquoi cette approche a été rejetée pour cette recherche. L’article de Su et al. [2010] relate l’usage de la méthode Canny pour faire le filtrage et la détection des contours de l’élément principal de l’image. Cependant, l’étude de l’usage de cette méthode faite par Sosik et Olson [2007] pour le traitement d’image de phytoplancton a démontré que la méthode Canny ne pouvait pas être optimisée pour éliminer le bruit et les variations d’intensité lumineuse présente autour de l’élément principal de l’image.
L’étude des autres pistes de solution permettant la détection de contour a permis d’iden-tifier le filtre Sobel comme étant une méthode fréquemment utilisée pour ce type de tâche. De plus, ce filtre fait également partie de la suite de traitements utilisée par la méthode Canny [Canny, 1986]. Ces observations ont donc favorisé l’adoption de cette approche pour cette étape de prétraitement.
Le filtre Sobel est un masque de convolution que l’on applique à une image par un balayage en X et Y , afin de calculer le gradient d’intensité lumineuse de chaque point. L’image résul-tante fait ressortir les changements abrupts de luminosité dans l’image originale, donc les
contours probables d’une ou plusieurs formes. La figure 3.1 présente les kernels définissant le filtre Sobel ainsi que l’opérateur mathématique permettant d’obtenir le gradient nor-malisé pour chaque point d’une image traité à l’aide de cet opérateur [Sobel et Feldman, 1968].
Figure 3.1 Kernel 3 x 3 du filtre Sobel
À titre comparatif au filtre Sobel, le filtre Prewitt [Prewitt, 1970] a également été étudié dans cette recherche due aux similitudes des kernels 3x3 utilisés par ce dernier versus ceux du filtre Sobel (voir figures 3.1 et 3.2).
Figure 3.2 Kernel 3 x 3 du filtre Prewitt
3.1.2
Seuillage d’image et retrait des particules distantes
Le seuillage d’une image en niveau de gris consiste à remplacer chacun de ses pixels par la valeur 0 (noir) ou 255 (blanc) en fonction d’un seuil déterminé. Cette méthode a été utilisée par Sosik et Olson [2007] pour obtenir une image où la surface du phytoplancton est en blanc et l’arrière-plan en noir. Le seuillage d’une image est défini selon l’opérateur mathématique suivant :
Si g(x, y) est la version de l’image après le seuillage de f (x, y) selon un seuil T alors,
g(x, y) = 255 si f (x, y) ≥ T 0 sinon (3.1)
La qualité de l’extraction de la forme d’un phytoplancton dans une image est fonction du seuil établi. Pour déterminer la valeur de ce seuil, il faut se référer aux étapes de traitement de la méthode de seuillage choisi. Parmi les méthodes de seuillage possible, la méthode hystérésis a été utilisée pour cette recherche en raison de la qualité du rendu
après seuillage [Pridmore, 2002], à son usage par la méthode Canny [Canny, 1986] et à sa capacité d’éliminer les particules distantes (tel que des poussières) présentent dans l’image .
La méthode de seuillage hystérésis a la particularité d’utiliser 2 seuils au lieu d’un seul, soit un seuil bas (Sb) et un seuil haut (Sh). Dans le cas du traitement d’image de phytoplancton,
le seuil du bas délimite le niveau de gris à partir duquel il y a une grande probabilité que les niveaux de gris inférieurs appartiennent au phytoplancton que l’on désire détecter. Puis, le seuil du haut délimite le niveau de gris à partir duquel les niveaux de gris inférieur ont une forte probabilité d’appartenir au phytoplancton. Ces deux seuils sont donc utilisés séparément sur l’image à traiter afin d’obtenir une première image basée sur le seuil Sb et
une seconde basée sur le seuil Sh. Ensuite, tous les pixels blancs de l’image de seuillage Sh qui touchent à un pixel blanc de l’image Sb sont ajoutés à l’image de seuillage Sb pour
obtenir une image résultante d’hystérésis. La figure 3.3 présente les résultats obtenus après chaque séquence de seuillage.
Figure 3.3 Séquence de seuillage de la méthode hystérésis1
Pour calculer les seuils Sb et Sh, la méthode employée fut inspirée de l’étude de Pridmore
[2002]. Voici les étapes du processus (voir la figure 3.4 comme référence) : 1. Tracer l’histogramme du niveau de gris de l’image à traiter.
2. Déterminer les niveaux de gris limite de l’histogramme, soit : le pixel ayant un niveau de gris le plus près du noir (Xi), le pixel ayant un niveau de gris le plus près du
blanc (Xf).
1Images obtenues à l’aide du logiciel Biotaxis (Ciliate, réf. 2006_052_145231_2649.png). À noter que
l’image de seuillage Sh présente un groupe de points blancs au-dessus de la forme du phytoplancton et
que ce groupe n’est pas présent dans l’image d’hystérésis, car aucun des points du groupe ne touche un point blanc de l’image de seuillage Sb.
3. Diviser en deux sections (bas et haut), selon le centre de Xf et Xi. Le centre étant
déterminé selon l’équation 3.2, les limites délimitant l’intervalle du bas sont donc Xi
et Xc et pour le haut Xc et Xf.
Xc= ((Xf − Xi)/2) + Xi (3.2)
4. Calculer la moyenne x et l’écart type σ de la section du bas et du haut. Pour la section du bas on calcule xb et xb et xh et σh pour le haut.
5. Déterminer les valeurs de seuil bas (Sb) et de seuil haut (Sh), selon les équations :
Sb = xb + 2σb (3.3)
Sh = xh− 2σh (3.4)
Figure 3.4 Démonstration du calcul des Seuils Sb et Sh2
Pour ce qui est des particules distantes, puisque leur niveau de gris est presque toujours supérieur au niveau de gris de l’élément principal (dans le contexte des images utilisées dans cette recherche), alors le seuillage de l’image est suffisant pour les retirer.
3.1.3
Tracé de contour de l’élément principal
Une fois les étapes de filtrage et seuillage de l’image complétées, le traitement suivant doit permettre de délimiter le contour de l’élément principal, donc de définir l’espace minimal requis permettant d’englober cet élément. Sosik et Olson [2007] ont utilisé le descripteur de Fourier pour tracer ce contour, mais ce descripteur nécessite beaucoup de calculs. L’étape suivante consiste donc à déterminer quelle méthode permettrait de tracée un contour plus rapidement que la méthode du descripteur de Fourier. Les études dans la littérature sur le
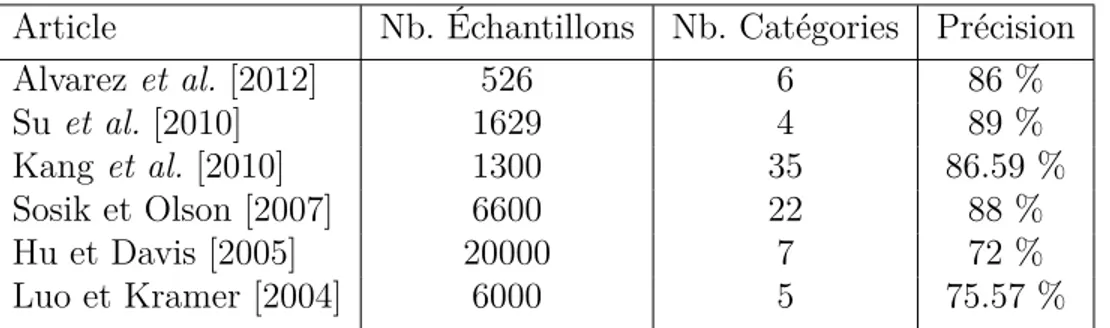
sujet ont permis d’identifier la méthode Convex Hull comme étant une solution simple et rapide pour obtenir une enveloppe de contour d’objet principal. L’exemple de la figure 3.5 présente le résultat obtenu lors de l’usage de ce processus à une image de phytoplancton ayant passé précédemment par une étape de seuillage.
Figure 3.5 Méthode Convex Hull appliquée à une image de phytoplancton3
À la figure 3.5, l’image 3 présente la surface de couverture du phytoplancton en blanc sur fond noir avec un tracé de contour représentant le résultat de l’application de la méthode
Convex Hull sur l’image 2. L’ouvrage de de Berg et al. [2008], présente une méthode pour
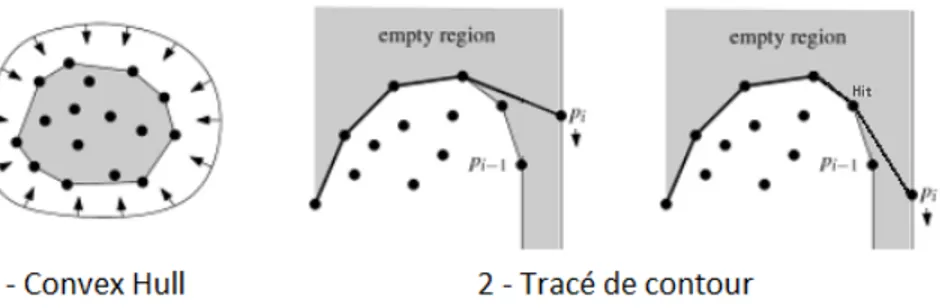
effectuer le tracé de contour d’un objet (voir la figure 3.6). Le principe consiste à trouver d’abord un point extrême A, soit par exemple le pixel le plus haut de l’objet, puis d’obtenir la coordonnée du point B positionné au-dessus du point A, soit à la limite supérieure de l’image (bordure). Ensuite, il faut tracer un vecteur entre les points A et B, en partant du point A. Si aucune bordure de l’objet n’a été touchée lors du tracé du vecteur, alors le vecteur est effacé, puis le point B est déplacé de un pixel dans le sens horaire. Un nouveau vecteur est alors tracé et effacé entre les points A et B tant que le vecteur ne touchera pas à une bordure de l’objet. Lorsqu’il y a un contact entre le vecteur et l’objet, le tracé entre le point A et le point de contact est conservé, puis la coordonnée du point de contact devient la nouvelle coordonnée du point A. La séquence de tracé du vecteur entre les points A et
B se poursuit jusqu’à ce que le point B ait complété une rotation complète de la bordure
de l’image (retour au point de départ). L’image 2 de la figure 3.6 présente le concept d’un déplacement de vecteur pour obtenir le tracé de contour d’un objet.
3.1.4
Acquisition des caractéristiques de l’élément principal
L’étape faisant suite à la méthode Convex Hull est l’acquisition des caractéristiques de la zone d’intérêt délimitée par le tracé de contour. L’ouvrage de Luo [1998] définit très bien les étapes et formules permettant de caractériser un objet à partir de la méthode Convex
Hull. Cette séquence d’étapes se présente comme suit :
3Images obtenues à l’aide du logiciel Biotaxis (Ciliate, réf. 2006_052_145231_2649.png) 4Référence : [de Berg et al., 2008]
Figure 3.6 Convex Hull et tracé de contour4
1. Trouver la plus longue diagonale (Dmax) du Convex Hull qui est obtenu par le calcul
de la distance entre tous les coins du tracé du Convex Hull.
Figure 3.7 Trouver la plus longue diagonale du Convex Hull
2. Appliquer une rotation sur l’objet afin que la plus longue diagonale soit sur un plan horizontal. Puis trouver les valeurs verticales supérieures (Vt) et inférieures (Vb),
calculer les largeurs maximales 1 et 2 (Wmax1 et Wmax2) représentant les distances
entre le plan horizontal et les valeurs Vtet Vb, puis calculer les distances entre l’origine
et les largeurs maximales (Wmax1 et Wmax2).
3. Ensuite, à l’aide des valeurs obtenues à l’étape 2, il est possible de calculer une série de caractéristiques propre à la zone d’intérêt. Le tableau 3.3 présente des exemples de caractéristiques pouvant être obtenues grâce à l’usage de la méthode Convex Hull. No. Caractéristique Définition Formule
1 Dmax Longueur de la diagonale maximale (en pixels)
2 Wmax1
Largeur maximale Wmax1 = Xmax1/Dmax
3 Wmax2 Wmax2 = Xmax2/Dmax
4 Ws Largeur de l’objet Ws = Wmax1+ Wmax2
5 Sh Symétrie horizontale Sh = Wmax2/Wmax1
6 Sv1
Demi-Symétrie verticale Sv1= Xmax1 − 1/2
7 Sv2 Sv2= Xmax2 − 1/2
8 Ac1 Aire du Convex Hull (partie
minimale et maximale) Ac1 = 1 R 0 g1(y1)dy1 9 Ac2 Ac2 = 0 R −1 g2(y2)dy2
10 Ac Aire totale du Convex Hull Ac= Ac1+ Ac2
11 Ap Aire de l’objet (soit la forme délimitée par le tracé de contour) 12 Pc Périmètre du Convex Hull Somme de la longueur des
vec-teurs de contour. 13 Rcr1 Ratio du Convex Hull versus
son rectangle convex
Rcr1 = Ac1− (Dmax∗ Wmax1)/2 (Dmax∗ Wmax1)/2 14 Rcr2 Rcr2 = Ac1− (Dmax∗ Wmax2)/2 (Dmax∗ Wmax2)/2 15 CDGa Déficience globale du Convex Hull CDGa= (Ac− Ap)/Ac
16 Rapc Facteur de l’aire de la
forme versus le périmètre du Convex Hull
Rapc = Ac/Pc2
17 Cmean Moyenne des courbes du
Convex Hull
Cmean = 1
N
PN
i=1Cur(vi)
18 Fdim1 Dimension fractale (selon
demi-périmètre haut et bas)
Fdim1 = log(peri1)/ log(Dmax)
19 Fdim2 Fdim2 = log(peri2)/ log(Dmax)
20 Corner Somme des coins du Convex Hull (max : 360 pour un cercle) Tableau 3.3 Caractéristiques obtenues à l’aide du Convex Hull d’un objet5
Note : les caractéristiques obtenues sont propres à l’objet d’intérêt, mais la représentativité de ces valeurs est fonction de l’image obtenue après les étapes de filtrage et de seuillage. Une image résultante n’ayant pas une représentation fidèle de la surface de l’objet d’intérêt
dû à une déformation ou la présence de particules causant un mauvais tracé du Convex
Hull, occasionnera l’acquisition de valeurs de caractérisation erronées.
3.2
Discrimination de catégories
Suite au prétraitement, certaines approches étudiées passent par une étape de discrimi-nation des catégories en fonction d’attributs spécifiques (caractéristiques) [Alvarez et al., 2012; Hu et Davis, 2005; Sosik et Olson, 2007]. Certains critères sont utilisés comme discriminant par plusieurs chercheurs et d’autres non. Pour cette recherche, ce sont les caractéristiques énumérées dans le tableau 3.3 qui sont utilisées comme discriminants. En analysant le tableau 3.3, on constate qu’il y a plusieurs discriminants possible. Chacun d’eux peut-être employé à la création d’un arbre décisionnel servant à départager les diffé-rentes catégories. En fonction des caractéristiques d’une image traitée, certaines catégories seront éliminées et d’autres conservées selon les résultats de comparaison. Par élimination, on désigne le retrait d’une catégorie comme candidat d’appartenance possible pour une image traitée, lorsque la valeur de l’une des caractéristiques de l’image traitée se situe à l’extérieur des limites minimale et maximale de cette même caractéristique définie pour une catégorie donnée (le calcul des limites est abordé à la sous-section 3.2.2) . Voici un exemple simplifié de cette logique :
Par exemple, si la caractéristique Dmax (diagonale maximale) d’une image traitée mesure
250 pixels. Alors si l’on remplace la variable X dans le tableau 3.4 par la valeur de 250, la logique décisionnelle sera :
Catégorie Limites pour Dmax Décision
Catégorie 1 100 ≤ X ≤ 230 +0 pour cette catégorie (Dmax est trop grand)
Catégorie 2 180 ≤ X ≤ 260 +1 pour cette catégorie (limites respectées) Catégorie 3 240 ≤ X ≤ 510 +1 pour cette catégorie (limites respectées)
Tableau 3.4 Discrimination de catégorie selon les limites d’une caractéristique
Pour cet exemple, selon le tableau 3.4 la catégorie 1 aurait un poids de 0 et les catégories 2 et 3 un poids respectif de +1. Au terme de l’analyse des caractéristiques, la ou les catégories ayant le plus grand poids sont conservées et la ou les autres catégories sont discriminées.
3.2.1
But recherché par la discrimination de catégories
L’aboutissement de cette étape de discrimination de catégories vise à simplifier et accé-lérer l’étape suivante qui est la classification d’images. En d’autres mots, moins il y a de catégories possibles pour une image donnée, moins il y a de traitements comparatifs à faire entre les catégories restantes (selon les principes d’un contre un ou un contre tous de Hsu et Lin [2002]). Le choix de la catégorie d’appartenance de l’image traitée (sa classification) se trouve alors simplifié.
3.2.2
Calcul des limites d’une caractéristique
Dans le cadre de cette recherche, le calcul de la limite minimale et maximale d’une caracté-ristique propre à une catégorie donnée est réalisé lors de l’entraînement du classificateur. L’étape d’entraînement consiste à fournir un groupe d’images ayant été préalablement identifié manuellement comme appartenant à une même catégorie. Chacune des images du groupe passe d’abord par l’étape de prétraitement de l’image (section 3.1) permettant de faire la caractérisation de celles-ci. Ensuite, toutes les caractéristiques sont regroupées par type et assignées à la catégorie d’appartenance. Au terme de ce traitement, chaque catégorie possède une liste de valeurs pour chacune des caractéristiques possibles.
La suite consiste à traiter chaque caractéristique indépendamment afin de calculer la moyenne (x) et l’écart-type (σ) de la liste de N valeurs obtenue à l’étape précédente. Pour cette recherche, il fut supposé que chacune des caractéristiques des images suivait une distribution selon la loi normale [Baillargeon, 1990]. C’est pourquoi les limites infé-rieure Xmin et supérieure Xmax furent calculées comme suit :
x = 1 N N X i=1 xi (3.5) σ = s PN i=1(xi− x)2 N − 1 (3.6) Xmin = x − (3σ) (3.7) Xmax = x + (3σ) (3.8)
Tel que spécifié dans l’ouvrage de Baillargeon [1990], l’usage de ces formules permet d’avoir une couverture de la distribution de la population de l’ordre de 99.75 %.
3.2.3
Traitement de discrimination
Deux approches possibles permettant la discrimination de catégories ont été considérées pour cette recherche. Soit une première possibilité qui est de comparer chaque caractéris-tique de l’image traitée avec chacune des limites de l’ensemble des catégories possibles. Et une seconde possibilité, qui est de définir un arbre décisionnel à intervalles pour cha-cune des caractéristiques possibles selon les intervalles minimaux et maximaux de chaque catégorie (chacune des caractéristiques de l’image traitée doit passer dans chacun des arbres décisionnels appropriés). Au terme des deux approches, la liste des catégories non discriminées (donc ayant le plus grand poids) est transmise au classificateur SVM.
Pour déterminer quelle est la meilleure approche de discrimination de catégories, une analyse du temps de traitement requis par approche a été réalisée. D’abord, le premier choix possible nécessite la comparaison de N caractéristiques d’une image en fonction de
M catégories, ce qui donne un nombre de comparaisons possibles par image de M × N
(donc le maximum de traitement possible en tout temps). Tandis que le second choix offre la possibilité de diminuer le nombre de comparaisons nécessaires par caractéristique. Par exemple, un objet d’intérêt ayant une très grande surface (aire) ne devrait pas être comparé avec toutes les catégories ayant une petite surface. Cette comparaison devrait se faire uniquement avec les catégories ayant une large surface. C’est pourquoi l’arbre décisionnel à intervalles a été retenu dans cette recherche pour cette partie du traitement. Tel que décrit dans l’article de Safavian et Landgrebe [1991], l’arbre décisionnel vise à diminuer le nombre de décisions nécessaire à l’atteinte d’une solution désirée, en regroupant les noeuds et/ou feuilles ayant des similitudes dans une même section d’un arbre.
3.2.4
Arbre décisionnel à intervalles
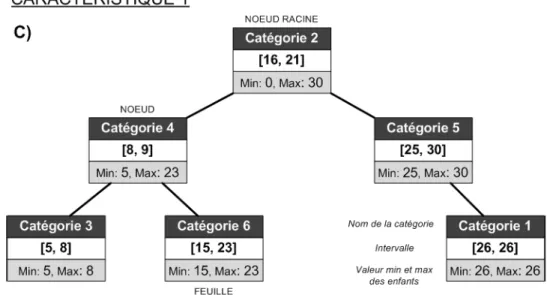
Un arbre décisionnel à intervalles (« Interval Tree ») est une structure organisée sous la forme d’un arbre inversé. Cette structure est composée de noeuds et de feuilles. Chaque noeud a deux enfants et un parent (sauf le noeud racine qui n’a pas de parent) et chaque feuille à un parent, mais n’a pas d’enfant(s). Le parent d’un noeud est le noeud précédent son atteinte et ses enfants sont les noeuds et/ou feuilles au bout de la ou des branches de ce noeud [Safavian et Landgrebe, 1991]. Les figures 3.9 et 3.10 présentent les étapes de création d’un arbre décisionnel à intervalles.
Figure 3.9 Étapes A et B de création d’un arbre décisionnel à intervalles6
Figure 3.10 Étape C de création d’un arbre à intervalles7
La première étape pour créer un arbre à intervalles consiste à regrouper les catégories et leur intervalle en fonction d’une caractéristique donnée A). Ensuite, il faut trier les catégories selon la limite minimale de leur intervalle, de la plus petite valeur à la plus grande B). Finalement, il y a la création de l’arbre à intervalles dont les noeuds et feuilles sont positionnés de gauche à droite en fonction de la limite inférieure de l’intervalle de
6Inspiré de [Cormen et al., 1989] 7Inspiré de [Cormen et al., 1989]
ces noeuds et feuilles C). Chaque intersection de l’arbre (noeud ou feuille), contient un identifiant qui est le nom de la catégorie représentée, un intervalle qui est la valeur minimale et maximale couverte par la caractéristique de la catégorie, puis une valeur minimale et maximale qui donne l’intervalle de couverture du noeud jumelée avec celles de ses enfants (s’il y a lieu) [Cormen et al., 1989].
Un exemple d’utilisation de l’arbre à intervalles de la figure 3.10 est décrite ci-après. Supposons qu’une image X a 26 comme valeur pour sa caractéristique 1. Donc la séquence de traitement sera la suivante :
1. Selon la valeur minimale et maximale du noeud racine est ce que la valeur 26 est à l’intérieure de l’intervalle ? Oui, [0, 30] donc on poursuit l’analyse.
2. Selon le noeud actuel (racine), est-ce que 26 est à l’intérieur de son intervalle ? Non, cette intervalle étant [16, 21] cette catégorie est rejetée.
3. Est-ce 26 est à l’intérieur de la valeur minimale et maximale de l’enfant de gauche de ce noeud ? Non, [5, 23] donc aucun passage à gauche.
4. Est-ce 26 est à l’intérieur de la valeur minimale et maximale de l’enfant de droite de ce noeud ? Oui, [17, 30] donc passage à l’enfant de droite.
5. Selon le noeud actuel, est-ce que 26 est à l’intérieur de son intervalle ? Oui, étant [25, 30] le poids de la catégorie 5 est augmenté de 1 (+1).
6. La suite consiste à répéter la séquence à partir de l’étape 3, jusqu’à ce qu’il n’y ait plus d’intervalles respectant la valeur analysée.
Tel que cité précédemment, un arbre décisionnel à intervalles doit être créé pour chacune des caractéristiques utilisées et le total final du poids de chaque catégorie, résultant du passage dans tous les arbres, détermine les catégories étant transférées au classificateur (voir la section 3.3).
3.3
Classification
Cette étape finale du processus de classification d’une image consiste à déterminer la catégorie à laquelle l’image de phytoplancton appartient. Telle que définie précédemment dans ce mémoire de recherche, la méthode de classification SVM a été choisie pour le projet Biotaxis.
Le développement d’un classificateur à généralement pour but de répondre à un problème spécifique. L’objectif premier est de classifier correctement différents objets, et ce, avec la plus grande précision possible [Abe, 2010]. L’idée directrice est donc de définir un ou des modèles décisionnels permettant d’obtenir la meilleure approximation de classification pour l’ensemble des objets observés.
3.3.1
Classification SVM linéaire
Le cas le plus simple pour l’élaboration d’un classificateur est lorsque l’on peut appliquer un séparateur linéaire. Donc, lorsqu’il est possible de séparer les 2 classes d’un univers de données Rn par un hyperplan linéaire. Le livre de Hamel [2009] décrit bien cette
pro-blématique et l’encadré, présenté ci-après, est une traduction d’un modèle général de représentation de la classification SVM linéaire décrit dans ce volume8 :
- Soit l’univers de données Rn, avec les vecteurs ~x ∈ Rn représentant les objets.
- Un échantillon de donné S, où S ⊂ Rn.
- Une fonction cible f : Rn → {+1, -1}.
- Un groupe de formation pré identifié D = {(~x,y) | ~x ∈ S et y = f(~x)}.
Sur cette base, il faut déterminer une fonction f : Rb n → {+1, -1} en utilisant D de
sorte que : f (~bx) ∼= f (~x), pour tous ~x ∈ Rn (donc que le modèle f soit une bonneb
approximation de la fonction f ).
Cet ensemble d’informations requiert de plus amples explications :
- D’abord, l’univers de données est représenté par l’espace Rn, où n représente les
différents plans de cet univers.
- L’échantillon de données S est constitué d’un groupe de données devant représenter adéquatement l’ensemble des données de l’univers. Leur sélection doit être faite de façon à ce que chacune des catégories possibles ({+1, -1} dans le cas présent) soit bien définie.
- Le modèle f définit la surface décisionnelle qui est représentée par une ligne, unb
plan ou un hyperplan séparant les différentes classes dans l’univers Rn.
- Dans le cas plus complexe d’une classification de classes multiples (multiclasses), les valeurs en sortie {+1, -1} sont alors remplacées par l’ensemble des valeurs possibles soient {1, 2, ..., M }.
8Le texte dans l’encadré est une traduction de l’anglais dont le contenu provient du livre de Hamel
Note : lorsqu’un groupe de formation préidentifiée D est utilisé pour définir un modèle
b
f , alors le modèle sera dit basé sur un apprentissage supervisé. Inversement, s’il n’y a pas de groupe de formation D utilisé alors l’apprentissage du modèle sera non supervisé.
9
En partant du groupe de formation prédéfini D, il est possible de projeter sur un graphique l’ensemble des vecteurs ~x qui le composent afin de trouver le modèle f . Voici l’exempleb
d’un graphique bidimensionnel représentant cette projection selon un classificateur SVM linéaire :
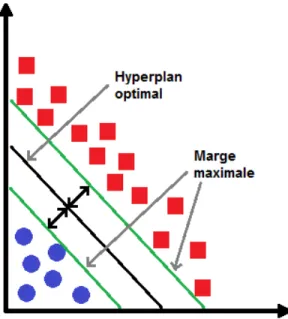
Figure 3.11 Classificateur SVM avec hyperplan optimal et marge maximale
Le graphique de la figure 3.11 présente des informations nécessitant une attention particu-lière. Pour débuter, on retrouve un ensemble de points représenté par deux formes (cercle et carré). Les cercles peuvent être remplacés par yi = +1 et les carrés par yi = −1, selon
l’équation D = {(~x,y) | ~x ∈ S et y = f (~x)} définie précédemment. Ensuite on retrouve
deux hyperplans de support (marge maximale), qui sont des traits parallèles distants de
2
kwk. Ceux-ci définissent les limites maximales de séparation entre les cercles (+1) et les
carrés (−1). à partir des équations représentant ces hyperplans, il est possible d’obtenir la forme générale suivante :
- Sachant que w · ~xi - b ≥ +1
- et que ~w · ~xi - b ≤ -1
- alors on obtient la formule générale : yi( ~w · ~xi - b) - 1 ≥ 0, pour i = 1..., M .
9Le texte dans l’encadré est une traduction de l’anglais dont le contenu provient du livre de Hamel
Le tracé nommé hyperplan optimal qui divise uniformément les deux hyperplans de sup-port est représenté par l’équation ~w · ~x - b = 0, qui représente la surface maximale de
décision. Le but recherché par l’ensemble de ce processus est de définir un hyperplan permettant d’obtenir la plus grande précision de classification possible.
3.3.2
Classification SVM non linéaire
Après la classification SVM linéaire vient la classification SVM non linéaire. Cette dernière survient lorsque le classificateur ne peut pas être défini directement par un hyperplan linéaire. Pour être en mesure de concevoir un classificateur non linéaire, il faut utiliser le « kernel trick » [Hamel, 2009]. L’idée de cette approche est de transformer un ensemble de données non séparable linéairement en un espace dimensionnel supérieur, appelé espace fonctionnel (feature space), où les données peuvent être séparées linéairement [Hamel, 2009]. La figure 3.12 illustre le but recherché par l’application du « kernel trick » :
Figure 3.12 Mappage d’un problème non linéaire vers un espace supérieur
En partant de la fonction décisionnelle f décrite précédemment et en la reconstruisantb
selon le mappage de l’espace initial vers l’espace fonctionnel Φ, on obtient alors [Hamel, 2009] :
b
f (~x) = sgn( ~w∗· Φ(~x) − b∗) avec Φ : Rn → Rm, où m ≥ n (3.9) Le vecteur normal ~w∗ de cette fonction décisionnelle peut-être représenté par la dualité suivante : ~w∗ = Pl
i=1α
∗
iyiΦ(~xi). En remplaçant ~w∗ par sa dualité dans l’équation 3.1, on
obtient la fonction décisionnelle :
b f (~x) = sgn( l X i=1 α∗iyiΦ(~xi) · Φ(~x) − b∗) (3.10)
Par définition la représentation d’une fonction kernel est donnée par k(~x, ~y), où ~x, ~y ∈ Rn.
de la fonction kernel, ce qui donne : b f (~x) = sgn( l X i=1 α∗iyik( ~xi, ~x) − b∗) (3.11)
Cette série d’étapes mathématiques représente l’application du « kernel trick ». Soit d’ap-pliquer un kernel à la fonction décisionnelle afin de faire le mappage direct des données vers l’espace fonctionnel. La décision restante est le choix du kernel à employer, soit d’établir celui qui via le mappage sépare le mieux les différentes classes afin de pouvoir les subdivi-ser par un hyperplan linéaire. Le tableau 3.5 présenté ci-après, énumère quelques-uns des
kernels pouvant être utilisés :
Nom du kernel Fonction du kernel
Kernel linéaire k(~x, ~y) = ~x · ~y Kernel polynomial homogène k(~x, ~y) = (~x · ~y)d Kernel polynomial non homogène k(~x, ~y) = (~x · ~y + c)d Kernel gaussien k(~x, ~y) = e−(|~x·~y|2/2β2)
Tableau 3.5 Quelques kernels fréquemment utilisés10
Il est important de souligner que parmi les différents articles référés traitants de la classi-fication d’images de phytoplancton, nombreux sont ceux qui ont utilisé le kernel gaussien (aussi appelé radial basis function kernel ou RBF kernel) avec la méthode SVM [Alvarez
et al., 2012; Luo et Kramer, 2004; Sosik et Olson, 2007; Su et al., 2010; Verikas et al.,
2012]. Dans le cadre de cette recherche, c’est le kernel gaussien qui a été employé. Ce choix étant appuyé par la citation de son usage dans les articles cités ci-haut et dû à l’article de Hsu et al. [2010] qui présente des motifs justifiant son utilisation (voir l’encadré ci-après).
L’article de Hsu et al. [2010], qui est un guide sur la classification SVM, cite que le RBF
kernel est un premier choix raisonnable dû aux raisons suivantes :
- Il peut être appliqué aux classes ayant une relation non linéaire (impossible avec le
kernels linéaire).
- Il a moins de paramètres à configurer que les kernels polynomiaux (donc moins complexe que ceux-ci).
- Il donne des valeurs comprises entre 0 et 1, comparativement aux kernels poly-nomiaux dont les valeurs varient entre 0 et l’infini positif (présentant moins de difficultés numériques).
3.3.3
Traitement de classes multiples
Il arrive parfois que l’on ait à effectuer la classification de classes multiples. L’une des approches les plus souvent utilisées consiste à décomposer le problème en plusieurs sous-problèmes de classification binaire. Cette représentation binaire peut se faire de deux façons différentes [Hamel, 2009] :
- La première option se nomme un contre tous (one-vs-all). Cette méthode consiste à séparer chaque classe du reste des autres classes. Ainsi pour un nombre de classes possible M , il y aura alors M classifications binaires à effectuer.
- La deuxième option se nomme un contre un (one-vs-one). Cette dernière consiste à établir toutes les combinaisons possibles de jumelage par pair entre chacune des classes disponibles. L’usage de cette approche requiert un total de M (M − 1)/2 classifications binaires.
L’étude de Hsu et Lin [2002] sur les deux méthodes énoncées ci-haut a démontré que dans de nombreuses situations, l’approche un contre un était plus rapide et plus précise que l’approche un contre tous lors de la résolution d’un problème de classification SVM. Luo et Kramer [2004] ont fait des tests similaires avec des images de phytoplancton et sont arrivés à la même conclusion que Hsu et Lin, c’est-à-dire que l’approche un contre un est préférable à l’approche un contre tous.
3.4
Précision et temps de traitement
À titre de rappel, les objectifs visés par cette recherche sont d’obtenir une précision de la classification de 87 % et un temps moyen de classification de 200 ms par image, ce qui équivaut à 5 images par seconde. Il est donc important d’élaborer sur les méthodes employées pour le calcul de ces deux valeurs.
Pour obtenir la valeur de la précision de la classification l’article de Fawcett [2006] a été utilisé comme référence. La figure 3.13 présente de quelle façon la précision est calculée.
Figure 3.13 Matrice de confusion et calcul de la précision11
Dans la figure 3.13, trois types d’assignation à une image classifiée requiert des explications pour pouvoir passer par la suite au deux équations présentes dans cette même figure. Ces trois types d’assignation sont les suivants :
- Le terme TP qui représente l« True Positive » (Vrai Positif) désigne une image dont on connaît la catégorie d’appartenance et qui a été classifiée dans la bonne catégorie. - FN qui signifie « False Negative » (Faux négatif) représente l’assignation d’une
catégorie autre que la catégorie réelle à une image donnée.
- Si une image est classifiée dans une autre catégorie que sa catégorie d’appartenance, alors celle-ci sera comptée comme « False Positive » FP (Faux Positif).
- Finalement, le terme TN représente une image n’ayant pas été assignée à une mau-vaise catégorie.
Dans le contexte présent, différents groupes d’images test, dont la catégorie d’appartenance était déjà connue, a été utilisée pour calculer la précision du classificateur. En se basant sur la formule du calcul de la précision énoncé dans la figure 3.13, la précision a été calculée par catégorie selon la moyenne de la précision de la classification de l’ensemble des images d’une catégorie. Puis globalement, par le calcul de la moyenne de la précision de la classification selon la valeur de la précision de l’ensemble des catégories.
La formule de précision de la figure 3.13 représente le ratio de bonne classification (TP) versus la sommation de bonne (TP) et de mauvaise classification dans des catégories autres que la catégorie réelle (FP).