HAL Id: hal-02800309
https://hal.inrae.fr/hal-02800309
Submitted on 5 Jun 2020HAL is a multi-disciplinary open access archive for the deposit and dissemination of sci-entific research documents, whether they are pub-lished or not. The documents may come from teaching and research institutions in France or abroad, or from public or private research centers.
L’archive ouverte pluridisciplinaire HAL, est destinée au dépôt et à la diffusion de documents scientifiques de niveau recherche, publiés ou non, émanant des établissements d’enseignement et de recherche français ou étrangers, des laboratoires publics ou privés.
Mécanismes impliqués lors de la récupération musculaire
suite à une immobilisation chez le rat
Julie Calonne
To cite this version:
Julie Calonne. Mécanismes impliqués lors de la récupération musculaire suite à une immobilisation chez le rat. [Stage] France. Université d’Auvergne - Clermont-Ferrand I (UdA), FRA. 2014, 23 p. �hal-02800309�
Master Sciences, Technologie et Santé Mention « Nutrition et Sciences des Aliments »
Spécialité « Nutrition Humaine et Santé » Promotion 2013-2014
Mécanismes impliqués lors de la
récupération musculaire suite à une
immobilisation chez le rat
Rapport de stage de M2
Etudiante : Julie CALONNE
Responsable du stage : Lydie COMBARET
Responsable de la spécialité : Didier ATTAIX Rapporteurs : Christelle GUILLET
Marie-Chantal FARGES Laboratoire d’accueil
Equipe protéolyse, UMR 1019, Unité de Nutrition Humaine INRA de Clermont-Theix
Master Sciences, Technologie et Santé Mention « Nutrition et Sciences des Aliments »
Spécialité « Nutrition Humaine et Santé » Promotion 2013-2014
Mécanismes impliqués lors de la
récupération musculaire suite à une
immobilisation chez le rat
Rapport de stage de M2
Etudiante : Julie CALONNE
Responsable du stage : Lydie COMBARET
Responsable de la spécialité : Didier ATTAIX Rapporteurs : Christelle GUILLET
Marie-Chantal FARGES Laboratoire d’accueil
Equipe protéolyse, UMR 1019, Unité de Nutrition Humaine INRA de Clermont-Theix
REMERCIEMENTS
Je tiens tout d’abord à remercier Didier ATTAIX, Directeur de Recherche pour m’avoir accueillie dans son équipe Protéolyse.
Mes remerciements vont ensuite vers Lydie COMBARET, Directeur de Recherche qui a été mon responsable durant ce stage. Je te remercie pour ta disponibilité, ta gentillesse et tes précieux conseils pour mener à bien ces travaux.
Je souhaite remercier également Christiane DEVAL, Ingénieur au sein de l’équipe, qui a répondu à toutes mes questions et qui m’a guidée et épaulée lors des manipulations.
Enfin, d’une manière générale, je souhaite remercier tous les membres de l’équipe protéolyse (Cécile POLGE, Agnès CLAUSTRE, Cécile COUDY, Daniel TAILLANDIER et Daniel BECHET) pour l’accueil qu’ils m’ont réservé, les conseils qu’ils ont pu me donner ainsi que pour leur gentillesse.
SOMMAIRE
Remerciements Liste des abréviations Liste des figures Liste des tableaux
INTRODUCTION...1
BIBLIOGRAPHIE...2
1. Le muscle squelettique...2
1.1 Structure et fonction...2
1.2 Altérations au cours de l’immobilisation et pendant la récupération...3
2. Rôle de l’autophagie dans le maintien de la masse musculaire...3
2.1 Mécanismes de la dégradation lysosomale-autophagie-dépendante...4
2.2 Implication de ce système pendant l’immobilisation et la récupération...5
3. La mitochondrie...5
3.1 Généralités...5
3.2 Remodelage mitochondrial...6
3.3 Le « turnover » des mitochondries...7
3.4 Altérations mitochondriales au cours de l’immobilisation et de la récupération...7
4. Objectifs de l’étude et stratégie expérimentale...8
MATERIELS & METHODES...9
1. Modèle d’étude...9
2. Techniques expérimentales...9
2.1 Mesure des niveaux d’ARNm par RT-qPCR...9
2.1.1 Dosage, traitements des ARN et rétrotranscription...9
2.1.2 Dessin des amorces et RT-qPCR...10
2.2 Analyse des niveaux protéiques par Western Blot...10
2.2.1 Préparation des échantillons...10
2.2.2 Mise au point des conditions d’hybridation...11
2.2.3 Western Blot...12
RESULTATS & DISCUSSION...14
1. Etude de l’autophagie au cours des phases d’immobilisation et de remobilisation musculaire dans le muscle tibialis anterior chez le rat...14
1.1 Phase d’initiation et de formation des autophagosomes...14
1.2 Phase de fusion...15
2. Etude des altérations mitochondriales au cours des phases d’immobilisation et de remobilisation musculaire dans le muscle tibialis anterior chez le rat...16
2.1 La biogenèse mitochondriale...16
2.2 La dynamique mitochondriale...17
2.3 La mitophagie...19
CONCLUSIONS & PERSPECTIVES...21
REFERENCES BIBLIOGRAPHIQUES...22
ABRÉVIATIONS
ADN : Acide DesoxiriboNucléique ANOVA : ANalysis Of VAriance ARNm : Acide RiboNucleique messager ATG : AuTophagy-related Gene
ATP : Adénosine TriPhosphate BSA : Bovine Serum Albumine
CaMKIα : Calcium/Calmodulin-dependent protein kinase α CT : Cycle Threshold
CTRL : patte controlatérale
DRP1 : Dynamin Related Protein 1
FIP200 : FAK family kinase-interacting protein of 200 kDa FIS1 : mitochondrial Fission Protein 1
GA : muscle Gastrocnemius
GABARAPL1 : GABA(A) receptor-associated protein like 1 GTP : Guanosine TriPhosphate
HR2 : Heat Repeat 2
HRP : Horseradich peroxidase HU : Hindlimb Unloading IMF : Inter myofribrillar IMM : patte immobilisée
LC3 : microtubule-associated-protein-Light-Chain3 MFF : Mitochondrial Fission Factor
MFN1/2 : Mitofusin 1/2
MiD49/51 : Mitochondrial Dynamic Protein of 49/51 kDa mTOR : mammalian Target Of Rapamycin
MyHC : Myosin Heavy Chain OPA1 : Optic Atrophy 1 PARKIN : Parkinson protein 2
PARL : Presenilin-Associated Rhomboid-Like serine protease PE : Phosphatidylinositol
PINK1 : Phosphatase and tensin homologue-Induced Putative Kinase 1 PI3K : Phosphatidyl Inositide 3 Kinase
PKA : Protein Kinase A
PLSD : Protected Least Significant Difference PVDF : PolyVinyl DiFluoride
RAB : Ras-related protein in brain RE : Réticulum endoplasmique RIN : RNA Integrity Number
RT- qPCR : Real Time quantitative Polymerase Chain Reaction SEM : Standard Error of Mean
SNARE : Soluble N-ethylmaleimide-sensitive fusion Attachment protein REceptors SS : SubSarcolemmal
TA : muscle Tibialis Anterior
TBST : Tris-Buffer Saline + Tween20 TGN : Trans Golgien Network
Ub : Ubiquitine
ULK1/2 : Unc-51 like autophagy activating kinase 1/2 UPS : Ubiquitin Proteasome-dependent System VSP34 : Vacuolar sorting protein-34
LISTE DES FIGURES
Figure 1 : Structure du muscle squelettique.
Figure 2 : Evolution de l’atrophie musculaire dans le Gastrocnemius et dans le Tibialis Anterior suite à l’immobilisation par plâtrage chez le rat adulte.
Figure 3 : L’autophagie : voie d’élimination de protéines et d’organelles déficientes. Figure 4 : Schémas des phénomènes de fusion (a) et fission (b) mitochondriale chez les mammifères.
Figure 5 : Illustration des différents modèles de fission mitochondriale chez les mammifères. Figure 6 : La mitophagie : voie d’élimination des mitochondries par un processus
autophagique.
Figure 7 : Schéma du protocole expérimental.
Figure 8 : Stratégies utilisées pour la mise en place des Western Blots.
Figure 9 : Régulation des phases d’initiation et de formation des autophagosomes de l’autophagie dans le tibialis anterior immobilisé et remobilisé.
Figure 10 : Régulation de la phase terminale de l’autophagie dans le tibialis anterior immobilisé et remobilisé.
Figure 11 : Régulation de la biogenèse mitochondriale dans le tibialis anterior immobilisé et remobilisé.
Figure 12 : Régulation de la fusion mitochondriale dans le tibialis anterior immobilisé et remobilisé.
Figure 13 : Régulation de la fission mitochondriale dans le tibialis anterior immobilisé et remobilisé.
Figure 14 : Régulation de la fusion mitochondriale dans le tibialis anterior immobilisé et remobilisé.
LISTE DES TABLEAUX
Tableau 1 : Amorces utilisées pour les RT-qPCR.
1
INTRODUCTION
Le muscle squelettique représente 40% des protéines corporelles et constitue le principal réservoir d’acides aminés mobilisables par l’organisme lors de situations cataboliques pour assurer un certain nombre de fonctions : la synthèse protéique des organes vitaux (cerveau, cœur, poumon, ...), la réponse immunitaire, la réponse inflammatoire, et la néoglucogenèse hépatique. Ceci entraîne une fonte musculaire qui conduit, lorsqu’elle perdure, à un affaiblissement généralisé des individus pouvant aller jusqu’à l’alitement. La baisse d’activité physique, voire l’immobilisation, qui en découle aggrave la fonte musculaire déjà existante. Les phases d’atrophie musculaire sont suivies de phases de récupération dont la durée dépend de l’état physiopathologique de l’individu et de la nature de l’agression. Si un second évènement catabolique survient avant la récupération complète de l’évènement précédent, une fonte musculaire additionnelle s’observe et impacte d’autant plus l’organisme. A terme, l’atrophie musculaire conduit à une baisse d’efficacité des traitements, une perte d’autonomie des patients, une augmentation de la morbidité, et contribue à élever les coûts de santé publique. Il est donc nécessaire de comprendre les mécanismes impliqués lors des phases d’atrophie et de récupération musculaire pour permettre le développement de stratégies visant à limiter ces altérations.
Les protéines musculaires sont en constant renouvellement et la masse musculaire dépend de l’équilibre entre la synthèse et la dégradation des protéines musculaires. L’atrophie musculaire survient lorsque la protéolyse devient supérieure à la synthèse protéique. Des données précédentes du laboratoire 1) indiquaient que l’atrophie musculaire observée dans le muscle tibialis anterior (TA) lors de l’immobilisation et dans les phases précoces de récupération résultait d’une activation persistante de la protéolyse ubiquitine-protéasome dépendante, mais 2) suggéraient également que l’autophagie pourrait jouer un rôle dans la période de récupération qui s’ensuit. En effet, parmi les différents systèmes protéolytiques, le système autophagique joue aussi un rôle clé dans le maintien de la masse musculaire car il permet non seulement de dégrader des protéines musculaires, mais également d’éliminer les mitochondries anormales ou déficientes. Quelques études montrent des altérations du réseau mitochondrial dans des modèles d’inactivité musculaire.
Mon stage a donc eu pour objectif de mieux comprendre le rôle de l’autophagie, ainsi que les altérations du réseau mitochondrial dans les phases d’atrophies du muscle tibialis anterior suite à l’immobilisation et à la remobilisation chez le rat.
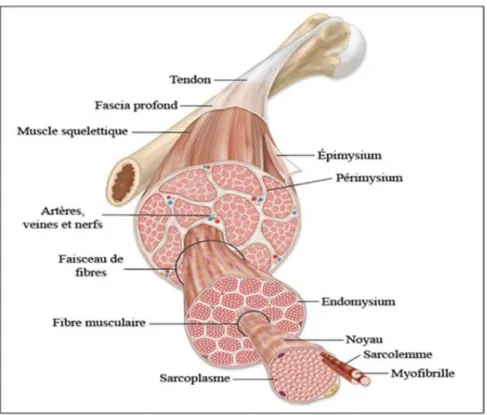
Figure 1 : Structure du muscle squelettique
Source : Thèse de Jean-Christophe DOMINIQUE, 2009 « Amélioration du taux de greffe de cellules myogéniques pour la dystrophie musculaire de Duchenne », Université de Laval, Canada, http://theses.ulaval.ca/archimede/fichiers/25962/25962.html.
2
BIBLIOGRAPHIE
1. Le muscle squelettique 1.1 Structure & Fonction
Il existe 3 types de tissus musculaires : le muscle squelettique, le muscle cardiaque, et le muscle lisse. Le muscle squelettique permet de réaliser les mouvements volontaires. Il joue également un rôle dans le maintien de la posture, dans la production d’énergie, dans la réponse immunitaire, et enfin dans la thermogenèse.
Le muscle squelettique (Figure 1) est constitué de plusieurs types de tissus : musculaire, nerveux, vasculaire, adipeux et conjonctif. Il est formé de fibres musculaires (également appelées cellules musculaires ou myocytes) qui peuvent atteindre plusieurs centimètres de long et jusqu’à 80-90 µm de diamètre. Chacune d’entre elles est entourée par le sarcolemme, qui constitue la membrane cellulaire. Ces cellules sont plurinucléées et les noyaux sont situés à leur périphérie. Le cytoplasme, nommé sarcoplasme contient les protéines contractiles (myofibrilles), de nombreuses mitochondries, le réticulum sarcoplasmique, etc…
Les fibres musculaires sont organisées en faisceaux et sont composées de myofibrilles qui contiennent les protéines contractiles (actine et myosines). Les myofibrilles sont composées de sarcomères qui constituent les éléments contractiles du muscle (Figure 1). La tête globulaire de la myosine porte l’activité ATPase et permet la contraction, alors que la queue se compose des 2 chaines lourdes de myosine (MyHC). L’élément déclenchant de la contraction musculaire est une élévation de la concentration calcique associée à la présence d’ATP dans la cellule musculaire. Il se produit alors un raccourcissement des sarcomères dû au glissement des filaments fins d’actine et des filaments épais de myosine. Ce phénomène se produisant dans l’ensemble des sarcomères présents dans la cellule, il en résulte un raccourcissement global des fibres musculaires.
Il existe différents types de fibres que l’on distingue par leurs propriétés métaboliques et contractiles. Les fibres de type I sont de petites fibres à contraction lente, majoritairement oxydatives car elles possèdent de nombreuses mitochondries. Ces fibres confèrent une grande résistance à des efforts physiques de longue durée. Les fibres de type IIB sont des fibres de grande taille qui, contrairement aux fibres de type I, sont plutôt glycolytiques et possèdent moins de mitochondries. Ces fibres sont davantage sollicitées pour des efforts de courte durée et d’intensité élevée. Il existe également des fibres de type IIA et IIX qui présentent des caractéristiques intermédiaires aux fibres I et IIB.
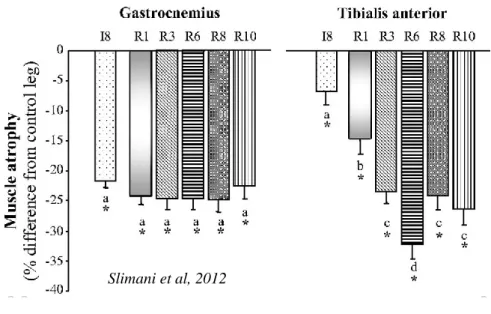
Figure 2 : Evolution de l’atrophie musculaire dans le Gastrocnemius et dans le Tibialis Anterior suite à l’immobilisation par plâtrage chez le rat adulte.
Les rats ont été immobilisés pendant 8 jours (I8) et placés en récupération pendant 1, 3, 6, 8 et 10 jours (R1-R10).
L’atrophie musculaire est exprimée en % des différences par rapport à la patte controlatérale. Les valeurs représentent des moyennes ± SEM pour n=9-11 rats/groupe.
Les barres d’histogramme qui portent des lettres différentes sont statistiquement différentes (ANOVA). *, P < 0,05 vs. Patte controlaterale (Test t apparié de Student).
3
1.2 Altérations au cours de l’immobilisation et pendant la récupération
Le muscle présente une plasticité qui se traduit par des phénomènes d’hypertrophie et d’atrophie. La perte de masse musculaire s’accompagne de changements structuraux, fonctionnels et métaboliques. Les périodes de récupération musculaire sont indispensables pour retrouver une fonction musculaire normale. L’inactivité musculaire se traduit par une perte de masse musculaire. Par exemple dans un modèle de suspension (HU), la masse du muscle soleus est réduite de 50% après 3 semaines de HU, alors qu’elle n’est plus que de 36% après 7 jours de désuspension (Liu et al. 2014). En revanche, dans un modèle d’immobilisation par plâtrage, l’atrophie des muscles gastrocnemius (GA) et tibialis anterior (TA) était respectivement de 25 et 16% après 8 jours d’immobilisation. Par contre, dès déplâtrage, l’atrophie du GA s’est stabilisée, alors que celle du TA s’est accentuée pour atteindre 32% après 6 jours de remobilisation (Figure 2) (Slimani et al. 2012). Ceci conduit à un retard de récupération de la masse du TA encore atrophié de 10% même après 40 jours de remobilisation (Vazeille et al. 2008). Les mécanismes qui interviennent pendant la période de remobilisation sont donc très variables d’un muscle à l’autre et d’un modèle à l’autre.
L’atrophie qui résulte de l’immobilisation implique une baisse de synthèse protéique (Magne et al. 2012, Savary-Auzeloux et al. 2013) et une augmentation de la protéolyse (Vazeille et al. 2008, Slimani et al. 2012). Plusieurs systèmes protéolytiques permettent de fournir les acides aminés à l’organisme. Cependant, 2 d’entre eux sont majoritairement responsables du contrôle de la masse musculaire : le système ubiquitine protéasome-dépendant (UPS) et l’autophagie (Bonaldo and Sandri. 2013). Une activation du système UPS a été largement décrite dans les muscles atrophiés suite à l’immobilisation (Vazeille et al. 2008, Slimani et al. 2012, Savary-Auzeloux et al. 2013 ; Magne et al. 2012) ou la suspension (Taillandier et al. 1996, Taillandier et al. 2003). Le rôle de l’autophagie a cependant été beaucoup moins décrit, alors qu’il pourrait jouer un rôle non seulement dans les processus d’atrophie, mais également de récupération musculaire.
2. Rôle de l’autophagie dans le maintien de la masse musculaire
L’autophagie a lieu à l’état basal dans toutes les cellules pour renouveler les protéines et certains organites. Dans le muscle squelettique, l’autophagie intervient dans le maintien de sa masse (Masiero et al. 2009). En effet, elle permet d’éliminer les organelles déficientes comme les mitochondries endommagées qui pourraient produire des radicaux libres par exemple. On appelle cette forme d’autophagie, la mitophagie (Cf Partie Bibliographie § 3.2.3 Le « turnover » des mitochondries).
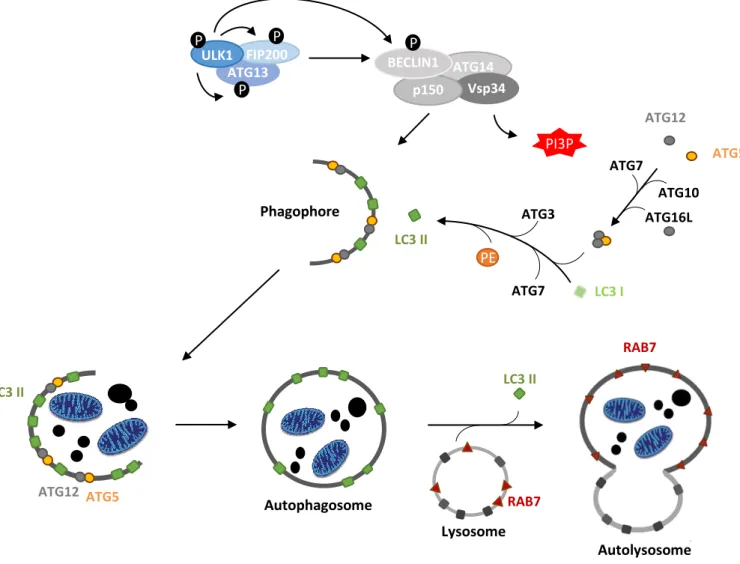
Figure 3 : L’autophagie : voie d’élimination de protéines et d’organelles déficientes
L’autophagie est initiée après activation de ULK1 qui phosphoryle alors ATG13 et FIP200, conduisant à la phosphorylation de BECLIN1 et au recrutement d’autres membres du complexe PI3K (ATG14, p150 et Vsp34). Vsp34 est indispensable à l’initiation de l’autophagie puisqu’elle induit la formation de PI3P qui permet le recrutement du complexe ATG5-ATG12. Celui-ci se forme via l’action d’ATG7 (enzyme E1-like) et ATG10 (enzyme E2-like), permettant le recrutement d’ATG16L, afin de former un complexe indispensable à la lipidation de LC3. LC3-II est la forme lipidée de LC3-I (protéine cytosolique) dont le précurseur est la protéine pro-LC3. Le clivage de pro-LC3 au niveau C-terminal par la protéase ATG4 aboutira à LC3-I. Un groupement phosphatidylethanolamine (PE) est ensuite fixé à LC3-I via l’action d’ATG7 et ATG3, pour aboutir à LC3-II. L’association des complexes ATG5-ATG12 et LC3-II va entrainer l’élongation de la membrane du phagophore via le recrutement de phospholipides intracellulaires pour former une vésicule à double membrane appelée autophagosome. Enfin, l’autophagosome va fusionner avec les endosomes (précoces puis tardifs) pour former les amphisomes, puis avec les lysosomes pour former les autolysosomes. Cette étape est médiée, entre autres, par RAB7 qui est une protéine GTPase.
ATG : Autophagy related gene ; ULK1/2 : Unc-51-like kinase 1/2 ; FIP200 : 200 kDa FAK-family interacting protein ; PE : phosphatidylethanolamine ; PI3P : Phosphatidyl Inositol 3 Phosphate ; LC3 : microtubule-associated-protein-Light-Chain3 ; FIP200 : FAK family kinase-interacting protein of 200 kDa ; VSP34 : Vacuolar sorting protein-34 ; RAB : Ras-related protein in brain
Autophagosome LC3 II ATG5 ATG12 LC3 II RAB7 Lysosome ATG13 FIP200 ULK1 ATG14 Vsp34 p150 BECLIN1 1 ATG12 LC3 II LC3 I ATG5 ATG3 ATG7 ATG16L Phagophore PI3P ATG10 ATG7 P PE P P P RAB7 Autolysosome
4
2.1 Mécanismes de la dégradation lysosomale-autophagie-dépendante
Il existe différents types d’autophagie mais la principale voie est la macroautophagie. Elle se déroule en plusieurs étapes, 1) la phase d’initiation, 2) la phase de formation des autophagosomes, et 3) la phase de fusion des autophagosomes avec les lysosomes (Mizushima et al. 2011) (Figure 3). L’autophagie est donc un ensemble d’étapes dans lesquelles de nombreux acteurs interviennent. Parmi eux, les gènes indispensables ATG (Autophagy related genes) d’abord découverts chez la levure (Klionsky et al. 2003), et hautement conservés chez les mammifères (Ashrafi et al. 2013).
Durant la phase d’initiation, les portions de cytoplasme destinées à être dégradées sont englobées par une membrane nommée phagophore, le tout initié par les complexes ULK (Unc-51-like kinase 1/2) et PI3K (Phosphatidylinositol 3 Kinase). Plusieurs hypothèses existent quant à l’origine de cette membrane (mitochondrie, réticulum endoplasmique (RE), membrane plasmique).
La présence de PI3P sur la membrane du phagophore permet le recrutement, dans un 1er
temps, du complexe ATG5-ATG12, puis dans un 2ème temps, de II sur ce complexe.
LC3-II a été la première protéine identifiée comme étant associée de manière spécifique à la membrane de l’autophagosome. L’évaluation de son abondance permet, d’estimer celle des autophagosomes formés (Kabeya et al. 2000).
Enfin, la dernière étape de l’autophagie est la fusion des autophagosomes avec les lysosomes. Cette fusion est médiée, entre autres, par des protéines de la famille des RAS-related protein in brain (RAB). Ces protéines RABs régulent les processus de transport des vésicules. Parmi toutes les protéines RABs connues à ce jour, certaines d’entre elles (RAB7, RAB9, RAB11, RAB24, RAB32 et RAB33) interviennent dans la formation et/ou la maturation des vacuoles autophagiques (Hirota et al. 2013). Par exemple, RAB7 et RAB9, situés sur la membrane des endosomes tardifs/lysosomes, seraient respectivement impliqués dans l’étape de fusion de l’autophagosome avec le lysosome (Gutierrez et al. 2004) et dans le transport des endosomes tardifs vers le réseau trans-golgien (TGN) (Barbero et al. 2002).
Les lysosomes contiennent les enzymes nécessaires (protéases, nucléases, lipases, ...) pour la dégradation du matériel séquestré, qu’il s’agisse de protéines, de lipides, de glucides ou encore d’acides nucléiques.
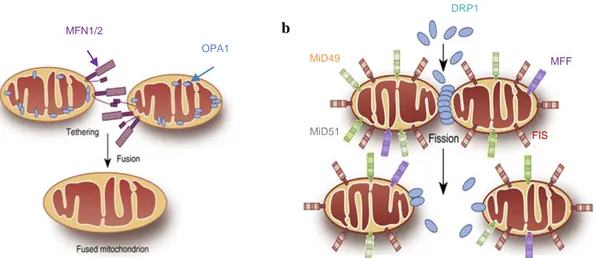
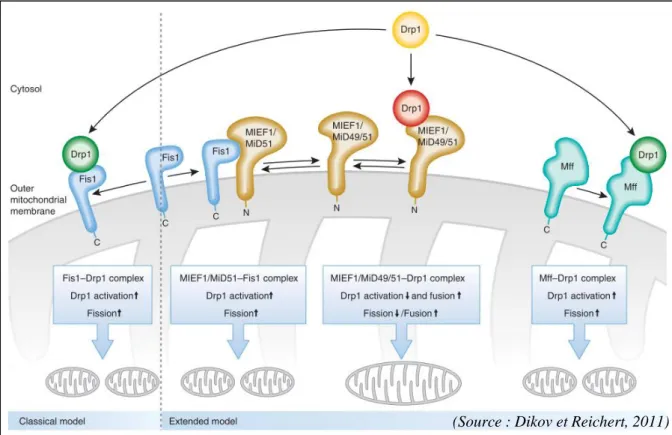
MFN1/2 OPA1 FIS 1 MFF MiD49 MiD51 DRP1 a b
Figure 4 : Schémas des phénomènes de fusion (a) et fission (b) mitochondriale chez les mammifères
(a) Les mitofusines MFN1/2 sont des protéines GTPases qui permettent la fusion des membranes externes des mitochondries. Elles induisent le rapprochement de 2 mitochondries via leur domaine HR2 (Heptad Repeat 2). La fusion des 2 membranes a ensuite lieu suite à l’hydrolyse de GTP via MFN1/2. OPA1 est également une protéine GTPase et permet la fusion des membranes internes des mitochondries. OPA1 permet de conserver la structure de crêtes.
(b) La fission mitochondriale fait intervenir de nombreux acteurs présents à la surface externe des mitochondries (FIS1, DRP1, MFF et MiD49/51). DRP1 peut être recrutée par différents facteurs (Figure 5). Lorsqu’elle est recrutée, les protéines DRP1 s’assemblent pour former des structures oligomériques en forme d’anneau. Ces structures s’enroulent autour des mitochondries et induisent la fission mitochondriale.
MFN1/2 : Mitofusins1/2 ; OPA1 : Optic Atrophy 1 ; DRP1 : Dynamin Related Protein 1 ; FIS1 : mitochondrial Fission Protein 1 ; MFF : Mitochondrial Fission Factor ; MiD49/51 : Mitochondrial Dynamic Protein of 49 or 51 kDa
5
2.2 Implication de ce système pendant l’immobilisation et la récupération
Concernant la régulation de l’autophagie pendant l’inactivité musculaire, le modèle de suspension a été le plus étudié. Après 2 jours de suspension, l’expression des ARNm de LC3, ATG7, et GABARAPL1 (GABA(A) receptor-associated protein like 1) est induite dans le muscle biceps femoris (Smith et al. 2014). Une augmentation du ratio LC3-II/LC3-I a été rapportée dans le soleus après 14 jours de suspension (Maki et al. 2012). Inversement, après 4 semaines de suspension, les taux protéiques de BECLIN1 et ATG5 diminuent de 40%, alors que ceux de de p62 augmentent de près de 40%, sans variation pour ATG7 et le ratio LC3-II/LC3-I (Liu et al. 2012). Dans un modèle de dénervation, les taux protéiques d’ULK1, ATG7 et LC3-II (x6, x2 et x2 respectivement) ont été induits dans le TA après 7 jours de dénervation (O’Leary et al. 2013). Enfin, dans un modèle de plâtrage chez l’Homme, les taux d’ARNm de GABARAPL1 augmentent uniquement après 14 jours d’immobilisation, alors que le ratio
LC3-II/LC3-I augmente du 1er au 4ème jour d’immobilisation (Suetta et al. 2012).
La phase de récupération a été beaucoup moins étudiée. L’expression des ARNm de LC3, ATG7, et GABARAPL1 induite dans le muscle biceps femoris lors de la suspension est normalisée après 7 jours de désuspension (Smith et al. 2014). On note également que dans deux études, les taux protéiques ou ARNm de BECLIN1 sont fortement induits après 5 jours de désuspension dans le soleus (+230%, Andrianjafiniony et al. 2010) ou 6 jours après le déplâtrage dans le TA (+260%, Slimani et al. 2012).
L’induction de marqueurs de l’autophagie lorsque l’atrophie est stabilisée après la désuspension ou le déplâtrage (Slimani et al. 2012), suggère que ce processus pourrait être impliqué non pas dans la dégradation de protéines musculaires, mais dans l’élimination d’organelles déficientes qui ont un effet délétère sur la masse musculaire (Masiero et al. 2009).
3. La mitochondrie 3.1 Généralités
L’origine des mitochondries repose sur la théorie endosymbiotique, et l’ancêtre des
mitochondries serait une -protéobactérie (Margulis. 1975). Les mitochondries sont des
organites cytoplasmiques à double membrane présentes uniquement chez les eucaryotes et dans tous les types cellulaires hormis les globules rouges. En moyenne on trouve entre 1000 et 3000 mitochondries dans les différents types cellulaires.
Les mitochondries ont pour principal rôle la production d’ATP, source d’énergie indispensable à de nombreux processus biologiques. Le muscle squelettique contient 2 types de populations de mitochondries : subsarcolemmales (SS : sous le sarcolemme) et inter-myofibrillaires (IMF :
Figure 5 : Illustration des différents modèles de fission mitochondriale chez les mammifères Dans le modèle classique (gauche) le recrutement de DRP1 à la surface mitochondriale par FIS1 active DRP1, qui, via une hydrolyse de GTP induit la fission mitochondriale. Dans le modèle « étendu » (droite), DRP1 est recruté par MFF induisant aussi l’activation de DRP1 et la fission. D’autres acteurs, MiD49 et MiD51, jouent un rôle dans la fission mitochondriale. MiD49 et MiD51 recruteraient DRP1 induisant sa séquestration et son inactivation, et donc réprimant la fission mitochondriale. Cependant, lorsque FIS1 est abondant, il va se lier à MiD51 (non démontré pour MiD49 à ce jour) pour empêcher la liaison MiD51-DRP1, libérant DRP1 et induisant la fission mitochondriale.
DRP1 : Dynamin Related Protein 1 ; FIS1 : mitochondrial Fission Protein 1 ; MFF : Mitochondrial Fission Factor ; MiD49/51 : Mitochondrial Dynamic Protein of 49 or 51 kDa
(Source : Dikov et Reichert, 2011)
6
le long des myofibrilles). Les mitochondries IMF représentent 80% du contenu mitochondrial et participent à la production d’énergie lors de la contraction musculaire, alors que les mitochondries SS ont un rôle dans la production d’énergie pour tous les évènements se produisant à la membrane mitochondriale (Powers et al. 2011, Romanello et Sandri. 2013).
La dynamique mitochondriale (fusion et fission), ainsi que le turnover des mitochondries (biogenèse et dégradation) sont des processus interconnectés essentiels au « contrôle qualité » des mitochondries.
3.2 Remodelage mitochondrial
La dynamique mitochondriale est fonction des phénomènes de fusion et fission mais les mécanismes qui régulent cette dynamique ne sont pas encore tous élucidés. Cependant, des marqueurs spécifiques de ces mécanismes ont été identifiés : DRP1 (Dynamin Related Protein 1), FIS1 (mitochondrial Fission Protein 1), OPA1 (Optic Atrophy 1), et MFN1/2 (Mitofusin 1/2).
La fusion des mitochondries permet d’assurer l’intégrité du réseau mitochondrial en rétablissant un potentiel de membrane adéquat via la réintroduction de mitochondries saines dans le pool de mitochondries. Dans ce processus, MFN1 et 2 permettent la fusion des membranes externes, alors qu’OPA1 permet la fusion des membranes internes. Ces 2 évènements sont indépendants mais hautement coordonnés (Youle et al. 2012) (Figure 4a).
La fission permet de séparer une mitochondrie « saine » d’une mitochondrie ayant un potentiel de membrane trop bas, l’orientant alors vers l’élimination par mitophagie (Youle et al. 2012). DRP1, protéine de la famille des dynamines, porte une activité GTPase, et est l’acteur majeur de la fission mitochondriale (Figure 4b et 5). Cependant, DRP1 peut être modifiée post-traductionnellement de diverses façons (phosphorylation, sumoylation, ubiquitination, S-nitrosylation) influençant son recrutement et/ou son activité. Par exemple, la phosphorylation de DRP1 par la kinase CaMKIα (Calcium/Calmodulin-dependent protein kinase α) induit la fission mitochondriale (Cribbs et al. 2007), alors que la phosphorylation par la PKA (Protein kinase A) inhibe la fission mitochondriale (Chang et al. 2007). Le recrutement de DRP1 au niveau de la membrane mitochondriale dépend de différents acteurs (FIS1, MFF, MiD49/51) dont l’équilibre conditionne l’induction ou la répression de la fission (Figure 5).
Concernant FIS1, il a été démontré récemment qu’elle aurait également un rôle dans la phase finale de l’autophagie, en inhibant l’action de RAB7, et par conséquent en inhibant la fusion entre le lysosome et l’autophagosome (Yamano et al. 2014) (Figure 3).
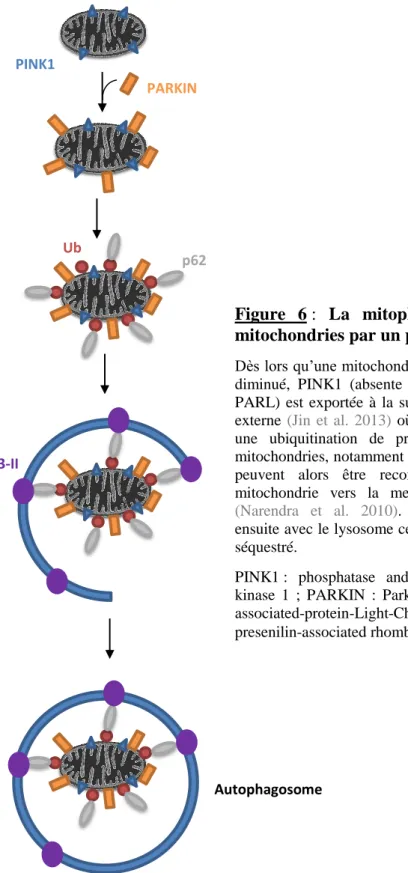
Figure 6 : La mitophagie : voie d’élimination des mitochondries par un processus autophagique
Dès lors qu’une mitochondrie présente un potentiel de membrane diminué, PINK1 (absente en condition basale car éliminée par PARL) est exportée à la surface de la membrane mitochondriale externe (Jin et al. 2013) où elle va recruter PARKIN. Il s’ensuit une ubiquitination de protéines présentes à la surface des mitochondries, notamment MFN2. Ces protéines polyubiquitinées peuvent alors être reconnues par p62, ce qui dirige la mitochondrie vers la membrane du phagophore via LC3-II
(Narendra et al. 2010). L’autophagosome formé fusionnera ensuite avec le lysosome ce qui permettra de dégrader le matériel séquestré.
PINK1 : phosphatase and tensin homologue-induced putative kinase 1 ; PARKIN : Parkinson protein 2 ; LC3 : microtubule-associated-protein-Light-Chain3 ; Ub : Ubiquitine ; PARL : presenilin-associated rhomboid-like LC3-II p62 Ub PINK1 PARKIN Autophagosome
7
La fission mitochondriale ainsi que la mitophagie semble être interconnectées. En effet, chez des mutants DRP1, la mitophagie est réprimée, suggérant que la fission permettrait l’adressage de mitochondries déficientes vers leur dégradation via la mitophagie (Twig et al. 2008).
3.3 Le « turnover » des mitochondries
Contrairement à d’autres organites, la biogenèse des mitochondries se fait à partir de mitochondries déjà existantes via leur élongation puis leur scission et dépend de PGC1α (Peroxysome proliferator-activated receptor gamma coactivator-1α).
La mitophagie est un processus dédié aux mitochondries ayant des défauts fonctionnels et a pour conséquence leur élimination ; de manière générale, la perte de potentiel de membrane de la mitochondrie entraîne l’exportation de PINK1 (phosphatase and tensin homologue-induced putative kinase 1) vers la membrane externe de la mitochondrie (Jin et al. 2013), permettant le recrutement de PARKIN (Parkinson protein 2, enzyme E3 ligase). Ceci est à l’origine d’une cascade d’évènements permettant l’adressage de la mitochondrie déficiente vers l’autophagosome (Narendra et al. 2010) (Figure 6)
3.4 Altérations mitochondriales au cours de l’immobilisation et de la récupération L’atrophie musculaire se traduit par une perte de cellules mais également par des altérations mitochondriales.
Durant les phénomènes d’atrophie musculaire, les mitochondries SS sont prioritairement touchées probablement du fait de leur localisation : par exemple, on observe une diminution de 64% des mitochondries SS dans le TA lors de la dénervation (Iqbal et al. 2013). Ceci est associé à une baisse du contenu mitochondrial (Iqbal et al. 2013, O’Leary et al. 2013), des modifications des fonctions mitochondriales et une augmentation de la production d’espèces réactives à l’oxygène (Powers et al. 2012, Qi et al. 2012). Dans la plupart des études, l’expression des ARNm ou protéique de PGC1α est fortement diminuée (Sandri et al. 2006, Wagatsuma et al. 2011, Liu et al. 2012, Suetta et al. 2012, Qi et al. 2012). De plus, le maintien de PGC1α permet de prévenir l’atrophie musculaire lors du jeûne ou de la dénervation (Sandri et al. 2006, Romanello et al. 2010).
L’activité de la citrate synthase ou le nombre de copies d’ADN mitochondrial diminuent dans les muscles GA ou soleus après dénervation ou suspension (Wagatsuma et al. 2011, Liu et al. 2014, Qi et al. 2012), suggérant une baisse de la biogenèse mitochondriale et donc de la densité mitochondriale.
8
Concernant la dynamique mitochondriale, les taux protéiques de MFN2, OPA1 et FIS1 sont fortement diminués (-70%, -84% et -46% respectivement) dans le TA après dénervation (Iqbal et al. 2013). Cependant, dans cette étude, aucune différence n’a été observée pour DRP1. A l’inverse, O’Leary et coll. (2013) n’observent aucun changement des niveaux protéiques de FIS1, OPA1 et MFN2 dans les fractions protéiques totales dans le TA après dénervation, mais ont rapporté une forte induction (x6) des niveaux protéiques de PARKIN dans les fractions de protéines mitochondriales. L’étude des altérations de la fusion/fission mitochondriale et de la mitophagie ont été réalisées principalement en utilisant le modèle de dénervation, et ne concernent pas les phases de récupération.
4. Objectifs de l’étude et stratégie expérimentale
A la vue de l’ensemble de ces données bibliographiques, il apparait que l’autophagie semble jouer un rôle important dans les variations de masse musculaire au cours de l’inactivité musculaire. Elle peut être activée pendant l’immobilisation et/ou plus tardivement pendant la phase de récupération. De plus, le réseau mitochondrial semble subir des modifications pendant l’atrophie induite par l’inactivité musculaire et la récupération qui s’ensuit. Des altérations mitochondriales pourraient conduire à l’accumulation d’organelles déficientes si elles ne sont pas éliminées correctement et concourir à l’atrophie ou à un retard de récupération. Tout cela pourrait alors jouer un rôle dès le déplâtrage dans le TA, notamment via l’élimination d’organelles déficientes telles que les mitochondries.
Les objectifs de mon stage de M2 ont donc été de caractériser le rôle de l’autophagie et des altérations mitochondriales pendant l’immobilisation et la remobilisation dans le TA.
Pour cela, nous avons étudié différentes étapes de l’initiation à la formation des autophagosomes, les processus de fusion et fission mitochondriales, ainsi que le turnover des mitochondries (biogenèse et mitophagie) dans un modèle d’immobilisation par plâtrage chez le rat.
Adaptation
Déplâtrage
Plâtrage
I8
R1
-21
I0
0
8
9
14
Immobilisation
Récupération
R6
18
R10
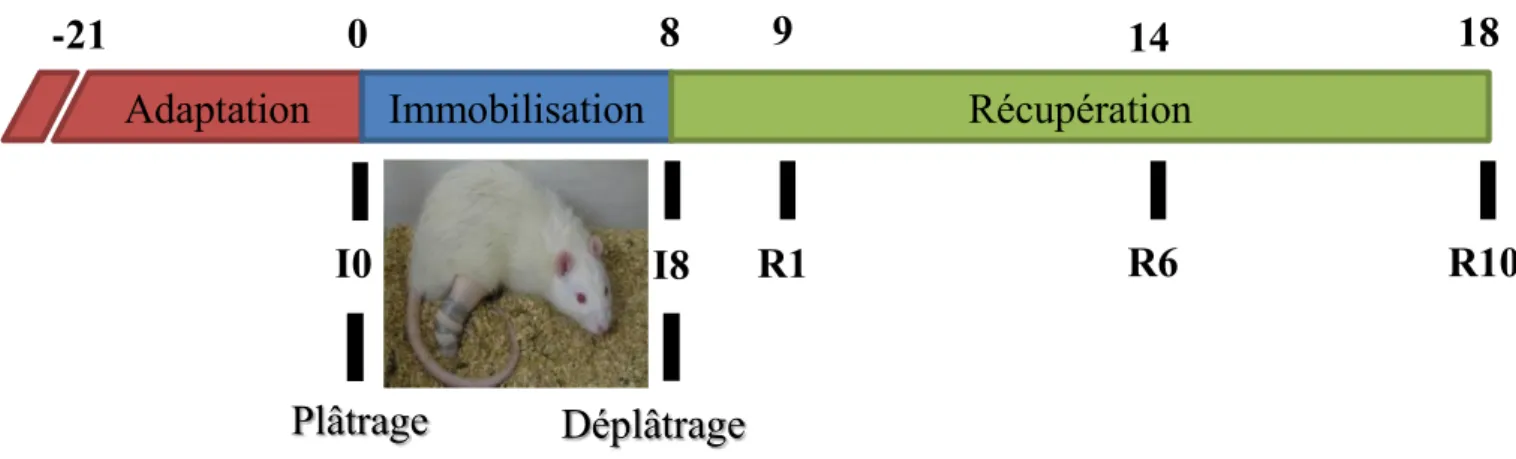
Figure 7 : Schéma du protocole expérimental
Après 21 jours d’adaptation, les rats ont été soit sacrifiés (I0, n=10), soit immobilisés. Après 8 jours d’immobilisation, les rats ont été déplâtrés. Certains ont été sacrifiés (I8, n=10), et les autres ont été placés en récupération et sacrifiés après 1 (R1, n=10), 6 (R6, n=10) ou 10 jours (R10, n=11).
9
MATERIELS & METHODES
1. Modèle d’étude
La présente étude a été réalisée à partir d’échantillons de muscle tibialis anterior (TA) issus d’un protocole animal (décrit ci-dessous) déjà réalisé.
Les expériences ont été conduites en accord avec la législation en vigueur en France sur l’expérimentation animale. Cinquante rats mâles adultes (6 mois) de souche Wistar (Charles River Laboratories, l’Arbresle, France) ont été mis en adaptation pendant 21 jours. Les animaux ont été hébergés dans les locaux de l’Installation Expérimentale de Nutrition de l’INRA de Clermont-Ferrand/Theix en cages individuelles sous conditions expérimentales contrôlées (température 22°C et cycle de lumière normal 8h – 20h). Ils ont reçu un régime standard de croquettes contenant 13% de protéines (Unité de Préparation des Aliments Expérimentaux, INRA, Domaine de Vilvert –Jouy en Josas) avec un libre accès à l’eau de boisson.
Dix rats ne subissent pas d’immobilisation et constituent le groupe témoin avant plâtrage (I0). Les autres animaux ont été soumis à une immobilisation unilatérale de la patte arrière droite par la pose d’un plâtre thermo-moulant micro-perforé (ORFIT, Soft micro-perforé, Gibaud) maintenu par un fil de fer protecteur (patte immobilisée : Imm). Après 8 jours d’immobilisation (I8, n=10), les animaux ont été déplâtrés et placés en remobilisation pendant 1 (R1, n=10), 6 (R6, n=10) et 10 (R10, n=11) jours (Figure 7).
2. Techniques expérimentales
2.1 Mesure des niveaux d’ARNm par RT-qPCR
2.1.1 Dosage, traitement des ARNs et rétrotranscription
Les ARN totaux des muscles TA précédemment extraits par le TRIZOL (Invitrogen), ont été quantifiés par spectrophotométrie à 260 nm grâce au NanoDrop 1000 (Thermo Scientific). Leur qualité a été évaluée avec un BioAnalyser 2100 (Agilent). Seuls les échantillons présentant un RIN (RNA Integrity Number) supérieur ou égal à 8, témoignant d’une bonne qualité, ont été utilisés et rétrotranscrits.
Un µg d’ARN a été traité à la DNAse I (Invitrogen) pendant 15 minutes à température ambiante dans le but d’éliminer l’ADN génomique résiduel. L’enzyme a ensuite été inactivée grâce à l’ajout d’EDTA (2,5 mM) et incubée à 65°C pendant 10 minutes. Ensuite, 0,5 µg
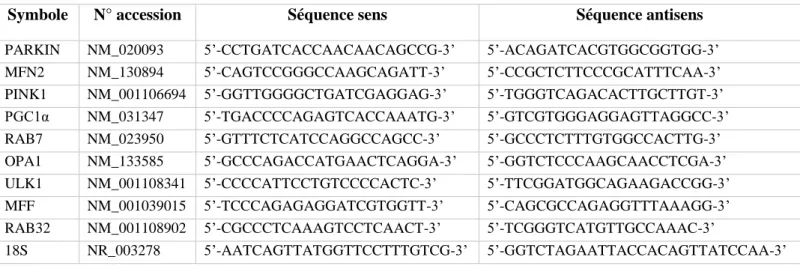
Symbole N° accession Séquence sens Séquence antisens
PARKIN NM_020093 5’-CCTGATCACCAACAACAGCCG-3’ 5’-ACAGATCACGTGGCGGTGG-3’
MFN2 NM_130894 5’-CAGTCCGGGCCAAGCAGATT-3’ 5’-CCGCTCTTCCCGCATTTCAA-3’
PINK1 NM_001106694 5’-GGTTGGGGCTGATCGAGGAG-3’ 5’-TGGGTCAGACACTTGCTTGT-3’
PGC1α NM_031347 5’-TGACCCCAGAGTCACCAAATG-3’ 5’-GTCGTGGGAGGAGTTAGGCC-3’
RAB7 NM_023950 5’-GTTTCTCATCCAGGCCAGCC-3’ 5’-GCCCTCTTTGTGGCCACTTG-3’
OPA1 NM_133585 5’-GCCCAGACCATGAACTCAGGA-3’ 5’-GGTCTCCCAAGCAACCTCGA-3’
ULK1 NM_001108341 5’-CCCCATTCCTGTCCCCACTC-3’ 5’-TTCGGATGGCAGAAGACCGG-3’
MFF NM_001039015 5’-TCCCAGAGAGGATCGTGGTT-3’ 5’-CAGCGCCAGAGGTTTAAAGG-3’
RAB32 NM_001108902 5’-CGCCCTCAAAGTCCTCAACT-3’ 5’-TCGGGTCATGTTGCCAAAC-3’
18S NR_003278 5’-AATCAGTTATGGTTCCTTTGTCG-3’ 5’-GGTCTAGAATTACCACAGTTATCCAA-3’
10
d’ARN traité à la DNase I ont été rétrotranscrits grâce au kit SuperScript II RT (Invitrogen) en présence d’amorces aléatoires (125 ng), de nucléotides (0,5 mM dNTP), et de DTT (10 mM
DTT) pendant 1 heure à 42°C. Les ADNc obtenus ont été dilués au 1/50ème et conservés à
-20°C jusqu’à utilisation.
2.1.2 Dessin des amorces et qPCR
Les amorces présentées dans le Tableau 1 ont été dessinées avec logiciel Primer3
(http://biotools.umassmed.edu/bioapps/primer3_www.cgi) à partir des séquences
nucléotidiques obtenues dans la banque de données Genomatix (http://www.genomatix.de). Le logiciel NCBI BLAST (http://blast.st-va.ncbi.nlm.nih.gov/Blast.cgi) a permis de vérifier la spécificité des séquences choisies.
Une première étape a consisté à mesurer l’efficacité des amplifications pour chaque couple d’amorces sélectionnées. Pour cela, une PCR en temps réel a été réalisée sur une gamme d’ADNc de dilution progressive d’un facteur 10 pour obtenir une courbe étalon correspondant au couple d'amorces utilisé (moyenne des efficacités obtenues = 97,3 %). Une courbe de fusion en fin de cycles a permis de contrôler la présence d’un seul produit d’amplification (amplicon). Les couples d’amorces pour l’amplification des transcrits codant pour MFN1 et ATG16L présentaient des valeurs de CT (Cycle Threshold) trop élevées (35) et n’ont pas été utilisées.
Les réactions de PCR ont été réalisées avec un thermocycleur CFX96TM (Biorad) selon le programme suivant : 3 minutes d’activation de la Taq Polymérase à 95 °C suivie des phases de dénaturation (40 cycles de 10 secondes à 95 °C), d’hybridation et d’élongation (20 secondes à 60 °C). 2 µL de chaque dilution d’ADNc ont été additionnés à 0,7 µL de chaque amorce (10 µM), 7 µL du mélange réactionnel contenant l’enzyme (PerfeCTA SYBR Green SuperMix,
Quanta Biosciences) et 3,6 µL d’H2O.
A chaque cycle d’amplification, la quantité d’ADN est mesurée grâce à un marqueur fluorescent (SYBR Green) dont l’émission est directement proportionnelle à la quantité d’amplicons produits. Le nombre de cycles à partir duquel le produit PCR est détectable est dénommé cycle seuil ou CT (Cycle Threshold). L'ARNr 18S exprimé de façon ubiquitaire a été utilisé comme référence. La méthode comparative des seuils de cycle (2ΔΔCT), a été utilisée pour comparer l'expression relative des ARNm entre chaque groupe. L'abondance relative de l'ARNm a été arbitrairement fixée à 1 pour le groupe I0.
2.2 Analyse des niveaux protéiques par Western Blot 2.2.1 Préparation des échantillons
11
Les muscles ont été réduits en poudre puis mis en présence de 2 mL de tampon d’extraction (Hepes 20mM, β-glycérophosphate 50mM, KCl 100mM, NaF 50mM, EGTA 2mM et EDTA 0,2mM, DTT 1mM, benzamidine 1mM, sodium vanadate 0,5mM, Triton X100 1%, PMSF 200mM, et un cocktail d’inhibiteurs de protéases (Sigma P8340) (pH=7,4). Les muscles ont ensuite été broyés au polytron, puis centrifugés à 14500 g pendant 12 minutes à 4°C. Un aliquot de chaque surnageant a été conservé à -80°C pour déterminer la concentration en protéines (Lowry et al. 1951).
Parmi les cibles étudiées, LC3 (sous sa forme lipidée, LC3-II) est très sensible à des agents tels que le SDS. Pour cette raison, les extraits protéiques ont été ajustés à 2,8µg/µL dans du tampon Laemli (1X final) exempt de SDS. Les extraits ont été conservés à -80°C jusqu’à utilisation. Le SDS (2% final) a été ajouté extemporanément aux échantillons avant leur dénaturation pendant 5 min à 95°C, et leur dépôt sur gel d’acrylamide.
2.2.2 Mise au point des conditions d’hybridation
Nous avons testé différentes dilutions des anticorps d’intérêt avec différentes concentrations de protéines séparées sur des gels d’acrylamide à 15%. Ceci nous a permis d’obtenir rapidement les conditions optimales pour 4 anticorps (MFN2, DRP1, anti-RAB7 et anti-FIS1) qui permettent de détecter de manière très sensible une seule bande (Cf Partie Résultats & Discussion, Figures 10A, 12A, 13A et 13C).
En ce qui concerne l’anticorps anti-PARKIN, nous avons testé 2 anticorps provenant de 2 fournisseurs différents (SIGMA et ABCAM). Seul celui provenant de chez SIGMA nous a permis de détecter une seule bande de manière très sensible (Cf Partie Résultats & Discussion,
Figure 14C).
Deux autres anticorps ont été testés (anti-MFN1 et anti-PINK1). Cependant les tests d’optimisation ne nous ont pas permis de détecter des signaux interprétables quelles que soient les conditions utilisées.
Enfin pour l’anticorps anti-LC3, les conditions avaient déjà été définies précédemment au laboratoire avec l’anticorps de chez SIGMA. Cet anticorps permet de détecter les 2 bandes correspondant à LC3-I et LC3-II. Cet anticorps présente une hybridation non spécifique assez importante au-dessus de 26 kDa. C’est la raison pour laquelle, la détection de LC3 a été réalisée sur des membranes dont la partie haute (> 26kDa) a été excisée. Ces parties hautes ont été utilisées pour les cibles MFN2, DRP1, PARKIN, dont le signal était par contre très spécifique.
Stratégie n°1
Stratégie n°2
Figure 8 : Stratégies utilisées pour la mise en place des Western Blots Ladder = échelle ; I0 = rat non plâtrés ; I8 = rats immobilisés 8 jours ; R1-R10 = rats remobilisés pendant 1 à 10 jours
Ctrl = patte controlatérale ; Imm = patte immobilisée Les illustrations ci-dessus sont des exemples :
1. pour la stratégie n°1, le même type de gel a été réalisé pour les échantillons des temps R1, R6 et R10.
2. pour la stratégie n°2, les 5 derniers puits ont été utilisés avec des échantillons I0, I8, R1, R6 ou R10 selon les besoins pour que tous les échantillons puissent être déposés sur les 4 gels.
12 2.2.3 Western blot
a) Stratégie d’analyse des échantillons
Pour chacun des rats, les muscles TA de la patte immobilisée (Imm) et de la patte controlatérale (Ctrl) ont été analysés, ce qui a conduit à l’analyse d’environ 100 échantillons.
Une première stratégie a consisté à grouper les dépôts des muscles issus des pattes Ctrl et Imm pour un même temps d’analyse sur des mini-gels (10 puits) (Figure 8). Pour pouvoir analyser l’effet « temps », un échantillon référent (pool d’échantillon I0) avait été inclus sur chacun des gels. Il s’est avéré que cette méthode d’analyse générait de très grosses variations d’un temps à l’autre qui pouvaient être attribuées à un effet cinétique réel, mais qui auraient également pu résulter d’un artéfact lié au fait que les échantillons analysés n’étaient pas déposés sur les mêmes gels.
Nous avons ensuite vérifié ceci en réalisant une nouvelle répartition des échantillons, et en choisissant de déposer 2 à 3 échantillons issus de la patte Imm et Ctrl des différents temps (I0 et I8, R1, R6, R10) sur chacun des gels de 25 puits (Figure 8). Quatre gels ont donc été réalisés pour pouvoir déposer l’ensemble des échantillons. Après analyse, les différences initialement détectées entre les temps étudiés ne semblaient pas refléter de réelles différences entre les échantillons, mais des effets « membranes ». Nous avons donc adopté cette seconde stratégie pour analyser l’ensemble des cibles décrites dans ce rapport.
b) Séparation et transfert
En raison 1) du nombre d’échantillons à tester, 2) de la nécessité de couper les gels au-dessus de 26kDa pour l’analyse de LC3, 3) de la très bonne spécificité des anticorps dirigés contre les cibles dont le poids moléculaire est supérieur à 26 kDa, et 4) de la nécessité d’adapter les conditions de transfert en fonction du poids moléculaire des cibles testées, nous avons traités les échantillons de la manière suivante :
- 30 µg de protéines ont été déposées et séparées par SDS-page sur des gels à 15 % d’acrylamide.
- La migration des protéines a été réalisée dans un tampon « running buffer 1X » (Annexe 1) à voltage constant (100 V).
- Les gels ont ensuite été coupés au niveau de la bande de 26 kDa dans l’optique de réaliser le transfert optimal des protéines de haut (MFN2, DRP1, PARKIN) ou bas (FIS1, LC3 et RAB7) poids moléculaire:
le transfert de la partie haute du gel a été réalisé dans un tampon de transfert contenant du SDS (0,2 %) (Annexe 1), la nuit à 150 mA à 4 °C, sur des membranes d’hybridation PVDF «Amersham Hybond-P PVDF» (activée au méthanol),
Anticorps IR Taille Description Référence Dilution anticorps IR
Dilution anticorps IIR
MFN2 (Abcam) 80 kDa Rabbit
monoclonal Ab124773 1/2000 (BSA 5%) 1/4000 (BSA 5%) DRP1 (Abcam) 82 kDa Mouse monoclonal Ab56788 1/2000 (BSA 5%) 1/4000 (BSA 5%) FIS1 (Abcam) 17 kDa Rabbit polyclonal Ab71498 1/1000 (BSA 5%) 1/2000 (BSA 5%)
LC3 I (Sigma) 16 kDa Rabbit
polyclonal L7543 1/1000 (LAIT 5%) 1/2000 (LAIT 5%) LC3 II (Sigma) 14 kDa PARKIN (Sigma) 50 kDa Mouse monoclonal P6248 1/10000 (BSA 5%) 1/20000 (BSA 5%) RAB7
(Cell Signaling) 23 kDa
Rabbit monoclonal 9367 (D95F2) 1/1000 (BSA 5%) 1/2000 (BSA 5%) Tableau 2 : Liste des anticorps et conditions d’utilisation
13
le transfert de la partie basse du gel a été réalisé dans un tampon de transfert sans SDS (Annexe 1), pendant 1H30 à 80V à 4°C, sur des membranes d’hybridation PVDF
«Immobilon-PSQ membrane» (activée au méthanol).
Les 4 demi-gels correspondant à la séparation de l’ensemble des échantillons ont été transférés sur une seule et même membrane.
Après transfert, les membranes ont été colorées avec le BLOT FastStain ce qui nous a permis de nous assurer que le transfert était correct, et de normaliser ensuite les données obtenus après révélation.
c) Immunoblots
Après transfert, les membranes ont été rincées (3 x 10 minutes à température ambiante) dans du TBST 1X (Annexe 1) avant d’être bloquées dans une solution de lait écrémé à 5 % dans du TBST 1X pendant 1h à température ambiante sous agitation.
Les membranes sont ensuite rincées (2 x 10 minutes à température ambiante) dans du TBST 1X
avant d’être hybridées avec l’anticorps primaire (IR
) (dilué dans de la BSA ou dans du lait à 5%) la nuit à 4 °C ou quelques heures à température ambiante sous agitation. (Tableau 2) Les membranes ont ensuite été rincées (2 x 10 minutes à température ambiante) dans du TBST
1X puis hybridées avec l’anticorps secondaire (IIR) correspondant (dilué dans de la BSA ou
dans du lait à 5%) (Tableau 2) pendant 1h à température ambiante sous agitation.
Après 3 lavages de 10 minutes (à température ambiante) dans du TBST 1X sous agitation, les signaux ont été détectés après incubation avec le substrat HRP (horseradish peroxidase) Luminata Crescendo ou Forte, et visualisés avec le détecteur G:BOX ChemiXT4 (XL1) (Syngene, MD, USA).
Les signaux ont ensuite été quantifiés avec le logiciel GeneTools (Syngene, MD, USA). Les données ont été normalisées par la quantité de protéines déposées sur les gels, visualisées grâce à la coloration BLOT FastStain, et quantifiées par le logiciel ImageJ pour corriger d’éventuelles différences de dépôt.
3. Analyses statistiques
Les données présentées sont les moyennes ± erreur standard à la moyenne (SEM) pour n=6-11 rats par groupe. Elles ont ensuite été analysées par ANOVA à 2 facteurs pour étudier l’effet « immobilisation » et « temps ». Lorsque des différences significatives ont été détectées par ANOVA, les comparaisons 2 à 2 ont ensuite été réalisées par le test PLSD (Protected Least Significant Difference) de « Fisher ». Les données sont considérées comme statistiquement différentes pour P < 0.05.
14 kDa 16 kDa LC3-II LC3-I I0 I8 R1 R6 R10 0 20 40 60 80 100 120 AR Nm ULK 1/18S Uni té s ar b itr air es % I0 a ab bcd ab abc de e cde cde I8 R1 R6 R10 I0 IMMOBILISATION REMOBILISATION 0 50 100 150 200 250 Rapp or t LC3 -II/ L C3 -I Uni té s ar b itr air es % I0 d ce bce a a e a ab abc 0 100 200 300 400 500 600 P roté in e L C3 -I U ni té s ar bi tr air es % I0 a a a a a b bc d c 0 50 100 150 200 250 P roté in e L C3 -II Uni té s ar b itr air es % I0 d cd bcd abc a ab a a ab IMMOBILISATION I8 R1 R6 R10 I0 I0 I8 R1 R6 R10
REMOBILISATION IMMOBILISATION REMOBILISATION
IMMOBILISATION REMOBILISATION
I8 R1 R6 R10
I0
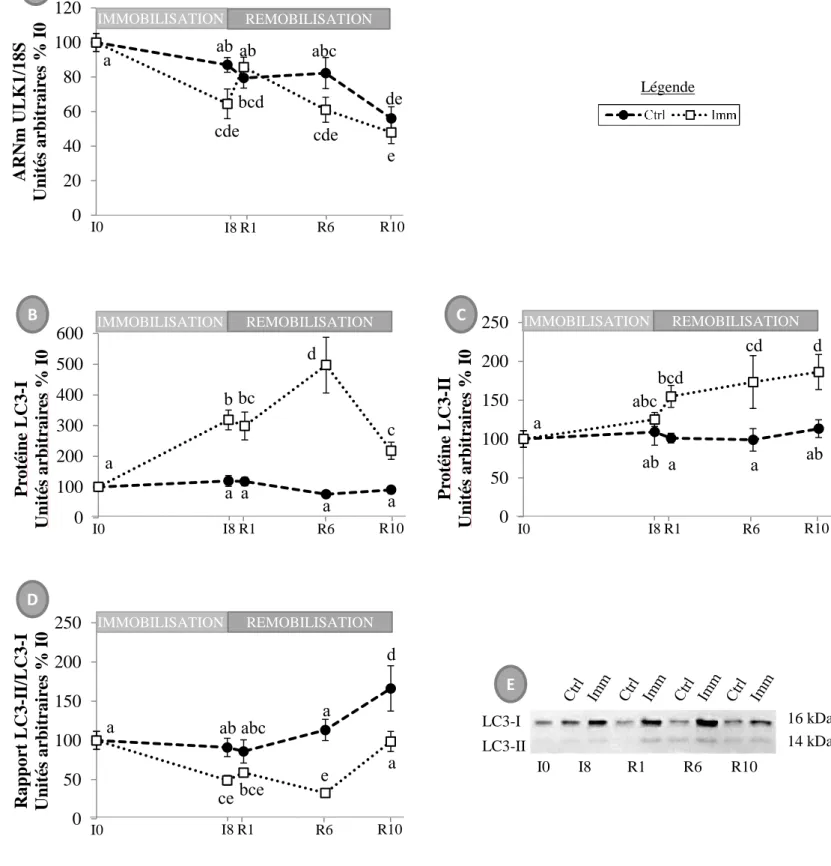
Figure 9 : Régulation des phases d’initiation et de formation des autophagosomes de l’autophagie dans le tibialis anterior immobilisé et remobilisé. (A) Les taux d’ARNm de ULK1 ont été déterminés par RT-qPCR. Les données ont été normalisées par rapport à l’ARN ribosomal 18S et exprimées en pourcentage de variation par rapport à I0. (B, C, D et E) Les niveaux d’expression protéiques de LC3-I et de LC3-II ont été déterminés par Western-Blots. Une image représentative est présentée (E). Après quantification, les signaux ont été normalisés par l’intensité de la coloration BLOT FastStain pour corriger d’éventuelles différences de dépôts.
Les données sont des moyennes ± SEM pour 6 à 11 rats/groupe. Les différences statistiques ont été testées par ANOVA, et les points affectés de lettres différentes sont significativement différents.
I0 = rats non plâtrés ; I8 = rats immobilisés 8 jours ; R1-R10 = rats remobilisés pendant 1 à 10 jours. Ctrl = patte controlatérale ; Imm = patte immobilisée
A
B C
D
E
14
RESULTATS & DISCUSSION
Dans le but de comprendre les mécanismes impliqués pendant la phase de remobilisation, nous avons étudié la régulation de l’autophagie (un des 2 systèmes protéolytiques prédominants dans le contrôle de la masse musculaire) puis l’intégrité mitochondriale ainsi que sa dynamique dans le muscle TA chez le rat après 8 jours d’immobilisation par plâtrage et 1 à 10 jours de remobilisation.
Pour de nombreux marqueurs étudiés, des effets dans le TA de la patte controlatérale (Ctrl) ont été observés, comme rapporté auparavant (Magne et al. 2013, Slimani et al. 2012 et données non publiées). Cela pourrait résulter 1) de la diminution de prise alimentaire qui caractérise les animaux pendant l’immobilisation mais qui se normalise rapidement après le déplâtrage (Slimani et al. 2012), 2) de dysfonctionnements qui se produisent dans la patte immobilisée (inflammation, stress oxydant, …) et se répercutent au niveau du corps entier et donc dans la patte Ctrl, et/ou 3), par une utilisation plus importante de la patte qui n’a pas été plâtrée. C’est pourquoi, nous avons choisi de présenter les données en comparant les effets dans la patte immobilisée ou controlatérale par rapport aux données obtenues dans le TA des animaux non plâtrés (I0).
1. Etude de l’autophagie au cours des phases d’immobilisation et de remobilisation dans le muscle tibialis anterior chez le rat :
L’autophagie est un processus biologique complexe à mettre en évidence car ce n’est pas un phénomène « statique » mais dynamique. Pour avoir une vue globale de l’induction ou non de l’autophagie, nous avons choisi d’étudier la phase d’initiation et de formation des autophagosomes, ainsi que la phase de fusion de l’autophagosome avec le lysosome.
1.1. Etude de la phase de formation des autophagosomes
ULK1 jouant un rôle très important dans l’étape de formation de l’autophagosome, nous avons mesuré son expression au niveau ARNm (Figure 9A). Les taux d’ARNm de ULK1 ont tout d’abord diminué dans le TA immobilisé à I8 (-32%, P < 0,05 vs. I0), puis ont augmenté dans le TA remobilisé entre I8 et R1 (ns, vs. I0), avant de diminuer à nouveau à R6 et R10 (-34 et -50% respectivement, P < 0,05) par rapport à I0. Les taux d’ARNm de ULK1 ont également diminué dans la patte controlatérale particulièrement à R10 (-44%, P < 0,05 vs. I0). La lipidation de LC3 sur les membranes des autophagosomes figure comme le principal marqueur
0 50 100 150 200 P roté in e RA B 7 Uni té s ar b itr air es % I0 0 20 40 60 80 100 120 AR Nm RA B 7/18S Uni té s ar b itr air es % I0 a ab ab ab c c c bc c ab b b ab ab ab b a c IMMOBILISATION REMOBILISATION I8 R1 R6 R10 I0 B 23 kDa RAB7 I0 I8 R1 R6 R10 I8 R1 R6 R10 I0 IMMOBILISATION REMOBILISATION A Légende :
Figure 10 : Régulation de la phase terminale de l’autophagie dans le tibialis anterior immobilisé et remobilisé. (A) Les niveaux d’expression protéique de RAB7 ont été déterminés par Western-Blot. Après quantification, les signaux ont été normalisés par l’intensité de la coloration BLOT FastStain pour corriger d’éventuelles différences de dépôts. Une image représentative a été inclue en-dessous du graphique. (B) Les taux d’ARNm de RAB7 ont été déterminés par RT-qPCR. Les données ont été normalisées par rapport à l’ARN ribosomal 18S et exprimées en pourcentage de variation par rapport à I0.
Les données sont des moyennes ± SEM pour 6 à 11 rats/groupe. Les différences statistiques ont été testées par ANOVA, et les points affectés de lettres différentes sont significativement différents.
I0 = rats non plâtrés ; I8 = rats immobilisés 8 jours ; R1-R10 = rats remobilisés pendant 1 à 10 jours. Ctrl = patte controlatérale ; Imm = patte immobilisée
15
de l’autophagie (Klionsky et al. 2012). Nous avons donc étudié les taux protéiques de LC3 sous sa forme cytosolique (LC3-I) et sous sa forme lipidée (LC3-II). Les Figures 9B et 9E montrent que les niveaux protéiques de LC3-I ont augmenté dans le TA dès la phase d’immobilisation (+220% à I8, P < 0,05) et ont atteint un maximum à R6 (+410%, P < 0,05) par rapport à I0. Ils ont ensuite diminué entre R6 et R10 (+210%, P < 0,05 vs. I0). En parallèle, les taux protéiques de LC3-II ont augmenté uniquement dans le TA remobilisé à partir de R1 et ce jusqu’à R10 (+56% et +81% respectivement, P < 0,05) par rapport à I0 (Figure 9C et 9E). La Figure 9D montre que le ratio LC3-II/LC3-I a diminué dans le TA immobilisé à I8 (-50%, P < 0,05), et dans le TA remobilisé à R1 et R6 (-38 et -65% respectivement, P < 0,05) par rapport à I0. Ce ratio a ensuite augmenté entre R6 et R10 pour atteindre des valeurs comparables à I0. Il est à noter que ce ratio a également augmenté dans le TA de la patte controlatérale à R10 (+65%, P < 0,05 vs. I0).
Ces données suggèrent que la phase d’initiation des autophagosomes est réprimée, comme en atteste la diminution des ARNm de ULK1 ainsi que la baisse du ratio LC3-II/LC3-I. Cependant, les données obtenues pour ULK1 ne sont pas corrélées pas avec les résultats observés précédemment pour BECLIN1 : en effet les niveaux d’ARNm de BECLIN1 étaient induits uniquement après 6 jours de remobilisation dans le TA (Slimani et al. 2012).
Cela peut s’expliquer par le fait que l’autophagie, comme de nombreux processus biologiques, est régulée aussi par des modifications post-traductionnelles. En effet, ULK1 et BECLIN 1 sont activées par phosphorylation (Figure 3) (Boya et al. 2013). De ce fait, l’étude des phosphorylations d’ULK1 et de BECLIN1 nous donnerait une image plus précise de la régulation de l’initiation de l’autophagie dans le TA immobilisé et remobilisé.
1.2. Etude de la phase de fusion des autophagosomes avec les lysosomes Pour étudier cette phase, nous avons mesuré les taux de protéine et d’ARNm de RAB7 dans le TA immobilisé et remobilisé. Les niveaux protéiques de RAB7 n’ont pas changé dans le TA immobilisée à I8 ou dans le TA remobilisé jusqu’à R6. Après R6, ces taux protéiques ont augmenté jusqu’à R10 dans la patte immobilisée (+63%, P<0,05 vs I0) (Figure 10A). Par contre, les niveaux d’ARNm de RAB7 ont diminué dans le TA immobilisé à I8 (-31%, P < 0,05), et sont restés bas dans le TA remobilisé jusqu’à R10 (-41%, P < 0,05) par rapport à I0 (Figure 10B). Enfin, les niveaux d’ARNm de RAB7 ont diminué dans le TA de la patte controlatérale à R10 (-41%, P < 0,05 vs. I0).
Les niveaux d’ARNm et de protéines concernant RAB7 ont évolué différemment. Cependant, les niveaux d’ARNm reflètent à un moment précis le bilan entre ce qui est transcrit et traduit.
0 20 40 60 80 100 120 140 AR Nm P G C1 α /18S Uni té s ar b itr air es % I0 c a a b a a bc bc a I8 R1 R6 R10 I0 IMMOBILISATION REMOBILISATION
Figure 11 : Régulation de la biogenèse mitochondriale dans le tibialis anterior immobilisé et remobilisé. Les taux d’ARNm de PGC1α ont été déterminés par RT-qPCR. Les données ont été normalisées par rapport à l’ARN ribosomal 18S et exprimées en pourcentage de variation par rapport à I0.
Les données sont des moyennes ± SEM pour 6 à 11 rats/groupe. Les différences statistiques ont été testées par ANOVA, et les points affectés de lettres différentes sont significativement différents.
I0 = rats non plâtrés ; I8 = rats immobilisés 8 jours ; R1-R10 = rats remobilisés pendant 1 à 10 jours. Ctrl = patte controlatérale ; Imm = patte immobilisée
16
Des variations des niveaux d’ARNm peuvent donc résulter de changement au niveau de la transcription et/ou de la traduction. Par exemple, les ARNm peuvent être dans un pool d’ARNm non traduits et migrer par la suite dans un pool d’ARNm traduits en réponse à une situation particulière. Cependant, cela ne se traduira pas par une modification des taux d’ARNm, mais pourrait avoir des conséquences sur la quantité de protéines produites. De plus, les taux d’ARNm peuvent également augmenter en réponse à un besoin accru lorsque la ½ vie de la protéine est réduite. Ceci peut donc conduire à une élévation des taux d’ARNm conjointement à une diminution des taux protéiques.
L’étude d’autres acteurs intervenant dans la phase de fusion entre les autophagosomes et les lysosomes, comme les protéines VAMP7 et VAMP8 qui appartiennent à la famille des protéines SNARE (soluble N-ethylmaleimide-sensitive fusion attachment protein receptors) (Moreau et al. 2013), pourrait permettre de préciser la régulation de cette étape terminale de l’autophagie.
L’ensemble de nos données suggèrent malgré tout que l’étape terminale de fusion entre les lysosomes et les autophagosomes pourrait être altérée. En effet, la faible augmentation de LC3-II pendant la remobilisation est plus en faveur d’une accumulation de LC3-LC3-II, plutôt que d’une conversion de LC3-I vers LC3-II. Ceci pourrait résulter d’une altération de la fusion des autophagosomes avec les lysosomes.
L’aggravation de l’atrophie musculaire observée dans le TA jusqu’à R6 (Slimani et al. 2012) pourrait donc s’expliquer, en partie, par une répression de l’autophagie. En effet, l’autophagie permet l’élimination des mitochondries déficientes par mitophagie, et des altérations de ce processus entraînent une atrophie musculaire (Masiero et al. 2009).
2. Etude des altérations mitochondriales au cours des phases d’immobilisation et de remobilisation musculaire dans le tibialis anterior chez le rat :
Pour étudier les fonctions mitochondriales, nous avons étudié des marqueurs de la biogenèse mitochondriale, de la dynamique mitochondriale (phénomènes de fission et de fusion), et enfin de la mitophagie.
2.1 La biogenèse mitochondriale
La biogenèse mitochondriale est régulée par le co-activateur transcriptionnel, PGC1α. La
Figure 11 montre que les taux d’ARNm de PGC1α ont fortement diminué dans le TA
immobilisé à I8 (-61%, P < 0,05, vs. I0). Après déplâtrage, ces taux d’ARNm sont progressivement revenus à des niveaux similaires à ceux de I0 à R6. On note une diminution
0 20 40 60 80 100 120 AR Nm OP A1/18S Uni té s ar b itr air es % I0 a b bc b d d bc cd cd IMMOBILISATION REMOBILISATION I8 R1 R6 R10 I0 C
Figure 12 : Régulation de la fusion mitochondriale dans le tibialis anterior immobilisé et remobilisé. (A) Les niveaux d’expression protéiques de MFN2 ont été déterminés par Western-Blots. Après quantification, les signaux ont été normalisés par l’intensité de la coloration BLOT FastStain pour corriger d’éventuelles différences de dépôts. Une image représentative a été inclue en-dessous des graphiques. (B et C) Les taux d’ARNm de MFN2 et OPA1 ont été déterminés par RT-qPCR. Les données ont été normalisées par rapport à l’ARN ribosomal 18S et exprimées en pourcentage de variation par rapport à I0.
Les données sont des moyennes ± SEM pour 6 à 11 rats/groupe. Les différences statistiques ont été testées par ANOVA, et les points affectés de lettres différentes sont significativement différents.
I0 = rats non plâtrés ; I8 = rats immobilisés 8 jours ; R1-R10 = rats remobilisés pendant 1 à 10 jours. Ctrl = patte controlatérale ; Imm = patte immobilisée
0 20 40 60 80 100 120 140 AR Nm M F N2/18S Uni té s ar b itr air es % I0 d ab c bc c d bc c a IMMOBILISATION REMOBILISATION I8 R1 R6 R10 I0 B 0 50 100 150 200 250 P roté in e M F N2 Uni té s ar b itr air es % I0 ab d ab ab b cd ac b ac ac I8 R1 R6 R10 I0 IMMOBILISATION REMOBILISATION A MFN2 80 kDa I0 I8 R1 R6 R10 Légende :