Caractérisation des inhibiteurs potentiels de DCIR, une
lectine de type C participant à la transmission du VIH-1
Mémoire
Thy-René NSIMBA BATOMENE
Maîtrise en Microbiologie - Immunologie
Maître ès sciences (M. Sc.)
Québec, Canada
Caractérisation des inhibiteurs potentiels de DCIR, une
lectine de type C participant à la transmission du VIH-1
Mémoire
Thy-René NSIMBA BATOMENE
Sous la direction de :
iii
Résumé
Le virus de l’immunodéficience humaine de type 1 provoque une infection définitive de l’organisme. Il entraine une déroute du système immunitaire depuis la primo-infection occasionnant ainsi, une déplétion massive des lymphocytes T CD4 (LTCD4). Le DCIR (Dendritic Cell Immuno Receptor) qui constitue le socle de notre travail, est une lectine de type C. Il est exprimé sur les cellules myéloïdes comme les cellules dendritiques mais aussi sur les cellules B, les LTCD4 infectés par le VIH-1 et apoptotiques ainsi que sur les LTCD4 polarisés en Th17. Il constitue un facteur d’attachement et d’internalisation du virus dans la cellule dendritique. Il permet son transfert aux LTCD4 dans les organes lymphoïdes secondaires, jouant ainsi un rôle dans la pathogenèse associée au VIH-1. En plus, le DCIR assure la régulation négative de la réponse cellulaire, favorisant ainsi la propagation et la réplication du virus au détriment de la réponse immunitaire contre le VIH-1. Le rôle que joue le DCIR est dépendant du sentier de signalisation induit à la suite de la phosphorylation des résidus tyrosine de son motif ITIM. Le blocage de DCIR par des inhibiteurs spécifiques pourrait empêcher cette phosphorylation et réduire l’attachement, l’internalisation et le transfert du virus. Nous avons montré que la stimulation des cellules dendritiques et des LTCD4 polarisés en Th17 avec un anticorps anti-DCIR générait un patron de phosphorylation des résidus
tyrosine des protéines. De plus, les inhibiteurs de la portion extracellulaire du DCIR inhibent cette activation. Afin de développer une mesure plus directe de l’interaction de DCIR avec ces inhibiteurs, nous avons purifié le DCIR à partir des cellules Raji-CD4-DCIR. En conclusion, ce projet de maitrise montre que l’activation directe de DCIR peut être renversée par des inhibiteurs montrant ainsi leurs spécificités. De plus, le profil d’activation de DCIR est spécifique pour chaque type cellulaire. A long terme, l’inactivation de DCIR par des inhibiteurs efficaces pourrait être une stratégie thérapeutique capable d’inhiber l’infection et de préserver une réponse immunitaire efficace.
iv
Table des matières
Résumé ... iii
Table des matières ... iv
Liste des figures ... vii
Liste des abréviations ... viii
Remerciement ... xii
Chapitre I : Introduction ... 1
1.1. Le virus de l’immunodéficience humaine de type I (VIH-1) ... 1
1.1.1. Structure et classification du VIH-1 ... 1
1.1.2. Organisation génomique du VIH-1 ... 2
1.1.3. Cycle de réplication du virus de l’immunodéficience humaine ... 4
1.1.3.1. Attachement/Fusion ... 4
1.1.3.2. Entrée du VIH-1 dans la cellule par endocytose ... 5
1.1.3.3. Décapsidation/Transcription inverse/Intégration nucléaire de l’ADN viral ... 5
1.1.3.4. Transcription ... 6
1.1.3.5. Traduction ... 7
1.1.3.6. Assemblage et bourgeonnement ... 7
1.1.4. Thérapie antirétrovirale du VIH-1 ... 8
1.1.5. Nouvelles approches thérapeutiques ... 9
1.1.6. Patients capables de contrôler la charge virale ... 11
1.1.7. Immunopathogénèse de l’infection à VIH-1 ... 12
1.1.7.1. Entrée du virus dans l’organisme ... 13
1.1.7.2. Perte de l’intégrité de la muqueuse intestinale ... 14
1.1.8. Immunopathogenèse du VIH-1 associée aux toll like receptors (TLRs) ... 16
1.2. Les lectines de type C et le VIH-1 ... 19
1.2.1. Les lectines ... 19
1.2.2. Classification des lectines ... 19
1.2.3. Les lectines de type C ... 20
1.2.3.1 Structure et classification des lectines de type C ... 20
1.2.3.2. Lectines de type C des cellules myéloïdes ... 21
1.2.3.3. Les lectines de type C de type II capable de lier le VIH-1 ... 24
v
1.2.3.3.2. Le DC-SIGN (CD209) ... 24
1.2.4. Le DCIR humain (CLEC4A) ... 26
1.2.4.1. Identification de DCIR ... 26
1.2.4.2. Expression de DCIR humain... 27
1.2.4.3. Structure du DCIR ... 28
1.2.4.4. Les ligands de DCIR ... 29
1.2.4.5. Fonctions du DCIR dans l’élaboration de la réponse immune ... 30
1.2.4.6. DCIR et VIH-1 ... 33
1.2.4.7. DCIR et LTCD4... 36
1.2.4.8. Les inhibiteurs de la portion extracellulaire de DCIR ... 38
Chapitre II : Hypothèse et objectifs ... 40
2.1. Hypothèse ... 40
2.2. Objectifs ... 41
Chapitre III : Matériels et méthodes ... 42
3.1. Réactifs ... 42
3.2. Anticorps ... 42
3.3. Cellules Raji-CD4-DCIR ... 43
3.4. Cellules dendritiques ... 43
3.5. Purification des LTCD4 et leur polarisation en Th17 ... 44
3.6. Évaluation de la phosphorylation des résidus tyrosine ... 45
3.7. Immunobuvardage ... 45
3.7.1. Migration sur gel de polyacrylamide 12% ... 45
3.7.2. Migration sur gel gradient de polyacrylamide 7,5 %- 20 % ... 46
3.8. Purification du DCIR et de HLA-DR par colonne d’affinité ... 47
3.8.1 Purification de DCIR... 47
3.8.1.1. Préparation de la colonne ... 47
3.8.1.2. Préparation de lysat cellulaire ... 48
3.8.1.3. Purification et élution ... 48
3.8.2. Purification de HLA-DR ... 48
3.9. Dosage des protéines ... 49
3.9.1. Dosage avec le Biodrop ... 49
3.9.2. Dosage avec le BCA ... 49
Chapitre IV : RESULTATS ... 50
vi
4.1.1. La présence de DCIR sur les Raji-CD4 ... 50
4.1.2. Détermination de la concentration saturante de l’anti-DCIR ... 51
4.1.3. Activation des Raji-CD4-DCIR et mesure du patron de phosphorylation des protéines sur leurs résidus tyrosine ... 52
4.2. Activation de DCIR dans les cellules primaires ... 54
4.2.1. Activation de DCIR sur les cellules dendritiques ... 54
4.2.2. Activation de DCIR sur les LTCD4 polarisés en Th17 ... 56
4.3. Évaluation de la liaison du DCIR avec les ligands ... 58
4.3.1. Détection de la protéine DCIR purifiée ... 58
Chapitre V : Discussion ... 59
5.1. Différence de phosphorylation dans les trois modèles cellulaires ... 60
5.2. Le rôle de DCIR dans la transmission de l’infection par le VIH-1 ... 60
5.3. Rôle du motif ITIM de DCIR dans l’infection par le VIH-1 ... 61
5.4. Les antirétroviraux versus les inhibiteurs de DCIR ... 62
5.5. Le DCIR dans le macrophage ... 63
5.6. Perspectives ... 64
Conclusions ... 66
vii
Liste des figures
Figure 1 : Structure morphologique du VIH-1………..3
Figure 2 : Organisation génomique du VIH-1………..3
Figure 3 : Cycle de réplication du VIH-1………...4
Figure 4 : Transmission du VIH-1 dans les muqueuses………..14
Figure 5 : Classification et structure des lectines de type C de type I et type II exprimées sur les cellules de la lignée myéloïde ………... 21
Figure 6 : Model d’un sentier de signalisation induite suite à l’activation des lectines de type C dectine-1, dectine-2 et Mincle dans la cellule myéloïde ………..23
Figure 7 : Les quatre formes de l’ARNm épissé de DCIR………28
Figure 8 : Voies de signalisation induite par le DCIR ………..33
Figure 9 : Rôle du motif ITIM dans la capture, internalisation et signalisation médiées par l’engagement de DCIR ……….36
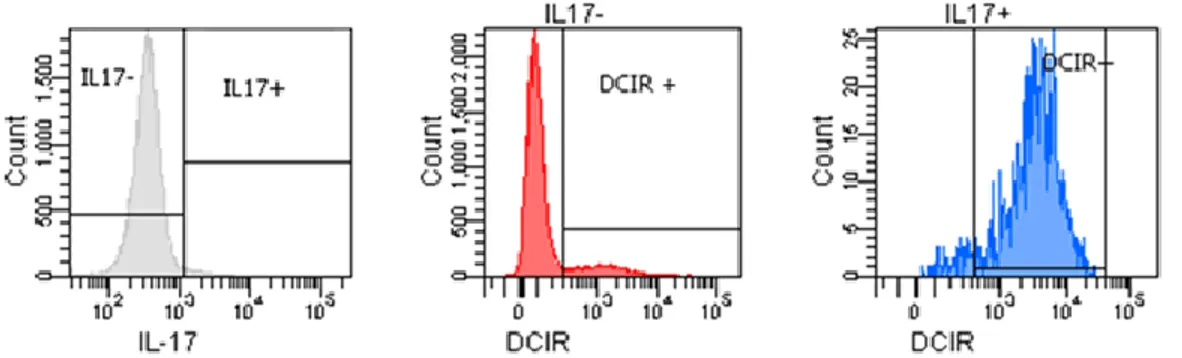
Figure 10 : Expression de l’IL-17 et de DCIR sur les LTCD4 polarisés en Th17……….37
Figure 11 : Inhibiteurs de la portion extracellulaires de DCIR obtenus après criblage virtuel…39 Figure 12 : Expression de DCIR sur les Raji-CD4………51
Figure 13 : Détermination de la concentration d’anticorps anti-DCIR saturante de DCIR…….52
Figure 14 : Augmentation de la phosphorylation des protéines sur leurs résidus tyrosine à la suite de l’activation de DCIR……….53
Figure 15 : Patron des protéines phosphorylées sur leurs résidus tyrosine à la suite de l’activation directe de DCIR sur les cellules dendritiques……….55
Figure 16 : Inhibition du patron de phosphorylation sur les cellules dendritiques par les inhibiteurs de DCIR………56
Figure 17 : Inhibition du patron de phosphorylation sur les LTCD4 Th17 par les inhibiteurs de DCIR……….57
viii
Liste des abréviations
ADN Acide désoxyribonucléique
ADNc Acide désoxyribonucléique complémentaire AP-1 Activator protein 1
APC Allophycocyanin
APOBEC3G Apolipoprotein B mRNA-editing enzyme-catalytic polypeptide-like-3G
ARN Acide ribonucléique
ARNm Acide ribonucléique messager ARNt Acide ribonucléique de transfert ARNdb Acide ribonucléique double brin ASGPR 1 et 2 Asialoglycoprotein receptor 1 et 2
BCA Acide Bicinchoninique
BCL B cell lymphoma 10
BCR B cell receptor, Recepteur de cellule B
BDCA-2 Blood dendritic cell antigen 2
BSA Bovine serum albumin
BTK Bruton’s tyrosine kinase
CA Capside
CARD9 Caspase recruitment domain family, member 9
CCR C-C chemokine receptor
CCL Chemokine (C-C motif) ligand
CD Cluster of differenciation
CLEC-1 et -2 C-type lectin domain family-1 et -2
CLEC-9A, 12B C-type lectin domain family-9A, 12B
CLECSF 6 et 8 C-type lectin superfamily member 6 et 8 CMH Complexe majeur d’histocompatibilité CNBr Cyanogen bromide CRD Carbohydrate recognition domain
CRF Circulating recombinant forms
CRISPR/Cas9 Clustered regularly interspaced short palindromic repeats/Caspase9
CTLA-4 Cytotoxic T-lymphocyte-associated antigen 4
CTLD C-type lectin-like domain
CXCR C-X-C chemokine receptor
CXCL Chemokine (C-X-C motif) ligand
DAMP Damage-associated molecular pattern
DAP-3 Death associated protein 3
DAP12 DNAX-activating protein of 12 kDa
DEC-205 Dendritic and epithelial cells, 205 kDa
Dectin-1 et -2 Dendritic cell-associated C-type lectin-1 et 2
DC Dendritic cell, Cellule dendritique
DCAR Dendritic cell immune-activating receptor
DCIR Dendritic cell immunoreceptor
DC-SIG N Dendritic cell-specific intercellular adhesion molecule-3-grabbing
ix
Env Enveloppe
EPN Glutamine-proline-asparagine EPS Glutamine proline-serine
ERK ½ Extracellular signal-regulated kinase 1/2
FcR Fc-receptor
Gag Groupe d’antigène
GALT Gut associated lymphoid tissue
gp glycoprotéine
GM-CSF Granulocyte macrophage colony-stimulating factor
HAART Highly active anti-retroviral therapy
HBSS Hank’s Buffered Salt Solution
Hck Hematopoietic cell kinase
hDCIR DCIR humain
HLA-DR Human leukocyte antigen–antigen D Related
HMGB High-mobility group protein
ICAM -2, -3 Intercellular adhesion molecule 2 et 3
IgA Immunoglobuline A IFN Interféron
IL Interleukine IN Intégrase
ITAM Immunoreceptor tyrosine-based activation motif
ITIM Immunoreceptor tyrosine-based inhibition motif
IRF3 Interferon regulatory factor 3
kDa Kilo dalton
KIR Killer immunoglobulin receptor
LARG Leukemia-associated Rho guanine nucleotide exchange factor
LPS Lipopolysaccharide
LTCD4/8 Lymphocyte T-cluster of differenciation 4/8
LTNP Long term non progressors
LTR Long terminal repeat
LRR Leucin-rich repeat
LOX-1 Lectin-like oxydized low-density lipoprotein receptor-1
MA Protéine de la matrice
MADCAM-1 Mucosale addressin cell adhesion molecule 1
MALT1 Mucosa-associated lymphoid tissue lymphoma translocation gene 1
MAPK Microtubule-associated protein kinase
MBP Mannose binding protein
mDCIR DCIR murin MGN Méganucléase
MICL Myeloid inhibitory C-type lectin-like receptor
MINCLE Macrophage-inducible C-type lectin
MMR Macrophage mannose receptor
MIP-1α et 1β Macrophage inflammatory protein 1 alpha et 1 beta
MyD88 Myeloid differentiation primary response gene 88
NC Nucléocapside Nef Negative factor
x NK Naturel killer
NKC Natural killer complex
NNRTI Non nucleoside reverse transcriptase inhibitors
NRTI Nucleoside reverse transcriptase inhibitors
PAK p21-activated kinase
PBMC Peripheral blood mononuclear cells
PD-1 Programmed cell death protein 1
PI3K Phosphoinositide-3-kinase
PIC Pre-integration complex
PKC Protein kinase C
PLC γ2 Phospholipase C gamma 2 Pol Polymérase
PR Protéase
PRR Pattern recognition receptor
RANTES Regulated on activation, normal T cell expressed and secreted
Rev Regulator of virion
RNAi RNA interference
SB Sample buffer
SDF-1 Stromal cell-derived factor 1
SIDA Syndrome d’immunodéficience humaine
SHP-1 et 2 Src homology-2 containing tyrosine phosphatase-1et 2
SHIP Src homology-2 (SH2)-containing tyrosine inositol phosphate
shRNA Small hairpin RNAs
siRNA Small interfering RNA
SU Protéine de surface (gp120) Syk Spleen tyrosine kinase
TALEN Transcription Activator-Like Effector Nuclease
Tat Trans-Activator of Transcription
TCR T-Cell receptor
TECK Thymus-expressed chemokine
TGF-β Tumor growth factor-beta
Th17 T helper 17
TIR Toll/IL-1 receptor
TIRAP TIR associated protein
TLR Toll like receptor
TM Protéine transmembranaire (gp41) TNF-α Tumor necrosis factor-alpha
TRAM TRIF lated adaptator molecule
TRIF TIR domain-containing adaptator protein induced IFN-β
VIH-1 Virus de l’immunodéficience humaine de type 1 Vpr Viral protein R
Vpu Viral protein U
xi
A mes parents, frères et soeurs A mon épouse et mes enfants Pour le soutien et encouragement
xii
Remerciement
Qu’il me soit permis au terme de mes études de maîtrise en microbiologie et immunologie, de remercier ma directrice de recherche, Dre Caroline Gilbert pour m’avoir accepté dans son laboratoire. Sa grande disponibilité en dépit de ses multiples occupations, sa passion pour la recherche et sa rigueur scientifique m’ont été d’un grand apport pour mon épanouissement scientifique. Le climat serein et l’esprit d’équipe qui règnent dans son laboratoire ont été déterminants pour mes recherches.
Je profite de cette opportunité pour exprimer ma gratitude aux professionnelles de recherche Alma Posvandzic et Myriam Vaillancourt pour leur encadrement. Mes remerciements vont également à Caroline Subra ainsi qu’à mes compagnons de lutte Audrey Hubert, Julien Pierre Vitry et Sofiane Berrazouane sans oublier Karianne St Gelais. Qu’ils trouvent ici, l’expression de ma reconnaissance.
Je voudrais enfin remercier le Programme Canadien de Bourses de la Francophonie (PCBF) et l’Agence Canadienne de Développement International (ACDI) ainsi que son agence d’exécution, l’Association des Universités et Collèges du Canada (AUCC) pour avoir pris en charge mes études de maîtrise et mon séjour au Canada. Je remercie enfin le gouvernement de la R.D.Congo pour m’avoir sélectionné.
1
Chapitre I : Introduction
1.1. Le virus de l’immunodéficience humaine de type I
(VIH-1)
1.1.1. Structure et classification du VIH-1
Le virus de l’immunodéficience humaine de type 1 (VIH-1) est un virus enveloppé à ARN monocaténaire, membre de la famille de retroviridae, de la sous famille des orthoretrovirinae et du genre lentivirus qui regroupe les virus cytopathogènes responsables d’infections persistantes et à évolution lente. Les rétrovirus possèdent une transcriptase inverse, une enzyme spécifique qui leur permet de rétrotranscrire l’ARN génomique viral en ADN double brin capable de s’intégrer dans le génome de la cellule hôte sous le nom de provirus, et de causer une infection définitive de l’organisme.
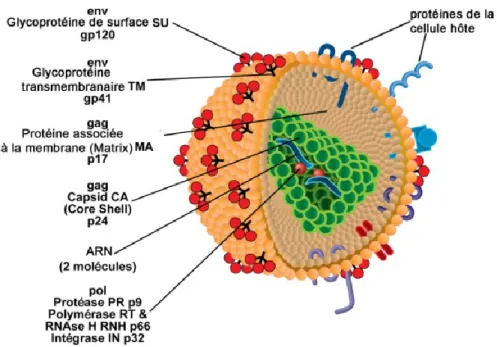
Tel que présenté à la figure 1, le VIH-1 a une forme sphérique et est doté d’une enveloppe formée d’une bicouche lipidique dérivée de la cellule hôte, comportant deux glycoprotéines de surface, la gp120 et la gp 41 ainsi que d’autres protéines issues de la membrane de la cellule hôte lors du bourgeonnement. Ces deux protéines forment à la surface du virus des complexes sous forme de spicules visibles au microscope électronique [1]. L’enveloppe virale entoure le core viral constitué d’une matrice et d’une capside à l’intérieur de laquelle on trouve deux molécules d’ARN monocaténaire, associées aux enzymes impliquées dans la réplication virale.
Le VIH-1 se classe en trois groupes dont le M (Major), O (Outlier) et le groupe N (virus appartenant ni au groupe M ni au groupe O). Le groupe M est le plus répandu et est responsable de la pandémie du sida dans 90% de cas. On trouve en son sein, neuf sous types ou clades définis de A à D, de F à H et de J à K dont les clades A, B, C et D sont en majorité responsables des infections par le VIH-1 [2]. Ces sous types sont inégalement répartis à travers le monde. L’infection d’un individu par plus d’un sous type viral peut engendrer des formes recombinantes en circulation (CRFs : Circulating recombinant forms) dont les plus répandus et impliqués dans la majorité des cas d’infections sont les recombinants CRF01-AE et CRF02-AG [2].
2
1.1.2. Organisation génomique du VIH-1
Le génome du VIH-1 comporte trois gènes de structure (gag, env et pol) impliqués dans la synthèse des protéines de structure, de l’enveloppe ainsi que des enzymes virales et six gènes accessoires et régulateurs de la réplication virale (tat, rev, nef, vif, vpr et vpu). Le gène
gag (groupe d’antigènes) code pour la protéine Gag (Pr55) dont le clivage par la protéase
virale amène à la synthèse des protéines de la matrice p17, des protéines de la capside p24 et des protéines de la nucléocapside p7 et p6 [3]. Le gène env (enveloppe) code pour la polyprotéine gp160 qui sera clivée par la protéase cellulaire pour générer les protéines membranaires gp120 et les protéines transmembranaires gp41 [4, 5], qui sont par la suite transportées vers la membrane plasmique de la cellule hôte pour y être incorporées lors du bourgeonnement. Le gène pol (polymérase) code pour les enzymes virales. Le génome du VIH-1 possède à ses extrémités 5’ et 3’ une séquence LTR (Long Terminal Repeat). Le 5’LTR est impliqué dans l’initiation de la transcription de l’ARN rétroviral et le 3’LTR sert de signal de coupure des gènes afin de donner naissance aux précurseur Gag, précurseur Gag-Pol et précurseur Env dont les deux premiers sont clivés par les protéases virales [6] et le troisième par les protéases cellulaires pour générer les protéines virales fonctionnelles. Les protéines codées par les gènes accessoires tat (Trans-Activator of Transcription), rev (Regulator of virion
expression), vpu (Viral protein U), vif (Viral infectivity factor), vpr (Viral protein R) et nef
(Negative factor), sont produites par la traduction des ARNm poly ou multi épissés et jouent un rôle dans le contrôle de l’expression des autres protéines virales ainsi que dans le cycle réplicatif du virus et la pathogenèse.
3
Figure 1 : Structure morphologique du VIH-1 (Adapté du Laboratoire Caroline Gilbert)
4
1.1.3. Cycle de réplication du virus de l’immunodéficience
humaine
Le cycle de la réplication du VIH-1 comprend de nombreuses étapes qui sont brièvement illustrées à la figure 3 et décrites ci-dessous.
Figure 3 : cycle de réplication du VIH-1 (Adaptée de [8])
1.1.3.1. Attachement/Fusion
Tout commence par la fixation de la glycoprotéine gp120 du VIH-1 au récepteur cellulaire CD4 présent sur les LTCD4, mais aussi présent à la surface membranaire des monocytes, des cellules dendritiques et des microglies dans le cerveau [1]. Ces cellules constituent les principales cibles du VIH-1. La gp120 à travers son site de liaison (CD4 Binding Site) interagit avec le domaine D1 du récepteur CD4. Il s’en suit un changement conformationnel de la gp120 permettant l’exposition de son domaine V3 qui contient le site de liaison pour les récepteurs de chimiokines (CCR5 ou CXCR4) [1, 9], appelés aussi corécepteurs. La liaison de la gp120 avec le CD4 et l’un de ces corécepteurs permet la fusion de la membrane virale avec celle de la cellule hôte. En effet, l’interaction entre la boucle V3 de la gp120 et le CCR5 (CD195) ou le CXCR4 (CD184) démasque la gp41 et rapproche la membrane du virus et celle
5
de la cellule hôte, permettant ainsi à la gp41 ancrée dans la bicouche lipidique virale d’engendrer la fusion des deux membranes grâce à son peptide de fusion. La capside virale est ainsi relâchée dans le cytoplasme.
Les récepteurs de chimiokines (CCR5 et CXCR4) découverts en 1996 [10] sont des protéines cellulaires avec sept domaines transmembranaires, associées aux protéines G. Elles sont exprimées sur les lymphocytes T, les macrophages/monocytes et les cellules dendritiques, mais à de différents niveaux. Les CCR5 est plus grandement exprimé sur les LTCD4 mémoire que sur les autres sous-types des lymphocytes T et le CXCR4 par contre, a une plus grande expression sur les LTCD4 naïfs [11]. Les ligands naturels de CCR5 sont le RANTES, le MIP-1α (CCL3) et le MIP-1β (CCL4) et celui de CXCR4 par contre, est le SDF-1 (CXCL12) [12, 13]. La spécificité de l’interaction de la région variable V3 de la gp120 avec le corécepteur indique le tropisme du virus [14-16].
1.1.3.2. Entrée du VIH-1 dans la cellule par endocytose
En dehors de la fusion qui se fait entre l’enveloppe virale à travers la gp120 et la membrane cytoplasmique à travers le CD4 et l’un des corécepteurs CCR5 ou CXCR4, le VIH-1 peut pénétrer dans la cellule hôte par endocytose. En effet, le VIH-1 peut se lier à d’autres récepteurs membranaires notamment les lectines de type C, et être internalisé dans les endosomes des cellules cibles comme la cellule dendritique, le monocyte et le macrophage. La voie d’endocytose permet aux cellules de séquestrer le virus dans les endosomes et d’assurer son transfert aux cellules avoisinantes [17, 18].
1.1.3.3. Décapsidation/Transcription inverse/Intégration nucléaire de l’ADN
viral
Une fois dans le cytoplasme, la déstabilisation de la capside à la suite de la phosphorylation des protéines de la matrice par les kinases associées au virus [19], favorise la libération de tout son contenu incluant les deux brins d’ARN et les enzymes virales. L’ARN viral simple brin est rétrotranscrit en ADN simple brin par la transcriptase inverse, en utilisant une amorce d’origine cellulaire liée au génome viral (ARNt Lys3 ). En même temps, la RNAse H de la
transcriptase inverse procède à la dégradation totale de l’ARN matriciel et laisse indemnes les courtes séquences riches en purines qui serviront d’amorces pour la synthèse du second brin
6
en utilisant comme matrice, l’ADN proviral simple brin. La transcriptase inverse est dépourvue d’une activité exonucléase 3’ vers 5' et elle peut fréquemment changer des matrices nécessaires pour la rétrotranscription du génome virale. Ceci fait que cette enzyme introduit des erreurs qui sont responsables de la grande variabilité génétique du VIH-1 et conséquemment de la résistance aux antirétroviraux et à l’échappement au système immunitaire [20].
L’ADN proviral double brin ainsi synthétisé, reste lié aux protéines virales et cellulaires formant ainsi, un complexe de pré-intégration (PIC : pre-integration complex) composé essentiellement de l'ADN proviral double brin, de l’intégrase, de la transcriptase inverse, des protéines de la matrice, de l’amorce ARNt Lys3,de la Vpr, et des protéines d’origine cellulaire comme HMG-I/Y
(High mobility group protein). Le mécanisme par lequel l’ADNc viral est transporté dans le noyau est encore mal connu et diffère en fonction du type cellulaire. Il est proposé que le PIC pénètre dans le noyau par les pores nucléaires soit grâce à la machinerie cellulaire d’import nucléaire associée aux protéines virales (Intégrase, protéines matricielles et Vpr) ayant des propriétés caryophiles, soit grâce à une structure inhabituelle (central DNA flap) contenue dans l’ADN viral [21, 22].
Dans le noyau, l’intégrase virale coupe quelques nucléotides dans chacune des extrémités de l’ADN viral et clive en même temps l’ADN chromosomique de la cellule infectée de manière à permettre la jonction de ces deux molécules. L’ADN viral est ainsi intégré dans le génome de la cellule sous la forme de provirus grâce à l’action de l’intégrase virale [23], et la réplication du virus commence ou sa latence s’installe.
1.1.3.4. Transcription
Une fois dans le noyau, l’ADN proviral est transcrit en ARNm viral par une enzyme cellulaire, l’ARN polymérase II. Cependant, cette transcription nécessite que la cellule soit dans un état d’activation. En effet, les facteurs de transcriptions présents dans la cellule activée notamment le NF-KB (Nuclear factor-kappa B), interagissent avec le promoteur viral situé sur le 5’LTR et permettent la transcription des gènes viraux en ARN viral génomique, ARNm partiellement épissé ou non épissé et en ARNm poly ou complètement épissé. À cette étape du cycle, la protéine Tat favorise la transcription virale efficiente en permettant à l’ARN polymérase II de
7
synthétiser des longs transcrits [24] et la protéine Rev assure l’exportation des ARNm viraux du noyau vers le cytoplasme [25].
1.1.3.5. Traduction
Dans le cytoplasme, les ARNm poly ou multi-épissés sont traduits en protéines régulatrices (Tat, Rev, Nef, Vif, Vpu, Vpr) et les ARNm peu ou non épissés sont traduits en polyprotéines Gag, Gag-Pol et Env. La polyprotéine Env est clivée par une protéase cellulaire pour donner naissance à deux protéines de l’enveloppe virale (gp120 et gp41) et les polyprotéines Gag et Gag-Pol par contre, vont subir un clivage protéolytique par la protéase virale, au moment du bourgeonnement pour aboutir aux protéines internes du virus (Matrice, Capside, Nucléocapside et les trois enzymes virales : Transcriptase inverse, Intégrase et Protéase).
1.1.3.6. Assemblage et bourgeonnement
Ils constituent les deux dernières étapes du cycle viral et interviennent au moment où la quasi-totalité des protéines virales est synthétisée et est présente dans le cytoplasme de la cellule infectée. Toutes les protéines virales y compris l’ARN génomique du virus s’assemblent au niveau de la face interne de la membrane plasmique sous le contrôle de la polyprotéine Gag myristoylée [26]. Le bourgeonnement se passe dans les corps vésiculaires pour les macrophages [27], et les virus sont ensuite libérés de la cellule après fusion de ces corps vésiculaire avec la membrane cytoplasmique.
La maturation des particules virales a lieu lorsque les virions sont relâchés dans l’environnement extracellulaire via la protéine Vpu. Par la suite, intervient le clivage des précurseurs de Gag et Gag-Pol par la protéase virale qui va générer les protéines constitutives internes (P6MA, P7MA, P17CA et P24NC) et les trois enzymes virales (transcriptase inverse, intégrase et protéase). La capside se condense et prend la forme en cône. La particule virale devient ainsi mature et infectieuse.
Les traitements antirétroviraux disponibles ciblent plusieurs de ces étapes du cycle réplicatif du virus.
8
1.1.4. Thérapie antirétrovirale du VIH-1
Les médicaments antirétroviraux actuellement sur le marché ne guérissent pas le sida, mais jouent un rôle important dans la prévention de la transmission du VIH-1 et dans l’augmentation de l’espérance de vie des patients. Généralement, une combinaison de trois molécules antirétrovirales est requise pour supprimer la réplication virale [28] afin de retarder la survenue du sida. Cependant, ce traitement ne restaure pas l’état de santé de patient et doit être maintenu à vie pour prolonger l’espérance de vie.
Les agents antirétroviraux sont répartis en six différentes classes en fonction de leur mécanisme et de leur cible. Ces classes sont : les inhibiteurs de la transcriptase inverse : NRTIs et NtRTIs (nucleoside and nucleotide analog reverse transcriptase inhibitors) et NNRTIs (Non-nucleoside reverse transcriptase inhibitors), les inhibiteurs d’intégrase, les inhibiteurs des protéases, les inhibiteurs de fusion et les inhibiteurs d’entrée (antagoniste de corécepteur CCR5) [29]. Chacun de ces agents pharmacologiques agit sur une cible spécifique dans les différents stades du cycle réplicatif du VIH-1. [30]. La réussite du traitement est mise en évidence par une élévation du nombre de LTCD4 et une diminution de la charge virale à un niveau indétectable (inférieur à 40 copies/ml) dans le sang et dans les sécrétions génitales [31, 32].
Bien qu’ayant un potentiel de supprimer la réplication virale, de réduire la transmission du virus et de retarder la survenue du sida, les antirétroviraux ne peuvent pas éliminer le virus latent dans les cellules. Il est donc important de chercher à comprendre davantage la physiopathologie du VIH-1, afin de découvrir des nouvelles cibles thérapeutiques et de développer des nouvelles approches thérapeutiques pour mieux lutter contre ce fléau. Il serait en outre intéressant que les nouvelles approches thérapeutiques puissent résoudre le problème d’échappement thérapeutique dû à la mutation virale et aux variants viraux, mais aussi le problème d’effets secondaires dus à la toxicité des médicaments. Une autre alternative serait d’aborder une approche qui puisse sortir le virus de sa latence. Ceci permettrait de guérir les patients de l’infection à VIH-1. Une autre approche serait de fournir une cure fonctionnelle, c’est-à-dire de contrôler le virus par immunothérapie.
9
1.1.5. Nouvelles approches thérapeutiques
Parmi les nouvelles approches thérapeutiques en cours de développement, la thérapie génique peut faire recours au RNAi (RNA interference) comme les shRNA (small hairpin
RNAs) et les siRNA (small interferingRNA) [33, 34], aux nucléases au doigt de zinc (ZFNs : Zinc Finger Nucleases) [35, 36], aux nucléases effectrices de type activateurs de transcription
(TALENs : Transcription Activator-Like Effector Nuclease) [37], au CRISPR/Cas9 (Clustered
Regularly interspaced Short Palindromic Repeats/Caspase9) [38, 39] ainsi qu’aux méganucléases (MGNs) et aux peptides d’acides nucléiques [40], pour modifier le gène cible. Ceci peut conduire vers une cure fonctionnelle. Par exemple, l’édition de gène peut réprimer de manière permanente le gène qui code la protéine CCR5 ou CXCR4 conférant ainsi à la cellule cible une résistance permanente à l’infection par le VIH-1 [37, 40-43]. La thérapie génique dirigée contre le VIH-1 peut constituer une approche personnalisée encore assez risquée et dispendieuse pour éradiquer le VIH-1. En effet, les scientifiques envisagent cette approche pour réduire au silence les gènes codant les protéines virales et ceux de la machinerie cellulaire impliqués dans la réplication virale, ainsi que ceux qui codent les protéines impliquées dans la liaison et la fusion du VIH-1. Ceci peut permettre de contrôler la réplication du VIH-1 et de bloquer l’infection dès les premières étapes du cycle viral, notamment la fusion et l’entrée du virus, la rétrotranscription et l’intégration de l’ADN viral dans la cellule hôte [28, 44]. Ainsi, le siRNA et le shRNA synthétiques et spécifiques au VIH-1 sont capables de réprimer l’expression post-transcriptionnelle des gènes viraux en ciblant des transcrits viraux tels que tat, rev, gag, pol, nef, env, vpr et LTR au niveau du cytoplasme [45]. Ces RNAi peuvent aussi cibler le génome viral intégré dans le génome de la cellule hôte [46-48] et le réduire au silence en dégradant l’ARNm viral. A l’instar des antirétroviraux, le virus peut s’échapper à cette approche thérapeutique suite aux multiples mutations occasionnées notamment par la transcriptase inverse. Ces mutations peuvent changer la structure des ARNm viraux et empêcher les RNAi de s’apparier à leurs séquences cibles et complémentaires [49-51]. Ainsi, il est important que le RNAi dirigé contre le VIH-1 puisse cibler les régions hautement conservées du génome viral de manière à éviter ou à réduire l’échappement thérapeutique [45]. La combinaison des RNAi (siRNA ou shRNA) ou la combinaison des siRNAs avec les antirétroviraux permet de cibler plusieurs sites de réplication virale et peut ainsi, contourner la résistance due aux mutations [49, 52, 53].
10
Les siRNA ou shRNA spécifiques au CCR5 semblent être intéressants, dans la mesure où ce récepteur n’est pas requis comme un facteur jouant une fonction immune essentielle de lymphocyte T [54]. En outre, il a été montré que les personnes déficientes du récepteur CCR5 à la surface des cellules progénitrices hématopoïétiques étaient résistantes à l’infection par le VIH-1, sans que le fonctionnement normal de ces cellules ne soit perturbé. Cependant, le CXCR4 est requis comme un facteur de maturation des lymphocytes T [55]. Sa suppression peut avoir des répercussions sur la maturation de ces cellules. Une autre limite de la thérapie génique est liée au potentiel immunoactivateur des vecteurs viraux et de RNAi, qui peuvent être reconnus par les TLRs endosomaux et induire une réponse inflammatoire suivie de la production des INF-α [45, 56]. Ces éléments doivent être pris en compte avant de tenter l’application des RNAi comme thérapie génique en clinique. Cependant, la plus grande difficulté reste le mode de livraison de RNAi dans la cellule ou le tissu cible [45], ceci implique l’utilisation d’un vecteur spécifique qui ne soit pas immunogène. La thérapie génique dirigée contre le VIH-1 est donc prometteuse, mais il faut encore beaucoup des recherches avant que son utilisation en clinique ne soit effective.
La latence virale constitue un grand obstacle qui empêche l’éradication définitive de l’infection à VIH-1. Ainsi, obtenir plutôt une cure fonctionnelle, une stratégie immunologique incluant le vaccin thérapeutique, les anticorps monoclonaux neutralisants et les inhibiteurs des immunomodulateurs serait plus réaliste. Par exemple, le vaccin thérapeutique jouerait le rôle d’augmenter la réponse immunitaire cytotoxique spécifique au VIH-1. Ceci permettrait le contrôle de l’infection par le système immunitaire. Les anticorps monoclonaux neutralisants dirigés contre le VIH-1 pourraient cibler et activer les cellules en latence virale de manière à stimuler la réplication du VIH-1 et la déplétion de ces cellules. Les immunorégulateurs, tel que la protéine PD-1 (Programmed cell death protein 1), jouent un rôle dans l’homéostasie immunitaire et leurs inhibitions contribueraient à augmenter le potentiel du système immunitaire pour faire efficacement face à l’infection [57, 58]. Pour parvenir à une cure fonctionnelle basée sur l’immunothérapie, une bonne compréhension du mécanisme par lequel les cellules maintiennent la latence virale et échappent au contrôle du système immunitaire [58] ainsi que des mécanismes immunitaires qui sont impliqués dans le contrôle du virus s’avère nécessaire. Dans ce sens, les observations et les études réalisées chez les
11
patients appelés contrôleurs élites du VIH-1 ont montré que ces derniers sont capables de contrôler leurs charges virales en dehors de tout traitement antiviral.
1.1.6. Patients capables de contrôler la charge virale
Les contrôleurs du VIH-1 ou Elite controllers en anglais, constituent un groupe de personnes infectées par le VIH-1 dont l’organisme est capable de contrôler de manière spontanée et durable la réplication du virus en l’absence de traitement antirétroviral [59]. Ces individus ne développent pas le sida. Ils maintiennent la charge virale plasmatique à des taux non détectables en clinique (inférieur à 50-75 copies /ml) pour une longue période. Le taux des LTCD4 reste par conséquent élevé, réduisant ainsi, le risque de progression vers la phase sida [59, 60]. Les contrôleurs élites représentent moins de 1% de l’ensemble de personnes infectées par le VIH-1 [60-62]. Ils constituent un modèle naturel du contrôle virologique par l’organisme.
Les contrôleurs élites sont différents des progresseurs lents du VIH-1 (LTNP : Long term non
progressors). En effet, ces derniers sont définis comme étant un groupe d’individus infectés
par le VIH-1, mais maintenant le taux de CD4 à un niveau élevé pendant plusieurs années en l’absence des traitements antirétroviraux [60, 63]. Ce sont donc des personnes ayant un phénotype avec une grande stabilité immunologique et dont la charge virale peut être basse ou modérée [60, 64]. Les deux groupes d'individus, bien que différents par leur définition, constituent deux phénotypes capables de gérer le VIH-1 sans développer le SIDA pendant plusieurs années. Une bonne compréhension du mécanisme biologique de ces deux modèles naturels peut permettre une mise en place d’un vaccin thérapeutique ou une thérapie antivirale les mimant et capable de contrôler la réplication virale [59].
Les contrôleurs élites et les progresseurs lents arrivent à contrôler la réplication virale grâce à leurs lymphocytes T cytotoxiques spécifiques aux antigènes viraux [65]. Chez certains contrôleurs élites, ces lymphocytes sont associés aux allèles du complexe majeur d’histocompatibilité du type I (CMH I) tels que le HLA B*27, le HLA B*57 et le HLA C. Ces facteurs immunogénétiques particuliers leur confèrent un potentiel de résistance contre la réplication du VIH-1. Les LTCD8 cytotoxiques associés à ces facteurs génétiques protecteurs,
12
possèdent une grande avidité aux épitopes du VIH-1 [66, 67], et sont capables de détruire les LTCD4 infectés par le virus. Une fois activés, les LTCD8 peuvent induire la production des enzymes cytolytiques comme les granzymes B et les perforines [68, 69], mais aussi la production des cytokines pro-inflammatoires comme l’IL-2 nécessaire à la prolifération cellulaire et l’IFN-γ ayant une activité antivirale. Tous ces éléments contribuent de manière naturelle au contrôle de la charge virale.
Un autre mécanisme permettant aux contrôleurs élites et aux progresseurs lents de contrôler la charge virale est l’immunité innée due par l’activation de cellule NK (Naturel killer). En effet, il a été montré in vitro que l’allèle HLA-Bw4 du HLA-B*57 était fortement associé aux récepteurs de la cellule NK, le KIR (killer immunoglobulin receptor) tels que KIR3DS1 et KIR3DSL1 [70-72]. Ces résultats ont montré que l’association entre le KIR et ses ligands, HLA-B HLA-Bw4 permettait le contrôle de la réplication virale et par conséquent empêchait la progression de l’infection vers la phase SIDA. Il a été également montré que l’inhibition de la réplication virale était plus efficace dans la cellule NK exprimant l’allèle KIR3DS1 que dans celle ne l’exprimant pas [72].
En plus des facteurs immunogénétiques décrits ci-dessus, il en existe d’autres notamment les facteurs intrinsèques de restriction ou les facteurs participant à l’immunité intrinsèque. C’est le cas de la protéine APOBEC3G qui est présente en plus grande quantité dans les LTCD4 des contrôleurs élites. Ceci permet une limitation de la réplication du VIH-1 sur les cellules cibles, contribuant ainsi, avec les autres facteurs cités ci-haut au contrôle de la charge virale [73]. Ces observations recueillies chez les contrôleurs élites et les progresseurs lents montrent qu’une réponse immunitaire efficace peut contrôler ce virus. Il est donc important de bien connaitre l’immunopathogénèse de l’infection à VIH-1.
1.1.7. Immunopathogénèse de l’infection à VIH-1
L’infection par le VIH-1 est caractérisée dès la primo-infection, par une déplétion massive et progressive des LTCD4 et par une dérégulation irréversible du système immunitaire.
13
1.1.7.1. Entrée du virus dans l’organisme
La barrière des muqueuses génitales ou digestives est la principale voie d’entrée du virus dans l’organisme. Dans la lamina propria, le virus va entrer en contact avec les cellules susceptibles à l’infection [74], notamment les cellules dendritiques myéloïdes, les LTCD4 à mémoire effectrice et les macrophages [75].
Les cellules dendritiques présentes dans la muqueuse constituent les premières cellules à reconnaitre les pathogènes et à provoquer l’induction de la réponse immunitaire adaptative par les lymphocytes T. Les cellules dendritiques qui reconnaissent les pathogènes à travers leurs PRR (pattern recognition receptors), migrent vers les organes lymphoïdes secondaires, grâce à leurs récepteurs de chimiokines CCR7 qui interagissent avec leurs ligands, CCL19 ou CCL21 produits par les cellules de stroma [76]. L’interaction entre l’antigène lié au CMHII et le complexe TCR/CD3, accompagnée de l’interaction des molécules de costimulation, provoquent l’activation des LTCD4 naïfs qui seront par la suite polarisés en LTCD4 mémoires et effecteurs (de type Th1, Th2, Th17) et en LTCD4 régulateurs (Treg).
Dans les tissus lymphoïdes de la muqueuse gastro-intestinale, les cellules dendritiques CD103+/- secrètent l’acide rétinoïque conférant aux lymphocytes T effecteurs un tropisme digestif en leur induisant l’expression de récepteurs de chimiokines CCR9 et des molécules d’adhésion α4β7 [77-79]. Ces molécules interagissent respectivement avec leurs ligands CCL25/TECK (Thymus-Expressed chemokines) et MADCAM-1 (Mucosal vascular addressin
cell adhesion molecule) et permettent la migration aussi bien des lymphocytes T effecteurs
que des lymphocytes B effecteurs ou plasmocytes sécréteurs d’IgA vers la muqueuse [77, 80, 81].
La cellule dendritique ayant capturé le VIH-1 grâce à ses PRR, notamment les lectines de types C comme le DC-SIGN, le DC205, le MMR, ou le DCIR, l’internalise dans les endosomes et migre dans les organes lymphoïdes secondaires, comme le ganglion lymphatique ou la cellule dendritique va transférer le virus aux LTCD4 lors des interactions cellulaires. Dans les organes lymphoïdes, la réplication du virus est très intense provoquant ainsi, la déplétion massive des LTCD4 et occasionnant la propagation du virus dans d’autres organes [82]. La plus grande déplétion de LTCD4 se passe dans le tractus gastro-intestinal, car cette muqueuse héberge un grand nombre de lymphocytes T mémoires activés ayant un phénotype
14
CD4+CCR5+, susceptibles à l’infection par le VIH-1 [83, 84]. Cette déplétion massive entraine
la détérioration du système immunitaire et de l’architecture de la muqueuse.
Figure 4 : Transmission du VIH-1 dans les muqueuses (Adapté de [85])
1.1.7.2. Perte de l’intégrité de la muqueuse intestinale
Les muqueuses constituent une barrière de protection contre les microorganismes présents dans l’environnement extérieur. Les tissus lymphoïdes qui s’y trouvent jouent un rôle crucial pour le contrôle de l’inflammation et de l’activation immunitaire, participant ainsi, à l’intégrité de la barrière mucosale [86]. Dans la muqueuse gastro-intestinale, les LTCD4 polarisés en Th17
15
contribuent à l’homéostasie de la muqueuse digestive. En effet, grâce à la sécrétion de l’IL-17 et l’IL-22, ils assurent le maintien de l’intégrité de la barrière mucosale.La cellule dendritique CD103+, en secrétant l’acide rétinoïque, permet à d’autres cellules comme les cellules lymphoïdes innées de sécréter l’IL-17 et l’IL-22, deux cytokines associées au maintien de l’intégrité structurale de la barrière épithéliale [87], protégeant ainsi les jonctions serrées entre les cellules épithéliales. Les LTCD4 Th17 sont des cellules très permissives à l’infection par le VIH-1 [88]. Ainsi, lors des premières étapes de l’infection par le VIH-1, on observe une déplétion massive de ces cellules au niveau des organes lymphoïdes secondaires du tractus gastro-intestinal [89], menant subséquemment à une diminution importante de production de l’IL-17 et l’IL-22. Ceci occasionne un dysfonctionnement et une perte de l’intégrité de la muqueuse, résultant par une translocation microbienne et des produits microbiens.
La perte de l’intégrité de la barrière mucosale est aussi observée suite au dysfonctionnement des cellules B durant l’infection par le VIH-1. Il mène à une diminution de production des immunoglobulines A (IgA). Il est cependant connu que l’IgA joue un rôle dans la protection de la muqueuse contre la translocation microbienne. Cet anticorps est capable de former des complexes avec les bactéries, leur empêchant ainsi d’être en contact avec l’épithélium. Il peut en outre assurer la neutralisation des produits microbiens [90]. Le dysfonctionnement des cellules B induit à la suite de l’infection par le VIH-1 incluant la diminution du nombre d’IgA contribue à la perte de l’intégrité de la barrière épithéliale [91, 92].
Après la perte de l’intégrité mucosale, on observe chez les personnes infectées, une augmentation des taux plasmatiques des produits microbiens notamment le lipopolysaccharide qui constitue un indice clé de la translocation microbienne et prédicteur de la progression de la maladie [93]. On observe en outre l’augmentation d’autres marqueurs associés à la translocation microbienne, mais aussi à la sévérité de la maladie, comme l’ADN ribosomique 16s bactérien [94] et le CD14 soluble [95]. Tous ces marqueurs peuvent persister et rester élevés dans le plasma après plusieurs années de traitement aux antirétroviraux [96, 97]. Les bactéries et les produits microbiens ayant subi la translocation peuvent être reconnus par les PRR comme les TLRs exprimés sur les cellules présentatrices d’antigènes. Ceci occasionne l’activation des certaines voies de signalisation cellulaires notamment le NF-kB résultant par la production des cytokines pro-inflammatoires [98]. Les LTCD4 sont activés par
16
la présentation antigénique, mais aussi par les cytokines pro-inflammatoires et cette activation du système immunitaire favorise la réplication du VIH-1.
1.1.8. Immunopathogenèse du VIH-1 associée aux toll like
receptors (TLRs)
Les TLRs (Toll Like Receptor) sont des PRRs qui reconnaissent les motifs conservés des pathogènes PAMP (Pathogen-Associated Molecular Patterns) et DAMP (Damage-Associated
Molecular Patterns) provoquant l’activation cellulaire et la production des cytokines
inflammatoires et de plusieurs molécules antivirales [99]. Les TLRs sont exprimés par la majorité des cellules de l’immunité innée y compris les cellules endothéliales. Certains parmi eux sont membranaires tels que le TLR1, le TLR2, le TLR4, le TLR5, le TLR6, le TLR10 et le TLR11. D’autres par contre sont localisés dans les compartiments endosomaux, il s’agit de TLR3, TLR7, TLR8 et de TLR9 [99-101] et sont impliqués dans la défense contre les virus [102, 103]. Ils contiennent dans leurs parties extracellulaires un domaine LRR (Leucin-Rich
Repeat) et dans la partie cytoplasmique une région hautement conservée, un domaine TIR
(Toll/IL-1 receptor). L’activation des TLRs par leurs ligands spécifiques induit une cascade des voies de signalisation dépendante ou indépendante de MyD88 (Myeloid differentiation primary
response gene 88) aboutissant à l’activation des facteurs de transcription tels que NF-Kb
(Nuclear factor-kappa B), AP-1 (Activator protein 1) et IRF3 (Interferon regulatory factor 3). La voie dépendant de MyD88 conduit à l’activation de facteur de transcription NF-kB qui favorise la production des cytokines pro-inflammatoires et au facteur IRF3 spécifique à la production d’IFN-β après activation de TLR7, TLR8 et TLR9. Cette voie est commune à tous les TLRs sauf le TLR3 dont la voie de signalisation est dépendante d’une protéine adaptatrice, le TRIF (TIR domain-containing adaptator protein induced IFN-β). L’activation de TLR4 par contre peut engager les deux voies et implique comme protéines adaptatrices, les TRAM (TRIF lated
adaptator molecule)/TRIF et les TIRAP (TIR associated protein)/MyD88 [103].
Les TLRs exprimés sur les cellules de l’immunité innée notamment les cellules myéloïdes tels que les monocytes, les macrophages et les cellules dendritiques contribuent de manière directe dans la stimulation du système immunitaire des personnes infectées par le VIH-1. Cette activation chronique du système immunitaire est en faveur de la réplication virale
17
intense, à la déplétion massive des LTCD4 [87, 104]et provoque la dérégulation de la réponse immunitaire [105], préparant ainsi la route vers l’étape sida.
La présence de LPS dans le sang de personnes infectées par le VIH-1 constitue un indice remarquable de la translocation microbienne. Ainsi, le TLR4 est capable de le reconnaitre et d’induire l’activation du système immunitaire inné ou adaptatif. L’activation des TLRs par les pathogènes ou les produits microbiens favorise la sécrétion des cytokines pro-inflammatoires notamment le TNF-α, l’IFN-α, l’IL-1, l’IL-6 et l’IL-18 qui, en dehors de leur rôle activateur du système immunitaire innée [106, 107] participent aussi à l’apoptose des cellules de l’immunité des personnes infectées par le VIH-1. La modulation de ces cytokines par l’immunothérapie peut constituer une approche thérapeutique [107].
En dehors de LPS, d’autres produits microbiens notamment l’ARN, l’ADN, la flagelline et le peptidoglycane sont susceptibles d’être reconnus par les TLRs et participent à l’activation chronique du système immunitaire des personnes infectées par le VIH-1. En effet, les TLRs 7/8 reconnaissent l‘ARN génomique du VIH-1, mais aussi les ARNs simples brins des autres pathogènes permettant ainsi l’activation directe du système immunitaire. Leur expression particulière sur les cellules dendritiques plasmacytoïdes permet la sécrétion d’INF-α, l’une des cytokines jouant un rôle non seulement dans l’immunité innée et adaptative [108, 109], mais aussi dans l’apoptose des LTCD4 [110]. Il en est de même pour le TLR9 localisé particulièrement dans le réticulum endoplasmique et le TLR3 qui se lient respectivement à leurs ligands, CpG-DNA et ARNdb et par conséquent participent à l’activation immunitaire. Le TLR2 constitue un bon détecteur de mycobactérium tuberculosis lors de la co-infection sida-tuberculose [111]. L’infection chronique à VIH-1 est associée à l’augmentation de l’expression des TLRs sur les cellules de l’immunité innée [112] notamment le TLR2 sur le monocyte, contribuant ainsi à l’inflammation et à la réplication virale.
Il a été montré que la pré-stimulation de TLR8 par l’ARN génomique du VIH-1 ou par un autre ligand qui lui est spécifique, entrainait l’augmentation de la production des cytokines pro-inflammatoires par le biais de TLR4 exprimé sur le monocyte, après sa stimulation par le LPS issu de la translocation microbienne [113]. La stimulation croisée des certains TLRs pendant
18
l’infection chronique par le VIH-1, peut donc amplifier l’activation immunitaire et accélérer la réplication virale.
La voie de TLRs impliquée dans l’activation chronique du système immunitaire peut être considérée comme une cible thérapeutique potentielle capable de réduire l’immunopathologie de l’infection par le VIH-1 [114]. Comme dit ci-haut, la stimulation des TLRs endosomaux induit une cascade des voies de signalisation aboutissant à une réponse immunitaire antivirale par la production des molécules antivirales notamment l’IFN-β, l’IFN-α et IFN-γ [99]. L’utilisation des agonistes de ces TLRs ou autres molécules qui miment l’ARN génomique du VIH-1 peut générer une réponse immunitaire anti-VIH-1 capable d’inhiber la réplication virale dans les premières étapes de l’infection [115]. Il a ainsi été montré que les agonistes de TLR7, comme le gardiquimod étaient capables de stimuler le TLR7 par une voie dépendante de la protéine adaptatrice MyD88 et d’induire la production d’une grande quantité d’IFN-α nécessaire pour inhiber la réplication du VIH-1 dans les PBMCs (Peripheral blood mononuclear cell) en culture. Le rôle des agonistes de TLRs comme adjuvant ont été montré dans le vaccin thérapeutique dirigé contre le VIH-1 chez la souris [116, 117] et chez les primates non humains [118, 119]. Ces expériences ont montré que les agonistes de TLRs étaient capables d’augmenter la réponse immunitaire anti-VIH-1 tant humorale que cellulaire induite suite à l’inoculation du vaccin.
Les agonistes des TLRs ou autres molécules mimant l’ARN génomique du VIH-1, constituent des outils thérapeutiques contre cette pandémie. Leur combinaison avec le vaccin thérapeutique peut s’avérer une approche intéressante capable d’inhiber la réplication virale, tout comme notre compréhension des mécanismes impliqués dans leurs régulations. Cette introduction au chapitre 2 est une excellente initiative. À ce sens, la lectine de type C, DCIR a été décrite comme un inhibiteur de l’activation des TLRs endosomaux TLR7/8 et TLR9 [120, 121]. Quels rôles jouent les lectines de type C dans la réponse immunitaire et l’infection au VIH-1 ? Ce sujet sera abordé dans le chapitre 2 de ce mémoire.
19
1.2. Les lectines de type C et le VIH-1
1.2.1. Les lectines
Les lectines sont des protéines ou glycoprotéines capables de reconnaitre et de se lier de manière spécifique et réversible aux saccharides, oligosaccharides et aux glycoprotéines. Les lectines sont présentes chez les animaux et sur les microorganismes notamment les bactéries et les virus. La plupart des lectines possèdent un domaine commun de reconnaissance de sucre (CRD : Carbohydrate Recognition Domain). La première lectine, extraite des graines de ricin (Ricinus communis), découverte en 1888 par Herman Silltmark était de nature végétale. Cette lectine avait la capacité de reconnaitre les sucres présents à la surface des érythrocytes et de provoquer leur agglutination [122].
1.2.2. Classification des lectines
Plusieurs critères existent pour classifier les lectines. Cette classification peut se faire en fonction de leurs origines (animale, virale, bactérienne ou végétale), de la séquence en acides aminés du domaine CRD de reconnaissance des glucides [123], de la spécificité vis-à-vis des saccharides ou en fonction de leurs structures moléculaires. Il existe plusieurs structures tridimensionnelles des lectines, regroupées en familles dont certaines sont d’origine animale.
Parmi les lectines animales, on trouve les calnexines, les lectines de type M, type C, type F, type I (Siglecs), type L, type P, type R, Lectines box F, galectines, lectines chitinase like, ficolines et intelectines. Certaines sont cytosoliques et jouent un rôle dans le repliement, le tri, la maturation et le trafic des protéines. Dans ce groupe, on retrouve notamment les calnexines, les galectines, les lectines de types M, P et L. D’autres par contre, sont extracellulaires telles que les lectines de type C et de type R, les siglecs et les galectines et sont impliquées dans la reconnaissance des motifs conservés des pathogènes et dans la signalisation et l’adhésion cellulaire [124, 125].
Dans ce projet de maitrise, les lectines de type C et particulièrement le DCIR a retenu notre attention et constitue l’objet de notre étude.
20
1.2.3. Les lectines de type C
1.2.3.1 Structure et classification des lectines de type C
Les lectines de type C constituent un groupe hétérogène de protéines transmembranaires caractérisées par un ou plusieurs domaines de reconnaissance des hydrates de carbone, CRD [126, 127]. Ce terme, lectine de type C a été désigné pour les différencier des autres lectines animales ne possédant pas le domaine CRD et dont la liaison avec le sucre ne dépendait pas du calcium [128]. Il est à noter par ailleurs que toute lectine de type C ne lie pas exclusivement le sucre. Ainsi, il existe des lectines de type C, possédant le domaine CRD, mais qui sont capables de lier d’autres ligands que le sucre, notamment les protéines, les lipides ou des substances non organiques tel que le venin du serpent [129]. D’autres encore peuvent lier le sucre, mais indépendamment du calcium. Ces lectines sont désignées lectine de type C like [126, 129].
Le domaine CRD des lectines de type C est doté d’une structure en forme de double boucle et possède 4 sites de liaison du calcium, dont deux sont requis pour la reconnaissance et la liaison des hydrates de carbone. Ces deux sites de liaison en l’occurrence le motif EPN (Gln-Pro-Asn), spécifique au mannose et au fucose et le motif QPD (Gln-Pro-Asp), spécifique au galactose et au glycane contenant le N-acetylgalactosamine [128, 130, 131], sont composés chacun d’un triplé d’acides aminés dont l’un est un résidu cystéine-proline qui sépare les chaines carbonyles latérales. Chez le DCIR, le résidu aspargine est remplacé par la sérine, ce qui résulte au motif EPS (Glutamate-Proline-Sérine) [132]. La liaison du domaine CRD/Ca2+ et
le monosaccharide forment un complexe dont les deux chaines carbonyles assurent la coordination de la liaison du CRD au calcium et la formation des ponts d’hydrogène en liaison avec le monosaccharide. Ces deux liaisons permettent de stabiliser le complexe et de déterminer la spécificité de la liaison du domaine de lectine de type C au sucre [133]. Les lectines de type C sont codées par un complexe de gènes des cellules NK (NKC : Natural
Killer Complex) localisé sur le chromosome 6F3 chez la souris et sur le chromosome 12p13
chez l’humain [134].
Les lectines de type C sont exprimées dans plusieurs types cellulaires, mais dans le cadre de ces travaux, nous allons plus nous concentrer sur les lectines membranaires de type C, particulièrement celles qui sont exprimées sur les cellules de la lignée myéloïde.
21
1.2.3.2. Lectines de type C des cellules myéloïdes
Selon le groupe de Figdor, on peut classer les récepteurs lectines de type C exprimées sur les cellules myéloïdes, particulièrement sur les cellules dendritiques et sur les cellules de langherans en deux grands groupes en fonction du nombre de domaines CRD présent dans la portion extracellulaire. Le premier groupe est celui des lectines de type C de type I comportant plusieurs domaines CRD. On retrouve dans ce groupe, les lectines comme le MMR (Macrophage mannose receptor) et le DEC 205 (Dendritic and epithelial cell 205 kDa). Le deuxième groupe par contre est composé des lectines de type C de type II possédant un seul domaine CRD [135].
Figure 5 : classification et structure des lectines de type C de type I et type II exprimées sur les cellules de la lignée myéloïde ( Adapté de [135]).
En nous référant à certains travaux, notamment ceux de Huysamen [136, 137], ceux de Susanne Sattler [138] et ceux de Bernhard Kerscher [139], on peut classer les lectines de type C de type II en deux grands blocs en fonction de l’organisation du domaine CRD et du motif de signalisation cytoplasmique: le groupe de dectine-1 et de dectine-2. Les 2 groupes possèdent dans leur portion extracellulaire, un seul domaine CRD, un domaine neck et une région transmembranaire. Dans leur portion cytoplasmique, on trouve un motif impliqué dans la transduction du signal qui peut être soit un ITAM (Immunoreceptor tyrosine-based activation
22
motif), soit un ITIM (Immunoreceptor tyrosine-based inhibition motif) ou un domaine riche en
proline ou encore un motif di-leucine [140]. Le groupe de dectine-1 comprend le MICL (Myeloid
Inhibitory C-type lectin-like receptor) ou CLEC12A, CLEC12B, CLEC9A (C-type lectin domain family-12A,12B, 9A), CLEC-1, CLEC-2 (C-type lectin family-1, 2), Dectin-1(Dendritic cell-associated C-type lectin-1) ou CLEC7A et LOX-1 (Lectin-like oxydized low-density lipoprotein receptor-1). Le groupe de dectin-2 est composé de BDCA (Blood dendritic cell antigen-2),
DCAR (Dendritic cell immuno-activating receptor), DCIR (Dendritic cell immunoreceptor), Dectin-2 (Dendritic cell-associated type lectin-2), CLECSF8 (C-type lectin superfamily 8) et Mincle (Macrophage-inducible C-type lectin) [136]. Les deux groupes présentent des grandes ressemblances dans leurs portions extracellulaires, ils se différencient cependant par la taille de leur queue cytoplasmique. En effet, le dectin-1 possède un domaine cytoplasmique long et suffisant pour induire directement une signalisation intracytoplasmique. Le groupe de dectin-2 par contre, contient une queue cytoplasmique courte et ne possède donc pas un domaine de signalisation dans leur portion cytoplasmique à l’exception de DCIR dont le motif cytoplasmique est long comme celui du groupe de dectin-1 [139]. Pour les autres membres, l’induction du signal intracytoplasmique nécessite au préalable l’association du récepteur avec une molécule adaptatrice comme le récepteur FcRγ [139] ou avec un partenaire de signalisation comme DAP12 (DNAX-activating protein of 12 kDa) [134]. Cette association se fait au niveau de la région transmembranaire grâce à une interaction entre un résidu chargé positivement dans la dectin-2 et un autre résidu chargé négativement dans la chaine gamma du FcR [139, 141].
Les lectines de type C jouent un rôle majeur dans le système immunitaire. En effet, à l’instar des TLRs, elles se comportent comme des PRR et sont capables de reconnaitre et de lier leurs ligands endogènes et exogènes, incluant les pathogènes comme les champignons, les bactéries et les virus et de participer à l’induction de l’immunité innée et adaptative, mais aussi à l’homéostasie du système immunitaire [142, 143]. Après activation, les lectines contenant le motif ITIM recrutent les tyrosines phosphatases SHP-1 et SHP-2 et sont capables d’inhiber certaines fonctions cellulaires [120, 121]. La phosphorylation des tyrosines du motif ITAM conduit généralement à une activation de la cellule. Elle peut cependant dans d’autres cas induire un signal négatif et inhiber l’activation de la cellule, tout comme un motif ITIM peut induire un signal positif [144, 145].
23
Dans les cellules myéloïdes, certaines lectines comme dectin-1, dectin-2 et MINCLE avec un motif ITAM sont phosphorylées par la tyrosine Src kinase à travers les tyrosines présentes dans leur motif de signalisation. Les lectines activées procèdent au recrutement et à l’activation de la kinase Syk qui à son tour active la PLCγ2 (Phospholipase C gamma 2). Il s’en suit une cascade d’activation des protéines impliquant le CARD9 (Caspase Recruitment
Domain family, member 9), le BCL10 (B Cell Lymphoma10) et le MALT1 (Mucosa-Associated lymphoid tissue lymphoma Translocation gene1) [146, 147] qui sont requis pour l’activation
des facteurs de transcription NF-kB et de la voie de MAPKinase [147]. Ceci se solde par l’activation des fonctions cellulaires. D’autres lectines, comme le BDCA-2 exprimé sur la cellule dendritique plasmacytoïde, bien que possédant un motif ITAM, sa phosphorylation par la Src kinase entraine l’inhibition de l’activation de la cellule et de la production des cytokines pro-inflammatoires notamment l’IFN-α en réponse au TLR9 [148, 149]. Cette lectine a été identifiée comme un récepteur reconnaissant et liant le mannose présent sur la gp120 du VIH-1 [VIH-150] et peut donc moduler les sentiers de signalisation induite par le TLR9 et jouer un rôle dans la pathogenèse du VIH-1.
Figure 6 : Model d’un sentier de signalisation induite suite à l’activation des lectines de type C dectine-1, dectine-2 et Mincle dans la cellule myéloïde (Adaptée de [147])
24
1.2.3.3. Les lectines de type C de type II capable de lier le VIH-1
Dans ce sous-titre, nous parlerons des lectines de type C type II, dectine 1 ou dectine 2 pouvant jouer un rôle dans l’endocytose, la phagocytose, la présentation antigénique, ainsi que celles ayant le potentiel de reconnaitre et lier le VIH-1. Il s’agit notamment de DC-SIGN, la langerin, le BDCA-2 et le DCIR. Le DCIR constitue le point focal de notre projet et sera par conséquent traité dans un sous-titre particulier.
1.2.3.3.1. La langerin (CD207)
Ce récepteur est exprimé à la surface des cellules de langerhans. Il possède dans sa partie extracellulaire un seul domaine CRD et dans sa partie intracellulaire, un domaine riche en proline. La langerin peut lier le mannose ou le galactose de manière dépendante du Ca+2,
cependant sa liaison avec le glycosaminoglycan serait indépendante de l’ion calcium [151]. La cellule de langerhans est caractérisée par les granules de birbeck qui sont constitutivement associées à la langerin dans le cytoplasme [152]. Le VIH-1 est lié à travers le mannose ou le galactose présent sur la gp120 par la langerin, qui internalise et dégrade directement dans les granules de birbeck le virus sans présentation antigénique. L’endocytose du VIH-1 provoquée par la langerin est dépendante de la cavéolin-1 et prévient non seulement l’infection à VIH-1 mais, aussi le transfert du virus aux LTCD4. La langerin constitue donc une sorte de barrière naturelle au niveau de la muqueuse contre la transmission et la propagation du VIH-1 [153].
1.2.3.3.2. Le DC-SIGN (CD209)
Le DC-SIGN (Dendritic Cell Specific Intercellular adhesion molecule-3-Grabbing Non-integrin) est une lectine de type C avec une région extracellulaire composée d’un domaine CRD en C-terminal avec un motif EPN de reconnaissance des résidus de mannose ou des résidus fucosylés incluant les antigènes Lewis [154, 155], un domaine neck avec 7 motifs complets et un motif incomplet répétés en tandem de 23 résidus amino-acides et un domaine transmembranaire. Il contient dans sa région intracellulaire une queue cytoplasmique en N-terminal contenant un motif di-leucine (LL), un motif à base de résidu tyrosine (YSKL) et un groupe tri-acidique (EEE), nécessaire pour la transduction du signal intracellulaire. Le domaine neck est nécessaire pour l’oligomérisation permettant ainsi au récepteur d’augmenter son
![Figure 3 : cycle de réplication du VIH-1 (Adaptée de [8])](https://thumb-eu.123doks.com/thumbv2/123doknet/6383655.168920/16.918.231.796.276.612/figure-cycle-réplication-vih-adaptée.webp)
![Figure 4 : Transmission du VIH-1 dans les muqueuses (Adapté de [85])](https://thumb-eu.123doks.com/thumbv2/123doknet/6383655.168920/26.918.255.706.198.814/figure-transmission-du-vih-dans-les-muqueuses-adapté.webp)
![Figure 5 : classification et structure des lectines de type C de type I et type II exprimées sur les cellules de la lignée myéloïde ( Adapté de [135])](https://thumb-eu.123doks.com/thumbv2/123doknet/6383655.168920/33.918.250.632.409.733/figure-classification-structure-lectines-exprimées-cellules-myéloïde-adapté.webp)
![Figure 6 : Model d’un sentier de signalisation induite suite à l’activation des lectines de type C dectine-1, dectine-2 et Mincle dans la cellule myéloïde (Adaptée de [147])](https://thumb-eu.123doks.com/thumbv2/123doknet/6383655.168920/35.918.171.701.598.957/signalisation-activation-lectines-dectine-mincle-cellule-myéloïde-adaptée.webp)
![Figure 7 : Les quatre formes de l’ARNm épissé de DCIR (Adapté de [140])](https://thumb-eu.123doks.com/thumbv2/123doknet/6383655.168920/40.918.246.756.656.1015/figure-les-quatre-formes-arnm-épissé-dcir-adapté.webp)
![Figure 8 : Voies de signalisation induite par le DCIR (Adaptée de [209])](https://thumb-eu.123doks.com/thumbv2/123doknet/6383655.168920/45.918.180.664.108.590/figure-voies-signalisation-induite-dcir-adaptée.webp)
![Figure 9 : Rôle du motif ITIM dans la capture, internalisation et signalisation médiées par l’engagement de DCIR (Adaptée de [209])](https://thumb-eu.123doks.com/thumbv2/123doknet/6383655.168920/48.918.172.771.105.534/figure-rôle-capture-internalisation-signalisation-médiées-engagement-adaptée.webp)
![Figure 11: Inhibiteurs de la portion extracellulaire de DCIR obtenus après criblage virtuel [228]](https://thumb-eu.123doks.com/thumbv2/123doknet/6383655.168920/51.918.180.746.105.534/figure-inhibiteurs-portion-extracellulaire-dcir-obtenus-criblage-virtuel.webp)