Étude du microbiome intestinal de Choristoneura
fumiferana, la tordeuse des bourgeons d’épinette
Mémoire
Mathieu Landry
Maîtrise en Microbiologie-Immunologie
Maître ès sciences (M. Sc.)
Résumé
La tordeuse des bourgeons d’épinette (Choristoneura fumiferana) est l'un des insectes ravageurs les plus destructeurs au Canada. La principale méthode de contrôle de cet insecte est l’insecticide à base de bactéries Bacillus thurigiensis (BT). La communauté bactérienne de l'intestin de la tordeuse des bourgeons de l'épinette pourrait jouer un rôle dans l'action insecticide de BT, en plus de son rôle potentiel dans le métabolisme de matière ligneuse. Cette étude visait à obtenir un premier aperçu des communautés bactériennes de l'intestin de C. fumiferana et de mesurer l'effet des changements de régime alimentaire sur la composition de ces communautés. Un groupe de larves a été élevé dans le laboratoire sur diète synthétique McMorran, tandis que les deux autres groupes ont été recueillis sur le terrain sur le sapin baumier et l'épinette noire. L'intestin moyen de larves a été extrait, broyé et l'ADN en a été extrait. La région V6-V8 des petites sous-unités ribosomiques 16S bactériennes a été amplifiée puis séquencée avec 454 GS FLX-titanium. Les séquences obtenues ont été traitées, regroupées et classées avec le logiciel mothur. Nous avons trouvé que le microbiote intestinal de la tordeuse était dominé par des Proteobactéries, principalement du genre Pseudomonas. En outre, le microbiote des larves élevées sur l'alimentation synthétique était beaucoup plus riche en termes de diversité taxonomique que pour les larves recueillies sur le terrain, et beaucoup plus uniforme entre chaque échantillon. Nous en avons donc conclu que différentes méthodes d’élevage favorisaient différentes communautés microbiennes dans l’intestin de C. fumiferana. Il reste à déterminer si cette différence est dûe au génotype de l’insecte, son alimentation ou son environnement, et quel impact ont ces différences sur les capacités métaboliques de l’insecte.
Table des matières
Résumé ... iii
Table des matières ... v
Liste des tableaux et figures ... vii
Remerciements ... ix
Avant-propos ... xi
1. Introduction ... 1
1.1. Le microbiome ... 2
1.1.1. Le microbiome humain ... 3
1.1.1.1. Le microbiome de l’intestin humain ... 5
1.1.2. Les microbiomes intestinaux d’insectes ... 6
1.1.2.1. Microbiome spécifique à chaque groupe ... 6
1.1.2.2. Fonctions du microbiome chez l’insecte ... 7
1.1.2.3. Le microbiome chez les lépidoptères ... 8
1.1.2.4. Le microbiome de C. fumiferana ... 8
1.2. La tordeuse des bourgeons de l’épinette ... 9
1.2.1. Description de l’insecte ... 9
1.2.1.1. Répercussions de C. fumiferana sur les forêts ... 11
1.2.1.2 Cycle saisonnier de C. fumiferana ... 13
1.2.1.3. Aire de répartition de C. fumiferana ... 14
1.2.2. Hôtes et alimentation ... 15
1.2.2.1. Le sapin baumier ... 15
1.2.2.2. L’épinette blanche ... 15
1.2.2.3. L’épinette noire ... 15
1.2.2.4. La composition chimique et nutritive des aiguilles de conifère ... 17
1.2.3. Régulation des populations de C. fumiferana ... 18
1.2.3.1. Agents pathogènes, prédateurs et climat ... 18
1.2.3.2. Lutte contre C. fumiferana ... 19
1.3.1.2. Culture des microorganismes ... 23
1.3.1.3. Séquençage nouvelle génération ... 24
1.3.1.4. Séquençage d’amplicons 16S ... 26
1.3.2. Statistiques et mesures reliées à l’étude du microbiome ... 29
1.3.2.1 Mesures de diversité ... 29
1.3.2.2. Arbres phylogénétiques ... 30
1.3.2.3. Mesure Unifrac ... 31
1.3.2.4. La distance Bray-Curtis... 32
1.4. Buts et motifs du présent travail de recherche ... 33
2. La composition des communautés bactériennes du mésentéron de Choristoneura fumiferana en fonction de son alimentation. ... 35
2.1. Résumé ... 35
Composition of the spruce budworm (Choristoneura fumiferana) midgut microbiota as affected by rearing conditions ... 36
ABSTRACT ... 37
INTRODUCTION ... 37
MATERIALS AND METHODS ... 38
Experimental insects ... 38
Midgut dissections and DNA extraction ... 39
Choice of primers for Roche 454 pyrosequencing ... 39
Pyrosequencing of the 16S rRNA gene ... 39
Sequence pre-processing ... 39
Data analysis ... 40
Statistical analysis ... 40
RESULTS ... 40
Variation in richness among microbiotas (α-diversity) ... 40
Overall composition of spruce budworm midgut microbiota ... 42
Compositional variation among microbiotas (β-diversity)... 43
DISCUSSION ... 47
ACKNOWLEDGMENTS ... 49
REFERENCES... 49
3. Conclusion ... 53
Liste des tableaux et figures
Tableau 1.1 – Associations entre problèmes de santé humaine et taxons bactériens d’après (Cho et Blaser, 2012) ... 4 Figure 1.1 – Larve de 6e stade de C. fumiferana (Ressources naturelles Canada, Service canadien des forêts) ... 10 Figure 1.2 – Adulte de C. fumiferana (Ressources naturelles Canada, Service canadien des forêts) ... 10 Figure 1.3 - Nature cyclique des épidémies (Blais 1983) – Les dommages causés par C. fumiferana varient grandement dans le temps. Les périodes de ravages importants, appelées épidémies, sont causées par plusieurs facteurs contribuant à la prolifération de la tordeuse et arrêtées par d’autres facteurs, dont
l’épuisement de la nourriture, ce qui amène la phase endémique. ... 12 Figure 1.4 Cycle saisonnier de C. fumiferana. Source : Michel Cusson, Service Canadien des Forêts. ... 12 Figure 1.5 - Aire de répartition de C. fumiferana au Canada, en Alaska et dans le nord-est des États-Unis (Lumley et Sperling, 2011) ... 14 Figure 1.6 - Déplacement des populations de tordeuse vers le nord (Régnière 2012) – Carte réprésentant les zones les plus touchées par C. fumiferana au 20e siècle (zone délimitée par les lignes noires). Les lignes bleues délimitent la zone touchée par C. fumiferana au 19ème siècle et les lignes rouges délimitent la zone qui
sera vraisemblablement touchée au 21ème siècle. ... 14
Figure 1.7 - Aire de répartition du sapin baumier au Canada et au nord des États-Unis (Ressources naturelles Canada, Service canadien des forêts) ... 16 Figure 1.8– Aire de répartition de l’épinette blanche au Canada, en Alaska et au nord des États-Unis
(Ressources naturelles Canada, Service canadien des forêts) ... 16 Figure 1.9 – Aire de répartition de l’épinette noire au Canada, en Alaska et au nord-est des États-Unis
(Ressources naturelles Canada, Service canadien des forêts) ... 17 Figure 1.10 – Arbre de la vie de Woese démontrant la séparation en phylums des organismes vivants et la distance génétique qui les sépare de l’ancêtre commun. Figure inspirée de (Woese et al., 1990) ... 22 Figure 1.11- Taux de variabilité selon la position de la base dans la séquence du gène de l’ARN 16S de Pseudomonas (d’après (Bodilis et al. 2012). Les pics identifiés V1 à V9 désignent les régions variables pouvant être séquencées à des fins d’identification taxonomique. Le taux de variabilité en axe des Y est mesuré en prenant en compte la fréquence moyenne dans une fenêtre de 50 bases de part et d’autre de chaque position sur l’axe des X. ... 22 Tableau 1.2 - Comparaison de quelques méthodes de séquençage, permettant de déterminer quelle méthode est la plus appropriée pour divers expériences. Source : Brian Boyle, coordonateur, plateforme d'analyse génomiques, IBIS, U. Laval ... 25 Figure 1.12- Amplification des fragments lors du séquençage 454 de Roche (Metzker 2010). Les billes, chacune dans leur microréacteur recouvert d’huile, sont soumises à une amplification PCR, recouvrant chaque bille de fragments d’ADN identiques. ... 25 Figure 1.13 – Amorces utilisées pour l’étude de biodiversité à partir du 16S bactérien. En bleu, séquences complémentaires avec des régions conservées du 16S bactérien. En rouge, séquences d’identification nécessaires au multiplexage. En vert, adaptateurs complémentaires aux amorces se trouvant sur les billes. En
Figure 1.15 – Exemple de résultat Unifrac (Lozupone et Knight 2005). La valeur D est la valeur Unifrac, qui mesure à quel point chaque communauté est spécifique à son environnement. Les boîtes rouges représentent des séquences provenant de l’environnement 1 alors que les boîtes bleues représentent les séquences provenant de l’environnement 2. Ce qui est important est la distinction entre les branches rouges ou bleus (uniques à un seul environnement et les branches violettes (communes aux deux environnements) ... 31 Figure 2.1 - OTU rarefaction curves for each experimental group. Each group represents the aggregate of 12 individuals at 2 460 reads each (total 29 520 reads per group). Note that the balsam fir and black spruce curves have overlapping 95% confidence intervals (i.e.: not significantly different). ... 41 Figure 2.2 - Taxonomic distribution of reads at the phylum level for each experimental group. ... 43 Figure 2.3 - Taxonomic distribution of reads in the Proteobacteria for each experimental group. Only genera comprising more than ~1% of the phylum are shown. ... 43 Figure 2.4 - Individual and group diversity patterns. Dendrogram of the Bray-Curtis distances between each larva (left) and Sørensen similarities (right) between or within the three groups. Note that the individuals within the two subclades of each fir and spruce group have high similarities (92-98% in parentheses) compared to each group taken as a whole (62 and 67%). ... 44 Table 2.1 List of top 10 OTUs within each experimental group, along with the names of the species they most likely represent (from BLAST similarity)... 44 Table 2.2 Sequences of primers used to amplify the 16S V6-V8 region for pyrosequencing (from Comeau et al. 2011). The dots are used to separate the different parts of the primers (Roche adaptor • multiplex identifier (barcode) • specific primer). Multiplex identifiers are only present in the forward primers, hence there is only one unique, shorter reverse primer. ... 46
Remerciements
Ces travaux de maîtrise n’auraient jamais vu le jour sans le support grandement apprécié de plusieurs personnes.
Tout d’abord, je remercie chaleureusement mon directeur de recherche Roger Levesque, sans qui rien de tout cela n’aurait été possible. Il m’a donné la chance de découvrir ce que c’était les études graduées. Son grand enthousiasme, sa constante disponibilité et son ouverture d’esprit ont rendu ce projet un véritable plaisir à accomplir. Je me considère chanceux d’être tombé sur un directeur d’une telle qualité.
Je tiens aussi à remercier Michel Cusson, qui a suivi ma formation scientifique depuis ma première année de baccalauréat. Il a su me communiquer son amour pour la science, et c’est, entre autres, ce qui m’a poussé vers les études graduées. Depuis qu’il m’a accueilli dans son laboratoire il y a de cela cinq ans, j’ai été impressionné par son immense compréhension et sa patience pour bien former ses étudiants.
Plusieurs autres personnes ont joué un rôle dans l’élaboration de ce projet. Nicolas Derome, qui a su guider le projet lorsqu’il était question d’écologie microbienne. Irena Kukavica-Ibrulj, qui m’a si souvent aidé en laboratoire et qui me supportait constamment dans mon projet. Bryan Boyle et son équipe, pour leur travail et leurs conseils techniques au niveau du séquençage. André Comeau, qui a eu la patience de m’enseigner comment utiliser le logiciel mothur et m’a accompagné dans mes premiers pas en bioinformatique. Catherine Béliveau m’a beaucoup aidé au niveau des techniques de PCR et de la conception d’amorces PCR. Deepa Pureswaran, qui a collecté une grande partie de mes échantillons sur le terrain. Finalement, Clothilde Bourgeois, qui m’a encouragé et supporté tout au long de ce projet. Un chaleureux merci à tous !
En dernier lieu, je tiens à remercier le CRSNG pour le financement de mes travaux dans le laboratoire de Roger Levesque et le Marine Biology Laboratory de Woodshole pour la bourse me permettant d’assister à la formation S.T.A.M.P.S.
Avant-propos
Les sections Matériel & Méthodes et Résultats de ce mémoire sont présentées sous forme de manuscrit d’article scientifique, au chapitre 2. Ce manuscrit, intitulé « La composition des communautés bactériennes du mésentéron de Choristoneura fumiferana » sera soumis très bientôt à un journal scientifique. La conclusion générale des expériences traitées dans le manuscrit terminera ce mémoire. L’article aura comme co-auteurs André M. Comeau, Nicolas Derome, Michel Cusson et Roger C. Levesque. Ma contribution à l’article s’est faite au niveau des manipulations, de l’analyse et de la rédaction. J’ai élevé les larves en laboratoire, les ai disséquées et en ai extrait l’ADN. Les larves ont été collectées sur le terrain par Deepa Pureswaran, une collaboratrice de Michel Cusson. Le séquençage a été fait par l’équipe de Bryan Boyle à la plate-forme de séquençage de l’IBIS. J’ai fait l’analyse bio-informatique sous la tutelle d’André Comeau. Tout ce travail a été fait sous la supervision de Roger Levesque et Michel Cusson, qui ont révisé l’article et le mémoire.
1. Introduction
Chaque année, au Canada, les ravageurs forestiers sont responsables de pertes considérables pour l’industrie forestière. Ces pertes se mesurent en termes de réduction de volume de bois récoltable et, par conséquent, peuvent représenter des diminutions importantes de revenus. La récente épidémie du dendroctone du pin ponderosa, sur la côte ouest, nous en fournit d’ailleurs un exemple très probant (Taylor et al. 2006).
La lutte contre ces ravageurs s’avère parfois difficile, et les scientifiques sont constamment à l’affût de stratégies plus efficaces et/ou plus respectueuses de l’environnement pour réduire les pertes engendrées par les insectes nuisibles. L’épandage d’insecticides de synthèse conventionnels n’étant plus préconisé en milieu forestier au Canada, les chercheurs tentent maintenant de mettre au point des outils de lutte biologique ainsi que des approches visant à perturber, de façon spécifique, la physiologie de l’insecte dont on espère réduire les dégâts. Par exemple, plusieurs initiatives de recherche actuelles visent le séquençage de génomes d’insectes ravageurs en vue d’identifier des gènes qui constituent des cibles prometteuses pour le développement de nouveaux produits antiparasitaires. La flore intestinale des insectes pourrait, elle aussi, s’avérer une cible intéressante pour la mise au point de nouveaux outils de lutte, étant donné l’impact important que ces micro-organismes sont soupçonnés avoir sur la biologie de leurs hôtes.
C’est dans ce contexte qu’a pris forme le présent projet de maîtrise. Ainsi, les travaux qui font l’objet de ce mémoire visaient la caractérisation du microbiote intestinal de la tordeuse des bourgeons de l’épinette, (Choristoneura fumiferana, un des plus importants ravageurs des forêts conifériennes de l’Amérique du Nord. Dans les lignes qui suivent, je me penche d’abord sur la définition et le concept de « microbiome » et je résume les connaissances actuelles sur les microbiomes d’insectes, en particulier ceux des Lépidoptères (groupe auquel appartient C. fumiferana). La deuxième section porte sur le ravageur ciblé par la présente étude, C. fumiferana: sa biologie, les dégâts qu’elle cause et les outils disponibles pour réduire son impact. Je traite subséquemment des différentes approches disponibles pour la caractérisation des microbiomes, puis je conclus en présentant les objectifs spécifiques à la présente étude.
1.1. Le microbiome
Les termes « microbiome » et « microbiote » définissent deux concepts très proches et sont souvent utilisés de façon interchangeable pour désigner l’ensemble des populations microbiennes habitant une niche écologique donnée. Par contre, le concept de microbiome désigne les tissus hôtes et les populations microbiennes alors que le concept de microbiote ne prend en compte que les populations microbiennes elles-mêmes (Whiteside et al. 2015).Dans le présent mémoire, le terme microbiome est utilisé dans la revue de littérature car c’est de ce concept que traitent la majorité des sources utilisées. Le terme de microbiote est utilisé pour l’expérience traitée dans ce mémoire, qui ne concerne pas vraiment les tissus hôtes des populations microbiennes étudiées. La niche occupée par le microbiote peut être un élément de l’environnement, comme un lac, ou faire partie d’un autre organisme, comme l’intestin d’un insecte ou le poumon d’un humain. Finalement, il est important de considérer que les populations désignées par ces termes disposent de leur propres génomes et capacités métaboliques, dont l’ensemble est appelé métagénome (Whiteside et al. 2015).
Le microbiome peut rendre de nombreux services à l’hôte. Dans l’intestin humain, par exemple, les microorganismes résidents ont un génome collectif de plus de ~250 000 gènes fonctionnels, constituant une importante boîte à outils, par rapport aux ~20 000 gènes humains fonctionnels (Gill et al. 2006). Ces populations occupent un espace et consomment des nutriments qui, autrement, pourraient être utilisés par des pathogènes plus transitoires. Par contre, si l’équilibre de ces populations est perturbé, le microbiome peut entrer en dysbiose, qui est un débalancement microbien. Ce phénomène peut favoriser l’émergence de pathogènes parmi ses populations et la perte de précieuses fonctions apportées à l’hôte.
1.1.1. Le microbiome humain
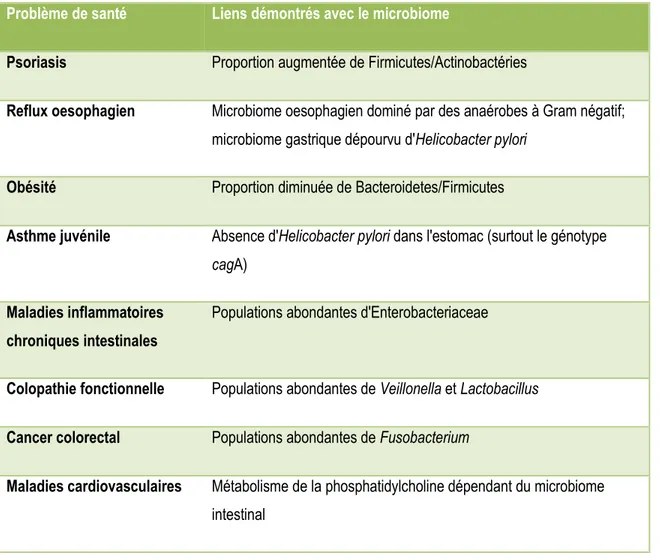
Avec l’avènement des technologies de séquençage haut-débit, l’intérêt pour les microorganismes habitant le corps humain s’est grandement développé. Le Human Microbiome Project, financé par le National Health Institute (NIH), a généré plus de 2.3 téraoctets de données sous la forme de 35 milliards de séquences d’amplicons ribosomiques 16S, couvrant 690 échantillons provenant de 300 Américains (Cho et Blaser 2012). D’autres projets visent à assembler le métagénome des populations microbiennes de l’humain et à comparer cet assemblage avec le génome humain pour connaître la contribution fonctionnelle de ces populations. Ces travaux ont pour but d’évaluer l’existence d’associations entre les maladies humaines et l’identité et la diversité des microorganismes habitant l’humain. Déjà, on a noté une association entre plusieurs maladies et des taxons bactériens particuliers (Tableau 1).
L’humain hérite d’une partie de la composition du microbiome de sa mère, influencé à la fois par l’accouchement, l’allaitement et le contact direct après l’accouchement. Une perturbation comme la prise d’antibiotique ou un changement de diète radical peut donc non seulement influencer le microbiome de la mère mais aussi celui de sa progéniture (Cho et Blaser 2012).
L’étude du microbiome est aussi très intéressante dans le cas de maladies liées à une immunodéficience partielle ou complète. Des bactéries jusqu’alors inoffensives prolifèrent hors de tout contrôle et mettent en danger la santé humaine. Un exemple intéressant est la fibrose kystique, causée par un défaut génétique rendant extrêmement difficile l’évacuation du mucus des poumons. Ceci crée un environnement idéal pour des taxons bactériens comme Pseudomonas qui utilisent ce mucus pour se protéger du système immunitaire. Une communauté microbienne importante se développe alors dans les poumons et forme un biofilm réduisant grandement l’efficacité des traitements antibiotiques (Fodor et al. 2012). Un exemple similaire est fourni par le développement du SIDA, au cours duquel l’épithélium du tractus intestinal est endommagé. Ceci permet aux bactéries du microbiome intestinal de se déplacer vers les tissus avoisinants et de mettre le système immunitaire en état d’alerte constant. Cette réaction immunitaire importante va contribuer grandement à l’épuisement du système immunitaire et mener à l’immunodéficience typique du SIDA (Cunningham-Rundles et al. 2011).
Tableau 1.1 – Associations entre problèmes de santé humaine et taxons bactériens d’après (Cho et Blaser, 2012) Problème de santé Liens démontrés avec le microbiome
Psoriasis Proportion augmentée de Firmicutes/Actinobactéries
Reflux oesophagien Microbiome oesophagien dominé par des anaérobes à Gram négatif; microbiome gastrique dépourvu d'Helicobacter pylori
Obésité Proportion diminuée de Bacteroidetes/Firmicutes
Asthme juvénile Absence d'Helicobacter pylori dans l'estomac (surtout le génotype cagA)
Maladies inflammatoires chroniques intestinales
Populations abondantes d'Enterobacteriaceae
Colopathie fonctionnelle Populations abondantes de Veillonella et Lactobacillus
Cancer colorectal Populations abondantes de Fusobacterium
Maladies cardiovasculaires Métabolisme de la phosphatidylcholine dépendant du microbiome intestinal
1.1.1.1. Le microbiome de l’intestin humain
Pour diverses raisons, l’intestin est l’organe dont le microbiome a été le mieux étudié chez l’humain. D’abord, c’est l’organe affichant la plus grande abondance bactérienne, contenant dix fois plus de cellules bactériennes que le corps humain ne contient de cellules humaines. C’est aussi un milieu facile à échantillonner, par les selles. Finalement, le microbiome de l’intestin joue un rôle important dans la digestion, mais influence aussi la sévérité de problèmes de santé tels l’obésité, le cancer colorectal et la maladie de Crohn (Kostic et al. 2014). Le facteur le plus significatif reliant la santé humaine et le microbiome intestinal est la stabilité de ce dernier (Backhed et al. 2012). Bien que les effets à long terme causés par les perturbations du microbiome n’aient pas encore été pleinement évalués, les effets immédiats se présentent sous la forme de diarrhées, de colites, de perturbations du métabolisme et de risques augmentés d’infection. Ces perturbations des populations du microbiome peuvent être causées par un changement drastique de diète, un traitement antibiotique ou bien d’autres facteurs extrinsèques.
A ce jour, il s’est avéré impossible d’identifier un microbiome intestinal "coeur" (c’est-à-dire espèces microbiennes communes à tous les individus) chez les humains en santé. Les variations liées à l’âge, la diète, le type d’accouchement, le génotype et le mode de vie sont trop grandes d’un individu à l’autre pour que des taxons communs puissent être identifiés. Par contre, des fonctions métaboliques attribuées au microbiome intestinal sont présentes chez tous les individus en santé (Backhed et al. 2012). Il est donc plus approprié de parler de métagénome microbien "coeur" pour les humains en santé. Ce métagénome se divise en deux groupes de fonctions principales, soit les interactions avec le système immunitaire et la métabolisation de la nourriture. Certains microbes habitant l’intestin jouent un rôle dans la conversion des composés nutritifs ou dans la détoxification de produits nocifs pour l’intestin humain. D’autres microbes interagissent avec le système immunitaire et contribuent à son développement et sa bonne régulation (Cho et Blaser 2012).
1.1.2. Les microbiomes intestinaux d’insectes
Qu’ils soient des producteurs, des ravageurs ou simplement nuisibles, les insectes sont d’une grande importance pour l’homme. Ils peuvent aussi servir de modèle pour l’étude de microbiomes d’organismes plus complexes. L’étude du microbiome des insectes est grandement facilitée par le fait qu’ils se développent rapidement, se reproduisent en grands nombres, sont faciles à élever et leur utilisation n’est pas soumise à des contraintes éthiques. Par contre, en raison de leur petite taille, l’échantillonnage signifie souvent la mort pour l’insecte étudié. Ceci empêche ainsi l’échantillonnage du même insecte à plusieurs points dans le temps. Comme la plupart des organismes vivants possédant un système digestif, les insectes ont eux aussi un microbiome intestinal. L’intestin des insectes est divisé en trois parties (Engel et Moran 2013). L’intestin antérieur (stomodéum) est d’origine ectodermique et peut servir de réservoir de nourriture. L’intestin moyen, ou mésentéron, est l’endroit où se produit en grande partie la digestion, c’est pourquoi l’expérience présente s’attarde particulièrement à cette portion de l’intestin. Il est d’origine endodermique. L’intestin postérieur (proctodéum) joue un rôle dans l’absorption de l’eau et des sels ainsi que dans l’expulsion des excréments et est d’origine ectodermique. L’intestin d’insecte est un milieu très instable pour le maintien d’un microbiome. Ceci est principalement dû au fait que l’intestin subit d’importantes perturbations causées par les changements développementaux que l’insecte traverse (Engel et Moran 2013). Les bactéries formant le microbiome intestinal d’insectes peuvent, comme chez l’humain, jouer un rôle dans la digestion des nutriments et la régulation du système immunitaire (Engel et Moran 2013). Ils joueraient aussi un rôle dans la protection contre les agents pathogènes chez une grande majorité des espèces d’insectes étudiées. Entraîner des perturbations supplémentaires de ce microbiome pourrait avoir des effets néfastes sur la santé de l’insecte, et serait possiblement un moyen de contrôle alternatif contre les insectes considérés comme nuisibles.
1.1.2.1. Microbiome spécifique à chaque groupe
Les insectes forment une des classes du vivant démontrant la plus grande diversité en termes d’espèces. Il n’est donc pas surprenant que les microbiomes d’insectes varient immensément d’un groupe d’insectes à l’autre. Le fait que les habitudes alimentaires varient énormément à l’intérieur de la classe des insectes explique en bonne partie ce phénomène (Engel et Moran 2013). Par exemple, les détritivores comme les termites, criquets et cafards possèdent les microbiomes avec la plus grande charge bactérienne (Cazemier et al. 1997). Les différents microbiomes peuvent aussi être reliés aux divers types spécifiques de système digestifs à chaque groupe d’insectes. Certains insectes possèdent un système digestif doté d’adaptations, permettant le maintien de populations microbiennes contribuant à leur survie. Parmi ces adaptations on compte les replis et les poches dans l’épithélium intestinal, permettant aux microbes de mieux résister aux
perturbations subies par le système digestif, ce qui améliore la stabilité du microbiome intestinal. Une autre adaptation intéressante est observée chez le termite, dont l’intestin est constitué de plusieurs compartiments, chacun contenant des populations microbiennes différentes. Ces compartiments permettent aux bactéries qui les habitent de maintenir les conditions physicochimiques (pH, ions) optimales à leur prolifération (Breznak et Brune 1994).
1.1.2.2. Fonctions du microbiome chez l’insecte
La présence d’un microbiome intestinal peut apporter de multiples bienfaits à l’insecte hôte. La contribution du microbiome à la digestion est le premier avantage qui vient à l’esprit. Le termite est encore une fois un excellent exemple, représentant un des partenariats insecte-microorganisme les mieux connus. L’appareil digestif du termite est formé de plusieurs compartiments contenant cette flore microbienne et jouant le rôle de microréacteurs ayant chacun sa part à jouer dans la digestion. Les principales fonctions métaboliques attribuées aux microorganismes habitant cet insecte sont l’hydrolyse de la cellulose et de l’hémicellulose, la fermentation de produits complexes en acides gras et finalement le recyclage de l’azote intestinal et sa fixation. Ces fonctions font intervenir des protozoaires, des champignons et des bactéries (Breznak et Brune 1994). En ce qui concerne ces dernières, la majorité des espèces de termites possèdent un microbiome dominé par les Protéobacteries, les Spirochètes, les Bacteroidetes et les bactéries à Gram positif à faible contenu en G+C (Kudo et al. 1998). Lors des premières avancées dans la caractérisation des microbiomes d’insectes, on supposait que la plupart des bactéries qui en faisaient partie avaient un rôle à jouer dans la digestion. Or, il s’avère maintenant que plusieurs insectes n’ont peu ou pas besoin des microbes dans leur système digestif pour digérer leur nourriture (Calderón-Cortés et al. 2012). Par contre, diverses autres fonctions ont été attribuées aux bactéries habitant les intestins d’insectes.
Le microbiome fournit une résistance à la colonisation des parasites et agents pathogènes tel que montré chez l’abeille, le moustique et certains criquets (Engel et Moran 2013). Chez la drosophile, le microbiome contribue au développement et au maintien de l’épithélium intestinal (Buchon et al. 2013). Certaines molécules toxiques ingérées avec la diète, des insecticides par exemple, peuvent être métabolisées et inactivées par des bactéries du microbiome, comme chez la punaise (Kikuchi et al. 2012). Finalement, les bactéries du microbiome peuvent fournir à certains insectes des molécules nécessaires à la communication, comme des phéromones (Dillon et al. 2002).
1.1.2.3. Le microbiome chez les lépidoptères
Les Lépidoptères sont un ordre d’insectes se nourrissant principalement de matière végétale. Ni des replis ni des poches n’ont été observés dans le système digestif des Lépidoptères, ce système se présentant sous la forme d’un tube assez simple, comparativement au système digestif d’autres insectes. Le pH à l’intérieur du mésentéron de larves de Lépidoptères se situe autour de 11-12, limitant sévèrement la diversité des bactéries pouvant le coloniser (Harrison 2001). Ce pH très élevé serait dû aux tannins présents dans la diète de la majorité des Lépidoptères (Berenbaum 1980). Le système digestif des insectes de cet ordre ne semble donc pas très bien adapté au maintien d’une communauté bactérienne stable et diverse.
Les études faites sur des larves de papillon démontrent que la majorité des bactéries composant leur microbiome intestinal sont acquises dans la nourriture (Broderick et al. 2004). La nature de la diète a donc un très grand impact sur la composition du microbiome intestinal de ces insectes. Par contre, certains taxons bactériens semblent présents chez tous les individus de la même espèce de papillon, indépendamment de la diète. Chez Lymantria dispar, Enteroccus faecalis domine le microbiome de tous les individus, ce qui pourrait suggérer une transmission verticale de ce microbe (Broderick et al. 2004).
Le rôle du microbiome intestinal chez les Lépidoptères n’a pas encore été élucidé. Il a été proposé que les bactéries du microbiome pouvaient neutraliser certaines molécules toxiques comme le tannin ou certains insecticides (Xia et al. 2013). Le taxon bactérien des Enterobacteriaceae, qui domine le microbiome de plusieurs Lépidoptères, jouerait un rôle dans la digestion des sucres (Anand et al. 2010). De plus, une souche d’Aeromonas, isolée chez Samia cynthia pryeri, possèderait le bagage génétique nécessaire à la digestion de polymères de xylane, qui est un composant important des membranes cellulaires végétales (Narayan Roy et al. 2003). Par contre, d’autres études tendent à démontrer que ces bactéries ne sont pas nécessaires à la digestion chez l’insecte (Calderón-Cortés et al. 2012).
1.1.2.4. Le microbiome de C. fumiferana
La caractérisation complète du microbiome de C. fumiferana, la tordeuse des bourgeons de l’épinette, n’a toujours pas été réalisée. Des expériences en culture réalisée auparavant ont montré une prédominance des espèces Enterococcus mundtii et Staphyloccocus succinus parmi les bactéries cultivables de l’intestin de C. fumiferana. Parmi les taxons moins abondants, on a répertorié Bacillus subtilis et des espèces appartenant aux genres Pseudomonas et Paenibacillus. Ces travaux ont aussi démontré que l’auréomycine présente dans la diète synthétique n’était pas assez efficace pour éliminer les bactéries de
l’intestin (van Frankenhuyzen et al. 2010). Une autre étude concernant le lien diète-microbiome chez C. fumiferana a démontré que les sucs intestinaux d’insectes se nourrissant de sapin baumier inhibaient la croissance bactérienne, contrairement aux sucs intestinaux d’insectes se nourrissant de diète synthétique (Pang 2010). Ainsi, on peut soupçonner que le microbiome de l’intestin de tordeuses se nourrissant de conifères est moins diversifié que si ces dernières se nourrissaient de diète synthétique. Ce milieu intestinal plus hostile à la croissance bactérienne est une explication potentielle pour laquelle l’insecticide Bt est moins efficace en champ qu’en laboratoire (Tabashnik 1994).
1.2. La tordeuse des bourgeons de l’épinette
C. fumiferana est l’un des ravageurs forestiers les mieux connus au Canada. Elle fait partie de la famille des Tortricidae et du complexe d’espèces des tordeuses, lequel compte plus de huit espèces de Choristoneura étroitement apparentées à C. fumiferana (Brown 2005). La larve de l’insecte se nourrit des jeunes pousses de sapin et d’épinette, causant d’importantes pertes en matière ligneuse. Ces pertes sont évaluées à 32 à 43 millions de m3 de bois chaque année dans la forêt boréale canadienne et sont le résultat de perte de
croissance ou de mortalité d’arbres trop affaiblis pour survivre (Fournier et al. 2010). De plus, les gouvernements et les compagnies forestières doivent dépenser des millions en épandage d’insecticide pour minimiser les dégâts causés par ce fléau.
1.2.1. Description de l’insecte
Les œufs sont adossés les uns aux autres sous forme de masses soyeuses aplaties, de couleur vert pomme passant au brun puis au blanc-soie après l'éclosion. La larve est une chenille de couleur brune, tachetée de blanc, ayant une longueur, au sixième (dernier) stade, d’environ 25 mm (Figure 1.1). La chrysalide est de couleur rougeâtre et mesure environ 20 mm. La forme adulte est un papillon de grosseur moyenne, de couleur gris-brun et tacheté de blanc, avec une envergure des ailes d’environ 22 mm (Figure 1.2) (Martineau 1985).
Figure 1.1 – Larve de 6e stade de C. fumiferana (Ressources naturelles Canada, Service canadien des forêts)
Figure 1.2 – Adulte de C. fumiferana (Ressources naturelles Canada, Service canadien des forêts)
1.2.1.1. Répercussions de C. fumiferana sur les forêts
Au stade larvaire, la tordeuse se nourrit d'aiguilles de conifères. Elle a une préférence pour les jeunes pousses et c'est pourquoi les premières cibles sont les bourgeons fraîchement débourrés. Si la population de tordeuses est faible, les bourgeons suffisent pour satisfaire l'appétit des larves et l'arbre ne perd que son feuillage de l’année courante. Par contre, si ceci se produit plusieurs années en succession, le feuillage s'éclaircit progressivement chaque année jusqu'au point où il n’est plus en quantité suffisante pour maintenir l'arbre en vie. Ceci se produit généralement suite à quatre années de défoliation consécutives. Avec des populations de tordeuse importantes, les arbres sont beaucoup plus à risque de mourir, car les chenilles s'attaquent aussi au feuillage des années précédentes. L'arbre peut ainsi être privé de la presque totalité de son feuillage. Même dans les cas où les dommages causés par les chenilles n’entraînent pas la mortalité, les arbres touchés accusent des pertes de croissance. Les arbres ayant survécu se développent avec une forme caractéristique de baïonnette, un des signes les plus évidents d'une invasion de C. fumiferana (McLintock 1955), outre l’abondance d’arbres morts.
Des invasions sévères de C. fumiferana se produisent tous les 30 à 50 ans et couvrent des territoires de superficie variable. La probabilité d’apparition d’une épidémie varie de façon cyclique au fil des ans (Figure 1.3). La fin des épidémies est, en partie, entraînée par la raréfaction de la nourriture pouvant supporter les populations importantes de chenilles. Une épidémie est présentement en cours au Québec, avec des millions d’hectares défoliés chaque année au Lac-Saint-Jean, sur la Côte-Nord et plus récemment dans le Bas-Saint-Laurent (Regnière 2014)
Figure 1.3 - Nature cyclique des épidémies (Blais 1983) – Les dommages causés par C.
fumiferana varient grandement dans le temps. Les périodes de ravages importants,
appelées épidémies, sont causées par plusieurs facteurs contribuant à la prolifération de la tordeuse et arrêtées par d’autres facteurs, dont l’épuisement de la nourriture, ce qui amène la phase endémique.
Figure 1.4 Cycle saisonnier de C. fumiferana. Source : Michel Cusson, Service Canadien des Forêts.
1.2.1.2 Cycle saisonnier de C. fumiferana
C. fumiferana a un cycle saisonnier assez simple qui dure un an. Les œufs sont pondus sur des aiguilles des arbres hôtes au mois de juillet/août. Une dizaine de jours plus tard, les larves éclosent et commencent à se chercher un abri pour passer l’hiver, souvent sur l’écorce de la cime des arbres où elles se tissent un hibernacle en soie à l’intérieur duquel elles muent au deuxième stade avant d’entrer en diapause pour l’hiver. Le printemps venu, les chenilles reprennent leur activité et commencent à arpenter les branches à la recherche de bourgeons nouvellement débourrés. Elles peuvent aussi se nourrir des aiguilles des années précédentes jusqu’à ce que les bourgeons débourrent. La larve pénètre alors dans le bourgeon et s’y réfugie pour le consommer de l’intérieur, ou bien se tisse un abri en soie autour des jeunes aiguilles pour les manger avec plus de facilité. Les chenilles continuent à se nourrir et traversent quatre autres stades larvaires jusqu’au cœur de l’été, moment auquel elles se transforment en chrysalides. Une à deux semaines plus tard, les papillons émergent. Ils ne se nourrissent pas et leur brève existence d’environ deux semaines ne consiste qu’à s’accoupler et à pondre des œufs (Martineau 1985).
1.2.1.3. Aire de répartition de C. fumiferana
L’aire de répartition de C. fumiferana couvre une très grande partie du territoire canadien (Figure 1.5). Cependant, on observe présentement un déplacement vers le nord des populations de tordeuse. Les changements climatiques et leur influence sur le cycle d’émergence des bourgeons de conifères est la principale raison avancée pour expliquer ce déplacement (Régnière 2012).
Figure 1.5 - Aire de répartition de C. fumiferana au Canada, en Alaska et dans le nord-est des États-Unis (Lumley et Sperling, 2011)
Figure 1.6 - Déplacement des populations de tordeuse vers le nord (Régnière 2012) – Carte
réprésentant les zones les plus touchées par C. fumiferana au 20e siècle (zone délimitée par les lignes noires). Les lignes bleues délimitent la zone touchée par C. fumiferana au 19ème
siècle et les lignes rouges délimitent la zone qui sera vraisemblablement touchée au 21ème
1.2.2. Hôtes et alimentation
C. fumiferana tire tous les nutriments nécessaires à sa survie de la matière végétale. Plus particulièrement, la larve se nourrit d’aiguilles de conifères. Elle privilégie les jeunes aiguilles des bourgeons fraîchement débourés, mais en situation d’indisponibilité, la larve s’attaque aux aiguilles plus âgées et aux fleurs mâles. Les hôtes principaux de la tordeuse sont le sapin baumier (Abies balsamea), l’épinette blanche (Picea glauca), l’épinette rouge, (Picea rubens) et dans une moindre mesure, l’épinette noire (Picea mariana) (http://www.rncan.gc.ca/forets/insectes-maladies/13404).
1.2.2.1. Le sapin baumier
Le sapin baumier est l’hôte de prédilection de C. fumiferana. C’est un conifère poussant dans divers sols et sous les climats observés dans les forêts septentrionales du centre et de l’est du Canada. Cet arbre peut atteindre une taille de 25 mètres et un âge de 150 ans. On l’observe dans des forêts consistant seulement de sapin baumier ou bien avec des peupliers faux-trembles, des bouleaux à papier, des épinettes blanches, des épinettes noires, des épinettes rouges et des pruches du Canada (figure 1.7). L’arbre est principalement utilisé pour les sapins de Noël, le bois d’œuvre ou les pâte et papiers (http://tidcf.nrcan.gc.ca/fr/arbres/fiche/38).
1.2.2.2. L’épinette blanche
L’épinette blanche est la deuxième espèce d’arbre la plus touchée par C. fumiferana. Le nom de tordeuse des bourgeons de l’épinette vient du fait que lors des premières invasions de cet insecte, l’industrie forestière utilisait principalement l’épinette blanche comme essence et voyait sa destruction comme le principal impact de C. fumiferana (Martineau 1985). On trouve cet arbre partout au Canada sauf sur la côte du Pacifique et dans le grand Nord (figure 1.8). L’épinette blanche peut atteindre 25 m de hauteur et peut vivre jusqu’à 200 ans. Son bois est utilisé pour les pâtes et papier et le bois d’œuvre (http://tidcf.nrcan.gc.ca/fr/arbres/fiche/38).
hauteur et peut vivre plus de 200 ans. On peut le différencier des autres espèces d’épinette par ses courtes aiguilles (0.5 à 1.5 cm), ses petits cônes et son port étroit. Son bois est utilisé pour les pâtes et papiers et le bois d’œuvre (http://tidcf.nrcan.gc.ca/fr/arbres/fiche/39).
Figure 1.7 - Aire de répartition du sapin baumier au Canada et au nord des États-Unis (Ressources naturelles Canada, Service canadien des forêts)
Figure 1.8– Aire de répartition de l’épinette blanche au Canada, en Alaska et au nord des États-Unis (Ressources naturelles Canada, Service canadien des forêts)
1.2.2.4. La composition chimique et nutritive des aiguilles de conifère
Comme les aiguilles de conifères sont la seule source de nourriture connue des larves de tordeuse, on peut supposer qu’elles contiennent tous les nutriments nécessaires à leur croissance. Des études faites sur le contenu chimique des aiguilles d’autres conifères comme l’épinette de Norvège ont révélé une forte teneur en lignine, cellulose et hémicellulose (Johansson 1995). Ces produits sont les principaux composants des cellules végétales et sont probablement la principale source de carbone des larves de tordeuse. Chez quelques insectes bien connus, la digestion de ces produits fait intervenir une symbiose avec un microorganisme se trouvant dans le système digestif de l’insecte (Calderón-Cortés et al. 2012). On ne peut donc exclure la possibilité que la digestion des aiguilles d’épinette et de sapin baumier se fasse, ne serait-ce qu’en partie, par l’intermédiaire de microorganismes habitant le tube digestif des larves de C. fumiferana.
Ces aiguilles contiennent aussi plusieurs nutriments inorganiques comme l’azote, le phosphore, le potassium, le calcium et le magnésium, tous en assez bonne quantité pour constituer une bonne source de nourriture (Johansson 1995). Les aiguilles contiennent aussi des composés phénoliques appelés « tannins » qui pourraient avoir un effet toxique sur les larves de tordeuse (Kumbaşlı 2005). Par contre, ces mêmes tannins pourraient aussi diminuer la survie de la bactérie B. thuringiensis et ainsi diminuer l’efficacité du
Figure 1.9 – Aire de répartition de l’épinette noire au Canada, en Alaska et au nord-est des États-Unis (Ressources naturelles Canada, Service canadien des forêts)
1.2.3. Régulation des populations de C. fumiferana
Étant donné les dommages importants causés par la tordeuse aux forêts canadiennes et à l’industrie forestière, ses populations font l’objet de mesures de répression. La tordeuse compte déjà de nombreux ennemis naturels (agents pathogènes, parasites et prédateurs) qui contribuent au maintien des populations à de faibles densités pendant la phase endémique et à leur chute à la fin de la phase épidémique. Toutefois, pendant les pullulations, il est nécessaire d’intervenir avec des pulvérisations insecticides afin de protéger les peuplements les plus touchés. En 2014, deux produits sont homologués au Canada pour le contrôle de la tordeuse, l’insecticide à base de Bacillus thuringiensis et le Mimic, un perturbateur de croissance, mais seul le premier est autorisé au Québec.
1.2.3.1. Agents pathogènes, prédateurs et climat
Comme bien des organismes pluricellulaires, la tordeuse est l’hôte de divers agents pathogènes qui peuvent mettre en péril sa survie et ainsi limiter sa prolifération. Parmi ces agents pathogènes se trouvent des microsporidies, des champignons, des virus et des bactéries. La vulnérabilité de la tordeuse à ces divers agents pathogènes varie en fonction de son stade de développement, de son alimentation et du climat (Morris 1963).
Les populations peuvent aussi être grandement diminuées par l’action des oiseaux prédateurs, qui se nourrissent des larves et des chrysalides et en éliminent ainsi un grand nombre. Des araignées et de nombreux insectes se nourrissent des œufs, des larves et des adultes de C. fumiferana. Parmi ces insectes, les guêpes parasitoïdes comptent parmi les ennemis naturels ayant le plus grand impact sur les populations de tordeuse. Ces guêpes pondent leurs œufs à l’intérieur de la cavité abdominale des embryons, des larves et des pupes de C. fumiferana (Sippell 1982). Parmi les espèces les plus efficaces, on compte Tranosema rostrale, Apanteles fumiferanae et Glypta fumiferanae. Certaines espèces ont été introduites par l’homme dans des zones touchées par la tordeuse, sans grand succès (Morris 1963).
Le climat joue aussi un grand rôle dans la régulation des populations de tordeuses. Les larves sont particulièrement vulnérables au climat à deux périodes de l’année. Après leur éclosion, elles doivent trouver un abri avant les premières gelées d’automne et sont donc à la merci de vents forts et de gelées hâtives. Lors de la sortie de leur hibernation, elles sont encore une fois vulnérables aux vents forts et aux gelées tardives du printemps, jusqu’à ce qu’elles se tissent un abri en soie pour se nourrir des aiguilles. La dispersion par le vent peut être responsable de la mort de jusqu’à 20% des larves chaque année. Des conditions climatiques
favorables à la prolifération de la tordeuse plusieurs années consécutives comptent parmi les facteurs présumés importants au déclenchement d’une épidémie (Morris 1963).
1.2.3.2. Lutte contre C. fumiferana
Comme les facteurs de régulation naturels ne suffisent pas à limiter les pullulations de C. fumiferana, les gouvernements provinciaux utilisent divers moyens pour combattre ce fléau. En détectant des signes avant-coureurs d’une épidémie, comme une défoliation hâtive dans les forêts d’arbres matures, il est possible de conserver une certaine quantité de bois en récoltant les peuplements les plus à risques avant l’arrivée de l’invasion. Pendant la période endémique, les peuplements d’arbres vulnérables sont aménagés de façon à ce que d’autres espèces d’arbres, résistantes à la tordeuse prolifèrent, augmentant la diversité du peuplement. Malgré tout, l’épandage d’insecticide est souvent la méthode permettant de mieux protéger la forêt lors d’invasions importantes. Le seul moyen ayant vraiment fait ses preuves est l’épandage aérien d’insecticide à base de Bacillus thuringiensis. Cet insecticide est épandu dans les zones les plus à risque à travers le Canada avec comme objectif de préserver au moins 50% du feuillage des zones touchées (Davidson 1999).
1.2.3.3. Bacillus thuringiensis
B. thuringiensis est une bactérie Gram positive sporulante du sol. On la trouve souvent dans des environnements habités par des insectes. Cette espèce est particulièrement connue en raison de sa capacité à produire un cristal, la delta-endotoxine, lors de la sporulation. Ce cristal a la capacité de créer des pores dans la paroi du tube digestif de nombreux insectes, dont les larves de papillons, de mouches, de moustiques et de Coléoptères (Cranshaw 2013). Les pores dans la paroi du tube digestif empêchent l’insecte de se nourrir, mais permettent aussi le passage des microorganismes de l’intestin vers l’hémocèle de l’insecte. La mort de l’insecte peut donc résulter d’une inanition et/ou d’une septicémie. Cette bactérie est l’un des insecticides biologiques les plus utilisés sur la planète. Dans le cas de la lutte contre la tordeuse, c’est la variété kurstaki, spécifique aux Lépidoptères, qui est utilisée (Davidson 1999). L’insecticide peut être très efficace, mais doit être utilisé dans des conditions spécifiques pour que les résultats soient satisfaisants. L’insecticide doit tomber directement sur les aiguilles et y rester; il ne doit donc pas être appliqué lors de pluies et de forts vents. Il doit être appliqué après la sortie des aiguilles, mais assez tôt pour pouvoir accomplir son action qui est assez lente. De plus, cet insecticide est assez coûteux, limitant la superficie des zones pouvant être protégées. Des études ont aussi démontré que le tannins contenus dans les aiguilles de conifères pouvaient inactiver la delta-endotoxine (Lüthy et al. 1985). Finalement, il peut aussi toucher des Lépidoptères non-ciblés, ce qui pourrait entraîner des effets indirects non-souhaités. 1.1.2.4. Bacillus thuringiensis et son lien avec le microbiome intestinal
La cause de la mort causée par B. thurigiensis et son lien potentiel avec le microbiome sont encore sujet à controverse. Les premières études sur le sujet concluaient que la présence de bactéries commensales dans l’intestin de l’insecte était nécessaire à l’action insecticide de B. thuringiensis (Broderick et al. 2006, Broderick et al. 2009). Des études subséquentes sont toutefois arrivées à des conclusions inverses, la toxine ayant entraîné la mort de larves de Lépidoptères axéniques (Johnston et Crickmore 2009, van Frankenhuyzen et al. 2010). Ultérieurement, une autre équipe s’est penchée sur le sujet et leurs résultats ont suggéré que les bactéries présentes dans l’intestin accéléraient la mort de l’insecte sans toutefois être essentielles (Mason et al. 2011). En effet, les larves axéniques touchées par la toxine cessent de se nourrir et meurent de faim. D’autre part, les pores intestinaux créés par la toxine permettent aux bactéries commensales d’accéder à l’hémocèle de l’insecte et d’accélérer sa mort par septicémie. Finalement, d’autres facteurs comme la diète et l’espèce d’insecte peuvent probablement influencer l’impact du microbiome sur l’efficacité de B. thuringiensis.
1.3. Analyse du microbiome
1.3.1. Méthodes d’analyse du microbiome
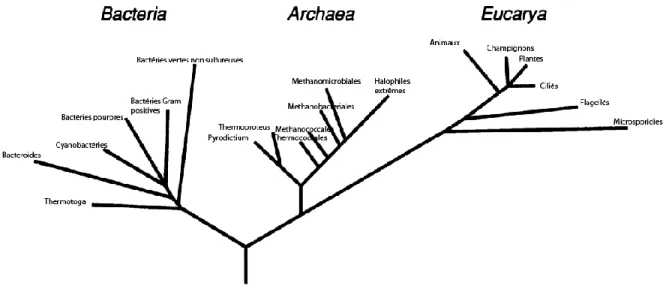
Comme le concept de microbiome est assez récent, peu de méthodes standardisées ont été développées à ce jour pour en faire l’analyse. Les premières études d’écologie microbienne consistaient en différentes étapes d’isolement, d’enrichissement et de croissance sur gélose suivies par différents tests biochimiques visant à classer les microorganismes dans un taxon connu. Des pionniers comme Carl Woese ont démontré l’utilité du gène codant pour l’ARN ribosomal 16S bactérien pour faire la classification bactérienne (Fox et al. 1980). Aux fins d’analyse, le gène en question était cloné puis soumis à une digestion avec des enzymes de restriction, générant un patron de restriction permettant l’identification du taxon. Mitchell Sogin a ensuite développé un protocole d’isolation d’ARN et de rétro-transcription suivi par du séquençage Sanger pour accélérer l’identification de bactéries (Lane et al. 1985). Suite à la découverte de la réaction d’amplification en chaîne par polymérisation (PCR), il devint possible d’amplifier le gène codant pour l’ARN ribosomal 16S chez les bactéries et l’ARN ribosomal 18S chez les eucaryotes. C’est à partir des résultats obtenus en utilisant ces techniques que Woese a pu proposer sa version de l’arbre phylogénétique de la vie (Woese et al. 1990) (Figure 10). Avec l’avènement du séquençage nouvelle génération, il devint possible de faire séquencer tous les gènes du 16S ou 18S contenus dans un échantillon. Il fut aussi possible de séquencer les génomes ou les ARN transcrits de tous les microorganismes contenus dans un échantillon.
1.3.1.1. Le gène ribosomique 16S
L’ARN 16S est un fragment d’ARN servant d’échafaudage à la petite sous-unité du ribosome bactérien. Le gène codant pour ce dernier est un des gènes les mieux conservés du domaine bactérien. Par contre, il est aussi constitué de régions hypervariables, comme l’illustre la Figure 1.11, où neuf régions affichent un taux de variation nettement plus élevé. Cette structure particulière permet à ce gène d’être utilisé comme marqueur de divergence phylogénétique ou simplement comme identificateur taxonomique. Les régions conservées (creux dans la figure) permettent la conception d’amorces universelles pour le domaine bactérien alors que les régions variables permettent de distinguer des bactéries au niveau de l’espèce. Chez les eucaryotes, l’ADN ribosomique qui porte les gènes codant pour les ARN ribosomiques 18S, 5.8S et 28S, ainsi que les deux espaces intergéniques ITS1 et ITS2 peut être utilisé aux mêmes fins (Hunt et al. 2004).
Figure 1.10 – Arbre de la vie de Woese démontrant la séparation en phylums des organismes vivants et la distance génétique qui les sépare de l’ancêtre commun. Figure inspirée de (Woese et al., 1990)
Figure 1.11- Taux de variabilité selon la position de la base dans la séquence du gène de l’ARN 16S de Pseudomonas (d’après (Bodilis et al. 2012). Les pics identifiés V1 à V9 désignent les régions variables pouvant être séquencées à des fins d’identification taxonomique. Le taux de variabilité en axe des Y est mesuré en prenant en compte la fréquence moyenne dans une fenêtre de 50 bases de part et d’autre de chaque position sur l’axe des X.
1.3.1.2. Culture des microorganismes
Avant l’avènement de la biologie moléculaire, la seule méthode d’identification des populations microbiennes dans un échantillon naturel était l’isolement en culture. L’échantillon était homogénéisé et étalé sur un milieu permettant à la majorité des organismes connus de croître, c’est-à-dire un milieu comportant des sources de carbone et d’énergie faciles à métaboliser. On isolait ensuite chaque type microbien retrouvé, soit par des méthodes physiques comme la striation en trois étapes, soit par étalement sur divers milieux sélectifs.
Une fois que l’on avait obtenu des cultures pures, il fallait utiliser divers tests biochimiques pour tenter de classer les microorganismes isolés selon leurs capacités physiologiques ou métaboliques. Parmi ces tests, on compte la dégradation des nitrites ou de certaines formes de glucose, les tests de motilité, la couleur des colonies sur milieu MacConkey et la coloration de Gram. Le résultat de ces tests donnait un bon aperçu des capacités métaboliques de la communauté microbienne de l’échantillon et permettent de classer certains de ses composants dans des taxons connus (Dance et al. 1989).
Suite au développement de la PCR et du séquençage Sanger, le gène codant pour l’ARN faisant partie de la petite sous-unité du ribosome devint un outil de choix permettant de classer les microbes en fonction de l’espèce. La séquence de ce gène permet effectivement de différencier des bactéries appartenant à différentes espèces. Donc, avec des amorces universelles 16S pour les bactéries ou 18S et ITS pour les eucaryotes, on obtient un amplicon dont la séquence indique, lorsque comparée à une base de données, à quel taxon appartient l’organisme en question. Pour les bactéries, il suffit donc d’isoler chaque organisme en culture comme mentionné précédemment, d’extraire son ADN génomique et de l’amplifier par PCR avec les amorces 16S. L’amplicon obtenu peut être séquencé par la méthode de Sanger et sa séquence peut être comparée par analyses BLAST aux séquences déposées dans diverses bases de données telles que Greengenes et Genebank (Lane et al. 1985).
une méthode extrêmement inefficace en termes de temps et d’argent. Chaque espèce présente dans l’échantillon doit être soumise à toutes les étapes du protocole. Donc si avant l’isolement on obtient 100 colonies différentes, cela veut dire 100 étalements et cultures pour chaque milieu sélectif utilisé, 100 réactions PCR différentes et 100 échantillons à faire séquencer, sans compter les réplicats et témoins.
1.3.1.3. Séquençage nouvelle génération
Le développement de nouvelles méthodes de séquençage au cours des dernières décennies a donné naissance à de nouveaux domaines de recherche et initié l’ère des technologies –omiques. Parmi les avantages des méthodes de séquençage haut débit se trouvent le coût moindre par base et la rapidité de séquençage (Tableau 1.2). Ces particularités ont grandement augmenté la faisabilité de projets de séquençage de génome et d’études de biodiversité pour des laboratoires n’ayant pas nécessairement les moyens de mener ces projets à terme par séquençage Sanger.
Toutefois, le principal avantage du séquençage haut débit pour l’étude des génomes et de la biodiversité est la possibilité qu’il offre de séquencer à partir d’un mélange de fragments d’ADN différents (contrairement au Sanger où chaque échantillon ne doit contenir que des fragments d’ADN identiques). Parmi les appareils de séquençage haut-débit les plus utilisés on compte le GS-FLX+ de Roche, utilisant la technologie de pyroséquençage 454, et les HiSeq et MiSeq d’Illumina utilisant la technologie « bridge amplification » (Metzker 2010). Par contre, au cours des dernières années, les technologies Illumina ont progressivement surpassé les capacités de la technologie 454 et sont maintenant le principal outil pour les études génomiques et d’écologie microbienne.
Tableau 1.2 - Comparaison de quelques méthodes de séquençage, permettant de déterminer quelle méthode est la plus
appropriée pour divers expériences. Source : Brian Boyle, coordonateur, plateforme d'analyse génomiques, IBIS, U. Laval
Sanger
454
Illumina
Appareil
384 capillaires
GS-FLX+
HiSeq2000
Longueur
moyenne (bases)
650-750
550-650
150
Longueur
maximale
(bases)
1000
1000
150
Quantité de
nucléotides par
analyse
(gigabases)
0.0003
0.6
600
Durée de
l’analyse
6 heures
2 jours
13 jours
Coût par
kilobase ($)
5
0.016
0.000032
Application la
mieux adaptée
Construction de
références
Séquençage de
novo et
amplicons
Séquençage et
transcriptomique
Figure 1.12- Amplification des fragments lors du séquençage 454 de Roche (Metzker 2010). Les billes, chacune dans leur
microréacteur recouvert d’huile, sont soumises à une amplification PCR, recouvrant chaque bille de fragments d’ADN identiques.
Le pyroséquençage (figure 1.12) est effectué en fragmentant l’ADN et en liant des adaptateurs aux extrémités de chaque fragment. Les fragments sont mélangés avec des billes couvertes d’amorces complémentaires aux adaptateurs. Le ratio billes/ADN est tel que chaque bille portera un seul fragment. Les billes, en solution dans l’eau sont alors couvertes d’huile de façon à ce que chaque bille soit contenue dans une gouttelette aqueuse entourée d’huile, un type d’émulsion. Une amplification PCR est alors réalisée et chaque gouttelette sert de microréacteur. Chaque bille est recouverte de fragments d’ADN identiques, simple brin, l’huile est éliminée et le mélange de billes est coulé sur une plaque contenant des puits permettant l’entrée d’une seule bille par puits pendant la centrifugation de la plaque. Celle-ci est subséquemment recouverte d’un mélange d’enzymes, dont une DNA polymérase, une ATP sulfurylase et une luciferase. Différents nucléotides sont appliqués de façon séquentielle et un ordinateur enregistre les signaux lumineux captés par un photorécepteur et déduit ainsi la séquence des fragments (Metzker 2010).
1.3.1.4. Séquençage d’amplicons 16S
L’étude de la biodiversité d’un échantillon par séquençage d’amplicons 16 S est une des premières méthodes d’écologie microbienne développées suite à l’avènement du séquençage haut débit. On extrait l’ADN bactérien de l’échantillon et on amplifie une portion de la séquence du gène codant pour l’ARN 16S. La technologie 454 est tout indiquée pour cette méthode, pouvant séquencer des fragments d’une longueur équivalente au tiers du gène 16S. Les amorces utilisées contiennent déjà les adapteurs nécessaires (A et B sur la Figure 1.13) et les amplicons obtenus sont mélangés avec les billes pour la poursuite de la procédure. Chaque séquence obtenue représente un fragment d’ADN matrice pouvant être considéré comme un individu bactérien, et ces séquences sont regroupées en "Operational Taxonomic Units" (OTUs). Ces derniers ne peuvent être considérés comme représentant un niveau taxonomique formellement défini mais, en pratique, ils permettent de ségréguer les organismes à un niveau se rapprochant de celui de l’espèce. Comme l’illustre la Figure 1.14, les séquences ayant plus de 97% d’identité entre elles sont considérées comme appartenant à un même OTU (Edgar 2013). Ainsi, le seuil de 97% d’identité est considéré comme le seuil permettant de rassembler les séquences provenant vraisemblablement d’un même taxon. Chaque OTU généré par l’analyse est donc considéré ici comme représentant un seul taxon.
Figure 1.13 – Amorces utilisées pour l’étude de biodiversité à partir du 16S bactérien. En bleu, séquences
complémentaires avec des régions conservées du 16S bactérien. En rouge, séquences d’identification nécessaires au multiplexage. En vert, adaptateurs complémentaires aux amorces se trouvant sur les billes. En gris, portion d’ADN à amplifier.
Les séquences sont comparées à celles déposées dans des bases de données d’ARN 16S bactérien afin d’évaluer l’identité des espèces bactériennes présentes dans l’échantillon. Cette méthode permet de faire un inventaire des espèces bactériennes présentes dans l’échantillon et de mesurer leur abondance relative. On peut ainsi déterminer quelles espèces de bactéries sont favorisées sous certaines conditions et d’identifier le microbiome « cœur » correspondant aux taxons présents dans tous les échantillons provenant de l’environnement choisi. Par contre, cette méthode ne donne pas d’information sur l’identité des gènes microbiens ou sur les voies métaboliques auxquelles ils participent.
1.3.2. Statistiques et mesures reliées à l’étude du microbiome
Au-delà de l’identification des microorganismes présents dans l’échantillon étudié, il est intéressant d’évaluer à quel point ces microorganismes sont proches génétiquement, et comment les populations bactériennes sont réparties parmi les taxons recensés. En raison d’un intérêt grandissant pour l’écologie microbienne, de nombreuses méthodes statistiques et informatiques ont été développées pour obtenir ce type d’information.
1.3.2.1 Mesures de diversité
La diversité est une valeur prenant en compte deux concepts extrêmement importants en écologie. Elle prend en compte la richesse, qui est la quantité de taxons différents retrouvés dans une population, mais elle prend aussi en compte l’abondance relative de chaque taxon, c’est-à-dire la proportion d’individus appartenant à chaque taxon de la population. La diversité est souvent reliée à la stabilité et à la productivité d’un environnement (Stirling et Wilsey 2001). Les deux indices les plus utilisés pour mesurer la diversité sont l’indice Shannon et l’indice Simpson.
Le « Shannon index » fut proposé par Claude Shannon pour mesurer l’entropie de caractères dans une ligne de texte. Ce concept repose sur le fait que plus il existe de caractères et plus ces caractères sont utilisés à la même fréquence, plus le prochain caractère utilisé sera difficile à prédire. L’indice est calculé comme suit :
Où R est la quantité de caractères (ou taxons) différents présents et pi est la proportion de caractères du type i
(abondance relative) dans la ligne de texte. Plus la valeur P varie parmi les divers caractères, plus l’indice se rapproche de 0, et plus le caractère est facile à prédire (Shannon 1997).
Où R est la richesse et pi est l’abondance relative du taxon i. Comme la valeur diminue en fonction de la
diversité de la population, l’indice inverse Simpson est plus souvent utilisé en écologie, en divisant 1 par l’indice Simpson obtenu (Simpson 1949).
1.3.2.2. Arbres phylogénétiques
Un arbre phylogénétique est une représentation graphique des relations de parenté entre organismes vivants. Il permet de visualiser la proximité évolutive entre divers individus ou espèces inférées à partir de critères biologiques telles la distance génétique. Ces arbres peuvent être construits à partir d’alignements de séquences ou bien avec une matrice de distance génétique entre des séquences provenant des individus de la population étudiée. « Clearcut » est un des logiciels les plus utilisés pour la production d’arbres phylogénétiques dans le contexte d’études de microbiomes. Il utilise une variante à stringence limitée, l’algorithme du neighbor-joining, qui consiste en l’utilisation de distances génétiques entre individus pour évaluer leur niveau de parenté (Evans et al. 2006).