Étude de la survie et de la virulence de Salmonella
enterica ssp. enterica dans des modèles
gastro-intestinaux humains
Thèse
Camille Cavestri
Doctorat en sciences et technologie des aliments
Philosophiæ doctor (Ph. D.)
Résumé
Salmonella enterica spp. enterica est un pathogène omniprésent responsable de toxi-infection
alimentaire pouvant potentiellement être mortelle pour l’humain. Afin de déclencher une infection, le pathogène doit survivre aux conditions stressantes rencontrées lors de son passage dans le tractus gastro-intestinal humain et coordonner l'expression de plusieurs facteurs de virulence. L’objectif de cette thèse est d’étudier le comportement de différentes souches de S. enterica dans le tractus gastro-intestinal humain en utilisant des approches in vitro. Les souches ont été sélectionnées en fonction de leur potentiel de virulence et de leur origine (clinique, alimentaire, animale, environnementale). En simulant le tractus gastro-intestinal supérieur avec le modèle in vitro TIM-1, une mortalité bactérienne est observée en compte viable dans l'estomac comparé aux résultats au PMA-qPCR. Cette différence suggère la présence de cellules viables mais non cultivables dans l’estomac. Une reprise de croissance est par la suite observée dans le duodénum et l’iléon. Après le passage dans le TIM-1, toutes les souches ont bien survécu, mais la survie de S. enterica dépendait de sa virulence. En effet, les souches de virulence élevée avaient significativement une survie plus élevée que celles de faible virulence. En condition iléale simulée dans le modèle PolyfermS, S. enterica est progressivement éliminée du milieu durant les douze premières heures, mais certaines souches maintiennent leur présence avec le lavage continu après 24 h. Les souches de S. enterica varient donc dans leur capacité à coloniser le système en présence du microbiote iléal. Par ailleurs, l’ajout de S.
enterica n’a eu aucun impact sur la composition du microbiote iléale ou sur son activité métabolique. S. enterica ne peut utiliser le système immunitaire pour provoquer une dysbiose dans ce modèle, ce
qui limite les mécanismes compétitifs. L’exposition des souches de S. enterica à une digestion gastro-intestinale en présence du microbiote iléal a induit l'expression de facteurs de virulence liés à l'adhésion et au gène ssaB du SPI-2, malgré l'absence de tissus hôtes. Cette induction était plus importante pour les souches à virulence élevée que pour les souches à faible virulence.
Ces résultats approfondissent les connaissances sur le comportement de S. enterica dans le tractus gastro-intestinal humain pour pouvoir établir des stratégies de prévention contre ce microorganisme pathogène et pour mieux gérer le risque lié à Salmonella.
Abstract
Salmonella enterica subsp. enterica is a ubiquitous pathogen responsible for food-borne infection,
potentially life-threatening to humans. In order to trigger infection, the pathogen must survive the stressful conditions of the human gastrointestinal tract and coordinate the expression of several virulence factors in response to this environment. The objective of this thesis is to study the behavior of S. enterica in the human gastrointestinal tract using in vitro approaches. S. enterica strains were selected according to their virulence potential and their origin (clinical, food, animal and environmental). By simulating the upper gastrointestinal tract with the in vitro model TIM-1, bacterial mortality was observed by viable count in the stomach, compared to PMA-qPCR results. This difference suggests the presence of viable but non-cultivable cells. A resumption of growth was subsequently observed in the duodenum and the ileum. After passage through the TIM-1, all strains survived but high virulence
S. enterica strains had significantly higher survival than low virulence strains. In simulated ileal
conditions in the PolyfermS model, S. enterica was gradually eliminated from the medium during the first twelve hours. After 24 h, most strains maintained their presence with continuous wash-out. S.
enterica strains vary in their capacity to colonize the system in the presence of the ileal microbiota.
Furthermore, S. enterica had no impact on the composition of the ileal microbiota or on its metabolic activity. S. enterica cannot use the immune system to induce dysbiosis in this model, limiting the competitive mechanisms. The exposure of S. enterica strains to gastrointestinal digestion in the presence of ileal microbiota induced expression of virulence factors linked to adhesion and SPI-2 gene (ssaB), despite the absence of host tissues. This induction was greater for high virulence strains than low virulence strains.
These results deepen our knowledge on the behavior of S. enterica in the human gastrointestinal tract, in order to establish prevention strategies against the pathogen and to better manage the risk linked to
Table des matières
Résumé……… ... ii
Abstract…………. ... iii
Table des matières ... iv
Liste des tableaux ... viii
Liste des figures…………. ... x
Liste des abréviations ... xii
Remerciements……... xv
Avant-propos….. ... xvii
Introduction……… ... 1
Chapitre 1. Revue de littérature ... 3
1.1. Caractéristique des infections à Salmonella ... 3
1.1.1. Diversité taxonomique et spectre d’hôte ... 3
1.1.2. Cycle d’infection... 4
1.1.3. Manifestations cliniques ... 5
1.1.4. Surveillances épidémiologiques des infections ... 7
1.1.5. Réservoir et transmission ... 8
1.2. Caractérisation du génome de Salmonella ... 9
1.2.1. Le pangénome ... 9
1.2.2. Facteur de virulence ... 10
1.2.2.1. Les îlots de pathogénicité ... 10
1.2.2.2. Flagelle ... 12
1.2.2.3. Fimbriae ... 13
1.2.2.4. Plasmide et phage ... 13
1.2.2.5. Lipopolysaccharide (LPS) ... 14
1.2.2.6. Régulation des facteurs de virulence ... 14
1.2.3. Relation diversité génomique et phénotypique ... 15
1.3. Salmonella et les interactions avec le tractus gastro-intestinal ... 17
1.3.1. Effets des paramètres abiotiques de l’intestin humain sur la survie et la virulence ... 18
1.3.1.1. Acidité gastrique ... 18
1.3.1.2. Sécrétions biliaires ... 21
1.3.1.3. Enzymes digestives ... 22
1.3.2. Effets des facteurs biotiques de l’intestin humain sur la survie et la virulence….. ... 24
1.3.2.1. Hormones de l’hôte et éthanolamine ... 24
1.3.2.2. Microbiote intestinal et leurs métabolites ... 25
1.3.3. Application à des modèles dynamiques intestinaux humain ... 27
1.3.3.1. Modèles multi-compartimentés mimant le tractus gastro-intestinal supérieur….. ... 28
1.3.3.2. Systèmes de fermentation ... 30
1.4. Consortium international « Salmonella Syst-OMICs »... 31
1.5. Hypothèse de recherche ... 32
1.6. Objectifs ... 32
Chapitre 2. Salmonella enterica subsp. enterica virulence potential can be linked to higher survival within a dynamic in vitro human gastrointestinal model ... 33
2.1 Résumé ... 34
2.2. Abstract ... 35
2.3. Introduction ... 36
2.4. Materials and Methods ... 37
2.4.1. Bacterial strains and growth conditions ... 37
2.4.2. Dynamic in vitro digestion ... 38
2.4.3. Determination of bacterial survival ... 41
2.4.4. Comparative analysis of selected S. enterica genes ... 42
2.4.5. Statistical analysis ... 42
2.5. Results ... 42
2.5.1. Acid resistance and viability state of S. enterica strains during simulated stomach passage . 42 2.5.2. Survival and growth of S. enterica strains during simulated duodenum passage… ... 43
2.5.3. The outgrowth of S. enterica strains in the simulated small intestine ... 43
2.5.4. Polymorphic sites in S. enterica genes involved in stress, bile and acid responses. ... 44
2.6. Discussion ... 49
2.7. Conclusion ... 52
2.8. Acknowledgments ... 52
Chapitre 3. Survival of Salmonella enterica subsp. enterica in competition with human ileal microbiota in the continuous PolyFermS model ... 53
3.1. Résumé ... 54
3.2. Abstract ... 55
3.3. Introduction ... 56
3.4. Materials and methods ... 57
3.4.1. Bacterial strains and growth conditions ... 57
3.4.2.1. Fecal collection and immobilization in gel beads ... 58
3.4.2.2. Fermentation set up and design ... 58
3.4.2.3. Propidium monoazide treatment and DNA extraction ... 60
3.4.2.4. PCR method for bacterial enumeration ... 60
3.4.2.5. HPLC analysis for short-chain fatty acids (SCFA) ... 61
3.4.2.6. Metagenomic analysis ... 62
3.4.3. Biofilm phenotype characterization ... 64
3.4.4. Biofilm Formation Assay ... 64
3.4.5. Statistical analysis ... 65
3.5. Results ... 66
3.5.1. Microbial composition by qPCR of donor feces and reactors after the stabilization period... 66
3.5.2. Effect of eight S. enterica strains on microbial composition, diversity by qPCR and metabolic activity…….. ... 66
3.5.3. Microbial community in effluents determined by 16S rRNA MiSeq sequencing…… ... 67
3.5.4. Survival of S. enterica in the ileal fermentation model ... 73
3.5.5. Biofilm phenotypes and formation assay ... 73
3.6. Discussion ... 75
3.7. Conclusion ... 79
3.8. Acknowledgment ... 79
Chapitre 4. Human ileal metabolome increases expression of Salmonella enterica subsp. enterica adhesion factors and modulates invasion factors ... 80
4.1. Résumé ... 81
4.2. Abstract ... 82
4.3. Introduction ... 83
4.4. Materials and methods ... 84
4.4.1. Bacterial strains and growth conditions ... 84
4.4.2. Exposure of S. enterica strains to static simulated human gastrointestinal conditions ... 85
4.4.2.1. S. enterica strains under stomach, duodenum and jejunum conditions ... 85
4.4.2.2. S. enterica strains under ileum conditions ... 87
4.4.2.2.1. Ileum fermentation model ... 87
4.4.2.2.2. S. enterica strains with ileal metabolome ... 87
4.4.2.3. Bacterial enumeration by PMA-qPCR ... 88
4.4.2.4. Expression of stress and virulence genes of S. enterica by RT-qPCR ... 89
4.4.3. Comparative analysis of fimH S. enterica gene ... 91
4.4.4. Statistical analysis ... 92
4.5. Results ... 92
4.5.1. Ileal microbiota composition in fermentation model and in simulated ileal conditions ... 92
4.5.2. Survival of S. enterica strains exposed to simulated gastrointestinal conditions ... 92
4.5.3. S. enterica stress and virulence gene expression after simulated gastrointestinal conditions 94 4.5.3.1. Regulation and stress genes ... 94
4.5.3.2. Genes determining adhesion ... 94
4.5.3.3. Virulence factors from Salmonella pathogenicity island 2,3 and -5 ... 95
4.5.4. Polymorphic site in the fimH gene of S. enterica ... 97
4.6. Discussion ... 97
4.7. Conclusion ... 101
4.8. Acknowledgment ... 102
Conclusion générale et perspective ... 103
Liste des tableaux
Chapitre 1
Tableau 1- 1 Prévalence des sérotypes de Salmonella responsables de maladies humaines de 2009
à 2017 au Canada (adapté du Programme national de surveillance des maladies entériques de l’Agence de la sante publique du Canada) ... 8
Chapitre 2
Tableau 2- 1 List and source of S. enterica strains used in this study ... 39 Tableau 2- 2 Parameters of the TIM-1 system during digestion of semi-solid meal by healthy adult
human ... 40
Tableau 2- 3 The survival rate and the log change of S. enterica strains after digestion of contaminated
lettuce in the TIM-1. The survival rate was determined using the non-log transformed cultivable counts in the pooled ileal effluents and the non-log transformed remaining cells after 300 min of digestion relative to the non-log transformed total cultivable counts at the start of digestion ± standard deviation (n =2). The log change was determined from the difference between the log of cultivable counts in the pooled ileal effluents with the log of remaining cells after 300 min of digestion and the log of total cultivable counts at the start of digestion ± standard deviation (n =2). ... 47
Tableau 2- 4 List of genes involved in resistance to acid and their non-synonymous SNPs between 13
strains and S. enterica Typhimirium LT2 (bp: base pairs). ... 48
Tableau 2- 5 List of genes involved in bile resistance and their non-synonymous SNPs between 13
strains and S. enterica Typhimirium LT2 (bp: base pairs). ... 49
Chapitre 3
Tableau 3- 1 Primer for specific bacterial group used in this study for qPCR ... 61 Tableau 3- 2 Biofilm Formation Assay Classification ... 65 Tableau 3- 3 Bacterial population in the fecal inoculum and in samples from the inoculum reactor (IR)
at the end of the stabilization period. ... 68
Tableau 3- 4 Organic acid content in TR reactors before and during S. enterica challenge (control/NC
and infected at 0, 4, 8 and 12 hours with S. enterica strains), determined by HPLC ... 69
Tableau 3- 5 Biofilm phenotype characterization and formation classification ... 75 Chapitre 4
Tableau 4- 1 S. enterica strains used in this study ... 85 Tableau 4- 2 Conditions used for the sequential simulation of the human gastrointestinal conditions,
adapted from De Sales et al. (2018). ... 86
Tableau 4- 3 Primer for specific bacterial group used in this study for qPCR ... 88 Tableau 4- 4 Genes and primers used for RT-qPCR ... 90 Tableau 4- 5 The bacterial population in the inoculum reactor (IR) at the end of the stabilization period
Tableau 4- 7 Single nucleotide polymorphism and amino acid variation in the fimH gene of S. enterica
Liste des figures
Chapitre 1
Figure 1- 1 Principales étapes de l’infection à Salmonella au niveau de l'épithélium intestinal: adhésion
et translocation à travers les cellules M, invasion directe des cellules épithéliales intestinales et capture par les cellules dendritiques. Adaptée de Sansonetti (2004). ... 5
Figure 1- 2 Facteur de virulence de S. enterica ssp. enterica. Adaptée de de Jong et al. (2012) ... 10 Figure 1- 3 Tractus gastro-intestinal d’un adulte en bonne santé et principaux processus physiques et
chimiques. T1/2: temps de demi-vidange gastrique, Modifié de Roussel 2019. ... 18
Figure 1- 4 Composants des mécanismes d’adaptation et de résistance à l’acide. Un faible pH interne
induit l'expression (a) des systèmes de décarboxylase d'acides aminés, (b) la synthèse des ASPs et (c) des modifications de la composition de la membrane. Adaptée de Álvarez-Ordóñez et al. (2011). ... 20
Figure 1- 5 Composition du microbiote résident d’un adulte sain, dans les différentes parties du tube
digestif. Modifiée de Fernandez 2014 ... 26
Chapitre 2
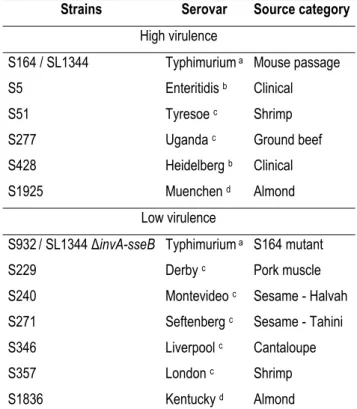
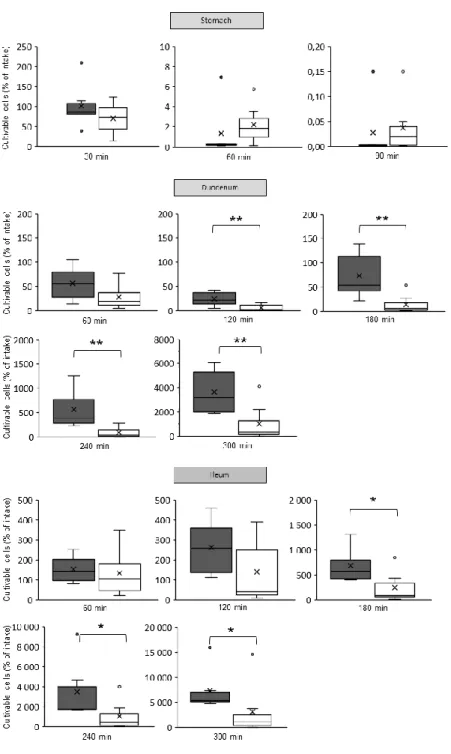
Figure 2- 1 Effect of gastric, duodenal and ileal secretions in the TIM-1 on the HV strains and the LV strains of S. enterica. Results are expressed as log CFU/ml ± mean absolute deviation (n=2) of cultivable count and were compared over time (only one value was obtained in ileum for strain S5). For each strain, different letters (a-d) indicate significant differences over time at p ≤ 0.05 with the Tukey-Kramer test ... 45 Figure 2- 2 Survival of S. enterica HV strains (grey) and LV strains (blanc) in the stomach, duodenum and ileum compartment of the TIM-1 model. Results are expressed as survival percentage of cultivable count. Boxes indicate 25th and 75th percentiles, black bars indicate medians, crosses indicate means and whiskers indicate minimum and maximum. Dots represent extreme outliers. Statistical differences between HV and LV strains are given at *p < 0.005 and **p < 0.001 with Wilcoxon-Mann-Whitney test……….46 Figure 2- 3. Comparison between PMA-qPCR (grey striped) and classical plating (black) at 90 min in the gastric compartment of the TIM-1 model for the (A) HV strains and (B) LV strains of S. enterica. Results are expressed as log10 S. enterica cells/ml ± mean absolute deviation (n=2) and statistical differences between the cross-compared methods are given at *p < 0.05 and **p < 0.005 ... 47
Chapitre 3
Figure 3- 1 Experimental set up (Tanner et al. 2014) and timeline of the continuous PolyFermS
fermentation, reproducing the human ileum. After the stabilization period of 13 days (A) and 17 days (B), the eight S. enterica strains were tested each in duplicate. (A) low-virulence strains (LV fermentation), (B) high-virulence strains (HV fermentation) IR: inoculum reactor, TR1-2: test reactor 1 and 2, F: flow rate, M: fresh MacFarlane medium supply, BC: bead colonization, S: stabilization, T: testing period, W: wash period ... 59
Figure 3- 2 Quantification by PMA-qPCR of the three main bacterial groups in TR reactors for 24h
after adding (A) low virulent strains S229 (black circle), S271 (black diamond), S346 (black triangle) and S1836 (black square) and (B) high virulent strains S5 (white diamond), S164 (white circle), S428 (white square) and S932 (white triangle). Each point represents the mean of two biological replicates
(log10 copy/ml effluent) and bars indicate standard deviations. Values were compared using the Kruskal-Wallis test and no significant differences were found (p > 0.05). ... 68
Figure 3- 3 Composition of the human ileal microbiota preceding and following S. enterica infection as
determined by 16 S rRNA gene amplicon sequencing. Stacked bars show the relative abundance of the 10 most abundant genera in (A) the LV fermentation and (B) HV fermentation before infection (negative control: NC; gray square represents no replicate) and at time 0, 2 and 4 hours after infection for each replicate (Rep 1, Rep 2). ... 71
Figure 3- 4 Effect of S. enterica on ileal microbial composition and diversity as determined by 16S
rRNA gene sequencing. (A) Simpson diversity index considers the number of species present and their relative abundance. The Simpson diversity index ranges between 0 (no diversity) and 1 (infinite diversity). Strains are represented in duplicate (Rep 1 and Rep 2). (B) PCoA analysis included weighted average scores of the six most abundant species in the microbial community indicated in blue and (C) db–RDA analysis of the ileal microbial community at the genus level with respect to treatment (control/NC vs infected at 0, 2 and 4 hours with S. enterica strains). The probabilities associated with the possible impact of each condition separately (donor and treatment time) on the microbial community are listed in the table. ... 72
Figure 3- 5 Survival of S. enterica strains by dd-qPCR during continuous flow in the presence of ileal
microbiota. (A) for low virulent strains S229 (black circle), S271 (black diamond), S346 (black triangle) and S1836 (black square) and (B) for high virulent strains S5 (white diamond), S164 (white circle), S428 (white square) and S932 (white triangle). Dotted lines indicate theoretical washout of an inert particle added at 107copy / ml and a detection limit of 2 log10 ttrA copy/ml. Each point represents the
mean of two biological replicates and bars indicate standard deviation. ... 74
Chapitre 4
Figure 4- 1 (A) Design of the experiment’s days. Stab: two days were required for the stabilization of
ileum microbiota in the reactor between each experiment. All Strains were passed in duplicate in the system. (B) Set up of the fermentation models with immobilized gut microbiota (Tanner et al., 2014) ... 86
Figure 4- 2 Transcriptomic analysis of high and low virulent strains of S. enterica based on (A)
regulation and stress response genes, (B) attachment structure and (C) virulent genes from SPI after passage through the simulated human gastrointestinal condition. Heatmaps were generated using the complexHeatmap package in R studio. All expression experiments were done in two biological replicates and three technical replicates. ... 96
Liste des abréviations
ADN, DNA : acide désoxyribonucléique ANOVA: analysis of variance
AR : résistances aux acides ARCOL: ARtificial COLon ARN, RNA : acide ribonucléique ASP: acid shock protein
ASV: amplicon sequence variant ATR : réponse de tolérance à l’acide CaCl2: Chlorure de calcium
CDC: Centers for Disease Control and Prevention CGH: Comparative Genomic Hybridization CO2: dioxyde de carbone
db-RDA: distance-based redundancy analysis ddPCR: droplet digital PCR
EDTA : acide éthylènediaminetétraacétique ESIN: Engineered Stomach and small INtestinal GBD: Global Burden of Disease
GI: gastro-intestinal HCL : acide chlorhydrique
HPLC: high performance liquid chromatography, chromatographie en phase liquide à haute performance
HV: high virulent
IBIS : Institut de biologie intégrative et des systèmes IR: inoculum reactor
KCl : Chlorure de potassium LB: Luria-Bertani
LPS: lipopolysaccharide LV: low virulent
MLVA: Multi Locus Variable-Numbers tandem-repeat Analysis NaCl: Chlorure de sodium
NaHCO3: Sodium bicarbonate NC: control négatif, negative control NTS : salmonelles non typhiques
OMS, WHO: Organisation Mondiale de la Santé, World Health Organization PCoA: Principle Coordinate Analysis
PFGE: Électrophorèse sur Gel à champ Pulsé PMA: propidium monoazide
qPCR: quantitative Polymerase Chain Reaction
SCFA, AGCC : short-chain fatty acids, acide gras volatiles à courte chaine, SHIME: Simulator of the Human Intestinal Microbial Ecosystem
SIMGI: SIMulator of the Gastro-Intestinal tract
SNP : Single Nucleotidic Polymorphisme, polymorphisme de simple nucléotide (
SPI : Salmonella Pathogenicity Islands, îlots de pathogénicité de Salmonella
SST3, T3SS : système de sécrétion de type 3, type 3 secretion system TIM-1: TNO Gastrointestinal Model 1
TR: test reactor
VBNC : viable but non cultivable VCS : vacuole contenant Salmonella VIH : virus de l'immunodéficience humaine
Remerciements
Comme tout projet, un doctorat ne peut se faire sans l'aide et le soutien de plusieurs personnes. Je tiens donc à exprimer toute ma reconnaissance et ma gratitude à ces personnes qui y ont contribué de différentes manières.
Mes premiers remerciements vont d'abord à ma directrice de thèse, Gisèle Lapointe, qui m'a accompagné dans ce long travail de manière inestimable, malgré la distance. Je la remercie de m'avoir donné la chance de faire partie de son équipe et de m'avoir fait confiance pour travailler sur ce projet passionnant et multidisciplinaire depuis le début. Cette confiance m'a permis de jouir d'une grande autonomie, afin de me développer sur le plan personnel et professionnel. Je la remercie également pour sa grande disponibilité, ses précieux conseils, ses encouragements et sa patience lors de nos nombreux échanges, qu'ils aient été manuscrits, virtuels ou réels. Tout ceci m'a permis de me motiver et de mieux avancer surtout dans les moments difficiles. Je tiens également à la remercier pour ses conseils et ses corrections lors de la rédaction de mon manuscrit de thèse.
Je remercie mon codirecteur, Ismail Fliss, pour sa gentillesse et de m'avoir introduit à la plateforme de digestion in vitro de l'INAF pour la réalisation d'une majeure partie de mes expériences dans le cadre de cette thèse. Je souhaite également remercier Julie Jean d'avoir accepté de faire la prélecture et d'avoir évalué l'ensemble de cette thèse. Je profite aussi pour remercier Sylvain Quessy et Roger Levesque d’avoir accepté d’évaluer ma thèse et de faire partie du jury de thèse.
J'adresse mes remerciements à Patricia Savard, pour son aide précieuse et sa grande contribution dans la mise au point des protocoles et l’utilisation des systèmes de digestion utilisés dans cette étude. Ton savoir-faire, ton dynamisme et ton optimisme m'ont été indispensables pour surmonter les nombreux caprices du TIM-1 et des fermenteurs. Il avait de quoi « virer fou » sans toi !
Un énorme merci à Jérémie Théolier qui s'est lancé en tant que support technique dans le début de l'aventure de la digestion in vitro, et qui malgré son changement de direction a toujours su m'épauler et me prêter main forte. Ton expertise en fermentation et ta disponibilité ont été un atout majeur lors de ce projet et ton apport dans l'avancement et la finalisation de la rédaction de cette thèse ont été très appréciés.
Cette thèse n’aurait pas pu être accomplie sans le consortium « Syst-OMICs Salmonella ». Je voudrais donc remercier tous les partenaires de ce projet notamment, Roger Levesque et son équipe : Jean-Guillaume Emond et Brian Boyle pour le séquençage des souches et leur aide pour les techniques moléculaires utilisées dans cette thèse. France Daigle et Danielle Malo et leurs équipes pour avoir effectué le phénotypage in vitro et in vivo des souches de Salmonella.
Je remercie également Mérilie, Alexandre et Charlène pour avoir participé à l’élaboration de cette thèse. Vous avez été d’une grande aide que ce soit au laboratoire, au niveau de la bio-informatique ou des statistiques. Merci pour votre enthousiasme et votre gentillesse à m’aider alors que vous n’aviez pas particulièrement le temps.
Je tiens à remercier Denis Roy pour m’avoir « adopté » dans son équipe, j’ai beaucoup apprécié ces réunions d’équipes qu’elles soient professionnelles ou personnelles. Pour nos échanges conviviaux, nos repas dans l’atrium de l’INAF ou encore nos sorties en dehors du cadre professionnel, un grand merci à mes collègues Joseph, Elena, Carolina, Anna-Lise, Attara, Virginie, Émilie, Véronique, Perrine, Anna-Sofia, Juan, Bénédicte et Frédérique.
Je ne remercierai jamais assez la personne au premier rang de mes journées durant ces dernières années, mon conjoint Bertrand. Merci d’avoir partagé à la fois mes joies et mes craintes, de m’avoir rassuré pour poursuivre cette aventure.
Pour finir, je voudrais particulièrement remercier ma famille et mes proches amis. Pour m’avoir soutenue et encouragée depuis le début et m’avoir comblée d’amour durant toutes ces années. Que ce soit au Québec ou en France, j’ai de la chance de vous avoir. Je sais que je ne vous ai pas rendu la tâche facile ... avec une mention spéciale à ma mère Sylvie.
Avant-propos
Cette thèse de doctorat est composée d’une introduction générale, d’une revue de littérature, de trois articles intégrant les résultats de cette thèse et d’une conclusion générale.
Tout d’abord, l’introduction générale présente la problématique de cette thèse. La revue de littérature qui suit (chapitre 1) est répartie en trois parties : en première partie, les caractéristiques des infections liées à Salmonella (données épidémiologiques, cliniques, pathologies...) sont compilées puis en seconde partie, les caractéristiques de son génome y sont exposées, incluant notamment ces facteurs de virulence. La troisième partie de ce chapitre est dédiée à la modulation de la survie et de la virulence de Salmonella par les principaux paramètres digestifs biotiques et abiotiques de l’humain. De même, cette partie introduit l’intérêt d’utiliser des modèles de digestion in vitro complexe, permettant de reproduire au mieux la physiologie de la digestion humaine. L’hypothèse de recherche et les objectifs spécifiques de cette thèse sont par la suite énoncés.
Le chapitre 2 intitulé “ Salmonella enterica subsp. enterica virulence potential can be linked to higher survival within a dynamic in vitro human gastrointestinal model ” a été soumis pour publication en Avril 2020 dans le journal Food Microbiology. Le chapitre 3 intitulé « Survival of Salmonella enterica subsp.
enterica in competition with human ileal microbiota in the continuous PolyFermS model » et le chapitre
4 intitulé « Human ileal metabolome increases expression of Salmonella enterica subsp. enterica adhesion factors and modulates invasion factors » vont être soumis dans les journaux Gut Microbiome et mBio au cours de l'année 2020. Les auteurs pour ces trois articles sont : Camille Cavestri 1, Patricia
Savard 1, Ismail Fliss 1, France Daigle 4, Daniel Malo 5, Sadjia Bekal 6, Linda J. Harris7, Roger Levesque 3, Lawrence Goodridge 2 et Gisèle LaPointe2
Cette thèse entre dans le cadre du consortium international « Salmonella Syst-OMICs », conçu et dirigé par Roger Levesque et Lawrence Goodridge. Toutes les expériences ont été réalisées à l’Institut sur la nutrition et les aliments fonctionnels (INAF) de l’Université Laval, sous la direction de Gisèle LaPointe et la codirection de Ismail Fliss. Durant ce travail, avec l’aide de Patricia Savard, j’ai réalisé l’ensemble des expériences qui portent sur le passage des souches de Salmonella dans les systèmes
in vitro : le système de digestion gastro-intestinal dynamique TIM-1 (chapitre 2), le modèle de
fermentation PolyfermS permettant de simuler les conditions de l’iléon terminal humain (chapitre 3) et le modèle de digestion gastro-intestinal statique en présence du métabolome iléal humain (chapitre 4).
Les prélèvements, la majorité des analyses et leurs traitements ainsi que la plus grande partie de la rédaction ont été réalisés par moi-même. Les souches utilisées dans nos travaux proviennent des laboratoires de France Daigle, Sadjia Bekal et de Linda J. Harris alors que les séquences génomiques entières ont été réalisées par l’équipe de Roger Levesque. Les équipes de France Daigle et de Danielle Malo ont déterminé la virulence de nos souches dans deux modèles d’infections in vitro (cellules épithéliales et macrophages humains) et dans un modèle d’infection in vivo (souris).
Pour le chapitre 3, le séquençage du gène bactérien ARNr 16S a été réalisé à l'Institut de biologie intégrative et des systèmes (IBIS) de l'Université Laval à l’aide de la technologie Illumina MiSeq. La partie relative à l’analyse des acides gras à courte chaîne a été effectuée sur la plateforme analytique de l’INAF par HPLC et les analyses par ddPCR ont été exécutées à l’Institut de recherche en immunologie et en cancérologie (IRIC) de l’Université de Montréal. Les tests portant sur la capacité des souches à produire des biofilms, ont été accomplis par l’équipe de Joel Weadge de l’Université Wilfrid Laurier.
1 Institute of Nutrition and Functional Foods, Université Laval, Québec, Canada, ² Department of
Food Science, University of Guelph, Guelph, Canada, 3 Institute for Integrative and Systems Biology,
Université Laval, Québec, Canada, 4 Département de microbiologie infectiologie et immunologie,
Université de Montréal, Montréal, Canada, 5 Department of Medicine, McGill University, Montréal,
Canada 6 Laboratoire de Santé Publique du Québec, Ste-Anne de Bellevue, Canada. 7 Department
Introduction
Notre corps est naturellement colonisé par de nombreuses espèces bactériennes présentes sur la peau, les muqueuses et le tube digestif. La plupart de ces bactéries ne provoquent pas de maladies chez leur hôte, et au contraire contribuent à nous maintenir en bonne santé. Ces bactéries vont former une barrière de protection ou encore remplir des fonctions biologiques, tel que la synthèse de vitamine ou la dégradation des aliments. Dans un autre côté, une faible portion de ces bactéries est considérée comme pathogène et peut causer des maladies diarrhéiques chez l'humain. Ces bactéries pathogènes constituent un problème majeur en santé publique puisque les maladies diarrhéiques représentent une des dix plus grandes causes de mortalité dans le monde (Organisation Mondiale de la Santé, 2018). Dans une majorité des cas, le déclenchement de la maladie résulte de la consommation d'aliments contaminés avec plus de 550 millions de personnes touchées par an et 230 000 morts (Organisation Mondiale de la Santé, 2019). Campylobacter, Vibrio cholerae, Escherichia coli entérohémorragique et
Salmonella figurent parmi les agents bactériens les plus couramment responsables d'infections
alimentaires chez l'homme. Malgré le développement de moyens pour freiner l’apparition de ses agents, ils restent encore aujourd’hui un défi de taille.
À elle seule, Salmonella enregistre plus de 93,8 millions de cas d’infection et 15 000 décès par an dans le monde (Majowicz et al., 2010), faisant d’elle la deuxième cause d’infection alimentaire chez l’homme derrière les norovirus. Parmi les 2 600 sérotypes distingués à ce jour, la manifestation la plus courante de l’infection est la gastro-entérite, mais elle peut également se déclarer sous forme de bactériémie ou de fièvre entérique. Outre l’aspect sanitaire, ces infections génèrent d’importantes pertes économiques au niveau mondial. L’infection à Salmonella résulte principalement de l’ingestion d’œufs ou de poulets contaminés par les sérotypes Enteritidis et Typhimurium, les plus communs. Néanmoins, ces dernières années, les données exposent l’émergence d’infection due à la consommation de fruits et de légumes crus associés à des sérotypes rares provenant de l’environnement (Callejón et al. 2015; Reddy et al. 2016; Public Health Agency of Canada 2010-2017). Ce changement épidémiologique pourrait indiquer une évolution de leur virulence, de leur contenu génomique ou encore être le résultat de la sélection naturelle (Zaidenstein et al., 2010), mais ces souches environnementales restent insuffisamment étudiées jusqu’à présent.
Une fois ingéré en quantité suffisante, de 101 à 1011 bactérie dépendant du sérotype et de l’hôte
grâce à un ensemble de facteurs de virulence. Or, il est connu que chaque souche est fortement influencée par son environnement actuel, mais également par son passé (Lianou et al. 2013; Begley et al. 2015). C’est pourquoi, l’exposition aux nombreuses conditions drastiques du tube digestif (acidité gastrique, sels biliaires, faible concentration en oxygène, peptides antimicrobiens, microbiote résident...) peut avoir un impact sur la survie de la bactérie ainsi que sur la régulation des facteurs de virulence (Álvarez-Ordóñez et al., 2011). La diversité phénotypique qui existe dans l’habilité de
Salmonella à survivre et persister dans le système gastro-intestinal est encore mal comprise, surtout
pour des questions d’éthique et le manque de modèles d’études adaptés. Au regard de ces données, il est nécessaire d’obtenir une meilleure compréhension du comportement de Salmonella dans le tractus gastro-intestinal humain, afin de développer des nouveaux outils de diagnostic ainsi que des traitements préventifs et/ou curatifs. Dans cette thèse, la survie et la virulence de plusieurs souches de Salmonella enterica seront évaluées dans des modèles in vitro dynamiques et/ou multi-compartimentés du tractus gastro-intestinal humain.
Chapitre 1. Revue de littérature
1.1.
Caractéristique des infections à Salmonella
1.1.1. Diversité taxonomique et spectre d’hôte
Faisant partie de la famille des Enterobacteriaceae, la bactérie Salmonella est découverte pour la première fois en 1880 par Ebert et cultivée en 1884 par Gaffky. C’est en 1896 que Widal met en évidence la diversité antigénique des souches de Salmonella grâce à un nouveau test appelé sérodiagnostique, qui permet d'identifier les anticorps spécifiques d'un agent pathogène (Brands, 2005). Au fils du temps et de l’évolution des techniques, c’est finalement la distance génétique mesurée par hybridation d’ADN (Crosa et al., 1973) qui permet de comprendre que la bactérie Salmonella comprend deux espèces reconnues par la « Judicial Commission of the International Committee for Systematics of Prokaryotes » : la première Salmonella bongori et la deuxième Salmonella enterica qui se divise en six sous-espèces: enterica, salamae, arizonae, diarizonae, houtenae et indica (Grimont and Weill, 2007). Ces espèces sont subdivisées en plus de 2 659 sérotypes sur la base de leurs antigènes somatique O, flagellaire H et capsulaire Vi, ce dernier étant retrouvé uniquement chez quelques sérotypes comme Salmonella Typhi ou Paratyphi (Tindall et al., 2005). À l’exception des 22 sérotypes appartenant à l’espèce S. bongori, tous appartiennent à S. enterica avec plus de 1 586 sérotypes regroupés chez la sous-espèce enterica (Issenhuth-Jeanjean et al., 2014).
Alors que les souches de S. enterica ssp. enterica infectent majoritairement les hôtes à sang chaud et sont responsables de plus de 99 % des infections humaines, les autres sous-espèces ainsi que S.
bongori infectent généralement les hôtes à sang froid (Baumler et al., 1998; CDC, 2011). S. enterica
ssp. enterica a la capacité d’infecter une large gamme d’hôtes mais l’adaptation de certains sérotypes à divers hôtes a conduit au développement d’une spécificité vis-à-vis de ces derniers par l’acquisition de facteurs de virulence ou la perte de fonction de gène (Singh, 2013). Ces sérotypes peuvent être classés selon trois types de spectres d’hôtes : (i) les généralistes, tels que les sérotypes S. Typhimurium et S. Enteritidis, qui ont la capacité d’infecter une large gamme d’hôtes, avec des pathologies distinctes dépendamment de l’hôte; (ii) les adaptés à un hôte, tels que les sérotypes S. Dublin (bovins) et S. Choleraesuis (porcs), qui possèdent un hôte préférentiel en déclenchant des infections systémiques chez celui-ci mais peuvent à l’occasion infecter d’autre hôtes de manière moins
grave ou asymptomatique; et (iii) les restreints, tels que les sérotypes S. Typhi et Paratyphi (humain) ou encore S. Gallinarium (volaille), qui n’infectent qu’un seul type d’hôte, en provoquant exclusivement des infections systémiques.
1.1.2. Cycle d’infection
La pathogénèse est l’étude des mécanismes impliqués dans le développement de la maladie. S.
enterica ssp. enterica est un pathogène puisqu’elle a la capacité d’envahir, de se répliquer et de
survivre dans les cellules de l’hôte humain, entrainant divers symptômes. Lorsqu’elles sont ingérées en quantité suffisante, les cellules pathogènes accèdent alors au tractus gastro-intestinal et celles qui survivent au pH acide de l’estomac et aux enzymes du petit intestin atteignent alors leur site d’invasion : l’iléon. Pour échapper à la compétition avec le microbiote en place, la principale stratégie de S. enterica est de traverser la muqueuse intestinale (Finlay, 1994). Les salmonelles interagissent et adhérent aux cellules épithéliales afin de coloniser la muqueuse intestinale (Figure 1-1). La présence de flagelles et de fimbriae sur la surface des cellules bactériennes, ainsi que d’autres facteurs comme les adhésines ou les lipopolysaccharides, contribue à cette étape (Wagner and Hensel, 2011). Cette adhésion facilite par la suite l’invasion des cellules épithéliales par les salmonelles et le passage de la barrière intestinale, via l’intermédiaire des cellules non phagocytaires, c’est-à-dire les entérocytes ou les cellules M. Puisque les cellules M communiquent avec les tissus lymphoïdes afin d’initier une réponse immunitaire, celles-ci peuvent transloquer les microorganismes de l’autre côté de la barrière intestinale. Après invasion, la salmonelle initie une modification complexe chez l’hôte afin de s’internaliser dans un compartiment spécial nommé la vacuole contenant Salmonella (VCS). La VCS permet la vie intracellulaire afin de survivre aux peptides antimicrobiens et à l’oxydation ainsi que de se répliquer efficacement dans la cellule hôte, ce qui est crucial pour la pathogénèse de Salmonella et sa virulence (Swearingen et al., 2012). Les gènes de virulence impliqués dans l’invasion et requis pour la survie intracellulaire sont majoritairement regroupés dans de grandes régions d'ADN chromosomique dénommées îlots de pathogénicité de Salmonella (Grassl and Finlay, 2008). Les cellules bactériennes sont phagocytées par les macrophages en contact avec les cellules M. Dans ces macrophages, elles induisent une réponse inflammatoire et peuvent être détruites par la suite. Même si l’utilisation des cellules M est favorisée par Salmonella, l’apoptose des entérocytes ou l’internalisation par les cellules phagocytaires de l’hôte permettent également de traverser la barrière intestinale (Vasquez-Torres et al., 1999). Occasionnellement, ou pour certains sérotypes adaptés à un hôte, cette infection peut se
généraliser au niveau systémique: les bactéries n’induisent pas de réponse immunitaire et rejoignent la voie sanguine grâce aux macrophages et accèdent à la vésicule biliaire, au foie et à la rate.
Figure 1- 1 Principales étapes de l’infection à Salmonella au niveau de l'épithélium intestinal: adhésion
et translocation à travers les cellules M, invasion directe des cellules épithéliales intestinales et capture par les cellules dendritiques. Adaptée de Sansonetti (2004).
1.1.3. Manifestations cliniques
Les infections à Salmonella varient considérablement selon le sérotype et l’hôte infecté: en pathologie humaine, les souches peuvent être divisées en deux groupes : les salmonelles typhiques et les salmonelles non typhiques (NTS). Les personnes les plus à risques de complications et d’infections graves sont les enfants, les femmes enceintes, les personnes âgées et les personnes immunodéprimées. Lors d’infection humaine, quatre types de manifestations cliniques liées à
Salmonella sont distingués:
(i) La fièvre entérique. Elle est causée par les salmonelles typhiques, soit les sérotypes S. Typhi et S. Paratyphi, et se manifeste au bout d’une longue période d’incubation, située entre 10 et 14 jours. Le risque d'infection est élevé dans les pays en voie de développement où l'accès à une nourriture et à une eau salubre est insuffisant, alors que dans les pays développés, la fièvre se déclare chez les voyageurs (Crump, Luby, and Mintz 2004). Ce type d’infection est invasif
et systémique, avec comme principaux symptômes des maux de tête, des douleurs abdominales, de la diarrhée, ainsi que l’apparition de fièvre faible à forte au bout de deux semaines (Eng et al. 2015). Outre ces symptômes, l’infection peut également provoquer bradycardie, hépatomégalie et splénomégalie, qui se définissent respectivement comme un rythme cardiaque lent, une augmentation du volume du foie et de la rate. Chez certains patients, des complications peuvent survenir dans 10 à 15 % des cas et entraîner des hémorragies, des perforations intestinales, voir le décès (Crump et al. 2015).
(ii) La gastro-entérite ou salmonellose. Elle est causée par les NTS et est la forme la plus courante lors d’infections humaines. La dose infectieuse chez un adulte en bonne santé est de 106 bactéries (Kothary and Babu, 2001) mais lorsque lorsqu’elle est ingérée avec de la nourriture, cette dose peut-être beaucoup plus basse avec environ 100 cellules (Gawande and Bhagwat, 2002). La période d’incubation varie de six à douze heures après ingestion avec l’apparition de frissons, nausée, diarrhée et vomissements comme symptômes les plus courants. En général, les symptômes disparaissent en 72 heures, mais en cas de diarrhée sanglante, ils peuvent perdurer jusqu’à 3 à 7 jours après l’infection.
(iii) La bactériémie. Dans 8 % des cas d’infection à Salmonella, celle-ci passe dans le sang après avoir envahi la barrière intestinale. La bactériémie peut être causée par tous les sérotypes de
Salmonella mais avec une forte prédominance pour ceux adaptés à un hôte comme S. Dublin
ou S. Choleraesuis : aux États-Unis d’Amérique, les sérotypes Enteritidis, Typhimurium, Choleraesuis et Dublin causent des bactériémies à hauteur de 5,7 %, 6,7 %, 56,4 % et 64 % respectivement (Zaidenstein et al. 2010). Le principal symptôme est une forte fièvre mais, sous certaines conditions, un choc septique entrainant la mort peut survenir. Cette septicémie touche le plus souvent les individus immunodéprimés, notamment les adultes infectés par le VIH et les enfants atteint du paludisme, du VIH et de malnutrition (Feasey et al., 2012). La plupart des bactériémies sont le plus souvent attribuables aux NTS (60 %) comparativement aux salmonelles typhiques (Wain et al., 2013).
(iv) L’état asymptomatique. Jusqu’à 10 % des patients atteints de fièvre entérique excrétaient encore la bactérie dans leurs selles durant les 3 mois suivants l’infection, et 1 à 4 % des patients continuaient de l’excréter 12 mois après (Gonzalez-Escobedo et al., 2011). Chez les patients
révélé que l’excrétion au-delà de 12 mois survenait chez 1 % des sujets (Buchwald and Blaser, 1984).
1.1.4. Surveillances épidémiologiques des infections
Les données épidémiologiques des infections associées à Salmonella varient selon le sérotype ainsi que le pays concerné. Comme dit précédemment, la fièvre entérique est généralement détectée dans les pays en voie de développement, alors que les infections aux NTS touchent le monde entier. Aujourd’hui, Salmonella spp. est parmi les 31 agents d’origines alimentaires affichant le plus grand potentiel de déclenchement de maladies intestinales ou systémiques chez l’humain (WHO, 2015) et parmi les quatre principales causes de maladies diarrhéiques (WHO, 2018). Salmonella a même été classée seconde cause mondiale d’infection chez l’humain derrière les norovirus en 2010 (Scallan 2011). En 2017, il a été estimé que les infections aux NTS avait été à l’origine de 95,1 millions de cas et 50 771 décès (GBD 2017 Causes of Death Collaborators, 2018; GBD 2017 Disease and Injury Incidence and Prevalence Collaborators, 2018) et que la fièvre entérique aurait entrainé 14,3 millions de cas pour 135 900 décès (GBD 2017 Typhoid and Paratyphoid Collaborators, 2019) dans le monde. Les chiffes de surveillances à l’échelle mondiale démontrent que les sérotypes S. Enteritidis et S. Typhimurium sont les plus incriminés lors de cas d’infections humaines (WHO, 2018).
Au Canada, Salmonella spp. est classée 4ème cause d’infection derrière les norovirus, Clostridium perfringens et Campylobacter, et elle est responsable de 88 000 cas d’infections alimentaires par
année (5 %). Elle est la deuxième cause d’hospitalisation avec 923 cas (24 %) et troisième cause de mort avec 17 décès (16 %) (Thomas et al., 2015, 2013). Chaque année en santé publique, il est estimé que ces infections à Salmonella spp. coutent environ 1 milliard de dollars CAD/année (Health Canada 2018).
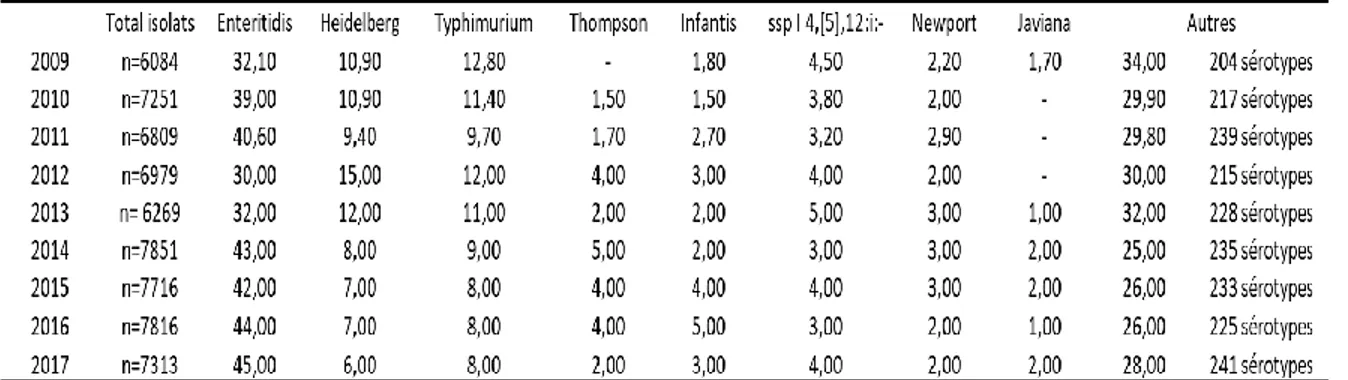
Tableau 1- 1Prévalence des sérotypes de Salmonella responsables de maladies humaines de 2009 à 2017 au Canada (adapté du Programme national de surveillance des maladies entériques de l’Agence de la sante publique du Canada)
Depuis 2009, les sérotypes prédominant lors de ces infections appartiennent à S. enterica ssp. enterica et sont principalement Enteritidis, Typhimurium et Heidelberg (Tableau 1-1). Au cours de ces 10 dernières années, un changement épidémiologique a été observé incluant l’augmentation de la prévalence de souches ainsi que la prévalence d’autre sérotypes (Infantis et Javiana) et la probable émergence de sérotypes rares de S. enterica. En 2009, 204 sérotypes étaient isolés, en plus des 8 sérotypes majeurs, alors que 241 sérotypes étaient isolés en 2017. Ces changements épidémiologiques pourraient être dus à une évolution continue au niveau génomique et contribueraient à l'augmentation de leur virulence et de leur antibiorésistance (Hurley et al., 2014).
1.1.5. Réservoir et transmission
Comme la plupart des Enterobacteriaceae, les mammifères, les reptiles, les amphibiens et les oiseaux sont les principaux réservoirs de Salmonella avec quelques exceptions près. Par exemple, les sérotypes S. Typhi et Paratyphi ne possèdent que l’humain comme réservoir. Via les excréments,
Salmonella peut être transmise par ingestion d’aliments ou d’eau contaminés, par consommation
d’animaux infectés ou par contact direct ou indirect avec des animaux. En effet, plusieurs cas d’infections par contact, via les animaux domestiques ou sauvages, que ce soit en milieu professionnel, en environnement agricole ou familial, ont été reportés dans la littérature scientifique (Rodriguez et al. 2006; Keen et al. 2007; Hoelzer et al. 2011). En plus de ces vecteurs, Salmonella est également capable de survivre et de persister dans un environnement non-hôte, via différents mécanismes, dont la formation de biofilm (Waldner et al., 2012). Dans la plupart des cas d’infections alimentaires, la
crus et la consommation de noix sont de plus en plus rapportées comme sources de contamination (Dewey-Matthia et al., 2018; Reddy et al., 2016). Malgré cette recrudescence, les souches de
Salmonella non-cliniques restent insuffisamment étudiées jusqu’à présent. Pourtant avoir une vue
globale sur toutes les souches de Salmonella permettrait d’apporter une meilleure évaluation des risques liés aux aliments et potentiellement apporter des nouvelles voies de contrôle.
1.2.
Caractérisation du génome de Salmonella
1.2.1. Le pangénome
Il existe aujourd’hui un grand nombre de génomes entiers de bactéries séquencés grâce aux technologies de biologie moléculaire et de bio-informatique. Ces méthodologies sont d’une grande aide en santé publique car elles permettent de caractériser et d’identifier des souches lors d’éclosions (Allard et al., 2012; Okoro et al., 2012; Sabat et al., 2013) et même d’apporter une meilleure compréhension du phénotype d’une souche, comme la spécificité d’hôte de certaines souches de
Salmonella ou encore les symptômes qu’elles déclenchent (Sabbagh et al., 2010). Une comparaison
de ces génomes permet de mettre en évidence des traits communs, comme par exemple entre des espèces d’une même famille comme Escherichia coli et Salmonella, qui sont étroitement liés génétiquement mais aussi d’observer des traits génétiques uniques. Le génome entier de Salmonella est appelé pangénome et est dit « ouvert » car cette dernière est capable d’occuper différents environnements, par l’intermédiaire de plusieurs mécanismes d’échange de matériel génétique. Le pangénome présente deux fractions, soient le génome fondamental et le génome accessoire (Porwollik, 2011) :
(i) La fraction fondamentale représente l’ensemble des familles de gènes présents chez toutes les souches de Salmonella et est impliquée dans la survie et la croissance de la bactérie. Ces gènes sont conservés et présentent généralement une évolution très lente puisqu’ils sont essentiels. (ii) La fraction accessoire représente les familles de gènes uniques à une espèce, à une sous-
espèce, à un sérotype ou même à un type de souche (Desai et al., 2013; Jacobsen et al., 2011). La plupart de ces gènes appartiennent à des ilots génomiques tels que les ilots de pathogénicité de Salmonella, des prophages, des plasmides ou des séquences insérées. Cette fraction accessoire marque de manière importante l’évolution de Salmonella puisque des transferts
horizontaux, ainsi que la recombinaison de gènes associés aux îlots de pathogénicité de
Salmonella et aux plasmides chez S. enterica, sont assez fréquents (Roer et al., 2016). Les
différences phénotypiques dans les lignées de Salmonella proviennent souvent de cette fraction accessoire.
1.2.2. Facteur de virulence
Salmonella a acquis dans son génome des facteurs de virulence, par transfert horizontal (Figure 1-2).
L’acquisition d’îlots de pathogénicité, d’opérons fimbriaires, de flagelles et de plasmides a conféré avantageusement à la bactérie son caractère virulent. La sécrétion de ces protéines, a permis à
Salmonella de survivre chez l'hôte, ou encore de persister plus longtemps et se disperser avec plus de
succès.
Figure 1- 2 Facteur de virulence de S. enterica ssp. Enterica. Adaptée de de Jong et al. (2012)
1.2.2.1. Les îlots de pathogénicité
Les îlots de pathogénicité (ou SPIs pour Salmonella Pathogenicity Islands) sont des blocs d’éléments génétiques mobiles sur le chromosome associés à l’adaptation à l’hôte, à son invasion et à la survie du pathogène chez l’hôte. Ils jouent un rôle important dans la virulence de la bactérie puisque ces îlots
littérature. Certains de ces SPIs codent pour le système de sécrétion de type III (SST3) qui fonctionne comme une seringue moléculaire et injecte les protéines du SST3 dans le cytosol de la cellule cible. Ces îlots codent également pour des effecteurs qui vont contribuer à l’invasion des cellules cible ou la survie intracellulaire de Salmonella, via des cascades signalétiques. Parmi les 23 SPIs, les SPIs 1 à 5 sont considérés comme les plus importants au regard de la virulence de Salmonella et sont également les plus décrits (Hensel, 2004; Hurley et al., 2014):
(i) SPI-1: c’est un locus chromosomique de 40 kb, qui code pour le SST3-1 afin d’induire un réarrangement du cytosquelette d'actine de la cellule hôte pour l’invasion de l'épithélium intestinal via les cellules non-phagocytaires (Que et al. 2013). Il participe également à la prolifération dans la VCS et à l’apoptose des macrophages. Il est retrouvé chez les espèces S. enterica et S. bongori et est conservé chez toutes les souches, bien que quelques variations soient notées parmi des isolats environnementaux (McWhorter and Chousalkar, 2015).
(ii) SPI-2: ce locus de 40 kb code pour le SST3-2. Il est requis pour la survie et la réplication intracellulaire de Salmonella dans les VCS et les macrophages, ainsi que pour les infections systémiques. SPI-2 est présent en deux portions : une de 25 kb qui est retrouvée seulement chez
S. enterica et qui est essentielle pour les infections systémiques, et une autre portion de 15 kb
présente chez S. bongori et S. enterica mais qui ne code pas pour le SST3 (Hensel et al. 1999). L’induction du SST3-2 est médiée par le système à deux composants phoP/phoQ (Deiwick et al., 1999) et est mis en place grâce aux gènes encodant l’aiguille (gènes ssa) et les régulateurs (gènes
ssrAB).
(iii) SPI-3: ce locus de 36 kb joue un rôle dans la survie intra-macrophage et la croissance dans un milieux pauvre en 𝑀𝑔2+ grâce à l’opéron mgtBC. Le SPI-3 code également pour l’adhésine MisL
impliquée dans la colonisation intestinale (Dorsey et al., 2005). Tandis que SPI-1 et SPI-2 sont conservés, l’îlot SPI-3 est présent en différentes versions chez les souches de Salmonella par l’intermédiaire de délétions et d’insertions (Jacobsen et al., 2011; Suez et al., 2013).
(iv) SPI-4: ce locus de 24 kb module l'adhérence aux cellules épithéliales, via le gène d’adhésion siiE et l’opéron siiABCDEF, et codant pour le SST1 (Wille et al. 2014; Kiss, Morgan et al. 2007). Le SST1 transloque les protéines effectrices ainsi que l’injection des adhésines dans la cellule hôte. Il pourrait également jouer un rôle dans la sécrétion de toxine ainsi que la survie intra-macrophage.
(v) SPI-5: ce locus de 8 kb semble associé à la pathogenèse et plus spécifiquement à l’inflammation, notamment en codant pour sopB (sécrété via SST3 de SPI-1) et pipB (par SST3 de SPI-2 à la VCS).
L’acquisition de certains fragments de SPI-7, qui était auparavant spécifique à S. Typhi, par S. Dublin et S. Paratyphi C (Jacobsen et al., 2011) ou encore de SPI-18, lui aussi spécifique auparavant à S. Typhi, par S. Montevideo (Suez et al., 2013) montre que ces ilots évoluent encore aujourd’hui. La diversité génétique de ces SPIs peut-être ainsi être associée au degré de sévérité du phénotype de
Salmonella comme la capacité d’invasion (Suez et al., 2013), l’hypervirulence d’une souche (Le Bars
et al., 2012), ou encore le potentiel d’une souche environnementale à infecter l’homme (Mcwhorter and Chousalkar, 2015). Ces associations entre les SPIs et les différentes formes phénotypiques de
Salmonella enterica ne sont pas encore totalement établies et restent à définir.
1.2.2.2. Flagelle
Le flagelle permet à la bactérie d’être motile et contribue à la virulence du pathogène en jouant un rôle important dans l’adhésion de la bactérie aux cellules hôtes. Les souches avec des flagelles paralysés, manquants ou dysfonctionnels ont présenté une diminution de leur capacité à infecter les cellules de l’hôte (Achouri et al., 2015). Elles sont considérées moins infectieuses en raison de leurs capacités limitées à initier un contact avec les cellules hôtes, par rapport à des souches de type sauvage (Porwollik, 2011). Chaque bactérie possède entre six à huit flagelles distribués à sa surface et leur synthèse nécessite l’expression de plus d’une cinquantaine de gènes distribués sur 17 opérons (Chilcott and Hughes, 2000). Salmonella possède deux antigènes flagellaires distincts, FliC et FljB, qui sont régulés de manière coordonnée de sorte qu'un seul antigène flagellaire est exprimé à tout moment, processus appelé «variation de phase» (Silverman, 1980). Cette variation pourrait jouer un rôle pour échapper au système de défense de l’hôte mais son rôle précis reste encore inconnue (Prozorov, 2001). Toutefois, il a été démontré que la phase FliC donne un avantage sélectif pour l’infection des souris, mais n’accorde aucun avantage concernant l’invasion des cellules épithéliales ou l’induction de l’entéropathogénicité de S. enterica.
1.2.2.3. Fimbriae
Les fimbriae sont des structures présentes à la surface des bactéries, principalement composées de protéines hélicoïdales. Elles jouent un rôle dans la formation de biofilm, l’adhésion aux cellules hôte et le déclenchement de la réponse pro-inflammatoire. Chez S. enterica, la grande variabilité dans la distribution phylogénétique de ces opérons fimbriaires semble indiquer qu’une association existe entre la possession d’un large répertoire fimbriaire et la colonisation de plusieurs espèces d’hôtes (Porwollik, 2011). La présence de pseudogènes dans les opérons fimbriaires entrainerait une spécificité d’hôte comme chez les sérotypes S. Typhi, Paratyphi et Gallinarium associés à des infections systémiques. Environ 12 opérons fimbriaires sont recensés par souche chez S. enterica, composés de quatre à quinze gènes, avec les opérons bcf, csg, fim, sth, stb,std,stc et peg retrouvés chez 90 % des souches de Salmonella (Dufresne and Daigle, 2017). Malgré les nombreux opérons fimbriaires dans les génomes de Salmonella, leurs fonctions sont très peu comprises, car les génomes s’expriment mal dans des conditions de croissance standard en laboratoire (Nuccio and Baumler, 2007). Les fimbriae de type 1, FimA et FimH, les plus étudiés à ce jour, joueraient un rôle dans l’adhésion via l’adaptation à un hôte. Par exemple, les variations de l’allèle FimH chez S. Newport permettraient de distinguer les souches capables d’infecter les bovines des autres souches (Yue et al., 2012). Les variants hautement adhésifs FimH, associés aux sérotypes invasifs, seraient capables de discriminer les cellules eucaryotes alors que les variants faiblement adhésifs FimH, associées aux gastro-entérites, ne font pas la distinction (Grzymajlo et al., 2010; Kisiela et al., 2012).
1.2.2.4. Plasmide et phage
La diversité génétique de Salmonella est principalement due à l’intégration de plasmides, de bactériophages ou de phages dans son génome. Le génome plasmidique est connu pour subir de fortes variations, grâce à la perte et au gain de gènes accessoires, ce qui serait la cause de la diversité phénotypique des Salmonella, ainsi que leur diversité de virulence (Williams et al., 2013). Le plasmide de virulence pSLT d’environ 90 kB portant l’opéron spv, qui participe à la survie intracellulaire et la virulence chez la souris, est retrouvé chez les NTS mais est absent chez S. Typhi (Sabbagh et al., 2010). Des plasmides de multirésistance aux antibiotiques sont également fréquemment retrouvés dans le génome plasmidique de Salmonella et auraient facilité l’émergence de plusieurs sérotypes comme S. Typhimurium, S. Heidelberg et S. Virchow ces dernières années (de Jong et al. 2012; Gal-Mor et al. 2014). Cette émergence pourrait aussi être associée à d’autre effecteurs de la virulence,
encodés dans des éléments phagiques comme les effecteurs du SST3-1 SopE, SopE2 et la superoxyde dismutase SodC-1. Ces effecteurs, non nécessaires à la virulence, sont retrouvés chez les sérotypes les plus communs tels que S. Dublin, S. Enteritidis, S. Heidelberg et S. Newport (Suez et al., 2013). L’intégration de ces plasmides et phages dans le génome pourrait donc donner un avantage sélectif à certains sérotypes et participer à leur émergence (Kuijpers et al., 2019).
1.2.2.5. Lipopolysaccharide (LPS)
Les LPS sont des composants majeurs de la membrane externe des bactéries Gram négatif et protègent la bactérie des composés toxiques tels que les antibiotiques ou les sels biliaires (Hernández et al., 2012). Ils sont composés de trois parties : le lipide A, le noyau oligosaccharide et la chaine externe O polysaccharide. les LPS contribuent également à la virulence de la bactérie: le lipide A contient l’endotoxine responsable du choc septique chez les hôtes et plusieurs travaux décrivent le rôle des LPS dans l’adhésion de Salmonella aux cellules hôtes ainsi que l’activation de la réponse inflammatoire (Bravo et al., 2011; Kong et al., 2011). La structure des LPS peut être modifiée afin d’améliorer la survie de la bactérie selon les environnements via par exemple l’opéron pmr et le régulateur PhoP-PhoQ (Gunn, 2008).
1.2.2.6. Régulation des facteurs de virulence
La plupart de ces facteurs de virulence sont contrôlés par des systèmes à deux composants. Ces systèmes, à la suite de la perception d’un signal environnemental reçu directement ou par le biais d’un intermédiaire, vont se phosphoryler (Mascher et al., 2006). Cette réaction va constituer un intermédiaire riche en énergie pour l’activation ou la répression de la transcription des gènes sous leur contrôle. Ces systèmes se composent d’une protéine transmembranaire, à titre de senseur, et une protéine cytoplasmique, à titre de régulateur de réponse (Stock et al., 2000). Les signaux perçus peuvent être un changement de température, de pH, de pression d’oxygène, de concentration d’ions, d’osmolarité, de nutriment, de potentiel redox, ou encore la présence de molécules antimicrobiennes ou du quorum
sensing (Beier and Gross, 2006; Laub and Goulian, 2007). Ces systèmes sont, donc, impliqués dans
l’adaptation aux différentes conditions rencontrées par la bactérie, comme celle-ci rencontré chez l’hôte. Le plus détaillé jusqu’à présent est le système PhoP/PhoQ, avec un rôle-clé dans le contrôle de la virulence et la résistance aux peptides antimicrobiens (Miller et al., 1989). Ce système active ou réprime les gènes pag ou prg impliqué dans la synthèse des protéines de la membrane externe de la
bactérie, suite à la présence d’Un pH acide, une faible concentration en Mg2+ et la présence de peptides
antimicrobiens (Spector and Kenyon, 2012). Un autre système à deux composants majeurs est le système OmpR/EnvZ situé sur la membrane interne. Il répond quant à lui au changement d’osmolarité et au pH acide, en régulant la production des porines majeurs de la membrane externe (Chakraborty and Kenney, 2018). Le système AcrAB est un régulateur global de la bactérie autant en conditions aérobiques qu’anaérobiques. Il est associé à la coordination de l’expression des gènes du métabolisme cellulaire, à la biosynthèse des flagelles et à la motilité (Evans et al., 2011; Morales et al., 2013). Parmi les autres systèmes à deux composantes discuter dans la prochaine section (1.3), on retrouve également le système Rcs impliqué dans la modification des LPS (Delgado et al., 2006) ou encore QseCB, activateur de la motilité (Bearson and Bearson, 2008).
1.2.3. Relation diversité génomique et phénotypique
La caractérisation des souches pour la surveillance et l’investigation de toxi-infections reliées à
Salmonella par typage moléculaire étaient effectuées jusqu’à ces dernières années par Électrophorèse
sur Gel à Champ Pulsé (PFGE), considérée comme le « gold-standard », ou encore par Multi Locus Variable-Numbers tandem-repeat Analysis (MLVA), Multi Locus Sequence Typing (MLST) et d’autres (Foley et al. 2007; Ruppitsch 2016). Ces techniques discriminantes ont toutefois démontré qu’il était parfois difficile de discriminer des souches à l’intérieur d’un même sérotype (Young et al. 2012) dû à une grande homogénéité génétique chez S. enterica.
Ces données génomiques ont cependant permis de comprendre certains mécanismes impliqués dans la variation des phénotypes de S. enterica : même si les souches de S. enterica sont génétiquement proches, leurs capacités à adhérer, envahir et survivre chez l’hôte et/ou dans l’environnement varient de manière hétérogène aussi bien au niveau inter-sérotype qu’intra-sérotype (Rosselin et al., 2011; Suez et al., 2013). Des expériences par hybridation génomique comparative (Comparative Genomic
Hybridization ou CGH) ont ainsi pu distinguer que l’absence d’éléments prophagiques et de certains
gènes de transport d’acide aminés chez S. Enteritidis ont pu rendre des souches moins compétitives dans le tractus gastro-intestinal et avoir un impact sur leur prévalence (Betancor et al., 2009; Pan et al., 2009). De la même manière, certains facteurs de virulence correspondant à S. Typhi C18 (fimbriae, lipoprotéines et régulateurs) étaient présents chez des souches invasives de S. Heidelberg, mais étaient absents des souches gastro-intestinales de S. Typhimurium (Bronowski and Winstanley, 2009).