Université de Sherbrooke
Oligonucléotides comme modulateurs de l'expression génique
Par
Samuel Rouleau Programme de biochimie
Thèse présentée à la Faculté de médecine et des sciences de la santé en vue de l’obtention du grade de philosophiae doctor (Ph.D.)
en biochimie
Sherbrooke, Québec, Canada Août, 2017
Membres du jury d’évaluation
Jean-Pierre Perreault, Département de biochimie Martin Bisaillon, Département de biochimie
Pascale Legault, Département de biochimie et médecine moléculaire, Faculté de médecine, Université de Montréal
François Boudreau, Département d’anatomie et de biologie cellulaire Éric Massé, Département de biochimie
faits pour moi, c’est ce qui m’a permis de me rendre aussi loin et d’accomplir tout le travail présenté ici.
Oligonucléotides comme modulateurs de l'expression génique Par
Samuel Rouleau Programme de biochimie
Thèse présentée à la Faculté de médecine et des sciences de la santé en vue de l’obtention du diplôme de philosophiae doctor (Ph.D.) en biochimie, Faculté de médecine et des sciences de la santé, Université de Sherbrooke, Sherbrooke, Québec, Canada, J1H 5N4 L’ARN est sans aucun doute la molécule biologique la plus versatile qui soit. Tout comme l’ADN, il peut contenir et transmettre de l’information génétique. Tout comme les protéines, il peut accomplir une multitude de fonctions biologiques. De plus, son rôle le plus connu demeure celui d’intermédiaire entre l’ADN et les protéines. L’ARN est donc au cœur d’un bon nombre de processus biologiques. Ceci lui confère un immense potentiel thérapeutique qui jusqu’à présent demeure largement inexploité. Pour accomplir ses fonctions, l’ARN doit adopter une structure tridimensionnelle précise qui est dépendante à la fois de sa séquence et de son environnement. Ainsi, en modifiant la structure d’un ARN, il est possible d’en moduler sa fonction. C’est l’objectif global des travaux présentés dans cette thèse. Pour y parvenir, de courts oligonucléotides antisens (OA) ont été utilisés. Cette stratégie revêt plusieurs avantages. Comme les OA s’apparient à leur cible en formant des paires de bases Watson-Crick, ils offrent une grande spécificité et leur design est facile. De plus, en se fiant aux données structurales et aux logiciels de prédictions de structures des ARN, on peut aisément identifier les régions à cibler avec les OA. Enfin, cette technique est versatile puisqu’on peut cibler différents motifs d’ARN. La première cible a été le ribozyme du virus de l’hépatite D. Cet ARN, qui catalyse une réaction d’auto-coupure, a été modifié afin que son activité devienne dépendante à la liaison d’OA. Plusieurs modules ont ainsi été créés et combinés afin d’obtenir des ribozymes qui répondaient à la présence d’un ou plusieurs OA. En insérant ces interrupteurs moléculaires dans les régions non traduites d’un ARNm, nous avons ainsi modulé l’expression de ce gène avec les OA. Cet outil a des applications intéressantes pour la régulation de gènes en biologie synthétique. Un autre motif ciblé a été le G-quadruplex (G4). Cette structure non canonique exerce de nombreuses fonctions biologiques et représente donc une cible thérapeutique intéressante. Lorsque présent dans la région 5’ non traduite d’un ARNm, le G4 mène généralement à une diminution de la traduction. En utilisant des OA qui empêchent la formation du G4, nous avons été en mesure d’augmenter la traduction du gène ciblé. De plus, il a été possible de développer des OA qui favorisent la formation d’un G4 dans le but de diminuer l’expression de la cible. Finalement, dans le dernier chapitre de cette thèse, il est démontré que les G4 présents dans les microARN primaires influencent leur maturation en microARN matures. Des OA ciblant ces G4 ont été utilisés afin de favoriser la maturation de microARN suppresseurs de tumeurs, ce qui présente un potentiel thérapeutique intéressant. En bref, les travaux présentés dans cette thèse démontrent clairement que les OA sont un outil de choix pour cibler et modifier la structure de motifs d’ARN spécifiques. Mots clés : ARN, oligonucléotide antisens, ribozyme VHD, G-quadruplex.
Summary
Oligonucleotides as Gene Expression Modulators By
Samuel Rouleau Biochemistry Program
Thesis presented at the Faculty of medicine and health sciences for the obtention of Doctor degree diploma philosophiae doctor (Ph.D.) in Biochemistry, Faculty of medicine and
health sciences, Université de Sherbrooke, Sherbrooke, Québec, Canada, J1H 5N4 RNA is a versatile biological molecule. Like DNA, it can contain and transmit genetic information. Like proteins, it can accomplish multiple biological functions. Also, its most known role remains that of intermediary between DNA and proteins. RNA is thus a key player in many biological processes. This gives it an immense therapeutic potential which remains largely untapped. To fulfill its functions, RNA must adopt a precise three-dimensional structure that is dependent on both its sequence and its environment. Thus, by modifying the structure of an RNA, it is possible to modulate its function. This is the overall objective of the work presented in this thesis. To achieve this, small antisense oligonucleotides (ASO) have been used. This strategy has several advantages. As ASO bind their target with Watson-Crick base pairs, they offer great specificity and their design is easy. Moreover, reliance on structural data and RNA structure prediction softwares makes it easy to identify the regions to be targeted with ASO. Finally, this technique is versatile since it is possible to target different RNA motifs. The first target was the HDV self-cleaving motif. This RNA, which catalyzes a self-cleaving reaction, has been modified so that its activity became dependent on the binding of ASO. Several modules were thus created and combined in order to obtain ribozymes which responded to the presence of one or more ASO. By inserting these molecular switches into an mRNA’s UTR, the expression of this gene was modulated with the ASO. This has interesting applications for the regulation of genes in synthetic biology. Another target motif was the G-quadruplex (G4). This non-canonical structure exerts many biological functions and therefore represents an interesting therapeutic target. When present in the mRNA’s 5’UTR, G4 generally lead to a decrease in translation. Using ASO that prevent G4 formation, we were able to increase the translation of the target gene. In addition, it has been possible to develop ASO which promote the formation of a G4 in order to decrease the expression of the target. Finally, in the last chapter of this thesis, it is demonstrated that the G4 present in the primary microRNAs influence their maturation in mature microRNAs. ASO targeting these G4 have been used in order to promote the maturation of tumor suppressor microRNAs, which has an interesting therapeutic potential. The work presented in this thesis clearly demonstrates that ASO are ideal for targeting and altering the structure of specific RNA motifs.
Résumé ... iv
Summary ... v
Table des matières ... vi
Liste des figures ... viii
Liste des tableaux ... x
Liste des abréviations ... xi
Introduction ... 1
1. La relation structure-fonction des ARN ... 1
2. Hypothèse de recherche ... 5
3. Les oligonucléotides antisens ... 5
4. Utiliser les oligonucléotides pour cibler des motifs structuraux ... 13
Résultats ... 34
Chapitre 1 : Développement de ribozymes VHD allostériques régulés par des oligonucléotides 34 Abstract ... 35
Introduction ... 35
Results and discussion ... 37
Methods ... 43
Supplementary informations ... 46
References ... 53
Chapitre 2: Moduler la traduction d’ARN messagers spécifiques en ciblant les G-quadruplexes 55 Abstract ... 56
Introduction ... 56
Materials and Methods ... 57
Results ... 62
Discussion ... 76
Supplementary informations ... 78
References ... 92
Chapitre 3 Moduler la biogénèse de microARN spécifiques en ciblant les G-quaduplexes ... 96
Abstract ... 97
Introduction ... 97
Results ... 99 Discussion ... 110 Supplementary Figures ... 115 References ... 123 Discussion ... 126 1. Cibler le ribozyme VHD ... 126
2. Cibler les G4 impliqués dans la traduction ... 128
3 Cibler les G4 impliqués dans la maturation des pri-miARN ... 130
4 Optimisation des OA ... 130 5. Applications potentielles ... 134 Conclusion ... 134 Remerciements ... 136 Références ... 137 Annexes ... 149
Introduction
Figure 1. Les différents mécanismes d’action des oligonucléotides antisens………7
Figure 2. Les modifications chimiques des oligonucléotides antisens...………...9
Figure 3. Utilisation des oligonucléotides pour modifier la structure de la cible…………13
Figure 4. Le ribozyme VHD……….15
Figure 5. Le G-quadruplex………18
Figure 6. Fonctions des G-quadruplexes dans les ARNm…...……….24
Figure 7. La biogénèse des microARN………...………27
Figure 8. Induction de G-quadruplex par un oligonucléotide………...………30
Résultats Chapitre 1 Figure 1 The HP18 ribozyme...……….35
Figure 2 Regulation of luciferase activity within HEK 293 cells……….38
Supplementary Figure 1. The Not and Nor gates………..43
Supplementary Figure 2. The loop IV YES gate………..44
Supplementary Figure 3. The AND gate……….……..45
Supplementary Figure 4. The OR gate……….……….46
Chapitre 2 Figure 1 Schematic representation of the oligonucleotide-based strategy to inhibit G4-structure formation…………..………....60
Figure 2 Characterization of the long-loop-2 Artificial G4...63
Figure 3 Characterization of naturally-occurring H2AFY G4………..66
Figure 4 Promoting G4 folding……….70
Figure 5 Targeting endogenously-expressed H2AFY………...72
Supplementary Figure 1. Comparative circular-dichroism analysis of G-Quadruplexes….75 Supplementary Figure 2. Anti-ArtG4 cannot disrupt pre-folded ArtG4………..76
Supplementary Figure 3. Modulation of G4 folding does not affect mRNA levels….…...77
Supplementary Figure 4. Dose response of the ArtG4 to Anti-ArtG4 ASO………78
Supplementary Figure 6. Structural context of the H2AFY G4………...80
Supplementary Figure 7. Characterization of the naturally-occurring AkG4………..81
Supplementary Figure 8. DsG4 structure……….………….82
Chapitre 3 Figure 1. Predicted secondary structures for pri-mir200C, pri-mir451a, and pri-mir497…98 Figure 2. RTS assays………...100
Figure 3. pri-miRNA overexpression assays………...………….…………..103
Figure 4. Modulating miRNA expression with ASO………..105
Figure 5. Screening for pri-miRNAs possessing a G4………107
Supplementary Figure 1. In line probing of the pri-miRNAs……….110
Résultats Chapitre 1
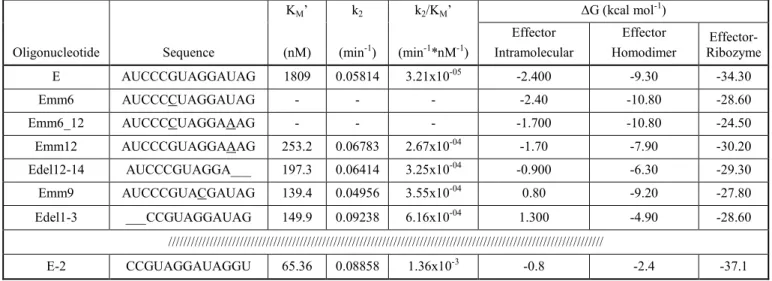
Table 1. Kinetic and thermodynamic parameters of the effector oligonucleotides for the HP18 ribozyme……….………..37 Supplementary Table 1. Ribozymes construction….……….………..47 Chapitre 2
Supplementary Table 1. G4 ASO………..………..83 Supplementary Table 2. Primers used for q-PCR………..………..84 Supplementary Table 3. Thermal denaturation analysis………..………85 Chapitre 3
Liste des abréviations 2’F 2’OMe 2’OMOE ARNm ARNt DMS G4 IRES LNA lncRNA miARN OA OH ORF PMO PNA pré-miRNA pri-miARN RNase SHAPE siRNA SOFA snRNA SSO TERC TERRA 2’-Fluore 2’-O-methyl 2’-O-methoxyethyl ARN messager ARN de transfert
Sulfate de dimethyl, Dimethyl Sulfate G-quadruplex
Site interne de liaison au ribosome, Internal Ribosome Entry Site Acide nucléique vérouillé, Locked Nucleic Acid
Longs ARN non codants, Long Non Coding RNA microARN
Oligonucléotide antisens Groupement hydroxyle
Cadre de lecture ouvert, Open Reading Frame Phosphorodiamade Morpholino Oligonucleotide Acide nucléique peptidique, Peptidic Nucleic Acid microARN précurseur, precursor microRNA microARN primaire, primary mircroRNA Ribonucléase
Analyse de l’acétylation sélective du 2’hydroxyl par extension d’amorce, Selective 2’Hydroxy Acetylation by Primer Extension Petit ARN interférant, small interfering RNA
Specific On-oFf Adaptor
Petits ARN nucléaire, Small Nuclear RNA
Oligonucléotide modificateur d’épissage, Splice Switching Oligonucleotide
ARN composant de la télomérase, Telomerase RNA Component ARN des répétitions télomériques, Telomeric Repetition Contaning RNA
VE VEB VHC VHD VHS VIH VPH UTR Virus Ebola Virus Epstein-Barr Virus de l’hépatite C Virus de l’hépatite D Virus Herpes Simplex
Virus de l’immunodéficience humaine Virus du papillome humain
1. La relation structure-fonction des ARN
L’un des principes de base de la biologie est la corrélation entre la structure et la fonction (Campbell and Reece 2002). En effet, l’anatomie d’un organisme et de ses tissus nous en dit long sur ses capacités. Par exemple, les ailes d’un oiseau sont idéales pour voler. Ce concept est applicable tant au niveau macroscopique que microscopique et même moléculaire.
Ainsi, la structure des acides nucléiques est une partie primordiale de la biologie moléculaire. En effet, cette branche moderne de la science a sans aucun doute été mise à l’avant-plan par la découverte de la double hélice d’ADN par Watson et Crick (Watson et Crick 1953). Cette double hélice, qu’on a plus tard classifié comme étant l’hélice B d’ADN, est la structure principalement adoptée par l’ADN dans la cellule bien que d’autres structures telles que l’hélice Z et le motif G-quadruplex (G4) ont pu être observées pour l’ADN in vivo (Du et Zhou 2013).
Dans la nature, l’ARN possède une plus grande diversité structurale que l’ADN (Batey et al. 1999). La présence du groupement hydroxyle (OH) sur le carbone 2 du ribose lui confère des propriétés structurales uniques. L’ARN n’est ainsi pas limité aux paires de bases Watson-Crick, 38 paires de bases possibles ont été répertoriées avec au moins deux ponts hydrogène (Lemieux et Major 2002; Aylett et Ban 2017; Mellin et Cossart 2015). De plus, en cellule, l’ADN est presque toujours en présence d’un brin complémentaire, ce qui favorise l’appariement des deux brins ensemble et l’adoption de la double hélice. L’ARN, lui, n’a généralement pas de brin complémentaire, ce qui lui permet d’adopter plus librement d’autres structures. Plusieurs structures d’ARN ou d’hybride ARN/ADN sont possibles : double hélice, triple hélice, pseudo-nœud, kissing loop, quadruplex, motif A mineur, ribose zipper, etc. (Hendrix et al. 2005).
Tous ces motifs sont un peu comme les ‘‘blocs de construction’‘ qui servent à bâtir la structure globale d’un ARN donné. La finalité de cette structure dépend de plusieurs facteurs : la séquence de l’ARN, les modifications chimiques qui lui sont apportées, la température, le pH, la présence d’ions métalliques, la liaison de métabolites ou de
structures distinctes. Différentes conditions cellulaires peuvent venir influencer l’équilibre et la proportion d’ARN qui adoptent une structure particulière (Bevilacqua et al. 2016). Comme il existe une étroite relation entre la structure et la fonction d’une molécule, en changeant la structure d’un ARN, on affecte aussi sa fonction. On peut donc ainsi l’empêcher d’exercer sa fonction normale ou même lui en conférer une nouvelle qui est toute autre.
1.1 Fonctions de l’ARN dépendantes de la structure
La nature a su profiter de cette réalité à de nombreuses occasions. En effet, l’adoption ou non d’une bonne structure est un moyen de réguler toute les fonctions et étapes de maturation de la plupart sinon tous les ARN. On n’a qu’à penser aux ARN ribosomaux et de transfert (ARNt), qui sont hautement modifiés : méthylation, pseudourydylation, acétylation, etc. (Sharma et Lafontaine 2015; Maraia et Arimbasseri 2017). Ces modifications permettent à ces ARN d’adopter une structure tertiaire adéquate à leurs fonctions dans la traduction des ARN messagers (ARNm). De plus, elles servent de points de contrôle de la qualité dans la maturation des ribosomes et des ARNt.
1.1.1 Ribo-régulateurs
Un autre exemple est l’utilisation des riboswitch ou riborégulateurs. Surtout présents dans la région 5’ non traduite (UTR) des ARNm procaryotes, les riborégulateurs sont composés de deux parties : un aptamère et une plateforme d’expression (Wachter 2014). L’aptamère permet la reconnaissance et la liaison d’un métabolite spécifique. La liaison du métabolite vient stabiliser une structure particulière de l’aptamère, ce qui entraîne un changement de structure dans la plateforme d’expression. Dépendamment des cas, ce changement dans la plateforme d’expression entraîne une inhibition ou une activation de la transcription et/ou de la traduction et/ou de la dégradation de l’ARNm (Beaudoin et Perreault 2013). Ce genre de système est le plus souvent utilisé pour réguler les gènes impliqués dans l’homéostasie du métabolite reconnu par le riborégulateur.
Un autre système très similaire, mais qui n’est pas un riborégulateur dans la définition stricte du terme, est le thermorégulateur (Tucker et Breaker 2005). Le principe est le même excepté qu’il n’y a pas d’aptamère. C’est plutôt un changement de température
qui fait varier la structure de la plateforme d’expression et vient ainsi réguler l’expression de l’ARNm.
1.1.2 Structures qui affectent la traduction chez les eucaryotes
Les eucaryotes ne sont pas en reste. La traduction de leurs ARNm peut elle aussi être grandement affectée par la structure de ceux-ci. De nombreux ARNm viraux, de même que certains ARNm cellulaires, utilisent une structure dite Internal Ribosome Entry Site (IRES) pour être traduits de manière indépendante de la coiffe (Terenin et al. 2017). Il est connu que normalement, la structure coiffe est essentielle au recrutement de facteurs d’initiation de la transcription, tel qu’eIF4E, qui à leur tour vont recruter la petite sous-unité du ribosome, qui pourra ainsi commencer à parcourir l’ARNm à la recherche d’un codon d’initiation (Aylett et Ban 2017). Les structures IRES permettent de subjuguer la fonction de l’un ou plusieurs de ces facteurs et ainsi de recruter le ribosome et initier la traduction de manière indépendante de la structure coiffe.
Si les structures IRES favorisent la traduction, de nombreuses autres structures ont plutôt l’effet inverse. En effet, il a été démontré que des mutations qui stabilisent les structures secondaires dans la région 5’UTR mènent à une diminution de la traduction (Svitkin et al. 2001). De surcroît, l’insertion d’aptamères liant le colorant Hoechst dans le 5’UTR d’un gène rapporteur induit une diminution de l’expression du rapporteur d’une manière dépendante de la concentration d’Hoechst (Werstuck et Green 1998). Finalement, il a également été démontré que la présence d’une structure G4 dans le 5’UTR d’un ARNm mène généralement à une diminution de sa traduction (Beaudoin et Perreault 2010; Bugaut et Balasubramanian 2012; Bennett et Swayze 2010). Dans tous ces cas, plus les structures secondaires et tertiaires adoptées par les 5’UTR sont stables, moins l’ARNm est traduit. Ceci s’explique par le fait que la petite sous-unité du ribosome doit parcourir l’ARNm à la recherche du codon d’initiation. Le ribosome utilise alors des hélicases pour déplier ces structures et avancer le long de l’ARNm. Ainsi, plus les structures sont stables, plus il est difficile pour la petite sous-unité de passer au travers, ce qui résulte en moins de ribosomes complets formés au codon d’initiation et donc moins d’ARNm pouvant être traduits.
1.1.3 La maturation de l’ARN
La traduction n’est pas le seul processus pouvant être affecté par la structure de l’ARN. En effet, à peu près toutes les étapes de la vie d’un ARNm peuvent être régulées de façon dépendante de la structure. L’épissage et la polyadénylation sont deux processus-clés dans la maturation d’un ARNm (Millevoi et Vagner 2010; Lee et Rio 2015). De plus, ces deux phénomènes peuvent se produire à plusieurs endroits sur la plupart des messagers, ce qui donne la possibilité à un même gène de produire plusieurs isoformes. Des structures telles que les G4 peuvent influencer et réguler l’épissage (Marcel et al. 2011; Perriaud et al. 2014; Ribeiro et al. 2015) ou la polyadénylation alternative. D’autre part, de nombreuses protéines influencent grandement ces processus et leur action est dépendante de la liaison de ces protéines à l’ARNm. Dans certains cas, des protéines aux fonctions antagonistes vont entrer en compétition pour la liaison à un même site. Il est connu que la structure de l’ARNm influence la liaison de beaucoup de ces facteurs (Li et al. 2010; Li et al. 2014).
1.1.4 L’interférence par l’ARN
Une autre façon de réguler l’expression des gènes de manière dépendante de l’ARN est l’ARN interférence. Ce phénomène a été décrit par Craig Mello et Andrew Fire (Fire et al. 1998). D’abord considéré comme un évènement inusité présent seulement chez le ver C. elegans, on s’est vite rendu compte qu’il s’agissait d’un très important mécanisme de régulation de l’expression génique conservé à travers tous les organismes supérieurs (Mello et Conte 2004). L’appariement de petits ARN, small interfering RNA (siRNA), ou microARN (miARN) à l’ARNm, favorisé par les protéines de la famille Ago, mène à une répression de la traduction et/ou à une dégradation de l’ARNm. Les petits ARN peuvent être parfaitement (siRNA) ou partiellement (miARN) complémentaires à leur cible. Les ARN interférents endogènes ciblent généralement la partie 3’UTR des ARNm (Bartel 2009), bien que des liaisons dans le 5’UTR (Ørom et al. 2008; Lee et al. 2009) et la région codante (Reczko et al. 2012) ont aussi été rapportées.
Dans tous les cas, il a été démontré que la disponibilité de la région appariée par le petit ARN est d’une grande importance (Schubert et al. 2005; Kertesz et al. 2007; Afonso-Grunz et Müller 2015). En effet, quand les régions ciblées sont peu structurées et donc plus accessibles, l’effet sur l’expression du gène est plus important.
2.Hypothèse de recherche
Tous ces exemples montrent clairement que la structure d’un ARNm influence grandement son destin et par le fait même l’expression de la protéine correspondante. Par conséquent, si on peut moduler la structure d’un ARNm, on peut aussi en moduler l’expression. Notre hypothèse est qu’il est possible d’y arriver en utilisant de courts oligonucléotides antisens (OA). Cette stratégie revêt plusieurs avantages. Comme les OA s’apparient à leur cible en formant des paires de bases de type Watson-Crick, ils offrent une grande spécificité et leur design est très facile. De plus, en se fiant aux données structurales et aux logiciels de prédictions de structures des ARN, on peut aisément identifier les régions à cibler avec les OA. Finalement, cette technique est très versatile puisqu’on peut cibler plusieurs types de motifs d’ARN.
3.Les oligonucléotides antisens
Le concept d’OA remonte à la fin des années 1970 (Stephenson and Zamecnik 1978). D’abord utilisés comme molécules antivirales, on s’est rapidement aperçu du grand potentiel de cette stratégie. En effet, de courts oligonucléotides, généralement entre 12 et 20 nucléotides, peuvent être utilisés pour cibler n’importe quel ARN cellulaire, qu’il soit d’origine endogène ou autre, de manière spécifique. L’appariement de paires de bases Watson-Crick assure cette spécificité et permet de prédire les cibles non spécifiques potentielles (Lucier et al. 2006).
3.1 Mécanismes d’action des oligonucléotides
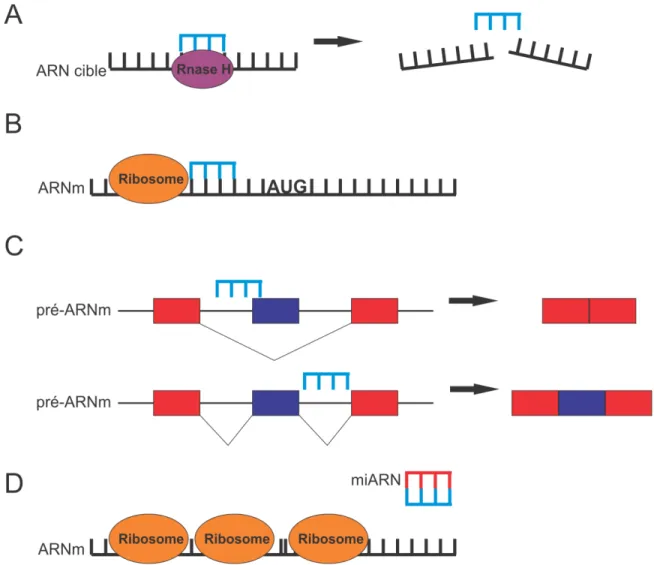
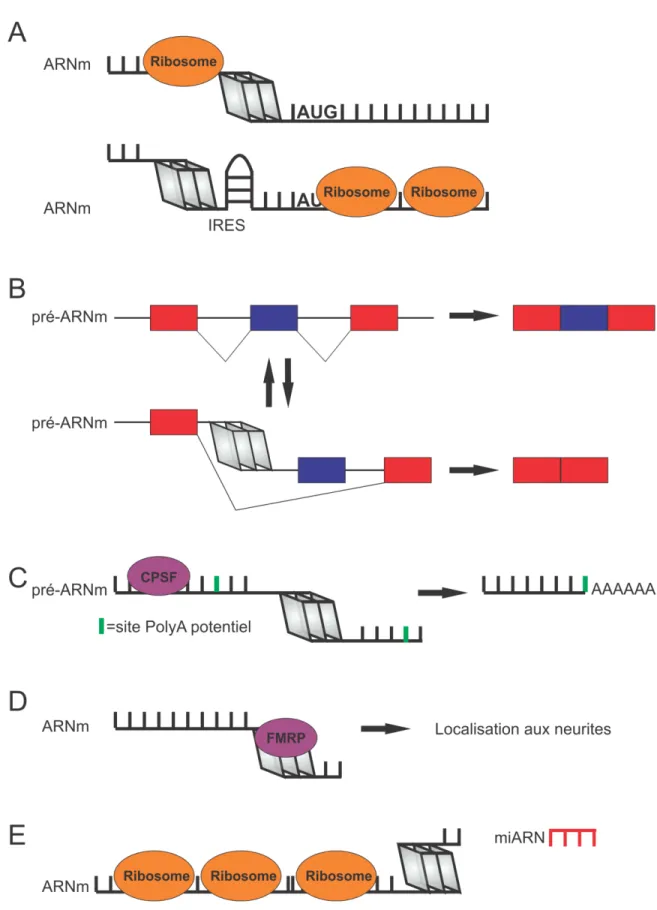
Dépendamment de la nature chimique des oligonucléotides utilisés et de l’endroit qui est ciblé sur l’ARN, les OA ont plusieurs mécanismes d’action (Bennett et Swayze 2010) (Figure 1). Chacun d’eux sera brièvement décrit dans les sections qui suivent.
3.1.1 L’hydrolyse par la ribonucléase H
L’utilisation d’OA d’ADN, ou d’autres dérivés s’en rapprochant, mène à la formation d’hétéroduplexes ADN-ARN. Ces hétéroduplexes sont reconnus par la ribonucléase (RNase) H, une riboendonucléase exprimée de façon ubiquitaire (Cerritelli et Crouch 2009). Elle clive l’ARN seulement lorsqu’il est apparié à l’ADN. La liaison de l’OA mène
l’ARN ciblé (Figure 1A). Si la cible est un ARNm, son expression s’en trouve alors diminuée. Alternativement, on peut plutôt cibler et dégrader un ARN non codant. La dégradation de l’ARN non codant entraîne alors une augmentation de l’expression des gènes normalement réprimés par celui-ci.
D’un autre côté, les oligonucléotides d’ARN, de même que d’autres dérivés, ne sont pas reconnus par la RNase H. Il n’y a donc pas coupure et dégradation des ARN ciblés par ces oligonucléotides. Ceux-ci agissent plutôt par encombrement stérique. Dépendamment de l’endroit ciblé par l’oligonucléotide, l’effet sera différent.
3.1.2 Le blocage de la traduction
Si la région ciblée se retrouve dans le 5’UTR ou la partie codante de l’ARNm, la présence de l’oligonucléotide peut venir empêcher le passage du ribosome. Ceci a pour effet de diminuer la traduction (Boiziau et al. 1991) (Figure 1B).
3.1.3 La modulation de l’épissage
Il est également possible de moduler l’épissage alternatif d’un ARNm avec des oligonucléotides. On parle alors de splice-switching oligonucleotide (SSO) (Bauman et al. 2009). L’épissage de l’ARNm est accompli par un complexe dynamique appelé le spliceosome (Matlin et Moore 2007). Le spliceosome se compose de cinq petits ARN non codants, les snRNA (small nuclear RNA), et d’un nombre variable de protéines. La nature et le nombre de ces protéines varient selon les tissus, les conditions cellulaires et la séquence de l’ARNm. Lors de l’épissage, un spliceosome doit se former à chacune des jonctions où les exons sont rejoints. Ces complexes sont déposés sur l’ARNm par la reconnaissance de motifs non strictement conservés : site d’épissage, point de branchement, séquences riches en polypyridines. La formation du spliceosome est de plus modulée par la présence de séquences introniques ou exoniques : les éléments d’épissage. Ces séquences sont reconnues par certaines protéines et peuvent venir activer l’épissage (splicing enhancer) ou l’inhiber (splicing silencer). L’épissage est dépendant de la compétition entre les sites d'épissage et les éléments d'épissage pour la liaison aux facteurs d'épissage et au spliceosome. Dans beaucoup de cas, cette compétition mène à un équilibre et plusieurs isoformes d’un même messager sont exprimées en même temps. Les SSO peuvent être
utilisés pour venir lier les éléments d’épissage ou les séquences reconnues par le spliceosome et ainsi déséquilibrer la compétition et donc changer le ratio d’expression des isoformes exprimées (Figure 1 C). Une autre stratégie pour moduler l’épissage est d’utiliser des oligonucléotides couplés à des protéines d’épissage, activateurs ou inhibiteurs, et ainsi de modifier la dynamique de formation du spliceosome à un site bien précis reconnu par l’oligonucléotide (Brosseau et al. 2014).
3.1.4 Effets sur les ARN non codants
On peut également cibler les miARN et ainsi augmenter l’expression de leurs messagers cibles sans qu’il y ait nécessairement dégradation du miARN (Esau 2008) (Figure 1D).
Blocage de la traduction. C) Modification de l’épissage pour favoriser l’exclusion ou l’inclusion d’un intron. D) Blocage du miARN ce qui permet à l’ARNm d’être traduit.
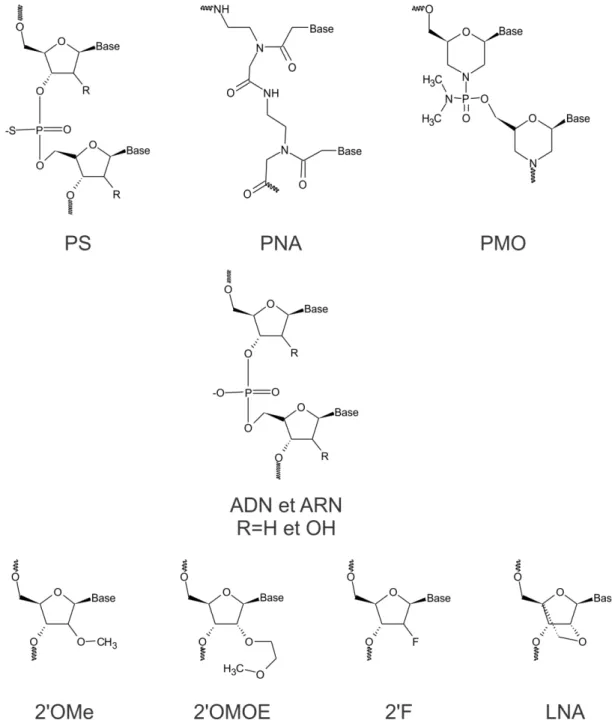
3.2 Les modifications chimiques des oligonucléotides
L’un des principaux obstacles à l’utilisation d’oligonucléotides est leur faible stabilité dans la circulation sanguine et à l’intérieur des cellules (Sharma et al. 2014). En effet, de nombreuses nucléases dégradent rapidement les oligonucléotides, ce qui en plus de diminuer leur effet entraîne une cytotoxicité en raison de la production de désoxyribonucléoside 5’-monophosphate (Moreno et Pêgo 2014). C’est pourquoi on a entrepris de modifier chimiquement le ribose et le squelette phosphodiesterique. Au fil des années, de nombreuses modifications chimiques ont été développées (Dirin et Winkler 2013; Sharma et al. 2014; Geary et al. 2015; Vorobyeva et al. 2016) (Figure 2). Il est important de noter qu’en plus de stabiliser les oligonucléotides, ces modifications vont généralement augmenter l’affinité et/ou la spécificité envers leur cible. On peut regrouper ces modifications en deux grandes catégories qui dépendent des deux régions de l’oligonucléotide pouvant être modifiées : le phosphosquelette et le ribose. Les modifications dans les différentes régions apportent des propriétés distinctes aux OA.
3.2.1 Modifications du phosphosquelette
Les modifications du phosphosquelette mènent généralement à une résistance aux nucléases. La modification la plus souvent utilisée est le phosphorothioate, où l’un des atomes d’oxygène lié au phosphore est substitué par un atome de soufre. D’autres modifications peuvent aussi servir à retirer les charges négatives du phosphosquelette, rendant ainsi plus facile le passage des OA à travers les membranes cellulaires et augmentant l’affinité pour la séquence ciblée. Pour ce faire, on remplace à la fois le phosphosquelette et le ribose par une chaîne peptidique (PNA, Peptidic Nucleic Acid) ou des cycles morpholines reliés entre eux par des groupements phosphorodiamidates (PMO, Phosphorodiamidate Morpholino Oligonucleotide).
3.2.2 Modifications du ribose
Pour modifier le ribose, on cible généralement le carbone 2 et l’on ajoute un groupement à la place de l’hydrogène (ADN) ou de l’hydroxyle (ARN). Ces modifications améliorent l’affinité à la cible de l’OA et peuvent parfois améliorer la résistance aux nucléases vu la proximité du groupement ajouté au phosphate 3’. Les modifications les plus souvent utilisées sont 2’-O-methyl (2’OMe), 2’-O-methoxyethyl (2’OMOE), 2’-Fluore (2’F) et Locked Nucleic Acid (LNA), où les carbones 2 et 4 du ribose sont reliés par un pont méthylène.
Notons que les modifications du squelette et du ribose peuvent être combinées ou distribuées de façon non uniforme le long de l’OA. De plus, ces modifications peuvent influencer l’interaction des OA avec les protéines cellulaires ce qui, dépendamment de leur mode d’action, peut avoir un grand impact sur leur efficacité. Par exemple, la RNase H ne peut pas reconnaître la plupart des modifications au groupement 2’OH du ribose, et donc ne pourra dégrader la cible de tels OA.
Figure 2. Les modifications chimiques des oligonucléotides antisens. Les modifications chimiques les plus couramment utilisées avec les oligonucléotides antisens sont représentées.
3.3 Les modes de livraison des oligonucléotides
Un autre obstacle majeur à l’utilisation d’OA comme molécules thérapeutiques est qu’il est difficile de les amener spécifiquement dans les cellules que l’on souhaite cibler. Au cours des années, de nombreuses techniques ont été utilisées pour pallier à ce problème, ce qui
fait en sorte qu’aujourd’hui, de plus en plus d’OA sont en phase clinique pour le traitement de nombreuses maladies (Kaczmarek et al. 2017). Ces techniques seront brièvement décrites dans cette section.
3.3.1 Les vecteurs viraux
Il est possible d’utiliser des virus modifiés afin qu’ils expriment des gènes d’intérêts dans les cellules qu’ils infectent (Imbert et al. 2017). Cette technique présente plusieurs avantages. Les virus sont des vecteurs naturels qui ont évolués pendant des millions d’années pour pouvoir ‘‘livrer’‘ leur matériel génétique aux cellules infectées. Ils sont donc adéquatement outillés pour pénétrer la barrière cellulaire et intégrer un ou plusieurs gènes aux cellules ciblées. Ainsi, il n’est pas nécessaire d’effectuer des traitements en continu afin que les gènes thérapeutiques continuent d’être exprimés. De plus, en utilisant une combinaison de protéines de l’enveloppe ou la capside virale et de promoteurs appropriés, on peut s’assurer que les gènes ne soient présents et exprimés que dans les cellules que l’on veut traiter.
Pour utiliser cette technique avec les OA, il faut trouver un vecteur d’expression puisque de courts ARN exprimés seuls seraient rapidement dégradés par la cellule. Plusieurs groupes de recherches ont démontré qu’il était possible de modifier les snRNA U1 (Gorman et al. 2000), U6 (Noonberg et al. 1994) et U7 (Gorman et al. 1998) afin qu’ils ne soient plus liés par les protéines du spliceosome et qu’ils contiennent les séquences antisens désirées. Cette stratégie a été appliquée pour corriger l’épissage de transcrits causant la dystrophie musculaire de Duchenne, ainsi que d’autres maladies neurodégénératives pour lesquelles il n’existe pas de remède présentement (Imbert et al. 2017).
Cependant, l’utilisation de vecteurs viraux comporte certains risques. Notamment, les particules virales peuvent parfois être reconnues par le système immunitaire et déclencher des réactions immunologiques non désirées (Bessis et al. 2004). De plus, il est difficile de contrôler l’endroit exact dans le génome où les gènes thérapeutiques s’intègrent, ce qui peut entraîner des effets néfastes, dont le cancer (Hacein-Bey-Abina et al. 2003).
3.3.2 Les aptamères
Les aptamères sont des séquences d’ADN ou d’ARN dont la structure tridimensionnelle leur permet de reconnaître et de lier spécifiquement leur cible (Vorobyeva et al. 2016). Leur affinité et spécificité sont souvent comparables ou supérieures à celles des anticorps (Jayasena 1999). Les aptamères peuvent donc être utilisés pour reconnaître et lier des protéines de surfaces présentes seulement chez les cellules que l’on veut cibler. De plus, cette liaison aux protéines de surfaces entraîne généralement une internalisation de l’aptamère. En liant chimiquement l’aptamère à un cargo, il est donc possible de livrer ce dernier aux cellules ciblées par l’aptamère (Catuogno et al. 2016). Ce cargo peut être une drogue, ou bien dans le cas qui nous intéresse, de courts ARN interférents ou bien des OA.
En plus d’offrir une grande spécificité de ciblage, les aptamères ont d’autres propriétés avantageuses : ils sont généralement non toxiques et ne déclenchent pas de réactions immunologiques non désirées (Song et al. 2012). Cependant, vu leur petite taille, ils sont facilement filtrés de l’organisme par les reins (Lee et al. 2006). De plus, ils sont facilement dégradés par les nucléases de l’organisme, ce qui peut réduire considérablement leur demi-vie in vivo. De plus, quand les aptamères et leur cargo sont internalisés dans la cellule, ils se retrouvent ensuite dans la voie endosomale/lysosomale et une grande partie du cargo sera dégradée avant de pouvoir exercer son effet thérapeutique. Des solutions existent cependant pour pallier à ces problèmes. Les modifications chimiques discutées précédemment pour les OA peuvent aussi être appliquées aux aptamères afin d’améliorer leur résistance aux nucléases. De plus, les aptamères peuvent être couplés à des nanoparticules (voir section suivante) ou d’autres molécules ce qui permet d’augmenter leur demi-vie in vivo et/ou d’échapper à la voie endosomale/lysosomale (Oh et al. 2014).
3.3.3 Les nanoparticules
Par définition, les nanoparticules ont une taille située entre 1 et 100 nm. Elles peuvent être composées de plusieurs différents matériaux. Diverses nanoparticules ont été utilisées par le passé pour la livraison d’OA (Lambert et al. 2001). Ces nanoparticules peuvent être composées de lipides, de métaux (par exemple or ou fer), de nanotubes de carbones, d’acides nucléiques ou d’autres bio-composés (Li et al. 2015). Pour être de bons agents de livraison, ces nanoparticules doivent améliorer la stabilité et la demi-vie des OA dans le
système sanguin. De plus, différentes molécules telles que des aptamères ou des protéines peuvent être ajoutées aux nanoparticules afin de cibler seulement les cellules désirées. Il faut cependant s’assurer que ces nanoparticules soient non toxiques et qu’elles ne posent pas, à long terme, de problèmes d’accumulation ou d’immunogénicité.
3.3.4 La conjugaison
Alternativement, les OA peuvent simplement être conjugués à de courtes chaînes lipidiques ou peptidiques (Ming et Liang 2015). Ces groupements ajoutés servent généralement à améliorer la stabilité et la biodisponibilité des OA. Ils peuvent de plus conférer une hydrophobicité qui permet aux OA de pénétrer à l’intérieur des cellules. Ces ajouts sont généralement non toxiques et non immunogènes. Cependant, pour l’instant, il est difficile de cibler spécifiquement des cellules ou des tissus, à l’exception du foie, qui semble avoir une propension naturelle à internaliser les OA présents dans la circulation sanguine.
En bref, il existe plusieurs façons de modifier les OA et de les livrer aux cellules que l’on désire cibler. De nombreux motifs d’ARN peuvent donc potentiellement être visés. 4. Utiliser les oligonucléotides pour cibler des motifs structuraux
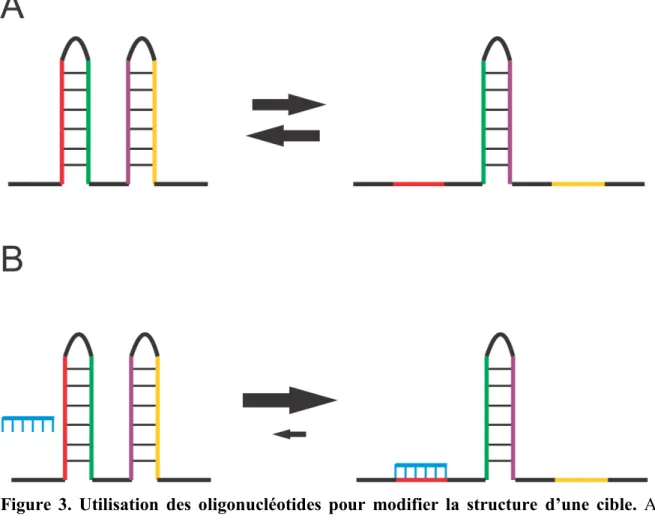
Bien qu’ils aient des modes d’action différents, les OA présentés jusqu’à maintenant ont tout de même un point en commun : on tente de viser des régions simple brin et facilement accessibles. Viser ces régions permet de recruter la RNase H et dégrader l’ARN ciblé ou bien les OA servent d’encombrement et empêche la liaison de protéines ou d’autres ARN à l’ARN ciblé. L’idée primordiale des travaux de cette thèse était d’utiliser des courts OA pour cibler spécifiquement des motifs structuraux d’ARN particuliers afin de modifier la structure globale de l’ARN. En effet, le repliement de l’ARN est un processus dynamique (Bugaut et al. 2012; Mirihana Arachchilage et al. 2015). Un seul ARN peut adopter de multiples conformations possibles, et ces conformations peuvent avoir des fonctions différentes (Gros et al. 2008). Nous avons postulé qu’en utilisant des OA, il était possible d’affecter l’équilibre entre les différentes structures et par conséquent d’affecter aussi les niveaux d’expression des gènes associés à ces motifs (Figure 3). Différents motifs ont ainsi été ciblés et ils seront décrits plus en détails dans les sections suivantes. Remarquons aussi que les travaux présentés dans cette thèse ont utilisés deux types de modifications : 2’OMe et LNA. Ces modifications ont été choisie pour diverses
ciblé ne soit dégradé par cet enzyme. De plus, ces modifications augmentent la résistance des OA, augmentant ainsi la durée de vie des OA en cellules. Finalement, ces modifications augmentent l’affinité des OA pour leur cible et la stabilité du duplex formé, ce qui devrait permettre un ciblage plus efficace.
Figure 3. Utilisation des oligonucléotides pour modifier la structure d’une cible. A) Équilibre entre deux structures adoptées par un même ARN. B) L’ajout d’un oligonucléotide fait varier cet équilibre.
4.1 Le ribozyme du virus de l’hépatite delta
Le terme ribozyme est une contraction des mots ribose et enzyme. Il désigne donc un ARN qui est capable de catalyser une réaction biochimique (Fedor et Williamson 2005). Plusieurs ribozymes ont été décrits à ce jour et le plus célèbre d’entre eux est certainement le ribosome. En effet, c’est l’ARN de la grande sous-unité du ribosome qui catalyse la formation du lien peptidique lors de la traduction des protéines (Cech 2000). Plusieurs ribozymes ont pour fonction de catalyser la coupure de leur propre phosphosquelette :
parmi ceux-ci, on retrouve les ribozymes de type hammerhead(de la Peña et al. 2017), hairpin (Müller et al. 2012), twister (Gebetsberger et Micura 2017), du virus de Varkud (Dagenais et al. 2017) et du virus de l’hépatite delta (VHD) (Riccitelli et Lupták 2013). Ce dernier type a été utilisé pour les travaux de cette thèse.
4.1.1 Le virus VHD
Le ribozyme VHD a été découvert dans l’ARN d’un virus satellite, le virus de l’hépatite D (Wu et al. 1989). Ce virus est dit satellite parce qu’il utilise les protéines d’un autre virus, celui de l’hépatite B, pour compléter son cycle réplicatif (Lempp et Urban 2017). Le virus VHD possède un génome d’ARN simple brin circulaire de polarité positive qui se réplique par un mécanisme de cercle roulant symétrique. Un seul long ARN comprenant plusieurs copies du brin négatif est donc produit à partir de l’ARN circulaire. Le ribozyme sert à l’auto-coupure de l’ARN ce qui sépare le long brin en copies uniques du génome de polarité négative. Ces brins sont ensuite ligués sur eux-mêmes pour former un ARN circulaire qui sert de matrice pour produire l’ARN de brin positif en utilisant le même mécanisme. Les deux polarités possèdent donc un motif de ribozyme VHD. Le motif du brin négatif a été utilisé comme base pour les travaux de cette thèse.
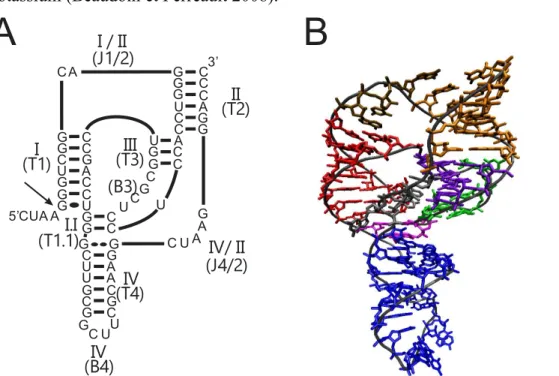
4.1.2 Structure du ribozyme VHD
La structure du ribozyme des deux polarités est très similaire(Reymond et al. 2010) (Figure 4). Elle est composée de cinq séries de nucléotides appariés (tiges I, I.I, II, III et IV) séparées par des régions simple brin. Ces régions sont des boucles (III et IV) ou des jonctions reliant deux tiges (I/II, I/IV et IV/II). La structure est également caractérisée par deux pseudonoeuds, le premier formé par les tiges I et II, le second par la tige I.I, qui est en fait composée de seulement deux nucléotides de la jonction I/IV appariés à deux nucléotides de la boucle III. Cette structure de deux pseudonoeuds imbriqués est caractéristique du ribozyme VHD et permet l’empilement des tiges I, I.I et IV ainsi que II et III. Cela confère une grande stabilité au ribozyme, ce qui explique pourquoi l’activité de coupure est toujours présente même en conditions extrêmes telles que des températures de 80oC ou des concentrations de 8 M d’urée, un puissant agent dénaturant (Rosenstein et Been 1990). Cette grande stabilité de la structure a nui au développement de ribozymes allostériques à partir de ribozymes VHD. Au moment de débuter mes études graduées, le
seul ribozyme allostérique développé avec la version antigénomique était le G-quartzyme, un ribozyme capable de former une structure G-Quadruplexe dont l’activité dépend du potassium (Beaudoin et Perreault 2008).
Figure 4. Le ribozyme VHD. A) Représentation de la structure secondaire du ribozyme VHD. La flèche indique l’endroit du clivage. Les différentes régions sont annotées (T=tige, J=jonction, B=boucle) et numérotées. B) Structure du crystal du ribozyme du brin positif VHD. Figure adaptée de (Reymond et al. 2010).
4.1.3 Mécanisme de catalyse du ribozyme VHD
Bien que de nombreux travaux aient été réalisés sur la structure et le repliement du ribozyme VHD, le mécanisme par lequel la coupure a lieu fait encore aujourd’hui l’objet de débats (Wei et al. 2007). Malgré tout, certaines caractéristiques ont été établies. La coupure requiert la présence d’un ion divalent (Suh et al. 1993), préférentiellement Mg2+ ou Ca2+ et
est effectuée par une cytosine catalytique, C75 ou C76 pour les versions de polarité positive et négative respectivement. La coupure se produit à la base de la tige I, juste avant la guanine 1 qui forme une paire de base de type wobble. Après la coupure, les deux nouveaux brins possèdent une extrémité 5’OH et une autre 2’-3’ phosphocyclique. Ces types d’extrémités sont très sensibles aux nucléases.
C U AA G G C G C G U A U A G C G C G U G G C U G U 3’ G C G C G C C G C G U A C G C G G C U A C G C C U U G C C G C U A G C G C CA A A U C G 5’ (T1)I (J1/2)I / II (T2)II (T3)III (J4/2)IV/ II (B4)IV (B3) (T1.1)I.I (T4)IV
A
B
4.1.4 Ribozyme VHD comme outil de régulation génique
Le ribozyme possède donc des propriétés intéressantes pour en faire un outil d’inactivation génique : il possède une structure stable, il a une catalyse rapide et il a évolué pour être actif dans les cellules humaines. Deux stratégies ont été employées pour les exploiter .
Premièrement, on a modifié le ribozyme pour qu’il coupe une autre molécule d’ARN plutôt que de s’auto-couper. Pour ce faire, il a suffi de tronquer le ribozyme en 5’ pour enlever le brin 5’ de la tige I et la jonction I/II. Le brin 5’ de la tige I sera remplacé par le substrat que l’on veut couper avec le ribozyme (Roy et al. 1999). On peut ensuite modifier le brin 3’ de la tige I dans le ribozyme pour qu’il soit complémentaire à l’ARN que l’on désire cibler. La seule restriction est que le site de coupure doit être d’une séquence HG (H=A, U ou C) suivi de sept nucléotides.
4.1.4.1 Le module SOFA
Le principal inconvénient de ce système est son manque de spécificité. En effet, la reconnaissance de la cible par 8 nucléotides seulement augmente les probabilités de cibler et couper des ARN autres que la cible désirée. Pour pallier à cette lacune, le ribozyme a été modifié avec l’ajout d’un module appelé SOFA (Specific On/oFf Adaptor) (Bergeron et Perreault 2005). Cette modification permet au ribozyme d’avoir deux conformations, l’une active et l’autre inactive. Deux parties du module SOFA déterminent la conformation du ribozyme. En absence de l’ARN ciblé, le bloqueur lie partiellement la tige I sans être coupé, rendant le ribozyme inactif. En présence de la cible, une autre partie, le biosenseur, se lie à la cible ce qui dégage le bloqueur de la tige I. Celle-ci peut alors elle aussi se lier à la cible et active le ribozyme. En utilisant différentes longueurs de biosenseurs, on peut ainsi atteindre le degré de spécificité désiré. De tels ribozymes ont été utilisés pour cibler des ARNm en cellules bactériennes (Fiola et al. 2006) et humaines (D’Anjou et al. 2011; Ben Aissa et al. 2012). Des ARN viraux du virus de l’hépatite C (VHC) (Lévesque et al. 2010), du virus de l’immunodéficience humaine (VIH) (Lainé et al. 2011; Scarborough et al. 2014) et de l’Influenza (Motard et al. 2011) ont aussi été ciblés.
Une seconde stratégie consiste à insérer un ribozyme capable d’auto-coupure dans un ARNm. Suite à la coupure, la partie codante sera privée soit de la coiffe en 5’ ou encore de la queue poly-A, ce qui en plus de réduire sa stabilité va diminuer sa capacité à être traduit (Auslander et al. 2010). De plus, on peut modifier le ribozyme pour qu’il devienne allostérique et ainsi réguler l’expression génique de manière dépendante d’un ligand. Cette stratégie a été appliquée avec succès sur différents ribozymes (Win et al. 2009; Auslander et al. 2010; Liang et al. 2011), mais s’est avérée difficilement superposable au ribozyme VHD.
Au moment de débuter mes études graduées, l’un des seuls exemples était basé sur la version retrouvée dans le brin positif de VHD et son activité était dépendante de la théophylline, un dérivé de la caféine (Kertsburg et Soukup 2002). Son activité avait cependant seulement été démontrée in vitro. Notre groupe avait tenté sans succès de développer un ribozyme similaire en se basant sur la version du brin négatif VHD (A. Nehdi, résultats non publiés). Un ancien étudiant avait également tenté de remplacer la tige boucle IV avec la tige boucle TAR du VIH afin de rendre l’activité du ribozyme dépendante de la liaison à la protéine virale Tat, sans succès (A. Nehdi, résultats non publiés).
Une autre tentative en ce sens a été de fusionner le ribozyme avec un motif G4 (Beaudoin et Perreault 2008). L’activité du ribozyme est ainsi devenue dépendante du repliement du G4, ce qui permet de contrôler l’activité du ribozyme en changeant la concentration d’ions K+, qui est nécessaire à la stabilité du G4. Le problème majeur de cette stratégie est qu’il est difficile de modifier la concentration intracellulaire de K+, ce qui rend difficile l’utilisation d’un tel ribozyme comme outil de régulation génique en cellules.
4.1.5 Objectif de recherche : Modifier le ribozyme VHD pour le cibler avec des oligonucléotides
Le premier objectif de recherche a donc été de modifier le ribozyme afin que son activité devienne dépendante de la liaison à de courts OA. Ainsi, en insérant ces ribozymes modifiés dans les régions non codantes d’un gène, il deviendrait possible de moduler l’expression de ce gène avec les OA. Le chapitre 1 de cette thèse est consacré à cet objectif.
4.2 Le G-quadruplex
Dans la deuxième partie des travaux de cette thèse, un second motif a été ciblé : le G4.
4.2.1 Structure
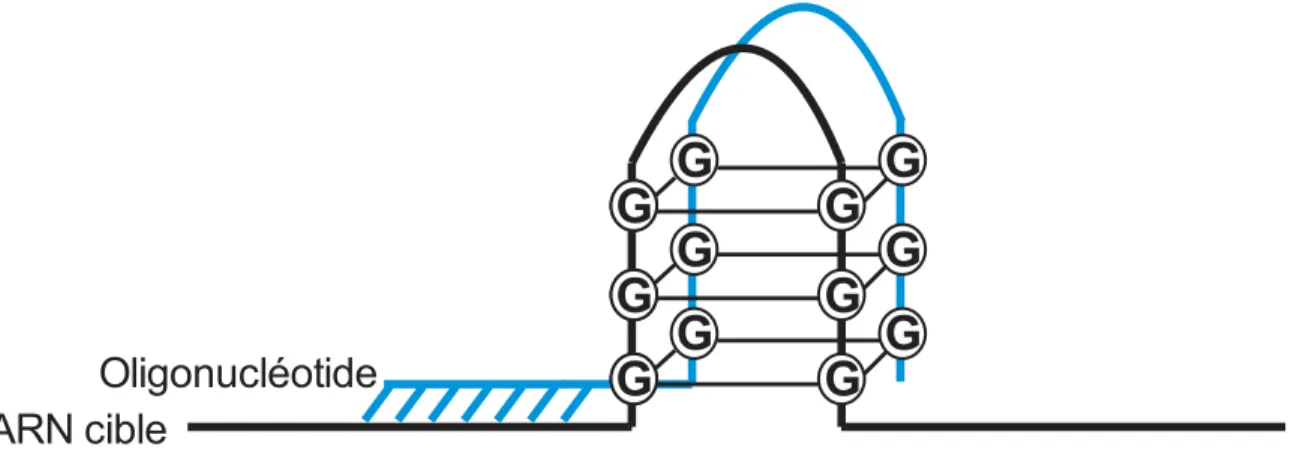
Les G4 sont des structures non-canoniques que peuvent adopter les séquences d’acides nucléiques, ADN ou ARN, qui sont riches en guanines (Huppert 2008). La structure G4 est en fait un empilement hélicoïdal de plusieurs tétrades de guanines (Figure 5). Comme son nom l’indique, la tétrade de guanines est formée de quatre guanines arrangées de manière coplanaire : chacune est reliée à deux autres guanines via sa face Watson-Crick et sa face Hoogsteen. Cette conformation fait en sorte que l’oxygène 6 de chaque guanine est orienté vers le centre de la tétrade. Comme ces 4 oxygènes sont chargés partiellement négativement, les G4 sont fortement stabilisés par la présence de cations monovalents, par exemple : K+, Na+, NH4+ (Williamson et al. 1989; Burge et al. 2006). Il est intéressant de
noter que le Li+, malgré qu’il soit aussi un cation monovalent, est beaucoup moins apte à
stabiliser les G4, étant donné que le noyau de cet atome est trop petit. Notons également que le K+ est le cation qui stabilise le mieux les G4.
D‘abord pris pour des artefacts de laboratoire, le nombre croissant de preuves de leur implication dans une myriade de processus biologiques force à arriver à la conclusion que ces structures sont bel et bien formées in vivo. De récents articles montrent d’ailleurs qu’il est possible de visualiser des G4 dans des cellules humaines (Biffi et al. 2013; Biffi et al. 2014).
Figure 5. Le G-quadruplex. A) La tétrade de G est constituées de quatre guanines, arrangées de manière coplanaire. Chacune des guanines est reliée à deux autres via sa face Watson-Crick et sa face Hoogsteen, Au centre, un cation (dans ce cas-ci un ion K+) est
requis pour stabiliser le tout. B) L’empilement de plusieurs tétrades en une structure hélicoïdale, le G-quadruplex.
4.2.2 Les G quadruplexes d’ADN
De nombreux rôles ont été attribués aux G4 d’ADN (Hänsel-Hertsch et al. 2017). Le plus connu et le plus étudié est sans aucun doute celui formé par les séquences télomériques (Lu et al. 2013). Les télomères sont présents au bout des chromosomes et sont composés de séquences répétées (TAAGGG pour l’humain). Les télomères possèdent une extrémité 3’ saillante simple brin, qui est raccourcie à chaque division cellulaire. Les télomères peuvent cependant être rallongés par la télomérase, un complexe riboprotéique qui se sert de sa sous-unité ARN comme matrice afin de synthétiser l’ADN télomérique. La télomérase est généralement inactive dans les cellules adultes saines, mais est réactivée dans la plupart des cancers (Armstrong et Tomita 2017). Il a été démontré que l’ADN télomérique peut former des structure G4 in vitro(Lu et al. 2013) et in cellulo (Biffi et al. 2013). De plus, la stabilisation de ces structures inhibe l’activité de la télomérase (Mergny et al. 2001; Read et al. 2001; Gomez et al. 2003). Cela donne donc un potentiel thérapeutique pour contrer le cancer aux molécules ciblant et stabilisant les G4.
De plus, de nombreux oncogènes possèdent un G4 d’ADN dans leur région promotrice et stabiliser ces G4 avec des ligands chimiques pourrait permettre de diminuer la transcription de ces gènes. En effet, ceci a été démontré pour les gènes MYC (Siddiqui-Jain et al. 2002), KRAS (Cogoi et Xodo 2006), VEGF (Sun et al. 2005) et KIT (Bejugam et al. 2007).
Plus récemment, les travaux de Tsai et al. ont suggéré que les G4 d’ADN retrouvés dans les introns pouvaient jouer un rôle dans l’épissage de l’ARNm (Tsai et al. 2014). Ils ont remarqué que les séquences G4 et d’autres structures non canoniques étaient enrichies près des sites de saut d’exon, l’un des mécanismes d’épissage alternatif. De plus ils ont noté que plus la structure prédite avait une grande stabilité anticipée, plus le phénomène de saut d’exon était fréquent. Aucune donnée expérimentale n'est toutefois disponible à ce jour pour infirmer ou confirmer leur hypothèse.
4.2.3 Les G-quadruplexes d’ARN
Les G4 d’ARN ont été quant à eux moins bien étudiés que leurs analogues d’ADN. Ils présentent quelques différences structurales, notamment, ils adoptent généralement une topologie parallèle et ont tendance à être plus stables que les G4 d’ADN(Joachimi et al. 2009). De plus, l’absence de brin complémentaire pour venir s’apparier laisse à penser qu’in vivo, les G4 d’ARN ont plus de probabilités de se former que ceux d’ADN.
Au cours des années, il a été démontré que les G4 d’ARN sont impliqués dans une multitude de processus biologiques (Agarwala et al. 2015; Fay et al. 2017; Rouleau et al. 2017b): maintenance des télomères, traduction, épissage alternatif, polyadénylation, localisation de l’ARNm, cycle de réplication virale, et rétrotransposition. La fonction du G4 d’ARN dépend de sa position dans l’ARNm et des protéines qui le reconnaissent.
4.2.3.1 Les G4 et la maintenance des télomères
Deux longs ARN non codant (lncRNA) sont impliqués dans la régulation de la longueur des télomères : TERC (Telomerase RNA Component) (Armstrong et Tomita 2017) et TERRA (Telomeric Repeat Containing RNA)(Martadinata et al. 2011). Les deux ont la capacité de former une structure G4. TERC est un ARN de 491 nucléotides qui, avec les protéines TERT, TEP1 et DKC1, forment le complexe ribonucléoprotéique de la
télomérase. TERC sert de matrice à la transcriptase inverse TERT, ce qui permet l’allongement des télomères. De plus, TERC sert d’échafaud moléculaire pour le complexe. Dans la cellule, TERC peut adopter plusieurs conformations. Il a été suggéré que tout de suite après la transcription, la partie 5’ de TERC se replie en G4. La formation du G4 stabiliserait TERC, mais elle interfère aussi avec le positionnement du brin matrice dans le complexe (Gros et al. 2008). La protéine hélicase RHAU viendrait ensuite se lier au G4 et permettrait de le déplier afin favoriser la formation de l’hélice P1a, ce qui rend la télomérase active (Booy et al. 2012).
TERRA est un lncRNA transcrit à partir du brin riche en cytosine des télomères. Il est donc composé de séquences répétées UAAGGG, est polyadénylé et présent dans les cellules dans des longueurs hétérogènes allant jusqu’à près de 6 000 nucléotides. Il a été démontré que TERRA peut se replier en plusieurs structures G4 in vitro (Martadinata et al. 2011), et probablement aussi in vivo (Hirashima et Seimiya 2015). Les différents G4 peuvent être arrangés indépendamment, comme des billes sur un fil, ou bien se regrouper pour former une suprastructure. Le rôle de TERRA est d’inhiber l’activité de la télomérase. Pour ce faire, il utilise plusieurs mécanismes. Il a été démontré que TERRA peut lier la télomérase, mais aussi l’ARN TERC et ainsi entrer en compétition avec les télomères pour la liaison (Redon et al. 2010). De plus, il semble que TERRA puisse former des G4 intermoléculaires hybrides ADN/ARN avec les télomères (Xu et al. 2008). La formation de ces hybrides inhibe l’activité de la télomérase.
4.2.3.2 Traduction
La présence de G4 dans le 5’UTR d’un gène mène généralement à une diminution de la traduction. Cela a été démontré pour de nombreux gènes (Kumari et al. 2007; Beaudoin et Perreault 2010; Lammich et al. 2011; Bugaut et Balasubramanian 2012). Plusieurs mécanismes ont été proposés pour expliquer cette diminution de la traduction. Dans le cas de NRAS, le G4 semble empêcher la liaison de la structure coiffe par le complexe eIF4F, ce qui diminue le recrutement de la sous-unité 40S du ribosome (Kumari et al. 2008). En effet, l’inhibition de la traduction par le G4 NRAS est dépendant de sa proximité à la structure coiffe. De plus, les auteurs ont démontré que peu importe la position du G4, plus celui-ci est stable, plus son effet sur la traduction est prononcé. Il semble que la structure G4 nuise
au passage de la petite sous-unité du ribosome lors de la recherche pour le codon d’initiation, ce qui diminue le nombre de ribosomes formés au codon d’initiation et réduit la traduction. Finalement, dans certains cas, comme pour BCL-2 (Shahid et al. 2010), le G4 est situé près du codon d’initiation. Le G4 pourrait alors nuire à la traduction en empêchant la reconnaissance du codon d’initiation par le ribosome. Cette hypothèse a été clairement démontrée chez les procaryotes (Wieland et Hartig 2007), mais pas encore chez les eucaryotes.
Dans d’autres cas, il semble que la structure G4 puisse faire partie d’une structure IRES. C’est le cas notamment pour les gènes VEGF (Morris et al. 2010) et TGFB2 (Agarwala et al. 2013). Dans ce cas-ci, la formation du G4 mène à une augmentation de la traduction, de manière indépendante de la structure coiffe. Une étude récente a d’ailleurs démontré que c’est le contexte dans lequel il se trouve et non le G4 lui-même qui détermine l’effet sur la traduction (Bhattacharyya et al. 2017). En effet, lorsqu’ils modifiaient le 5’UTR de VEGF et remplaçait le G4 par celui de NRAS ou de M3-MMP, deux G4 reconnus pour inhiber la traduction de leur ARNm respectifs, l’expression d’un gène rapporteur était augmentée par rapport à un contrôle ou le G4 était absent. D’une manière similaire, les G4 de VEGF et TGFB2 diminuaient la traduction lorsque placés dans le contexte du 5’UTR de M3-MMP.
Même s’ils sont enrichis dans les UTRs, il arrive que des G4 se retrouvent dans les régions codantes, ORF (open reading frame). Ces G4 peuvent affecter la traduction de protéines de plusieurs façons. Endoh et al. ont étudié la formation de G4 dans l’ORF des gènes de la bactérie Escherischia coli. Ils ont prouvé que les ORF des gènes eutE, gudP, rnlA et yebZ contenaient tous des séquences pouvant former des G4 in vitro. De plus, ces séquences causaient des arrêts de la traduction lorsqu’insérées dans des systèmes rapporteurs lors d'une expérience de traduction in vitro ainsi que dans deux lignées de cellules humaines (Endoh et al. 2013a; Endoh et al. 2013b).
Ce même groupe a aussi rapporté un autre mécanisme par lequel les G4 peuvent réguler la traduction. Ils ont démontré qu’une séquence présente dans le gène ESR1 pouvait se replier en G4 (Endoh et al. 2013c). La formation du G4 mène à un ralentissement de la translocation du ribosome pendant la traduction, ce qui favorise le clivage de la protéine
ERα. Ils ont aussi montré par des mutations synonymes que le clivage était proportionnel à la stabilité du G4 formé.
Une autre étude, à laquelle j’ai participé, a démontré que la protéine AVEN reconnait et lie des G4 dans la séquence codante des gènes MLL1 et MLL4 et aide à la traduction de ces messagers, probablement en recrutant DHX36, une hélicase reconnue pour déplier les structures G4 (Thandapani et al. 2015).
Finalement, notons que la traduction de certains gènes peut aussi être inhibée par des G4 situés dans le 3’UTR. C’est le cas de PIM1 (Arora et Suess 2011) et TP53 (Decorsiere et al. 2011). Le mécanisme par lequel l’inhibition a lieu n’est pas connu.
4.2.3.3 Épissage alternatif
L’épissage des gènes BACE1 (Fisette et al. 2012), FMR1 (Didiot et al. 2008), TP53 (Marcel et al. 2011), PAX9 (Ribeiro et al. 2015), et TERT (Gomez et al. 2004) a été montré pour être régulé par une structure G4. La structure G4 sert de signal de recrutement à différents facteurs d’épissage : hnRNP F/H (Decorsiere et al. 2011) de même que les protéines FMRP, FXR1 et FXR2 (Evans et al. 2012; Blice-Baum et Mihailescu 2014) sont capables de lier les séquences riches en guanines. Si certaines d’entre elles reconnaissent spécifiquement la structure G4, d’autres ont plutôt tendance à lier des séquences d’ARN simple brin et ne peuvent lier les G4 (Samatanga et al. 2013). De plus, les G4 situés près d’autres éléments régulateurs de l’épissage peuvent venir nuire à la liaison des facteurs d’épissage. Ainsi la formation ou non du G4 affecte le recrutement de facteurs d’épissage, ce qui affecte le recrutement du spliceosome et donc l’épissage de l’ARNm.
4.2.3.4 Polyadénylation et terminaison de la transcription
La polyadénylation est une étape primordiale dans la biogénèse des ARNm eucaryotes (Proudfoot 2016). Elle consiste en l’ajout d’une queue de poly-adénines à l’extrémité 3’ de l’ARN. La longueur de cette queue est variable et influence le transport, la traduction et la stabilité de l’ARNm. La polyadénylation s’effectue en plusieurs étapes. D’abord il y a reconnaissance du signal de polyadénylation par l’enzyme CPSF. Le signal préférentiellement reconnu est AAUAAA, bien que d’autres séquences similaires puissent être reconnues et utilisées. Il y a ensuite clivage de l’ARN de 10 à 30 nucléotides en aval du
site du signal. Cette coupure mène à un arrêt de la transcription et à l’ajout d’adénines à l’extrémité 3’.
Chez l’humain, environ 70% des ARNm possèdent au moins deux sites possibles de polyadénylation, et environ 50% en possèdent 3 ou plus (Elkon et al. 2013). L’utilisation de sites alternatifs mène donc à plusieurs isoformes de 3’UTR ce qui peut affecter la traduction, la stabilité et la localisation de l’ARNm. Il a été démontré que la présence d’un G4 en aval du signal de polyadénylation peut influencer la fréquence à laquelle celui-ci est utilisé (Beaudoin et Perreault 2013). En effet, la mutation du G4 mène à la diminution de l’utilisation du site de polyadénylation en amont du G4.
4.2.3.5 Localisation cellulaire
La localisation de certains ARNm est un phénomène essentiel pour la compartimentation de certaines cellules spécialisées (Chin et Lécuyer 2017). Elle permet que certaines protéines ne soient traduites qu’à l’endroit précis où elles doivent exercer leur action. Plusieurs mécanismes peuvent mener à cette distribution asymétrique des ARNm. Parmi ceux-ci, notons la présence de signaux de localisation dans le 3’UTR. Par exemple, dans les neurones, il a été démontré que la présence de G4 dans le 3’UTR des ARNm des gènes DLG4 et CAMK2A leur permet d’être localisés aux neurites (Subramanian et al. 2011). Les protéines FMRP et TDP-43 (Ishiguro et al. 2016) semblent être responsables de la reconnaissance du G4 et de la localisation des ARNm.
Figure 6. Fonctions des G-quadruplexes dans les ARNm. Quelques exemples de fonctions des G4 retrouvés dans les ARNm. A) Les G4 dans le 5’UTR peuvent inhiber ou, quand ils font partie d’un IRES, activer la traduction. B) Influence du G4 sur l’épissage
alternatif. C) Le G4 favorise l’utilisation d’un site de polyadénylation alternatif situé en amont. D) Le G4 est reconnu part des protéines qui permettent une localisation cellulaire spécifique de l’ARNm. E) Le G4 empêche la liaison du miARN, ce qui permet la traduction de l’ARNm.
4.2.3.6 Les longs ARN non codants
Les lncRNAs sont des ARN longs de plus de 200 nucléotides qui ne codent pour aucune protéine mais régulent l’expression d’autres gènes par divers mécanismes, notamment en modifiant le remodelage de la chromatine (Engreitz et al. 2016). Plus de 125 000 lncRNAs ont été annotés dans le génome humain. Cependant, la fonction de la plupart d’entre eux n’est pas encore connue. Récemment, Jayaraj et al. ont étudié la présence de G4 potentiels dans les lncRNAs (Jayaraj et al. 2012). Ils ont découvert que près de 2 400 lncRNAs possédaient au moins une séquence capable de former un G4. Ils ont aussi remarqué que les G4 étaient enrichis dans les lncRNAs d’une taille allant de 200 à 300 nucléotides et que les G4 possédant des boucles de 1 ou 2 nucléotides, donc plus stables, étaient enrichis par rapport à des G4 à plus longues boucles. De plus, ils ont démontré qu’une partie des séquences identifiées étaient capables de former un G4 in vitro. Le rôle des G4 dans les lncRNAs demeure cependant toujours inconnu. Les auteurs de cette étude ont suggéré qu’ils pourraient servir d’éponge et entrer en compétition avec des ARNm pour lier les protéines reconnaissant les G4.
4.2.3.7 Les microARN
Les miARN sont de petits ARN, généralement longs de 22 nucléotides, qui répriment l’expression de leurs gènes cibles en s’appariant généralement dans la région 3’UTR des ARNm (Bartel 2009). L’ARNm ciblé est ensuite dégradé ou bien voit sa traduction grandement diminuée. Chez les mammifères, l’appariement entre le miARN et sa cible n’a pas besoin d’être parfait pour que la répression ait lieu. En effet, le plus important est que la seed region du miARN (les nucléotides en positions 2 à 8) soit parfaitement appariée, le reste du miARN étant partiellement complémentaire à sa cible, pour que la répression ait lieu. Ce phénomène fait en sorte qu’un seul miARN peut avoir des dizaines, voire des centaines de cibles potentielles et rend la prédiction de ces cibles très difficile. Les miARN
sont donc considérés comme des acteurs majeurs dans la régulation de l’expression génique et une dérégulation de leur expression peut mener à plusieurs pathologies telles que des maladies cardiovasculaires (Small et Olson 2011), neurodégénératives (Abe et Bonini 2013) et des cancers (Farazi et al. 2013).
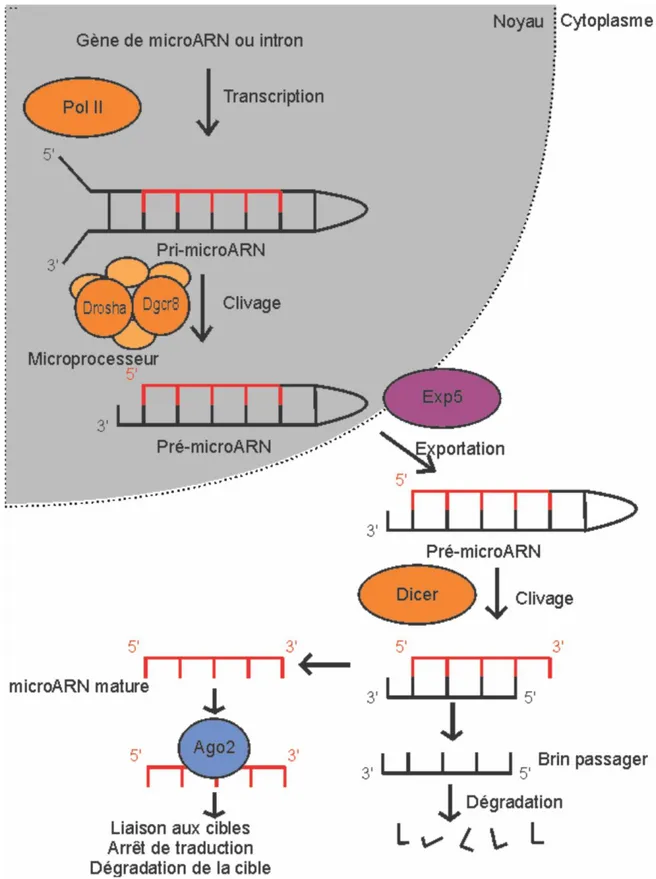
L’expression des miARN est donc un processus finement contrôlé dans la cellule et présente un intéressant potentiel thérapeutique (Ha et Kim 2014). La plupart des miARN sont produits à partir de longs ARN non codants transcrits par la polymérase II, appelés primary-microRNA (pri-miARN). Certains sont également produits à partir des introns de certains ARNm et sont parfois appelées mirtrons (Curtis et al. 2012). Tous ces précurseurs de miARN ont cependant un point en commun: ils adoptent une structure en tige boucle qui est reconnue dans le noyau par un complexe protéique appelé le microprocesseur. Ce complexe comprend deux protéines principales, dgcr8 et drosha, ainsi que plusieurs protéines accessoires qui peuvent varier d’un miARN à l’autre (Nguyen et al. 2015). Drosha, une endonucléase de type III, est considérée comme l’enzyme principale du micro-processeur puisqu’elle reconnaît la structure tige-boucle et clive l’ARN. Le produit de cette coupure est une tige-boucle d’environ 60 nucléotides appelée precursor microRNA (pré-miARN).
Les pré-miARN sont ensuite reconnus par des protéines de transport telles qu’exp5 et transportés dans le cytoplasme. Ils peuvent ensuite servir de substrat à dicer, une autre endonucléase de type III. Le clivage par dicer donne un duplex de petits ARN, généralement de 22 nucléotides. L’un deux, appelé le brin passager, est dégradé, tandis que l’autre devient le miARN et est associé à une protéine argonaute, nécessaire à sa fonction. Il est intéressant de noter que pour certains duplexes, les deux brins peuvent être utilisés comme miARN et que les mécanismes de sélection entre le brin passager et le miARN ne sont pas très bien compris, bien que certains travaux tendent à démontrer que la stabilité du duplex à l’extrémité 3’ du miARN joue un rôle important (Hu et al. 2009). La biogénèse des miARN est donc un processus complexe et finement régulé. Plusieurs étapes peuvent être influencées par les G4.