GUILLAUME J. BILODEAU
DÉTECTION ET GÉNOMIQUE DE PHYTOPHTHORA
RAMORUM AGENT CAUSAL DE LA MORT SUBITE
DU CHÊNE (L’ENCRE DES CHÊNES ROUGES)
Thèse présentée
à la Faculté des études supérieures de l’Université Laval dans le cadre du programme de doctorat en microbiologie
pour l’obtention du grade de Philosophiæ doctor (Ph.D)
DÉPARTEMENT DE BIOCHIMIE ET MICROBIOLOGIE FACULTÉ DES SCIENCES ET DE GÉNIE
UNIVERSITÉ LAVAL QUÉBEC
2008
Résumé
Le Phytophthora ramorum Werres est responsable de la mort de dizaines de milliers de chênes sur la côte ouest-californienne depuis 1995. On nomme cette maladie l’encre des chênes rouges ou la mort subite du chêne. Cet organisme appartient au même genre que le
Phytophthora infestans, agent causal de la famine irlandaise de la pomme de terre du 19e
siècle. Il fut découvert en 1993, infectant des rhododendrons et des viornes en Europe. Depuis, il a été démontré qu’il pouvait infecter plus d’une centaine d’espèces de plantes, non seulement sur la côte ouest américaine, mais également au Canada et dans plusieurs pays d’Europe. Des mesures de quarantaine ont été mises en place, essentiellement en pépinières où sa prolifération est la plus efficace, afin d’éviter sa propagation dans d’autres États américains ou d’autres pays. Il est donc nécessaire de développer des outils de détection, et d’identification.
La détection moléculaire et le génotypage peuvent être des outils importants pour découvrir et mieux comprendre la biologie de ces populations et les mouvements de cet agent pathogène. La disponibilité des séquences complètes du génome du P. ramorum, depuis 2004, apporte une nouvelle ressource pour l’identification de gènes portant du polymorphisme et la conception d’outils pour les études de populations. Les objectifs de ce projet de recherche proposés sont : de développer des outils moléculaires pour identifier le
P. ramorum et le différencier des autres Phytophthora; de découvrir des loci différenciant
le polymorphisme intra-spécifique du P. ramorum; de réaliser des études de populations entre les populations européennes et nord-américaines connues avec les différents marqueurs développés.
Par le biais d’analyses bioinformatiques et de séquençage d’ADN, la méthodologie employée consiste au développement d’outils de diagnostic tels que la PCR en temps réel utilisant des sondes et amorces spécifiques au P. ramorum sur trois loci et de les distinguer des autres espèces de Phytophthora connues. D’ailleurs, ces analyses ont permis d’identifier plusieurs polymorphismes de nucléotides simples (SNPs) (Single Nucleotide
codons. Dans une collection d’isolats provenant d’Europe et d’Amérique du nord, des profils de SNPs distincts et fortement corrélés avec l’origine géographique ont été identifiés. Les populations du P. ramorum en Californie et Oregon, présentent généralement trois profils uniques de SNPs et semblent plus être dérivés d’un ou quelques clones, quelques individus nouvellement trouvés présentant des insertions et délétions. En Europe plusieurs génotypes ont été retrouvés et les gènes sélectionnés semblent avoir des homologies avec des protéines de la paroi cellulaire et donc pourraient jouer un rôle dans l’adaptation. Cette thèse présente donc l’utilisation et la découverte de nouveaux gènes présentant du polymorphisme permettant de détecter et différencier le P. ramorum des autres espèces de Phytophthora mais également de connaître les origines et une identification des individus par leurs polymorphismes.
Abstract
Phytophthora ramorum Werres is responsible of mortality of ten thousand of oak
trees on the California coast since 1995. This disease is called sudden oak death, Ramorum blight, canker. This organism is on the same genus than Phytophthora infestans, the causal agent of Irish potato famine on the 19th century. P. ramorum was discovered in 1993 infecting Rhododendron and Viburnum in Europe. Since, it was demonstrated that it could infect more than one hundred plant species, not only on the American west coast but also in Canada and many European countries. Quarantine measures were placed in effect, principally in plant nurseries where the spread is more efficient to prevent propagation in other American States or other countries. That is necessary to develop detection and identification tools.
The molecular detection and genotyping could be important tools to discover and understand le biology of the population and movement of this pathogen. The availability of complete sequences from the P. ramorum genome since 2004 permitted to use new resources for identification of gene sharing potential polymorphism and conception of tools for population studies. The objectives of this research project were: to develop molecular tools for identification of P. ramorum and differentiate it from other Phytophthora species; to discover loci differentiating intra-specific polymorphism of P. ramorum; and finally to realize population studies between European and North American population known with the different markers developed.
With utilisation of bioinformatics and DNA sequencing, the method used was to develop diagnosis tool with real-time PCR using specific probes and primers for P.
ramorum on three different loci and distinguish it from the other Phytophthora species
known. Moreover, these analyses allowed identification of multiple single nucleotide polymorphisms (SNPs), in thirteen genes for a total of 6.3 kb based on codon volatility. In a collection of isolates from Europe and North America, distinct SNPs profiles and correlated with the geographic origin were identified. P. ramorum populations in California and Oregon present generally three unique SNPs profiles and seem to drift from one or few clones, new individual newly discovered with insertion-deletion mutation. In Europe, many
genotypes were found and the selected genes had homologies with proteins implicated in cell wall. This could have an implication on adaptation and evolution. This thesis presents utilisation and discovery of new genes sharing polymorphisms allowing to detect and differentiate P. ramorum form other Phytophthora species and moreover to known the origin and identification of individuals by their polymorphisms.
Avant-Propos
Les quatre chapitres présentés dans cette thèse sont présentés sous forme d’articles publiés, soumis ou qui seront soumis dans différents journaux scientifiques. L’emploi de la langue anglaise a donc été utilisé pour ces chapitres pour la diffusion dans des revues internationales. L’introduction, la discussion et la conclusion générale de la thèse, sont quant à elles rédigées en français. Un résumé en français de même que les références de chaque chapitre ont été ajoutés à chaque section afin de rendre la lecture plus facile. Chaque chapitre, issu d’un article scientifique, est également muni d’un résumé en anglais et français, ainsi que les noms des coauteurs. L’ordre des chapitres ne se représente pas nécessairement selon l’ordre chronologique de publication mais plutôt selon l’ordre chronologique du projet de recherche et des liens entree les différents chapitres. Des précisions sont fournies ainsi que la contribution des différents coauteurs des quatre articles formant le corps de la thèse :
Le premier chapitre de cette thèse fut publié dans la revue Phytopathology, de la Société américaine de Phytopathologie, en mai 2007 :
Bilodeau G.J., Levesque C.A., de Cock, A.W.A.M., Duchaine C., Brière S., Uribe P., Martin F. N., and R. C. Hamelin. (2007). Molecular detection of Phytophthora ramorum
by real-time polymerase chain reaction using TaqMan, SYBR Green, and molecular beacons. Phytopathology 97, 632-642.
Il présente le développement et la comparaison de différents tests moléculaires utilisant la PCR en temps réel et permettant la détection de P. ramorum. J’ai réalisé l’ensemble des travaux et la rédaction de cet article. Drs. Hamelin, Lévesque et Duchaine, ont supervisé l’ensemble des travaux et ont participé à la rédaction de cet article. Les quatre autres coauteurs ont récolté et distribué le matériel nécessaire aux expérimentations de même qu’à la rédaction de l’article. Plusieurs communications orales et sous forme d’affiches ont d’ailleurs été présentées dans des congrès nationaux et internationaux depuis
le début du projet. De plus, une note d’application utilisant ses sondes PCR, développées en lecture finale (endpoint), à été réalisée et est distribuée par la compagnie Thermo electron
corporation.
Le second chapitre se réfère toujours au diagnostic du P. ramorum mais cette fois ci dans le développement d’un test de détection PCR en temps réel, mais en multiplex. Plusieurs sondes ciblant différents gènes du P. ramorum ainsi qu’une sonde contrôle d’extraction d’ADN, Phytophthora, Oomycete et plante ont été développées. Ces travaux ont été soumis à la revue : Applied and Environmental Microbiology, sous :
Bilodeau, G. J., Pelletier, G., Pelletier, F., Lévesque, C. A., and R. C. Hamelin. Multiplex
Real-Time PCR for detection of Phytophthora ramorum, the causal agent of sudden oak death.
J’ai réalisé la majorité des travaux et la rédaction de cet article. Drs. Richard C. Hamelin, et André Lévesque, ont supervisé les travaux et ont participé à la rédaction de cet article. Les deux autres coauteurs ont participé aux développements et expérimentations du projet, de même qu’à la rédaction de l’article. Les résultats de ce chapitre ont également été présentés lors de congrès internationaux.
Le troisième chapitre de cette thèse fut publié dans la Revue canadienne de
phytopathologie (Canadian Journal of Plant Pathology), de la Société canadienne de
Phytopathologie, en décembre 2007 :
Bilodeau, G. J., Lévesque, C. A., de Cock, A.W.A.M., Brière, S.C, and R. C. Hamelin. (2007). Differentiation of European and North-American genotype of Phytophthora
ramorum by Real-time polymerase chain reaction primer extension. Can. J. Plant Pathol. 29, 408-420.
Cet article présente un test en PCR en temps réel permettant le génotypage et la différenciation des isolats européens et nord-américains du P. ramorum. J’ai réalisé l’ensemble des travaux et la rédaction de cet article. Drs. Richard C. Hamelin, et André Lévesque, ont supervisé l’ensemble des travaux et ont participé à la rédaction de cet article.
Les deux autres coauteurs ont récolté et distribué du matériel nécessaire aux expérimentations et ont participé à la rédaction de l’article.
Le dernier chapitre porte sur le génotypage et l’étude des populations du P.
ramorum utilisant plusieurs gènes présentant du polymorphisme SNPs et sélectionnés
utilisant la notion de volatilité du génome. Cet article porte le nom:
Bilodeau, G. J., Lévesque, C. A., and R. C. Hamelin. SNP Discovery and Multilocus
Strain Genotyping in Phytophthora ramorum.
Il sera soumis après le dépôt de la thèse. Deux communications orales ont d’ailleurs été présentées sur ce chapitre. J’ai réalisé l’ensemble des travaux et la rédaction de cet article. Drs. Richard C. Hamelin, et C. André Lévesque, ont supervisé l’ensemble des travaux et ont participé à la rédaction de cet article.
Remerciements…
Cette thèse a été réalisée grâce au soutien de mon comité aviseur composé de Dr Caroline Duchaine, ma directrice, Dr Richard Hamelin, mon codirecteur et Dr André Lévesque. Merci pour leur soutien et leurs conseils tout au long de ce projet de doctorat et dans l’élaboration et la poursuite des travaux. Je tiens tout d’abord à remercier Dr Richard Hamelin pour m’avoir accueilli dans son laboratoire et proposé ce projet de doctorat. Ce travail avec lui et son équipe, m’a permis d’évoluer dans le monde de la recherche avec autonomie, esprit scientifique mais surtout plaisir et enthousiasme. Merci également au Dr André Lévesque pour le support et l’expertise et ses précieux conseils. Merci au Dr Caroline Duchaine pour son soutient ses conseils et son encadrement pour la réalisation de ce doctorat. Je souhaite à tout étudiant un comité aviseur aussi agréable et enrichissant.
Je remercie chaleureusement l’équipe du Dr Hamelin au Centre de Foresterie des Laurentides (CFL), particulièrement ceux qui ont mis main à la pâte au projet, Gervais Pelletier, Josée Grondin et Françoise Pelletier. Merci également à Dr Danny Rioux pour sont aide et collaboration au projet. Un grand merci aussi à l’équipe du Dr Lévesque d’Agriculture et Agroalimentaire Canada, Ottawa pour leur aide, conseils et surtout pour le plaisir que j’ai eu de travailler avec eux. Plus précisément, Nicole Desaulniers, Hélène Rocheleau et Tharcisse Barasubyiet pour leur aide si précieuse.
Un gros merci aussi aux nombreux collaborateurs qui ont su me fournir en matériel (ADN), équipement et expertise : Stéphan Brière et son équipe à l’ACIA, Dr Arthur deCock, Dr Frank Martin, Dr Micheal Coffey, Dr Claude Husson, Dr Sabine Werres, Dr Anne Chandelier, et Dr Alenka Munda. Merci à toutes les collaborations de près et de loin qui ont permis d’échanger sur le monde de la recherche, des Oomycetes mais également de développer des amitiés.
Merci aux fonds CBS (Stratégique en biotechnologie canadienne) et aux fonds IRTC 04-0045RD (Initiative en recherche et technologie du CBRNE) pour leur support financier au cours de ce projet de recherche.
Et finalement, un gros merci à mon épouse et ma famille pour leur support moral, encouragements qu’ils ont su me manifester avec fierté tout au long de mes études et qui m’ont toujours poussé à aller jusqu’au bout et plus loin …
Table des matières
Résumé...i
Abstract... iii
Avant-Propos ...v
Table des matières ...x
Liste des tableaux...xiv
Liste des figures ...xvi
Introduction générale ...1
Introduction générale ...1
1. La maladie...2
1.1 La découverte...2
1.2 Les hôtes du P. ramorum...2
1.3 Localisation...4 1.4 Impact économique...5 1.5 Mesures...6 2. Biologie...7 2.1 Phytophthora ...7 2.2 Phytophthora ramorum...8
2.2.1 Caractères morphologiques du P. ramorum ...8
2.2.2 La pathogénicité et variabilité morphologique et moléculaire du P. ramorum ...10
2.2.3 Le génome du P. ramorum ...10
3. Identification et détection du P. ramorum ...12
3.1 L’isolement par culture...12
3.2 Les méthodes moléculaires...12
3.2.1 L’ELISA ...13
3.2.2 La technique de PCR ...14
3.2.3 La technique de PCR en temps réel ...14
3.2.4 Génotypage des populations du P. ramorum...15
4.0 Problématique (objectifs et hypothèses de recherche)...16
5.0 Références...28
Chapitre I ...36
Molecular Detection of Phytophthora ramorum by Real Time- Polymerase Chain Reaction Using TaqMan, SYBR®Green and Molecular Beacons ...36
I.1 Résumé/Abstract...38
I.1.1 Résumé...38
I.1.2 Abstract...39
I.2 Introduction...40
I.3 Materials and methods ...42
I.3.1 Isolates ...42
I.3.2 DNA sequencing...43
I.3.4 Design of molecular beacon and TaqMan probes ...44
I.3.5 PCR amplification...45
I.3.5.a SYBR® Green ...46
I.3.5.b TaqMan® probe...46
I.3.5.c Molecular beacon...46
I.3.5.d Standard curves...47
I.3.6 DNA extraction and pathogen isolation from infected plant material...47
I.4 Results...48
I.4.1 Phytophthora sequence divergence ...48
I.4.2 β-tubulin PCR assays with TaqMan, molecular beacon, and SYBR® Green..49
I.4.3 Comparison of ITS, β-tubulin and elicitin TaqMan probes using real-time PCR 50 I.4.4 Detection of Phytophthora ramorum in infected samples...51
I.5 Discussion...52
I.6 Acknowledgements...57
I.7 References...67
Chapitre II ...72
Multiplex Real-Time PCR for detection of Phytophthora ramorum, the causal agent of sudden oak death...72
II.1 Résumé/Abstract...74
II.1.1 Résumé...74
II.1.2 Abstract...75
II.2 Introduction...76
II.3 Materials and methods ...78
II.3.1 Isolates from culture collection...78
II.3.2 Isolates from field ...78
II.3.3 DNA isolation from ELISA lysates ...79
II.3.4 Design of primers and probes ...79
II.3.5 Multi-gene P. ramorum multiplex, targeting Phytophthora ramorum and Phytophthora genus. ...80
II.3.6 Hierarchical multiplex, targeting Phytophthora ramorum, Phytophthora genus, oomycetes and plants...81
II.3.7 Standard curves...82
II.4 Results...82
II.4.1 Multiplex assay targeting P. ramorum and Phytophthora genus ...82
II.4.2 P. ramorum and Phytophthora genus multiplex assay on infected host material 84 II.4.3 P. ramorum and Phytophthora genus multiplex assay using ELISA lysate DNA 85 II.4.4 Hierarchical and RuBisCO multiplexing...85
II.5 Discussion...87
II.6 Acknowledgements...93
II.7 References...108
Chapitre III...116
Differentiation of European and North-American genotype of Phytophthora ramorum by Real-time polymerase chain reaction primer extension...116
III.1 Résumé/Abstract...118
III.1.1 Résumé...118
III.1.2 Abstract...119
III.2. Introduction...120
III.3. Materials and methods ...123
III.3.1 Isolates and DNA extraction...123
III.3.2 Primer design for DNA sequencing...124
III.3.3 DNA sequencing and ASO primer extension...125
III.3.4 Allele-specific genotyping...126
III.3.5 Allele-specific genotyping of field samples ...127
III.4 Results...127
III.4.1 DNA polymorphisms...127
III.4.2 Genotyping...128
III.4.5 Genotyping of field samples ...128
III.5 Discussion...130
III.6 Acknowledgements...134
III.7 References...150
Chapitre IV ...153
SNP Discovery and Multilocus Strain Genotyping in Phytophthora ramorum. ...153
IV.1 Résumé/Abstract...155
IV.1.1 Résumé...155
IV.1.2 Abstract...156
IV.2 Introduction...157
IV.3. Materials and methods ...159
IV.3.1 Isolates and DNA extraction...159
IV.3.2 Codon volatility genome determination ...160
IV.3.3 Selection of genes with high and low codon volatility...160
IV.3.4 Primer design ...161
IV.3.5 PCR amplification...161
IV.3.6 PCR-Single-strand conformation polymorphism (SSCP) ...162
IV.3.7 Sequence analysis, SNP, and haplotype determination ...162
IV.3.8 Sequence and population analyses...163
IV.4 Results...164
IV.4.1 Phytophthora ramorum codon volatility ...164
IV.4.2 Primers and PCR amplification ...165
IV.4.3 Sequences...165
IV.5 Discussion...168
IV.6 Acknowledgements...174
IV.7 References...189
IV.8 Supplement material ...194
Discussion générale et conclusion ...199
Références (Discussion générale et conclusion)...209
Annexes ...211
Annexe 1 ...212
Tableaux des hôtes naturels confirmés et possibles du Phytophthora ramorum...212
Annexe 2 ...241
Cycle de vie probable de Phytophthora ramorum...241
Annexe 3 ...242
Liste des tableaux
Introduction
Boite 1.0 Milieu de culture PARP...12 Boite 2.0 Qu’est-ce que la volatilité? ...19
Chapitre I
Table 1. Isolates of Phytophthora species from different culture collections used in this study...58 Table 2. Primers and probes used for PCR assays targeting Phytophthora spp. and P.
ramorum...60
Table 3. Isolates of Phytophthora species sequenced in this study and GenBank accession numbers for β-tubulin and elicitin genes ...61 Table 4. Number of cycles before fluorescence is detected in Phytophthora ramorum
isolates tested by real-time PCR using three different gene regions (ITS, β-tubulin and elicitin) with TaqMan ...62 Table 5. Detection of Phytophthora ramorum in infected plant material by real-time PCR
using three gene regions with TaqMan and comparison with other methods ...63 Chapitre II
Table 1. Isolates of Phytophthora and other oomycete species used in this study...94 Table 2. Primers and probes used in the multiplex PCR assays targeting Phytophthora
ramorum, Phytophthora genus, oomycetes and plants...95
Table 3a. Result of the multiplex real-time PCR assay comprising three P. ramorum specific TaqMan probes and one Phytophthora genus TaqMan tested on a collection of Phytophthora ramorum from pure cultures...96 Table 3b. Results of the multiplex real-time PCR assay comprising three P. ramorum
specific TaqMan probes and one Phytophthora genus TaqMan tested on a collection of Phytophthora and Pythium from pure cultures ...97 Table 4. Results of the multiplex real-time PCR assay comprising three P. ramorum
specific TaqMan probes and one Phytophthora genus TaqMan tested with field
samples from a ring trial ...98 Table 5. Results of the multiplex real-time PCR assay comprising three P. ramorum
specific TaqMan probes and one Phytophthora genus TaqMan. tested with field
samples from ELISA lysate, survey 2006 CFIA ...101 Table 6. Multiplex real-time PCR assay combining TaqMan probes targeting plant,
oomycete, Phytophthora and P. ramorum tested with samples from different hosts infected by various species of Phytophthora...103 Supplemental Table 1. Alignment of the internal transcribed spacer 1, 5.8S ribosomal RNA
gene, and internal transcribed spacer 2 sequences with oomycetes, ascomycetes and basidiomycetes...113
Supplemental Table 2. Alignment of RuBisCO (ribulose 1,5-bisphosphate carboxylase) sequences with different kind of the chloroplast plant species...115
Chapitre III
Table 1. Isolates of Phytophthora ramorum from different culture collections used in this study...135 Table 2. Primers used for PCR assays targeting Phytophthora spp. and P. ramorum. ...137 Table 3. ASO genotyping of β-tubulin gene in a selection of Phytophthora ramorum from
different geographic origins and mating types. ...138 Table 4. ASO genotyping of CBEL gene in a selection of Phytophthora ramorum from
different geographic origins and mating types. ...140 Table 5. Allele-specific oligonucleotide genotyping of field samples (nurseries). ...142 Table 5 (suite). Allele-specific oligonucleotide genotyping of field samples (nurseries). .145
Chapitre IV
Table 1. Isolates of Phytophthora ramorum from different culture collections used in this study with genotypes bases on SNP profiles. ...175 Table 2. Primers used for amplification and DNA sequencing ...178 Table 3. Characteristics of Phytopthora ramorum genes sequenced...180 Table 4. Comparison of divergence in European and North American populations of P.
ramorum...181
Table 5. Polymorphism comparison between North American and European populations of
P. ramorum ...182
Table 6: Multilocus genotypes of P. ramorum from Europe and North America at 13 loci ...183 Table 7: Genotype frequencies and heterozygosis at 13 different loci in and between
European and North-American population...184 Supplement material 1. Isolates of Phytophthora ramorum used in this study with the
Genbank accession number. ………..197
Annexe I
Tableau annexe 1a ...212 Tableau annexe 1b ...221
Liste des figures
Introduction
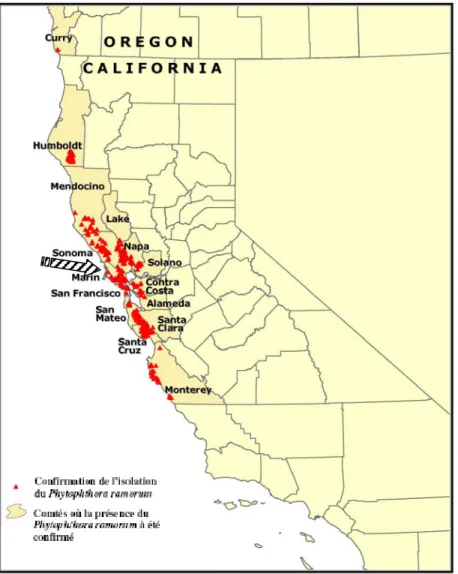
Figure 1. Distribution du Phytophthora ramorum en Californie et en Oregon le 15 février
2008. ...20
Figure 2 (a et b). Aperçu des dommages causés par l’encre des chênes rouges dans les collines du « China Camp State Park », en Californie. Les arbres en gris sont morts ou atteints par la maladie. ...21
Figure 3. Symptômes de la maladie de l’encre des chênes rouges ...23
Figure 4. Localisation du P. ramorum en Europe...24
Figure 5. Localisation du P. ramorum en Amérique du Nord...25
Figure 6. Structures sexuées et asexuées chez Phytophthora ...26
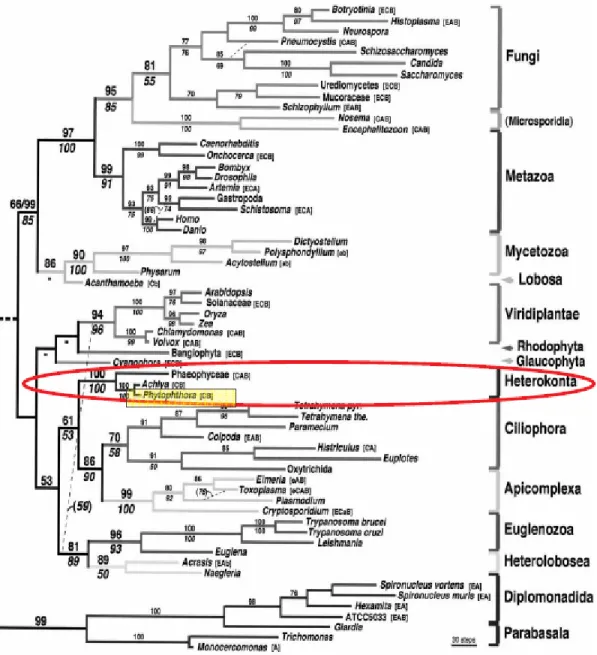
Figure 7. Arbre phylogénétique du monde du vivant basé sur des séquences d’ADN ribosomiaux. ...27
Figure 8. Deux exemples de calculs de la volatilité des codons...19
Chapitre I Figure 1. Real-time PCR of Phytophthora species assayed with TaqMan (■), molecular beacons (●) and SYBR® Green (▲), targeting the β-tubulin gene. ...65
Figure 2. Standard curves based on dilution of Phytophthora ramorum DNA for TaqMan assays targeting ITS, β-tubulin, and elicitin. ...66
Chapitre II Figure 1. Standard curves based on dilution of Phytophthora ramorum DNA for TaqMan assays targeting P. ramorum on ITS, β-tubulin, and elicitin and Phytophthora species on β-tubulin...106
Figure 2. Standard curves shape based on dilution of Phytophthora ramorum DNA for multiplex and simplex TaqMan assays targeting P. ramorum. ...107
Chapitre III Figure 1. SNPs observed on the chromatogram sequence of β-tubulin and CBEL:...148
Figure 2. Allele-specific oligonucleotide (ASO) genotyping using real-time PCR amplification with SYBR® Green on SNP at position 279 of β-tubulin...149
Chapitre IV Figure 1. Distribution of volatility of 10252 genes of P. ramorum...185
Figure 2. SSCP profiles of eight P. ramorum isolates, four from Europe and four from North America, with different migration profiles...186
Figure 3. Agarose gel electrophoresis of the PCR amplicon of the gene 83987 with primers 83987-87U and 83987-594L on 1.5% agarose gel, TAE buffer 1X, stained with
ethidium bromide...187 Figure 4. Unweighted pair group dendrogram presenting the different genotypes. ...188
Annexe II
Introduction générale
Les organismes pathogènes des plantes du genre Phytophthora ont depuis longtemps occupé une place de choix dans les priorités de recherche des phytopathologistes. Au XIXe siècle, la famine irlandaise, causée par la destruction des cultures de pommes de terre par le Phytophthora infestans (Mont.) de Bary, a provoqué de grands changements économiques et sociologiques dans le pays ainsi que l’émigration de près d’un quart de la population vers les États-Unis d’Amérique. Le Phytophthora
cinnamomi Rands a d’autre part causé de sérieux problèmes dans les forêts australiennes.
Une épidémie causée par cet agent pathogène, qui peut s’attaquer à plus de neuf cents plantes hôtes, a détruit une grande partie du couvert forestier et des arbustes dans les forêts d’eucalyptus (Eucalyptus marginata Sm.) de l’ouest du pays (Shearer et Tippett, 1989). Étant donné l’importance de l’industrie du bois d’eucalyptus, cette épidémie a eu des effets néfastes non seulement sur les écosystèmes, mais aussi sur ce secteur de l’économie (Stukely et al., 2007).
Plus récemment, le Phytophthora ramorum Werres, a causé « la mort subite du chêne » ou « l’encre des chênes rouges ». Cette maladie affecte principalement les espèces du groupe des chênes rouges ainsi que le lithocarpe de Californie (tanoak) sur la côte ouest californienne depuis les années 1990. Également problématique en pépinière, ce microorganisme affecte, tout comme le P. cinnamomi, une grande variété de plantes hôtes et cause de sérieux soucis aux exportateurs et importateurs de végétaux potentiellement sensibles à cet agent pathogène. Depuis sa découverte, plusieurs interceptions de cet organisme, qui fait l’objet de mesures de quarantaine, ont été rapportées en pépinière chez différentes plantes et dans plusieurs pays. Une revue des connaissances et des concepts de cette maladie et des méthodes d’identification et de détections sera discutée dans cette introduction afin d’exposer les problématiques présentées dans cette thèse.
1. La maladie
1.1 La
découverte
Depuis 1994-1995, on dénombre la mortalité de milliers de lithocarpes et de chênes dans le comté Marin de la Californie (Fig. 1) (McPherson et al., 2001; Svihra, 1999), particulièrement le Lithocarpus densiflorus (Hook.et Arn.) Rehder et le Quercus agrifolia Née, un représentant appartenant à la famille des chênes rouges. Cette maladie appelée « encre des chênes rouges » ou « mort subite du chêne », traduction de l’anglais sudden oak
death ou SOD, a décimé environ 80% des chênes de la Californie depuis son apparition
(Hansard, 2003). Sujet d’actualité dans plusieurs journaux comme le New York Times, cette maladie fut classée à la 43e position des 100 meilleures histoires scientifiques en 20021. Elle cause des chancres saignants ou suintants sur le tronc des chênes et finit par mener à la mort de l’arbre (Fig. 2). Le nom « mort subite du chêne » fut donné par différents média américains après qu’on eut rapporté que la cime des arbres affectés semblait mourir très rapidement, la couleur du feuillage tournant d’un vert vigoureux à brun dans une période de quelques semaines (Rizzo et al., 2002; Svihra, 1999). Ce n’est qu’en 2000 qu’on réalise que l’organisme en cause appartient au genre Phytophthora. Il est nommé officiellement en 2001 Phytophthora ramorum (Werres et al., 2001).
1.2 Les
hôtes
du
P. ramorum
Jusqu’à maintenant, en faisant le décompte à partir de sites Web européens, américains et canadiens, on dénombre plus de 120 espèces végétales, réparties dans quelque 70 genres et 35 familles, sensibles à cet agent pathogène. En plus des chênes rouges (Lithocarpus densiflorus, Quercus agrifolia, Q. kellogii Newberry), on retrouve le séquoia (Sequoia sempervirens (D.Don.)Endl.), le sapin de Douglas (Pseudotsuga menziesii var.
menziesii (Mirb.) Franko.), deux essences commerciales très importantes, ainsi que
plusieurs arbres et arbustes ornementaux parmi les hôtes potentiels du P. ramorum. Une liste répertoriant les hôtes naturels et possibles de P. ramorum a été dressée dans le PRA (« Pest Risk Assesment ») de l’Agence canadienne d’inspection des aliments (ACIA) en 2006 (Rioux, et al., 2006) (Cf. Annexe 1). Quant au chêne des marais (Quercus palustris Münchh) et au chêne rouge (Q. rubra L.), que l’on retrouve plus au nord-est des États-Unis et également au Québec (spécifiquement le Q. rubra), ils ont été infectés artificiellement et ont présenté les mêmes symptômes que les chênes atteints en Californie, ce qui laisse croire à un risque potentiel de maladie chez ces hôtes (Tooley et al., 2004). Plus précisément au Québec, une étude de la sensibilité de différentes espèces d’arbres de l’est du Canada est en cours (Simard et al., 2007).
Les symptômes sont assez variables d’un hôte à l’autre. Sur les chênes de la Californie, les principaux symptômes se caractérisent par l’apparition de chancres accompagnée d’un saignement brun foncé à noir (Fig. 3a, b) qui peut survenir exceptionnellement jusqu’à une hauteur de 18,3 m (60 pieds) dans les arbres, ainsi que par le brunissement (Fig. 3c) et la perte des feuilles en plein été. Pour les autres hôtes, on peut retrouver des taches ou nécroses sur les feuilles (Fig 3d), les aiguilles ou les tiges des plantes infectées (Garbelotto et al., 2002a). La dissémination des spores se fait majoritairement par le vent et les éclaboussures lors des pluies ou mouvements dans les flaques d’eau sur les hôtes environnants. Les symptômes varient énormément dépendamment du type d’hôte (le dépérissement, la flétrissure et des lésions) ce qui rend parfois le diagnostic de la maladie très difficile. De plus, sur certaines plantes, l’infection semble parfois négligeable bien que ces plantes puissent toutefois servir de vecteur (réservoir d’inoculum) pour l’inoculation sur un hôte adjacent, un bon exemple étant le laurier de Californie (Umbellularia californica (Hook et Arn.) Nutt.) (Davidson et al., 2002). De plus, ils peuvent contribuer grandement à la propagation de l'agent pathogène, par les mouvements naturels dans l'environnement et par le commerce des végétaux provenant de pépinières infestées dont l’infection serait passée inaperçue.
1.3 Localisation
Jusqu’à présent, l’agent pathogène est retrouvé en pépinière en Europe (Fig. 4) dans les pays suivants : Allemagne, Belgique, Danemark, Espagne, Finlande, France, Irlande, Italie, Norvège, Pays-Bas, Pologne, Royaume-Uni, Slovénie, Suède, République Tchèque et Suisse (Brasier, 2004; De Merlier, 2003; Garbelotto et al, 2002a; Hansen et al., 2003; Herrero et al., 2006; Husson et al., 2007; Lane et al., 2003; Lane et al., 2004; Lilja et al., 2007; Moralejo et Werres, 2002; Werres, 2002; Werres et al., 2001). En Amérique du Nord (Fig. 5), la maladie est aussi présente en pépinière au Canada ainsi qu’aux États-Unis (dans 23 États américains en 2004 (Cave et al., 2007)). En décembre 2007, on répertoriait un total de 21 pépinières situées dans 6 États américains dans lesquelles le P. ramorum avait pu être détecté2. Au Canada, le P. ramorum a seulement été retrouvé en pépinière en Colombie-Britannique près de Vancouver depuis 2003 (CFIA, 2003). Il est resté confiné à ces endroits depuis ce temps là.
Il a été également retrouvé en milieu naturel en Californie et en Oregon ainsi qu’au Royaume-Uni et aux Pays-Bas, où il s’attaque à certains chênes ainsi qu’à d’autres espèces [par exemple le hêtre (Fagus sylvatica L.) et le châtaignier (Castanea sativa Mill.)] depuis janvier 2004 (DEFRA, 2004a; DEFRA, 2004b). Le RAPRA (« Risk Analysis for
Phytophthora ramorum ») estime que le P. ramorum a été retrouvé hors pépinière
(outdoor) dans 7 pays (RAPRA, 2008).
1.4 Impact
économique
L’impact économique du P. ramorum n’est pas facile à déterminer, mais semble être majeur. Les conséquences pour l’industrie forestière pourraient être considérables si les forêts canadiennes venaient à être infectées par cette maladie comme c’est le cas en Californie et en Oregon. Si l’exportation de bois de sciage venait à être réduite, des sommes importantes seraient en jeu puisque le Canada en exporte annuellement pour plus de 20 milliards de dollars3.
Ce sont les impacts indirects envers les pépinières et les entreprises paysagistes dans les zones infectées ou à risque qui sont pour l’instant les plus problématiques. Ainsi, le Canada a fermé son marché à la plupart des cultures végétales des États de l'Oregon et de la Californie en 2001. Si l'accès au marché n'avait pas été rétabli, les pépinières de l'Oregon auraient à elles seules perdu des exportations au Canada d'une valeur de 15 à 20 millions de dollars. En Colombie-Britannique, la valeur estimée du secteur des pépinières et de la floriculture est de 500 millions de dollars et les exportations vers les États-Unis sont estimées à 170 millions de dollars. De plus, si l’exportation de conifères vers l’Asie venait à être interrompue, c’est une industrie de 150 millions de dollars qui serait affectée4. Depuis 2003 et jusqu’en 2006, les coûts en Colombie-Britannique ont été estimés à plus de 3 millions de dollars pour la perte de plants et les ventes perdues et à environ 500 000 $ pour le nettoyage et l’élimination des zones infestées (Kristjansson et Miller, 2008). Seulement en 2007, une enquête à travers différentes pépinières du Canada a permis de tester plus de 46 000 échantillons ce qui a conduit à la détection de 164 échantillons positifs en Colombie-Britannique entre le 1er avril et le 14 décembre 2007 (communication personnelle Stéphan Brière, ACIA, Ottawa).
3 Statistique Canada, CANSIM, Exportations de biens sur la base de la balance des paiements, selon le produit.2006, 228-0003 ; and BC STATS (exportation by province, 1997-2006, Exports to all countries 2006) 4 La Gazette du Canada, Vol. 141, no 14, 7 avril 2007, Règlement sur l'indemnisation relative au
1.5 Mesures
Des mesures de quarantaine ont été mises en place dans plusieurs pays pour éviter une propagation et/ou une introduction de l’agent pathogène. Pour déterminer la présence ou l’absence de cet organisme dans la gamme d’hôtes qui ne cesse de s’élargir, des méthodes de détection doivent être mises en place. Lorsqu'il faut confirmer que les arbres d’un secteur donné sont atteints par la maladie, il est très important de s’assurer que l’agent pathogène en cause est vraiment celui recherché. Pour cela on utilise différentes méthodes de détection comme la mise en culture de l’agent pathogène sur des milieux spécifiques ou des méthodes moléculaires ciblant l’ADN de l’organisme (Cf. introduction section 3). De tels outils permettent alors de mieux contrôler les plants et les zones infestées et ainsi éviter la propagation.
Lors des mesures de quarantaine, un protocole d’éradication5 est enclenché. Tous les plants de la pépinière infectée qui sont des hôtes connus du P. ramorum sont mis en quarantaine. Une zone d’échantillonnage est déterminée selon la nature des hôtes et ces derniers se retrouvant dans les zones infestées (block) sont alors détruits (par brûlage et par fumigations (sols)). Des échantillons d’eau et de sol sont ensuite prélevés pour analyse de la présence du P. ramorum (infestation). Les zones sélectionnées mais non détruites sont mises en quarantaines pour 90 jours. Les plants sont ensuite ré-inspectés et testés de nouveau pour confirmer que le P. ramorum n’est plus présent. Si c’est le cas, les pépinières sont libérées, sinon une autre ronde d’éradication et quarantaine est amorcée. Les pépinières libérées sont toujours surveillées, mais ne sont plus sous quarantaine.
5 Official Regulatory Protocol for Wholesale and Production Nurseries Containing Plants Infected with
Phytophthora ramorum, Confirmed Nursery Protocol: Version 8.0, Revised: July 20, 2007;
United States Department of Agriculture (USDA), Animal Plant Health Inspection Service (APHIS), Plant Protection and Quarantine (PPQ), Center for Plant Health Science and Technology (CPHST), Emergency and Domestic Programs (EDP), Eastern Region (ER), Western Region (WR)
2. Biologie
2.1 Phytophthora
Le nom Phytophthora dérivant du grec phyto (plante) et phthora (destructeur), caractérise bien ce genre car les différentes espèces qu’il regroupe s’attaquent uniquement aux plantes et causent beaucoup de dégâts tant dans les milieux agricoles que forestiers (Erwin et Ribeiro, 1996). C’est d’ailleurs l’apparition d’une maladie causée par le P.
infestans (Mont.) De Bary, entraînant une importante famine en Irlande au milieu du XIXe
siècle, qui marqua la naissance de la phytopathologie (Large, 1940), et ce 20 ans avant que Louis Pasteur étudie la théorie des germes comme agents infectieux (Cooke, 2007). Depuis plus de 150 ans, les scientifiques étudient avec intérêt les organismes appartenant à ce genre, sans cesser de découvrir de nouvelles espèces : à ce jour, plus de 80 espèces ont été identifiées. Les Phytophthora sont les agents pathogènes les plus dévastateurs chez les dicotylédones (Erwin et Ribeiro, 1996). Ils font partie des Oomycètes car ils produisent une spore sexuée appelée oospore (Fig 6a), qui résulte de la fusion entre les deux gamètes [oogone (femelle) et anthéridie (mâle)]. Des structures asexuées sont aussi présentes chez cet organisme, telles les chlamydospores (Fig 6b) et les zoospores (Fig. 6c). La chlamydospore possède une paroi épaisse qui permet de résister longtemps à des conditions difficiles (c’est donc une structure de survie). Les zoospores sont des spores asexuées et biflagellées, qui nagent dans des milieux aqueux (Moore-Landeker, 1982) et sont attirées par les champs électromagnétiques (électrotaxi) des racines végétales. On retrouve chez le genre Phytophthora des espèces homothalliques et hétérothalliques. Autrefois, les Oomycètes étaient classés dans le règne des Fungi. Il font maintenant partie des Straménopiles qui se rapprochent beaucoup plus, par la présence de spores motiles à deux flagelles inégaux et par phylogénie de l’ADN ribosomal, aux diatomées et aux algues brunes (Baldauf et al., 2000; Cavalier-Smith et al., 1994; Van der Auwera et De Wachter, 1997) (Fig. 7). Les Straménopiles, (Stramenopila : « organismes plumeux », ou hétérokontes (Heterokonta : « organismes à deux flagelles différents ») (Fig. 6c) constituent
un sous-règne des Eucaryotes et sont parfois classés dans un règne supérieur, les Chromistes (Cavalier-Smith, 1998; Cavalier-Smith et al., 1994). Les Straménopiles auraient donc perdu leur capacité à faire la photosynthèse. En taxinomie, on peut classer le
P. ramorum comme étant : Eukaryota; Stramenopiles; Oomycetes; Peronosporales; Phytophthora.
On les classait autrefois, parmi les Fungi, car ils présentent des similitudes avec ce règne notamment la formation d’un thalle mycélien. Par contre, les Oomycètes ne possèdent pas ou très peu de chitine dans leur paroi alors que l’on retrouve de bonnes quantités de cellulose et de β-glucanes dans celles-ci. Finalement, ils sont généralement diploïdes, dans leur phase végétative contrairement aux champignons vrais comme les Ascomycètes ou aux Basidiomycètes qui sont généralement haploïdes (Rossman, 2006.).
2.2 Phytophthora ramorum
C’est en 2001 que le nom Phytophthora ramorum (Werres et al, 2001) est donné à l’agent pathogène infectant les branches des rhododendrons et des viornes (Viburnum spp.). Le nom d’espèce ramorum vient du mot ramus qui signifie en latin « branche » et fait référence à la prédilection de cette espèce à s’attaquer aux branches et aux pousses de ses hôtes.
2.2.1 Caractères morphologiques du P. ramorum
Chez le P. ramorum, l’oogone et l’anthéridie ne sont pas produites en culture simple, ce qui suggère qu’il serait autostérile et hétérothallique. Actuellement, comme la reproduction sexuée est peu probable, le cycle d’infection de P. ramorum (C.F. Annexe 2) est possible grâce à la reproduction asexuée. Les différentes populations échantillonnées ne semblent pas, pour l’instant, indiquer de signes de recombinaison. Le P. ramorum peut alors produire des oospores (reproduction sexuée) seulement lorsque les deux types de
compatibilité sexuelle (mating type) (A1 et A2) sont présents. Les deux types sexuels du P.
ramorum ne se retrouvent pas souvent en nature dans un même territoire. La production
d’oospores peut aussi survenir lorsque des espèces différentes, de types sexuels opposés (de sexes différents), croissent ensemble sur un même milieu (Erwin et Ribeiro, 1996). Par exemple, lorsqu’on met en culture le Phytophthora cryptogea Pethybridge et Lafferty, ou alors le Phytophthora cambivora (Petri) Buisman, avec un type sexuel différent du P.
ramorum, l’obtention des structures sexuées est possible (Werres et al., 2001). Des études
de croisements (Brasier et Kirk, 2004; Werres, et Zielke, 2003) ont montré que les populations du P. ramorum provenant d’Europe (EU) présentaient un type sexuel A1 et que celles provenant de l’Amérique du Nord (NA) étaient du type A2 (Brasier, 2003; Werres, Zielke, 2003). Ainsi, initialement, le type sexuel semble être lié à l’origine géographique. Cependant, des études récentes rapportent pour la première fois la présence d’individus de type sexuel A2 en Europe (Belgique). Ce type sexuel semblait jusque là être exclusivement lié aux populations d’Amérique du Nord (Werres et De Merlier, 2003). De même, le type A1 a été retrouvé récemment en Amérique du Nord, plus précisément au Canada et dans les états de Washington et de l’Oregon aux États-Unis (Hansen et al., 2003). Cette nouvelle découverte est importante car des phénomènes de croisements et de recombinaisons entre les deux types sexuels pourraient avoir lieu. Une nouvelle descendance présentant des variations de profils génétiques et de la pathogénicité pourrait changer dramatiquement l’épidémiologie de cet agent pathogène (Brasier, 2003; Erwin et Ribeiro, 1996). Pour s’en convaincre, on peut penser au P. infestans qui, de cette façon, a engendré une plus grande diversité génétique et une augmentation de sa virulence et de sa résistance aux fongicides (Drenth et Goodwin, 1999). Quand la formation d’oogones se produit chez le P. ramorum, elles sont, la plupart du temps, en position terminale et quelquefois latéralement sessiles, d’un diamètre variant de 24 - 40 µm (moyenne de 29.8 - 33µm). Les oospores plérotiques sont de 20 à 36 µm de diamètre. L’anthéridie est de forme arrondie et sa taille approximative est de 12-22 X 15-18 µm (Werres et al., 2001).
2.2.2 La pathogénicité et variabilité morphologique et moléculaire du P. ramorum
La pathogénicité des isolats du P. ramorum provenant d’Amérique du Nord et d’Europe a été étudiée et comparée (Brasier et al., 2003; Pogoda et Werres, 2002). L’analyse des empreintes d’ADN (AFLP) et microsatellites des populations de P. ramorum en Amérique du Nord et en Europe semble démontrer des profils assez différents (Garbelotto et al., 2002; Ivors et al., 2006; Ivors et al., 2004). Cependant, ces analyses indiquent que celles-ci seraient plus variables en Europe qu’en Amérique du Nord et ne permettent pas de cerner la source potentielle de l’épidémie. Par contre, les populations nord-américaines semblent démontrer une plus grande variabilité dans leur morphologie (Werres et Kaminski, 2005). Cela peut suggérer que les populations nord-américaines et européennes sont différentes sur le plan adaptatif (Brasier, 2003). Néanmoins, une étude de prédiction des origines et risques de l’encre des chênes rouges (Kluza et al., 2007) complémente les études réalisées précédemment (Guo et al., 2007; Guo et al., 2005; Meentemeyer et al., 2004). Cette étude basée sur des modèles de niches écologiques, tenant compte des climats (les niveaux de la température et de l’humidité sont très important pour l’établissement de la maladie), des topographies, des types d’hôtes (surtout d’origine asiatique), permet aux auteurs de prédire que l’origine du P. ramorum pourrait se situer dans l’est de l’Asie.
2.2.3 Le génome du P. ramorum
En avril 2004, le JGI (Joint Genome Institute) rendait disponible la séquence
complète du génome du P. ramorum (
http://genome.jgi-psf.org/Phyra1_1/Phyra1_1.home.html) (Tyler et al., 2006). Dans la version 1.0 de 2004, les séquences de ce génome ont pu être obtenues grâce à la technique de séquençage à haut débit « shotgun » et l’assemblage a été réalisé en utilisant le logiciel JGI assembler dénommé « Jazz » pour une couverture de sept fois le génome. Après nettoyage des séquences vectrices et de faibles qualités, plus de 1 million de lectures ont été assemblées
en 2576 structures (scaffolds), pour une couverture totalisant 66,6 Mpb. La moitié de la séquence du génome brut de P. ramorum est contenue dans 63 structures d’au moins 308 Kpb de longueur. Ces assemblages ont été annotés en utilisant la plateforme d’annotation de JGI. Des modèles de gènes, les prédictions associant différents transcrits et/ou protéines ainsi que la création de cartes, ont été réalisés avec des méthodes utilisant les ADNc et des homologies de protéines. De plus, le génome du Phytophthora sojae (86 Mpb), séquencé en 2004, http://genome.jgi-psf.org/Physo1_1/Physo1_1.info.html, a montré une certaine conservation avec celui du P. ramorum apportant un support additionnel à son annotation. Les fonctions des gènes ont été assignées automatiquement basées sur les homologies des gènes connus. En 2006, l’annotation version v1.1, a pu déterminer que le génome contenait 15743 modèles de gènes. L’annotation est toujours en cours, et une partie est disponible sur la base de données du NCBI (GenBank) depuis la fin 2006. Depuis, le génome du
Phytophthora infestans (190 Mpb) est disponible via le site internet du Broad Institute
(http://www.broad.mit.edu/annotation/genome/phytophthora_infestans). Le génome du P.
capsici sera également disponible sous peu après utilisation d’un mélange des technique de
séquençage Sanger et 454 (454 Life Sciences, Branford, CT) par le JGI (http://www.jgi.doe.gov/sequencing/why/CSP2006/Pcapsici.html). Celui d’un autre Oomycète, le Pythium ultimum, est en cours de séquençage par le consortium TIGR (http://cpgr.tigr.org).
3. Identification et détection du P. ramorum
3.1 L’isolement par culture
Pour confirmer que des plants sont infestés par la maladie il est très important de s’assurer que l’organisme en cause est vraiment celui recherché. L’approche traditionnelle (classique) pour identifier cet agent pathogène est sa mise en culture (Garbelotto et al., 2001; Werres et al., 2001a). Il s’agit de faire croître l’agent pathogène sur des milieux sélectifs, comme le PARP-V8 (Jeffers et Martin, 1986) permettant sa croissance et l’identification par ses traits morphologiques caractéristiques.
3.2 Les méthodes moléculaires
Dépendamment de la saison, du type d’hôte, de l’utilisation de fongicides, la culture des organismes pathogènes à partir des plantes n’est pas toujours facile. Elle demande beaucoup de temps, une bonne expertise et les risques de contamination sont élevés. Ces
Boite 1.0 Milieu de culture PARP
Le PARP (pimaricine-ampicilline-rifampicine-pentachloronitrobenzène) est un milieu sélectif pour les oomycètes. La pimaricine et le pentachloronitrobenzène (PCNB) (Erwin et Ribeiro, 1996; Jeffers et Martin, 1986) sont des fongicides et l’ampicilline et la rifampicine sont des antibiotiques contre les bactéries.
On rajoute ces composés généralement à des milieux V8, CMA (corn meal agar) ou PDA (potato dextrose agar). Les Phytophthora et les Pythium y poussent très bien mais l’ajout de hémexazol permet généralement d’inhiber la croissance des Pythium.
raisons expliquent pourquoi des méthodes moléculaires ont été développées au cours des dernières années (Martin et al., 2000). Des méthodes de détection à partir d’ADN sont possibles grâce à la réaction de polymérisation en chaîne (PCR) qui permet d’amplifier des copies multiples d’une portion ciblée de l’ADN. Cette technique offre la possibilité d’être utilisée directement sur les tissus de l’hôte pour y détecter la présence de l’agent pathogène. De plus, ces méthodes permettent d’obtenir des résultats en quelques heures, contrairement à plusieurs jours pour la croissance sur milieux de culture (Garbelotto et al., 2001; Levesque, 1997).
3.2.1 L’ELISA
La première méthode utilisée dans le cas du P. ramorum est l’immunodosage. La technique ELISA ("enzyme-linked immunosorbent assay") (Gaastra, 1984) fait appel aux anticorps spécifiques aux protéines du Phytophthora. Ces anticorps sont couplés à des marqueurs chémiluminescents. En raison de leur très grande spécificité, les anticorps sont souvent utilisés pour détecter et mesurer une protéine spécifique. L’intensité lumineuse de l’enzyme peut être évaluée en fonction de l’absorbance. Cette méthode peu coûteuse et rapide est utilisée dans le cas du P. ramorum. Utilisée en première analyse, un résultat positif avec ce procédé permet de confirmer la présence d’un Phytophthora dans l’échantillon visé. L’ACIA (l’Agence canadienne d’inspection des aliments) utilise plus spécifiquement l’enzyme alcaline phosphatase conjuguée à l’anticorps «
anti-Phytophthora » (anti-Phytophthora Reagent Set Instructions, numéro de catalogue SRA 92600,
Agdia Inc. Elkhart, IN). Il est à noter que cette méthode présente certaines limites. Elle détecte seulement le genre Phytophthora et non l’espèce P. ramorum. De plus, elle donne parfois des faux négatifs (Osterbauer et Trippe, 2005) selon le seuil de détection fixé par les expérimentateurs (Stéphan Brière communication personnelle). Une fois des résultats positifs obtenus (pour éliminer ainsi, le plus possible d’échantillons qui seraient négatifs pour la présence de Phytophthora), des tests faisant appel à la technique de PCR spécifique au P. ramorum sont utilisés sur ces échantillons pour confirmer la présence de l’agent pathogène.
3.2.2 La technique de PCR
Un protocole d’amplification d’une région de l’ADN ribosomal ITS (« Internal
Transcribed Spacer ») a été développé par PCR imbriquée (« nested ») au début de la
découverte de l’agent pathogène (Davidson et al., 2003). La PCR imbriquée consiste à ré-amplifier un amplicon. L’avantage de cette technique est qu’elle permet un meilleur rendement d’amplification et offre une plus grande sensibilité. Elle est toutefois loin d’être parfaite. Les risques de contamination par de l’ADN (amplicons) sont très grands, ce qui demande de manipuler avec la plus grande précaution. De plus, l’utilisation de la région ITS de l’ADN ribosomal nucléaire, ne différencie pas toujours très bien le P. ramorum du
P. lateralis Tucker et Milbrath, l’espèce de Phytophthora qui montre le plus de
ressemblance phylogénétiquement (Garbelotto et al., 2002) et qui est aussi retrouvée sur la côte ouest américaine. Il a donc fallu trouver d’autres régions ou gènes présentant plusieurs sites polymorphes ou SNP (« Single nucleotide polymorphism ») entre ces deux espèces et les autres espèces appartenant au genre Phytophthora. De récentes phylogénies basées sur des régions de l’ADN mitochondrial (la Cytochrome oxydase II) (Martin et Tooley, 2003) et nucléaire (la β-tubuline et le facteur d’élongation Iα ) (Kroon et al., 2004) ont permis de concevoir des amplifications PCR pour la détection de Phytophthora, incluant le P.
ramorum (Ioos et al., 2006; Martin et Tooley, 2004; Martin et al., 2004).
3.2.3 La technique de PCR en temps réel
La PCR en temps réel (Real Time-PCR) ou quantitative (qPCR), une autre technique de biologie moléculaire a été développée ces dernières années afin de faire la distinction entre des polymorphismes (variations de séquences) et de quantifier des molécules (Schaad, Frederick, 2002). Elle utilise le principe d’hybridation d’un allèle spécifique (ASH) (Jenkins, 2001), par la détection d’acides nucléiques par SYBRGreen ou par des sondes fluorescentes internes. Le SYBRGreen est une molécule fluorescente, agent intercalant non spécifique, qui lorsque l’ADN est double brin, s’insère entre ceux-ci. Il est donc possible de
lire la fluorescence durant l’extension des brins d’ADN par l’ADN polymérase à chaque cycle (Giglio et al., 2003). Cependant, cette molécule est non spécifique et toutes fausses amplifications ou dimérisations d’amorces peuvent produire un signal. Néanmoins, des courbes de dissociation sont disponibles afin de vérifier si d’autres produits d’amplification ont été formés. Le phare moléculaire (« molecular beacon ») (Marras et al., 2003; Tyagi et Kramer, 1996; Tyagi et al., 2000) et le TaqMan (Holland et al., 1991; Lee et al., 1993; Livak, 2003) sont des sondes internes d’acides nucléiques qui s’hybrident avec l’ADN-cible (séquence) lorsqu’elles sont complémentaires, émettant alors une fluorescence. La lecture du signal émis par le phare moléculaire se fait durant l’appariement des amorces et la molécule n’est pas dégradée. Le TaqMan quant à lui se lit durant l’élongation du brin, dégradant ainsi la molécule par l’activité exonucléase de la polymérase et conduisant à l’émission d’une fluorescence en séparant le fluorochrome de l’absorbeur (quencher). Ces techniques peuvent donc s’avérer très utiles pour une détection rapide des agents pathogènes en autant que des régions polymorphiques soient connues (Böhm et al., 1999; Schaad et Frederick, 2002; Weller et al., 2000). Dans la détection du P. ramorum, certaines variations de la technique de la PCR en temps réel ont été développées au cours de ces dernières années (Hayden et al., 2006; Hughes et al., 2006a; Hughes et al., 2006b; Schena
et al., 2006; Tomlinson et al., 2007; Tomlinson et al., 2005; Tooley et al., 2006).
3.2.4 Génotypage des populations du P. ramorum
Comme mentionné plus tôt à la section 2.2.2, les analyses des populations du P.
ramorum au moyen de marqueurs AFLP et microsatellites semblent démontrer des profils
européens et américains assez différents (Garbelotto et al., 2002; Ivors, 2006; Ivors et al., 2004). Les variations semblent plus fréquentes en Europe qu’en Amérique du Nord, ce qui suggère que les populations ont évolué en goulot d’étranglement. Les populations étant séparées, ont donc subi une réduction de la taille effective ainsi qu’une réduction importante de la diversité allélique. Ces analyses démontrent également qu’un autre génotype unique fut découvert en Amérique. D’autres études de populations ont été réalisées dernièrement utilisant les microsatellites en Californie et en Oregon (Cooke, 2007;
Prospero et al., 2004; Prospero et al., 2007). Dans un premier temps, ces études montraient que les populations de l’Oregon en forêt et en pépinière semblaient provenir d’origines indépendantes. De plus, les isolats prélevés en forêt provenaient principalement d’un seul génotype qui s’est diversifié avec le temps.
4.0 Problématique (objectifs et hypothèses de recherche)
Les différents concepts qui viennent d’être évoqués permettent de réaliser la problématique et l’ampleur de la mort subite du chêne et de l’organisme qu’est le
Phytophthora ramorum. Mon projet de doctorat commença en 2002, peu après
l’identification de l’agent pathogène causant l’encre des chênes rouges comme étant le P.
ramorum (Werres et al., 2001). À cette époque, seule la méthode de culture et la technique
de PCR imbriquée (Garbelotto et al., 2002b) permettaient d’identifier l’organisme en cause. Cependant, comme discuté dans l’introduction, ces méthodes ont leurs limites et ne permettent pas toujours de bien distinguer les deux espèces très apparentées se retrouvant dans la même niche écologique. Le P. ramorum avait déjà été retrouvé en Californie et en Oregon, ainsi que dans quelques pays Européens. Comme la maladie n’était toujours pas rapportée au Canada, son introduction à la suite d’échanges commerciaux était crainte par les responsables de l’ACIA. La maladie a été détectée pour la première fois au Canada en mars 2003 et elle est toujours présente aujourd’hui dans certaines pépinières de la Colombie-Britannique. Si l’organisme venait à s’échapper et parvenait à s’établir dans des milieux naturels, les coûts financiers seraient considérables et persistants. Pour éviter ce risque, une demande fut faite par l’ACIA visant l’obtention d’un outil de détection qui permettrait de révéler rapidement et efficacement la présence de l’agent pathogène. Pour bien répondre à cette demande, il est de mise de considérer les deux hypothèses suivantes : 1- le P. ramorum présente des différences inter-spécifiques par rapport aux autres
Phytophthora spp. et 2- il existe du polymorphisme intra-spécifique à l’intérieur même de
Les différents chapitres de cette thèse permettent de répondre aux différents objectifs que nous nous étions fixés. Tout d’abord, les deux premiers chapitres visent à développer des outils moléculaires pour identifier le P. ramorum et le différencier des autres Phytophthora. Le chapitre 1 traite de la conception d’un outil de diagnostic permettant de détecter rapidement et efficacement le P. ramorum. Comme le génome du P.
ramorum n’était pas connu au début de ce doctorat (Cf. introduction 2.2.3), les régions
disponibles sur Genbank concernant les Phytophthora (par exemple celles des ITS, β-tubuline et élicitine) ont donc été utilisées pour la création d’amorces amplifiant ces régions du génome des Phytophthora spp. Par la suite, après séquençage des espèces se rapprochant le plus du P. ramorum, les séquences ont été alignées pour déterminer le polymorphisme du
P. ramorum en comparaison avec les autres Phytophthora. Des amorces et sondes
spécifiques ont ainsi été conçues.
Le deuxième chapitre est une suite logique du premier chapitre. Il partage le même objectif et utilise les mêmes régions génomiques. Par contre, ce chapitre décrit essentiellement la création d’un test de détection mais ici avec une réaction multiplexe, c’est-à-dire que toutes les réactions se réalisent dans le même tube. Les résultats obtenus démontrent que l’utilisation de plus d’une région du génome augmentait la fiabilité du test par rapport à un test utilisant seulement une région. Cette réaction multiplexe permet de gagner du temps mais également de réduire les coûts de détection, deux éléments non négligeables puisque des dizaines de milliers d’échantillons doivent être analysés. De plus, afin d’assurer une plus grande fiabilité du test, une autre sonde et des amorces spécifiques au genre Phytophthora sont incluses dans la réaction permettant d’avoir un contrôle supplémentaire.
Les chapitres 3 et 4 ont pour objectifs de découvrir des loci différenciant le polymorphisme intra-spécifique du P. ramorum, de mener des études visant à différencier les populations européennes et nord-américaines connues tout en développant des outils permettant de détecter ces polymorphismes. Dans le chapitre 3, comme la séquence du génome du P. ramorum n’était pas encore disponible, des différences entre les séquences des souches du P. ramorum provenant d’Europe et d’Amérique du Nord ont pu être mises
en évidence en utilisant les séquences du gène de la β-tubuline et de CBEL (« cellulose
binding elicitor lectin »). Ces différences (SNPs) ont donc permis de distinguer les
populations du P. ramorum entre les deux continents. La détermination de la provenance (Europe vs Amérique du nord) du P. ramorum permet de mieux connaître l’ampleur de cette épidémie et d’en prévenir la propagation. De plus, afin de détecter plus rapidement ces types, des amorces spécifiques ASO (« Allele Specific Oligonucleotide ») ont été développées pour génotyper les individus rapidement et sans séquençage. Cette méthode permet ainsi de génotyper des individus du P. ramorum à partir d’échantillons provenant de cultures pures mais aussi d’échantillons environnementaux, ce qui est particulièrement intéressant lors d’enquêtes en pépinière visant à mieux cibler le génotype et l’origine de l’agent pathogène.
Finalement, le quatrième chapitre vise au génotypage plus exhaustif des différentes populations de P. ramorum. Des études préliminaires avaient montré que plusieurs populations étaient distinctes à la suite d’analyses avec les AFLP ou microsatellites (Cf. introduction 2.2.2). Comme vu au chapitre précédent, deux gènes présentant du polymorphisme intraspécifique (SNPs) avaient été identifiés. Cependant, depuis 2004, la séquence complète du génome du P. ramorum est disponible avec plus de 16000 régions transcrites rendues publiques. Dans ce chapitre, nous avons tenté de répondre à plusieurs questions comme : Quels gènes portent des variations intra-spécifiques? Est-ce que ces polymorphismes occasionnent des changements dans la séquence de la protéine? Quelle est l’ampleur de ces changements et dans quelles populations? Pour déterminer le choix des gènes sélectionnés pour le génotypage (plus de 16000), nous avons utilisé le critère de volatilité du génome (Cf. Boite 2.0) (Plotkin et al., 2004). Cette volatilité a permis de sélectionner les gènes à étudier parmi les 16000 disponibles, ayant comme hypothèse que les gènes les plus volatiles présenteraient plus de polymorphismes que les moins volatiles. De plus, plusieurs nouveaux gènes présentant du polymorphisme intraspécifique ont alors pu être détectés et utilisés pour l’étude des populations du P. ramorum en Amérique du Nord et en Europe.
Boite 2.0 Qu’est-ce que la volatilité?
La volatilité est un concept utilisé pour comparer la sélection des gènes par rapport à son génome entier. Cette méthode suggère une façon de détecter rapidement une différence de pression sélective sur les gènes par inspection des séquences du génome, pour une empreinte des substitutions non synonymes. Si une séquence nucléotidique pour une région codant une protéine subit un nombre excessif de substitutions d’acides aminés, la région va en moyenne contenir une surabondance de codons volatiles comparés avec le génome entier. Pour chacun des 61 codons, on définit la volatilité par une proportion de ses points de mutations voisins qui encodent différents acides aminés. La volatilité de ce codon sera donc utilisée pour quantifier la probabilité que la plus récente mutation sur ce codon cause une substitution d’acide aminé (Fig 8). Ainsi, la probabilité pour chaque codon est calculée pour chaque séquence du génome. Cette probabilité P-value permet donc de classer les différentes séquences selon leur probabilité de volatilité, soit les plus et les moins volatiles.
Figure 8 : Deux exemples de calculs de la volatilité des codons. La volatilité de chaque codon dépend de la structure du code génétique. Le codon CGA, codant pour l’arginine, possède huit codons ancestraux potentiels, ainsi qu’un codon-stop, qui diffère de CGA par un seul point de mutation. Quatre de ces codons ancêtres de CGA encodent pour des acides aminés autres que l’arginine (R). Ainsi, la volatilité de CGA est de 4/8. Le codon AGA aussi encode pour l’arginine mais avec une volatilité de 6/8. Il y a 22 codons qui ont au moins un synonyme avec une volatilité différente. La volatilité est utilisée pour quantifier la probabilité que la plus récente mutation acceptée à un site cause un changement d’acide aminé.
Figure 1. Distribution du Phytophthora ramorum en Californie et en Oregon le 15 février 2008. Les différents comtés infectés sont représentés. Le comté de Marin est identifié par une flèche hachurée. Les données provenant de pépinières ne sont pas illustrées ici. Les données proviennent des pathologistes du CDFA et de l’UC Davis/UC Berkeley (adapté d’une carte produite par le UCB GIIF : http://kellylab.berkeley.edu/SODmonitoring/).
Figure 2 (a et b). Aperçu des dommages causés par l’encre des chênes rouges dans les collines du « China Camp State Park », en Californie. Les arbres en gris sont morts ou atteints par la maladie. Photos provenant de Shane Sela, ACIA.
a
a
Figure 3. Symptômes de la maladie de l’encre des chênes rouges : a) chancre rouge suintant sur le lithocarpe de Californie (tanoak); b) l’écorce est prélevée sur ce chancre et on observe la marge de progression de l’agent pathogène; c) brunissement des feuilles d’un lithocarpe de Californie malade et d) exemple de tâches foliaires que l’on peut observer sur les autres hôtes que le chêne, ici un rhododendron. Photos a et c, provenant de Guillaume Bilodeau et Danny Rioux, SCF et photos b et d, de Shane Sela, ACIA.
c
Figure 4. Localisation du P. ramorum en Europe. Les zones (pays) où le P. ramorum a été retrouvé en pépinière sont en gris. Les pays où le P. ramorum a été détecté en nature ont un cercle noir.
Figure 5. Localisation du P. ramorum en Amérique du Nord. Les zones (pays) où le P.
ramorum a été retrouvé en pépinière sont en gris. Les États où le P. ramorum a été détecté
en nature ont un cercle noir. CB
a
b
c
Figure 6. Structures sexuées et asexuées chez Phytophthora : a) oospore (fusion de l’anthéridie et de l’oogone); b) chlamydospores sur mycélium et c) schéma d’une zoospore. Figure adaptée de a) http://www.apsnet.org/education/illustratedglossary/ , b)
http://www.livingharbour.net/partners/popups/dieback04.htm, et c)
Figure 7. Arbre phylogénétique du monde du vivant basé sur des séquences d’ADN ribosomiaux. Les Straménopiles sont encerclés en rouge. On peut voir que les Straménopiles, auxquels appartiennent les Oomycètes (encadré jaune), dont les