Estimation de la contribution des différents stocks
reproducteurs identifiés aux nourriceries chez le Flétan
du Groenland (Reinhardtius hippoglossoides)
Mémoire
Émilie Carrier
Maîtrise en biologie - avec mémoire
Maître ès sciences (M. Sc.)
Estimation de la contribution des différents
stocks reproducteurs identifiés aux
nourriceries chez le Flétan du Groenland
(Reinhardtius hippoglossoides)
Mémoire
Emilie Carrier
Sous la direction de :
Louis Bernatchez, directeur de recherche
Pascal Sirois, codirecteur de recherche
Résumé
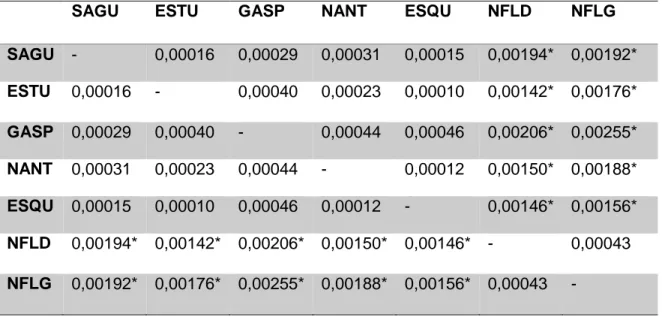
L'identification des stocks et la quantification de leur contribution relative au recrutement sont des objectifs majeurs pour améliorer la gestion et la conservation des espèces marines exploitées. Le séquençage de nouvelle génération permet d’analyser des milliers de marqueurs génomiques et d’apporter la résolution nécessaire pour répondre à ces questions chez les espèces marines qui sont faiblement différenciées. Le Flétan du Groenland (Reinhardtius hippoglossoides) est un poisson plat largement exploité, particulièrement dans le golfe du St-Laurent, au Canada. On y retrouve 2 nourriceries connues, dont la contribution des juvéniles au renouvellement des différents stocks adultes reste inconnue à ce jour. Les buts de ce projet sont de i) déterminer la structure des populations de flétan du Groenland du Saint-Laurent et ii) d’estimer la contribution des différents stocks reproducteurs identifiés aux différentes nourriceries. Pour ce faire, nous avons échantillonné 100 juvéniles par nourricerie et 50 adultes de sites s’étendant du fjord du Saguenay jusqu’au large de Terre-Neuve, avec quelques sites échantillonnés sur 2 années consécutives pour évaluer la stabilité temporelle de cette contribution. Nos résultats montrent qu’après avoir retiré les marqueurs liés au sexe, la région de l’Estuaire/Golfe du Saint-Laurent forme une population distincte de l’Atlantique près de Terre-Neuve (Fst = 0.00146, p-value = 0.001). Les analyses d’assignation populationnelle
montrent que le recrutement dépend largement du stock du Saint-Laurent. Par ailleurs, on retrouve une contribution variable du stock de Terre-Neuve, variant de 1% pour la première année à 33% pour la deuxième, ce qui suggère un transport interannuel de larves variable selon la force des courants profonds. Cette étude sert de modèle pour l’identification des stocks pour les ressources halieutiques, dans un contexte où le milieu marin offre peu de barrières à la dispersion, en plus de démontrer l’importance des marqueurs liés au sexe et des répliques temporelles en génomique des populations.
Abstract
The identification of stocks and quantifying their relative contribution to recruitment are major objectives toward improving the management and conservation of marine exploited species. Next-generation sequencing allows to analyze thousands of genomic markers which provide the resolution needed to address these questions in marine species with weakly differentiated populations. Greenland Halibut (Reinhardtius hippoglossoides) is one the most important exploited demersal species throughout the North Atlantic, and in particular in the Gulf of St. Lawrence, Canada. There, two nurseries are known, the St. Lawrence Estuary and the northern Anticosti Island, but their contribution to the renewal of stocks remains unknown. The goals of this study were i) to document the genetic structure and ii) to estimate the contribution of the different identified breeding stocks to nurseries. We sampled 100 juveniles per nursery and 50 adults from seven sites ranging from Saguenay fjord to offshore Newfoundland, with some sites sampled over two consecutive years in order to evaluate the temporal stability of the contribution. Our results show that after removing of sex-linked markers, the Estuary/Gulf of St. Lawrence represent a stock which is genetically distinct from the Atlantic around Newfoundland (Fst = 0.00146, p-value
= 0.001). Population assignment showed that recruitment in both nurseries is largely predominantly associated with the St. Lawrence stock. However, we found that the relative contribution of both stocks to the nurseries is temporally variable with 1% contribution of the Newfoundland stock one year but up to 33% for the second year, which may hypothetically be caused by year-to-year variation in larval transport into the Gulf of St. Lawrence. This study serves as a model for the identification of stocks for fisheries resources in a context where few barriers to dispersal occurs, in addition to demonstrating the importance of considering sex-linked markers and temporal replicates in studies of population genomics.
Table des matières
Résumé ... ii
Abstract ... iii
Table des matières ... iv
Liste des figures ... vi
Liste des tableaux ... vii
Remerciements ... viii
Avant-propos ... ix
Introduction ... 1
Problématique générale ... 1
La génomique des populations au service de la gestion des pêches ... 3
Structure des populations ... 3
Population et forces évolutives ... 4
Marqueurs moléculaires et méthode de séquençage ... 5
Méthodes de séquençage ... 8
Le Flétan du Groenland ... 9
Biologie de l’espèce ... 9
Pêche commerciale au Canada ... 10
Problématique ... 11
Contexte du projet ... 12
Objectifs ... 13
Chapter 1: ... 14
Résumé ... 15
Abstract ... 16
Introduction ... 17
Material and methods ... 20
Sampling ... 20
DNA extraction ... 22
Bioinformatics and sequencing ... 22
Identifying sex-linked and selected markers ... 24
Population structure ... 25
Assignment of juveniles ... 26
Results ... 26
Selection of SNPs ... 27
F-statistics ... 27
Effect of sex-linked markers on population structure ... 28
Assignment of juveniles to identified stocks ... 30
Discussion ... 33
Population structure: effect of sex-linked markers ... 33
Assignment of juveniles to identified stocks ... 34
Comparison of SNPs and microhaplotypes ... 35
Conclusion and perspective ... 36
Conclusion ... 38
Bibliographie ... 47
Liste des figures
Figure 1. Map of sampling sites in Saguenay fjord (SAGU), St. Lawrence Estuary (ESTU,
ESTJ), Gaspé (GASP), North Anticosti Island (NANT, ANTJ), Esquiman (ESQU) and Atlantic Ocean (NFLD, NFLG). The Estuary and northern Anticosti Island nurseries (ESTJ and ANTJ) are defined by the triangles and adult populations of the St. Lawrence and Atlantic (SAGU, NFLD, ESTU, NANT, GASP and ESQU) by circles. Sites that are sampled for a single year are colored in pink for the first year of sampling and in blue for the second. Those sampled for the two consecutive years are colored in green……...……….……….………20
Fig 2. Population structure for Greenland Halibut from the Estuary/Gulf of St. Lawrence
(Saguenay fjord SAGU, St. Lawrence Estuary ESTU, St. Lawrence Estuary nursery ESTJ, Gaspé GASP, North Anticosti Island NANT, North Anticosti Island nursery ANTJ and Esquiman ESQU) and Newfoundland (NFLD and NFLG) using ADMIXTURE: a) for dataset including sex-linked markers and b) for dataset without sex-linked markers. Results are shown for K = 2. Sites are classified from west to east and individuals in alphanumerical order. Sites sampled for two consecutive years are combined……….……….28
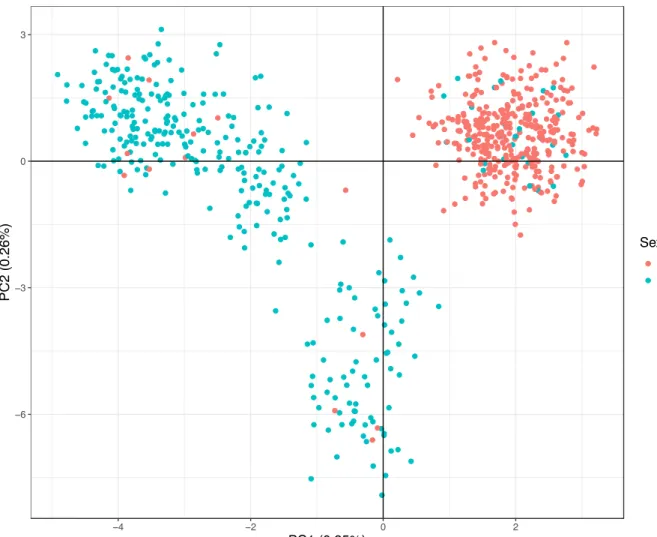
Fig 3. Principal component analysis (PCA) with dataset including sex-linked markers.
Females are colored in pink and males in blue.………29
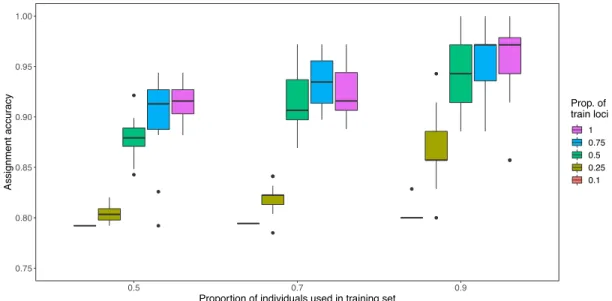
Fig 4. Monte Carlo cross-validation test for SNPs dataset using different proportions of
training loci (0.1, 0.25, 0.5, 0.75 and 1) and individuals in training sets. Boxplots were generated for overall sources populations (left) and for both populations separately (GSL in center and NFL to the right). Selected SNPs were ranked in decreasing values of differentiation index when using a different proportion then 1……….30
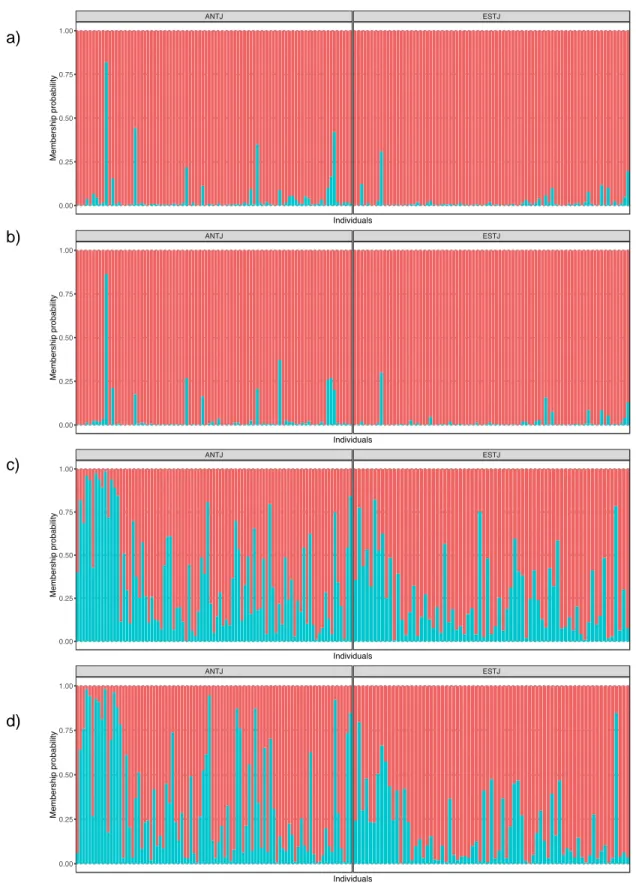
Fig 5. Individual assignment test using SNPs: a) for the first year and b) for the second
year, and for haplotypes: c) the first year of sampling and d) for the second year of sampling to reference stocks of Gulf of St. Lawrence (GSL) and Newfoundland (NFL). All
loci and individuals were kept for the
Liste des tableaux
Table 1. Number and percentage of remaining loci after every filtering step of the for: a)
SNPs and b) microhaplotype datasets………23
Table 2. Pairwise differentiation index (Fst) of Greenland Halibut from 8 sampling sites
distributed from the St. Lawrence Estuary to offshore Newfoundland for 2 consecutive years of sampling. Significant Fst (p-value < 0.05) are marked with an
Remerciements
J’aimerais tout d’abord remercier mon directeur de recherche, Louis Bernatchez, pour m’avoir fait confiance dans la réalisation de ce projet et de m’avoir permis de développer des compétences qui me seront utiles dans mon parcours professionnel, notamment en communication scientifique. Je garde avec moi une expérience enrichissante du laboratoire Bernatchez en raison de ses membres et de l’ambiance conviviale qui y règne. Pour l’aide technique en laboratoire pour l’extraction et le séquençage de l’ADN, je remercie particulièrement Alysse Perrault-Payette, Damien Boivin-Delisle et Louis Deslauriers. Pour son support tout au long de ma maîtrise, tant pour résoudre des problèmes techniques que l’interprétation des résultats, je remercie Anne-Laure Ferchaud. Pour leur aide au niveau des analyses bio-informatiques, je remercie Éric Normandeau et Yann Dorant. Je voudrais également remercier mon co-directeur, Pascal Sirois, pour m’avoir apporté un retour à différentes étapes du projet.
Je remercie les professionnels et les pêcheurs volontaires sans qui le projet n’aurait pas pu voir le jour, particulièrement Rénée Gagné et Céline Audet de l’Université du Québec à Rimouski qui ont coordonné les opérations.
Je tiens à remercier Ressources Aquatiques Québec, qui est un regroupement de chercheurs et d’étudiants interuniversitaire pour le développement durable de l’industrie aquacole et des pêches commerciales et récréatives dans la région de Québec, pour m’avoir donné l’opportunité de présenter lors des réunions annuelles, en plus de m’accorder un soutien financier lors de voyages pour des communications scientifiques à l’international.
Pour terminer, je voudrais remercier spécialement ma famille et mes amis, qui m’ont toujours encouragée à poursuivre dans la voie professionnelle qui allait me rendre la plus heureuse et accomplie, malgré toutes les épreuves pouvant survenir en cours de route.
Avant-propos
Le projet qui a mené à la rédaction de ce mémoire a été financé par le Conseil de recherches en sciences naturelles et en génie (CRSNG), le Fonds de recherche du Québec – Nature et technologies (FRQNT) et par le regroupement interuniversitaire Ressources Aquatiques Québec (RAQ).
Ce mémoire est essentiellement constitué d’un article « Estimating contribution of reproductive stocks of Geenland Halibut (Reinhardtius hippoglossoides) to nurseries by means of RADseq », qui a été soumis à la revue Evolutionnary Applications le 3 octobre 2019 et qui est en processus de révision par les pairs. Prochainement, ces connaissances seront transférées dans la production d’un second article mettant en commun les récentes avancés sur la migration des juvéniles, la connectivité transatlantique, ainsi que les effets de la température et du sexe sur la croissance des juvéniles du Flétan du Groenland dans tout l’est du Canada.
Je suis l’auteure principale de cet article. Il a été écrit en collaboration avec madame Anne-Laure Ferchaud, monsieur Éric Normandeau, mon co-directeur Pascal Sirois et mon directeur Louis Bernatchez. Pour le second article, je serai une des coauteurs.
Anne-Laure Ferchaud a participé aux analyses statistiques et à l’interprétation de celles-ci et Éric Normandeau a effectué la préparation et la transformation des données bio-informatique. L’échantillonnage a été réalisé par des équipes du ministère Pêches et Océans Canada et des pêcheurs volontaires de la région, sous la coordination de Céline Audet. J’ai réalisé les manipulations en laboratoire, analysé les données et interprété les résultats avec l’assistance des différents coauteurs, ainsi que rédigé l’article en tant qu’auteure principale. Tous les coauteurs ont participé à la révision du manuscrit.
Introduction
Problématique générale
La pêche commerciale occupe une importance autant sur le plan économique, culturel et politique pour divers peuples à l’échelle mondiale. En effet, de nombreuses communautés côtières dépendent de cette industrie comme source principale de protéine et de revenus (FAO, 2018). Les ressources halieutiques subissent plus que jamais des pressions anthropiques, en raison de la surpêche, des captures accidentelles et des changements climatiques.
Selon la définition du dictionnaire Larousse, une espèce est en surpêche lorsque le taux d’exploitation est au-delà de son taux de renouvellement pour une période de temps donné. On observe à ce jour plusieurs cas de pêcheries commerciales dont les populations ont diminué drastiquement en quelques années, allant jusqu’au seuil de l’extinction. Notamment, au Canada, on peut mentionner le cas de la morue de l’Atlantique (Gadus morhua), espèce anadrome répartie dans tout l’est du Canada. La Morue est composée de quatre stocks génétiquement distincts : les populations de l’Arctique, de Terre-Neuve-et-Labrador, du Nord Laurentien et des Maritimes. Les scientifiques ont observé une augmentation fulgurante du nombre d’effectifs dans tous les stocks (à l’exception de celui de Terre-Neuve-et-Labrador) au début des années 80, ce qui a influencé l’émission d’un nombre de permis et des quotas de pêche plus élevés dans les zones exploitables (MPO, 2003). Par ailleurs, après 5 ans de pêche abondante, les stocks ont chuté drastiquement : on note, par exemple, une diminution de 97% de l’abondance de poissons (nombre d’individus matures) entre 1968 et 2001 pour le stock de Terre-Neuve-et-Labrador. Ces observations imposent l’implantation d’un moratoire sur la pêche de la Morue de l’Atlantique par les gestionnaires et l’interdiction d’utilisation d’engins mobiles pour les poissons de fonds au début des années 90 (MPO, 2003). Cette diminution importante du nombre d’effectifs a non seulement eu un impact au niveau de la taille des différents stocks, mais également au niveau de leur composition phénotypique. On observe un taux de croissance et un âge à la maturité moindres, ainsi qu’un taux de mortalité supérieur après l’effondrement des populations (Smedbol et al. 2002).
Tout comme la surpêche, les prises accidentelles causent des diminutions d’effectifs dans les stocks. Celles-ci correspondent à l’ensemble des espèces non désirées (que ce soient de mammifères marins, des oiseaux, des tortues marines, des poissons ou encore des invertébrés), qu’elles soient d’intérêt commercial ou non, capturées lors des activités de pêche (Hall, 1996). Bien que la plupart des méthodes de pêche aient le potentiel de capturer des espèces non désirées (Northridge 1991), la pêche à la palangre et aux filets maillants sont les deux techniques les plus dommageables selon une méta-analyse des pêches commerciales situées sur la côte est américaine (Zollett 2009). L’ampleur des effets néfastes à l’échelle écosystémique dépend de la quantité d’individus récoltés, du statut de vulnérabilité (particulièrement pour les espèces longévives ayant un temps de génération élevé) et du rôle dans la chaîne alimentaire des espèces accidentellement visées (Lewison et al., 2011). À une échelle plus locale, les conséquences des prises accidentelles pour les pêcheurs comprennent le bris d’équipement, la perte monétaire, en plus de potentiellement nuire à leur sécurité (dans les cas où l’animal piégé peut être dangereux). Pour les individus capturés, la pêche peut résulter en des blessures irréversibles, voire mortelles.
Selon les plus récentes statistiques citées par le Groupe d’experts intergouvernemental sur le climat (GIEC), si le développement se maintient à un niveau similaire, on prévoit qu’à chaque cycle de 30 ans, la température de l’air augmentera entre 0,3 et 0,7 degré Celsius (Kirtman et al. 2013). Les conséquences des changements climatiques sur les milieux marins peuvent se classer en deux catégories : les changements physiques directs, ainsi que les réponses biologiques et écologiques aux changements physiques directs (FAO, 2016). On compte parmi les conséquences physiques directes l’augmentation du dioxyde de carbone atmosphérique (qui contribue à l’acidification des océans), l’augmentation de la température de l’eau (qui influence la modification de l’aire de répartition d’espèces aquatiques, en plus de causer une hausse de la mortalité) et l’augmentation de la fréquence d’évènements climatiques extrêmes (qui peuvent contribuer à la destruction d’habitats côtiers et rendent plus risquées les pêches en haute mer) (FAO, 2016). Pour ce qui est des impacts indirects des changements climatiques, il est plus difficile d’en prédire les conséquences puisqu’il est plus difficile de les tester en milieu contrôlé (FAO, 2016).
Tous ces facteurs risquent de modifier l’équilibre entre le taux de mortalité et le taux de renouvellement des espèces exploitées, ce qui causerait un déclin des effectifs (FAO,
2012). Pour contrer cette problématique, il est primordial d’établir un plan de gestion adapté aux caractéristiques biologiques de l’espèce en question, des mutations de son milieu, ainsi que de son taux d’exploitation.
La génomique des populations au service de la gestion des
pêches
Structure des populations
La définition d’une population et la détermination de sa structure génétique revêtent d’une importance particulière dans le cadre de mise en œuvre d’un plan de gestion. Cela permet de comprendre les causes écologiques de la structuration lorsqu’on met en relation ces informations avec des renseignements écologiques, d’en optimiser l’exploitation (dans le cas des ressources naturelles) et de prédire les effets d’une perturbation sur l’espèce en question. Une population correspond à un ensemble d’individus vivant à une proximité suffisante pour que tous aient une probabilité de rencontre égale à des fins de reproduction (Waples et Gaggiotti, 2006). Lorsque ce concept est appliqué au domaine de la gestion des pêches, on emploie plutôt le terme de stocks. Tout comme une population, un stock répond à des contraintes spatiales et reproductrices; les individus d’un même stock doivent avoir des traits d’histoire de vie similaires et doivent présenter un certain isolement reproducteur (Hilborn et Walters, 2013). Par contre, la distinction majeure entre ces concepts réside dans leur application. On définit un stock dans le but de mettre en place une politique de gestion pour préserver l’espèce exploitée à une échelle plus appropriée que celle de l’espèce (Begg et al. 1999). Différentes approches permettent d’identifier les stocks ou les populations d’une espèce. On peut les diviser en différentes catégories, soit la capture-recapture, les données de débarquements, les traits d’histoire de vie, la composition et de l’abondance des parasites, la chimie des otolithes, les traits morphologiques et la génétique (Begg et al. 1999). Néanmoins, selon Elliott et collaborateurs (1995) la combinaison de différentes approches permet d’augmenter la fiabilité de l’inférence des unités de gestion.
Population et forces évolutives
Il est possible d’étudier la structure des populations, c’est-à-dire la façon dont la variation génétique est distribuée au sein d’un ensemble d’individus, en fonction des forces évolutives qui agissent sur celle-ci, soit la dérive génétique, la mutation, la migration et la sélection. Les forces évolutives, qu’elles soient neutres (dérive génétique, mutation et migration) ou non (sélection), ont une action différente sur la diversité génétique. Parmi toutes ces forces, la migration (ou la dispersion) a des conséquences importantes sur la dynamique démographique des populations observées. On peut définir la dispersion comme étant le mouvement d’individu(s) d’une population vers une autre de façon permanente ou suffisamment longue pour contribuer à enrichir le patrimoine génétique de la nouvelle population (Lowe et Allendorf 2010). Cette dispersion peut soit être passive (provoquée par divers facteurs environnementaux) (Bohonak et Jenkins 2003) ou active (mouvement volontaire d’individus) (Van Dyck et Baguette 2005). La dispersion est bénéfique lorsque les coûts liés au déplacement et à l’acclimatation au nouvel habitat sont inférieurs aux bénéfices projetés en termes d’augmentation de succès reproducteur (Cote
et al. 2010). Les populations d’une espèce sont panmictiques lorsque l’espèce est
composée d’une seule population génétique (ou métapopulation), c’est-à-dire qu’il y a assez de migration entre les « sous-populations » pour observer la même structure génétique chez chacune d’elles (Waples et Gaggiotti, 2006). Au contraire, lorsqu’il n’y a pas de flux génétique entre les populations d’une même espèce (autrement dit, il n’y a pas de dispersion), les populations sont isolées les unes des autres. Il existe une gamme de variation de dépendance démographique entre ces 2 états en fonction du taux de dispersion entre les sous-populations.
Par ailleurs, la mesure directe du taux de dispersion comporte des difficultés techniques et logistiques. Il est particulièrement difficile de suivre les espèces de petite taille, les espèces vivant dans des habitats ouverts et celles qui sont très mobiles (Dingle et Drake 2007). De plus, comme la durée de vie des batteries est limitée, il n’est parfois pas possible de monitorer le mouvement d’individus jusqu’au moment de la dispersion effective. C’est particulièrement le cas pour des espèces ayant une longévité importante et où les mouvements vers une population différente se font occasionnellement (Moore et al. 2017). Les méthodes indirectes impliquent, quant à elles, l’utilisation de marqueurs
moléculaires (Paetkau et al. 1995, Rannala et Mountain 1997, Hellberg et al. 2002, Waples et Gaggiotti 2006). Les outils moléculaires offrent une possibilité de mieux répondre à des questions écologiques, évolutives et de conservation, dans un contexte d’évolution dynamique des populations naturelles (Manel et al. 2005). Ils permettent notamment d’identifier les migrants, sans avoir besoin de suivre les déplacements d’individus marqués. Autrement dit, il est possible de déterminer la proportion les migrants présents dans les populations par leurs différences génétiques qui les distinguent de la nouvelle population dans laquelle ils se trouvent (Paetkau et al. 1995, Waser et Strobeck 1998). L’ensemble des méthodes qui utilisent les informations génétiques pour déterminer l’appartenance d’un individu à une population se nomme l’assignation populationnelle. Celle-ci permet de répondre à plusieurs questions biologiques, telles que l’origine d’un individu, la structure d’une population, le taux de dispersion et le taux d’hybridation (Waser et Strobeck 1998, Manel et al. 2005). La puissance d’assignation est dépendante du niveau de différenciation des sous-populations étudiées, du nombre de marqueurs utilisés et de la taille d’échantillonnage (Cornuet et al. 1999). Plus les populations sont similaires, plus le nombre de marqueurs est faible ou plus l’échantillon est petit, moins grande sera la puissance du test statistique. Ainsi, il devient difficile de faire la distinction entre un faible niveau de différenciation causé soit par une population de grande taille effective (dérive génétique) et une grande dispersion, ou soit par une divergence récente (Allendorf et al. 2010). Ces outils d’assignation sont particulièrement utiles dans le domaine de la gestion des ressources halieutiques. Comme il est difficile de suivre la dispersion des larves et des œufs, l’assignation des juvéniles à un stock adulte est problématique (Begg et al. 1999). De plus, chez la plupart des espèces marines, des contraintes font en sorte que les distributions des différentes unités de conservation sont continues. Elles incluent un nombre réduit de barrières à la dispersion en milieu marin, la phase larvaire pélagique qui provoque un flux génique important et des tailles de population élevées (Gagnaire et al. 2015, Riginos et al. 2016). Il devient alors difficile de faire une gestion individuelle de ces entités (Waples et al. 2008).
Marqueurs moléculaires et méthode de séquençage
Le choix du type de marqueur utilisé pour les études de génétique des populations dépend principalement de trois critères principaux : le niveau de polymorphisme, la facilité de
générer les données, ainsi que le coût par individu. Les Polymorphismes de Nucléotide Simple ou Single Nucleotide Polymorphism (SNP) sont des marqueurs bi-alléliques répartis sur l’ensemble du génome (Collins et al. 1997) et correspondent à ce jour au marqueur de choix en génomique des populations. Avec le développement du séquençage de nouvelle génération, ces marqueurs sont de plus en plus utilisés dans divers domaines de recherche en raison de leur polyvalence en génétique des populations (Andrews et al. 2016, Ekblom et Galindo 2011, McCormack et al. 2013, Narum et al. 2013), ainsi que leur faible coût de développement (De Wit et al. 2015). On retrouve de nombreux exemples dans la littérature de détection de structure de population chez des espèces marines à l’aide de ce type de marqueur. Les espèces marines concernées incluent le Homard d’Amérique (Homarus americanus) (Benestan et al. 2015), le Holothurie rouge de Californie (Parastichopus californicus) (Xuereb et al. 2018) et la Morue de l’Atlantique (Gadus morhua) (Barth et al. 2017). Par exemple, l’étude de Benestan et collaborateurs (2015) a démontré la présence d’une différenciation significative entre le nord et le sud de l’aire de répartition du Homard d’Amérique (Homarus americanus). Bien que la puissance d’analyse soit suffisante pour de nombreuses espèces aquatiques, les SNPs peuvent s’avérer impuissants pour résoudre la structure de population d’espèces marines. En effet, les contraintes du cycle de vie, qui sont répandues chez de nombreuses espèces, limitent la différenciation. On observe un taux de dispersion élevé, en raison principalement de barrières physiques modérées, ainsi que de grandes tailles de populations (Gagnaire et al., 2015). Par ailleurs, il est possible de combiner plusieurs SNPs en déséquilibre de liaison, ce qui permet d’augmenter le nombre de génotypes. Cette approche multi-SNPs se nomme micro-haplotypes (Kidd et
al. 2013, 2014). En effet, lorsqu’on analyse différents SNPs séparément, la probabilité
d’obtenir au hasard la même valeur de SNPs entre 2 individus (et ainsi inférer une même unité populationnelle) est plus élevée que la combinaison des plusieurs SNPs. L’utilisation des micro-haplotypes a comme avantage de ne nécessiter aucune méthode de séquençage particulière, de demander peu de traitement bio-informatique pour faire la transformation, en plus d’être compatible avec les logiciels d’assignation populationnelle qui peuvent traiter les microsatellites. Plus particulièrement dans le domaine des pêches commerciales, on retrouve deux exemples récents qui illustrent l’utilité de ces micro-haplotypes pour répondre à une problématique de conservation. Dans leur étude publiée en 2017, McKinney et collaborateurs ont comparé la puissance des marqueurs SNPs et des micro-haplotypes pour déterminer la contribution relative des différents stocks chez le
Saumon quinnat (Oncorhynchus tshawytscha) aux différentes populations de l’ouest de l’Alaska. Pour les groupes les plus étroitement liés, les auteurs ont démontré que les micro-haplotypes composés de plusieurs SNPs augmentent la précision des analyses de stock mixtes de 7% à partir d’un total de 500 marqueurs. Celle-ci monte jusqu’à 17% pour l’assignation d’individus inconnus à leur population d’origine. Il est également possible d’utiliser les micro-haplotypes afin de performer une analyse l’assignation parentale de larves d’origine inconnue à un couple de géniteurs potentiels. Ce type d’analyse peut être employé notamment pour estimer le taux de dispersion des juvéniles des espèces qui sont sédentaires à maturité (Christie 2010). De plus, cette méthode permet de détecter les mouvements récents, plutôt que le flux génique historique, que fournit l’indice de différenciation. C’est ce qu’a réalisé l’équipe de Baetscher et collaborateurs (2019) avec le Sébaste du varech (Sebastes atrovirens), une espèce résidante des milieux côtiers de Californie et dont une partie de l’aire de répartition est intégrée dans plusieurs aires marines protégées. Sur les 6000 échantillons récoltés, ils ont pu identifier significativement 8 familles (couple et rejeton), en addition à 25 fratries à l’aide de 96 micro-haplotypes hautement différenciés. Avec ces groupements, ils ont découvert qu’une partie de celles qui naissent à l’intérieur d’une certaine zone protégée peut migrer en fonction des courants marins pour s’établir soit dans une autre zone protégée à proximité, ou encore dans des régions où la pêche est permise.
En addition au choix du type de marqueur, il est important de sélectionner les régions génomiques à analyser. Particulièrement pour l’étude de populations d’espèces marines, une étude de Benestan et collaborateur (2015) a démontré qu’une attention particulière doit être mise sur les marqueurs associés au sexe. Ces marqueurs sont présents sur les chromosomes sexuels (pour les espèces qui en possèdent) ou dans une région associée à l’expression de gènes impliqués dans la différenciation sexuelle pour des traits morphologiques, physiologiques et/ou comportementaux (Bell 1982), chez les espèces ayant un déterminisme génétique sur la différenciation entre les sexes (Gamble et Zarkower 2014). Ils peuvent à eux seuls amplifier, voire créer un signal de structuration recherché ou le masquer si le ratio entre le nombre de mâles et de femelles n’est pas balancé, surtout pour des espèces possédant un faible niveau de variation génétique. Par exemple, chez le Homard d’Amérique (Homarus americanus), seulement 12 SNPs (sur un total de 1717) peuvent créer une structuration significative (Fst moyen de 0,0800 pour les
et le milieu marin, en manipulant le nombre d’individus de chaque sexe dans ces deux groupes (Benestan et al. 2015). À ce jour, peu d’études se sont intéressées à l’influence des marqueurs associés au sexe pour répondre à des problématiques de biologie de la conservation (Jackson et al., 2018, Messmer et al. 2018, Ogata et al., 2018).
Méthodes de séquençage
L’amélioration des méthodes de séquençage de nouvelle génération depuis le milieu des années 2000 est un tournant dans la génétique des populations, en permettant de définir des différenciations subtiles à des échelles locales fines (Metzker 2010). Bien qu’il soit maintenant possible de séquencer l’entièreté du génome de toutes les espèces, il devient fastidieux et dispendieux d’utiliser cette pratique pour génotyper des centaines ou des milliers d’individus pour déterminer la structure de populations. Parmi toutes les approches de séquençage, le « Génotype par Séquençage » (GBS) permet d’analyser des milliers de SNPs chez plusieurs organismes à la fois (Davey et al. 2011). Le GBS a déjà été utilisé pour de nombreux projets de génétique des populations, et, selon certains auteurs, cette méthode a révolutionné les études dans le domaine par sa facilité d’utilisation et sa polyvalence (Narum et al. 2013). Le protocole du GBS peut se diviser en trois grandes étapes décrites dans le papier d’Elshire et al. (2011). La première étape est d’extraire l’ADN et de distribuer les échantillons dans les puits d’une plaque. La deuxième étape consiste à préparer des librairies de séquençage. Des enzymes de restriction sont ajoutées afin de fragmenter le génome. Les endonucléases choisies sont non-spécifiques pour créer des milliers de morceaux d’ADN génomique de toutes les longueurs. Des séquences code-barres de quelques nucléotides qui sont spécifiques aux individus sont ajoutées afin de les distinguer subséquemment. Les échantillons sont groupés et amplifiés par PCR. La troisième étape au protocole est de séquencer les échantillons et identifier le génotype. Il existe quelques variantes au protocole selon le nombre d’enzymes de restriction utilisées et le moment auquel ils sont incorporés aux échantillons, la méthode de sélection de fragments selon leur taille et le moment dans le protocole où sont incorporés les adaptateurs utilisés pour se fixer à la matrice de séquençage (Li et Wang, 2017). Le principal avantage de cette méthode est de réduire la charge de matériel génétique à séquencer, puisque l’étape de la PCR amplifie les fragments ayant une longueur optimale. Cela permet de limiter le temps et les coûts des projets, d’autant plus
qu’il n’est souvent pas nécessaire de séquencer l’ensemble du génome. Le GBS offre également la possibilité de séquencer des espèces qui ne sont pas des modèles biologiques, ce qui s’avère souvent le cas dans les études de génétique de populations (Narum et al. 2013).
Le Flétan du Groenland
Biologie de l’espèce
Le flétan du Groenland (Reinhardtius hippoglossoides) est un poisson plat de la famille des Pleuronectidae. Son aire de répartition est circumpolaire et l’espèce se retrouve à la fois dans l’océan Atlantique et Pacifique. Dans l’océan Atlantique, l’espèce s’étend depuis la mer de Barents à l’est, jusqu’à la terre de Baffin à l’ouest et à au sud jusqu’en Nouvelle-Écosse (Bowering et Brodie 1995). On retrouve le Flétan du Groenland à des profondeurs moyennes de 200 m jusqu’à 375 m dans le golfe du Saint-Laurent (MPO 2019) et ses températures préférentielles varient de 1,3 à 2,7 °C (Peklova et al. 2012). Dans le golfe du Saint-Laurent, la fraie a lieu l’hiver durant les mois de janvier jusqu’à mars. Pour les poissons plats bentho-pélagiques tels que le flétan du Groenland, on observe un niveau élevé de dispersion en raison de la phase larvaire pélagique prolongée, ce qui peut conduire à une panmixie de la population (Bailey 1997, Hoarau et al. 2002). Les juvéniles se retrouvent en majorité dans l’estuaire du Saint-Laurent et au nord de l’île d’Anticosti regroupés dans deux nourriceries (MAPAQ 2014). Une nourricerie correspond à un rassemblement de juvéniles (poissons ayant franchi le stade larvaire, mais dont les organes génitaux ne sont pas encore développés), dans un lieu où la densité est supérieure à la moyenne de l’aire de répartition de l’espèce. Cet habitat leur permet de s’y nourrir et de se développer pour atteindre la forme adulte (Beck et al. 2001, Dahlgren et al. 2006). Hors du golfe du Saint-Laurent, seules deux autres nourriceries ont été identifiées à ce jour. La première est située plus au nord, dans le détroit de Davis et aurait pour origine les poissons adultes de la baie de Baffin et du nord-ouest du Groenland (Jørgensen et Arboe 2013). La deuxième serait située dans la mer de Svalbald, au large des côtes norvégiennes (Albert et Vollen 2014). Dans le golfe, cette espèce est sujette à des modifications considérables de son environnement qui affectent l’état des stocks. En raison des changements climatiques, les valeurs moyennes de température de l’eau sont
à la hausse. En effet, on a observé une valeur moyenne inférieure à 6°C dans les régions du centre du golfe et du chenal Esquiman en 2013 (Galbraith et al. 2017). Une des conséquences entraînée par la hausse des températures de l’eau est la diminution de la quantité d’oxygène dissous. Bien que cette espèce soit relativement résistante à l’anoxie (entre 18% et 25% en moyenne, Gilbert et al. 2005), il a été démontré que l’hypoxie affecte les juvéniles (Dupont-Prinet et al. 2013). Une équipe de Rimouski a démontré que les adultes ont un seuil critique d’oxygène significativement supérieur à celui des juvéniles à une température de 5°C, qui simule les valeurs actuelles approximatives (11% de saturation chez les individus matures contre 15% chez les juvéniles). De plus, ces mêmes auteurs ont étudié les effets de la rareté en oxygène sur le métabolisme. Ils ont observé chez les juvéniles une baisse du métabolisme, qui a pour conséquence le ralentissement de la digestion et une réduction approximative de 58% du métabolisme aérobie. Ces résultats suggèrent que l’hypoxie dans le golfe pourrait affecter négativement le recrutement et ainsi menacer à moyen ou à long la pêche commerciale (Dupont-Prinet et
al. 2013). Présentement, la gestion dans le Saint-Laurent est divisée en trois régions
distinctes : l’ouest du golfe (avec ou sans l’estuaire), le nord de l’île d’Anticosti et le chenal Esquiman, qui sont soumises à des dynamiques environnementales ou des pratiques de pêches différentes (MPO 2010).
Pêche commerciale au Canada
Depuis le déclin de la Morue de l’Atlantique dans les eaux canadiennes, en plus des baisses du nombre d’effectifs chez plusieurs espèces de poissons plats (NAFO, 1994), l’exploitation du flétan du Groenland a été le sujet de plusieurs questionnements, particulièrement en raison de son impact économique. Les industries de la pêche et de la transformation, particulièrement dans les régions du golfe du Saint-Laurent, de Terre-Neuve et de l’Arctique, bénéficient considérablement de l’abondance du Flétan du Groenland dans les eaux canadiennes. Les débarquements de flétan du Groenland représentaient 39,8% du tonnage de poissons de fonds pour la province de Québec et 23,8% pour la province de Terre-Neuve et du Labrador en 2018. En raison du prix élevé de sa chair et de son abondance, cette pêche représente plus de 50% de la valeur des pêches commerciales de poissons de fond pour ces deux régions (DFO, 2019).
Problématique
Malgré l’importance de cette espèce pour l’économie canadienne, on connaît peu sur son écologie de même que sur sa structure des populations. Par ailleurs, plusieurs auteurs affirment que la sous-population de flétan présente dans le Saint-Laurent serait isolée des stocks de l’Atlantique. Les évidences proviennent d’études portant sur le polymorphisme d’allozymes (Fairbairn, 1981) et sur l’abondance et la diversité des parasites (Khan et al. 1982, Arthur et Albert 1993). Fairbairn (1981) a estimé que le stock du Nord-Est de l’Atlantique, représenté par un site près des côtes ouest islandaises et un dans la mer de Barents, serait isolé des stocks du Nord-Ouest en raison de fréquences d’allèles uniques à un locus pour les poissons provenant de la mer de Béring. Également, une différence observée à deux allozymes entre les stocks du nord-ouest de l’Atlantique et le golfe du Saint-Laurent suggère qu’ils sont partiellement isolés. Une autre équipe a obtenu des résultats similaires en étudiant deux protozoaires vivant dans le sang des flétans (Khan, 1982). Selon les occurrences d’infections par les différentes espèces de parasites, ils ont conclu qu’il existe trois stocks indépendants dans l’ouest : celui du Nord-Ouest, celui du Sud-Ouest de Terre-Neuve dans la baie Fortune et celui du golfe du Saint-Laurent. Arthur et collaborateurs (1993) se sont intéressés aux flétans du Groenland de cours d’eau à proximité du golfe pour déterminer s’il y a présence d’une structure à une échelle plus locale. La présence significativement plus importante de certains parasites dans les flétans du Saint-Laurent contrairement à ceux du fjord du Saguenay et au stock de l’Atlantique les distingueraient de ces derniers. Leurs résultats suggèrent donc que tout le cycle vital des flétans du Saint-Laurent se produit à l’intérieur du golfe. Plus récemment Roy et collaborateurs (2014) ont démontré à l’aide de marqueurs microsatellites que la population du golfe serait plutôt panmictique et que cette population comprendrait aussi les flétans retrouvés à l’Est du Labrador et près de Terre-Neuve. Ils proposent comme explication que la différenciation locale observée provient de nouvelles recrues et de colonisateurs issus du même fond génétique, ce qui est en accord avec les résultats précédents qui montrent une différenciation à grande échelle. Bien que ces résultats apportent une résolution plus locale de la connectivité, ils ne permettent pas de démontrer avec certitude l’existence d’une structure des stocks de flétan du Groenland à l’intérieur du Saint-Laurent. Cela permet donc de se questionner quant à la présence d’une telle
structure et de le vérifier avec des marqueurs plus précis tels que les SNPs.
En plus de l’incertitude concernant les populations adultes, on ne retrouve pas d’information sur l’appartenance des juvéniles aux populations adultes. Les biologistes de Pêches et Océan Canada s’intéressent principalement au taux de renouvellement, qu’ils mesurent par le nombre de juvéniles, soit l’abondance des poissons de longueur totale entre 12 et 21 cm, correspondant aux poissons de 1 an (MPO 2010). Cette mesure permet de prédire l’abondance du stock de poissons exploitable et, par le fait même, les rendements possibles de la pêche commerciale 4 ans plus tard. Ils ont pu observer que depuis le début des années 90, l’abondance des juvéniles est très variable d’une année à l’autre. Par exemple, la cohorte de 2003 avait un niveau de recrutement relatif faible (l’indice d’abondance est d’environ 25 millions d’individus), ce qui a dû se traduire par une saison de pêche difficile pour l’année 2010, soit l’année médiane d’entrée de cette cohorte dans la pêche. Au contraire, la cohorte de 1999 est une des plus abondantes mesurées par le ministère (plus de 250 millions d’individus), ce qui suggère que la saison de pêche a été prolifique pour l’année 2006. Le taux de renouvèlement donne des informations utiles à la gestion, mais cet indice ne tient pas compte de la proportion de l’approvisionnement des différents stocks adultes adjacents à chacune des deux nourriceries. Par conséquent, l’origine des juvéniles présents dans le Saint-Laurent reste à ce jour inconnue, d’autant plus qu’il s’agit des uniques nourriceries présentes dans tout l’ouest de l’aire de répartition de cette espèce.
Contexte du projet
Le projet global dans lequel s’inscrit ma maîtrise a pour objectif principal de documenter l’état de la population exploitable de flétan du Groenland dans tout l’est du Canada, afin de contribuer à en faire une gestion durable. Les 4 objectifs spécifiques sont i) identifier les populations adultes ; ii) déterminer la connectivité génétique entre les populations de l’Est et de l’ouest de l’Atlantique ; iii) estimer la contribution des différents stocks reproducteurs dans le Saint-Laurent, et iv) déterminer le niveau d’activité migratoire par la chimie des otolithes et de déterminer les effets du sexe, de la température et de l’origine sur la croissance des juvéniles. Les différentes approches couvertes par les différents objectifs permettront de mettre en commun des informations complémentaires sur les dynamiques
qui influencent la croissance, l’abondance et la connectivité des populations de Flétan du Groenland. L’approche holistique de ce projet permettra au ministère Pêches et Océans Canada, qui assure la gestion des pêches commerciales du flétan du Groenland, d’intégrer différents aspects de la biologie de cette espèce dans son plan de gestion pour les années futures.
Objectifs
Le but de mon projet correspond au deuxième objectif spécifique cité au paragraphe précédent, c’est-à-dire d’estimer la contribution des différents stocks reproducteurs identifiés du Flétan du Groenland (Reinhardtius hippoglossoides) aux nourriceries présentes dans le Saint-Laurent. De façon plus spécifique, mon projet se divise en 2 objectifs principaux : 1) définir la structure des populations de flétans adultes présentes dans le golfe du Saint-Laurent et 2) déterminer l’appartenance des juvéniles aux stocks définis au premier objectif. Dans le cadre du premier objectif, nous comparons l’effet des marqueurs liés au sexe sur la structure de populations observée. Pour ce qui est du deuxième objectif, je compare la différence de proportion d’assignation entre les marqueurs de type SNPs et les micro-haplotypes, en plus d’en évaluer la stabilité temporelle. D’ailleurs, comme il s’agit des deux seules nourriceries identifiées dans la région sud-ouest de toute l’aire de répartition pour cette espèce, cette étude permettra de déterminer si des populations plus éloignées de l’Atlantique contribuent au renouvellement des stocks du Saint-Laurent.
Chapter 1:
Estimating the contribution of Greenland Halibut (Reinhardtius hippoglossoides)
stocks to nurseries by means of Genotyping-By-Sequencing: sex and time matter.
Chapitre 1 : Estimation de la contribution des stocks de Flétan du Groenland
(Reinhardtius hippoglossoides)
aux nourriceries à l’aide du
Génotypage-par-séquençage : le sexe et le temps sont important.
Résumé
L'identification des stocks et la quantification de leur contribution relative au recrutement sont des objectifs majeurs pour améliorer la gestion et la conservation des espèces marines exploitées. Le séquençage de nouvelle génération permet d’analyser des milliers de marqueurs génomiques et d’apporter la résolution nécessaire pour répondre à ces questions chez les espèces marines qui sont faiblement différenciées. Le Flétan du Groenland (Reinhardtius hippoglossoides) est un poisson plat largement exploité, particulièrement dans le golfe du St-Laurent, au Canada. On y retrouve 2 nourriceries connues, dont la contribution des juvéniles au renouvellement des différents stocks adultes reste inconnue à ce jour. Les buts de ce projet sont de i) déterminer la structure des populations de flétan du Groenland du Saint-Laurent et ii) d’estimer la contribution des différents stocks reproducteurs identifiés aux différentes nourriceries. Pour ce faire, nous avons échantillonné 100 juvéniles par nourricerie et 50 adultes de sites s’étendant du fjord du Saguenay jusqu’au large de Terre-Neuve, avec quelques sites échantillonnés sur 2 années consécutives pour évaluer la stabilité temporelle de cette contribution. Nos résultats montrent qu’après avoir retiré les marqueurs liés au sexe, la région de l’Estuaire/Golfe du Saint-Laurent forme une population distincte de l’Atlantique près de Terre-Neuve (Fst = 0.00146, p-value = 0.001). Les analyses d’assignation populationnelle
montrent que le recrutement dépend largement du stock du Saint-Laurent. Par ailleurs, on retrouve une contribution variable du stock de Terre-Neuve, variant de 1% pour la première année à 33% pour la deuxième, ce qui suggère un transport interannuel de larves variable selon la force des courants profonds. Cette étude sert de modèle pour l’identification des stocks pour les ressources halieutiques, dans un contexte où le milieu marin offre peu de barrières à la dispersion, en plus de démontrer l’importance des marqueurs liés au sexe et des répliques temporelles en génomique des populations.
Abstract
Identification of stocks and quantification of their relative contribution to recruitment are major objectives toward improving management and conservation of marine exploited species. Next-generation sequencing allows to analyze thousands of genomic markers which provide the resolution needed to address these questions in marine species with weakly differentiated populations. Greenland Halibut (Reinhardtius hippoglossoides) is one the most important exploited demersal species throughout the North Atlantic, in particular in the Gulf of St. Lawrence, Canada. There, two nurseries are known, the St. Lawrence Estuary and the northern Anticosti Island, but their contribution to the renewal of stocks remains unknown. The goals of this study were i) to document the genetic structure and ii) to estimate the contribution of the different identified breeding stocks to nurseries. We sampled 100 juveniles per nursery and 50 adults from seven sites ranging from Saguenay fjord to offshore Newfoundland, with some sites sampled over two consecutive years in order to evaluate the temporal stability of the contribution. Our results show that after removing of sex-linked markers, the Estuary/Gulf of St. Lawrence represent a stock which is genetically distinct from the Atlantic around Newfoundland (Fst = 0.00146, p-value = 0.001). Population assignment showed that recruitment in both nurseries is largely predominantly associated with the St. Lawrence stock. However, we found that the relative contribution of both stocks to the nurseries is temporally variable with 1% contribution of the Newfoundland stock one year but up to 33% for the second year, which may hypothetically be caused by year-to-year variation in larval transport into the Gulf of St. Lawrence. This study serves as a model for the identification of stocks for fisheries resources in a context where few barriers to dispersal occur, in addition to demonstrating the importance of considering sex-linked markers and temporal replicates in studies of population genomics.
Introduction
The use of genomic tools for conservation and management offers the opportunity to better understand the dynamics and demography of natural populations (Manel et al. 2005). Population genomics allows answering conservation issues, such as assessing reproductive success (Ford et al. 2015), testing for local adaptation (O’Malley et al. 2007), documenting migratory patterns of exploited and/or threatened populations (Larson et al. 2013) and defining management units (Palumbi 2004). In the case of marine species, the lack of statistical power is a main issue when trying to define stocks of exploited species, where exchange of genetic material is high due to minimal dispersal constraints and differentiation between stocks is further limited because of large effective population sizes (Waples et al. 2008). However, the improvement of next-generation sequencing since the mid-2000s has allowed the definition of subtle differentiations at fine local scales in order to delineate populations (i.e. stocks) by making it possible to genotype thousands of markers on hundreds of individuals in a relatively short time (Metzker 2010), as exemplified by recent studies of various marine organisms such as the American lobster (Homarus americanus) (Benestan et al. 2015), the giant California sea cucumber (Parastichopus californicus) (Xuereb et al. 2018) or the Atlantic cod (Gadus morhua) (Barth et al. 2017), to name a few.
Nevertheless, increasing the number of markers is not always sufficient to resolve population structure for species with large populations and/or high connectivity such as marine species (Gagnaire et al. 2015). For reduced-representation sequencing data, it is known that filtering parameters have a major impact on results (Puebla et al. 2014, Rodriguez-Ezpeleta et al. 2016; 2017, Shafer et al. 2017). For example, as shown by Gagnaire et al. (2015), removing SNPs under directional selection from assessment of migratory pattern and connectivity of marine species populations might result in elimination of informative loci with high Fst between “real” populations and thus creates false negative
interpretation. Sex-linked markers consist of genomic markers located in genomic regions associated to sex determinism such as sex chromosomes. These markers may also influence population structure results. As shown in Benestan at al. (2017), removing SNPs linked to sex resulted in a lower and more realistic estimation of differentiation for the anadromous species Arctic charr (Salvelinus alpinus) and in American lobster (Homarus
americanus). Despite these results, little to no attention has been made to evaluate the
importance of this type of marker in other species.
One specific application of genetic stocks definition is to determine their relative contribution in a context of mixed stock fisheries. In particular, this issue may be addressed using assignment methods, which consist of determining the most probable origin (or reference stock in the case of fisheries) of an individual or a group of individuals from unknown sources based on multi-locus genotype information (Dann et al. 2013, Ensing et al. 2013, Beacham et al. 2017). The statistical power of assignment tests highly depends on the level of differentiation between putative source populations (Pritchard et
al. 2000). When using markers such as single-nucleotide polymorphism (SNP), one way to
increase assignment success besides augmenting the number of SNPs is to combine multiple SNPs in linkage disequilibrium to form microhaplotypes (Kidd et al. 2013, 2014). For instance, McKinney et al. (2017) showed that using SNP microhaplotypes increased significantly the success of population assignment for Chinook salmon (Oncorhynchus
tshawytscha) of unknown origin to their native river, rising up to 14% compared with 6% for
the single SNPs per loci approach, using the same total number of genomic markers. Despite their potential to improve assignment success, the efficiency of microhaplotypes have yet to be tested on more species, particularly so in weakly structured marine species. Greenland Halibut (Reinhardtius hippoglossoides), also named turbot, is a flatfish of the Pleuronectidae family with a circumpolar distribution throughout the northern hemisphere. In the Gulf of St. Lawrence (Canada), spawning occurs every year during the winter months from January to March. Following emergence, larvae drift for a few months and then settle in a nursery area until growth is complete (Sohn et al. 2010). A nursery can be defined as an area where the density of juveniles (fish older than larvae, but in which gonads are not yet mature) is above average compared to the range of the species. These habitats are also usually characterized by an abundance of smaller prey and a relatively low predation rate (Beck et al. 2001, Dahlgren et al. 2006). There are two known nurseries of Greenland Halibut in the St. Lawrence system, one being located in the Estuary and the other in the Gulf north of Anticosti Island (Youcef et al. 2013). Benthopelagic fishes such as Greenland Halibut are characterized by a high potential to disperse due to the prolonged pelagic larval phase, which increases connectivity between populations, thus complicating the definition of genetically distinct population and management units (Bailey 1997, Hoarau et al. 2002). Several studies have aimed at documenting the extent of
genetic connectivity of the species in the western Atlantic with different outcomes. First, several authors proposed that Greenland halibut from the Gulf of St. Lawrence are comprise in a single population being distinct from other stocks in the Atlantic. This has been supported from studies on allozymes (Fairbairn 1981) and prevalence of parasites (Khan et al., 1982, Arthur and Albert 1993). Arthur and Albert (1993) looked more precisely within the Gulf of St. Lawrence and nearby Atlantic Ocean and further suggested that Halibut from the Saguenay Fjord was distinct from the rest of St. Lawrence system. In contrast to those previous studies, Roy et al. (2014) suggested based on the analysis of 12 microsatellite markers Greenland halibut from that the Estuary/Gulf of St. Lawrence, eastern Labrador and Newfoundland comprised a single panmictic population. It is possible that these contradictory results stem from the limited resolution offered by previous types and number of markers that were used previously to assess population structure in the Greenland Halibut, thus supporting the need to revisit the population genetics of the species using a more powerful method, such as Genotyping-by-Sequencing (GBS). Moreover, no study to date have attempted to document the contribution of different putative halibut populations to the juveniles that use the two known nurseries within the St. Lawrence system.
The first goal of this study was to test the null hypothesis of the absence of population structure within Greenland Halibut from the Estuary/Gulf of St. Lawrence and Atlantic sampling locations near Newfoundland. The second goal was to determine the origin of juveniles from the two nurseries located within the Estuary/Gulf of St. Lawrence To do so, we used GBS to genotype a total of 850 adults from 7 localities distributed from Saguenay Fjord to offshore of Newfoundland and 200 juveniles from both nurseries (two annual temporal replicates of 100 for each nursery) at 26 965 filtered genomic SNPs. We also assessed the effect of sex-linked markers on the definition of population structure. We also compared the efficiency of single SNPs versus microhaplotypes on the success of population assignment being achieved. In the light of our results, we discuss i) the importance of using a large number of markers to refine the resolution of population structure in weakly structured marine species, ii) the importance of sexing individuals being genotyped in order to be able to sort out sex-linked markers for the data set being used to perform accurate population structure analysis, and iii) the importance of performing temporal replicates to assess the temporal stability of patterns being
documented, namely pertaining to the estimate of different populations to mixed samples (e.g. on nursery grounds).
Material and methods
Sampling
Fisheries and Oceans Canada (DFO) and volunteer fishermen from the area collaborated to achieve the sampling (Fig. 1). Every fish was measured and its sex identified at the time of capture. Fish larger than 31 cm were considered mature, whereas those under were classified as juveniles (Morin and Bernier 2003, DFO 2012). The nurseries sampled in the St. Lawrence are located in the estuary (ESTJ) (49.0993, -67.3247) and the North of Anticosti Island (ANTJ) (49.7913, -62.4102). A total of 100 individuals/year from each nursery were collected in late summer of 2016 and 2017, totalling 400 juvenile samples. We also sampled seven adult localities, namely: Saguenay (SAGU) (48,339722; -70,849,722), Estuary (ESTU) (48,9548; -67,9288), northern Anticosti Island (NANT) (49.7807; 62.5422), Gaspé (GASP) (49.533333; 64.85), Esquiman (ESQU) (49.9485, 59.516), Labrador (NFLD) (53.218056, 53.600556) and Newfoundland (NFLG) (47.04; -48). Presumed adult stocks of Greenland Halibut were selected based on their proximity to the two nurseries and to provide longitudinal coverage of its range in the St. Lawrence system, with the assumption that this stock might be partially or completely isolated. We also included two sampling sites outside the St. Lawrence to test whether the Atlantic stock is genetically connected to the St. Lawrence region. Fifty adult individuals for each site, with ESTA, ESQU sampled twice, that is late summer of 2016 and 2017 (total of 450 adult samples). Part of the pelvic fin was recovered and stored in 95% ethanol (EtOH) until DNA extraction.
Fig 1. Map of sampling sites in Saguenay fjord (SAGU), St. Lawrence Estuary (ESTU,
ESTJ), Gaspé (GASP), North Anticosti Island (NANT, ANTJ), Esquiman (ESQU) and Atlantic Ocean (NFLD, NFLG). The Estuary and northern Anticosti Island nurseries (ESTJ and ANTJ) are defined by the triangles and adult populations of the St. Lawrence and Atlantic (SAGU, NFLD, ESTU, NANT, GASP and ESQU) by circles. Sites that are sampled for a single year are colored in pink for the first year of sampling and in blue for the second. Those sampled for the two consecutive years are colored in green.
DNA extraction
The genetic material was extracted according to the protocol detailed in Aljanabi and Martinez (1997). Samples were migrated on a 1% agarose gel to ensure genomic DNA was sufficiently abundant for sequencing and the solution of acceptable purity. Subsequently, DNA concentration was determined by colorimetry using a spectrophotometer (NanoDrop).
Bioinformatics and sequencing
Following DNA extraction, every sample was diluted to a concentration of 10ng/L in 10 L to normalize at 100 ng of DNA per individual. A total of ten 96-pools chips with a mean of 75 individuals were sequenced. Double digest RAD-sequencing libraries were prepared using a protocol inspired by Mascher et al. (2013). Two non-specific endonucleases (namely PstI and MspI) and a digestion buffer (NEB4) were combined to cut genomic DNA for a period of two hours at 37°C. Chips were then incubated at 65°C for 20 minutes to stop enzymatic reaction. DNA fragments were combined to adaptors at both ends using a ligation master mix and a T4 ligase. All samples were incubated at 22°C for two hours. Prepped DNA fragments were pooled in a 96-plex and cleaned using a QIAquick PCR purification kit. Every sampled individual was barcoded with a unique six nucleotide sequence. After the amplification of pooled fragments was complete using PCR, the samples were sequenced on the platform ION PROTON™ System of the company Life Technologies located at the Institut de Biologie Intégrative et des Systèmes (Laval University, Québec, Canada) that specifically targets 100 bp DNA fragments.
The resulting dataset was checked for overall quality using FASTQC (http://www.bioinformatics.babraham.ac.uk/projects/fastqc/). Adapters were removed from reads using cutadapt v1.8.1 (Martin 2011). Then, they were demultiplexed and truncated to 80 pb with the process_radtags module implemented in the software STACKS v1.48 (Catchen 2013). Cleaned DNA reads were aligned to a mostly complete reference genome of a mature female Halibut from the Estuary of St. Lawrence that was assembled at McGill University and Genome Quebec Innovation Center (Ferchaud et al. 2019, in preparation) with the package software BWA (Li et al. 2009) with parameters fixed at 500 for the
maximum occurrence of a read (c = 500), no gap open penalty (O = 0), a gap extension penalty of 2.2 (E = 2.2) and alignments with score lower than 0 were discarded (T = 0). SNPs were identified at each locus with pstacks with a minimum depth of coverage to report a stack set to 1 (m = 1). Then, cstacks was selected to construct a reference catalog with default parameters. Individual SNPs were called using the catalog with sstacks also with parameter left as is. The minimum depth was fixed to 7 (m = 7) using populations module in STACKS set with the minimum number percentage of individuals in a population required to process a locus set to 0.6 (r = 0.6), the minimum number of populations a locus must be present to process it fixed at 6 (p = 6) and the maximum p-value to keep a Fst
measurement set to 0.1. After, the ddRADseq dataset was filtered for SNPs calling at over 70% of genotyped individuals in every population (see details in table 1). Following filtering
was performed with stacks_workflow available online
(https://github.com/enormandeau/stacks_workflow). Reads were filtered for a minimum coverage depth of 10X and a maximum coverage depth of 100X. This step ensures working with markers that have enough coverage to limit false positives and also remove those that could be located in repetitive segments in the genome, which causes SNP overrepresentation. Next, we removed SNPs that were genotyped in less than 70% of overall individuals. Observed heterozygosity was set to be superior than 0.5 (Ho > 0.5) within samples to pull out of the dataset potentially homeologs markers (Davey et al. 2011). Both individuals and loci with a proportion of more than 0.1% of missing genotype overall were discarded. Minor allele frequency was set to more than 0.001 (MAF > 0.001) for all of the sampling sites to make sure any sequencing error from the dataset was removed. Finally, a relatedness analysis was performed to remove individuals closely related (relatedness = 1) using VCFtools (Danecek et al., 2011). When a pair of individuals with a high relatedness coefficient originated from the same sampling site, one was selected randomly to be kept in the dataset, if not, both individuals were discarded. One SNP per read was kept based on which one had the highest minor allele frequency.