HAL Id: tel-03207729
https://tel.archives-ouvertes.fr/tel-03207729
Submitted on 26 Apr 2021HAL is a multi-disciplinary open access archive for the deposit and dissemination of sci-entific research documents, whether they are pub-lished or not. The documents may come from teaching and research institutions in France or abroad, or from public or private research centers.
L’archive ouverte pluridisciplinaire HAL, est destinée au dépôt et à la diffusion de documents scientifiques de niveau recherche, publiés ou non, émanant des établissements d’enseignement et de recherche français ou étrangers, des laboratoires publics ou privés.
L’éditosome du chloroplaste : questions, éléments de
réponses et digressions
Kévin Baudry
To cite this version:
Kévin Baudry. L’éditosome du chloroplaste : questions, éléments de réponses et digressions. Géné-tique. Université Paris Saclay (COmUE), 2019. Français. �NNT : 2019SACLE036�. �tel-03207729�
N N T : 2 0 1 9 S A C L E 0 3 6
L’éditosome du chloroplaste
questions, éléments de réponses et digressions
Thèse de doctorat de l'Université Paris-Saclay Préparée à l’Université d’Evry Val d’Essonne (UEVE) et à
l’Institut des Sciences des Plantes Paris-Saclay (IPS2)
École doctorale n°577 Structure et Dynamique des Systèmes Vivants (SDSV)
Spécialité de doctorat: Sciences de la vie et de la santé
Thèse présentée et soutenue à Gif-sur-Yvette, le 17/12/2019, par
Kévin BAUDRY
Composition du Jury : Ian SMALL
Professeur, University of Western Australia Rapporteur
Philippe GIEGÉ
DR, CNRS (– IBMP) Rapporteur
Bénédicte STURBOIS
Professeur, UEVE (– IPS2) Présidente
Dominique RUMEAU
CR, CNRS (– CEA Cadarache) Examinatrice
Francis-André WOLLMAN
DR, CNRS (– IBPC) Examinateur
Claire LURIN
DR, INRA (– IPS2) Directrice de thèse
Wojciech MAJERAN
Page | 2 A mon grand-père, Jean Corblin, qui m’a appris à se battre pour réaliser ses rêves,
Page | 4
‘Science is much more than a body of knowledge.
It is a way of thinking.’
– Carl SAGAN, 1990
Dans Why we need to understand Science, Skeptical Inquirer (1990).Page | 6
Remerciements
Merci ! Après deux stages et une thèse au sein de l’équipe Organellar Gene Expression, des liens ont forcément été tissés ; il y a donc plein de personnes que j’aimerais remercier. Tout d’abord, je tiens à remercier l’équipe OGE entière, ainsi que l’équipe de Jean et de Marie-Laure, pour les discussions sérieuses mais surtout pour la bonne ambiance, la sympathie et les bons moments passés avec vous tous. De la même façon, je remercie toutes ces personnes qui ont réussi à supporter mon caractère dans les périodes difficiles, je sais que ça n’a pas dû être facile. Également, je tiens à remercier ma famille, mes amis et les pilotes du Volant Montalbanais, pour leur soutien moral durant l’ensemble de ma thèse.
Je tiens à remercier Claire, Wojciech et Etienne, pour m’avoir donné ma chance (en stage puis en thèse), leur patience infinie, leur encadrement et leurs encouragements, chacun à sa manière. Pour les mêmes raisons, je remercie aussi, Damien et Benoît, qui ont su m’écouter, me conseiller et me permettre d’avancer dans une ambiance « bienveillante ». Plus particulièrement, je remercie Etienne pour son implication dans ma formation mais surtout pour avoir été une oreille très attentive lors de mes moments de doute et d’angoisse.
Benoît, grâce à toi j’ai appris que les « plantes supérieures sont des procaryotes comme les autres » ! Et finalement, non, je ne t’ai pas mis dans les non-remerciements !
Je remercie également Bastien et Livia qui m’ont beaucoup aidé et hébergé quand j’ai perdu mon appartement.
Aussi, je remercie Dario, pour sa bonne humeur quotidienne et sa sympathie.
Je remercie Emmanuelle, pour avoir pris le temps de me former sur de nombreuses expériences et pour sa collaboration sur l’ensemble du projet CBSX. Je remercie aussi Jean B. pour m’avoir formé sur les Western Blot et les pull-down. Également, je remercie Marie-Laure et Guillem pour m’avoir patiemment enseigné les stats. Je remercie Julien, pour avoir su me rappeler (très) régulièrement que la date de rendu du manuscrit approchait.
De même, je remercie les stagiaires, les thésards et post-doc, que j’ai eu la chance de côtoyer et avec qui j’ai passé de super moment aussi bien scientifiquement que amicalement. En particulier, je ne peux pas ne pas remercier Bastien, Houda, Tiffany et Linda !
Enfin, je remercie tous ceux qui m’ont apporté quelque chose professionnellement ou humainement durant ces trois années de thèse !
Page | 7
Abréviations
A : Adénine aa : acide aminé
AD : Activation Domain
ADN : Acide Désoxyribonucléique ADNc : ADN complémentaire ADN-T : ADN de transfert
AGI : Arabidopsis Genome Initiative AIM : Arabidopsis Interactome Mapping AMP : Adénosine Monophosphate ARN : Acide Ribonucléique ARNm : ARN messager ARNr : ARN ribosomal ARNt : ARN de transfert At : Arabidopsis thaliana ATP : Adénosine Triphosphate
BiFC : Bimolecular Fluorescence
Complementation
BS : Bundle Sheath (gaine péri-vasculaire) C : Cytosine
CBBC : cycle de Calvin-Benson-Basham CBS : Cystathionine-β-Synthase
CDCP : CBS Domain Containing Protein
Col0 : Arabidopsis thaliana écotype
“Columbia 0”
DB : DNA Binding domain
dNTP : désoxyribonucléotide Triphosphate DO : Densité optique
G : Guanine
IPS2 : Institute of Plant Science Paris-Saclay IR : inverted repeat
LB : Lysogeny broth MS : Mésophylle
MEF : Mitochondrial Editing Factor
MORF : Multiple Organellar RNA editing Factor
MS : Milieu Murashige et Skoog NaCl : Chlorure de sodium
NEP : Nuclear Encoded RNA Polymerase ORF : Open Reading Frame (cadre ouvert de lecture)
ORRM : Organelle RNA Recognition Motif-containing
pb : paire de bases
PCR : Polymerase Chain Reaction (réaction de polymérisation en chaine)
PEP : Plastid-Encoded RNA Polymerase POPS : Plateforme Transcriptomique de l’IPS2
PPR : Pentatricopeptide Repeat PS : Photosystème
Page | 8 RIP : RNA editing factor Interacting Protein
RNAseq : Séquençage des ARN ROS : Reactive Oxygen Species RRM : RNA Recognition Motifs
qRT-PCR : Rétro-transcription suivie d’une PCR quantitative
T : Thymine
TAIR: The Arabidopsis Information
Resource
TMM : Trimmed Mean of M values TPR : Tetratricopeptide Repeat TRX : Thiorédoxine
U : Uracile
UTR : Untranslated region Wt : Wild type
YFP : Yellow Fluorescent Protein Y2H : Yeast Two Hybride
Page | 9
Sommaire
Remerciements ... 6 Abréviations ... 7 Sommaire ... 9 Définitions ... 16 Introduction ... 18Le chloroplaste : un organite spécialisé dans la photosynthèse issu de l’endosymbiose d’une cyanobactérie ... 20
Aspect et organisation membranaire du chloroplaste ... 23
Généralités sur la photosynthèse oxygénique ... 24
Phase lumineuse, chaine de transport d’électrons ... 24
Phase sombre, fixation du carbone atmosphérique ... 28
Photosynthèse en C4 : spécialisation chloroplastique ... 32
Le génome plastidial, un chromosome réduit au sein du nucléoïde lié au génome nucléaire ... 36
Transfert de gènes endosymbiotiques et ses conséquences ... 36
La cellule végétale : un système à trois génomes complémentaires ... 37
Organisation du génome plastidial ... 40
Le Nucléoïde : siège du génome plastidial ... 43
Points de contrôle de la photosynthèse ... 47
Transcription... 47
Modifications post-transcriptionnelles, maturation des transcrits primaires ... 49
Contrôle de l’activité au niveau post-traductionnel ... 56
Edition C vers U des ARN du Chloroplaste ... 61
L’édition des ARN, généralités ... 64
L’édition des ARN des organites chez les embryophytes... 68
Éditosome, machinerie de l’édition C vers U des ARN du chloroplaste ... 71
Objectifs de la Thèse ... 78
Résultats ... 80
Chapitre 1er : Le domaine DYW est-il indispensable dans l’éditosome ? ... 82
Page | 10
CRR4 et DYW1, deux protéines PPR pour un seul site d’édition ... 84
CRR4DYW1, une PPR chimérique fonctionnelle ... 85
DYW2 et NUWA, deux membres centraux de l’éditosome des PPR-E+ ... 87
CLB19, une PPR- E+ du chloroplaste ... 87
DYW2, NUWA et l’éditosome des PPR-E+ ... 89
MEF8 et les autres DYW1-like ... 92
Famille de PPR DYW1-like ... 92
DYW3 et DYW4 ... 93
MEF8 et MEF8S ... 93
Le modèle « unifié », une intégration des modèles d’éditosome ... 94
Bilan des trois modèles existants ... 94
Discussion du modèle proposé ... 95
Étude des interactions PPR-PPR par double hybride chez la levure ... 96
Protéines d’intérêt ... 96
Matrice d’interaction ... 97
NUWA, stabilisateur de l’interaction entre DYW2 et les PPR-E+ ? ... 98
Approche en levure, par un système « Triple-hybride » ... 98
Approche par complémentation fonctionnelle grâce à une fusion CLB19DYW2 ... 99
Conclusion ... 107
DYW1 et DYW2, des garants de l’édition dans les organites ? ... 108
Introduction ... 108
Inversion des domaines C-terminaux entre CRR4 et CLB19 ... 110
Étude de PPR-E2 et –E+ de novo ... 113
Conclusion ... 117
Rôle du domaine E/E+ chez les PPR-DYW canonique ... 118
Le domaine E/E+, une région conservée ... 118
Mutations ponctuelles dans le domaine E/E+, construction CRR287A ... 119
Conclusion ... 120
Discussion ... 120
Chapitre 2 : Recherche de nouveaux membres de l’éditosome ... 124
Page | 11
Recherche de candidats par crible Y2H ... 127
Crible double hybride ... 128
Détermination du réseau d’interaction ... 130
Étude des candidats facteur d’édition ... 133
Étude de mutants d’insertion en conditions standard de culture ... 133
Éditotype partiel des mutants... 134
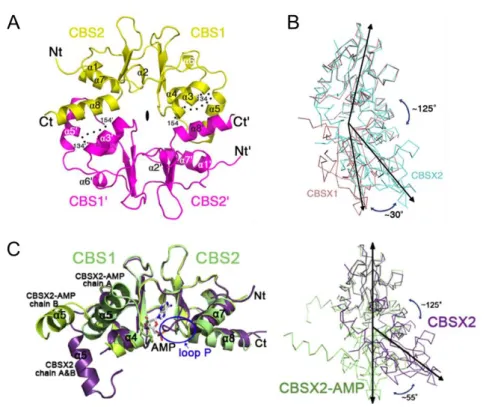
Étude approfondie de CBSX2 ... 136
Trois CBSX adressées aux organites ... 136
Étude du double-mutant cbsx1cbsx2 ... 140
L’interaction entre DYW2 et CBSX2 est probablement un artefact... 142
Discussion ... 144
Chapitre 3 : Les CBSX chloroplastiques, régulateurs de l’activité de thiorédoxines ... 146
Connaissances bibliographiques concernant CBSX1 et CBSX2 ... 148
Les CBSX interagissent avec les TRX ... 148
Les CBSX activent les TRX plastidiales ... 148
Les sur-expresseurs des CBSX sont stériles ... 149
Mise au point des conditions expérimentales ... 150
Production des CBSX recombinantes ... 150
Test d’activation MDH ... 151
Article : CBSX2, régulateur de TRX dans le chloroplaste ... 156
Manuscrit de l’article ... 157
Résultats supplémentaires ... 192
CBSX2-TRXm, un complexe dynamique ... 192
Mesure de l’activité MDH in planta ... 193
NTRC, un interactant de CBSX2 impliqué dans le stress oxydant ... 195
Conclusions ... 197
Chapitre 4 : Expression des gènes plastidiaux au cours de la différenciation des chloroplastes chez les plantes en C4 ... 198
Mise en contexte ... 200
Analyse du transcriptome associé aux nucléoïdes ... 201
Page | 12
Étude des données préliminaires ... 203
Conclusion ... 213
Étude de mutants de candidats impliqués dans la différenciation ... 213
CBSPPR : une PPR du nucléoïde à domaine CBS de fonction inconnue ... 214
PPR66L : une PPR de la gaine impliquée dans l’épissage de ndhA ... 216
PPR287 : une PPR du mésophylle impliquée dans la stabilité des ARNr ... 219
Discussion ... 221
Discussion générale ... 224
Bilan des résultats obtenus durant la thèse ... 226
L’éditosome, un complexe réel ou purement théorique ? ... 228
Les éditosomes, de petits complexes site-spécifiques ... 228
Les éditosomes, de petits complexes dynamiques et transitoires, se formant autour d’un cœur structurel et catalytique ... 229
L’éditosome n’existerait peut-être pas ... 230
Conclusion ... 231
Rôle de l’édition dans l’expression des gènes des organites ... 231
Hétérogénéité de l’édition, à toutes les échelles ... 231
L’édition, un processus de correction de la séquence peptidique et structure secondaire de l’ARN ... 232
Traduction des transcrits édités ... 233
L’édition peut-elle être une fonction régulatrice de l’expression des gènes ? ... 234
Une origine possible d’une édition neutre ... 235
L’éditosome, une complexification incessante expliquée par la théorie CNE ... 236
Conclusion ... 237
Matériels et Méthodes ... 238
Matériel biologique utilisé durant la thèse ... 240
Matériel végétal et conditions de culture ... 240
Souches bactériennes ... 241
Souches de levures ... 242
Plasmides utilisés ... 242
Méthodes ... 243
Page | 13
Transformations ... 244
PCR ... 248
Clonage ... 249
Double hybride chez la levure (Y2H) ... 251
Extraction d’ADN génomique ... 252
Transcriptomique ... 253
Production de protéines recombinantes ... 255
Activités TRX ... 257
Pull-down CBSX-TRXm1 ... 259
Dosage des pigments photosynthétiques ... 259
Préparation de BS/MS et Purification de Nucléoïdes ... 260
Liste des amorces utilisées ... 262
Annexes ... 266
Annexe S1 : carte du plastome de Zea mays B73 ... 268
Annexe S2 : Gènes du plastome d’A. thaliana classés par fonction biologique ... 269
Annexe S3 : Alignement des motifs post-E2 des PPR-E2 avec les domaines E+ des PPR-E+ d’A. thaliana ... 270
Annexe S4 : Article « Two interacting PPR proteins are major Arabidopsis editing factors in plastid and mitochondria », PNAS 2017 ... 271
Annexe S5 : particularités de la sous-famille DYW1-like ... 278
Annexe S6 : Sites d’édition affectés chez mef8-2 ... 279
Annexe S7 : Séquences produites par synthèse de gènes pour le Y2H ... 281
Annexe S8 : Alignement des domaines E+ des 58 PPR-E+ d’A. thaliana ... 282
Annexe S9 : Séquences des PPR produites par synthèse de gènes pour les complémentations fonctionnelles ... 283
Annexe S10 : Alignement des différentes versions de CRR28 utilisées ... 287
Annexe S11 : Effet du NaCl sur la croissance d’A. thaliana Col0 ... 288
Annexe S12 : Différents stress abiotiques sur les mutants cbsx ... 289
Annexe S13 : Stress oxydant sur le double-mutant cbsx1cbsx2 ... 290
Annexe S14 : Script pour le calcul de 96 pentes d’activité MDH ... 291
Annexe S15 : Taux d’épissage des introns dans les échantillons W22 ... 293
Page | 14
Annexe S17 : Comptage par gène pour les échantillons W22 ... 295
Annexe S18 : Analyse de couverture pour les échantillons W22 ... 297
Annexe S19 : Couverture de l’opéron des ARNr des échantillons W22 ... 298
Annexe S20 : Localisation subcellulaire de AtCBSPPR1... 299
Table des Figures ... 300
Table des Tableaux ... 302
Page | 16
Définitions
Afin d’éviter toute confusion à la lecture de ce manuscrit, la définition utilisée pour certains termes est précisée dans cette section. De même, il a été choisi une nomenclature pour l’écriture des noms de gènes, transcrits, protéines, mutants et sites d’édition.
Nomenclature d’écriture : Noyau : - Gène : CRR4, DYW1 - Protéine : CRR4, DYW1 - mutant : crr4, dyw1 - double-mutant : crr4dyw1 Organite : - Gène : ClpP, RpoA, NdhD - ARN : clpP, rpoA, ndhD - Protéine : ClpP, RpoA, NdhD - Site d’édition : ndhD_117166
(Gène & position génomique du site)
Organite : Le terme « organite » ne désigne dans ce manuscrit que les organites contenant un génome, c’est-à-dire les mitochondries et les plastes.
Édition des ARN : L’édition des ARN est un terme vague (défini précisément et discuté dans la partie « L’édition des ARN, le problème de la définition » page 66). Sauf mention contraire, dans ce manuscrit, il est employé pour parler de la modification spécifique d’une Cytidine en une Uridine dans un ARN d’un organite.
Site d’édition : Un site d’édition est une Cytidine éditée en Uridine au niveau de l’ARN. On définit un site d’édition par l’analyse de données RNAseq, comme étant une position ayant une couverture d’au moins 20 reads et un taux d’édition d’au moins 5%.
Page | 18
Page | 20 Cette thèse intitulée « L’éditosome du chloroplaste : questions, éléments de réponses et digressions » porte principalement sur l’étude de l’édition C vers U des ARN du chloroplaste et du complexe réalisant cette étape de maturation des transcrits. Le chloroplaste, issu de l’endosymbiose d’une cyanobactérie, est l’organite réalisant la photosynthèse. Nous commencerons cette introduction par un rappel sur la photosynthèse. Ensuite, les principales caractéristiques du génome plastidial et du nucléoïde, le complexe organisant ce génome, seront exposées. La mise en place des complexes photosynthétiques dans le chloroplaste est contrôlée en plusieurs points depuis la transcription des gènes plastidiaux jusqu’aux modifications post-traductionnelles de ces protéines. Ces points de contrôle et les protéines impliquées seront abordés dans la troisième partie. Enfin, dans la dernière partie, nous détaillerons l’édition des ARN et l’éditosome, puis nous énoncerons les objectifs de la thèse.
Le chloroplaste : un organite spécialisé dans la photosynthèse issu de
l’endosymbiose d’une cyanobactérie
La « vie » sur Terre, serait apparue il y a environ 3.5 milliards d’années et les eucaryotes, il y a 2 milliards d’années (López-García and Moreira, 2015; López-García et al., 2017). Les analyses phylogénétiques montrent que l’on peut classer les eucaryotes en différents super-groupes, dont celui des Archaeplastida ou Plantae (Baldauf, 2003; Burki, 2014) (Fig. 1). L’ensemble des eucaryotes possède un organite particulier, la mitochondrie. Cet organite résulte de l’endosymbiose d’une α-protéobactérie, par une cellule dite hôte (Dyall et al., 2004). Les plantes sont des eucaryotes très particuliers possédant, en plus d’une mitochondrie, un plaste issu d’une endosymbiose primaire (Encadré n°1).
Encadré n°1 : L’émergence de la lignée verte, endosymbiose primaire d’une cyanobactérie
La lignée verte correspond au super-groupe des Archaeplastida (ou Plantae) et regroupe les Glaucophytes, les Rhodophytes (algues rouges) et les Viridiplantae. Les Viridiplantae regroupent les algues vertes (uni- et pluricellulaires) et les Embryophytes. Enfin l’embranchement des Embryophytes regroupe les bryophytes, les fougères, les gymnospermes et les angiospermes (Fig. 1).
Page | 21 Ce super-groupe des Plantae se définit également comme regroupant les organismes possédant un plaste résultant directement de l’endosymbiose d’une cyanobactérie (Burki, 2014). L’endosymbiose dite primaire d’une cyanobactérie aurait eu lieu il y a 1,2 à 1,5 milliards d’années (Dyall et al., 2004). Les plastes des Plantae ont donc une origine commune, étant tous issus de cette endosymbiose primaire. Toutefois, les Plantae ne sont pas les seules eucaryotes possédant des plastes ou étant photosynthétiques. En effet, il existe de nombreux eucaryotes possédant des plastes dit secondaires, voire tertiaires (Archibald, 2009). Une endosymbiose secondaire est l’endosymbiose d’un eucaryote portant un endosymbiote primaire, comme une algue verte. Une fois acquis, l’endosymbiote secondaire peut réduire jusqu’à ce qu’il ne reste plus que son plaste (par exemple chez Euglena, une Excavata), ou bien le noyau peut subsister sous une forme réduite nommée nucléomorphe (par exemple chez Chlorarachniophytes du groupe des Rhizaria). Également, un plaste secondaire peut perdre ses fonctions photosynthétiques et devenir un apicoplaste comme chez Plasmodium (Apicomplexans) (Dyall et al., 2004; Archibald, 2009). Enfin, certains eucaryotes pratiquent la kleptoplastie, c’est la rétention temporaire des plastes des proies. C’est le cas par exemple du dinoflagellé Dinophysis caudata ou du gastéropode marin Elysia chlorotica, qui sont capables de retenir respectivement les plastes de Cryptophytes et de Stramenopiles pendant plusieurs mois (Kim and Archibald, 2010).
Après plus d’un milliard d’années d’évolution, l’endosymbiote primaire, issu d’une cyanobactérie, a permis la formation d’une famille d’organites, les plastes. Ils se différencient selon la fonction de la cellule hôte en différents types spécialisés, tels que le chromoplaste, l’amyloplaste ou le chloroplaste. Par exemple, l’amyloplaste, présent dans les racines ou dans les rhizomes, est spécialisé dans le stockage de l’amidon (Jarvis and López-Juez, 2013). L’ensemble des travaux effectués durant cette thèse a été réalisé sur le chloroplaste, type spécialisé dans la photosynthèse. Nous allons à présent aborder la structure du chloroplaste et faire un rappel général sur la fonction principale qu’il remplit : la conversion d’énergie lumineuse en énergie métabolique via la photosynthèse.
Page | 22
Figure 1 : Arbre phylogénétique des eucaryotes
Représentation des super-groupes d’eucaryotes : Excavates, Opisthochontes, Amoebozoa, SAR (Stramenopiles (i.e. hétérochontes), Alvéolates et Rhizaria) et Archaeplastida. Le super-groupe des Archeaplastida (ou Plantae ou lignée verte) contient les Glaucophytes, les Rhodophytes (algues rouges) et les Viridiplantea. Ces derniers se séparent en algues vertes et Embryophytes. Adaptée de Burki (2014).
Il est à noter que la suite de cette partie a été rédigée avec l’aide de la récente réédition de l’ouvrage « La Photosynthèse : Processus physiques, moléculaires et physiologiques » des éditions Quae (Farineau and Morot-Gaudry, 2018). Ainsi, sauf mention contraire, toutes les informations de cette partie sont issues de cet ouvrage.
Page | 23
Aspect et organisation membranaire du chloroplaste
Le chloroplaste est un organite mesurant de 5 à 10 µm. Il est entouré par une enveloppe constituée de deux membranes, une interne et une externe. Au sein du chloroplaste se trouve un réseau membranaire complexe, les thylacoïdes. Les vésicules constituant ce réseau forment par endroit des empilements nommés grana. Les différents grana sont reliés entre eux par des thylacoïdes non-granaires, parfois appelés lamelles (lamellae). L’espace interne des thylacoïdes est le lumen (Fig. 2).
La chaîne de transport d’électrons convertissant l’énergie des photons en énergie chimique, est située au sein de la membrane des thylacoïdes. Cette chaîne est constituée de nombreux complexes protéiques associés à des chlorophylles et caroténoïdes dont la composition et le fonctionnement seront détaillés dans cette partie. Le stroma entoure les thylacoïdes, il contient notamment toutes les enzymes du cycle de Calvin-Benson-Basham, les autres enzymes solubles des voies métaboliques du chloroplaste, ainsi que les grains d’amidon. Le stroma contient également le génome du chloroplaste organisé sous forme de nucléoïdes.
Figure 2 : Aspect microscopique d'un chloroplaste et des thylacoïdes
En haut à gauche, schéma d’une cellule végétale (N : noyau ; M : mitochondrie ; TIC/TOC : système d’import protéique du chloroplaste ; PM : membrane plasmique). En bas à gauche, électronographie d’un chloroplaste de Pisum sativum (IE : membrane interne ; OE : membrane externe ; IES : espace intermembranaire). A droite, schéma et électronographie des thylacoïdes (bleu : PSII ; violet : cytochrome b6f ; rouge : PSI ; vert : ATP synthase). Adaptée de Soll et Schleiff (2004).
Page | 24
Généralités sur la photosynthèse oxygénique
Les plantes vertes (Viridiplantae) sont capables de convertir l’énergie solaire véhiculée par les photons en une énergie organique métabolisable via un processus biophysique ayant lieu dans leurs chloroplastes : la photosynthèse. On distingue deux grands types de photosynthèse : la photosynthèse anoxygénique, effectuée en particulier par les bactéries pourpres et la photosynthèse oxygénique, effectuée par les cyanobactéries et les plantes. Cette photosynthèse se caractérise par une émission de dioxygène et une consommation d’eau, selon l’équation globale : 6 CO2 + 12 H2O + h C6H12O6 + 6 H2O + 6 O2, où h représente l’énergie photonique. Au travers de cette équation, on voit la conversion du dioxyde de carbone en une molécule organique : le glucose (C6H12O6) qui sera par la suite
stocké sous forme d’amidon ou utilisé pour produire de l’énergie via la respiration mitochondriale.
L’équation globale de la photosynthèse est très simple mais masque un mécanisme complexe qui requiert de très nombreux acteurs protéiques. En particulier, la photosynthèse se découpe en deux phases. La phase photochimique, ou phase lumineuse, met en jeu le transport d’électrons au sein de la membrane des thylacoïdes et crée un gradient de protons au travers de cette dernière grâce à l’énergie lumineuse. Cette phase produit un déchet, le dioxygène, ainsi que des intermédiaires photosynthétiques, le NADPH et l’ATP. Par la suite, ces intermédiaires sont consommés au cours de la phase sombre, ou non-photochimique, dans le stroma, pour réduire le carbone minéral (CO2) en carbone organique (glucide).
Phase lumineuse, chaine de transport d’électrons
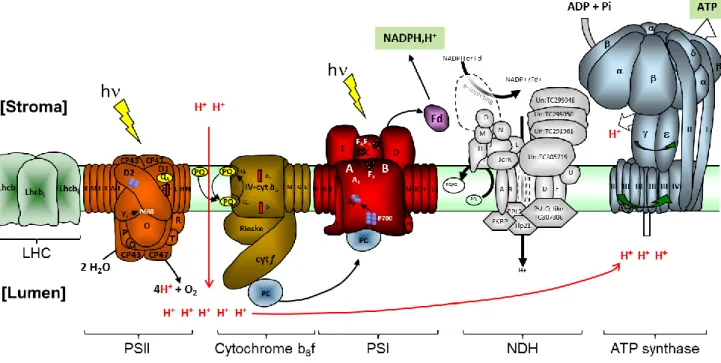
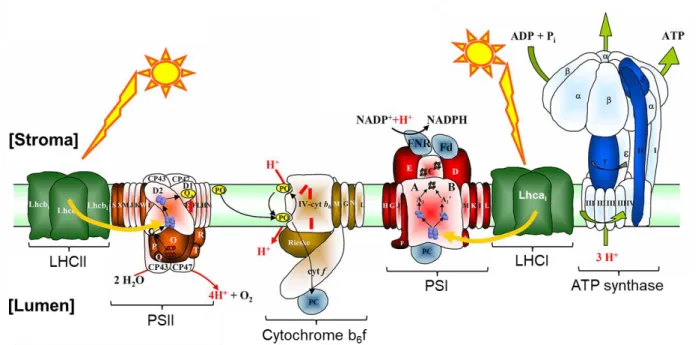
Cette phase est lumière-dépendante et prend place dans la membrane des thylacoïdes. Un ensemble de complexes protéiques permet le transport des électrons de la molécule d’eau vers un accepteur final pour créer du pouvoir réducteur sous forme de NADPH grâce à une succession de réaction d’oxydo-réduction (redox). Le transport des électrons génère également un gradient de protons entre le lumen et le stroma qui sera utilisé pour produire de l’ATP. L’ensemble des complexes impliqués est nommé la chaîne de transport d’électrons. Afin de capter l’énergie lumineuse permettant les transferts d’électrons le long de la chaîne, les protéines sont associées à des pigments, les chlorophylles et les caroténoïdes.
Page | 25 Fonctionnement de la chaine, le transfert linéaire d’électrons
La chaîne de transport d’électrons est composée de plusieurs complexes protéiques ancrés dans la membrane des thylacoïdes. Dans cette partie, le fonctionnement de la chaîne sera succinctement décrit. Elle est composée de deux photosystèmes (PS), nommés PSI et PSII, associés à des antennes collectrices périphériques, les LHC (Light Harvest Complex). Le cytochrome b6f permet de relier les deux PS via le réservoir de plastoquinones et la
plastocynanine. Deux autres complexes membranaires composent la chaîne : le complexe NDH (NADPH déshydrogénase) et l’ATP synthase (Fig. 3).
Les LHCI et LHCII sont des complexes mobiles s’associant respectivement au PSI et au PSII afin de réguler leurs activités. Ces complexes riches en pigments permettent de capter des photons et de transmettre leur énergie. Il a été montré que le LHCII pouvait se dissocier du PSII et migrer jusqu’au PSI afin d’augmenter son activité et de stopper l’alimentation de la chaîne en électrons par extinction du PSII (cf partie « Transfert cyclique d’électrons »).
Figure 3 : Schéma général de la chaîne de transport d'électrons du chloroplaste
Schéma des différents complexes protéiques. En vert, les antennes collectrices ou LHC (Light harvest
complex). En orange, le PSII. En marron, le cytochrome b6f. En rouge, le PSI. En gris, le complexe
NDH (NADPH déshydrogénase). En bleu, l’ATP synthase. PQ : plastoquinones ; PC : plastocyanine ; Fd : Ferrédoxine.
Les deux photosystèmes sont des complexes protéiques associés à des chlorophylles et caroténoïdes. Ils possèdent chacun un centre réactionnel (P700 dans le PSI et P680 dans le
Page | 26 PSII) composé de deux protéines similaires hébergeant une paire de chlorophylle a dite « spéciale ». Ces chlorophylles du centre réactionnel libèrent un électron (séparation des charges) en recevant l’énergie « excitonique » captée et transmise par les LHC, alimentant ainsi la chaîne redox.
Le PSII est le premier complexe de la chaîne qu’il alimente en électrons. Il s’accumule dans les zones granaires des thylacoïdes. Le centre réactionnel du PSII est composé de deux protéines, D1 et D2 (aussi nommées respectivement PsbA et PsbD, suivant le nom des gènes qui les codent). Au centre est associé une antenne interne (CP47 (PsbB) et CP43 (PsbC)), un cytochrome b559 (PsbE et PsbF) ainsi que le complexe de libération de l’oxygène, l’OEC
(Oxygen Evolving Complex, PSBO, PSBQ et PSBP) (Choquet and Vallon, 2000). Lorsque le P680 reçoit l’énergie excitonique, il libère un électron qui est transporté le long de la chaîne redox interne du PSII, jusqu’à QA une quinone associée à D2. Cet électron est alors transféré à
QB, une quinone du réservoir temporairement associé à D1. Cette dernière quinone est un
transporteur à 2 électrons, qui va ensuite se détacher du PSII et rejoindre le cytochrome b6f,
sous forme protonée (PQH2). Un groupe à 4 atomes de manganèse situé côté lumenal proche
de l’OEC, permet de régénérer le P680 en lui cédant un à un des électrons, via une tyrosine particulière de D1 (nommée YZ). Le complexe à manganèse se régénère à son tour par
l’oxydation de deux molécules d’H2O, libérant alors 4 protons dans le lumen ainsi qu’une
molécule d’O2 (Joliot et al., 1969; Joliot and Kok, 1975).
Le cytochrome b6f, est présent dans les zones granaires et lamellaires. Le cœur de ce
complexe est composé de 4 protéines, les cytochromes b6 et f, la protéine Fe-S de Rieske et la
sous-unité IV (Choquet and Vallon, 2000). La plastoquinone réduite libère ses deux électrons au niveau du site Qo du cytochrome b6, ainsi que ses deux protons dans le lumen. Un des deux
électrons est pris en charge par la protéine de Rieske et le cytochrome f avant d’être transféré sur la plastocyanine, une petite protéine à cuivre. Le deuxième électron peut être transféré de nouveau sur une plastoquinone, au niveau du site Qi du cytochrome b6 proche du stroma où
des protons sont captés pour protoner la quinone. La quinone QH2 ainsi formée au niveau de
Qi sera ensuite prise en charge par Qo. Ce recyclage d’électrons par le cytochrome b6 entre les
sites Qi et Qo est nommé le « cycle Q », il permet de transférer quatre protons du stroma au
lumen pour 4 électrons émis par le PSII.
Le PSI est situé dans les zones inter-granaires des thylacoïdes. Son centre réactionnel est composé des protéines A et B (respectivement PsaA et PsaB) et abrite le P700. Côté stromal, trois protéines (C, D et E) sont associées au centre réactionnel, elles finissent la
Page | 27 chaîne membranaire de transport d’électrons. Lorsque le centre réactionnel P700 est excité il libère un électron dans la chaîne redox interne du PSI, qui est transféré à la protéine C portant deux centres fer-soufre. L’électron est alors transféré à la ferrédoxine, une protéine stromale liée au PSI. La régénération du P700 est assurée par la libération de l’électron de la plastocyanine au niveau de la face lumenale du PSI (Choquet and Vallon, 2000).
Le fonctionnement de la chaîne de transport d’électron ainsi décrite correspond à un transfert linéaire d’électrons, du P680 jusqu’à la ferrédoxine, permettant la production couplée des intermédiaires photosynthétiques.
Production des intermédiaires photosynthétiques
La phase photochimique produit deux intermédiaires photosynthétiques qui seront consommés lors de la phase sombre, le NADPH et l’ATP. Le NADP+ est l’accepteur final d’électrons de la chaîne de transport. La conversion du NADP+
en NADPH est effectuée par la ferrédoxine NADP réductase (FNR) en captant les électrons de la ferrédoxine. L’oxydation de l’eau et le fonctionnement du « cycle Q », crée un gradient de protons au travers de la membrane des thylacoïdes. Cette force protomotrice est utilisée par l’ATP synthase pour produire le second intermédiaire, l’ATP. Ce complexe protéique est composé de deux parties, le CF0 membranaire et le CF1 stromal. Le CF0 est composé de quatre sous-unité (I à IV). Les
sous-unités I, II extra-membranaires et IV intra-membranaire forment un stator qui s’associe avec un anneau de sous-unités III formant le rotor. Cette structure est mise en mouvement grâce au gradient de protons qui sont pris en charge par les sous-unités III et IV pour rejoindre le stroma. Le CF1 composé de cinq sous-unités (α à ε) fonctionne grâce à la rotation du rotor
transmise par la sous-unité γ (Choquet and Vallon, 2000). Le mouvement de cette sous-unité induit des changements de conformation des sous-unités et , permettant la production d’ATP à partir d’ADP et de phosphate inorganique. On considère qu’il faut 3 à 4 protons pour produire un ATP.
Transfert cyclique d’électrons
Il existe un autre mode de transfert d’électrons, le transfert cyclique. Il se caractérise par une production d’ATP découplée de celle du NADPH et d’une absence d’émission d’O2.
Dans ce cas, les électrons captés par la ferrédoxine sont envoyés au cytochrome b6f via le
Page | 28 par les plastoquinones et le cytochrome b6f, permet le fonctionnement du « cycle Q » et donc
le maintien du gradient de protons nécessaire à la synthèse d’ATP (Rochaix, 2011). Les électrons de la ferrédoxine peuvent suivre différentes voies pour rejoindre le réservoir de plastoquinones. Une d’entre elles fait intervenir le complexe NDH (NADPH déshydrogénase). Toutefois, le fonctionnement de ce complexe n’est que très peu compris. Une autre voie fait intervenir deux protéines PGR5 et PGRL1 (PGR5-like protein 1) (Johnson, 2011; Shikanai, 2014).
Le changement de mode de transfert (linéaire vers cyclique et inversement) s’accompagne d’une transition d’état. On parle de transition d’un état I où le LHCII est associé au PSII, favorisant le transfert linéaire, vers un état II dans lequel, après phosphorylation du LHCII, celui-ci est associé au PSI, favorisant le transfert cyclique. Cette transition permet d’adapter la taille des antennes de chaque PS afin d’adapter leurs activités en fonction des radiations perçues et des besoins (Rochaix, 2011). Le transfert cyclique d’électrons permet de découpler la synthèse d’ATP et de NADPH, c’est-à-dire la production d’ATP sans production de NADPH. Ce découplage permet au chloroplaste de pouvoir ajuster le ratio ATP/NADPH produit en fonction des besoins pour la phase sombre de la photosynthèse (Rochaix, 2011; Johnson, 2011).
Phase sombre, fixation du carbone atmosphérique
Cette phase tire son nom du fait qu’elle n’est pas directement dépendante de la lumière pour fonctionner. Durant cette phase, les intermédiaires photosynthétiques sont consommés afin de produire des composés carbonés réduits (glucides) à partir du carbone minéral (CO2).
Cette conversion de carbone se fait dans le stroma par un ensemble de réactions enzymatiques, regroupées sous l’appellation de cycle de Calvin-Benson-Basham.
Rubisco, une enzyme bipolaire
La Rubisco (Ribulose-1,5-bisphosphate carboxylase/oxygénase) est une des enzymes du cycle de Calvin-Benson-Basham. Elle est composée de deux sous-unités, une grande (RbcL) codée par le génome chloroplastique et une petite (RBCS) codées par le génome nucléaire. La Rubisco est formée par l’association de 8 grandes et de 8 petites sous-unités (L8S8) (Bracher et al., 2017). C’est elle qui réalise l’incorporation du CO2 par une réaction de
Page | 29 catalyser une autre réaction, l’oxygénation du RuBP. Le même site actif incorpore de l’O2 au
lieu du CO2, sur le squelette carboné du RuBP. Cette réaction produit du 2-phosphoglycolate,
un composé toxique qui doit être éliminé (cf partie « Photorespiration »). Ces deux fonctions, carboxylase et oxygénase, sont en compétition ce qui tend à diminuer le rendement de la photosynthèse en termes de carbone fixé.
Cycle de Calvin-Benson-Basham
Le cycle de Calvin-Benson-Basham (CBBC) regroupe l’ensemble des réactions biochimiques permettant l’incorporation du CO2 sur du RuBP produisant du
3-phosphoglycérate (3PGA), ainsi que la régénération de l’accepteur de carbones. Le cycle est souvent découpé en 3 grandes étapes : la carboxylation du RuBP, la réduction du 3PGA et la régénération du RuBP (Fig. 4).
La première étape consiste en une seule réaction, l’ajout d’un CO2 sur le RuBP, une
molécule en C5 (i.e. à 5 carbones). Cette carboxylation produit un intermédiaire en C6 instable qui se clive en deux molécules en C3, le 3PGA. La deuxième étape du cycle est la réduction du 3PGA en G3P (glycéraldéhyde-3-phosphate). Cela requiert deux réactions, une phosphorylation du 3PGA consommant de l’ATP ; suivie de la réduction du produit phosphorylé en consommant du NADPH. Le G3P a plusieurs devenirs, une partie sera utilisée comme « brique de base » pour produire un glucide en C6 (glucose), stocké sous forme de polymère en amidon ; une autre partie sera utilisée pour finir le cycle en régénérant le RuBP.
La dernière étape du CBBC comprend plusieurs réactions consistant en une série d’associations, séparations et réarrangements moléculaires dans le but de produire du RuBP. De façon schématique et simplifiée, l’association de deux G3P permet la production de fructose-6-phosphate (F6P). Le F6P réagit avec du G3P, formant du xylulose et de l’érythrose. La réaction de ce dernier avec du G3P forme un composé en C7, réagissant avec un autre G3P ce qui produit du ribose et du xylulose. L’action d’une isomérase et d’une épimérase permet la conversion de ces derniers composés en ribulose-5-phosphate (RuP). Enfin le RuBP est régénéré par la phosphorylation du RuP en consommant un autre ATP (Fig. 4) (Michelet et al., 2013). Ainsi la fixation d’un CO2 requiert la consommation de 3 ATP et
Page | 30
Figure 4 : Cycle de Calvin-Benson-Basham
Schéma du cycle de Calvin-Benson-Basham, découpé en 3 étapes : carboxylation du RuBP (vert), la réduction du 3-PGA (violet) et la régénération du RuBP (jaune). En rouge, le sixième G3P produit par le cycle, pouvant être utilisé pour produire de l’amidon. Les enzymes sont représentées par des ovales. Abréviations : Enzymes : Rubisco (ribulose-1,5-bisphosphate Carboxylase/Oxygénase) ; PGK (phosphoglycérate kinase) ; GAPDH (glyceraldéhyde-3-phosphate déshydrogénase) ; TPI (triose phosphate isomérase) ; FBA bisphosphate aldolase) ; FBPase (fructose-1,6-bisphosphatase) ; SBPase (sédoheptulose-1,7-(fructose-1,6-bisphosphatase) ; RPE (ribulose-5-phosphate 3-épimérase) ; RPI (ribose-5-phosphate isomérase) ; PRK (phosphoribulokinase). Métabolites : RuBP (ribulose-1,5-bisphosphate) ; 3-PGA (3-phosphoglycérate) ; 1,3-PGA (1,3-bisphosphoglycérate) ; G3P (glycéraldéhyde-3-phosphate) ; DHAP (dihydroxyacétone phosphate) ; F1,6P (fructose-1,6-bisphosphate) ; F6P (fructose-6-phosphate); X5P (xylulose-5-phosphate) ; E4P (érythrose-4-phosphate) ; S1,7P (sédoheptulose-1,7-bis(érythrose-4-phosphate) ; S7P (sédoheptulose-7-(érythrose-4-phosphate) ; R5P (ribose-5-phosphate) ; RuP (ribulose-5-phosphate). Adaptée de Michelet et al (2013).
Bien que le mécanisme soit globalement cyclique, on ne peut pas vraiment considérer le CBBC comme une succession de réactions en chaîne. En effet, l’image de « cycle » renvoie l’idée que l’entrée d’un input (ici le CO2) dans le cycle permet d’effectuer un tour complet de
celui-ci. Comme la régénération de 3 RuBP nécessite 5 G3P (i.e. 1,6 G3P par RuBP), le CBBC ne peut avoir un tel fonctionnement. Ainsi le CBBC fonctionne plutôt par multiples de trois, l’assimilation de 3 CO2 produit six G3P, dont 5 sont utilisés pour la troisième étape et le
Page | 31 sixième est incorporé dans le métabolisme carboné du chloroplaste et permet par exemple le stockage sous forme d’amidon.
La Photorespiration
L’activité oxygénase de la Rubisco peut également incorporer un O2 sur le RuBP (au
lieu d’un CO2), celui-ci va se scinder en un 3PGA et un 2PG (2-phosphoglycolate). Le 2PG, toxique pour la cellule doit être éliminé. Pour cela, il est converti en 3PGA via une voie métabolique impliquant le chloroplaste, le peroxysome et la mitochondrie, la voie du glycolate (Fig. 5).
Figure 5 : Photorespiration
Schéma de la voie du glycolate. Flèche en bleu, réaction de carboxylation du RuBP. Flèches en rouge, réaction d’oxygénation du RuBP, produisant le 2PG. Abréviations : Enzymes : GLYK (D-glycérate 3-kinase) ; PGP (2PG phosphatase) ; GOX (glycolate oxydase) ; CAT (catalase) ; HPR1/HPR2 (hydroxypyruvate réductase) ; GGT (glutamate:glyoxylate aminotransférase) ; GDC (glycine décarboxylase) ; SHM (serine transhydrométhyltransférase) ; AGT1 (glyoxylate aminotransférase). Métabolites : RuBP (ribulose-1,5-bisphosphate) ; 3PGA (3-phosphoglycérate) ; 2PG (2-phosphoglycolate) ; 2OG (2-oxo-glutarate) ; THF (tétrahydrofolate) ; CH2-THF (méthylène-tétrahydrofolate). Adaptée de Orf et al (2016).
Page | 32 Schématiquement, le 2PG est exporté vers le peroxysome où il est oxydé puis aminé en glycine. L’étape d’oxydation produit du peroxyde d’hydrogène (H2O2). La glycine est
transférée dans la mitochondrie. Dans cet organite, 2 molécules de glycine seront utilisées pour produire une sérine. Pour cela, une des deux glycines est désaminée et décarboxylée, libérant un CO2 et un NH3. L’ammonium produit sera réassimilé dans la voie de synthèse de
la glutamine du chloroplaste (Bauwe et al., 2010). De nouveau dans le peroxysome, la serine est désaminée puis réduite en glycérate. Enfin, le glycérate sera phosphorylé en 3PGA dans le chloroplaste, et peut entrer dans le CBBC (Bauwe et al., 2010; Orf et al., 2016).
Cette voie permet de convertir le 2PG issue de l’oxygénation du RuBP en 3PGA. Toutefois, elle reste extrêmement couteuse pour la cellule. En effet, pour deux O2 incorporés
par la Rubisco, deux 3PGA sont formés directement ainsi que deux 2PG. Ils sont alors convertis en un troisième 3PGA, en consommant un ATP et un O2 et en produisant un CO2.
La photorespiration constitue donc une perte de carbone pour la cellule, pour 10 carbones engagés (2 RuBP) seulement 9 (trois 3PGA) rentrent dans le CBBC, ce qui permet de ne régénérer que 1,8 RuBP. Néanmoins, cette voie est primordiale lorsque les stomates sont fermés. Dans ce cas, le taux d’O2 augmente, elle permet donc de consommer de l’oxygène et
de produire du CO2. Cette voie inverse ainsi le ratio O2/CO2, et permet de maintenir un CBBC
fonctionnel et d’éliminer l’excès énergétique de la chaîne de transfert d’électrons. Sans photorespiration, l’accumulation d’O2 conduirait à une production excessive d’espèces
réactive de l’oxygène (ROS), à la destruction du chloroplaste et à la mort cellulaire programmée de la cellule hôte.
Photosynthèse en C4 : spécialisation chloroplastique
Le fonctionnement de la chaîne de transport d’électrons et de l’assimilation du CO2
décrit précédemment correspond au cas des plantes effectuant une photosynthèse dite « en C3 » comme A. thaliana. Toutefois, certaines plantes réalisent une photosynthèse dont le fonctionnement varie légèrement. Par exemple, le maïs (Zea mays) ou le sorgho réalisent une photosynthèse dite « en C4 », alors que d’autres plantes, comme les cactées, ont un métabolisme CAM (Crassulacean acid metabolism). Une partie des travaux de ma thèse ayant été effectués sur le maïs, le mécanisme de la photosynthèse en C4 sera décrit succinctement.
Page | 33 Différenciation morphologique des chloroplastes chez les plantes en C4 Dans le cas de la photosynthèse en C3, toutes les réactions se passent au sein du même chloroplaste. A l’inverse, chez les plantes effectuant une photosynthèse en C4, on peut distinguer deux types de chloroplastes dans deux tissus des feuilles : le mésophylle (MS) et la gaine péri-vasculaire (BS). La mise en place de ces tissus au cours du développement foliaire s’accompagne en effet de la différenciation des chloroplastes notamment sur l’aspect de leur réseau de thylacoïdes. Les chloroplastes du mésophylle présentent de très nombreux et épais grana, alors que les chloroplastes de la gaine ne présentent que très peu de grana composés d’un faible nombre de couches (Fig. 6) (Majeran et al., 2010). Le réseau de thylacoïdes étant différent, la composition de la chaîne de transport d’électron est également différente. En effet, les chloroplastes de la gaine (pauvres en grana), ne contiennent que très peu de PSII mais contiennent beaucoup plus de PSI et de complexe NDH, par comparaison à ceux du mésophylle (riches en grana) (Majeran et al., 2008; Majeran and van Wijk, 2009). L’absence de PSII dans la gaine induit une très faible émission d’O2 et un transport d’électrons
principalement cyclique. Au contraire, dans le mésophylle, la chaine de transport étant complète, les deux types de transport d’électrons sont actifs (linéaire et cyclique). Cette différenciation MS/BS n’impacte pas que les complexes de transport d’électrons, les enzymes du CBBC sont aussi différentiellement accumulées. En effet, la Rubisco est principalement présente dans la gaine, alors que la phosphoglycérate kinase (PGK), phosphorylant le 3PGA dans la deuxième étape du CBBC, s’accumule préférentiellement dans le mésophylle (Majeran et al., 2005).
Figure 6 : Observations microscopiques de chloroplaste de Z. mays
Observation de l’extrémité de feuille de maïs à 9 jours, b : gaine péri-vasculaire ; m : mésophylle. (A) coupe de feuille en microscopie optique, barre = 25µm. (B) chloroplastes de BS et MS en microscopie électronique à transmission, barre = 1µm. Adaptée de Majeran et al (2010).
Page | 34 Fixation du carbone par les plantes en C4, cas du maïs
Chez A. thaliana (plante en C3), le CO2 atmosphérique est directement incorporé dans
le CBBC par la Rubisco, formant une molécule à trois carbones. Chez le maïs (plante en C4), en revanche, le CO2 est fixé deux fois, une première dans le mésophylle, puis une seconde
fois dans la gaine. Cette fixation en deux étapes fait intervenir une navette entre les deux types de chloroplastes, la navette malate – pyruvate (Fig. 7). La carboxylation primaire prend ainsi place dans le cytosol des cellules du mésophylle et consiste en l’incorporation du CO2
atmosphérique sur du phosphoénol pyruvate, formant un composé à quatre carbones, l’oxaloacétate (OAA). Cet OAA est réduit en malate dans le chloroplaste du mésophylle, avant d’être transporté dans les chloroplastes de la gaine. L’enzyme malique des chloroplastes de la gaine décarboxyle le malate en pyruvate, ce qui libère du CO2 avec une production
associée de NADPH. Le pyruvate est phosphorylé en PEP dans le chloroplaste du mésophylle, bouclant ainsi la navette. La carboxylation secondaire a lieu dans le chloroplaste de la gaine et est effectuée par la Rubisco qui fixe un CO2 sur du RuBP et produit du 3PGA. Le CBBC se
déroule de la même façon que décrite précédemment. Toutefois, la deuxième étape du cycle (réduction du 3PGA) a lieu dans le mésophylle, les deux autres étapes (carboxylation et régénération du RuBP) ont lieu dans la gaine.
Page | 35
Figure 7 : Fixation du carbone chez les plantes en C4
Schéma du mécanisme d’assimilation du CO2 chez les C4, cas du maïs. En haut, une cellule du MS.
En bas, une cellule du BS. Les enzymes sont dans des boîtes colorées. En rouge, les protéines enrichies dans le BS. En bleu, celles enrichies dans le MS. Adaptée de Majeran et Van Wijk (2009).
Conséquences de la photosynthèse en C4
Ce type de photosynthèse en C4 est apparue plusieurs fois indépendamment au cours de l’évolution lors de la décroissance du taux de CO2 atmosphérique, il y a environ 25
millions d’années (Sage et al., 2012). Il existe donc des variantes de la photosynthèse en C4 présentée ci-dessus. On peut noter par exemple qu’il existe d’autres plantes, comme les amarantes, effectuant une photosynthèse en C4 mais impliquant l’enzyme malique mitochondriale. Il existe également de nombreuses plantes présentant une photosynthèse intermédiaire entre C3 et C4.
L’absence de PSII dans la gaine et l’action de la navette malate – pyruvate permettent de créer un environnement enrichi en CO2 dans les chloroplastes de la gaine. Ainsi, chez les
plantes en C4, la photorespiration est extrêmement réduite (mais existe néanmoins) ce qui améliore le rendement photosynthétique (en termes de fixation de carbone) (Majeran and van Wijk, 2009; Sage et al., 2012). Toutefois, cette concentration n’est pas sans coût. La régénération du PEP par la pyruvate phosphate dikinase (PPDK) consomme 2 ATP (la phosphorylation produit un AMP, régénéré en ADP par un second ATP), augmentant ainsi le coût énergétique de la fixation de carbone.
Page | 36
Le génome plastidial, un chromosome réduit au sein du nucléoïde lié au
génome nucléaire
Les plastes ont la particularité, comme la mitochondrie, de posséder un génome. Ce génome dérive de celui de la cyanobactérie capturée lors de l’endosymbiose et s’est réduit au cours du temps. Il en résulte que la cellule végétale contient trois génomes. Dans cette partie, nous allons aborder la réduction du génome plastidial par transfert de gènes et la notion de complémentarité entre les génomes nucléaire et cytoplasmique. Le génome plastidial ainsi que le complexe le contenant, le nucléoïde, seront ensuite développés.
Transfert de gènes endosymbiotiques et ses conséquences
Au cours de l’évolution, les endosymbiotes ont été « domestiqués » et sont devenus des organites à génome, les plastes et les mitochondries. Une des conséquences de cette domestication est la réduction du génome de l’endosymbiote. En effet, on observe une diminution de la taille du chromosome et du nombre de gènes. Par exemple, le génome de la cyanobactérie Synechocystis sp. PCC6803 comprend environ 3573kb et code pour environ 3200 protéines (Kaneko and Tabata, 1997) alors que le génome du chloroplaste d’Arabidopsis thaliana a une taille d’environ 154kb et porte environ 130 gènes (Sato et al., 1999) (cf partie « Description du bagage génétique du plastome d’A. thaliana »). La réduction du nombre de gènes et de la taille du génome de l’endosymbiote s’accompagne de transferts de gènes. En effet, de nombreux gènes sont transférés de l’endosymbiote vers le noyau, où ils seront adaptés à une expression par la machinerie nucléaire. Ces transferts ont permis de domestiquer l’endosymbiote, en le rendant dépendant du noyau. Ils ont aussi forgé le génome nucléaire des eucaryotes, de nombreux fragments d’ADN originaire des organites sont en effet observés dans le génome nucléaire de levures, d’humains et de plantes (Timmis et al., 2004). L’analyse du génome nucléaire d’A. thaliana montre la présence de 11kb d’ADN d’origine chloroplastique (ADN Pt) et de 627kb d’ADN d’origine mitochondriale (ADN Mt) répartis respectivement sur 17 et 14 insertions (The Arabidopsis Genome Initiative, 2000). Il est à noter qu’une des 14 insertions d’ADN Mt, localisée à proximité du centromère sur le bras court du chromosome 2, a une taille de 620kb et contient l’ensemble du chromosome mitochondrial (Lin et al., 1999; Stupar et al., 2001). On ne sait toutefois pas si ces copies sont exprimées. En plus de ces copies présentes à la fois dans le noyau et l’organite, certains gènes
Page | 37 n’existent plus que dans le noyau. C’est le cas par exemple de RBCS, gène codant pour la petite sous-unité de la Rubisco.
Suite à ces transferts de gènes, certaines capacités métaboliques de l’endosymbiote se trouvent altérées. Par exemple, la perte du gène RbcS du génome de l’endosymbiote l’empêche de fixer du carbone, du moins tant que cette protéine est absente dans l’endosymbiote. Par conséquent, il faut « complémenter » le bagage protéique du plaste. Pour cela, les protéines codées par ces gènes transférés sont alors traduites dans le cytoplasme et importées dans l’organite. Ces protéines d’origine procaryotique ne sont toutefois pas les seules à être adressées à l’organite. Le noyau code et adresse également des protéines, d’origine eucaryotique, s’assurant de la bonne coordination entre les différents compartiments subcellulaires. Il est estimé qu’environ 3000 protéines sont importées dans le chloroplaste, soit environ 95% du protéome chloroplastique (en nombre de protéines différentes) (Soll and Schleiff, 2004; Cline and Dabney-Smith, 2008). Cet import requiert un mécanisme de transport de protéines au travers des deux membranes de l’enveloppe des organites. Cette fonction est assurée par les complexes protéiques TIC (translocon at the inner envelope membrane of chloroplasts) et TOC (translocon at the outer envelope membrane of chloroplasts) (Soll and Schleiff, 2004). L’import dans la mitochondrie est réalisé par des complexes similaires : TIM et TOM (Truscott et al., 2003). Comme vu précédemment, le chloroplaste comporte différents sous-compartiments : enveloppe externe, espace inter membranaire, enveloppe interne, stroma, membrane du thylakoïde ou lumen. Une signalisation est donc requise afin que les protéines soient correctement adressées. Le peptide d’adressage situé en N-terminal de la protéine remplit généralement ce rôle, il permet sa reconnaissance par le complexe TOC pour son import. Ce peptide est ensuite clivé par la peptidase SPP (stroma processing peptidase). Les protéines adressées aux thylakoïdes ou au lumen, possèdent un peptide supplémentaire permettant la prise en charge par différents complexes d’import au niveau de la membrane du thylakoïde (Shi and Theg, 2013; Cline and Dabney-Smith, 2008).
La cellule végétale : un système à trois génomes complémentaires
Malgré la réduction de la taille du génome des endosymbiotes, les plastes et mitochondries possèdent toujours un génome. Ainsi les cellules des angiospermes possèdent 3 génomes distincts. Ces trois génomes cohabitent et co-évoluent depuis plus d’un milliard d’années. On peut constater que certains transferts de gènes sont spécifiques de certaines
Page | 38 espèces. Par exemple, le gène chloroplastique InfA qui code pour un facteur d’initiation de la traduction est présent dans le génome chloroplastique de nombreuses angiospermes dont Zea mays. Toutefois, chez A. thaliana et d’autres angiospermes, il a été transféré dans le noyau, où le gène est exprimé et la protéine adressée au chloroplaste (Millen et al., 2007). En plus des transferts, des mutations sont apparues et ont fait évoluer ces génomes. De cette cohabitation résulte une forme de complémentarité entre le génome nucléaire et celui des organites, et donc la notion de compatibilité nucléo-cytoplasmique. En effet, le développement et le fonctionnement de la cellule ne peut se faire correctement que si l’union des produits de l’expression de ces trois génomes est « complète ». On définit le génotype comme étant l’information génétique issue du noyau, et le plasmotype comme étant l’information génétique contenue dans le cytoplasme, donc issue des chloroplastes et des mitochondries. Le cytoplasme est généralement un héritage maternel. Ainsi, suite à un croisement, il est possible de générer un conflit nucléo-cytoplasmique si le génotype et le plasmotype sont incompatibles. L’incompatibilité peut-être dû aussi bien au chloroplaste qu’à la mitochondrie (Encadré n°2).
Encadré n°2 : Incompatibilité nucléo-cytoplasmique
Incompatibilités liées aux chloroplastes
Les incompatibilités entre génome chloroplastique et nucléaire aboutissent généralement à des défauts photosynthétiques et ont été très étudié chez le genre Oenothera (Greiner and Bock, 2013). En effet, il existe dans ce genre trois génotypes stables à l’état homo ou hétérozygote, soit 6 génotypes possibles. On distingue 5 types de génome chloroplastique (plastome). Parmi les 30 combinaisons génome-plastome, seulement 12 sont compatibles (c’est-à-dire conduisant à des descendants verts et photosynthétiques), alors qu’au moins 6 sont létales et les autres produisent des phénotypes intermédiaires (plantes vert-jaunes ou albinos) (Stubbe, 1989). Également, le cybride (hybride cytoplasmique) contenant le noyau d’Atropa belladona et les chloroplastes de Nicotiana tabacum est albinos et ne peut pousser qu’en présence de sucre in vitro ; alors que le cybride réciproque (noyau de N. tabacum et chloroplaste de A. belladona) ne présente aucun phénotype particulier. L’analyse du cybride albinos montre un défaut d’édition d’un site spécifique du tabac sur le transcrit atpA (Schmitz-Linneweber, Kushnir, et al., 2005).
Page | 39 Incompatibilités liées à la mitochondrie
Concernant la mitochondrie, l’exemple d’incompatibilité la mieux connue est la stérilité mâle cytoplasmique (CMS). La CMS peut être décrite comme l’incapacité pour la plante de produire un pollen fonctionnel suite à une incompatibilité entre le génome nucléaire et mitochondrial. Généralement, et de façon schématique, le produit d’un gène toxique mitochondrial est bloqué par une protéine codée par le génome nucléaire, nommée facteur de restauration (Rf). Selon les cas, le blocage peut être au niveau ARN ou au niveau protéique (Chen and Liu, 2014). Par exemple, la CMS-WA du riz (Oryza sativa ssp. indica) est causée par un gène mitochondrial codant pour la protéine WA352. Deux protéines Rf, peuvent contrecarrer cette CMS : Rf4 peut agit au niveau du transcrit wa352, tandis que Rf3 agit sur la protéine WA352 (Luo et al., 2013). En absence de gène Rf, l’action de la protéine mitochondriale empêche la production d’un grain de pollen fonctionnel. La CMS résulte donc d’une incompatibilité entre les génomes nucléaire et mitochondrial. Un autre exemple est celui du croisement entre les écotypes Shahdara (Sha) et Monte-Rosso (Mr-0) d’A. thaliana qui révèle une CMS lorsque l’écotype Sha est utilisé comme receveur de pollen, c’est-à-dire quand la descendance possède les mitochondries de l’écotype Sha (Gobron et al., 2013).
Double origine génétique de la chaine photosynthétique
La chaine de transport d’électrons, est composée de nombreuses protéines. Le PSII est composé de plus de 20 protéines différentes et le PSI de plus de 10 protéines. Or le plastome d’Arabidopsis thaliana ne compte que 15 gènes Psb (PSII) et 5 gènes Psa (PSI) (Annexe S2). Parmi les protéines codées par le génome nucléaire et importées dans le chloroplaste, on trouve de nombreuses sous-unités des complexes photosynthétiques. Les quatre complexes majeurs, le PSI, PSII, cytochrome b6f et l’ATP synthase, sont composés de protéines codées
par ces deux génomes (Choquet and Vallon, 2000). A l’inverse, l’ensemble des protéines composant les LHCI/II sont codées par le noyau. Toutefois, on peut remarquer que les protéines des centres réactionnels des deux photosystèmes, D1 et D2 (respectivement PsbA et PsbD) pour le PSII ainsi que PsaA et PsaB pour le PSI, sont toutes codées par le génome chloroplastique (Fig. 8).
Page | 40 Les gènes codés par le génome plastidial sont donc cruciaux pour la mise en place de l’appareillage photosynthétique. Nous allons donc décrire le génome plastidial et plus particulièrement celui de l’espèce modèle A. thaliana.
Figure 8 : Origine génétique des protéines des complexes photosynthétiques
Les protéines sont colorées en fonction du complexe et de leur origine génétique. En vert, les LHC ; en orange : PSII ; en marron : cytochrome b6f ; en rouge : PSI ; en bleu : ATP synthase. En clair, les
protéines codées par le plastome (exemple D1). En sombre, celles codées par le génome nucléaire (exemple PSBO). Adaptée de Choquet et Vallon (2000).
Organisation du génome plastidial
Le Plastome, un petit chromosome avec une structure conservée
Le génome plastidial ou plastome, n’est composé que d’un seul chromosome, localisé dans le stroma du chloroplaste. Ce chromosome, généralement représenté sous forme circulaire, est en réalité majoritairement sous forme linéaire in vivo (~70% de chromosome linéaire) (Bendich, 2004). Le plastome des plantes terrestres est relativement petit, avec une taille généralement comprise entre 120 et 160 kb et peu de variations autour de cette plage de taille. En effet, le plus petit génome chloroplastique a une taille de 107kb (Cathaya argyrophylla) et le plus grand est à peine deux fois plus long avec 217kb (Pelargonium). Le chromosome chloroplastique porte un peu plus d’une centaine de gènes codant pour des protéines, des ARNr et des ARNt (Bock, 2007; Daniell et al., 2016).
Page | 41 Comme sa taille et son nombre de gènes, le plastome présente également une organisation conservée. On distingue 4 régions le long du chromosome : deux régions dont les séquences sont identiques mais en sens inverse, IRA et IRB (inverted repeat regions A and B), séparées par deux régions de tailles différentes présentes en une seule copie, SSC et LSC (short and large single-copy regions). Bien que cette structure soit conservée, les extrémités des régions inversées-répétées varient d’une espèce à l’autre (Bock, 2007; Jansen and Ruhlman, 2012). Par exemple, chez A. thaliana, les gènes Ycf1 et Rpl2 bordent les IR, alors que chez Z. mays, ce sont les gènes Rps15 et Rps19. De plus, la synténie n’est pas toujours identique. Par exemple, chez A. thaliana, Rps15 se trouve dans le SSC entre NdhH et Ycf1 et non dans l’IR comme chez le maïs. Également, le groupe de gènes de AtpA à PsbM précède le groupe de PsbD à PsbZ chez A. thaliana alors qu’il est inversé et situé après chez Z. mays (Fig. 9 et Annexe S1).
Description du bagage génétique du plastome d’A. thaliana
Dans cette partie, nous allons décrire plus en détails l’ensemble des gènes portés par le plastome d’A. thaliana pour deux raisons principales, 1) il est à présent très bien annoté et 2) une partie conséquente des travaux effectués durant cette thèse l’a été sur cette espèce modèle. Séquencé en 1999, le plastome de l’écotype Columbia (Col0) d’A. thaliana contient 154 478 pb (LSC : 84 170pb ; SSC : 17 780pb ; IR : 26 264pb) et 132 gènes (Sato et al., 1999). La région IRA s’étend de la base 84 172 à la base 110 434, l’IRB correspond à la région entre la base 128 216 et la base 154 478, c’est-à-dire la « fin » du chromosome (Fig. 9). Le nombre de gènes mérite d’être nuancé. En effet, l’annotation TAIR10 propose 133 gènes chloroplastiques mais distingue les gènes de chaque IR, les comptant donc deux fois. En réalité, en supprimant les doublons, on dénombre 114 gènes, sans prendre en compte l’IRB (de la base 128 216 à 154 478). Sans être réellement regroupés en opérons, les gènes sont souvent regroupés physiquement par fonction biologique ; on peut dénombrer 4 grandes catégories : 1) la chaine photosynthétique, comprenant les gènes Psa, Psb, Pet, Ndh et Atp ; 2) la transcription, gènes Rpo ; 3) la traduction, avec les gènes Rpl, Rps, ARNr et ARNt ; 4) les autres, gènes Ycf, RbcL, ClpP, MatK … L’annexe S2 donne la liste complète des gènes du chloroplaste d’A. thaliana classés par catégories, en indiquant ceux ayant des introns et/ou localisés dans les IR.
Page | 42
Figure 9 : Carte du plastome d'A. thaliana écotype Columbia (Col0)
Représentation graphique du génome chloroplastique d’A. thaliana écotype Col0 (identifiant GenBank : AP000423.1). Les gènes sont colorés par rapport à leur fonction. Les gènes vers l’intérieur sont codés par le brin -, ceux vers l’extérieur sont codés par le brin +. IRA/IRB (inverted repeat
regions A and B). SSC/LSC (short and large single-copy regions). Carte créée avec le logiciel
OGDraw (Lohse et al., 2007).
Encadré n°3 : Similarité du génome mitochondrial avec le plastome
La mitochondrie est l’organite résultant de l’endosymbiose d’une α-protéobactérie, il y a environ 2 milliards d’années (Dyall et al., 2004). Comme le plaste, cet organite a subi une réduction chromosomique. Le chromosome mitochondrial d’A. thaliana écotype C24 a été séquencé en 1997 et a une taille d’environ 367kb (Unseld et al., 1997). Plus récemment, celui de l’écotype Col0 a aussi été séquencé et annoté (Sloan et al., 2018). Ce génome comporte