HAL Id: hal-02799722
https://hal.inrae.fr/hal-02799722
Submitted on 5 Jun 2020
HAL is a multi-disciplinary open access archive for the deposit and dissemination of sci-entific research documents, whether they are pub-lished or not. The documents may come from teaching and research institutions in France or abroad, or from public or private research centers.
L’archive ouverte pluridisciplinaire HAL, est destinée au dépôt et à la diffusion de documents scientifiques de niveau recherche, publiés ou non, émanant des établissements d’enseignement et de recherche français ou étrangers, des laboratoires publics ou privés.
Facteurs cellulaires de la plante impliqués dans la
transmission du cauliflower mosaic virus
Marie Ducousso
To cite this version:
Marie Ducousso. Facteurs cellulaires de la plante impliqués dans la transmission du cauliflower mosaic virus. Biologie végétale. 2015. �hal-02799722�
MINISTÈRE DE L'ENSEIGNEMENT SUPÉRIEUR ET DE LA RECHERCHE
ÉCOLE PRATIQUE DES HAUTES ÉTUDES
Science de la Vie et de la Terre
MÉMOIRE
présenté
par
Marie DUCOUSSO
pour l'obtention du diplôme de l'École Pratique des Hautes Études
FACTEURS CELLULAIRES DE LA PLANTE IMPLIQUÉS DANS LA
TRANSMISSION DU CAULIFLOWER MOSAIC VIRUS
soutenu le 17 Décembre 2015
devant le jury suivant :
Mireille ROSSEL – Président
Martin DRUCKER – Tuteur scientifique
Thierry DUPRESSOIR – Tuteur pédagogique
Véronique BRAULT – Rapporteur
Cécile DESBIEZ – Examinateur
Mémoire préparé sous la direction de :
Martin DRUCKER
Directrice : Claire NEEMA
UMR Biologie et Génétique des Interaction Plante-Parasite (BGPI)
INRA-CIRAD-Supagro
Campus International de Baillarguet
34398 Montpellier Cedex 5
et de
Thierry DUPRESSOIR
Directrice : Anne-Nathalie VOLKOFF
UMR Diversité Génomes et Interactions Micro-organismes/Insecte
Université Montpellier 2 - Place Eugène Bataillon - CC 101
MINISTÈRE DE L'ENSEIGNEMENT SUPÉRIEUR ET DE LA RECHERCHE
ÉCOLE PRATIQUE DES HAUTES ÉTUDES
Science de la Vie et de la Terre
MÉMOIRE
présenté
par
Marie DUCOUSSO
pour l'obtention du diplôme de l'École Pratique des Hautes Études
FACTEURS CELLULAIRES DE LA PLANTE IMPLIQUÉS DANS LA
TRANSMISSION DU CAULIFLOWER MOSAIC VIRUS
soutenu le 17 Décembre 2015
devant le jury suivant :
Mireille ROSSEL – Président
Martin DRUCKER – Tuteur scientifique
Thierry DUPRESSOIR – Tuteur pédagogique
Véronique BRAULT – Rapporteur
Cécile DESBIEZ – Examinateur
Mémoire préparé sous la direction de :
Martin DRUCKER
Directrice : Claire NEEMA
UMR Biologie et Génétique des Interaction Plante-Parasite (BGPI)
INRA-CIRAD-Supagro
Campus International de Baillarguet
34398 Montpellier Cedex 5
et de
Thierry DUPRESSOIR
Directrice : Anne-Nathalie VOLKOFF
UMR Diversité Génomes et Interactions Micro-organismes/Insecte
Université Montpellier 2 - Place Eugène Bataillon - CC 101
REMERCIEMENTS
Je souhaite remercier dans un premier temps, Claire Neema pour l'accueil au sein de l'UMR BGPI ainsi que Stéphane Blanc pour l'accueil au sein de l'équipe VIP.
Un grand merci à Martin Drucker, tuteur scientifique, pour m'avoir suivie pendant 2 ans et pour sa disponibilité. Je remercie également, Thierry Dupressoir, mon tuteur pédagogique, pour m'avoir poussée à obtenir ce diplôme et m'avoir encouragée. Merci à vous 2 d’avoir corrigé ce mémoire. Je tiens à remercier tout particulièrement l'équipe VIP pour leur gentillesse et la bonne humeur qui régnait. Un énorme merci à Martine Granier et Marilyne Uzest pour leur précieuse aide dans l'organisation et le fonctionnement du L64 et L69 qui n'est pas une mince affaire. Un immense merci à Jean-Luc Macia et Sophie Le Blaye pour leur travail en serre, leur soutien et nos discussions sympathiques. Merci à Jean-Louis Zeddam, Michel Yvon et Craig Webster pour avoir été de supers colocataires de laboratoire ainsi que « Monsieur » Daniel Gargani pour m'avoir écoutée et initiée à la microscopie électronique.
Je remercie Loup Rimbaud et Gaël Thébaud pour m'avoir prodiguée de précieux conseils pour les analyses statistiques ainsi que Marie-Stef Vernerey pour ses discussions constructives et ses connaissances en microscopie.
Pour finir, je tiens à remercier Franz Durandet et mes parents pour leur soutien et leurs remarques judicieuses ainsi que Dominique Durandet pour avoir corrigé ce mémoire.
TABLE DES MATIÈRES
REMERCIEMENTS...3
LISTE DES ABRÉVIATIONS...7
LISTE DES FIGURES ET TABLES...11
1. Figures...11
2. Tableaux...12
INTRODUCTION...13
1. Présentation du Cauliflower mosaic virus...16
1.1.Composition du génome viral et de ses protéines...16
1.1.1.Protéine P1 ou protéine de mouvement (MP)...17
1.1.2.Protéine P2 ou facteur assistant de la transmission (FAT)...17
1.1.3.Protéine P3 ou protéine associée au virion (VAP)...18
1.1.4.Protéine P4 ou protéine de capside (CP)...18
1.1.5.Protéine P5 ou reverse transcriptase (RT)...19
1.1.6.Protéine P6 ou transactivator/viroplasmin (TAV)...19
1.1.7.Protéine P7...20
1.2.Cycle infectieux du CaMV...21
2. Transmission des virus de plante...23
2.1.Généralités sur la transmission...23
2.1.1.Transmission persistante...24
2.1.2.Transmission non persistante et semi-persistante...24
2.2.Transmission du CaMV...25
2.2.1.Complexe transmissible et corps à transmission...25
2.2.2.Mécanismes d'activation du corps à transmission...27
3. Projet de recherche...30
RÉSULTATS...32
PARTIE 1 :...33
Recherche de partenaires du Corps à Transmission (CT) par purification en
gradient discontinu...33
1. Mise au point de la purification des CT-TC...35
2. Conclusion...36
PARTIE 2 : ...37
Expression de la protéine P2::GFP dans A. thaliana...37
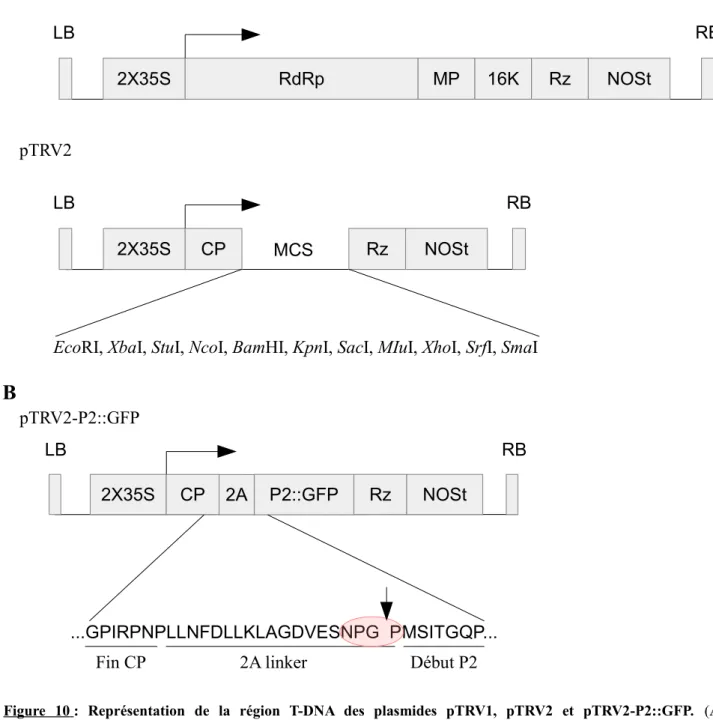
1. Constructions des vecteurs TRV pour agroinoculation...39
2. Expression de la protéine P2::GFP dans A. thaliana...40
3. Conclusion...42
PARTIE 3 : ...43
Approche génomique pour identifier des partenaires cellulaires interagissant
avec la protéine P2 directement in planta...43
1. Construction de la banque d'ADNc d'A. thaliana dans pCK-DUO et
caractérisation...46
2. Validation de la technique Duolink® en utilisant l'interaction connue entre la
protéine P2 et la tubuline...47
3. Optimisation du système de criblage entre la banque d'ADNc d'A. thaliana et
la protéine P2...49
3.1.Transfection de la banque dans des protoplastes de navet B-JI...49
3.2.Test de la technique Duolink® avec la banque d'ADNc et la protéine P2...49
3.3.Duolink® sur des sous-pools de banque d'ADNc contre la protéine P2...51
4. Conclusion et perspectives...53
DISCUSSION...56
MATÉRIEL & MÉTHODES...60
1. Plantes, virus et inoculations...61
1.1.Virus...61
1.2.Plantes...61
1.3.Inoculation mécanique d'A. thaliana et B. rapa avec le CaMV et le TuMV...61
2. Préparation des protoplastes et transfection...63
2.1.Préparation des protoplastes de navet...63
2.2.Transfection transitoire des protoplastes de navet...64
3. Préparation du TRV et agroinoculation...65
3.1.Préparation du TRV...65
3.2.Agroinoculation des A. thaliana Col-0...66
4. Méthodes d'analyse des protéines...67
4.1.Anticorps...67
4.2.Électrophorèse en condition dénaturante : SDS-PAGE...67
4.3.Coloration des gels SDS-PAGE en bleu de Coomassie...67
4.4.Western Blot...68
4.5.Purification des Corps à transmission (CT)...69
5. Immunofluorescences...70
5.1.Immunofluorescence (ou Immunomarquage) indirecte...70
5.2.Proximity Ligation Assay : Duolink®...71
6. Microscopies...72
6.1.Microscope à épifluorescence...72
6.2.Microscope confocal...72
6.3.Loupe binoculaire à fluorescence...72
7. Méthodes d'analyses des acides nucléiques...73
7.1.Extraction de l'ADN plasmidique...73
7.2.Extraction d'ARN totaux à partir de tissus végétaux...74
7.3.Amplification de l'ADN par réaction en chaîne par polymérase (PCR) et électrophorèse...74
7.4.Purification de l'ADN sur gel d'agarose...75
7.5.Digestion par enzymes de restriction...75
7.6.Ligation...76
7.7.Mutagénèse dirigée...76
7.8.Préparation des bactéries compétentes DH5α...76
7.10.Criblage des clones obtenus par transformation...78
7.11.Amplification de la banque d'ADNc normalisée d'A. thaliana...78
7.12.Préparation des sous-pools de banque d'ADNc...79
8. Statistiques...80
RÉFÉRENCES BIBLIOGRAPHIQUES...81
ANNEXES...88
Annexe I : Données brutes permettant les calculs des facteurs d'enrichissement pour les fractions aliquotes F et E...89
Annexe II : Préparation de la banque d'ADNc d'A. thaliana normalisée...90
Annexe III : Tableaux de séquençage des 96 clones aléatoires de la banque d'ADNc d'A. thaliana...92
Annexe IV : Données brutes pour les calculs des taux de transfection afin de déterminer la quantité de banque optimale à transfecter dans les protoplastes de navet...94
Annexe V : Données brutes pour les calculs des taux de transfection de la banque et les taux d'interaction entre les protéines de banque et la protéine P2...95
Annexe VI : Données brutes permettant de calculer les taux de transfection de la banque complète et des sous-pools ainsi que les taux d'interaction en Duolink® pour comparaison. ...96
LISTE DES ABRÉVIATIONS
(-) moins
(+) plus
< inférieur
2X 35S double Promoteur CaMV 35 S
3D tridimensionnel
A. thaliana Arabidopsis thaliana
A. tumefaciens Agrobacterium tumefaciens
aa acides aminés
ADN acide désoxyribonucléique
ADNc ADN complémentaire
ARN acide ribonucléique
db ARN ARN double brin
sb ARN ARN simple brin
ARNm ARN messager
BCIP 5-Bromo-4-Chloro-3-Indolyl Phosphate
BET Bromure d'Ethidium
BiFC Bimolecular Complementation Fluorescence
BQ ou Bq banque
B. rapa ou navet(s) Brassica rapa
C-terminale ou C-ter carboxy-terminale
CaCl2 chlorure de calcium
CaMV Cauliflower mosaic virus
CHUP1 Chloroplaste Insolite Positioning 1
Col-0 Columbia 0
CP Coat Protein ou protéine de capside
CPC Cystéine Proline Cystéine
CT Corps à Transmission
DAPI 4',6'-diamidino-2-phénylindole
dNTP 4 désoxyribonucléotides
DO600 Densité Optique à 600 nm
dpi jours post-infections ou jours post-inoculation suivant le contexte
DTT dithiothréitol
E ou Ech Échantillon
E. coli Esherichia coli
FAT Facteur Assistant de la Transmission
Fc Fragment constant
FMDV Foot and Mouth Disease Virus ou virus de la fièvre aphteuse
GFP Green Fluorescent Protein
HCPro Helper Component Protease
HEPES 4-(2-hydroxyethyl)-1-piperazineethanesulfonic acid
ICTV International Commitee of Taxonomy of Viruses
KCl chlorure de potassium
KH2PO4 di-hydrogénophosphate de potassium
KO Knock Out
LB Lysogeny Broth ou Luria-Bertani Broth
LB Left Border
LIR Large Intergenic Region ou grande région intergénique
MCS Multiple Cloning Site ou site multiple de clonage
MES 2-(N-morpholino)ethanesulfonic acid.
MgSO4 sulfate de magnésium
MLO2 barley Mildew resistance Locus O2 protein
MnCl2 chlorure de manganèse
MOPS 3-(N-morpholino)propanesulfonic acid
MP Movement Protein ou protéine de mouvement
myc-tag étiquette c-myc
N-terminale ou N-ter amino-terminale
Na2HPO4 di-hydrogénophosphate de sodium
NaCl chlorure de sodium
NBT NitroBlue Tetrazolium chloride
NES Nuclear Export Signal ou signal d'export nucléaire
NLS Nuclear Localization Signal ou signal de localisation nucléaire
NOSt Terminateur de la nopaline synthase
NZ caseine hydrolysate
ORF Open Reading Frame ou cadre ouvert de lecture
PBS Phosphate Buffered Saline
PCR Polymerase Chain Reaction
PDLP1 Plasmodesma-Localised Protein 1
PEG PolyEthylène Glycol
PEST Proline, Glutamate, Aspartate, Serine et Thréonine
PFA ParaFormAldéhyde
Pfu Pyrococcus furiosus
PLA Proximity Ligation Assay
PR2 β-glucosidase PR2
proteine 16K protéine riche en cystéine de 16 kDa
PTGS Post-Transcriptional Gene Silencing ou extinction de gène au niveau
post-transcriptionnel
PVDF PolyFluorure De Vinylidène
RB Right Border
RbCl2 chlorure de rubidium
RdRp ARN polymérase dépendante ARN
RFP Red Fluorescent Protein
RNase H RiboNucléase H
RT Reverse Transcriptase ou transcriptase inverse
Rz ribozyme
SDS-PAGE Sodium Dodecyl Sulfate-PolyAcrylamide Gel Electrophoresis
SIR Small Intergenic Region ou petite région intergénique
SSC Saline Sodium Citrate
T- Témoin négatif
T+ Témoin positif
Taq Thermus aquaticus
TBS Tris Buffered Saline
TBS-M TBS-milk
TC TétraCystéine
ter terminateur de transcription
TH Tampon d'Homogénéisation
Tp Tampon protoplastation
TP Tampon
Triton X-100 Polyéthylène glycol mono [4-(1,1,3,3-tétraméthylbutyl) phényl] éther
TRV Tobacco rattle virus
tub tubuline
TuMV Turnip mosaic virus
Tween-20 polysorbate 20
UV Usine Virale
VAP Virion Associated Protein ou protéine associée au virion
VIGS Virus Induced Gene Silencing
wt wild type
Unités :
nm nanomètre µm micromètre % pourcentage mL millilitre L litre µL microlitre kg kilogramme ng nanogramme mM millimolaire M molaire µM micromolaire kDa kilodalton Å Angström kV kilovolt Ω ohmrpm tour par minute
g accélération
h heure
s seconde
pb paire de base
LISTE DES FIGURES ET TABLES
1. Figures
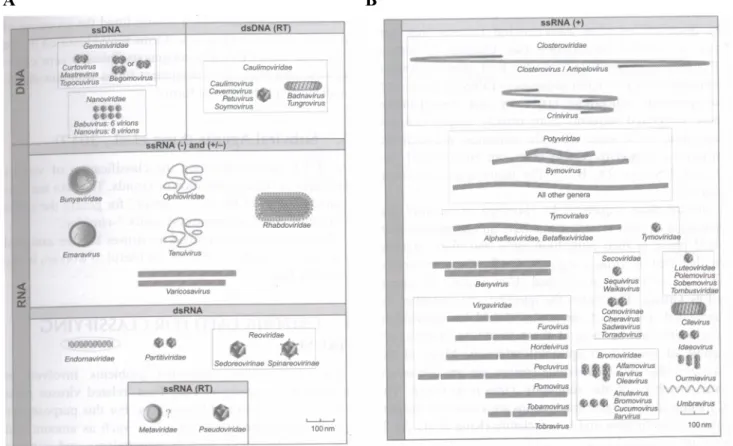
Figure 1 : Taxonomie des virus de plante.
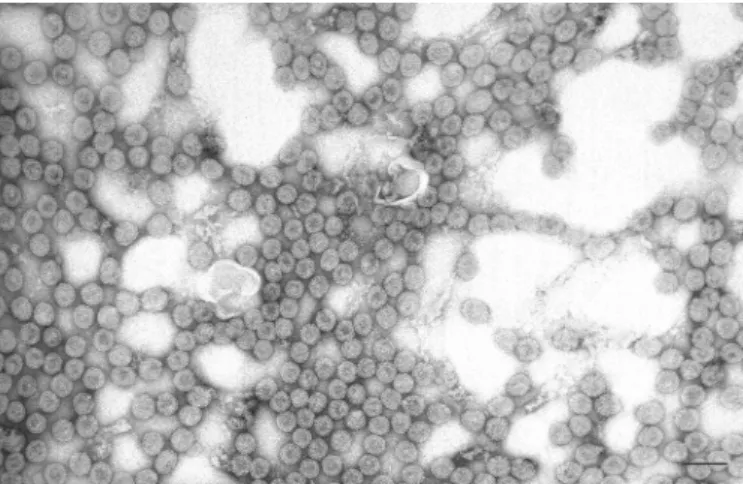
Figure 2 : Symptômes provoqués par l'infection du CaMV sur des feuilles de navet et d'A. thaliana. Figure 3 : Micrographe des virions du CaMV.
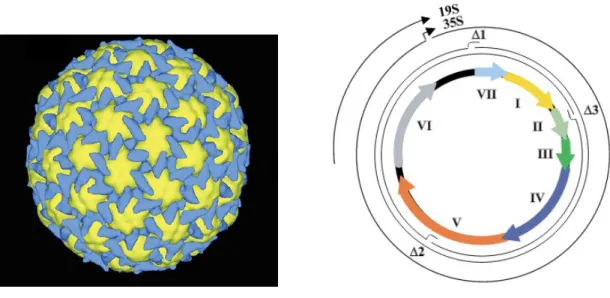
Figure 4 : Reconstitution 3D du complexe P3/virion et schéma de l'organisation du génome du
CaMV.
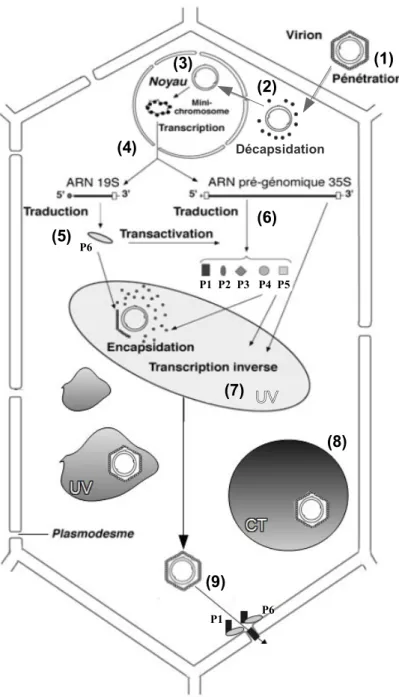
Figure 5 : Structure de l'extrémité N-terminale de la protéine P3. Figure 6 : Cycle infectieux du CaMV.
Figure 7 : Représentation schématique de la transmission d'un virus par puceron.
Figure 8 : Représentation du complexe transmissible et du modèle d'acquisition du CaMV.
Figure 9 : Purification des corps à transmission du CaMV à partir des feuilles d'A. thaliana
infectées par les mutants TC et ΔP2.
Figure 10 : Représentation de la région T-DNA des plasmides pTRV1, pTRV2 et pTRV2-P2::GFP. Figure 11 : Expression de la protéine P2::GFP avec le système TRV dans A. thaliana Col-0. Figure 12 : Présentation des différentes étapes du Duolink®.
Figure 13 : Schéma de la cassette d'expression du vecteur pCK-DUO-banque.
Figure 14 : Association in situ entre la protéine P2 et la tubuline dans des cellules de navet. Figure 15 : Cinétique d'amplification du signal PLA sur l'interaction P2/tubuline.
Figure 16 : Détermination de la quantité optimale de banque à transfecter dans des protoplastes de
navet infectés par le CaMV B-JI.
Figure 17 : Interactions in situ entre la protéine P2 et des protéines de la banque dans des
protoplastes de navet.
Figure 18 : Comparaison entre le taux de transfection de la banque et le taux d'interaction
P2/banque.
Figure 19 : Criblage avec pool I, II et III de clones d'ADNc pour les interactions avec la protéine
P2.
Figure 20 : Schéma du criblage entre les protéines de la banque d'ADNc et la protéine P2.
Figure 21 : Organigramme récapitulatif des étapes de purification des CT-TC en gradient de
2. Tableaux
Tableau I : Caractéristiques des différents anticorps utilisés dans les expériences. Tableau II : Plan de plaque pour la formation de sous-pools de banque d'ADNc.
Les phytovirus sont des parasites obligatoires c’est-à-dire qu’ils ont besoin de facteurs de l'hôte pour réaliser toutes les étapes de leur cycle infectieux, de la réplication au mouvement de cellule à cellule jusqu'à l'infection systémique.
Pour permettre une meilleure identification, les phytovirus comme les virus d' animaux, sont classés en ordres, familles et genres. Initialement, une taxonomie des phytovirus a été établie, basée sur les caractères morphologiques des particules virales ainsi que sur les caractères biologiques descriptifs comme le spectre d'hôte. Puis, avec les avancées scientifiques, ils ont été classés suivant la composition de leur génome et de leur protéine. La classification des virus de plantes présentée dans la Figure 1 suit ce schéma. Elle est réalisée à partir de la nature des acides nucléiques (ARN ou ADN), leur structure (monocaténaire ou bicaténaire, linéaire ou circulaire) et pour les ARN si ils sont messagers (+) ou anti-messagers (-). Il s'avère, que la plupart des phytovirus (90 %) comporte un génome à ARN monocaténaire de polarité positive (Hull, 2014).
Le cycle viral des phytovirus se passe en 3 étapes : – l'inoculation
– l'établissement de l'infection locale dans un nouvel hôte, qui se caractérise par la réplication et le mouvement de cellule à cellule.
– l'établissement de l'infection systémique qui se distingue par le mouvement à longue distance dans les vaisseaux, suivi par la réplication et le mouvement de cellule à cellule dans les tissus de destination comme lors de l'infection locale.
Pour être inoculé sur une nouvelle plante, ils ont besoin de traverser la paroi rigide pecto-cellulosique des cellules végétales qui est différente de celle des cellules animales où les virus peuvent pénétrer de manière autonome. Pour cela, les phytovirus sont amenés le plus souvent par un vecteur. Après cette étape de transmission, les phytovirus doivent se répliquer pour établir l'infection dans les cellules inoculées et se propager dans toute la plante. La propagation se fait d'abord d'une cellule à l'autre puis de manière systémique. Le déplacement dans l'hôte se fait sur deux niveaux : de cellule à cellule et le mouvement à long distance dans le phloème qui transporte les virus dans les autres organes et parties de la plante.
Au niveau de cellule à cellule, les virus se déplacent à travers les plasmodesmes sous forme de virion ou sous forme de complexes nucléoprotéiques avec des protéines virales ainsi que des protéines cellulaires de l'hôte. Il est nécessaire que des protéines virales de mouvement soient impliquer pour dilater les plasmodesmes qui normalement ont un goulot trop petit pour laisser passer des grandes molécules (Niehl and Heinlein, 2011).
Pour que les virus puissent atteindre la sève élaborée (phloème) de la plante sans rester cantonnés aux cellules de l’épiderme et du mésophylle, ils entrent, toujours par des plasmodesmes, dans le
système vasculaire en franchissant le parenchyme phloémien et les cellules compagnes et toujours en impliquant des protéines de mouvement virales spécifiques. Après le transport dans la sève à un tissu cible, les plasmodesmes qui relient les tubes criblés aux cellules compagnes permettent aux virus de sortir du phloème et d'établir un nouveau foyer d'infection dans des tissus éloignés (Collectif and Lepoivre, 2003; Hipper et al., 2013). C'est ce que nous appelons le mouvement à longue distance.
Les plasmodesmes du système vasculaire sont différents des plasmodesmes reliant le mésophylle et l'épiderme. Ces différences se situent au niveau morphologique et protéique ce qui expliquerait pourquoi certains mutants de virus sont toujours capables de se déplacer dans le mésophylle, mais incapable d'envahir le phloème. Après la distribution et la multiplication dans toute la plante, les virus de plantes pourront être une nouvelle fois acquis et transmis par leur vecteur permettant de les transporter dans une autre plante. Cette étape doit être achevée avant que la plante ne récupère de l'infection ou n'en meurt. La transmission sera décrite plus en détails dans la partie 3 de l'introduction.
1. Présentation du Cauliflower mosaic virus
Cette partie de l'introduction sera consacrée à la description du génome, la réplication ainsi que le cycle infectieux du Cauliflower mosaic virus (CaMV) qui est le virus étudié dans ce projet. Le CaMV est le membre type de la famille des Caulimoviridae. Cette famille est subdivisée par l’International Committee of Taxonomy of Viruses (ICTV) en sept genres : Badnavirus,
Tungrovirus, Petuvirus, Cavemovirus, Soymovirus, Solendovirus et Caulimovirus. Le CaMV a été
largement décrit dans la littérature depuis sa découverte en 1937 (Tompkins, 1937). La mosaïque du chou-fleurs qui est la maladie caractéristique provoquée par ce virus, a peu d'impact économique et agricole. Cependant par effet de niche, le CaMV peut détruire des cultures entières car il est très virulent.
Le CaMV contient un génome en ADN double-brin circulaire. La ressemblance avec un plasmide bactérien facilite son clonage dans des plasmides ainsi que la mutagenèse. De plus, le génome viral peut être libéré du plasmide par digestion et il est directement infectieux par inoculation mécanique. C'est pour cela que le CaMV est devenu un virus modèle pour les plantes et par ailleurs, c'est le premier phytovirus à ADN à être entièrement séquencé (Franck et al., 1980).
Le CaMV infecte les plantes dicotylédones surtout dans des familles des Brassicacae et des
Solanacae (Broadbent et al., 1953; Schoelz et al., 1986)
.
Ce virus provoque par exemple surArabidopsis thaliana (A. thaliana) ou Brassica rapa (B. rapa ou navet) des marbrures sur les
feuilles ainsi qu'un jaunissement de la nervure centrale et un nanisme de la plante (Fig.2). Les symptômes peuvent largement varier selon la souche virale et l'espèce et variété végétale concernées.
1.1. Composition du génome viral et de ses protéines
Le CaMV possède un virion de symétrie icosaèdrique de 50 nm de diamètre (Fig.3). La capside non enveloppée est constituée de 420 sous-unités protéiques, organisées dans une symétrie d’ordre T = 7 (Plisson et al., 2005) (Fig.4A). Elle contient une molécule d'ADN double brin circulaire d'environ 8000 nucléotides avec trois discontinuités sur les deux brins (Franck et al., 1980). La réplication du virus se fait par transcription inverse à partir d' un ARN pré-génomique matrice. Sachant qu'il a un mode de réplication particulier, le CaMV est répertorié avec le virus de l' hépatite B, qui est classé dans le groupe non phylétique des pararetrovirus. Le génome possède sept cadres de lecture ouverts majeurs (ORFs) codant sept protéines et deux régions non codantes, dénommé small intergenic region (SIR) et large intergenic region (LIR) (Haas et al., 2002). Sur ces
sept protéines, six sont exprimées de manière détectable dans la plante à partir de deux ARN messagers. L'ARN 35S code le génome entier avec en plus une petite partie redondante, tandis que l'ARN 19S code seulement la protéine P6 (Fig4.B) (Haas et al., 2002). Ces sept protéines ont des fonctionnalités différentes pour permettre la réplication, le mouvement intra-hôte et la transmission du virus.
1.1.1. Protéine P1 ou protéine de mouvement (MP)
La protéine P1 possède un poids moléculaire de 40 kDa et est impliquée dans le mouvement de cellule à cellule. Elle forme des tubules au niveau de plasmodesmes permettant le passage des virions via les plasmodesmes (Perbal et al., 1993). Il a été démontré que la protéine P1 s'auto-assemble dans des trimères et interagit avec la protéine P3 lorsque cette protéine est liée au virion (Stavolone et al., 2005). Lorsque la protéine P1 est modifiée dans sa partie N-terminale (se liant à la protéine P3), le virus ne peut plus établir une infection systémique sans pour autant avoir de répercutions sur sa réplication (Thomas et al., 1993).
La protéine P1 est traduite dans le cytoplasme au niveau des usines virales. Pour permettre son adressage vers les plasmodesmes, elle est transportée d'abord par un mécanisme inconnu à la membrane plasmique. Puis elle est internalisée par endocytose via des vésicules à clathrine et semble circuler entre l'endosome et la membrane plasmique, pour atteindre les plasmodesmes (Carluccio et al., 2014). Pour ceci, la protéine P1 interagit avec la µA-adaptin, un composant des puits et vésicules à clathrine, grâce à 3 motifs d'adressage tyrosine, qui sont aussi utilisés par des milliers de protéines cellulaires (Carluccio et al., 2014).
1.1.2. Protéine P2 ou facteur assistant de la transmission (FAT)
La protéine P2 dont la taille est de 18 kDa est un élément clé dans la transmission du CaMV par le puceron car elle fait la liaison entre le virion et le vecteur. La partie C-terminale forme une structure tertiaire de super-hélice dite coiled-coils, qui interagit avec les coiled-coils de deux autres molécules de P2 pour former un trimère (Hébrard et al., 2001). Ces super-hélices interagissent avec la protéine virale P3 lorsque celle-ci est associée à la capside (Drucker et al., 2002; Leh et al., 2001) en formant un complexe transmissible pouvant être acquis par le puceron. Au point de vue structural, la partie N-terminale de la protéine P2 n'a pas été éditée. Dans le complexe transmissible, composé des protéines P2, P3 et du virion, la partie N-terminale de la protéine P2 semble impliquée dans l'interaction avec le vecteur (Moreno et al., 2005) et plus précisément elle peut se lier à un récepteur dans l'acrostyle du puceron qui est situé dans le canal commun des stylets maxillaires
(Uzest et al., 2010, 2007).
Après traduction, la protéine P2 est localisée en totalité dans des inclusions spécifiques, dénommées corps à transmission (CT) présentes dans le cytoplasme des cellules infectées (Martinière et al., 2009b). Son fonctionnement dans la transmission est détaillé dans la partie 3 de l'introduction.
1.1.3. Protéine P3 ou protéine associée au virion (VAP)
La protéine P3 a un poids moléculaire de 15 kDa. L'extrémité C-terminale de la protéine P3 est impliquée dans la liaison à l'ARN et l'ADN. La région centrale de la protéine P3 composée de 20 acides aminés (aa) résiduels non requis pour l'infection par le CaMV (Jacquot et al., 1998). L'extrémité N-terminale de la protéine P3 contient 3 hélices a qui peuvent former des coiled-coils responsables de l'auto-assemblage en tétramère de la protéine (Leclerc et al., 1998). La protéine P3 soluble peut former un tétramère parallèle contenant 2 domaines coiled-coils de latéralité inversée qui créent un phénomène de tension dans le tétramère (Hoh et al., 2010). Après ces 2 domaines, un motif Cystéine Proline Cystéine (CPC) peut former des ponts disulfures, suivi par une 3ème hélice très courte (Fig.5). Grâce à ce motif CPC, la protéine P3 pourrait réagir aux changements du potentiel d'oxydoréduction de la cellule (Hoh et al., 2010). La fonction de cette structure est cependant méconnue. La partie C-terminale de la protéine P3 n'est pas structurée. La protéine P3 peut interagir avec les protéines P1, P2 et la protéine de capside P4. Quand la protéine P3 est associée au virion, elle forme un réseau, composé de triskéles, autour des capsomères (Plisson et al., 2005). La partie C-terminale de la protéine P3 est ancrée dans la capside virale, probablement en interagissant avec l'ADN viral, tandis que la partie N-terminale, toujours structurée en coiled-coils, forme des dimères qui sont exposés en surface et peuvent interagir avec d'autres protéines virales. Ces associations en dimères interagissent avec la protéine P1 (Stavolone et al., 2005) ainsi qu'avec l'extrémité C-terminale de la protéine P2 (Hébrard et al., 2001; Leh et al., 1999). Les structures coiled-coils , du tétramère seraient « fermées » et ne pourraient plus interagir avec les protéines P2 et P1.
1.1.4. Protéine P4 ou protéine de capside (CP)
Les isoformes de la protéine CP dérivent d'une protéine précurseur de 57 kDa. Cette protéine précurseur est clivée - grâce à l'activité protéolytique de la protéine P5 - en trois protéines de tailles différentes : 44 kDa, 39 kDa et 37 kDa. Ces trois protéines possèdent la même séquence centrale mais ont des différences au niveau des extrémités (Chapdelaine et al., 2002).
La protéine CP de 44 kDa possède à son extrémité C-terminale un motif en doigt de zinc qui interagit avec les acides nucléiques permettant la formation de particules virales. La partie N-terminale contient une séquence signal de localisation au noyau (NLS). Cette extrémité contient aussi un motif de dégradation par le protéasome caractérisé par des résidus Proline, Glutamate, Aspartate, Serine et Thréonine (PEST). Ce motif est présent sur la protéine CP précurseur et de 44 kDa (Karsies et al., 2001).
Les extrémités N-terminale et C-terminale contiennent plusieurs sites de phosphorylation par la caséine kinase II cellulaire qui sont impliqués dans l'infectivité du CaMV. Lorsque ces extrémités sont mutées, le virus devient non-infectieux (Chapdelaine et al., 2002).
1.1.5. Protéine P5 ou reverse transcriptase (RT)
C'est une polyprotéine de 78 kDa qui est impliquée dans la réplication du génome viral. L'extrémité N-terminale comporte une activité de protéinase à acide aspartique qui autocatalyse le clivage du N-terminal en polypeptides de 20 et 22 kDa. Il s'auto-mature et joue un rôle dans la maturation de la protéine de capside P4 (Torruella et al., 1989). L'extrémité C-terminale est le domaine qui possède l'activité transcriptase inverse (RT) pour la réplication du génome et aussi l'activité de ribonucléase H (RNase H). Dans un premier temps, la protéine P5 est traduite en une protéine précurseur inactive puis, dans un second temps, elle s'active par processus auto-protéolytique grâce à l'activité de la protéinase à acide aspartique.
1.1.6. Protéine P6 ou transactivator/viroplasmin (TAV)
La protéine P6 est une protéine multifonctionnelle de 62 kDa. Au niveau de l'extrémité N-terminale, dans sa partie centrale, il y a un signal d'export nucléaire (NES) ainsi que deux signaux NLS, lui permettant de circuler entre le noyau et le cytoplasme (Haas et al., 2005). La protéine P6 interagit avec la protéine CP et la protéine P1 (MP) et avec les deux protéines nécessaires à la transmission, P2 et P3 (Lutz et al., 2012).
La protéine P6 constitue la matrice des usines virales qui servent de réservoir pour les virions. Les usines virales formées grâce à l'auto-assemblage de P6 se déplacent au niveau intracellulaire le long des filaments d'actine. Ces microfilaments d'actine sont nécessaires pour l'infectivité du CaMV (Harries et al., 2009).
Mis à part, le fait que la protéine P6 est la composante principale des usines virales, elle joue de nombreux rôles dans le cycle infectieux du CaMV. Elle possède la fonction de suppresseur de
silencing post-transcriptionnel de l'ARN (PTGS) et joue un rôle dans la mort cellulaire et dans la
sévérité des symptômes (Haas et al., 2008; Hull, 2014).
Elle est nécessaire pour la traduction des protéines virales à partir de l'ARN 35S. Cet ARN est polycistronique. Les ORF qui sont présents en amont de l'ORF 1 ne sont pas lus par les ribosomes eucaryotes. En interagissant avec plusieurs facteurs du ribosome et du complexe d'initiation de la traduction, la protéine P6 permet le « saut » du ribosome à travers la région 5' de l'ARN 35S qui contient des tiges boucles extrêmement stables et la ré-initiation de la traduction après chaque ORF. La protéine P6 possède de nombreuses interactions avec différents partenaires que ce soit avec des protéines virales ou des facteurs cellulaires de l'hôte. Récemment, il a été décrit que la protéine P6 jouerait un rôle dans le mouvement intracellulaire des virions. Il a été démontré que la P6 interagit avec une protéine de plante qui est la Chloroplast Insolite Positioning 1 (CHUP1). CHUP1 est localisée sur la membrane externe des chloroplastes et est impliquée dans le mouvement des chloroplastes sur les microfilaments ainsi que dans le mouvement intracellulaire du CaMV en interagissant avec la protéine P6 (Angel et al., 2013).
Nous savons que le complexe P3/virion se déplace vers les plasmodesmes indépendamment de la MP (P1) (Stavolone et al., 2005), et il a été suggéré que la protéine P6 pourrait être impliquée dans la « livraison » des virions aux plasmodesmes (Schoelz et al., 2011). Plus récemment, il a été montré que la protéine P6 interagit avec le domaine C2 de la protéine AtSRC2.2, qui est un domaine structural protéique impliqué dans le ciblage des protéines de la membrane cellulaire (Rodriguez et al., 2014). AtSRC2.2 est localisée au niveau de la membrane plasmique et est associée aux plasmodesmes. Cette protéine co-localise avec la protéine MP et la protéine de plasmodesme Plasmodesma-Localised Protein 1 (PDLP1). Les auteurs suggèrent que des petites inclusions de P6 contenant des virions migrent à l'aide des filaments d'actine vers les plasmodesmes et y déposent les virions, avec l'implication de AtSRC2.2, qui elle-même peut interagir avec MP. Ainsi des virions seront transférés de P6 vers la MP aux plasmodesmes (Rodriguez et al., 2014).
1.1.7. Protéine P7
Cette protéine est très peu décrite dans la littérature. La protéine P7 peut être produite dans la levure. Cependant elle ne peut pas être détectée dans les plantes infectées (Wurch et al., 1990). Il a été démontré que la séquence de l'ORF VII ainsi que la région intergénique entre l'ORF VII et l'ORF I n'est pas nécessaire pour l'infection par le CaMV. Cependant l'emplacement des codons
start et stop dans cette région affecte l'infectivité du CaMV (Dixon and Hohn, 1984).
1.2. Cycle infectieux du CaMV
Le cycle infectieux du CaMV (Fig.6) comporte plusieurs phases : la réplication où le virus détourne la cellule pour produire des virus descendants, la propagation de l'infection dans les cellules/tissus voisins et finalement dans toute la plante hôte et la transmission par son vecteur puceron.
Le cycle du CaMV débute par l'entrée du virus dans le cytoplasme d'une cellule hôte, grâce à son vecteur puceron ou via les plasmodesmes. Puis la réplication virale peut débuter. Elle comporte deux phases (Fig.6) (Hull, 2014) :
– transcription des ARN viraux à partir du génome à ADN dans le noyau.
– traduction des protéines virales et transcription inverse à partir de l'ARN 35S en ADN génomique dans le cytoplasme.
Dans la première phase de réplication, la particule virale est adressée au noyau par le NLS situé en N-terminal de la protéine CP. Elle est retenue aux pores nucléaires, probablement par l'interaction avec l'importin a (Karsies et al., 2001) et est décapsidée. L'ADN double brin migre dans le noyau où ses discontinuités de séquences sont réparées. Cet ADN s'associe aux histones nucléaires pour former un mini-chromosome. Ce mini-chromosome est utilisé comme matrice pour la transcription de deux ARN messagers viraux majeurs, l'ARN monocistronique 19S et l'ARN polycistronique 35S. La transcription a lieu grâce à l'ARN polymérase II dépendante de l'ADN de l'hôte. Ces deux ARN migrent dans le cytoplasme. L'ARN 19S est traduit en protéine P6 qui va, entre autres, s'accumuler dans des foyers, et former la matrice des usines virales futures. Elles sont présentes tout au long de l'infection et croissent jusqu'à atteindre des tailles variables supérieures ou égales à 10 µm. Dans ces usines ont lieu la transcription inverse et la traduction des autres protéines virales. Dans la deuxième phase de la réplication, l'ARN pré-génomique 35S sert de matrice pour la transcription inverse. L'ADN négatif est transcrit en premier à partir de l'ARN 35S pré-génomique en utilisant comme amorce l'ARN de transfert-méthionine cytoplasmique. Après synthèse du brin d'ADN négatif, la RNase H dégrade l'amorce ARN en épargnant une petite séquence riche en purine qui va servir d'amorce pour la synthèse du brin positif d'ADN. La phase d'élongation du brin positif est bloquée quand la polymérase arrive à l’extrémité 5’ du brin d’ADN négatif et ne peut se poursuivre qu’après appariement avec l’extrémité 3’ du brin négatif pour permettre la circularisation de l'ADN viral. Trois discontinuités dans l'ADN néoformé témoignent de ce mode particulier de transcription. En même temps ou peu après, l'ADN viral est encapsidé en s'associant aux capsomères de la protéine P4.
En parallèle de la réplication virale dans les usines virales, la protéine P6 produite à partir de l'ARN 19S permet d'activer la traduction des protéines P1 à P5 à partir de l'ARN 35S. L'accumulation des
différentes protéines au cours de l'infection commence donc par la production de P6, puis les protéines P1 et P5, suivies par les protéines P3 et P4 et tardivement par la protéine P2 (Maule et al., 1989). Le contrôle de l'expression des protéines virales et surtout de la protéine P2 se fait grâce à un mécanisme d'épissage alternatif de l'ARN 35S (Froissart et al., 2004).
Les nouveaux virions sont stockés majoritairement dans les usines virales puis certains relocalisent soit dans les CT , soit vers les plasmodesmes.
Après avoir répliqué son génome viral dans la cellule hôte, le virus se propage sous forme de virion de cellule à cellule et par les tubes criblés afin d'infecter toute la plante. Comme décrit précédemment, le CaMV est acheminé vers les plasmodesmes avec l'aide de la protéine P6 (Carluccio et al., 2014). Les plasmodesmes sont dilatés grâce à la protéine P1 (Stavolone et al., 2005) et permettent le passage du virion. Pour établir l'infection systémique où le CaMV infecte la quasi-totalité des organes de la plante incluant les racines, les virions doivent franchir les plasmodesmes entre les cellules compagnes et les tubes criblés et sortir par la même voie. Nous supposons que la protéine P1 est aussi impliquée dans le mouvement systémique. Cependant, à ma connaissance, les études menées sur le mouvement de la protéine P1 ne font pas la distinction entre les plasmodesmes du système vasculaire et ceux du parenchyme alors qu'ils sont différents. Ainsi, l'infection se répand dans toute la plante de manière systémique.
2. Transmission des virus de plante
2.1. Généralités sur la transmission
Pour que les phytovirus assurent leur survie dans l'environnement, ils doivent maintenir leur population virale en étant transmis à la descendance de la plante infectée, ou en contaminant d'autres individus ou d'autres espèces végétales. Pour cela, il existe deux modes de transmission : verticale et horizontale.
La transmission verticale permet à quelques virus d'être transmis par passage direct d'une génération de plante à la suivante, par multiplication végétative (greffage, taille etc...), ou via les semences et le pollen (Hull, 2014).
La transmission la plus représentée chez les phytovirus est la transmission horizontale. Très souvent, la transmission de plante à plante est effectuée par des vecteurs invertébrés de types insectes piqueurs-suceurs, nématodes et quelques champignons. L'utilisation de ces vecteurs s'explique du fait, que les plantes sont immobiles, et que la paroi pecto-cellulosique des plantes est une barrière, que les virus ne peuvent pas franchir seuls.
70 % des phytovirus utilisent comme moyen de transport les insectes piqueurs-suceurs comme les pucerons de la super-famille des Aphidoidea. Ces insectes peuvent disséminer de manière spatiale les virus permettant d'infecter des plantes relativement éloignées. Grâce à leurs pièces buccales, les piqueurs-suceurs peuvent franchir la paroi végétale et acquérir les virus puis les inoculer facilement dans une autre plante hôte. De plus, les insectes piqueurs-suceurs contrairement aux insectes phytophages broyeurs (par exemple Spodoptera sp.) ont un comportement alimentaire non destructif c'est à dire que leur piqûre n'est pas mortelle pour les cellules cibles de l'hôte. Ces piqûres permettent de micro-injecter des virus dans les cellules vivantes pour ainsi obtenir une transmission efficace.
Il existe aussi quelques rares cas de transmission horizontale (par exemple les Tobamovirus) où les virus sont transmis de manière mécanique par contact entre deux plantes voisines ou par l'intermédiaire de l'homme.
Les différentes étapes de la transmission par vecteur sont décrites ci-dessous (Astier et al., 2001) : – l'acquisition est le temps nécessaire pour que le vecteur ingère du virus à partir d'une plante
infectée.
– L'inoculation est le temps nécessaire pour que le puceron transmette le virus sur une nouvelle plante saine.
– la latence est la période entre l’acquisition du virus et sa transmission. 23
– La rétention est le temps pendant lequel le puceron à la capacité de transmettre le virus. Selon le temps nécessaire pour les différentes étapes de transmission, les phytovirus transmis par pucerons et autres vecteurs, ont pu être répertoriés dans trois catégories : persistant, non persistant et semi-persistant.
2.1.1. Transmission persistante
Les phases d'acquisition et de latence du vecteur sont très longues (heures) car l'acquisition des particules virales est le plus souvent faite dans la sève élaborée du phloème (Fig.7A). Les virus concernés effectuent un cheminement dans le corps du vecteur en passant du tube digestif dans l'hémocèle pour arriver aux glandes salivaires et être injectés avec la salive dans une nouvelle plante hôte. Il existe deux modes différents. Lorsque, le virus a la possibilité de se répliquer dans le vecteur, nous parlons de transmission persistante multipliante comme pour les Rhabdoviridae. Quand le virus passe dans le vecteur sans se répliquer, la transmission est dite persistante non multipliante comme pour le Tomato yellow leaf curl virus (TYLCV). La phase de rétention dure toute la vie du vecteur. Une autre caractéristique de ce mode de transmission est le fait que les pucerons peuvent encore transmettre après leurs mues successives. Parce que la transmission persistante est caractérisée par un passage du virus dans le vecteur, elle est aussi appelée transmission circulante.
2.1.2. Transmission non persistante et semi-persistante
La transmission dite non-persistante ou semi-persistante est caractérisée par le fait que les virus ne sont pas internalisés par les vecteurs. Ce mode de transmission est aussi appelée transmission non-circulante. Dans ce type de transmission, des piqûres d'essai (durant quelques secondes à une minute) (Fig7.B) suffisent pour récupérer le virus, qui s'accroche sur une partie spécifique des pièces buccales du vecteur, comme l'acrostyle des stylets pour le CaMV (Uzest et al., 2010, 2007) ou le precibarium des Bemisia tabaci pour le Lettuce infectious yellows virus (LIYV) (Chen et al., 2011).
Les particules virales sont retenues par les récepteurs suivant deux stratégies dites de capside ou
helper. Lors de la stratégie capside, la capside virale se lie directement au récepteur dans les stylets
maxillaires ou le precibrium tandis que la stratégie helper fait intervenir une protéine complémentaire permettant de faire un pont entre le virion et le récepteur du vecteur (Pirone and Blanc, 1996). Dans les deux cas, le virus est transmissible sans phase de latence. La phase de rétention est assez réduite de l'ordre de plusieurs heures à quelques jours et après que le vecteur ait
mué, le virus n'est plus transmis . Il existe des différences entre la transmission non persistante et semi-persistante. Lors de la transmission semi-persistante, la phase de rétention est plus longue dans le vecteur que lors de la transmission non persistante. L'acquisition du virus se fait pendant des piqûres tests dans l'épiderme et le mésophylle pour la transmission non persistante, alors que les virus ayant une transmission semi-persistante peuvent aussi être acquis dans le phloème. Le CaMV qui est notre virus d'étude, est un exemple de la transmission semi-persistante suivant la stratégie
helper.
2.2. Transmission du CaMV
Le CaMV est transmis par des vecteurs piqueurs suceurs de type pucerons. Au moins une trentaine d'espèces de pucerons transmettent le CaMV (Kennedy et al., 1962), les meilleurs vecteurs sont Myzus persicae et Brevicorynae brassicae (Markham et al., 1987).
Le CaMV forme dans la cellule infectée deux sortes de corps d'inclusion : les usines virales et le corps à transmission (CT). Les usines virales sont abondantes dans la cellule tandis que généralement un seul CT est présent par cellule. Les usines virales sont le lieu du stockage des virions, de la réplication virale et de la traduction et sont localisées en général à proximité du réticulum endoplasmique (Bak et al., 2014). Le CT quant à lui est dédié à la transmission virale. Leur différence n'est pas seulement fonctionnelle mais aussi phénotypique, les usines virales ont des formes et des tailles variables (Fig.8B) tandis que le CT a une morphologie sphérique de taille peu variable. Le CaMV forme des complexes impliqués dans la transmission qui sont observables dans les cellules végétales ainsi que dans le puceron.
2.2.1. Complexe transmissible et corps à transmission
Comme précédemment décrit, le CaMV suit la stratégie helper du mode semi-persistant pour être transmis dans une nouvelle plante hôte. Ce mode implique la formation d'un complexe transmissible et son accrochage dans les pièces buccales du puceron au niveau de l'acrostyle (Uzest et al., 2010, 2007). Ce complexe transmissible est composé de la protéine P2 et du complexe P3/virion (Leh et al., 1999). La protéine P2 est considérée comme un Helper Component (HC) ou facteur assistant de la transmission (Blanc et al., 1993) ayant une fonction commune avec la protéine HCPro des Potyvirus, qui fait le lien moléculaire entre le virion et le vecteur (Blanc et al., 1997) (Fig.8A). La formation du complexe transmissible est obligatoire pour la transmission du CaMV.
Pour identifier et comprendre les mécanismes impliqués dans l'acquisition du CaMV dans les cellules infectées, il faut connaître le comportement alimentaire du puceron. La composition chimique intrinsèque suivant l'espèce de plante et les substances allélochimiques produites par les plantes jouent un rôle important dans le choix alimentaire du puceron. En revanche, il y a aussi une part d'aléatoire dans l'arrivée des pucerons sur les plantes hôtes. Lorsque le puceron se retrouve sur une feuille, il procède d'abord, à une série de piqûres d'essai dans l’épiderme de la plante avec ses stylets. Puis, pour se nourrir, il réalise des prélèvements dans le phloème (Powell et al., 2006). L'injection de la salive et l'aspiration des composants végétaux au moment de prélever permet d'identifier la plante hôte. C'est à dire que des stimulus tels que le pH, la teneur en sucres et acides aminés permettent d'identifier le phloème correspondant à sa plante d’intérêt. L'hypothèse de départ était que la protéine P2 et les virions étaient aspirés ensemble avec du cytoplasme lors de ces piqûres d'essai. Or il s'est avéré que le phénomène est plus complexe.
Il a été démontré que lorsque des pucerons sont d'abord nourris sur membrane (Parafilm M®)
contenant la protéine P2, puis avec le complexe P3/virion purifiés, il y a transmission du CaMV. Cela indique que le puceron peut dans un premier temps, acquérir de la protéine P2 libre lors d'une piqûre d'essai, puis dans un second temps, des complexes P3/virion ce qui suggère que les complexes transmissibles se forment dans le puceron vecteur (Drucker et al., 2002). Cette acquisition en deux phases est appelée séquentielle. Le fait que la protéine P2 produite est préalablement récupérée par le vecteur, permet d'assister la transmission des virions produits dans une autre cellule ou dans une autre plante. Ce phénomène est appelé trans-complémentation et pourrait augmenter la biodiversité des virus transmis (Froissart et al., 2002).
L'utilisation de technique de marquage et de microscopie électronique a permis d'identifier dans les cellules végétales, un type d'inclusion claire nommée aujourd'hui CT, contenant la totalité des protéines P2 alors que 95 % des virions sont stockés dans les usines virales (Fig.8Ba) (Drucker et al., 2002). Nous savons que toutes les cellules végétales infectées par le CaMV contiennent des CT sauf les tubes criblés qui possèdent seulement les virions. Il s'est avéré que lors des tests de transmission, leur efficacité est doublée lorsque le puceron peut réaliser des piqûres préliminaires dans le parenchyme puis dans la sève élaborée du phloème ce qui témoigne du rôle important de ce mode séquentiel dans la transmission du CaMV (Palacios et al., 2002).
Un mutant CaMV exprimant une protéine P2 mutée sur l'aa 94 (P2-94) a été utilisé pour déterminer le rôle des CT dans la transmission. Ce mutant ne possède plus la capacité d'être transmis de plante à plante cependant une accumulation de la P2-94 dans les plantes infectées est observable sous forme de paracristaux et non pas de CT. Dans la publication (Khelifa et al., 2007), des tests de transmission ont été réalisés en utilisant un broyât de plantes infectées avec le mutant P2-94 pour permettre l’acquisition de ce virus par les pucerons, à travers des membranes. Puis ces pucerons ont
été placés sur des plantes saines. Après cette acquisition, ils ont observé, de la transmission. En revanche, lorsque ils ont utilisé ce mutant en tests de transmission de plantes infectées à plantes saines (sans broyage), ils n'ont observé aucune transmission (Khelifa et al., 2007). Les auteurs ont conclu que la simple présence de P2 dans une cellule n'est pas suffisante pour la transmission, mais que la protéine P2 doit être stockée dans un CT pour permettre la transmission. Nous savons depuis une vingtaine d'années que la protéine P2 possède une affinité pour la tubuline, sans savoir pourquoi (Blanc et al., 1996). Plus récemment, il a été montré, pour qu'une formation de CT ait lieu, il faut que les protéine P2, P3 et les virions puissent circuler via les réseaux de microtubules (composés de tubuline) pour passer des usines virales au CT. Il y a une véritable cinétique de formation de CT (Martinière et al., 2009a). Ces résultats suggèrent que les CT ne sont pas de simples structures remplies de protéine P2 inerte et que la transmission du CaMV n'est pas un événement aléatoire.
2.2.2. Mécanismes d'activation du corps à transmission
Tout un mécanisme d'activation est déclenché dans la cellule hôte pour permettre l’acquisition du complexe transmissible par le puceron. Il a été démontré que l'utilisation de différents traitements changeait la morphologie du CT (Martinière et al., 2013). En effet, un choc thermique et un stress mécanique provoquent par contact d'une feuille avec une micro-électrode, l'entrée de tubuline dans le CT. En revanche, lorsqu'une blessure est réalisée au scalpel sur une feuille infectée avec le CaMV, ou qu'elle est incubée avec dudioxyde de carbone, il y a dissociation du CT et relocalisation de la protéine P2 et des virions sur les réseaux de microtubules appelées réseaux mixtes. Lorsque la cellule est non stressée, les CT se reforment. Il a été démontré que la forme réseaux mixtes était la plus adaptée pour la transmission du CaMV. Effectivement, le taux de transmission est doublé pour les structures réseaux mixtes par rapport aux cellules témoins présentant des CT seuls. L'immunomarquage de feuilles infectées où des pucerons ont été placés, a révélé que la présence seule des pucerons est suffisante pour induire des réseaux mixtes près des sites de piqûre de ces pucerons. Puis, lorsque les pucerons sont retirés des feuilles, les CT se reforment. L'ensemble de ces données a permis d'identifier que le CaMV peut « percevoir » la présence du puceron sur la plante et déclencher une réponse immédiate en induisant des réseaux mixtes dans les cellules. Ces réseaux permettent d'augmenter les chances d’acquisition du virus (Martinière et al., 2013).
Un modèle d'acquisition du CaMV (Fig.8C) a été décrit (Martinière et al., 2013) permettant d'expliquer la réaction des CT qui est liée à l'insertion des stylets du puceron dans la cellule infectée.
Tout d’abord, les CT sont en stand-by (Fig.8C1 et Bb) lorsqu’il n’y a pas de piqûre de l’insecte aux alentours de la cellule infectée. Lorsque le puceron induit par sa piqûre un stress mécanique (mouvement des stylets) et/ou chimique (par exemple un composant de la salive), il est perçu par la cellule végétale ce qui provoque un afflux très rapide de tubuline (en quelques secondes) (Fig.8C2) dans le CT. Cette forme est dite activée. Puis, le CT se dissocie et la protéine P2 et quelques virions se relocalisent (quelques secondes) sur les microtubules pour y former des réseaux mixtes (Fig.8C3
et Bb). Sur ces réseaux, tous les composants du complexe transmissible sont réunis et distribués de
manière homogène dans toute la cellule. Cette répartition homogène des complexes permet d'augmenter les chances de récupération du virus par son vecteur ainsi que d'augmenter sa transmission lors des brèves piqûres d'essai ou le vecteur ingère très peu de volume de la cellule infectée. Après que le puceron ait rétracté ses stylets (Fig.8C1), la cellule infectée reprend son état initial et un nouveau CT en stand-by est généré à partir des réseaux mixtes. C'est la phase de réversion. Le CT est prêt pour un nouveau cycle de transmission déclenché par la piqûre d'un autre puceron. Les virions qui sont présents en grande quantité sur les réseaux mixtes ont été identifiés récemment comme provenant des usines virales (Bak et al., 2013). En effet, les virions peuvent rapidement - en parallèle de la dissociation des CT - sortir des usines virales et s'accumuler sur les réseaux mixtes. Lors de la réversion du CT, ces virions peuvent retourner aux usines virales ou être incorporés dans le CT en stand by. Ces observations suggèrent que les usines virales du CaMV sont des structures dynamiques qui participent, comme le CT, activement à la transmission en libérant et en réabsorbant de manière contrôlée des virions au cours de la réaction du CT (Bak et al., 2013). Les rôles de la protéine P2 et des virions dans l'activation de la transmission sont bien décrits mais l'implication de la protéine P3 reste floue. Il a été démontré que la protéine P3 existe dans les CT sous deux formes différentes : associée au virion ou libre (Drucker et al., 2002). La forme associée au virion est requise pour la formation des complexes transmissibles, alors que la forme libre de la protéine P3 pourrait empêcher la précipitation de la protéine P2 en paracristaux inactifs en transmission (Blanc et al., 1993; Khelifa et al., 2007). Cette forme permettrait de maintenir la protéine P2 dans un état fonctionnel dans le CT. Lors de la thèse d'Aurélie Bak, un autre rôle de la protéine P3 a été mis en évidence. Le motif CPC de la protéine P3 n'est pas impliqué dans la transformation du CT. En revanche, il pourrait jouer un rôle dans le relargage des virions par les usines virales. C'est à dire que ce motif pourrait permettre ou non l'accrochage des particules virales dans les usines virales (Bak, 2013).
Pour conclure cette partie, certaines hypothèses sont énoncées pour expliquer pourquoi le CaMV utilise des structures particulières impliquées dans l'activation de la transmission.
Une des hypothèses est que la formation de CT permettrait d'éviter la dégradation de la protéine P2 par la plante. En effet, la comparaison des cinétiques d'accumulation de la protéine P2 sauvage et de
la protéine P2-94 mutée ne formant pas de CT dans les cellules infectées, a montré que tôt dans l'infection les deux protéines s'accumulent de manière identique. Plus tardivement, le niveau de P2 sauvage reste stable tandis que le niveau de P2-94 chute en étant rapidement indétectable (Khelifa et al., 2007; Nakayashiki et al., 1993). Une autre hypothèse concerne la toxicité présumée de la protéine P2 (Froissart et al., 2004) qui serait stockée dans le CT pour protéger la cellule de sa toxicité. D'autres hypothèses se concentrent sur le phénomène d'activation de la transmission. Par exemple, il serait aussi envisageable que des complexes transmissibles soient toujours présents dans les cellules infectées. Cependant, le fait de former des complexes transmissibles à l'arrivé du puceron est moins coûteux pour le virus qui peut mettre à profit les ressources utilisées dans d'autres activités comme la réplication, le mouvement de cellule à cellule, etc... Il est aussi possible que l'activation de la transmission pourrait être impliquée dans la trans-complémentation des virus à stratégie helper. Récemment, des résultats préliminaires d'une étudiante du laboratoire, Edwige Berthelot, indiquent que le phénomène de l'activation de la transmission s'applique aussi sur le
Turnip mosaic virus (TuMV) qui est un potyvirus totalement différent du CaMV sauf au niveau de
sa transmission qui suit la stratégie helper. E. Berthelot a montré que le TuMV réagirait à la présence du vecteur en formant des oligomères de la protéine HC-Pro (la protéine assistante de la transmission pour ce virus) qui pourraient être impliqués dans l'activation de la transmission du TuMV. Ces résultats suggèrent que d'autres virus que le CaMV peuvent utiliser l'activation de la transmission pour augmenter leur chance d'être propagés. Cependant, il n'a pas encore été défini si ce phénomène est spécifique des virus à stratégie helper ou applicable aux virus utilisant la stratégie capside. Si c'est le cas, cela pourrait avoir une importance agronomique ainsi que des répercussions sur la virologie en général.
3. Projet de recherche
La forte représentation des phytovirus dans l'environnement a des conséquences sur les cultures en causant des dommages considérables ce qui a pour impact une baisse de la productivité. Actuellement, nous ne disposons d'aucun traitement chimique qui permette de lutter directement contre les virus. Des stratégies de lutte basées sur la biologie des virus ont donc été mises en place. Pour cela, des méthodes prophylactiques comme le contrôle qualité des semences et du terrain, la réduction de la dissémination des virus en utilisant des traitements phytosanitaires chimiques contre les vecteurs aériens, sont utilisées pour lutter contre les maladies provoqués par les phytovirus. Cependant, l'utilisation d'insecticide est néfaste pour l'environnement et inefficace contre les virus non circulants. En plus des méthodes prophylactiques, il existe aussi la sélection de variétés de plantes naturellement résistantes à certains phytovirus ou la création de plantes modifiées par génie génétique, conférant une protection partielle puisque ces résistances peuvent être contournées par les souches de phytovirus ayant subi des modifications génomiques. Le développement de ces plantes est complexe et demande un temps considérable. Une lutte alternative serait d'arrêter de manière biologique la transmission des virus par leur vecteur. Pour cela, il est donc primordial de comprendre les mécanismes de transmission pour combattre les épidémies liées aux virus de plante. Dans le passé, les recherches sur la transmission étaient plutôt centrées sur les virus et leurs vecteurs. Depuis peu de temps, la compréhension des mécanismes de transmission inclut l'hôte végétal, par exemple (Drucker et al., 2002). Cette approche a permis la découverte du phénomène d'activation de la transmission chez le CaMV qui pourrait être appliqué à d'autres virus de plante et permettre à plus long terme, le développement de stratégies d’inhibition au niveau de certaines de étapes de la transmission par vecteurs. Actuellement, nous disposons d'un modèle purement descriptif : l'équipe d'accueil a identifié les différentes étapes de la transformation du CT seulement de manière phénotypique et aucun interactant de l'hôte n'est connu mise à part la tubuline. Une des suites du projet de recherche de l'équipe, auquel mon travail participe, est l'identification de facteurs de l'hôte pouvant interagir avec les protéines du CT. Pour cela, nous avons choisi différentes approches de biochimie et de génétique.
La première approche est purement biochimique et consiste à purifier le CT et analyser son contenu par protéomique. Des résultats préliminaires avaient été obtenus par Aurélie Bak mais la méthode de purification n'était pas encore mise au point. Nous souhaitons donc améliorer la purification et analyser le contenu des CT pour déterminer les protéines de l'hôte pouvant interagir avec les CT. La seconde approche est aussi biochimique et consiste à exprimer la protéine P2 étiquetée avec la
Green Fluorescent Protein (GFP) dans A. thaliana pour la purifier avec ses partenaires cellulaires
par une technique de purification par affinité, le GFP-Trap. L'identification de ces facteurs cellulaires s'effectuerait comme pour la première approche par protéomique.
En parallèle, la troisième approche que nous développons, est un criblage génétique, basé sur la technique de Duolink® qui est une méthode immunologique permettant dans notre cas la détection des interactions directement in planta, entre les protéines d'une banque d'ADNc d'A. thaliana et la protéine P2.
PARTIE 1 :
Recherche de partenaires du Corps à
Transmission (CT) par purification en gradient
discontinu
Pour purifier des complexes de protéines avec ses partenaires ex-planta, nous pouvons utiliser des méthodes de biochimie telles que la centrifugation différentielle, par gradient, la filtration, différentes méthodes de chromatographie, etc... L'avantage de ces approches, qui permettent la purification des simples complexes de protéines et celle des organites, est qu'elles ne nécessitent aucune modification de la protéine d'intérêt. C'est donc dans un premier temps la plus simple à mettre en œuvre. Puisque les CT sont des inclusions, nous avons choisi de réaliser une méthode de purification basée sur la technique de purification des viroplasmes décrite par Al Ani et al (Al Ani et al., 1980) qui utilise la centrifugation en gradient discontinu de saccharose.
L'objectif était de purifier des CT pour identifier des facteurs de plante associés à ceux-ci par des techniques de protéomique. La purification des CT du CaMV sauvage s'est avérée impossible car les CT sont instables. En effet, les CT se dissocient lors de l'homogénéisation des tissus et la protéine P2 précipite sous forme de paracristaux (Drucker et al., 2002; Hébrard et al., 2001). Nous avons donc eu recours au mutant CaMV-P2 TC (TC) qui possède une insertion de 7 acides aminés incluant un tag tétracystéines (Griffin et al., 1998) à la position 100 de la protéine P2. Le mutant TC forme des CT (CT-TC) sphériques d'environ 1-2 µm de diamètre (Martinière et al., 2013) beaucoup plus stables que les CT sauvages. Le virus muté est non-transmissible mais conserve quelques unes des caractéristiques principales du CT comme l'entrée de tubuline dans le CT lors d'un stress. Nous avons profité de la stabilité du mutant TC pour tenter de purifier les CT-TC.
1. Mise au point de la purification des CT-TC
La purification des CT-TC est réalisée à partir de broyat de feuilles d'A. thaliana infectée par le CaMV soit TC soit par un second mutant, le mutant Cabb-B-JI ΔP2 (ΔP2) qui possède une délétion du gène P2 sur le génome du CaMV. Cette délétion entraîne naturellement une incapacité de formation des CT (Fig.9D) (Martinière et al., 2009a). Le mutant ΔP2 est un témoin pour estimer la qualité de purification des CT-TC puisque la seule différence entre « TC » et « ΔP2 » est l'absence de la protéine P2 et donc des CT. En comparant des purifications à partir de « TC » et « ΔP2 », nous pouvons faire de la protéomique différentielle en visualisant, si il y en a, des différences dans la composition protéique des purifications qui devraient être dues à la protéine P2. En effet, les éventuelles protéines supplémentaires observables uniquement pour « TC » pourraient être des partenaires cellulaires liés au CT.
Dans la figure 9, nous montrons une purification de CT-TC optimisée à partir du protocole décrit par Al.Ani (Al Ani et al., 1980). Un schéma de la purification présentant les différentes étapes ainsi que les récupérations des fractions aliquotes est placé dans la partie 4.5 du Matériel et Méthodes. Nous avons analysé les différentes fractions de purification par SDS-PAGE colorés en bleu de Coomassie pour visualiser les profils protéiques totaux et par Western Blots pour révéler la protéine P2 marqueur des CT-TC et la protéine P6 marqueur des usines virales. Les profils protéiques en bleu de Coomassie sont très similaires pour les deux purifications (Fig.9A et C). Les Western Blots révèlent que les deux purifications contiennent des quantités semblables de protéine P6 (62 KDa théorique) ainsi qu'un enrichissement de celle-ci au cours de la purification (Fig.9B et 9D). En revanche, seule la purification TC montre une bande pour la protéine P2 (18 KDa théorique) qui est aussi enrichie dans les dernières fractions (Fig.9B). Au niveau de la protéine P6, nous observons des bandes entre 40 et 70 KDa qui sont, soit des produits de clivage, soit des formes dégradées de la protéine P6. La similarité des profils protéiques entre « TC » et « ΔP2 » suggère que la plupart des protéines visibles sont issues des usines virales ou des protéines contaminantes. Une étape supplémentaire de filtration 5 µm par centrifugation a été introduite pour retenir les usines virales (souvent de taille supérieure aux CT du mutant TC) sur le filtre et laisser passer les CT. Or, nous n'observons aucune différence sur la rétention des usines virales ce qui montre que les CT sont pour une bonne partie agrégés entre-eux et que les usines virales sont partiellement retenues. Une différence d'enrichissement est observable entre les fractions aliquotes 5F, 5E et 7F, 7E (Fig.9E). Il y a une perte d'enrichissement d'environ 21 % entre la fraction aliquote 5F et 7F et de 50 % entre la fraction aliquote 5E et 7E. Il y a probablement eu une solubilisation partielle des protéines qui sont restées dans le surnageant.
2. Conclusion
Nous avons essayé plusieurs modifications du protocole de base proposé par (Al Ani et al., 1980) pour obtenir en quantité et en pureté convenables des CT-TC. Cependant le protocole optimisé qui est présenté en (Fig.9) montre toujours la présence de beaucoup de protéines contaminantes. Nous avons tenté d'autres modifications par exemple, d'utiliser un composé, le Ludox HS-40, qui est un milieu aqueux contenant des particules de silice permettant de réaliser des gradients de densité (Xu et al., 2010) et de séparer les CT-TC des autres inclusions et organites par centrifugation à densité isopycnique. Or nous avons obtenu les mêmes profils protéiques qu' avec l'utilisation du gradient discontinu de saccharose (données non présentées) et donc pas d'amélioration de la purification : la densité des CT était trop grande pour les retenir. Une alternative que nous n'avons pas testé serait d'utiliser des gradients en CsCl2, qui présentent une densité apparente plus importante que le saccharose.
Tous nos essais de purification n'ont été que partiels. Malheureusement, aucune des différentes modifications apportées au protocole n’ont permis d’améliorer la purification.