Sélection et génomique de souches naturelles
provenant de fromages du Québec. Génomique
comparative de Staphylococcus equorum.
Mémoire
Gabrielle Jaquemet
Maîtrise en sciences des aliments - microbiologie alimentaire - avec mémoire
Maître ès sciences (M. Sc.)
Sélection et génomique de souches naturelles provenant
de fromages du Québec
Génomique comparative de Staphylococcus equorum
Mémoire
Gabrielle Jaquemet
Sous la direction de :
Steve Labrie, directeur de recherche
Éric Dugat-Bony, codirecteur de recherche
Résumé
Les souches microbiennes naturelles des fromages sont souvent caractérisées pour étudier leur contribution au développement des propriétés sensorielles (goût, odeur, texture) des fromages affinés. Une meilleure compréhension de ce rôle lors de l’affinage du fromage est essentielle afin de mieux contrôler la qualité de ceux-ci. Peu d’analyses génomiques en détails ont été effectuées sur les microorganismes de la microflore naturelle des fromages (microorganismes non inoculés), alors qu’un plus grand intérêt est porté sur les ferments inoculés. La caractérisation du génome complet de souches bactériennes provenant du terroir québécois permet d’en révéler le contenu en gènes et de prédire les voies métaboliques associées. Ceci permet également d’établir l’apport individuel de celles-ci à la production de composés aromatiques.
Dans le cadre de ce travail, le génome complet de quatre souches Staphylococcus equorum ont été séquencés. Ensuite, une analyse détaillée du génome de ces quatre souches a été réalisée en effectuant l’assemblage et l’annotation fonctionnelle des ~2700 gènes prédits dans chacune des souches à l’étude. Des gènes impliqués potentiellement dans la protéolyse, la lipolyse et la dégradation du lactose, ont été retrouvés dans toutes les souches de S. equorum, révélant leur potentiel métabolique important dans le fromage. Plusieurs attributs intéressants des S. equorum ont également été identifiés par des analyses de génomique comparative. D’abord, la relation entre le regroupement phylogénétique des souches avec leurs sources d’isolement indique une possibilité d’adaptation des souches à leur niche écologique. La présence de gènes uniques ou peu partagés est aussi une caractéristique identifiable dans les génomes des souches étudiées et pouvant avoir un impact sur les métabolismes des souches. La caractérisation du génome de souches de S. equorum et les analyses phylogénomiques fournissent de nouvelles informations sur son rôle dans le fromage et des indices sur son potentiel métabolique. Les données génomiques obtenues permettront, lors de validations futures, de sélectionner des souches ayant des propriétés désirables en fonction des variétés fromagères afin d’obtenir des fromages de qualité optimale.
Abstract
The natural microbiota of cheese has often been characterized to study their potential participation in the development of sensorial properties (taste, odour, texture) of the ripened cheeses. A better understanding of their role during cheese ripening is therefore essential in order to have a better control of its quality. Few in-depth genomic analyzes have been carried out on microorganisms of the natural microflora of cheese (non-inoculated microorganisms), while there is a greater interest for inoculated ferments. The genes possessed by bacterial strains of the Quebec terroir can be revealed by the characterization of their complete genome, leading to the prediction of the associated metabolic pathways. This also allows the establishment of the individual contribution of each strain to the production of aromatic compounds.
As part of this work, the complete genome of four strains of Staphylococcus equorum were sequenced. Then, a detailed analysis of the genome of these four strains was completed by their assembly and the functional annotation of the ~ 2700 genes predicted in each of the studied strains. Genes potentially implicated in proteolysis, lipolysis and lactose degradation, were found in all S.
equorum strains, revealing their potential metabolisms important for cheese. Several interesting
attributes of S. equorum were also identified by comparative genomic analyses. First, the relation in between the phylogenetic grouping and the source of isolation of the strains, indicates a possible adaptation of the strains to their ecological niche. The presence of unique or barely shared genes is also a distinguishable characteristic of the studied strains and can have an impact on the metabolisms of the strains. The characterization of the genome of S. equorum strains and the phylogenomic analyzes have provided new information on their role in cheese and clues about their metabolic potential. The genomic data collected will allow during future validations the selection of strains with desirable properties in function of cheese variety to yield cheeses of optimal quality.
Table des matières
Résumé ... ii
Abstract ... iii
Table des matières ... iv
Liste des figures ... vi
Liste des tableaux ... vii
Liste des abréviations, sigles, acronymes ... ix
Remerciements ... xii
Avant-propos ... xiii
Introduction ... 1
Chapitre 1 - Revue de littérature ... 3
1.1 Le fromage ... 3
1.1.1 Les variétés de fromage ... 3
1.1.2 Défi de la constance du goût du fromage ... 3
1.2 Les microorganismes de l’écosystème du fromage ... 4
1.2.1 Les ferments ... 6
1.2.2 Les souches de la microflore naturelle du fromage ... 9
1.3 Méthodes d’analyse sur microbiote du fromage ... 12
1.3.1 Microbiologie classique – Méthodes dépendant de la culture des microorganismes ... 13
1.3.2 Méthodes moléculaires - Méthodes indépendantes de la culture des microorganismes .. 16
1.3.3 La nécessité d’études génomiques sur les souches naturelles ... 26
1.4 Les analyses génomiques portant sur Staphylococcus sp. ... 27
1.4.1 La génomique comparative des Staphylococcus equorum ... 27
Chapitre 2 - Problématique, hypothèses de recherche et objectifs ... 29
2.1 Problématique ... 29
2.2 Hypothèse de recherche ... 29
Objectif 1 ... 29
Objectif 2 : ... 29
Chapitre 3 - Génomique comparative de souches de Staphylococcus equorum isolées fromage du terroir québécois ... 30
3.1 Résumé ... 30
3.2 Abstract ... 31
3.3 Introduction ... 31
3.4 Results ... 34
3.4.1 Genome assembly and characteristics ... 34
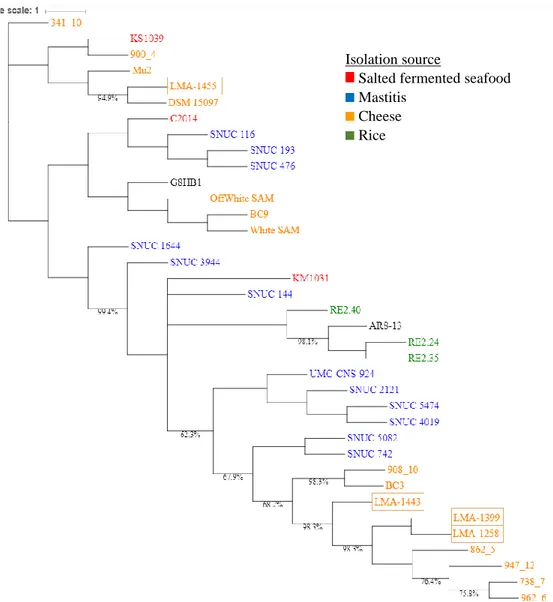
3.4.2 Phylogenomic relationship of Staphylococcus equorum strains ... 34
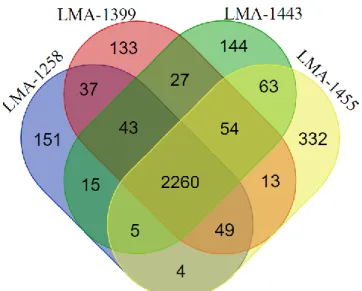
3.4.3 Pan/core-genome analysis ... 35
3.4.4 Prophages ... 37
3.4.5 Unique genes relevant for cheese ripening ... 37
3.5 Discussion ... 46
3.6 Conclusion ... 49
3.7 Material and Methods... 49
3.7.1 Isolation of Staphylococcus equorum strains and DNA extraction ... 49
3.7.2 Library preparation and genome sequencing ... 50
3.7.3 Genome assembly, gene prediction and functional annotation ... 50
3.7.4 Comparative genomic analysis ... 51
3.7.5 Sequence Accession numbers ... 51
Chapitre 4 – Discussion générale ... 52
4.1 Retour sur l’assemblage des génomes et leur annotation fonctionnelle ... 53
4.2 Retour sur la génomique comparative des Staphylococcus equorum ... 54
Conclusion... 57
Bibliographie ... 58
Liste des figures
Figure 1. Phylogenomic tree of Staphylococcus equorum based on a core-genome alignment of the 37 genomes generated by parsnp from the Harvest suite and contsructed with RAxML ... 35
Figure 2. Venn diagram representing the distribution of the gene clusters calculated by PGAP in between the four newly sequenced Staphylococcus equorum genomes. ... 36
Figure 3. Functional annotation and distribution of the unique genes predicted within four strains of S. equorum, using the Clusters of Orthologous Groups (COG v2014) database. ... 37
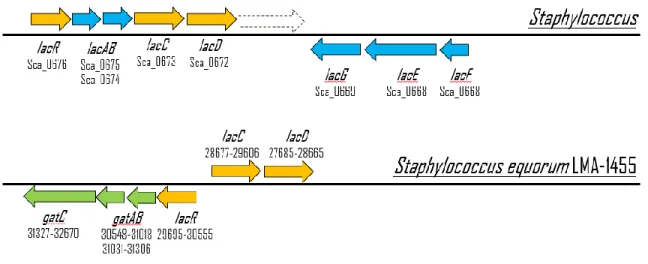
Figure 4. Illustration of the general genetic organization of the genes involved in the tagatose-6-phosphate pathway of lactose degradation for Staphylococcus possessing this pathway and the genetic organization of the genes potentially involved in the tagatose-6-phosphate pathway of lactose degradation for S. equorum LMA-1455. ... 43
Figure S1. Proposed volatile sulfur compound formation through methionine catabolism in Staphylococcus equorum LMA 1258, S. equorum LMA 1399, S. equorum LMA 1443, S. equorum
Liste des tableaux
Tableau 1. Les différents types de ferments d’affinage, la variété de fromage dans laquelle ils sont ajoutés et leurs fonctions principales dans le fromage [12]. ... 9
Table 2. Genomic characteristics of the four Staphylococcus equorum strains genomes investigated in this study. ... 34
Table 3. List of genes encoding membrane associated proteases detected in the four Staphylococcus equorum genomes sequenced in this study. ... 38
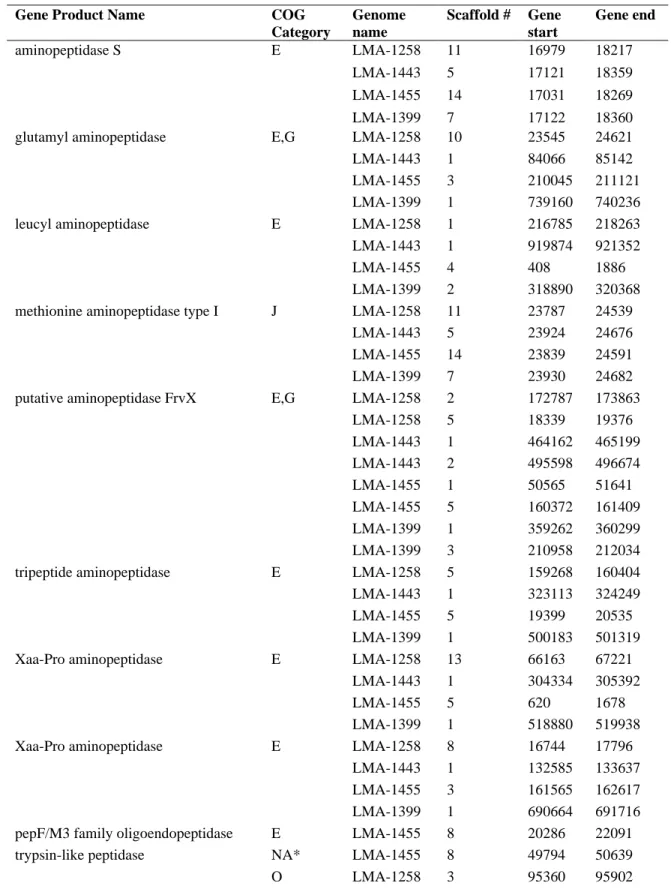
Table 4. List of genes encoding peptidase detected in the four Staphylococcus equorum genomes sequenced in this study. ... 40
Table 5. List of genes encoding lipase and esterase detected in the four Staphylococcus equorum genomes sequenced in this study. ... 42
Table 6. List of the genes encoding enzymes involved in the catabolism of methionine detected in the four Staphylococcus equorum genomes sequenced in this study. ... 45
Table S1. List of the isolates used in the phylogeny of the species Staphylococcus equorum and their isolation sources. ... 82
Table S2. Descriptions of potential functions determined by COG2014 annotation of the unique genes found in the Staphylocohccus equorum LMA strains of this study, categorized by COG categories. A) LMA-1258, B) LMA-1399, C) LMA-1443, D) LMA-1455. Sca. Stands for scaffold. ... 83
Table S3. For the Leloir pathway found in the S. equorum LMA strains, the blastp analysis results for detection of genes and list of all the genes found. ... 99
Table S4. Strain specific genes potentially part of the tagatose-6-P pathway for lactose degradation found in S. equorum LMA. ... 100
Table S5. Results of the blastp analysis against enzymes part of the tagatose pathway for lactose degradation found in other Staphylococcus to confirm their absence in S. equorum LMA strains. 101
Liste des abréviations, sigles, acronymes
DGGE : Denaturing Gradient Gel Electrophoresis - Électrophorèse sur gel avec gradient dénaturant EMA : ethidium monoazide
ITS : Internal transcribed spacer - Espace interne transcrit
MLST : multilocus sequence typing - typage par séquençage multilocus
MRS : (MRS agar) De Man, Rogosa and Sharpe agar - Gélose de Man, Rogosa, Sharpe PCR : polymerase chain reaction - Réaction en chaîne par polymérase
PFGE : Pulsed-field gel electrophoresis - Électrophorèse sur gel en champ pulsé PMA : propidium monoazide - monoazide de propidium
qPCR : PCR quantitative, PCR en temps réel
RuBisCO : ribulose-1,5-bisphosphate carboxylase/oxygénase
SMS : single molecule sequencing - séquençage d’une seule molécule
TGGE : Temperature Gradient Gel Electrophoresis - Électrophorèse sur gel avec gradient de température
Je dédie ce mémoire à mes parents, pour leurs
encouragements tout au long de mes études.
Plus j’étudie la nature, plus je suis émerveillé
par le travail du Créateur. Dans ses plus
petites créatures, Dieu a placé des propriétés
extraordinaires… - Louis Pasteur
Remerciements
Sans le support de nombreuses personnes et organismes subventionnaires, ce mémoire de maitrise n’aurait pas été possible. Tout d’abord, un grand merci à mon directeur, Steve Labrie, qui m’a offert la possibilité d’entreprendre ce projet de recherche très intéressant. Il a su me motiver et restructurer les objectifs du projet au besoin, de manière très honnête et réaliste. J’aimerais aussi remercier Éric Dugat-Bony, le co-directeur de ce projet, qui m’a beaucoup aidée lors de problèmes bio-informatiques et avec la rédaction de l’article scientifique de ce projet, son support et son encouragement ont été très appréciés. Merci à Marie-Hélène Lessard, professionnelle de recherche de l’équipe de recherche, source de conseils scientifiques et d’encouragements. Merci à mes collègues dans le LMA, tout spécialement Vincent Perkins et Annick Raymond-Fleury, qui ont été des collaborateurs importants et motivants lors de l’apprentissage de la bioinformatique.
J’aimerais aussi remercier les organismes subventionnaires pour leur soutien financier : Novalait, CRIBIQ, CSRNG, FRQNT, CTAQ, la Fondation INITIA et l’INAF. Merci aussi à Calcul Québec pour l’accès aux plateformes de calcul bioinformatique Graham et Cedar, ainsi que pour leur support bio-informatique. Merci à Dre Sylvie Turgeon pour la correction de ce mémoire et le temps consacré à son évaluation.
Enfin, un grand merci à Jérémie, Maman, Papa, Marie, Donald et les autres membres de ma famille, pour leur soutien et leurs encouragements lors de la rédaction de ce mémoire. Je suis reconnaissante pour leur capacité à me motiver à livrer mon plein potentiel lors de mes études postsecondaires, me permettant de découvrir l’aspect captivant du monde de la recherche.
Avant-propos
Ce mémoire est divisé en quatre chapitres précèdes d’une introduction au projet de recherche présenté dans ce mémoire. Le premier chapitre est un résumé de la littérature soutenant le sujet de recherche et porte sur la microbiologie de souches microbiennes naturelles présentes dans le fromage du terroir, ainsi que sur différentes méthodes permettant de mieux comprendre leurs rôles dans le fromage, en concluant avec une présentation des connaissances déjà acquises par la génomique comparative. Le deuxième chapitre présente l’hypothèse et les objectifs de recherche de ce projet. Le chapitre trois est
présenté sous la forme d’article scientifique et est rédigé en anglais. Cet article présente les résultats
de l’analyse comparative de génomes de Staphylococcus equorum. L’article sera soumis au journal
BMC Genomics pour publication dès la remise de ce mémoire.
La majorité des travaux effectués en laboratoire, les analyses bio-informatiques ainsi que la rédaction de l’article ont été réalisés par la première auteure de ce mémoire, Gabrielle Jaquemet. Le deuxième auteur est Dr Éric Dugat Bony, co-directeur du projet, qui a participé à la rédaction et à la correction de l’article ainsi qu’aux analyses bio-informatiques. La troisième auteure est Dre Marie-Hélène Lessard, professionnelle de recherche, qui a participé aux corrections de l’article. Le quatrième auteur est Dr Steve Labrie, le directeur de recherche de ce projet; il a obtenu les fonds de recherche, a supervisé le travail de recherche et a fait des corrections pour l’article et le mémoire.
Le quatrième chapitre est une discussion générale du mémoire et les avancements obtenus à la suite des analyses, ainsi que les contraintes rencontrées lors du projet. Les perspectives générales et les avenues de recherche futures sont présentées dans la conclusion de ce mémoire.
Introduction
En 2016, 227 806 tonnes métriques de fromages ont été produits au Québec, ce qui représente plus de la moitié de la production fromagère canadienne la même année [1]. La participation à de nouveaux accords commerciaux internationaux par le Canada [2] dans les dernières années a créé un environnement commercial dynamique et compétitif [3], obligeant l’industrie fromagère québécoise à mettre en place de nouvelles stratégies afin de se démarquer de la concurrence. Les fromages de spécialité d’origine québécoise, qui comprennent les fromages à pâte molle, semi-ferme ou ferme, et affinés en surface [1], répondent à la demande croissante des consommateurs québécois pour des produits de flaveur optimale, de qualité et en provenance du terroir [4]. Les Québécois sont aussi les plus grands producteurs de fromages de spécialité au pays (30 234 tonnes métriques) [5], lesquels sont fréquemment récompensés au niveau national, mais aussi internationalement [6]. Pour maintenir la qualité supérieure de ces variétés de fromages et mettre en avant les caractéristiques liées au terroir québécois, une meilleure compréhension des variables qui influencent les caractéristiques organoleptiques des fromages (plus spécifiquement les microorganismes présents) serait bénéfique pour les fromagers.
Les variétés de fromages peuvent se différencier de multiples façons, par exemple, par la microflore sélectionnée pour l’affinage, par le type de lait utilisé (vache, chèvre, brebis, etc.), par le traitement thermique appliqué au lait (cru, pasteurisé ou thermisé) et par les paramètres utilisés lors de la fabrication (pH, humidité, température d’affinage, concentration de sel…) [7, 8]. La microflore des fromages joue un rôle très important dans le développement des caractéristiques sensorielles uniques des variétés de fromages, telles que la couleur, la flaveur et la texture [9]. Les ferments lactiques et d’affinage (aussi appelés microflore primaire) sont ajoutés pendant la production par le fromager et sont respectivement responsables de l’acidification du lait et du développement des propriétés sensorielles typiques pendant l’affinage. La microflore secondaire (souches naturelles) qui se développe dans les fromages est très complexe et sa diversité évolue constamment lors de la production et l’affinage des fromages [10]. Elle provient naturellement du lait et de l’environnement. Cette microflore secondaire se compose de bactéries lactiques (Non Starter Lactic Acid Bacteria; NSLAB), ainsi que d’autres bactéries, de levures et de moisissures qui sont présentes en différentes proportions selon les variétés de fromages. Même en étant moins abondante que la microflore inoculée (primaire) au début de la production fromagère, la microflore non inoculée (dite « naturelle » ou « secondaire ») devient progressivement dominante lors de l’affinage de plusieurs variétés de fromages. Les enzymes produites par la microflore secondaire participent à la production de composés aromatiques [11] et contribuent à la flaveur du fromage [8, 12]. Pour comprendre les rôles
spécifiques de la microflore secondaire lors de l’affinage et sur la typicité des fromages, il est nécessaire de l’identifier, puis de la caractériser [13]. La compréhension du rôle de l’écosystème microbien secondaire des fromages pourra ainsi améliorer le contrôle de l’affinage de ceux-ci.
Étant moins dominantes en début d’affinage et plus difficiles à analyser [14], encore peu d’espèces de la microflore secondaire ont été analysées en profondeur comparativement à l’effort de caractérisation réalisé pour la microflore primaire. Pourtant, plusieurs études phénotypiques démontrent que la microflore secondaire participe au développement des propriétés sensorielles (goût, odeur, texture) des fromages et serait responsable de la formation de notes aromatiques spécifiques [15]. Des analyses du génome complet de quelques genres bactériens ont permis d’identifier des gènes participant potentiellement dans les voies métaboliques menant à la génération de composés aromatiques, tels que des composés volatils soufrés qui sont des composés clés dans la flaveur de plusieurs variétés de fromages [16–18]. Lors de la réalisation des présents travaux de maîtrise, quatre génomes complets de souches de Staphylococcus equorum seront rendus disponibles dans les bases de données publiques. Également, l’analyse fonctionnelle du génome de ces souches améliorera la compréhension de la participation des membres de cette espèce dans la production potentielle de composés de flaveur dans le fromage par leurs activités métaboliques. Éventuellement, ces connaissances contribueront à la sélection de souches et l’élaboration de ferments d’affinage, dans le but d’optimiser la production de fromage québécois de qualité.
Chapitre 1 - Revue de littérature
1.1 Le fromage1.1.1 Les variétés de fromage
Connu à travers le monde sous de multiples formes, le fromage est à la base le résultat de la coagulation du lait. Les variétés de fromage multiples sont dues à de nombreux facteurs, parmi lesquels, les plus importants sont le type de lait transformé, la méthode de production, l’environnement et son microbiote. Le lait peut provenir de différents mammifères incluant, parmi les plus fréquents, la vache, la chèvre et la brebis. Lors des premières étapes de production, le lait peut être laissé cru ou traité thermiquement de deux manières différentes, pasteurisé ou thermisé. La matrice obtenue est acidifiée (acidification de type lactique, présure ou mixte), ce qui mène à la coagulation [19]. Des étapes de découpe, de synérèse et d’égouttage permettent la formation d’un caillé. En fonction du type de fromage, d’autres étapes de transformation sont utilisées parmi lesquelles on retrouve le moulage, la cuisson, le retournement, le pressage et le salage [7, 20]. Certains fromages peuvent être consommés immédiatement, tandis que d’autres nécessitent un temps d’affinage, qui peut normalement varier de quelques jours à plusieurs années. En somme, la production de tout fromage conduit à la déshydratation du lait et à l’augmentation des teneurs en gras et caséines par un facteur de six à douze fois [7]. Les variations réalisées pour chacune de ces étapes par les fromagers mènent à de nombreuses variétés de fromage. Parmi les fromages de spécialité, on peut retrouver des fromages à pâte molle, à pâte semi-ferme, à pâte ferme ou persillée et ayant une croûte soit fleurie, naturelle ou lavée [19]. Les particularités de ces fromages résultent de leur méthode de production, du contrôle de l’affinage, de la microflore présente dans le fromage et des composants du lait utilisé [21].
1.1.2 Défi de la constance du goût du fromage
L’affinage du fromage, d’une durée variable, est une étape essentielle pour le développement des caractéristiques organoleptiques des fromages [21]. Entre autres, la modification des interactions entre les protéines et l’eau du fromage induite par le sel a un effet sur les propriétés organoleptiques du fromage, sur son affinage et sur sa qualité [21, 22]. Le sel a aussi un effet inhibiteur sur la croissance de certains microorganismes et peut être utilisé pour le contrôle de la microflore lors de l’affinage. En fonction de la variété de fromage, le sel peut être ajouté dans la masse ou en surface seulement, ce qui amène une modulation différente de l’écosystème du fromage [23]. L’affinage optimal de chaque type de fromage requiert des intervalles de températures et d’humidité relative contrôlés et spécifiques, ainsi qu’une composition des gaz de l’atmosphère stable. Ces paramètres
dans le hâloir d’affinage ont un impact important sur la texture, la couleur et l’odeur du fromage [21, 24, 25]. Plusieurs études ont démontré que ces paramètres ont un impact direct sur la croissance et l’activité métabolique des microorganismes [24–26], ce qui mène à la synthèse de produits métaboliques influençant, une fois de plus, le goût et la qualité du fromage produit [4, 25, 27]. L’emballage non seulement permet la vente au détail, mais protège le fromage de la croissance de bactéries indésirables en permettant la poursuite normale de l’affinage [21, 28]. Le fromage et son microbiote étant sensibles aux changements des paramètres d’affinage, il est important que ces paramètres soient les plus constants possible et adaptés à la microflore à favoriser.
Il est important pour les fromagers de conserver et même d’accroître leur part de marché. La constance du goût du fromage contribue assurément à maintenir cette part de marché [29]. Afin de se distinguer parmi le large éventail de fromages disponibles, et de rencontrer les attentes des consommateurs fidèles, un fromage doit être non seulement de bonne qualité et sécuritaire, mais aussi posséder une homogénéité de flaveur d’un lot à l’autre [21]. De plus, les acheteurs vont sélectionner des produits ayant un profil aromatique intéressant, contrairement à un produit fade ne procurant aucun plaisir [30]. Une grande partie du contrôle de la nature dynamique du fromage et l’optimisation de sa flaveur se fait par l’entremise des ferments ajoutés et de la microflore naturelle se développant dans le produit. Les ferments lactiques ont un rôle dans le goût de base du fromage. Afin de diversifier la gamme aromatique du fromage, l’utilisation des ferments d’affinage et la présence de la microflore naturelle, responsables des goûts distincts de fromage artisanaux, sont avantageuses [31]. Une meilleure compréhension des métabolismes associés à la production de flaveur par les souches sélectionnées permettra de mieux les contrôler dans le fromage et potentiellement favoriser le développement de produits innovants. Les attentes des consommateurs pourront être surpassé lorsque les caractéristiques qui rendent le fromage agréable en bouche sont produites de façon constantes. 1.2 Les microorganismes de l’écosystème du fromage
Tel qu’indiqué précédemment l’origine du lait, les méthodes de production, et l’environnement de production sont tous des facteurs qui mènent à la présence de microorganismes spécifiques dans chaque fromage, contribuant à sa signature spécifique [32]. Dans l’histoire humaine, la production d’aliments fermentés a exploité les caractéristiques des microorganismes pour leur contribution à l’arôme de ces produits, sans que leur présence soit connue [29]. Par contre, les défauts du fromage sont souvent le résultat de la contamination du produit par des microorganismes indésirables, par exemple une décoloration bleue de fromages pourrait être la résultante d’une contamination par
des interactions entre les microorganismes de l’écosystème pendant l’affinage [29]. Par exemple, la présence de bactéries coliformes mène au gonflement du produit par la production de gaz et à la production de saveur amère [34]. L’ajout de ferments contenant des souches, du genre Penicillium, peut créer un déséquilibre du système protéolytique présent dans un fromage, produisant des peptides amers, nécessitant l’identification et l’élimination de ces souches de ferments ajoutés au fromage, afin d’obtenir un fromage sans amertume
[3, 35].
Également, un déséquilibre de la communauté microbienne peut provoquer plusieurs défauts, dont l’acidification trop importante qui engendre des défauts de texture, au craquelage de la surface et à la synérèse du fromage [36]. Ainsi un excellent contrôle des facteurs menant à l’équilibre du microbiote du fromage est nécessaire. Également, l’étude des interactions entre les microorganismes et la matrice fromagère est primordiale afin de déterminer le rôle précis de chacun des membres de l’écosystème et sa sensibilité aux différents paramètres de fabrication.Le fromage possède une composition physicochimique très variée selon les variétés, offrant ainsi la possibilité d’abriter simultanément de nombreuses espèces de microorganismes [37]. Généralement, dans le fromage, on retrouve les phylums bactériens Firmicutes, Proteobacteria, Actinobacteria,
Bacteroidetes et les phylums de levures Ascomycota, Zygomycota, Basidiomycota et des
champignons filamenteux [38–40]. Cette microflore générale pourra varier en diversité et en proportion en fonction de la région où est produit le fromage [41]. Par exemple, dans une étude sur des fromages mexicains, les genres bactériens les plus abondants étaient des Streptococcus,
Lactococcus, Lactobacillus, et Marinomonas, les levures Galactomyces, Saccharomyces et Scheffersomyces [39]. Les genres Corynebacterium, Leucobacter, Glutamicibacter, Enterococcus, Lactobacillus, Lactococcus, Streptococcus, Halomonas, Psychrobacter et Pseudoalteromonas furent
retrouvés dans tous les échantillons d’une étude sur douze fromages français [38]. Une étude sur des fromages irlandais trouva qu’en plus des Lactococcus dominants, les genres Lactobacillus,
Leuconostoc, Pseudomonas, Psychrobacter, Staphylococcus, Glutamicibacter et Faecalibacterium
étaient retrouvés dans tous les types de fromages, qu’ils soient à pâte molle, mi-dure ou dure [10]. Globalement, certains genres microbiens sont retrouvés dans tous les fromages, tels que les
Lactococcus et Lactobacillus. Les autres genres de la communauté microbienne du fromage vont être
plus ou moins nombreux dans les différents types de fromage. Certaines espèces de microorganismes semblent plus adaptées à certaines variétés de fromage comme Mycetocola reblochoni dans le Reblochon [42] et Mucor lanceolatus dans le fromage Saint-Nectaire [38, 43]. Dans la croûte du fromage, plusieurs genres sont fréquemment détectés, telles que les levures Debaryomyces, Yarrowia,
Streptococcus, les actinobactéries Brevibacterium, Corynebacterium et Glutamicibacter et les
protéobactéries Psychrobacter, Halomonas, Pseudoalteromonas et Vibrio [40]. Le lien entre la signature spécifique de l’écosystème microbien d’un fromage et la qualité de celui-ci est encore difficile à établir, mais certaines espèces indésirables sont toutefois facilement associées à des produits de mauvaise qualité.
1.2.1 Les ferments
L’écosystème microbien évolue constamment lors du procédé de fabrication fromagère. Le lait pasteurisé, l’ingrédient principal, contient relativement peu de microorganismes viables comparativement à la concentration finale de microorganismes retrouvés dans le fromage [44]. Le lait, soit cru ou pasteurisé, est généralement dominé par des bactéries lactiques, des Pseudomonas, des Acinetobacter et des Staphylococcus [41, 44]. La production et l’affinage d’un fromage impliquent très souvent l’ajout de ferments et de cultures d’appoint, mais aussi l’action de microorganismes provenant de sources environnementales (le microbiote naturel) [45, 46]. Ainsi l’abondance des microorganismes dans un fromage affiné sont beaucoup plus grandes comparativement à la microflore du lait [47]. Pendant l’affinage du fromage, les ratios des différentes espèces évoluent progressivement. Après une croissance rapide pour atteindre un seuil limite, les bactéries lactiques meurent et lysent graduellement lors de l’affinage. Dans plusieurs fromages de spécialité, les levures et moisissures colonisent la surface des fromages, permettant une désacidification progressive du caillé par la consommation de l’acide lactique et la production d’ammoniac [48]. Ceci favorise le développement de bactéries sensibles aux acides et tolérantes au sel [40]. Les ferments d’affinage non-lactiques et le microbiote naturel qui sont peu abondants en début de transformation, lors de la croissance des ferments lactiques, vont croître lors du déclin des bactéries acidifiantes [15], devenant ainsi les microorganismes viables dominants [49]. Dans chaque type de fromage, l’évolution de l’équilibre microbien sera différente [20, 46]. Par exemple pour un fromage à croûte lavée (smear ripened cheese), durant le stade précoce d’affinage, les bactéries lactiques sont présentes en grande concentration. Ensuite, à mi-parcours, la croissance de
Staphylococcus saprophyticus est importante alors qu’à la fin de l’affinage, différentes espèces de Corynebacterium et Agrococcus dominent [45]. Pour un fromage de type Cheddar affiné en surface,
les bactéries lactiques dominent aussi au début, puis à la fin de l’affinage (30 jours), Glutamicibacter
Les caractéristiques organoleptiques fromagères sont fortement influencées par l’écosystème microbien présent dans le fromage [39]. Le rôle et l’origine des microorganismes dans le fromage permettent de les répartir dans deux catégories, les ferments et le microbiote naturel. Les ferments sont constitués par l’ensemble des microorganismes qui sont ajoutés au lait, donc les ferments lactiques et les ferments d’affinage. Le microbiote naturel, quant à lui, est adventice et n’est pas volontairement introduit dans le lait; il s’y retrouve naturellement [11, 13]. Les ferments et le microbiote naturel forment un écosystème diversifié et dynamique [11] qui va influencer la fabrication du fromage de multiples façons.
1.2.1.1 Les ferments lactiques
L’ajout de ferments en début de production fromagère est très commun. La production d’acide lactique par les ferments lactiques ainsi que la présence de présure fait précipiter les caséines ce qui forme un caillé. Les ferments le plus souvent utilisés appartiennent aux genres Lactococcus,
Lactobacillus, Streptococcus [9]. Les ferments lactiques métabolisent plusieurs composants retrouvés
dans le lait (lactose, galactose, citrate, azote, caséines, etc.) [51] et produisent principalement de l’acide lactique, de l’acétaldéhyde et du diacétyle [52]. Les galactosidases de plusieurs bactéries lactiques produisent, au travers du métabolisme du lactose, selon qu’elles soient homofermentaires ou hétérofermentaires, des quantités variables de molécules de lactate [14, 51]. Leuconostoc sp. et certains Lc. lactis ssp. lactis métabolisent le citrate en diacétyle et en acétate, des composés aromatiques importants qui confèrent au fromage des arômes lactiques (beurre), fruités et floraux [53]. Lactococcus lactis peut hydrolyser les caséines en acides aminés et en courts peptides. Ces molécules sont importantes pour la croissance des microorganismes et sont précurseurs des composés aromatiques des fromages [31, 51, 54]. Des lipases et des estérases sont aussi synthétisées par les ferments lactiques [51] et contribuent à la lipolyse initiale [14]. Ayant déjà fait l’objet de multiples études, le rôle des espèces du microbiote primaire (ferments lactiques) est relativement bien compris [3, 11, 14, 55–58].
1.2.1.2 Les ferments d’affinage
Les caractéristiques organoleptiques des fromages sont le plus souvent modulées par les ferments d’affinage [15]. Auparavant, l’utilisation de « ferments d’affinage » se limitait aux moisissures, et était souvent considérée comme superflue puisque tous les fromages étaient produits à partir de lait cru, ce qui permettait en quelque sorte d’obtenir un ensemencement naturel du fromage via la matière première [12]. Avec l’avènement de la pasteurisation et des pratiques de travail plus hygiéniques, les fromages ont perdu une partie de cette microflore naturelle et aussi une partie de leurs propriétés
sensorielles avec elle [4, 59]. Pour remédier à ce problème, les fromagers ont commencé à intégrer l’utilisation des ferments d’affinage de façon contrôlée dans leur procédé de fabrication. Ces ferments sont composés de microorganismes provenant, à l’origine, de la microflore naturelle de fromages au lait cru, sélectionnés pour leur contribution à la formation de caractéristiques organoleptiques intéressantes lors l’affinage du fromage [12]. Ils sont inoculés dans le lait ou sur la surface du fromage [14]. Leur croissance et leur activité métabolique seront pleinement déployées seulement lors de l’affinage, quand cette action est nécessaire. Les ferments d’affinage peuvent être soit des levures, telles que Geotrichum candidum, Debaryomyces hansenii et Klyuveromyces lactis, soit des moisissures, telles que Penicillium camemberti et P. roqueforti, soit des bactéries, les principales appartenant aux familles des Brevibacteriaceae (Brevibacterium sp.), Micrococacceae (Micrococcus sp. et Staphylococcus sp.) et des Corynebacteriaceae (Glutamicibacter sp. (anciennement
Arthrobacter), Brachybacterium sp. et Corynebacterium sp.) [60] et les lactobacilles
hétérofermentaires [12]. Les microorganismes ajoutés varient selon la variété de fromage à produire et ont également différentes fonctions (Tableau 1) [12]. Par exemple, la couleur orange produite par
Brevibacterium linens dans le fromage est fréquemment exploitée pour colorer les fromages à croûte
lavée [12]. Dans le Cheddar, plusieurs lactobacilles peuvent être utilisés comme ferment d’affinage d’appoint (adjunct culture). L’augmentation de la protéolyse qui en résulte crée une flaveur boisée et de noisette plus prononcée, des caractéristiques organoleptiques désirables pour le Cheddar vieilli [61–63]. Utilisées fréquemment en fromagerie pour la formation d’une mince croûte blanche, les
Geotrichum candidum contribuent aussi à la flaveur du fromage par son activité protéolytique [64].
Cette levure a aussi une activité alcalinisante essentielle. Souvent développés à partir de microorganismes naturellement présents dans le fromage, les ferments d’affinage ont un rôle très important dans la production du fromage.
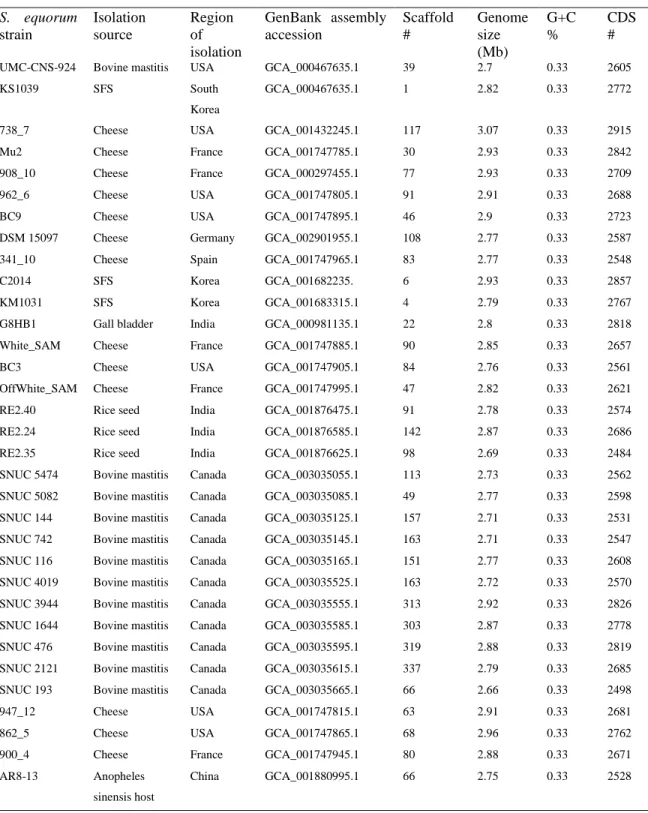
Tableau 1. Les différents types de ferments d’affinage, la variété de fromage dans laquelle ils sont ajoutés et leurs fonctions principales dans le fromage [12].
Microorganismes Type de fromage Fonctions principales et activités métaboliques Levures Fromages affinés en
surface
Activité lipolytique
Composés aromatiques produits: éthanol, aldéhydes, esters, ammoniac, acides cétoniques
Favorisent la croissance d’autres microorganismes (G. candidum)
Moisissures Fromage affiné en surface
Fromages de chèvre
Confèrent une apparence duveteuse aux fromages Alcalinisation
Activité protéolytique et lipolytique
Composés aromatiques produits : éthanol, aldéhydes, esters, ammoniac, méthylcétones, composés volatiles Interactions avec autres microorganismes
Corynébactéries et staphylocoques
Fromages à croûte lavée et pâte molle/ mi-ferme
Activités protéolytique, peptidolytique, lipolytique Catabolisme d’acides aminés
Activité antimicrobienne. Propioniobactéries Fromages de type
Suisse
Métabolisme du lactate
Activités protéolytique et lipolytique Catabolisme d’acides aminés, Possibles propriétés probiotiques Lactobacilles
hétérofermentaires
Fromages à pâte ferme ou semi-ferme
Activités protéolytique et lipolytique, catabolisme des acides aminés
Certains ont des propriétés probiotiques
1.2.2 Les souches de la microflore naturelle du fromage
La présence de la microflore secondaire dans le fromage est dite « naturelle » car elle n’est pas volontairement introduite par l’humain dans le fromage. Lors de la production du fromage et de l’affinage, des microorganismes sont introduits dans le fromage, les sources pouvant être le lait, les moules, les surfaces, l’équipement utilisé, l’air de l’atelier de production ou les travailleurs [13, 41, 65, 66]. La composition des microorganismes dans le lait varie selon plusieurs facteurs, tels que l’environnement, la saison et l’état physiologique de la vache [41, 65, 67]. Elle provient de sources multiples : du lait lors de la traite (pis de la vache, paille dans le lait, machines à traire) ou lors de son entreposage (tubulures, cuves). La croûte du fromage, étant constamment exposée à l’oxygène et à diverses particules présentes dans l’environnement, possède une microflore distincte de la pâte du fromage [10, 68]. Leur niveau de population est habituellement au maximum de concentration à la fin de l’affinage du fromage, ainsi leur action se manifeste plutôt lors de l’étape d’affinage. Bien que les fonctions précises de cette microflore soient encore mal connues, plusieurs études portant sur ces microorganismes permettent de mieux caractériser l’impact de certaines espèces dans le développement des caractéristiques organoleptiques des fromages de spécialité [4, 56, 65, 69]. En
comprenant mieux les fonctions précises de chaque espèce, nous pourrons avoir une meilleure idée du fonctionnement global du processus d’affinage et de leur impact sur la signature spécifique des fromages [40].
1.2.2.1 Diversité microbienne des souches naturelles
La diversité microbienne du microbiote naturel a été répertoriée dans plusieurs études, afin de déterminer la signature spécifique de plusieurs variétés de fromage [38] et afin d’identifier quels microorganismes seraient potentiellement d’intérêt fonctionnel (ou unique) dans le fromage. Les études démontrent que le microbiote adventice domine la surface du fromage [40], l’une d’elles estimant que 60 % des bactéries présents sur la surface des fromages ne provenaient pas de ferments [13]. N’étant sous aucun contrôle, autre que les facteurs imposés par le fromager lors de la transformation, ce microbiote naturel peut inclure une variété de taxons indéfinis, et même des taxons déjà utilisés comme ferments. Afin d’évaluer l’abondance relative des taxons distincts retrouvés dans les différents fromages, des méthodes de séquençage ont été utilisées. Spécifiquement, le gène codant ARNr 16s est séquencé pour les bactéries, et les ITS de l’ARNr sont séquencé pour les levures. L’abondance relative des genres diffère beaucoup pour chaque variété de fromage. Il a par exemple été estimé à partir de l’étude de trois fromages français de technologies différentes que Debaryomyces pouvait représenter entre 0,23 % [13], 51% et 97% [38] de la flore fongique. Dans certains fromages, quelques genres sont présents en grande quantité, laissant supposer un rôle important. La levure
Cyberlindnera, représente 77 % de la flore fongique dans un fromage québécois pasteurisé à croûte
lavée (Raymond-Fleury, Communication personnelle). L’abondance relative bactérienne des
Psychrobacter dans un fromage français à pâte molle à croute lavée était de 66 % [38], et dans un
fromage québécois thermisés à croûte lavée, les Staphylococcus représentent 67 % de la flore bactérienne (Raymond-Fleury, Communication personnelle). Ces microorganismes différents des ferments traditionnels, mais toutefois présents dans l’écosystème, ont le potentiel de contribuer à la qualité du produit.
1.2.2.2 Rôle des souches naturelles dans le fromage 1.2.2.2.1 Les levures
Parmi les fromages artisanaux du même type (même croûte, pâte, sorte de lait, procédés de fabrication), il est possible de distinguer des notes aromatiques distinctes développées par l’action du microbiote présent dans les fromages [19]. Le rôle du microbiote secondaire du fromage est encore relativement peu compris, mais certaines études microbiologiques ont démontré quelques rôles et
propriétés intéressants de ce microbiote naturel. Parmi les levures, l’espèce Debaryomyces hansenii domine au début de l’affinage du fromage et a des propriétés d’intérêt pour sa production [50]. Elle a une haute tolérance au sel et une capacité à produire des enzymes protéolytiques et lipolytiques [70]. Cette levure est aussi liée à la production d’acides carboxyliques et des alcools, des composés volatils menant à la formation d’arômes [50]. Une étude utilisant des caillés modèles a révélé qu’elle produit des arômes acides, fruités et de fromage [71]. Elle est maintenant souvent incluse dans des ferments d’affinage. Le genre Pichia, démontrait une activité protéolytique faible, mais pouvait utiliser le lactate et ainsi augmenter le pH [71]. Les levures de ce genre possèdent des activités lipolytiques qui favorisent la formation de composés volatils [72]. Également, l’activité de souches de Rhodotorula dans du fromage de brebis a été analysée. Ce genre est présent au début de l’affinage, mais disparaît lorsque le fromage se déshydrate [73]. Peu de souches Rhodotorula ont la capacité d’assimiler le lactose, mais presque toutes avaient une activité estérasique et un effet alcalinisant, ce qui contribue à augmenter le pH du fromage [73]. La présence de Kazachstania servazzii a été révélée dans des fromages [74] et son rôle d’exhausteur d’arômes a été examiné [75], mais aucune fonction spécifique dans le fromage n’a encore été confirmée [75]. La présence de Cyberlindnera jadinii a été décelée dans des fromages [38, 76] et est analysée pour sa contribution à la saveur du fromage et son potentiel en tant que ferment d’affinage [50]. Par contre encore aucune étude ne lui donne un rôle spécifique [77]. En résumé, plusieurs travaux sont encore nécessaires afin de préciser la contribution (positive, négative ou neutre) de plusieurs espèces de levures dans l’écosystème du fromage.
1.2.2.2.2 Les bactéries
De façon similaire, des bactéries peu connues, mais néanmoins présentes, telles que Halomonas sont présentes dans l’écosystème fromager. La présence de Marinomonas [78], de Cobetia [13, 40, 79, 80] et de Halomonas (dans certains cas en grande abondance) [81] a été détectée plusieurs fois dans différents fromages, toutefois sans aucune association avec les caractéristiques du fromage [81, 82]. Des analyses effectuées sur d’autres bactéries du microbiote secondaire fromager étaient plus développées. Les Psychrobacter peuvent cataboliser des aldéhydes à chaines ramifiées (3 méthyl-butanal) et des cétones [83, 84], précurseurs de nombreux composés aromatiques du fromage. Non seulement ils augmenteraient la diversité des composés aromatiques, mais ils produiraient aussi une relativement grande quantité de composés volatils ayant un seuil olfactif bas, tels que les composés soufrés. Ainsi, ils donneraient au fromage des notes soufrées, fruitées et de « fromage » [84]. Les
Glutamicibacter sont souvent associés à une couleur jaune de la croûte [85, 86]. Ils ont une haute
les produits peuvent contribuer à l’arôme du fromage [87]. L’analyse des caractéristiques dans le fromage de certains genres bactériens du microbiote secondaire est bien amorcée, mais plusieurs questionnements demeurent, même pour les espèces dont le rôle dans le fromage a déjà été partiellement élucidé.
1.2.2.2.2.1 Les bactéries de l’espèce Staphylococcus equorum
Les Staphylococcus equorum sont généralement retrouvés naturellement dans le fromage, faisant partie du microbiote secondaire [46, 88]. Ayant une croissance peu rapide, il a néanmoins été démontré dans une étude portant sur plusieurs fromages que S. equorum peut devenir l’espèce dominante à la fin de la période d’affinage du fromage [89] et donc, contribuer à cet affinage. Les caractéristiques de cette espèce varient, dépendamment des conditions environnementales [90, 91], mais aussi en fonction de la souche [92]. La fermentation de plusieurs hydrates de carbone et d’autres activités enzymatiques sont dépendantes de la souche présente [90, 93–96]. Les activités protéolytiques et lipolytiques sont souvent faibles parmi les S. equorum [97–104]. Cependant plusieurs études ont souligné leur contribution aux caractéristiques organoleptiques du fromage via le catabolisme des acides aminés [90, 93, 97, 105, 106]. Cette formation de composés ayant un effet potentiel sur l’arôme du fromage est encore une fois, majoritairement dépendante de la souche [84, 90, 93, 103, 105–111]. Plusieurs études ont aussi souligné que la présence de cette espèce mène à une désacidification du caillé plus rapide [112–114] et à un fromage avec une meilleure texture [113]. Les
S. equorum sont donc pour toutes ces raisons des candidats prometteurs pour développer de nouveaux
ferments d’affinage [85, 93]. D’un point de vue phénotypique, plusieurs caractéristiques et activités ont pu être attribuées à cette espèce dans le fromage. Cependant peu de données génomiques sont disponibles pour celle-ci. Or, la génomique peut permettre de révéler les voies métaboliques reliées à ces phénotypes, et ainsi permettre de mieux connaitre le fonctionnement des microorganismes. Ces analyses plus approfondies permettent aussi de déceler des phénotypes potentiels non détectables (ou non soupçonnés) en laboratoire. Cependant, aucune étude génomique sur les Staphylococcus equorum isolés de fromages n’a encore été réalisée. Une telle étude nous permettrait de mieux comprendre les caractéristiques de cette espèce en relation avec cet environnement et l’aspect souche-dépendant de celles-ci.
1.3 Méthodes d’analyse sur microbiote du fromage
L’étude des microorganismes dans le fromage a beaucoup évolué lors du siècle dernier. Les premières études de l’écosystème microbien du fromage ont d’abord été réalisées par des méthodes de microbiologie classiques, soit la culture sur milieu de culture et la caractérisation par méthodes
biochimiques. Dans le dernier quart de siècle, les efforts de séquençage de l’ADN et l’émergence de la biologie moléculaire ont permis plusieurs avancées dans la compréhension des caractéristiques particulières des microorganismes. L’optimisation de l’analyse de l’ADN au travers des années, tel que les méthodes de PCR et de séquençage, a permis de recueillir les caractéristiques génomiques de microorganismes [115] provenant de multiples environnements, et ce, de manière efficace et précise. Ces nouvelles données, reliées aux données phénotypiques, nous donnent une vue d’ensemble de leurs rôles potentiels. Les prochaines sections comparent les méthodes classiques de caractérisation des microorganismes et les méthodes moléculaires et font l’état des avantages et des désavantages de chacune de ces approches.
1.3.1 Microbiologie classique – Méthodes dépendantes de la culture des microorganismes
1.3.1.1 Tests phénotypiques
Différentes méthodes de microbiologie classique, telles que des tests phénotypiques et enzymatiques, ont été et continuent d’être utilisées pour caractériser et identifier les microorganismes retrouvés dans le fromage et permettent aussi d’en établir la fonction dans l’écosystème. En visualisant les microorganismes sous microscope, leur morphologie peut être décrite [90, 93, 116, 117]. Pour déterminer la capacité de fermentation de sucres spécifiques (ex : rhamnose, cellobiose, melibiose, mannitol, ribose, raffinose, mannose), il est possible de mesurer la concentration de sucres produites ainsi que le pH lors de la croissance d’espèce dans un milieu de culture (production d’acide) [116]. La présence d’enzymes, par exemple de lipases et de protéases, peut être analysée en détectant l’utilisation de substrat des microorganismes étalés sur des milieux de culture spécifiques [99, 102]. La susceptibilité aux antibiotiques peut être déterminée par la méthode de diffusion sur agar en présence d’antibiotiques d’intérêt [90, 93]. Ces caractéristiques sont utilisées pour différencier des microorganismes étudiés, par exemple la différenciation d’espèces de Lactobacillus fut réalisée en déterminant pour chaque souche les capacités de fermentation de sucres et le type de fermentation du lactose [116]. Il est également possible d’identifier les microorganismes par l’analyse de plusieurs phénotypes dans des systèmes miniaturisés, tels que Microlog (Biolog) [118] ou les galeries d’identification API (bioMérieux) [119]. Préalablement à l’utilisation de méthodes génétiques pour différencier les sous-espèces, l’identification de sous-espèces se faisait par des tests phénotypiques [120]. Ces méthodes d’analyse du phénotype ont été utilisées afin de distinguer deux sous-espèces de Staphylococcus equorum du fromage [93]. Cependant l’utilisation ultérieure des nouvelles méthodes génomiques n’ont pas permis de justifier la création de ces sous-espèces de S. equorum [90]. Lors de l’étude, les caractéristiques phénotypiques avaient des expressions variables ce qui
survient fréquemment à l’intérieur d’une espèce donnée. Ceci illustre que l’utilisation seule des tests phénotypiques n’est pas suffisante pour décrire précisément tous les microorganismes [99, 121] et qu’ils bénéficieraient d’analyses supplémentaires, notamment par des approches génomiques.
1.3.1.2 Tests enzymatiques
Afin de caractériser les voies métaboliques utilisées par les microorganismes, la présence des enzymes impliquées dans ces métabolismes (par exemple la fermentation de sucres variés) peut être mise en évidence au laboratoire par des tests enzymatiques et ainsi en mesurer le niveau d’activité. La réaction entre un substrat et un extrait cellulaire est mesurée par la détection d’un produit de réaction. En analysant séparément l’extrait cellulaire et le milieu de croissance, il est possible de différencier l’activité des enzymes intracellulaire et extracellulaire, par exemple, l’activité protéolytique intra- et extracellulaire [104, 122]. Plusieurs méthodes de mesure sont utilisées pour ces analyses, telles que l’absorbance [99, 106] ou la fluorimétrie [104]. Par exemple, l’activité de la cystathionine lyase est mesurée par la quantité de thiols formés à partir de la cystathionine [103]. La présence de la cystéine permet de différencier entre l’activité de la cystathionine-γ-lyase (CγL) et de la cystathionine-β-lyase (CβL), indiquant l’activité de la CγL. Le taux de formation de l’acide L-glutamique est utilisée pour analyser l’activité de la L-méthionine aminotransférase [103, 106]. L’activité des aminopeptidases a été mesurée avec des substrats acides amine p-nitroanilide (PNA) [102, 103] et aussi aminoacyl-AMC [104]. Le niveau subsistant du substrat azocaseine peut être utilisée pour mesurer l’activité protéolytique d’un microorganisme [102]. Le substrat caséine-FITC permet de mesurer l’activité spécifique de la protéinase [104] et celle des dipeptidase et tripeptidase en mesurant le réactif Cd-ninhydrine [103, 123]. La mesure des dérivés β-naphtyle [103] ou ρ-nitrophenyl [104] d’acides gras permet de déterminer l’activité des estérases.
Afin de comprendre l’effet des conditions d’affinage sur l’activité des enzymes, l’environnement dans lequel ces réactions sont conduites peut être modifié, par exemple en modifiant le pH, la concentration en sel et la température. Si le taux de formation ou le taux de catalyse du substrat est modifié, l’impact de ces conditions d’affinage peut être évalué [103, 106]. Ces analyses permettent de repérer quelles souches ou quelles espèces ont des enzymes spécifiques et leur niveau d’activité. Ainsi les souches d’intérêt, ayant un niveau d’activité élevé qui est d’importance dans son contexte, peuvent être découvertes et analysées plus en profondeur. Ces données ne nous permettent pas de détailler spécifiquement les voies métaboliques dont font partie ces enzymes, et des analyses génomiques peuvent permettre d’obtenir une vision plus complète du potentiel métabolique des souches étudiées [102].
1.3.1.3 Évaluation des composés volatils
Les fonctions des microorganismes retrouvés dans le fromage peuvent aussi être prédites en évaluant le métabolome de ces espèces dans l’environnement fromager. Plus spécifiquement, l’évaluation des composés volatils aromatiques, le volatilome [124], permet d’évaluer l’aspect aromatique de leur activité. Lors d’études métabolomiques, différents types de méthodes chromatographiques sont utilisées pour évaluer quels types d’arômes sont produits. Les composés volatils identifiés lors de ces études proviennent de métabolismes variés, soit dérivés du catabolisme des acides aminés ou des carbohydrates [111], et incluent des acides carboxyliques libres, des composés soufrés, des composés carbonyles, des alcools et des esters [125]. Les niveaux et les variétés d’arômes produits sont mesurés afin d’évaluer la contribution de chaque souche. De ce fait, les espèces et même les souches ayant une production moindre ou plus élevée de certaines molécules peuvent être identifiées. Ces caractéristiques peuvent ensuite être considérées lors de la composition de ferments d’intérêts et être analysées plus en détail [111]. Par exemple, une étude a identifié une souche de Proteus vulgaris produisant une grande quantité et une large variété de composés soufrés, associés aux arômes fromagers [84]. D’autres études ont évalué la formation de composés aromatiques dans des milieux fromagers par leur microbiote. Des associations statistiques couplées aux analyses du niveau de production d’arômes ont permis d’identifier les souches potentiellement productrices d’arômes dans le fromage, ainsi que leurs niveaux de production [125, 126]. Une étude a pu différencier les souches d’une même espèce, certaines produisant de plus hautes concentrations de composés volatils spécifiques, déterminant ainsi l’importance de la contribution de chacune à la flaveur globale du fromage [127]. Malgré cela, des méthodes complémentaires sont souvent nécessaires pour mieux comprendre les voies métaboliques utilisées par les microorganismes lors de la production de composés aromatiques. La génomique et la transcriptomique peuvent, entre autres, s’avérer utiles afin de mieux comprendre les mécanismes moléculaires impliqués dans la formation des arômes.
1.3.1.4 Conclusions sur la microbiologie classique
L’analyse des microorganismes dans le fromage avec des méthodes de microbiologie classique (dépendantes de la culture des microorganismes) nous permet de décrire leur phénotype, de caractériser leurs enzymes et d’établir leurs niveaux d’activité. Il est également possible de déterminer leur capacité à produire certaines molécules d’intérêt comme, par exemple, des composés volatils. Même si ces méthodes ont été améliorées pour augmenter leur efficacité, la croissance en milieux de culture exige un certain délai ainsi que beaucoup de manipulations en laboratoire et ne permet pas d’étudier toutes les souches, car certaines sont non cultivables. Aussi, la nature de ces expériences ne
permet pas d’apprécier pleinement l’aspect dynamique des communautés microbiennes dans le fromage, leur milieu naturel [128].
1.3.2 Méthodes moléculaires - Méthodes indépendantes de la culture des microorganismes
Les méthodes indépendantes de la culture des microorganismes (culture-independent methods) éliminent l’obligation de faire croître les microorganismes sur milieu de culture et permettent d’analyser directement les microorganismes dans le fromage [10, 55, 129]. L’identification des microorganismes, l’analyse de la biodiversité microbienne, la génomique de ces espèces et leur analyse transcriptomique sont toutes des méthodes qui ne dépendent pas directement de la croissance de souches en milieux de culture afin de les analyser. Dans le cas des écosystèmes complexes, les données moléculaires obtenues sont analysées afin d’identifier précisément la majorité des espèces/genres présents [99, 130] et de mieux comprendre les voies métaboliques présentes dans les génomes.
1.3.2.1 Les méthodes moléculaires
L’avènement des technologies basées sur l’étude des acides nucléiques (ADN et ARN) a permis d’acquérir de nouvelles connaissances sur les microorganismes d’intérêt laitier, incluant les espèces qui n’avaient précédemment pas pu être isolées et cultivées en microbiologie traditionnelle [14]. Les méthodes ciblant l’ADN incluent la génomique, la métagénomique, le métabarcoding, la PCR et l’électrophorèse. L’analyse du matériel génétique des souches permet de les identifier et de caractériser leur potentiel métabolique. L’analyse, par des méthodes telles que la transcriptomique et la qPCR de l’ARNm exprimé par les souches dans un environnement spécifique, vérifie leur activité et détermine des niveaux d’expression.
1.3.2.2 La génomique
La génomique, d’abord définie comme l’étude des séquences d’ADN, a été étendue à l’étude de profils d’expression et des rôles des gènes [128]. Plusieurs outils génomiques ont été développés et utilisés dans le domaine laitier.
1.3.2.2.1 Les méthodes à base de PCR
Jusqu’ici, les méthodes d’analyse par PCR classique et par PCR quantitative (qPCR) ont été les plus populaires pour respectivement identifier et quantifier des microorganismes d’intérêt [69]. Ces
méthodes sont basées sur l’amplification, par une succession de cycles de température, de fragments d’ADN spécifique ciblés par des amorces. Des amorces spécifiques sont utilisées pour différencier des sous-espèces, par exemple Lactobacillus delbrueckii subsp. bulgaricus et L. delbrueckii subsp.
lactis [131]. Des gènes spécifiques, tels que des gènes d’entérotoxine, peuvent être ciblés par des
amorces pour détecter leur présence dans des souches spécifiques [132]. La quantification d’espèces par des amorces spécifiques peut se réaliser en temps réel par la qPCR, ce qui permet d’observer les dynamiques changeantes des membres de communautés microbiennes dans un environnement, tel que des modèles de fromages [133].
1.3.2.2.1.1 Les régions variables de l’ADNr servant de cible à la PCR
Lors d’études nécessitant l’identification du genre et de l’espèce des microorganismes, les régions variables de l’ADN ribosomique (ADNr) sont souvent utilisées comme cible PCR. L’ADNr est une région de l’ADN génomique codant pour des éléments structuraux des ribosomes. Cette région est caractérisée par des séquences de très haute variabilité qui sont entourées de séquences hautement conservées. Les régions conservées permettent l’hybridation des amorces universelles sélectionnées pour la PCR ce qui permet d’amplifier les régions de haute variabilité. Puisque les régions variables sont spécifiques pour une espèce donnée il est possible, par séquençage d’effectuer l’identification des microorganismes étudiés [67, 69]. La localisation des régions utilisées à l’intérieur de l’opéron des gènes ribosomiques peut différer en fonction des espèces qui sont à l’étude. Par exemple, pour les bactéries, le gène 16S de l’ADNr est le plus utilisé. Pour la plupart des eucaryotes, le gène 26S de l’ADNr peut être analysé [69], tandis que plus spécifiquement pour les mycètes, les ITS (des espaces intergéniques situés entre les gènes codant pour les ARNs) sont de plus en plus ciblées et séquencées [65, 134]. L’utilisation de la région de l’ADNr n’est pas la seule utilisée chez les eucaryotes. Les animaux et protistes sont fréquemment identifiés avec une portion du gène cytochrome c oxydase
sous-unité 1 (COX1), un gène mitochondrial et les plantes avec le gène codant pour la RuBisCO, une
enzyme intervenant dans la photosynthèse [135, 136].
1.3.2.2.1.2 La PCR couplée avec la séparation sur gel contenant un gradient dénaturant
La PCR couplée aux techniques d’empreinte (fingerprinting) et d’électrophorèse est utilisée pour identifier les microorganismes présents dans un écosystème. L’ADN extrait d’une communauté mixte est utilisé comme matrice pour les amplifications par PCR. Les amorces universelles de PCR s’apparient de part et d’autre des régions variables de l’ADNr, générant des fragments spécifiques pour chaque espèce. Les acides nucléiques amplifiés sont de taille similaire, mais ont des séquences
différentes. Puisque les forces d’attachement entre les brins d’ADN dépendent de la composition en bases du fragment amplifié, il est possible d’utiliser cette propriété pour séparer les fragments sur un gel d’agarose dénaturant. Ainsi, les fragments peuvent être séparés selon des gradients chimiques (DGGE) ou de température (TGGE) [137, 138]. Les résultats obtenus à la suite de ces techniques sont des gels montrant des profils de bande pour lesquels chaque bande correspond à une espèce particulière. Afin d’associer l’espèce microbienne à la bande générée sur gel, il est ensuite nécessaire d’isoler la bande, d’en purifier l’ADN et d’effectuer le séquençage. Les méthodes basées sur la PCR ne représentent qu’une portion de celles qui sont utilisées pour identifier des microorganismes dans le fromage. D’autres techniques, telles que le PFGE (électrophorèse sur gel en champ pulsé), sont utilisées pour la caractérisation spécifique de souches pures. Dans cette dernière, de longs fragments d’ADN génomiques sont coupés par des enzymes de restrictions et sont séparés sur gel d’agarose en utilisant un champ électrique pulsé changeant périodiquement d’orientation [138, 139].
Plusieurs études ont utilisé des méthodes de PCR couplées aux techniques de fingerprinting pour déterminer la composition des communautés bactériennes dans des fromages ou des modèles de fromages [11, 140]. Dans une étude récente, la PCR-DGGE a été appliqué sur des échantillons prélevés au cours de l’affinage d’un fromage produit sans ferments et a permis le suivi des changements structuraux de la microflore du fromage [140]. L’utilisation de la PCR-DGGE a aussi permis de différencier la diversité de microorganismes dans du fromage fabriqué dans trois régions montagneuses à différentes altitudes [141]. Ces méthodes sont surtout performantes pour l’identification des microorganismes dominants dans les écosystèmes, car l’amplification des séquences, qui sont dominées par celles des populations dominantes, dissimule le peu de séquences des populations microbiennes non dominantes [11, 67, 142, 143].
1.3.2.2.1.3 PCR ciblant des gènes spécifiques
Afin de différencier des espèces parmi d’autres présentes dans un écosystème, des PCR utilisant des amorces ayant une séquence spécifique à une espèce ont été développées. Cette méthode est utilisée lorsque les espèces sont proches génétiquement et sont difficiles à différencier lors d’analyse du microbiote [144]. Par exemple, lors de l’analyse du microbiote d’un fromage au lait de brebis cru, les isolats bactériens étaient d’abord identifiés par leur phénotype et, afin de déterminer précisément l’espèce, des gènes spécifiques furent amplifiés par PCR. Par exemple, des amorces ciblant le gène
recA, codant pour une protéine fondamentale, entre autres impliquée dans la recombinaison de
![Tableau 1. Les différents types de ferments d’affinage, la variété de fromage dans laquelle ils sont ajoutés et leurs fonctions principales dans le fromage [12]](https://thumb-eu.123doks.com/thumbv2/123doknet/2916859.76124/23.918.129.787.157.599/tableau-ferments-affinage-variété-fromage-ajoutés-fonctions-principales.webp)