Caractérisation de la localisation et de l'activité du
complexe effecteur des microARN
Thèse
Pascale Michaud
Doctorat en biologie cellulaire et moléculaire
Philosophiæ doctor (Ph. D.)
Résumé
Les microARN sont de courtes molécules d’ARN qui jouent un rôle important dans la régulation post-transcriptionnelle des gènes. Ces molécules ont d’abord été découvertes chez le nématode
Caenorhabditis elegans mais sont maintenant reconnues comme des régulateurs importants de
l’expression génique chez plusieurs animaux et plantes. Pour accomplir cette fonction, les microARN doivent s’associer à un complexe protéique nommé le miRISC (microRNA-induced silencing complex) qui est composé principalement d’une protéine Argonaute et des protéines GW182. Les microARN sont liés directement par les protéines Argonautes et peuvent ainsi guider celles-ci à la région 3’ non-traduite d’un ARNm cible par complémentarité de base. Le miRISC peut ensuite induire différents mécanismes de répression allant de la répression traductionnelle à la dégradation de la cible. À ce jour, les mécanismes qui modulent l’activité du miRISC ainsi que les interacteurs de ce complexe sont peu caractérisés. L’objectif principal de mon doctorat était donc d’étudier les processus qui régulent l’activité du complexe effecteur des miARN, le miRISC.
Dans un premier temps, nous avons évalué l’importance de composantes connues du miRISC, soit les protéines GW182. Pour ce faire, nous avons aboli l’interaction entre les protéines Argonautes et GW182 et nous avons étudié l’effet de cette perte d’interaction sur la fonction du miRISC au cours du développement de C. elegans. Nous avons ainsi démontré que l’association de GW182 au miRISC n’était pas nécessaire pour le développement embryonnaire de l’animal. Nous avons par la suite confirmé que certaines cibles de microARN embryonnaires n’étaient pas affectées par l’absence de GW182. Nous avons ainsi conclu que le miRISC pouvait exister et fonctionner sous différentes formes et que les protéines GW182 étaient dispensables à l’activité du miRISC dans certains contextes. Dans un deuxième temps, nous avons tenté d’identifier de nouveaux facteurs qui contribuent à l’activité du miRISC en réalisant un criblage génétique. Nous avons ainsi identifié la RabGAP tbc-11 comme un nouvel acteur contribuant à la fonction des microARN. Nous avons démontré que tbc-11 agit sur la petite GTPase rab-6 et que la régulation de celle-ci est importante pour assurer la localisation intracellulaire adéquate de la protéine Argonaute ALG-1. Nos résultats ont permis de démontrer qu’une localisation inadéquate de la protéine ALG-1 engendre un défaut dans la répression de cibles de
microARN. Nous avons ainsi pu conclure que le transport vésiculaire joue un rôle important dans la fonction des microARN.
Les travaux réalisés au cours de mon doctorat ont permis de démontrer que la localisation et la composition du miRISC contribuent à la modulation de sa fonction. Ces résultats ont pu approfondir nos connaissances sur les mécanismes utilisés par la cellule pour moduler l’activité des microARN, et ouvrent ainsi la voie à d’autres recherches pouvant étudier les fonctions des microARN dans divers contextes.
Abstract
MicroRNAs are small RNA molecules that play an important role in post-transcriptional gene silencing. These molecules were first discovered in the nematode Caenorhabditis elegans but are now known as important regulators of gene expression in most animals and plants. To accomplish this function, microRNAs interact with a protein complex called the miRISC (microRNA-induced silencing complex) which is formed by an Argonaute protein and GW182 proteins. MicroRNAs interact directly with Argonaute and can guide them to the 3’ UTR region of a target mRNA by base pairing. The miRISC will then induce several repression mechanisms, ranging from translational repression to target decay. To this day, the mechanisms that modulate miRISC activity as well as the proteins that interact with the complex are poorly characterized. The main objective of my PhD was therefore to study the processes that regulate the activity of the effector complex of microRNAs, the miRISC.
First, we evaluated the importance of a known component of the miRISC; the GW182 proteins. To accomplish this, we abolished the interaction between Argonaute and GW182 proteins and we studied the effect of this loss of interaction on miRISC function during C. elegans development. We have shown that the association of GW182 to the miRISC is dispensable for the animal’s embryonic development. We then confirmed that certain embryonic microRNA targets were not affected by the loss of GW182. These results allowed us to conclude that the miRISC can exist and function under different forms and that GW182 proteins are dispensable for the miRISC activity under certain conditions.
Second, we wanted to identify new factors that contribute to miRISC silencing by performing a forward genetic screen. This allowed us to identify the RabGAP tbc-11 as a new factor contributing to microRNA function. We have shown that tbc-11 acts on the small GTPase rab-6 and that its regulation is important for proper intracellular localization of the Argonaute ALG-1. Our results have shown that an improper localization of ALG-1 leads to defects in microRNA target repression. We have therefore concluded that vesicular transport plays an important role in microRNA function.
Our results have shown that the localization and composition of the miRISC contributes to the modulation of its activity. This study has deepened our knowledge on the mechanisms that modulate microRNA activity and opens the door for other research on microRNA function in different contexts.
Table des matières
Résumé ... ii
Abstract ... iv
Table des matières ... v
Liste des figures ... vii
Liste des tableaux ... viii
Liste des abréviations ... ix
Remerciements ... xii
Avant-propos ... xiv
Introduction ... 1
Les microARN ... 2
La biogénèse des microARN ... 2
Les microARN dans un contexte physiologique ... 5
Le complexe effecteur des microARN (miRISC) ... 8
Les protéines Argonautes ... 8
Les protéines GW182 ... 11
Les modes de répression du miRISC ... 13
La régulation du miRISC ... 18
Le transport vésiculaire ... 23
Les petites GTPase Rab ... 24
Le réticulum endoplasmique ... 27
L’appareil de Golgi ... 28
Caenorhabditis elegans comme modèle d’étude ... 30
Description du nématode ... 30
Les microARN chez C. elegans ... 33
Problématique et objectifs ... 38
Chapitre 1. GW182-free microRNA silencing complex controls post-transcriptional gene expression during Caenorhabditis elegans embryogenesis ... 40
1.1Résumé ... 41
1.2Abstract ... 42
1.3 Introduction ... 43
1.4 Results ... 45
ALG-1 tryptophan-binding pockets are essential for the interaction with GW182 proteins AIN-1 and AIN-2 ... 45
The interaction between ALG-1 and AIN proteins is not essential for miRISC formation ... 50
The alteration of tryptophan-binding pockets leads to defective miRNA-mediated regulation in the somatic tissues ... 53
The embryos miRISC does not require GW182 proteins to control gene expression ... 60
1.5 Discussion ... 64
1.7 Acknowledgments and contributions ... 68
1.8 References ... 69
Chapitre 2. Criblage génétique pour identifier de nouveaux acteurs importants pour la répression par les microARN ... 75
2.1 Introduction ... 75
2.2 Conception du criblage génétique et isolation de mutants ... 76
2.3 Identification du mutant ... 81
Chapitre 3. The RabGAP TBC-11 controls Argonaute localization for proper microRNA function in C. elegans ... 87
3.1 Résumé ... 88
3.2 Abstract ... 89
3.3 Introduction ... 90
3.4 Results ... 92
The Rab GTPase activating protein tbc-11 contributes to miRNA function ... 92
TBC-11 acts on RAB-6 to affect miRNA function ... 99
The RabGAP TBC-11 affects the association of ALG-1 to endomembranes ... 101
TBC-11 affects the association of ALG-1 to microRNAs ... 103
miRNA unloaded ALG-1 accumulates in a high molecular weight complex at the perinuclear region in tbc-11(qbc24) animals ... 106
3.5 Discussion ... 113
3.6 Material and methods ... 115
3.7 Acknowledgments and contributions ... 118
3.8 References ... 118
Conclusion ... 125
Comment le miRISC fonctionne sans GW182 ? ... 126
Un nouvel interacteur du miRISC sans GW182 ... 129
L’importance du transport vésiculaire pour l’activité du miRISC ... 135
L’implication de TBC-11 et RAB-6 sur la localisation des protéines Argonautes ... 138
Conclusion générale ... 140
Liste des figures
Figure I. Voie de biogénèse des miARN ... 4
Figure II. Représentation des domaines des protéines Argonautes ... 10
Figure III. Représentation des domaines des protéines GW182 ... 13
Figure IV. Mécanismes de répression employés par le miRISC ... 18
Figure V. Modes de régulation du miRISC ... 22
Figure VI. Activation et désactivation des petites GTPase Rab ... 26
Figure VII. Cycle de vie de C. elegans ... 32
Figure VIII. Cellules de couture et alae de C. elegans ... 36
Figure 1.1 Identification of the Tryptophan-binding pockets of ALG-1 ... 46
Figure 1.2 Purification of recombinant ALG-1(TPmut) ... 47
Figure 1.3 Co-Immunoprecipitation of ALG-1(TPmut) with AIN-1 and AIN-2 in vivo ... 49
Figure 1.4 MicroRNA levels in alg-1(TPmut) animals ... 51
Figure 1.5 Levels of microRNA associated to ALG-1(TPmut) ... 52
Figure 1.6 Phenotypic rescue of alg-1 null animals ... 54
Figure 1.7 Seam cell division and fusion in ALG-1(TPmut) expressing animals ... 56
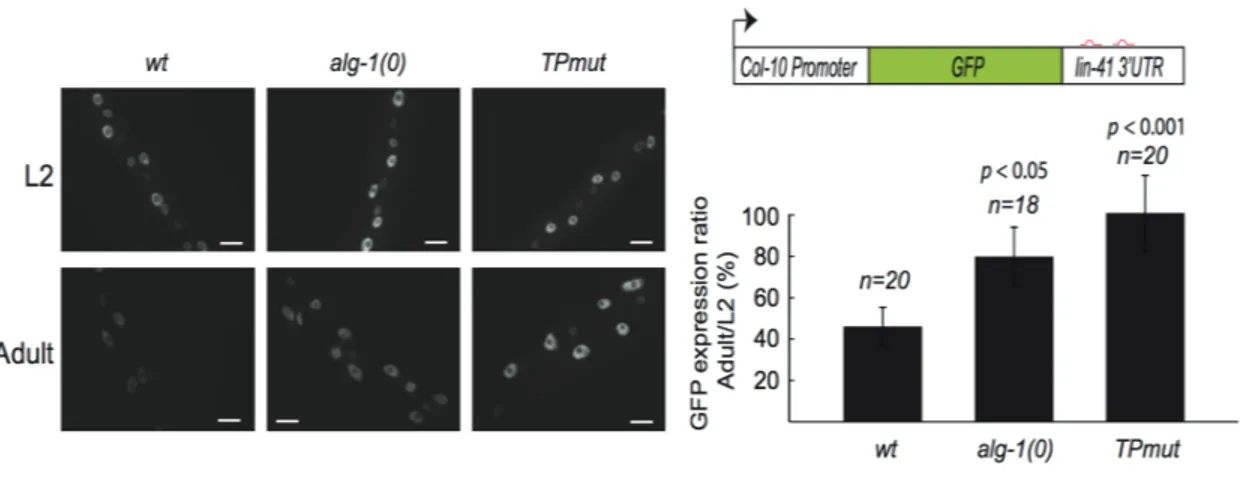
Figure 1.8 Effect of ALG-1(TPmut) on a let-7 microRNA activity reporter ... 57
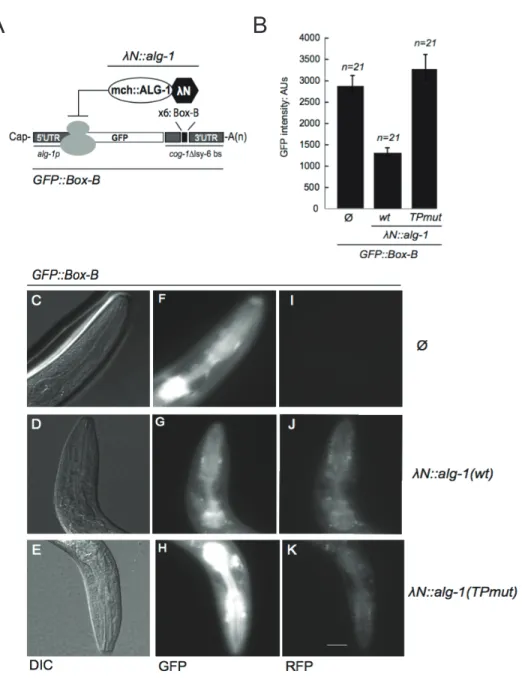
Figure 1.9 Tethered ALG-1 tryptophan-binding pockets mutant cannot repress gene expression ... 59
Figure 1.10 Synthetic lethality of alg-1(TPmut) with alg-2 ... 61
Figure 1.11 Dispensability of ain-1 and ain-2 for C. elegans embryonic development ... 63
Figure 1.12 Effect of ALG-1(TPmut) on embryonic sex determination controlled by miR-35 family ... 64
Figure 2.1 Constructions utilisées pour générer le système rapporteur GFP ... 78
Figure 2.2 Conception et étapes du criblage génétique ... 80
Figure 2.3 Quantification de la GFP dans le pharynx des mutants tard ... 81
Figure 2.4 Schéma de la méthodologie pour l’identification de la mutation d’intérêt ... 83
Figure 2.5 Identification de tbc-11 comme gène responsable des défauts d’alae chez tard-10 ... 86
Figure 3.1 Identification of tbc-11(qbc24) allele ... 93
Figure 3.2 Sequence characteristics of tbc-11 ... 94
Figure 3.3 Seam cell division and fusion in tbc-11 animals ... 95
Figure 3.4 tbc-11 affects the function of let-7 and the repression of its target, LIN-41 ... 97
Figure 3.5 tbc-11 affects the function of miR-228 miRNA ... 98
Figure 3.6 TBC-11 is likely a GTPase activating protein for RAB-6.1 and RAB-6.2 ... 101
Figure 3.7 Protein levels of ALG-1 and AIN-1 in tbc-11 mutant extracts ... 102
Figure 3.8 tbc-11 affects microRNA levels ... 104
Figure 3.9 tbc-11 affects microRNA loading ... 105
Figure 3.10 Intracellular localization of ALG-1 in the seam cells of tbc-11 animals ... 107
Figure 3.11 Co-localization of ALG-1 and the Golgi marker MANS ... 108
Figure 3.12 ALG-1 associates with a high molecular weight complex in tbc-11(qbc24) animals ... 110
Figure 3.13 High molecular weight ALG-1 is not loaded with miRNA in tbc-11(qbc24) animals ... 112
Figure 3.14 Model for the implication of tbc-11 in ALG-1 localization ... 112
Figure IX. Létalité des animaux alg-1(TPmut);dpf-3 sur ARNi alg-2 ... 131
Figure X. Létalité des animaux alg-1(0);dpf-3 sur ARNi alg-2 ... 132
Figure XI. Létalité à la transition L4-adulte des animaux alg-1(0);dpf-3 ... 133
Liste des tableaux
Tableau 2.1 Gènes mutés chez le candidat tard-10 ... 84 Tableau I Interacteurs protéiques de ALG-1(TPmut) chez les embryons ... 130
Liste des abréviations
A : adénine
ADN : acide désoxyribonuclétique Ago : Argonaute
AIN : ALG-1 interacting protein ALG : argonaute-like gene ARN : acide ribonucléique ARNm : ARN messager ATP : adénosine triphosphate
CCR4 : carbon catabolite repression 4
C. elegans : Caenorhabditis elegans
DIC : differential interference contrast eIF : eukaryotic initiation factor EMS : ethyl methanesulfonate GAP : GTPase activating protein
GARP : Golgi-associated retrograde protein Gb : gigabase
GDI : GDP dissociation inhibitor GDP : guanosine diphosphate
GEF : guanine nucleotide exchange factor GERp95 : Golgi ER protein 95 kDa GFP : green fluorescent protein GTP : guanosine triphosphate Hsc : heat shock cognate Hsp : heat shock protein
iCLIP : individual-nucleotide resolution cross-linking immunoprecipitation kDa : kilodalton
Mb : mégabase miARN : microARN Mid : middle
miRISC : microRNA-induced silencing complex mm : millimètre
MVB : multivesicular bodies NOT : negative on tata less nt : nucléotides
PABP : poly-A binding protein PAN : poly-A nuclease Paz : Piwi-Argonaute-Zwille
Piwi : p-element induced wimpy testis pré-miARN : microARN précurseur pri-miARN : microARN primaire
RabGGTase : Rab geranylgeranyltransferase RE : réticulum endoplasmique
REP : Rab escort protein RRM : RNA recognition motif siARN : small interfering ARN
SNP : single nucleotide polymorphism
tard : tethering alg-1 repression defect
TBC : Tre2-Bub2-Cdc16
TNRC6 : trinucleotide repeat-containing gene 6 UBA : ubiquitine-associated
unc : uncoordinated
Remerciements
Au cours de mes 5 années passées au laboratoire, j’ai eu la chance de recevoir l’appui de nombreuses personnes que je tiens à remercier. Tout d’abord, merci à mon directeur de recherche, Martin, pour le dévouement dont tu fais preuve envers tes étudiants. Merci pour ta patience, ta disponibilité, ta porte de bureau toujours ouverte, et par-dessus tout : ton optimisme légendaire. Dans les moments les plus difficiles, tu as toujours cru en moi et en mon projet, même quand je n’y croyais plus moi-même. Je suis confiante que je repars du laboratoire avec la formation et les compétences nécessaires pour accomplir tous mes projets futurs.
Merci aux stagiaires d’été et aux collaborateurs qui ont participés aux deux publications qui se retrouvent dans cette thèse. Votre aide fut très appréciée et importante pour la réalisation de cette thèse. Je tiens également à remercier les membres de mon jury, Dr. Marc-Etienne Huot, Dre. Amélie Fradet-Turcotte et Dr. Christian Rocheleau pour avoir accepté d’évaluer ma thèse.
Merci aux membres du laboratoire, les anciens et les nouveaux. Merci à Guillaume et Sandra qui m’ont tout appris dans mes débuts au laboratoire. Merci à Lucile qui a été, et qui est toujours, une très bonne amie. Merci spécial à mes voisins de bureau, mes ‘boys’, Pierre-Marc et Louis-Mathieu. Même si j’ai dû endurer vos conversations de soccer et de Civilization trop souvent, vous avez rendu les dernières années au laboratoire beaucoup plus agréables. Merci d’avoir été des très bons amis et confidents. Merci aussi à François pour ta grande sagesse et pour le temps que tu va mettre pour la complétion de mon article. À toutes les autres personnes que j’ai rencontrées au laboratoire ; merci pour toutes les discussions et les rires, autour du ‘bench’ comme autour d’une bière.
Je souhaite aussi remercier plusieurs personnes hors du laboratoire. Premièrement, merci à mes amies Vanessa et Caroline. C’est assez spécial d’avoir des amies d’enfance qui ont le même parcours et qui comprennent la frustration d’un western blot qui ne fonctionne jamais. Merci pour tous les rires, les pleurs, les soupers, les nombreux spectacles de musique et d’humour, mais surtout les nombreuses bouteilles de vin. Merci d’avoir été mes piliers à Québec.
Merci également à ma petite famille ; mes parents, Arthur et Josée, et mon frère Fred. J’ai toujours pu compter sur vous, même si vous étiez à plus de 300 km de distance. Je remercie spécialement mes parents qui, même à ce jour, font passer le bonheur de leurs enfants avant tout (alors que nous sommes tous des adultes maintenant). J’ai une chance incroyable d’avoir une famille qui m’apporte autant de soutien.
Finalement, un énorme merci à mon plus grand supporter, David. Tu as été d’une écoute et d’un positivisme incroyable pendant les 3 dernières années et je te remercie énormément de m’avoir aidé à passer à travers les périodes plus difficiles. Tu m’as appris que les relations à distance c’est possible si on le veut vraiment. Merci d’être venu me voir à Québec aussi souvent pendant 2 ans et demi. Merci de m’avoir accueilli chez toi pendant l’écriture de cette thèse. Et merci pour ta patience pendant ces années, on peut maintenant commencer notre vie d’adultes.
Avant-propos
Les résultats présentés dans cette thèse représentent les travaux que j’ai effectués au cours des 5 dernières années dans le laboratoire du Dr. Martin Simard. Tout d’abord, lors de ma maîtrise, j’ai assisté Dr. Guillaume Jannot dans la caractérisation de l’importance des protéines GW182 pour la fonction des microARN au cours du développement de C. elegans. Ces travaux ont fait l’objet d’une publication dans le journal Plos Genetics, en décembre 2016, intitulée : GW182-Free microRNA
Silencing Complex Controls Post-transcriptional Gene Expression during Caenorhabditis
elegans Embryogenesis (Guillaume Jannot, Pascale Michaud, Miguel Quévillon-Huberdeau, Louis Morel-Berryman, James A. Brackbill, Sandra Piquet, Katherine McJunkin, Kotaro Nakanishi et Martin J. Simard). Mon statut sur cette publication est celui de 2e auteur. J’ai réalisé plusieurs figures présentées dans cet article en plus d’avoir réalisé les expériences nécessaires lors des rondes de révisions (j’ai contribué ou réalisé les figures 1E, 4, 5, 6 et S6). Cette publication est présentée au
Chapitre 1 de cette thèse.
Au cours de ma maîtrise, j’ai aussi réalisé un criblage génétique afin d’identifier de nouveaux facteurs importants pour l’activité du miRISC. Les détails de ce criblage sont présentés au Chapitre 2. La souche utilisée pour ce criblage fut générée par Dr. Guillaume Jannot avant mon arrivée au laboratoire. J’ai par la suite réalisé le criblage génétique et isolé plusieurs mutants. Au début de mon doctorat, j’ai reçu l’aide d’une stagiaire d’été, Rachel Rioux, afin d’identifier le gène muté chez l’un des candidats du criblage. C’est ainsi que j’ai identifié la RabGAP tbc-11 comme un nouveau facteur contribuant à l’activité du miRISC.
Lors de mon doctorat, j’ai caractérisé le rôle de tbc-11 sur la fonction des microARN. Ces travaux ont mené à un article intitulé : The RabGAP TBC-11 controls Argonaute localization for proper
microRNA function in C. elegans (Pascale Michaud, Pauline Adjibade, Rachel Rioux, Camille
Lavoie-Ouellet, Weifeng Gu, Rachid Mazroui et Martin J. Simard). Cet article a été soumis au journal Plos Genetics en mars 2020. Mon statut sur ce manuscrit est celui de premier auteur. J’ai réalisé toutes les expériences qui y sont présentées, et j’ai écrit l’article en collaboration avec Dr. Martin Simard. Ces travaux sont présentés au Chapitre 3.
Le chapitre 1 est précédé d’une revue de la littérature permettant d’expliquer les concepts importants pour la compréhension des travaux présentés dans la thèse. Les chapitres 1 et 3 sont présentés sous forme d’article. L’ordre et la numérotation des figures ont été modifiés afin de mieux s’intégrer à cette thèse.
Introduction
Les nombreuses cellules qui composent un organisme doivent contrôler finement leur expression génique afin d’exercer leurs fonctions respectives et répondre aux stimuli qu’elles reçoivent. Pour ce faire, plusieurs mécanismes sont mis en place et peuvent s’effectuer à toutes les étapes allant de la transcription d’un gène sur l’ADN jusqu’à la production d’une protéine fonctionnelle.
L’un de ces mécanismes, appelé interférence à l’ARN, repose sur l’action de courtes molécules d’ARN non-codantes qui jouent un rôle important dans la régulation post-transcriptionnelle des gènes. Bien que l’ARN fût longtemps perçu comme un simple intermédiaire entre l’ADN et les protéines, la découverte de l’interférence à l’ARN a ouvert la voie à un tout nouveau champ d’étude. Aujourd’hui, il est bien accepté que des ARN non-codants peuvent jouer divers rôles allant de la régulation de l’expression génique à la catalyse de réactions enzymatiques.
Parmi ces ARN non-codants, on retrouve une classe nommée les microARN. Ces derniers ont suscité un intérêt particulier au cours des dernières années grâce à leur capacité d’inhiber l’expression de nombreux ARNm en protéine ainsi que leur implication dans diverses pathologies humaines. Dans cette introduction, les concepts importants portant sur la fonction des microARN et sur leur complexe effecteur chez les animaux sont présentés. En plus, des concepts portant sur le transport vésiculaire et sur l’utilisation de C. elegans comme modèle d’étude sont abordés pour faciliter la compréhension des résultats présentés aux Chapitres 1 à 3.
Les microARN
Les microARN (miARN) sont une classe de courts ARN non codants impliqués dans la régulation de l’expression génique. Les miARN se lient à des ARNm cibles par complémentarité de base pour réprimer leur expression par un mécanisme appelé interférence à l’ARN. Il est estimé que 60% des gènes codants pour des protéines chez l’humain contiennent des sites de liaison de miARN (Friedman
et al., 2009). Les miARN régulent ainsi l’expression de gènes impliqués dans diverses fonctions
biologiques tel que la prolifération, le développement, la différenciation et la réponse au stress. D’autant plus, leur dérégulation est associée à diverses pathologies humaines, incluant le cancer (Lin et Gregory, 2015).
Le premier miARN a été découvert lors d’un criblage génétique chez C. elegans (Lee et al., 1993). Les auteurs de cette étude cherchaient à identifier de nouveaux gènes impliqués dans la synchronisation du développement de l’animal. Ils ont ainsi identifié lin-4, un ARN qui ne code pour aucune protéine et qui réprime l’expression de la protéine LIN-14. Quelques années plus tard, la découverte du miARN
let-7, lui aussi impliqué dans la synchronisation du développement de C. elegans, a pu suggérer qu’il
existait plusieurs autres miARN (Reinhart et al., 2000). Enfin, c’est la découverte de la conservation de
let-7 qui a permis de mettre en évidence que ce mécanisme n’était pas exclusif au nématode et qu’il
s’agissait d’un mode de régulation important chez l’humain également (Pasquinelli et al., 2000).
La biogénèse des microARN
Les miARN sont transcrits au noyau par l’ARN polymérase II (Lee et al., 2004). Ils sont transcrits sous forme de miARN primaire (pri-miARN) à partir de locus spécifiques ou encore à partir d’introns ou d’exons de gènes (Bartel, 2004). Les miARN primaires sont des transcrits de plus de 1 kb qui adoptent une structure tige-boucle avec des extrémités 5’ et 3’ simple brin. Si les séquences de plus d’un miARN mature sont contenues dans un miARN primaire, on appelle ceux-ci des miARN polycistroniques. Suite à leur transcription, les pri-miARN sont clivés une première fois dans le noyau par un complexe nommé le Microprocesseur. Ce dernier, composé de la ribonucléase de type III Drosha et du cofacteur DGCR8,
permet le clivage des extrémités simple brin du miARN primaire et forme ainsi une structure tige-boucle nommée miARN précurseur (pré-miARN) (Lee et al., 2003 ; Denli et al., 2004). Le pré-miARN est ensuite exporté au cytoplasme par Exportin-5, une protéine dépendante du RanGTP (Yi et al., 2003 ; Bohnsack et al., 2004 ; Lund et al., 2004). Par contre, des mécanismes d’export alternatifs entrent en jeu puisque la déplétion de l’Exportin-5 n’affecte pas la localisation cytoplasmique de tous les pré-miARN (Kim et al., 2016). De plus amples études seront nécessaires pour identifier ces mécanismes alternatifs et clarifier le rôle de l’Exportin-5 dans la biogénèse des miARN.
Une fois exportés au cytoplasme, les pré-miARN subissent un second clivage par une deuxième ribonucléase de type III, Dicer (Zhang et al., 2004). Celle-ci reconnait les deux nucléotides flottants à l’extrémité 3’ du pré-miARN qui ont été générés par Drosha et clive dans la structure tige-boucle pour former un duplex de miARN d’environ 22 nt (MacRae et al., 2007 ; Park et al., 2011). Les cofacteurs TRBP et PACT peuvent s’associer avec Dicer et sembleraient améliorer la précision du clivage pour certains miARN, mais leur rôle exact demeure toujours sous investigation (Daugaard et Hansen, 2017). Suite au clivage par Dicer, le duplex de miARN peut être pris en charge par les protéines Argonautes. Le complexe de chargement, composé de l’Argonaute, Hsc70, Hsp90 et du duplex de miARN est alors formé (Naruse et al., 2018). Les protéines chaperons Hsp90 et Hsc70 hydrolysent l’ATP et sont nécessaires pour le chargement des miARN dans l’Argonaute. Le duplex de miARN est ensuite séparé et le brin possédant l’extrémité 5’ la moins stable sera conservé dans l’Argonaute et deviendra le brin guide (Schwarz et al., 2003). Le second brin, nommé brin passager, est expulsé de l’Argonaute et dégradé. Le brin guide du miARN et la protéine Argonaute forme ainsi le complexe miRISC minimal (microRNA-induced silencing complex) (Figure I).
Figure I. Voie de biogénèse des miARN
La voie de biogénèse des miARN débute au noyau par la transcription d’un miARN primaire (pri-miARN) par l’ARN polymérase II. Le pri-miARN est ensuite clivé en miARN précurseur (pré-miARN) par l’enzyme Drosha et son cofacteur DGCR8. Le pré-miARN est exporté au cytoplasme par l’Exportin-5 ou par d’autres mécanismes d’export non caractérisés. Le pré-miARN est clivé par Dicer au cytoplasme et le duplex de pré-miARN formé est chargé dans la protéine Argonaute à l’aide des protéines Hsp90 et Hsc70. Le brin passager est expulsé et dégradé alors que le brin guide est chargé dans l’Argonaute pour former le miRISC.
ARN polymérase II pri-miARN Drosha DGCR8 pré-miARN Exportin-5 Autre mécanisme d’export Dicer Duplex miARN Argonaute Hsc70 Hsp90 Brin passager Argonaute miRISC minimal Noyau Cytoplasme
Les miARN matures sont d’une longueur d’environ 22 nt et peuvent s’associer à des ARNm cibles, généralement dans leur région 3’ non-traduite, par complémentarité de base partielle. En effet, alors que les siARN s’apparient parfaitement avec leurs cibles, les miARN dépendent plutôt de leur région ‘seed’ pour s’apparier à leurs cibles. Cette séquence représente les nucléotides 2 à 8 de l’extrémité 5’ du miARN et se lie parfaitement avec l’ARNm cible (Lewis et al., 2003). Les miARN possédants la même séquence ‘seed’ sont classifiés comme des membres de la même famille. Ceux-ci ont alors le potentiel de réguler les mêmes cibles. En plus de la séquence ‘seed’, la région 3’ du miARN peut contribuer à l’interaction avec l’ARNm cible. En effet, de plus en plus d’études démontrent que l’appariement entre la région 3’ du miARN et la cible peut affecter la spécificité et la régulation des cibles, et même avoir un effet sur la stabilité du miARN (Chipman et Pasquinelli, 2019).
Les microARN dans un contexte physiologique
Puisque les miARN peuvent contrôler l’expression de nombreux gènes dans la cellule, il n’est pas étonnant que leur dérégulation soit impliquée dans de nombreuses pathologies chez l’humain. Tout d’abord, deux gènes étant connus comme des agents causaux de deux maladies humaines sont des acteurs de la voie des miARN. C’est le cas de la protéine FMR1P, qui est responsable du syndrome du X fragile. Celle-ci interagit avec la protéine Argonaute et avec certains miARN in vivo, et cette interaction est perdue chez les patients atteint du syndrome du X fragile (Jin et al., 2004). Deuxièmement, la protéine DGCR8, une composante importante de la voie de biogénèse des miARN, est déplétée chez les patients atteints du syndrome cardio-faciale de DiGeorge (Shiohama et al., 2003). En plus de ces syndromes associés directement à la perte d’un interacteur de la voie des miARN, plusieurs protéines impliquées dans la voie de biogénèse des miARN se retrouvent mutées dans différents cancers. En effet, on retrouve de nombreuses mutations germinales et somatiques dans les gènes Drosha, DGCR8, Exportin-5, Dicer et TRBP chez différents cancers. On retrouve particulièrement un grand nombre de mutations dans les domaines catalytiques RNase IIIB de Dicer et Drosha ainsi que dans les domaines de liaison à l’ARN double-brin des protéines TRBP et DGCR8 (Lin et Gregory, 2015).
De plus, plusieurs études ont démontré que les niveaux d’expression des facteurs impliqués dans la voie de biogénèse des miARN sont affectés chez différents cancers. Dans le cas de Dicer, on retrouve un niveau réduit de son expression dans certains cancers tel que ceux du poumon, du sein, de la peau et des ovaires. De façon opposée, on retrouve une surexpression de Dicer dans les métastases du cancer de la prostate et du cancer colorectal, entre autres (Foulkes et al., 2014 ; Lin et Gregory, 2015). La même tendance est observée pour Drosha, où certains cancers présentent une surexpression de cette protéine alors que d’autres cancers présentent plutôt une diminution de l’expression de Drosha. Dans certains cas, ces changements d’expression de Dicer et Drosha sont corrélés avec un mauvais pronostic ou avec la progression de la maladie. Par contre, l’effet de leur dérégulation reste à élucider dans d’autres cas (Foulkes et al., 2014).
Alors que les protéines importantes pour la voie des miARN peuvent être impliquées dans différentes pathologies, incluant les cancers, certains miARN sont eux-mêmes responsables de diverses maladies. La première évidence de l’implication de miARN dans le cancer est apparue lorsqu’une étude a démontré que le loci contenant miR-15 et miR-16 était déplété chez plusieurs patients atteints de leucémies lymphoïdes chroniques des cellules B (Calin et al., 2002). Par la suite, plusieurs autres études ont permis de mettre en évidence que le profil d’expression de miARN était affecté dans différents cancers (Calin et Croce, 2006). En effet, environ 50% des gènes codants pour des miARN chez l’humain se retrouvent dans des régions fragiles, c’est-à-dire des régions qui sont susceptibles aux translocations, aux amplifications et aux délétions dans les cellules cancéreuses (Calin et al., 2004). Certains miARN se retrouvent dans des régions amplifiées dans les cancers et fonctionnent comme des oncogènes. Ceux-ci sont alors nommés oncomiR. C’est le cas par exemple des miARN polycistroniques miR-17-92 qui sont surexprimés dans les lymphomes de cellules B et qui promeuvent le développement de tumeurs (He et al., 2005). De façon opposée, certains miARN se retrouvent dans des régions déplétées dans les cancers et sont ainsi classifiés comme suppresseurs de tumeur. Ces miARN ont comme cibles des oncogènes qui ne peuvent plus être réprimés lorsque les miARN suppresseurs de tumeur sont déplétés, favorisant ainsi la progression tumorale (Esquela-Kerscher et Slack, 2006).
Dans certains cas, le rétablissement de l’expression d’un seul miARN est suffisant pour inhiber la croissance tumorale. C’est le cas par exemple de miR-21, un miARN surexprimé dans les lymphomes
de cellules B. Chez un modèle de souris, la surexpression de miR-21 est suffisante pour induire un phénotype de cellules B malignes, confirmant son rôle comme oncogène. De façon intéressante, lorsque l’expression de miR-21 est abolie, les tumeurs induites régressent complètement en quelques jours, indiquant que l’expression de miR-21 est nécessaire à leur survie chez ce modèle (Medina et
al., 2010). Un autre exemple est celui du miARN miR-335, dont l’expression est diminuée dans les
cancers du sein. Le rétablissement de l’expression de miR-335 chez les souris ayant reçu une greffe de cellules mammaires cancéreuses permet de diminuer les métastases au poumon, confirmant le rôle de miR-335 comme suppresseur de tumeur (Tavazoie et al., 2008).
Il est maintenant accepté que les miARN ainsi que les protéines nécessaires à leur biogénèse sont impliqués dans diverses maladies, particulièrement dans le cancer. Aujourd’hui, plusieurs études démontrent que certains miARN sont impliqués dans la progression tumorale, mais leurs différentes contributions semblent être spécifiques à certains types de cancer. Il en va de soi que moduler l’expression des miARN dans les cellules cancéreuses peut être une thérapie prometteuse. Par contre, de plus amples études sont nécessaires pour atteindre cet objectif. Plusieurs questions telles que la méthode optimale pour délivrer un miARN à un type cellulaire particulier et les rôles de différents miARN dans divers types cellulaires restent à élucider.
Le complexe effecteur des microARN (miRISC)
Les courts ARN doivent s’associer à divers complexes protéiques afin d’effectuer leurs fonctions. Ces complexes sont nommés RISC pour RNA-induced silencing complex. Les protéines Argonautes sont les principaux acteurs des complexes RISC, liant directement les courts ARN. Les courts ARN peuvent ainsi guider l’Argonaute à un ARN messager cible grâce à une complémentarité de base. Dans le cas des miARN, le complexe effecteur est nommé miRISC (microRNA-induced silencing complex) et est composé typiquement des protéines Argonautes et des protéines d’échafaudage GW182. Ces dernières permettent le recrutement des complexes de déadénylation et décoiffage, permettant ainsi d’induire la dégradation de l’ARNm. De plus, le miRISC peut induire la répression traductionnelle de sa cible. Par contre, l’ordre dans lequel les différents mécanismes de répression sont effectués demeure un sujet débattu. Il est probable que ce choix soit dépendant du contexte. En effet, la localisation intracellulaire de l’ARNm cible ainsi que les différentes protéines qui s’associent au miRISC pourraient déterminer quel mécanisme de répression est favorisé.
Les protéines Argonautes
Les protéines Argonautes ont été identifiées pour la première fois lors d’un criblage génétique réalisé chez la plante Arabidopsis thaliana (Bohmert et al., 1998). Dans cette étude, elles ont été décrites comme étant importantes pour le développement normal de la plante. Les feuilles des mutants observés présentaient un phénotype qui rappelait la forme de la pieuvre Argonauta argo, d’où le nom Argonaute choisi par les auteurs. Par contre, à cette époque, la fonction moléculaire de ces protéines demeurait inconnue.
Suite à la découverte de l’interférence à l’ARN, plusieurs équipes se sont intéressées à l’identification de protéines impliquées dans ce processus. C’est ainsi que le rôle des protéines Argonautes dans la répression post-transcriptionnelle des gènes fut élucidé (Tabara et al., 1999 ; Hammond et al., 2000 ; Hammond et al., 2001 ; Grishok et al., 2001 ; Martinez et al., 2002). Plusieurs études ont permis de
mettre en évidence que les protéines Argonautes lient les courts ARN pour former le RISC, et sont ensuite guidées à un ARNm cible où elles peuvent induire son clivage ou sa répression.
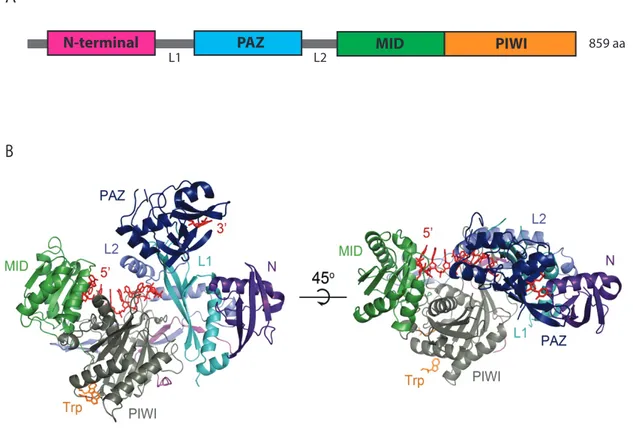
Les protéines Argonautes ont été bien conservées au cours de l’évolution. En effet, on retrouve des gènes codants pour des Argonautes chez la levure jusqu’à l’humain. Leur nombre est par contre très variable ; chez l’humain on compte 8 gènes codants pour des Argonautes, 5 chez la drosophile et un impressionnant 26 chez le nématode C. elegans (Hutvagner et Simard, 2008). On distingue 3 types de de protéines Argonautes, soit les Argonaute-like (similaire à AGO1 chez la plante), les PIWI-like (similaire à PIWI chez la drosophile) et finalement les Argonautes spécifique à C. elegans, les WAGO (worm-specific Argonaute). Les différentes Argonautes sont impliquées dans tous les types de régulation génique médiée par les courts ARN et peuvent ainsi effectuer des fonctions variées. On retrouve quatre domaines distincts qui se divisent en deux lobes chez les protéines Argonautes. Le premier est composé des domaines N-terminal et PAZ alors que le deuxième lobe contient les domaines MID et PIWI (Meister, 2013) (Figure II). Le domaine N-terminal est requis pour la séparation des deux brins du miARN mature lors du chargement de celui-ci dans l’Argonaute (Kwak et Tomari, 2012). Le domaine PAZ lie l’extrémité 3’ des courts ARN en formant une poche hydrophobique dans laquelle les deux nucléotides flottants peuvent s’insérer (Lingel et al., 2004). Le domaine MID, quant à lui, permet la liaison de l’extrémité 5’ des courts ARN en formant une poche de liaison qui lie l’extrémité phosphate du court ARN (Boland et al., 2010). Finalement, le domaine PIWI est celui qui possède une activité enzymatique lui permettant de cliver les ARNm cibles (Song et al., 2004). En effet, ce domaine étant similaire aux ribonucléases H, il contient un motif DDH (aspartate-aspartate-histidine) qui lui confère une activité de clivage. Celle-ci est importante pour la voie des siARN où le RISC induit un clivage de l’ARNm cible. Cette fonction est principalement attribuée à Ago2 chez les mammifères (Liu
et al., 2004), mais Ago3 semblerait aussi posséder cette activité catalytique (Park et al., 2017). Chez
le nématode C. elegans, le motif DDH des protéines Argonautes impliquées spécifiquement dans la voie des miARN (ALG-1 et ALG-2) semble être important pour la biogénèse de certains miARN (Bouasker et Simard, 2012). En plus de son activité catalytique, le domaine PIWI possède des poches de liaison au tryptophane qui permettent le recrutement des protéines d’échafaudage GW182 (Schirle et MacRae, 2012 ; Pfaff et al., 2013).
Les protéines Argonautes sont bien connues comme étant le corps du miRISC puisqu’elles lient directement les miARN. Par contre, puisqu’elles n’ont pas recours à leur activité de clivage pour les cibles de miARN, elles doivent recruter et interagir avec divers facteurs afin d’accomplir leurs fonctions de répression. Les protéines interagissant avec les Argonautes, les modes de répression employés par le miRISC ainsi que la régulation des Argonautes et du miRISC seront décrits dans les prochaines sections.
Figure II. Représentation des domaines des protéines Argonautes
(A) Schéma des domaines présents chez la protéine humaine Ago2. L1 et L2 : séquences
linker 1 et 2. PAZ : Piwi-Argonaute-Zwille. MID : middle. PIWI : p-element induced wimpy testis. La protéine contient 859 acides aminés. (B) Structure cristalline de la protéine Ago2. Les deux lobes contenant les domaines PAZ et N ainsi que MID et PIWI y sont représentés. Un court ARN est représenté en rouge. Les poches de liaison au tryptophane du domaine PIWI sont indiquées en orange. Figure tirée de Schirle et MacRae 2012, avec permission de l’AAAS.
859 aa
N-terminal PAZ MID PIWI
L1 L2
A
Les protéines GW182
Bien que les protéines Argonautes forment le corps du miRISC, d’autres facteurs se lient au complexe afin d’assurer sa fonction. Parmi ceux-ci, les mieux caractérisés sont sans doute les protéines GW182. Celles-ci ont d’abord été découvertes par une étude visant à identifier de nouveaux complexes protéines/ARN (Eystathioy et al., 2002). Pour ce faire, les auteurs ont utilisé des auto-anticorps humains dérivés de sérum de patients puisque ceux-ci vont reconnaitre et marquer plusieurs structures et macromolécules de la cellule. En effet, cette technique a permis de mieux caractériser les ARNm associés à diverses protéines telle que eIF4-E et PABP (Tenenbaum et al., 2000). C’est donc en utilisant un sérum auto-immun sur des cellules HeLa que les auteurs ont identifié une nouvelle structure formant des foci cytoplasmiques distincts. Ils ont par la suite déterminé que ces foci contenaient une nouvelle protéine de 182 kDa qui présentait de nombreuses répétitions glycine-tryptophane (GW) d’où le nom GW182. Ils ont aussi détecté la présence de nombreux ARNm dans ces foci, qu’ils ont par la suite nommé ‘GW bodies’ (Eystathioy et al., 2002). Une deuxième étude du même groupe a par la suite identifié que les ‘GW bodies’ co-localisaient avec les protéines hDcp1 et hLSm4 (Eystathioy et
al., 2003). Celles-ci étant impliquées dans le décoiffage des ARNm, la fonction des GW bodies dans
la dégradation des ARNm commençait alors à se dessiner.
Quelques études ont par la suite permis d’élucider le rôle des protéines GW182 dans la voie des miARN. Tout d’abord, une étude a démontré que l’Argonaute humaine Ago2 co-localise avec les GW bodies (Sen et Blau, 2005). Par la suite, un orthologue des protéines GW182 chez C. elegans fut identifié dans un criblage génétique et son association avec l’Argonaute fut démontrée (Ding et al., 2005). Cette interaction fut ensuite confirmée chez l’humain et il fut observé que l’inhibition de l’expression de GW182 induit des défauts de répression chez des rapporteurs de miARN (Meister et
al., 2005 ; Liu et al., 2005). Tous ces résultats suggéraient alors que les protéines GW182 sont une
composante importante du miRISC.
Les protéines GW182 sont retrouvées seulement chez les métazoaires. Chez l’humain, on retrouve trois paralogues de GW182 ; soit TNRC6A, TNRC6B et TNRC6C (trinucleotide repeat-containing gene
6) (Braun et al., 2013). Chez la drosophile, on retrouve seulement une protéine GW182 nommée
GW182 ont été identifiés, soit AIN-1 et AIN-2 (ALG-1 interacting protein) (Ding et al., 2005 ; Zhang et
al., 2007). Par contre, bien que ces deux protéines interagissent avec l’Argonaute ALG-1, seulement
AIN-1 a la capacité de recruter les complexes de déadénylation, suggérant alors que AIN-1 est le seul orthologue fonctionnel de GW182 chez C. elegans (Kuzuoğlu-Öztürk et al., 2012).
Chez les humains et la drosophile, les protéines GW182 contiennent seulement deux domaines annotés. On retrouve un domaine d’association à l’ubiquitine (UBA) ainsi qu’un domaine de reconnaissance de l’ARN (RRM) qui sont tous les deux entourés de régions prédites comme étant non-structurées (Ding et Han, 2007 ; Eulalio et al., 2009a) (Figure III). Les différents membres de la famille GW182 possèdent différents nombres de répétitions GW qui se retrouvent majoritairement dans la région N-terminal de la protéine. Cette région correspond aussi au site de liaison avec les protéines Argonautes (Behm-Ansmant et al., 2006a). Dans les parties Mid et C-terminal de la protéine, on retrouve une région impliquée dans le recrutement des complexes de déadénylation et dans la répression des ARNm cibles. Cette région est nécessaire et suffisante pour induire la répression et la dégradation d’un ARNm (Eulalio et al., 2009b ; Zipprich et al., 2009 ; Lazzaretti et al., 2009). En effet, le recrutement artificiel de la région C-terminal de TNRC6 à un ARNm est suffisant pour induire sa répression, démontrant bien l’importance de ce domaine.
Chez C. elegans, la protéine AIN-1 diverge grandement des autres membres de la famille GW182. En effet, les domaines UBA et RRM ne sont pas retrouvés chez celle-ci. On y retrouve seulement des répétitions GW dans la région N-terminal de la protéine (Figure III). Par contre, nous pouvons tout de même conclure que AIN-1 un orthologue des protéines GW182 puisque celle-ci se lie aux protéines Argonautes et est importante pour la répression des cibles de miARN in vivo (Ding et al., 2005 ; Zhang
et al., 2007 ; Ding et Grosshans, 2009).
Le domaine de liaison de l’Argonaute à GW182 se retrouve dans la partie N-terminal de la protéine, où se retrouvent la majorité des répétitions GW. On retrouve dans cette région trois motifs de liaison à l’Argonaute (Takimoto et al., 2009). Alors que les poches de liaison au tryptophane de la protéine Argonaute permettent la liaison d’une seule protéine GW182, les motifs GW des protéines GW182 permettent la liaison de trois protéines Argonautes à la fois (Takimoto et al., 2009 ; Elkayam et al.,
2017). Ceci suggère donc qu’il pourrait y avoir formation de larges complexes contenant plusieurs ARNm cibles et que plusieurs RISC pourraient coopérer à travers une seule protéine GW182.
Figure III. Représentation des domaines des protéines GW182
Schémas des séquences primaires de la protéine humaine TNRC6C (A) et de la protéine de
C. elegans AIN-1 (B). Le nombre d’acides aminés (aa) contenu dans chaque protéine est
indiqué. UBA : domaine d’association à l’ubiquitine. RRM : motif de reconnaissance de l’ARN.
Les modes de répression du miRISC
Les miARN ont comme rôle de guider le miRISC à la région 3’ non traduite des ARNm cibles par complémentarité de base. Le miRISC peut ensuite induire divers mécanismes de répression. Le complexe peut dégrader sa cible par des mécanismes de déadénylation et de décoiffage (decapping), et/ou inhiber la traduction de l’ARNm en protéine (Figure IV). Par contre, les détails moléculaires de ces mécanismes ainsi que la cinétique de ceux-ci demeurent des sujets débattus. Cette section présente différentes études qui se sont attardées à ces questions ainsi que les conclusions contradictoires que certaines d’entre elles ont obtenues.
1690 aa RRM UBA A B Répétitions GW
Domaine liaison Ago Domaine répression
Répétitions GW
Domaine liaison Ago
Le mécanisme de répression le mieux caractérisé est sans doute la déadénylation de l’ARNm. Le modèle accepté repose sur le recrutement des complexes de déadénylation CCR4-NOT et PAN2-PAN3 par les protéines GW182. Ces deux complexes agissent de façon successive afin d’enlever la queue poly-A des ARNm (Yamashita et al., 2005). En effet, le complexe PAN2-PAN3 est impliqué dans les premières étapes de la déadénylation et son activité est stimulée par les protéines PABPC, qui lient directement la queue poly-A. Le complexe CCR4-NOT est ensuite recruté et permet de compléter la déadénylation de l’ARNm. Chez les cibles de miARN, les protéines GW182 sont essentielles à la déadénylation de l’ARNm (Behm-Ansmant et al., 2006a ; Eulalio et al., 2008). Elles recrutent les complexes de déadénylation par interaction directe entre la région C-terminale de GW182 et les protéines NOT1 et PAN3 (Braun et al., 2011). De plus, les protéines GW182 lient directement les protéines PABPC, ce qui pourrait contribuer au recrutement des complexes PAN2-PAN3 et CCR4-NOT et ainsi promouvoir les mécanismes de déadénylation (Hutzinger et al., 2010 ; Flamand et al., 2016). Une fois dépourvu de queue poly-A, le transcrit est considéré instable et une dégradation 3’ – 5’ peut être induite (Figure IV).
Un autre mode de répression employé par le miRISC est le décoiffage des ARNm. En effet, le complexe DCP1-DCP2 est recruté aux ARNm ciblés par le miRISC et permet d’enlever la coiffe de l’ARNm. Les protéines GW182 sont importantes pour le recrutement de ce complexe (Behm-Ansmant et al., 2006a). La protéine DCP2 est la sous-unité catalytique du complexe et celle-ci est assistée par les activateurs de décoiffage DCP1, Me31B et HPat (Nishihara et al., 2013). Ces protéines recrutées par le miRISC induisent alors la dégradation 5’ – 3’ irréversible de l’ARNm. L’activateur de décoiffage Ge-1 semble aussi être important pour la répression de certaines cibles de miARN (Eulalio et al., 2007a). De plus, l’activateur de décoiffage DDX6 est aussi recruté aux cibles de miARN par une interaction directe avec la protéine NOT1 du complexe de déadénylation CCR4-NOT (Rouya et al., 2014). Ceci suggère que les mécanismes de déadénylation et de décoiffage sont couplés, ce qui pourrait expliquer l’importance de GW182 dans le décoiffage des cibles de miARN (Figure IV).
Le mécanisme de répression suscitant le plus de contradictions est sans doute l’inhibition de la traduction. Premièrement, l’étape de la traduction qui est inhibée par le miRISC est un sujet débattu. Certaines études suggèrent que les miARN bloquent la traduction des ARNm au niveau de l’initiation. En effet, la traduction des cibles de miARN serait inhibée en ciblant le complexe de liaison à la coiffe
eIF4F (Mathonnet et al., 2007 ; Fukaya et al., 2014). Une autre étude a suggéré que la protéine 4EHP, un homologue de la protéine de liaison à la coiffe eIF4E, est importante pour l’inhibition de l’initiation de la traduction chez les cibles de miARN. Le complexe CCR4-NOT pourrait recruter 4EHP et ce dernier serait en compétition avec eIF4E pour lier la coiffe, ce qui permettrait d’inhiber l’initiation de la traduction (Chapat et al., 2017). Une autre étude a démontré qu’on retrouvait une séquence similaire au domaine de liaison à la coiffe de eIF4E à l’intérieur du domaine Mid de la protéine Ago2. Cette séquence permettrait la liaison directe de l’Argonaute à la coiffe, ce qui empêcherait la liaison du complexe eIF4F et induirait une répression de la traduction (Kiriakidou et al., 2007). Ces observations demeurent toutefois à confirmer.
Certaines études suggèrent plutôt que la traduction est inhibée après l’étape d’initiation. Une première étude se concentrant sur le mode d’action du miARN lin-4 a établi que celui-ci réprimait l’expression de lin-14 après l’étape d’initiation puisque l’association du transcrit de lin-14 aux polysomes était inchangée en présence de lin-4 (Olsen et Ambros, 1999). De plus, d’autres études ont démontré que les ARNm ciblés par des miARN sont associés aux polysomes, suggérant encore une fois que l’initiation de la traduction n’est pas réprimée par les miARN (Maroney et al., 2006 ; Petersen et al., 2006 ; Nottrot et al., 2006). Une expérience de marquage métabolique a aussi permis de suggérer que la répression par les miARN s’effectue avant que la chaine polypeptidique naissante soit complétée (Petersen et al., 2006). Toutes ces études suggèrent donc que les miARN pourraient réprimer leurs cibles au niveau de l’élongation de la traduction.
Bien qu’il soit connu que les miARN peuvent induire une répression traductionnelle et une dégradation des transcrits, il est toujours incertain si ces mécanismes sont successifs et si l’un d’entre eux engendre le second. Plusieurs études suggèrent que l’inhibition de la traduction précède la déadénylation (Mathonnet et al., 2007 ; Fabian et al., 2009 ; Bazzini et al., 2012 ; Djuranovic et al., 2012). Par contre, d’autres études suggèrent que ces mécanismes sont parfois indépendants et qu’il est possible que certains miARN répriment leur cible par un seul mécanisme. En effet, il fut observé que les miARN peuvent réprimer la traduction de leurs cibles de façon indépendante de la déadénylation (Fukaya et Tomari, 2011). De plus, une autre étude a observé que certaines cibles de miARN n’étaient pas dégradées suite au recrutement du miRISC, suggérant alors que certains ARNm pouvaient être maintenus dans un état réprimé de façon temporaire (Behm-Ansmant et al., 2006b). Cette tendance
fut aussi observée chez les embryons de C. elegans, où des cibles de miARN sont déadénylées et leur traduction est réprimée mais les transcrits sont tout de même stables (Wu et al., 2010). De plus, une étude a déterminé qu’en absence de traduction active, certaines cibles de miARN étaient stabilisées alors que d’autres étaient dégradées, suggérant alors qu’il existe différents mécanismes employés par le miRISC pour réprimer ses cibles (Eulalio et al., 2007a).
Certaines études affirment que les protéines GW182 sont nécessaires pour la déadénylation ainsi que pour la répression de la traduction des cibles de miARN (Eulalio et al., 2008 ; Ding et Grosshans, 2009). Par contre, d’autres études suggèrent que le miRISC peut fonctionner sans les protéines GW182. En effet, une étude chez la drosophile a démontré qu’en absence de GW182, le miRISC ne peut plus effectuer la déadénylation de ses cibles mais peut toujours induire une répression de la traduction. Cette étude suggère aussi qu’il pourrait y avoir différents mécanismes de répression de la traduction, certains dépendants de GW182 et d’autres non (Fukaya et Tomari, 2012). De plus, une autre étude chez la drosophile a démontré qu’en absence de facteurs mitogènes, on retrouve majoritairement un miRISC sans les protéines GW182. Ce dernier est principalement associé aux ARNm qui se retrouvent aux polysomes et pourrait tout de même inhiber leur traduction (Wu et al., 2013).
De façon intéressante, certaines études ont démontré que les miARN pouvaient plutôt stimuler la traduction d’ARNm cible dans certaines conditions. En effet, ce phénomène fut observé pour certains ARNm chez les cellules de mammifères quiescentes (phase G0) et chez les oocytes de xénope (Vasudevan et al., 2007 ; Vasudevan et al., 2008 ; Mortensen et al., 2011). Ce mécanisme requiert l’activité des protéines Argonautes et FXR1, mais est indépendant des protéines GW182 (Vasudevan
et al., 2007 ; Truesdell et al., 2012).
Chaque miARN a le potentiel de réguler plusieurs ARNm qui contiennent une séquence d’appariement à leur séquence ‘seed’. De façon inverse, plusieurs miARN peuvent réguler simultanément un même ARNm (Doench et Sharp, 2004). Une étude chez les embryons de C. elegans a aussi démontré qu’un complexe miRISC lié à un ARNm cible peut faciliter le recrutement d’un deuxième complexe miRISC. En effet, ce deuxième complexe contient un miARN qui reconnait la cible sans sa séquence ‘seed’ et qui ne peut se lier à l’ARNm à lui seul. Par contre, une interaction directe entre les protéines Argonautes
permettrait de recruter ce deuxième miRISC qui possède une interaction miARN-ARNm non canonique (Flamand et al., 2017).
Somme toute, plusieurs études tirent des conclusions divergentes et parfois même contradictoires quant aux mécanismes qui sont employés par le miRISC pour réprimer ses cibles. Il est clair que différents mécanismes sont employés, mais la séquence dans laquelle ceux-ci sont induits est débattue. Une hypothèse permettant d’harmoniser toutes ces observations est que le choix de mécanisme de répression est dépendant du contexte. En effet, le miRISC pourrait induire différents mécanismes selon la nature de la cible, le type cellulaire ou les protéines qui interagissent avec le complexe par exemple.
AAAAAAAAAAA AGO PABP PABP GW182 PAN3 PAN2 NOT1 CCR4 DDX6 DCP1 DCP2 eIF4F
Figure IV. Mécanismes de répression employés par le miRISC
Le miRISC est composé d’un miARN (en rouge), d’une protéine Argonaute (AGO), et des protéines GW182 (région des répétitions GW représentée par la ligne rose). Le complexe peut induire la déadénylation des ARNm par interaction avec PABP et les complexe PAN2-PAN3 et CCR4-NOT. La coiffe de l’ARNm est représentée par un cercle noir. Celle-ci peut être enlevée par le complexe DCP1-DCP2. L’activité de ce dernier est stimulée par l’activateur de décoiffage DDX6. Le miRISC peut aussi induire une répression de la traduction ; soit par compétition avec le complexe de liaison à la coiffe eIF4F ou par inhibition de l’élongation (représenté par le ribosome en gris et la chaine polypeptidique naissante en orange). Les complexes CCR4-NOT et eIF4F sont composés de plusieurs autres protéines qui n’ont pas été représentées par soucis d’encombrement de la figure.
La régulation du miRISC
Les cellules emploient divers mécanismes afin de réguler l’activité du miRISC. Tout d’abord, les protéines Argonautes sont la cible de nombreuses modifications post-traductionnelles qui permettent de moduler leur activité. De plus, différentes protéines interagissant avec le miRISC ont été identifiées comme des modulateurs de l’activité de celui-ci. Finalement, la localisation intracellulaire du miRISC pourrait aussi jouer un rôle sur son activité. Ces trois différents modes de régulation seront présentés dans cette section.
Plusieurs modifications post-traductionnelles peuvent être ajoutées aux protéines Argonautes afin de moduler rapidement leur activité (Figure V). La plus caractérisée est sans doute la phosphorylation. Par exemple, la sérine 387 de l’Argonaute humaine Ago2 peut être phosphorylée par la voie de réponse au stress p38-MAPK. Cette phosphorylation semble importante pour induire la répression traductionnelle des cibles au profit de la dégradation de l’ARNm (Horman et al., 2013). De façon intéressante, ce même site de phosphorylation fut ensuite démontré comme étant important pour le recrutement de LIMD1, une protéine qui facilite l’interaction entre l’Argonaute et les protéines GW182 (Bridge et al., 2017). La phosphorylation de la tyrosine 393 de Ago2 par EGFR, quant à elle, est
importante pour l’interaction entre l’Argonaute et Dicer. Elle affecte ainsi la maturation des pré-miARN en miARN mature et influence alors le chargement des miARN dans l’Argonaute (Shen et al., 2013 ; Yang et al., 2014). La phosphorylation de l’Argonaute peut aussi affecter directement la liaison du miARN ; c’est le cas de la tyrosine 529 qui affecte la liaison de l’extrémité 5’ du miARN (Rüdel et al., 2011). Finalement, deux études ont démontré qu’on retrouve un groupe de résidus phosphorylables dans la région C-terminale de la protéine humaine Ago2 et de ALG-1 chez C. elegans. La phosphorylation de ces résidus est importante pour contrôler l’association du miRISC aux cibles (Golden et al., 2017 ; Quévillon Huberdeau et al., 2017).
En plus de la phosphorylation, on retrouve d’autres types de modifications post-traductionnelles sur les protéines Argonautes qui peuvent influencer leur activité. C’est le cas par exemple de l’hydroxylation. Une étude a démontré que la protéine Ago2 est hydroxylée sur la proline 700 et que cette modification est importante pour la stabilité de la protéine (Qi et al., 2008). De plus, l’ajout de poly(ADP-ribose) sur l’Argonaute permet d’inhiber son activité en condition de stress (Leung et al., 2011). Finalement, les protéines Argonautes peuvent aussi être SUMOylées. En effet, l’ajout d’un groupement SUMO à la lysine 402 de Ago2 induit une déstabilisation de la protéine (Sahin et al., 2014). Ces exemples illustrent bien la pertinence des modifications post-traductionnelles des protéines Argonautes dans la régulation de l’activité du miRISC. Il s’agit en effet d’une façon rapide de moduler l’activité générale des miARN en réponse à divers stimuli ou en fonction du contexte cellulaire. Plusieurs protéines interagissant avec le miRISC sont aussi des modulateurs de l’activité de celui-ci (Figure V). En effet, bien que les protéines GW182 sont les partenaires des Argonautes les mieux caractérisés, d’autres protéines ont été identifiées comme étant importante pour l’activité du miRISC. Plusieurs de ces facteurs ont été identifiés chez C. elegans puisqu’on y retrouve des protéines Argonautes dédiées spécifiquement à la voie des miARN. La caractérisation de leurs interacteurs est donc un bon indice quant à leur fonction dans la voie des miARN. Tout d’abord, les hélicases NHL-2 et CGH-1 ont été montrées comme étant importantes pour l’activité du miRISC chez C. elegans. Ces dernières n’affectent pas la biogénèse des miARN mais sont plutôt importantes pour la répression de cibles de miARN (Hammell et al., 2009). De plus, la protéine casein kinase II (CK2) est aussi importante pour la répression de cibles de miARN puisque celle-ci phosphoryle directement CGH-1 (Alessi et al., 2015). Les hélicases semblent jouer un rôle important dans la fonction du miRISC puisqu’une autre
étude a identifié l’hélicase GLH-1 comme un facteur important pour la répression médiée par le miRISC dans la lignée germinale de C. elegans (Dallaire et al., 2018). De plus, la protéine ribosomale RACK-1 joue aussi un rôle dans la répression des cibles de miARN. Celle-ci promeut le recrutement du miRISC au site de traduction des ARNm, autant chez C. elegans que chez les cellules humaines (Jannot et al., 2011a). Finalement, la protéine de liaison à l’ARN Staufen (STAU-1) affecte aussi la fonction du miRISC. Par contre, celle-ci agit plutôt comme un régulateur négatif de l’activité du miRISC. Il fut proposé que Staufen entre en compétition avec le miRISC pour lier les mêmes régions d’un ARNm (Ren et al., 2016).
Un autre mécanisme qui semble important pour moduler l’activité du miRISC est la régulation de sa localisation intracellulaire (Figure V). La localisation intracellulaire des miARN a d’abord été associée aux corps P (P bodies) (Chan et Slack, 2006). Ces foci cytoplasmiques sont des agrégats composés d’ARNm réprimés ainsi que de protéines impliquées dans la répression traductionnelle et la dégradation des ARN. En plus de la présence des facteurs de déadénylation et de décoiffage interagissant avec le miRISC, les protéines Argonautes et GW182 co-localisent elles aussi avec les corps P (Parker et Sheth, 2007). Ces foci ont alors été proposés comme étant un site de répression et de dégradation des cibles de miARN. Par contre, une étude a démontré que la répression par les miARN pouvait avoir lieu même en absence des corps P. En effet, c’est plutôt la formation des corps P qui est dépendante de l’activité des miARN. Ces résultats suggèrent donc que la formation des corps P est une conséquence de la répression par les miARN et non pas une nécessité pour leur fonction (Eulalio et al., 2007b).
Plusieurs études démontrent aussi un lien entre le transport vésiculaire et la voie des miARN. Tout d’abord, des études réalisées dans les cellules de mammifères et chez la drosophile ont associé les corps multivésiculaires (‘multivesicular bodies’ ou MVB) à la fonction des miARN. Ils ont observé que les protéines Argonautes, GW182 et certains miARN s’associent avec les MVB et que la déplétion de ces structures affecte la fonction des miARN. Ils ont ainsi proposé que les MVB étaient importants pour le recyclage et le transport des composantes du miRISC (Gibbings et al., 2009 ; Lee et al., 2009). D’autres études ont fait un lien entre le système endomembranaire, tel que le réticulum endoplasmique (RE) et l’appareil de Golgi, et les miARN. Premièrement, un criblage génétique réalisé chez C. elegans
visant à identifier de nouveaux facteurs impliqués dans la voie des miARN a identifié cogc-4, un gène du complexe oligomérique du Golgi, comme un facteur contribuant à la fonction des miARN (Parry et
al., 2007). Un autre criblage réalisé chez C. elegans a permis d’identifier vps-52, une composante du
Golgi-associated retrograde protein (GAPR) complex, comme un facteur important pour la fonction des miARN. Cette étude a proposé que ce complexe participerait au recyclage des protéines GW182 (Vasquez-Rifo et al., 2013). Par la suite, il fut observé que le réticulum endoplasmique semble aussi être important pour la fonction des miARN. Une première étude a démontré que les protéines Argonautes, Dicer, TRBP et PACT co-sédimentent avec le RE rugueux et que cette association est nécessaire pour le chargement des miARN dans l’Argonaute, la reconnaissance de la cible ainsi que la répression de la cible (Stalder et al., 2013). Une autre étude a suggéré que les ARNm doivent être transportés au RE rugueux avant d’être reconnus par le miRISC (Barman et Bhattacharyya, 2015). Chez la plante Arabidopsis thaliana, une étude a démontré que l’inhibition de la traduction des cibles de miARN s’effectue au RE et que la protéine AMP1, une protéine membranaire se retrouvant au RE, est requise pour ce mécanisme (Li et al., 2013b). D’autres études chez la plante ont aussi démontré que les miARN et les protéines Argonautes sont associés avec les polysomes liés aux membranes plutôt que les polysomes libres (Li et al., 2016 ; Sjögren et al., 2018). Toutes ces observations suggèrent donc que le système endomembranaire joue un rôle important dans la fonction des miARN. Alors que les corps P semblent être un lieu où les cibles de miARN s’accumulent afin d’être dégradées, les MVB, le RE et le Golgi semblent plutôt impliqués dans le chargement des miARN, la répression traductionnelle des cibles et le recyclage des composantes du miRISC.
Certaines études ont suggéré que les cibles de miARN sont réprimées au RE rugueux. Par contre, ces études ont été réalisées à l’aide de rapporteurs artificiels et ne représentent donc pas la dynamique complexe de véritables cibles de miARN in vivo. En effet, il est plutôt probable que le lieu de répression soit dépendant de chaque cible. Ceci est bien illustré chez les neurones, où il fut démontré que
miR-134 doit être transporté aux dendrites pour réprimer localement sa cible Limk1 afin d’assurer le bon
développement du neurone (Schratt et al., 2006). De façon intéressante, c’est la forme précurseur du miARN qui est transporté aux dendrites pour que sa biogénèse soit effectuée au même endroit où le miARN mature doit exercer sa fonction (Bicker et al., 2013). Ce transport de pré-miARN aux dendrites fut aussi observé pour miR-181 (Sambandan et al., 2017).
Toutes ces études soulèvent ainsi la nécessité de transporter les miARN et les composantes du miRISC à l’intérieur de la cellule. En effet, ce transport semble important afin de coordonner le chargement des miARN, la reconnaissance de la cible, la répression des cibles, la dégradation des cibles, ainsi que le recyclage et la dégradation des miARN et des composantes du miRISC.
Figure V. Modes de régulation du miRISC
Plusieurs mécanismes sont utilisés pour moduler l’activité du miRISC. Les protéines Argonautes subissent de nombreuses modifications post-traductionnelles (en jaune) qui affectent leur activité. Différentes protéines (en grises) interagissent avec le miRISC pour moduler son activité. Celles-ci peuvent lier directement le miRISC (Y) ou interagir via l’ARN (X). Finalement, l’association des miARN et du miRISC aux corps multivésiculaires, au Golgi, au réticulum endoplasmique et aux corps P semble aussi réguler leur activité.
GW182 AGO AAAAAAAAAAAA