HAL Id: tel-00447215
https://tel.archives-ouvertes.fr/tel-00447215
Submitted on 14 Jan 2010
HAL is a multi-disciplinary open access
archive for the deposit and dissemination of sci-entific research documents, whether they are pub-lished or not. The documents may come from teaching and research institutions in France or abroad, or from public or private research centers.
L’archive ouverte pluridisciplinaire HAL, est destinée au dépôt et à la diffusion de documents scientifiques de niveau recherche, publiés ou non, émanant des établissements d’enseignement et de recherche français ou étrangers, des laboratoires publics ou privés.
Cytochromes P450
Thien-An Nguyen
To cite this version:
Thien-An Nguyen. Relations structure - Fonction dans la superfamille des Cytochromes P450. Infor-matique [cs]. Université Paris-Diderot - Paris VII, 2007. Français. �tel-00447215�
Résumé
Les cytochromes P450 (CYP) sont des enzymes responsables de la biotransformation de composés exogènes, aussi bien dans les phénomènes de détoxication que d’intoxication par formation d’entités réactives. La forme hépatique humaine la plus abondante (CYP3A4) est responsable du métabolisme de plus de 60 % des médicaments utilisés actuellement, entraînant de nombreuses interactions médicamenteuses indésirables. La connaissance des mécanismes moléculaires de fonctionnement de ces CYPs au moyen de modèles prédictifs est d’un intérêt primordial pour les industriels. L’obtention de ces modèles par modélisation comparative est toutefois pénalisée par la dispersion en séquences dans cette superfamille.
Une méthode originale de reconstruction des CYPs basée sur l’identification au sein de cette famille des blocs structuralement conservés (CSB) est proposée ici. Ces CSBs définissent un repliement commun aux CYPs et sont considérés comme la signature structurale de la superfamille. Les CSBs sont codés en termes d’informations statistiques (profile) puis alignés sur les séquences de CYP de structure inconnue, par un outil d’alignement multiple (Caliseq) créé pour produire l’alignement multiple optimal pour les reconstructions par modélisation comparative. Caliseq sert aussi à détecter des séquences originales de CYP dans une banque ou un génome.
Le modèle structural obtenu permet de suggérer des mutations pour observer les modifications du comportement de la protéine vis-à-vis de ses substrats spécifiques. Le cas du CYB2B6 est un exemple concret où le modèle a suggéré des mutations permettant d’augmenter l’affinité de l’enzyme pour un substrat spécifique utilisé en chimiothérapie.
Mots clef : Cytochrome P450, Modélisation comparative à bas taux d’identité, Génomique exploratoire à partir de signature structurale, CYP2B6.
Abstract
Cytochromes P450 (CYP) are monoxygenase enzymes involved in biotransformations of exogenous compounds in detoxification processes, but also in intoxication by generation of reactive species. CYP3A4, the most abundant isoform present in human liver, is responsible for the metabolisation of more than 60% of drugs used in therapy, leading to unwanted drug-drug interactions. The knowledge and understanding of mechanisms underlying substrate recognition and transformation is of outstanding interest for setting up reliable predictive tools in pharmaceutical companies. Achievement of CYP models by homology modeling is however limited by the high diversity of sequences in this superfamily.
An original method based on the identification of Common Structural Blocks (CSB) within the family, is proposed here to rebuild structural models despite their low sequence identity. CSBs define a common fold of the CYPs and are used as a structural signature of the superfamily. A multiple alignment tool able to align successive profiles (calculated in each CSB) on the CYP sequences of unknown structure, has been developed to make use of the structural information beneath the CSBs, in one hand to give reliable multiple alignments used in homology modeling, and in the other hand, to search for new CYP sequences in databanks or genomes.
When the structural model is obtained, in silico mutation experiments can be performed to monitor changes in the behavior of the protein towards its specifics substrates. A successful example is shown through CYP2B6 case, in which mutations suggested by the model led to significant increase of its affinity towards a specific substrate used in chemotherapy.
Keywords: Cytochrome P450, low identity Homology Modeling, Genomic exploration using structural signature, CYP2B6.
i
Table des matières
INTRODUCTION GENERALE 1
ÉTAT DES CONNAISSANCES 9
1 CYTOCHROMES P450 : POURQUOI UN TEL INTERET ? 11
1.1 A LA DECOUVERTE D’UNE SUPERFAMILLE D’ENZYME… 12
1.2 NOMENCLATURE,CLASSIFICATION ET PHYLOGENIE 23
1.3 OCCURRENCE,DISTRIBUTION ET LOCALISATION 31
1.4 UN REPLIEMENT TRES CONSERVE 36
1.5 PROPRIETES CHIMIQUES DES P450S 52
1.6 AU CENTRE D’UN SYSTEME DE DETOXICATION 58
1.7 CONCLUSION 64
2 LES OUTILS BIOINFORMATIQUES : VERS DE NOUVELLES SOLUTIONS 67
2.1 BREVE PRESENTATION DE LA DISCIPLINE ET OBJECTIFS 68
2.2 LES PROTEINES : DESCRIPTION GEOMETRIQUE DU SQUELETTE PEPTIDIQUE 69
2.3 BANQUES DE SEQUENCES ET DE STRUCTURES : LES DONNEES SOURCES 72
2.4 TRAITEMENT DES DONNEES SOURCES 82
2.5 CONSTRUCTION D’UN MODELE 112
2.6 EXPLOITATION DU MODELE 120
2.7 CONCLUSION 127
NOUVELLES METHODES 129
3 MODELISATION COMPARATIVE ADAPTEE POUR LES SUPERFAMILLES 131
3.1 INTRODUCTION A LA METHODE 132
3.2 RECHERCHE DES ELEMENTS STRUCTURAUX CONSERVES 133
3.3 POSITIONNEMENT DES CSBS SUR LA SEQUENCE CIBLE 140
3.4 CONSTRUCTION DES MODELES DE CYTOCHROME P450 148
3.5 ÉVALUATION, SELECTION ET RAFFINEMENT DES MODELES 152
ii
4.1 P ’ 160
4.2 FONCTIONNEMENT DE CALISEQ 160
4.3 LES DIFFERENTES EVOLUTIONS DE CALISEQ 163
4.4 CONCLUSION 172
RESULTATS 175
5 COMPARAISON DES METHODES 177
5.1 INTRODUCTION 178
5.2 LES CSBS, UNE UTILISATION INNOVANTE ? 179
5.3 CALISEQET LES AUTRES METHODES D’ALIGNEMENT 205
5.4 CONSTRUCTION DE MODELES DE P450S, QUELLE METHODE ADOPTER ? 217
5.5 VERS L’OBTENTION D’UNE BANQUE DE P450S 223
5.6 CONCLUSION ET PERSPECTIVES 234
6 DU VIRTUEL AU CONCRET : APPLICATIONS SUR LE CYP2B6 237
6.1 INTRODUCTION 238
6.2 DIFFERENTES APPROCHES EXPLOITEES POUR UN SEUL MODELE FINAL 239
6.3 DOCKING MANUEL ET MUTATIONS IN SILICO 243
6.4 SIMULATION DE MD 247
6.5 CONCLUSION 260
6.6 ARTICLE 261
CONCLUSION ET PERSPECTIVES 263
7 BILAN ET NOUVELLES IDEES 265
BIBLIOGRAPHIES 271
ANNEXES 289
ANNEXE 1 LE MONDE MERVEILLEUX DES PROTEINES 291
ANNEXE 2 FICHIER,FORMAT ET EXEMPLES 301
ANNEXE 3 CALISEQ : DISSECTION D’UN PROGRAMME 315
ANNEXE 4 MODELLER :STRATEGIE ADAPTEE AUX BLOCS 343
iii
Abréviations et acronymes
ADN = Acide Désoxyribonucléique
CASP = Critcal Assessment of protein Structure Prediction
CATH = Class Architecture Topology Homology
CPA = Chlorophosphamide
CPI = 4-(4-chlorophenyl)imidazole
CSB = Common Structural Block (fr. block structuralement commun ou conserve)
CYP = Cytochrome P450
Da = Dalton
EMBL = European Molecular Biology Laboratory
EMX = Enzymes du Métabolisme des Xénobiotiques
GDEPT = Gene-Directed Enzyme Prodrug Therapy
HMM = Hidden Markov Model (fr. modèle de Markov caché)
indel = Insertion Délétion
Kd = Constante de Dissociation
Km = Constante de Michaelis
M = Molaire
MD = Molecular Dynamics (fr. Simulation de Dynamique Moléculaire)
P450 = Cytochrome P450
PDB = Protein Data Bank
PSSM = Position Specific Scoring Matrix ou Profil
RMN = Résonance Magnétique Nucléaire
RMSD = Root Mean Square Deviation (fr. écart quadratique moyen) SCOP = Structural Classification Of Proteins
SRS = Substrate Recognition Site (fr. Site de reconnaissance du substrat) SSE = Structural Secondary Element (fr. élément de structure secondaire)
1
Introduction générale
Introduction générale
Introduction générale
Introduction générale
En l’espace d’un demi-siècle, le Cytochrome P450, est devenu un véritable emblème dans la communauté scientifique des biologistes tant l’engouement que cette protéine suscite est intense. Depuis sa découverte dans les années 50, elle ne cesse de poser de nouvelles questions aux chercheurs. En réalité, il n’existe pas un Cytochrome P450, mais toute une variété : ces cytochromes P450s correspondent en effet à une large superfamille d’hémoprotéines mono-oxygénases, présente à la fois chez les procaryotes et les eucaryotes. Ces enzymes jouent un rôle important dans le métabolisme oxydatif d’une grande diversité de substrats pour la plupart hydrophobe, aussi bien d’origine endogène qu’exogène. Ainsi, chez les cellules de mammifère, ils sont notamment responsables de la métabolisation et de la détoxication des composés exogènes, également appelés xénobiotiques. On les trouve aussi impliqués dans les phénomènes d’intoxication par formation d’entités réactives : époxydes, radicaux, etc. Ces enzymes de phase I du métabolisme, assurent une modification du potentiel redox de ces xénobiotiques par mono-oxygénation ou réduction, modifiant leur liposolubilité et rendant possible leur excrétion après conjugaison ou non par des enzymes de phase II. Les P450s, de 400 à 500 acides aminés environ, contiennent une proto-porphyrine IX de fer (l'hème) qui joue le rôle de catalyseur d'oxydo-réduction, et peuvent être solubles ou membranaires. Les réactions d'oxydo-réduction entraînent l’activation de l’oxygène qui est transférée sur le substrat positionné entre 3 et 6 Å du fer (Johnson, 2003).
En raison de leurs propriétés physico-chimiques et de leur implication dans les réactions de détoxication, les cytochromes P450 constituent donc un enjeu à la fois économique et pharmaceutique majeur : les industriels pharmaceutiques doivent en effet faire face à ces enzymes qui reconnaissent et dégradent la majeure partie de leurs médicaments actuellement mis sur le marché. De plus, cette dégradation peut entraîner des interactions médicamenteuses non désirées capable de provoquer des effets toxiques. Les laboratoires de recherche doivent pourtant composer avec ces protéines qui constituent une des premières barrières naturelles aux agressions extérieures. C’est pourquoi il est impératif d’étudier et de comprendre les mécanismes impliqués dans la reconnaissance du substrat par ces CYPs, pour pouvoir espérer soit contourner cette barrière par diminution les doses de médicaments prescrits, soit agir directement sur ces enzymes et les modifier à notre avantage, par des expériences de mutation.
La thématique des P450s depuis, tout particulièrement les P450s impliqués dans la cascade de détoxication chez l’homme est une des activités majeures de l’équipe dirigée par M. Delaforge et F. André au sein de laquelle j’ai effectué mon travail de thèse. Elle concerne aussi bien l’activité catalytique de ces enzymes (multi-spécificité et coopérativité) que leurs relations structure-activité
vis-à-vis de composés hydrophobes (médicaments, pesticides, mycotoxines, ….). Fort de leurs connaissances et de leurs savoir-faire expérimentaux, les membres de cette équipe ont décidé d’aborder la problématique de la compréhension des mécanismes de reconnaissance par des approches nouvelles : celles de la bioinformatique. En effet, la bioinformatique est devenue aujourd’hui un outil puissant et complémentaire des autres outils qu’on utilisait jusqu’alors en biologie pour étudier le fonctionnement des protéines (études spectroscopies, études enzymologiques d’activités et mutagénèse, etc.). Le développement d’algorithmes de plus en plus nombreux et la montée en flèche de la puissance de calcul des ordinateurs, ont fait de l’informatique un outil parfaitement adapté à la biologie dont on attend beaucoup, en particulier dans le domaine de l’ingénierie et la conception de médicaments. C’est dans ce contexte que j’ai pris mes fonctions dans cette équipe en Octobre 2004, pour explorer in silico les mécanismes généraux de reconnaissance par les CYPs, pour expliquer à la fois la sélectivité à l’intérieur d’une famille de substrats, la multi-spécificité et les effets coopératifs entre substrats de famille chimique différente (interaction médicamenteuse).
En bioinformatique structurale, le point de départ avant toute investigation est la disposition d’un support de travail qui est soit la structure de la protéine, soit un modèle. Même si aujourd’hui un nombre important de structure de CYPs est disponible, il demeure insignifiant comparé au nombre de gènes connus dans cette superfamille. À noter que la plupart des structures de P450s connus correspondent à celles qui ont un intérêt clinique ou pharmaceutique. Bref, pour étudier des P450s de structure inconnue, il est nécessaire de produire des modèles. Toutefois, la diversité en séquence (principalement en partie N-terminale, et à un degré moindre en C-terminale) qui existe au sein de cette superfamille est un vrai frein à l’obtention d’un modèle 3D par modélisation comparative.
Le premier point sur lequel j’ai travaillé durant mes trois années de thèse, a consisté à développer une méthodologie fiable de reconstruction de P450s à bas taux d’identité. Pour cela, j’ai conduit mes expériences sur un cas particulier des P450s : celui du CYP 3A4. En effet, chez l'homme, la famille de P450 la plus abondante est celle du CYP3A, qui comprend notamment l'isoforme P450 3A4 (CYP3A4, 503 acides aminés). Celui-ci est majoritaire dans le foie, responsable à lui seul du métabolisme de près de 60% des médicaments utilisés actuellement (Lewis, 2003), mais aussi de nombreux produits présents dans notre environnement : pesticides, mycotoxines, additifs alimentaires, etc. Des composés endogènes tels que la testostérone ou des petits dérivés peptidiques (Delaforge et al., 1997) sont aussi reconnus et oxydés par ce cytochrome. Les substrats pris en charge sont
généralement hydrophobes, mais de taille, de nature chimique, et de poids moléculaire très variables : de 150 à 1400 Da (Rendic, 2002, Ekins et al., 2003).
Un premier modèle moléculaire du CYP3A4 a été développé au laboratoire au cours du travail de thèse de N. Loiseau (Loiseau, 2002). Il a été obtenu par modélisation comparative basée sur les six structures de cytochromes P450 disponibles à l’époque dans la PDB (Protein Data Bank), dont cinq d'espèces bactériennes solubles, avec une moyenne de 20 % d'identité. Ces isozymes partagent une homologie fonctionnelle (activité mono-oxygénase), mais reconnaissent et métabolisent des substrats très différents, parfois spécifiques (camphre par exemple, pour le P450cam ou Camphor 5-monoxygenase, le plus étudié). Un autre modèle de la famille 3A, l’isoforme CYP3A7 qui présente 88% d’identité avec le CYP3A4, et est exprimée principalement dans le foie durant la période fœtale et néonatale (Lacroix et al., 1997, de Wildt et al ., 1999), a été construit en 2003 par M. Cottevieille avec un jeu réduit à quatre structures PDB de cytochrome P450, dont trois bactériennes. Le modèle de CYP3A7 devait permettre de comparer les mécanistiques 3A4/3A7 chez l’homme, et d’utiliser leurs propriétés opposées dans le métabolisme des stéroïdes, afin de réaliser des validations croisées des modèles. Toutefois, les modèles obtenus avec pourtant la même stratégie de modélisation, ne sont pas en accord. Ce désaccord provient de l’étape d’alignement des séquences de référence sur la séquence cible, étape cruciale en modélisation comparative. Dans les deux cas pourtant, l’information structurale spécifique des CYPs était utilisée pour améliorer l’alignement multiple. En effet, en dépit de la diversité en séquence, on observe une bonne conservation du repliement global dans la superfamille des P450, au travers des différentes structures déposées dans la PDB qui sont d’origines diverses (bactérienne, fongique ou de mammifère). Cette information structurale servait d’ancrage à l’alignement multiple.
J’ai donc été amené à étudier cette information de conservation structurale au sein de la superfamille des CYPs, puis vérifier et valider l’intérêt de son utilisation pour améliorer l’alignement multiple. Pour cela, il a fallu mettre en place et développer un outil fiable d’alignement multiple (Caliseq) permettant de positionner correctement les séquences des P450s de structures connus, sur la famille des 3A. Les alignements produits par cette méthode servent de points de départ aux reconstructions de modèles 3D de CYP 3A et par extension à d’autres P450s ne disposant pas d’autres homologues à plus de 30% d’identité (limite acceptable pour la reconstruction de modèles par des méthodes classiques).
Par ailleurs, le programme développé m’a également permis d’aborder la bioinformatique génomique : en effet, ce logiciel initialement mis au point pour produire les alignements multiples était également en mesure d’exploiter l’information structurale comme signature spécifique des P450s pour des expériences de génomique exploratoire. Caliseq permet en effet de détecter des séquences de P450s (dans la mesure où l’information de conservation structurale est tirée de la superfamille des P450s) dans des banques de séquences. Cet aspect de génomique exploratoire présente un intérêt dans la construction d’une banque de séquences spécifique au P450s. L’utilisation de cette banque, combinée à celle de la reconstruction de modèles pourrait théoriquement aboutir à une constitution d’une base de modèle de site actif, sur laquelle les chimiothèques pourraient être confrontées. Les résultats des criblages obtenus, apporteront probablement des éléments de réponses, ou du moins une idée sur les mécanismes de reconnaissance du substrat par ces enzymes.
Outre l’aspect prédictif de la bioinformatique, j’ai été également conduit au cours de ma thèse à travailler sur une application concrète de la bioinformatique structurale dans un traitement par thérapie génique, impliquant une autre P450, la CYP 2B6. Ce travail a été initié par l’équipe de Toxicologie Moléculaire à la Faculté de Médecine de Paris V qui a développé les techniques de thérapie génique autour d’une prodrogue, dont le produit de dégradation par le CYP présente un effet chimiothérapique. Pour améliorer l’affinité et l’activité enzymatique du CYP 2B6 pour ce substrat, des expériences de mutations dirigées ont été réalisées. En raison du nombre important de mutants opérés, cette équipe a sollicité notre aide pour lui fournir un support bioinformatique en vue de tester in silico les résultats. Ce fut donc pour moi l’occasion d’aborder un autre aspect de la bioinformatique apportant une véritable finalisation au travail in silico, à savoir utiliser les ressources et les informations disponibles pour fournir une base destinée et une description moléculaire à des expériences biochimiques.
Ce travail de collaboration m’a ainsi permis de confronter des résultats obtenus (mutations in silico, docking de la prodrogue dans le site actif, simulations de dynamique moléculaire…) sur des modèles de CYP 2B6 par des résultats expérimentaux et au plan plus général, d’apporter (ou de confirmer) des éléments de réponse aux mécanismes de reconnaissances de substrat par les P450s.
L’ensemble de tous mes travaux est présenté au travers de ce manuscrit, agencé de la manière suivante : dans un premier chapitre, j’exposerai l’état des connaissances actuelles disponibles, aussi bien sur les P450s que sur les méthodes bioinformatiques classiques d’étude de protéine. Je décrirai dans un second chapitre les méthodes que j’ai développées pour compléter les méthodes
bioinformatiques existantes, et en dernier chapitre, je présenterai les résultats obtenus par cette méthode et ceux issus de la collaboration avec l’équipe de Paris V.
9
Première Partie
Première Partie
Première Partie
Première Partie
État
État
État
11
CHAPITRE
1
1
Cytochromes P450 : pourquoi un tel intérêt ?
« Nous ne connaissons pas le vrai si nous ignorons les causes » Aristote (384-322 av JC)
1.1 A la découverte d’une superfamille d’enzyme…
1.1.1 Présentation d’une protéine hors norme
Le cytochrome P450 constitue un des sujets de recherche les plus en vogue au cours de ce dernier demi-siècle, aussi bien en biochimie qu’en biologie moléculaire. Il doit également son principal essor au développement de ressources de l’industrie pharmaceutique mondiale. Sous le terme général de cytochrome P450, se regroupe une superfamille multigénique de protéines enzymatiques dites «hemo-thiolate» à propriétés redox. Aussi connu sous le terme de mono-oxygénases, il a la capacité d’activer les molécules de dioxygène en des entités hautement réactives (ROS) et d’insérer ensuite l’oxygène moléculaire dans un nombre important et varié de substrats tant au niveau d’un atome de carbone, que d’azote ou de soufre. Cette activation du dioxygène est rendue possible par la présence d’un atome de fer inclus dans l’hème qu’il porte. Ces cytochromes P450 (CYPs ou P450s) sont des enzymes ubiquitaires qu’on retrouve dans tous les organismes vivants à l’exception près de certains micro-organismes primitifs qui auraient évolué il y a plus de 3,5 milliards d’années. De même, substrats et réactions générés par ces enzymes sont multiples et ubiquitaires : les CYPs sont responsables du métabolisme oxydatif de molécules très diverses, comprenant aussi bien des substances endogènes (hormones stéroïdiennes, acides gras, vitamines D, prostanoïdes, alkanoïdes, terpènes et autres pytoalexines…) que les xénobiotiques (médicaments, pesticides polluants, toxiques, cancérigènes, carcinogènes…). Les réactions de biotransformation des xénobiotiques catalysées par les CYPs s’inscrivent en effet dans un processus de détoxication évitant l’accumulation de substances potentiellement toxiques dans l’organisme. Ce sont d’ailleurs ces mêmes substrats qui auto induisent leurs propres réactions de détoxication. Paradoxalement, les CYPs peuvent parfois catalyser l’activation chimique de certains composés (procarcinogènes…) et produire des métabolites toxiques, mutagènes voir cancérogènes (génération de ROS qui endommagent l’ADN). Cette ambivalence et les conséquences majeures qui en découlent ont conduit à s’intéresser aux cytochromes P450, tant du point de vue structural que fonctionnel.
1.1.2 Identification et caractérisation des CYPs
Cela fait à présent cinquante ans que Garfinkel et Kligenberg (1958) ont reporté pour la première fois l’apparition d’un pigment jaune orangé dans des fractions microsomales hépatiques de rat et de cochon en présence de monoxyde de carbone. Ce pigment fut alors dans un premier temps caractérisé comme une hémoprotéine, identifiée à un cytochrome de type b peu commun, puis deviendra plus tard
ce qu’on connaît sous la désignation « P450 ». Cette désignation de « P450 » a été attribué en raison de sa propriété d’absorption maximal à 450 nm dans le spectre UV (cf. Figure 1-1) lorsque le fer de l’hème, à l’état réduit, est complexé au monoxyde de carbone (Omura et Sato, 1962). Le ‘P’ de P450 fait référence quant à lui au « pigment » d’où le terme P450 pour Pigment à 450 nm. La fonction biochimique des cytochromes P450 fut alors établie vers le début des années 60 aussi bien dans la biotransformation des stéroïdes (Estrabook et al., 1963) que dans l’oxydation des composés exogènes (Cooper et al., 1965).
Figure 1-1 Spectre d’absorption du P450 complexé à du monoxyde de carbone montrant le pic caractéristique aux alentours de 450 nm
C’est donc, d’une part l’observation d’une pigmentation cellulaire jaune-orangé apparue à la suite d’une complexion de préparation microsomale avec un monoxyde de carbone et d’autre part, la présence d’un pic distinct à 450 nm en spectre UV visible d’absorption (Garfinkel, 1958 ; Klingenberg, 1958) qui ont permis la découverte de l’enzyme. Les origines phylogéniques des P450s quant à elles, demeurent discutables et restent encore à démontrer. On pense toutefois que le système des P450s aurait évolué de façon constante il y a plus de 3,5 milliards d’années, avant même l’arrivée de l’oxygène atmosphérique (Wickramasinghe et Villee, 1975). Au départ, il semble que les organismes présents se servaient du potentiel chimique des molécules d’oxygène pour traiter les composés organiques, grâce à la formation contrôlée d’entités réactives (i.e. superoxydes, peroxydes, etc.). Ces mécanismes de transformation de l’oxygène seraient rendus possibles par la médiation d’un complexe fer-porphyrine dont la structure est similaire de la protoporphyrine IX (ou hème cf. Figure 1-1). La porphyrine est en effet une entité appropriée pour la régulation de l’équilibre de l’état de spin du fer (Frausto da Sliva et Williams, 1991), occupant donc un rôle important dans les échanges électroniques du fer (et donc par la même occasion, dans la catalyse par les hémoprotéines). Ce type de réaction peut être de plus favorisé par l’influence d’une cystéine proximale (thiolate) présente dans l’apoprotéine du P450 qui se charge, d’acheminer les molécules d’eau, les éléments réducteurs (i.e. proton/électrons)
nécessaires à la réaction (Lewis et Pratt, 1998) et les substrats organiques à lier. De toute évidence, l’apoprotéine P450 semblerait avoir évolué de façon à incorporer la partie héminique et à « orienter » les substrats potentiels vers un métabolisme oxydatif d’une position particulière. L’apport en électron peut être obtenu quant à lui par transfert à partir de partenaires redox pouvant appartenir à un système immergé dans une bicouche phospholipidique (comme celle de la membrane du réticulum endoplasmique dans le cas des P450s hépatiques microsomaux).
Figure 1-2 Protoporphyrine IX de fer constituant l'hème des cytochromes P450
Au final, tous les P450s identifiés à ce jour désignent des hémoprotéines constituées d’un hème relié à l’apoprotéine par un groupement cystéinate complexant le fer de groupe prosthétique (cf. Figure 1-3). Schématiquement, on a coutume de représenter les CYPs comme une grosse hémoprotéine constitué d’une chaîne polypeptidique unique, l’apoprotéine (d’un poids moléculaire compris entre 45 et 60 kDa) et d’un groupement prosthétique central, l’hème (catalyseur de la réaction enzymatique). Ce dernier est lié de façon « non covalente » à l’apoprotéine par l’intermédiaire d’une cystéine. Le site catalytique de l’enzyme est hydrophobe, permettant l’accueil de substrats.
- La diversité des P450s est naturellement liée à la séquence primaire en acides aminés de l’apoprotéine.
- L’hème des CYPs est constitué d’une protoporphyrine XI de Fer. Au sein des P450s, ce métal est lié aux quatre azotes pyrroliques de la porphyrine et au soufre de la cystéine axiale. Cette cinquième liaison de coordination du fer est réalisée avec le groupement thiolate de la cystéine présente dans la région C-terminale. Le fer peut être hexacoordonné, le sixième ligand étant par exemple, O2, H2O, CO ou une autre molécule. Dans le P450, le plan de l’hème définit
deux régions : (a) la face proximale située du côté du ligand cystéinate et (b) la face distale contenant le site actif de l’enzyme.
Figure 1-3 Représentation schématique d'un CYP
1.1.3 Diversité des cytochromes P450
Depuis sa découverte, l’intérêt que cette superfamille suscite n’a cessé d’augmenter de façon considérable : aujourd’hui encore, le CYP reste un challenge scientifique majeur. Les trois décennies qui séparent la première purification d’un P450 en 1970 (Yu et Gunsalus, 1970) de la première résolution d’une structure d’un P450 de mammifère en 2000 (Williams et al., 2000b), ont ainsi permis d’appréhender efficacement la complexité et la diversité de cette superfamille.
1.1.3.1 Multiplicité en gènes
On remarque tout d’abord chez cette superfamille une multiplicité en gènes : Lewis reportait déjà en 2000 plus de 1200 gènes individuels de P450. Selon lui, beaucoup d’autres nouveaux gènes viendront grossir les rangs dans les années à venir, tant l’état de connaissance dans ce domaine est vaste, profond ne demandant qu’à être exploré. Ses intuitions se sont révélées exactes, puisque aujourd’hui (2007), nous atteignons déjà 7700 gènes répartis entre les bactéries, végétaux, champignons, insectes et animaux. De plus, d’une espèce à l’autre, le nombre de gènes codant pour les P450s est très variable.
À partir d’informations basées sur des alignements de séquences et d’arbres phylogéniques d’évolution Nelson a émis l’hypothèse d’un gène ancestral des P450s qui serait apparu il y a environ 3.5 milliard d’années chez la bactérie (Nelson, 1999; Nelson et al., 1993, 1996). Ce dernier serait très proche du seul P450 présent dans tous les organismes : le CYP51. Ce P450 catalyse la
14-α-N N N N FeIII S Cystéine Apoprotéine Hème (protoporphyrine) Site catalytique (zone hydrophobe pour le substrat) Face proximale Face distale
déméthylation du lanostérol, un intermédiaire qui intervient dans les premières phases de la chaîne de synthèse des stérols. La diversification des CYPs pourrait donc être expliquée par une complexification de la biosynthèse des stérols au cours de l’évolution. Par ailleurs, l’apparition d’une « menace oxygène » dans le monde végétal ou animal expliquerait également la différentiation des CYPs, spécialisés dans la détoxication des xénobiotiques. Malgré l’importante diversité génique des cytochromes, cette superfamille est caractérisée par deux séquences protéiques consensus :
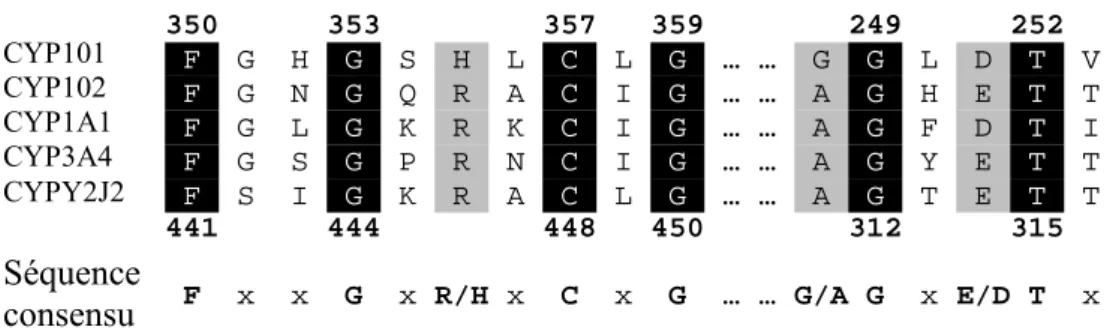
- Une séquence consensus située du côté proximal de l’hème FxxGx(R/H)CxG (il s’agit là du motif décrit de façon historique). Appelé « Cys-Pocket », elle contient la cystéine ligand du fer et est considérée comme la signature peptidique de la superfamille des P450s. D’ailleurs, ce motif est également désigné « signature cytochrome P450 – hème » et est utilisé pour identifier les P450s dans la base PROSITE (voir le paragraphe sur Prosite page 79) pour la recherche des P450s. Le motif de la base PROSITE s’écrit sous cette forme : [FW]-[SGNH]-x-[GD]-{F}-[RKHPT]-{P}-C-[LIVMFAP]-[GAD]. Cette séquence participe au maintien de l’hème au sein de l’apoprotéine.
Figure 1-4 Séquences consensus « Cys-Pocket » caractéristiques des cytochromes P450 (source : thèse de P. Lafite)
- Une séquence consensus distale (G/A)Gx(E/D)T moins bien conservée au sein des P450s, elle joue un rôle dans l’activation de l’oxygène moléculaire lors de la catalyse.
En raison du nombre important de P450s exprimés dans le monde vivant, une nomenclature en famille et sous famille basée sur l’identité en séquence a été élaborée. Cette nomenclature sera détaillée la section 1.2.1.
1.1.3.2 Localisation hétérogène
Les CYPs sont représentés dans tous les organismes vivants, à différents niveaux de localisation cellulaire : dans le cas des procaryotes, les CYPs sont des protéines cytosoliques, tandis que chez les eucaryotes, ils sont membranaires, localisés dans différents compartiments cellulaires. Ainsi, on
350 353 357 359 249 252 F G H G S H L C L G … … G G L D T V F G N G Q R A C I G … … A G H E T T F G L G K R K C I G … … A G F D T I F G S G P R N C I G … … A G Y E T T F S I G K R A C L G … … A G T E T T 441 444 448 450 312 315 Séquence consensu s F x x G x R/H x C x G … … G/A G x E/D T x CYP101 CYP102 CYP1A1 CYP3A4 CYPY2J2
distingue les P450s mitochondriaux, les P450s microsomaux, fixés aux membranes du réticulum endoplasmique (RE), et les cytochromes plastidiaux chez les végétaux.
1.1.3.3 Variabilité en forme
On pensait initialement que le P450 n’était qu’un simple et unique cytochrome. Très vite, on se rendit compte que cette enzyme pouvait exister sous de multiples formes, chacune possédant des propriétés différentes qui dépendent à la fois de leur sélectivité au substrat et de certaines caractéristiques physicochimiques (Omura et al., 1993). A titre d’exemple, des variations distinctes sont relevées autour du pic d’absorption des 450 nm pour des enzymes isolées de sources différentes, ou issues de préparations microsomales provenant d’animaux traités par des composés chimiques différents (Ruckpaul et Rein, 1984). Cette multiplicité en formes de cytochromes P450 n’est en fait qu’une conséquence d’une multiplicité en gènes (Gonzalez, 1988). Elle a ainsi conduit l’enzyme dans un premier temps, à l’appellation d’oxydase à « fonction mixte » puis, plus récemment, à celle de « mono-oxygénase » pour décrire sa capacité à insérer un atome d’oxygène dans une large variété de substrats et classes structurales de composés (Porter et Coon, 1991 ; Okita et Masters, 1992 ; Ortiz de Montellano, 1986, 1995). Encore largement utilisée, l’appellation de « cytochrome » n’est pourtant plus considérée comme la plus appropriée pour décrire ces hémoprotéines : non seulement les P450s diffèrent significativement des autres cytochromes en de nombreux points, ils sont également plus reconnus comme des enzymes dites « hemo-thiolates » que des protéines redox (cas des cytochromes a, b ou c par exemple) en dépit du fait qu’ils possèdent, un potentiel redox, bien plus bas que leurs homologues (de -420 mV).
1.1.3.4 Diversité des réactions catalysées
Une des particularités des CYPs est leur capacité à catalyser une multitude de réaction de monooxgénation, dont l’hydroxylation est la plus répandue. Ils réalisent également des réactions d’époxydation, de déshydratation, d’isomérisation ou de réductions (Mansuy et Battioni, 2000 ; Guengerich, 2001 ; Ortiz de Montellano, 2005) sur des motifs carbonés aliphatiques ou aromatiques en transformant un atome d’oxygène à partir de dioxygène et nécessitant quelques protons et un donneur d’électrons.
Figure 1-5 Réactions d’oxydation généralement catalysées par les P450s. D’après (Mansuy et Battioni, 2000)
1.1.3.5 Rôles multiples
Cette variété de réactions catalysées confère aux P450s un rôle important aussi bien dans la transformation de molécules endogènes qu’exogènes. Selon les organismes dans lesquels ils sont exprimés, les CYPs ne jouent pas le même rôle. Comme il a été déjà évoqué, les P450s sont les principales enzymes impliquées dans le métabolisme des xénobiotiques. Ils participent entre autre à l’élimination des composés exogènes comme les médicaments, les toxines ou les polluants en complétant l’action défensive du système immunitaire destiné à prendre en charge les macromolécules. Dans certains cas, pourtant, la dégradation de certains xénobiotiques peut former des métabolites réactifs électrophiles et devenir source de toxicité pour le P450 ou d’autres protéines cellulaires (cf. paragraphe 1.6.2). Par ailleurs, les CYPs occupent une place importante dans la biosynthèse et la biodégradation des composés endogènes. Chez les procaryotes, certains CYPs participent à la
biosynthèse de composés endogènes tels que les stérols ou des antibiotiques assurant la défense du microorganisme. Chez les eucaryotes, c’est également les P450s qui assurent la synthèse des stérols nécessaires aux membranes plasmiques comme le cholestérol ou l’ergostérol, ainsi que la biosynthèse des hormones stéroïdiennes, des eïcosanoïdes, des rétinoïdes et de certaines vitamines (Guenguerich, 2005).
1.1.3.6 Un large spectre de substrats
Les CYPs sont en mesure de catalyser un nombre très important et varié de substrats : les CYPs participant à des chaînes de biosynthèse endogènes sont généralement spécifiques à un seul substrat, tandis que ceux impliqués dans le métabolisme de xénobiotiques montrent une très faible spécificité de substrat. Ces derniers sont capables de métaboliser des substrats de polarité ou taille très variable. L’exemple qu’on a coutume de citer pour illustrer ce phénomène est celui du CYP3A4, cytochrome humain qui à lui seul est en mesure de métaboliser jusqu’à 50% des médicaments prescrits chez l’homme (Rendic, 2002). Il reconnaît des substrats allant du paracétamol (151 Da) pour le plus petit, à la cyclosporine (1,2 kDa) pour la plus volumineuse. À noter qu’un substrat peut être oxydé de plusieurs façons différentes au sein d’un même P450 et que la même activité d’oxydation d’un substrat peut être catalysée par plusieurs P450s.
1.1.3.7 Un corpus de connaissance croissant
Enfin, outre la diversité en gènes, en formes, en réaction et en rôle, le corpus de connaissance dans ce domaine est lui même profond et étendu : le nombre de publications sur les P450s (ou relatives aux P450s) ne cesse d’augmenter depuis cette dernière décennie, de façon régulière (voir exponentielle) et avoisinant la barre des 1000 publications scientifiques par an (Estabrook, 1996 ; Koymans et al., 1993). La Figure 1-6 montre justement les différents domaines de recherche et autres champs d’application des P450s.
Figure 1-6 Domaines de recherches du P450 et champs d'application (Adapté de Bernhardt, 1996 ; récupéré de Lewis, 2001)
En résumé, il est facile de comprendre toute cette agitation « cérébrale » autour des CYPs, tant leurs fonctions, leurs implications, leurs mécanismes sont importants et variés. Ils présentent non seulement une véritable multiplicité en gènes, une grande diversité en réactions catalytiques mais montrent également des rôles variés, pouvant aller du métabolisme de xénobiotiques à la biosynthèse et biodégradation des composés endogènes. Dans cette diversité pourtant, le mécanisme sous-jacent semble imposer une contrainte à cette superfamille : sa structure très conservée.
1.1.4 Conséquences structurales
Étonnamment et en dépit de leur diversité, les CYPs semblent partager en commun un repliement très conservé. En effet, compte tenu de l’évolution fonctionnelle des CYPs, la structure tertiaire des
Métabolisme de phytoalexins, alkanoïdes, terpènes et couleur des fleurs Biotransformation de médicaments/métabolisme Pharmacologie Toxicologie Sciences des plantes Cytochromes P450 Sciences de environnement Biosenseurs Biosynthèses ciblées Conversion des herbicides et insecticides Microbiologie Activation métabolique de procarcinogènes
Physiologie Détoxication de xénobiotiques
Dégradation métabolique de terpènes, alkanes et d’acides gras Stéréo- et Régio-sélectivité d’oxygénation Biochimie Biotechnologie Chimie Polymorphisme génétique Métabolisme endogènique et biosynthèse de stéroïdes, d’acides gras, prostanoïdes, eicosanoïdes vitamines D et acides biliaires Bioremédiation Biomonitoring
P450s telle que nous la connaissons1 au travers des différents cristaux publiés dans la Protein Data Bank (PDB) (http://www.rcsb.org) résulterait de contraintes apportées par divers éléments nécessaires à sa fonction catalytique (cf. Tableau 1.1) : liaison au substrat, hème, oxygène et partenaire(s) redox.
Tableau 1.1 Rôles structuraux de l'apoprotéine P450 (source Lewis, 2001)
Fonction du P450 Dispositifs structuraux impliqués
1. Liaison à l’hème Hélice I et L, cystéine invariante et usuellement deux résidus
basiques pour la liaison ionique avec les propionates de l’hème 2. Liaison et activation de l’oxygène La région de l’hélice I distale à la partie héminique
3. Liaison des partenaires redox Résidus basiques pour les liaisons ioniques
4. Transfert du proton à l’oxygène Liaisons ioniques internes établies par les résidus avoisinant l’hème 5. Liaison des différents substrats Les hélices B’, F et I et les brins β1 et β4 (Région SRS)
6. Régulation de ces activités à
l’intérieur de la membrane Peptide N-terminal de 30-40 résidus de long
SRS = Substrate recognition site (Gotoh, 1992)
Ces contraintes liées à la fonction sembleraient exercer une certaine pression conduisant à la conservation structurale des P450s : en effet, d’un P450 à un autre, la structure des P450s est inchangée, et ce en dépit de leurs localisations environnementales, que ce soit chez la bactérie (où le P450s est cytoplasmique), les mitochondries ou encore dans les systèmes microsomaux (où les P450s sont ancrés à la membrane) (Degtyarenko et Archakov, 1993). Il existe bien entendu quelques exceptions connues à cette conservation structurale. C’est le cas des P450s dont la fonction est quelque peu inhabituelle, comme par exemple l’allène oxyde synhtase (AOS), la prostacyclin synthase (PGIS) ou encore la thromboxane synthase (TXAS). En fait, les différentes fonctionnalités montrées par ces enzymes peuvent être associées à de petits changements dans la séquence (par exemple sur l’hélice I), mais suffisamment cruciaux (cf. Tableau 1.2) à l’intérieur du centre catalytique du P450 pour entraîner une légère modification de la structure de la protéine.
1
De 16 à 18 hélices α numérotées de A à L avec quelques variations et 4 à 6 feuillets β numérotés de 1 à 6. La structure des CYPs sera vue plus loin.
Tableau 1.2 Comparaison de l'hélice I entre plusieurs P450s.
Espèces CYP Abréviation Séquence Type de réaction
enzymatique
↓↓↓↓ ↓↓↓↓ ↓↓↓↓ ↓↓↓↓
Humain 3A4 Nif F I F A G Y E T T S Nifedipine N-Oxidase
Humain 5A1 TXS F L I A G Y E I I T Thromboxane synthase
Rat 5A1 TXS F L I A G H E I T T Thromboxane synthase
Souris 5A1 TXS F L I A G H E V I T Thromboxane synthase
Cochon 5A1 TXS F L I A G Y E I I T Thromboxane synthase
Lin 74 AOS N S W G G F K I L L Allène oxyde synthase
Guayule 74 AOS N T F G G V K I L F Allène oxyde synthase
Humain 19 Arom M L I A A P D T M S Estrogène synthase
Vache 19 Arom M L I A A P D T M S Estrogène synthase
Rat 19 Arom M L I A A P D T M S Estrogène synthase
Souris 19 Arom M L I A A P D T M S Estrogène synthase
Poulet 19 Arom M L I A A P D T L S Estrogène synthase
Truite 19 Arom M V I A A P D T L S Estrogène synthase
Levure 51 14DM V L M G G Q Q T S A Lanosterol 14α-démétylase
Rat 51 14DM L L L A G Q Q T S S Lanosterol 14α-démétylase
Humain 51 14DM L L L A G Q Q T S S Lanosterol 14α-démétylase
Candida 51 14DM V L M G G Q Q T S A Lanosterol 14α-démétylase
Note :
1. La séquence habituelle de l’hélice distale est montrée par la séquence AGxET du CYP3A4 (ou x est n’importe quel résidu
2. CYP5A1 ne possède pas la thréonine distale conservée
3. CYP74 ne possède ni la thréonine distale conservée ni le résidu acide précédent 4. CYP19 possède une proline avant l’aspartate
5. CYP51 ne possède pas de résidu acide avant la thréonine distale 6. ↓. Pointe les résidus normalement conservés
Références : Nelson et al., 1996 ; Mansuy et Renaud, 1995, Tableau tiré de Lewis, 2001
En conclusion, ce qui a été un jour vu en tant qu’un simple enzyme est désormais connu pour sa multiplicité de formes, disposant de fonctionnalités variées et d’un vaste nombre de substrats potentiels (Porter et Coon, 1991 ; Guengerich, 1991a et b ; Stegeman et Livingstone, 1998 ; Mansuy, 1998; Nelson, 1999).
1.2 Nomenclature, Classification et Phylogénie
Vu l’importance en nombre et l’extrême diversité des P450s exprimés dans le monde vivant, sans méthodologie stricte, on risquerait de se perdre dans un brouillard d’informations. Ainsi, il a été convenu de répertorier chaque P450 selon une nomenclature particulière. Cette dernière a été arbitrairement choisie, en fonction du degré d’identité entre chaque P450. Un autre regroupement a pu également être décrit, reposant cette fois-ci sur la fonctionnalité des P450s, et plus précisément, la manière dont ces derniers reçoivent les électrons nécessaires à leurs activités. On parlera alors de classification. Bien évidemment, ces deux éléments permettent d’apporter suffisamment d’informations pertinentes à la réalisation d’un modèle d’évolution, qui peut être décrit par la phylogénie.
1.2.1 Nomenclature
Depuis 1989, une appellation systématique des P450s a été mise en place, périodiquement mise à jour depuis (Nebert et al., 1989a et b, 1991a ; Nelson et al., 1993, 1996). Cette nomenclature utilise le symbole CYP (ou Cyp chez la souris et la drosophile) comme abréviation pour le terme cytochrome P450 (aussi bien ADNc, ARNm que protéine), symbole qu’on retrouve en italique (CYP) lorsqu’il fait référence au gène. La lettre P qui est parfois trouvé après un gène informe quant à lui qu’on a affaire à un pseudogène. Il a donc été convenu que les CYPs soient classés en fonction de leur degré de similitude dans leur séquence primaire2 (séquence en acides aminés) (voir aussi le site internet de David Nelson à l’adresse http://drnelson.utmem.edu/CytochromeP450.html) comme suit :
- Appartiennent à la même famille (désigné par un chiffre arabe, CYP1, CYP2, …), les CYPs ayant un degré de similitude supérieur à 40% ;
- A la même sous-famille (représentée par une lettre majuscule, CYP2B, CYP2C, …), les CYPs ayant un degré de similitude supérieur à 55%
- Enfin, les isoformes appartenant à une même sous famille sont différenciés par un chiffre arabe (CYP2C8, CYP2C9,…).
2
Il existe des cas particuliers pour les CYP responsable du métabolisme des stéroïdes où l’ancienne nomenclature subsiste : le nombre juste après CYP correspond alors au numéro de l’atome du substrat où s’opère la réaction de monoxygénation. C’est le cas pour le CYP 19 par exemple.
Alors que ce système fut initialement mis en place de façon arbitraire, ce choix de classification par degré de similitude s’est révélé être assez judicieux et approprié, compte tenu qu’il permet de séparer les différentes familles et sous-familles de façon satisfaisante dans la majorité des cas. Bien évidemment, certains regroupements demeurent inexacts.
En se basant sur cette similitude entre les séquences et dans certains cas en tenant compte des activités catalytiques, il a donc été possible de déterminer si deux gènes étaient issus ou non d’une duplication de gènes phylogénétiques. En raison du nombre de familles devenu très important, un niveau supplémentaire de regroupement est requis. Les clans font alors leur apparition et rassemblent les familles qui appartiennent à un même groupe (issu d’un même gène ancestral) d’après de nombreux arbres phylogénétiques établis auparavant (Nelson, 1999). Ces clans sont désignés par le chiffre le plus petit des familles qu’ils regroupent ou par le chiffre de la famille majoritaire. Ainsi, le clan 2 regroupe la famille de CYP2 de même que les familles CYP1, 17, 18 et 21.
Outre cette classification standard, les divers CYP se distinguent aussi par deux autres critères : leur spécificité de substrat et la classe de molécules entraînant l’augmentation de l’expression. Néanmoins, bien que les CYPs puissent se distinguer par leur spécificité au substrat, cette dernière caractéristique peut être chevauchante comme il a été déjà mentionné précédemment : un CYP peut ainsi métaboliser plusieurs substrats et un substrat peut être métabolisé par plusieurs CYPs. On peut donner à titre d’exemple le cas de la morphine qui peut être soit métabolisée par les CYP3A4 et 2C8 (Projean et al., 2003). Un aperçu des CYPs et de leurs substrats est disponible sur le site Internet
http://www.edhayes.com/startp450.html.
1.2.2 Classification
Les CYPs catalysant principalement des réactions de mono-oxygénation, un apport d’électron au substrat est donc nécessaire. Ces électrons sont fournis par le NADPH ou le NADH. Ainsi, les CYPs ont été divisés en plusieurs grandes classes selon différents critères, tels que la façon dont les électrons sont transportés du NAD(P)H au site catalytique. A ce jour, deux nomenclatures sont utilisées pour différencier ces CYPs : (a) une ancienne nomenclature comprenant seulement 4 classes, adoptée de tous, et (b) une plus récente décrit par Hannemann en 2006 qui distingue 10 classes. L’intérêt de cette dernière nomenclature est discutable, mais semble être plus rigoureuse pour départager certains regroupements qui hébergent des cas particuliers. Néanmoins, dans ce manuscrit, c’est la classification en 4 classes qui sera utilisée.
1.2.2.1 Classification à 4 classes
Figure 1-7 Partenaires de transfert d'électrons des CYPs (d’après Paine et al., 2005). La classe I comprend les P450s mitochondriaux (A), la majorité des P450s solubles (B) et certains systèmes fusionnés, dont le CYP RhF est un représentant (C). La classe II comprend les P450s du RE (D) et les P450s qui incluent la ferredoxine à deux flavines comme P450BM3 (E) Fx : ferredoxine, FxR : ferredoxine réductase, CPR : CYP réductase
Dans la classification originale, on distingue deux classes majoritaires de transfert d’électrons (Paine et al., 2005) (cf. Figure 1-7). Deux autres classes sont également rencontrées, mais beaucoup plus rarement.
- La classe I fait intervenir dans la chaîne de transfert d’électrons des enzymes à centre fer-soufre. Les électrons du NAD(P)H sont prélevés par une ferrodoxine réductase à flavine (FAD) puis transmis à une ferrodoxine à centre fer-soufre Fe2S2. Cette ferrodoxine transfère ensuite
les électrons au P450 pour permettre la catalyse. Cette classe contient la très grande majorité des P450s bactériens et les P450s mitochondriaux. Il conviendrait d’ajouter à cette classe des nouveaux systèmes identifiés récemment comme le P450RhF (Roberts et al., 2002), qui est une protéine de fusion contenant une réductase à flavine et FMN, une protéine type ferredoxine à centre fer-soufre et le cytochrome P450.
- La classe II des P450s est la plus commune chez les eucaryotes, à l’origine de réactions catalytiques extrêmement diverses et variés. Les CYPs de cette classe sont trouvés au niveau du RE et requièrent une NADPH-Cytochrome P450 réductase (CPR) qui contient les groupements prosthétiques FAD et FMN. Cette dernière est issue d’une fusion de deux protéines ancestrales : elle montre une partie N-terminale homologue avec une flavodoxine bactérienne à FMN et une partie C-terminale homologue avec une ferrodoxine NADP+
réductase à FAD et avec une NADPH-cytochrome b5 réductase. Dans le système microsomal de P450s, en plus de la CPR précédemment décrite, on retrouve aussi un autre système qui n’est pas spécifique des P450s, faisant intervenir un cytochrome b5. Ce dernier est en mesure de transférer lui aussi des électrons au P450, après avoir reçu ces électrons soit par la CPR soit par la NADH-cytochrome b5 réductase. Dans la nomenclature à 4 classes, le P450BM3 issu de
Bascillus megaterium et la NO-synthase apparaissent toutes deux dans cette classe, ce qui n’est pas le cas dans la nomenclature à 10 classes.
- Le P450nor catalyse la réduction du monoxyde d’azote NO et reçoit ses électrons directement
du NADH, sans protéine intermédiaire (Takaya et al., 1999). Ce P450 est le seul représentant de la classe IV.
- D’autres P450 catalysent des isomérisations ou des déshydratations, et ne nécessitent pas d’apport d’électrons extérieurs ou d’oxygène moléculaire. Les substrats transformés sont généralement riches en électrons, comme des hydroperoxydes ou des endoperoxydes. Bien qu’aucun système de transfert d’électrons ne soit présent, la classe III est tout de même défini et comprend ces systèmes. La récente P450PCIS humaine (qui est une isomérase) fait partie de
cette classe (Strushkevich et al., en attente de publication).
1.2.2.2 Classification à 10 classes
La classification précédemment décrite est la plus connue, et admise de toute la communauté scientifique. Néanmoins, il existe des cas pour lesquels le regroupement tel qu’il a été décrit ne semble pas des plus appropriés, notamment pour des CYPs bactériens. Ceux-ci ont amené Hannemann à proposer une autre classification (Hannemann et al., 2006), toujours basée sur le transfert d’électrons, mais plus adaptée pour ces cas particuliers. Les principaux changements observés, outre le nombre de classes, est principalement la sortie de certains de ces P450s de procaryotes d’une certaine classe (des classes I et II) pour former une nouvelle classe à eux seuls.
Le Tableau 1.3 et la Figure 1-8 qui suivent, résument et illustrent les principales caractéristiques permettant de regrouper les P450s selon cette nouvelle classification.
Tableau 1.3 Classes des systèmes P450s, classées selon la topologie des partenaires redox impliqués dans le transfert d’électrons au CYP (d’après Hannermann, 2006).
Classe / Source Chaîne de transport d’électron Localisation / Remarques
Classe I Bactérienne Mitochondriale
NAD(P)H > [FdR] > [Fdx]a> [P450]
NADPH > [FdR] > [Fdx] > [P450] Cytosolique, soluble P450 : membrane interne des mitochondries
FdR : membrane associée
Fdx : matrice mitochondriale, soluble Classe II Bactérienne Microsomale A Microsomale B Microsomale C NADH > [CPR] > [P450] NADPH > [CPR] > [P450] NADH > [CPR] > [cytb5] > [P450] NADH > [cytb5Red] > [cytb5] > [P450]
Cytosolique, soluble ; Streptomyces carbophilus
Ancrée à la membrane, ER Ancrée à la membrane, ER Ancrée à la membrane, ER Classe III
Bactérienne NAD(P)H > [FdR] > [Fldx] > [P450] Cytosolique, soluble ; Citrobacter braakii
Similaire à la classe II, mais sans l’entité CPR
Classe IV
Bactérienne Pyruvate, CoA > [OFOR] > [Fdx] > [P450] Cytosolique, soluble ; Sulfolobus tokadaii
Classe des P450s thermophiles (ex CYP 119) Classe V
Bactérienne NADPH > [FdR] > [Fdx–P450] Cytosolique, soluble ; Methylococcus
capsulatarus Classe VI
Bactérienne NAD(P)H > [FdR] > [Fldx–P450] Cytosolique, soluble ; Rhodococcus
rhodochrous strain 11Y Classe VII
Bactérienne NADH > [PFOR–P450] Cytosolique, soluble ; Rhodococcus sp. strain
NCIMB 9784, Burkholderia sp., Ralstonia metallidurans
Cas du P450RhF Classe VIII
Bactérienne, fungique NADPH > [CPR–P450] Cytosolique, soluble ; Bacillus megaterium,
Fusarium oxysporum Cas du P450BM3 Classe IX
Seule NADH
dépendant, fungique
NADH > [P450] Cytosolique, soluble ; Fusarium oxysporum
Seul représentant : P450nor Classe X
Indépendant chez la plante/mammifère
[P450] Liée à la membrane, ER
Anciennement la classe III
Abréviation pour les partenaires contenant les centres redox suivants : Fdx (complexe fer-sulfure) ; FdR, Ferrodoxine réductase (FAD); CPR, cytochrome P450 réductase (FAD, FMN) ; Fldx, Flavodoxine (FMN) ; OFOR, 2-oxyacid:ferrodoxine oxydoréductase (complexe thiamine phosphate, [4Fe-4S]) ; PFOR, phthalate-family oxygénase réductase (FMN, complexe [2Fe-2S]).
Figure 1-8 Organisation schématique des différents systèmes de P450s. (A) Classe I, système bactérien ; (B) Classe I, système mitochondrial ; (C) Classe II, système microsomal ; (D) Classe III, système bactérien, ex. du P450cin ; (E) Classe IV, système bactérien thermophile ; (F) Classe V, système bactérien à [Fdx]–[P450] fusionné ; (G) Classe VI, système bactérien à [Fldx]–[P450] fusionné ; (H) Classe VII, système bactérien à [PFOR]–[P450] fusionné ; (I) Classe VIII, système bactérien à [CPR]–[P450] fusionné ; (J) Classe IX, système eucaryote P450nor ; (K) Classe X, système eucaryote indépendant, cas du P450TxA (source Hannemann et al., 2006)
1.2.3 Phylogénie
Pour remonter aux origines des P450s, les chercheurs ont essayé de retracer leur évolution au moyen de techniques phylogéniques. Cependant, la construction d’arbres phylogénétiques n’est pas aisée pour cet enzyme car elle se base sur des matrices de distances calculées à partir d’un alignement multiple en séquences primaires (MSA). Cet alignement dépend fortement du degré de similarité entre séquences utilisées. En raison des divergences en séquence primaire, il est facile de comprendre qu’un alignement multiple qui prendrait en compte tous les P450s serait difficilement réalisable. La classification des P450s en famille et sous famille, basée sur la similitude en séquence, a rendu plus abordable dans un premier temps, la construction d’arbres phylogénétiques ainsi que d’autres formes d’analyses qui étudient l’évolution des relations entre gènes de P450s.
Un nombre très varié de méthodes a été utilisé pour comparer les séquences de P450s conduisant la plupart du temps à des arbres assez proches pour un gène ancestral commun à 2 milliards d’années. La méthode largement utilisée pour investir l’évolution et les relations entre P450s est celle de l’UPGMA (Unweighted Pair Group Method of Analysis) (Nelson et Strobel, 1987 ; Gotoh et Fujii-Kuriyama, 1989 ; Nebert et al., 1991 ; Nebert et Gonzalez, 1987 ). Nebert et Nelson ont également exploré les variations au sein des arbres phylogénétiques produits par le Neighbor Joining (NJ) et l’UPGMA pour 39 séquences, où ils trouvèrent une légère différence entre les deux méthodes principalement en raison des familles de CYP1 et CYP2. Toutes les méthodes d’analyse de séquences ont montré que la forme bactérienne CYP102 (P450BM3) du Bascillus megaterium se regroupe avec les
P450s d’eucaryotes, tandis que les autres formes de procaryotes ségrégent clairement dans des classes différentes. Une des raisons évoquées serait liée à la similitude des partenaires redox partagés par CYP102 et les P450s microsomaux d’eucaryotes comme il a été vu précédemment lors des classifications à 4 formes.
Au final, un arbre simplifié (cf. Figure 1-9) a été proposé où partant d’un gène ancestral commun (il y a 2 milliards d’années) qui se serait dupliqué. Les gènes ainsi créés auraient alors divergé, faisant apparaître progressivement une multitude de familles, sous-familles et d’isoformes de CYP. Au cours de l’évolution, ces gènes qui se sont considérablement diversifiés, ont vraisemblablement participé à l’adaptation des organismes aux changements de biotope. Cette diversification est corrélée au passage d’une atmosphère originellement réductrice à une atmosphère plus oxydante sous l’effet de la production de dioxygène par les organismes photosynthétiques. La pression actuelle en oxygène a
ainsi crû rapidement de façon exponentielle avec le temps. Ceci serait une cause probable de la non linéarité de l’évolution des CYPs.
Figure 1-9 Arbre phylogénique simplifié des cytochromes P450 (d'après Lewis, 2001). Les CYPs (CYP1 à 4) qui interviennent dans le métabolisme des xénobiotiques sont annotés en rouge (où EMX signifie Enzyme du métabolisme des Xénobiotiques). Il est admis que ces familles CYPs ont évolué puis divergé vers le monde animal pour permettre une adaptation et/ou une protection vis-à-vis de composés nouveaux et/ou toxiques synthétisés par les végétaux. Les CYP11A, 11B, 17,19 et 21 interviennent dans la synthèse d’hormones stéroïdiennes en catalysant des réactions d’hydroxylations.
1.3 Occurrence, Distribution et Localisation
1.3.1 Occurrence et Distribution
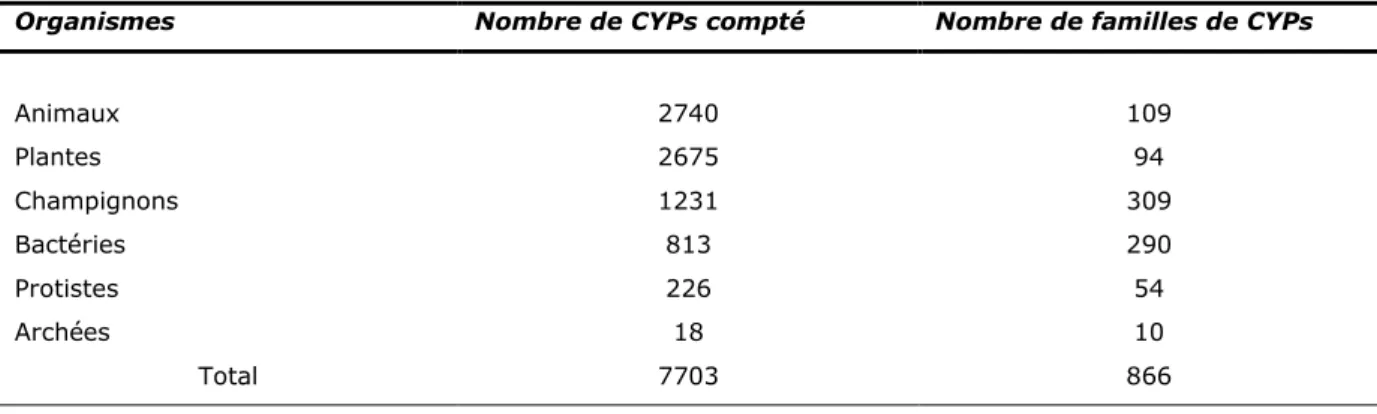
Comme mentionné précédemment, les CYPs sont des enzymes ubiquitaires, largement représentées dans tous les organismes vivants, de la bactérie aux mammifères, en passant par la plante et les champignons (Nerbert et al., 1989a et b), bien qu’il semble que certaines espèces primitives de bactéries en soient dépourvues. La répartition de ces CYPs au sein des différents organismes est consultable sur le site (http://drnelson.utmem.edu/CytochromeP450.html) de David Nelson, site complet qui est tenu régulièrement à jour par l’auteur de façon bénévole depuis plus de 10 ans. On y retrouve donc non seulement la répartition entre organismes et les statistiques sur les P450s, mais également tout un ensemble d’informations relatives aux P450s. Il permet ainsi à n’importe quel utilisateur d’avoir accès aux connaissances actuellement disponibles sur les P450s telles que les bases de données ou encore les séquences de P450s. Concernant la répartition, à la date du 7 septembre 2007, Nelson a répertorié pas moins de 7703 séquences de CYPs provenant de 866 familles différentes qui se répartissent entre les animaux, les plantes, les champignons, les bactéries, les protistes et les archées. Ces données sont issues d’un comptage et d’une vérification manuelle par l’auteur du site (cf. Tableau 1.4 et Figure 1-10). Elles mettent d’ailleurs en évidence une proportion très élevée de CYPs chez la plante, phénomène qui s’explique en partie par le fait que de nombreux génomes ont été séquencés chez la plante.
Tableau 1.4 Répartitions des CYPs dans les différents organismes vivants (source de Nelson)
Organismes Nombre de CYPs compté Nombre de familles de CYPs
Animaux 2740 109 Plantes 2675 94 Champignons 1231 309 Bactéries 813 290 Protistes 226 54 Archées 18 10 Total 7703 866
Figure 1-10 Répartition des séquences (a) et des familles (b) de CYPs dans les différents organismes vivants. On remarque que les CYPs sont largement représentés dans chez la plante et l’animal. Cela est lié à l’effort des investigations dans chaque organisme.
Même si les P450s ont été caractérisés dans de nombreuses espèces telles que les oiseaux, les poissons, les reptiles, les insectes, les mollusques, les arthropodes, les crustacés, les champignons, les plantes et les bactéries (Schenkman et Griem, 1993 ; Pinot et al., 1999 ; Gorman et al., 1998 ; Chapple, 1998 ; Scott et al., 1998 ; Walker, 1998 ; Hallahan et al., 1993 ; Hallahan et West, 1995 ; Nelson, 1998) (Une revue spéciale de « Comparative Biochemestry and Physiology » (Volume 121C, Nos. 1–3, 1998) traite justement de ces P450s non mammifères), c’est pourtant chez les mammifères que le P450 suscitent le plus d’intérêt, en particulier les formes humaines de l’enzyme (Guenguerich, 1989a et b, 1992a–d, 1994, 1995a ; Guenguerich et al., 1998, 1992 ; Rendic et DiCarlo, 1997). En conséquence, les P450s humains ont été intensément investis et étudiés, et les systèmes d’expression hétérologues sont devenus à présent un moyen fiable d’analyse pour les interactions enzyme–substrat de P450s isolés (Estabrook et al. 1991 ; Gonzalez et Korzerkwa, 1995).
1.3.2 Localisation
Chez les mammifères, les P450s sont présents dans la plupart des tissus, mais ils sont particulièrement abondants en quantité et en diversité au niveau du foie (cf. Figure 1-11). Pour l’être humain, le foie est un organe très important tant au niveau de sa corpulence physique que son rôle fonctionnel. C’est le premier organe, après l’intestin grêle, à être en contact avec les xénobiotiques via le système porte hépatique. Il représente en effet à lui seul 1/50e du poids total du corps humain. Il se compose pour 70% de cellules parenchymateuse ou hépatocytes et pour 30% de cellules diverses dont les cellules de Ito, les cellules composant les canicules biliaires et les cellules de Küpffer. Ses fonctions sont nombreuses et variées : fondamentalement, le foie est impliqué dans la sécrétion et l’évacuation de la bile par les canalicules biliaires, mais du fait qu’il soit extrêmement vascularisé et qu’il soit situé à l’interface de l’appareil digestif et l’appareil circulatoire, il joue également un rôle
dans la distribution de nombreux métabolites issus de la nutrition. En outre, le foie est capable d’assurer un grand nombre de fonctions métaboliques comme la synthèse des protéines plasmatiques, des lipides (dont le cholestérol), de l’urée et le stockage et la libération du glucose dans le sang, la production de substrats à haut potentiel énergétique, mais aussi le stockage de nombreuses vitamines. Enfin, il joue un rôle central au cours du processus de détoxication.
Figure 1-11 Répartition des CYPs dans le foie Humain (d'après Guenguerich, 2005)
Ces deux dernières (fonctions de synthèse et de détoxication par le foie) ne sont en fait que le reflet macroscopique des acteurs principaux de l’échelle microscopique : les CYPs. Les CYPs sont donc en nombre abondant dans le foie, et plus précisément localisés dans les membranes du réticulum endoplasmique (RE) des hépatocytes (Ruckpaul et Rein, 1984 ; Stier, 1976). Le RE est en effet une structure lipidique particulière, très favorable aux échanges, et disposant d’une surface étonnamment large (comprenant 7 à 11 m²/g du poids du foie) comparé à son volume, ce qui fait de lui la structure idéale pour héberger les enzymes de Phase I comme les P450s (Lewis et Pratt, 1998 ; Gibson et Skett, 1994). Sachant que 12 à 15% du RE est composé de cytochromes P450, on peut estimer approximativement que les P450s comptent pour un peu moins d’1% du poids total d’un hépatocyte (Ruckpaul et Rein, 1984).
Figure 1-12 Localisation dans le réticulum endoplasmique lisse (source Nebert, 1987). Six à huit P450s se regroupe autour d’une réductase qui leur apporte les électrons nécessaires au métabolisme des substrats.
Au sein de la membrane du RE, des études stœchiométrique (Finch et Stier, 1991 ; Schwarz, 1991) ont suggéré que les P450s se regrouperaient par 6 ou par huit autour d’une réductase, un partenaire fournisseur en électrons. Ces unités hexamèriques (ou octamèriques) subissent alors une diffusion transversale à l’intérieur de cette membrane phospholipidique du RE, elle-même sujette à une diffusion latérale au moyen du « flip-flop ». Par conséquent, les substrats relativement lipophiles se retrouvent alors facilement acheminés vers le centre réactionnel des P450s afin d’y être métabolisés.
Bien que majoritairement concentrés au niveau du foie, les P450s sont également exprimés dans d’autres organes et tissus (cf. Tableau 1.5) tels que le rein, les seins, la prostate (Williams et al., 2000a) la peau, l’épithélium nasal, le placenta, le cerveau, le poumon, la rate, le pancréas et la région gastro-intestinale (Waterman, 1992 ; Schenkmann et Griem, 1993 ; Hellmold et al., 1998 ; Hakkola et al., 1998, 1996). Toutefois, même si les P450s sont identifiés dans ces autres organes, ils demeurent en concentration faible et leur participation dans les biotransformations des xénobiotiques n’est pas bien définie. Certains CYP (CYP11A et CYP11B) sont également présents du côté du feuillet externe de la membrane nucléaire et dans la membrane interne des mitochondries (Montoliu et al., 1995). Ces isoformes sont relativement peu impliquées dans la biotransformation des xénobiotiques mais elles jouent un rôle important dans le métabolisme des composés endogènes comme les stéroïdes, le cholestérol, la vitamine D3, et les acides biliaires.